一种大豆基因拷贝数变异分析方法与流程
本发明涉及遗传育种技术领域,具体涉及基因拷贝数变异研究方法。
背景技术:
拷贝数变异(copy number variants,CNVs)是生物基因组中一种重要的遗传变异形式。研究发现CNVs与作物的许多性状相关,Cook等(2012)研究发现大豆胞囊线虫抗病位点Rhg1作用机制为31kbp基因组片段的拷贝数变异。因此研究作物种质资源个体表型差异、群体拷贝数表型变异规律以及基因组进化具有重要意义。目前拷贝数变异的检测技术包括微阵列比较基因杂交(array-based comparative genomic hybridization,aCGH)、单核苷酸多态性(single nucleotide polymorphisms,SNPs)、芯片代表性寡核苷酸微阵列分析(respectively oligonucleotide microarray analysis,ROMA)、多重连接探针扩增(multiplex ligation.dependent probe amplification;MLPA)、多重可扩增探针杂交(multiplex amplifiable probe hybridization,MAPH)、新一代测序技术以及荧光原位杂交技术(fluorescence in situ hybridization,FISH)检测等,虽然CNV检测技术较多,其主要用于人类疾病的检测,针对于作物的研究,受到每种方法的技术手段、成本及操作难度的限制难于在常规分子实验室中推广应用,尤其当被测样本容量大的情况下,如进行作物重组群体的表型检测难度很大。
目前拷贝数变异的检测技术包括微阵列比较基因杂交(array-based comparative genomic hybridization,aCGH)、单核苷酸多态性(single nucleotide polymorphisms,SNPs)、芯片代表性寡核苷酸微阵列分析(respectively oligonucleotide microarray analysis,ROMA)、多重连接探针扩增(multiplex ligation.dependent probe amplification;MLPA)、多重可扩增探针杂交(multiplex amplifiable probe hybridization,MAPH)、新一代测序技术以及荧光原位杂交技术(fluorescence in situ hybridization,FISH)检测,以上检测方法存在明显的不足之处,难于在常规分子实验室中广泛应用,其中aCGH、ROMA、MLPA和MAPH等方法对芯片及杂交探针的需求及探针回收率低、实验成本高等问题;新一代测序技术对CNV的检测因高测序深度而导致高成本及对大样本群体CNV分析的限制;FISH技术检测价格昂贵,且对研究人员操作手法和分析经验要求极高,无法进行大规模重复性分析且周期长、不适合做大样本分析。
技术实现要素:
为解决上述问题,本发明提供一种大豆基因拷贝数变异分析方法,具体方案包括如下步骤:
1)基因组DNA的提取和浓度均一化:采取SDS法提取大豆基因组DNA,将基因组DNA浓度调整为50ng/L,-20℃保存备用。
2)被测基因引物设计:设计被测基因引物,设计模板为被测基因CDS序列;3)RT-qPCR:以β-actin为内参基因,用内参基因引物与步骤2)涉及的被测基因引物分别对基因组DNA模板进行荧光定量PCR反应;荧光定量PCR检测方法反应体系为20μL,包括:2×荧光定量预混试剂(SuperRealPreMix Plus(2×))10.0μl,浓度为10μmol·L-1的PCR上下游引物各0.6μL,基因组DNA模板2μl,灭菌蒸馏水6.8μl;PCR程序包括:预变性95℃、15min;变性95℃、10s,退火55-60℃、20s,延伸72℃、20s,40个循环;
内参引物:
上游:5’GATCTACCATGTTCCCAAGT 3’,
下游:5’ATAGAGCCACCAATCCAGAC 3’;
4)利用RT-qPCR获得被测基因和内参基因β-actin的Ct值计算被测基因在不同大豆品种中的相对丰度值,通过被测基因在不同品种大豆中的相对丰度值之间的比值判断被测基因在不同品种大豆中的拷贝数变异关系。
上述方法具体包括以下步骤:
1)基因组DNA的提取和浓度均一化:采取SDS法提取品种A大豆基因组DNA,采用1%琼脂糖凝胶电泳检测和 Lite紫外分光光度计联合检测DNA浓度并通过稀释实现浓度均一化,将基因组DNA浓度调整为50ng/L,-20℃保存备用。
2)被测基因引物设计:设计被测基因引物,设计模板为被测基因CDS序列;
3)RT-qPCR:以β-actin为内参基因,用内参基因引物与步骤2)涉及的被测基因引物分别对基因组DNA模板进行荧光定量PCR反应;荧光定量PCR检测方法反应体系为20μL,包括:2×荧光定量预混试剂(SuperRealPreMix Plus(2×))10.0μl,浓度为10μmol·L-1的PCR上下游引物各0.6μL,基因组DNA模板2μl,灭菌蒸馏水6.8μl;PCR程序包括:预变性95℃、15min;变性95℃、10s,退火55-60℃、20s,延伸72℃、20s,40个循环;
内参引物:
上游:5’GATCTACCATGTTCCCAAGT 3’,
下游:5’ATAGAGCCACCAATCCAGAC 3’;
4)以RT-qPCR获得被测基因和内参基因β-actin的Ct值为基础数据,按公式2-(Ct(目的基因)-Ct(内参基因))计算被测基因相对丰度值,设被测基因相对丰度值为a;
5)取品种B大豆,重复上述步骤1)-4),设被测基因相对丰度值为b';
6)根据a/b的值判定被测基因在品种A中的拷贝数与被测基因在品种B中的拷贝数的多少:当a/b>2.5时,被测基因在品种A中的拷贝数显著大于被测基因在品种B中的拷贝数。
优选的,步骤2)所述引物设计遵循以下原则:a)PCR产物长度90bp-200bp;b)引物对在大豆基因组上具有唯一匹配位置。
优选的,用Primer 5.0软件设计引物,设计出的引物要以大豆基因组DNA为模板进行PCR扩增以测试基因引物特异性,以扩增产物唯一为准;并在退火温度55-60℃范围内进行温度梯度PCR确定引物的最佳退火温度,以扩增产物量最大为准;步骤3)中的退火温度选取步骤2)确定的最佳退火温度。
上述引物设计遵循以下原则:
(1)PCR产物长度90bp-200bp;
(2)GC含量40%-60%;
(3)避免形成发夹结构和引物二聚体;
(4)产物避免跨内含子区;
(5)引物设计完成后通过与大豆基因组序列的在线比对(www.phetozome.net),确保引物对在大豆基因组上具有唯一匹配位置。
上述步骤4)-6)中的数据处理方法为:
以被测基因PCR产物相对丰度作为基因拷贝数变异基础数据,被测基因PCR产物相对丰度的计算采用内参基因法,具体通过公式2-ΔΔCt计算。其中ΔΔCt=Ct(目的基因)-Ct(内参基因),对于相对定量ΔΔCt方法,Ct值是由三次试验重复取平均数得,相对拷贝数,即被测基因PCR产物相对丰度由2-ΔΔCt计算得到,利用不同品种的PCR产物相对丰度之间的倍数关系判断基因拷贝数差异。具体操作如下:
(1)对不同被测样本内参基因Ct值进行F测验,在无显著差异情况下进行下一步分析;
(2)对不同被测样本目的基因Ct值进行F测验,同一基因在不同样本间存在显著或极显著差异的情况下进行下一步分析;
(3)计算相对拷贝数比值,即基因PCR产物相对丰度比值,具体为:相对拷贝数比值=2-ΔΔCt样本n/2-ΔΔCt样本n+1,该比值大于2.5作为拷贝数差异阈值。
本发明所提供的大豆基因组基因拷贝数变异分析方法是基于荧光定量PCR结合DNA浓度均一化处理的分析方法,该方法具有低成本及技术难度低等特点,克服现有基因拷贝数变异分析方法的高成本、技术要求高和操作难度大等缺点,可用于大豆基因组任何编码基因或区段的拷贝数变异定性分析和品种基因间拷贝数差异比较,便于实验室常规操作,辅助判断因基因拷贝数变异引起的相应基因组区段功能差异。
有益效果
1基因组DNA用量较高通量测序、微阵列法和原位杂交技术用量少103倍;
2与现有技术相比,无需进行测序和杂交探针合成,成本低廉;
3操作简单,只需进行DNA提取,浓度均一化及PCR反应即可获得数据,对实验人员技术和经验要求低,掌握基本的实验技能即可运用本发明提供的方法,且周期短,可以实现大样本分析;
4理论上可对任意基因组区段进行拷贝数定性分析及品种间比较;
5适用于大样本容量重组群体的拷贝数变异表型分析。
附图说明
图1模板DNA浓度均一化;A:DNA提取浓度检测;B:RNA酶消化后DNA浓度检测;
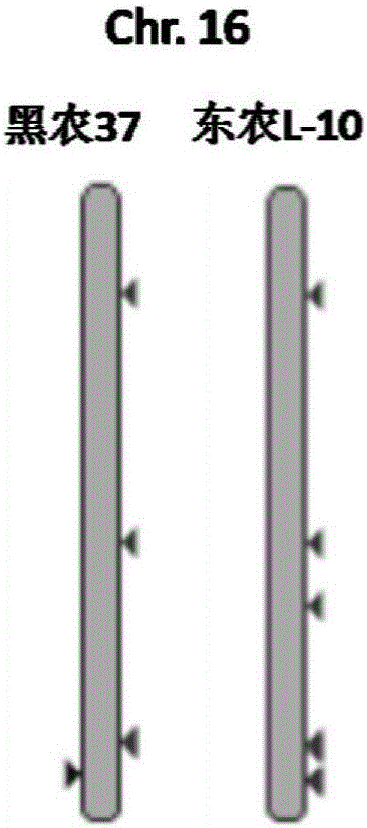
图2基于高通量测序的拷贝数变异区;注:染色体左侧三角代表缺失区段(相对于参考基因组拷贝数低);染色体右侧三角代表获得区段(相对于参考基因组拷贝数高);
图3目标基因引物特异性检测;
A:温度梯度PCR检测;B,C:引物特异性检测。
具体实施方式
实施例1
利用本方法对高通量测序获得的拷贝数变异区进行验证
1)植物材料及DNA提取
采取SDS法提取大豆基因组DNA,采用1%琼脂糖凝胶电泳检测和Lite紫外分光光度计联合检测DNA浓度并通过稀释实现浓度均一化,将基因组DNA浓度调整为50ng/μL,-20℃保存备用。大豆胞囊线虫多小种抗性大豆种质东农L-10为参照材料,感病大豆品种黑农37、绥农10和绥农14为参照材料。
2)目标基因引物设计及特异性分析
通过大豆基因组重测序(测序深度30x),获得抗病品种东农L-10和感病品种黑农37之间拷贝数差异区(图2,表1)。
表1基于基因组重测序的拷贝数变异区预测
结合主效QTL区段,选取拷贝数变异区内27个候选基因进行方法建立和验证试验(表2)。根据其CDS序列进行基因特异性引物设计,以东农L-10为模板,通过设定6个温度梯度PCR来确定最适退火温度(图3A),其中56℃引物和模板结合较好,产物特异性较好,确定为最适退火温度;设置退火温度为56℃,对27对基因特异性引物进行4个模板DNA的PCR分析,经琼脂糖凝胶电泳检测,获得22个特异性好的引物,用于拷贝数变异定性分析(图3B-C)。
表2实时定量PCR引物序列
3)RT-qPCR分析
以β-actin为内参基因,用内参基因引物与步骤2)涉及的被测基因引物分别对基因组DNA模板进行荧光定量PCR反应,与上述引物对分别对四种大豆基因组DNA进行荧光定量PCR;荧光定量PCR检测方法反应体系为20μL,包括:2×荧光定量预混试剂(SuperRealPreMix Plus(2×))10.0μl,浓度为10μmol·L-1的PCR上下游引物各0.6μL,PCR Reverse Primer(10μmol·L-1)0.6μL,基因组DNA模板2μl,灭菌蒸馏水6.8μl;PCR程序包括:预变性95℃、15min;变性95℃、10s,退火56℃、20s,延伸72℃、20s,40个循环。
通过分析RT-qPCR获得被测基因和内参基因β-actin的Ct值,可知,四个品种内参基因的Ct值在26–29之间,其中东农L-10的CT值在26.07–29.62之间,其平均Ct值为26.64;黑农37的Ct值在26.03-28.94之间,平均Ct值为27.07;绥农10的Ct值在26.04-29.68之间,平均Ct值为27.68;绥农14的Ct值在26.18-28.47之间,平均Ct值为27.01;四个品种内参基因Ct值方差分析结果表明在P=0.05水平和P=0.01水平上均不显著(表3)。由此可见,内参基因β-actin在四个品种模板DNA浓度均一化后PCR产物丰度一致性较好,说明不同品种之间内参基因β-actin在基因组模板DNA的初始含量一致,间接说明该基因在被测的不同品种间无拷贝数的差异,可以作为被测目标基因的对照。
表3内参基因β-actin Ct值方差分析
22个目的基因在四个品种中的Ct值差异较大,整体在13–32之间,其中东农L-10的Ct值在13.46–31.78之间,其平均CT值为17.74;黑农37的Ct值在15.47-32.85之间,平均CT值为19.47;绥农10的CT值在14.86-28.88之间,平均Ct值为20.79;绥农14的CT值在15.19-28.17之间,平均Ct值为19.58(表4)。以上结果说明在四个品种模板DNA浓度均一化后不同基因在特定品种当中PCR产物丰度存在较大差异,间接说明不同基因在基因组DNA中初始含量不同,PCR产物丰度差异可以通过q RT-PCR方法检测。
表4目的基因Ct值分析
4)拷贝数变异分析
通过F测验,对同一品种不同基因以及同一基因不同品种进行差异显著性分析,结果表明目的基因18、21在抗病品种东农L-10与感病品种绥农14的中PCR产物丰度差异不显著,目的基因1、2、17、23在东农L-10与黑农37中的PCR产物丰度差异达到显著水平,目的基因22在东农L10与绥农10中的PCR产物丰度差异达到显著水平,其他的15个目的基因的PCR产物丰度在抗病品种与感病品种间均呈极显著差异,由此可以推断这15个目的基因在抗感品种基因组中可能存在拷贝数变异(表5)。
表5 PCR产物丰度显著性分析F测验值
注:F crit0.05=7.71;F crit0.01=21.20
表6 PCR产物相对丰度值
表7 PCR产物相对丰度比较值
在22个基因中,对PCR产物丰度在抗感品种间存在极显著差异的15个基因进行PCR产物相对丰度值的估算(表6),并进行抗病品种东农L-10和其他三个感病品种的PCR产物相对丰度值进行比较(表7),目的基因4分别在东农L-10和绥农14,东农L-10和绥农10,东农L-10和绥农14中的PCR产物相对丰度比值较低,说明目的基因4在东农L-10中拷贝数低于绥农14、绥农10、黑农37的拷贝数;目的基因15,24分别在东农L-10和绥农10、东农L-10和黑农37,东农L-10和绥农14、东农L10和绥农10中的PCR产物相对丰度比值较低,说明目的基因15、24在东农L-10中拷贝数低于在绥农10、绥农14,黑农37。目的基因8、11、27在东农L-10和黑农37的PCR产物相对丰度值较低,说明目的基因8、11、27在东农L-10中拷贝数低于在黑农37中的拷贝数,其他的9个目的基因在东农L-10和黑农37,东农L-10和绥农10,东农L-10和绥农14中的PCR产物相对丰度比值均大于2.5,说明这9个目的基因在东农L-10中的拷贝数高于在黑农37,绥农10,绥农14中的拷贝数,可定性为这9个目的基因在这四个品种中存在拷贝数变异(表7)。
SEQUENCE LISTING
<110> 东北农业大学
<120> 一种大豆基因拷贝数变异分析方法
<130>
<160> 56
<170> PatentIn version 3.5
<210> 1
<211> 20
<212> DNA
<213> CNV1-F
<220>
<221> DNA
<222> (1)..(20)
<400> 1
tcaatcactt tgcctttctg 20
<210> 2
<211> 20
<212> DNA
<213> CNV1-R
<220>
<221> DNA
<222> (1)..(20)
<400> 2
atccgaaaca ttatgaaccc 20
<210> 3
<211> 21
<212> DNA
<213> CNV2-F
<220>
<221> DNA
<222> (1)..(21)
<400> 3
agtcctctgc cacagcgtca c 21
<210> 4
<211> 20
<212> DNA
<213> CNV2-R
<220>
<221> DNA
<222> (1)..(20)
<400> 4
tccaccaaag ctccatctcc 20
<210> 5
<211> 20
<212> DNA
<213> CNV3-F
<220>
<221> DNA
<222> (1)..(20)
<400> 5
atgggtatca agtatgtgga 20
<210> 6
<211> 21
<212> DNA
<213> CNV3-R
<220>
<221> DNA
<222> (1)..(21)
<400> 6
ggttcattat agtgagggag t 21
<210> 7
<211> 20
<212> DNA
<213> CNV4-F
<220>
<221> DNA
<222> (1)..(20)
<400> 7
gggacaggat cagtgagtta 20
<210> 8
<211> 22
<212> DNA
<213> CNV4-R
<220>
<221> DNA
<222> (1)..(22)
<400> 8
aaatagaaga gggaaacacc aa 22
<210> 9
<211> 20
<212> DNA
<213> CNV5-F
<220>
<221> DNA
<222> (1)..(20)
<400> 9
tgacctcctt gactcacctc 20
<210> 10
<211> 21
<212> DNA
<213> CNV5-R
<220>
<221> DNA
<222> (1)..(21)
<400> 10
ccacatactt gatacccatt c 21
<210> 11
<211> 20
<212> DNA
<213> CNV6-F
<220>
<221> DNA
<222> (1)..(20)
<400> 11
tggtgcatca gggaacaatt 20
<210> 12
<211> 20
<212> DNA
<213> CNV6-R
<220>
<221> DNA
<222> (1)..(20)
<400> 12
gaaagctggg acctaactgc 20
<210> 13
<211> 20
<212> DNA
<213> CNV7-F
<220>
<221> DNA
<222> (1)..(20)
<400> 13
attctcaaag acagggatga 20
<210> 14
<211> 20
<212> DNA
<213> CNV7-R
<220>
<221> DNA
<222> (1)..(20)
<400> 14
tagaaattga acgtgccaga 20
<210> 15
<211> 20
<212> DNA
<213> CNV8-F
<220>
<221> DNA
<222> (1)..(20)
<400> 15
atgggtatca agtatgtgga 20
<210> 16
<211> 21
<212> DNA
<213> CNV8-R
<220>
<221> DNA
<222> (1)..(21)
<400> 16
ggttcattat agtgagggag t 21
<210> 17
<211> 20
<212> DNA
<213> CNV9-F
<220>
<221> DNA
<222> (1)..(20)
<400> 17
tcatggcggt atgcctattt 20
<210> 18
<211> 22
<212> DNA
<213> CNV9-R
<220>
<221> DNA
<222> (1)..(22)
<400> 18
acatcggtgt attctaagca ac 22
<210> 19
<211> 20
<212> DNA
<213> CNV10-F
<220>
<221> DNA
<222> (1)..(20)
<400> 19
cactgctcta acgaggcaca 20
<210> 20
<211> 20
<212> DNA
<213> CNV10-R
<220>
<221> DNA
<222> (1)..(20)
<400> 20
gcacaatttg ggaagaaaca 20
<210> 21
<211> 24
<212> DNA
<213> CNV11-F
<220>
<221> DNA
<222> (1)..(24)
<400> 21
agaaggtatt ggtaatatgg gatc 24
<210> 22
<211> 20
<212> DNA
<213> CNV11-R
<220>
<221> DNA
<222> (1)..(20)
<400> 22
gcagtttatg ggcagtggtg 20
<210> 23
<211> 21
<212> DNA
<213> CNV12-F
<220>
<221> DNA
<222> (1)..(21)
<400> 23
gaatgggtat caagtatgtg g 21
<210> 24
<211> 20
<212> DNA
<213> CNV12-R
<220>
<221> DNA
<222> (1)..(20)
<400> 24
gtgggtcaaa gaaggaagag 20
<210> 25
<211> 19
<212> DNA
<213> CNV13-F
<220>
<221> DNA
<222> (1)..(19)
<400> 25
gcctggtgat gaagacagg 19
<210> 26
<211> 22
<212> DNA
<213> CNV13-R
<220>
<221> DNA
<222> (1)..(22)
<400> 26
ttcccagtat tctttgatgt tc 22
<210> 27
<211> 20
<212> DNA
<213> CNV14-F
<220>
<221> DNA
<222> (1)..(20)
<400> 27
aaacaggcag ggcagcagta 20
<210> 28
<211> 20
<212> DNA
<213> CNV14-R
<220>
<221> DNA
<222> (1)..(20)
<400> 28
agtgacgttg tggcagagga 20
<210> 29
<211> 20
<212> DNA
<213> CNV15-F
<220>
<221> DNA
<222> (1)..(20)
<400> 29
ccatgaaggc caacaagaag 20
<210> 30
<211> 20
<212> DNA
<213> CNV15-R
<220>
<221> DNA
<222> (1)..(20)
<400> 30
agaagggaga ccaacagcaa 20
<210> 31
<211> 20
<212> DNA
<213> CNV16-F
<220>
<221> DNA
<222> (1)..(20)
<400> 31
tttatcggca acaatctatg 20
<210> 32
<211> 20
<212> DNA
<213> CNV16-R
<220>
<221> DNA
<222> (1)..(20)
<400> 32
aaggagcaat cactatccag 20
<210> 33
<211> 22
<212> DNA
<213> CNV17-F
<220>
<221> DNA
<222> (1)..(22)
<400> 33
aatggataag agttgtaccg tg 22
<210> 34
<211> 20
<212> DNA
<213> CNV17-R
<220>
<221> DNA
<222> (1)..(20)
<400> 34
gcaaattgct gtaccttagg 20
<210> 35
<211> 20
<212> DNA
<213> CNV18-F
<220>
<221> DNA
<222> (1)..(20)
<400> 35
cccattccca tttgcttata 20
<210> 36
<211> 20
<212> DNA
<213> CNV18-R
<220>
<221> DNA
<222> (1)..(20)
<400> 36
aactgtctgt ccattcaccc 20
<210> 37
<211> 24
<212> DNA
<213> CNV19-F
<220>
<221> DNA
<222> (1)..(24)
<400> 37
ctcttatcag agtttatagc atat 24
<210> 38
<211> 20
<212> DNA
<213> CNV19-R
<220>
<221> DNA
<222> (1)..(20)
<400> 38
tttgttactg tcatcatccc 20
<210> 39
<211> 21
<212> DNA
<213> CNV20-F
<220>
<221> DNA
<222> (1)..(21)
<400> 39
aaggaggttc acaacgcaaa g 21
<210> 40
<211> 24
<212> DNA
<213> CNV20-R
<220>
<221> DNA
<222> (1)..(24)
<400> 40
gctcagatat gaatgatacc caaa 24
<210> 41
<211> 20
<212> DNA
<213> CNV21-F
<220>
<221> DNA
<222> (1)..(20)
<400> 41
gctggcaacc atgacagaag 20
<210> 42
<211> 20
<212> DNA
<213> CNV21-R
<220>
<221> DNA
<222> (1)..(20)
<400> 42
acaggtcgat aagccgcagt 20
<210> 43
<211> 20
<212> DNA
<213> CNV22-F
<220>
<221> DNA
<222> (1)..(20)
<400> 43
tatggctgaa aggaagagga 20
<210> 44
<211> 20
<212> DNA
<213> CNV22-R
<220>
<221> DNA
<222> (1)..(20)
<400> 44
atgccttgtg gaatatgacc 20
<210> 45
<211> 20
<212> DNA
<213> CNV23-F
<220>
<221> DNA
<222> (1)..(20)
<400> 45
ttggacacgg tgatggtaat 20
<210> 46
<211> 21
<212> DNA
<213> CNV23-R
<220>
<221> DNA
<222> (1)..(21)
<400> 46
tctcgcaagt ggtcaagata a 21
<210> 47
<211> 21
<212> DNA
<213> CNV24-F
<220>
<221> DNA
<222> (1)..(21)
<400> 47
tgttgcttag aatacaccga t 21
<210> 48
<211> 20
<212> DNA
<213> CNV24-R
<220>
<221> DNA
<222> (1)..(20)
<400> 48
ctttacatcc caaaacagaa 20
<210> 49
<211> 20
<212> DNA
<213> CNV25-F
<220>
<221> DNA
<222> (1)..(20)
<400> 49
gtgaagaaag cggaggtaga 20
<210> 50
<211> 19
<212> DNA
<213> CNV25-R
<220>
<221> DNA
<222> (1)..(19)
<400> 50
cccttgaatt tctggagga 19
<210> 51
<211> 20
<212> DNA
<213> CNV26-F
<220>
<221> DNA
<222> (1)..(20)
<400> 51
gtctgccacc actcctatta 20
<210> 52
<211> 23
<212> DNA
<213> CNV26-R
<220>
<221> DNA
<222> (1)..(23)
<400> 52
agggtttgta tctgaagagt tta 23
<210> 53
<211> 20
<212> DNA
<213> CNV27-F
<220>
<221> DNA
<222> (1)..(20)
<400> 53
aaccctccat tctcaacctc 20
<210> 54
<211> 20
<212> DNA
<213> CNV27-R
<220>
<221> DNA
<222> (1)..(20)
<400> 54
caaaggtcag gtgtaatccc 20
<210> 55
<211> 20
<212> DNA
<213> 内参引物上游
<220>
<221> DNA
<222> (1)..(20)
<400> 55
gatctaccat gttcccaagt 20
<210> 56
<211> 20
<212> DNA
<213> 内参引物下游
<220>
<221> DNA
<222> (1)..(20)
<400> 56
atagagccac caatccagac 20
- 还没有人留言评论。精彩留言会获得点赞!