一种富集BRCA1和BRCA2基因目标区域的引物、方法、试剂盒及其应用与流程
本发明涉及生物技术领域,具体地涉及一种利用多重pcr富集brca1和brca2基因区域的引物、方法、试剂盒及其应用。
背景技术:
brca1和brca2是重要的抑癌基因,参与染色体损伤修复过程。brca1和brca2的突变可能造成基因功能的降低或缺失,进而增加细胞癌变的风险。现有的数据显示,这两个基因的胚系突变是造成遗传性乳腺及卵巢癌的最重要因素,突变携带者一生中罹患乳腺癌的几率可高达87%,卵巢癌的几率可高达44%。因此,对高危人群进行brca1/2基因突变检测,对于癌症的预防,发现和治疗有着重要的指导意义。
brca1和brca2已发现的突变大部分是单个碱基的突变或少量碱基的缺失和插入。这些突变可以造成基因产物氨基酸序列的改变、翻译的提前终止、或信使rna加工的异常,进而影响蛋白的功能。为了检测这些突变,常规方法是pcr扩增得到感兴趣的基因片段后,以sanger法进行序列测定。全面扫描两个基因所有编码外显子通常需要进行80个以上的pcr反应,扩增片段通常采用正反双向测序以保证准确性,工作量较大,成本较高。
越来越多的实验室开始采用二代测序技术来检测brca1和brca2的突变。二代测序技术由于其高通量的特性,可以覆盖更大的基因组区域,并且在一次测序中可以完成多个样本的突变检测。基因组目的区域的富集是这一技术的关键。大型实验室多采用杂交捕获技术或微滴pcr、微流体pcr等技术。这些技术各有其优点,但在成本和仪器要求上均有较高要求。
例如,申请号201610334650.9公开的用于扩增人乳腺癌易感基因brca1和brca2编码序列的pcr引物,其主要利用引物进行两轮pcr获得,虽然可以得到brca1和brca2基因突变的检测结果,但是由于其扩增出的基因片段不能完全覆盖到目的基因的全部,即其覆盖率不能达到100%,因此可能存在不能准确检测出每个突变位点的技术问题。
长片段扩增(long-rangepcr)技术操作简单,成本低廉,适应性强,同样也是目前使用较广的目的区域富集技术。现有的应用方式,是使用primestar等适用于长片段扩增的dna聚合酶,以10kb左右的扩增子大小覆盖brca1和brca2整个基因组区域,共需要15-20对引物。这种方法每对引物单独扩增,每个样品所需的pcr反应管数较多,不利于大批量操作,由于扩增子存在重合,且扩增长度的设置决定了多重扩增产物中的各个片段无法用简单的方式,如琼脂糖凝胶电泳,进行分析,所以也不适于以多重pcr的方式缩减反应管数。
技术实现要素:
本发明针对现有技术存在的问题,舍弃了部分大跨度内含子的中间区域,设计了一套共26对扩增范围涵盖brca1和brca2所有外显子,和部分内含子区域,互不重叠长度在2kb至8kb之间,分组同时进行多重pcr,每个片段均能通过普通的琼脂糖凝胶电泳进行区分。
为实现本发明的目的,本发明第一方面提供一种富集brca1和brca2基因目标区域的引物,包括:
从其扩增区段包含brca1和brca2基因所有外显子和外显子侧翼的内含子区域的多对引物中得到的五组引物对;
其中,所述五组引物对是根据所述多对引物对brca1和brca2基因扩增得到的扩增片段的大小进行分组得到的,以便将pcr反应数降低到5个;
其中,在对待测样品中的brca1和brca2基因进行扩增时,利用所述五组引物对中的每组引物对分别对待测样品中的brca1和brca2基因进行多重pcr处理,得到5组的扩增产物,实现待测样品中的brca1和brca2基因目标区域的100%富集;
其中,每组扩增产物的扩增片段之间长度有明显区别,
其中,所述引物组合具有如seqidno.1、seqidno.52所示的序列。
其中,所述五组引物对包括:
如seqidno.13、seqidno.14、seqidno.17、seqidno.18、seqidno.23、seqidno.24、seqidno.33、seqidno.34、seqidno.43、seqidno.44所示的第一组引物对,其中,所述seqidno.13和seqidno.14、seqidno.17和seqidno.18、seqidno.23和seqidno.24、seqidno.33和seqidno.34、seqidno.43和seqidno.44为五对互为上下游引物;
如seqidno.7、seqidno.8、seqidno.11、seqidno.12、seqidno.21、seqidno.22、seqidno.27、seqidno.28、seqidno.37、seqidno.38所示的第二组引物对,其中,所述seqidno.7和seqidno.8、seqidno.11和seqidno.12、seqidno.21和seqidno.22、seqidno.27和seqidno.28、seqidno.37和seqidno.38为五对互为上下游引物;
如seqidno.1、seqidno.2、seqidno.31、seqidno.32、seqidno.35、seqidno.36、seqidno.39、seqidno.40、seqidno.41、seqidno.42、seqidno.49、seqidno.50所示的第三组引物对,其中,所述seqidno.1和seqidno.2、seqidno.31和seqidno.32、seqidno.35和seqidno.36、seqidno.39和seqidno.40、seqidno.41和seqidno.42、seqidno.49和seqidno.50为六对互为上下游引物;
如seqidno.5、seqidno.6、seqidno.15、seqidno.16、seqidno.19、seqidno.20、seqidno.25、seqidno.26、seqidno.45、seqidno.46、seqidno.47、seqidno.48所示的第四组引物对,其中,所述seqidno.5和seqidno.6、seqidno.15和seqidno.16、seqidno.19和seqidno.20、seqidno.25和seqidno.26、seqidno.45和seqidno.46、seqidno.47和seqidno.48为六对互为上下游引物;
如seqidno.3、seqidno.4、seqidno.9、seqidno.10、seqidno.29、seqidno.30、seqidno.51、seqidno.52所示的第五组引物对,其中,所述seqidno.3、seqidno.4、seqidno.9、seqidno.10、seqidno.29、seqidno.30、seqidno.51、seqidno.52为四对互为上下游引物。
为实现本发明的技术目的,本发明第二方面提供一种富集brca1和brca2基因目标区域的方法,包括:
设计其扩增区段包含brca1和brca2基因所有外显子和外显子侧翼的内含子区域的多对引物;
根据所述多对引物对brca1和brca2基因扩增处理得到的扩增片段的大小,将所述多对引物分成五组引物对,以便将pcr反应数降低到5个;
利用所述5个引物组中的每个引物组分别对待测样品中的brca1和brca2基因进行多重pcr处理,得到五组扩增产物,实现待测样品中的brca1和brca2基因目标区域的100%富集。
其中,所述五组扩增产物中的每组扩增产物的扩增片段之间的长度有明显区别,使得扩增产物质量易于监控。
其中,所述多对引物具有如seqidno.1-seqidno.52所示的序列。
其中,所述五组引物对包括:
如seqidno.13、seqidno.14、seqidno.17、seqidno.18、seqidno.23、seqidno.24、seqidno.33、seqidno.34、seqidno.43、seqidno.44所示的第一组引物对,其中,所述seqidno.13和seqidno.14、seqidno.17和seqidno.18、seqidno.23和seqidno.24、seqidno.33和seqidno.34、seqidno.43和seqidno.44为五对互为上下游引物;
如seqidno.7、seqidno.8、seqidno.11、seqidno.12、seqidno.21、seqidno.22、seqidno.27、seqidno.28、seqidno.37、seqidno.38所示的第二组引物对,其中,所述seqidno.7和seqidno.8、seqidno.11和seqidno.12、seqidno.21和seqidno.22、seqidno.27和seqidno.28、seqidno.37和seqidno.38为五对互为上下游引物;
如seqidno.1、seqidno.2、seqidno.31、seqidno.32、seqidno.35、seqidno.36、seqidno.39、seqidno.40、seqidno.41、seqidno.42、seqidno.49、seqidno.50所示的第三组引物对,其中,所述seqidno.1和seqidno.2、seqidno.31和seqidno.32、seqidno.35和seqidno.36、seqidno.39和seqidno.40、seqidno.41和seqidno.42、seqidno.49和seqidno.50为六对互为上下游引物;
如seqidno.5、seqidno.6、seqidno.15、seqidno.16、seqidno.19、seqidno.20、seqidno.25、seqidno.26、seqidno.45、seqidno.46、seqidno.47、seqidno.48所示的第四组引物对,其中,所述seqidno.5和seqidno.6、seqidno.15和seqidno.16、seqidno.19和seqidno.20、seqidno.25和seqidno.26、seqidno.45和seqidno.46、seqidno.47和seqidno.48为六对互为上下游引物;
如seqidno.3、seqidno.4、seqidno.9、seqidno.10、seqidno.29、seqidno.30、seqidno.51、seqidno.52所示的第五组引物对组,其中,所述seqidno.3、seqidno.4、seqidno.9、seqidno.10、seqidno.29、seqidno.30、seqidno.51、seqidno.52为四对互为上下游引物。
特别是,所述利用五组引物对中的每组引物对分别对待测样品中的brca1和brca2基因进行多重pcr处理是利用包含长片段dna聚合酶的混合液,并以待测样本的dna为模板,以所述五组引物对中的每组引物对为扩增引物分组进行。
其中,所述多重pcr处理的反应退火温度为62-64℃。延伸时间以最长的扩增子(8kb)为基准。
具体的,所述多重pcr处理的优选反应条件是:95℃预变性5分钟,32个循环98℃变性10秒,62℃退火15秒,68℃延伸9分钟。
特别是,所述长片段dna聚合酶是指具有长片段扩增能力的dna聚合酶。
其中,所述包含长片段dna聚合酶的混合液,为现有技术中常用的dna聚合酶试剂盒,可以通过市售得到,例如takara公司的primestargxl产品,其中包含了缓冲液、dntp、dna聚合酶等成分。
为实现本发明的技术目的,本发明第三方面提供一种用于富集brca1和brca2基因目标区域的试剂盒,其包括第一方面所述的引物,以及具有长片段扩增能力的dna聚合酶;
其中,所述引物的五组引物对中,每个引物的浓度不完全相同。
其中,所述每个引物组的用量为:在20ul的pcr反应体系中,其用量为3.2ul。
特别是,使用所述试剂盒的退火温度为58-64℃,延伸时间以最长的扩增子(8kb)为基准。
优选地,所述退火温度为62-64℃。
优选地,所述具有长片段扩增能力的dna聚合酶为takaraprimestargxl酶
具体的,使用所述试剂盒的反应条件优选为:95℃预变性5分钟,98℃变性10秒,62℃退火15秒,68℃延伸9分钟,32个循环。
为实现本发明的技术目的,本发明第四方面提供一种将第一方面所述的引物用于brca1和brca2基因序列突变检测的用途。
其中,所述将第一方面所述的引物用于brca1和brca2基因序列突变检测包括:
提取待测样本中的dna,获得扩增模板;
以第一方面所述的五组引物对中的每组引物对为扩增引物,分别与所述扩增模板混合,并加入包含长片段dna聚合酶的混合液,得到五组反应液;
将五组反应液在pcr反应仪器中进行多重pcr反应,获得五组扩增产物;
纯化所述五组扩增产物,进行文库构建,经高通量测序后,与数据库比对后,获得brca1和brca2基因序列突变信息。
尤其是,所述每个引物组中的每个引物的浓度不完全相同。
其中,所述五组扩增产物中的每组扩增产物的片段大小不均有明显区别,可经琼脂糖凝胶电泳分离,观察每个片段的扩增结果。
其中,所示五组反应液中每组反应液的浓度使用比例为:
其中,所述多重pcr反应的反应条件为95℃预变性5分钟,32个循环98℃变性10秒,62℃退火15秒,68℃延伸9分钟。
为实现本发明的技术目的,本发明第五方面提供一种将第三方面所述的方法用于brca1和brca2基因目标区域扩增产物的质量监控的用途。
由于所述扩增产物富集了brca1和brca2基因的所有目标区域,并且,扩增产物中的不同片段分别反映了brca1和brca2基因上的不同编码区,因此,将扩增产物进行琼脂糖凝胶电泳就可以直接观察获得待测样本的扩增产物是否符合要求,实时进行质量监控,操作简便,成本低廉。
为实现本发明的技术目的,本发明第六方面提供一种检测brca1和brca2基因序列突变的方法,包括:
利用第二方面所述的方法获得100%富集了brca1和brca2基因目标区域的扩增产物;
将扩增产物纯化后进行文库构建,经测序后,将测序结果与数据库比对分析,获得brca1和brca2基因序列突变信息。
其中,所述测序方法采用高通量测序。
其中,所述数据库为人类基因组数据库,例如人类基因组hg19数据库。
为实现本发明的技术目的,本发明第七方面提供一种检测brca1和brca2基因序列突变的试剂盒,包括第一方面所述的引物,以及具有长片段扩增能力的dna聚合酶;
其中,所述引物的五组引物对中,每组引物对中的每对引物的浓度不完全相同。
本发明所称的待测样本为血液,唾液,口腔拭子,新鲜组织,培养细胞等,不适用于甲醛固定石蜡包埋组织。
有益效果:
1、本发明提供的引物可以与brca1和brca2基因的目标区域特异性结合,扩增出brca1和brca2基因的所有外显子及外显子侧翼的内含子,全面覆盖了具有临床意义的突变位点,实现brca1和brca2基因的目标区域的富集。
2、由于本发明提供的引物为五个引物组,仅需要5个反应数(即使用5个反应管)就可以实现目标区域的富集,而现有长片段扩增技术最低也需高达17个反应,因此,本发明的提供的富集方式成本低,效率高;而且利用本发明的每个引物组对待测样本进行检测,所获得的扩增产物中的每个扩增子均可利用琼脂糖凝胶电泳进行分离,查看每个区域均成功富集后,再进行文库制备和测序,流程更加可靠。
3、本发明提供的富集方法简单、结果可靠、效率高、成本低廉,普适性广,有利于本领域技术人员对brca基因进行多态性分析或序列突变检测或其他研究。
4、本发明提供的试剂盒成本低廉、富集效率高,可靠性好。
5、本发明所提供的富集方法只需5个pcr反应即可保证对brca基因编码区100%的均匀覆盖,提高了检测效率、降低了检测成本。
附图说明
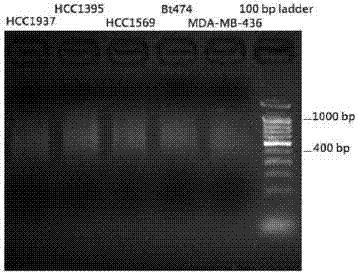
图1是brca1和brca2基因富集产物琼脂糖凝胶电泳图;
图2是二代测序文库琼脂糖凝胶电泳图;
图3是26对brca1和brca2引物扩增区域示意图;
图4是brca1与brca2各外显子的平均覆盖率图。
具体实施方式
为了能够更清楚地理解本发明的上述目的、特征和优点,下面结合附图和具体实施方式对本发明进行进一步的详细描述。
在下面的描述中阐述了很多具体的细节以便于充分理解本发明,但是,本发明还可以采用其他不同于在此描述的其他方式来实施,因此,本发明并不限于下面公开的具体实施例的限制。
实施例1引物
1、本发明采用常规的引物设计方法设计扩增区段包含brca1和brca2基因所有外显子和外显子侧翼内含子区域的引物,该引物有多个,经过常规筛选,获得26个引物,引物信息如表1所示:
表1本发明提供的引物信息
其中,表1中“f”表示上游引物,“r”表示下游引物。
2、将筛选获得的26个引物对brca1和brca2基因扩增处理得到的扩增片段的大小,将所述26个引物分成5组引物对,以便将pcr反应数降低到5个。发明人经过大量的试验调整,成功将26个引物分成了5组引物对,而且每组引物对中的每个引物浓度都不完全相同。
特别是,使用本发明提供的引物的退火温度为62-64℃,延伸时间以最长的扩增子(8kb)为基准。
实施例2试剂盒
本发明提供的试剂盒包括了上述5组引物对,还包括了具有长片段扩增能力的dna聚合酶。
其中,所述具有长片段扩增能力的dna聚合酶为市售的长片段dna聚合酶试剂盒,在本发明的一个实施例中,采用的是例如takara公司的primestargxl产品,其中包含了缓冲液、dntp、dna聚合酶等成分。
其中,试剂盒中每个引物组的用量比例为:在总体积为20μl的反应液中,每个引物组为3.2μl。
具体的,试剂盒中各个成分的比例优选为:
特别是,试剂盒的使用条件是:95℃预变性5分钟;98℃变性10秒,62℃退火15秒,68℃延伸9分钟,32个循环。
其中,试剂盒中每个引物组中的每个引物之间的浓度都不完全相同,如表2所示:
表2引物浓度信息
以下,结合具体的实施步骤进一步说明本发明的特点。
本发明采用已知的5株乳腺癌细胞系作为待测样本,分别为bt-474,hcc1395,hcc1569,mda-mb-361,mda-mb-436,hcc1937,其相关信息如表3所示:
表3乳腺癌细胞系样本
表3中已知突变信息来源为ccle,检测方法为杂交捕获测序。
实施例3brca1和brca2基因目标区域的富集
1、样品的处理和基因组dna提取
从atcc购买的细胞系为帖壁细胞,在t25细胞培养瓶进行贴壁培养后,采用全式金公司的离心柱式基因组dna提取试剂盒easypuremicrogenomicdnakit提取基因组dna,具体步骤按试剂盒操作说明进行提取,得到dna提取液,将dna提取液以100微升洗脱缓冲液洗脱,并经nanodrop2000检测提取质量,并确定浓度,得到最终260/280值大于1.8,浓度均在30至60ng/μl之间的dna溶液。
2、扩增目的基因片段
建立扩增反应体系:
将反应体系在反应条件为95℃预变性5分钟,98℃变性10秒,62℃退火15秒,68℃延伸9分钟,32个循环中进行扩增,得到五组扩增产物,每组扩增产物均各取5μl,在0.8%的琼脂糖凝胶上进行电泳检测,得到图1所示的电泳图,根据电泳图确认条带。
如图1所示的电泳图,每个片段均能表现出明亮的条带,可见,使用本发明提供的引物特异性好,通过普通的琼脂糖凝胶电泳就可以对每个dna片段进行确认。
3、测序
3.1、纯化处理
将每个样品的5组扩增产物等体积混合成一管,组合成一个完整的待测样本。取30μl,加入24μl磁珠(axypreppcrclean-upkit),按说明书进行纯化。纯化后,用nanodrop2000测定浓度,并稀释到10ng/μl。
3.2、高通量测序文库构建
采用诺唯赞公司的trueprepdnalibraryprepkitv2forillumina试剂盒构建文库。该试剂盒构建文库包含两个主要步骤,首先是利用转座酶将步骤2.3获得的目的基因片段打断,然后在打断后的片段两端加上适用于illuminahiseq测序平台的接头,并富集文库,具体步骤遵循试剂盒操作说明,得到构建后的测序文库。
向构建的文库中加入0.6倍体积的磁珠(axypreppcrclean-upkit)进行磁珠纯化,去除小于300bp的片段,得到如图2所示的电泳图。
3.3、高通量测序
在illuminahiseq2000平台上对5个文库测序,获得每个文库的数据,与样本的已知序列信息比对,发现所的序列覆盖了brca1和brca2基因上的所有外显子及外显子侧翼的内含子,如图3所示。
根据图3所示的26对brca1和brca2引物扩增区域示意图可知,扩增子完全覆盖了brca1和brca2基因上的所有外显子,有些甚至覆盖了外显子之间的内含子。
由此可见,本发明提供的方法可以实现brca1和brca2基因所有外显子及外显子侧翼内含子的富集。
4数据分析
采用bcbio-nextgen流程对高通量测序结果数据进行分析。高通量测序得到的reads通过bwa工具与人类基因组hg19数据库比对,通过freebayes工具进行多态位点预测,并以snpeff或varianteffectpredictor(vep)对多态进行注释。最后生成的vcf文件,在ncbi网站上与clinvar数据库交叉对比,得到样本测序结果的总read数、gc含量%、文库插入片段中间值、pcr重复、pcr重复比例%、成功比对read数、成功比对比例%、脱靶read数、脱靶比例%,得到如表4所示的样本测序结果指标以及图4所示的brca1与brca2各外显子的平均覆盖率图。
表4样本测定指标结果
根据表4所示的测定结果中的“总read数”与“成功比对read数”的比例计算值可知,超过99%的read可以比对到hg19基因组上,可见,本发明获得样本生物信息准确。文库插入片段大小中间值差异较小,说明文库构建方法稳定。pcr重复比例,符合预期,脱靶比例最高仅为4.1%,说明目的基因片段的多重扩增特异性满足要求。
图4为brca1与brca2各外显子的平均覆盖率。纵轴表示覆盖乘数,以2的对数展示。横轴1-27依次对应brca2的1-27号外显子,28-50依次对应brca1的23-1号外显子(依据两基因在染色体上的位置和方向依次排列)。在各个外显子上,获得了较为均匀的覆盖,可见,本发明提供的引物组合实现了brca1和brca2基因外显子的富集。
实施例4brca1和brca2基因序列突变的检测
应用实施例3的方法获得的测序结果,与人类基因组hg19数据库比对,获得分析结果,其中,五个细胞系中检测到的有临床意义的突变如表5所示。表中包括了可能对蛋白氨基酸序列产生影响,或临床致病性有文献报道的基因突变,其中可能存在临床致病性的突变黑体标出。
如表5所示的突变检测结果可知,五个细胞系中已知的致病性突变均成功检出,与样本的突变信息结果一致,符合率100%。
本发明利用多重pcr,扩增brca1和brca2所有外显子和毗邻的内含子区域,得到的pcr产物,利用得到的pcr产物构建高通量测序文库,经高通量测序后,一次性检测所有可能存在临床意义的brca1和brca2点突变。
需要说明的是本发明将引物组合应用于brca1和brca2序列突变的检测,虽然在某些情况下,对这些基因的检测对于相关癌症的诊断、预防和治疗有重要的辅助意义,但这些基因的突变不是必然导致所述疾病的发生,如表5所示的检测结果,因此对这些基因的检测并不一定涉及疾病的诊断或治疗方法。
以上所述仅为本发明的优选实施例而已,并不用于限制本发明,对于本领域的技术人员来说,本发明可以有各种更改和变化。凡在本发明的精神和原则之内,所作的任何修改、等同替换、改进等,均应包含在本发明的保护范围之类。
序列表
<110>北京明谛生物医药科技有限公司
<120>一种富集brca1和brca2基因目标区域的引物、方法、试剂盒及其应用
<160>5
<170>patentinversion3.5
<210>1
<211>26
<212>dna
<213>人工序列
tgaagggcaagggagaggatggttat26
<210>2
<211>20
<212>dna
<213>人工序列
tctccggcgcttttcgccca20
<210>3
<211>26
<212>dna
<213>人工序列
actcaagctgacaagaaaacatacag26
<210>4
<211>30
<212>dna
<213>人工序列
tgtttttacaaagtaatcacaaaccaaaca30
<210>5
<211>24
<212>dna
<213>人工序列
agctccaaagcaaggaagggccta24
<210>6
<211>24
<212>dna
<213>人工序列
ccctacctagtcacccccttcacc24
<210>7
<211>27
<212>dna
<213>人工序列
agctcttcttaaaaggcttcctcatct27
<210>8
<211>24
<212>dna
<213>人工序列
aggagtgctggggttttattgtca24
<210>9
<211>23
<212>dna
<213>人工序列
gccttgaccattaggtccagaaa23
<210>10
<211>22
<212>dna
<213>人工序列
aggcagaggtgagtggatacat22
<210>11
<211>29
<212>dna
<213>人工序列
actcacaccagatttagggagaaaaaggc29
<210>12
<211>24
<212>dna
<213>人工序列
tggatccttccctctcacaggtcc24
<210>13
<211>28
<212>dna
<213>人工序列
ggccaaataccacatttgatgccaaaca28
<210>14
<211>25
<212>dna
<213>人工序列
acttcctagaaggcagtggcacaaa25
<210>15
<211>27
<212>dna
<213>人工序列
tgcccggccatgcaattatttttatta27
<210>16
<211>26
<212>dna
<213>人工序列
gcacccggcctgtactcttattcttt26
<210>17
<211>23
<212>dna
<213>人工序列
tccacctcaccagtgtagtccca23
<210>18
<211>22
<212>dna
<213>人工序列
ctggcggagggtggtagcaaaa22
<210>19
<211>25
<212>dna
<213>人工序列
acagaagttctcagatggctgttct25
<210>20
<211>22
<212>dna
<213>人工序列
gggctccagaaagcagacgagt22
<210>21
<211>24
<212>dna
<213>人工序列
acttagggagctgagaaagcagcc24
<210>22
<211>24
<212>dna
<213>人工序列
aagtcccagtgaggtgaaaagccg24
<210>23
<211>22
<212>dna
<213>人工序列
gctgcgcaactccagaggtaag22
<210>24
<211>24
<212>dna
<213>人工序列
caatgaggagaggcagaacctggc24
<210>25
<211>23
<212>dna
<213>人工序列
gacatggggctgcttttactgtc23
<210>26
<211>23
<212>dna
<213>人工序列
ggggtgagttagtcaaccttcgt23
<210>27
<211>24
<212>dna
<213>人工序列
gtttactgctcaagcaccttctgg24
<210>28
<211>26
<212>dna
<213>人工序列
agccagacattaaagagaactgcaaa26
<210>29
<211>24
<212>dna
<213>人工序列
aaatgtgccagcagttttacccag24
<210>30
<211>24
<212>dna
<213>人工序列
gccttgtctattggtccctttcag24
<210>31
<211>24
<212>dna
<213>人工序列
gctcacagcctagtagggggaaaa24
<210>32
<211>27
<212>dna
<213>人工序列
agtcatgattagccaaatctcagctac27
<210>33
<211>24
<212>dna
<213>人工序列
actgggccagatcattgtttctca24
<210>34
<211>25
<212>dna
<213>人工序列
ggacagagttacaccacggagatgc25
<210>35
<211>26
<212>dna
<213>人工序列
tcaggctgttagtatatggaccctgt26
<210>36
<211>23
<212>dna
<213>人工序列
gcaagccaacacttactgaatgc23
<210>37
<211>24
<212>dna
<213>人工序列
acttagggagctgagaaagcagcc24
<210>38
<211>24
<212>dna
<213>人工序列
gcctggcctcaattcaccatcttt24
<210>39
<211>24
<212>dna
<213>人工序列
ccgtggtgtgccatctcagagtag24
<210>40
<211>25
<212>dna
<213>人工序列
gggggaggaatctaccataaggcca25
<210>41
<211>22
<212>dna
<213>人工序列
gatcgttggccttgtaggcagc22
<210>42
<211>24
<212>dna
<213>人工序列
gaatgaggatggggagggttgtgc24
<210>43
<211>24
<212>dna
<213>人工序列
ctggctgcgtctgctcagtattc23
<210>44
<211>23
<212>dna
<213>人工序列
aaattgccacaactgccccaacc23
<210>45
<211>25
<212>dna
<213>人工序列
gactccatgatccatgggccacaaa25
<210>46
<211>23
<212>dna
<213>人工序列
gttacagagtggctgggcaaggc23
<210>47
<211>23
<212>dna
<213>人工序列
ttgaggagaacgtaggcatccat23
<210>48
<211>29
<212>dna
<213>人工序列
acaataaaatgtgtgaaaagagtcatcca29
<210>49
<211>24
<212>dna
<213>人工序列
ccctgccctagtcttggaatcagc24
<210>50
<211>30
<212>dna
<213>人工序列
gtcaacacagctttctttatacagctactc30
<210>51
<211>27
<212>dna
<213>人工序列
agtttctaaattaggggtgggagttgt27
<210>52
<211>24
<212>dna
<213>人工序列
ctttccctagctgacccctgaaat24
- 还没有人留言评论。精彩留言会获得点赞!