核酸的精确识别方法与流程
本公开涉及生物识别领域,尤其涉及一种核酸的精确识别方法。
背景技术:
为了研究细胞中核酸分子的结构和相互作用,需要对核酸分子的有无、位置、形态等进行识别。一种常见的方法是荧光原位杂交法(fluorescenceinsituhybridization,fish)。在该方法中,使探针与目标核酸基于序列的高度互补性而杂交,然后依靠直接或间接标记探针的荧光团而成像,从而对特定序列的目标核酸进行识别和定位(非专利文献1)。
然而,由于衍射极限的限制,传统的光学显微法只能获得侧向约200~300nm、轴向约600nm的成像分辨率,因而无法分辨更小尺度的细胞器或分子结构。为了克服这一问题,近年来提出了多种超分辨成像法。例如,非专利文献2提出用含有2.1kbhaeiii片段的dna探针对含有重复序列的染色体区域进行标记,通过光启动定位显微法(photo-activatedlocalizationmicroscopy,palm)获得分辨率约50nm的图像。然而,非重复的核酸区域,特别是长度较短的核酸区域,其标记和识别是非常困难的。
专利文献1公开了一种超分辨成像方法。在该方法中,使对接链与待识别目标分子杂交,再用荧光标记的成像链与对接链结合,然后通过激活成像链上的荧光团而进行成像。该方法需要使用至少两种功能性分子(对接链和成像链),因而操作繁琐,并且对目标分子标记的效率不利。
例如,在专利文献1提供的实例中,以“dna涂料”(dnapaint)作为对接链。dna涂料是沿着待识别染色体区域的全长而将其标记的标志物(专利文献2)。用户需要建立复杂的系统来制备dna涂料,并且在用pcr制备dna涂料的过程中,由于序列偏好,制得的dna涂料的终浓度可发生偏差,导致fish标记的不稳定。
特别地,dna涂料与待测样品可产生非特异性结合,导致成像质量下降。为了减轻非特异结合,需提高dna涂料与样品杂交时的温度。然而温度的升高又导致所需的特异性杂交的强度降低,因而不得不需要更多数量的成像链,才足以有效地标记待测目标分子。这妨碍了分辨率的进一步提高,因而难以实现对更短的目标分子区域的高分辨成像。
引证文件列表
专利文献
专利文献1:wo2015/017586
专利文献2:us2010304994(a1)
非专利文献
非专利文献1:langer-saferpr,levinem,warddc.immunologicalmethodformappinggenesondrosophilapolytenechromosomes.procnatlacadsciusa.1982jul;79(14):4381-5.
非专利文献2:weiland,y.,lemmer,p.,cremer,c.2011.combiningfishwithlocalisationmicroscopy:super-resolutionimagingofnucleargenomenanostructures.chromosomeres,19,5-23.
非专利文献3:rouillard,j.m.,zuker,m.&gulari,e.2003.oligoarray2.0:designofoligonucleotideprobesfordnamicroarraysusingathermodynamicapproach.nucleicacidsres,31,3057-62.
非专利文献4:markham,n.r.&zuker,m.2008.unafold:softwarefornucleicacidfoldingandhybridization.methodsmolbiol,453,3-31.
非专利文献5:huang,b.,wang,w.,bates,m.&zhuang,x.2008.three-dimensionalsuper-resolutionimagingbystochasticopticalreconstructionmicroscopy.science,319,810-3.
技术实现要素:
在本文中使用时,除非另有说明,下列术语/词语具有下列含义:
“dna”表示脱氧核糖核酸。
“rna”表示核糖核酸。
“分子信标(molecularbeacon)”表示一种茎环结构的寡核苷酸片段,5’端和3’端分别缀合荧光团和淬灭团,所述分子信标在下列两种状态下形态不同,从而产生可识别的信号:(i)分子信标没有结合于靶核酸而游离存在,(ii)分子信标与靶核酸结合。
“tm值”表示双链核酸的双螺旋结构解离一半时的温度。
发明要解决的问题
本公开的目的是提供一种核酸的识别方法,该方法不仅可简化核酸识别的步骤,而且可高效地对靶核酸分子进行标记,提高识别的效率和准确性。
用于解决问题的方案
本公开提供一种核酸的识别方法,所述识别方法包括以下步骤:
a.使样品与杂交探针接触,
b.任选地,将未与靶核酸结合的杂交探针洗脱,和
c.识别所述杂交探针是否与靶核酸结合;
其中,所述杂交探针包含报告基团,且所述杂交探针在下列两种情况下的状态不同,从而通过所述报告基团产生可识别的信号:(i)所述杂交探针处于游离状态,(ii)所述杂交探针与所述靶核酸结合。
根据本公开的一方面,识别方法的步骤c通过荧光分光光度计读取或荧光显微成像而进行,优选所述荧光显微成像为超分辨成像。
根据本公开的一方面,杂交探针包含结合区域和解离区域,当所述杂交探针处于游离状态时,所述解离区域自杂交,所述报告基团不发出可识别的信号;当所述杂交探针通过所述结合区域与所述靶核酸的靶序列片段发生特异性结合时,所述解离区域解离,所述报告基团发出可识别的信号。
根据本公开的一方面,所述报告基团包括荧光团和淬灭团,所述可识别的信号为荧光;优选所述荧光团为alexa-647,和/或所述淬灭团为bhq3。
根据本公开的一方面,所述杂交探针包含充当所述结合区域的环结构,和充当所述解离区域的至少两个臂结构,其中所述至少两个臂结构分别位于所述环结构两侧。
根据本公开的一方面,所述环结构的长度为20nt~60nt,优选25nt~55nt,更优选30nt~50nt,更优选35nt~45nt,还进一步优选40nt~44nt,和/或所述臂结构的长度为4nt~10nt,优选5nt~9nt,优选6nt~8nt,更优选7nt。
根据本公开的一方面,所述环结构与所述靶核酸的靶序列片段之间的序列同一性为90%~100%,优选95%~100%,更优选98%~100%。
根据本公开的一方面,所述靶核酸的长度小于4.9kb;优选地,所述靶核酸的长度为3.3kb以下,更优选2.5kb以下。
根据本公开的一方面,所述靶核酸不含重复序列。
根据本公开的一方面,所述识别方法的步骤a按照以下方法进行:使样品与杂交探针在70℃~80℃、优选73℃~77℃、更优选75℃的温度下共同温育,然后在10℃~42℃、更优选12℃~38℃、更优选14℃~34℃、更优选16℃~30℃、更优选18℃~26℃、更优选22℃的温度下杂交。
根据本公开的一方面,所述超分辨成像在x-y方向上的单分子重复定位精度的标准差为50nm以下,优选20nm以下,进一步优选9nm以下;或者,所述超分辨成像在x-y方向上的单分子重复定位精度的半高全宽为120nm以下,优选50nm以下,进一步优选20nm以下。
根据本公开的一方面,所述超分辨成像在z方向的单分子重复定位精度的标准差为100nm以下,优选40nm以下,进一步优选20nm以下;或者,所述超分辨成像在z方向的单分子重复定位精度的半高全宽为250nm以下,优选100nm以下,进一步优选50nm以下。
根据本公开的一方面,激发所述荧光团时使用的激光功率为0.80kw/cm2~2.00kw/cm2,优选0.90kw/cm2~1.45kw/cm2,进一步优选1.00kw/cm2。
根据本公开的一方面,识别所述荧光时的采样帧频为10hz以上,优选50hz以上,更优选60hz以上,进一步优选85hz以上。
本公开还提供杂交探针在靶核酸的荧光分光光度法读取或荧光显微成像中的用途,其中,所述杂交探针包含报告基团,且所述杂交探针在下列两种情况下的状态不同,从而通过所述报告基团产生可识别的信号:(i)所述杂交探针处于游离状态,(ii)所述杂交探针与所述靶核酸结合。
在根据本公开的用途中,优选所述荧光显微成像为超分辨成像;优选靶核酸的长度小于4.9kb,例如长度为3.3kb以下,例如2.5kb以下;优选靶核酸不含重复序列;优选超分辨成像在x-y方向上的单分子重复定位精度的标准差为50nm以下,例如20nm以下,例如9nm以下;或者,所述超分辨成像在x-y方向上的单分子重复定位精度的半高全宽为120nm以下,例如50nm以下,例如20nm以下;或者,优选所述超分辨成像在z方向的单分子重复定位精度的标准差为100nm以下,例如40nm以下,例如20nm以下;或者,优选所述超分辨成像在z方向的单分子重复定位精度的半高全宽为250nm以下,例如100nm以下,例如50nm以下。
发明的效果
本公开的核酸的识别方法可简化核酸识别的操作步骤,高效地对靶核酸分子进行标记,降低探针与靶核酸以外的核酸的非特异性结合,提高识别的效率和准确性,还特别地有利于长度较短和/或非重复序列的核酸的识别,特别适合于获得靶核酸的超分辨图像。
特别地,通过使用本公开的核酸识别方法,消除了选择杂交温度的两难问题,可在提高特异性杂交强度的同时减弱非特异性结合,从而实现了对更短(例如小于4.9kb)的靶核酸片段的高分辨成像。
另一方面,本公开的核酸识别方法简化了操作步骤和对试剂的要求,无需使用者建立昂贵而复杂的核酸涂料生成系统,不仅节约成本,核酸涂料及探针质量稳定可控,而且操作上更方便可行。
附图说明
包含在说明书中并且构成说明书的一部分的附图与说明书一起示出了本公开的示例性实施例、特征和方面,并且用于解释本公开的原理。
图1a示出实施例1中制备的插入细胞基因组的靶核酸片段的结构。
图1b示出实施例1中制备的细胞样品(i)的pcr检测结果,显示出阳性样品包含靶核酸片段,阴性样品不含靶核酸片段。
图2示出实施例1中的3.3kb的靶核酸片段的反义链的序列。
图3a示出杂交探针mb1至mb29与相应的互补或不互补的寡核苷酸在室温(22℃)下共同反应后的荧光发射读数。
图3b示出杂交探针mb1至mb29与相应的互补或不互补的寡核苷酸在不同温度下共同温育后的荧光发射读数。
图3c示出用不同的采样帧频和激光功率进行成像时,记录到的事件数随着时间推移的变化。
图4a示出对实施例4中制备的阳性样品进行storm成像时所获得的1378个位置信息。
图4b示出图4a中的位置信息在x、y、z方向上的分布。
图5示出对实施例4中制备的阳性样品,用传统光学显微镜和storm超分辨成像法重构后获得的3个示例性靶核酸的图像。
图6示出图5中的三个通过storm法获得的示例性视野图像中红线所在区域的强度归一化分布。
图7示出实施例7中制备的细胞样品(ii)的pcr检测结果,显示出阳性样品包含靶核酸片段,阴性样品不含靶核酸片段。
图8示出实施例7中的2.5kb的靶核酸片段的有义链的序列。
图9a示出杂交探针mb30至mb63与相应的互补或不互补的寡核苷酸在室温(22℃)下共同反应后的荧光发射读数。
图9b示出杂交探针mb30至mb63与相应的互补或不互补的寡核苷酸在不同温度下共同温育后的荧光发射读数。
图10示出对实施例10中制备的阳性样品,用传统光学显微镜和storm超分辨成像法重构后获得的3个示例性靶核酸的图像。
图11示出图10中的三个通过storm法获得的示例性视野图像中红线所在区域强度归一化分布。
图12示出根据本公开的示例性实施方式的原理示意图。
具体实施方式
以下将参考附图详细说明本公开的各种示例性实施例、特征和方面。在这里专用的词“示例性”意为“用作例子、实施例或说明性”。这里作为“示例性”所说明的任何实施例不必解释为优于或好于其它实施例。
另外,为了更好地说明本公开,在下文的具体实施方式中给出了众多的具体细节。本领域技术人员应当理解,没有某些具体细节,本公开同样可以实施。在一些实例中,对于本领域技术人员熟知的方法、手段、试剂和设备未作详细描述,以便于凸显本公开的主旨。
本公开提供一种核酸的识别方法,所述识别方法包括以下步骤:
a.使样品与杂交探针接触,
b.任选地,将未与靶核酸结合的杂交探针洗脱;和
c.识别所述杂交探针是否与靶核酸结合;
其中,所述杂交探针包含报告基团,且所述杂交探针在下列两种情况下的状态不同,从而通过所述报告基团产生可识别的信号:(i)所述杂交探针处于游离状态,(ii)所述杂交探针与所述靶核酸结合。
样品
用本公开的识别方法识别的样品可以是天然或者合成的核酸,也可以是取自生物体的组织或细胞、培养的细胞等,例如,样品可以为组织冰冻切片、石蜡切片、细胞爬片、组织/细胞裂解物等。样品的前处理可参照通常的原位杂交样品的前处理方法进行。
杂交探针
杂交探针包含报告基团,且所述杂交探针在下列两种情况下的状态不同,从而通过所述报告基团产生可识别的信号:(i)所述杂交探针处于游离状态,(ii)所述杂交探针与所述靶核酸结合。满足上述条件时,杂交探针的具体结构没有特别限制。例如,杂交探针可包含结合区域和解离区域,当所述杂交探针处于游离状态时,所述解离区域自杂交,所述报告基团不发出可识别的信号;当所述杂交探针通过所述结合区域与所述靶核酸的靶序列片段发生特异性结合时,所述解离区域解离,所述报告基团发出可识别的信号。
杂交探针的结合区域和解离区域的相互关系可以是,例如,解离区域包含在结合区域中,或者解离区域与结合区域部分重叠,或者解离区域位于结合区域以外。
优选地,所述报告基团包括荧光团和淬灭团,所述可识别的信号为荧光。即,当所述杂交探针处于游离状态时,所述解离区域自杂交,所述荧光团被所述淬灭团淬灭,从而报告基团不发出荧光;当所述杂交探针通过所述结合区域与所述靶核酸的靶序列片段发生特异性结合时,所述解离区域解离,所述淬灭团对所述荧光团的淬灭作用解除,从而报告基团发出荧光。
杂交探针可以是dna探针、rna探针、经化学修饰的寡核苷酸核酸探针、核酸类似物等。优选杂交探针为单链dna探针或单链rna探针。
在本公开的一种优选的实施方式中,杂交探针具有茎-环状结构,例如发卡状结构。例如,杂交探针包含充当结合区域的环结构,和充当解离区域的至少两个臂结构,其中至少两个臂结构分别位于环结构两侧,当杂交探针没有与靶核酸接触时,位于环结构5’侧的臂结构(以下简称5’臂结构)和位于环结构3’侧的臂结构(以下简称3’臂结构)互补结合形成稳定的茎状结构;荧光团和淬灭团分别位于5’臂结构的5’端和3’臂结构的3’端,并且荧光团和淬灭团的位置可以互相交换,即,可以是荧光团位于5’臂结构的5’端而淬灭团位于3’臂结构的3’端,或者也可以是荧光团位于3’臂结构的3’端而淬灭团位于5’臂结构的5’端。
环结构的长度没有特别限制,只要在杂交温度下环结构与靶核酸有充分的结合强度即可。出于设计难度、制备探针的成本等多方面的考虑,优选环结构的长度为20~60个核苷酸(即,20nt~60nt),更优选25nt~55nt,更优选30nt~50nt,更优选35nt~45nt,还进一步优选40nt~44nt。
关于环结构的具体核酸序列,本领域技术人员可根据待识别的靶核酸的序列,考虑序列的gc含量、tm值,序列特异性等因素而适当地进行设计,例如按照非专利文献3中介绍的方法进行,在此通过引用将该文献并入本公开。优选地,环结构与靶核酸的反向互补序列之间的序列同一性为90%~100%,优选95%~100%,优选98%~100%。最优选环结构与靶核酸的反向互补序列之间的序列同一性为100%,即,环结构与靶核酸反向互补。
臂结构的长度和核酸序列没有特别限制,臂结构的设计可参照非专利文献4介绍的方法进行,在此通过引用将该文献并入本公开。优选地,臂结构的长度为4nt~10nt,优选5nt~9nt,优选6nt~8nt,更优选7nt。
在本公开的一种优选的实施方式中,杂交探针可以是由以下通式(1)表示的单链核酸:
5’-x1x2…xmy1y2…ynx’1x’2…x’m-3’(1)
其中,x1至xm、y1至yn、x’1至x’m表示任意的核苷酸,
由y1y2…yn表示的核苷酸链片段与靶核酸反向互补,
由x1x2…xm表示的核苷酸链片段与由x’1x’2…x’m表示的核苷酸链片段反向互补,
由y1y2…yn表示的核苷酸链片段与靶核酸结合的tm值高于由x1x2…xm表示的核苷酸链片段与由x’1x’2…x’m表示的核苷酸链片段结合的tm值,
核苷酸x1上缀合有荧光团且核苷酸x’m上缀合有淬灭团,或者核苷酸x1上缀合有淬灭团且核苷酸x’m上缀合有荧光团,
m为选自4~10、优选5~9、优选6~8的整数,更优选m=7,
n为选自20~60、优选25~55、优选30~50、优选35~45、更优选40~44的整数。
优选地,由y1y2…yn表示的核苷酸链片段满足以下条件的至少之一:i)tm值不低于70℃,ii)当样品中包含基因组核酸时,由y1y2…yn表示的核苷酸链片段的序列与样品中包含的基因组非目标核酸的相似序列长度不超过25nt,iii)不包含6个或更多个连续重复的核苷酸,iv)在等于或高于杂交温度的温度下没有二级结构形成。
优选地,由x1x2…xm表示的核苷酸链片段和由x’1x’2…x’m表示的核苷酸链片段满足以下条件的至少之一:i)由x1x2…xm表示的核苷酸链片段与由x’1x’2…x’m表示的核苷酸链片段为高gc含量片段(75%~100%),ii)由x1x2…xm表示的核苷酸链片段与由x’1x’2…x’m表示的核苷酸链片段结合的tm值在50℃~60℃的范围内,iii)由x1x2…xm表示的核苷酸链片段和由x’1x’2…x’m表示的核苷酸链片段均不与由y1y2…yn表示的核苷酸链片段形成二级结构。
优选地,缀合有荧光团的核苷酸中的碱基为c(胞嘧啶)。
在本公开的一种优选的实施方式中,杂交探针为分子信标。
荧光团和淬灭团
杂交探针的报告基团为可识别标记物,优选地包括荧光团和淬灭团。优选荧光团的最大发射波长与淬灭团的最大吸收波长接近,以获得优化的淬灭效果。
按照本公开的内容使用的荧光团的实例包括但不限于,荧光素类、得克萨斯红、硝基苯并-2-氧杂-1,3-二唑-4-基(nbd)、香豆素类、丹磺酰氯和罗丹明等。优选的荧光团为,例如由thermofisher以molecular
按照本公开的内容使用的淬灭团的实例包括但不限于,4-(4'-二甲基氨基偶氮苯基)苯甲酸(dabcyl)、黑洞淬灭剂-1(blackholequencher1,bhq-1)、黑洞淬灭剂-2(bhq-2)、黑洞淬灭剂-3(bhq-3)等。
靶核酸
靶核酸即为欲通过本公开的方法识别的核酸或核酸片段。靶核酸可为,例如,单链或双链dna,包括但不限于基因组dna、cdna等,或者单链或双链rna,包括但不限于信使rna、核糖体rna、microrna、病毒rna等。
优选靶核酸的长度小于4.9kb;更优选地,所述靶核酸的长度为3.3kb以下,进一步优选2.5kb以下,特别优选2.0kb~2.5kb。优选地,靶核酸不包含重复序列。现有技术中,对于长度小于4.9kb的不含重复序列的靶核酸,并没有成功标记的先例。此外,优选靶核酸含有增强子序列或启动子序列。
本公开的方法对标记识别多种形态的靶核酸是普遍适用的,无论靶核酸是否包含重复序列。在本公开中,重复序列定义为基因组中不同位置或同一大范围位置中多次出现的相同单元或对称性片段,包括但不限于轻度、中度、高度重复序列如alu家族、着丝粒、端粒等;非重复序列定义为在整个基因组中独有的一段序列,没有与它相似的重复序列(在多倍体,如二倍体中,非重复序列可分别出现在配对染色体中)。不过,如果用于识别不包含重复序列的靶核酸,则本公开的优点变得更加明显。现有技术中对不含重复序列的短靶核酸的标记存在技术障碍,对于长度小于4.9kb的不含重复序列的靶核酸的标记未见报道。当不含重复序列的靶核酸的长度较短,例如小于4.9kb时,受限于靶核酸长度、序列特性及每个杂交探针结合区域核酸序列长度,与靶核酸结合的杂交探针的总数量较少,进而导致成功与靶核酸结合的杂交探针上的报告基团数量较少,总信号微弱,难以与背景区分,阻碍了识别精度的进一步提高。而本公开的核酸识别方法成功实现了对长度小于4.9kb的非重复靶核酸短片段的高分辨率成像,如下文的实施例6、11所例示。
自淬灭探针与样品的接触
使样品与杂交探针接触的方法和条件没有特别限制,只要使杂交探针与样品中可能存在的靶核酸结合即可。例如,可使样品与自淬灭探针在70℃~80℃、优选73℃~77℃、更优选75℃的温度下共同温育,然后在10℃~42℃、更优选12℃~38℃、更优选14℃~34℃、更优选16℃~34℃、更优选18℃~26℃、更优选22℃的温度下杂交。
识别自淬灭探针是否与靶核酸结合的方式
在本公开的核酸识别方法中,可根据样品、待测自淬灭探针和靶核酸的特性,依照识别的目的,适当地选择识别自淬灭探针是否与靶核酸结合的方式。例如,可采用荧光分光光度计读取,或采用荧光显微成像,如普通荧光显微成像、共聚焦显微成像或荧光超分辨成像等。当报告基团包括荧光团和淬灭团时,激发和识别荧光所用的波长可根据荧光团自身的性质适当地选择。
虽然多种识别自淬灭探针是否与靶核酸结合的方式都适合在本公开的核酸识别方法中使用(例如上文举出的非限制性实例),但如果结合荧光超分辨成像,则本公开的优越性表现得明显。当结合荧光超分辨成像方法时,本公开的核酸识别方法所实现成像分辨率,若以单分子重复定位精度的标准差计,在x-y方向上达到50nm以下,特别地为9nm,和/或在z方向上达到40nm以下,特别地为22nm;或者以单分子重复定位精度的半高全宽计,在x-y方向上达到50nm以下,特别地为22nm,和/或在z方向上达到100nm以下,特别地为52nm。这是现有技术所难以实现的高分辨率成像,特别是对于长度低于4.9kb的非重复靶核酸短片段,现有技术中未见对其进行高分辨率成像的报道。
激发时使用的激光功率可根据样品的状态、荧光团的种类等条件适当地进行调整。优选地,激发时使用的激光功率为0.80kw/cm2~2.20kw/cm2。当荧光团为alexa-647时,优选激发时使用的激光功率为0.80kw/cm2~2.00kw/cm2,更优选0.90kw/cm2~1.45kw/cm2,进一步优选1.00kw/cm2。采用优选范围的激发时的激光功率,一方面可以激发荧光团从而获得足够强的荧光信号,另一方面有助于延缓荧光团被光漂白的进程。
识别荧光时采用的采样频率可根据对成像质量的需求、成像设备的性能等条件而适当地进行调整。优选识别荧光时采用的采样帧频为10hz以上,更优选50hz以上,更优选60hz以上,进一步优选85hz以上。识别荧光时,采用优选范围的激发光激发功率和采样帧频,有助于提高成像质量,获得高分辨率、低噪声的图像。
下面将结合实施例对本公开的实施方案进行详细描述,但是本领域技术人员将会理解,下列实施例仅用于说明本公开,而不应视为对本公开的范围的限定。实施例中未注明具体条件者,按照常规条件或制造商建议的条件进行。所用试剂或仪器未注明生产厂商者,均为可以通过市购获得的常规产品。
实施例1细胞样品(i)的制备
用经改造的基于pll3.7的慢病毒载体感染人sk-n-sh细胞。感染后,用流式细胞仪(bdfacsariasorpcellsorter)筛选egfp表达阳性的细胞。在含有10%胎牛血清的mem培养基(gibco)中,在盖玻片(fisherbrandtmcoverglassforgrowthtmcoverglasses,货号12-545-82)上培养上述egfp表达阳性的细胞(阳性细胞)或未被慢病毒感染的人sk-n-sh细胞(阴性对照)。
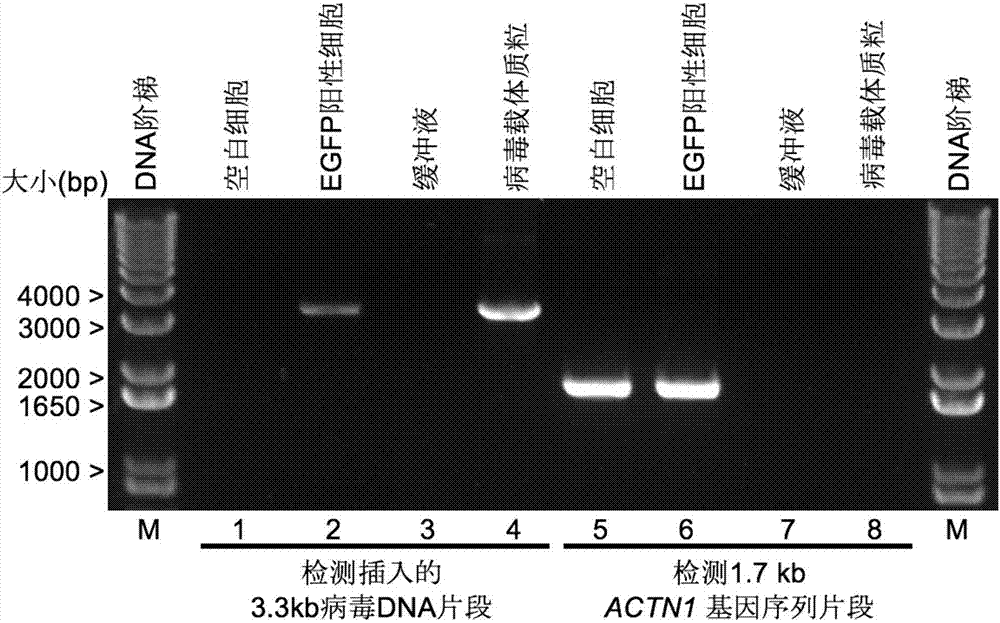
在阳性细胞中,上述慢病毒载体的一段长度约3.3kb的病毒rna片段经逆转录整合进sk-n-sh细胞的基因组。整合的3.3kb的片段(seqidno:30)包含由cmv启动子启动表达的编码egfp的序列。3.3kb的片段中的一段长度约2.5kb的片段作为示例性的靶核酸片段,在下文详述的根据本公开的识别方法中被识别(图1a)。作为识别的阳性样品的细胞基因组中包含上述靶核酸片段,并且在每个整合位点只有1个靶核酸片段的拷贝。作为识别的阴性样品的细胞不含上述靶核酸片段。通过pcr证实了阳性样品和阴性样品的上述特性(图1b)。
实施例2杂交探针mb1至mb29的设计和合成
本实施例中提供的示例性杂交探针为56nt的单链寡核苷酸,包含42nt的环结构,和环结构两侧的各7nt的臂结构。杂交探针的一端缀合作为荧光团的alexa-647,另一端缀合作为淬灭团的bhq3。
设计了29条杂交探针,记为mb1至mb29,由lifetechnology合成。这29条杂交探针的序列记载在序列表中(seqidno:1~29)。环结构的序列与实施例1中的靶核酸片段的反义链的一部分反向互补(即,与靶核酸片段的有义链的一部分相同),29条杂交探针可通过其各自的环结构沿着靶核酸的链将靶核酸片段标记。各探针中的两个臂结构的序列相互反向互补。因此,当杂交探针没有与靶核酸片段结合时,alexa-647的荧光被bhq3淬灭;当杂交探针结合于靶核酸片段时,探针的alexa-647可受激发出荧光且荧光不被bhq3影响。
实施例1中的3.3kb的靶核酸片段的反义链的序列示于图2,靶核酸片段中被杂交探针mb1至mb29靶向的序列片段用下划线显示。可见,29条杂交探针可对靶核酸片段的非重复序列进行标记。另外,29条杂交探针的序列、环结构tm值、臂结构tm值于表1。探针序列中,环结构用大写字母表示,臂结构用小写字母表示。
表1识别实施例1中的靶核酸片段的杂交探针的序列和参数
实施例3杂交探针mb1至mb29与靶核酸的特异性结合
在含有50mmnacl、1mmedta、10mmtris(ph7.4)的缓冲液中,溶解杂交探针、与杂交探针互补的寡核苷酸(cs)、以及与杂交探针序列不互补的寡核苷酸(ncs)。
在室温下将80nm杂交探针(mb1至mb29)与相应的cs(1600nm)或ncs(1600nm)共同在2×ssc、50%甲酰胺的杂交液体中反应30min。此后,使用荧光分光光度计以647nm的激光激发,在665nm读取荧光发射读数。各杂交探针的读数示于图3a。对于各杂交探针,与cs共同反应后的荧光发射读数显著高于与ncs共同反应后的荧光发射读数,表明各杂交探针与靶序列的特异性结合。
在不同的温度(42℃、38℃、34℃、30℃、26℃、22℃、18℃、14℃)下将80nm杂交探针与相应的cs(1600nm)或ncs(1600nm)共同反应30min。此后,以647nm的激光激发,在665nm读取荧光发射读数。结果示于图3b。
在现有的基于非自淬灭的线状寡核苷酸的原位杂交中,本领域技术人员公知,随着杂交温度的降低,寡核苷酸对目标序列的特异性结合和对非目标序列的非特异性结合同时增强。为了消除非特异性结合的不利影响,需要提高杂交温度,例如选择在37℃至47℃的温度下进行杂交。然而,较高的杂交温度也会对所需的特异性结合的强度带来负面影响。这是选择杂交温度面临的两难问题。
而在本公开中,如图3b所示,随着杂交温度从42℃降低至14℃,特异性结合增强;此外还发现,随着杂交温度降低,非特异性结合出乎意料地减弱。这表明在使用杂交探针的本公开中,选择杂交温度的两难问题被消除。不受任何理论限制,认为该现象可能与杂交探针的结构使得探针免于与ncs相互作用有关。
实施例4细胞样品(i)与杂交探针的接触
实施例1中的盖玻片上的细胞生长至80%汇合时,用4%多聚甲醛-pbs固定10min,浸润于pbs中2min,用1mg/ml硼氢化钠处理7min,浸润于ddh2o2min。接着细胞在25%甘油-pbs中浸泡40~50min,用液氮冷冻,解冻,重复冻融循环3次。然后在37℃下用rnasea(100μg/ml)处理细胞1hr。用pbs洗涤后,用pbs浸润细胞5分钟。细胞在75℃的2×ssc缓冲液中预热5min,然后将2×ssc缓冲液换成2×ssc缓冲液、80%去离子甲酰胺继续在75℃处理3min。紧接着细胞用冷乙醇浸润(浓度75%,90%,100%)处理各2min。用1.5%fish封闭缓冲液(roche)、50%甲酰胺和2×ssc缓冲液在室温(22℃)下将细胞封闭过夜。在与杂交探针反应3~6小时前,用新配置的1.5%fish封闭缓冲液(roche)、50%甲酰胺和2×ssc缓冲液在42℃下将细胞继续封闭至杂交前。配制总浓度为714nm实施例2中合成的杂交探针混合物(包含所有探针)、1.5%fish封闭缓冲液、50%甲酰胺和2×ssc缓冲液的混合物,用14μl该混合物与各载玻片上的细胞在22℃下杂交16~20小时。最后,在室温下用含50%甲酰胺和2×ssc的缓冲液洗涤细胞40~50min,用4%多聚甲醛-pbs固定5~10min。得到的阳性和阴性待测样品可在4℃浸润于0.25×ssc缓冲液保存。
实施例5细胞样品(i)识别条件的优化
用下述方法对实施例4中得到的样品进行识别。用配有100×油浸物镜(uplansapo,n.a.1.40,olympus)的倒置光学显微镜(ix-71,olympus),以随机光学重建显微法(storm)进行成像。对于杂交探针上标记的alexa-647,用641nm激光激发alexa647态辐射荧光,并将alexa-647转换为暗态,并用405nm激光重新活化。每次从亮态到暗态的转换记为一次开/关事件。
由于杂交探针标记的是靶核酸的非重复序列,标记密度相对比较稀疏,荧光的识别可能受到细胞自发荧光的干扰。为了优化成像条件,对激光功率、采样帧频进行调整,进行多组实验,各组的具体条件如表2所示。假阳性率(fdr)记为相同条件下从阴性样品中记录到的开/关事件数目与从阳性样品中记录到的开/关事件数目之比。各条件下的平均fdr也示于表2。
表2样品storm成像使用的条件和假阳性率
由上表可见,条件ix、x、xi、xii的平均假阳性率相对较低,是用本公开的杂交探针进行storm成像的较优条件。
进一步地,对于在条件ix、x、xi、xii下进行的实验,进一步观察记录到的开关事件数随着时间推移的变化,如图3c所示。图3c中的x轴表示时间推移,以5秒为单位;y轴表示亮态分子数目比,以开始记录起最初5秒内记录到的亮态分子数为标准,将记录过程中每5秒的时间段内记录到的亮态分子数比例化。由图3c可见,亮态分子数比在首90秒内迅速降低,并在随后的90s内降速缓慢。与条件ix、x、xi相比,当使用条件xii时,在整个成像过程中记录到的亮态分子数比例保持较高,这表明条件xii有助于降低永久性光漂白效应,有助于获得高质量的图像。
实施例6细胞样品(i)的识别
在实施例5的条件xii下,对实施例4中制备的阳性样品进行storm成像。通过对alexa-647进行亮暗转换,获得多个荧光单分子重复定位形成的团簇分布。从53个团簇累计的1378次定位分布示于图4a。重复定位满足高斯分布,在横向上半高全宽为22nm,在轴向上为52nm,表明在x-y方向上获得了约20~30nm的分辨率,在z方向上获得了约50~60nm的分辨率(图4b)。
根据非专利文献5记载的方法进行storm图像重建。图5示出3个细胞核示例性视野的图像。图5中,由上至下,栏一、二、三为常规光学显微法获得的图像,可见无法辨别出标记靶核酸的荧光点。栏四为栏一、二、三中绿色方框区域中由storm成像方法获得的超分辨彩色图像,栏五为栏四图像中白色方框区域的放大,更清楚地展现超分辨图像中靶核酸的形貌,并在右下角标注靶核酸在成像过程中收集的常态分子数。超分辨彩色图像的颜色代表了z方向上的单分子定位(-350至350nm)。图6示出图5中三个通过storm成像方法获得的靶核酸超分辨结构中红线所在区域(i、ii、iii)的强度归一化分布,展现出成像精度能分辨结构中相距44nm的微结构(i)和33-36nm的单独结构(ii、iii)。可见本公开的方法对于超分辨成像应用有特别的优势。
实施例7细胞样品(ii)的制备
用crispr/cas9基因编辑技术在小鼠cj9干细胞敲除一段约3kb的片段(mm9小鼠全基因组坐标位点,chr6:122612566–122615608,seqidno:66),经过筛选后,得到纯合子敲除细胞作为阴性对照。在mes完全培养液中,在盖玻片(fisherbrandtmcoverglassforgrowthtmcoverglasses,货号12-545-82)上培养野生型小鼠cj9干细胞(阳性细胞)或上述通过crispr/cas9技术得到的纯合子敲除细胞(阴性对照)。
在阳性细胞中,基因组中6号染色体中的一段长度约2.5kb的片段(mm9小鼠全基因组坐标位点,chr6:122612623-122615179)作为示例性的靶核酸片段(seqidno:65),在下述详细的根据本公开的识别方法中被识别。作为识别的阳性样品的细胞基因组中包含上述靶核酸片段,并且在整个基因组中只有6号染色体上有1个靶核酸片段的拷贝。作为识别的阴性样品的细胞不含上述靶核酸片段,因一段包含2.5kb的靶核酸的3.3kb核酸片段已通过crispr/cas9基因编辑技术敲除。通过pcr证实了阳性样品和阴性样品的上述特性(图7)。
实施例8杂交探针mb30至mb63的设计和合成
本实施例中提供的示例性杂交探针为56nt的单链寡核苷酸,包含42nt的环结构,和环结构两侧的各7nt的臂结构。杂交探针的一端缀合作为荧光团的alexa-647,另一端缀合作为淬灭团的bhq3。
设计了34条杂交探针,记为mb30至mb63,由lifetechnology合成。这34条杂交探针的序列记载在序列表中(seqidno:31~64)。其中,24条杂交探针(mb30、31、33、35、36、37、38、39、41、42、44、45、47、49、51、52、53、55、56、58、59、61、62、63)的环结构的序列与实施例7中的靶核酸片段的反义链的一部分反向互补(即,与靶核酸片段的有义链的一部分相同),其余10条杂交探针(mb32、34、40、43、46、48、50、54、57、60)的环结构的序列与实施例7中的靶核酸片段的有义链的一部分反向互补(即,与靶核酸片段的反义链的一部分相同)。这34条杂交探针可通过其各自的环结构沿着靶核酸的链将靶核酸片段标记。各探针中的两个臂结构的序列相互反向互补。因此,当杂交探针没有与靶核酸片段结合时,alexa-647的荧光被bhq3淬灭;当杂交探针结合于靶核酸片段时,探针的alexa-647可受激发出荧光且荧光不被bhq3影响。
实施例7中的2.5kb的靶核酸片段的有义链的序列示于图8,靶核酸片段中被杂交探针mb30至mb63靶向的序列片段以下划线或加粗显示。当中,下划线表示该杂交探针靶向靶核酸的反义链,加粗表示该杂交探针靶向靶核酸的有义链。可见,34条杂交探针可对靶核酸片段的非重复序列进行标记。这34条杂交探针的序列、环结构tm值、臂结构tm值于表3。探针序列中,环结构用大写字母表示,臂结构用小写字母表示。
表3识别实施例7中的靶核酸片段的杂交探针的序列和参数
实施例9杂交探针mb30至mb63与靶核酸的特异性结合
在含有50mmnacl、1mmedta、10mmtris(ph7.4)的缓冲液中,溶解杂交探针、与杂交探针互补的寡核苷酸(cs)、以及与杂交探针序列不互补的寡核苷酸(ncs)。
在室温下将80nm杂交探针(mb30至mb63)与相应的cs(1600nm)或ncs(1600nm)共同在2×ssc、50%甲酰胺的杂交液体中反应30min。此后,使用荧光分光光度计以647nm的激光激发,在665nm读取荧光发射读数。各杂交探针的读数示于图9a。对于各杂交探针,与cs共同反应后的荧光发射读数显著高于与ncs共同反应后的荧光发射读数,表明各杂交探针与靶序列的特异性结合。
在不同的温度(46℃、42℃、38℃、34℃、30℃、26℃、22℃、18℃、14℃)下将80nm杂交探针与相应的cs(1600nm)或ncs(1600nm)共同反应30min。此后,以647nm的激光激发,在665nm读取荧光发射读数。结果示于图9b。
在现有的基于非自淬灭的线状寡核苷酸的原位杂交中,本领域技术人员公知,随着杂交温度的降低,寡核苷酸对目标序列的特异性结合和对非目标序列的非特异性结合同时增强。为了消除非特异性结合的不利影响,需要提高杂交温度,例如选择在37℃至47℃的温度下进行杂交。然而,较高的杂交温度也会对所需的特异性结合的强度带来负面影响。这是选择杂交温度面临的两难问题。
而在本公开中,如图9b所示,随着杂交温度从46℃降低至14℃,特异性结合增强;此外还发现,随着杂交温度降低,非特异性结合出乎意料地减弱。这表明在使用杂交探针的本公开中,选择杂交温度的两难问题被消除。不受任何理论限制,认为该现象可能与杂交探针的结构使得探针免于与ncs相互作用有关。
实施例10细胞样品(ii)与杂交探针的接触
实施例7中的盖玻片上的细胞生长至80%汇合时,用4%多聚甲醛-pbs固定10min,浸润于pbs中2min,用1mg/ml硼氢化钠处理7min,浸润于ddh2o2min。接着细胞在25%甘油-pbs中浸泡40~50min,用液氮冷冻,解冻,重复冻融循环3次。然后在37℃下用rnasea(100μg/ml)处理细胞1hr。用pbs洗涤后,用pbs浸润细胞5分钟。细胞在75℃的2×ssc缓冲液中预热5min,然后将2×ssc缓冲液换成2×ssc缓冲液、80%去离子甲酰胺继续在75℃处理3min。紧接着细胞用冷乙醇浸润(浓度75%,90%,100%)处理各2min。用1.5%fish封闭缓冲液(roche)、50%甲酰胺和2×ssc缓冲液在室温(22℃)下将细胞封闭过夜。在与杂交探针反应3~6小时前,用新配置的1.5%fish封闭缓冲液(roche)、50%甲酰胺和2×ssc缓冲液在42℃下将细胞继续封闭至杂交前。配制总浓度为714nm实施例2中合成的杂交探针混合物(包含所有探针)、1.5%fish封闭缓冲液、50%甲酰胺和2×ssc缓冲液的混合物,用14μl该混合物与各载玻片上的细胞在22℃下杂交16~20小时。最后,在室温下用含50%甲酰胺和2×ssc的缓冲液洗涤细胞40~50min,用4%多聚甲醛-pbs固定5~10min。得到的阳性和阴性待测样品可在4℃浸润于0.25×ssc缓冲液保存。
实施例11细胞样品(ii)的识别
在实施例5的条件xii下,对实施例10中制备的阳性样品进行storm成像。通过对alexa-647进行亮暗转换,获得多个荧光单分子重复定位形成的团簇分布。
根据非专利文献5记载的方法进行storm图像重建。图10示出3个细胞核示例性视野的图像。图10中,由上至下,栏一、二、三为常规光学显微法获得的图像,可见无法辨别出标记靶核酸的荧光点。栏四为栏一、二、三中绿色方框区域中由storm成像方法获得的超分辨彩色图像,栏五为栏四图像中白色方框区域的放大,更清楚地展现超分辨图像中靶核酸的形貌,并在右下角标注靶核酸在成像过程中收集的常态分子数。超分辨彩色图像的颜色代表了z方向上的单分子定位(-350至350nm)。图11示出图10中三个通过storm成像方法获得的靶核酸超分辨结构中红线所在区域(i、ii、iii)的强度归一化分布,展现出成像精度能分辨结构中相距58nm(i)和37nm(ii)的微结构和25-34nm(iii)的单独结构。可见本公开的方法对于超分辨成像应用有特别的优势。
以上已经描述了本公开的各实施例,上述说明是示例性的,并非穷尽性的,并且也不限于所披露的各实施例。在不偏离所说明的各实施例的范围和精神的情况下,对于本技术领域的普通技术人员来说许多修改和变更都是显而易见的。本文中所用术语的选择,旨在最好地解释各实施例的原理、实际应用或对市场中的技术改进,或者使本技术领域的其它普通技术人员能理解本文披露的各实施例。
序列表
<110>倪燕翔
<120>核酸的精确识别方法
<130>6519-170019i
<160>66
<170>patentin版本3.5
<210>1
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb1的序列
<400>1
cggcctggcctccccgcatcgataccgtcgacctcgatcgagacctagacaggccg56
<210>2
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb2的序列
<400>2
cgtgcgctacgtcccttcggccctcaatccagcggaccttccttcccgcgcgcacg56
<210>3
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb3的序列
<400>3
cggcacgtgggcactgacaattccgtggtgttgtcggggaaatcatcgtcgtgccg56
<210>4
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb4的序列
<400>4
cggccgattgccaccacctgtcagctcctttccgggactttcgctttcctcggccg56
<210>5
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb5的序列
<400>5
cgggcagggccgcgactctagatcataatcagccatacaattcgtcgagctgcccg56
<210>6
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb6的序列
<400>6
cggcgcacgtgctgctgcccgacaaccactacctgagcacccagtccgctgcgccg56
<210>7
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb7的序列
<400>7
cgtcgggtgcagctcgccgaccactaccagcagaacacccccatcggcgcccgacg56
<210>8
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb8的序列
<400>8
cgcgggtaaggtgaacttcaagatccgccacaacatcgaggacggcagcacccgcg56
<210>9
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb9的序列
<400>9
cggtgcgccacaacgtctatatcatggccgacaagcagaagaacggcatcgcaccg56
<210>10
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb10的序列
<400>10
cgaccggcaccatcttcttcaaggacgacggcaactacaagacccgcgcccggtcg56
<210>11
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb11的序列
<400>11
cgcggctcacatgaagcagcacgacttcttcaagtccgccatgcccgaaagccgcg56
<210>12
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb12的序列
<400>12
cgctcggaccctcgtgaccaccctgacctacggcgtgcagtgcttcagcccgagcg56
<210>13
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb13的序列
<400>13
cgctggctgaccctgaagttcatctgcaccaccggcaagctgcccgtgcgccagcg56
<210>14
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb14的序列
<400>14
cggacgcgcccatcctggtcgagctggacggcgacgtaaacggccacaagcgtccg56
<210>15
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb15的序列
<400>15
cgcgcgtaccggtcgccaccatggtgagcaagggcgaggagctgttcacacgcgcg56
<210>16
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb16的序列
<400>16
cgccgtgagcttcgaattctgcagtcgacggtaccgcgggcccgggatccacggcg56
<210>17
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb17的序列
<400>17
cgcgtcggtgaaccgtcagatccgctagcgctaccggactcagatctcgcgacgcg56
<210>18
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb18的序列
<400>18
cgcggtcgcaaatgggcggtaggcgtgtacggtgggaggtctatataaggaccgcg56
<210>19
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb19的序列
<400>19
cgagggctcaacgggactttccaaaatgtcgtaacaactccgccccattgccctcg56
<210>20
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb20的序列
<400>20
cgggcgtcaagtctccaccccattgacgtcaatgggagtttgttttggcacgcccg56
<210>21
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb21的序列
<400>21
cgtcggcggcagtacatcaatgggcgtggatagcggtttgactcacggggccgacg56
<210>22
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb22的序列
<400>22
cgggtccgggactttcctacttggcagtacatctacgtattagtcatcgggacccg56
<210>23
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb23的序列
<400>23
cggcagcccacttggcagtacatcaagtgtatcatatgccaagtacgccgctgccg56
<210>24
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb24的序列
<400>24
cgctggggacgtatgttcccatagtaacgccaatagggactttccattgcccagcg56
<210>25
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb25的序列
<400>25
cggtgcctaacttacggtaaatggcccgcctggctgaccgcccaacgacggcaccg56
<210>26
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb26的序列
<400>26
cgtggcgttacggggtcattagttcatagcccatatatggagttccgcgcgccacg56
<210>27
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb27的序列
<400>27
cgagccgattacagggacagcagagatccagtttggttagtaccgggcccggctcg56
<210>28
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb28的序列
<400>28
cgtcgcgattagtgaacggatcggcactgcgtgcgccaattctgcagaccgcgacg56
<210>29
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb29的序列
<400>29
cgcgcagcagggatattcaccattatcgtttcagacccacctcccaaccctgcgcg56
<210>30
<211>3243
<212>dna
<213>人工序列
<220>
<223>细胞样品(i)中插入的靶核酸片段
<400>30
tctaggtctcgatcgaggtcgacggtatcgatgcggggaggcggcccaaagggagatccg60
actcgtctgagggcgaaggcgaagacgcggaagaggccgcagagccggcagcaggccgcg120
ggaaggaaggtccgctggattgagggccgaagggacgtagcagaaggacgtcccgcgcag180
aatccaggtggcaacacaggcgagcagccaaggaaaggacgatgatttccccgacaacac240
cacggaattgtcagtgcccaacagccgagcccctgtccagcagcgggcaaggcaggcggc300
gatgagttccgccgtggcaatagggagggggaaagcgaaagtcccggaaaggagctgaca360
ggtggtggcaatgccccaaccagtgggggttgcgtcagcaaacacagtgcacaccacgcc420
acgttgcctgacaacgggccacaactcctcataaagagacagcaaccaggatttatacaa480
ggaggagaaaatgaaagccatacgggaagcaatagcatgatacaaaggcattaaagcagc540
gtatccacatagcgtaaaaggagcaacatagttaagaataccagtcaatctttcacaaat600
tttgtaatccagaggttgattatcgataagcttgatatcgaattgtacctagtggaaccg660
gaacccttaaacatgtataacttcgtataatgtatgctatacgaagttattaggtccctc720
gacgaattgtatggctgattatgatctagagtcgcggccgctttacttgtacagctcgtc780
catgccgagagtgatcccggcggcggtcacgaactccagcaggaccatgtgatcgcgctt840
ctcgttggggtctttgctcagggcggactgggtgctcaggtagtggttgtcgggcagcag900
cacggggccgtcgccgatgggggtgttctgctggtagtggtcggcgagctgcacgctgcc960
gtcctcgatgttgtggcggatcttgaagttcaccttgatgccgttcttctgcttgtcggc1020
catgatatagacgttgtggctgttgtagttgtactccagcttgtgccccaggatgttgcc1080
gtcctccttgaagtcgatgcccttcagctcgatgcggttcaccagggtgtcgccctcgaa1140
cttcacctcggcgcgggtcttgtagttgccgtcgtccttgaagaagatggtgcgctcctg1200
gacgtagccttcgggcatggcggacttgaagaagtcgtgctgcttcatgtggtcggggta1260
gcggctgaagcactgcacgccgtaggtcagggtggtcacgagggtgggccagggcacggg1320
cagcttgccggtggtgcagatgaacttcagggtcagcttgccgtaggtggcatcgccctc1380
gccctcgccggacacgctgaacttgtggccgtttacgtcgccgtccagctcgaccaggat1440
gggcaccaccccggtgaacagctcctcgcccttgctcaccatggtggcgaccggtggatc1500
ccgggcccgcggtaccgtcgactgcagaattcgaagcttgagctcgagatctgagtccgg1560
tagcgctagcggatctgacggttcactaaaccagctctgcttatatagacctcccaccgt1620
acacgcctaccgcccatttgcgtcaatggggcggagttgttacgacattttggaaagtcc1680
cgttgattttggtgccaaaacaaactcccattgacgtcaatggggtggagacttggaaat1740
ccccgtgagtcaaaccgctatccacgcccattgatgtactgccaaaaccgcatcaccatg1800
gtaatagcgatgactaatacgtagatgtactgccaagtaggaaagtcccataaggtcatg1860
tactgggcataatgccaggcgggccatttaccgtcattgacgtcaatagggggcgtactt1920
ggcatatgatacacttgatgtactgccaagtgggcagtttaccgtaaatactccacccat1980
tgacgtcaatggaaagtccctattggcgttactatgggaacatacgtcattattgacgtc2040
aatgggcgggggtcgttgggcggtcagccaggcgggccatttaccgtaagttatgtaacg2100
cggaactccatatatgggctatgaactaatgaccccgtaattgattactattaataacta2160
actagagcgggcccggtactaaccaaactggatctctgctgtccctgtaataaacccgaa2220
aattttgaatttttgtaatttgtttttgtaattctttagtttgtatgtctgttgctatta2280
tgtctactattctttcccctgcactgtaccccccaatccccccttttcttttaaaattgt2340
ggatgaatactgccatttgtctgcagaattggcgcacgcagtgccgatccgttcactaat2400
cgaatggatctgtctctgtctctctctccaccttcttcttctattccttcgggcctgtcg2460
ggtcccctcggggttgggaggtgggtctgaaacgataatggtgaatatccctgcctaact2520
ctattcactatagaaagtacagcaaaaactattcttaaacctaccaagcctcctactatc2580
attatgaataattttatataccacagccaatttgttatgttaaaccaattccacaaactt2640
gcccatttatctaattccaataattcttgttcattcttttcttgctggttttgcgattct2700
tcaattaaggagtgtattaagcttgtgtaattgttaatttctctgtcccactccatccag2760
gtcgtgtgattccaaatctgttccagagatttattactccaactagcattccaaggcaca2820
gcagtggtgcaaatgagttttccagagcaaccccaaatccccaggagctgttgatccttt2880
aggtatctttccacagccaggattcttgcctggagctgcttgatgccccagactgtgagt2940
tgcaacagatgctgttgcgcctcaatagccctcagcaaattgttctgctgctgcactata3000
ccagacaataattgtctggcctgtaccgtcagcgtcattgacgctgcgcccatagtgctt3060
cctgctgctcccaagaacccaaggaacaaagctcctattcccactgctcttttttctctc3120
tgcaccactcttctctttgccttggtgggtgctactcctaatggttcaatttttactact3180
ttatatttatataattcacttctccaattgtccctcatatctcctcctccaggtctgaag3240
atc3243
<210>31
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb30的序列
<400>31
cgcctgggtttaaacttgtaaaactccagtcgtgggctaaactgtctggccaggcg56
<210>32
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb31的序列
<400>32
cgctcggctactgtgttcacggacaggtgccagcccagtgacccagagcccgagcg56
<210>33
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb32的序列
<400>33
cgccggtcccagggcagtgctaggtttcgggagacaactgaaagggaataccggcg56
<210>34
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb33的序列
<400>34
cggcgtgcctggagcccatctcagtctgccagcgattcttttcaaacaacacgccg56
<210>35
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb34的序列
<400>35
cgggctgtcttgtagcaactcatagatctgcaaatacatgaacctgtggcagcccg56
<210>36
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb35的序列
<400>36
cgggtcggccgctccctggatagcgatgaatccttcagcctactgtccacgacccg56
<210>37
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb36的序列
<400>37
cggctgcaggaggccaagcgccttgtccgctccgcaccacccacccatcgcagccg56
<210>38
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb37的序列
<400>38
cgtccgggcgggtggtgataaaccggtgcacctcgcggcctcccgaattccggacg56
<210>39
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb38的序列
<400>39
cggctcgcaggaggtcccaactgtccttactcacagttcattccaagcccgagccg56
<210>40
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb39的序列
<400>40
cggtgcggaaaaggccttcctagcccaagggtgggaatgttaaggctggcgcaccg56
<210>41
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb40的序列
<400>41
cggtggcgacctaggagagcccagtcacccatctgtgtgaggtttcccagccaccg56
<210>42
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb41的序列
<400>42
cgtgcgcctctgacacctctgggtcttgcaaaccgggtgctccacatatgcgcacg56
<210>43
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb42的序列
<400>43
cgtcgggatagatttggaattaaccgtacagctggcatggcagtgttggcccgacg56
<210>44
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb43的序列
<400>44
cgtgccgtctgaccttcctcttgctaatccaccttgaagatctcacatccggcacg56
<210>45
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb44的序列
<400>45
cgtcgcggacctttcagcaagcaacccttcaactgtgagttgctagttacgcgacg56
<210>46
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb45的序列
<400>46
cgcgcgtaagggcaaactggcgacagtaaacataattagctgacccttcacgcgcg56
<210>47
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb46的序列
<400>47
cgtggcgggcctttgagatagcagattgccgacggactttatttcctttcgccacg56
<210>48
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb47的序列
<400>48
cgcgggtgccagccttaactcctgcaaagttaagcccacagtacttgaaacccgcg56
<210>49
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb48的序列
<400>49
cggtcgccgtgctccgtctcctcgagccaacaaaggaagaaataattaagcgaccg56
<210>50
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb49的序列
<400>50
cgccgtgccagcaggacaggtgtcatgtcctcctcagcaaaagcttcaacacggcg56
<210>51
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb50的序列
<400>51
cgtgggcagagcccgggaagatggaaaaccagtttcctccggtctctgcgcccacg56
<210>52
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb51的序列
<400>52
cggtcggcagccttgtgcttcgcgtgtgctgttggagcgatttcgttaaccgaccg56
<210>53
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb52的序列
<400>53
cgtgcggccgatcttatgtcacctcacctgaggacccagtttgatcattccgcacg56
<210>54
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb53的序列
<400>54
cgctgcggaatagtacttttcaccatgggtaaacttgaacccaggtctgcgcagcg56
<210>55
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb54的序列
<400>55
cgcggtgggctgtacagggcagtgaagagccgtgaggctgagcatggagcaccgcg56
<210>56
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb55的序列
<400>56
cgctgggccacacatctgcacccttgccttgtaaacatctggcataagacccagcg56
<210>57
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb56的序列
<400>57
cgtcggctgaagggacagacacttaaagggtcttactgcccagatacttgccgacg56
<210>58
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb57的序列
<400>58
cggtgccgggaagtcactctgcagttcattcctggtgataggatggttgggcaccg56
<210>59
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb58的序列
<400>59
cgggtgcaacaggatagatattggtgactgaggttggaagacaggcctggcacccg56
<210>60
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb59的序列
<400>60
cgggctccatgtcttctgtggtgtccaattctgttctcatgcatttgcagagcccg56
<210>61
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb60的序列
<400>61
cggcgtcggtaaatggtacccaggcccttccggccatctcagctctctggacgccg56
<210>62
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb61的序列
<400>62
cgcggtctctgaggccggaaggagacatcgcaagcatcttagagtgcaggaccgcg56
<210>63
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb62的序列
<400>63
cgggtccaacttccatattgatttcacaattcttctgggagtagctctcggacccg56
<210>64
<211>56
<212>dna
<213>人工序列
<220>
<223>杂交探针mb63的序列
<400>64
cgctggcattctctctgcttatccaattatcctcaggtatgaaagaggagccagcg56
<210>65
<211>2556
<212>dna
<213>小鼠(musmusculus)
<400>65
gtttaaacttgtaaaactccagtcgtgggctaaactgtctggagcaggttctgtctctta60
ctgtccaagatttctcaatgctcgtgggtctggaagaaagagttctgcctactgtgttca120
cggacaggtgccagcccagtgacccagagcaatgtaggcccattccctttcagttgtctc180
ccgaaacctagcactgccctggggctgcctgctcagcaccctcacctctctcccctggag240
cccatctcagtctgccagcgattcttttcaaacaaaggtttctactgtggtttgaatgtg300
aaacgtccccccacaggttcatgtatttgcagatctatgagttgctacaagaaggctggc360
cgctccctggatagcgatgaatccttcagcctactgtccaggaagtgaaccggcttcaga420
ctgagaccaaagtgccactcccaaggtgaactgggcaggaggccaagcgccttgtccgct480
ccgcaccacccacccatcccccttgcagctgggcctctttgatgctgctgctgagagtct540
atagcgggtggtgataaaccggtgcacctcgcggcctcccgaattcagcttctggagaca600
atccactcgctagaaacaagggcgctttccccaggaggtcccaactgtccttactcacag660
ttcattccaagccctaacacagccctaataaaccagtctgctcttcacctcccagaagag720
aaaaggccttcctagcccaagggtgggaatgttaaggctgggggataacagcgcatttgc780
tgtggagtactgttcctaaaaagccgtgggaaacctcacacagatgggtgactgggctct840
cctaggtcccctctgctggctttttttcctctgacacctctgggtcttgcaaaccgggtg900
ctccacatataccggggatccgctgtgacacccccaccaccaccacctgtgaccgtgtta960
gtgactgaatttaccttccatagatttggaattaaccgtacagctggcatggcagtgttg1020
gggcagacaaggagtcggggagatgtgagatcttcaaggtggattagcaagaggaaggtc1080
agactttcaaactgagacctttcagcaagcaacccttcaactgtgagttgctagttagtt1140
aaaaatgaacccaaagggcaactggggccttaaagtggccacaaagctaaagggcaaact1200
ggcgacagtaaacataattagctgacccttcctggcccaaagctaacacaaacttaaagg1260
aaataaagtccgtcggcaatctgctatctcaaaggcccctcctgagttggggccagcctt1320
aactcctgcaaagttaagcccacagtacttgaatgcagttaattatttcttcctttgttg1380
gctcgaggagacggagcacgtttttgacccccaccctcacccccaggctcacatgagccc1440
cagcaggacaggtgtcatgtcctcctcagcaaaagcttcaatgcagagaccggaggaaac1500
tggttttccatcttcccgggctctgcagccttgtgcttcgcgtgtgctgttggagcgatt1560
tcgttaaaagcccctttagcctttcttctggactctttgggttttttttgtctgagaagg1620
tgctgcgggggggggggggccctccccgatcttatgtcacctcacctgaggacccagttt1680
gatcattcagtaaggatagtacttttcaaaaaaaagaaagaaagaaagaaagaaagaaag1740
aaagaaagaaagaaagaaagaaagaaagaaagaaagaaagaaaagaatagtacttttcac1800
catgggtaaacttgaacccaggtctgtctccatgctcagcctcacggctcttcactgccc1860
tgtacagcctacaccccagcccgcccacacatctgcacccttgccttgtaaacatctggc1920
ataagaacatcaaatgtttcataaacttgagtccagctacagcatctcaaccttgaaggg1980
acagacacttaaagggtcttactgcccagatacttttaaaaaattgaggcttgcaaccat2040
cctatcaccaggaatgaactgcagagtgacttcccattttccaagagtggcaacaggata2100
gatattggtgactgaggttggaagacaggcctgatttcctcatctgaccaggtaagcact2160
cagacagaggtgtgacttacagaaccccatgtcttctgtggtgtccaattctgttctcat2220
gcatttgcaaatgtgaatgtgaggagccagtgcaggaggagcaggcagagagctgagatg2280
gccggaagggcctgggtaccatttacccaggcacataagtctgaggccggaaggagacat2340
cgcaagcatcttagagtgcagtcctttggtcttgaatttctcacttctctgggactgtat2400
cctttttccgaacttccatattgatttcacaattcttctgggagtagctctctacttctc2460
ccaaggtcatctatccctgagtgcccactttctcacttctctgccctctaccgaattctc2520
tctgcttatccaattatcctcaggtatgaaagagga2556
<210>66
<211>3043
<212>dna
<213>小鼠(musmusculus)
<400>66
cagcacgtgcgccggcacacacagacacacccacaacaataataatcaatatatattagt60
ttaaacttgtaaaactccagtcgtgggctaaactgtctggagcaggttctgtctcttact120
gtccaagatttctcaatgctcgtgggtctggaagaaagagttctgcctactgtgttcacg180
gacaggtgccagcccagtgacccagagcaatgtaggcccattccctttcagttgtctccc240
gaaacctagcactgccctggggctgcctgctcagcaccctcacctctctcccctggagcc300
catctcagtctgccagcgattcttttcaaacaaaggtttctactgtggtttgaatgtgaa360
acgtccccccacaggttcatgtatttgcagatctatgagttgctacaagaaggctggccg420
ctccctggatagcgatgaatccttcagcctactgtccaggaagtgaaccggcttcagact480
gagaccaaagtgccactcccaaggtgaactgggcaggaggccaagcgccttgtccgctcc540
gcaccacccacccatcccccttgcagctgggcctctttgatgctgctgctgagagtctat600
agcgggtggtgataaaccggtgcacctcgcggcctcccgaattcagcttctggagacaat660
ccactcgctagaaacaagggcgctttccccaggaggtcccaactgtccttactcacagtt720
cattccaagccctaacacagccctaataaaccagtctgctcttcacctcccagaagagaa780
aaggccttcctagcccaagggtgggaatgttaaggctgggggataacagcgcatttgctg840
tggagtactgttcctaaaaagccgtgggaaacctcacacagatgggtgactgggctctcc900
taggtcccctctgctggctttttttcctctgacacctctgggtcttgcaaaccgggtgct960
ccacatataccggggatccgctgtgacacccccaccaccaccacctgtgaccgtgttagt1020
gactgaatttaccttccatagatttggaattaaccgtacagctggcatggcagtgttggg1080
gcagacaaggagtcggggagatgtgagatcttcaaggtggattagcaagaggaaggtcag1140
actttcaaactgagacctttcagcaagcaacccttcaactgtgagttgctagttagttaa1200
aaatgaacccaaagggcaactggggccttaaagtggccacaaagctaaagggcaaactgg1260
cgacagtaaacataattagctgacccttcctggcccaaagctaacacaaacttaaaggaa1320
ataaagtccgtcggcaatctgctatctcaaaggcccctcctgagttggggccagccttaa1380
ctcctgcaaagttaagcccacagtacttgaatgcagttaattatttcttcctttgttggc1440
tcgaggagacggagcacgtttttgacccccaccctcacccccaggctcacatgagcccca1500
gcaggacaggtgtcatgtcctcctcagcaaaagcttcaatgcagagaccggaggaaactg1560
gttttccatcttcccgggctctgcagccttgtgcttcgcgtgtgctgttggagcgatttc1620
gttaaaagcccctttagcctttcttctggactctttgggttttttttgtctgagaaggtg1680
ctgcgggggggggggggccctccccgatcttatgtcacctcacctgaggacccagtttga1740
tcattcagtaaggatagtacttttcaaaaaaaagaaagaaagaaagaaagaaagaaagaa1800
agaaagaaagaaagaaagaaagaaagaaagaaagaaagaaaagaatagtacttttcacca1860
tgggtaaacttgaacccaggtctgtctccatgctcagcctcacggctcttcactgccctg1920
tacagcctacaccccagcccgcccacacatctgcacccttgccttgtaaacatctggcat1980
aagaacatcaaatgtttcataaacttgagtccagctacagcatctcaaccttgaagggac2040
agacacttaaagggtcttactgcccagatacttttaaaaaattgaggcttgcaaccatcc2100
tatcaccaggaatgaactgcagagtgacttcccattttccaagagtggcaacaggataga2160
tattggtgactgaggttggaagacaggcctgatttcctcatctgaccaggtaagcactca2220
gacagaggtgtgacttacagaaccccatgtcttctgtggtgtccaattctgttctcatgc2280
atttgcaaatgtgaatgtgaggagccagtgcaggaggagcaggcagagagctgagatggc2340
cggaagggcctgggtaccatttacccaggcacataagtctgaggccggaaggagacatcg2400
caagcatcttagagtgcagtcctttggtcttgaatttctcacttctctgggactgtatcc2460
tttttccgaacttccatattgatttcacaattcttctgggagtagctctctacttctccc2520
aaggtcatctatccctgagtgcccactttctcacttctctgccctctaccgaattctctc2580
tgcttatccaattatcctcaggtatgaaagaggaataaaaataaataattttaaaaagac2640
atgaaaaagtgtgtggctggctataggtcaactattcatttctttttaaaagatttattt2700
gcagccgggcgtggtggcatacacctttaatcccaacactcgggaggcagaggcaggcag2760
atttctgagttcgaggccagcctggtctacaaagtgagttccaggatagccagagctaca2820
cagagaaaccctgactcaaaaaaacaaaaacaaacaaaaaaaagagagagggttttcatt2880
tttgttttgaaattatgatataattacaactttttccatttcctccaaactctcccatat2940
accccacctttctaccttttaaattcatggcctcttttaaaataactgttattacacgca3000
tattatattcttaaatatgaccttctcggtctgtatgatgtca3043
- 还没有人留言评论。精彩留言会获得点赞!