在细胞内产生点突变的融合蛋白、其制备及用途的制作方法
本发明涉及在细胞内产生点突变的融合蛋白、其制备及用途。
背景技术:
基因型与表型间存在密切关系。自然界中,自发突变会引起基因型的改变,从而产生多种表型。实验室中,仍然通过突变,使基因多样化,产生多种表型,从而筛选出功能突变体,研究基因与功能的相关,获得功能更强的蛋白质。自然界中,自发突变频率极低。常见生物中,人类基因组的自发突变率为5.0×10-10,小鼠基因组自发突变率为1.8×10-10,大肠杆菌基因组的自发突变率为5.4×10-10,hiv的自发突变率为3×10-5,随着生物基因组的减小,生物体的自发突变频率增高〔holmesec.thecomparativegenomicsofviralemergence[j].proceedingsofthenationalacademyofsciences,2010,107(4):1742-1746〕。但这种低水平的基因突变频率不能产生足够数量的表型,用以研究基因、表型与功能的关系。
为了提高基因突变频率,实验室现有手段主要分体内突变方法和体外突变方法。体内点突变方法:1.物理方法:紫外辐射,突变频率为1×10-10〔packerms,liudr.methodsforthedirectedevolutionofproteins[j].naturereviewsgenetics,2015〕。2.化学方法:enu是一种烷化剂,将乙基转移到dna的氧和氮原子上,引起错配,碱基置换或者缺失,突变频率为1-1.5×10-5〔filby.zebrafish:methodsandprotocols.methodsinmolecularbiology‐byg.j.lieschke,a.coatesandk.kawakami.[j].journaloffishbiology,2010,76(7):1874-1876〕。虽然enu易于获得,但它对光、热、ph都很敏感,限制了它的应用。这两种方法均可以通过剂量改变其突变频率,但引起的点突变是随机的,突变频率低,突变图谱不均一,对生物体有害〔guénetjl.chemicalmutagenesisofthemousegenome:anoverview[j].genetica,2004,122(1):9-24〕。3.生物方法:转座子,染色体dna上可自主复制和位移的基本单位,可引起插入突变,可以通过基因的插入导致基因敲除,基因激活,并可以通过选择不同载体来选择不同的插入位点,但其突变亲率比enu低,在每一细胞周期中,只能发生3×10-5插入事件,并且需要host同时表达转座酶来完成转座〔kitadak,ishishitas,tosakak,etal.transposon-taggedmutagenesisintherat.[j].naturemethods,2007,4(2):131-133〕。
而在免疫系统,生发中心的b细胞,可以通过体细胞高频突变产生多样性抗体,抵抗病原的入侵〔odegardvh,schatzdg.targetingofsomatichypermutation.[j].naturereviewsimmunology,2006,6(8):573-583〕。体细胞高频突变指的是免疫球蛋白重轻链可变区的非模板点突变,与b细胞亲和成熟有关〔odegardvh等,同前〕。而介导这一过程重要的酶是激活诱导的胞嘧啶脱氨酶(activationinducedcytosinedeaminase,aid)。aid是一种胞嘧啶脱氨酶,属于apobec家族,一种rna编辑酶家族:n端有核定位信号,c端有核输出信号,其催化结构域为apobec家族所共有〔zhenmingx,hongz,poneej,etal.immunoglobulinclass-switchdnarecombination:induction,targetingandbeyond.[j].naturereviewsimmunology,2012,12(7):517-31〕。一般认为n端结构为shm所必须。aid的表达局限于生发中心的b细胞,其发挥点突变功能是有条件的,必须作用于单链的dna,并且具有序列偏好性,hotspot结构域为rgyw〔kiyotsuguy,il-mio,tomonorie,etal.aidenzyme-inducedhypermutationinanactivelytranscribedgeneinfibroblasts[j].science,2002,296(5575):2033-2036〕。r代表a/g,y代表c/t,w代表a/t,可见aid发挥功能与dna的一级结构有关。首先将单链dna上的胞嘧啶脱氨基变为u,形成u-g错配,如果u-g未修复,在dna复制过程中会形成c-tg-a的转换突变。此外,u可被ung(尿嘧啶dna糖苷酶)切除,形成无嘧啶位点,将四种碱基随机参入〔odegardvh等,同前〕。以上过程产生的点突变对于体细胞高频突变意义重大,可以产生多样性的抗体。但其在体内引起的点突变频率为1×10-4-1×10-3,且位点具有随机性〔masatoshia,nesreenh,andres,etal.accumulationofthefactcomplex,aswellashistoneh3.3,servesasatargetmarkerforsomatichypermutation.[j].proceedingsofthenationalacademyofsciencesoftheunitedstatesofamerica,2013,110(19):7784-7789〕,仍无法满足实验筛选突变体所需。
技术实现要素:
本文第一方面提供一种融合蛋白,所述融合蛋白含有胞嘧啶脱氨酶和核酸酶活性缺失、保留了解旋酶活性的cas酶。
在一个或多个实施方案中,所述融合蛋白由胞嘧啶脱氨酶和核酸酶活性缺失、保留了解旋酶活性的cas酶形成。
在一个或多个实施方案中,所述cas酶选自:cas1、cas1b、cas2、cas3、cas4、cas5、cas6、cas7、cas8、cas9(也称为csn1和csx12)、cas10、csy1、csy2、csy3、cse1、cse2、csc1、csc2、csa5、csn2、csm2、csm3、csm4、csm5、csm6、cmr1、cmr3、cmr4、cmr5、cmr6、csb1、csb2、csb3、csx17、csx14、csx10、csx16、csax、csx3、csx1、csx15、csf1、csf2、csf3、csf4、其同源物或其修饰形式。
在一个或多个实施方案中,所述cas酶的核酸酶活性部分缺失,使得所述cas酶仅能造成dna单链断裂;或所述cas酶的核酸酶活性全部缺失,能引起dna双链断裂。
在一个或多个实施方案中,所述cas酶为cas9酶,选自:来自化脓链球菌的cas9(spcas9)、来自金黄色葡萄球菌的cas9(sacas9),以及来自嗜热链球菌的cas9(st1cas9)。
在一个或多个实施方案中,所述cas酶为cas9酶,该酶的两个核酸内切酶催化结构域ruvc1和/或hnh发生突变,导致该酶核酸酶活性缺失、保留了解旋酶活性。
在一个或多个实施方案中,所述cas9酶的ruvc1和hnh都发生突变,导致该酶核酸酶活性缺失、保留了解旋酶活。
在一个或多个实施方案中,所述cas9酶的第10个氨基酸天冬酰胺突变为丙氨酸或其它氨基酸,第841位氨基酸组氨酸突变为丙氨酸或其它氨基酸。
在一个或多个实施方案中,所述cas9酶的氨基酸序列如seqidno:2第42-1452所示,或如seqidno:72第42-1419位氨基酸残基所示。
在一个或多个实施方案中,所述胞嘧啶脱氨酶为全长胞嘧啶脱氨酶或其片段,其中所述片段至少包括胞嘧啶脱氨酶的nls结构域、催化结构域和apobec样结构域。
在一个或多个实施方案中,所述胞嘧啶脱氨酶在第10位、82位和156位氨基酸残基发生取代突变。
在一个或多个实施方案中,所述取代突变为k10e、t82i和e156g。
在一个或多个实施方案中,所述片段至少包含aid的第9-182位氨基酸残基,例如至少包含aid第1-182位氨基酸残基。
在一个或多个实施方案中,所述胞嘧啶脱氨酶的氨基酸序列如seqidno:2第1457-1654位氨基酸所示,或如seqidno:68第1447-1629位氨基酸残基所示。
在一个或多个实施方案中,所述片段至少包含seqidno:2的第1465-1638位氨基酸残基,例如至少包含seqidno:2第1457-1638位氨基酸残基。
在一个或多个实施方案中,所述片段由第1-182位氨基酸残基组成,由第1-186位氨基酸残基组成,或由第1-190位氨基酸残基组成。
在一个或多个实施方案中,所述融合蛋白还包含以下序列中的一种或多种:接头,核定位序列,以及为了构建融合蛋白、促进重组蛋白的表达、获得自动分泌到宿主细胞外的重组蛋白、或利于重组蛋白的纯化而引入的氨基酸残基或氨基酸序列。
在一个或多个实施方案中,所述融合蛋白的氨基酸序列如seqidno:2、4、66、68、70或72所示,或如seqidno:2第26-1654位氨基酸所示,或如seqidno:4第26-1638位所示,或如seqidno:68第26-1629位氨基酸所示,或如seqidno:70第26-1629位氨基酸所示,或如seqidno:72第26-1638位氨基酸所示。
本文第二方面提供一种多核苷酸序列,选自:
(1)编码本文第一方面所述的融合蛋白的多核苷酸序列;和
(2)(1)所述序列的互补序列。
本发明第三方面提供核酸构建物,所述核酸构建物含有本文第二方面所述的多核苷酸序列。
在一个或多个实施方案中,所述核酸构建物是表达载体,用于在宿主细胞中表达本文所述的融合蛋白。
本发明第四方面提供一种宿主细胞,所述宿主细胞含有本文所述的融合蛋白、其编码序列或核酸构建物。
本文第五方面提供一种在细胞内产生点突变的方法,所述方法包括在所述细胞中表达本文所述的融合蛋白和sgrna的步骤。
在一个或多个实施方案中,所述方法包括将本文所述的融合蛋白或其表达载体和sgrna或其表达载体转入所述细胞内,然后筛选获得所需要的突变核酸序列的步骤。
在一个或多个实施方案中,所述sgrna包括靶标结合区和cas蛋白识别区,所述靶标结合区能特异性结合待突变的核酸序列,所述cas蛋白识别区能被所述融合蛋白中的cas酶识别并结合。
在一个或多个实施方案中,所述sgrna的靶标结合区与待突变的核酸序列的模板链特异性结合,模板链上sgrna结合区域的对侧区紧邻该cas蛋白所识别的前间区序列邻近基序,或隔开10个以内的碱基。
在一个或多个实施方案中,所述待突变的基因编码功能蛋白。
在一个或多个实施方案中,所述功能蛋白包括疾病的发生、发展和转移中涉及的蛋白,细胞分化、增殖与凋亡中涉及的蛋白,参与新陈代谢的蛋白,发育相关的蛋白,以及各种药物靶点等等。
在一个或多个实施方案中,所述功能蛋白选自:抗体、酶、脂蛋白、激素类蛋白、运输和贮存蛋白、运动蛋白、受体蛋白、和膜蛋白。
本发明第六方面提供一种试剂盒,所述试剂盒含有本文所述的融合蛋白、多核苷酸序列或核酸构建物。
本发明第七方面提供本文所述的融合蛋白、多核苷酸序列或核酸构建物在在细胞内产生点突变中的应用,或在制备用于在细胞内产生点突变的组合物或试剂盒中的应用。
附图说明
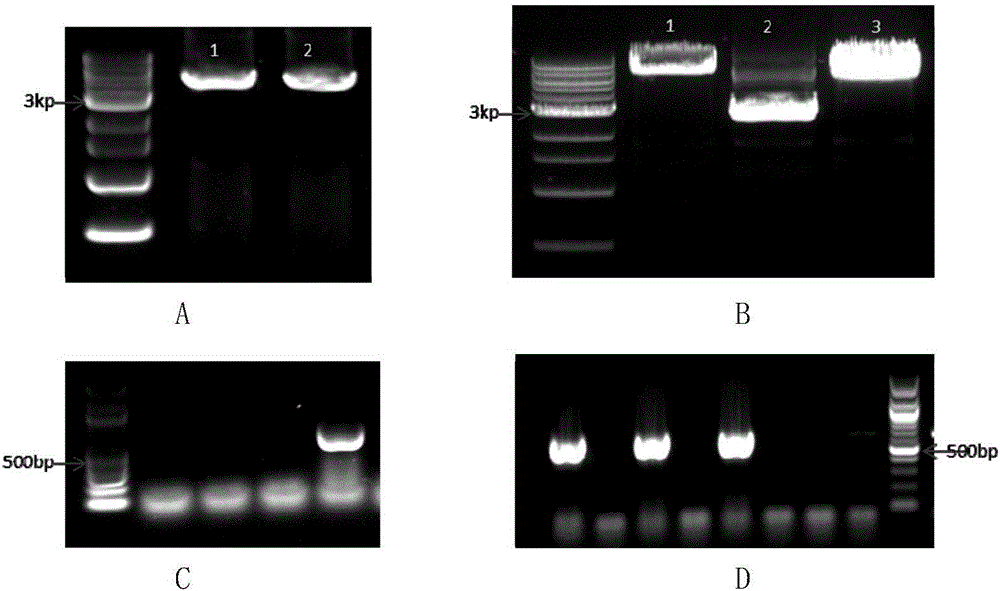
图1:a和c分别为pcr扩增出的aid(泳道1)及aidx片段(泳道1);b为pentr11-dcas9-aid质粒琼脂糖凝胶图,其中1道为pentr11空载质粒,2道为pentr11-dcas9质粒,3-7道为pentr11-dcas9-aid质粒;d为pentr11-dcas9-aidx质粒菌液pcr结果,扩增出的片段是aidx。d中1-5泳道分别代表5个不同的阳性克隆,6号是空载质粒,作为阴性对照。
图2:a,1道和2道分别为pcr扩增出的dcas9-aid及dcas9-aidx片段;b,酶切mo91空载质粒,其中1道为bglⅱ单酶切,2道为mo91空载质粒,3道为bglⅱ和xhoⅰ双酶切;c,mo91-dcas9-aidx质粒菌液pcr结果,扩增出的片段是aidx;d,mo91-dcas9-aid质粒菌液pcr结果,扩增出的片段是aid。
图3:a,1道为pcr扩增出的3*flag+nls片段,2道及3道分别为bglⅱ单酶切mo91-dcas9-aid质粒和mo91-dcas9-aidx质粒,4道为mo91-dcas9-aid质粒对照;b,1-4道为mo91-dcas9(3*flag,nls)-aid质粒,5道为mo91-dcas9-aid质粒,6-9道为mo91-dcas9(3*flag,nls)-aidx质粒。
图4:egfp报告子的序列,终止密码子以粗体表示。设计的sgrna用箭头表示。
图5:报告质粒的模式示意图。
图6:流式检测报告细胞系。三条曲线从左到右分别表示未染色对照、报告子阴性细胞和报告子阳性细胞的thy1.1表达水平。
图7:dcas9-aid,dcas9-aidx,aid和aidx点突变效率在报告细胞中的比较。
图8:dcas9-aid点突变效率在报告细胞中的优化。a,dcas9-aid诱导gfp表达;b,不同aid变体的示意图以及其诱导点突变的效率;c,dcas9-aidx诱导点突变需要aid的胞嘧啶脱氨酶活性。
图9:dcas9-aidx和aid对egfp和cmyc基因造成的点突变频率分布。
图10:dcas9-aidx将c和g碱基随机突变为其他三种碱基。a,碱基突变类型的统计;b,dcas9-aidx诱导点突变的机制。
图11:ugi提高dcas9-aidx系统的碱基置换频率,揭示dcas9-aidx在基因上的作用轨迹,并使碱基突变方向更加单一化。
图12:dcas9-aidx不仅可以对外源性基因起作用,同时可以作用于内源性基因。
图13:aid的结构功能域。
图14:将dcas9-aidx应用于k562bcr-abl基因的gleevec耐药性筛选的实验过程(a)及结果(b-d)。
图15:tam(靶向胞嘧啶脱氨酶aid介导基因突变技术)突变抗hel-igg1可变区的氨基酸。
图16:tam诱导抗hel-igg1可变区的碱基突变(上图),且可重复地诱导igg1cdr的碱基突变(下图)。
图17:突变后的抗体对hel的亲和力增强了10倍以上。
图18:ncas9-aidx在细菌中的表达结果。方框框出的条带为ncas9-aidx融合蛋白的条带。
图19:不同融合蛋白的功能测试结果。对每一组数据,从左到右三根柱子依次代表mo91-aidx-xten-dcas9、mo91-dcas9-xten-aidx和mo91-dcas9-aidx的结果。
图20:不同融合蛋白的功能测试结果。对每一组数据,从左到右三根柱子依次代表mo91-dcas9-aidx、mo91-dcas9-xten-aidx(k10et82ie156g)和mo91-dcas9-xten-aidx的结果。
图21:ncas9-aidx融合蛋白的功能验证结果。
具体实施方式
本文涉及核酸酶活性缺失的cas蛋白与胞嘧啶脱氨酶aid或其突变体的融合蛋白。在sgrna的指引下,所述融合蛋白被招募到特定的dna序列,aid或其突变体对胞嘧啶脱氨基,产生尿嘧啶,而后在dna修复过程中,被随机突变成其它碱基,从而在实现定点突变的同时获得高的突变效率。
关于cas/sgrna的内容,除本文下文所述外,还可参见cn201380049665.5和cn201380072752.2,本文将其全部内容以引用的方式纳入本文。
cas蛋白
crispr(clusteredregularlyinterspacedshortpalindromicrepeats)是细菌抵御病毒侵袭或躲避哺乳动物免疫反应的基因编辑系统。该系统经过改造和优化,目前已被广泛应用在体外生化反应、细胞与个体的基因编辑中。
通常,具有核酸内切酶活性的cas蛋白与其特异性识别的sgrna形成的复合物通过sgrna的配对区与靶标dna中的模板链进行互补配对,由cas在特定位置将双链dna切断。应理解,本文中,“cas蛋白”与“cas酶”可互换使用。
本文利用cas/sgrna的上述特性,即利用sgrna与靶标的特异性结合而将cas定位到期望的位置,在该位置由融合蛋白中的aid或其突变体对胞嘧啶脱氨基。适用于本发明的核酸酶活性部分或完全缺失,尤其是核酸内切酶活性部分或完全缺失、但保留了解旋酶活性的cas蛋白可以衍生自本领域周知的各种cas蛋白及其变异体,包括但不限于cas1、cas1b、cas2、cas3、cas4、cas5、cas6、cas7、cas8、cas9(也称为csn1和csx12)、cas10、csy1、csy2、csy3、cse1、cse2、csc1、csc2、csa5、csn2、csm2、csm3、csm4、csm5、csm6、cmr1、cmr3、cmr4、cmr5、cmr6、csb1、csb2、csb3、csx17、csx14、csx10、csx16、csax、csx3、csx1、csx15、csf1、csf2、csf3、csf4、其同源物或其修饰形式。
在一些实施方案中,使用核酸酶活性缺失的cas9酶和其特异性识别的单链sgrna。cas9酶可以是来自不同物种的cas9酶,包括但不限于来自化脓链球菌的cas9(spcas9)、来自金黄色葡萄球菌的cas9(sacas9),以及来自嗜热链球菌的cas9(st1cas9)等。可以使用cas9酶的各种变体,只要该cas9酶能特异性识别它的sgrna,并缺失核酸酶活性即可。
可采用本领域周知的方法制备核酸酶活性缺失的cas蛋白,这些方法包括但不限于使cas蛋白中核酸内切酶的整个催化结构域缺失或使该结构域中的一个或数个氨基酸发生突变,从而产生核酸酶活性缺失的cas蛋白。突变可以是一个或数个(例如2个以上、3个以上、4个以上、5个以上、10个以上,至整个催化结构域)氨基酸残基的缺失或取代,或一个或数个新氨基酸残基(例如1个以上、2个以上、3个以上、4个以上、5个以上、10个以上,或者1~10个、1~15个不等)的插入。可采用本领域常规的方法进行上述结构域的缺失或氨基酸残基的突变,以及检测突变后的cas蛋白是否还具有核酸酶活性。例如,对于cas9,可将它的两个核酸内切酶催化结构域ruvc1和hnh分别突变,例如将该酶的第10个氨基酸(位于ruvc1结构域中)天冬酰胺突变为丙氨酸或其它氨基酸,将第841位氨基酸(位于hnh结构域中)组氨酸突变为丙氨酸或其它氨基酸。这两处突变使cas9失去核酸内切酶活性。优选的是,cas酶完全无核酸酶活性。在一个或多个实施方案中,本文使用的无核酸酶活性的cas9酶的氨基酸序列如seqidno:2第42-1452所示。在其他实施方案中,本文使用的cas酶部分缺失核酸酶活性,即该cas酶可引起dna单链断裂。这类cas酶的代表性例子可如seqidno:72第42-1419位氨基酸残基所示。
cas/sgrna复合物行使功能需要在dna的非模板链(3’到5’)有前间区序列邻近基序(protospaceradjacentmotif,pam)。不同cas酶,其对应的pam并不完全相同。例如,针对spcas9的pam通常是ngg;针对sacas9酶的pam通常是nngrr;针对st1cas9酶的pam通常是nnagaa;其中,n为a、c、t或g,r为g或a。
在某些优选的实施方式中,针对sacas9酶的pam是nngrrt。在某些优选的实施方式中,针对spcas9的pam是tgg。
sgrna
sgrna通常包括两部分:靶标结合区和cas蛋白识别区。靶标结合区与cas蛋白识别区通常以5’到3’的方向连接。
靶标结合区的长度通常为15~25个碱基,更通常为18~22个碱基,如20个碱基。靶标结合区与dna的模板链特异性结合,从而将融合蛋白招募到预定位点。通常,dna模板链上sgrna结合区域的对侧区紧邻pam,或者隔开数个碱基(例如10个以内,或8个以内,或5个以内)。因此,在设计sgrna时,通常先根据所用的cas酶确定该酶的pam,然后在dna的非模板链上寻找可作为pam的位点,之后将该非模板链(3’到5’)pam位点下游紧邻该pam位点或与该pam位点隔开10个以内(例如8个以内、5个以内等)的长15~25个碱基、更通常长18~22个碱基的片段作为sgrna的靶标结合区的序列。
sgrna的cas蛋白识别区则根据所使用的cas蛋白而确定,这为本领域所技术人员所掌握。
因此,本文的sgrna的靶标结合区的序列为含所选cas酶识别的pam位点的dna链下游紧邻该pam位点或与该pam位点隔开10个以内(例如8个以内、5个以内等)的长15~25个碱基、更通常长18~22个碱基的片段;其cas蛋白识别区为所选cas酶所特异性识别。
可采用本领域常规的方法制备sgrna,例如,采用常规的化学合成方法合成。sgrna也可经由表达载体转入细胞,在细胞内表达出该sgrna。可采用本领域周知的方法构建sgrna的表达载体。
激活诱导的胞嘧啶脱氨酶(aid)
aid是一种胞嘧啶脱氨酶,属于apobec家族,一种rna编辑酶家族:n端有核定位信号,c端有核输出信号,其催化结构域为apobec家族所共有。一般认为n端结构为体细胞超变(shm)所必须。aid的功能是对胞嘧啶脱氨基,将胞嘧啶变成尿嘧啶,随后的dna修复可以将尿嘧啶变成其它碱基。应理解的是,本领域周知的胞嘧啶脱氨酶或其保留了对胞嘧啶脱氨基、将胞嘧啶变成尿嘧啶的生物学活性的片段或突变体均可用于本文。
如图14显示了aid的结构功能域。其中氨基酸9-26为核定位(nls)结构域,尤其是氨基酸13-26参与了dna的结合,氨基酸56-94为催化结构域,氨基酸109-182为apobec样结构域,氨基酸193-198为核输出(nes)结构域,氨基酸39-42与连环蛋白样蛋白1(ctnnbl1)相互作用,氨基酸113-123是hotspot识别环。
本文可使用aid的全长序列(如seqidno:2第1457-1654位氨基酸所示),也可使用aid的片段。优选的是,所述片段至少包括nls结构域、催化结构域和apobec样结构域。因此,在某些实施方案中,所述片段至少包含aid第9-182位氨基酸残基(即seqidno:2第1465-1638位氨基酸残基)。在其他实施方案中,所述片段至少包含aid第1-182位氨基酸残基(即seqidno:2第1457-1638位氨基酸残基)。例如,在某些实施方案中,本文使用的aid片段由第1-182位氨基酸残基组成,由第1-186位氨基酸残基组成,或由第1-190位氨基酸残基组成。因此,在某些实施方案中,本文使用的aid片段由seqidno:2第1457-1638位氨基酸残基、seqidno:2第1457-1642位氨基酸残基,或由seqidno:2第1457-1646位氨基酸残组成。
本文还可使用aid的保留了其胞嘧啶脱氨酶活的变体。例如,这样的变体相当于aid的野生型序列可具有1-10个,如1-8个,1-5个或1-3个氨基酸变异,包括氨基酸的缺失、取代和突变。优选的是,这些氨基酸变异不发生在上述nls结构域、催化结构域和apobec样结构域内,或即便发生在这些结构域内也不影响到这些结构域原本的生物学功能。例如,优选的是,这些变异不发生在aid氨基酸序列的第24、27、38、56、58、87、90、112、140等位置上。在某些实施方案中,这些变异也不发生在氨基酸39-42、氨基酸113-123之内。因此,例如,变异可发生在氨基酸1-8、氨基酸28-37、氨基酸43-55和/或氨基酸183-198之中。在某些实施方案中,变异发生在第10、82和156位。例如,在第10、82和156位发生取代突变,这类取代突变可以是k10e、t82i和e156g。在这些实施方案中,示例性的aid突变体的氨基酸序列含有如seqidno:68第1447-1629位所示的氨基酸序列,或由如seqidno:68第1447-1629位所示的氨基酸残基组成。
融合蛋白
本文提供融合蛋白,其含有cas酶与aid。本文的融合蛋白,cas酶通常在融合蛋白氨基酸序列的n端,aid在c端。在某些实施方案中,本文提供主要由cas酶和aid形成的融合蛋白。应理解的是,本文所述的“主要由……形成”的融合蛋白或类似表述并不意指融合蛋白仅包括cas酶和aid,该限定应理解为融合蛋白可仅包括cas酶和aid,或还可含有其他不影响到该融合蛋白中的cas酶的靶向作用及aid突变靶序列的功能的部分,包括但不限于各种接头序列、核定位序列以及如下文所述因基因克隆操作、和/或为了构建融合蛋白、促进重组蛋白的表达、获得自动分泌到宿主细胞外的重组蛋白、或利于重组蛋白的检测和/或纯化等而在融合蛋白中引入的氨基酸序列。
cas酶可通过接头与aid融合。接头可以是3~25个残基的肽,例如3~15、5~15、10~20个残基的肽。肽接头的适合的实例是本领域中公知的。通常,接头含有一个或多个前后重复的基序,该基序通常含有gly和/或ser。例如,该基序可以是sggs、gssgs、gggs、ggggs、ssssg、gsgsa和ggsgg。优选地,该基序在接头序列中是相邻的,在重复之间没有插入氨基酸残基。接头序列可以包含1、2、3、4或5个重复基序组成。在某些实施方案中,接头序列是多甘氨酸接头序列。接头序列中甘氨酸的数量无特别限制,通常为2~20个,例如2~15、2~10、2~8个。除甘氨酸和丝氨酸来,接头中还可含有其它已知的氨基酸残基,例如丙氨酸(a)、亮氨酸(l)、苏氨酸(t)、谷氨酸(e)、苯丙氨酸(f)、精氨酸(r)、谷氨酰胺(q)等。在某些实施方案中,接头序列为xten,其氨基酸序列如seqidno:66第183-198位氨基酸残基所示。
作为例子,接头可由以下氨基酸序列组成:g(sgggg)2sggglgstef(seqidno:21)、rstsglgggs(ggggs)2g(seqidno:22)、qltsglgggs(ggggs)2g(seqidno:23)、gggs(seqidno:24)、ggggs(seqidno:25)、ssssg(seqidno:26)、gsgsa(seqidno:27)、ggsggggggsggggsggggs(seqidno:28)、ssssgssssgssssg(seqidno:29)、gsgsagsgsagsgsa(seqidno:30)、ggsggggsggggsgg(seqidno:31)、seqidno:72第1420-1456位氨基酸残基等。
应理解,在基因克隆操作中,常常需要设计合适的酶切位点,这势必在所表达的氨基酸序列末端引入了一个或多个不相干的残基,而这并不影响目的序列的活性。为了构建融合蛋白、促进重组蛋白的表达、获得自动分泌到宿主细胞外的重组蛋白、或利于重组蛋白的纯化,常常需要将一些氨基酸添加至重组蛋白的n-末端、c-末端或该蛋白内的其它合适区域内,例如,包括但不限于,适合的接头肽、信号肽、前导肽、末端延伸等。因此,本文融合蛋白的氨基端或羧基端还可含有一个或多个多肽片段,作为蛋白标签。任何合适的标签都可以用于本文。例如,所述的标签可以是flag(dykddddk,seqidno:32),ha,ha1,c-myc,poly-his,poly-arg,strep-tagii,au1,ee,t7,4a6,ε,b,ge以及ty1。这些标签可用于对蛋白进行纯化。
本文的融合蛋白还可含有核定位序列(nls)。可使用本领域周知的各种来源和各种氨基酸组成的核定位序列。这类核定位序列包括但不限于:sv40病毒大t抗原的nls,其具有氨基酸序列pkkkrkv(seqidno:33);来自核质蛋白的nls,例如,具有序列krpaatkkagqakkkk(seqidno:34)的核质蛋白二分nls;来自c-myc的nls,其具有氨基酸序列paakrvkld(seqidno:35)或rqrrnelkrsp(seqidno:36);来自hrnpa1m9的nls,其具有序列nqssnfgpmkggnfggrssgpyggggqyfakprnqggy(seqidno:37);来自输入蛋白-α的ibb结构域的序列rmrizfknkgkdtaelrrrrvevsvelrkakkdeqilkrrnv(seqidno:38);肌瘤t蛋白的序列vsrkrprp(seqidno:39)和ppkkared(seqidno:40);小鼠c-abliv的序列salikkkkkmap(seqidno:41);流感病毒ns1的序列drlrr(seqidno:42)和pkqkkrk(seqidno:43);肝炎病毒δ抗原的序列rklkkkikkl(seqidno:44);小鼠mx1蛋白的序列rekkkflkrr(seqidno:45);人聚(adp-核糖)聚合酶的序列krkgdevdgvdevakkkskk(seqidno:46);以及类固醇激素受体(人)糖皮质激素的序列rkclqagmnlearktkk(seqidno:47);等。在某些具体实施方案中,本文使用seqidno:2第26-33位氨基酸残基所示的序列作为nls。nls可位于融合蛋白的n端、c端;也可位于融合蛋白序列中,例如位于融合蛋白中cas9酶的n端和/或c端,或位于融合蛋白中的aid的n端和/或c端。
可以通过任何适合的技术检测本发明融合蛋白在细胞核中的积聚。例如,可将检测标记融合到cas酶上,使得在与检测细胞核的位置的手段(例如,对于细胞核特异的染料,如dapi)相结合时融合蛋白在细胞内的位置可以被可视化。在某些实施方案中,本文使用3*flag作为标记,该肽段序列可如seqidno:2第1-23位氨基酸残基所示。应理解,通常,若存在标记序列时,标记序列通常在融合蛋白的n端。标记序列与nls之间可直接连接,也可通过适当的接头序列连接。nls序列可直接与cas酶或aid连接,也可通过适当的接头序列与cas酶或aid连接。
因此,在某些实施方案中,本文的融合蛋白由cas酶和aid组成。在其它实施方案中,本文的融合蛋白由cas酶通过接头与aid连接而成。在某些实施方案中,本文的融合蛋白nls、cas酶、aid以及cas酶和aid之间的任选的接头序列组成。在某些具体实施方案中,融合蛋白中的cas酶是前文所述的cas9酶。在某些具体实施方案中,融合蛋白中的aid的氨基酸序列如seqidno:2第1457-1654位氨基酸残基所示。在其它具体实施方案中,融合蛋白中的aid的氨基酸序列如seqidno:4第1457-1646位氨基酸残基所示。在其它具体实施方案中,融合蛋白中的aid的氨基酸序列如seqidno:68第1447-1629位氨基酸残基所示。
在某些实施方案中,本文的融合蛋白的氨基酸序列如seqidno:2、4、66、68、70或72所示,或如seqidno:2第26-1654位氨基酸所示,或如seqidno:4第26-1638位所示,或如seqidno:68第26-1629位氨基酸所示,或如seqidno:70第26-1629位氨基酸所示,或如seqidno:72第26-1638位氨基酸所示。
多核苷酸序列、宿主和蛋白表达
本文包括编码本文融合蛋白的的多核苷酸序列。本文的多核苷酸可以是dna形式或rna形式。dna形式包括cdna、基因组dna或人工合成的dna。dna可以是单链的或是双链的。dna可以是编码链或非编码链。
本文所述的核苷酸序列通常可以用pcr扩增法获得。具体而言,可根据本文所公开的核苷酸序列,尤其是开放阅读框序列来设计引物,并用市售的cdna库或按本领域技术人员已知的常规方法所制备的cdna库作为模板,扩增而得有关序列。当序列较长时,常常需要进行两次或多次pcr扩增,然后再将各次扩增出的片段按正确次序拼接在一起。例如,在某些实施方案中,编码本文所述融合蛋白的多核苷酸序列如seqidno:1、3、65、67、79或71所示,或如seqidno:1第73-4965位碱基所示,或如seqidno:3第73-4917位碱基所示,或如seqidno:67第76-4890位碱基所示,或如seqidno:70第76-4890位碱基所示,或如seqidno:72第76-4917位碱基所示。
本文也包括包含所述多核苷酸的核酸构建物。该核酸构建物含有本文所述的融合蛋白的编码序列,以及与这些序列操作性连接的一个或多个调控序列。本发明所述的融合蛋白的编码序列可以多种方式被操作以保证所述蛋白的表达。在将核酸构建物插入载体之前可根据表达载体的不同或要求而对核酸构建物进行操作。利用重组dna方法来改变多核苷酸序列的技术是本领域已知的。
调控序列可以是合适的启动子序列。启动子序列通常与待表达蛋白的编码序列操作性连接。启动子可以是在所选择的宿主细胞中显示转录活性的任何核苷酸序列,包括突变的、截短的和杂合启动子,并且可以从编码与该宿主细胞同源或异源的胞外或胞内多肽的基因获得。
调控序列也可以是合适的转录终止子序列,由宿主细胞识别以终止转录的序列。终止子序列与编码该多肽的核苷酸序列的3’末端操作性连接。在选择的宿主细胞中有功能的任何终止子都可用于本发明。
调控序列也可以是合适的前导序列,对宿主细胞翻译重要的mrna的非翻译区。前导序列与编码该多肽的核苷酸序列的5′末端可操作连接。在选择的宿主细胞中有功能的任何终止子都可用于本发明。
在某些实施方案中,所述核酸构建物是载体。例如,可将本文的多核苷酸序列插入到重组表达载体中。术语“重组表达载体”指本领域熟知的细菌质粒、噬菌体、酵母质粒、植物细胞病毒、哺乳动物细胞病毒如腺病毒、逆转录病毒或其它载体。只要能在宿主体内复制和稳定,任何质粒和载体都可以用。表达载体的一个重要特征是通常含有复制起点、启动子、标记基因和翻译控制元件。表达载体还可包括翻译起始用的核糖体结合位点和转录终止子。本文所述的多核苷酸序列可操作性地连接到表达载体中的适当启动子上,以经由该启动子指导mrna合成。这些启动子的代表性例子有:大肠杆菌的lac或trp启动子;λ噬菌体pl启动子;真核启动子包括cmv立即早期启动子、hsv胸苷激酶启动子、早期和晚期sv40启动子、反转录病毒的ltrs和其它一些已知的可控制基因在原核或真核细胞或其病毒中表达的启动子。标记基因可用于提供用于选择转化的宿主细胞的表型性状,包括但不限于真核细胞培养用的二氢叶酸还原酶、新霉素抗性以及绿色荧光蛋白(gfp),或用于大肠杆菌的四环素或氨苄青霉素抗性。当本文所述的多核苷酸在高等真核细胞中表达时,如果在载体中插入增强子序列,则将会使转录得到增强。增强子是dna的顺式作用因子,通常大约有10到300个碱基对,作用于启动子以增强基因的转录。
本领域一般技术人员清楚如何选择适当的载体、启动子、增强子和宿主细胞。可采用本领域技术人员熟知的方法构建含本文所述的多核苷酸序列和合适的转录/翻译控制信号的表达载体。这些方法包括体外重组dna技术、dna合成技术、体内重组技术等。
可将本文所述的载体转化适当的宿主细胞,以使其能够表达本文所述的融合蛋白。宿主细胞可以是原核细胞,如细菌细胞;或是低等真核细胞,如酵母细胞;丝状真菌细胞、或是高等真核细胞,如哺乳动物细胞。宿主细胞还可以是植物细胞。宿主细胞的代表性例子有:大肠杆菌;链霉菌属;鼠伤寒沙门氏菌的细菌细胞;真菌细胞如酵母、丝状真菌;植物细胞;果蝇s2或sf9的昆虫细胞;cho、cos、293细胞、或bowes黑素瘤细胞的动物细胞等。除用于表达融合蛋白的细胞外,其它的含本文所述多核苷酸序列或载体以及sgrna或其表达载体的细胞,例如用于制备点突变蛋白的细胞,也在本文所述的宿主细胞的范围之内。
用重组dna转化宿主细胞可用本领域技术人员熟知的常规技术进行。当宿主为原核生物如大肠杆菌时,能吸收dna的感受态细胞可在指数生长期后收获,用cacl2法处理,所用的步骤在本领域众所周知。另一种方法是使用mgcl2。如果需要,转化也可用电穿孔的方法进行。当宿主是真核生物,可选用如下的dna转染方法:磷酸钙共沉淀法,常规机械方法如显微注射、电穿孔、脂质体包装等。
转化宿主细胞后,获得的转化子可以用常规方法培养,以允许其表达本文所述的融合蛋白。根据所用的宿主细胞,培养中所用的培养基可选自各种常规培养基。可利用本领域已知的各种分离方法分离和纯化本文的重组融合蛋白。这些方法是本领域技术人员所熟知的,包括但并不限于:常规的复性处理、用蛋白沉淀剂处理(盐析方法)、离心、渗透破菌、超处理、超离心、分子筛层析(凝胶过滤)、吸附层析、离子交换层析、高效液相层析(hplc)和其它各种液相层析技术及这些方法的结合。
因此,本文也包括含本文所述融合蛋白、其编码序列或表达载体和任选的sgrna或其表达载体的宿主细胞。这种宿主细胞可组成型表达本文所述的融合蛋白,也可在一定的诱导条件下表达本文所述的融合蛋白。如何使宿主细胞组成型表达或在诱导条件下表达本发明融合蛋白的方法是本领域周知的。例如,在某些实施方案中,使用诱导型启动子构建本发明的表达载体,从而实现融合蛋白的诱导表达。
组合物、试剂盒
本文的融合蛋白、其编码序列或表达载体,和/和sgrna、其编码序列或表达载体可以组合物的形式提供。例如,组合物可含有本文的融合蛋白和sgrna或sgrna的表达载体,或可含有本文融合蛋白的表达载体和sgrna或sgrna的表达载体。在组合物中,融合蛋白或其表达载体、或sgrna或其表达载体可以混合物的形式提供,或者可单独包装。组合物可以是溶液的形式,也可以是冻干形式。
组合物可提供在试剂盒中。因此,本文提供含有本文所述组合物的试剂盒。或者,本文也提供一种试剂盒,该试剂盒含有本文的融合蛋白和sgrna或sgrna的表达载体,或含有本文融合蛋白的表达载体和sgrna或sgrna的表达载体。试剂盒中,融合蛋白或其表达载体、或sgrna或其表达载体可独立包装,或以混合物的形式提供。试剂盒中还可包括例如用于将所述融合蛋白或其表达载体和/或sgrna或其表达载体转入细胞的试剂,以及指导技术人员进行所述转入的说明书。或者,试剂盒还可包括指导技术人员采用试剂盒所含成分实施本文所述的各种方法和用途的说明书。试剂盒中还包括其它的试剂,例如用于pcr的试剂等。
方法和用途
本文第三方面提供一种在细胞内产生点突变的方法,所述方法包括在所述细胞内表达本文所述的融合蛋白和sgrna的步骤。在某些实施方案中,将本发明的融合蛋白或其表达载体和sgrna或其表达载体转入所述细胞内。在细胞组成型表达本文所述融合蛋白的情况下,可仅将相应的sgrna或其表达载体转入细胞中。在细胞诱导型表达本文所述融合蛋白的情况下,在转入sgrna之后,还可用诱导剂孵育细胞,或对细胞施与相应的诱导措施(例如光照)。可采用常规的转染方法将所述融合蛋白或其表达载体和/或sgrna或其表达载体转入细胞中。例如,在某些实施方案中,转染时,首先制备质粒dna-脂质体复合物,然后将该质粒dna-脂质体复合物和相应的sgrna共同转染细胞。获得产生了点突变的细胞之后,可在适于该细胞生长并表达所需蛋白的条件下培育该细胞,并通过各种常规方法(例如高通量方法)分离、分析所产生的突变体。
因此,本文所述的在细胞内产生点突变的方法也可用于产生突变体文库,然后利用常规的技术手段对文库中的突变体进行分离和筛选,获得具有所需生物学功能的突变体。因此,本发明也提供一种构建突变体文库的方法,所述方法包括在所述细胞内表达本文所述的融合蛋白和sgrna的步骤。
可针对同一待突变位点设计一种或多种sgrna。当设计多种sgrna时,所设计的多种sgrna的靶标结合区不同,但具有相同的cas蛋白识别区。然后可将该一种或多种sgrna与相应的融合蛋白一同转入细胞中。
细胞可以是任意感兴趣的细胞,包括原核细胞和真核细胞,例如植物细胞、动物细胞、微生物细胞等。尤其优选的是动物细胞,例如哺乳动物细胞、啮齿类动物细胞,包括人、马、牛、羊、鼠、兔等等。微生物细胞包括本领域周知的来自各种微生物种类的细胞,尤其是那些具有医疗研究价值、生产价值(例如燃料如乙醇的生产、蛋白质生产、油脂如dha生产)的微生物种类的细胞。细胞还可以是各种器官来源的细胞,例如来自人肝脏、肾脏、皮肤等处的细胞。细胞还可以是目前在售的各种成熟的细胞系,例如293细胞、cos细胞。在某些实施方案中,细胞是来自健康个体的细胞;在其他实施方案中,细胞是来自患病个体的患病组织的细胞,例如来自炎症组织的细胞、肿瘤细胞,诱导型多能干细胞等。细胞还可以是经基因工程改造过,以使其具有某种特定功能(例如生产感兴趣的蛋白)或产生感兴趣的表型的细胞。换言之,待突变的基因或核酸序列对于该细胞而言可以是天然就存在于该细胞内的(内源性)基因或核酸序列,也可以是外来转入的(外源性的)基因或核酸序列。外来转入的基因或核酸序列可整合入细胞的基因组序列中,也可独立于基因组之外并稳定表达。
针对不同的细胞,可采用已知技术设计表达本文融合蛋白和sgrna的表达载体,以使这些表达载体适于在该细胞中表达。例如,可在表达载体中提供利于在该细胞中启动表达的启动子以及其他相关的调控序列。这些都可由技术人员根据实际情况加以选择和实施。
期待产生点突变的核酸序列可以是任何感兴趣的核酸序列,例如基因序列,尤其是各种与疾病相关,或与各种感兴趣的蛋白质的生产相关,或各种与感兴趣的生物学功能相关的基因或核酸序列。这类感兴趣的基因或核酸序列包括但不限于编码各种功能蛋白的核酸序列。本文中,功能蛋白指能够完成生物体的生理功能的蛋白质,包括催化蛋白、运输蛋白、免疫蛋白和调节蛋白等。在某些具体实施方式中,所述功能蛋白包括但不限于:疾病的发生、发展和转移中涉及的蛋白,细胞分化、增殖与凋亡中涉及的蛋白,参与新陈代谢的蛋白,发育相关的蛋白,以及各种药物靶点等等。例如,功能蛋白可以是抗体、酶、脂蛋白、激素类蛋白、运输和贮存蛋白、运动蛋白、受体蛋白、膜蛋白等。因此,可利用本文所述的融合蛋白、多核苷酸、核酸构建物、细胞和方法等构建突变体文库,并进一步筛选获得具有新功能或更强功能的蛋白质,例如抗体、酶或其它功能蛋白等。
利用本文所述的方法可在感兴趣的核酸序列上产生随机突变,或在感兴趣核酸序列的特定位点上产生突变。对于前者,可根据所用cas酶寻找模板链上的pam位点,以该pam位点下游紧邻该pam位点或与该pam位点隔开10个以内(如8个以内、5个以内或3个以内)的长15~25个碱基、更通常长18~22个碱基的片段作为sgrna的靶标识别区设计该cas酶识别的sgrna。对于后者,可在该特定位点附近寻找可作为pam的位点,根据该pam选择能识别该pam的cas酶,并依本文所述设计、制备含该cas酶的本发明融合蛋白以及相应的sgrna。
本文的方法可以是体外方法,也可以是体内方法。当体内实施时,可采用本领域周知的手段将本文的融合蛋白或其表达载体和sgrna或其表达载体转入实验对象体内,如相应的组织细胞内,并通过观察动物表型变化筛选出感兴趣的功能变体。应理解,体内实验时,实验对象可以是各种非人动物,尤其是本领域惯常采用的各种非人模式生物。体内实验也应满足伦理要求。
下文将以具体实施例的方式阐述本发明。应理解,这些实施例仅仅是示例性的,而非限制本发明的范围。下列实施例中未注明具体条件的实验方法,通常按照常规条件如sambrook&russell所著的molecularcloning:alaboratorymanual(分子克隆实验指南第三版)中所述的条件,或按照制造厂商所建议的条件。除非另行定义,文中所使用的所有专业与科学用语与本领域熟练人员所熟悉的意义相同。此外,任何与所记载内容相似或均等的方法及材料皆可应用于本发明中。文中所述的较佳实施方法与材料仅作示范之用。
实施例1:pentr11-dcas9-aid质粒和pentr11-dcas9-aidx质粒的构建
1、以a20细胞株〔购买于中国科学院典型培养物保藏委员会细胞库〕rna反转录出的cdna为模板,利用seqidno:5和6所示引物及seqidno:5和7所示引物分别扩增出aid全长序列和aidx片段(从第183位氨基酸残基起截短)(见图1,a和c);
2、构建pentr11-dcas9-tet1cd质粒:
(1)利用pcr从dcas9质粒(addgene)扩增出dcas9目的基因片段;
(2)利用限制性内切酶bamhⅰ和ncoⅰ对dcas9目的基因片段及pentr11质粒(invitrogen)酶切,回收上述片段;
(3)将酶切后的dcas9片段及pentr11载体连接,然后将连接产物转化到top10感受态细胞中;
(4)挑选阳性克隆,抽提质粒并送测序验证,至此完成了pentr11-dcas9质粒的构建;
(5)利用pcr扩增出tet1cd目的基因片段;
(6)利用限制性内切酶bamhⅰ和xhoⅰ对pentr11-dcas9质粒酶切,并回收片段;
(7)利用gibsonassembly方法将tet1cd克隆到pentr11-dcas9质粒中,至此完成了pentr11-dcas9-tet1cd质粒的构建;
3、利用限制性内切酶bamhⅰ和xhoⅰ对pentr11-dcas9-tet1cd质粒、aid、aidx片段进行酶切,然后回收pentr11-dcas9载体及aid、aidx片段;
4、分别将酶切后的aid、aidx片段与pentr11-dcas9载体连接,然后将连接产物转化到top10感受态细胞中;
5、挑选阳性克隆,抽提质粒并送测序验证,至此完成了pentr11-dcas9-aid及pentr11-dcas9-aidx质粒的构建(图1,b和d)。
实施例2:mo91-dcas9-aid质粒和mo91-dcas9-aidx质粒的构建
1、利用seqidno:8和9所示引物从pentr11-dcas9-aid质粒和pentr11-dcas9-aidx质粒扩增出dcas9-aid片段和dcas9-aidx片段(图2,a);
2、利用限制性内切酶bglⅱ和xhoⅰ对mo91质粒(addgeneplasmid#19755)及aid、aidx片段进行酶切,然后回收载体、aid片段和aidx片段(图2,b);
3、分别将酶切后的aid片段、aidx片段与mo91载体连接,然后将连接产物转化到stbl3感受态细胞中;
4、挑选阳性克隆,抽提质粒并送测序验证,至此完成了mo91-dcas9-aid及mo91-dcas9-aidx质粒的构建(图2,c和d)。
实施例3:mo91-dcas9(3*flag,nls)-aid质粒和mo91-dcas9(3*flag,nls)-aidx质粒的构建
以pcw-cas9质粒(武汉淼灵生物科技有限公司)为模板,设计引物pcr扩增出3*flag+nls片段,利用gibsonassembly方法将3*flag+nls片段分别克隆到mo91-dcas9-aid质粒和mo91-dcas9-aidx质粒的dcas9n端,构建得到mo91-dcas9(3*flag,nls)-aid质粒和mo91-dcas9(3*flag,nls)-aidx质粒(图3)。
实施例4:建立指示aid点突变效率的有效的报告系统
在基因组水平造成的点突变水平需要通过简单直观的方法检测,本发明主要采用流式分析技术在蛋白水平间接检测点突变水平。egfp基因中人为插入终止密码子(tag),egfp无法正常表达。当本文的融合蛋白作用于egfp基因中的终止密码子时,使终止密码子点突变,使egfp基因突变正常表达。因此,egfp表达水平越高,点突变的效率越高。
本实施例将含终止密码子的egfp基因(序列如图4所示)插入到mo405-thy1.1质粒(addgene)中,mscv启动基因表达。使用该质粒包毒感染293t,具体包括:
1、铺板293t,包毒时细胞密度达到90%;
2、24h后包毒,包毒方法和转染一样;
3、包毒后24h换液;
4、包毒后24h,第一次收毒,加入聚凝胺1ug/ml,800g,90min,6-8h后换液;
5、包毒后48h,第二次收毒,加入聚凝胺1ug/ml,800g,90min,6-8h后换液;
6、待细胞长到足够数量后,流式染色(pe-thy1.1),分选th1.1阳性细胞作为报告细胞。结果如图6所示。报告细胞的模式示意图显示在图5中。
实施例5:sgrna的制备
1、寻找20bp的靶标序列。如果该20bp的靶标序列的起始碱基不是g,需将一个g加到其5’端以使其能被rna聚合酶iiiu6启动子有效转录。需注意的是该靶标序列不能含有xhoi或nhei的识别位点。
2、将sgrna克隆到plx(addgene50662)中,获得plxsgrna。需如下4个引物,其中r1和f2是sgrna特异性的:
f1:aaactcgagtgtacaaaaaagcaggctttaaag(seqidno:10)
r1:rc(gn19)ggtgtttcgtcctttcc(seqidno:11)
f2:gn19gttttagagctagaaatagcaa(seqidno:12)
r2:aaagctagctaatgccaactttgtacaagaaagctg(seqidno:13)
其中,gn19=新的靶标序列,rc(gn19)=新靶标序列的反向互补序列。
3、分别使用f1+r1和f2+r2扩增plxsgrna;
4、凝胶纯化两次扩增获得的产物,合并,用于f1+r2进行第三次pcr;
5、使用nhei和xhoi消化步骤4进行的pcr获得的产物;和
6、连接和转化,从而制备得到sgrna的表达载体。
四条sgrna的靶标结合区的碱基序列如下所示:
gcatgcccgaaggctacgtcc(seqidno:14);
gcaactagtatacccgcgccg(seqidno:15);
gcctcgaacttcacctcggcg(seqidno:16);
gtcagctcgatgcggttcacc(seqidno:17)。
实施例6:crispr-cas9提高aid点突变效率
培养实施例4所构建的报告细胞至70-90%的汇合度时进行转染。转染时,首先制备质粒dna-脂质体复合物,包括将四倍量的
结果如图7所示,aid与aidx的%egfp+分别为0.14%和0.30%,而dcas9-aid+sgrna和dcas9-aidx+sgrna的%egfp+分别为2.14%和4.36%。
结果表明,将aid或aidx与dcas9融合,在sgrna的导向作用下,会使aid在sgrna的靶向作用下,在aid的点突变功能局限在特异的部位,同时提高其作用浓度,提高其突变效率。
实施例7:crispr-cas9提高aid点突变效率及优化
采用与实施例6相同的方法,在实施例4构建的报告细胞中共转sgrna和dcas9-aid的表达载体。其中sgrna分两组,一组是针对aavs1的对照sgrna,其靶标结合区分别如下:gattcccagggccggttaatg(seqidno:18);gtcccctccaccccacagtg(seqidno:19);和ggggccactagggacaggat(seqidno:20)。另外一组是针对egfp的sgrna组(seqidno:14-17)。同时设置对照组在报告细胞中单转aid。对照sgrna的表达载体如实施例5所述方法构建。
在转染后第8天测facs,aid组的egfp%+只有0.13%,而dcas9-aid+sgrna组的egfp%+达到2.1%(图8,a),egfp%+有了16倍提高。为了进一步优化dcas9-aid系统的效率,将dcas9与不同的aid突变体融合:aid-fl(全长),aid-cd(仅含催化结构域),p182x(从第183位氨基酸残基起截短),r186x(从第187位氨基酸残基起截短),r190x(从第191位氨基酸残基起截短)。在报告细胞中共转各dcas9-aid表达载体和sgrna,其中dcas9-r186x的效率最高(图8,b和c)。因此采用dcas9-r186x进行实施例8-13的试验,在这些实施例中,将dcas9-r186x简称为dcas9-aidx。
为了证明在dcas9-aid体系中确实是由aid与dcas9融合后,才使整个系统具有碱基置换功能,在报告细胞中分别共转cas9,dcas9,dcas9-aidx的功能突变体〔r186x(e58q)〕,dcas9-aidx和sgrna,只有dcas9-aidx和sgrna组具有egfp%+,而其他组均为0(图8,c)。也就证明确实是由aid与dcas9融合后,才使整个系统具有碱基置换功能。
实施例8:crispr-cas9将aid点突变功能局限在sgrna靶向部位
为研究crispr-cas9是否能将aid点突变功能局限在sgrna靶向部位,以实施例4构建的报告系统的基因组dna为模板,对含终止密码子的egfp进行pcr,构建文库,并将cmyc作为对照基因,进行miseq测序。结果如图9所示。由报告细胞的测序结果可知,miseq虽然测序通量高,滤去低质量的读数(reads)后,但仍有测序基底突变频率,egfp为0.25%,cmyc为0.15%。但即使有基底水平干扰,仍可观察到dcas9-aidx+sgrna组的egfp基因点突变频率明显高于aidx组,同样证明crispr-cas9提高aid点突变效率。并且这些高频突变位点主要集中在sgrna的靶向位点,而在cmyc基因中几乎没有发生点突变。证明dcas9与aid融合之后,sgrna将dcas9-aid靶向到sgrna的靶向位点,使aid只会对sgrna的靶向位点发挥作用,产生点突变,而不会对其他基因位点造成很大改变;并且能够大幅提高点突变频率。
实施例9:dcas9-aidx将c和g碱基随机突变为其他三种碱基
aidx本身会将c突变为t,将g突变为a。将dcas9与aidx融合之后,与aidx组对比,c和g的突变方向变得更加均一化。
同时aid本身的作用是依赖于hotspot基序的wrcy(w代表a/t,r代表a/c,y代表c/t),其中最偏好的基序是agct。而将dcas9与aidx融合之后,这种基序的偏好性会明显消失。因此本发明人提出一种假设,正常情况下,aid会将胞嘧啶脱氨基,形成尿嘧啶,通过dna复制修复,将这种u-g错配保留,发生c到t、g到a的突变,另外可以通过碱基切除修复方式,将u碱基切除,随即插入四种碱基。所以dcas9与aid的融合很有可能抑制dna复制这条途径,促进碱基切除修复,使突变方向更加均一化(图10,b)。
此外,对miseq数据进行统计分析,aidx和dcas9-aidx+sgrna组在egfp上的造成点突变类型基本上与报道一致,c和g碱基突变占主要部分,a和t所占比例较少。并且g主要突变向t,c突变向a。但在dcas9-aidx组,g突变向t和c的比例增加,c突变向g或a的比例增加。因此,dcas9-aidx可以产生更均一的突变类型(图10,a)。
实施例10:ugi提高dcas9-aidx系统的碱基置换频率,揭示dcas9-aidx在基因上的作用轨迹,并使碱基突变方向更加单一化。
ugi是ung的抑制剂,是一种噬菌体蛋白,当噬菌体入侵大肠杆菌时,可以保护自身的基因组免受宿主ung的修复(图11,a)。在报告细胞中共转三种质粒,分别表达dcas9-aidx、单条sgrna(靶标结合区为gcctcgaacttcacctcggcg,seqidno:16)和ugi(蛋白序列:uniprotkb-p14739),用以提高在整个体系中单条sgrna的突变效率。结果显示,最高点突变效率有10倍提高(图11,b)。
除此之外,加入ugi后,整个体系的突变方向更加单一,c到t,g到a。同时统计了dcas9-aidx的作用轨迹,整个体系在pam序列前后造成的突变频率。图11(c)是根据针对egfp位点设计的4个sgrna的数据进行的统计。都是以pam序列中ngg中的n为第一位碱基。其上游为-,下游为+,两组数据的统计结果一致,都是对pam的上游20bp也就是在原型间隔序列区域造成突变,而且突变最高点是在pam的-12/-13位。ugi可以增加aid的整体突变频率,但会使碱基置换的比例增加,转换比例减少(图11,d)。
实施例11:dcas9-aidx不仅可以对外源性基因起作用,同时可以作用于内源性基因。以上的实验均是在报告细胞中进行,本实施例选用内源性基因aavs1作为靶标位点,设计3个sgrna(seqidno:18-20),在293t中共转表达dcas9-aid和针对aavs1的三个sgrna的载体(如实施例7所述)。
结果如图12所示。dcas9-aid系统同样可以对内源性基因aavs1产生碱基置换,并且这种突变也是集中在sgrna靶标位点。
实施例12:将dcas9-aidx应用于k562bcr-abl基因的gleevec耐药性筛选
k562是来源于慢性髓样白血病人的白血病细胞系。在这种细胞中存在着一种染色体,叫做ph染色体。该染色体是由第9号和第22号染色体的长臂转座而成。第9号染色体上的abl基因含有酪氨酸激酶活性中心,在正常状态下处于低活性状态,而当转座到bcr基因座中后,会具有很高的活性。会引起一系列信号转导,引发癌症,因此bcr-abl是一种原癌基因,常用的药物就是gleevec(格列卫,活性成分是甲磺酸依马替尼),其主要作用机制是gleevec可以竞争性与abl结合atp,从而使abl基因处于低活性。但在病人样本中发现在酪氨酸激酶活性结构域中,会发生点突变,如t315i,使结构域失去结合gleevec的能力,产生gleevec耐药性。除此之外,其它位点的碱基置换也会导致gleevec耐药性。可以使用dcas9-aidx系统来筛选gleevec耐药性位点及具体突变类型,作为设计下一代抑制剂的基础。
首先,为了获得稳定表达dcas9-aidx的k562细胞,我们利用目的质粒mscv-dcas9-aid-p182x-ires-thy1.1与病毒包装质粒pcl-10a1共同转染293t细胞。在六孔板的一孔中提前12-24小时铺好1x106的293t细胞,并用2ml无抗10%fbs的dmem培养过夜,次日待细胞长至80%密度时,转染3ug目的质粒和1ug病毒包装质粒,以及10ul转染试剂lipo2000。转染24小时后用2ml有抗培液培养,分别在48小时、72小时收集病毒。收集好的病毒立即1000rpm离心5分钟去除细胞碎片,取上清加入2ul10mg/mlpolybrene感染1x105的k562细胞,37℃、900g转速甩板90分钟。感染后4小时离心细胞,取沉淀用有抗培液培养。经过两天连续感染后的k562细胞需要再继续培养两天,再利用流式染色,将表达thy1.1表面分子的细胞标记为pe+(抗体1:200稀释),并利用单细胞分选技术获得两块96孔板pe-thy1.1+的k562单细胞。经过两周的培养,收集由各个单细胞克隆产生的细胞群的rna,分别进行rt-qpcr实验。其中dcas9-aidx表达最高的细胞株用以进行后续对gleevec耐药性位点及突变类型的筛选。
同时,为了筛选出gleevec耐药性的位点,我们针对abl基因第六号外显子exon6所在基因组区域进行了sgrna的设计。共设计了16个sgrna(靶标区序列分别如seqidno:49-64所示),其中6个靶向到与外显子exon6相邻的内含子区域,10个直接靶向到exon6区域,并覆盖了83%的外显子序列。由于t315i的突变已被公认为是造成gleevec耐药性的最主要突变之一,我们设计的sgrna中有且仅有1个能够覆盖到t315i突变的位点(944c),能够作为阳性对照。同时,我们针对与gleevec耐药性无关的aavs1基因的基因组序列设计了3个sgrna作为阴性对照(靶标区序列如seqidno:18-20所示)。这些sgrna序列都是通过化学合成,利用bamh1和hindiii双酶切,最终被克隆于携带h1启动子的psuper-sgrna载体中。我们利用苯酚氯仿-乙醇沉降法对等量混合的16个exon6的sgrna质粒或3个aavs1的sgrna质粒进行沉降,使混合质粒的最终浓度在1.5ug/ul以上。随后,将稳定表达dcas9-aidx的k562细胞株分别用abl-exon6、aavs1混合好的sgrna库进行电转,仪器使用美国lifetechnology公司neo电转仪。电转前12-24小时,先以无抗10%fbs的imdm培养液培养k562细胞,电转当天以1000v电压、单脉冲、50ms电击时间为条件,对两份1.2x106的k562细胞分别转染8ug等量混合的abl-exon6或aavs1的sgrna。由于psuper-sgrna质粒载体携带有嘌呤霉素抗性基因,故在转染后24小时,加入2ug/ml嘌呤霉素对表达sgrna的细胞进行筛选。嘌呤霉素处理48小时后撤去,k562细胞继续扩大培养。转染后第六天收集2x105的细胞dna和rna进行高通量测序并作为input对照,将其余细胞分成两份,分别用10umgleevec药物或与其等体积的dmso处理。每三天进行一次ficoll,除去死亡细胞,直到细胞数低于2x104时为止。在gleevec药物处理下,转染进aavs1sgrna的对照组细胞在7-10天左右基本全部死亡,而转染进abl-exon6sgrna的实验组细胞能够继续增殖。在转染后第36-40天左右,实验组细胞增殖到107数量级(图14,b)。同时收集gleevec处理和dmso处理的细胞的dna和rna,进行高通量测序分析。测序结果表明,在30%的细胞中有t315i的突变,而此突变是已知的在病人中发现的耐药性突变,除此之外,还发现多个未报道过的点突变(图14,c和d)。
实施例13:将dcas9-aidx应用于体外提高抗体的亲和力和特异性
抗体可以特异性的识别抗原,作为治疗多种疾病的药物蛋白。抗体的亲和力与其在体内生发中心产生的体细胞突变成正比,一般而言,高亲和力的抗体都具有多个体细胞高频突变。因此,可以使用dcas9-aidx来针对抗体基因进行突变,筛选亲和力更强或具有其它特征(如特异性更好等)的抗体。
使用方案如下,在293t细胞表面稳定表达抗体分子,而后针对抗体基因,设计sgrna,和dcas9-aidx同时转染293t细胞,而后进行细胞表面的染色,染色越强的细胞,其突变的抗体分子具有更强的亲和力。
本实施例采用invitrogen公司的稳定表达一个lacz-zeocintm融合基因座的flp-intm-293细胞。首先合成低亲和力的抗鸡卵溶菌酶(hel)的小鼠igg1抗体(kd=2.78e-09m)的cdna序列,并连接上h2kk蛋白跨膜区序列的编码序列,以在抗体末端加入h2kk蛋白的跨膜区序列,将所得dna序列克隆如pcdna5/frt/goi载体(lifesciencetechnology,usa)中。将该载体转入flp-intm-293细胞中,利用该flp-intm-293细胞所含的flp-intm系统将含flp重组靶位点的该igg1编码序列通过flp重组酶整合到lacz-zeocintm融合基因座上。没有整合成功的细胞能够表达抗zeocin的蛋白;而整合成功后,抗zeocin的蛋白由于缺少起始密码子atg而不能表达,但能够表达抗潮霉素的蛋白。因此,利用潮霉素抗生素来筛选出igg1整合成功的293细胞,在这类细胞中,每个细胞只表达一个拷贝的抗hel-igg1基因。
接着,分别针对igg1重链和轻链的各3个cdrs选择16个合适的pam序列设计如下所示的sgrna(seqidno:73-88),使每个重链或轻链的cdr至少有2条sgrna覆盖:
igh
cdr1_1:tccctcacctgttctgtcac(seqidno:73);
cdr1_2:gctccagtaatcactggtga(seqidno:74);
cdr1_3:gatccagctccagtaatcac(seqidno:75);
cdr1_4:gtgattactggagctggatc(seqidno:76);
cdr2_1:atggggtacgtaagctacag(seqidno:77);
cdr2_2:gagattcgacttttgagaga(seqidno:78);
cdr3_1:tattactgtgcaaactggga(seqidno:79);
cdr3_2:caaactgggacggtgattac(seqidno:80);
cdr3_3:gacggtgattactggggcca(seqidno:81);
igl
cdr1_1:gttgttgccaatactttggc(seqidno:82);
cdr1_2:atagcgtcagtctttcctgc(seqidno:83);
cdr1_3:gtattggcaacaacctacac(seqidno:84);
cdr2_1:aggggatcccagagatggac(seqidno:85);
cdr2_2:tatgcttcccagtccatctc(seqidno:86);
cdr3_1:tctgtcaacagagtaacagc(seqidno:87);
cdr3_2:gtcccccctccgaacgtgta(seqidno:88)。
然后将sgrna序列克隆到psuper-puro质粒载体(addgene)中。将实施例3构建的mo91-dcas9(3*flag,nls)-aidx质粒和sgrna库(即16个sgrna按等量混合在一起)或对照基因aavs1的sgrna共转染到前文获得的表达igg1的293细胞中,经过嘌呤霉素和杀稻瘟菌素抗生素筛杀后,于转染后第7天进行pe抗小鼠igg和alex647-hel表面染色后进行流式分选,分选出igg强度不变而和hel抗原结合增加的细胞。经培养增殖后,首先对dna上的突变进行高通量测序分析,其结果和本文对abl基因或gfp基因的突变基本一致(图15)。dcas9-aidx诱导了抗heligg1可变区的碱基突变并可重复地诱导igg1cdr的碱基突变(图16)。
而后,用pe抗小鼠igg1和647-hel表面染色在流式细胞仪上检测突变后的细胞,发现一小群细胞的igg1表达不变而和hel结合增加。而后对这群细胞进行流式分选,分选扩增后,和突变前的细胞进行比较,发现突变后的抗体对hel的亲和力增强了10倍以上(图17)。
然后收取适量细胞抽取基因组dna进行测序,发现其亲和力增加的主要原因是由轻链的52位的甘氨酸突变为天冬氨酸(碱基为ggt改变为gat,图15)。
实施例14:其它融合蛋白的制备
1、质粒的构建
(1)利用基因合成合成xten接头序列;
(2)利用限制性内切酶对实施例2构建获得的mo91-dcas9-aidx质粒进行酶切,回收载体、aidx片段和dcas9片段;
(3)分别将酶切后的aidx片段、dcas9片段、xten接头序列与mo91载体连接,然后将连接产物转化到stbl3感受态细胞中;
(4)挑选阳性克隆,抽提质粒并送测序验证,至此完成了mo91-dcas9-xten-aidx质粒的构建;
可参照上述步骤以及实施例1和2的方法构建质粒mo91-aidx-xten-dcas9,mo91-dcas9-xten-aidx(k10et82ie156g)以及mo91-ncas9-aidx。
在需要克隆入3*flag和/或nls片段时,可参照实施例3的方法在上述质粒中克隆入3*flag和/或nls片段,获得分别表达seqidno:66、68、70和72所示融合蛋白的质粒。这些融合蛋白中的aidx为从第183位氨基酸残基起截短的aid片段或其突变体。
2、重组蛋白的表达和纯化
(1)按常规方法构建质粒pet-ncas9-aidx-6his,然后用该质粒转化大肠杆菌bl21star-感受态细胞;
(2)将所得表达菌株在含有100μg/ml卡那霉素的lb培养基中在37℃下生长过夜。将细胞以1:100稀释到2xyt培养基中,并在37℃下生长至od600=~0.6。培养物在2小时内冷却至4℃,加入iptg0.5mm,诱导蛋白表达~16h;
(3)通过在4000g离心15分钟收集细胞,并重悬于裂解缓冲液中;
(4)细胞用细胞破碎剂(union)在800巴下裂解5分钟,离心后分离裂解物上清15分钟;
(5)将裂解物在4℃下与ni-nta(1ml浆液/l细菌)(dp101,transgen)一起温育1小时以捕获his-标记的融合蛋白;将树脂转移到柱中,用冷洗涤缓冲液(使用考马斯g250不能观察到颜色变化的程度)广泛洗涤;
(6)his标记的融合蛋白在洗脱缓冲液中洗脱,并通过超滤(amicon-millipore,100kda分子量截留)浓缩至1ml总体积;
(7)将蛋白质在缓冲液a中稀释至20ml,并加载到hi-trapsp柱(29051324,gehealthcare)上并用100mm-1mnacl梯度洗脱;
(8)将含有ncas9-aidx的洗脱级分浓缩至约1ml,并通过使用superdex20010/300gl柱(17517501,ge医疗);
(9)将洗脱的蛋白质浓缩至约3mg/ml,在液氮中快速冷冻并储存在-80℃。
在细菌中诱导ncas9-aidx表达的电泳图谱见图18。
3、不同融合蛋白的功能测试
采用与实施例10相同的方法测试本实施例不同融合蛋白的功能。结果如图19-21所示。
序列表
<110>中国科学院上海生命科学研究院
<120>在细胞内产生点突变的融合蛋白、其制备及用途
<130>162593z1
<160>95
<170>patentinversion3.3
<210>1
<211>4989
<212>dna
<213>人工序列
<220>
<223>人工序列的描述:dcas9-aid的编码序列
<400>1
atggactataaggaccacgacggagactacaaggatcatgatattgattacaaagacgat60
gacgataagatggccccaaagaagaagcggaaggtcggtatccacggagtcccagcagct120
accatggacaagaagtattctatcggactggccatcgggactaatagcgtcgggtgggcc180
gtgatcactgacgagtacaaggtgccctctaagaagttcaaggtgctcgggaacaccgac240
cggcattccatcaagaaaaatctgatcggagctctcctctttgattcaggggagaccgct300
gaagcaacccgcctcaagcggactgctagacggcggtacaccaggaggaagaaccggatt360
tgttaccttcaagagatattctccaacgaaatggcaaaggtcgacgacagcttcttccat420
aggctggaagaatcattcctcgtggaagaggataagaagcatgaacggcatcccatcttc480
ggtaatatcgtcgacgaggtggcctatcacgagaaatacccaaccatctaccatcttcgc540
aaaaagctggtggactcaaccgacaaggcagacctccggcttatctacctggccctggcc600
cacatgatcaagttcagaggccacttcctgatcgagggcgacctcaatcctgacaatagc660
gatgtggataaactgttcatccagctggtgcagacttacaaccagctctttgaagagaac720
cccatcaatgcaagcggagtcgatgccaaggccattctgtcagcccggctgtcaaagagc780
cgcagacttgagaatcttatcgctcagctgccgggtgaaaagaaaaatggactgttcggg840
aacctgattgctctttcacttgggctgactcccaatttcaagtctaatttcgacctggca900
gaggatgccaagctgcaactgtccaaggacacctatgatgacgatctcgacaacctcctg960
gcccagatcggtgaccaatacgccgaccttttccttgctgctaagaatctttctgacgcc1020
atcctgctgtctgacattctccgcgtgaacactgaaatcaccaaggcccctctttcagct1080
tcaatgattaagcggtatgatgagcaccaccaggacctgaccctgcttaaggcactcgtc1140
cggcagcagcttccggagaagtacaaggaaatcttctttgaccagtcaaagaatggatac1200
gccggctacatcgacggaggtgcctcccaagaggaattttataagtttatcaaacctatc1260
cttgagaagatggacggcaccgaagagctcctcgtgaaactgaatcgggaggatctgctg1320
cggaagcagcgcactttcgacaatgggagcattccccaccagatccatcttggggagctt1380
cacgccatccttcggcgccaagaggacttctacccctttcttaaggacaacagggagaag1440
attgagaaaattctcactttccgcatcccctactacgtgggacccctcgccagaggaaat1500
agccggtttgcttggatgaccagaaagtcagaagaaactatcactccctggaacttcgaa1560
gaggtggtggacaagggagccagcgctcagtcattcatcgaacggatgactaacttcgat1620
aagaacctccccaatgagaaggtcctgccgaaacattccctgctctacgagtactttacc1680
gtgtacaacgagctgaccaaggtgaaatatgtcaccgaagggatgaggaagcccgcattc1740
ctgtcaggcgaacaaaagaaggcaattgtggaccttctgttcaagaccaatagaaaggtg1800
accgtgaagcagctgaaggaggactatttcaagaaaattgaatgcttcgactctgtggag1860
attagcggggtcgaagatcggttcaacgcaagcctgggtacctaccatgatctgcttaag1920
atcatcaaggacaaggattttctggacaatgaggagaacgaggacatccttgaggacatt1980
gtcctgactctcactctgttcgaggaccgggaaatgatcgaggagaggcttaagacctac2040
gcccatctgttcgacgataaagtgatgaagcaacttaaacggagaagatataccggatgg2100
ggacgccttagccgcaaactcatcaacggaatccgggacaaacagagcggaaagaccatt2160
cttgatttccttaagagcgacggattcgctaatcgcaacttcatgcaacttatccatgat2220
gattccctgacctttaaggaggacatccagaaggcccaagtgtctggacaaggtgactca2280
ctgcacgagcatatcgcaaatctggctggttcacccgctattaagaagggtattctccag2340
accgtgaaagtcgtggacgagctggtcaaggtgatgggtcgccataaaccagagaacatt2400
gtcatcgagatggccagggaaaaccagactacccagaagggacagaagaacagcagggag2460
cggatgaaaagaattgaggaagggattaaggagctcgggtcacagatccttaaagagcac2520
ccggtggaaaacacccagcttcagaatgagaagctctatctgtactaccttcaaaatgga2580
cgcgatatgtatgtggaccaagagcttgatatcaacaggctctcagactacgacgtggac2640
gccatcgtccctcagagcttcctcaaagacgactcaattgacaataaggtgctgactcgc2700
tcagacaagaaccggggaaagtcagataacgtgccctcagaggaagtcgtgaaaaagatg2760
aagaactattggcgccagcttctgaacgcaaagctgatcactcagcggaagttcgacaat2820
ctcactaaggctgagaggggcggactgagcgaactggacaaagcaggattcattaaacgg2880
caacttgtggagactcggcagattactaaacatgtcgcccaaatccttgactcacgcatg2940
aataccaagtacgacgaaaacgacaaacttatccgcgaggtgaaggtgattaccctgaag3000
tccaagctggtcagcgatttcagaaaggactttcaattctacaaagtgcgggagatcaat3060
aactatcatcatgctcatgacgcatatctgaatgccgtggtgggaaccgccctgatcaag3120
aagtacccaaagctggaaagcgagttcgtgtacggagactacaaggtctacgacgtgcgc3180
aagatgattgccaaatctgagcaggagatcggaaaggccaccgcaaagtacttcttctac3240
agcaacatcatgaatttcttcaagaccgaaatcacccttgcaaacggtgagatccggaag3300
aggccgctcatcgagactaatggggagactggcgaaatcgtgtgggacaagggcagagat3360
ttcgctaccgtgcgcaaagtgctttctatgcctcaagtgaacatcgtgaagaaaaccgag3420
gtgcaaaccggaggcttttctaaggaatcaatcctccccaagcgcaactccgacaagctc3480
attgcaaggaagaaggattgggaccctaagaagtacggcggattcgattcaccaactgtg3540
gcttattctgtcctggtcgtggctaaggtggaaaaaggaaagtctaagaagctcaagagc3600
gtgaaggaactgctgggtatcaccattatggagcgcagctccttcgagaagaacccaatt3660
gactttctcgaagccaaaggttacaaggaagtcaagaaggaccttatcatcaagctccca3720
aagtatagcctgttcgaactggagaatgggcggaagcggatgctcgcctccgctggcgaa3780
cttcagaagggtaatgagctggctctcccctccaagtacgtgaatttcctctaccttgca3840
agccattacgagaagctgaaggggagccccgaggacaacgagcaaaagcaactgtttgtg3900
gagcagcataagcattatctggacgagatcattgagcagatttccgagttttctaaacgc3960
gtcattctcgctgatgccaacctcgataaagtccttagcgcatacaataagcacagagac4020
aaaccaattcgggagcaggctgagaatatcatccacctgttcaccctcaccaatcttggt4080
gcccctgccgcattcaagtacttcgacaccaccatcgaccggaaacgctatacctccacc4140
aaagaagtgctggacgccaccctcatccaccagagcatcaccggactttacgaaactcgg4200
attgacctctcacagctcggaggggatgagggagctcccaagaaaaagcgcaaggtaggt4260
agttccggatctccgaaaaagaaacgcaaagttggtagtgatgctttagacgattttgac4320
ttagatatgcttggttcagacgcgttagacgacttcggtggaggatccatggacagcctc4380
ttgatgaaccggaggaagtttctttaccaattcaaaaatgtccgctgggctaagggtcgg4440
cgtgagacctacctgtgctacgtagtgaagaggcgtgacagtgctacatccttttcactg4500
gactttggttatcttcgcaataagaacggctgccacgtggaattgctcttcctccgctac4560
atctcggactgggacctagaccctggccgctgctaccgcgtcacctggttcacctcctgg4620
agcccctgctacgactgtgcccgacatgtggccgactttctgcgagggaaccccaacctc4680
agtctgaggatcttcaccgcgcgcctctacttctgtgaggaccgcaaggctgagcccgag4740
gggctgcggcggctgcaccgcgccggggtgcaaatagccatcatgaccttcaaagattat4800
ttttactgctggaatacttttgtagaaaaccatgaaagaactttcaaagcctgggaaggg4860
ctgcatgaaaattcagttcgtctctccagacagcttcggcgcatccttttgcccctgtat4920
gaggttgatgacttacgagacgcatttcgtacttggggacgtgattacaaagacgatgac4980
gataagtga4989
<210>2
<211>1662
<212>prt
<213>人工序列
<220>
<223>人工序列的描述:dcas9-aid的氨基酸序列
<400>2
metasptyrlysasphisaspglyasptyrlysasphisaspileasp
151015
tyrlysaspaspaspasplysmetalaprolyslyslysarglysval
202530
glyilehisglyvalproalaalathrmetasplyslystyrserile
354045
glyleualaileglythrasnservalglytrpalavalilethrasp
505560
glutyrlysvalproserlyslysphelysvalleuglyasnthrasp
65707580
arghisserilelyslysasnleuileglyalaleuleupheaspser
859095
glygluthralaglualathrargleulysargthralaargargarg
100105110
tyrthrargarglysasnargilecystyrleuglngluilepheser
115120125
asnglumetalalysvalaspaspserphephehisargleugluglu
130135140
serpheleuvalglugluasplyslyshisgluarghisproilephe
145150155160
glyasnilevalaspgluvalalatyrhisglulystyrprothrile
165170175
tyrhisleuarglyslysleuvalaspserthrasplysalaaspleu
180185190
argleuiletyrleualaleualahismetilelyspheargglyhis
195200205
pheleuilegluglyaspleuasnproaspasnseraspvalasplys
210215220
leupheileglnleuvalglnthrtyrasnglnleupheglugluasn
225230235240
proileasnalaserglyvalaspalalysalaileleuseralaarg
245250255
leuserlysserargargleugluasnleuilealaglnleuprogly
260265270
glulyslysasnglyleupheglyasnleuilealaleuserleugly
275280285
leuthrproasnphelysserasnpheaspleualagluaspalalys
290295300
leuglnleuserlysaspthrtyraspaspaspleuaspasnleuleu
305310315320
alaglnileglyaspglntyralaaspleupheleualaalalysasn
325330335
leuseraspalaileleuleuseraspileleuargvalasnthrglu
340345350
ilethrlysalaproleuseralasermetilelysargtyraspglu
355360365
hishisglnaspleuthrleuleulysalaleuvalargglnglnleu
370375380
proglulystyrlysgluilephepheaspglnserlysasnglytyr
385390395400
alaglytyrileaspglyglyalaserglnglugluphetyrlysphe
405410415
ilelysproileleuglulysmetaspglythrglugluleuleuval
420425430
lysleuasnarggluaspleuleuarglysglnargthrpheaspasn
435440445
glyserileprohisglnilehisleuglygluleuhisalaileleu
450455460
argargglngluaspphetyrpropheleulysaspasnargglulys
465470475480
ileglulysileleuthrpheargileprotyrtyrvalglyproleu
485490495
alaargglyasnserargphealatrpmetthrarglyssergluglu
500505510
thrilethrprotrpasnpheglugluvalvalasplysglyalaser
515520525
alaglnserpheilegluargmetthrasnpheasplysasnleupro
530535540
asnglulysvalleuprolyshisserleuleutyrglutyrphethr
545550555560
valtyrasngluleuthrlysvallystyrvalthrgluglymetarg
565570575
lysproalapheleuserglygluglnlyslysalailevalaspleu
580585590
leuphelysthrasnarglysvalthrvallysglnleulysgluasp
595600605
tyrphelyslysileglucyspheaspservalgluileserglyval
610615620
gluaspargpheasnalaserleuglythrtyrhisaspleuleulys
625630635640
ileilelysasplysasppheleuaspasnglugluasngluaspile
645650655
leugluaspilevalleuthrleuthrleuphegluaspargglumet
660665670
ileglugluargleulysthrtyralahisleupheaspasplysval
675680685
metlysglnleulysargargargtyrthrglytrpglyargleuser
690695700
arglysleuileasnglyileargasplysglnserglylysthrile
705710715720
leuasppheleulysseraspglyphealaasnargasnphemetgln
725730735
leuilehisaspaspserleuthrphelysgluaspileglnlysala
740745750
glnvalserglyglnglyaspserleuhisgluhisilealaasnleu
755760765
alaglyserproalailelyslysglyileleuglnthrvallysval
770775780
valaspgluleuvallysvalmetglyarghislysprogluasnile
785790795800
valileglumetalaarggluasnglnthrthrglnlysglyglnlys
805810815
asnserarggluargmetlysargileglugluglyilelysgluleu
820825830
glyserglnileleulysgluhisprovalgluasnthrglnleugln
835840845
asnglulysleutyrleutyrtyrleuglnasnglyargaspmettyr
850855860
valaspglngluleuaspileasnargleuserasptyraspvalasp
865870875880
alailevalproglnserpheleulysaspaspserileaspasnlys
885890895
valleuthrargserasplysasnargglylysseraspasnvalpro
900905910
serglugluvalvallyslysmetlysasntyrtrpargglnleuleu
915920925
asnalalysleuilethrglnarglyspheaspasnleuthrlysala
930935940
gluargglyglyleusergluleuasplysalaglypheilelysarg
945950955960
glnleuvalgluthrargglnilethrlyshisvalalaglnileleu
965970975
aspserargmetasnthrlystyraspgluasnasplysleuilearg
980985990
gluvallysvalilethrleulysserlysleuvalseraspphearg
99510001005
lysasppheglnphetyrlysvalarggluileasnasntyrhis
101010151020
hisalahisaspalatyrleuasnalavalvalglythralaleu
102510301035
ilelyslystyrprolysleuglusergluphevaltyrglyasp
104010451050
tyrlysvaltyraspvalarglysmetilealalysserglugln
105510601065
gluileglylysalathralalystyrphephetyrserasnile
107010751080
metasnphephelysthrgluilethrleualaasnglygluile
108510901095
arglysargproleuilegluthrasnglygluthrglygluile
110011051110
valtrpasplysglyargaspphealathrvalarglysvalleu
111511201125
sermetproglnvalasnilevallyslysthrgluvalglnthr
113011351140
glyglypheserlysgluserileleuprolysargasnserasp
114511501155
lysleuilealaarglyslysasptrpaspprolyslystyrgly
116011651170
glypheaspserprothrvalalatyrservalleuvalvalala
117511801185
lysvalglulysglylysserlyslysleulysservallysglu
119011951200
leuleuglyilethrilemetgluargserserpheglulysasn
120512101215
proileasppheleuglualalysglytyrlysgluvallyslys
122012251230
aspleuileilelysleuprolystyrserleuphegluleuglu
123512401245
asnglyarglysargmetleualaseralaglygluleuglnlys
125012551260
glyasngluleualaleuproserlystyrvalasnpheleutyr
126512701275
leualaserhistyrglulysleulysglyserprogluaspasn
128012851290
gluglnlysglnleuphevalgluglnhislyshistyrleuasp
129513001305
gluileilegluglnileserglupheserlysargvalileleu
131013151320
alaaspalaasnleuasplysvalleuseralatyrasnlyshis
132513301335
argasplysproilearggluglnalagluasnileilehisleu
134013451350
phethrleuthrasnleuglyalaproalaalaphelystyrphe
135513601365
aspthrthrileasparglysargtyrthrserthrlysgluval
137013751380
leuaspalathrleuilehisglnserilethrglyleutyrglu
138513901395
thrargileaspleuserglnleuglyglyaspgluglyalapro
140014051410
lyslyslysarglysvalglyserserglyserprolyslyslys
141514201425
arglysvalglyseraspalaleuaspasppheaspleuaspmet
143014351440
leuglyseraspalaleuaspasppheglyglyglysermetasp
144514501455
serleuleumetasnargarglyspheleutyrglnphelysasn
146014651470
valargtrpalalysglyargarggluthrtyrleucystyrval
147514801485
vallysargargaspseralathrserpheserleuaspphegly
149014951500
tyrleuargasnlysasnglycyshisvalgluleuleupheleu
150515101515
argtyrileserasptrpaspleuaspproglyargcystyrarg
152015251530
valthrtrpphethrsertrpserprocystyraspcysalaarg
153515401545
hisvalalaasppheleuargglyasnproasnleuserleuarg
155015551560
ilephethralaargleutyrphecysgluasparglysalaglu
156515701575
progluglyleuargargleuhisargalaglyvalglnileala
158015851590
ilemetthrphelysasptyrphetyrcystrpasnthrpheval
159516001605
gluasnhisgluargthrphelysalatrpgluglyleuhisglu
161016151620
asnservalargleuserargglnleuargargileleuleupro
162516301635
leutyrgluvalaspaspleuargaspalapheargthrtrpgly
164016451650
argasptyrlysaspaspaspasplys
16551660
<210>3
<211>4941
<212>dna
<213>人工序列
<220>
<223>人工序列的描述:dcas9-aidx的编码序列
<400>3
atggactataaggaccacgacggagactacaaggatcatgatattgattacaaagacgat60
gacgataagatggccccaaagaagaagcggaaggtcggtatccacggagtcccagcagct120
accatggacaagaagtattctatcggactggccatcgggactaatagcgtcgggtgggcc180
gtgatcactgacgagtacaaggtgccctctaagaagttcaaggtgctcgggaacaccgac240
cggcattccatcaagaaaaatctgatcggagctctcctctttgattcaggggagaccgct300
gaagcaacccgcctcaagcggactgctagacggcggtacaccaggaggaagaaccggatt360
tgttaccttcaagagatattctccaacgaaatggcaaaggtcgacgacagcttcttccat420
aggctggaagaatcattcctcgtggaagaggataagaagcatgaacggcatcccatcttc480
ggtaatatcgtcgacgaggtggcctatcacgagaaatacccaaccatctaccatcttcgc540
aaaaagctggtggactcaaccgacaaggcagacctccggcttatctacctggccctggcc600
cacatgatcaagttcagaggccacttcctgatcgagggcgacctcaatcctgacaatagc660
gatgtggataaactgttcatccagctggtgcagacttacaaccagctctttgaagagaac720
cccatcaatgcaagcggagtcgatgccaaggccattctgtcagcccggctgtcaaagagc780
cgcagacttgagaatcttatcgctcagctgccgggtgaaaagaaaaatggactgttcggg840
aacctgattgctctttcacttgggctgactcccaatttcaagtctaatttcgacctggca900
gaggatgccaagctgcaactgtccaaggacacctatgatgacgatctcgacaacctcctg960
gcccagatcggtgaccaatacgccgaccttttccttgctgctaagaatctttctgacgcc1020
atcctgctgtctgacattctccgcgtgaacactgaaatcaccaaggcccctctttcagct1080
tcaatgattaagcggtatgatgagcaccaccaggacctgaccctgcttaaggcactcgtc1140
cggcagcagcttccggagaagtacaaggaaatcttctttgaccagtcaaagaatggatac1200
gccggctacatcgacggaggtgcctcccaagaggaattttataagtttatcaaacctatc1260
cttgagaagatggacggcaccgaagagctcctcgtgaaactgaatcgggaggatctgctg1320
cggaagcagcgcactttcgacaatgggagcattccccaccagatccatcttggggagctt1380
cacgccatccttcggcgccaagaggacttctacccctttcttaaggacaacagggagaag1440
attgagaaaattctcactttccgcatcccctactacgtgggacccctcgccagaggaaat1500
agccggtttgcttggatgaccagaaagtcagaagaaactatcactccctggaacttcgaa1560
gaggtggtggacaagggagccagcgctcagtcattcatcgaacggatgactaacttcgat1620
aagaacctccccaatgagaaggtcctgccgaaacattccctgctctacgagtactttacc1680
gtgtacaacgagctgaccaaggtgaaatatgtcaccgaagggatgaggaagcccgcattc1740
ctgtcaggcgaacaaaagaaggcaattgtggaccttctgttcaagaccaatagaaaggtg1800
accgtgaagcagctgaaggaggactatttcaagaaaattgaatgcttcgactctgtggag1860
attagcggggtcgaagatcggttcaacgcaagcctgggtacctaccatgatctgcttaag1920
atcatcaaggacaaggattttctggacaatgaggagaacgaggacatccttgaggacatt1980
gtcctgactctcactctgttcgaggaccgggaaatgatcgaggagaggcttaagacctac2040
gcccatctgttcgacgataaagtgatgaagcaacttaaacggagaagatataccggatgg2100
ggacgccttagccgcaaactcatcaacggaatccgggacaaacagagcggaaagaccatt2160
cttgatttccttaagagcgacggattcgctaatcgcaacttcatgcaacttatccatgat2220
gattccctgacctttaaggaggacatccagaaggcccaagtgtctggacaaggtgactca2280
ctgcacgagcatatcgcaaatctggctggttcacccgctattaagaagggtattctccag2340
accgtgaaagtcgtggacgagctggtcaaggtgatgggtcgccataaaccagagaacatt2400
gtcatcgagatggccagggaaaaccagactacccagaagggacagaagaacagcagggag2460
cggatgaaaagaattgaggaagggattaaggagctcgggtcacagatccttaaagagcac2520
ccggtggaaaacacccagcttcagaatgagaagctctatctgtactaccttcaaaatgga2580
cgcgatatgtatgtggaccaagagcttgatatcaacaggctctcagactacgacgtggac2640
gccatcgtccctcagagcttcctcaaagacgactcaattgacaataaggtgctgactcgc2700
tcagacaagaaccggggaaagtcagataacgtgccctcagaggaagtcgtgaaaaagatg2760
aagaactattggcgccagcttctgaacgcaaagctgatcactcagcggaagttcgacaat2820
ctcactaaggctgagaggggcggactgagcgaactggacaaagcaggattcattaaacgg2880
caacttgtggagactcggcagattactaaacatgtcgcccaaatccttgactcacgcatg2940
aataccaagtacgacgaaaacgacaaacttatccgcgaggtgaaggtgattaccctgaag3000
tccaagctggtcagcgatttcagaaaggactttcaattctacaaagtgcgggagatcaat3060
aactatcatcatgctcatgacgcatatctgaatgccgtggtgggaaccgccctgatcaag3120
aagtacccaaagctggaaagcgagttcgtgtacggagactacaaggtctacgacgtgcgc3180
aagatgattgccaaatctgagcaggagatcggaaaggccaccgcaaagtacttcttctac3240
agcaacatcatgaatttcttcaagaccgaaatcacccttgcaaacggtgagatccggaag3300
aggccgctcatcgagactaatggggagactggcgaaatcgtgtgggacaagggcagagat3360
ttcgctaccgtgcgcaaagtgctttctatgcctcaagtgaacatcgtgaagaaaaccgag3420
gtgcaaaccggaggcttttctaaggaatcaatcctccccaagcgcaactccgacaagctc3480
attgcaaggaagaaggattgggaccctaagaagtacggcggattcgattcaccaactgtg3540
gcttattctgtcctggtcgtggctaaggtggaaaaaggaaagtctaagaagctcaagagc3600
gtgaaggaactgctgggtatcaccattatggagcgcagctccttcgagaagaacccaatt3660
gactttctcgaagccaaaggttacaaggaagtcaagaaggaccttatcatcaagctccca3720
aagtatagcctgttcgaactggagaatgggcggaagcggatgctcgcctccgctggcgaa3780
cttcagaagggtaatgagctggctctcccctccaagtacgtgaatttcctctaccttgca3840
agccattacgagaagctgaaggggagccccgaggacaacgagcaaaagcaactgtttgtg3900
gagcagcataagcattatctggacgagatcattgagcagatttccgagttttctaaacgc3960
gtcattctcgctgatgccaacctcgataaagtccttagcgcatacaataagcacagagac4020
aaaccaattcgggagcaggctgagaatatcatccacctgttcaccctcaccaatcttggt4080
gcccctgccgcattcaagtacttcgacaccaccatcgaccggaaacgctatacctccacc4140
aaagaagtgctggacgccaccctcatccaccagagcatcaccggactttacgaaactcgg4200
attgacctctcacagctcggaggggatgagggagctcccaagaaaaagcgcaaggtaggt4260
agttccggatctccgaaaaagaaacgcaaagttggtagtgatgctttagacgattttgac4320
ttagatatgcttggttcagacgcgttagacgacttcggtggaggatccatggacagcctc4380
ttgatgaaccggaggaagtttctttaccaattcaaaaatgtccgctgggctaagggtcgg4440
cgtgagacctacctgtgctacgtagtgaagaggcgtgacagtgctacatccttttcactg4500
gactttggttatcttcgcaataagaacggctgccacgtggaattgctcttcctccgctac4560
atctcggactgggacctagaccctggccgctgctaccgcgtcacctggttcacctcctgg4620
agcccctgctacgactgtgcccgacatgtggccgactttctgcgagggaaccccaacctc4680
agtctgaggatcttcaccgcgcgcctctacttctgtgaggaccgcaaggctgagcccgag4740
gggctgcggcggctgcaccgcgccggggtgcaaatagccatcatgaccttcaaagattat4800
ttttactgctggaatacttttgtagaaaaccatgaaagaactttcaaagcctgggaaggg4860
ctgcatgaaaattcagttcgtctctccagacagcttcggcgcatccttttgcccgattac4920
aaagacgatgacgataagtga4941
<210>4
<211>1646
<212>prt
<213>人工序列
<220>
<223>人工序列的描述:dcas9-aidx的氨基酸序列
<400>4
metasptyrlysasphisaspglyasptyrlysasphisaspileasp
151015
tyrlysaspaspaspasplysmetalaprolyslyslysarglysval
202530
glyilehisglyvalproalaalathrmetasplyslystyrserile
354045
glyleualaileglythrasnservalglytrpalavalilethrasp
505560
glutyrlysvalproserlyslysphelysvalleuglyasnthrasp
65707580
arghisserilelyslysasnleuileglyalaleuleupheaspser
859095
glygluthralaglualathrargleulysargthralaargargarg
100105110
tyrthrargarglysasnargilecystyrleuglngluilepheser
115120125
asnglumetalalysvalaspaspserphephehisargleugluglu
130135140
serpheleuvalglugluasplyslyshisgluarghisproilephe
145150155160
glyasnilevalaspgluvalalatyrhisglulystyrprothrile
165170175
tyrhisleuarglyslysleuvalaspserthrasplysalaaspleu
180185190
argleuiletyrleualaleualahismetilelyspheargglyhis
195200205
pheleuilegluglyaspleuasnproaspasnseraspvalasplys
210215220
leupheileglnleuvalglnthrtyrasnglnleupheglugluasn
225230235240
proileasnalaserglyvalaspalalysalaileleuseralaarg
245250255
leuserlysserargargleugluasnleuilealaglnleuprogly
260265270
glulyslysasnglyleupheglyasnleuilealaleuserleugly
275280285
leuthrproasnphelysserasnpheaspleualagluaspalalys
290295300
leuglnleuserlysaspthrtyraspaspaspleuaspasnleuleu
305310315320
alaglnileglyaspglntyralaaspleupheleualaalalysasn
325330335
leuseraspalaileleuleuseraspileleuargvalasnthrglu
340345350
ilethrlysalaproleuseralasermetilelysargtyraspglu
355360365
hishisglnaspleuthrleuleulysalaleuvalargglnglnleu
370375380
proglulystyrlysgluilephepheaspglnserlysasnglytyr
385390395400
alaglytyrileaspglyglyalaserglnglugluphetyrlysphe
405410415
ilelysproileleuglulysmetaspglythrglugluleuleuval
420425430
lysleuasnarggluaspleuleuarglysglnargthrpheaspasn
435440445
glyserileprohisglnilehisleuglygluleuhisalaileleu
450455460
argargglngluaspphetyrpropheleulysaspasnargglulys
465470475480
ileglulysileleuthrpheargileprotyrtyrvalglyproleu
485490495
alaargglyasnserargphealatrpmetthrarglyssergluglu
500505510
thrilethrprotrpasnpheglugluvalvalasplysglyalaser
515520525
alaglnserpheilegluargmetthrasnpheasplysasnleupro
530535540
asnglulysvalleuprolyshisserleuleutyrglutyrphethr
545550555560
valtyrasngluleuthrlysvallystyrvalthrgluglymetarg
565570575
lysproalapheleuserglygluglnlyslysalailevalaspleu
580585590
leuphelysthrasnarglysvalthrvallysglnleulysgluasp
595600605
tyrphelyslysileglucyspheaspservalgluileserglyval
610615620
gluaspargpheasnalaserleuglythrtyrhisaspleuleulys
625630635640
ileilelysasplysasppheleuaspasnglugluasngluaspile
645650655
leugluaspilevalleuthrleuthrleuphegluaspargglumet
660665670
ileglugluargleulysthrtyralahisleupheaspasplysval
675680685
metlysglnleulysargargargtyrthrglytrpglyargleuser
690695700
arglysleuileasnglyileargasplysglnserglylysthrile
705710715720
leuasppheleulysseraspglyphealaasnargasnphemetgln
725730735
leuilehisaspaspserleuthrphelysgluaspileglnlysala
740745750
glnvalserglyglnglyaspserleuhisgluhisilealaasnleu
755760765
alaglyserproalailelyslysglyileleuglnthrvallysval
770775780
valaspgluleuvallysvalmetglyarghislysprogluasnile
785790795800
valileglumetalaarggluasnglnthrthrglnlysglyglnlys
805810815
asnserarggluargmetlysargileglugluglyilelysgluleu
820825830
glyserglnileleulysgluhisprovalgluasnthrglnleugln
835840845
asnglulysleutyrleutyrtyrleuglnasnglyargaspmettyr
850855860
valaspglngluleuaspileasnargleuserasptyraspvalasp
865870875880
alailevalproglnserpheleulysaspaspserileaspasnlys
885890895
valleuthrargserasplysasnargglylysseraspasnvalpro
900905910
serglugluvalvallyslysmetlysasntyrtrpargglnleuleu
915920925
asnalalysleuilethrglnarglyspheaspasnleuthrlysala
930935940
gluargglyglyleusergluleuasplysalaglypheilelysarg
945950955960
glnleuvalgluthrargglnilethrlyshisvalalaglnileleu
965970975
aspserargmetasnthrlystyraspgluasnasplysleuilearg
980985990
gluvallysvalilethrleulysserlysleuvalseraspphearg
99510001005
lysasppheglnphetyrlysvalarggluileasnasntyrhis
101010151020
hisalahisaspalatyrleuasnalavalvalglythralaleu
102510301035
ilelyslystyrprolysleuglusergluphevaltyrglyasp
104010451050
tyrlysvaltyraspvalarglysmetilealalysserglugln
105510601065
gluileglylysalathralalystyrphephetyrserasnile
107010751080
metasnphephelysthrgluilethrleualaasnglygluile
108510901095
arglysargproleuilegluthrasnglygluthrglygluile
110011051110
valtrpasplysglyargaspphealathrvalarglysvalleu
111511201125
sermetproglnvalasnilevallyslysthrgluvalglnthr
113011351140
glyglypheserlysgluserileleuprolysargasnserasp
114511501155
lysleuilealaarglyslysasptrpaspprolyslystyrgly
116011651170
glypheaspserprothrvalalatyrservalleuvalvalala
117511801185
lysvalglulysglylysserlyslysleulysservallysglu
119011951200
leuleuglyilethrilemetgluargserserpheglulysasn
120512101215
proileasppheleuglualalysglytyrlysgluvallyslys
122012251230
aspleuileilelysleuprolystyrserleuphegluleuglu
123512401245
asnglyarglysargmetleualaseralaglygluleuglnlys
125012551260
glyasngluleualaleuproserlystyrvalasnpheleutyr
126512701275
leualaserhistyrglulysleulysglyserprogluaspasn
128012851290
gluglnlysglnleuphevalgluglnhislyshistyrleuasp
129513001305
gluileilegluglnileserglupheserlysargvalileleu
131013151320
alaaspalaasnleuasplysvalleuseralatyrasnlyshis
132513301335
argasplysproilearggluglnalagluasnileilehisleu
134013451350
phethrleuthrasnleuglyalaproalaalaphelystyrphe
135513601365
aspthrthrileasparglysargtyrthrserthrlysgluval
137013751380
leuaspalathrleuilehisglnserilethrglyleutyrglu
138513901395
thrargileaspleuserglnleuglyglyaspgluglyalapro
140014051410
lyslyslysarglysvalglyserserglyserprolyslyslys
141514201425
arglysvalglyseraspalaleuaspasppheaspleuaspmet
143014351440
leuglyseraspalaleuaspasppheglyglyglysermetasp
144514501455
serleuleumetasnargarglyspheleutyrglnphelysasn
146014651470
valargtrpalalysglyargarggluthrtyrleucystyrval
147514801485
vallysargargaspseralathrserpheserleuaspphegly
149014951500
tyrleuargasnlysasnglycyshisvalgluleuleupheleu
150515101515
argtyrileserasptrpaspleuaspproglyargcystyrarg
152015251530
valthrtrpphethrsertrpserprocystyraspcysalaarg
153515401545
hisvalalaasppheleuargglyasnproasnleuserleuarg
155015551560
ilephethralaargleutyrphecysgluasparglysalaglu
156515701575
progluglyleuargargleuhisargalaglyvalglnileala
158015851590
ilemetthrphelysasptyrphetyrcystrpasnthrpheval
159516001605
gluasnhisgluargthrphelysalatrpgluglyleuhisglu
161016151620
asnservalargleuserargglnleuargargileleuleupro
162516301635
asptyrlysaspaspaspasplys
16401645
<210>5
<211>28
<212>dna
<213>人工序列
<220>
<223>人工序列的描述:引物
<400>5
gcggatccatggacagcctcttgatgaa28
<210>6
<211>54
<212>dna
<213>人工序列
<220>
<223>人工序列的描述:引物
<400>6
actcgagtcacttatcgtcatcgtctttgtaatcacgtccccaagtacgaaatg54
<210>7
<211>55
<212>dna
<213>人工序列
<220>
<223>人工序列的描述:引物
<400>7
gactcgagtcacttatcgtcatcgtctttgtaatcgggcaaaaggatgcgccgaa55
<210>8
<211>34
<212>dna
<213>人工序列
<220>
<223>人工序列的描述:引物
<400>8
gcagatctaccatggacaagaagtattctatcgg34
<210>9
<211>35
<212>dna
<213>人工序列
<220>
<223>人工序列的描述:引物
<400>9
gactcgagtcacttatcgtcatcgtctttgtaatc35
<210>10
<211>33
<212>dna
<213>人工序列
<220>
<223>人工序列的描述:引物
<400>10
aaactcgagtgtacaaaaaagcaggctttaaag33
<210>11
<211>37
<212>dna
<213>人工序列
<220>
<223>人工序列的描述:引物
<220>
<221>misc_feature
<222>(2)..(20)
<223>n为a,c,g或t
<400>11
gnnnnnnnnnnnnnnnnnnnggtgtttcgtcctttcc37
<210>12
<211>42
<212>dna
<213>人工序列
<220>
<223>人工序列的描述:引物
<220>
<221>misc_feature
<222>(2)..(20)
<223>n为a,c,g或t
<400>12
gnnnnnnnnnnnnnnnnnnngttttagagctagaaatagcaa42
<210>13
<211>36
<212>dna
<213>人工序列
<220>
<223>人工序列的描述:引物
<400>13
aaagctagctaatgccaactttgtacaagaaagctg36
<210>14
<211>21
<212>dna
<213>人工序列
<220>
<223>人工序列的描述:sgrna的靶标结合区
<400>14
gcatgcccgaaggctacgtcc21
<210>15
<211>21
<212>dna
<213>人工序列
<220>
<223>人工序列的描述:sgrna的靶标结合区
<400>15
gcaactagtatacccgcgccg21
<210>16
<211>21
<212>dna
<213>人工序列
<220>
<223>人工序列的描述:sgrna的靶标结合区
<400>16
gcctcgaacttcacctcggcg21
<210>17
<211>21
<212>dna
<213>人工序列
<220>
<223>人工序列的描述:sgrna的靶标结合区
<400>17
gtcagctcgatgcggttcacc21
<210>18
<211>21
<212>dna
<213>人工序列
<220>
<223>人工序列的描述:sgrna的靶标结合区
<400>18
gattcccagggccggttaatg21
<210>19
<211>20
<212>dna
<213>人工序列
<220>
<223>人工序列的描述:sgrna的靶标结合区
<400>19
gtcccctccaccccacagtg20
<210>20
<211>20
<212>dna
<213>人工序列
<220>
<223>人工序列的描述:sgrna的靶标结合区
<400>20
ggggccactagggacaggat20
<210>21
<211>21
<212>prt
<213>人工序列
<220>
<223>人工序列的描述:接头
<400>21
glyserglyglyglyglyserglyglyglyglyserglyglyglyleu
151015
glyserthrgluphe
20
<210>22
<211>21
<212>prt
<213>人工序列
<220>
<223>人工序列的描述:接头
<400>22
argserthrserglyleuglyglyglyserglyglyglyglysergly
151015
glyglyglysergly
20
<210>23
<211>21
<212>prt
<213>人工序列
<220>
<223>人工序列的描述:接头
<400>23
glnleuthrserglyleuglyglyglyserglyglyglyglysergly
151015
glyglyglysergly
20
<210>24
<211>4
<212>prt
<213>人工序列
<220>
<223>人工序列的描述:接头
<400>24
glyglyglyser
1
<210>25
<211>5
<212>prt
<213>人工序列
<220>
<223>人工序列的描述:接头
<400>25
glyglyglyglyser
15
<210>26
<211>5
<212>prt
<213>人工序列
<220>
<223>人工序列的描述:接头
<400>26
sersersersergly
15
<210>27
<211>5
<212>prt
<213>人工序列
<220>
<223>人工序列的描述:接头
<400>27
glyserglyserala
15
<210>28
<211>20
<212>prt
<213>人工序列
<220>
<223>人工序列的描述:接头
<400>28
glyglyserglyglyglyglyglyglyserglyglyglyglysergly
151015
glyglyglyser
20
<210>29
<211>15
<212>prt
<213>人工序列
<220>
<223>人工序列的描述:接头
<400>29
serserserserglyserserserserglysersersersergly
151015
<210>30
<211>15
<212>prt
<213>人工序列
<220>
<223>人工序列的描述:接头
<400>30
glyserglyseralaglyserglyseralaglyserglyserala
151015
<210>31
<211>15
<212>prt
<213>人工序列
<220>
<223>人工序列的描述:接头
<400>31
glyglyserglyglyglyglyserglyglyglyglyserglygly
151015
<210>32
<211>8
<212>prt
<213>人工序列
<220>
<223>人工序列的描述:flag标签
<400>32
asptyrlysaspaspaspasplys
15
<210>33
<211>7
<212>prt
<213>人工序列
<220>
<223>人工序列的描述:核定位序列
<400>33
prolyslyslysarglysval
15
<210>34
<211>16
<212>prt
<213>人工序列
<220>
<223>人工序列的描述:核定位序列
<400>34
lysargproalaalathrlyslysalaglyglnalalyslyslyslys
151015
<210>35
<211>9
<212>prt
<213>人工序列
<220>
<223>人工序列的描述:核定位序列
<400>35
proalaalalysargvallysleuasp
15
<210>36
<211>11
<212>prt
<213>人工序列
<220>
<223>人工序列的描述:核定位序列
<400>36
argglnargargasngluleulysargserpro
1510
<210>37
<211>38
<212>prt
<213>人工序列
<220>
<223>人工序列的描述:核定位序列
<400>37
asnglnserserasnpheglyprometlysglyglyasnpheglygly
151015
argserserglyprotyrglyglyglyglyglntyrphealalyspro
202530
argasnglnglyglytyr
35
<210>38
<211>42
<212>prt
<213>人工序列
<220>
<223>人工序列的描述:核定位序列
<400>38
argmetargileglxphelysasnlysglylysaspthralagluleu
151015
argargargargvalgluvalservalgluleuarglysalalyslys
202530
aspgluglnileleulysargargasnval
3540
<210>39
<211>8
<212>prt
<213>人工序列
<220>
<223>人工序列的描述:核定位序列
<400>39
valserarglysargproargpro
15
<210>40
<211>8
<212>prt
<213>人工序列
<220>
<223>人工序列的描述:核定位序列
<400>40
proprolyslysalaarggluasp
15
<210>41
<211>12
<212>prt
<213>人工序列
<220>
<223>人工序列的描述:核定位序列
<400>41
seralaleuilelyslyslyslyslysmetalapro
1510
<210>42
<211>5
<212>prt
<213>人工序列
<220>
<223>人工序列的描述:核定位序列
<400>42
aspargleuargarg
15
<210>43
<211>7
<212>prt
<213>人工序列
<220>
<223>人工序列的描述:核定位序列
<400>43
prolysglnlyslysarglys
15
<210>44
<211>10
<212>prt
<213>人工序列
<220>
<223>人工序列的描述:核定位序列
<400>44
arglysleulyslyslysilelyslysleu
1510
<210>45
<211>10
<212>prt
<213>人工序列
<220>
<223>人工序列的描述:核定位序列
<400>45
argglulyslyslyspheleulysargarg
1510
<210>46
<211>20
<212>prt
<213>人工序列
<220>
<223>人工序列的描述:核定位序列
<400>46
lysarglysglyaspgluvalaspglyvalaspgluvalalalyslys
151015
lysserlyslys
20
<210>47
<211>17
<212>prt
<213>人工序列
<220>
<223>人工序列的描述:核定位序列
<400>47
arglyscysleuglnalaglymetasnleuglualaarglysthrlys
151015
lys
<210>48
<211>644
<212>dna
<213>智人(homosapiens)
<400>48
acaagttcagcgtgtctggcgagggcgagggcgatgccacctacggcaagctgaccctga60
agttcatctgcaccaccggcaagctgcccgtgccctggcccaccctcgtgaccaccctga120
cctacggcgtgcagtgcttcagccgctaccccgaccacatgaagcagcacgacttcttca180
agtccgccatgcccgaaggctacgtccaggagcgcaccatcttcttcaaggacgacggca240
actagtatacccgcgccgaggtgaagttcgagggcgacaccctggtgaaccgcatcgagc300
tgaagggcatcgacttcaaggaggacggcaacatcctggggcacaagctggagtacaact360
acaacagccacaacgtctatatcatggccgacaagcagaagaacggcatcaaggcgaact420
tcaagatccgccacaacatcgaggacggcagcgtgcagctcgccgaccactaccagcaga480
acacccccatcggcgacggccccgtgctgctgcccgacaaccactacctgagcacccagt540
ccgccctgagcaaagaccccaacgagaagcgcgatcacatggtcctgctggagttcgtga600
ccgccgccgggatcactctcggcatggacgagctgtacaagtaa644
<210>49
<211>20
<212>dna
<213>人工序列
<220>
<223>人工序列的描述:sgrna的靶标结合区
<400>49
tagacagttgtttgttcagt20
<210>50
<211>20
<212>dna
<213>人工序列
<220>
<223>人工序列的描述:sgrna的靶标结合区
<400>50
gtcctcgttgtcttgttggc20
<210>51
<211>20
<212>dna
<213>人工序列
<220>
<223>人工序列的描述:sgrna的靶标结合区
<400>51
gttggcaggggtctgcaccc20
<210>52
<211>20
<212>dna
<213>人工序列
<220>
<223>人工序列的描述:sgrna的靶标结合区
<400>52
tcactgagttcatgacctac20
<210>53
<211>20
<212>dna
<213>人工序列
<220>
<223>人工序列的描述:sgrna的靶标结合区
<400>53
catgacctacgggaacctcc20
<210>54
<211>20
<212>dna
<213>人工序列
<220>
<223>人工序列的描述:sgrna的靶标结合区
<400>54
cctgagggagtgcaaccggc20
<210>55
<211>20
<212>dna
<213>人工序列
<220>
<223>人工序列的描述:sgrna的靶标结合区
<400>55
ccggcaggaggtgaacgccg20
<210>56
<211>20
<212>dna
<213>人工序列
<220>
<223>人工序列的描述:sgrna的靶标结合区
<400>56
cgccgtggtgctgctgtaca20
<210>57
<211>20
<212>dna
<213>人工序列
<220>
<223>人工序列的描述:sgrna的靶标结合区
<400>57
ctcgtcagccatggagtacc20
<210>58
<211>20
<212>dna
<213>人工序列
<220>
<223>人工序列的描述:sgrna的靶标结合区
<400>58
aaaaacttcatccacaggta20
<210>59
<211>20
<212>dna
<213>人工序列
<220>
<223>人工序列的描述:sgrna的靶标结合区
<400>59
agcctgcgccatggagtcac20
<210>60
<211>20
<212>dna
<213>人工序列
<220>
<223>人工序列的描述:sgrna的靶标结合区
<400>60
ggagtcacagggcgtggagc20
<210>61
<211>20
<212>dna
<213>人工序列
<220>
<223>人工序列的描述:sgrna的靶标结合区
<400>61
acaacgaggacttcaacacg20
<210>62
<211>20
<212>dna
<213>人工序列
<220>
<223>人工序列的描述:sgrna的靶标结合区
<400>62
tcagtgatgatatagaacgg20
<210>63
<211>20
<212>dna
<213>人工序列
<220>
<223>人工序列的描述:sgrna的靶标结合区
<400>63
tgcactccctcaggtagtcc20
<210>64
<211>20
<212>dna
<213>人工序列
<220>
<223>人工序列的描述:sgrna的靶标结合区
<400>64
gccctgtgactccatggcgc20
<210>65
<211>4731
<212>dna
<213>人工序列
<220>
<223>人工序列的描述:aidx-xten-dcas9的编码序列
<400>65
atggacagcctcttgatgaaccggaggaagtttctttaccaattcaaaaatgtccgctgg60
gctaagggtcggcgtgagacctacctgtgctacgtagtgaagaggcgtgacagtgctaca120
tccttttcactggactttggttatcttcgcaataagaacggctgccacgtggaattgctc180
ttcctccgctacatctcggactgggacctagaccctggccgctgctaccgcgtcacctgg240
ttcacctcctggagcccctgctacgactgtgcccgacatgtggccgactttctgcgaggg300
aaccccaacctcagtctgaggatcttcaccgcgcgcctctacttctgtgaggaccgcaag360
gctgagcccgaggggctgcggcggctgcaccgcgccggggtgcaaatagccatcatgacc420
ttcaaagattatttttactgctggaatacttttgtagaaaaccatgaaagaactttcaaa480
gcctgggaagggctgcatgaaaattcagttcgtctctccagacagcttcggcgcatcctt540
ttgcccagcggcagcgagactcccgggacctcagagtccgccacacccgaaagtgataaa600
aagtattctattggtttagccatcggcactaattccgttggatgggctgtcataaccgat660
gaatacaaagtaccttcaaagaaatttaaggtgttggggaacacagaccgtcattcgatt720
aaaaagaatcttatcggtgccctcctattcgatagtggcgaaacggcagaggcgactcgc780
ctgaaacgaaccgctcggagaaggtatacacgtcgcaagaaccgaatatgttacttacaa840
gaaatttttagcaatgagatggccaaagttgacgattctttctttcaccgtttggaagag900
tccttccttgtcgaagaggacaagaaacatgaacggcaccccatctttggaaacatagta960
gatgaggtggcatatcatgaaaagtacccaacgatttatcacctcagaaaaaagctagtt1020
gactcaactgataaagcggacctgaggttaatctacttggctcttgcccatatgataaag1080
ttccgtgggcactttctcattgagggtgatctaaatccggacaactcggatgtcgacaaa1140
ctgttcatccagttagtacaaacctataatcagttgtttgaagagaaccctataaatgca1200
agtggcgtggatgcgaaggctattcttagcgcccgcctctctaaatcccgacggctagaa1260
aacctgatcgcacaattacccggagagaagaaaaatgggttgttcggtaaccttatagcg1320
ctctcactaggcctgacaccaaattttaagtcgaacttcgacttagctgaagatgccaaa1380
ttgcagcttagtaaggacacgtacgatgacgatctcgacaatctactggcacaaattgga1440
gatcagtatgcggacttatttttggctgccaaaaaccttagcgatgcaatcctcctatct1500
gacatactgagagttaatactgagattaccaaggcgccgttatccgcttcaatgatcaaa1560
aggtacgatgaacatcaccaagacttgacacttctcaaggccctagtccgtcagcaactg1620
cctgagaaatataaggaaatattctttgatcagtcgaaaaacgggtacgcaggttatatt1680
gacggcggagcgagtcaagaggaattctacaagtttatcaaacccatattagagaagatg1740
gatgggacggaagagttgcttgtaaaactcaatcgcgaagatctactgcgaaagcagcgg1800
actttcgacaacggtagcattccacatcaaatccacttaggcgaattgcatgctatactt1860
agaaggcaggaggatttttatccgttcctcaaagacaatcgtgaaaagattgagaaaatc1920
ctaacctttcgcataccttactatgtgggacccctggcccgagggaactctcggttcgca1980
tggatgacaagaaagtccgaagaaacgattactccatggaattttgaggaagttgtcgat2040
aaaggtgcgtcagctcaatcgttcatcgagaggatgaccaactttgacaagaatttaccg2100
aacgaaaaagtattgcctaagcacagtttactttacgagtatttcacagtgtacaatgaa2160
ctcacgaaagttaagtatgtcactgagggcatgcgtaaacccgcctttctaagcggagaa2220
cagaagaaagcaatagtagatctgttattcaagaccaaccgcaaagtgacagttaagcaa2280
ttgaaagaggactactttaagaaaattgaatgcttcgattctgtcgagatctccggggta2340
gaagatcgatttaatgcgtcacttggtacgtatcatgacctcctaaagataattaaagat2400
aaggacttcctggataacgaagagaatgaagatatcttagaagatatagtgttgactctt2460
accctctttgaagatcgggaaatgattgaggaaagactaaaaacatacgctcacctgttc2520
gacgataaggttatgaaacagttaaagaggcgtcgctatacgggctggggacgattgtcg2580
cggaaacttatcaacgggataagagacaagcaaagtggtaaaactattctcgattttcta2640
aagagcgacggcttcgccaataggaactttatgcagctgatccatgatgactctttaacc2700
ttcaaagaggatatacaaaaggcacaggtttccggacaaggggactcattgcacgaacat2760
attgcgaatcttgctggttcgccagccatcaaaaagggcatactccagacagtcaaagta2820
gtggatgagctagttaaggtcatgggacgtcacaaaccggaaaacattgtaatcgagatg2880
gcacgcgaaaatcaaacgactcagaaggggcaaaaaaacagtcgagagcggatgaagaga2940
atagaagagggtattaaagaactgggcagccagatcttaaaggagcatcctgtggaaaat3000
acccaattgcagaacgagaaactttacctctattacctacaaaatggaagggacatgtat3060
gttgatcaggaactggacataaaccgtttatctgattacgacgtcgatgccattgtaccc3120
caatcctttttgaaggacgattcaatcgacaataaagtgcttacacgctcggataagaac3180
cgagggaaaagtgacaatgttccaagcgaggaagtcgtaaagaaaatgaagaactattgg3240
cggcagctcctaaatgcgaaactgataacgcaaagaaagttcgataacttaactaaagct3300
gagaggggtggcttgtctgaacttgacaaggccggatttattaaacgtcagctcgtggaa3360
acccgccaaatcacaaagcatgttgcacagatactagattcccgaatgaatacgaaatac3420
gacgagaacgataagctgattcgggaagtcaaagtaatcactttaaagtcaaaattggtg3480
tcggacttcagaaaggattttcaattctataaagttagggagataaataactaccaccat3540
gcgcacgacgcttatcttaatgccgtcgtagggaccgcactcattaagaaatacccgaag3600
ctagaaagtgagtttgtgtatggtgattacaaagtttatgacgtccgtaagatgatcgcg3660
aaaagcgaacaggagataggcaaggctacagccaaatacttcttttattctaacattatg3720
aatttctttaagacggaaatcactctggcaaacggagagatacgcaaacgacctttaatt3780
gaaaccaatggggagacaggtgaaatcgtatgggataagggccgggacttcgcgacggtg3840
agaaaagttttgtccatgccccaagtcaacatagtaaagaaaactgaggtgcagaccgga3900
gggttttcaaaggaatcgattcttccaaaaaggaatagtgataagctcatcgctcgtaaa3960
aaggactgggacccgaaaaagtacggtggcttcgatagccctacagttgcctattctgtc4020
ctagtagtggcaaaagttgagaagggaaaatccaagaaactgaagtcagtcaaagaatta4080
ttggggataacgattatggagcgctcgtcttttgaaaagaaccccatcgacttccttgag4140
gcgaaaggttacaaggaagtaaaaaaggatctcataattaaactaccaaagtatagtctg4200
tttgagttagaaaatggccgaaaacggatgttggctagcgccggagagcttcaaaagggg4260
aacgaactcgcactaccgtctaaatacgtgaatttcctgtatttagcgtcccattacgag4320
aagttgaaaggttcacctgaagataacgaacagaagcaactttttgttgagcagcacaaa4380
cattatctcgacgaaatcatagagcaaatttcggaattcagtaagagagtcatcctagct4440
gatgccaatctggacaaagtattaagcgcatacaacaagcacagggataaacccatacgt4500
gagcaggcggaaaatattatccatttgtttactcttaccaacctcggcgctccagccgca4560
ttcaagtattttgacacaacgatagatcgcaaacgatacacttctaccaaggaggtgcta4620
gacgcgacactgattcaccaatccatcacgggattatatgaaactcggatagatttgtca4680
cagcttgggggtgactctggtggttctcccaagaagaagaggaaagtctaa4731
<210>66
<211>1576
<212>prt
<213>人工序列
<220>
<223>人工序列的描述:aidx-xten-dcas9的氨基酸序列
<400>66
metaspserleuleumetasnargarglyspheleutyrglnphelys
151015
asnvalargtrpalalysglyargarggluthrtyrleucystyrval
202530
vallysargargaspseralathrserpheserleuasppheglytyr
354045
leuargasnlysasnglycyshisvalgluleuleupheleuargtyr
505560
ileserasptrpaspleuaspproglyargcystyrargvalthrtrp
65707580
phethrsertrpserprocystyraspcysalaarghisvalalaasp
859095
pheleuargglyasnproasnleuserleuargilephethralaarg
100105110
leutyrphecysgluasparglysalagluprogluglyleuargarg
115120125
leuhisargalaglyvalglnilealailemetthrphelysasptyr
130135140
phetyrcystrpasnthrphevalgluasnhisgluargthrphelys
145150155160
alatrpgluglyleuhisgluasnservalargleuserargglnleu
165170175
argargileleuleuproserglysergluthrproglythrserglu
180185190
seralathrprogluserasplyslystyrserileglyleualaile
195200205
glythrasnservalglytrpalavalilethraspglutyrlysval
210215220
proserlyslysphelysvalleuglyasnthrasparghisserile
225230235240
lyslysasnleuileglyalaleuleupheaspserglygluthrala
245250255
glualathrargleulysargthralaargargargtyrthrargarg
260265270
lysasnargilecystyrleuglngluilepheserasnglumetala
275280285
lysvalaspaspserphephehisargleuglugluserpheleuval
290295300
glugluasplyslyshisgluarghisproilepheglyasnileval
305310315320
aspgluvalalatyrhisglulystyrprothriletyrhisleuarg
325330335
lyslysleuvalaspserthrasplysalaaspleuargleuiletyr
340345350
leualaleualahismetilelyspheargglyhispheleuileglu
355360365
glyaspleuasnproaspasnseraspvalasplysleupheilegln
370375380
leuvalglnthrtyrasnglnleupheglugluasnproileasnala
385390395400
serglyvalaspalalysalaileleuseralaargleuserlysser
405410415
argargleugluasnleuilealaglnleuproglyglulyslysasn
420425430
glyleupheglyasnleuilealaleuserleuglyleuthrproasn
435440445
phelysserasnpheaspleualagluaspalalysleuglnleuser
450455460
lysaspthrtyraspaspaspleuaspasnleuleualaglnilegly
465470475480
aspglntyralaaspleupheleualaalalysasnleuseraspala
485490495
ileleuleuseraspileleuargvalasnthrgluilethrlysala
500505510
proleuseralasermetilelysargtyraspgluhishisglnasp
515520525
leuthrleuleulysalaleuvalargglnglnleuproglulystyr
530535540
lysgluilephepheaspglnserlysasnglytyralaglytyrile
545550555560
aspglyglyalaserglnglugluphetyrlyspheilelysproile
565570575
leuglulysmetaspglythrglugluleuleuvallysleuasnarg
580585590
gluaspleuleuarglysglnargthrpheaspasnglyserilepro
595600605
hisglnilehisleuglygluleuhisalaileleuargargglnglu
610615620
aspphetyrpropheleulysaspasnargglulysileglulysile
625630635640
leuthrpheargileprotyrtyrvalglyproleualaargglyasn
645650655
serargphealatrpmetthrarglysserglugluthrilethrpro
660665670
trpasnpheglugluvalvalasplysglyalaseralaglnserphe
675680685
ilegluargmetthrasnpheasplysasnleuproasnglulysval
690695700
leuprolyshisserleuleutyrglutyrphethrvaltyrasnglu
705710715720
leuthrlysvallystyrvalthrgluglymetarglysproalaphe
725730735
leuserglygluglnlyslysalailevalaspleuleuphelysthr
740745750
asnarglysvalthrvallysglnleulysgluasptyrphelyslys
755760765
ileglucyspheaspservalgluileserglyvalgluaspargphe
770775780
asnalaserleuglythrtyrhisaspleuleulysileilelysasp
785790795800
lysasppheleuaspasnglugluasngluaspileleugluaspile
805810815
valleuthrleuthrleuphegluaspargglumetileglugluarg
820825830
leulysthrtyralahisleupheaspasplysvalmetlysglnleu
835840845
lysargargargtyrthrglytrpglyargleuserarglysleuile
850855860
asnglyileargasplysglnserglylysthrileleuasppheleu
865870875880
lysseraspglyphealaasnargasnphemetglnleuilehisasp
885890895
aspserleuthrphelysgluaspileglnlysalaglnvalsergly
900905910
glnglyaspserleuhisgluhisilealaasnleualaglyserpro
915920925
alailelyslysglyileleuglnthrvallysvalvalaspgluleu
930935940
vallysvalmetglyarghislysprogluasnilevalileglumet
945950955960
alaarggluasnglnthrthrglnlysglyglnlysasnserargglu
965970975
argmetlysargileglugluglyilelysgluleuglyserglnile
980985990
leulysgluhisprovalgluasnthrglnleuglnasnglulysleu
99510001005
tyrleutyrtyrleuglnasnglyargaspmettyrvalaspgln
101010151020
gluleuaspileasnargleuserasptyraspvalaspalaile
102510301035
valproglnserpheleulysaspaspserileaspasnlysval
104010451050
leuthrargserasplysasnargglylysseraspasnvalpro
105510601065
serglugluvalvallyslysmetlysasntyrtrpargglnleu
107010751080
leuasnalalysleuilethrglnarglyspheaspasnleuthr
108510901095
lysalagluargglyglyleusergluleuasplysalaglyphe
110011051110
ilelysargglnleuvalgluthrargglnilethrlyshisval
111511201125
alaglnileleuaspserargmetasnthrlystyraspgluasn
113011351140
asplysleuilearggluvallysvalilethrleulysserlys
114511501155
leuvalseraspphearglysasppheglnphetyrlysvalarg
116011651170
gluileasnasntyrhishisalahisaspalatyrleuasnala
117511801185
valvalglythralaleuilelyslystyrprolysleugluser
119011951200
gluphevaltyrglyasptyrlysvaltyraspvalarglysmet
120512101215
ilealalyssergluglngluileglylysalathralalystyr
122012251230
phephetyrserasnilemetasnphephelysthrgluilethr
123512401245
leualaasnglygluilearglysargproleuilegluthrasn
125012551260
glygluthrglygluilevaltrpasplysglyargasppheala
126512701275
thrvalarglysvalleusermetproglnvalasnilevallys
128012851290
lysthrgluvalglnthrglyglypheserlysgluserileleu
129513001305
prolysargasnserasplysleuilealaarglyslysasptrp
131013151320
aspprolyslystyrglyglypheaspserprothrvalalatyr
132513301335
servalleuvalvalalalysvalglulysglylysserlyslys
134013451350
leulysservallysgluleuleuglyilethrilemetgluarg
135513601365
serserpheglulysasnproileasppheleuglualalysgly
137013751380
tyrlysgluvallyslysaspleuileilelysleuprolystyr
138513901395
serleuphegluleugluasnglyarglysargmetleualaser
140014051410
alaglygluleuglnlysglyasngluleualaleuproserlys
141514201425
tyrvalasnpheleutyrleualaserhistyrglulysleulys
143014351440
glyserprogluaspasngluglnlysglnleuphevalglugln
144514501455
hislyshistyrleuaspgluileilegluglnilesergluphe
146014651470
serlysargvalileleualaaspalaasnleuasplysvalleu
147514801485
seralatyrasnlyshisargasplysproilearggluglnala
149014951500
gluasnileilehisleuphethrleuthrasnleuglyalapro
150515101515
alaalaphelystyrpheaspthrthrileasparglysargtyr
152015251530
thrserthrlysgluvalleuaspalathrleuilehisglnser
153515401545
ilethrglyleutyrgluthrargileaspleuserglnleugly
155015551560
glyaspserglyglyserprolyslyslysarglysval
156515701575
<210>67
<211>4890
<212>dna
<213>人工序列
<220>
<223>人工序列的描述:dcas9-xten-aidx(k10et82ie156g)的编码序列
<400>67
atggactataaggaccacgacggagactacaaggatcatgatattgattacaaagacgat60
gacgataagatggccccaaagaagaagcggaaggtcggtatccacggagtcccagcagct120
accatggacaagaagtattctatcggactggccatcgggactaatagcgtcgggtgggcc180
gtgatcactgacgagtacaaggtgccctctaagaagttcaaggtgctcgggaacaccgac240
cggcattccatcaagaaaaatctgatcggagctctcctctttgattcaggggagaccgct300
gaagcaacccgcctcaagcggactgctagacggcggtacaccaggaggaagaaccggatt360
tgttaccttcaagagatattctccaacgaaatggcaaaggtcgacgacagcttcttccat420
aggctggaagaatcattcctcgtggaagaggataagaagcatgaacggcatcccatcttc480
ggtaatatcgtcgacgaggtggcctatcacgagaaatacccaaccatctaccatcttcgc540
aaaaagctggtggactcaaccgacaaggcagacctccggcttatctacctggccctggcc600
cacatgatcaagttcagaggccacttcctgatcgagggcgacctcaatcctgacaatagc660
gatgtggataaactgttcatccagctggtgcagacttacaaccagctctttgaagagaac720
cccatcaatgcaagcggagtcgatgccaaggccattctgtcagcccggctgtcaaagagc780
cgcagacttgagaatcttatcgctcagctgccgggtgaaaagaaaaatggactgttcggg840
aacctgattgctctttcacttgggctgactcccaatttcaagtctaatttcgacctggca900
gaggatgccaagctgcaactgtccaaggacacctatgatgacgatctcgacaacctcctg960
gcccagatcggtgaccaatacgccgaccttttccttgctgctaagaatctttctgacgcc1020
atcctgctgtctgacattctccgcgtgaacactgaaatcaccaaggcccctctttcagct1080
tcaatgattaagcggtatgatgagcaccaccaggacctgaccctgcttaaggcactcgtc1140
cggcagcagcttccggagaagtacaaggaaatcttctttgaccagtcaaagaatggatac1200
gccggctacatcgacggaggtgcctcccaagaggaattttataagtttatcaaacctatc1260
cttgagaagatggacggcaccgaagagctcctcgtgaaactgaatcgggaggatctgctg1320
cggaagcagcgcactttcgacaatgggagcattccccaccagatccatcttggggagctt1380
cacgccatccttcggcgccaagaggacttctacccctttcttaaggacaacagggagaag1440
attgagaaaattctcactttccgcatcccctactacgtgggacccctcgccagaggaaat1500
agccggtttgcttggatgaccagaaagtcagaagaaactatcactccctggaacttcgaa1560
gaggtggtggacaagggagccagcgctcagtcattcatcgaacggatgactaacttcgat1620
aagaacctccccaatgagaaggtcctgccgaaacattccctgctctacgagtactttacc1680
gtgtacaacgagctgaccaaggtgaaatatgtcaccgaagggatgaggaagcccgcattc1740
ctgtcaggcgaacaaaagaaggcaattgtggaccttctgttcaagaccaatagaaaggtg1800
accgtgaagcagctgaaggaggactatttcaagaaaattgaatgcttcgactctgtggag1860
attagcggggtcgaagatcggttcaacgcaagcctgggtacctaccatgatctgcttaag1920
atcatcaaggacaaggattttctggacaatgaggagaaagaggacatccttgaggacatt1980
gtcctgactctcactctgttcgaggaccgggaaatgatcgaggagaggcttaagacctac2040
gcccatctgttcgacgataaagtgatgaagcaacttaaacggagaagatataccggatgg2100
ggacgccttagccgcaaactcatcaacggaatccgggacaaacagagcggaaagaccatt2160
cttgatttccttaagagcgacggattcgctaatcgcaacttcatgcaacttatccatgat2220
gattccctgacctttaaggaggacatccagaaggcccaagtgtctggacaaggtgactca2280
ctgcacgagcatatcgcaaatctggctggttcacccgctattaagaagggtattctccag2340
accgtgaaagtcgtggacgagctggtcaaggtgatgggtcgccataaaccagagaacatt2400
gtcatcgagatggccagggaaaaccagactacccagaagggacagaagaacagcagggag2460
cggatgaaaagaattgaggaagggattaaggagctcgggtcacagatccttaaagagcac2520
ccggtggaaaacacccagcttcagaatgagaagctctatctgtactaccttcaaaatgga2580
cgcgatatgtatgtggaccaagagcttgatatcaacaggctctcagactacgacgtggac2640
gccatcgtccctcagagcttcctcaaagacgactcaattgacaataaggtgctgactcgc2700
tcagacaagaaccggggaaagtcagataacgtgccctcagaggaagtcgtgaaaaagatg2760
aagaactattggcgccagcttctgaacgcaaagctgatcactcagcggaagttcgacaat2820
ctcactaaggctgagaggggcggactgagcgaactggacaaagcaggattcattaaacgg2880
caacttgtggagactcggcagattactaaacatgtagcccaaatccttgactcacgcatg2940
aataccaagtacgacgaaaacgacaaacttatccgcgaggtgaaggtgattaccctgaag3000
tccaagctggtcagcgatttcagaaaggactttcaattctacaaagtgcgggagatcaat3060
aactatcatcatgctcatgacgcatatctgaatgccgtggtgggaaccgccctgatcaag3120
aagtacccaaagctggaaagcgagttcgtgtacggagactacaaggtctacgacgtgcgc3180
aagatgattgccaaatctgagcaggagatcggaaaggccaccgcaaagtacttcttctac3240
agcaacatcatgaatttcttcaagaccgaaatcacccttgcaaacggtgagatccggaag3300
aggccgctcatcgagactaatggggagactggcgaaatcgtgtgggacaagggcagagat3360
ttcgctaccgtgcgcaaagtgctttctatgcctcaagtgaacatcgtgaagaaaaccgag3420
gtgcaaaccggaggcttttctaaggaatcaatcctccccaagcgcaactccgacaagctc3480
attgcaaggaagaaggattgggaccctaagaagtacggcggattcgattcaccaactgtg3540
gcttattctgtcctggtcgtggctaaggtggaaaaaggaaagtctaagaagctcaagagc3600
gtgaaggaactgctgggtatcaccattatggagcgcagctccttcgagaagaacccaatt3660
gactttctcgaagccaaaggttacaaggaagtcaagaaggaccttatcatcaagctccca3720
aagtatagcctgttcgaactggagaatgggcggaagcggatgctcgcctccgctggcgaa3780
cttcagaagggtaatgagctggctctcccctccaagtacgtgaatttcctctaccttgca3840
agccattacgagaagctgaaggggagccccgaggacaacgagcaaaagcaactgtttgtg3900
gagcagcataagcattatctggacgagatcattgagcagatttccgagttttctaaacgc3960
gtcattctcgctgatgccaacctcgataaagtccttagcgcatacaataagcacagagac4020
aaaccaattcgggagcaggctgagaatatcatccacctgttcaccctcaccaatcttggt4080
gcccctgccgcattcaagtacttcgacaccaccatcgaccggaaacgctatacctccacc4140
aaagaagtgctggacgccaccctcatccaccagagcatcaccggactttacgaaactcgg4200
attgacctctcacagctcggaggggatgagggagctcccaagaaaaagcgcaaggtaggt4260
agttccggatctccgaaaaagaaacgcaaagttagcggcagcgagactcccgggacctca4320
gagtccgccacacccgaaagtatggacagcctcttgatgaaccggagggagtttctttac4380
caattcaaaaatgtccgctgggctaagggtcggcgtgagacctacctgtgctacgtagtg4440
aagaggcgtgacagtgctacatccttttcactggactttggttatcttcgcaataagaac4500
ggctgccacgtggaattgctcttcctccgctacatctcggactgggacctagaccctggc4560
cgctgctaccgcgtcacctggttcatctcctggagcccctgctacgactgtgcccgacat4620
gtggccgactttctgcgagggaaccccaacctcagtctgaggatcttcaccgcgcgcctc4680
tacttctgtgaggaccgcaaggctgagcccgaggggctgcggcggctgcaccgcgccggg4740
gtgcaaatagccatcatgaccttcaaagattatttttactgctggaatacttttgtagaa4800
aaccatggaagaactttcaaagcctgggaagggctgcatgaaaattcagttcgtctctcc4860
agacagcttcggcgcatccttttgccctga4890
<210>68
<211>1629
<212>prt
<213>人工序列
<220>
<223>人工序列的描述:dcas9-xten-aidx(k10et82ie156g)的氨基酸序列
<400>68
metasptyrlysasphisaspglyasptyrlysasphisaspileasp
151015
tyrlysaspaspaspasplysmetalaprolyslyslysarglysval
202530
glyilehisglyvalproalaalathrmetasplyslystyrserile
354045
glyleualaileglythrasnservalglytrpalavalilethrasp
505560
glutyrlysvalproserlyslysphelysvalleuglyasnthrasp
65707580
arghisserilelyslysasnleuileglyalaleuleupheaspser
859095
glygluthralaglualathrargleulysargthralaargargarg
100105110
tyrthrargarglysasnargilecystyrleuglngluilepheser
115120125
asnglumetalalysvalaspaspserphephehisargleugluglu
130135140
serpheleuvalglugluasplyslyshisgluarghisproilephe
145150155160
glyasnilevalaspgluvalalatyrhisglulystyrprothrile
165170175
tyrhisleuarglyslysleuvalaspserthrasplysalaaspleu
180185190
argleuiletyrleualaleualahismetilelyspheargglyhis
195200205
pheleuilegluglyaspleuasnproaspasnseraspvalasplys
210215220
leupheileglnleuvalglnthrtyrasnglnleupheglugluasn
225230235240
proileasnalaserglyvalaspalalysalaileleuseralaarg
245250255
leuserlysserargargleugluasnleuilealaglnleuprogly
260265270
glulyslysasnglyleupheglyasnleuilealaleuserleugly
275280285
leuthrproasnphelysserasnpheaspleualagluaspalalys
290295300
leuglnleuserlysaspthrtyraspaspaspleuaspasnleuleu
305310315320
alaglnileglyaspglntyralaaspleupheleualaalalysasn
325330335
leuseraspalaileleuleuseraspileleuargvalasnthrglu
340345350
ilethrlysalaproleuseralasermetilelysargtyraspglu
355360365
hishisglnaspleuthrleuleulysalaleuvalargglnglnleu
370375380
proglulystyrlysgluilephepheaspglnserlysasnglytyr
385390395400
alaglytyrileaspglyglyalaserglnglugluphetyrlysphe
405410415
ilelysproileleuglulysmetaspglythrglugluleuleuval
420425430
lysleuasnarggluaspleuleuarglysglnargthrpheaspasn
435440445
glyserileprohisglnilehisleuglygluleuhisalaileleu
450455460
argargglngluaspphetyrpropheleulysaspasnargglulys
465470475480
ileglulysileleuthrpheargileprotyrtyrvalglyproleu
485490495
alaargglyasnserargphealatrpmetthrarglyssergluglu
500505510
thrilethrprotrpasnpheglugluvalvalasplysglyalaser
515520525
alaglnserpheilegluargmetthrasnpheasplysasnleupro
530535540
asnglulysvalleuprolyshisserleuleutyrglutyrphethr
545550555560
valtyrasngluleuthrlysvallystyrvalthrgluglymetarg
565570575
lysproalapheleuserglygluglnlyslysalailevalaspleu
580585590
leuphelysthrasnarglysvalthrvallysglnleulysgluasp
595600605
tyrphelyslysileglucyspheaspservalgluileserglyval
610615620
gluaspargpheasnalaserleuglythrtyrhisaspleuleulys
625630635640
ileilelysasplysasppheleuaspasngluglulysgluaspile
645650655
leugluaspilevalleuthrleuthrleuphegluaspargglumet
660665670
ileglugluargleulysthrtyralahisleupheaspasplysval
675680685
metlysglnleulysargargargtyrthrglytrpglyargleuser
690695700
arglysleuileasnglyileargasplysglnserglylysthrile
705710715720
leuasppheleulysseraspglyphealaasnargasnphemetgln
725730735
leuilehisaspaspserleuthrphelysgluaspileglnlysala
740745750
glnvalserglyglnglyaspserleuhisgluhisilealaasnleu
755760765
alaglyserproalailelyslysglyileleuglnthrvallysval
770775780
valaspgluleuvallysvalmetglyarghislysprogluasnile
785790795800
valileglumetalaarggluasnglnthrthrglnlysglyglnlys
805810815
asnserarggluargmetlysargileglugluglyilelysgluleu
820825830
glyserglnileleulysgluhisprovalgluasnthrglnleugln
835840845
asnglulysleutyrleutyrtyrleuglnasnglyargaspmettyr
850855860
valaspglngluleuaspileasnargleuserasptyraspvalasp
865870875880
alailevalproglnserpheleulysaspaspserileaspasnlys
885890895
valleuthrargserasplysasnargglylysseraspasnvalpro
900905910
serglugluvalvallyslysmetlysasntyrtrpargglnleuleu
915920925
asnalalysleuilethrglnarglyspheaspasnleuthrlysala
930935940
gluargglyglyleusergluleuasplysalaglypheilelysarg
945950955960
glnleuvalgluthrargglnilethrlyshisvalalaglnileleu
965970975
aspserargmetasnthrlystyraspgluasnasplysleuilearg
980985990
gluvallysvalilethrleulysserlysleuvalseraspphearg
99510001005
lysasppheglnphetyrlysvalarggluileasnasntyrhis
101010151020
hisalahisaspalatyrleuasnalavalvalglythralaleu
102510301035
ilelyslystyrprolysleuglusergluphevaltyrglyasp
104010451050
tyrlysvaltyraspvalarglysmetilealalysserglugln
105510601065
gluileglylysalathralalystyrphephetyrserasnile
107010751080
metasnphephelysthrgluilethrleualaasnglygluile
108510901095
arglysargproleuilegluthrasnglygluthrglygluile
110011051110
valtrpasplysglyargaspphealathrvalarglysvalleu
111511201125
sermetproglnvalasnilevallyslysthrgluvalglnthr
113011351140
glyglypheserlysgluserileleuprolysargasnserasp
114511501155
lysleuilealaarglyslysasptrpaspprolyslystyrgly
116011651170
glypheaspserprothrvalalatyrservalleuvalvalala
117511801185
lysvalglulysglylysserlyslysleulysservallysglu
119011951200
leuleuglyilethrilemetgluargserserpheglulysasn
120512101215
proileasppheleuglualalysglytyrlysgluvallyslys
122012251230
aspleuileilelysleuprolystyrserleuphegluleuglu
123512401245
asnglyarglysargmetleualaseralaglygluleuglnlys
125012551260
glyasngluleualaleuproserlystyrvalasnpheleutyr
126512701275
leualaserhistyrglulysleulysglyserprogluaspasn
128012851290
gluglnlysglnleuphevalgluglnhislyshistyrleuasp
129513001305
gluileilegluglnileserglupheserlysargvalileleu
131013151320
alaaspalaasnleuasplysvalleuseralatyrasnlyshis
132513301335
argasplysproilearggluglnalagluasnileilehisleu
134013451350
phethrleuthrasnleuglyalaproalaalaphelystyrphe
135513601365
aspthrthrileasparglysargtyrthrserthrlysgluval
137013751380
leuaspalathrleuilehisglnserilethrglyleutyrglu
138513901395
thrargileaspleuserglnleuglyglyaspgluglyalapro
140014051410
lyslyslysarglysvalglyserserglyserprolyslyslys
141514201425
arglysvalserglysergluthrproglythrsergluserala
143014351440
thrproglusermetaspserleuleumetasnargarggluphe
144514501455
leutyrglnphelysasnvalargtrpalalysglyargargglu
146014651470
thrtyrleucystyrvalvallysargargaspseralathrser
147514801485
pheserleuasppheglytyrleuargasnlysasnglycyshis
149014951500
valgluleuleupheleuargtyrileserasptrpaspleuasp
150515101515
proglyargcystyrargvalthrtrppheilesertrpserpro
152015251530
cystyraspcysalaarghisvalalaasppheleuargglyasn
153515401545
proasnleuserleuargilephethralaargleutyrphecys
155015551560
gluasparglysalagluprogluglyleuargargleuhisarg
156515701575
alaglyvalglnilealailemetthrphelysasptyrphetyr
158015851590
cystrpasnthrphevalgluasnhisglyargthrphelysala
159516001605
trpgluglyleuhisgluasnservalargleuserargglnleu
161016151620
argargileleuleupro
1625
<210>69
<211>4890
<212>dna
<213>人工序列
<220>
<223>人工序列的描述:dcas9-xten-aidx的编码序列
<400>69
atggactataaggaccacgacggagactacaaggatcatgatattgattacaaagacgat60
gacgataagatggccccaaagaagaagcggaaggtcggtatccacggagtcccagcagct120
accatggacaagaagtattctatcggactggccatcgggactaatagcgtcgggtgggcc180
gtgatcactgacgagtacaaggtgccctctaagaagttcaaggtgctcgggaacaccgac240
cggcattccatcaagaaaaatctgatcggagctctcctctttgattcaggggagaccgct300
gaagcaacccgcctcaagcggactgctagacggcggtacaccaggaggaagaaccggatt360
tgttaccttcaagagatattctccaacgaaatggcaaaggtcgacgacagcttcttccat420
aggctggaagaatcattcctcgtggaagaggataagaagcatgaacggcatcccatcttc480
ggtaatatcgtcgacgaggtggcctatcacgagaaatacccaaccatctaccatcttcgc540
aaaaagctggtggactcaaccgacaaggcagacctccggcttatctacctggccctggcc600
cacatgatcaagttcagaggccacttcctgatcgagggcgacctcaatcctgacaatagc660
gatgtggataaactgttcatccagctggtgcagacttacaaccagctctttgaagagaac720
cccatcaatgcaagcggagtcgatgccaaggccattctgtcagcccggctgtcaaagagc780
cgcagacttgagaatcttatcgctcagctgccgggtgaaaagaaaaatggactgttcggg840
aacctgattgctctttcacttgggctgactcccaatttcaagtctaatttcgacctggca900
gaggatgccaagctgcaactgtccaaggacacctatgatgacgatctcgacaacctcctg960
gcccagatcggtgaccaatacgccgaccttttccttgctgctaagaatctttctgacgcc1020
atcctgctgtctgacattctccgcgtgaacactgaaatcaccaaggcccctctttcagct1080
tcaatgattaagcggtatgatgagcaccaccaggacctgaccctgcttaaggcactcgtc1140
cggcagcagcttccggagaagtacaaggaaatcttctttgaccagtcaaagaatggatac1200
gccggctacatcgacggaggtgcctcccaagaggaattttataagtttatcaaacctatc1260
cttgagaagatggacggcaccgaagagctcctcgtgaaactgaatcgggaggatctgctg1320
cggaagcagcgcactttcgacaatgggagcattccccaccagatccatcttggggagctt1380
cacgccatccttcggcgccaagaggacttctacccctttcttaaggacaacagggagaag1440
attgagaaaattctcactttccgcatcccctactacgtgggacccctcgccagaggaaat1500
agccggtttgcttggatgaccagaaagtcagaagaaactatcactccctggaacttcgaa1560
gaggtggtggacaagggagccagcgctcagtcattcatcgaacggatgactaacttcgat1620
aagaacctccccaatgagaaggtcctgccgaaacattccctgctctacgagtactttacc1680
gtgtacaacgagctgaccaaggtgaaatatgtcaccgaagggatgaggaagcccgcattc1740
ctgtcaggcgaacaaaagaaggcaattgtggaccttctgttcaagaccaatagaaaggtg1800
accgtgaagcagctgaaggaggactatttcaagaaaattgaatgcttcgactctgtggag1860
attagcggggtcgaagatcggttcaacgcaagcctgggtacctaccatgatctgcttaag1920
atcatcaaggacaaggattttctggacaatgaggagaaagaggacatccttgaggacatt1980
gtcctgactctcactctgttcgaggaccgggaaatgatcgaggagaggcttaagacctac2040
gcccatctgttcgacgataaagtgatgaagcaacttaaacggagaagatataccggatgg2100
ggacgccttagccgcaaactcatcaacggaatccgggacaaacagagcggaaagaccatt2160
cttgatttccttaagagcgacggattcgctaatcgcaacttcatgcaacttatccatgat2220
gattccctgacctttaaggaggacatccagaaggcccaagtgtctggacaaggtgactca2280
ctgcacgagcatatcgcaaatctggctggttcacccgctattaagaagggtattctccag2340
accgtgaaagtcgtggacgagctggtcaaggtgatgggtcgccataaaccagagaacatt2400
gtcatcgagatggccagggaaaaccagactacccagaagggacagaagaacagcagggag2460
cggatgaaaagaattgaggaagggattaaggagctcgggtcacagatccttaaagagcac2520
ccggtggaaaacacccagcttcagaatgagaagctctatctgtactaccttcaaaatgga2580
cgcgatatgtatgtggaccaagagcttgatatcaacaggctctcagactacgacgtggac2640
gccatcgtccctcagagcttcctcaaagacgactcaattgacaataaggtgctgactcgc2700
tcagacaagaaccggggaaagtcagataacgtgccctcagaggaagtcgtgaaaaagatg2760
aagaactattggcgccagcttctgaacgcaaagctgatcactcagcggaagttcgacaat2820
ctcactaaggctgagaggggcggactgagcgaactggacaaagcaggattcattaaacgg2880
caacttgtggagactcggcagattactaaacatgtagcccaaatccttgactcacgcatg2940
aataccaagtacgacgaaaacgacaaacttatccgcgaggtgaaggtgattaccctgaag3000
tccaagctggtcagcgatttcagaaaggactttcaattctacaaagtgcgggagatcaat3060
aactatcatcatgctcatgacgcatatctgaatgccgtggtgggaaccgccctgatcaag3120
aagtacccaaagctggaaagcgagttcgtgtacggagactacaaggtctacgacgtgcgc3180
aagatgattgccaaatctgagcaggagatcggaaaggccaccgcaaagtacttcttctac3240
agcaacatcatgaatttcttcaagaccgaaatcacccttgcaaacggtgagatccggaag3300
aggccgctcatcgagactaatggggagactggcgaaatcgtgtgggacaagggcagagat3360
ttcgctaccgtgcgcaaagtgctttctatgcctcaagtgaacatcgtgaagaaaaccgag3420
gtgcaaaccggaggcttttctaaggaatcaatcctccccaagcgcaactccgacaagctc3480
attgcaaggaagaaggattgggaccctaagaagtacggcggattcgattcaccaactgtg3540
gcttattctgtcctggtcgtggctaaggtggaaaaaggaaagtctaagaagctcaagagc3600
gtgaaggaactgctgggtatcaccattatggagcgcagctccttcgagaagaacccaatt3660
gactttctcgaagccaaaggttacaaggaagtcaagaaggaccttatcatcaagctccca3720
aagtatagcctgttcgaactggagaatgggcggaagcggatgctcgcctccgctggcgaa3780
cttcagaagggtaatgagctggctctcccctccaagtacgtgaatttcctctaccttgca3840
agccattacgagaagctgaaggggagccccgaggacaacgagcaaaagcaactgtttgtg3900
gagcagcataagcattatctggacgagatcattgagcagatttccgagttttctaaacgc3960
gtcattctcgctgatgccaacctcgataaagtccttagcgcatacaataagcacagagac4020
aaaccaattcgggagcaggctgagaatatcatccacctgttcaccctcaccaatcttggt4080
gcccctgccgcattcaagtacttcgacaccaccatcgaccggaaacgctatacctccacc4140
aaagaagtgctggacgccaccctcatccaccagagcatcaccggactttacgaaactcgg4200
attgacctctcacagctcggaggggatgagggagctcccaagaaaaagcgcaaggtaggt4260
agttccggatctccgaaaaagaaacgcaaagttagcggcagcgagactcccgggacctca4320
gagtccgccacacccgaaagtatggacagcctcttgatgaaccggaggaagtttctttac4380
caattcaaaaatgtccgctgggctaagggtcggcgtgagacctacctgtgctacgtagtg4440
aagaggcgtgacagtgctacatccttttcactggactttggttatcttcgcaataagaac4500
ggctgccacgtggaattgctcttcctccgctacatctcggactgggacctagaccctggc4560
cgctgctaccgcgtcacctggttcacctcctggagcccctgctacgactgtgcccgacat4620
gtggccgactttctgcgagggaaccccaacctcagtctgaggatcttcaccgcgcgcctc4680
tacttctgtgaggaccgcaaggctgagcccgaggggctgcggcggctgcaccgcgccggg4740
gtgcaaatagccatcatgaccttcaaagattatttttactgctggaatacttttgtagaa4800
aaccatgaaagaactttcaaagcctgggaagggctgcatgaaaattcagttcgtctctcc4860
agacagcttcggcgcatccttttgccctga4890
<210>70
<211>1629
<212>prt
<213>人工序列
<220>
<223>人工序列的描述:dcas9-xten-aidx的氨基酸序列
<400>70
metasptyrlysasphisaspglyasptyrlysasphisaspileasp
151015
tyrlysaspaspaspasplysmetalaprolyslyslysarglysval
202530
glyilehisglyvalproalaalathrmetasplyslystyrserile
354045
glyleualaileglythrasnservalglytrpalavalilethrasp
505560
glutyrlysvalproserlyslysphelysvalleuglyasnthrasp
65707580
arghisserilelyslysasnleuileglyalaleuleupheaspser
859095
glygluthralaglualathrargleulysargthralaargargarg
100105110
tyrthrargarglysasnargilecystyrleuglngluilepheser
115120125
asnglumetalalysvalaspaspserphephehisargleugluglu
130135140
serpheleuvalglugluasplyslyshisgluarghisproilephe
145150155160
glyasnilevalaspgluvalalatyrhisglulystyrprothrile
165170175
tyrhisleuarglyslysleuvalaspserthrasplysalaaspleu
180185190
argleuiletyrleualaleualahismetilelyspheargglyhis
195200205
pheleuilegluglyaspleuasnproaspasnseraspvalasplys
210215220
leupheileglnleuvalglnthrtyrasnglnleupheglugluasn
225230235240
proileasnalaserglyvalaspalalysalaileleuseralaarg
245250255
leuserlysserargargleugluasnleuilealaglnleuprogly
260265270
glulyslysasnglyleupheglyasnleuilealaleuserleugly
275280285
leuthrproasnphelysserasnpheaspleualagluaspalalys
290295300
leuglnleuserlysaspthrtyraspaspaspleuaspasnleuleu
305310315320
alaglnileglyaspglntyralaaspleupheleualaalalysasn
325330335
leuseraspalaileleuleuseraspileleuargvalasnthrglu
340345350
ilethrlysalaproleuseralasermetilelysargtyraspglu
355360365
hishisglnaspleuthrleuleulysalaleuvalargglnglnleu
370375380
proglulystyrlysgluilephepheaspglnserlysasnglytyr
385390395400
alaglytyrileaspglyglyalaserglnglugluphetyrlysphe
405410415
ilelysproileleuglulysmetaspglythrglugluleuleuval
420425430
lysleuasnarggluaspleuleuarglysglnargthrpheaspasn
435440445
glyserileprohisglnilehisleuglygluleuhisalaileleu
450455460
argargglngluaspphetyrpropheleulysaspasnargglulys
465470475480
ileglulysileleuthrpheargileprotyrtyrvalglyproleu
485490495
alaargglyasnserargphealatrpmetthrarglyssergluglu
500505510
thrilethrprotrpasnpheglugluvalvalasplysglyalaser
515520525
alaglnserpheilegluargmetthrasnpheasplysasnleupro
530535540
asnglulysvalleuprolyshisserleuleutyrglutyrphethr
545550555560
valtyrasngluleuthrlysvallystyrvalthrgluglymetarg
565570575
lysproalapheleuserglygluglnlyslysalailevalaspleu
580585590
leuphelysthrasnarglysvalthrvallysglnleulysgluasp
595600605
tyrphelyslysileglucyspheaspservalgluileserglyval
610615620
gluaspargpheasnalaserleuglythrtyrhisaspleuleulys
625630635640
ileilelysasplysasppheleuaspasngluglulysgluaspile
645650655
leugluaspilevalleuthrleuthrleuphegluaspargglumet
660665670
ileglugluargleulysthrtyralahisleupheaspasplysval
675680685
metlysglnleulysargargargtyrthrglytrpglyargleuser
690695700
arglysleuileasnglyileargasplysglnserglylysthrile
705710715720
leuasppheleulysseraspglyphealaasnargasnphemetgln
725730735
leuilehisaspaspserleuthrphelysgluaspileglnlysala
740745750
glnvalserglyglnglyaspserleuhisgluhisilealaasnleu
755760765
alaglyserproalailelyslysglyileleuglnthrvallysval
770775780
valaspgluleuvallysvalmetglyarghislysprogluasnile
785790795800
valileglumetalaarggluasnglnthrthrglnlysglyglnlys
805810815
asnserarggluargmetlysargileglugluglyilelysgluleu
820825830
glyserglnileleulysgluhisprovalgluasnthrglnleugln
835840845
asnglulysleutyrleutyrtyrleuglnasnglyargaspmettyr
850855860
valaspglngluleuaspileasnargleuserasptyraspvalasp
865870875880
alailevalproglnserpheleulysaspaspserileaspasnlys
885890895
valleuthrargserasplysasnargglylysseraspasnvalpro
900905910
serglugluvalvallyslysmetlysasntyrtrpargglnleuleu
915920925
asnalalysleuilethrglnarglyspheaspasnleuthrlysala
930935940
gluargglyglyleusergluleuasplysalaglypheilelysarg
945950955960
glnleuvalgluthrargglnilethrlyshisvalalaglnileleu
965970975
aspserargmetasnthrlystyraspgluasnasplysleuilearg
980985990
gluvallysvalilethrleulysserlysleuvalseraspphearg
99510001005
lysasppheglnphetyrlysvalarggluileasnasntyrhis
101010151020
hisalahisaspalatyrleuasnalavalvalglythralaleu
102510301035
ilelyslystyrprolysleuglusergluphevaltyrglyasp
104010451050
tyrlysvaltyraspvalarglysmetilealalysserglugln
105510601065
gluileglylysalathralalystyrphephetyrserasnile
107010751080
metasnphephelysthrgluilethrleualaasnglygluile
108510901095
arglysargproleuilegluthrasnglygluthrglygluile
110011051110
valtrpasplysglyargaspphealathrvalarglysvalleu
111511201125
sermetproglnvalasnilevallyslysthrgluvalglnthr
113011351140
glyglypheserlysgluserileleuprolysargasnserasp
114511501155
lysleuilealaarglyslysasptrpaspprolyslystyrgly
116011651170
glypheaspserprothrvalalatyrservalleuvalvalala
117511801185
lysvalglulysglylysserlyslysleulysservallysglu
119011951200
leuleuglyilethrilemetgluargserserpheglulysasn
120512101215
proileasppheleuglualalysglytyrlysgluvallyslys
122012251230
aspleuileilelysleuprolystyrserleuphegluleuglu
123512401245
asnglyarglysargmetleualaseralaglygluleuglnlys
125012551260
glyasngluleualaleuproserlystyrvalasnpheleutyr
126512701275
leualaserhistyrglulysleulysglyserprogluaspasn
128012851290
gluglnlysglnleuphevalgluglnhislyshistyrleuasp
129513001305
gluileilegluglnileserglupheserlysargvalileleu
131013151320
alaaspalaasnleuasplysvalleuseralatyrasnlyshis
132513301335
argasplysproilearggluglnalagluasnileilehisleu
134013451350
phethrleuthrasnleuglyalaproalaalaphelystyrphe
135513601365
aspthrthrileasparglysargtyrthrserthrlysgluval
137013751380
leuaspalathrleuilehisglnserilethrglyleutyrglu
138513901395
thrargileaspleuserglnleuglyglyaspgluglyalapro
140014051410
lyslyslysarglysvalglyserserglyserprolyslyslys
141514201425
arglysvalserglysergluthrproglythrsergluserala
143014351440
thrproglusermetaspserleuleumetasnargarglysphe
144514501455
leutyrglnphelysasnvalargtrpalalysglyargargglu
146014651470
thrtyrleucystyrvalvallysargargaspseralathrser
147514801485
pheserleuasppheglytyrleuargasnlysasnglycyshis
149014951500
valgluleuleupheleuargtyrileserasptrpaspleuasp
150515101515
proglyargcystyrargvalthrtrpphethrsertrpserpro
152015251530
cystyraspcysalaarghisvalalaasppheleuargglyasn
153515401545
proasnleuserleuargilephethralaargleutyrphecys
155015551560
gluasparglysalagluprogluglyleuargargleuhisarg
156515701575
alaglyvalglnilealailemetthrphelysasptyrphetyr
158015851590
cystrpasnthrphevalgluasnhisgluargthrphelysala
159516001605
trpgluglyleuhisgluasnservalargleuserargglnleu
161016151620
argargileleuleupro
1625
<210>71
<211>4917
<212>dna
<213>人工序列
<220>
<223>人工序列的描述:ncas9-aidx的编码序列
<400>71
atggactataaggaccacgacggagactacaaggatcatgatattgattacaaagacgat60
gacgataagatggccccaaagaagaagcggaaggtcggtatccacggagtcccagcagct120
accatggacaagaagtattctatcggactggccatcgggactaatagcgtcgggtgggcc180
gtgatcactgacgagtacaaggtgccctctaagaagttcaaggtgctcgggaacaccgac240
cggcattccatcaagaaaaatctgatcggagctctcctctttgattcaggggagaccgct300
gaagcaacccgcctcaagcggactgctagacggcggtacaccaggaggaagaaccggatt360
tgttaccttcaagagatattctccaacgaaatggcaaaggtcgacgacagcttcttccat420
aggctggaagaatcattcctcgtggaagaggataagaagcatgaacggcatcccatcttc480
ggtaatatcgtcgacgaggtggcctatcacgagaaatacccaaccatctaccatcttcgc540
aaaaagctggtggactcaaccgacaaggcagacctccggcttatctacctggccctggcc600
cacatgatcaagttcagaggccacttcctgatcgagggcgacctcaatcctgacaatagc660
gatgtggataaactgttcatccagctggtgcagacttacaaccagctctttgaagagaac720
cccatcaatgcaagcggagtcgatgccaaggccattctgtcagcccggctgtcaaagagc780
cgcagacttgagaatcttatcgctcagctgccgggtgaaaagaaaaatggactgttcggg840
aacctgattgctctttcacttgggctgactcccaatttcaagtctaatttcgacctggca900
gaggatgccaagctgcaactgtccaaggacacctatgatgacgatctcgacaacctcctg960
gcccagatcggtgaccaatacgccgaccttttccttgctgctaagaatctttctgacgcc1020
atcctgctgtctgacattctccgcgtgaacactgaaatcaccaaggcccctctttcagct1080
tcaatgattaagcggtatgatgagcaccaccaggacctgaccctgcttaaggcactcgtc1140
cggcagcagcttccggagaagtacaaggaaatcttctttgaccagtcaaagaatggatac1200
gccggctacatcgacggaggtgcctcccaagaggaattttataagtttatcaaacctatc1260
cttgagaagatggacggcaccgaagagctcctcgtgaaactgaatcgggaggatctgctg1320
cggaagcagcgcactttcgacaatgggagcattccccaccagatccatcttggggagctt1380
cacgccatccttcggcgccaagaggacttctacccctttcttaaggacaacagggagaag1440
attgagaaaattctcactttccgcatcccctactacgtgggacccctcgccagaggaaat1500
agccggtttgcttggatgaccagaaagtcagaagaaactatcactccctggaacttcgaa1560
gaggtggtggacaagggagccagcgctcagtcattcatcgaacggatgactaacttcgat1620
aagaacctccccaatgagaaggtcctgccgaaacattccctgctctacgagtactttacc1680
gtgtacaacgagctgaccaaggtgaaatatgtcaccgaagggatgaggaagcccgcattc1740
ctgtcaggcgaacaaaagaaggcaattgtggaccttctgttcaagaccaatagaaaggtg1800
accgtgaagcagctgaaggaggactatttcaagaaaattgaatgcttcgactctgtggag1860
attagcggggtcgaagatcggttcaacgcaagcctgggtacctaccatgatctgcttaag1920
atcatcaaggacaaggattttctggacaatgaggagaaagaggacatccttgaggacatt1980
gtcctgactctcactctgttcgaggaccgggaaatgatcgaggagaggcttaagacctac2040
gcccatctgttcgacgataaagtgatgaagcaacttaaacggagaagatataccggatgg2100
ggacgccttagccgcaaactcatcaacggaatccgggacaaacagagcggaaagaccatt2160
cttgatttccttaagagcgacggattcgctaatcgcaacttcatgcaacttatccatgat2220
gattccctgacctttaaggaggacatccagaaggcccaagtgtctggacaaggtgactca2280
ctgcacgagcatatcgcaaatctggctggttcacccgctattaagaagggtattctccag2340
accgtgaaagtcgtggacgagctggtcaaggtgatgggtcgccataaaccagagaacatt2400
gtcatcgagatggccagggaaaaccagactacccagaagggacagaagaacagcagggag2460
cggatgaaaagaattgaggaagggattaaggagctcgggtcacagatccttaaagagcac2520
ccggtggaaaacacccagcttcagaatgagaagctctatctgtactaccttcaaaatgga2580
cgcgatatgtatgtggaccaagagcttgatatcaacaggctctcagactacgacgtggac2640
catatcgtccctcagagcttcctcaaagacgactcaattgacaataaggtgctgactcgc2700
tcagacaagaaccggggaaagtcagataacgtgccctcagaggaagtcgtgaaaaagatg2760
aagaactattggcgccagcttctgaacgcaaagctgatcactcagcggaagttcgacaat2820
ctcactaaggctgagaggggcggactgagcgaactggacaaagcaggattcattaaacgg2880
caacttgtggagactcggcagattactaaacatgtagcccaaatccttgactcacgcatg2940
aataccaagtacgacgaaaacgacaaacttatccgcgaggtgaaggtgattaccctgaag3000
tccaagctggtcagcgatttcagaaaggactttcaattctacaaagtgcgggagatcaat3060
aactatcatcatgctcatgacgcatatctgaatgccgtggtgggaaccgccctgatcaag3120
aagtacccaaagctggaaagcgagttcgtgtacggagactacaaggtctacgacgtgcgc3180
aagatgattgccaaatctgagcaggagatcggaaaggccaccgcaaagtacttcttctac3240
agcaacatcatgaatttcttcaagaccgaaatcacccttgcaaacggtgagatccggaag3300
aggccgctcatcgagactaatggggagactggcgaaatcgtgtgggacaagggcagagat3360
ttcgctaccgtgcgcaaagtgctttctatgcctcaagtgaacatcgtgaagaaaaccgag3420
gtgcaaaccggaggcttttctaaggaatcaatcctccccaagcgcaactccgacaagctc3480
attgcaaggaagaaggattgggaccctaagaagtacggcggattcgattcaccaactgtg3540
gcttattctgtcctggtcgtggctaaggtggaaaaaggaaagtctaagaagctcaagagc3600
gtgaaggaactgctgggtatcaccattatggagcgcagctccttcgagaagaacccaatt3660
gactttctcgaagccaaaggttacaaggaagtcaagaaggaccttatcatcaagctccca3720
aagtatagcctgttcgaactggagaatgggcggaagcggatgctcgcctccgctggcgaa3780
cttcagaagggtaatgagctggctctcccctccaagtacgtgaatttcctctaccttgca3840
agccattacgagaagctgaaggggagccccgaggacaacgagcaaaagcaactgtttgtg3900
gagcagcataagcattatctggacgagatcattgagcagatttccgagttttctaaacgc3960
gtcattctcgctgatgccaacctcgataaagtccttagcgcatacaataagcacagagac4020
aaaccaattcgggagcaggctgagaatatcatccacctgttcaccctcaccaatcttggt4080
gcccctgccgcattcaagtacttcgacaccaccatcgaccggaaacgctatacctccacc4140
aaagaagtgctggacgccaccctcatccaccagagcatcaccggactttacgaaactcgg4200
attgacctctcacagctcggaggggatgagggagctcccaagaaaaagcgcaaggtaggt4260
agttccggatctccgaaaaagaaacgcaaagttggtagtgatgctttagacgattttgac4320
ttagatatgcttggttcagacgcgttagacgacttcggtggaggatccatggacagcctc4380
ttgatgaaccggaggaagtttctttaccaattcaaaaatgtccgctgggctaagggtcgg4440
cgtgagacctacctgtgctacgtagtgaagaggcgtgacagtgctacatccttttcactg4500
gactttggttatcttcgcaataagaacggctgccacgtggaattgctcttcctccgctac4560
atctcggactgggacctagaccctggccgctgctaccgcgtcacctggttcacctcctgg4620
agcccctgctacgactgtgcccgacatgtggccgactttctgcgagggaaccccaacctc4680
agtctgaggatcttcaccgcgcgcctctacttctgtgaggaccgcaaggctgagcccgag4740
gggctgcggcggctgcaccgcgccggggtgcaaatagccatcatgaccttcaaagattat4800
ttttactgctggaatacttttgtagaaaaccatgaaagaactttcaaagcctgggaaggg4860
ctgcatgaaaattcagttcgtctctccagacagcttcggcgcatccttttgccctga4917
<210>72
<211>1638
<212>prt
<213>人工序列
<220>
<223>人工序列的描述:ncas9-aidx的氨基酸序列
<400>72
metasptyrlysasphisaspglyasptyrlysasphisaspileasp
151015
tyrlysaspaspaspasplysmetalaprolyslyslysarglysval
202530
glyilehisglyvalproalaalathrmetasplyslystyrserile
354045
glyleualaileglythrasnservalglytrpalavalilethrasp
505560
glutyrlysvalproserlyslysphelysvalleuglyasnthrasp
65707580
arghisserilelyslysasnleuileglyalaleuleupheaspser
859095
glygluthralaglualathrargleulysargthralaargargarg
100105110
tyrthrargarglysasnargilecystyrleuglngluilepheser
115120125
asnglumetalalysvalaspaspserphephehisargleugluglu
130135140
serpheleuvalglugluasplyslyshisgluarghisproilephe
145150155160
glyasnilevalaspgluvalalatyrhisglulystyrprothrile
165170175
tyrhisleuarglyslysleuvalaspserthrasplysalaaspleu
180185190
argleuiletyrleualaleualahismetilelyspheargglyhis
195200205
pheleuilegluglyaspleuasnproaspasnseraspvalasplys
210215220
leupheileglnleuvalglnthrtyrasnglnleupheglugluasn
225230235240
proileasnalaserglyvalaspalalysalaileleuseralaarg
245250255
leuserlysserargargleugluasnleuilealaglnleuprogly
260265270
glulyslysasnglyleupheglyasnleuilealaleuserleugly
275280285
leuthrproasnphelysserasnpheaspleualagluaspalalys
290295300
leuglnleuserlysaspthrtyraspaspaspleuaspasnleuleu
305310315320
alaglnileglyaspglntyralaaspleupheleualaalalysasn
325330335
leuseraspalaileleuleuseraspileleuargvalasnthrglu
340345350
ilethrlysalaproleuseralasermetilelysargtyraspglu
355360365
hishisglnaspleuthrleuleulysalaleuvalargglnglnleu
370375380
proglulystyrlysgluilephepheaspglnserlysasnglytyr
385390395400
alaglytyrileaspglyglyalaserglnglugluphetyrlysphe
405410415
ilelysproileleuglulysmetaspglythrglugluleuleuval
420425430
lysleuasnarggluaspleuleuarglysglnargthrpheaspasn
435440445
glyserileprohisglnilehisleuglygluleuhisalaileleu
450455460
argargglngluaspphetyrpropheleulysaspasnargglulys
465470475480
ileglulysileleuthrpheargileprotyrtyrvalglyproleu
485490495
alaargglyasnserargphealatrpmetthrarglyssergluglu
500505510
thrilethrprotrpasnpheglugluvalvalasplysglyalaser
515520525
alaglnserpheilegluargmetthrasnpheasplysasnleupro
530535540
asnglulysvalleuprolyshisserleuleutyrglutyrphethr
545550555560
valtyrasngluleuthrlysvallystyrvalthrgluglymetarg
565570575
lysproalapheleuserglygluglnlyslysalailevalaspleu
580585590
leuphelysthrasnarglysvalthrvallysglnleulysgluasp
595600605
tyrphelyslysileglucyspheaspservalgluileserglyval
610615620
gluaspargpheasnalaserleuglythrtyrhisaspleuleulys
625630635640
ileilelysasplysasppheleuaspasngluglulysgluaspile
645650655
leugluaspilevalleuthrleuthrleuphegluaspargglumet
660665670
ileglugluargleulysthrtyralahisleupheaspasplysval
675680685
metlysglnleulysargargargtyrthrglytrpglyargleuser
690695700
arglysleuileasnglyileargasplysglnserglylysthrile
705710715720
leuasppheleulysseraspglyphealaasnargasnphemetgln
725730735
leuilehisaspaspserleuthrphelysgluaspileglnlysala
740745750
glnvalserglyglnglyaspserleuhisgluhisilealaasnleu
755760765
alaglyserproalailelyslysglyileleuglnthrvallysval
770775780
valaspgluleuvallysvalmetglyarghislysprogluasnile
785790795800
valileglumetalaarggluasnglnthrthrglnlysglyglnlys
805810815
asnserarggluargmetlysargileglugluglyilelysgluleu
820825830
glyserglnileleulysgluhisprovalgluasnthrglnleugln
835840845
asnglulysleutyrleutyrtyrleuglnasnglyargaspmettyr
850855860
valaspglngluleuaspileasnargleuserasptyraspvalasp
865870875880
hisilevalproglnserpheleulysaspaspserileaspasnlys
885890895
valleuthrargserasplysasnargglylysseraspasnvalpro
900905910
serglugluvalvallyslysmetlysasntyrtrpargglnleuleu
915920925
asnalalysleuilethrglnarglyspheaspasnleuthrlysala
930935940
gluargglyglyleusergluleuasplysalaglypheilelysarg
945950955960
glnleuvalgluthrargglnilethrlyshisvalalaglnileleu
965970975
aspserargmetasnthrlystyraspgluasnasplysleuilearg
980985990
gluvallysvalilethrleulysserlysleuvalseraspphearg
99510001005
lysasppheglnphetyrlysvalarggluileasnasntyrhis
101010151020
hisalahisaspalatyrleuasnalavalvalglythralaleu
102510301035
ilelyslystyrprolysleuglusergluphevaltyrglyasp
104010451050
tyrlysvaltyraspvalarglysmetilealalysserglugln
105510601065
gluileglylysalathralalystyrphephetyrserasnile
107010751080
metasnphephelysthrgluilethrleualaasnglygluile
108510901095
arglysargproleuilegluthrasnglygluthrglygluile
110011051110
valtrpasplysglyargaspphealathrvalarglysvalleu
111511201125
sermetproglnvalasnilevallyslysthrgluvalglnthr
113011351140
glyglypheserlysgluserileleuprolysargasnserasp
114511501155
lysleuilealaarglyslysasptrpaspprolyslystyrgly
116011651170
glypheaspserprothrvalalatyrservalleuvalvalala
117511801185
lysvalglulysglylysserlyslysleulysservallysglu
119011951200
leuleuglyilethrilemetgluargserserpheglulysasn
120512101215
proileasppheleuglualalysglytyrlysgluvallyslys
122012251230
aspleuileilelysleuprolystyrserleuphegluleuglu
123512401245
asnglyarglysargmetleualaseralaglygluleuglnlys
125012551260
glyasngluleualaleuproserlystyrvalasnpheleutyr
126512701275
leualaserhistyrglulysleulysglyserprogluaspasn
128012851290
gluglnlysglnleuphevalgluglnhislyshistyrleuasp
129513001305
gluileilegluglnileserglupheserlysargvalileleu
131013151320
alaaspalaasnleuasplysvalleuseralatyrasnlyshis
132513301335
argasplysproilearggluglnalagluasnileilehisleu
134013451350
phethrleuthrasnleuglyalaproalaalaphelystyrphe
135513601365
aspthrthrileasparglysargtyrthrserthrlysgluval
137013751380
leuaspalathrleuilehisglnserilethrglyleutyrglu
138513901395
thrargileaspleuserglnleuglyglyaspgluglyalapro
140014051410
lyslyslysarglysvalglyserserglyserprolyslyslys
141514201425
arglysvalglyseraspalaleuaspasppheaspleuaspmet
143014351440
leuglyseraspalaleuaspasppheglyglyglysermetasp
144514501455
serleuleumetasnargarglyspheleutyrglnphelysasn
146014651470
valargtrpalalysglyargarggluthrtyrleucystyrval
147514801485
vallysargargaspseralathrserpheserleuaspphegly
149014951500
tyrleuargasnlysasnglycyshisvalgluleuleupheleu
150515101515
argtyrileserasptrpaspleuaspproglyargcystyrarg
152015251530
valthrtrpphethrsertrpserprocystyraspcysalaarg
153515401545
hisvalalaasppheleuargglyasnproasnleuserleuarg
155015551560
ilephethralaargleutyrphecysgluasparglysalaglu
156515701575
progluglyleuargargleuhisargalaglyvalglnileala
158015851590
ilemetthrphelysasptyrphetyrcystrpasnthrpheval
159516001605
gluasnhisgluargthrphelysalatrpgluglyleuhisglu
161016151620
asnservalargleuserargglnleuargargileleuleupro
162516301635
<210>73
<211>20
<212>dna
<213>人工序列
<220>
<223>人工序列的描述:sgrna的靶标结合区
<400>73
tccctcacctgttctgtcac20
<210>74
<211>20
<212>dna
<213>人工序列
<220>
<223>人工序列的描述:sgrna的靶标结合区
<400>74
gctccagtaatcactggtga20
<210>75
<211>20
<212>dna
<213>人工序列
<220>
<223>人工序列的描述:sgrna的靶标结合区
<400>75
gatccagctccagtaatcac20
<210>76
<211>20
<212>dna
<213>人工序列
<220>
<223>人工序列的描述:sgrna的靶标结合区
<400>76
gtgattactggagctggatc20
<210>77
<211>20
<212>dna
<213>人工序列
<220>
<223>人工序列的描述:sgrna的靶标结合区
<400>77
atggggtacgtaagctacag20
<210>78
<211>20
<212>dna
<213>人工序列
<220>
<223>人工序列的描述:sgrna的靶标结合区
<400>78
gagattcgacttttgagaga20
<210>79
<211>20
<212>dna
<213>人工序列
<220>
<223>人工序列的描述:sgrna的靶标结合区
<400>79
tattactgtgcaaactggga20
<210>80
<211>20
<212>dna
<213>人工序列
<220>
<223>人工序列的描述:sgrna的靶标结合区
<400>80
caaactgggacggtgattac20
<210>81
<211>20
<212>dna
<213>人工序列
<220>
<223>人工序列的描述:sgrna的靶标结合区
<400>81
gacggtgattactggggcca20
<210>82
<211>20
<212>dna
<213>人工序列
<220>
<223>人工序列的描述:sgrna的靶标结合区
<400>82
gttgttgccaatactttggc20
<210>83
<211>20
<212>dna
<213>人工序列
<220>
<223>人工序列的描述:sgrna的靶标结合区
<400>83
atagcgtcagtctttcctgc20
<210>84
<211>20
<212>dna
<213>人工序列
<220>
<223>人工序列的描述:sgrna的靶标结合区
<400>84
gtattggcaacaacctacac20
<210>85
<211>20
<212>dna
<213>人工序列
<220>
<223>人工序列的描述:sgrna的靶标结合区
<400>85
aggggatcccagagatggac20
<210>86
<211>20
<212>dna
<213>人工序列
<220>
<223>人工序列的描述:sgrna的靶标结合区
<400>86
tatgcttcccagtccatctc20
<210>87
<211>20
<212>dna
<213>人工序列
<220>
<223>人工序列的描述:sgrna的靶标结合区
<400>87
tctgtcaacagagtaacagc20
<210>88
<211>20
<212>dna
<213>人工序列
<220>
<223>人工序列的描述:sgrna的靶标结合区
<400>88
gtcccccctccgaacgtgta20
<210>89
<211>4
<212>prt
<213>人工序列
<220>
<223>人工序列的描述:接头的重复基序
<400>89
serglyglyser
1
<210>90
<211>5
<212>prt
<213>人工序列
<220>
<223>人工序列的描述:接头的重复基序
<400>90
glyserserglyser
15
<210>91
<211>4
<212>prt
<213>人工序列
<220>
<223>人工序列的描述:接头的重复基序
<400>91
glyglyglyser
1
<210>92
<211>5
<212>prt
<213>人工序列
<220>
<223>人工序列的描述:接头的重复基序
<400>92
glyglyglyglyser
15
<210>93
<211>5
<212>prt
<213>人工序列
<220>
<223>人工序列的描述:接头的重复基序
<400>93
sersersersergly
15
<210>94
<211>5
<212>prt
<213>人工序列
<220>
<223>人工序列的描述:接头的重复基序
<400>94
glyserglyserala
15
<210>95
<211>5
<212>prt
<213>人工序列
<220>
<223>人工序列的描述:接头的重复基序
<400>95
glyglyserglygly
15
- 还没有人留言评论。精彩留言会获得点赞!