一种真核启动子及其制备方法和应用与流程
本发明属于生物技术领域,具体涉及一种真核启动子及其制备方法和应用。
背景技术:
外源基因在哺乳动物细胞中的表达对于转基因治疗试验、dna疫苗以及药物产品的生产和细胞生物学的基础研究是必需的。哺乳动物表达系统中外源基因的表达水平主要与表达载体中启动子的转录强度相关。因此,真核细胞中外源基因的表达,具有恰当的启动子至关重要。这些启动子在医学、药物及生物医学的药品生产、基因治疗,以及生命科学基础研究中具有重要应用价值。真核启动子的研发在基因治疗和药品生产领域具有特别重要的价值。
三十年前,研究人员在人巨细胞病毒(cmv)的基因组dna(gdna)中鉴定了出了强转录增强子/启动子(以下称为启动子),这个启动子序列位于重要的极早期(ie)基因的转录起始位点(tss)的上游,是已知最强的启动子序列之一。
通常,cmvie启动子和人延伸因子α(ef-1α)启动子是各种哺乳动物细胞系中转录活性最强的两个启动子。在一些研究中,小鼠巨细胞病毒ie启动子启动效率也许会更高一些。然而,在人细胞(例如293ebna或cho-k1-s细胞)中,带有内含子a的cmv启动子在瞬时和稳定转染中蛋白质表达水平都是最高的。有研究在cho细胞中使用lonza'sgs系统(工业中用于哺乳动物基因表达的常用载体之一)表达单克隆抗体(mab)来比较两种启动子的表达能力,结果显示,小鼠cmv启动子比人cmv启动子效率要低。研究人员已经确认人cmv启动子在很多细胞中具有高转录活性,而且该转录调控元件在转基因动物的组织表达中具有广泛的转录活性。在早期和最近的研究中,cmv启动子也已经被普遍用作各种基因表达载体的基础元件。
全长人cmv启动子含有多个转录因子结合位点(tfbss),其中许多顺式作用元件多次重复出现,包括多个nf-κb/rel、creb/atf、ap1、视黄酸受体(rar)、sp-1、血清应答因子(srf)和elk-1的dna结合位点。四个nf-κb结合位点与tss的距离各不同。一些研究表明nf-κb/rel位点不仅负责调节人cmv-ie增强子的活性,而且控制启动子基本的转录活性。然而也有一些研究人员认为,四个nf-κb结合位点对于cmv启动子激活是可有可无的。但以上结论并不是绝对的,该研究中,cmv启动子顺式作用元件的研究是在全病毒基因组感染细胞的背景下进行的,而实际上,cmv启动子通常用作载体系统中的转录调节元件。一些研究通过tnfα和ie1对cmv启动子的刺激,得出以下结论,cmv启动子中nf-κb结合位点与tss和基因序列的距离不同,其对转录活性的影响会有区别。位点4和2发挥最主要的作用,位点3发挥的作用次之,而序列中保守但远离tss的位点1对启动子的调节没有影响。但也有研究表明位点2和3分别表现为主要作用位点和低影响力的位点。很难得出确定的结论,这些顺式作用元件中的哪一个负责启动子的活化或抑制。重要的是,这些研究表明,cmv启动子是通过各种功能的顺式作用元件之间的相互作用来调节整体的转录功能。这些顺式作用元件形成了跨越整个增强子的功能网络。
启动子的设计和改造通常是为了获得各种不同的功能性启动子,包括高转录活性和持续表达能力,或产生各种不同强度的基因表达。可以通过去除对表达有不利影响的序列或引入有利于基因表达的因子来改造启动子。例如,人cmv启动子中的pdx1结合位点显示为阻抑物,去除该位点后,瞬时荧光素酶实验中表达增加四倍。在cmv启动子上游插入zfp-2392v的结合位点之后也观察到基因表达增强。去除由于dna甲基化而倾向于基因沉默的cpg二核苷酸位点也可以改善转基因的表达稳定性。商业生产和文献报道中,用不同调节元件组合成的启动子已广泛用于各种表达载体,从而使哺乳动物细胞高水平生产重组蛋白。已经有报道称,第一外显子和内含子a在瞬时和稳定转染中对基因表达具有积极效应。融合不同增强子和核心启动子的杂合启动子也可以增强转录活性或调节表达。已经知道cmv启动子是迄今为止鉴定的最强的转录控制元件,一些研究在细胞启动子的5'末端添加了cmv增强子区域以提高其转录活性。增强启动子活性的另一种方法是将随机地或已知的随机顺式作用元件进行串联延伸,构建合成增强子和核心启动子。在一项研究中,研究人员将10个碱基的dna序列进行随机组合,通过微阵列打印技术串联重复序列,组装成100个碱基对的合成增强子。发现在hela细胞中,其中一个增强子的转录活性达到人cmv增强子的两倍。此外,合成生物学也已经成为启动子设计和改造的重要手段。但是现有技术中野生型人巨细胞病毒启动子(wild-typecmv,wtcmv),活性单一不能灵活控制合成生物学中的基因表达输出量(output),并且缺乏高活性启动子。特别是目前生物技术高效大量制备药物蛋白,需要高活性真核启动子。
技术实现要素:
发明目的:针对现有技术存在的问题,本发明提供一种真核启动子,该真核启动子不仅包括发明高转录活性启动子,还包括一系列具有各种转录活性的工程化哺乳动物启动子,可以用于灵活控制合成生物学中的基因表达输出量。
本发明还提出了一种真核启动子的制备方法,作为一种新方法用于制造具有不同转录活性的启动子。
最后本发明还提供真核启动子在生物及医学领域的应用。
技术方案:为了实现上述目的,如本发明所述一种真核启动子,包括14种具有不同转录活性的突变cmv启动子:t1p2、t1p24、s12、s124、t1、t2、t3、t4、p1、p2、p3、p4、p12或p12s34中的任意一种,所述14种启动子分别命名为mcmv1~mcmv14,其碱基序列分别如seqidno.1~seqidno.14所示。其中,所述启动子为基于人野生型cmv(人wtcmv)启动子改造获得的具有不同转录活性的突变cmv(mcmw)启动子。
其中,所述启动子t1p2、t1和p2为高转录活性真核启动子。
本发明所述真核启动子的制备方法,包括如下步骤:将人wtcmv启动子中的nf-κb结合位点以不同的组合方式替换人工碱基序列,以此获得具有不同转录活性的突变cmv启动mcmv1~mcmv14。
其中,所述nf-κb结合位点有4个,其碱基序列和碱基位置分别为:wtbs1:gggactttcc(-422~-413,指该位点位于转录起始位点(tss)上游422~413处);wtbs2:gggactttcc(-271~-262,指该位点位于转录起始位点(tss)上游271~262处);wtbs3:ggggatttcc(-166~-157,指该位点位于转录起始位点(tss)上游166~157处);wtbs4:gggactttcc(-103~-94,指该位点位于转录起始位点(tss)上游103~94处);所述人工碱基序列为bs-t:ggggtttccc;bs-p:ggggattccc;bs-s:tagtaacgcc。
作为优选,所述mcmv1用bs-t替代人wtcmv启动子中的wtbs1,用bs-p替代人wtcmv启动子中的wtbs2。
作为优选,所述mcmv2用bs-t替代人wtcmv启动子中的wtbs1,用bs-p分别替代人wtcmv启动子中的wtbs2和wtbs4。
作为优选,所述mcmv3用bs-s分别替代人wtcmv启动子中的wtbs1和wtbs2。
作为优选,所述mcmv4用bs-s分别替代人wtcmv启动子中的wtbs1、wtbs2和wtbs4。
作为优选,所述mcmv5用bs-t替代人wtcmv启动子中的wtbs1。
作为优选,所述mcmv6用bs-t替代人wtcmv启动子中的wtbs2。
作为优选,所述mcmv7用bs-t替代人wtcmv启动子中的wtbs3。
作为优选,所述mcmv8用bs-t替代人wtcmv启动子中的wtbs4。
作为优选,所述mcmv9用bs-p替代人wtcmv启动子中的wtbs1。
作为优选,所述mcmv10用bs-p替代人wtcmv启动子中的wtbs2。
作为优选,所述mcmv11用bs-p替代人wtcmv启动子中的wtbs3。
作为优选,所述mcmv12用bs-p替代人wtcmv启动子中的wtbs4。
作为优选,所述mcmv13用bs-p分别替代人wtcmv启动子中的wtbs1和wtbs2。
作为优选,所述mcmv14的制备方法为:用bs-p分别替代人wtcmv启动子中的wtbs1和wtbs2,用bs-s分别替代人wtcmv启动子中的wtbs3和wtbs4。
上述启动子mcmv1~mcmv14分别命名为mcmv1=t1p2、mcmv2=t1p24、mcmv3=s12、mcmv4=s124、mcmv5=t1、mcmv6=t2、mcmv7=t3、mcmv8=t4、mcmv9=p1、mcmv10=p2、mcmv11=p3、mcmv12=p4、mcmv13=p12、mcmv14=p12s34
本发明所述真核启动子在生物及医学领域的应用。cmv启动子是目前基因工程领域广泛使用的转录活性最强的启动子,被广泛地用于基因工程药品生产、基因治疗、基因疫苗、基因编辑等领域。因此,本发明所筛选出的高转录活性启动子在这些生物医学领域具有潜在的重要应用价值。此外,本发明还产生了一系列具有各种转录活性的工程化哺乳动物启动子,可以用于灵活控制合成生物学中的基因表达输出量(output)。
外源基因在哺乳动物细胞中的表达对于转基因治疗试验、dna疫苗以及药物产品的生产和细胞生物学的基础研究是必需的。哺乳动物表达系统中外源基因的表达水平主要与表达载体中启动子的转录强度相关。cmv启动子是目前基因工程领域广泛使用的转录活性最强的启动子。但根据基因表达调控的分子机理,该启动子的活性还有进一步提高的可能。因此,本发明基于对基因表达调控分子机理的研究,改造该启动子上转录因子nf-κb的dna结合位点,以期获得转录活性更高的突变型新型cmv启动子。在本发明中,通过人工选择的高亲和力序列取代人wtcmv中不同的gggactttcc位点,制造了多个人突变型cmv(mcmv)启动子。对这些突变的cmv启动子进行了系统的转录活性评价。结果成功地鉴定了比wtcmv启动子转录活性更高的三个mcmv启动子。这些启动子可广泛应用于外源蛋白在哺乳动物细胞中的表达,例如基因工程和基因治疗。同时其他mcmv启动子的转录活性比wtcmv启动子更低且各不相同。
本发明通过电泳迁移率变动分析(electrophoreticmobilityshiftassay,emsa)的指数富集配体系统进化(systematicevolutionofligandsbyexponentialenrichment,selex)-测序(sequencing,seq)技术(简称selex-seq技术),用于表征转录因子蛋白nf-κb与所有可能结合的16碱基双链dna(dsdna)序列的相对结合亲和力,其中鉴定了许多10个碱基的dsdna序列,这些序列与天然nf-κb结合位点(5'-gggactttcc-3')相比,具有更高的结合亲和力。而在最广泛使用的人cmv启动子中就有四个nf-κb的结合位点,其中三个位点的序列是5'-gggactttcc-3',因此将这些天然结合位点用鉴定的高亲和力序列替代,则人cmv启动子的转录活性可能进一步增强。
有益效果:与现有技术相比,本发明具有如下优点:
本发明制备得到一系列新的哺乳动物启动子,其中三种启动子(t1p2、t1和p2)具有比人wtcmv启动子更强的转录活性,这三种启动子高转录活性启动子在这些生物医学领域具有潜在的重要应用价值。此外,本发明还产生了一系列具有各种转录活性的工程化哺乳动物启动子(t1p24、s12、s124、t2、t3、t4、p1、p3、p4、p12和p12s34),可以用于灵活控制合成生物学中的基因表达输出量(output)。本发明真核启动子的制备方法,作为一种新方法用于制造具有不同转录活性的启动子,简单方便,成功率高,并有利于筛选出高活性启动子。
在发明中,为了充分评估启动子的转录活性,使用三个不同的报告系统,一个细胞内表达报告蛋白系统(双色荧光素酶检测系统)和两种分泌表达报告蛋白系统(单分泌酶检测系统、双分泌酶检测系统)来对启动子进行检测。与检测细胞内表达的荧光素酶或egfp结果相比,三种高活性mcmv启动子(t1p2、t1和p2)在所有检测的细胞中,分泌型报告基因检测结果中启动子的转录活性差异更大。例如,在所有细胞内表达的报告基因检测中,p2启动子的转录活性并没有全部超过人wtcmv启动子的2倍;然而,在几乎所有分泌表达报告基因的检测中,p2启动子的转录活性都是人wtcmv启动子的2倍。结果显示,p2启动子的转录活性是人wtcmv启动子的大约2.4~4.4倍。这些数据再次证明,在所有使用的细胞系中,mcmv启动子具有比人wtcmv启动子更高的转录活性。同时本发明了的这三种mcmv启动子的高转录活性完全可以应用于分泌型蛋白的生产。所有分泌蛋白表达检测,单一分泌蛋白表达检测(gaussia荧光素酶报告测定)或双分泌蛋白表达检测(secrete-pairdualluminescenceassay),都表明三个mcmv启动子(t1p2、t1和p2)相同条件下比人wtcmv启动子转录活性都要高。动力学转录活性检测结果显示这三个mcmv启动子具有比人wtcmv启动子可持续性更好的转录活性。
附图说明
图1为本发明启动子的示意图;其中闭合框代表人wtcmv增强子/启动子序列中nf-κb结合位点的序列和相对位置;其中:(a)本研究中使用的人wtcmv启动子和试验性mcmv启动子;(b)研究中构建的其余mcmv启动子;p、t和s,用于构建mcmv启动子的三个序列;该图中site1=wtbs1、site2=wtbs2、site3=wtbs3、site4=wtbs4;mcmv1=t1p2、mcmv2=t1p24、mcmv3=s12、mcmv4=s124、mcmv5=t1、mcmv6=t2、mcmv7=t3、mcmv8=t4、mcmv9=p1、mcmv10=p2、mcmv11=p3、mcmv12=p4、mcmv13=p12、mcmv14=p12s34;t=bs-t、p=bs-p、s=bs-s;本发明以下附图说明及实施例中均使用本图中使用的启动子名称;
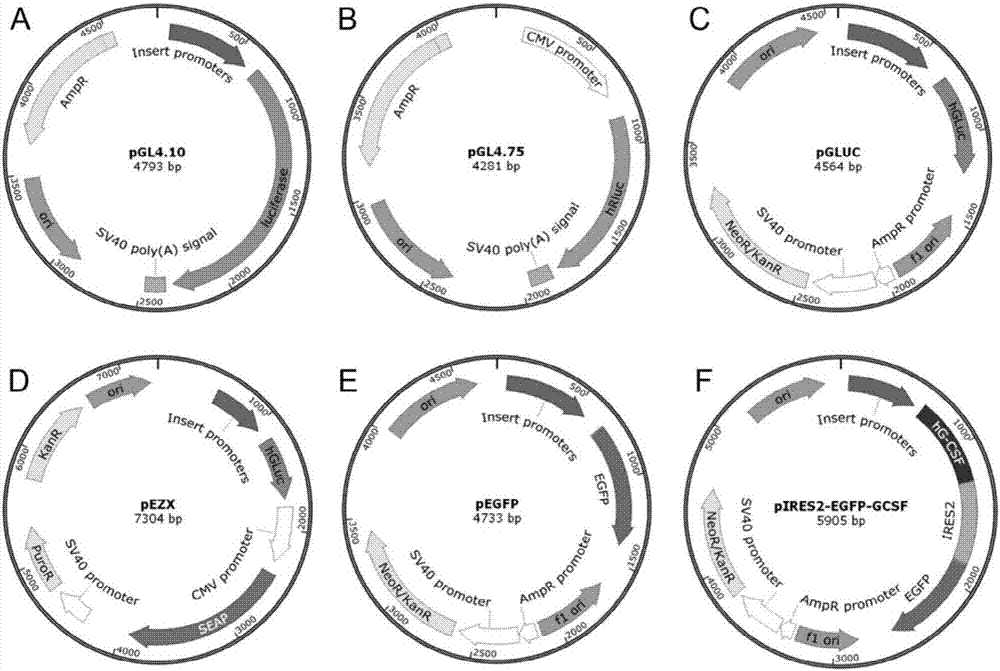
图2为质粒示意图;其中(a)用于双荧光素酶报告基因测定系统中的质粒:pgl4.10质粒用于插入待检测启动子;(b)pgl4.75质粒用作共转染的对照载体;(c)用于gaussia萤光素酶报告基因测定系统中的质粒:pgluc质粒用于插入待检测启动子;(d)用于双分泌报告基因测定系统中的质粒:pezx质粒用于插入待检测启动子;(e)用于绿色荧光检测中的质粒:使用pegfp质粒插入待检测启动子;(f)用于表达hg-csf的质粒pires-egfp-gcsf;
图3为hepg2细胞中启动子的转录活性分析示意图;其中(a)启动子的转录活性分析:带有各种启动子的pgl4.10质粒和pgl4.75质粒共转染细胞,并用双荧光素酶报告基因测定系统检测转染后24小时相对荧光素酶活性(rla),进行五个独立的生物重复(br)实验,结果显示三个技术重复的平均值;(b)启动子的动力学转录活性分析:带有各种启动子的pgl4.10质粒和pgl4.75质粒共转染细胞,并且用双荧光素报告基因测定系统检测转染后不同时间的rla,结果显示三次独立实验的平均值;foldrla的计算方法是mcmv启动子的rla与人wtcmv启动子(wt)的rla的比值;rc,报告载体;nc,阴性对照(没有插入启动子序列的报告载体);
图4为hepg2细胞中多种启动子转录活性分析示意图;其中(a)相对转录活性(rla)检测:带有各种启动子的pgl4.10质粒和pgl4.75质粒共转染细胞,用双荧光素酶报告基因测定系统检测转染后24小时的rla,结果显示三次独立实验的平均值;(b)mcmv启动子的rla相对于人wtcmv的倍数变化:foldrla的计算方法是mcmv启动子的rla与人wtcmv启动子的rla的比值;(c)根据foldrla将启动子分选;rc,报告载体;wt,人wtcmv启动子;nc,阴性对照(没有插入启动子序列的报告载体);
图5为不同细胞系中启动子活性分析示意图;其中(a)双荧光素酶报告基因测定系统分析不同细胞中的启动子活性:带有各种启动子的pgl4.10质粒和pgl4.75质粒共转染细胞;(b)用gaussia荧光素酶报告基因测定系统检测不同细胞中启动子活性:带有各种启动子的pgluc质粒转染细胞,在转染后24小时检测rla,结果显示三次独立实验的平均值;foldrla的计算方法是mcmv启动子的rla与人wtcmv启动子(wt)的rla比值;rla,相对荧光素酶活性;rc,报告载体;nc,阴性对照(没有插入启动子序列的报告载体);
图6为cho细胞中启动子活性分析示意图;其中(a)用双荧光素酶报告基因测定系统分析启动子活性:用带有各种启动子的pgl4.10质粒和pgl4.75质粒共转染细胞,并在转染后24小时检测相对荧光素酶活性(rla),该图显示相对转录活性(rla)(左),rla相对于人wtcmv的倍数变化(中)和根据它们的foldrla进行启动子分选(右);(b)用双荧光素酶报告基因测定系统分析高活力启动子活性:带有不同启动子的pgl4.10质粒和pgl4.75质粒共转染细胞;(c)用gaussia荧光素酶报告基因测定系统检测高活力启动子活性:带有不同启动子的pgluc质粒转染细胞;(d)用双分泌型报告基因测定系统分析启动子动力学活性:带有各种启动子的pezx质粒转染细胞,在转染后不同时间检测rla,结果显示三次独立实验的平均值;foldrla的计算方法是mcmv启动子的rla与人wtcmv启动子(wt)的rla比值;rc,报告载体;nc,阴性对照(没有插入启动子序列的报告载体);
图7为使用egfp作为报告基因分析启动子活性示意图;带有不同启动子的pegfp质粒转染hepg2和cho细胞,荧光显微镜(带有冷电荷耦合器件dp71的ix51)200倍观察拍摄细胞,显示了在明场(左)和荧光(右)下的相同细胞的图像,wt,人wtcmv启动子;con,对照(未转染的细胞);
图8为egfp表达的流式分析示意图;其中(a)在hepg2细胞中用流式细胞仪检测egfp荧光强度:带有不同启动子的pegfp质粒转染细胞,通过胰蛋白酶消化收集细胞,并用流式细胞仪(bdaccuric6)检测,荧光检测的柱形图展示在下面,mfi,平均荧光强度;rc,报告载体;wt,野生型cmv(wtcmv)启动子;br,生物学重复;(b)在cho细胞中用流式细胞仪检测egfp荧光强度:带有不同启动子的pegfp质粒转染细胞,通过胰蛋白酶消化收集细胞并用流式细胞仪(aceanovocyte)检测,荧光检测的柱形图展示于下面,mfi,平均荧光强度;rc,报告载体;wt,人wtcmv启动子;
图9为nf-κb对启动子活性的特异性影响关系图;其中(a)用双荧光素酶报告基因测定系统分析启动子活性;(b)用gaussia荧光素酶报告基因测定系统检测启动子活性:hepg2和cho细胞用tnfα诱导nf-κb或用bay11-7082(bay)抑制nf-κb活性,细胞处理1小时。未处理的细胞用作对照(con),然后用带有不同启动子的pgl4.10质粒和pgl4.75质粒共转染细胞,pgluc质粒单独转染细胞;foldrla的计算方法是mcmv启动子的相对荧光素酶活性(rla)与人wtcmv启动子(wt)的相对荧光素酶活性的比值,结果显示三次独立实验的平均值;
图10为通过hg-csf的分泌表达分析启动子活性示意图;其中(a)hg-csf定量:用带有不同启动子的hg-csf表达载体转染cho细胞,检测收集的培养基中hg-csf表达水平;**,p<0.01(与wt比较);(b)hg-csf的生物活性测定:收集的培养基根据检测的hg-csf浓度进行稀释,统一浓度为15ng/ml,用稀释后的培养基进行nfs-60细胞培养后,检测细胞增殖,cell组中使用的培养基来自于空白对照cho细胞培养基,总蛋白含量与cm组的培养基总蛋白量相同;(c)hg-csf的生物活性测定:将转染不同表达载体后的cho细胞培养基按照相同的体积添加到基础培养基中进行nfs-60细胞培养;cell,添加相同体积的空白对照cho细胞培养基;bm,无hg-csf的基础培养基;cm,含有15ng/ml大肠杆菌来源的hg-csf的完全培养基;nc,阴性对照(cm培养基,无细胞)。**,p<0.01(与cm相比);
图11为表达的hg-csf生物活性测定示意图;其中(a)和(b)为图10c的两个额外的独立生物学重复;将转染过带有不同启动子(wt、t1p2、p2和t1)的质粒后收集的cho细胞的培养基,按照相同的体积添加到不含hg-csf的基础培养基中进行nfs-60细胞培养;cell,基础培养基添加相同体积的空白对照组cho细胞培养基;bm,无hg-csf的基础培养基;cm,含有15ng/ml大肠杆菌来源的hg-csf的完全培养基;nc,阴性对照(cm培养基,无细胞);**,p<0.01(与cm相比)。
具体实施方式
以下结合附图和实施例对本发明作进一步说明。
实施例1
启动子和报告基因表达质粒的构建
启动子构建:通过使用改进的selex-seq方法,成功地表征了nf-κb的dna结合特异性(plosone,2013,8(10):e76109)。通过分析selex-seq数据,发现对nf-κb二聚体具有高亲和力的序列。将pegfp-n1质粒(clontech公司)中的人cmvie启动子用来进行改造,使用在selex-seq实验中发现的高亲和力序列替代其天然nf-κb结合位点。人wtcmv启动子序列seqidno.15含有四个nf-κb结合位点,如表1中的seqidno.16~seqidno.19所示。在改造中使用两个selex选择的高亲和力序列(t和p),如表2中seqidno.20和seqidno.21所示。使用不含有已知tfbs的命名为s的序列,表2中seqidno.22所示作为nf-κb结合位点敲除对照(图1)。
研究中使用pegfp-n1质粒(clontech公司)中的人wtcmv启动子为模板通过融合pcr扩增改造mcmv启动子,pcr引物见表3中的seqidno.23~seqidno.42所示。引物cmv-1-for和cmv-1-re,酶切位点为kpni和hindiii,cmv-2-for和cmv-2-re,酶切位点为asei和nhei,设计用于扩增完整的cmv启动子序列,也用于融合pcr扩增。例如,使用cmv-2-for和t1-re获得前半段的启动子序列,使用cmv-2-re和t1-for获得另一半,随后,将两个pcr产物与引物cmv-2-for和cmv-2-re共同进行pcr得到完整的启动子序列。在最终产物中,第一个nf-κb结合位点被序列t替代。通过使用不同的引物用相同的策略构建了其他mcmv启动子。使用takaraextaqhs聚合酶时,pcr条件如下:在94℃初始变性3分钟,随后94℃变性40秒,57℃退火40秒和72℃延伸30秒,共30个循环,最后72℃延伸5分钟。使用primestarhsdna聚合酶(takara)时,pcr条件如下:95℃变性10秒,68℃退火和延伸1分钟,共30个循环。
具体步骤1:通过selex-seq方法(plosone,2013,8(10):e76109)用于表征转录因子蛋白nf-κb与所有可能结合的碱基的dsdna序列的相对结合亲和力,鉴定出3个人工碱基序列为bs-t:ggggtttccc;bs-p:ggggattccc;bs-s:tagtaacgcc,以下简称t、p和s(表2)。
步骤2:根据文献报道(journalofcellularbiochemistry,2001,83(4):563-573),人wtcmv启动子中有四个nf-κb结合位点,其碱基序列和碱基位置分别为:wtbs1:gggactttcc(-422~-413);wtbs2:gggactttcc(-271~-262);wtbs3:ggggatttcc(-166~-157);wtbs4:gggactttcc(-103~-94)(表1)。
步骤3,mcmv1、mcmv2、mcmv3、mcmv4及mcmv14启动子采用全长化学合成法合成;其他mcmv用融合pcr技术构建,具体扩增方案如下:
mcmv1启动子,用bs-t替代人wtcmv启动子中的wtbs1,用bs-p替代人wtcmv启动子中的wtbs2。mcmv1启动子全长序列由南京金斯瑞公司合成。
mcmv2启动子,用bs-t替代人wtcmv启动子中的wtbs1,用bs-p分别替代人wtcmv启动子中的wtbs2和wtbs4。mcmv2启动子全长序列由南京金斯瑞公司合成。
mcmv3启动子,用bs-s分别替代人wtcmv启动子中的wtbs1和wtbs2。mcmv3启动子全长序列由南京金斯瑞公司合成。
mcmv4启动子,用bs-s分别替代人wtcmv启动子中的wtbs1、wtbs2和wtbs4。mcmv4启动子全长序列由南京金斯瑞公司合成。
mcmv5启动子,用bs-t替代人wtcmv启动子中的wtbs1。通过融合pcr方法获得。wtcmv启动子为模板,使用miep-2-for作为上游引物,t1-re作为下游引物进行pcr获得前半段的启动子序列,使用t1-for作为上游引物,miep-2-re作为下游引物进行pcr获得序列的另一半;随后,将两次pcr的产物同时作为模板,miep-2-for作为上游引物和miep-2-re作为下游引物进行pcr得到完整的启动子序列。
mcmv6启动子,用bs-t替代人wtcmv启动子中的wtbs2。通过融合pcr方法获得。wtcmv启动子为模板,使用miep-2-for作为上游引物,t2-re作为下游引物进行pcr获得前半段的启动子序列,使用t2-for作为上游引物,miep-2-re作为下游引物进行pcr获得序列的另一半;随后,将两次pcr的产物同时作为模板,miep-2-for作为上游引物和miep-2-re作为下游引物进行pcr得到完整的启动子序列。
mcmv7启动子,用bs-t替代人wtcmv启动子中的wtbs3。通过融合pcr方法获得。wtcmv启动子为模板,使用miep-2-for作为上游引物,t3-re作为下游引物进行pcr获得前半段的启动子序列,使用t3-for作为上游引物,miep-2-re作为下游引物进行pcr获得序列的另一半;随后,将两次pcr的产物同时作为模板,miep-2-for作为上游引物和miep-2-re作为下游引物进行pcr得到完整的启动子序列。
mcmv8启动子,用bs-t替代人wtcmv启动子中的wtbs4。通过融合pcr方法获得。wtcmv启动子为模板,使用miep-2-for作为上游引物,t4-re作为下游引物进行pcr获得前半段的启动子序列,使用t4-for作为上游引物,miep-2-re作为下游引物进行pcr获得序列的另一半;随后,将两次pcr的产物同时作为模板,miep-2-for作为上游引物和miep-2-re作为下游引物进行pcr得到完整的启动子序列。
mcmv9启动子,用bs-p替代人wtcmv启动子中的wtbs1。通过融合pcr方法获得。wtcmv启动子为模板,使用miep-2-for作为上游引物,p1-re作为下游引物进行pcr获得前半段的启动子序列;使用p1-for作为上游引物,miep-2-re作为下游引物进行pcr获得序列的另一半;随后,将两次pcr的产物同时作为模板,miep-2-for作为上游引物和miep-2-re作为下游引物进行pcr得到完整的启动子序列。
mcmv10启动子,用bs-p替代人wtcmv启动子中的wtbs2。通过融合pcr方法获得。wtcmv启动子为模板,使用miep-2-for作为上游引物,p2-re作为下游引物进行pcr获得前半段的启动子序列;使用p2-for作为上游引物,miep-2-re作为下游引物进行pcr获得序列的另一半;随后,将两次pcr的产物同时作为模板,miep-2-for作为上游引物和miep-2-re作为下游引物进行pcr得到完整的启动子序列。
mcmv11启动子,用bs-p替代人wtcmv启动子中的wtbs3。通过融合pcr方法获得。wtcmv启动子为模板,使用miep-2-for作为上游引物,p3-re作为下游引物进行pcr获得前半段的启动子序列;使用p3-for作为上游引物,miep-2-re作为下游引物进行pcr获得序列的另一半;随后,将两次pcr的产物同时作为模板,miep-2-for作为上游引物和miep-2-re作为下游引物进行pcr得到完整的启动子序列。
mcmv12启动子,用bs-p替代人wtcmv启动子中的wtbs4。通过融合pcr方法获得。wtcmv启动子为模板,使用miep-2-for作为上游引物,p4-re作为下游引物进行pcr获得前半段的启动子序列;使用p4-for作为上游引物,miep-2-re作为下游引物进行pcr获得序列的另一半;随后,将两次pcr的产物同时作为模板,miep-2-for作为上游引物和miep-2-re作为下游引物进行pcr得到完整的启动子序列。
mcmv13启动子,用bs-p分别替代人wtcmv启动子中的wtbs1和wtbs2。通过融合pcr方法获得。mcmv9启动子为模板,使用miep-2-for作为上游引物,p2-re作为下游引物进行pcr获得前半段的启动子序列;使用p2-for作为上游引物,miep-2-re作为下游引物进行pcr获得序列的另一半;随后,将两次pcr的产物同时作为模板,miep-2-for作为上游引物和miep-2-re作为下游引物进行pcr得到完整的启动子序列。
mcmv14启动子,用bs-p分别替代人wtcmv启动子中的wtbs1和wtbs2,用bs-s分别替代人wtcmv启动子中的wtbs3和wtbs4。mcmv14启动子全长序列由南京金斯瑞公司合成。
质粒构建:质粒pgl4.10表达萤火虫荧光素酶(promega公司),使用kpni和hindiii(fermentas公司)进行酶切,插入实施例1制备得到的不同启动子,得到带有各种启动子的不同的pgl4.10质粒,用于用双荧光素酶报告测定系统(promega)评价启动子活性。用来自pgluc-basic载体(neb公司)的gluc基因序列替换质粒pires-egfp(clontech公司)的ires和egfp序列,构建了表达分泌蛋白gaussia荧光素酶(gluc)的质粒pgluc。使用asei和nhei(takara公司)酶切位点插入改造后的启动子,用于gaussia荧光素酶报告基因测定系统评估启动子的转录活性。质粒pezx-epg04(genecopoeia公司)是能够表达两种分泌蛋白(gaussia荧光素酶(gluc)和碱性磷酸酶(seap))的载体。gluc用于在检测改造启动子控制下的报告基因,seap用作内参对照。通过使用ecori和hindiii(takara公司)酶切位点将改造后的启动子插入载体中,用于双分泌报告基因测定系统评估启动子的转录活性。质粒pegfp-n1(clontech公司)通过asei和nhei(takara公司)酶切位点插入改造后的启动子,用于细胞内表达egfp来评价启动子活性。使用asei和nhei(takara公司)酶切位点将启动子插入pires-egfp载体,通过bamhi和ecori酶切位点将带有6×his标签的人粒细胞集落刺激因子(hg-csf)克隆到pires-egfp质粒中。得到的质粒命名为pires-egfp-gcsf,不同启动子用来控制hg-csf基因的表达。通过测序验证所有质粒的序列。
表1人wtcmv启动子中的nf-κb结合位点
表2用于突变人wtcmv启动子中nf-κb结合位点的序列
表3用于启动子构建的pcr引物
结果:
在wtcmv启动子中有四个nf-κb结合位点(图1)。为了找到具有高转录活性的人工启动子,将不同位置的天然nf-κb结合位点用两个人工选择的高亲和力序列(t和p)进行改造。为了评估nf-κb结合位点对人wtcmv启动子转录活性的贡献,一些nf-κb结合位点也突变为没有已知tfbs的序列(s)。突变策略示意图如图1所示。为了充分评估所制造的启动子的转录活性,使用了四种不同的报告系统。将启动子克隆到pgl4.10,pgluc,pezx和pegfp质粒中,分别用双荧光素酶报告基因测定系统,gaussia荧光素酶报告基因测定系统,双分泌报告基因测定系统和egfp系统分析启动子活性(图2)。构建质粒pires-egfp-gcsf用于表达药物蛋白质hg-csf(图2)。
实施例2
使用hepg2细胞评价启动子
方法:
细胞培养:hepg2细胞使用dmem培养基培养。培养基含有10%(v/v)胎牛血清,100units/ml青霉素和100g/ml链霉素。细胞在37℃,5%(v/v)co2中培养。细胞来自中国科学院上海生命科学研究院细胞资源中心。
胞质表达的报告基因系统评价启动子活性(双荧光素酶检测):将细胞以1×105细胞/孔的密度接种在24孔板中并培养12小时以上。然后用带有各种启动子的pgl4.10质粒(0.5μg/孔)和内参质粒pgl4.75(0.05μg/孔)共转染细胞。将不含插入启动子序列的质粒pgl4.10和内参质粒pgl4.75共转染作为阴性对照。将细胞与lipofectamine2000和共转染质粒dna复合物孵育5小时。然后用不含转染试剂的培养基替换转染培养基,并将细胞继续培养。在转染固定时间点测量荧光素酶活性。将细胞用pbs洗涤两次,收获,并根据双荧光素酶检测试剂盒(promega)说明进行双荧光素酶检测。使用酶标仪(synergyht,biotek)测量萤火虫和海肾荧光素酶的活性。用海肾荧光素酶活性结果对萤火虫荧光素酶活性进行校正从而评估启动子活性。每次试验包括三个实验重复。
结果:
通过用selex选择的具有nf-κb高结合亲和力的序列替换了人wtcmv中1、2和3处天然的nf-κb结合位点,构建突变启动子(t1p2和t1p24)。然后使用双荧光素酶报告基因测定系统在转染后24小时进行检测。结果表明,其中一个突变(t1p2)增强了cmv启动子的活性,但另一个降低了cmv启动子的活性(图3a)。为了证实这些突变对cmv启动子转录活性的影响,通过不具有已知tfbs的序列改变t1p2和t1p24启动子中相应的突变位点,构建了两个新的启动子(s12和s124)。结果显示s12突变显著降低了t1p2活性,使得s12启动子活性变得低于人wtcmv启动子。而s124突变增加了t1p24活性,但s124启动子的活性仍然低于人wtcmv启动子的活性。
此外,还检测了启动子在hepg2细胞中的动力学转录活性。在转染后6小时,12小时,24小时,36小时,48小时和72小时测量相对荧光素酶活性(rla)。结果表明,t1p2启动子在所有时间点都具有最高的转录活性。其活性始终高于人wtcmv启动子(图3b)。t1p24启动子转录活性始终低于人wtcmv启动子。在该动态检测中,s12和s124启动子和人wtcmv启动子的活性之间没有显著差异。结果发现各种启动子的转录活性在转染后24小时达到最大,因此在随后的实验中在该时间点不断测量其他启动子的转录活性。这些初步实验表明,改变nf-κb结合位点可以调节cmv启动子的转录活性,并且以这种方式可以获得具有优异转录活性的新启动子。
实施例3
用hepg2细胞评价其他启动子
方法:
细胞培养:hepg2细胞使用dmem培养基培养。培养基含有10%胎牛血清,100units/ml青霉素和100g/ml链霉素。细胞在37℃,5%co2中培养。细胞来自中国科学院上海生命科学研究院细胞资源中心。
胞质表达的报告基因系统评价启动子活性(双荧光素酶检测):将细胞以1×105细胞/孔的密度接种在24孔板中并培养12小时以上。然后用带有各种启动子的pgl4.10质粒(0.5μg/孔)和内参质粒pgl4.75(0.05μg/孔)共转染细胞。将不含插入启动子序列的质粒pgl4.10和内参质粒pgl4.75共转染作为阴性对照。将细胞与lipofectamine2000和共转染质粒dna复合物孵育5小时。然后用不含转染试剂的培养基替换转染培养基,并将细胞继续培养。在转染固定时间点测量荧光素酶活性。将细胞用pbs洗涤两次,收获,并根据双荧光素酶检测试剂盒(promega)的说明进行双荧光素酶检测。使用酶标仪(synergyht,biotek)测量萤火虫和海肾荧光素酶的活性。用海肾荧光素酶活性结果对萤火虫荧光素酶活性进行校正从而评估启动子活性。每次试验包括三个实验重复。
结果:
基于启动子改造和转录活性测定的初步结果,我们通过系统地改变不同位置的nf-κb结合位点制造了更多的mcmv启动子,分别命名为t1,t2,t3,t4,p1,p2,p3,p4,p12和p12s34。同时在hepg2细胞中检测了这些新的启动子与t1p2启动子的转录活性。用带有各种启动子的pgl4.10质粒和pgl4.75质粒共转染细胞,并检测rla。结果表明,新的启动子t1和p2具有比人wtcmv启动子更高的活性,其中p2启动子活性最强(图4)。试验结果显示,t1p2启动子活性也高于人wtcmv。其他mcmv启动子活性比人wtcmv启动子低,其中t3启动子活性最低,约降低了60%。我们得到了三个比人wtcmv启动子转录活性更高的人工合成启动子,特别是p2启动子,转录活性提升最大。
实施例4
不同细胞系中启动子评价
方法:
细胞培养:hepg2,hela和293t细胞使用dmem培养基培养。cho细胞使用dmem/f121:1培养基。k562细胞。所有培养基含有10%胎牛血清,100units/ml青霉素和100g/ml链霉素。所有细胞在37℃,5%co2中培养。所有细胞均来自中国科学院上海生命科学研究院细胞资源中心。
胞质表达的报告基因系统评价启动子活性(双荧光素酶检测):将细胞以1×105细胞/孔的密度接种在24孔板中并培养12小时以上。然后用带有各种启动子的pgl4.10质粒(0.5μg/孔)和内参质粒pgl4.75(0.05μg/孔)共转染细胞。将不含插入启动子序列的质粒pgl4.10和内参质粒pgl4.75共转染作为阴性对照。将细胞与lipofectamine2000和共转染质粒dna复合物孵育5小时。然后用不含转染试剂的培养基替换转染培养基,并将细胞继续培养。在转染固定时间点测量荧光素酶活性。将细胞用pbs洗涤两次,收获,并根据双荧光素酶检测试剂盒(promega)的说明进行双荧光素酶检测。使用酶标仪(synergyht,biotek)测量萤火虫和海肾荧光素酶的活性。用海肾荧光素酶活性结果对萤火虫荧光素酶活性进行校正从而评估启动子活性。每次试验包括三个实验重复。
启动子活性评价与单分泌报告基因系统(单分泌酶检测):将细胞以1×105细胞/孔的密度接种在24孔板中并培养12小时以上。用带有不同启动子的质粒pgluc(0.8μg/孔)转染细胞,不含任何插入启动子序列的质粒pgluc作为阴性。将细胞与lipofectamine2000和质粒dna复合物孵育5小时。然后用新鲜培养基替换转染培养基,并将细胞继续培养。在转染固定时间点测量荧光素酶活性。使用双荧光素酶报告测定系统(promega)中包含的用于检测海肾萤光素酶活性的试剂检测细胞培养基中gluc的活性。根据说明书,通过bradford蛋白测定试剂盒(beyotime公司)测定相应培养基的总蛋白浓度。通过酶标仪synergyht(biotek)读取光密度。用总蛋白浓度标对gluc活性进行校正,评估启动子活性。每次试验包括三个实验重复。
结果:
人wtcmv启动子在不同的细胞系中基本上没有细胞特异性,为了确定mcmv启动子是否也没有细胞特异性,首先检测了5种哺乳动物细胞系(包括hepg2,hela,k562,cho和293t细胞)中t1p2,p2,t1和人wtcmv启动子的转录活性。
首先用双荧光素酶报告基因测定系统检测启动子的活性。结果表明,在5个不同的细胞中,三个mcmv启动子比人wtcmv启动子具有更强的转录活性(图5a)。在这些启动子中,p2启动子在所有检测的细胞中显示出最高的转录活性。与其他细胞相比,cho和k562细胞中p2的转录活性增强效果更显著。在hela和293t细胞中,mcmv启动子增强转录活性约10~20%。但在hepg2细胞中,mcmv启动子增加了约50~80%。在k562和cho细胞中,p2启动子具有超过人wtcmv启动子2倍以上的活性,t1p2启动子增强约50%,t1启动子增强50%~100%。在cho细胞中,与人wtcmv启动子相比,mcmv启动子转录活性显著增强。这些数据揭示,在所有测量的细胞中,mcmv启动子都比人wtcmv启动子的转录活性更高。
考虑到分泌蛋白在生产中应用性更广,并进一步评估启动子,通过使用gaussia荧光素酶作为报告基因构建了t1p2、p2、t1和人wtcmv启动子的分泌表达载体。用gaussia荧光素酶报告基因测定系统(单分泌酶检测)检测了这些启动子在多个细胞中的转录活性。结果显示三个mcmv启动子在所有细胞中转录活性高于人wtcmv启动子(图5b)。值得注意的是,p2启动子在所有细胞中显示出最高的转录活性。与使用双荧光素酶报告基因检测获得的结果相比,所有mcmv启动子转录活性比人wtcmv启动子高得多。结果显示,p2启动子的转录活性是人wtcmv启动子的大约2.4~4.4倍。这些数据再次证明,在检测的不同细胞系中,mcmv启动子具有比人wtcmv启动子更高的转录活性,且没有明显的细胞特异性。这些数据还说明mcmv启动子可以应用于分泌蛋白的高效表达。
实施例5
cho细胞评价启动子
方法:
细胞培养:cho细胞使用dmem/f12(体积比)1:1培养基。培养基含有10%胎牛血清,100units/ml青霉素和100g/ml链霉素。细胞在37℃,5%co2中培养。细胞来自中国科学院上海生命科学研究院细胞资源中心。
胞质表达的报告基因系统评价启动子活性(双荧光素酶检测):将细胞以1×105细胞/孔的密度接种在24孔板中并培养12小时以上。然后用带有各种启动子的pgl4.10质粒(0.5μg/孔)和内参质粒pgl4.75(0.05μg/孔)共转染细胞。将不含插入启动子序列的质粒pgl4.10和内参质粒pgl4.75共转染作为阴性对照。将细胞与lipofectamine2000和共转染质粒dna复合物孵育5小时。然后用不含转染试剂的培养基替换转染培养基,并将细胞继续培养。在转染固定时间点测量荧光素酶活性。将细胞用pbs洗涤两次,收获,并根据双荧光素酶检测试剂盒(promega公司)的说明进行双荧光素酶检测。使用酶标仪(synergyht,biotek)测量萤火虫和海肾荧光素酶的活性。用海肾荧光素酶活性结果对萤火虫荧光素酶活性进行校正从而评估启动子活性。每次试验包括三个实验重复。
启动子活性评价与单分泌报告基因系统(单分泌酶检测):将细胞以1×105细胞/孔的密度接种在24孔板中并培养12小时以上。用带有不同启动子的质粒pgluc(0.8μg/孔)转染细胞,不含任何插入启动子序列的质粒pgluc作为阴性。将细胞与lipofectamine2000和质粒dna复合物孵育5小时。然后用新鲜培养基替换转染培养基,并将细胞继续培养。在转染固定时间点测量荧光素酶活性。使用双荧光素酶报告测定系统(promega)中包含的用于检测海肾萤光素酶活性的试剂检测细胞培养基中gluc的活性。根据说明书,通过bradford蛋白测定试剂盒(beyotime公司)测定相应培养基的总蛋白浓度。通过酶标仪synergyht(biotek)读取光密度。用总蛋白浓度标对gluc活性进行校正,评估启动子活性。每次试验包括三个实验重复。
双分泌报告基因测定系统进行启动子活性评价(双分泌酶检测):将细胞以1×105细胞/孔的密度接种在24孔板中并培养12小时以上。带有各种启动子的质粒pezx((0.8μg/孔)转染细胞。不含任何插入启动子序列的质粒pezx作为阴性对照。将细胞与lipofectamine2000和共转染质粒dna复合物孵育5小时。然后用不含转染试剂的培养基替换转染培养基。在转染后固定时间点测量荧光素酶活性。根据说明书,使用secrete-pairtm双发光测定试剂盒(genecoporia公司)检测细胞培养基中gluc和seap的活性。通过酶标仪synergyht(biotek)读取吸光度。使用seap活性结果对gluc活性进行校正,评估启动子活性。每次试验包括三个实验重复。
结果:
许多应用领域中,cho细胞是蛋白质真核表达的首选细胞株。因此,通过不同的检测系统评估启动子在该细胞中的转录活性。
首先用双荧光素酶报告基因测定系统评价了启动子在cho细胞中的活性,用带有各种启动子的pgl4.10质粒,包括t1、t2、t3、t4、p1、p2、p3、p4、p12、p12s34、t1p2和人wtcmv,以及pgl4.75质粒共转染细胞,并在转染后24小时测量rla。结果表明,t1、t1p2和p2启动子比wt启动子转录活性更高,其中p2启动子的转录活性最高(图6a)。其他mcmv启动子的活性低于人wtcmv启动子的活性,其中t3启动子活性最低,约降低了47%。不同启动子在细胞中的相对活性与在hepg2细胞中的结果相似。
通过双荧光素酶报告基因测定系统检测启动子的动力学转录活性,用带有t1p2,p2,t1和wtcmv启动子的pgl4.10质粒和pgl4.75质粒共转染细胞。然后在转染后6小时,12小时,24小时,36小时,48小时和72小时测量rla(图6b)。结果显示三个mcmv启动子的活性恒定地高于wtcmv启动子。在不同的时间点,p2启动子的rla都是最高的。转染后24小时,所有突变启动子的rla达到最高值。结果显示p2启动子在转染后24小时显示出了超过2倍的wtcmv启动子活性。
通过gaussia荧光素酶报告基因测定系统(单分泌酶检测)检测了启动子的动力学转录活性,用带有t1p2,p2,t1和wtcmv启动子的pgluc质粒转染细胞。然后在转染后6小时,12小时,24小时,36小时,48小时和72小时测量细胞培养基中的gaussia萤光素酶活性和总蛋白浓度(图6c)。rla计算方法为gaussia荧光素酶活性与总蛋白浓度的比值。结果显示,转染后24小时,所有突变启动子的蛋白表达水平都比人wtcmv启动子高。转染后48小时,所有突变启动子的rla达到最高值。转染后24小时,所有mcmv启动子的蛋白表达水平都是人wtcmv启动子的2倍以上。转染后72小时,所有mcmv启动子的蛋白表达水平高达人wtcmv启动子的6倍。有趣的是,在本评价系统中,三个mcmv启动子的转录活性之间没有显著差异,三个mcmv启动子的蛋白表达水平都比人wtcmv启动子更高。
最后,通过用双分泌报告基因测定系统检测启动子的动力学转录活性。使用带有t1p2,p2,t1和人wtcmv启动子的pezx质粒转染cho细胞。在转染后6小时,12小时,24小时,36小时,48小时和72小时,用双分泌报告基因测定系统检测启动子活性。结果表明,mcmv启动子的活性在转染后24小时显著增强(图6d)。所有mcmv启动子的rla在转染后72小时达到最高值,但是三个启动子的foldrla在转染后12小时达到最高值。此时,t1和p2启动子的活性分别达到人wtcmv启动子的2.2和2.8倍,同时,t1p2启动子转录活性增强约60%。在所有检测的时间范围内,p2启动子都具有最高的转录活性,t1启动子的活性高于t1p2启动子。显然,启动子的活性特征不同于使用单分泌报告系统得到的结果,但是,用双分泌报告基因测定系统的检测也证明了这些启动子对于表达分泌蛋白具有重要的应用价值。
实施例6
用egfp报告基因可视化启动子活性检测
方法:
细胞培养:hepg2细胞使用dmem培养基培养。cho细胞使用dmem/f121:1培养基。所有培养基含有10%胎牛血清,100units/ml青霉素和100g/ml链霉素。所有细胞在37℃,5%co2中培养。所有细胞均来自中国科学院上海生命科学研究院细胞资源中心。
细胞处理:将细胞以1×105细胞/孔的密度接种在24孔板中并培养12小时以上。用带有不同启动子的pegfp-n1载体(0.8μg)转染细胞。将细胞与lipofectamine2000和共转染质粒dna复合物孵育5小时。然后用不含转染试剂的新鲜培养基替换转染培养基,并将细胞继续培养。在转染后固定时间点用egfp的表达情况评价启动子活性。用荧光显微镜ix51(olympus)和激光扫描共焦显微镜(leicasp8)以200倍放大率对细胞进行拍照。通过胰蛋白酶消化收集细胞,并用流式细胞仪accuric6(bd)或novocyte(acea)测量平均荧光强度(mfi)。
结果:
为了用可视化形式进一步评估启动子活性,通过egfp作为报告基因,在pegfp-n1载体中用t1p2,p2和t1启动子替换wt启动子,使用带有不同启动子的pegfp-n1质粒转染hepg2和cho细胞。结果表明egfp在两个细胞中高水平表达(图7)。通过流式细胞仪定量检测了egfp表达水平。两个测量的结果都表明,t1p2,p2和t1启动子可以在两个细胞中高水平表达(图8)。然而,尽管这些mcmv启动子在细胞中具有更高的egfp表达水平,但是这些启动子和人wtcmv启动子之间没有统计学上的显著差异。这与在上述双荧光素酶报告基因测定中观察到的结果类似。综上所述,这些数据表明t1p2,p2和t1启动子在哺乳动物细胞中具有高转录活性。
实施例7
nf-κb对启动子活性影响的特异性
方法:
细胞培养:hepg2细胞使用dmem培养基培养。cho细胞使用dmem/f121:1培养基。所有培养基含有10%胎牛血清,100units/ml青霉素和100g/ml链霉素。所有细胞在37℃,5%co2中培养。所有细胞均来自中国科学院上海生命科学研究院细胞资源中心。
细胞处理:将细胞以1×105细胞/孔的密度接种在24孔板中并培养12小时以上。用nf-κb诱导剂tnfα(promega公司)(5ng/ml)和nf-κb抑制剂bay11-7082(sigma-aldrich公司)(10μm)处理细胞1小时。除去培养基,向培养物中加入新鲜的基础培养基。然后用带有不同启动子的pgl4.10质粒和pgl4.75质粒共转染细胞,或用带有不同启动子的pgluc质粒转染细胞。将细胞与lipofectamine2000和共转染质粒dna复合物孵育5小时。然后用不含转染试剂的培养基替换转染培养基,并将细胞继续培养。在转染后固定时间点测量荧光素酶活性。通过双荧光素酶报告基因测定系统或gaussia萤光素酶报告基因测定系统评价启动子活性。
结果:
因为通过改变wtcmv启动子中的nf-κb结合位点来构建了新型mcmv启动子,说明这些启动子具有一定的nf-κb特异性。为此,通过用两种公认的nf-κb活性调节剂,nf-κb诱导剂tnfα和nf-κb抑制剂bay11-7082处理细胞来操纵细胞内nf-κb活化水平。细胞首先用tnfα或bay11-7082处理,然后用带有各种启动子的pgl4.10质粒和pgl4.75质粒共转染。用双荧光素酶报告基因分析启动子活性。结果表明,tnfα明显增强了hepg2细胞中p2,t1和人wtcmv启动子和cho细胞中所有启动子的转录活性(图9a)。结果还显示,所有启动子的转录活性在两个细胞中几乎完全被bay11-7082抑制(图9a)。
为了进一步证实这些启动子的nf-κb特异性,使用gaussia荧光素酶报告基因测定系统进行了类似的检测。带有不同启动子的pgluc质粒转染用tnfα和bay11-7082预处理的细胞。然后用gaussia萤光素酶报告系统检测启动子活性。在两个细胞中观察到类似于双荧光素酶报告基因测定的结果(图9b)。然而,通过tnfα刺激,启动子活性没有显着增强。这些数据证明了nf-κb在启动子活性变化中的特异性影响。
实施例8
通过hg-csf的表达评价启动子活性
方法:
细胞培养:cho细胞使用dmem/f121:1培养基。nfs-60细胞,使用1640培养基添加15ng/mlhg-csf(大肠杆菌来源)。所有培养基含有10%胎牛血清,100units/ml青霉素和100g/ml链霉素。所有细胞在37℃,5%co2中培养。所有细胞均来自中国科学院上海生命科学研究院细胞资源中心。
细胞处理:cho细胞在细胞培养皿(100mm×20mm)中培养,使用lipofectamine2000(invitrogen公司)转染带有不同启动子的pires-egfp-gcsf质粒。在转染后48小时收集培养基。使用hg-csfelisa试剂盒(boster公司),直接elisa定量法检测培养基中的hg-csf蛋白浓度。使用bradford试剂测定培养基中的总蛋白含量。用来进行elisa定量的培养基中总蛋白含量一致(100μg)。使用酶标仪(biotek)读取试验数据,测定hg-csf的浓度。通过小鼠骨髓白血病细胞(nfs-60)的增殖活性评价hg-csf的生物活性。先在37℃,5%co2条件下培养nfs-60细胞,使用rpmi1640培养基含有10%胎牛血清和15ng/mlhg-csf(大肠杆菌来源)。收集细胞,使用血细胞计数器计数,按照5×103细胞数量分装,并离心沉淀。然后,将细胞用不同成分的培养基悬浮。将细胞一式三份接种在96孔板中并培养48小时。通过cck-8细胞增殖-毒性测定试剂盒(dojindo公司)检测细胞的增殖活性。
结果:
最后,进一步通过药物蛋白hg-csf的分泌表达评价启动子活性。通过将全长hg-csf基因克隆到pires-egfp质粒中构建了hg-csf的分泌表达载体。为了纯化表达的hg-csf蛋白,将6×his标签融合至n端。将各种启动子序列克隆到pires-egfp-gcsf中。带有各种启动子的pires-egfp-gcsf质粒转染cho细胞。转染后48小时,用elisa对培养基中的hg-csf蛋白进行定量。结果表明,p2启动子转染的细胞产生最高浓度的hg-csf(88.1ng/ml),wt启动子产生最低浓度的hg-csf(45.8ng/ml)(图10a)。由带有t1p2和t1启动子的pires-egfp-gcsf质粒转染的细胞中hg-csf浓度分别为63.3ng/ml和69.6ng/ml。显然,三个突变的cmv启动子比人wtcmv启动子具有更高的生产力。与其他评价系统中相同,p2启动子显示出比人wtcmv启动子高约两倍的蛋白表达能力(p<0.01)。
接下来使用hg-csf药物工业中检测的标准方法,用nfs-60细胞检测了各种启动子产生的hg-csf蛋白的生物活性。通过两种方式评估hg-csf活性,首先,用相同蛋白浓度的由各种启动子产生的含有hg-csf蛋白的培养基,以及含有商业化hg-csf蛋白(peprotech公司)的培养基分别培养nfs-60细胞。nfs-60细胞增殖检测表明,由各种启动子产生的hg-csf蛋白具有与纯化的hg-csf蛋白相似的生物活性(图10b)。然后,用含有商业化hg-csf蛋白的培养基,以及含有相同体积的各种cho细胞培养基的不同培养基进行nfs-60细胞培养。结果显示由各种启动子产生的hg-csf蛋白在三种不同的检测剂量下显示相似的生物活性(图10c),表明由这些启动子产生的hg-csf蛋白具有较高的生物活性。本实施例还发现在三种不同剂量的三次生物重复(图10c和图11)中,在各种cho细胞培养基中存在恒定的生物活性差异,这与在各种cho细胞培养基中检测的hg-csf浓度一致(图10a)。
sequencelisting
<110>东南大学
<120>一种真核启动子及其制备方法和应用
<130>2017
<160>42
<170>patentinversion3.3
<210>1
<211>596
<212>dna
<213>人工合成
<400>1
tagttattaatagtaatcaattacggggtcattagttcatagcccatatatggagttccg60
cgttacataacttacggtaaatggcccgcctggctgaccgcccaacgacccccgcccatt120
gacgtcaataatgacgtatgttcccatagtaacgccaataggggtttcccattgacgtca180
atgggtggagtatttacggtaaactgcccacttggcagtacatcaagtgtatcatatgcc240
aagtacgccccctattgacgtcaatgacggtaaatggcccgcctggcattatgcccagta300
catgaccttatggggattccctacttggcagtacatctacgtattagtcatcgctattac360
catggtgatgcggttttggcagtacatcaatgggcgtggatagcggtttgactcacgggg420
atttccaagtctccaccccattgacgtcaatgggagtttgttttggcaccaaaatcaacg480
ggactttccaaaatgtcgtaacaactccgccccattgacgcaaatgggcggtaggcgtgt540
acggtgggaggtctatataagcagagctggtttagtgaaccgtcagatccgctagc596
<210>2
<211>596
<212>dna
<213>人工合成
<400>2
tagttattaatagtaatcaattacggggtcattagttcatagcccatatatggagttccg60
cgttacataacttacggtaaatggcccgcctggctgaccgcccaacgacccccgcccatt120
gacgtcaataatgacgtatgttcccatagtaacgccaataggggtttcccattgacgtca180
atgggtggagtatttacggtaaactgcccacttggcagtacatcaagtgtatcatatgcc240
aagtacgccccctattgacgtcaatgacggtaaatggcccgcctggcattatgcccagta300
catgaccttatggggattccctacttggcagtacatctacgtattagtcatcgctattac360
catggtgatgcggttttggcagtacatcaatgggcgtggatagcggtttgactcacgggg420
atttccaagtctccaccccattgacgtcaatgggagtttgttttggcaccaaaatcaacg480
gggattcccaaaatgtcgtaacaactccgccccattgacgcaaatgggcggtaggcgtgt540
acggtgggaggtctatataagcagagctggtttagtgaaccgtcagatccgctagc596
<210>3
<211>596
<212>dna
<213>人工合成
<400>3
tagttattaatagtaatcaattacggggtcattagttcatagcccatatatggagttccg60
cgttacataacttacggtaaatggcccgcctggctgaccgcccaacgacccccgcccatt120
gacgtcaataatgacgtatgttcccatagtaacgccaatatagtaacgccattgacgtca180
atgggtggagtatttacggtaaactgcccacttggcagtacatcaagtgtatcatatgcc240
aagtacgccccctattgacgtcaatgacggtaaatggcccgcctggcattatgcccagta300
catgaccttattagtaacgcctacttggcagtacatctacgtattagtcatcgctattac360
catggtgatgcggttttggcagtacatcaatgggcgtggatagcggtttgactcacgggg420
atttccaagtctccaccccattgacgtcaatgggagtttgttttggcaccaaaatcaacg480
ggactttccaaaatgtcgtaacaactccgccccattgacgcaaatgggcggtaggcgtgt540
acggtgggaggtctatataagcagagctggtttagtgaaccgtcagatccgctagc596
<210>4
<211>596
<212>dna
<213>人工合成
<400>4
tagttattaatagtaatcaattacggggtcattagttcatagcccatatatggagttccg60
cgttacataacttacggtaaatggcccgcctggctgaccgcccaacgacccccgcccatt120
gacgtcaataatgacgtatgttcccatagtaacgccaatatagtaacgccattgacgtca180
atgggtggagtatttacggtaaactgcccacttggcagtacatcaagtgtatcatatgcc240
aagtacgccccctattgacgtcaatgacggtaaatggcccgcctggcattatgcccagta300
catgaccttattagtaacgcctacttggcagtacatctacgtattagtcatcgctattac360
catggtgatgcggttttggcagtacatcaatgggcgtggatagcggtttgactcacgggg420
atttccaagtctccaccccattgacgtcaatgggagtttgttttggcaccaaaatcaact480
agtaacgccaaaatgtcgtaacaactccgccccattgacgcaaatgggcggtaggcgtgt540
acggtgggaggtctatataagcagagctggtttagtgaaccgtcagatccgctagc596
<210>5
<211>596
<212>dna
<213>人工合成
<400>5
tagttattaatagtaatcaattacggggtcattagttcatagcccatatatggagttccg60
cgttacataacttacggtaaatggcccgcctggctgaccgcccaacgacccccgcccatt120
gacgtcaataatgacgtatgttcccatagtaacgccaataggggtttcccattgacgtca180
atgggtggagtatttacggtaaactgcccacttggcagtacatcaagtgtatcatatgcc240
aagtacgccccctattgacgtcaatgacggtaaatggcccgcctggcattatgcccagta300
catgaccttatgggactttcctacttggcagtacatctacgtattagtcatcgctattac360
catggtgatgcggttttggcagtacatcaatgggcgtggatagcggtttgactcacgggg420
atttccaagtctccaccccattgacgtcaatgggagtttgttttggcaccaaaatcaacg480
ggactttccaaaatgtcgtaacaactccgccccattgacgcaaatgggcggtaggcgtgt540
acggtgggaggtctatataagcagagctggtttagtgaaccgtcagatccgctagc596
<210>6
<211>596
<212>dna
<213>人工合成
<400>6
tagttattaatagtaatcaattacggggtcattagttcatagcccatatatggagttccg60
cgttacataacttacggtaaatggcccgcctggctgaccgcccaacgacccccgcccatt120
gacgtcaataatgacgtatgttcccatagtaacgccaatagggactttccattgacgtca180
atgggtggagtatttacggtaaactgcccacttggcagtacatcaagtgtatcatatgcc240
aagtacgccccctattgacgtcaatgacggtaaatggcccgcctggcattatgcccagta300
catgaccttatggggtttccctacttggcagtacatctacgtattagtcatcgctattac360
catggtgatgcggttttggcagtacatcaatgggcgtggatagcggtttgactcacgggg420
atttccaagtctccaccccattgacgtcaatgggagtttgttttggcaccaaaatcaacg480
ggactttccaaaatgtcgtaacaactccgccccattgacgcaaatgggcggtaggcgtgt540
acggtgggaggtctatataagcagagctggtttagtgaaccgtcagatccgctagc596
<210>7
<211>596
<212>dna
<213>人工合成
<400>7
tagttattaatagtaatcaattacggggtcattagttcatagcccatatatggagttccg60
cgttacataacttacggtaaatggcccgcctggctgaccgcccaacgacccccgcccatt120
gacgtcaataatgacgtatgttcccatagtaacgccaatagggactttccattgacgtca180
atgggtggagtatttacggtaaactgcccacttggcagtacatcaagtgtatcatatgcc240
aagtacgccccctattgacgtcaatgacggtaaatggcccgcctggcattatgcccagta300
catgaccttatgggactttcctacttggcagtacatctacgtattagtcatcgctattac360
catggtgatgcggttttggcagtacatcaatgggcgtggatagcggtttgactcacgggg420
tttcccaagtctccaccccattgacgtcaatgggagtttgttttggcaccaaaatcaacg480
ggactttccaaaatgtcgtaacaactccgccccattgacgcaaatgggcggtaggcgtgt540
acggtgggaggtctatataagcagagctggtttagtgaaccgtcagatccgctagc596
<210>8
<211>596
<212>dna
<213>人工合成
<400>8
tagttattaatagtaatcaattacggggtcattagttcatagcccatatatggagttccg60
cgttacataacttacggtaaatggcccgcctggctgaccgcccaacgacccccgcccatt120
gacgtcaataatgacgtatgttcccatagtaacgccaatagggactttccattgacgtca180
atgggtggagtatttacggtaaactgcccacttggcagtacatcaagtgtatcatatgcc240
aagtacgccccctattgacgtcaatgacggtaaatggcccgcctggcattatgcccagta300
catgaccttatgggactttcctacttggcagtacatctacgtattagtcatcgctattac360
catggtgatgcggttttggcagtacatcaatgggcgtggatagcggtttgactcacgggg420
atttccaagtctccaccccattgacgtcaatgggagtttgttttggcaccaaaatcaacg480
gggtttcccaaaatgtcgtaacaactccgccccattgacgcaaatgggcggtaggcgtgt540
acggtgggaggtctatataagcagagctggtttagtgaaccgtcagatccgctagc596
<210>9
<211>596
<212>dna
<213>人工合成
<400>9
tagttattaatagtaatcaattacggggtcattagttcatagcccatatatggagttccg60
cgttacataacttacggtaaatggcccgcctggctgaccgcccaacgacccccgcccatt120
gacgtcaataatgacgtatgttcccatagtaacgccaataggggattcccattgacgtca180
atgggtggagtatttacggtaaactgcccacttggcagtacatcaagtgtatcatatgcc240
aagtacgccccctattgacgtcaatgacggtaaatggcccgcctggcattatgcccagta300
catgaccttatgggactttcctacttggcagtacatctacgtattagtcatcgctattac360
catggtgatgcggttttggcagtacatcaatgggcgtggatagcggtttgactcacgggg420
atttccaagtctccaccccattgacgtcaatgggagtttgttttggcaccaaaatcaacg480
ggactttccaaaatgtcgtaacaactccgccccattgacgcaaatgggcggtaggcgtgt540
acggtgggaggtctatataagcagagctggtttagtgaaccgtcagatccgctagc596
<210>10
<211>596
<212>dna
<213>人工合成
<400>10
tagttattaatagtaatcaattacggggtcattagttcatagcccatatatggagttccg60
cgttacataacttacggtaaatggcccgcctggctgaccgcccaacgacccccgcccatt120
gacgtcaataatgacgtatgttcccatagtaacgccaatagggactttccattgacgtca180
atgggtggagtatttacggtaaactgcccacttggcagtacatcaagtgtatcatatgcc240
aagtacgccccctattgacgtcaatgacggtaaatggcccgcctggcattatgcccagta300
catgaccttatggggattccctacttggcagtacatctacgtattagtcatcgctattac360
catggtgatgcggttttggcagtacatcaatgggcgtggatagcggtttgactcacgggg420
atttccaagtctccaccccattgacgtcaatgggagtttgttttggcaccaaaatcaacg480
ggactttccaaaatgtcgtaacaactccgccccattgacgcaaatgggcggtaggcgtgt540
acggtgggaggtctatataagcagagctggtttagtgaaccgtcagatccgctagc596
<210>11
<211>596
<212>dna
<213>人工合成
<400>11
tagttattaatagtaatcaattacggggtcattagttcatagcccatatatggagttccg60
cgttacataacttacggtaaatggcccgcctggctgaccgcccaacgacccccgcccatt120
gacgtcaataatgacgtatgttcccatagtaacgccaatagggactttccattgacgtca180
atgggtggagtatttacggtaaactgcccacttggcagtacatcaagtgtatcatatgcc240
aagtacgccccctattgacgtcaatgacggtaaatggcccgcctggcattatgcccagta300
catgaccttatgggactttcctacttggcagtacatctacgtattagtcatcgctattac360
catggtgatgcggttttggcagtacatcaatgggcgtggatagcggtttgactcacgggg420
attcccaagtctccaccccattgacgtcaatgggagtttgttttggcaccaaaatcaacg480
ggactttccaaaatgtcgtaacaactccgccccattgacgcaaatgggcggtaggcgtgt540
acggtgggaggtctatataagcagagctggtttagtgaaccgtcagatccgctagc596
<210>12
<211>596
<212>dna
<213>人工合成
<400>12
tagttattaatagtaatcaattacggggtcattagttcatagcccatatatggagttccg60
cgttacataacttacggtaaatggcccgcctggctgaccgcccaacgacccccgcccatt120
gacgtcaataatgacgtatgttcccatagtaacgccaatagggactttccattgacgtca180
atgggtggagtatttacggtaaactgcccacttggcagtacatcaagtgtatcatatgcc240
aagtacgccccctattgacgtcaatgacggtaaatggcccgcctggcattatgcccagta300
catgaccttatgggactttcctacttggcagtacatctacgtattagtcatcgctattac360
catggtgatgcggttttggcagtacatcaatgggcgtggatagcggtttgactcacgggg420
atttccaagtctccaccccattgacgtcaatgggagtttgttttggcaccaaaatcaacg480
gggattcccaaaatgtcgtaacaactccgccccattgacgcaaatgggcggtaggcgtgt540
acggtgggaggtctatataagcagagctggtttagtgaaccgtcagatccgctagc596
<210>13
<211>596
<212>dna
<213>人工合成
<400>13
tagttattaatagtaatcaattacggggtcattagttcatagcccatatatggagttccg60
cgttacataacttacggtaaatggcccgcctggctgaccgcccaacgacccccgcccatt120
gacgtcaataatgacgtatgttcccatagtaacgccaataggggattcccattgacgtca180
atgggtggagtatttacggtaaactgcccacttggcagtacatcaagtgtatcatatgcc240
aagtacgccccctattgacgtcaatgacggtaaatggcccgcctggcattatgcccagta300
catgaccttatggggattccctacttggcagtacatctacgtattagtcatcgctattac360
catggtgatgcggttttggcagtacatcaatgggcgtggatagcggtttgactcacgggg420
atttccaagtctccaccccattgacgtcaatgggagtttgttttggcaccaaaatcaacg480
ggactttccaaaatgtcgtaacaactccgccccattgacgcaaatgggcggtaggcgtgt540
acggtgggaggtctatataagcagagctggtttagtgaaccgtcagatccgctagc596
<210>14
<211>596
<212>dna
<213>人工合成
<400>14
tagttattaatagtaatcaattacggggtcattagttcatagcccatatatggagttccg60
cgttacataacttacggtaaatggcccgcctggctgaccgcccaacgacccccgcccatt120
gacgtcaataatgacgtatgttcccatagtaacgccaataggggattcccattgacgtca180
atgggtggagtatttacggtaaactgcccacttggcagtacatcaagtgtatcatatgcc240
aagtacgccccctattgacgtcaatgacggtaaatggcccgcctggcattatgcccagta300
catgaccttatggggattccctacttggcagtacatctacgtattagtcatcgctattac360
catggtgatgcggttttggcagtacatcaatgggcgtggatagcggtttgactcactagt420
aacgccaagtctccaccccattgacgtcaatgggagtttgttttggcaccaaaatcaact480
agtaacgccaaaatgtcgtaacaactccgccccattgacgcaaatgggcggtaggcgtgt540
acggtgggaggtctatataagcagagctggtttagtgaaccgtcagatccgctagc596
<210>15
<211>596
<212>dna
<213>人工合成
<400>15
tagttattaatagtaatcaattacggggtcattagttcatagcccatatatggagttccg60
cgttacataacttacggtaaatggcccgcctggctgaccgcccaacgacccccgcccatt120
gacgtcaataatgacgtatgttcccatagtaacgccaatagggactttccattgacgtca180
atgggtggagtatttacggtaaactgcccacttggcagtacatcaagtgtatcatatgcc240
aagtacgccccctattgacgtcaatgacggtaaatggcccgcctggcattatgcccagta300
catgaccttatgggactttcctacttggcagtacatctacgtattagtcatcgctattac360
catggtgatgcggttttggcagtacatcaatgggcgtggatagcggtttgactcacgggg420
atttccaagtctccaccccattgacgtcaatgggagtttgttttggcaccaaaatcaacg480
ggactttccaaaatgtcgtaacaactccgccccattgacgcaaatgggcggtaggcgtgt540
acggtgggaggtctatataagcagagctggtttagtgaaccgtcagatccgctagc596
<210>16
<211>10
<212>dna
<213>人工合成
<400>16
gggactttcc10
<210>17
<211>10
<212>dna
<213>人工合成
<400>17
gggactttcc10
<210>18
<211>10
<212>dna
<213>人工合成
<400>18
ggggatttcc10
<210>19
<211>10
<212>dna
<213>人工合成
<400>19
gggactttcc10
<210>20
<211>10
<212>dna
<213>人工合成
<400>20
ggggtttccc10
<210>21
<211>10
<212>dna
<213>人工合成
<400>21
ggggattccc10
<210>22
<211>10
<212>dna
<213>人工合成
<400>22
tagtaacgcc10
<210>23
<211>29
<212>dna
<213>人工合成
<400>23
ggggtaccccagtaatcaattacggggtc29
<210>24
<211>30
<212>dna
<213>人工合成
<400>24
cccaagcttggggctagcggatctgacggt30
<210>25
<211>28
<212>dna
<213>人工合成
<400>25
cccattaatagtaatcaattacggggtc28
<210>26
<211>30
<212>dna
<213>人工合成
<400>26
ctagctagctaggctagcggatctgacggt30
<210>27
<211>43
<212>dna
<213>人工合成
<400>27
tagtaacgccaataggggtttcccattgacgtcaatgggtgga43
<210>28
<211>44
<212>dna
<213>人工合成
<400>28
attgacgtcaatgggaaacccctattggcgttactatgggaaca44
<210>29
<211>45
<212>dna
<213>人工合成
<400>29
tacatgaccttatggggtttccctacttggcagtacatctacgta45
<210>30
<211>45
<212>dna
<213>人工合成
<400>30
atgtactgccaagtagggaaaccccataaggtcatgtactgggca45
<210>31
<211>44
<212>dna
<213>人工合成
<400>31
tggatagcggtttgactcacggggtttcccaagtctccacccca44
<210>32
<211>43
<212>dna
<213>人工合成
<400>32
aatggggtggagacttgggaaaccccgtgagtcaaaccgctat43
<210>33
<211>44
<212>dna
<213>人工合成
<400>33
ttggcaccaaaatcaacggggtttcccaaaatgtcgtaacaact44
<210>34
<211>44
<212>dna
<213>人工合成
<400>34
agttgttacgacattttgggaaaccccgttgattttggtgccaa44
<210>35
<211>43
<212>dna
<213>人工合成
<400>35
tagtaacgccaataggggattcccattgacgtcaatgggtgga43
<210>36
<211>44
<212>dna
<213>人工合成
<400>36
attgacgtcaatgggaatcccctattggcgttactatgggaaca44
<210>37
<211>45
<212>dna
<213>人工合成
<400>37
tacatgaccttatggggattccctacttggcagtacatctacgta45
<210>38
<211>44
<212>dna
<213>人工合成
<400>38
atgtactgccaagtgggaatccccataaggtcatgtactgggca44
<210>39
<211>44
<212>dna
<213>人工合成
<400>39
tggatagcggtttgactcacggggattcccaagtctccacccca44
<210>40
<211>43
<212>dna
<213>人工合成
<400>40
aatggggtggagacttgggaatccccgtgagtcaaaccgctat43
<210>41
<211>44
<212>dna
<213>人工合成
<400>41
ttggcaccaaaatcaacggggattcccaaaatgtcgtaacaact44
<210>42
<211>44
<212>dna
<213>人工合成
<400>42
agttgttacgacattttgggaatccccgttgattttggtgccaa44
- 还没有人留言评论。精彩留言会获得点赞!