配子发生的制作方法
本发明涉及活体外诱导配子发生的方法。还提供用于本发明的所述方法中的试剂和试剂盒。本发明适用于医学领域,特定来说适用于不孕症的研究和治疗。
背景技术:
配子发生是借以产生配子的过程。在动物中,配子发生通过性腺(雄性为睪丸;雌性为卵巢)中的减数分裂胜任生殖母细胞的分裂和分化进行。在雄性中,精子发生在睪丸中发生,以在涉及减数分裂和有丝分裂的多步骤过程中由精原干细胞(spermatogonialstemcell,ssc)产生精子。ssc源自产后睪丸中的生殖母细胞,且这些生殖母细胞转而源自原始生殖细胞(primordialgermcell,pgc;phillips等人,2010),其在胚胎发生期间迁移至生殖嵴。
在小鼠中,pgc(生殖母细胞的胚胎前体)的特化在胚胎第6.25天(e6.25)开始1。在特化之后,新生pgc进行明显的全域表观遗传变化2-9,包括基因组5-甲基胞嘧啶(5mc)的全域降低3,6,7,10。pgc在跨发育中胚胎各个期间的迁移之后,一旦到达发育中胚胎性腺,则继续进行包含全域dna去甲基化的表观遗传再程序化。性腺pgc的这个dna去甲基化中牵涉的分子机制已成为重点研究的焦点3,4,6,12-19,21,且近年来公开的观测表明,5mc加氧酶tet1是参与性腺pgc中正确进展dna去甲基化的关键因子12,14,16,17。然而,这个表观遗传再程序化的精确性质仍然难以理解。最新研究已显示(hill等人,2018),性腺表观遗传再程序化关键性地参与pgc-生殖母细胞的转化,所述转化是产生减数分裂胜任生殖母细胞(且因此允许配子发生的起始)所需要的。重要的是,性腺再程序化过程代表一种直到近年来才仅在性腺体细胞环境下被克服的屏障5,24,25,27。
最新研究已报道通过若干生殖系相关基因的诱导性表达将体细胞前体细胞转化为减数分裂胜任细胞(medrano等人,2016)。其它研究已将tet1鉴定为在雌性配子发生的活化期间调控某些生殖系相关基因方面的关键因子16。然而,尚未显示对tet1表达的操纵足以将体细胞前体细胞转化为减数分裂胜任细胞。
在人类中,不孕症是一个重大健康问题。举例来说,雄性不孕症影响7%的群体,其中约10%的不育男性为无精子症(galdon等人,2016)。减数分裂胜任细胞的提供代表活体外再现配子发生的一个重要步骤,其将应用于研究和医学,特定来说在不孕症的情形下。
技术实现要素:
本发明人已发现,有效活化从pgc进展为生殖系发育的生殖母细胞阶段所需的一组基因(所述基因在本文中和在hill等人2018中称为“生殖系再程序化应答基因(germlinereprogramming-responsive(grr)基因)”)需要两个不同生物化学条件。这些基因(也为体细胞前体细胞、多能细胞或早期生殖细胞转化为减数分裂胜任细胞所需的)可通过首先减少dna甲基化且其次去除多梳蛋白驱动的抑制来活化。一旦这些生物化学条件就位,则包含表观遗传活化因子tet1的转录因子和活化因子能够驱动grr基因表达。转录活化因子(如tet1)的募集和/或grr基因的表达指示前体(体细胞)细胞转化为减数分裂胜任细胞。
因此,在第一方面中,本发明提供一种生成减数分裂胜任细胞的活体外方法,所述方法包括:
(i)提供前体细胞,
(ii)抑制所述前体细胞的基因组dna的甲基化,
(iii)用多梳蛋白抑制复合物的抑制剂处理所述前体细胞,且随后
(iv)使所述前体细胞繁殖一段时间且在适合于所述前体细胞变为减数分裂胜任细胞的培养条件下繁殖;
其中步骤(ii)和步骤(iii)可以同时或按任一次序依序进行。
在一些实施例中,前体细胞是来源于已从个体获得的样本。前体细胞可以是干细胞、原始生殖细胞样细胞(primordialgermcell-likecell,pgclc)或早期生殖细胞。在一些实施例中,干细胞是诱导性多能干细胞(inducedpluripotentstem(ips)细胞)或精原干细胞
在一些实施例中,抑制步骤(ii)和处理步骤(iii)使得在繁殖步骤(iv)期间诱导前体细胞表达生殖系再程序化应答(grr)基因。grr基因的表达可能与转录活化因子(例如tet1)的募集相关或通过转录活化因子的募集来诱导。tet1可由前体细胞表达,和/或tet1可外源地提供(例如通过递送外源地表达tet1的核酸、通过增强或刺激tet1的内源性表达和/或通过以外源性蛋白质形式提供tet1)。外源提供的tet1可呈靶向一个或多个特定基因组区域的融合构建体形式。举例来说,tet1融合构建体可靶向参与表达本文所公开的一个或多个grr基因中的的启动子或增强子序列。提供有效水平的tet1作为转录活化因子增强grr基因的表达。本发明方法使得能够使grr基因表达得到增强,且这些方法可包含增加或诱导tet1表达和/或将tet1靶向一个或多个grr基因。
本发明方法也可包含检测和/或量化细胞中的一个或多个grr基因的表达水平。grr基因在表1中列出。用于检测和/或量化表达水平的方法为本领域所熟知的。举例来说,可例如通过rt-pcr测量基因的mrna水平。可例如通过如elisa的测试测量蛋白质表达水平。可在前体细胞转化为减数分裂胜任细胞之前、期间或之后测量一个或多个grr基因的表达。优选地,测量在步骤(iv)之后减数分裂胜任细胞中的一个或多个grr基因的表达。待测量的grr基因可以是以下中的一个或多个:dazl、hormad1、sycp2、sycp3、mae1、fkbp6(参见表1)。在本发明的一些实施例中,多梳蛋白抑制复合物的抑制剂是prc1抑制剂(意味着prc1复合物被选择性地抑制)。在本发明的其它实施例中,多梳蛋白抑制复合物的抑制剂是prc2抑制剂(意味着prc2复合物被选择性地抑制)。在又其它实施例中,多梳蛋白抑制复合物的抑制剂抑制prc1和prc2两个。
在一些实施例中,多梳蛋白抑制复合物的抑制剂是prt4165。在其它实施例中,多梳蛋白抑制复合物的抑制剂是rnai分子,其选择性地降低多梳蛋白抑制复合物的组分(例如prc1或prc2的组分)的表达。
在本发明的一些实施例中,通过用降低基因组dna甲基化的药剂处理前体细胞,来进行dna甲基化的抑制(所述方法的步骤(ii))。在本公开的情形下,‘处理’细胞应理解成意味着‘接触’细胞,即使细胞暴露于药剂。此外,‘抑制’包含‘降低’和‘完全阻止’两个。举例来说,可用dna甲基转移酶抑制剂、用阻止dna甲基化沉积的药剂或用抑制dna甲基化维持的药剂,来处理(接触)前体细胞。5-氮杂-2-脱氧胞苷(5-aza-dc)是一种抑制dna甲基化且还抑制dna甲基化维持的药剂。
在其中降低基因组dna甲基化的药剂是dna甲基转移酶抑制剂的实施例中,dna甲基转移酶抑制剂可以是dnmt1抑制剂。举例来说,dna甲基转移酶抑制剂可以是sgi1027或5-氮杂胞苷。或者,dna甲基转移酶抑制剂可以是rnai分子,其降低dna甲基化机制的组分的表达。rnai分子可以是sirna分子或mirna分子(或任一个的前体)。
在其他实施例中,可通过使用使dna甲基转移酶基因失活的技术,如基因编辑,来进行dna甲基化的抑制(所述方法的步骤(ii))。因此,抑制dna甲基化的多种手段可用于生成减数分裂胜任细胞。举例来说,可使用基因敲除甲基化机制或化学阻断甲基化机制。
在第二方面中,本发明提供一种通过本文所描述的方法所生成的减数分裂胜任细胞。可用视黄酸处理减数分裂胜任细胞。已知视黄酸诱导减数分裂胜任细胞中的配子发生。
因此,在第三方面中,本发明提供一种通过用视黄酸处理本发明的减数分裂胜任细胞而诱导其中的配子发生的方法。在一些实施例中,配子发生是精子发生。在其它实施例中,配子发生是卵子发生。
在另一方面,本发明提供一种用于活体外生成减数分裂胜任细胞的试剂盒。本发明试剂盒包括甲基化抑制剂和多梳蛋白抑制复合物的抑制剂。在一些实施例中,试剂盒也包括视黄酸。试剂盒也可包括用于本发明方法的适当硬件,例如试管、培养板等。
在又一方面中,本发明提供一种评定哺乳动物的生育力的方法。在本发明的此方面中,测定已从哺乳动物获得的细胞中的一个或多个生殖系再程序化应答(grr)基因的核酸序列和/或表观遗传状态和/或基因表达水平。
在相关方面中,本发明提供一种测定细胞的减数分裂胜任能力的方法,所述方法包括测定所述细胞的基因组dna中的一个或多个生殖系再程序化应答(grr)基因的核酸序列、表观遗传状态和/或表达。本发明还提供具有探针群组的试剂盒和/或分析板,所述群组由检测如表1中所示的一个或多个grr基因的表达或表观遗传状态或表达的探针组成或基本上由其组成。
表1-grr基因
n.d.:无数据;l.c.c.:低置信度分类
如本文中所描述,本发明的一些实施例涉及检测grr基因(例如序列、表观遗传状态或表达水平)或诱导grr表达。这些实施例可涉及检测或诱导包含以下各者、由以下各者组成或主要由以下各者组成的基因群组:选自表1的一个或多个grr基因;例如选自表1的任何2个、任何3个、任何4个、任何5个、任何6个、任何7个、任何8个、任何9个、任何10个、任何11个、任何12个、任何13个、任何14个、任何15个、任何16个、任何17个、任何18个、任何19个或任何20个基因。在一些实施例中,选自表1的基因可包含以下中的一个或更多个:dazl、hormad1、sycp2、sycp3、mae1、fkbp6。在其它实施例中,选自表1的基因可排除以下中的任一个或全部:dazl、hormad1、sycp2、sycp3、mae1、fkbp6。
治疗性应用
本发明的方法和产物具有治疗性应用,尤其在不孕症治疗中。举例来说,如本文中所描述,可诱导通过本发明方法生成的减数分裂胜任细胞进行配子发生,例如通过用视黄酸(ra)处理。以这种方式生成的配子母细胞(即精母细胞;卵母细胞)构成本发明的其它方面。本发明的配子母细胞例如在过继转移到不育个体中具有治疗性应用:可以设想,可将本发明的精母细胞过继转移到雄性不孕症患者的睪丸。可以设想,可将本发明的卵母细胞过继转移到雌性不孕症患者的卵巢。这些配子母细胞可来源于患者的自身细胞,例如通过对于来源于患者细胞的ips细胞、精原干细胞(ssc)或pgclc进行本发明的方法。这个方法允许将配子母细胞自体过继转移到患者。
在本发明的其它方面,配子来源于上述活体外本发明的配子母细胞。以这种方式,本发明提供可治疗性使用的雄性配子,精子(spermatozoa/sperm)和雌性配子,卵细胞(卵子)。举例来说,本发明的配子可用于活体外受精(ivf)应用中。
前体细胞
如本文中所阐释,本发明方法能够将体细胞前体细胞转化为减数分裂胜任细胞。这个小节论述可用作前体细胞的多种细胞类型。
在自然界中,前体到减数分裂胜任生殖母细胞为原始生殖细胞。旨在产生pgc样细胞(pgclc)的目前活体外系统5,24-26可成功地仅再现pgc发育的早期阶段,其中性腺再程序化仍呈现可仅在性腺体细胞环境的情形下克服和执行5,24,25,27的屏障。在本发明的一些实施例中,前体细胞是通过前述现有技术方法得到的pcglc。
在本发明的其它实施例中,前体细胞是干细胞,例如胚胎干细胞。人类胚胎干细胞表示一种前体细胞类型。本领域已知可在不破坏人类胚胎的情况下获得人类胚胎干细胞(chung等人,2008)。小鼠胚胎干细胞也表示有效证明本发明功效的前体细胞的一种类型。本发明人已发现,pgc中的grr基因的表观遗传调控与血清生长的小鼠胚胎干细胞中的grr基因的表观遗传调控极其相似。
还可使用不是胚胎来源的多能干细胞作为本发明方法中的前体细胞。可通过包含以下的方法来获得多能干细胞:
通过核转移的再程序化.这个技术涉及将细胞核从体细胞转移到卵母细胞或受精卵中。在一些情况下,这可导致产生动物-人类杂交细胞。举例来说,细胞可通过人类体细胞与动物卵母细胞或受精卵的融合或人类卵母细胞或受精卵与动物体细胞的融合来产生。
通过与胚胎干细胞融合的再程序化.这个技术涉及体细胞与胚胎干细胞的融合。这个技术也可导致产生如上1中的动物-人类杂交细胞。
通过培养进行的自发性再程序化.这个技术涉及在长期培养之后从非多能细胞产生多能细胞。举例来说,已通过长期培养原始生殖细胞(pgc)来产生多能胚胎生殖(embryonicgerm,eg)细胞(matsui等人,1992)。也已报道在长时间培养骨髓源性细胞之后发育多能干细胞(jiang等人,2002)。他们称这些细胞为多能成年祖细胞(mapc)。shinohara等人也证实,可在培养来自新生小鼠睪丸的生殖系干(gs)细胞的过程期间产生多能干细胞,称其为多能生殖系干(multipotentgermlinestem,mgs)细胞(kanatsu-shinohara等人,2004)。
通过限定因子的再程序化.举例来说,通过反转录病毒介导的引入转录因子(如oct-3/4、sox2、c-myc和klf4)到小鼠胚胎或成年纤维母细胞中来产生ips细胞,例如如kaji等人,2002所描述,也描述单一多蛋白表达载体的非病毒转染,所述表达载体包括与2a肽连接的c-myc、klf4、oct4和sox2的编码序列,其可再程序化小鼠和人类纤维母细胞。用这个非病毒载体生成的ips细胞显示多能性标记的稳固表达,指示通过活体外分化分析和成年嵌合小鼠的形成功能性确认的再程序化状态。他们成功地从稳固表达多能性标记的胚胎纤维母细胞建立再程序化的人类细胞系。诱导性多能干细胞具有以下优点,可通过不导致破坏胚胎的方法,更具体地说通过不导致破坏人类或哺乳动物胚胎的方法来获得所述诱导性多能干细胞。
多能干细胞也可从终止分裂且未能活体外发育桑椹体和囊胚的停滞胚胎获得,所述停滞胚胎通过单性生殖获得或来源于来自单一分裂球或活检分裂球的hesc系。
因此,可通过使用细胞来进行或实践本发明的方面,所述细胞不仅仅通过必然涉及破坏那些细胞所源自的人类或动物胚胎的方法所制备。这个任选限制尤其意图考虑欧洲专利局扩大上诉委员会(theenlargedboardofappealoftheeuropeanpatentoffice)的2008年11月25日的决策g0002/06。
在其它实施例中,配原细胞(配子干细胞)可用作前体细胞。举例来说,精原干细胞(ssc)是一种优选的前体细胞类型以供本发明方法使用。ssc可提取自睪丸,例如提取自睪丸活检体。睪丸抽吸物是含有ssc的细胞制剂(提取物)的一个来源。可以设想,可对于这类睪丸提取物直接进行本发明方法,或可对于已进行富集、选择和/或纯化的ssc实施本发明方法。
可从个体获得前体细胞。个体可以是哺乳动物个体,例如人类个体。在本发明的一些实施例中,个体是不孕症患者。
rna干扰(rnai)
本发明也包含本领域已知的技术的用途,其用于治疗性下调多梳蛋白抑制复合物的组分或dna甲基化机制的组分。这些包含使用rna干扰(rnai)。
可使用小rna分子来调控基因表达。这些包含通过小干扰rna(sirna)靶向降解mrna、转录后基因沉默(ptg)、通过微小rna(mirna)发育性调控mrna的序列特异性翻译抑制和靶向转录基因沉默。
也已证实rnai机制和小rna在靶向异染色质复合物和特定染色体基因座处的表观遗传基因沉默方面的作用。双链rna(dsrna)依赖性转录后沉默,也称为rna干扰(rnai),是一种其中dsrna复合物可在短时间段内靶向同源性特定基因以沉默的现象。其充当促进具有序列一致性的mrna降解的信号。20-核苷酸sirna一般足够长以诱导基因特异性沉默,但足够短以避开宿主反应。靶向基因产物的表达降低可为广泛的,其中90%沉默是通过几个sirna分子诱导。
在本领域中,视其来源而定,这些rna序列称为“短或小干扰rna”(sirna)或“微小rna”(mirna)。两种类型的序列均可用于通过与互补rna结合以及触发mrna消除(rnai)或遏制mrna翻译为蛋白质来下调基因表达。通过处理长双链rna来得到sirna。微小干扰rna(mirna)是通过对短发夹(shorthairpin)的处理所得到的内源性编码的小非编码rna。sirna和mirna均可抑制携带部分互补靶序列的mrna的翻译而无需rna裂解,且降解携带完全互补序列的mrna。
因此,本发明提供一种这些序列的用途,其用于下调多梳蛋白抑制复合物(例如prc1和/或prc2)的组分的表达。
sirna通常是双链,且为了使rna介导的下调靶基因的功能的效果达到最佳,优选的是,选择sirna分子的长度以确保介导由mrna标靶的sirna识别的risc复合物正确识别sirna,且因此sirna足够短以降低宿主反应。
mirna通常是单链且具有部分地互补使得mirna能够形成发夹的区。mirna是从dna转录但未转译成蛋白质的rna基因。编码mirna基因的dna序列比mirna长。这个dna序列包含mirna序列和大致反向互补序列。当这个dna序列转录成单链rna分子时,mirna序列和其反向互补序列碱基对形成部分双链的rna区段。微小rna序列的设计是本领域中已知的。
附图说明
现在将借助于实例参考附图描述本发明的实施例:
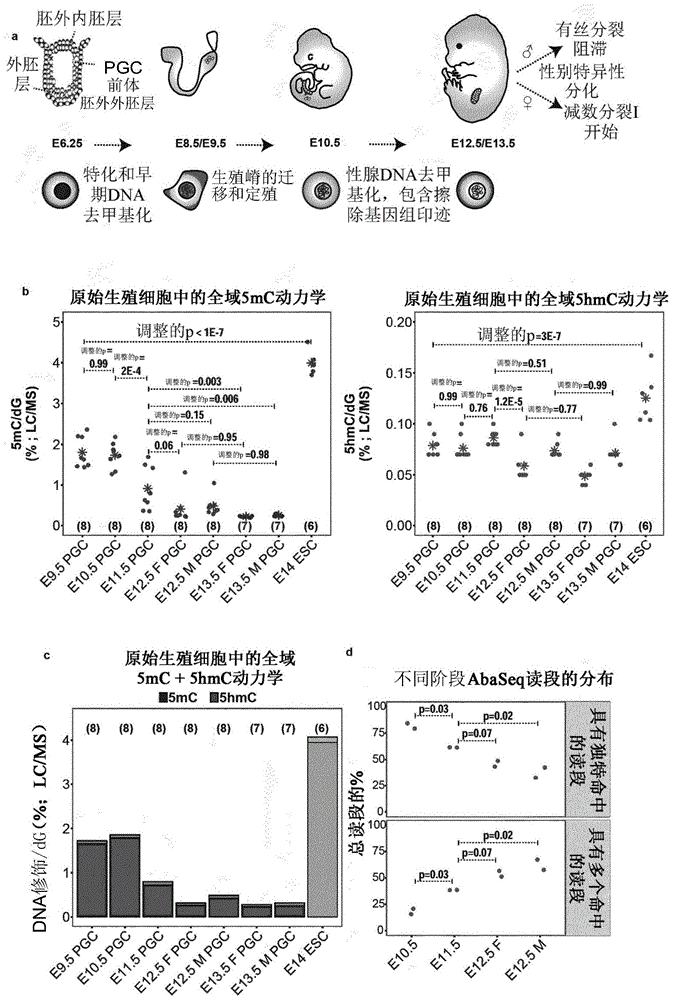
图1-在表观遗传再程序化期间的5mc和5hmc动力学.a)在小鼠pgc发育期间的关键事件。b-c)mesc且e9.5到e13.5pgc中的个别5mc(b,左侧)和5hmc(b,右侧)和组合5mc/5hmc(c)水平(lc/ms)。(b)中的星号是指平均值。已调整的p值是基于方差分析和tukey事后测试。(c)中的条形图描绘(b)中描绘的生物学重复的中值。d)从基因组的独特定位部分到e10.5与e12.5之间的重复元件的5hmc的再分布。p值是基于组合性方差分析和tukey事后测试。e)e10.5和e12.5pgc中的代表性5hmc免疫染色。比例尺表示10μm。关于样本大小和如何收集样本的详情可见于统计学和再现性章节。
图2-tet1保护但不驱动dna去甲基化.a-b)e13.5野生型和tet1-kopgc中的针对5hmc(a)或5mc(b)的代表性免疫染色。比例尺表示10μm。c-d)野生型和tet1-kopgc中的全域5hmc(c)和5mc(d)水平(lc/ms)。在曲线上指示样本数量。星号是指平均值。p值是基于双侧史都登氏t测试(student'st-test)。e)上图:e14.5tet1-kopgc中的差异甲基化区的比例(p<0.05,>10%甲基化差异;p值是从rnbeads软件得出)。下图:对于所有e14.5高度甲基化的2kb窗口,e12.5(中间)和e14.5(下部)tet1-ko(红色)和野生型(蓝色)pgc中的组合5mc/5hmc水平(rrbs)。也显示来自e10.5野生型pgc的dna修饰水平(上图)。通过垂直线表示中值组合5mc/5hmc水平。关于样本大小和如何收集样本的详情可见于统计学和再现性章节。
图3-生殖系再程序化应答(grr)基因.a)hcp基因簇的pgc发育的连续阶段中的组合启动子5mc/5hmc水平(右侧)、启动子5hmc水平(中心)或基因表达水平(右侧)(参见方法)。上部和下部铰链对应于第一和第三四分位数,且中间线对应于中值,且最大值和最小值分别对应于1.5×四分位数内范围内的最高或最低值。b)基于野生型pgc中的e10.5与e14.5之间的上调显著性排名的甲基化和去甲基化hcp(簇3,图3a)的以tss为中心的基因组序列。各水平线表示一个基因;红色的强度指示在各栏顶部处显示的特征的相对富集。显示tss+/-5kb。c)与生殖系再程序化应答(grr)基因相关的基因本体(go)术语;调整p值(adj.p-value)是基于david软件。关于样本大小和如何收集样本的详情可见于统计学和再现性章节。
图4-grr基因活化的表观遗传原理.a)tet1-kopgc中的grr基因表达动力学;p值是基于双侧配对威尔科克森测试(two-sidedpairedwilcoxontest)。b)e12.5或e14.5tet1-ko(红色)和野生型(蓝色)pgc中的grr基因处的组合5mc/5hmc水平(rrbs)。对于比较来说,显示mesc中的组合5mc/5hmc水平30(%;wgbs)。p值是基于配对双侧威尔科克森测试。c-d)grr基因和其它相关基因集合的以下的log2(倍数变化):dnmt-tko(绿色)或tet1-kodnmt-tko与野生型mesc之间(c)、或野生型+6hprt4165处理(紫色)、dnmt-tko+6hdmso处理(绿色)或dnmt-tko+6hprt4165处理(黄色)和野生型+6hdmso处理mesc之间(d)。fwer调整的p值是基于gsea软件(详情参见方法)。关于样本大小和如何收集样本的具体详情见于统计学和再现性章节。对于所有盒形图,上部和下部铰链对应于第一和第三四分位数,且中间线对应于中值,且最大值和最小值分别对应于1.5×四分位数内范围内的最高或最低值。
图5-wgbs数据集的特征和abaseq法的验证.a)各对称cpg的wgbs覆盖率的分布。对于盒形图,上部和下部铰链对应于第一和第三四分位数,且中间线对应于中值,且最大值和最小值分别对应于1.5×四分位数内范围内的最高或最低值。b)abaseq法的概述15。c-e)显示如通过以下计算的e14mesc中的在所有2kb窗口(最小4个对称cpg)下的5hmc水平之间的相关性的密度热图:(c)tab-seq35(x轴)和abaseq15(y轴);(d)tab-seq35(x轴)和hmedip36(y轴);或(e)abaseq15(x-轴)和hmedip36(y轴)。对于(c-e),显示皮尔森相关系数(pearsoncorrelationcoefficient)(ρ)。关于样本大小和如何收集样本的具体详情见于统计学和再现性章节。
图6-e10.5pgc中的5hmc水平的进一步分析.a)显示e10.5pgc(y轴)和e14mescs15(y轴)的每2kb窗口(最小4个cpg)的5hmc水平的密度热图。显示皮尔森相关系数(ρ)。b)e10.5pgc(左侧)或e14mesc中的各种调控元件处的5hmc水平(abaseq)15。p值是基于方差分析(anova)和dunnett事后测试。对于盒形图,上部和下部铰链对应于第一和第三四分位数,且中间线对应于中值,且最大值和最小值分别对应于1.5×四分位数内范围内的最高或最低值。c)显示在e10.5pgc中不同表达水平的基因的e10.5pgc中的5hmc水平(上图,abaseq)和组合5mc/5hmc水平(下图,wgbs)的metagene曲线。d-e)显示在整个cpg岛(d)中或在整个假定的活性增强子(e)中的e10.5pgc中的5hmc水平(上图,abaseq)和组合5mc/5hmc水平(下图,wgbs)的metagene曲线。f)显示如通过tab-seq35(%;浅绿色)或abaseq15(读段计数;墨绿色)测定的e14mesc中,或如通过abaseq(读段计数;橙色)测定的e10.5pgc中的icr处的5hmc水平的条形图。关于样本大小和如何收集样本的具体详情见于统计学和再现性章节。
图7-pgc中的5mc和5hmc动力学的进一步分析.a)e10.5与e12.5之间的pgc中的基因组的独特定位部分内的各种特征处的组合5mc/5hmc(wgbs;左侧)或5hmc(abaseq;右侧)水平。上部和下部铰链对应于第一和第三四分位数,且中间线对应于中值,且最大值和最小值分别对应于1.5×四分位数内范围内的最高或最低值。b)e10.5与e12.5之间的pgc中的各种共有重复元件处的组合5mc/5hmc(wgbs;左侧)或5hmc(abaseq;右侧)水平。星号是指平均值。关于样本大小和如何收集样本的具体详情,参见统计学和再现性章节。
图8-在小鼠性腺pgc中的dna去甲基化之后,5hmc靶向新的低甲基化区(也参见图9a)显示e10.5生物学重复(左侧)、e10.5与e11.5pgc(中间)和e10.5与e12.5pgc(右侧)的5hmc水平之间的皮尔逊相关性(ρ)的密度热图。b)描绘标准化为在跨阶段的5hmc(橙色,abaseq)或组合5mc/5hmc(灰色,wgbs)的平均水平的各阶段的5hmc(橙色,abaseq)和组合5mc/5hmc(灰色,wgbs)水平的平均z评分。所显示的平均值的标准误差太小而难以看见。c-f)显示e10.5(c,e)或e11.5(d,f)pgc中的总(c,d;y轴:abaseq读段计数)或相对(e,f;y轴:(abaseq读段计数)/(%;wgbs)的比率)5hmc水平与具有e10.5pgc中的最小20%组合5mc/5hmc的所有2kb窗口的这两个阶段之间的pgc中的组合5mc/5hmc水平的变化(x轴:%;wgbs)之间的相关性的密度热图。g)显示e11.5pgc中的相对5hmc水平(y轴:(abaseq读段计数)/(%;wgbs)的比率)与具有e10.5pgc中的最小20%组合5mc/5hmc的所有2kb窗口的e11.5pgc中的组合5mc/5hmc水平(x轴:%;wgbs)之间的相关性的密度热图。h)显示具有e10.5pgc中的最小20%总dna修饰的2kb窗口的e10.5与e11.5之间的pgc中的组合5mc/5hmc水平降低的密度曲线,其1)富集e10.5或e11.5处的总5hmc水平(绿色,上尾调整的泊松p值<0.05)或2)消耗e10.5和e11.5两处的总5hmc(红色,下尾调整的泊松p值<0.05)。i)具有e10.5pgc中的最小20%组合5mc/5hmc的2kb窗口的e10.5与e11.5pgc中的组合5mc/5hmc水平,其1)富集e10.5或e11.5处的总5hmc水平(绿色,上尾调整的泊松p值<0.05)或2)消耗e10.5和e11.5两处的总5hmc(红色,下尾调整的泊松p值<0.05)。对于所有盒形图,上部和下部铰链对应于第一和第三四分位数,且中间线对应于中值,且最大值和最小值分别对应于1.5×四分位数内范围内的最高或最低值。p值是基于双侧威尔科克森测试。对于密度热图应注意:1)显示斯皮尔曼相关性(spearmancorrelation)(ρs);和2)红线表示如通过一般化加性模型测定的平滑平均值。关于样本大小和如何收集样本的具体详情见于统计学和再现性章节。
图9-涉及性腺pgc的dna去甲基化中的5mc氧化的推荐模型.a)氧化,随后被动稀释的模型,其预测两个阶段之间的组合5mc/5hmc水平降低程度(即%;wgbs)与紧接在这个降低的前后两个阶段处的总5hmc水平之间的正相关性。b)涉及通过活化机制触发dna去甲基化中的5mc氧化的模型,其预测两个阶段之间的组合5mc/5hmc水平降低程度(即%;wgbs)与紧接在这个降低前的阶段中的相对5hmc水平之间的正相关性,这是由于5hmc进一步氧化为5fc是5mc完全氧化为5cac中的速率限制步骤39。c)涉及在dna去甲基化的主波(majorwave)之后保护dna低甲基化中的5mc的氧化的模型,其预测,在两个阶段之间已丧失大部分dna去甲基化的区(即新的低甲基化的那些区)将在紧接在dna去甲基化的主波后的阶段中具有高的相对5hmc水平,以去除残余甲基化和/或异常新生甲基化。因此,也可看见两个阶段之间的组合5mc/5hmc水平降低程度(即%,wgbs)与紧接在这个降低后的阶段中的相对5hmc水平之间的有限相关性。
图10-在表观遗传再程序化期间tet1-kopgc中的tet1-3表达和基因座特异性dna甲基化.a)e12.5tet1-ko和野生型pgc中的tet1总转录物(左侧)或缺失外显子4(右侧)的表达。通过deseq2计算的调整p值(左侧)和通过史都登氏t测试计算的p值(右侧)。星号是指平均值。b)针对e12.5野生型和tet1-kopgc中的tet1蛋白的n端的代表性免疫染色。比例尺表示10μm。c)e12.5tet1-ko和野生型pgc中的tet2和tet3的表达。通过deseq2计算的调整p值。星号是指平均值。d-e)雌性(d)或雄性(e)e12.5和e14.5tet1-ko和野生型pgc中的称为在e14.5tet1-kopgc中高度甲基化的icr和生殖系基因启动子的平均组合5hmc/5mc水平(rrbs)。通过rnbeads软件计算平均dna修饰水平和p值(详情参见方法)。f-g)e12.5(f)和e13.5(g)雌性tet1-ko和野生型pgc中的dazl启动子(左侧)、peg3icr(中间)和ig-dmricr(右侧)的基因座特异性亚硫酸氢盐测序。关于样本大小和如何收集样本的具体详情见于统计学和再现性章节。
图11-在生殖系再程序化期间的启动子dna甲基化聚类分析.a)通过对其启动子区处的组合5mc/5hmc动力学的k-平均值聚类进行分组的所有基因的pgc发育的连续阶段中的组合启动子5mc/5hmc水平(wgbs,右侧)、启动子5hmc水平(abaseq,中心)或基因表达水平(rna-seq,右侧)。b-c)描绘通过其启动子区处的组合5mc/5hmc动力学的k-平均值聚类进行分组的pgc发育的连续阶段中的具有低cpg启动子(lcp;b)或中等cpg启动子(icp;c)的三个基因簇的组合启动子5mc/5hmc水平(wgbs,右侧)、启动子5hmc水平(abaseq,中心)或基因表达水平(rna-seq,右侧)的盒形图。对于所有盒形图,上部和下部铰链对应于第一和第三四分位数,且中间线对应于中值,且最大值和最小值分别对应于1.5×四分位数内范围内的最高或最低值。关于样本大小和如何收集样本的具体详情见于统计学和再现性章节。
图12-在通常与表观遗传再程序化并行活化的反转录转座子处的野生型和tet1-kopgc中的dna修饰和表达动力学.a-b)在野生型pgc中的e10.5与e14.5之间以性别非依赖性方式(a)、以雄性特异性方式(b,蓝色盒)或以雌性特异性方式(b,粉红色盒)显著地上调(调整p值<0.05;sleuth)的代表性重复元件的野生型pgc中的组合5mc/5hmc动力学(%;wgbs;最左侧)、野生型pgc中的相对5hmc动力学(标准化为e10.5的abaseq读段计数;中心左侧)、野生型或tet1-kopgc中的表达动力学(每百万的转录物(transcriptspermillion,tpm);rna-seq;中心右侧)和野生型与tet1-kopgc中的组合5mc/5hmc动力学(%;rrbs;最右侧)。在所有情况下显示平均值。e14.5野生型与tet1-kopgc之间的差异重复表达分析的调整p值是基于sleuth软件。关于样本大小和如何收集样本的具体详情见于统计学和再现性章节。
图13-通过pgc和mesc中的tet1和5mc表征grr基因调控.a)在grr基因启动子和其它相关启动子处的cpg密度;p值是基于双侧威尔科克森测试。b)在pgc中的grr基因启动子和未活化的甲基化与去甲基化hcp处的平均5hmc动力学;p值是基于双侧配对威尔科克森测试。c)grr基因和其它相关基因集合的tet1-ko与野生型e14.5雄性(蓝色)或雌性(粉红色)pgc之间的log2(倍数变化)。fwer调整的p值是基于gsea软件(详情参见方法)。d)grr基因和其它相关基因集合的dnmt1-cko24与野生型mesc(绿色)之间或e14.5雌性(粉红色)或雄性(蓝色)野生型pgc与e10.5野生型pgc之间的log2(倍数变化)。fwer调整的p值是基于gsea软件(详情参见方法)。e)grr启动子处的组合5mc/5hmc水平差异(x轴;tet1-ko(rrbs;%)-wt(rrbs;%))与e12.5(右侧)和e14.5(左侧)tet1-kopgc中的grr基因表达变化(y轴;log2(tet1-ko/wt))之间的相关性。显示斯皮尔曼相关性。f)显示野生型、dnmt-tko和tet1-kodnmt-tkomesc中的tet1和laminb蛋白质表达的代表性免疫印迹。对于所有盒形图,上部和下部铰链对应于第一和第三四分位数,且中间线对应于中值,且最大值和最小值分别对应于1.5×四分位数内范围内的最高或最低值。对于所有图,关于样本大小和如何收集样本的具体详情见于统计学和再现性章节。关于样本大小和如何收集样本的具体详情见于统计学和再现性章节。
图14-mesc中的grr基因启动子的表观遗传特征.a)以以下基因的tss为中心的基因组序列:在含血清的介质中生长的野生型mesc中的e10.5与e14.5之间的雄性和雌性pgc中的grr基因、活化的非grr基因和非grr甲基化和去甲基化hcp基因。各水平线表示一个基因;红色的强度指示在各栏顶部处显示的特征的相对富集。显示tss和在tss上游与下游5kb的序列。b-f)描绘以下水平的盒形图:在含血清的介质中生长的野生型mesc中的grr基因或其它相关基因集合的启动子处的(b)组合5mc/5hmc水平(wgbs)30;(c)5hmc(abaseq)15;(d)tet1(chip-seq)21;(e)ring1b(chip-seq)38和(f)h2aub水平(chip-seq)37。对于所有盒形图,上部和下部铰链对应于第一和第三四分位数,且中间线对应于中值,且最大值和最小值分别对应于1.5×四分位数内范围内的最高或最低值。p值是基于双侧威尔科克森测试。g)描绘在含有血清的介质中生长的野生型和tet1-komesc中的grr基因(左侧)和非grrhcp基因(右侧)的tss附近的中值h3k4me3水平(chip-seq)30的metagene曲线,所述非grrhcp基因最初也甲基化且随后在pgc再程序化期间去甲基化。p值是基于对tss-1kb/+500bp的区进行的配对双侧威尔科克森测试。关于样本大小和如何收集样本的具体详情见于统计学和再现性章节。
图15-通过pgc和mesc中的prc1和5mc表征grr基因调控.a)grr基因与相较于野生型在e11.5和/或e12.5prc1条件性基因敲除pgc中显著上调的基因之间的重叠26。p值是基于超几何测试。b)显示野生型或dnmt-tkomesc+6hdmso和野生型或dnmt-tkomesc+6hprt4165(prc1抑制剂)中的h2aub和h2a水平的代表性免疫印迹。c)根据mesc中的5mc和/或prc1再程序化的其依赖性的grr基因分类(详情参见方法)。关于样本大小和如何收集样本的具体详情见于统计学和再现性章节。
图16-内源性pgc转化为生殖母细胞的模型
涉及pgc转化为生殖母细胞和成功配子发生的生殖系再程序化应答(grr)基因的及时且有效活化需要起始全域dna去甲基化、tet1募集和去除prc1介导的抑制之间的相互作用。tet1的dna去甲基化依赖性(保护免于异常残余/新生启动子dna甲基化)和非依赖性(如将ogt潜在募集到基因启动子36,因此促进通过set1/compass38沉积h3k4me3)功能对于grr基因活化均是重要的。
图17-响应于视黄酸的基因表达变化
用视黄酸(ra)处理小鼠胚胎干细胞(mesc)。用j1细胞系与缺乏dna甲基化机制的j1“tko”细胞(借助于dnmt1/dnmt3a/dnmt3b三重基因敲除)相比较。黑色条柱分别显示相较于j1对照(均不用ra处理)的tko细胞中的dazl-hormad1表达和mae1表达的倍数变化。灰色条柱分别显示用ra处理的j1细胞中相较于未用ra处理的j1细胞的的dazl、hormad1和mae1表达的倍数变化。白色条柱分别显示用ra处理的tko细胞相较于未用ra处理的j1细胞中的dazl、hormad1和mae1表达的倍数变化。
具体实施方式
虽然本发明已结合上文所描述的示范性实施例加以描述,但是当给出本发明时,本领域的技术人员将清楚许多等效修改和变化形式。因此,上文所阐述的本发明的示范性实施例被视为是说明性的而不是限制性的。可以在不脱离本发明的精神和范围的情况下对所描述实施例作出各种改变。
表观遗传再程序化使得原始生殖细胞能够转化为生殖母细胞
配子是可通过其产生全能受精卵的能力产生下一代的高度特化细胞。在小鼠中,在胚胎第(e)6.251天开始,生殖细胞在发育胚胎中首先特化为原始生殖细胞(pgc)(图1a)。在后续迁移到发育中性腺中之后,pgc在e10.5/e11.5进行一波广泛表观遗传再程序化2-11,包含5-甲基胞嘧啶(5mc)的全基因组缺失2-5,7-11(图1a)。这个过程的潜在分子机制仍为神秘的,导致我们不能在活体外再现生殖系发育的这个步骤12-14。本发明人使用整合方法显示,这个复杂再程序化过程涉及启动子序列特征、dna(去)甲基化、多梳蛋白(prc1)复合物和tet1的dna去甲基化依赖性与非依赖性功能之间的协同相互作用,以使得涉及配子产生和减数分裂的生殖系再程序化应答(grr)基因的关键集合能够活化。我们的结果也出乎意料地揭示tet1在保护但不驱动性腺pgc中的dna去甲基化方面的作用。总体来说,我们的研究揭示了性腺生殖系再程序化的基本生物学作用且鉴定了pgc转化为生殖母细胞的表观遗传原理,其将对于活体外再现完全配子发生具有启发性。
为了解决性腺生殖系再程序化的潜在作用和潜在分子机制,本发明人首先开始研究5mc与5-羟甲基胞嘧啶(5hmc)的动力学和5mc与5-羟甲基胞嘧啶(5hmc)之间的关系,其先前牵涉于pgc中的dna去甲基化3,6,9-11。对此,本发明人使用与全基因组亚硫酸氢盐测序(wgbs,图5a)和abaseq(图5b-e)15偶联的液相色谱/质谱法(lc/ms)在单碱基分辨率下定量性地完成。wgbs提供关于5mc和5hmc的组合水平的信息16,而abaseq15能够进行稳固的位点特异性量化和给定样本内和在与lc/ms组合时样本之间的的全基因组5hmc水平的准确比较(参见方法,图5b-e)。
通过lc/ms,本发明人观测到,基因组5mc的全域水平在迁移的(e9.5)与早期的性腺(e10.5)pgc之间保持稳定,随后在e10.5与e11.5之间显著降低,且在e11.5与e13.5之间多许多的有限dna去甲基化(图1b)。关于5hmc,lc/ms分析出乎意料地显示,pgc中的全域水平低于在含血清的培养条件中生长的小鼠胚胎干细胞(mesc)中的那些(图1b)。此外,pgc中的全域5hmc水平在e9.5与e13.5之间相对恒定,在雌性中在e12.5开始具有略微降低(图1b)。重要的是,5hmc水平始终比在e10.5的总5mc水平或在e10.5与e11.5之间的5mc缺失量低一个数量级(图1b-c),记录dna去甲基化并不全域地伴有5hmc水平的反向增加,如先前已暗示3,17(图9a)。
与我们的lc/ms测量一致,wgbs分析显示,在e10.5与e11.5之间基因组的独特定位区内的特征处的组合5mc/5hmc几乎完全缺失,其中在e11.5与e12.5之间观测到有限的进一步dna去甲基化(图7a)。也在共有重复序列处观测到dna甲基化的缺失,尽管一些重复元件(如line-1a和erv-iap反转录转座子)在e12.5pgc中保留相对较高水平的组合5mc/5hmc,如先前所暗示8(图7b)。通过abaseq在e10.5pgc中的5hmc定位的详细分析显示,尽管全域水平更低(图1b),但pgc中的5hmc定位明显类似于血清生长的mesc的全域水平,即使在印记控制区(imprintcontrolregion,icr;图6a,b,f)处。总之,5hmc在假定的活性增强子处富集,存在于基因间区域和基因体中,在启动子处消耗,且不存在于绝大部分cpg岛上(图6b-f)。关于转录,在启动子区处的5mc和5hmc与基因表达水平显示反向关系(图6c)。在基因体内,5mc和5hmc在已表达基因处相较于无可检测表达的基因处明显富集,但观测到5hmc与基因表达的非线性关系,而组合5mc/5hmc水平显示明显的正相关性(图6c)。
在整个检查的发育阶段中的5hmc模式的详细分析揭示,从基因组的独特定位区缺失大部分5hmc且再定位到重复元件(图1d,图7a-b)。这个再定位也通过免疫荧光染色而显而易见(图1e)。因此,我们的数据显示,在pgc的基因组的整个独特定位区中缺失5mc和5hmc,尽管5hmc的不同动力学显示更多的逐渐降低(图8b)。然而,这与通过细胞分裂进行的5hmc的被动稀释3不一致,如通过阶段间的不佳皮尔逊和斯皮尔曼相关性所展现(图8a、9a)。相反,本发明人总结,5hmc是pgc中的动态标记。
我们接下来研究对于所有最初甲基化2kb窗口(即在e10.5最小20%甲基化),在e10.5与e12.5之间性腺pgc中的5hmc沉积与dna去甲基化之间的关系。涉及5hmc中间物的dna去甲基化预测在5hmc出现与5mc缺失之间的直接相关性(图9a-b)。令本发明人出人意料的是,观测到在e10.5或e11.5的总或相对5hmc水平间无相关性,且这些阶段间的组合5mc/5hmc水平的程度降低(图8c-f)。然而,对于所有最初甲基化2kb窗口,观测到在e11.5的相对5hmc水平与组合5mc/5hmc水平之间的负相关性(图8g)。因此,5hmc表示在于e11.5时新的低甲基化的区域处比例高得多的组合5mc/5hmc水平,不管其初始dna甲基化水平如何。尽管在e11.5,5hmc耗尽区比5hmc富集区含有略微更多的5mc,但e10.5和e11.5pgc中5hmc耗尽的序列仍在这两个阶段之间进行大量的dna去甲基化(图8h-i),指示存在可检测的5hmc不是性腺pgc中5mc缺失的前提条件。因此,我们的观测暗示5hmc参与生殖细胞中的dna去甲基化后基因座特异性5mc水平的调控而非参与全域dna去甲基化的初始波(图9c)。
为了扩大这个观测,本发明人使用先前公开的tet1-ko小鼠模型18(图10a-c)。初始lc/ms分析显示,tet1缺失引起e10.5tet1-ko生殖细胞中大致50%的全域5hmc水平降低(图2c)。与在e12.5的高水平tet1表达一致3,9,11(图10a-c),lc/ms分析确认,tet1表示去甲基化pgc中的初级5mc加氧酶,其中在e14.5tet1-ko生殖细胞中观测到全域5hmc水平大致85%降低(图2a,c)。重要的是,tet1-ko和野生型pgc两个的基因组均在e13.5达到几乎完全的5mc耗尽(图2b,d),突显tet1介导的5mc氧化不直接造成性腺pgc中的大批dna去甲基化。
在我们的lc/ms测量的支持中,通过简化表示的亚硫酸氢盐测序(reducedrepresentationbisulphitesequencing)(rrbs),在e14.5tet1-kopgcs中检测到仅有限数量的差异甲基化区(图2e)。有趣的是,这些区最初在tet1-ko和野生型pgc中进行广泛dna去甲基化,随后在e12.5与e14.5之间tet1-kopgc中的5mc水平特异性地后续增加(图2e)。相比之下,在野生型生殖细胞中的这些阶段之间,5mc水平保持稳定和/或进行略微进一步降低(图2e)。在先前报道9,10的生殖系基因启动子和通过rrbs发现在e14.5tet1-kopgc中高度甲基化的icr的几个实例处,也观测到相同的dna去甲基化/再甲基化动力学(图10d-e)。尽管通过去甲基化pgc中的靶向亚硫酸氢盐测序,确实在dazl启动子处观测到5mc的显著富集,但实际上在peg3和ig-dmricr处观测到的高度甲基化的程度是极其有限的(图10f-g)。此外,对于所有三个区,极少的克隆保留完全甲基化,而多个克隆具有与随机未能去除tet1-kopgc中的异常残余/新生dna甲基化一致的甲基化模式(图10f-g)。
我们接下来结合来源于e10.5-e14.5pgc的rna-seq数据集来分析所观测到的5mc和5hmc动力学(图11)。基于其启动子dna甲基化动力学的所有基因的初始聚类分析显示,尽管大部分启动子变得完全去甲基化,但存在在全域dna去甲基化期间保留高水平的5mc/5hmc的小子集的转录沉默的启动子(簇2,图11a)。这些启动子与含有可能决定这个表观遗传状态的内源性逆转录病毒的line1和ltr(p值分别=9.5×10-24和7.2×10-83,超几何测试)明显重叠(图7b)。总之,尽管高水平的启动子5mc和5hmc与e10.5再程序化前pgc中的转录抑制相关,但缺失这些标记一般不导致转录活化(图11a)。
由于已在哺乳动物中显示5mc对于基因的转录活性的影响高度取决于启动子cpg含量19,所以本发明人在具有高cpg(hcp)、中等cpg(icp)或低cpg(lcp)启动子的基因处进行特异性聚类分析19(图3a和图11b-c)。有趣的是,这产生一群组的hcp基因,其在生殖系表观遗传再程序化过程期间变成dna去甲基化的,且显示进展性转录活化(簇3;图3a)。差异表达分析确认,这些基因显示在与pgc中的表观遗传再程序化并行上调的所有基因中显著富集(p值<0.001,超几何测试),其中45个基因通常在两个性别中均被活化(图3a-c)。考虑其启动子甲基化动力学和其活化时序,本发明人把这45个基因叫做‘生殖系再程序化应答’(grr)基因(图3c)。有趣的是,grr基因显示参与配子产生和减数分裂的因子的显著富集,所述因子包含dazl、sycp1-3、mael、hormad1和rad51c(图3c)。
考虑到grr基因(n=45)占进行dna去甲基化的hcp基因的整个子集(n=226;图3a-c)不到25%,dna去甲基化可能是用于甲基化hcp的转录活化的一种重要因子,以及另外需要的其它因子。实际上,相较于其它甲基化和去甲基化hcp,grr基因启动子显示格外高的cpg密度和5hmc水平(图13a-b)。也注意到,对于启动子罕见的是,紧接在dna去甲基化的主波后,在pgc中的grr基因启动子处的5hmc水平短暂增加(图7a,13b)。另外,且与其高cpg密度和5hmc水平一致20,21,已显示grr基因启动子被mescs21和pgcs9中的tet1结合(图3b)。
所观测到的tet1结合是功能性相关的,这是由于grr基因上调的程度在tet1-kopgc中显著较低(图4a,图13c)。尽管grr基因启动子在截至e12.5在不存在tet1下进行正常dna去甲基化,其显示在tet1-kopgce14.5的后期略微高度甲基化(图4b)。然而,这个有限dna高度甲基化与降低表达仅显示弱的相关性(图13e)。此外,tet1-ko生殖细胞中的grr基因的较低表达已在处e12.5在不存在任何甲基化差异下显而易见(图4a-b,图13e),表明tet1潜在地充当其5mc去除作用外的转录调控子21,22。除grr基因以外,转座因子(te)显示在性腺表观遗传再程序化期间积累5hmc(图7b,图12)。与dna甲基化的降低一起,一些te显示与表观遗传再程序化并行的转录活化,尤其与进化的初期反转录转座子(图12)。有趣的是,缺乏tet1似乎也降低通常活化的te的转录活化程度(图12)。
为了进一步在机理上探查表观遗传再程序化与grr基因活化之间的因果关系,本发明人转向活体外模型。血清生长的mesc代表一种理想系统,这是由于这些细胞不是生殖系限制的而在grr基因启动子处观察到的表现遗传修饰与在再程序化前性腺pgc中活体内观测到的高度类似(图14a-d)。与本发明人活体内观测到的一致,启动子dna去甲基化也表示一种活体外grr基因活化的显性表观遗传再程序化事件。dnmt-tko23mesc显示grr基因的表达增加(图4c)。然而,即使在完全不存在dna甲基化下,这关键取决于tet1的存在,这是由于tet1-kodnmt-tkomesc未能将grr基因作为一群组活化(图4c,图13f)。
尽管这些活体外观测明显支持我们的关于5mc和tet1的作用的活体内数据,但dnmt-tkomesc(图4c)或已经受通过条件性缺失dnmt1(dnmt1-cko)24的早熟dna去甲基化的e10.5pgc(图13d)中grr基因上调的程度相对轻微。因此,本发明人假设,其它因子,包含潜在地其它表观遗传屏障,可调控grr基因表达。在这种情形下,性腺表观遗传再程序化先前已与在各种不同水平下的表观遗传信息的擦除相关4,25,其中先前显示去除多梳蛋白抑制复合物1(prc1)来协调dna去甲基化的e11.5/e12.5pgc中的减数分裂起始的时序26。明显地,在pgc中的prc1缺失之后异常上调的基因显示显著富集grr基因(图15a),且富集血清生长的mesc中的grr基因的启动子以用于ring1b结合和h2ak119ub(图14a,e,f)。考虑到这个,本发明人在dnmt-tkomesc的情形下使用prc1的高度特异性化学抑制同时消除dna甲基化和prc1活性,来测试dna甲基化和prc1在grr基因调控中的作用,以此模拟性腺表观遗传再程序化。和prt416527一起培养mesc在仅6h培养之后引起对prc1介导的h2a泛素化的显著抑制(图15b)。5mc/prc1抑制的双重抑制惊人地引起45个grr基因中33个grr基因的活化,其中25个基因和10个基因分别在5mc或prc1抑制的单一抑制后活化(图4d,图15c)。组合起来,这些观测显示,性腺表观遗传再程序化表观遗传系统的复合擦除4,25以增强grr基因的表达。
我们的研究已鉴定出对于配子发生的正确进展关键的生殖系再程序化应答(grr)基因集合。这些基因具有独特的启动子序列特征,具有高水平的5mc和5hmc,且是tet1和prc1的靶标。本公开显示,dna甲基化和prc1抑制的组合缺失是grr基因活化独特性需要的,其中这个表观遗传准备状态进一步需要tet1来增强完全和有效活化。tet1似乎在雌性pgc9中尤其重要,其在完成表观遗传再程序化不久之后开启减数分裂早期,因此要求这些基因的及时高表达。重要的是,尽管本发明人观测到在e14.5tet1-kopgc中的grr基因启动子处的略微高度甲基化,我们的研究清楚地记录,tet1也通过dna去甲基化非依赖性机制刺激grr基因的转录21,22。在这种情形下,先前研究已显示,tet1将ogt募集到基因启动子22,因此促进通过set1/compass28的h3k4me3沉积,导致转录活化。在进一步支持中,mesc中的grr基因启动子被低但可检测的h3k4me3标记,其水平在不存在tet1下显著降低而dna甲基化无变化(图4b,图14g)。tet1可另外通过调控非启动子顺式元件(如增强子)处的5mc/5hmc水平来增强转录。最后但同样重要的是,我们的研究显示,tet1不直接参与在性腺pgc中的表观遗传再程序化期间的全域dna去甲基化的起始,但相反本发明人确定tet1在后续去除异常残余和/或新生dna甲基化方面的关键作用(图16)。这暗示tet3驱动的5mc氧化在保护免于在受精卵dna去甲基化29期间的新生dna甲基化方面的作用,表明全域再程序化事件需要有效保护免于在去除5mc之后的新生dna甲基化以使新获得的表观遗传状态稳定。总体来说,我们的研究性腺表观遗传再程序化需要复合擦除表观遗传信息4的想法,且表明这个过程的中心功能是确定grr基因的及时且有效活化,因此使得能够朝向配子发生进展(图16)。
方法
统计学和再现性
所有统计学测试在图式图例中和/或在方法章节中清楚地描述,且精确p值或调整p值在可能的地方给出。对于wgbs数据(图3a-b,5a,6c-e,7a-b,8,11,12),数据是来源于来自n=1(e10.5pgc样本)或n=2(所有其它样本)个生物学重复的细胞,其中来自合并胚胎的各重复(e10.5:n=39胚胎/4仔(litter);e11.5:n=8胚胎/1仔;e12.5m/f:n=4胚胎/1仔)。对于abaseq数据(图1d,3a-b,5c-e,6a-f,7a-b,8,11,12,13b),数据是来源于来自n=2个生物学重复的细胞,其中来自合并胚胎的各重复(e10.5:n=40胚胎/4仔;e11.5:n=8胚胎/1仔;e12.5m/f:n=4胚胎/1仔)。对于mesc的rna-seq,样本是来源于对应于来自n=1细胞系的n=2独立培养的样本的n=2个生物学重复。对于pgclc/ms、rna-seq和rrbs数据,请参见关于作为样本来源的胚胎/仔的数量的完全详情。进行三次免疫印迹(图13f,15b),具有类似结果,且显示代表性印迹。所有免疫染色(图1e,2a-b,图10b)进行两次,具有类似结果,且显示代表性图像。进行传统亚硫酸氢盐测序(图10f-g)两次,且显示代表性甲基化概况。对于先前公开的来自mesc的wgbs(图14a-b)、tab-seq(图5c-e)、abaseq(图5c-e,6b,14a,14c)和chip-seq(图3b,图14a,14c-g)数据集(登录号参见方法)的分析,除h2aubchip-seq数据集(其中n=1)以外,对生物重复进行组合(显示)和单独(未显示)分析以确保分析的再现性。
小鼠
按照且根据英国家庭办公项目许可证(ukhomeofficeprojectlicense)在家庭办公指示设施(home-officedesignatedfacility)中进行所有动物实验。除与tet1-kopgc直接比较以外,从通过远亲杂交mf1雌性与混合背景的gof18δpe-egfp5转基因雄性产生的胚胎分离野生型pgc。通过视觉检查性腺确定从e12.5开始的胚胎的性别。对于tet1-kopgc的研究,tet1基因敲除小鼠品系(b6;129s4-tet1tm1.1jae/j)18是购自杰克逊实验室(jacksonlaboratory)且培育到gof18δpe-egfp5转基因小鼠系上。从由tet1-杂合gof18δpe-egfp-纯合雌性与雄性之间杂交产生的胚胎分离野生型和tet1-kopgc。对于通过tet1-杂合gof18δpe-egfp-纯合雄性和雌性杂交产生的胚胎的基因分型,始终使用两组不同的引物(参见下文)进行两次pcr以确认外显子4缺失。从e12.5开始的胚胎的性别通过视觉检查性腺来确定且另外通过对sry进行pcr来确认。在所有情况下,以在正午出现阴道塞定义为e0.5的方式来给交配计时。
分子生物学
在本研究中使用以下基因分型引物:tcagggagctcatggagacta(tet1正向引物1);aactgattcccttcgtgcag(tet1正向引物2);ttaaagcatgggtgggagtc(tet1反向引物);ttgtctagagagcatggagggccatgtcaa(sry正向引物);ccactcctctgtgacactttagccctccga(sry反向引物)。
通过流式细胞测量术进行pgc分离
如先前所描述4进行pgc分离。简言之,使用0.05%胰蛋白酶-edta(1×)(吉毕科(gibco))或trypleexpress(赛默(thermo)),在37℃下消化胚胎干(e10.5)或生殖嵴(e11.5-e14.5)3min。酶促消化后,用含有15%胎儿牛血清(吉毕科)的dmem/f-12(吉毕科)中和,且通过移液手动解离。在离心之后,将细胞再悬浮于补充有透明质酸酶(300μg/ml;西格玛(sigma))的dmem/f-12中,且通过手动移液产生单细胞悬浮液。在离心之后,将细胞再悬浮于补充有聚乙烯醇(10μg/ml)和egta(0.4mg/ml,西格玛)的冰冷pbs中。使用ariaiiu(bd生物科学(bdbioscience))或ariaiii(bd生物科学)流式细胞仪分离gfp阳性细胞,且将所述细胞分选到补充有聚乙烯醇(10μg/ml)和egta(0.4mg/ml,西格玛)的冰冷pbs中。
tet1-kodnmt-tkomesc的产生
通过crispr/cas9介导的基因组编辑,产生tet1-kodnmt-tkomesc系。使用脂染胺3000,在5×106个dnmt-tkomescs23中用报告子gfp质粒共转染具有sgrna靶向tet131(ggctgctgtcagggagctca)的px330(爱德基因(addgene),#42230)。第二天,通过在96孔板中facs(bdfacsariaiii)分选gfp阳性细胞。在冷冻和提取gdna之前,培养细胞一周。使用测量器分析(来自艾迪悌公司(idt)的测量器突变检测试剂盒,和来自凯杰(qiagen)的taqdna聚合酶),来筛选菌落的突变。通过基因型测序进一步分析tet1-kodnmt-tkomesc选择的克隆,其确认框移突变的存在。通过rna-seq和免疫印迹验证tet1的缺失。使用以下引物用于基因型测序和测量器分析:5'ttgttctctcctctgactgc3'和5'tgattgatcaaataggcctgc3'。
mesc细胞培养
在0.1%明胶的不具有喂料的fcs/lif介质中培养j1(野生型)、dnmt-tko23和tet1-kodnmt-tkomesc。fcs/lif介质由gmem(吉毕科)和小鼠lif(esgro,密理博)组成,所述gmem补充有10%fcs、0.1mmmem非必需的氨基酸、2mml-谷氨酰胺、1mm丙酮酸钠和0.1mm2-巯基乙醇。对抑制剂实验,以1.5×104/cm2的密度涂铺mesc且留置隔夜。第二天上午,用含有50μmprc1抑制剂prt4165(ismail等人,2013)或dmso对照的fcs/lif介质交换介质,且在所指示时间处集结细胞以用于分析。
abaseq文库制备
使用qiaampdna微小试剂盒(凯杰),从10,000个分选的pgc分离总dna。如先前所描述15,构建用于5hmc分析的abaseq文库。简单来说,对基因组dna葡萄糖基化,随后通过abasi酶(neb)消化。将生物素标记的p1衔接子接合到abasi消化的dna上,随后按照制造商说明书使用科瓦里斯s2超声发生器(科瓦里斯(covaris))进行片段化。随后,根据制造商的说明书,通过与dynabeadsmyone抗生蛋白链菌素c1珠粒(生命技术公司(lifetechnologies))混合,来捕获断裂的p1接合的dna。通过分别在20℃和37℃下使用nebnext末端修复模块(neb)和nebnext加da尾模块(neb)30min,在珠粒上进行末端修复和加da尾。将p2衔接子与加da尾的dna的随机断裂末端接合。最终,使用phusiondna聚合酶(neb),添加300nm正向引物(pcr_i)和300nm反向引物(pcr_iipe),扩增全dna16个循环。使用ampurexp珠粒(贝克曼-库尔特)纯化文库,且在illuminahiseq2000仪器上测序。
全基因组亚硫酸氢盐测序(wgbs)文库制备
使用qiaampdna微小试剂盒(凯杰),从10,000个分选的pgc分离总dna。在一些情况下,在dna分离之后,掺入未甲基化的λ噬菌体dna(普洛麦格(promega)),以评定亚硫酸氢盐转化率。使用科瓦里斯s2超声发生器(科瓦里斯(covaris)),按照制造商说明书,将dna片段化。按照nebnext文库制备型方案,用甲基化的衔接子和以下修饰来制备文库:衔接子接合后,使用imprintmodification试剂盒(西格玛)进行亚硫酸氢盐转化;和使用nextflex亚硫酸氢盐-seq试剂盒illumina测序(biooscientific)主混合物和nebnext文库制备型通用和索引引物(neb)进行pcr富集16个循环。通过ampurexp珠粒(贝克曼-库尔特)来纯化文库。在illuminahiseq2000或2500仪上对文库进行测序。
简化表示的亚硫酸氢盐测序(rrbs)文库制备
使用zr-duetdna-rnaminiprep试剂盒(兹莫(zymo))从自个别tet1-ko或野生型胚胎分离的facs分选的pgc分离总dna,且将来自相同基因型、阶段和性别的两个到六个胚胎(相当于1,000到8,000个细胞)的dna合并且使用savantspeedvac浓缩器(赛默)且按照制造商说明书浓缩到26μl最终体积。通过在37℃下含20单位的mspi酶(neb)的neb缓冲液2消化基因组dna3小时,且使用ampurexp珠粒(贝克曼-库尔特)纯化消化的dna。按照nebnextultradna文库制备型方案,用甲基化的衔接子和以下修饰来制备文库:衔接子接合后,使用imprintmodification试剂盒(西格玛)进行亚硫酸氢盐转化;和使用kapauracil+dna聚合酶主混合物(卡帕生物系统(kapabiosystems))和nebnext文库制备型通用和索引引物(neb)进行pcr富集18个循环。通过ampurexp珠粒(贝克曼-库尔特)来纯化文库。如先前所描述32,在illuminahiseq2500仪上,使用‘暗测序’方案,对合并文库进行测序。
rna-seq文库制备
对于tet1-kopgc的研究,使用zr-duetdna-rnaminiprep试剂盒(兹莫)从自个别tet1-ko或野生型胚胎分离的分选的pgc分离总rna,且将来自相同基因型、阶段和性别的两个到六个胚胎(相当于1,000到8,000个细胞)的rna合并且使用rna清洁和浓缩器5试剂盒(兹莫)浓缩到6μl最终体积。对于从通过mf1雌性与gof18δpe-egfp雄性杂交产生的胚胎分离的野生型pgc的研究,使用nucleospinrnaxs试剂盒(马歇雷-纳格尔(macherey-nagel))从600-1,000个分选的e10.5pgc分离总rna。用smarter超低输入rna试剂盒(克隆科技公司(clontech)),使用100pg与3ng之间的总rna且按照制造商说明书,进行cdna合成和扩增(15个循环)。通过科瓦里斯s2超声发生器(科瓦里斯)且按照制造商说明书,将扩增的cdna片段化。使用nebnextdna文库制备型试剂盒(neb),按照制造商说明书且使用15个扩增循环,将断裂cdna转化成测序文库。对于mesc的研究,使用zr-duetdna-rnaminiprep试剂盒(兹莫),分离总rna。以500ng总rna起始,按照制造商说明书,使用nebnextultra文库制备型试剂盒(neb)和nebnextpoly(a)mrna磁性隔离模块(neb),进行cdna合成和文库制备。使用ampurexp珠粒(贝克曼-库尔特)来纯化所有文库,且在illuminahiseq2500仪器上测序。
生物信息学
全基因组亚硫酸氢盐测序(wgbs)和tet辅助的亚硫酸氢盐测序(tab-seq)比对和下游分析
首先使用trimgalore(版本0.3.1),与--配对的--trim1选择,修整原始读段。用bismark(版本0.13.0)与-n1参数,对小鼠基因组(mm9,ncbi建构37)进行比对;在适当时,添加λ噬菌体基因组作为额外染色体。用deduplicate_bismark对比对的读段去重。在适当时,使用与λ噬菌体基因组比对的读段且使用methpipe(版本3.3.1)的to-mr脚本(参数:-mbismark)和bsrate脚本(参数:-n),来计算亚硫酸氢盐转化率。使用bismark甲基化提取器,从去重后的定位输出提取cpg甲基化响应。使用bismark2bedgraph和coverage2cytosine来提取cpg内容中的甲基化和未甲基化胞嘧啶的数量。用定制r脚本合并对称cpg。对于所有下游分析,仅使用具有最小8×覆盖率的对称cpg。对于来自合并生物学重复的数据进行所有wgbs分析。对于评定特定重复元件处的dna修饰水平,使用bismark(版本0.14.4)来针对用-n1参数集合的repbase构筑的共有序列定位来自各数据集合的所有读段。使用bismark甲基化提取器(版本0.14.4),从定位输出提取cpg甲基化响应。
使用bedtools(版本2.24.0)的mapbed功能来计算以下基因组特征的组合5mc/5hmc水平:1)所有2kb窗口(含有最小4个对称cpg);2)基因启动子(如ensembl67基因起始位点-1kb/+500bp所定义);3)基因体(如含于ensembl67基因起始和基因终止位点内的区所定义);4)第6天pgclc33中的假定的活性增强子;5)印记控制区;6)cpg岛(ucsc);7)基因间区域。对于metagene图,使用bedtools(版本2.24.0)将基因组特征分成同等大小的组,包含:1)基因体(如含于ensembl67基因起始和基因终止位点内的区所定义)+/-0.5×基因体长度(100个组);2)第6天pgclc33中的假定的活性增强子+/-1*假定的活性增强子长度(90个组);和3)cpg岛(ucsc)+/-1*cpg岛长度(90个组)。在所有情况下,组合5mc/5hmc水平表述为个别cpg位点的平均值。
对于组合平均值5mc/5hmc水平的k-平均值聚类,高cpg(hcp)、中等cpg(icp)和低cpg(lcp)启动子,如使用与先前公开19,34的相同参数定义。简言之,lcp不含有cpg比率>0.45的500-bp窗口;hcp含有cpg比率>0.65且gc含量>55%的至少一个500-bp窗口;icp不满足前述标准。
对于测定含血清介质中生长的野生型mesc中的基因座特异性甲基化水平,从gse4851930下载原始wgbs读段且如上进行处理。从gse3617335下载e14mesc的tab-seq读段且如上进行处理,不同之处在于仅使用具有最小12×覆盖率的对称cpg。
abaseq比对和下游分析
对于基因组的独特可定位部分,如先前所描述15来处理abaseq读段。简单来说,使用trimgalore,针对衔接子序列和低质量碱基,对原始测序读段进行修整。使用具有参数的-n1-l25--best--strata-m1的bowtie(版本0.12.8),将修整的读段定位到小鼠基因组(mm9,ncbi建构37)。5hmc的响应(calling)是基于使用定制perl脚本的abasi酶(5′-cn11-13↓n9-10g-3′/3′-gn9-10↓n11-13c-5′)的识别序列和裂解模式。对于评定重复元件和非重复元件处的5hmc的相对富集,将abaseq比对分成两个群组:独特(单一最佳比对)和不明确的(定位到具有相同比对评分的多个位置)。随后,将两个群组各自定位到由mm9(ucsc基因组浏览器)的repeatmasker轨迹定义的重复元件。对于与mesc中的5hmc水平的比较,从gse4289815下载abaseq读段且以相同方式进行比对。
对于基因组的独特定位部分中的对称cpg处的相对5hmc水平的量化,将给定样本的每一对称cpg的计数数量标准化为既定文库的独特定位和不明确定位的读段的组合数量,且随后进一步乘以基于各阶段的平均5hmc水平的阶段特异性标准化因子,所述各阶段的平均5hmc水平由lc/ms计算(e14esc=1.64;e10.5=1.0;e11.5=1.13;e12.5f=0.76;e12.5m=1.0)。将在小鼠(mm9)encode项目黑名单上的属于基因组间隔内的所有对称cpg排除在所有进一步下游分析外。除非另外陈述,否则所有abaseq分析在来自合并生物学重复的数据上进行。
如对wgbs数据集(参见上文)进行的那样,使用bedtools(版本2.24.0)的mapbed功能来计算相同基因组特征的5hmc水平。在所有情况下,5hmc水平表述为个别cpg位点的平均值。
为了鉴定e10.5和e11.5pgc中的5hmc富集或耗尽区,将mm9基因组首先分成2kb窗口(最小4个对称cpg),且使用bedtools(版本2.24.0)来计算各窗口的平均5hmc水平。为了确定各2kb窗口中的5hmc富集的显著性,使用ppois(x,λ)来计算上尾(用以测定5hmc富集区)或下尾(用以测定5hmc耗尽区)poisson机率p值,其中x是所观测到的各2kb窗口的5hmc平均值,且λ是在e10.5时所有2kb窗口的5hmc平均数值的平均值。随后,将benjamini-hochberg校正应用于校正多个测试,得到各2kb窗口的最终调整的上尾和下尾p值。调整的上尾p值<0.05的窗口视为5hmc相对富集的,而调整的下尾p值<0.05的窗口视为5hmc相对耗尽的。
对于评定特定重复元件处的5hmc的相对富集,使用bowtie来针对从具有参数-n1-m1--strata-best的repbase构筑的共有序列定位来自各数据集合的所有读段。将定位到既定样本内的各序列的读段的数量首先标准化为那个特定样本的文库大小,且随后标准化为基于各阶段的平均5hmc水平的阶段特异性标准化因子,所述各阶段的平均5hmc水平由lc/ms计算所得(e10.5=1.0;e11.5=1.13;e12.5f=0.76;e12.5m=1.0)和定位到e10.5pgc中的既定序列的读段的平均比例。
简化表示的亚硫酸氢盐测序(rrbs)比对和下游分析
使用具有--rrbs参数的trimgalore(版本0.3.1),首先修整原始rrbs读段。用具有-n1参数的bismark(版本0.13.0),进行相对于小鼠基因组(mm9,ncbi建构37)的比对。使用bismark甲基化提取器(版本0.13.0),从定位输出提取cpg甲基化响应。使用bismark2bedgraph来提取cpg内容中的甲基化和未甲基化胞嘧啶的数量。
使用rnbeads(版本1.0.0)和rnbeads.mm9(版本0.99.0)来鉴定以下基因组特征的两个测试组之间的差异甲基化区,其中filtering.missing.value.quantile设定为0.95且filtering.missing.coverage.threshold设定为8:1)所有2kb窗口(含有最小4个对称cpg);2)基因启动子(如ensembl67基因起始位点-1kb/+500bp所定义);和3)印记控制区(mm9基因组)。从rnbeads的输出提取以下:1)各通常覆盖率的测试区的各群组(即阶段、性别和/或基因型)的平均甲基化水平;2)各通常覆盖率测试区的两个群组之间的甲基化平均值的差异;和3)表示各通常覆盖率测试区的两个群组之间的甲基化平均值的差异的显著性的p值。差异甲基化区鉴定为p值<0.05且两个群组之间的甲基化平均值的差异大于10%的区。
对于评定特定重复元件处的dna修饰水平,使用bismark(版本0.14.4)来针对用-n1参数集合的repbase构筑的共有序列定位来自各数据集合的所有读段。使用bismark甲基化提取器(版本0.14.4),从定位输出提取cpg甲基化响应。使用bismark2bedgraph和coverage2cytosine来提取cpg内容中的甲基化和未甲基化胞嘧啶的数量。差异甲基化的共有重复序列鉴定为p值<0.05(如通过双侧史都登氏t测试计算)且两个群组之间的甲基化平均值的差异大于10%的区。
hmedip比对和下游分析
从gse2850036下载e14mesc的原始hmedip-seq和输入读段,且用具有参数-n2-l25-m1的bowtie(版本0.12.8)与小鼠基因组(mm9,ncbi建构37)进行比对。使用bedtoolsmulticov来鉴定重叠各2kb窗口(含有最小4个对称cpg)的hmedip和的输入读段的数量。通过首先用重叠输入读段的数量(标准化为文库大小)标准化重叠hmedip读段的数量(标准化为文库大小),且随后使这个值除以2kb窗口内所含的对称cpg的数量,来确定各2kb窗口的最终5hmc水平。
chip-seq比对和下游分析
对于假定的活性增强子响应,从gse6020433下载第6天pgc样细胞(pgclc)中的h3k4me3、h3k27me3和h3k27ac的原始chip-seq读段,且从gse4851930下载野生型mesc中的h3k4me3、h3k27me3、h3k4me1和h3k27ac的原始chip-seq读段。用具有参数-n2-l25-m1和-c(在适当时)的bowtie(版本0.12.8或版本1.0.0),将读段与小鼠基因组(mm9,ncbi建构37)进行比对。对于来自合并生物学重复的数据进行后续chip-seq分析。为了鉴定假定的活性增强子,本发明人首先使用chromhmm产生8-状态染色质模型。假定的活性增强子定义为不重叠任何潜在启动子区(ensembl67基因起始位点-1kb/+500bp)且含于第6天pgclc中的(h3k27ac+/h3k4me3-/h3k27me3-)染色质状态或野生型mesc中的(h3k4me1+/h3k27ac+/h3k4me3-/h3k27me3-)内的所有区。
对于转录起始位点(ensembl67)附近的表观遗传修饰和修饰物的分析,以下的原始chip-seq读段:从gse2484321下载野生型血清生长的mesc中的tet1结合;从gse3452037下载野生型血清生长的mesc中的h2ak119ub1水平;从erp00557538下载野生型血清生长的mesc中的ring1b结合;和从gse4851930下载野生型和tet1-ko血清生长的mesc中的h3k4me3。用具有参数-n2-l25-m1的bowtie(版本0.12.8或版本1.0.0),将读段与小鼠基因组(mm9,ncbi建构37)进行比对。对于来自合并生物学重复的数据进行后续chip-seq分析。对于计算转录起始位点(tss)附近的chip-seq信号,使用bedtoolsmakewindows,将ensembl67基因起始位点+/-5kb(或2kb)附近的基因组间隔分成100(或40)个同等大小的组。随后,使用bedtoolsmulticov来计算重叠各组的测试和对照读段的数量。将各样本的每组的测试和对照读段的总数标准化为适当的文库大小,且通过使标准化的chip-seq测试样本读段的数量除以标准化的chip-seq对照样本读段的数量来确定各组的富集倍数。对于计算基因启动子处的chip-seq信号,ensembl67基因起始位点+500bp/-1kb附近的基因组间隔为
rna-seq比对和下游分析
对于tet1-ko和tet1-wtpgc的研究,首先使用trimmomatic来修整来自测序读段的illumina和smart-seq衔接子。对于其它rna-seq文库,由下一代测序的输出产生的fastq文件直接用于比对。用bowtie(版本0.12.8)和tophat(版本2.0.2),与选择-n2--b2-极-敏感的--b2-l25,将rna-seq读段与小鼠基因组(mm9,ncbi建构37)进行比对。来自ensemblgene版本67的注解用作tophat的基因模型。使用htseq(版本0.5.3p9)计算每个注释的基因的读段计数,且通过使用定制r脚本计算每千碱基每百万读段检测的片段(fpkm)数量,来量化各基因的表达水平。基于两个生物学重复的平均fpkm值,将基因归为表达水平组。使用deseq2(版本1.6.3)进行差异表达分析,且调整p值<0.05的基因视为差异表达的。对于测定野生型和dnmt1-条件性基因敲除和匹配野生型e10.5pgc中的基因表达水平,从gse7493824下载原始rna-seq读段且如上进行处理。
基于e10.5和e14.5pgc中的基因表达之间的活化显著性(α)(图4b),对在表观遗传再程序化期间的pgc中的甲基化和去甲基化的hcp(簇3,图4a)进行排名。在其中β表示倍数变化的方向性(即如果log2(fc)<0,β=-1,那么β=+1)且γ表示如通过deseq2计算的调整p值的情况下,α=β×(1-γ)。对于比较以下中的grr基因集合的表达水平:1)野生型、dnmt-tko和tet1-kodnmt-tkomesc(图6a);2)野生型+6hdmso处理、dnmt-tko+6hdmso处理、野生型+6hprt4165处理、dnmt-tko+6hprt4165处理(图6c);3)tet1-koe14.5pgc相对于野生型e14.5pgc(图5b);或3)dnmt1-ckoe10.5pgc相对于野生型e10.5pgc(图13g),最初通过deseq2进行各条件相对于彼此条件的成对差异表达分析。对于各成对差异表达测试,对各基因分配统计α,其中如果β表示log2(fc)且γ表示如通过deseq2计算的调整p值,那么α=β×(1-γ)。随后,使用基于α的排名基因列表来进行基因集富集分析(gsea)以用于测试组合grr基因集合和gsea标志基因集合的通常上或下调,且随后使用gseafwer调整的p值。对于pgc中的生殖系再程序化应答基因与prc1所抑制的基因之间的重叠(图6b),从26下载称为在e11.5和/或e12.5prc1-kopgc中被上调的基因列表。
对于grr基因(图14,表1)的分类,首先进行成对差异表达分析。5mc再程序化依赖性grr基因定义为进行以下的基因:1)dnmt-tko相对于wt、dnmt-tko+prc1抑制剂相对于wt和dnmt-tko+prc1抑制剂相对于wt+prc1抑制剂上调;和2)wt+prc1抑制剂相对于wt不上调。prc1再程序化依赖性grr基因定义为进行以下的基因:1)wt+prc1抑制剂相对于wt、dnmt-tko+prc1抑制剂相对于wt和dnmt-tko+prc1抑制剂相对于dnmt-tko上调;和2)dnmt-tko相对于wt不上调。5mc/prc1再程序化依赖性grr基因定义为进行以下任一个的基因:1)wt+prc1抑制剂相对于wt、dnmt-tko相对于wt、dnmt-tko+prc1抑制剂相对于wt、dnmt-tko+prc1抑制剂相对于dnmt-tko和dnmt-tko+prc1抑制剂相对于wt+prc1抑制剂上调;或2)dnmt-tko+prc1抑制剂相对于wt、dnmt-tko+prc1抑制剂相对于dnmt-tko和dnmt-tko+prc1抑制剂相对于wt+prc1抑制剂上调,且wt+prc1抑制剂相对于wt和dnmt-tko相对于wt不上调。5mc/prc1再程序化非依赖性或不足grr基因定义为dnmt-tko相对于wt、dnmt-tko+prc1抑制剂相对于wt、和dnmt-tko+prc1抑制剂相对于wt+prc1抑制剂以及wt+prc1抑制剂相对于wt不上调的基因。确实归为这五个类别中的一个的基因被描述为低置信度分类(l.c.c.)基因。
通过免疫荧光进行tet1和5mc/5hmc检测
首先将胚胎干(e10.5)或生殖嵴(e12.5/e13.5)在2%pfa(于pbs中)中4℃下固定30min。在固定之后,在pbs中洗涤组织三次持续10min,且随后在含15%蔗糖的pbs中培育隔夜。在用含1%bsa的pbs冲洗之后第二天,将组织包埋于oct包埋基质(赛默科技雷蒙德兰姆(thermoscientificraymondlamb))中且使用液氮冷冻。随后将样本存储在-80℃下。使用leicacm1950低温恒温器来从冷冻包埋组织切割下10μm切片。将切片置于聚赖氨酸载玻片(赛默科技)上且用含2%pfa的pbs后固定3分钟。
对于tet1的检测,用pbs洗涤切片三次持续5min。在室温下于含有0.1%tritonx-100的1%bsa/pbs中培育30min之后,将切片与所列举的一级抗体于相同缓冲液中在4℃下一起培育隔夜。随后,在含有0.1%tritonx-100的1%bsa/pbs中洗涤切片三次持续5min,且与二级抗体在相同缓冲液中在室温下一起暗培育1小时。随后,用pbs对二级抗体培育物进行三次5min洗涤。随后,用dapi(100ng/ml)对dna进行染色。在pbs中最后洗涤10min之后,用vectashield(载体实验室(vectorlaboratories))装设切片。
对于5hmc/5mc的检测,用pbs洗涤切片三次持续5min。固定后切片首先用0.5%tritonx-100(于1%bsa/pbs中)渗透30min,且随后在37℃下用含rna酶a(10mg/ml;罗奇(roche))的1%bsa/pbs处理1小时。在用pbs进行三次5min洗涤之后,将切片与4nhcl在37℃下一起培育10-20min以使基因组dna变性,随后用pbs进行三次10min洗涤。在室温下于含有0.1%tritonx-100的1%bsa/pbs中培育30min之后,将切片与所列举的一级抗体于相同缓冲液中在4℃下一起培育隔夜。随后,在含有0.1%tritonx-100的1%bsa/pbs中洗涤切片三次持续5min,且与二级抗体在相同缓冲液中在室温下一起暗培育1小时。随后,用pbs对二级抗体培育物进行三次5min洗涤。随后,用碘化丙锭(propidiumiodide,pi)(0.25mg/ml)对dna进行染色。在pbs中最后洗涤10min之后,用vectashield(载体实验室(vectorlaboratories))装设切片。
在本研究中使用以下一级抗体:抗ssea1(p.beverly博士通过g.durcovahills博士赠予);抗mvh(艾博抗(abcam)27591或艾博抗13840);抗5hmc(活性基序公司(activemotif)39791)、抗5mc(戴格诺德(diagenode)c15200081-100);抗tet1(基因特克斯(genetex)gtx125888);抗gfp(艾博抗5450)。在本研究中使用以下二级抗体:alexafluor647山羊抗小鼠igm(英杰公司(invitrogen)a21238);alexafluor488山羊抗兔igg(英杰公司a11008);alexafluor405山羊抗小鼠igg1:300(英杰公司a31553);alexafluor488山羊抗小鼠igg1:300(英杰公司a11001);alexafluor405山羊抗兔igg1:300(英杰公司a31556);alexafluor568驴抗兔igg(英杰公司a10042);alexafluor488驴抗山羊igg(英杰公司a11055)。
基因座特异性亚硫酸氢盐测序
使用imprintdna修饰试剂盒(西格玛)进行基因组dna的亚硫酸氢盐处理。使用以下引物来进行dazl启动子的半嵌套式扩增:f1:gatttttgttattttttagtttttttaggat;f2:tttatttaagttattattttaaaaatggtatt;r:agaaacaagctaggccagctgagagaattct。使用以下引物来进行ig-dmricr的半嵌套式扩增:f1:gtgttaaggtatattatgttagtgttagg;f2:atattatgttagtgttaggaaggattgtg;r:tacaacccttccctcactccaaaaatt。使用以下引物来进行peg3icr的嵌套式扩增:f1:tttttagattttgtttgggggtttttaata;f2:ttgataatagtagtttgattggtagggtgt;r1:aatccctatcacctaaataacatccctaca;r2:atctacaaccttatcaattacccttaaaaa。通过quma,使用默认设置来评定甲基化水平,其中重复亚硫酸氢盐序列被排除。
质谱法
使用zr-duetdna/rnaminiprep试剂盒(兹莫研究(zymoreasearch)按照制造商说明书来从100到2,000个facs分选的pgc提取基因组dna,且在lc/ms级水中洗脱。使用neb提供的消化酶混合物,将dna消化为核苷。对用已知量的合成核苷和消化dna制备的稀释系列外加类似量的同位素标记的核苷(t.carell博士(lmu,德国)提供),且通过使用uhplc1290系统(安捷伦(agilent))和安捷伦6490三重四极杆质谱仪在安捷伦rrhdeclipseplusc182.1×100mm1.8μ柱上分开。为了计算个别核苷的数量,生成表示未标记的核苷相对于同位素标记核苷的比率的标准曲线,且用于将峰面积值(peak-areavalues)转化为对应数量。量化的阈值是高于10的信噪比(用峰-峰法计算)。
免疫印迹
通过在ripa缓冲液(150mm氯化钠、1.0%tritonx-100、0.5%脱氧胆酸钠、0.1%十二烷基硫酸钠、50mmtrisph8.0)和蛋白酶-抑制剂混合物(罗奇,11697498001)中超声处理来使mesc裂解。通过以14000g在4℃下离心5min来去除细胞碎片。使用bca蛋白质分析(赛默,23227)来量化蛋白质。将2μg(h2a和h2aub)或20μg(tet1)的各蛋白质提取物负载到15%或8%sds聚丙烯酰胺凝胶上,且在电泳之后转移到pvdf膜。膜用5%bsa阻断1小时,且随后在4℃下与一级抗体以以下稀释一起培育隔夜:抗h2a抗体(艾博抗,18255)1:2000;抗泛素h2a抗体(细胞信号传导(cellsignalling)8240)1:2000;抗tet1抗体[n1](基因特克斯gtx125888)1:1000;抗laminb抗体(c20)(圣克鲁兹生物技术(santacruzbiotechnologies),sc-6216)1:10000。在室温下培育驴抗兔igg-hrp(圣克鲁兹生物技术,sc-2077)或驴抗山羊igg-hrp(圣克鲁兹生物技术,sc-2056)二级抗体1h。通过使用luminatacrescendowesternhrp底物(伊艾姆帝密里博(emdmilipore)来使印迹显色。
参考文献
按字母顺序排列的参考文献:
chungetal.,2008humanembryonicstemcelllinesgeneratedwithoutembryodestruction.cellstemcell.2(2)113-117.epub2008jan10
galdonetal.invitrospermatogenesis:howfarfromclinicalapplication?currurolrep17;49(2016)
hilletal.epigeneticreprogrammingenablestheprimordialgermcell-to-gonocytetransition,nature;555(7696):392–396;15mar2018.
jiangetal.,pluripotencyofmesenchymalstemcellsderivedfromadultmarrow.nature418,41–49,2002
kajietalvirus-freeinductionofpluripotencyandsubsequentexcisionofreprogrammingfactors.nature.onlinepublication1march2009
kanatsu-shinoharaetal.,generationofpluripotentstemcellsfromneonatalmousetestis.cell119,1001–1012,2004
phillipsetal.spermatogonialstemcellregulationandspermatogenesis.philtransrsocb365,1663-1678(2010)
matsuietal.,derivationofpluripotentialembryonicstemcellsfrommurineprimordialgermcellsinculture.cell70,841–847,1992
medranoetal.humansomaticcellssubjectedtogeneticinductionwithsixgermline-relatedfactorsdisplaymeioticgermcell-likefeatures.scientificreports6:24956:10.1038(2016)
编号的参考文献(主要):
1lesch,b.&page,d.geneticsofgermcelldevelopment.natrevgenet13,781-794(2012).
2guibert,s.,forné,t.&weber,m.globalprofilingofdnamethylationerasureinmouseprimordialgermcells.genomeres22,633-641(2012).
3hackett,j.etal.germlinednademethylationdynamicsandimprinterasurethrough5-hydroxymethylcytosine.science339,448-452(2013).
4hajkova,p.etal.chromatindynamicsduringepigeneticreprogramminginthemousegermline.nature452,877-881(2008).
5hajkova,p.etal.epigeneticreprogramminginmouseprimordialgermcells.mechdev117,15-23(2002).
6hill,p.,amouroux,r.&hajkova,p.dnademethylation,tetproteinsand5-hydroxymethylcytosineinepigeneticreprogramming:anemergingcomplexstory.genomics104,324-333(2014).
7lee,j.etal.erasinggenomicimprintingmemoryinmousecloneembryosproducedfromday11.5primordialgermcells.development129,1807-1817(2002).
8seisenberger,s.etal.thedynamicsofgenome-widednamethylationreprogramminginmouseprimordialgermcells.molcell48,849-862(2012).
9yamaguchi,s.etal.tet1controlsmeiosisbyregulatingmeioticgeneexpression.nature492,443-447(2012).
10yamaguchi,s.,shen,l.,liu,y.,sendler,d.&zhang,y.roleoftet1inerasureofgenomicimprinting.nature504,460-464(2013).
11hajkova,p.etal.genome-widereprogramminginthemousegermlineentailsthebaseexcisionrepairpathway.science329,78-82(2010).
12hayashi,k.etal.offspringfromoocytesderivedfrominvitroprimordialgermcell-likecellsinmice.science338,971-975(2012).
13hayashi,k.,ohta,h.,kurimoto,k.,aramaki,s.&saitou,m.reconstitutionofthemousegermcellspecificationpathwayinculturebypluripotentstemcells.cell146,519-532(2011).
14hikabe,o.etal.reconstitutioninvitrooftheentirecycleofthemousefemalegermline.nature539,299-303(2016).
15sun,z.etal.high-resolutionenzymaticmappingofgenomic5-hydroxymethylcytosineinmouseembryonicstemcells.cellrep3,567-576(2013).
16huang,y.etal.thebehaviourof5-hydroxymethylcytosineinbisulfitesequencing.plosone5,e8888(2010).
17yamaguchi,s.etal.dynamicsof5-methylcytosineand5-hydroxymethylcytosineduringgermcellreprogramming.cellres23,329-339(2013).
18dawlaty,m.etal.tet1isdispensableformaintainingpluripotencyanditslossiscompatiblewithembryonicandpostnataldevelopment.cellstemcell9,166-175(2011).
19weber,m.etal.distribution,silencingpotentialandevolutionaryimpactofpromoterdnamethylationinthehumangenome.natgenet39,457-466(2007).
20tahiliani,m.etal.conversionof5-methylcytosineto5-hydroxymethylcytosineinmammaliandnabymllpartnertet1.science324,930-935(2009).
21williams,k.etal.tet1andhydroxymethylcytosineintranscriptionanddnamethylationfidelity.nature473,343-348(2011).
22vella,p.etal.tetproteinsconnecttheo-linkedn-acetylglucosaminetransferaseogttochromatininembryonicstemcells.molcell49,645-656(2013).
23tsumura,a.etal.maintenanceofself-renewalabilityofmouseembryonicstemcellsintheabsenceofdnamethyltransferasesdnmt1,dnmt3aanddnmt3b.genescells11,805-814(2006).
24hargan-calvopina,j.etal.stage-specificdemethylationinprimordialgermcellssafeguardsagainstprecociousdifferentiation.devcell39,75-86(2016).
25mansour,a.etal.theh3k27demethylaseutxregulatessomaticandgermcellepigeneticreprogramming.nature488,409-413(2012).
26yokobayashi,s.etal.prc1coordinatestimingofsexualdifferentiationoffemaleprimordialgermcells.nature495,236-240(2013).
27ismail,i.,mcdonald,d.,strickfaden,h.,xu,z.&hendzel,m.asmallmoleculeinhibitorofpolycombrepressivecomplex1inhibitsubiquitinsignalingatdnadouble-strandbreaks.jbiolchem288,26944-26954(2013).
28deplus,r.etal.tet2andtet3regulateglcnacylationandh3k4methylationthroughogtandset1/compass.emboj32,645-655(2013).
29amouroux,r.etal.denovodnamethylationdrives5hmcaccumulationinmousezygotes.natcellbiol18,225-233,doi:10.1038/ncb3296(2016).
30hon,g.etal.5mcoxidationbytet2modulatesenhanceractivityandtimingoftranscriptomereprogrammingduringdifferentiation.molcell56,286-297(2014).
31yang,h.etal.one-stepgenerationofmicecarryingreporterandconditionalallelesbycrispr/cas-mediatedgenomeengineering.cell154,1370-1379(2013).
32boyle,p.etal.gel-freemultiplexedreducedrepresentationbisulfitesequencingforlarge-scalednamethylationprofiling.genomebiol13,r92(2012).
33kurimoto,k.etal.quantitativedynamicsofchromatinremodelingduringgermcellspecificationfrommouseembryonicstemcells.cellstemcell16,517-532(2015).
34borgel,j.etal.targetsanddynamicsofpromoterdnamethylationduringearlymousedevelopment.natgenet42,1093-1100(2010).
35yu,m.etal.base-resolutionanalysisof5-hydroxymethylcytosineinthemammaliangenome.cell149,1368-1380(2012).
36xu,y.etal.genome-wideregulationof5hmc,5mc,andgeneexpressionbytet1hydroxylaseinmouseembryonicstemcells.molcell42,451-464(2011).
37brookes,e.etal.polycombassociatesgenome-widewithaspecificrnapolymeraseiivariant,andregulatesmetabolicgenesinescs.cellstemcell10,157-170(2012).
38cooper,s.etal.targetingpolycombtopericentricheterochromatininembryonicstemcellsrevealsaroleforh2ak119u1inprc2recruitment.cellrep7,1456-1470(2014).
39hashimoto,h.etal.structureofanaegleriatet-likedioxygenaseincomplexwith5-methylcytosinedna.nature506,391-395(2014).
上文提及的所有参考文献均以引用的方式并入本文中。
- 还没有人留言评论。精彩留言会获得点赞!