猪6号染色体上与瘦肉率、眼肌面积、眼肌厚度相关的SNP分子标记与应用的制作方法
本发明属于分子生物技术和分子标记技术领域,具体涉及一种猪6号染色体上与瘦肉率、眼肌面积、眼肌厚度相关的SNP分子标记与应用。
背景技术:
近些年,随着人民生活水平的提高以及对猪肉消费的需求,瘦肉型猪占据了猪肉消费很大的市场份额。因此,优质瘦肉型猪的选育成为育种工作中的重点,影响猪生长的相关性状是重要的育种目标。猪的瘦肉率是指猪含有的瘦肉重量占整个胴体重量的百分比,是衡量猪生长性能的一个重要指标。长期以来,受益于人们对瘦肉的不断追求,瘦肉率取得了显著提高。眼肌面积和眼肌厚度在背膘厚度和瘦肉率的测量中扮演着重要的角色。因此,眼肌面积和眼肌厚度的改良对评估猪的生长性能是重要的参考因素。
近百年来,以杜洛克为代表的瘦肉型种猪大多以提高瘦肉率和降低背膘厚度为重要的育种目标。理论上来说,增大眼肌面积和眼肌厚度,背膘厚度则会降低。然而,瘦肉率、眼肌面积和眼肌厚度都是复杂的数量性状,由多基因控制,并且表型不易直接观测。因此采用常规育种方法难以快速实现预期的育种目标。而利用分子生物学的方法,通过先找到同时影响瘦肉率、眼肌面积和眼肌厚度的DNA分子标记,进而筛选优势等位基因用于性状改良,则有望提高瘦肉型种猪的瘦肉率和降低背膘厚度,从而提高猪的生长性能。
过去,人们通过候选基因法(Candidate gene approach)和QTL定位(QTL Mapping)进行QTL检测,并且已鉴别到部分影响生长性状的候选基因,如IGF2基因等。候选基因法虽然具有方法简单、操作方便等优点,但只能针对生物学功能已知的基因;而QTL定位方法所鉴定的候选位置基因的置信区域同样较大,这极大地限制了复杂性状分子标记在家畜遗传育种中的应用。随着高通量测序技术的发展及全基因组芯片的出现,使得全基因组关联分析(Genome-wide Association Study,GWAS)在复杂性状遗传解析中效果显著,也打破了猪生长性状分子标记鉴别的瓶颈。
技术实现要素:
为了克服现有技术的不足和缺点,本发明的首要目的在于提供一种猪6号染色体上与瘦肉率、眼肌面积、眼肌厚度相关的SNP分子标记。
本发明的另一目的在于提供上述猪6号染色体上与瘦肉率、眼肌面积、眼肌厚度相关的SNP分子标记的应用。
本发明的再一目的在于提供一种鉴定上述SNP分子标记的引物对。
本发明的第四个目的在于提供上述引物对的应用。
本发明的第五个目的在于提供一种猪的遗传改良方法。
本发明的目的通过下述技术方案实现:
一种猪6号染色体上与瘦肉率、眼肌面积、眼肌厚度相关的SNP分子标记,其SNP位点对应于国际猪基因组11.1版本参考序列6号染色体上第47357966位T>C突变;
所述的猪6号染色体上与瘦肉率、眼肌面积、眼肌厚度相关的SNP分子标记的核苷酸序列如SEQ ID NO:1所示,其中序列中的M是T或C,导致猪瘦肉率、眼肌面积、眼肌厚度的差异;
所述的猪6号染色体上与瘦肉率、眼肌面积、眼肌厚度相关的SNP分子标记,其SNP位点为SEQ ID NO:1序列标注位置为333位的T333-C333的核苷酸突变;
所述的猪6号染色体上与瘦肉率、眼肌面积、眼肌厚度相关的SNP分子标记在鉴定猪瘦肉率、眼肌面积、眼肌厚度相关形状和猪遗传育种中的应用;
利用上述猪6号染色体上与瘦肉率、眼肌面积、眼肌厚度相关的SNP分子标记筛选高瘦肉率、眼肌面积、眼肌厚度性状的猪品种的方法,包含如下步骤:
检测上述猪6号染色体上与瘦肉率、眼肌面积、眼肌厚度相关的SNP分子标记,所述分子标记的5’端第333位单核苷酸是T还是C,淘汰C保留T;
所述的猪为杜洛克其合成系;
所述的猪优选为美系杜洛克及其合成系;
一种用于鉴定上述猪6号染色体上与瘦肉率、眼肌面积、眼肌厚度相关的SNP分子标记的引物对,包含引物P001-F和引物P002-R,其核苷酸序列如下所示:
P001-F:5’-ACTCTTGCCTCCGACTTCTC-3’,
P002-R:5’-AGACCTGGTGACATAGTTGATGA-3’;
所述的引物对在鉴定猪瘦肉率、眼肌面积、眼肌厚度相关性状中的应用;
所述的引物对在猪分子标记辅助育种中的应用;
所述的引物对在提高猪瘦肉率、眼肌面积、眼肌厚度中的应用;
一种猪的遗传改良的方法,包含如下步骤:
确定种猪核心群中的种猪的上述猪6号染色体上与瘦肉率、眼肌面积、眼肌厚度相关的SNP分子标记的位点,并根据所述分子标记做出相应的选择:在所述种猪核心群中选择国际猪参考基因组11.1版本6号染色体上第47357966位点为TC、TT基因型的种猪个体,淘汰在第47357966位点为CC基因型的种猪个体,以逐代提高该位点的等位基因T的频率,从而提高后代猪的瘦肉率、眼肌面积、眼肌厚度;
所述的猪为杜洛克其合成系;
所述的猪优选为美系杜洛克及其合成系;
本发明相对于现有技术具有如下的优点及效果:
(1)本发明研究并确定影响猪瘦肉率、眼肌面积、眼肌厚度相关的分子标记位于猪的6号染色体上的核苷酸序列上,验证其对瘦肉率、眼肌面积、眼肌厚度性状的影响效应,最终建立高效准确的分子标记辅助育种技术,将其应用于种猪提高瘦肉率、眼肌面积、眼肌厚度的遗传改良中,从而提高后代猪的生长性能,提高企业经济利润,增加核心竞争力。通过优选该SNP的优势等位基因,能够逐代增加优势等位基因频率,提高种猪瘦肉率、增大眼肌面积和眼肌厚度,协同选育上述三个性状的优良种猪,加快猪遗传改良进展从而有效提高种猪育种的经济效益。
(2)本发明提供一种鉴定上述猪6号染色体上与瘦肉率、眼肌面积、眼肌厚度相关的SNP分子标记的引物对,通过该引物对,可建立高效准确的分子标记辅助育种技术,快速准确地对肉质品质进行选育,加快育种进程。
附图说明
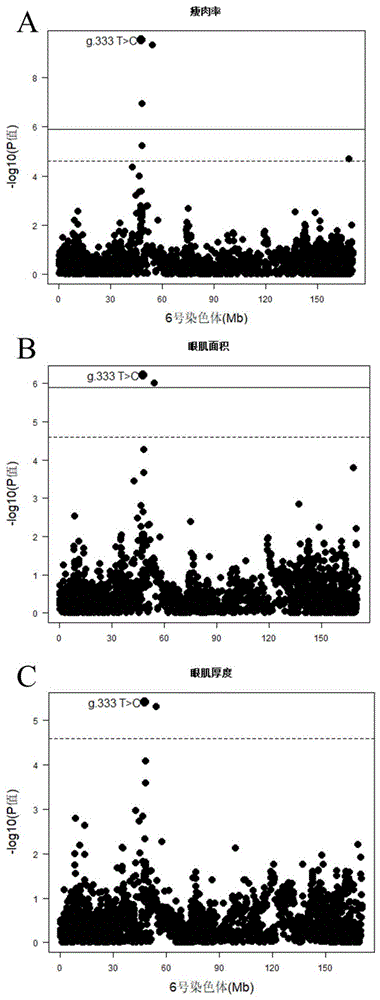
图1是美系杜洛克猪在6号染色体上关于瘦肉率、眼肌面积、眼肌厚度性状的全基因组关联分析(GWAS)曼哈顿图;其中:横坐标表示猪的染色体编号;纵坐标表示-logP值;A:瘦肉率,B:眼肌面积,C:眼肌厚度。
图2是不同基因型猪的瘦肉率、眼肌面积、眼肌厚度的表型比例结果分析图;其中,A:瘦肉率,B:眼肌面积,C:眼肌厚度。
具体实施方式
下面结合实施例及附图对本发明作进一步详细的描述,但本发明的实施方式不限于此。
实施例1
(1)实验动物
本发明所使用的实验猪群群体为温氏食品集团股份有限公司种猪分公司的纯种美系杜洛克3916头,为种猪分公司核心群。
本实验共选取该资源群体中美系杜洛克种猪,猪群自由采食、饮水,整个饲喂方式、饲养条件等始终保持一致,为常规方法。
(2)样本采集
收集上述仔猪断尾及耳组织浸泡于体积分数为75%的乙醇溶液中,置于-20℃冰箱保存备用。
(3)猪全基因组50K SNP判型
从上述资源群体中选取的3916头杜洛克种猪中的每个个体采集的耳组织或者断尾组织,用标准苯酚-氯仿法提取全基因组DNA,经Nanodrop2000/2000C核酸蛋白检测仪准确测定每一份样品的DNA的浓度和OD比值(OD260/280、OD260/230)。经NanoDrop2000/2000C核酸蛋白检测仪检测合格的DNA样品,按照检测的浓度将DNA稀释至50ng/μL左右。再将6μL已提取好的待测DNA样品与2μL Loading Buffer混合,上样到质量体积比为1%的琼脂糖凝胶中,150V电压下电泳25min,在紫外分光光度仪和凝胶成像设备下观察并拍照,观察DNA的完整性。
DNA样品送纽勤生物科技(上海)有限公司,在Illumina Beadstration平台上根据公司标准流程进行猪全基因组50K SNP芯片(Illumina,美国)基因型判定。利用R语言GenABEL包中checkmarker对所有样本50K芯片扫描分型数据进行质量控制,剔除检出个体率低于90%、家系孟德尔错误率高于0.1、最小等位基因频率小于0.05且哈代-温伯格平衡显著性水平高于10-6的SNP,最终得到38790个SNP的有效基因型数据。
(4)全基因组关联(GWAS)分析
为了消除群体层化效应,本发明采用线性混合模型单点回归分析并结合R语言GenABEL软件包进行GWAS分析,分析模型中利用个体间基因组的相似度校正层化效应。采用Bonferrini法确定SNP与有效总乳头数性状关联程度的显著性阈值,基因组水平显著阈值为0.05除以有效SNP位点数量,即基因组显著水平阈值为1.29e-6,即0.05/38790(有效SNP数量);染色体水平显著阈值为1除以有效SNP位点数量,即染色体显著水平阈值为2.58e-5,即1/38790(有效SNP数量)。
GWAS分析结果如图1所示。从图1可知,在杜洛克中,在6号染色体中存在显著影响瘦肉率、眼肌面积、眼肌厚度的位点,最强关联的SNP为g.333T>C(P值分别为3.00e-10、6.05e-7、3.88e-6)。
(5)不同基因型与瘦肉率、眼肌面积、眼肌厚度表型的关联性分析
根据表1可知,分子标记的SNP位点g.333T>C(SEQ NO.1中第333位核苷酸,即对应于国际猪基因组11.1版本参考序列6号染色体上第47357966位T>C突变)与瘦肉率性状极显著相关(P<0.001),说明此分子标记显著影响猪的瘦肉率性状,可以通过对猪的此SNP位点的辅助选择,从而提高该群体的瘦肉率,进而加快育种进程。
根据表2可知,分子标记的SNP位点g.333T>C(SEQ NO.1中第333位核苷酸,即对应于国际猪基因组11.1版本参考序列6号染色体上第47357966位T>C突变)与眼肌面积性状极显著相关(P<0.001),说明此分子标记显著影响猪的眼肌面积性状,可以通过对猪的此SNP位点的辅助选择,从而提高该群体的眼肌面积,进而加快育种进程。
根据表3可知,分子标记的SNP位点g.333T>C(SEQ NO.1中第333位核苷酸,即对应于国际猪基因组11.1版本参考序列6号染色体上第47357966位T>C突变)与眼肌厚度性状极显著相关(P<0.001),说明此分子标记显著影响猪的眼肌厚度性状,可以通过对猪的此SNP位点的辅助选择,从而提高该群体的眼肌厚度,进而加快育种进程。
另外根据表1~3及图2可知,TT型、TC型比CC型的瘦肉率、眼肌面积、眼肌厚度大,并且三个性状间协同变化。对于廋肉率性状,TT型个体表型平均值比CC型个体表型平均值大0.62;对于眼肌面积性状,TT型个体表型平均值比CC型个体表型平均值大1.28。对于眼肌厚度性状,TT型个体表型平均值比CC型个体表型平均值大0.76。因此,在育种中逐步保留TT、TC基因型的种猪,以逐代提高该位点的等位基因T的频率,可以显著提高瘦肉率、眼肌面积和眼肌厚度,为养殖企业带来更多的经济效益。
表1分子标记的SNP位点g.333T>C与瘦肉率的相关性分析
表2分子标记的SNP位点g.333T>C与眼肌面积的相关性分析
表3分子标记的SNP位点g.333T>C与眼肌厚度的相关性分析
实施例2目的DNA序列扩增及测序
(1)引物设计
通过Ensembl网站(http://asia.ensembl.org/index.html)下载猪的6号染色体上SEQ ID NO:1的DNA序列。并利用引物设计软件primer premier 6.0设计引物。设计的引物的DNA序列如下所示:
P001-F:5’-ACTCTTGCCTCCGACTTCTC-3’,
P002-R:5’-AGACCTGGTGACATAGTTGATGA-3’;
(2)PCR扩增
10μL的反应体系中加入DNA模板1μL,双蒸水3.4μL,2×Tag PCR StanMix with Loading Dye 5μL,引物P001-F和P002-R各0.3μL。PCR反应条件为:94℃预变性5min后,94℃变性30s、55℃退火30s、72℃延伸45s,35个循环,最后72℃延伸5min。
(3)DNA序列测定
DNA序列测序鉴定:在深圳华大基因科技有限公司进行,基因片段测正反两个反应。将所测得的序列与NCBI基因组序列对比,得出对应SNP位点的突变。测序结果如下所示:
注:序列表中标注的M为突变位点,用标有下划线显示(括号中为突变碱基,为等位基因突变),在该序列的首尾加粗显示为设计引物序列位置。
实施例3分子标记的SNP位点g.333 T>C效应分析
根据表1~3可知,对于瘦肉率、眼肌面积和眼肌厚度,SNP位点g.333T>C优势等位基因型(TT)的效应比CC型表型平均值分别显著提高0.62、1.28和0.76。因此,通过分子标记辅助选择,逐步对群体内基因型为CC的猪进行淘汰,则能显著提高等位基因T的等位基因频率,提高群体的瘦肉率、眼肌面积、眼肌厚度,使猪生长及生产性能得到改善,可产出更多优质商品瘦肉型猪肉以满足人们对高品质猪肉的需求,从而带动了猪肉销量的增长,这将给企业带来巨大的经济收益。
本发明通过对SEQ ID NO:1序列中的第333个位碱基突变位点进行检测,初步进行其基因型与猪的瘦肉率、眼肌面积、眼肌厚度性状之间的关联分析的应用,为猪的分子标记辅助选择提供了一个新的分子标记。
上述实施例为本发明较佳的实施方式,但本发明的实施方式并不受上述实施例的限制,其他的任何未背离本发明的精神实质与原理下所作的改变、修饰、替代、组合、简化,均应为等效的置换方式,都包含在本发明的保护范围之内。
SEQUENCE LISTING
<110> 华南农业大学
<120> 猪6号染色体上与瘦肉率、眼肌面积、眼肌厚度相关的SNP分子标记与应用
<130> 1
<160> 3
<170> PatentIn version 3.3
<210> 1
<211> 422
<212> DNA
<213> Artificial
<220>
<223> 猪6号染色体上与瘦肉率、眼肌面积、眼肌厚度相关的SNP分子标记的核苷酸
序列
<400> 1
actcttgcct ccgacttctc accccttgct cccgtctctc ctttcctcct ctgctgatgc 60
ccgatcccat ccctcacagc cccctgcgtc tcaccagacc tttctctttg accttgatct 120
ccctgtgtca tccctgacct tcccgctttc accacctctt ctcagtcaca tccccacctc 180
ccaccctggg acatcatcct tctggcttcc caccctgggt cttccatgga ccacaccctc 240
cccgcaagtg ccctcacacc ttgacctctg accttgaccc ctaggtgctg gatgtcctgt 300
gttccctgtg tgtgtgcaat ggtgtggccg tgmgctccaa ccaagatctc attactgaga 360
acttgctccc tggccgcgag cttctgctgc agacaaacct catcaactat gtcaccaggt 420
ct 422
<210> 2
<211> 20
<212> DNA
<213> Artificial
<220>
<223> 引物P001-F
<400> 2
actcttgcct ccgacttctc 20
<210> 3
<211> 23
<212> DNA
<213> Artificial
<220>
<223> 引物P002-R
<400> 3
agacctggtg acatagttga tga 23
- 还没有人留言评论。精彩留言会获得点赞!