一种新型转基因植物标记基因构建及应用的制作方法
本发明属于植物生物技术领域,具体地说利用fmdv2a连接肽串联标记基因的方法,通过对串联的基因分别进行验证,以确定该植株为转基因阳性。
背景技术:
外源基因转化到植物需要通过不同阶段的筛选标记来确认,不同筛选标记容易获得真实的转基因植株,但是植物基因表达需要启动子和终止子配套,因此多个标记基因构建比较繁琐。在植物基因表达载体构建过程中,往往由于载体表达组件的缺少或多克隆位点酶切位点的稀少而使目的基因植物表达载体构建受到限制。为了在植物表达载体中使用多个标记基因,增加转基因植物的筛选可信度,本项目利用口蹄疫病毒(fmdv)2a/2b连接区分别将潮霉素抗性基因(hpt)与bar抗草胺膦抗性基因串联构成双筛选标记的融合基因,将潮霉素抗性基因(hpt)、bar抗草胺膦抗性基因以及绿色荧光蛋白基因(egfp)串联构成双筛选标记和报告基因融合的3基因系统。
目前多基因表达策略主要包括表达融合蛋白,构建多顺反子表达载体等。利用内部核糖体进入位点ires(internalribosomeentrysite)或fmdv2a连接两个或多个基因,可以在真核系统中实现基因的共表达。利用ires介导的多顺反子表达结构较大,其应用常受到载体容量的限制,当ires被置于相邻基因中间时有助于双顺反子蛋白的表达,但ires对位于其后的基因翻译起始能力相对较弱。在多顺反子蛋白的表达中可以用fmdv2a来代替ires。
fmdv2a蛋白约18个氨基酸残基,具有自我剪切活性,其共翻译剪切活性是在自己的c端切割fmdv多聚蛋白,通过核糖体跳跃使2a蛋白和它的下游蛋白发生不连续的翻译(donnelly,j.gen.virol,2001,82:1027-1041;j.gen.virol,2001,82:1013-1025),2a片段的氨基酸连接到上游蛋白c端,一个脯氨酸连接到下游蛋白的n端(hasegawa,stemcells,2006,24:2649-2660)。目前已在哺乳动物细胞、植物和酵母中观察到2a介导的对多聚蛋白的自我剪切作用。2a介导的相邻基因表达产物通过翻译跳跃来实现分离。
序列分析发现,fmdv2a的氨基酸序列高度保守,多肽序列中含有的基序为“-dxexnpgp-”,最后三个氨基酸(-npg-)则完全保守,并且2b蛋白n端第一个氨基酸pro也完全保守,该保守元件在2a起始剪切中起到关键作用。研究表明2a和2b总是在gly和pro之间切开,动力学和结构模型分析表明,gly和pro之间的肽键实际上并未形成(doronina,mol.cell.biol.,2008,28:4227-4239)。在翻译过程中,2a的高级结构对核糖体肽基转移酶中心造成空间排阻,使肽基(2a)-trna酯键无法形成。由于空间排阻使pro-trna氨基氮亲核进攻无法完成,代之则是肽基(2a)-trna酯键的水解作用,形成与2a的融合蛋白,同时核糖体能继续翻译下游蛋白2b,整个过程不需要任何蛋白酶参与(chinnasamyvirolj,2006,3:14)。可见,2a能起到类似蛋白水解酶的作用,在2a和2b位点将其顺式切开,fmdv2a独特的剪切机制决定了2a只有在真核翻译系统中有活性,而在原核翻译系统中无活性。fmdv基因表达产物中不存在未剪切的多聚蛋白前体,因此fmdv2a的天然剪切活力可达到100%,(szymczak,natbiotechnol,2004,22:589–594),fmdv2a对体外构建的多顺反子剪切效率也能达到85%-95%。
体外构建fmdv2a多顺反子的特点是:基因之间以2a序列连接,同时去除上游基因的终止密码子以形成一个长的开放读码框。翻译时多聚蛋白可在编码2a区域的c端被2a切割开,释放出融合了2a多肽尾巴的上游蛋白,以及完整的下游蛋白(刘必胜,生物工程学报,2007,23(5):765-769)。目前,fmdv2a多顺反子系统已被应用于腺病毒(adenovirus)、逆转录病毒(retrovirus)、慢病毒(lentivirus)、腺相关病毒(adeno-associatedvirus)等载体的构建(osborn,molther,2005,12:569–574)。
技术实现要素:
本发明利用fmdv2a连接肽串联多个筛选标记基因和报告基因。首先按照植物偏爱密码,我们将口蹄疫病毒(fmdv)含有2a蛋白和2b蛋白区llnfdllklagdvesnpgp19个氨基酸重新设计引物合成,合成的核苷酸序列为:ctgttgaatttcgatcttcttaagcttgctggtgatgttgaatccaacccaggtcca(seqidno.2所示)。
本发明通过连续延伸pcr,利用该合成序列将筛选标记基因潮霉素抗性基因(hpt)、草胺膦抗性基因bar和gfp报告基因片断串联hygfpbar。
本发明利用bsteii和bglii双酶切植物表达载体pcambia1301,切除其中的gus报告基因,通过dna末端平滑试剂盒处理后,连接获得植物表达载体pcambia1301-gus。xhoi酶切融合标记基因hygfpbar,通过t4dna连接酶将融合标记基因hygfpbar插入植物表达载体pcambia1301-gus,替换其中的潮霉素抗性基因,获得植物表达载体phygfpbar。
本发明利用电击法将质粒导入根癌农杆菌中。农杆菌介导方法将三价基因表达载体转化单子叶模式植物水稻(clough1998,植物学杂志)和双子叶模式植物拟南芥,通过检测,获得了转基因植株具有潮霉素和草胺膦抗性,转基因植株组织能表达荧光蛋白。
附图说明
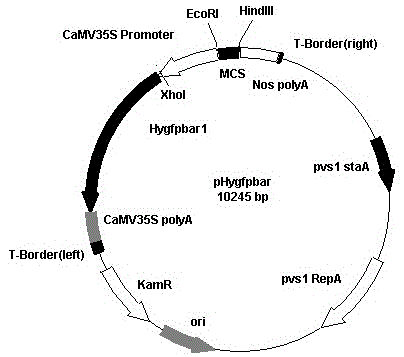
图1三价标记基因融合基因植物表达载体phygfpbar构建图谱。
图2转基因水稻愈伤组织的潮霉素抗性,水稻幼苗的草胺膦抗性及组织荧光检测。
图3转基因拟南芥种子潮霉素抗性、幼苗的草胺膦抗性及组织荧光检测。
本发明有益效果
该标记基因转化植物后,具有多重抗性,可以通过愈伤组织、苗期和成熟植株的检测,有利于加快转基因材料的纯合,同时该载体可以用于多基因表达载体构建。
具体实施方式
实施例1:标记基因hygfpbar构建
利用重叠延伸pcr串联潮霉素抗性基因(hpt)、绿色荧光蛋白基因egfp和草胺膦抗性基因bar。设计的引物为:hptz1:5’-ccgctcgagatgaaaaagcctgaactcaccg-3’(seqidno.3所示);hptf1:5’-acatcaccagcaagcttaagaagatcgaaattcaacagctatttctttgccctcggacg-3’(seqidno.4所示);gfpz:5’-cttaagcttgctggtgatgttgaatccaacccaggtccaatggtgagcaagggcgaggag-3’(seqidno.5所示);gfpf:5’-acatcaccagcaagcttaagaagatcgaaattcaacagtcagatctcggtgacgggcag-3’(seqidno.6所示);barz:5’-cttaagcttgctggtgatgttgaatccaacccaggtccaatgagcccagaacgacgcccg-3’(seqidno.7所示);barf:5’-ccgctcgagtcagatctcggtgacgggcag-3’(seqidno.8所示)。
在50µl反应体系中,利用引物hptz1和hptf1从pcambia1301扩增hpt基因;利用引物barz和barf从ppzp-rcs2-bar(novagen,madison,美国)中扩增bar基因;利用引物gfpz和gfpf从plegfp-c1(clontech公司,美国)中扩增egfp基因,回收上述3个扩增基因片段,然后利用引物hptz1和barf1将3个扩增片断通过重叠延伸方法串联,pcr扩增条件为:94℃预热1min;94℃,30s,50℃,30s,72℃,150s,使用的taqdna聚合酶为kodfxtaq酶(toyobo公司,日本),共25个循环。pcr产物进行1%琼脂糖胶回收,取10µl直接与平端克隆载体相连(大连宝生物公司)。4℃连接过夜,高效转化dh5α感受态中,获得阳性克隆,测序鉴定获得序列正确的新型标记基因hygfpbar。
实施例2:植物表达载体的构建
首先将植物表达载体pcambia1301利用bglii和bsteii双酶切,加入末端平滑试剂盒中的t4dna聚合酶(takara宝生物),然后加入5ut4dna连接酶,通过pcr鉴定载体gus基因的存在,筛选去除gus基因的载体作为新型标记基因表达载体。xhoi酶切pcambia1301-gus载体和hygfpbar片断,回收载体和片断以1:10以上的摩尔比例连接。利用pcr鉴定插入基因的方向,找到潮霉素抗性基因在启动子一侧的克隆进行进一步鉴定,构建成由camv35s启动子控制的新型标记基因植物表达载体phygfpbar。
实施例3:水稻和拟南芥转化
所用菌株为根癌农杆菌。质粒经电击法导入农杆菌中。挑取单菌到25mlyeb培养基(50mg/l利福平)培养过夜,取5ml菌液转接到100mlyeb培养基(50mg/l利福平),培养至od600=0.7-0.8,菌液冰上放置10分钟,5000rpm离心10min,4℃,收集菌体,加入100ml无菌双蒸水清洗两次。加入2ml10%甘油悬浮菌体,转到50ml离心管。5500rpm离心10min,4℃。收集菌体,加入500µl10%甘油悬浮菌体,转到1.5ml离心管。取70µl感受态细胞,分别加入1µl重组质粒phygfpbar。用去头的200µl枪头混匀,转到0.1cm电击杯中。电击参数:200ω,1.7kv,2.5f,电击后立即加入800µlsoc培养液。培养1小时后,取100µl涂抗性板筛选转化子,28℃培养。
水稻转化以n6培养基为基本培养基。去壳的种子,授粉后12-15天的幼胚,经表面消毒后接种到n6d2培养基中诱导愈伤组织(n6培养基,水解乳蛋白500mg/l,蔗糖30g/l,2,4-d2mg/l,植物凝胶2.5g/l,ph5.8);培养4-7天后取愈伤组织进行转化。农杆菌培养od600=0.8-1.0后离心5000g离心8分钟,去离子h2o清洗一次,等体积ms培养液悬浮侵染8分钟后,吸干放置在ms+naa1mg/l+ba2mg/l的培养基中,22度共培养3天。然后转入筛选培养基(加入头孢cb(500µg/ml)和潮霉素hyg(50µg/ml),转化后的愈伤组织在含有抗性培养基上培养3-4代,转入分化培养基中(2mg/lkt);幼芽长至2mm转移到生根培养基(1/2ms+0.5mg/liba)。以上培养基中分别加入500mg/l酶水解乳蛋白(ch),0-700mg/l谷氨酰胺或精氨酸,蔗糖30-80g/l,琼脂6g,ph5.8。继代周期为25d。将淡黄色的胚性愈伤组织转入分化培养基中,30d左右分化出芽。光照强度1500-2000lx,12-14h/d。
拟南芥转化采用蘸花法。含目的质粒的农杆菌菌株单菌落接菌在5毫升含对应抗生素的lb培养基中28℃培养2天。将5毫升菌液转到500毫升的液体lb培养基中28℃培养16-24小时(od=1.5-2.0)。室温下离心收集菌体,4000g离心10分钟。用等体积5%的新鲜蔗糖溶液悬浮。加入0.02%的silwet-77混匀后转移到烧杯中。每个菌株用300毫升转化,转2-3钵。隔7天后再转化1次。将拟南芥倒置后浸入菌液中10秒钟。莲座和花序都要侵染。侵染后将转化植株菌液空干3-5秒。用保鲜膜将转化植株圈好,平放16-24小时。转化后不要放置在高温和强光下。揭开保鲜膜,保持一定湿度,再生长1个月后收种子。
实施例4:转基因植物的验证
hyg抗性的表达:
作为新型筛选标记基因,hygfpbar具有潮霉素抗性,潮霉素抗性基因hpt是一种较理想的检测转基因植株及其后代的报告基因。水稻在抗性愈伤筛选过程中采用50mg/l潮霉素b作为筛选压,可以区别转基因和非转基因愈伤组织。拟南芥种子可以通过潮霉素筛选获得转基因植株。
另外利用水稻的潮霉素抗性检测叶片法,也可以检测转基因植株对潮霉素的抗性。剪取约2cm长的叶片放入含有检测溶液(1.0mg/l的6-苄氨基嘌呤(6-ba)和30mg/l的潮霉素b+1%silwet-l77)的平皿中,silwet-l77是一种高效有机硅表面活性剂,能够极大的降低水的表面张力(水的表面张力为72.4mn/m,0.1%的silwet-l77系列有机硅溶液的表面张力约为21mn/m),这使silwet-l77有机硅溶液可轻易湿润水稻的叶面,相对于传统助剂,显著提高了在靶标生物的覆盖面。在16h光照,8h黑暗,25℃条件下培养,定期观察叶片变色情况,以判断其抗性水平。到第5天时,在含潮霉素b的检测液中,对照组水稻叶片段均产生褐斑甚至枯死,而转基因水稻在含潮霉素的检测溶液中的叶片仍保持鲜绿色,形成鲜明对比。
bar除草剂抗性基因的表达:
转基因水稻种子(未脱壳),浸于蒸馏水中,置于25℃,16h光照、8h黑暗。七天后将萌发一致的水稻苗移栽到土里。22℃,16h光照、8h黑暗,待到水稻苗长出四个叶左右时,分别喷洒100-500mmol/l等不同浓度的草铵膦除草剂,五天后,观察发现,喷洒草铵膦浓度为100mmol/l、200mmol/l、300mmol/l的水稻苗没有发生变黄或者死亡的现象,有个别出现褐色斑点,但是对照组和转基因水稻对比不是很明显。喷洒浓度为500mmol/l的草铵膦的对照组水稻和转基因水稻植株均出现死亡现象,但是对照组死亡现象更严重。喷洒浓度为400mmol/l的草铵膦除草剂的对照组水稻发黄现象严重甚至死亡,而三种转基因水稻植株只有叶片出现个别发黄,大体生长状态较好。
转基因拟南芥草铵膦浓度为100mmol/l。
转基因gfp基因的表达:
用荧光显微镜分别观察1,3两个含有gfp标记基因的转基因水稻和拟南芥根尖的绿色荧光蛋白的表达。结果显示转基因植物表达了绿色荧光蛋白。
序列表
<110>上海市农业科学院
<120>一种新型转基因植物标记基因构建及应用
<130>2019
<160>8
<170>siposequencelisting1.0
<210>1
<211>2412
<212>dna
<213>artificialsequence
<400>1
atgaaaaagcctgaactcaccgcgacgtctgtcgagaagtttctgatcgaaaagttcgac60
agcgtctccgacctgatgcagctctcggagggcgaagaatctcgtgctttcagcttcgat120
gtaggagggcgtggatatgtcctgcgggtaaatagctgcgccgatggtttctacaaagat180
cgttatgtttatcggcactttgcatcggccgcgctcccgattccggaagtgcttgacatt240
ggggagtttagcgagagcctgacctattgcatctcccgccgtgcacagggtgtcacgttg300
caagacctgcctgaaaccgaactgcccgctgttctacaaccggtcgcggaggctatggat360
gcgatcgctgcggccgatcttagccagacgagcgggttcggcccattcggaccgcaagga420
atcggtcaatacactacatggcgtgatttcatatgcgcgattgctgatccccatgtgtat480
cactggcaaactgtgatggacgacaccgtcagtgcgtccgtcgcgcaggctctcgatgag540
ctgatgctttgggccgaggactgccccgaagtccggcacctcgtgcacgcggatttcggc600
tccaacaatgtcctgacggacaatggccgcataacagcggtcattgactggagcgaggcg660
atgttcggggattcccaatacgaggtcgccaacatcttcttctggaggccgtggttggct720
tgtatggagcagcagacgcgctacttcgagcggaggcatccggagcttgcaggatcgcca780
cgactccgggcgtatatgctccgcattggtcttgaccaactctatcagagcttggttgac840
ggcaatttcgatgatgcagcttgggcgcagggtcgatgcgacgcaatcgtccgatccgga900
gccgggactgtcgggcgtacacaaatcgcccgcagaagcgcggccgtctggaccgatggc960
tgtgtagaagtactcgccgatagtggaaaccgacgccccagcactcgtccgagggcaaag1020
aaatagctgttgaatttcgatcttcttaagcttgctggtgatgttgaatccaacccaggt1080
ccaatggtgagcaagggcgaggagctgttcaccggggtggtgcccatcctggtcgagctg1140
gacggcgacgtaaacggccacaagttcagcgtgtccggcgagggcgagggcgatgccacc1200
tacggcaagctgaccctgaagttcatctgcaccaccggcaagctgcccgtgccctggccc1260
accctcgtgaccaccctgacctacggcgtgcagtgcttcagccgctaccccgaccacatg1320
aagcagcacgacttcttcaagtccgccatgcccgaaggctacgtccaggagcgcaccatc1380
ttcttcaaggacgacggcaactacaagacccgcgccgaggtgaagttcgagggcgacacc1440
ctggtgaaccgcatcgagctgaagggcatcgacttcaaggaggacggcaacatcctgggg1500
cacaagctggagtacaactacaacagccacaacgtctatatcatggccgacaagcagaag1560
aacggcatcaaggtgaacttcaagatccgccacaacatcgaggacggcagcgtgcagctc1620
gccgaccactaccagcagaacacccccatcggcgacggccccgtgctgctgcccgacaac1680
cactacctgagcacccagtccgccctgagcaaagaccccaacgagaagcgcgatcacatg1740
gtcctgctggagttcgtgaccgccgccgggatcactctcggcatggacgagctgtacaag1800
taactgttgaatttcgatcttcttaagcttgctggtgatgttgaatccaacccaggtcca1860
atgagcccagaacgacgcccggccgacatccgccgtgccaccgaggcggacatgccggcg1920
gtctgcaccatcgtcaaccactacatcgagacaagcacggtcaacttccgtaccgagccg1980
caggaaccgcaggagtggacggacgacctcgtccgtctgcgggagcgctatccctggctc2040
gtcgccgaggtggacggcgaggtcgccggcatcgcctacgcgggcccctggaaggcacgc2100
aacgcctacgactggacggccgagtcgaccgtgtacgtctccccccgccaccagcggacg2160
ggactgggctccacgctctacacccacctgctgaagtccctggaggcacagggcttcaag2220
agcgtggtcgctgtcatcgggctgcccaacgacccgagcgtgcgcatgcacgaggcgctc2280
ggatatgccccccgcggcatgctgcgggcggccggcttcaagcacgggaactggcatgac2340
gtgggtttctggcagctggacttcagcctgccggtaccgccccgtccggtcctgcccgtc2400
accgagatctga2412
<210>2
<211>57
<212>dna
<213>artificialsequence
<400>2
ctgttgaatttcgatcttcttaagcttgctggtgatgttgaatccaacccaggtcca57
<210>3
<211>31
<212>dna
<213>artificialsequence
<400>3
ccgctcgagatgaaaaagcctgaactcaccg31
<210>4
<211>59
<212>dna
<213>artificialsequence
<400>4
acatcaccagcaagcttaagaagatcgaaattcaacagctatttctttgccctcggacg59
<210>5
<211>60
<212>dna
<213>artificialsequence
<400>5
cttaagcttgctggtgatgttgaatccaacccaggtccaatggtgagcaagggcgaggag60
<210>6
<211>59
<212>dna
<213>artificialsequence
<400>6
acatcaccagcaagcttaagaagatcgaaattcaacagtcagatctcggtgacgggcag59
<210>7
<211>60
<212>dna
<213>artificialsequence
<400>7
cttaagcttgctggtgatgttgaatccaacccaggtccaatgagcccagaacgacgcccg60
<210>8
<211>30
<212>dna
<213>artificialsequence
<400>8
ccgctcgagtcagatctcggtgacgggcag30
- 还没有人留言评论。精彩留言会获得点赞!