用于表达限制性内切酶ApaLI的甲基化保护菌株的筛选方法与流程
本发明涉及一种菌株的筛选方法,特别是一种用于表达限制性内切酶apali的甲基化保护菌株的筛选方法。
背景技术:
在生物体内有一类能将外来的dna切断的酶,它能够限制异源dna的侵入并使之失去活力,但对自身的dna却无损害作用,这样就可以起到保护细胞原有的遗传信息的作用。由于这种切割作用是在dna分子内部进行的,故名限制性内切酶(简称限制酶)。其发现促使dna重组技术的诞生从而极大推动现代分子生物学和基因工程的发展,是当代基因工程研究不可或缺的基础工具。
由于细菌体内存在“限制-修饰”现象,即自身的dna被甲基化酶修饰,从而避免所产生的限制酶对自身dna片段的切割,达到抵御外来噬菌体侵染的目的。因此在原核表达体系中单一的重组表达限制酶,会使宿主因dna被切割而死亡。而甲基转移酶在识别序列上的特异性修饰可防止限制性内切酶的切割。限制-修饰基因常紧密连锁,细菌通过调控两者的表达,使自身dna首先受到甲基化保护,保护完成后再表达限制酶,而入侵的异源dna并没有受到保护因而被限制性内切酶降解。即胞内相关的甲基转移酶对与其配对限制性内切酶识别的相同特定序列进行甲基化修饰,并使修饰的dna具有抵抗其限制性内切酶的切割的特性,保护宿主dna而降解未被甲基化的外源dna(geofferyg.w.,nucleicacidsres,2539-2564.1991)。
关于限制-修饰系统在原核细胞中表达的研究表明,系统的建立和维持是以甲基转移酶的保护作用为前提。甲基化修饰是蛋白质翻译后修饰的重要形式之一,参与了原核生物和真核生物的多种细胞进程。基于dna分子的结构组成和特性,自然界中主要存在三类dna甲基转移酶,分别是c5胞嘧啶甲基酶、n4胞嘧啶甲基酶和n6腺嘌呤甲基酶。其中,n4胞嘧啶和n6腺嘌呤甲基酶属于氨基-甲基转移酶(maloneetal.j.mol.biol.253:618-632.1995)。dna在甲基转移酶作用下,催化底物s-腺苷甲硫氨酸,转移一个甲基至胞嘧啶的c5位置,生成5-甲基胞嘧啶的过程。当一个有限制酶识别位点的dna被甲基转移酶修饰后,这个dna分子就会具有抵抗相应限制酶的酶切的特性。即通常情况下,如果dna识别序列相同(或识别序列包含在内)并且甲基化酶修饰位点和方式也一致,那么这种经过甲基转移酶特异性甲基化修饰的dna就具有可以抵抗相似识别序列的限制性内切酶切割的作用,从而保护宿主细胞的dna免受破坏。例如alui的识别序列是5’agct3’,包含在saci的识别序列之中(5’gagctc3’),而且其甲基化位点和修饰方式一致(c5胞嘧啶甲基酶),因而可以通过在宿主菌内表达m.alui来抵抗限制酶saci对dna的酶切。
随着限制修饰现象被发现,科学家wernerarben,danielnathans和hamiltonsmith发现了限制性内切酶,阐明了限制性内切酶在分子基因问题上的作用,之后,越来越多的限制性内切酶被发现、提纯并获得应用。关于限制性内切酶的研究,多年来着重于它们作为工具酶的实用价值,包括新酶的发现与筛选、高产工程菌株的构建、酶纯化技术与程序的优化改进等。其中apali是一种应用较为广泛的限制性内切酶,可识别六个碱基的核酸序列(5’g^tgcac3’),不仅可作为普通的限制性内切酶应用于分子克隆,在生物领域的其他方向也具有重要的应用价值及研究潜力。但由于表达量低、分离纯化程序繁琐、蛋白得率低等问题,目前市售的apali限制酶不仅价格高,而且也限制了它的广泛应用及深入研究。同时,以上提及的一系列问题也限制了其他很多应用广泛的限制性内切酶的深入研究。
限制性内切酶具有能够切割dna的特性,一旦表达将切割宿主细胞dna,造成宿主细胞的损伤甚至死亡。因此,必须对宿主细胞的dna进行特异性的甲基化修饰后才能实现其重组表达。现有技术一的方案中,利用了apali对应的天然特异性甲基转移酶m.apali进行宿主dna的保护。具体步骤包括:提取天然来源菌株acetobacterpasteurianusmogenes染色体dna并制备dna文库;利用m.apali保护dna不被apali切割的特性,从文库中筛选出m.apali基因;将带有m.apali基因的质粒转化进入表达宿主工程菌株中。在获得dna受到甲基化保护的表达宿主工程菌株后,将apali限制酶基因构建于表达载体,转化进入上述菌株,诱导apali的重组表达。表达后的纯化步骤涉及亲和层析、离子交换层析、疏水层析、反相层析和凝胶分子排阻层析等纯化过程。其缺点在于:对宿主细胞dna的甲基化保护在通过繁琐的dna文库构建和筛选后,所获得的甲基化保护菌株只能特异性保护dna免于apali限制酶的切割,少有能用于其他限制酶的表达,应用范围极为狭窄。
技术实现要素:
本发明所要解决的技术问题是针对现有技术的不足,提供一种可以用于表达限制性内切酶apali的新型甲基化保护宿主菌株。同时依据限制性内切酶对dna切割需要首先对识别序列进行识别的原理,获得对同一类具有类似识别序列的限制性内切酶进行甲基化保护的菌株,即一种甲基化保护菌株可进行多种限制性内切酶表达。
本发明所要解决的技术问题是通过以下的技术方案来实现的。本发明是
一种用于表达限制性内切酶apali的甲基化保护菌株的筛选方法,其特点是,
(1)甲基转移酶m.cviri的重组表达菌株的建立
a、甲基转移酶m.cviri基因的获取
甲基转移酶m.cviri基因的获得采用pcr扩增、基因合成或者提取染色体dna并建立文库方法,从菌株chlorellaviruss的基因组中使用甲基转移酶m.cviri基因的特异性引物获得了甲基转移酶m.cviri的基因;
b、构建甲基转移酶m.cviri的重组表达菌株
将m.cviri基因序列以酶切-连接的方式插入低拷贝表达载体pacyc184质粒中,连接产物转化进入克隆菌株dh5α并涂布于氯霉素抗性平板进行培养;
c、甲基化保护菌株的筛选
对转化后菌株进行甲基化保护程度的筛选;
d、限制性内切酶apali基因的获取
限制酶apali基因的获得采用pcr扩增、基因合成或者提取染色体dna并建立文库的方法,从菌株acetobacterpasteurianusmogenes的基因组中使用限制酶apali基因的特异性引物获得限制酶apali的基因;
e、构建限制性内切酶apali的重组表达菌株
使用阿拉伯糖操纵子表达系统,将apali基因序列以酶切-连接的方式插入pbad表达质粒中,连接产物转化进入dh5α表达菌株并涂布于氨苄霉素抗性平板进行培养;通过测序,确认重组质粒的构建成功后转化进入r.apali特异性保护菌株[pacyc184-m.cviri,er2566],构建限制酶apali的重组表达菌株;
(2)限制酶apali的重组表达
将测序正确的重组表达质粒pbad-r.apali转化感受态细胞[pacyc184-m.cviri,er2566],涂布于含氨苄霉素和氯霉素抗生素的平板上筛选培养,获得限制酶apali的重组表达菌株;该菌株经扩大培养后,以阿拉伯糖诱导,获得限制酶apali的重组蛋白,并确认细菌蛋白粗提液具有限制酶apali的活性。
本发明所要解决的技术问题还可以通过以下的技术方案来进一步实现。以上所述的一种用于表达限制性内切酶apali的甲基化保护菌株的筛选方法,其特点是,步骤(1)中,从菌株chlorellaviruss的基因组中使用甲基转移酶m.cviri基因的特异性引物pcr扩增获得了甲基转移酶m.cviri的基因;获得甲基转移酶m.cviri基因的方法如下:
菌株chlorellaviruss平板培养菌落以去离子水重悬后,95℃温育10min,作为pcr模板;pcr扩增的引物序列基于甲基转移酶m.cviri基因的上游引物5’-端序列及下游引物3’-端反向互补序列进行设计合成;
合成的引物序列如下所示:
正向引物:
反向引物:
这一对引物用于特异性扩增m.cviri基因,同时引入ndei与xhoi的酶切位点,方便后续酶切-连接以及筛选实验;pcr扩增反应使用monhi-fidnapolymerase,按照标准pcr反应流程进行,获得了与理论值大小一致的m.cviri基因片段序列1;上述pcr扩增产物通过酶切-连接的方式与pacyc184载体形成重组表达质粒。
本发明所要解决的技术问题还可以通过以下的技术方案来进一步实现。以上所述的一种用于表达限制性内切酶apali的甲基化保护菌株的筛选方法,其特点是,步骤(1)中,步骤(1)中,制备m.cviri的pacyc184表达载体按照以下流程进行:
pacyc184质粒以flashcuttmndei及flashcuttmxhoi限制酶双酶切后,再以monclonetmfastap进行去磷酸化处理,酶切产物进行琼脂糖凝胶电泳,并割胶回收线性化的pacyc184载体片段,溶于tebuffer中;
使用monhi-fidnapolymerase对m.cviri基因进行pcr扩增,对获得的pcr产物进行琼脂糖凝胶电泳,确认获得特异性扩增片段后,用flashcuttmndei及flashcuttmxhoi限制酶双酶切后与线性化的pacyc184载体片段连接,按照摩尔质量比1:3的浓度,经monclonetmfastt4dnaligase于22℃下反应1h后使用化学转化法转化进入dh5α感受态细胞中;在氯霉素筛选条件下进行细胞培养,获得了m.cviri的重组表达质粒pacyc184-m.cviri;质粒pacyc184-m.cviri序列的正确性经过测序确认。
本发明所要解决的技术问题还可以通过以下的技术方案来进一步实现。以上所述的一种用于表达限制性内切酶apali的甲基化保护菌株的筛选方法,其特点是,步骤(1)中,m.cviri保护菌株的筛选按照以下流程进行:将测序正确的质粒pacyc184-m.cviri通过化学转化法转化进入大肠杆菌菌株er2566,在氯霉素筛选条件下进行细胞培养;挑选单克隆,在含有氯霉素抗生素的琼脂平板上划线,并进行相应编号后于37℃,200rpm培养16h,收取菌液将质粒重新提出;在10μl反应体系中,取0.2μl限制性内切酶apali,在flashone缓冲条件下,与100ng重组质粒pacyc184-m.cviri在37℃温育1h,然后使用0.8%琼脂糖凝胶电泳检测底物被apali消化的情况;确认该重组质粒在宿主菌体内受到m.cviri甲基转移酶的完全保护,默认宿主菌基因组dna同样可受到pacyc184-m.cviri所表达的m.cviri甲基转移酶保护;经再次验证保护性后将平板上相应编号的菌株使用cacl2处理,制备成感受态细胞[pacyc184-m.cviri,er2566]。
本发明所要解决的技术问题还可以通过以下的技术方案来进一步实现。以上所述的一种用于表达限制性内切酶apali的甲基化保护菌株的筛选方法,其特点是,步骤(1)中,限制性内切酶apali基因的pcr扩增方法如下:
菌株acetobacterpasteurianusmogenes平板培养菌落以去离子水重悬后,95℃温育10min,作为pcr模板;pcr扩增的引物序列基于限制酶apali基因的上游引物5’-端序列及下游引物3’-端反向互补序列;
合成的引物序列如下所示:
正向引物:
反向引物:
这一对引物用于特异性扩增r.apali基因,并引入ndei、hindiii酶切位点,方便后续酶切-连接;pcr扩增反应使用monhi-fidnapolymerase,按照标准pcr反应流程进行,获得了与理论值大小一致的r.apali基因片段序列2;上述pcr扩增产物通过酶切-连接的方式与pbad载体形成重组表达质粒。
本发明所要解决的技术问题还可以通过以下的技术方案来进一步实现。以上所述的一种用于表达限制性内切酶apali的甲基化保护菌株的筛选方法,其特点是,步骤(1)中,制备apali的pbad表达载体按照以下流程进行:
pbad质粒以flashcuttmndei及flashcuttmhindiii限制酶双酶切后,再以monclonetmfastap进行去磷酸化处理;酶切产物进行琼脂糖凝胶电泳,并割胶回收线性化的pbad载体片段,溶于tebuffer中;
使用monhi-fidnapolymerase对r.apali基因进行pcr扩增,对获得的pcr产物进行琼脂糖凝胶电泳,确认获得特异性扩增片段后,用flashcuttmndei及flashcuttmhindiii限制酶双酶切后与线性化的pbad载体片段连接,按照摩尔质量比1:3的浓度,经monclonetmfastt4dnaligase于22℃下反应1h后使用化学转化法转化进入dh5α感受态细胞中;在氨苄青霉素抗性筛选条件下进行细胞培养,获得了r.apali的重组表达质粒pbad-r.apali;质粒pbad-r.apali序列的正确性经过测序确认。
本发明所要解决的技术问题还可以通过以下的技术方案来进一步实现。以上所述的一种用于表达限制性内切酶apali的甲基化保护菌株的筛选方法,其特点是,步骤(2)中,限制酶apali的重组表达方法如下:将测序正确的重组表达质粒pbad-r.apali转化[pacyc184-m.cviri,er2566]感受态细胞,涂布于含氨苄青霉素和氯霉素抗生素的平板上筛选培养,获得限制酶apali的重组表达菌株;在lb培养基中接种限制酶apali的重组表达菌株后,在37℃摇床以200rpm培养至od0.8,然后加入诱导剂阿拉伯糖,终浓度0.2%,并在37℃摇床以200rpm培养16h。
本发明所要解决的技术问题还可以通过以下的技术方案来进一步实现。以上所述的一种用于表达限制性内切酶apali的甲基化保护菌株的筛选方法,其特点是,步骤(2)中,限制性内切酶apali的活力检测方法如下:对经纯化后得到的限制性内切酶apali进行梯度稀释后,在1xflashonetmbuffer缓冲条件下、20μl反应体系中,与底物λdna在37℃温育15min,然后以1%琼脂糖凝胶电泳检测底物的酶切情况;限制性内切酶apali的酶活定义为:37℃下,在20μl反应体系中,1μl能够在15min内完全消化1μgλdna。
本发明方法为克隆表达重组限制性内切酶提供了一种高效可行的新方法和思路,成功制备重组限制性内切酶apali也为限制性内切酶生产工艺的提升提供了有价值的借鉴。与现有技术相比,本发明技术方案带来的有益效果是:本发明方法实现了限制性内切酶apali的高效重组表达;利用阿拉伯糖操纵子表达系统严苛控制限制性内切酶apali的本底表达水平;抵抗限制性内切酶apali对宿主基因组dna的酶切,相对无甲基化保护的表达系统,可稳定且高效进行限制酶apali的重组表达;筛选出的m.cviri甲基化保护菌株同样可应用于其他具有类似识别位点的限制酶的重组表达,比如cviri,hindiii,pvuii。
附图说明
图1为pacyc184-m.alui的载体图谱;
图2为pbad-r.apali的载体图谱;
图3为限制酶apali甲基化保护菌株筛选,图3中:
泳道1:apali酶切质粒puc19;泳道2:对照质粒puc19;泳道3:apali酶切质粒pacyc184-m.cviri;泳道4:对照质粒pacyc184-m.cviri
图4为apali的活力检测电泳图,图4中:
泳道11为mark,泳道10为λdna(hindiiidigest);泳道1为对标品酶切图;泳道2-9为分别是将apali原液进行不同梯度稀释的特异性酶切图,最终活力定为32u/ul。
2:稀释4倍;3:稀释8倍;4:稀释16倍;5:稀释32倍;6:稀释64倍;7:稀释128倍;8;稀释256倍;9:稀释512倍。
具体实施方式
以下参照附图,进一步描述本发明的具体技术方案,以便于本领域的技术人员进一步地理解本发明,而不构成对其权利的限制。
1、材料
1.1菌株与质粒
chlorellaviruss、acetobacterpasteurianusmogenes
菌株均来自美国菌种保藏中心(atcc);
dh5α购自北京全式金生物技术有限公司;
er2566购自赛默飞世尔科技有限公司(thermofisherscientific);
pbad、pacyc184、puc19购自淼灵生物质粒平台;
酶活检测底物λdna(hindiiidigest)购自赛默飞世尔科技有限公司(thermofisherscientific);
1.2仪器与试剂
jn-02c低温超高压连续流细胞破碎仪:购自广州聚能纳米生物科技股份有限公司;
蛋白分子量标准:page-masterproteinstandardplus购自金斯瑞生物科技有限公司
限制性内切酶flashcuttmndei,flashcuttmhindiii,flashcuttmxhoi(购自莫纳生物科技有限公司);apali购自newenglandbiolabs(neb)
monhi-fidnapolymerase(购自莫纳生物科技有限公司);
monclonetmfastt4dnaligase(购自莫纳生物科技有限公司);
monclonetmfastap(购自莫纳生物科技有限公司);
10×flashonetmbuffer购自莫纳生物科技有限公司。
2.2甲基转移酶m.cviri的重组表达菌株的建立
本发明按照下文描述的方法建立了甲基转移酶m.cviri的重组表达菌株。
2.2.1甲基转移酶m.cviri基因的获取
来源于菌株chlorellaviruss的甲基转移酶m.cviri的基因序列已公开(genebank:aaa42900.110.1)。甲基转移酶m.cviri基因的获得可以采用pcr扩增、基因合成、提取染色体dna并建立文库等行业内公知的技术。本发明本发明采用pcr扩增的方法,从菌株chlorellaviruss的基因组中使用甲基转移酶m.cviri基因的特异性引物进行pcr扩增获得了甲基转移酶m.cviri的基因。基因合成、提取染色体dna并建立文库等其它技术也能达到同样的目的。
实验1:获得甲基转移酶m.cviri基因
菌株chlorellaviruss平板培养菌落以去离子水重悬后,95℃温育10min,作为pcr模板。pcr扩增的引物序列基于甲基转移酶m.cviri基因的5’-端序列(上游引物)及3’-端反向互补序列(下游引物)进行设计合成。
合成的引物序列如下所示:
正向引物:
反向引物:
这一对引物可特异性扩增m.cviri基因,同时引入ndei与xhoi的酶切位点,方便后续酶切-连接以及筛选实验。pcr扩增反应使用monhi-fidnapolymerase,按照标准pcr反应流程进行,获得了与理论值大小一致的m.cviri基因片段(附件:序列1)。上述pcr扩增产物通过酶切-连接的方式与pacyc184载体形成重组表达质粒。2.2.2构建甲基转移酶m.cviri的重组表达菌株
m.cviri的识别序列为5’tgca3’,对第3位胞嘧啶进行甲基化修饰。apali对dna特异性识别的序列为5’gtgcac3’,切割位点在tc碱基间。m.cviri的识别序列包含在sfli,xcpi,halii,csti,cfli,apali的识别序列中,当m.cviri对序列5’tgca3’进行甲基化修饰后,有可能抵抗cviri,hindiii,pvuii,apali对该甲基化修饰序列的切割。本发明中利用了m.cviri对5’tgca3’中碱基a的特异性甲基化修饰,同样可达到对宿主基因组dna进行甲基化保护的目的,避免表达apali后致死宿主。但过高的甲基化酶的表达和修饰对于宿主同样具有致死效果,故而本发明将m.cviri基因序列以酶切-连接的方式插入低拷贝表达载体pacyc184质粒中,连接产物转化进入克隆菌株dh5α并涂布于氯霉素抗性平板进行培养。
实验2:构建甲基转移酶m.cviri的重组表达菌株
制备m.cviri的pacyc184表达载体按照以下流程进行。pacyc184质粒以flashcuttmndei及flashcuttmxhoi限制酶双酶切后,再以碱性磷酸酶monclonetmfastap进行去磷酸化处理。酶切产物进行琼脂糖凝胶电泳,并割胶回收线性化的pacyc184载体片段,溶于tebuffer中。
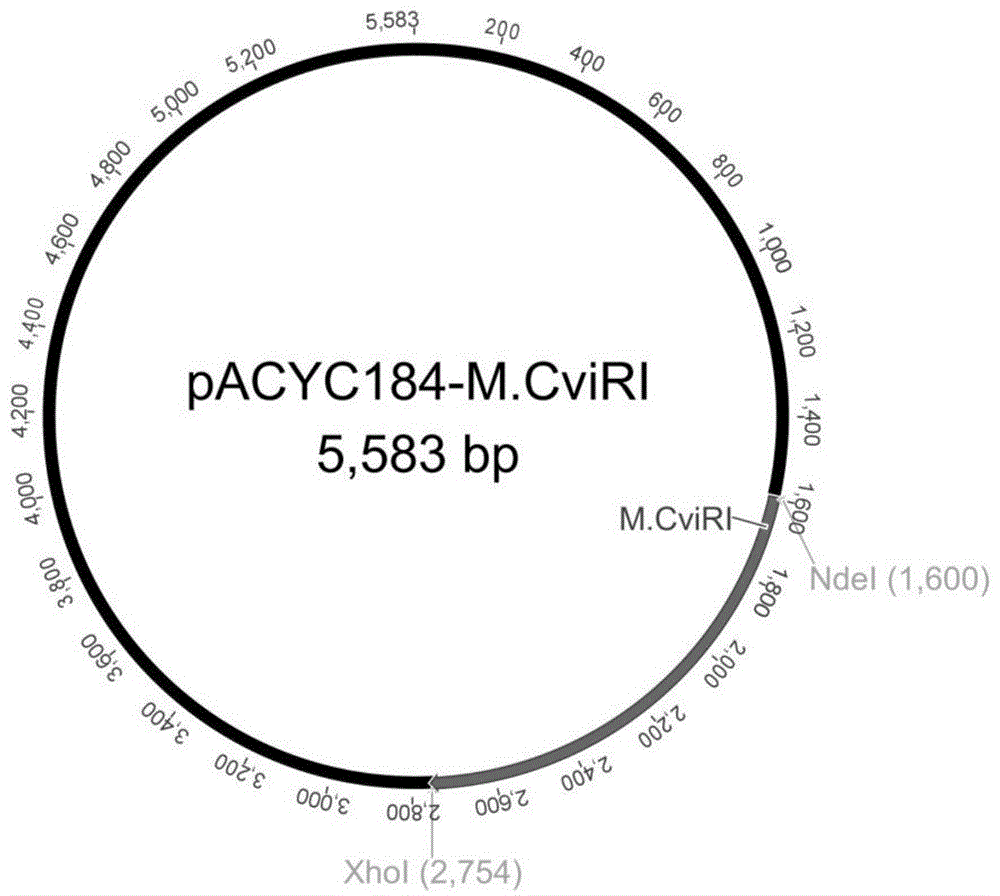
使用monhi-fidnapolymerase对m.cviri基因进行pcr扩增,对获得的pcr产物进行琼脂糖凝胶电泳,确认获得特异性扩增片段后,用flashcuttmndei及flashcuttmxhoi限制酶双酶切后与线性化的pacyc184载体片段连接,按照摩尔质量比1:3的浓度,经monclonetmfastt4dnaligase于22℃下反应1h后使用化学转化法转化进入dh5α感受态细胞中。在氯霉素筛选条件下进行细胞培养,获得了m.cviri的重组表达质粒pacyc184-m.cviri(参照图1)。质粒pacyc184-m.cviri序列的正确性经过测序确认(金唯智生物科技有限公司)。
2.2.3甲基化保护菌株的筛选
本发明中使用的载体pacyc184为低拷贝质粒,因而构建其上的m.cviri基因处于低水平表达状态。为了后续过程中限制性内切酶的高水平表达不致死宿主,需要对转化后菌株进行甲基化保护程度的筛选。
实验3:
m.cviri保护菌株的筛选按照以下流程进行:将测序正确的质粒pacyc184-m.cviri通过化学转化法转化进入大肠杆菌菌株er2566,在氯霉素筛选条件下进行细胞培养。挑选单克隆,在含有35μg/ml氯霉素抗生素的琼脂平板上划线,并进行相应编号后于37℃,200rpm培养16h,收取菌液将质粒重新提出。在10μl反应体系中,取0.2μl限制性内切酶apali,在
2.2.4限制性内切酶apali基因的获取
来源于菌株acetobacterpasteurianusmogenes的限制性内切酶apali的基因序列已公开(genebank:af_027867.1)。限制酶apali基因的获得可以采用pcr扩增、基因合成、提取染色体dna并建立文库等行业内公知的技术。本发明采用pcr扩增的方法,从菌株acetobacterpasteurianusmogenes的基因组中使用限制酶apali基因的特异性引物进行pcr扩增获得了限制酶apali的基因。基因合成、提取染色体dna并建立文库等其它技术也能达到同样的目的。
实验4:限制性内切酶apali基因的pcr扩增
菌株acetobacterpasteurianusmogenes平板培养菌落以去离子水重悬后,95℃温育10min,作为pcr模板。pcr扩增的引物序列基于限制酶apali基因的5’-端序列(上游引物)及3’-端反向互补序列(下游引物)。这一对引物可特异性扩增限制酶apali的基因。合成的引物序列如下所示:
正向引物:
反向引物:
这一对引物可特异性扩增r.apali基因,并引入ndei、hindiii酶切位点,方便后续酶切-连接。pcr扩增反应使用了monhi-fidnapolymerase,并按照标准pcr反应流程进行,获得了与理论值大小一致的r.apali基因片段(附件:序列2)。上述pcr扩增产物通过酶切-连接的方式与pbad载体形成重组表达质粒。
2.2.5构建限制性内切酶apali的重组表达菌株
本发明中使用乳糖操纵子表达系统,将apali基因序列以酶切-连接的方式插入pbad表达质粒中,连接产物转化进入dh5α表达菌株并涂布于氨苄霉素抗性平板进行培养。通过测序,确认重组质粒的构建成功后转化进入r.apali特异性保护菌株[pacyc184-m.cviri,er2566],构建限制酶apali的重组表达菌株。
实验5:构建限制性内切酶r.apali的重组表达菌株
制备apali的pbad表达载体按照以下流程进行:pbad质粒以flashcuttmndei及flashcuttmhindiii限制酶双酶切后,再以monclonetmfastap进行去磷酸化处理。酶切产物进行琼脂糖凝胶电泳,并割胶回收线性化的pbad载体片段,溶于tebuffer中。
使用monhi-fidnapolymerase(莫纳生物科技有限公司)对r.apali基因进行pcr扩增,对获得的pcr产物进行琼脂糖凝胶电泳,确认获得特异性扩增片段后,用ndei及hindiii限制酶(莫纳生物科技有限公司)双酶切后与线性化的pbad载体片段连接,按照摩尔质量比1:3的浓度,经monclonetmfastt4dnaligase于22℃下反应1h后使用化学转化法转化进入dh5α感受态细胞中。在氨苄霉素抗性筛选条件下进行细胞培养,获得了r.apali的重组表达质粒pet28b-r.apali(参照图2)。质粒pbad-r.apali序列的正确性经过测序确认(金唯智生物科技有限公司)。
2.3限制酶apali的重组表达
将测序正确的重组表达质粒pbad-r.apali转化感受态细胞[pacyc184-m.cviri,er2566],涂布于含氨苄霉素和氯霉素抗生素的平板上筛选培养,获得限制酶apali的重组表达菌株。该菌株经扩大培养后,以阿拉伯糖诱导,获得限制酶apali的重组蛋白,并确认细菌蛋白粗提液具有限制酶apali的活性。
实验6:限制酶apali的重组表达
将测序正确的重组表达质粒pbad-r.apali转化[pacyc184-m.apali,er2566]感受态细胞,涂布于含氨苄霉素和氯霉素抗生素的平板上筛选培养,获得限制酶apali的重组表达菌株。在lb培养基中接种限制酶apali的重组表达菌株后,在37℃摇床以200rpm培养至od0.8,然后加入诱导剂阿拉伯糖,终浓度0.2%,并在16℃摇床以200rpm培养16h。
2.4限制性内切酶apali的活力检测限制性内切酶apali的酶活定义为:37℃下,在20μl反应体系中,1μl能够在15min内完全消化1μgλdna(hindiiidigest)。本发明对经纯化后得到的限制酶apali进行梯度稀释后,在1xflashonetmbuffer(莫纳生物科技有限公司)缓冲条件下、20μl反应体系中,与底物λdna(hindiiidigest)在37℃温育15min,然后以1%琼脂糖凝胶电泳检测底物的酶切情况(参照图4)。经测试,纯化获得的限制酶apali具有良好的酶切活力,比活力约为900,000u/mg的诱导菌液。
序列表
<110>莫纳(连云港)生物科技有限公司
江苏海洋大学
<120>用于表达限制性内切酶apali的甲基化保护菌株的筛选方法
<160>6
<170>siposequencelisting1.0
<210>1
<211>379
<212>prt
<213>人工序列(artificialsequence)
<400>1
metlysleuphegluasplysservalgluphehislysargleuser
151015
lyslysgluargseraspglyglyvalphephethrprolysaspile
202530
argaspilevalpheglugluleuglyaspphegluprothrasnile
354045
leugluprothrcysglythrglyglupheileseraspcysarglys
505560
valtyrlysasnserargileileglyvalgluileaspproargser
65707580
alagluleualaargaspglyserlysasngluileilevalhisasp
859095
phemetthrtrpaspthraspglulyspheaspleuileileglyasn
100105110
proprotyrphethrargprothrglyphelyshisaspproserval
115120125
vallyscysargserasnilecysilegluvalleuhislyscysile
130135140
thrarghisleualaaspasnglymetleualametvalleuproval
145150155160
serileleuasnserlysphetyrthrprothrileaspleuilethr
165170175
aspthrmetaspvalvalseralaargalailelyslysasnasnphe
180185190
metglythrasnvalargvalmetvalpheileilearglysargthr
195200205
proglyphevalserlystyrthrphelysthrserleuglylysval
210215220
ileileasnproaspglygluargleuglyserilevalserglylys
225230235240
lysthrileglyserleuasnvalasnileserpheglyvalthrleu
245250255
alaservallysglutyrphevalasplyssercysserglyserphe
260265270
proleuilecystyrasnasnilealalyslysglyaspleuleuphe
275280285
valserasplystyrserlyslysargpheasnglyargalaileleu
290295300
ileproargglytyralahisglyasptyrserpheasnpheileasp
305310315320
tyrthrasnasptyrpheileilegluasnhisvalilealailethr
325330335
glygluaspcysvalleuaspileilealalysserphealaasphis
340345350
argthrarggluphecysargleuleucysserserglyaspileser
355360365
lysasptyrvallysgluileprovalphegly
370375
<210>2
<211>375
<212>prt
<213>人工序列(artificialsequence)
<400>2
metthrthrargglnargleuseralagluargserglnglnleuthr
151015
argleuleuthrilethrlysthralaasnmetargalaleumetglu
202530
alasergluleualalysvalilealaleuvalalavalaspilegly
354045
lysseraspglumetalaargalapheprovalleutrpprolysile
505560
serproglnglnglutyrtyralathralavalasptrpphethrasn
65707580
proaspgluthrvalthrserpheaspvalvalaspmetleuaspala
859095
glythrserleuaspglnaspphemetthrtyrleulyscysleuthr
100105110
gluleuhislysargargarglystyrglyleuileleuglnarggln
115120125
proleuprothrmetvalglnvalserproargalaleumetglutyr
130135140
glyproasppheproproglualaleualasertrpleuthrtrparg
145150155160
lysphephetyraspleuaspasnargseralaglngluthrglytyr
165170175
leuphegluproileleualaalaalaileglyglyglualalysser
180185190
alaarggluargvalvalargargthraspaspprothrlysglyarg
195200205
glnvalaspcystrplysvalleuproaspglythrproleualatyr
210215220
gluleulysleuargvalthrilealaalaserglyglnglyargphe
225230235240
glyglugluleuserphealaargaspcysserserserglyalalys
245250255
proileleuvalvalleuaspprothrgluasnasplysleuthrgly
260265270
leuglnalaalatyrarggluvalglyglyalaalatyrvalglyasp
275280285
alaalatrpalahisleugluaspglualaglyalathrmetalaser
290295300
pheilegluargtyrvalargvalprovalalaservalserserphe
305310315320
gluargvalilegluglyaspalathrlysargserleuileleugln
325330335
aspleuglnalaargleuaspglyasngluleuthrileserleugly
340345350
glyhisglnargleuvalgluarghisgluaspglnserleualaala
355360365
aspglyaspaspaspserglu
370375
<210>3
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>3
aaacagaccatgcatatggtgcac24
<210>4
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>4
gtgcacctcgagcataaagg20
<210>5
<211>40
<212>dna
<213>人工序列(artificialsequence)
<400>5
ggctaacaggaggaattacatatgaccacccgtcaacgtc40
<210>6
<211>39
<212>dna
<213>人工序列(artificialsequence)
<400>6
gatgacgatagcgaataagcttggctgttttggcggatg39
- 还没有人留言评论。精彩留言会获得点赞!