一种生产亚精胺的工程菌
本发明涉及一种生产亚精胺的工程,属于生物工程
技术领域:
。
背景技术:
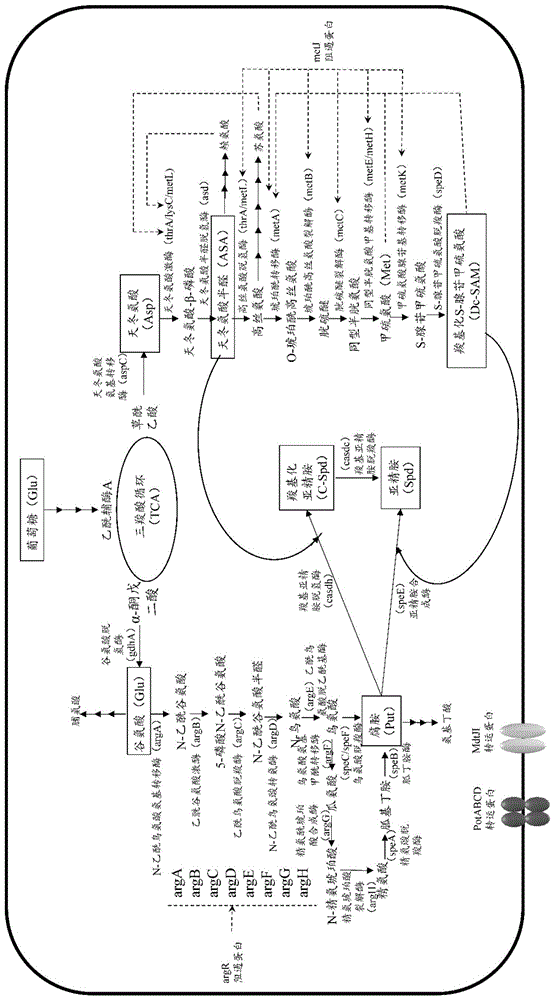
:亚精胺(spermidine),线性分子式为nh2(ch2)3nh(ch2)4nh2,广泛存在于微生物、植物和动物体内,是一种重要的生理活性物质。亚精胺具有延长动物寿命的功效,并抵消与年龄相关的疾病,如心血管疾病,神经退行性疾病和癌症等。如图1所示,生物体中主要有两条途径合成亚精胺:(1)经由羧基化腺苷甲硫氨酸和腐胺在亚精胺合成酶的作用下直接合成亚精胺,羧基化腺苷甲硫氨酸可由甲硫氨酸(met),经过相关酶的腺苷化和脱羧化催化得到,该途径在动物、植物和微生物体内,是比较常见的传统亚精胺合成的途径;(2)经由天冬氨酸-β-半醛和腐胺在羧基亚精胺脱氢酶和脱羧酶的催化下合成亚精胺,天冬氨酸-β-半醛可经常见氨基酸如天冬氨酸(asp)经相关酶的磷酸化和脱氢化催化合成,该途径为新发现的替代合成途径,主要存在于一些细菌中,包括重要的人类病原体,肠道菌群等。在大肠杆菌中仅存在途径1。这些途径中的酶受到高度调控,动植物体的亚精胺含量较低,目前也未发现可以大量生产亚精胺的微生物。技术实现要素:[技术问题]本发明要解决的技术问题是构建一种经改造的大肠杆菌基因工程菌,能够实现亚精胺的高效生产。[技术方案]本发明提供了一种大肠杆菌基因工程菌,是以大肠杆菌为宿主,敲除了编码dna结合转录抑制因子的metj基因、编码dna结合双转录调节因子的argr基因,并对编码高丝氨酸o-琥珀酰转移酶的meta基因进行突变构建负反馈突变体,以解除甲硫氨酸对高丝氨酸o-琥珀酰转移酶的反馈抑制。更进一步地,本发明还利用强启动子替代编码甲硫氨酸腺苷基转移酶的基因metk、编码鸟氨酸脱羧酶的基因spec、编码s-腺苷甲硫氨酸脱羧酶和亚精胺合成酶的基因spede的天然启动子。例如,利用强启动子trc替代metk、spec、spede的天然启动子,以强化表达甲硫氨酸腺苷基转移酶、鸟氨酸脱羧酶、s-腺苷甲硫氨酸脱羧酶、亚精胺合成酶。更进一步地,本发明还敲除了将亚精胺转移到胞内的转运蛋白potabcd,并强化了将亚精胺转移到胞外的转运蛋白mdtji的表达。例如,敲除了转运蛋白的atp结合亚基pota,并利用强启动子trc替代mdtj、mdti的天然启动子,以强化表达大肠杆菌将亚精胺转运出细胞的蛋白。更进一步地,本发明还进一步敲除了编码二氨基庚酸脱羧酶的lysa基因,或者,在敲除了lysa基因的基础上进一步敲除编码高丝氨酸激酶和苏氨酸合成酶的thrb和thrc基因,或者,在敲除了lysa、thrb和thrc基因的基础上进一步敲除编码吡咯啉-5-羧酸还原酶的proc基因。更进一步地,本发明还进一步敲除了编码谷氨酸-腐胺连接酶的puua基因。更进一步地,本发明还进一步引入外源的合成亚精胺的途径。例如,本发明还进一步表达了外源的羧基亚精胺脱氢酶基因、外源羧基亚精胺脱羧酶基因。本发明提供的基因工程菌可以用于生产亚精胺,尤其是用于高密度培养生产亚精胺。本发明的基因工程菌可以选择的宿主包括大肠杆菌bl21(de3)、大肠杆菌jm109、大肠杆菌dh5α、大肠杆菌top10或大肠杆菌mg1655。构建本发明的基因工程菌时,可以利用同源重组原理进行基因的整合表达,敲除基因时可以采用同源重组敲除方法或者crispr/cas9敲除方法。[有益效果]本发明基于合成生物学方法构建了一种以葡萄糖为原料高产亚精胺的大肠杆菌,生产过程简单且原料易得成本低,具有良好的工业化应用前景。本发明解除了反馈抑制和阻遏,如图1所示,甲硫氨酸的合成是从高丝氨酸开始的多级酶联反应,移除甲硫氨酸对meta编码的高丝氨酸o-琥珀酰转移酶的抑制作用是必须的。metj和argr则分别为蛋氨酸(met)和丁二胺(put)途径的阻遏蛋白。本发明解除了甲硫氨酸的反馈抑制,并解除了metj和argr对途径的阻遏。本发明强化了关键酶的表达。spee基因编码的亚精胺合成酶,是put和dc-sam缩合形成亚精胺(spd)的关键酶,metk编码的蛋氨酸腺苷转移酶则是合成dc-sam的关键酶,强化这两个酶的表达,可以提高亚精胺的产量。本发明改善了亚精胺的转运途径,potabcd和mdtji蛋白分别是亚精胺转运入和转运出细胞的蛋白,将potabcd敲除,将mdtji强化表达,可以提高亚精胺的产量。本发明弱化了亚精胺过程的竞争途径,在亚精胺合成过程中的天冬半醛中间体有多个氨基酸生成的竞争途径,如lys、thr/iso;谷氨酸中间体也存在竞争途径,如pro的合成,将这几个途径弱化,可以提高亚精胺的产量。本发明敲除相关分解途径,大肠杆菌中存在利用和降解亚精胺合成的中间体和产物的酶,如astc编码的琥珀酰鸟氨酸转氨酶会降解鸟氨酸;speg编码的丁二胺乙酰转移酶会将丁二胺转化为乙酰化的形式,同时还有puua介导的丁二胺降解途径。因此,敲除这几个相关的分解途径,可以提高亚精胺的产量。本发明引入外源途径,大肠杆菌内传统的亚精胺合成途径,经过羧基化-s-腺苷甲硫氨酸和腐胺在亚精胺合成酶作用下合成亚精胺,羧基化-s-腺苷甲硫氨酸的合成途径较长。因此,引入了外源casdh/casdc合成途径,在大肠杆菌中实现亚精胺的双途径合成,可以进一步提高亚精胺的产量。附图说明图1两种现有的合成亚精胺的途径具体实施方式1、本发明所涉及的菌株及质粒购自novagen公司的prsfduet-1、ptrc99a、pkd46、pcp20、t19载体质粒,pmd18-t载体质粒、escherichiacolibl21(de3)、escherichiacolijm109、escherichiacolidh5α、escherichiacolitop10、escherichiacolimg1655。2、发酵条件本发明验证菌株产量时所采用的摇瓶发酵培养基配方为:每升含有10g葡萄糖,10g酵母提取物,20gnacl,50%(v/v)的r/2培养基,5ml微量金属元素原液;r/2培养基成分为:每升含有2g(nh4)2hpo4、6.75gkh2po4,、0.85g柠檬酸、0.7gmgso4·7h2o,.ph6.86;微量金属元素原液成分为:每升含有5mhcl、10gfeso4·7h2o、2.25gznso4·7h2o、1gcuso4·5h2o、0.5gmnso4·5h2o、0.23gna2b4o7·10h2o、2gcacl2·2h2o、0.1g(nh4)6mo7o24.。摇瓶发酵控制温度37℃和初始ph为7,装液量为摇瓶体积的1/5,转速控制200rpm,摇瓶发酵时间24小时。3.亚精胺含量的测定方法制备opa衍生试剂:采用uren法和karababa(15)制备opa试剂。0.20gopa溶于9.0ml甲醇中,加入1.0mlof0.40m(ph9.0)硼酸盐缓冲液和160μl2-巯基乙醇(还原剂)。opa试剂4℃保存。亚精胺首先由邻苯二甲醛(opa;sigma,st.louis,mo)衍生化。将50μl样品和450μl水加入400μl甲醇中。加入100μlopa试剂后,将混合物通过0.2-μm滤膜过滤,并立即将20.0μl滤液注入hplc仪器中。hplc检测条件:参考文献(qian,z.g.,x.x.xia,ands.y.lee,metabolicengineeringofescherichiacolifortheproductionofputrescine:afourcarbondiamine.biotechnolbioeng,2009.104(4):p.651-662)中的方法对亚精胺进行检测:使用在25℃和0.8ml/min流动相下操作的luna5-μmc18(2)100a柱(250×4.6mm;phenomenex)用于所有样品分离。流动相由溶剂a(在0.1m乙酸钠中的55%甲醇,ph7.2)和溶剂b(甲醇)组成。应用以下梯度:1-6分钟,100%a;6-10分钟,b的线性梯度从0%到30%;10-15分钟,b的线性梯度从30%到50%;15-19分钟,b的线性梯度从50%到100%;19-23分,100%b;23-25分钟,b的线性梯度从100%到30%;25-28分钟,b的线性梯度从30%到0%(均以体积%计)。4、基因的整合表达方案采用同源重组原理进行基因的整合表达。具体地,利用扩增引物“kz-f/r”扩增出目的基因,通过同源重组的方式整合到t19载体质粒的多克隆位点处。以目的基因上下1000bp处为模版,扩增出同源臂左臂(la)和右臂(ra),连接到t19载体质粒上的目的基因的两端,构建同源臂。提取连接好左臂右臂的质粒:t19-la-trc-目的基因-ra,以此为模板,利用相应的引物,扩增目的片段:la-trc-目的基因-ra,pcr产物经纯化后,利用限制性内切酶dpni消除模板效应。酶切后的产物继续纯化,纯化后转入大肠杆菌感受态细胞中,利用卡钠霉素抗性标记在抗性平板上筛选阳性转化子,为验证是否发生重组,如果发生重组,利用相应的引物“目的基因-yz-f”和“目的基因-yz-r”可以扩增出1200bp左右的片段,反之,应无法扩增出任何片段。同时,用氨苄卡钠双抗平板点相应的pcr验证菌,确定得到正确的转化子e.coli-pkd46(kanar)。对卡钠霉素抗性进行消除,首先cacl2法制备e.coli-pkd46(kanar)感受态,将pcp20质粒转化到e.coli-pkd46(kanar)感受态中后,利用无抗性平板和有抗性平板筛选抗性消除转化子,菌落pcr鉴定转化子,筛选出整合了目的基因和消除抗性的转化子。应用上述整合表达的方法和整合操作步骤,将突变的基因和替换的启动子序列等整合到基因组所需的位置上,对菌株进行相应的代谢工程改造。酶切反应体系:10×qcutbuffer5μl,dna20μl,ddh2o33μl,内切酶-11μl,内切酶-21μl,混匀,37℃水浴加热0.5~1h。连接反应体系:闪电克隆酶5μl,双酶切质粒(10-100ng)1-2μl,同源目的片段4-5μl,ddh2o补足至10μl,混匀,50℃水浴加热30min。dpni消除模板效应反应体系(60μl):dpni3μl,pcr纯化产物20μl,10×fastdigestbuffer6μl,ddh2o31μl。pcr扩增目的基因反应体系(50μl):dna模板1μl,5×primestarbuffer10μl,dntpmixture4μl,扩增引物1μl,扩增引物1μl,primestarhsdnapolymerase0.5μl,ddh2o32.5μl。pcr扩增程序:30循环(98℃10s,55℃10s,72℃1kb/min),72℃10min,4℃保存。菌落pcr反应体系:2×tagmastermix10μl,上游引物(浓度10μm)1μl,下游引物(浓度10μm)1μl,ddh2o8μl,少量菌落或菌液做模板。实施例1:解除途径中重要酶的反馈抑制及阻遏蛋白对途径启动子的抑制在脱羧s-腺苷甲硫氨酸和腐胺合成的重要中间体过程中,存在着重要的阻遏蛋白,抑制中间产物和终产物的合成,metj基因编码的dna结合转录抑制因子能够抑制met系列基因的表达,最终抑制甲硫氨酸的过量合成;argr基因编码的dna结合双转录调节因子,抑制arg系列基因的表达,抑制所需重要中间体鸟氨酸和精氨酸的过量合成,因此,为了保证各中间体稳定高速率的合成,将编码该两个阻遏蛋白的基因删除,以提高代谢通量。在脱羧s-腺苷甲硫氨酸合成的代谢途径中,meta编码的高丝氨酸o-琥珀酰转移酶会受产物甲硫氨酸的反馈抑制,为了提高该酶的活性,在敲除metj基因、argr基因的基础上,进一步将编码该酶的基因进行碱基突变后构建负反馈突变体,解除反馈抑制,得到工程菌e.colimetafbrδmetjδargr,将之命名为e1。具体过程如下:(1)阻遏蛋白metj和argr的敲除引物设计如下表1:表1(seqidno.1~10)引物名称引物序列5’-3’apai-la-metjgagattgggcccgcagcagatagctgtcaaacmetj-la-ecoricacggagaattcgagatacttaatcctcttcgkpni-ra-metjctcggtaccagcaaaaaagagcggcgcggmetj-ra-saligacgatgtcgacgaaaatccgctccaccgttgla-yz-ftgaattagaactcggtacgcgcggala-yz-rtagagaataggaacttcgaactgcara-yz-faacttcgaagcagctccagcctacara-yz-ratgattacgccaagtttgcacgcctmetj-yz-fagttttttcaccaacggctgggtttmetj-yz-rcgccctgagtgcttgcggcagcgtg利用质粒提取试剂盒对同源重组酶表达pkd46质粒进行提取,经纯化后转化到感受态细胞e.colibl21中,构建e.colibl21-pkd46。cacl2法制备e.colibl21-pkd46感受态细胞后,-80℃保存备用。利用质粒提取试剂盒,提取同源重组片段模板质粒t19载体,并用割胶回收试剂盒进行纯化回收。设计引物apai-la-metj和metj-la-ecori,kpni-ra-metj和metj-ra-sali扩增相应的同源臂la和ra片段,并利用割胶回收试剂盒进行纯化回收。利用apai和ecori对质粒t19载体进行双酶切,电泳检测酶切结果,酶切后大小为5041bp,酶切产物进行纯化回收。t19载体经双酶切后的产物与左臂la片段进行连接。将连接好的产物转化进jm109感受态,涂布在卡钠抗性平板上,筛选阳性克隆菌株,利用引物la-yz-f和la-yz-r进行菌落pcr验证结果,条带大小为1100bp左右,验证成功的菌株送去测序,进一步确认片段连接成功。利用kpni和sali对连接好la片段的t19质粒进行双酶切,采取上述同样的方法,连接上ra片段,对连接的质粒进行验证。提取连接好la和ra片段的t19载体质粒,并以此为模板,以引物apai-la-metj和metj-ra-sali进行pcr,得到目的片段metj-la-frt-kan-frt-ra。电泳鉴定条带大小为3324bp左右。pcr产物经纯化后,利用限制性内切酶dpni消除模板效应。限制性内切酶dpni酶切后的目的片段metj-la-frt-kan-frt-rapcr产物继续纯化,纯化后转入e.colibl21-pkd46感受态细胞中,利用卡钠霉素抗性标记在抗性平板上筛选阳性转化子,为验证筛选得到的阳性转化子是否的确发生了重组,以阳性转化子为模版,利用引物metj-yz-f和metj-yz-r可以扩增出1100bp左右的片段则可以判断是发生了重组,反之,如果无法扩增出任何片段则可以判断是假阳性。在pcr扩增验证是否发生了重组的同时,用氨苄卡钠平板点相应的pcr验证菌。将经确定确实发生了重组的正确的转化子e.colibl21-pkd46(kanar)进行卡钠霉素抗性消除,首先利用cacl2法制备e.colibl21-pkd46(kanar)感受态,提取的pcp20质粒转化到e.colibl21-pkd46(kanar)感受态中后,利用无抗性平板和有抗性平板筛选抗性消除转化子,菌落pcr鉴定转化子,筛选出敲除metj基因和消除抗性的转化子e.colibl21-△metj。阻遏蛋白argr的敲除方式同上,引物设计如下表2。筛选出正确的敲除菌株e.colibl21△metj△argr。表2(seqidno.11~30)(2)metafbr突变体的构建对于meta基因,受到目的产物甲硫氨酸的反馈抑制,研究发现,meta基因的79位碱基c突变为t,所编码的精氨酸变为半胱氨酸后,显著提高甲硫氨酸的产量,因此,我们对该位点进行突变,将其整合到质粒prsfduet-1质粒上。具体过程如下:引物设计如下表3:表3(seqidno.31~34)引物名称引物序列5’-3’f-meta-扩增引物ggagatatacatatgatgccgattcgtgtgccggr-meta-扩增引物ggtttctttaccagactcgagttaatccagcgttggattcatgtgccgmeta-tb-fgtgatgacaacttcttgtgcgtctgmeta-tb-raagaagttgtcatcacaaagacgtt首先,利用质粒提取试剂盒提取prsfduet-1(高拷贝)质粒,选择bamhⅰ和hindⅲ进行双酶切,酶切体系(50μl)如下:10×qcutbuffer5μl,质粒20μl,bamhi1μl,hindⅲ1μl,ddh2o33μl,酶切后进行纯化。其次,提取野生型e.colibl21基因组,以基因组为模板,扩增meta基因片段,pcr产物验证后,连接到酶切后的prsfduet-1质粒上。cacl2法制备e.colijm109感受态细胞,将连接好meta片段的prsfduet-1质粒转化进入感受态中,挑选阳性转化子,菌落pcr鉴定后为转化成功的菌株e.colijm109-meta。提取e.colijm109-meta中的质粒prsfduet1-meta,以该质粒为模板,进行meta的定点突变。利用fastmutangenesissystem试剂盒进行突变基因的设计和提取,以提取的prsfduet1-meta质粒为模板,以meta突变体引物meta-tb-f/r扩增metafbr片段,其中,pcr扩增体系(50μl)如下:质粒模板3μl,meta-tb-f1μl,meta-tb-r1μl,flypcrsupermix25μl,ddh2o20μl。pcr扩增程序为:94℃2-5min,20-25循环(94℃20s,55℃20s,72℃2-6kb/min),72℃10min,4℃保存。对得到的pcr产物,加入1-3μldmt酶进行消化,混匀,37℃孵育1h。将得到的dmt酶消化产物,利用pcr产物纯化试剂盒进行纯化后,取2-5μl加入到50μljm109感受态细胞中,进行转化操作。挑选阳性转化子,经菌落pcr初步鉴定,以及公司测序后筛选出正确的转化子jm109-metafbr。提取转化子jm109-metafbr中的质粒prsfduet1-metafbr,按照前述“4、基因的整合表达方案”,将基因片段metafbr整合到敲除菌株e.colibl21△metj△argr上,构建菌株e.colimetafbrδmetjδargr,将之命名为e1,以此为基础菌株,进行后续的研究。摇瓶培养验证e1亚精胺的产量,测得摇瓶上清液中亚精胺浓度为4.8g/l。实施例2:强化表达途径关键酶在合成脱羧s-腺苷甲硫氨酸和腐胺重要中间体的代谢过程中,一些基因作为限速酶,严格调控整个代谢途径的通量表达,metk基因编码的甲硫氨酸腺苷基转移酶作为甲硫氨酸代谢合成途径的限速酶,在很大程度上抑制甲硫氨酸的过量合成,因此,我们利用强启动子trc替换其天然启动子,强化其表达,促进甲硫氨酸代谢通量的合成。其中,trc启动子(序列为:5’-ttgacaattaatcatccggctcgtataatg-3’)来源于ptrc99a质粒。sped基因编码的s-腺苷甲硫氨酸脱羧酶,催化s-腺苷甲硫氨酸合成脱羧-s腺苷甲硫氨酸,随后被spee编码的亚精胺合成酶催化和腐胺共同合成目标产物亚精胺(spd),为了加强该代谢途径的酶活以及代谢通量,利用强启动子trc替换其天然启动子,强化其表达。spec编码的鸟氨酸脱羧酶,受到产物腐胺,亚精胺的抑制,通过替换强trc启动子,促使其过量表达,减轻反馈抑制,积累更多的目标产物。以e1菌为出发菌,进一步强化表达合成通路关键酶metk,sped,spec以及spee,利用强trc启动子替代该基因天然启动子后,得到工程菌e1-ptrcmetkptrcspedptrcspecptrcspee菌株。具体过程如下:根据ncbi中e.colibl21的metk,采用primerpremier5设计相关的片段引物(表4)。以e.colibl21基因组为模板,metk-kz-f/r为引物,pcr扩增目的基因片段metk,在上游下游分别带上酶切位点ecori和hindⅲ,纯化pcr片段。利用ecori和hindⅲ对ptrc99a质粒进行双酶切,酶切后的质粒进行纯化,和纯化后的metk片段进行连接和转化e.colijm109感受态,挑选阳性转化子,利用验证引物metk-99a-yz-f/r进行菌落pcr和测序,验证其正确性。提取转化成功的质粒ptrc-metk,以此为模板,以bamhi-trc-metk-f和trc-metk-saci-r为引物,扩增出目的片段trc-metk序列,经跑胶验证为1253bp左右,并进行纯化。利用bamhi和saci两种酶对t19载体质粒进行双酶切,经纯化后与trc-metk片段进行连接和转化,利用验证引物trc-metk-t19-yzf/yzr经过菌落pcr和公司测序进行验证,挑选正确的阳性转化子t19-trc-metk。以e.colibl21中metk基因组为模板,以metk-la-apai和ecori-metk-la为引物设计扩增左臂片段la,以metk-ra-kpni和sali-metk-ra为引物扩增右臂片段ra,经过纯化后,分别连接到双酶切并纯化的质粒t19-trc-metk左臂和右臂上,利用验证引物metk-la-yz-f和metk-la-ya-r,metk-ra-yz-f和metk-ra-ya-r进行菌落pcr初步验证,并送公司测序,找到正确连接左臂和右臂的质粒t19-la-trc-metk-ra。提取连接好左臂右臂的质粒t19-la-trc-metk-ra,以此为模板,以metk-la-apai和sali-metk-ra为引物,扩增目的片段la-trc-metk-ra,大小为3700bp左右,pcr产物经纯化后,利用限制性内切酶dpni消除模板效应。酶切后的产物继续纯化,纯化后转入e1-pkd46(利用质粒提取试剂盒对同源重组酶表达质粒pkd46进行提取,经纯化后转化到感受态细胞e1中,构建e1-pkd46)感受态细胞中,利用卡钠霉素抗性标记在抗性平板上筛选阳性转化子,为验证是否发生重组,利用引物metk-yz-f和metk-yz-r可以扩增出1200bp左右的片段,反之,应无法扩增出任何片段。同时,用氨苄卡钠板点相应的pcr验证菌。确定正确的转化子e1-pkd46(kanar)。对卡钠霉素抗性进行消除,首先cacl2法制备e1-pkd46(kanar)感受态,将pcp20质粒转化到e1-pkd46(kanar)感受态中后,利用无抗性平板和有抗性平板筛选抗性消除转化子,菌落pcr鉴定转化子,筛选出替换metk基因启动子和消除抗性的转化子e1-metkptrc。依照上述方法,依次替换基因spec、sped以及spee基因的启动子。由e.coli基因组分析可知,spee和sped位于同一基因簇上,由同一个启动子进行诱导,因此改变共有的启动子即可。构建菌株e1-metkptrcspecptrcspeedptrc,将其命名为e2。所用引物如下表4:表4(seqidno.35~88)摇瓶培养验证e2亚精胺的产量,测得摇瓶上清液中亚精胺浓度为6.3g/l。实施例3:改善转运途径potabcd是大肠杆菌将亚精胺吸收入细胞的转运蛋白,因此将它敲除;由于potabcd蛋白为基因簇pota,potb,potc,potd基因编码的四个亚基所组成,pota编码atp结合亚单位,为转运提供必须的能量,potb编码abc转运膜亚基potb,potc编码abc转运膜亚基potc,potd编码abc转运蛋白周质结合蛋白,操纵子的四个基因对该操纵子功能的正确表达是必须的,任何一个基因的缺失必会导致功能的失活,因此,本研究中删除atp结合亚基pota,削弱转入蛋白运输亚精胺的能力,在e3的基础上构建改善转运途径的菌株。按照实施例1中red同源重组的方法对potabcd进行敲除,得到菌株e3△potabcd。mdtji是大肠杆菌将亚精胺转运出细胞的蛋白,由基因簇mdtj和mdti组成的基因簇所编码,因此,利用强trc启动子将之强化表达。按照实施例2所述的方法,替换其启动子,得到转运途径改善的菌株e3△potabcdptrcmdtji,并将其命名为e3。表5(seqidno.89~116)摇瓶培养验证e3亚精胺的产量,测得摇瓶上清液中亚精胺浓度为8.5g/l。实施例4:弱化竞争途径(1)lys竞争途径:中间体物质天冬氨酸半醛经过dap系列的基因和lysa基因编码的二氨基庚酸脱羧酶进一步转化为赖氨酸,为了解除该竞争代谢途径的分流,按照实施例1所述的方法,以菌株e3为基础,利用red同源重组的方法删除基因lysa,构建菌株e3△lysa。将得到菌株命名为e4-1。(2)thr/iso竞争途径:删除thrb或thrbc操纵子;thrb和thrc基因分别编码的高丝氨酸激酶和苏氨酸合成酶,将高丝氨酸转化苏氨酸和异亮氨酸,为该两种氨基酸合成途径的关键基因,为了减弱该分支途径的影响,使更多的高丝氨酸流向甲硫氨酸合成的代谢途径中,按照实施例1所述的方法,以菌株e3△lysa为基础,利用red同源重组的方法敲除操纵子基因thrbc,构建菌株e3△lysa△thrbc。将得到菌株命名为e4-2。(3)glu存在竞争途径:经prob编码的谷氨酸激酶,proa编码的谷氨酸半醛脱氢酶形成谷氨酸半醛,谷氨酸半醛可自发地转化为吡咯啉-5-羧酸,该物质经proc编码的吡咯啉5-羧酸还原酶转化为脯氨酸(pro),因此为了阻止竞争途径的代谢流,我们将删除基因proc,弱化该途径。按照实施例1所述的方法,以菌株e3△lysa△thrbc为基础,利用red同源重组的方法删除表达竞争途径基因proc,构建菌株e4△lysa△thrbc△proc。将得到菌株命名为e4-3。所需的引物如下表6。表6(seqidno.117~146)摇瓶培养验证e4-1,e4-2,e4-3亚精胺的产量,测得摇瓶上清液中亚精胺浓度为9.3g/l,9.7g/l,11.3g/l。实施例5:敲除相关分解途径put存在利用和降解途径(竞争途径):(1)put经过puu途径转化为氨基丁酸。首先,腐胺由puua基因编码的谷氨酸-腐胺连接酶催化转化为γ-谷氨酸-腐胺,随之被puub编码的γ-谷氨酸-腐胺氧化酶氧化为γ-谷氨酸-γ-丁醛,并被puuc编码的γ-谷氨酸-γ-丁醛脱氢酶进一步氧化为γ-谷氨酸-γ-氨基丁酸,该物质随后被puud编码的γ-谷氨酸-γ-氨基丁酸水解酶水解为谷氨酸和氨基丁酸(gaba),puu基因簇介导的puu途径伴随了谷氨酰胺化中间体的反应过程。(2)此外,还存在ygjg介导的非puu途径,ygig编码的腐胺:酮戊二酸氨基转移酶和ydcw编码的氨基丁酸脱氢酶将腐胺降解为gaba而没有谷氨酰胺化的传统途径,此途径的中间体γ-氨基丁醛很不稳定,易自发地形成△1-吡咯啉。puu途径中的谷氨酰胺化稳定了中间体物质,且研究发现,非puu途径中ygjg活性比较低,因此,为降低代谢工程菌的损伤,我们弱化了主要的put利用的途径,即删除puua基因,弱化了puu途径。根据实施例1所述的方法,以菌株e4-3为该代谢途径的出发菌株,利用red同源重组的方法删除基因puua,阻止腐胺的进一步降解,使更多的代谢流流向亚精胺的合成,得到菌株e5(e4-3△puua,或称为e.hzh01)。所需的引物如下表7:表7(seqidno.147~156)摇瓶培养验证e5亚精胺的产量,测得摇瓶上清液中亚精胺浓度为14.7g/l。实施例6:双途径合成亚精胺天冬氨酸半醛是细胞内的重要中间代谢产物,是甲硫氨酸和脱羧s-腺苷甲硫氨酸合成的直接前体物质,也是亚精胺合成的重要前体物质。从天冬氨酸半醛到脱羧s-腺苷甲硫氨酸合成的代谢途径中复杂多变,代谢途径较长,且涉及多种氨基酸中间体的代谢,对其代谢途径进行过度改造后,容易影响菌体的生长。基于上述原因,我们将来自不同微生物菌株中的casdh/casdc途径引入到e.coli中进行异源表达。casdh编码的羧基化亚精胺脱氢酶将直接利用天冬氨酸半醛和腐胺作为直接前体物质合成羧基化亚精胺,而后在casdc编码的羧基化亚精胺脱羧酶作用下直接脱羧合成亚精胺,该代谢途径只包含两个基因,在很大程度上缩短代谢途径,减少代谢流的分散,更大程度的合成亚精胺。pand编码的l-天冬氨酸脱羧酶将天冬氨酸脱羧转化为β-丙氨酸,为了减少该代谢途径,增加天冬氨酸流向甲硫氨酸的代谢途径,我们将casdh/casdc基因整合到该位点上,同时替换敲除该基因。因此,根据实施例1采用的同源重组方式,外源整合casdh/casdc途径。具体过程如下:利用扩增引物从ruminococcuscallidusatcc27760、porphyromonascatoniaeatcc51270、rhodobactersphaeroidesatccbaa-808、clostridiumsymbiosumatcc14940的基因组中克隆得到羧基亚精胺脱氢酶基因rccsdh、pccsdh、rscsdh、cscsdh。氨基酸序列在ncbi上accessionno为erj96771.1、ewc93538.1、yp_351518.1、eri79986.1。从bacteroidescellulosilyticusdsm14838、clostridiumsymbiosumatcc14940、campylobacterjejunisubsp.jejuniatcc700819、rhodobactersphaeroidesatccbaa-808的基因组中克隆得到羧基亚精胺脱羧酶基因bccsdc、cscsdc、cjcsdc、rscsdc,氨基酸序列在ncbi中的编号分别是eef87925.1、eri79985.1、yp_002344893.1、yp_351517.1。将克隆得到的两种基因分别连接到prsfduet-1质粒上,即可得到强化表达2个基因的重组大肠杆菌。以rccsdh-bamhi-f和rccsdh-hindⅲ-r为引物,ruminococcuscallidusatcc27760基因组为模板,扩增出目的片段rccsdh;利用双酶bamhi和hindⅲ酶酶切处理质粒prsfduet-1的第一个多克隆位点,纯化酶切产物后,将切开的质粒与扩增得到的目的片段rccsdh进行连接,连接产物prsfduet-1-rccsdh转入到jm109感受态细胞中,于抗性平板卡钠平板中挑选阳性克隆菌,菌落pcr进一步验证其正确性;以bccsdc-kpni-f和bccsdc-xhoi-r为引物,bacteroidescellulosilyticusdsm14838基因组为模板,扩增出目的片段bccsdc,利用双酶kpni和xhoi酶切质粒prsfduet-1-rccsdh的第二个多克隆位点,纯化酶切产物后,与扩增的目的片段bccsdc进行连接,连接产物prsfduet-1-rccsdh-bccsdc转化至jm109感受态细胞中,于抗性平板卡钠平板中挑选阳性克隆菌,菌落pcr进一步验证其正确性。挑选出连接正确的菌株prsfduet-1-rccsdh-bccsdc,提取质粒prsfduet-1-rccsdh-bccsdc转化至e5感受态细胞中,构建e5-rccsdh-bccsdc亚精胺合成菌株。其余菌株的构建方法和e5-rccsdh-bccsdc相同,分别将来自不同菌株的羧基亚精胺脱氢酶和羧基亚精胺脱羧酶连接到prsfduet-1质粒的两个多克隆位点上。其它基因的连接,方式和上述相同。所用引物序列如表8:表8(seqidno.157~160)引物名称序列5’-3’rccsdh-bamhi-fccacagccaggatccgatgaacatgggaaaagcgttrccsdh-hindⅲ-rgcattatgcggccgcaagcttttaatctaccagcaccggatbccsdc-kpni-fgcgatcgctgacgtcggtaccttggttaaaaaagaaaatatbccsdc-xhoi-rggtttctttaccagactcgagtcaactcattctgtcgcgat将重组大肠杆菌按体积比为2%的量转接到lb培养基(蛋白胨10g/l、酵母粉5g/l、nacl10g/l)中,当细胞od600达到0.6-0.8后,加入终浓度为0.4mm的iptg,在20℃诱导表达培养12h。诱导表达结束后,4℃、8000rpm、20分钟离心收集上清。利用hplc检测上清液中亚精胺浓度,如表9所示。表9应用表达载体过表达亚精胺合成的关键基因,会给菌体带来代谢负担影响菌体的生长,表达载体在菌体繁殖过程中的结构不稳定、易丢失以及在细胞中的拷贝数的不稳定,添加抗生素影响菌体生长且污染环境,iptg诱导造成的成本增加等因素限制了生产规模的扩大,因此,挑选诱导表达合成亚精胺效果最好的菌株e5-rccsdh-bccsdc,根据实施例1所示的方法,将其基因整合到e5(或称为e.hzh01)菌株中,构建双重合成途径菌株e.hzh02。具体地,以质粒prsfduet-1-rccsdh-bccsdc为模板,扩增出带有pand基因(编码天冬氨酸1-脱羧酶酶原的基因,分解天冬氨酸变为丙氨酸和二氧化碳)上下同源臂(50bp)的kana完整基因,连接到pmd18-t载体质粒上,构建得到pmd18-k。继续以prsfduet-1-rccsdh-bccsdc为模板,扩增出rccsdh-bccsdc两基因,与pmd18-k质粒相连接,得到pmd18-k-rccsdh-bccsdc。利用内切酶pvuⅱ酶切,得到目的片段pandf-kan-rccsdh-bccsdc-pandr,电转化至表达宿主菌株e5/pkd46感受态(按照实施例1中制备感受态方法制备),在重组酶的作用下,按照同源重组的原理,外源pandf-kan-rccsdh-bccsdc-pandr目的片段与染色体上pand对应区域发生同源重组,并将该基因删除,替换为kan-rccsdh-bccsdc片段,获得整合上rccsdh-bccsdc目的基因的菌株,并命名为e.hzh02,e.hzh02是整合了rccsdh-bccsdc基因的菌株。以pand基因上下游序列为引物,e.hzh02基因组dna为模板进行pcr,扩增产物结果表明rccsdh-bccsdc成功整合到基因组上。摇瓶培养验证e.hzh01,e.hzh02亚精胺的产量,测得摇瓶上清液中亚精胺浓度为14.7g/l,18.5g/l。实施例7各类整合表达菌株及高密度培养比较进一步分别在大肠杆菌jm109、dh5α、top10、mg1655中进行如实施例1-7的相同操作,得到类似的双途径整合亚精胺生产途径菌株e.jm、e.dh、e.to、e.mg,并行24小时高密度培养,培养基组成及流加操作如文献所示(simplefed-batchtechniqueforhighcelldensitycultivationofescherichiacoli.journalofbiotechnology1995,39:59-65),菌体量及亚精胺产如表10所示。将e.jm、e.dh、e.to、e.mg分别进行摇瓶培养,亚精胺的产量分别为17.3、21.3、22.4、25.6g/l。表10不同菌株高密度培养时的结果菌株菌体湿重(g/l)亚精胺浓度e.hzh02118196g/le.jm114178g/le.dh131193g/le.to119200g/le.mg140223g/l虽然本发明已以较佳实施例公开如上,但其并非用以限定本发明,任何熟悉此技术的人,在不脱离本发明的精神和范围内,都可做各种的改动与修饰,因此本发明的保护范围应该以权利要求书所界定的为准。sequencelisting<110>江南大学<120>baa190787a<130>一种生产亚精胺的工程菌<160>160<170>patentinversion3.3<210>1<211>32<212>dna<213>人工序列<400>1gagattgggcccgcagcagatagctgtcaaac32<210>2<211>32<212>dna<213>人工序列<400>2cacggagaattcgagatacttaatcctcttcg32<210>3<211>29<212>dna<213>人工序列<400>3ctcggtaccagcaaaaaagagcggcgcgg29<210>4<211>32<212>dna<213>人工序列<400>4gacgatgtcgacgaaaatccgctccaccgttg32<210>5<211>25<212>dna<213>人工序列<400>5tgaattagaactcggtacgcgcgga25<210>6<211>25<212>dna<213>人工序列<400>6tagagaataggaacttcgaactgca25<210>7<211>25<212>dna<213>人工序列<400>7aacttcgaagcagctccagcctaca25<210>8<211>25<212>dna<213>人工序列<400>8atgattacgccaagtttgcacgcct25<210>9<211>25<212>dna<213>人工序列<400>9agttttttcaccaacggctgggttt25<210>10<211>25<212>dna<213>人工序列<400>10cgccctgagtgcttgcggcagcgtg25<210>11<211>47<212>dna<213>人工序列<400>11cgcgcggatcttccagagattgggcccgcagcagatagctgtcaaac47<210>12<211>47<212>dna<213>人工序列<400>12aagcttgacgtccaggtgcctcttaaggagatacttaatcctcttcg47<210>13<211>47<212>dna<213>人工序列<400>13cacgctagcggatccgagctcggtaccagcaaaaaagagcggcgcgg47<210>14<211>47<212>dna<213>人工序列<400>14cgtgcggacggcaagctgctacagctggaaaatccgctccaccgttg47<210>15<211>25<212>dna<213>人工序列<400>15tgaattagaactcggtacgcgcgga25<210>16<211>25<212>dna<213>人工序列<400>16tagagaataggaacttcgaactgca25<210>17<211>25<212>dna<213>人工序列<400>17aacttcgaagcagctccagcctaca25<210>18<211>25<212>dna<213>人工序列<400>18atgattacgccaagtttgcacgcct25<210>19<211>25<212>dna<213>人工序列<400>19agttttttcaccaacggctgggttt25<210>20<211>25<212>dna<213>人工序列<400>20cgccctgagtgcttgcggcagcgtg25<210>21<211>47<212>dna<213>人工序列<400>21cgcgcggatcttccagagattgggcccacctgtgacagcagcggcag47<210>22<211>47<212>dna<213>人工序列<400>22aagcttgacgtccaggtgcctcttaagaagtcacccgatatggtggt47<210>23<211>47<212>dna<213>人工序列<400>23cacgctagcggatccgagctcggtacctctctgccccgtcgtttctg47<210>24<211>47<212>dna<213>人工序列<400>24cgtgcggacggcaagctgctacagctgcgatgcacggcagaactcgc47<210>25<211>25<212>dna<213>人工序列<400>25tgaattagaactcggtacgcgcgga25<210>26<211>25<212>dna<213>人工序列<400>26tagagaataggaacttcgaactgca25<210>27<211>25<212>dna<213>人工序列<400>27aacttcgaagcagctccagcctaca25<210>28<211>25<212>dna<213>人工序列<400>28atgattacgccaagtttgcacgcct25<210>29<211>25<212>dna<213>人工序列<400>29agacccgccaccggccttcgcttca25<210>30<211>25<212>dna<213>人工序列<400>30cgccctgagtgcttgcggcagcgtg25<210>31<211>34<212>dna<213>人工序列<400>31ggagatatacatatgatgccgattcgtgtgccgg34<210>32<211>48<212>dna<213>人工序列<400>32ggtttctttaccagactcgagttaatccagcgttggattcatgtgccg48<210>33<211>25<212>dna<213>人工序列<400>33gtgatgacaacttcttgtgcgtctg25<210>34<211>25<212>dna<213>人工序列<400>34aagaagttgtcatcacaaagacgtt25<210>35<211>28<212>dna<213>人工序列<400>35tggaattcatggcaaaacacctttttac28<210>36<211>29<212>dna<213>人工序列<400>36gccaagcttttacttcagaccggcagcat29<210>37<211>25<212>dna<213>人工序列<400>37tcactgcataattcgtgtcgctcaa25<210>38<211>25<212>dna<213>人工序列<400>38cgctactgccgccaggcaaattctg25<210>39<211>41<212>dna<213>人工序列<400>39agcctacacgctagcggatccttgacaattaatcatccggc41<210>40<211>41<212>dna<213>人工序列<400>40agtatctcaggtaccgagctcttacttcagaccggcagcat41<210>41<211>25<212>dna<213>人工序列<400>41tcttctaataaggggatcttgaagt25<210>42<211>25<212>dna<213>人工序列<400>42cgctgttgcggatcatctccagcgt25<210>43<211>47<212>dna<213>人工序列<400>43cgcgcggatcttccagagattgggcccgcgagcttccgggacgtccg47<210>44<211>47<212>dna<213>人工序列<400>44aagcttgacgtccaggtgcctcttaagatttaatatcacctaaagag47<210>45<211>47<212>dna<213>人工序列<400>45cacgctagcggatccgagctcggtacctctttcttcacctgcgttca47<210>46<211>47<212>dna<213>人工序列<400>46cgtgcggacggcaagctgctacagctgctggcaggaagaagacacca47<210>47<211>25<212>dna<213>人工序列<400>47tgaattagaactcggtacgcgcgga25<210>48<211>25<212>dna<213>人工序列<400>48tagagaataggaacttcgaactgca25<210>49<211>25<212>dna<213>人工序列<400>49aacttcgaagcagctccagcctaca25<210>50<211>25<212>dna<213>人工序列<400>50atgattacgccaagtttgcacgcct25<210>51<211>25<212>dna<213>人工序列<400>51ggaacgcaaacggtgctgcaggatc25<210>52<211>25<212>dna<213>人工序列<400>52cgccctgagtgcttgcggcagcgtg25<210>53<211>41<212>dna<213>人工序列<400>53aggaaacagaccatggaattcatgaaatcaatgaatattgc41<210>54<211>41<212>dna<213>人工序列<400>54tccgccaaaacagccaagcttttacttcaacacataaccgt41<210>55<211>25<212>dna<213>人工序列<400>55tcactgcataattcgtgtcgctcaa25<210>56<211>25<212>dna<213>人工序列<400>56cgctactgccgccaggcaaattctg25<210>57<211>41<212>dna<213>人工序列<400>57agcctacacgctagcggatccttgacaattaatcatccggc41<210>58<211>44<212>dna<213>人工序列<400>58atcagtatctcaggtaccgagctcttacttcaacacataaccgt44<210>59<211>25<212>dna<213>人工序列<400>59tcttctaataaggggatcttgaagt25<210>60<211>25<212>dna<213>人工序列<400>60cgctgttgcggatcatctccagcgt25<210>61<211>47<212>dna<213>人工序列<400>61cgcgcggatcttccagagattgggcccggttgccagcagcaataaac47<210>62<211>47<212>dna<213>人工序列<400>62aagcttgacgtccaggtgcctcttaagaagcgcaaacccgtttcggg47<210>63<211>47<212>dna<213>人工序列<400>63cacgctagcggatccgagctcggtaccgaataaaaaaaacgggtcac47<210>64<211>47<212>dna<213>人工序列<400>64cgtgcggacggcaagctgctacagctgtcgtttgcttatatgccaac47<210>65<211>25<212>dna<213>人工序列<400>65tgaattagaactcggtacgcgcgga25<210>66<211>25<212>dna<213>人工序列<400>66tagagaataggaacttcgaactgca25<210>67<211>25<212>dna<213>人工序列<400>67aacttcgaagcagctccagcctaca25<210>68<211>25<212>dna<213>人工序列<400>68atgattacgccaagtttgcacgcct25<210>69<211>25<212>dna<213>人工序列<400>69caaaccaggcggtccaggcggcgga25<210>70<211>25<212>dna<213>人工序列<400>70cgccctgagtgcttgcggcagcgtg25<210>71<211>41<212>dna<213>人工序列<400>71aggaaacagaccatggaattcatggccgaaaaaaaacagtg41<210>72<211>41<212>dna<213>人工序列<400>72tccgccaaaacagccaagcttttaggacggctgtgaagcca41<210>73<211>25<212>dna<213>人工序列<400>73tcactgcataattcgtgtcgctcaa25<210>74<211>25<212>dna<213>人工序列<400>74cgctactgccgccaggcaaattctg25<210>75<211>41<212>dna<213>人工序列<400>75agcctacacgctagcggatccttgacaattaatcatccggc41<210>76<211>44<212>dna<213>人工序列<400>76atcagtatctcaggtaccgagctcttaggacggctgtgaagcca44<210>77<211>25<212>dna<213>人工序列<400>77tcttctaataaggggatcttgaagt25<210>78<211>25<212>dna<213>人工序列<400>78cgctgttgcggatcatctccagcgt25<210>79<211>47<212>dna<213>人工序列<400>79cgcgcggatcttccagagattgggccctcaccgagcgcttgccacct47<210>80<211>47<212>dna<213>人工序列<400>80aagcttgacgtccaggtgcctcttaaggggttgatacctcctttgtt47<210>81<211>47<212>dna<213>人工序列<400>81cacgctagcggatccgagctcggtaccggagaagataagaaattgaa47<210>82<211>47<212>dna<213>人工序列<400>82cgtgcggacggcaagctgctacagctggaggtgatggttcgcgccaa47<210>83<211>25<212>dna<213>人工序列<400>83tgaattagaactcggtacgcgcgga25<210>84<211>25<212>dna<213>人工序列<400>84tagagaataggaacttcgaactgca25<210>85<211>25<212>dna<213>人工序列<400>85aacttcgaagcagctccagcctaca25<210>86<211>25<212>dna<213>人工序列<400>86atgattacgccaagtttgcacgcct25<210>87<211>25<212>dna<213>人工序列<400>87atggccgaaaaaaaacagtggcatg25<210>88<211>25<212>dna<213>人工序列<400>88cgccctgagtgcttgcggcagcgtg25<210>89<211>47<212>dna<213>人工序列<400>89cgcgcggatcttccagagattgggcccggctttcatccgccggaact47<210>90<211>47<212>dna<213>人工序列<400>90aagcttgacgtccaggtgcctcttaaggtaaacgcaacggatggctt47<210>91<211>47<212>dna<213>人工序列<400>91cacgctagcggatccgagctcggtaccgttccagaatgtagtgattg47<210>92<211>47<212>dna<213>人工序列<400>92cgtgcggacggcaagctgctacagctgtgtcgttgttcatcagcagg47<210>93<211>25<212>dna<213>人工序列<400>93tgaattagaactcggtacgcgcgga25<210>94<211>25<212>dna<213>人工序列<400>94tagagaataggaacttcgaactgca25<210>95<211>25<212>dna<213>人工序列<400>95aacttcgaagcagctccagcctaca25<210>96<211>25<212>dna<213>人工序列<400>96atgattacgccaagtttgcacgcct25<210>97<211>25<212>dna<213>人工序列<400>97cggtcgaaatcacggatgatgtagt25<210>98<211>25<212>dna<213>人工序列<400>98cgccctgagtgcttgcggcagcgtg25<210>99<211>41<212>dna<213>人工序列<400>99aggaaacagaccatggaattcatgtatatttattggatttt41<210>100<211>41<212>dna<213>人工序列<400>100tccgccaaaacagccaagctttcaggcaagtttcaccatga41<210>101<211>25<212>dna<213>人工序列<400>101tcactgcataattcgtgtcgctcaa25<210>102<211>25<212>dna<213>人工序列<400>102cgctactgccgccaggcaaattctg25<210>103<211>41<212>dna<213>人工序列<400>103agcctacacgctagcggatccttgacaattaatcatccggc41<210>104<211>41<212>dna<213>人工序列<400>104agtatctcaggtaccgagctctcaggcaagtttcaccatga41<210>105<211>25<212>dna<213>人工序列<400>105tcttctaataaggggatcttgaagt25<210>106<211>25<212>dna<213>人工序列<400>106cgctgttgcggatcatctccagcgt25<210>107<211>53<212>dna<213>人工序列<400>107cgcgcggatcttccagagattgggccccccggggataatgagaaacactgtca53<210>108<211>47<212>dna<213>人工序列<400>108aagcttgacgtccaggtgcctcttaagtgtccttctcctgcaagaga47<210>109<211>47<212>dna<213>人工序列<400>109cacgctagcggatccgagctcggtacctgaagacgctgcccgcgctg47<210>110<211>53<212>dna<213>人工序列<400>110cgtgcggacggcaagctgctacagctggggcccttttatgcgcactgattacc53<210>111<211>25<212>dna<213>人工序列<400>111tgaattagaactcggtacgcgcgga25<210>112<211>25<212>dna<213>人工序列<400>112tagagaataggaacttcgaactgca25<210>113<211>25<212>dna<213>人工序列<400>113aacttcgaagcagctccagcctaca25<210>114<211>25<212>dna<213>人工序列<400>114atgattacgccaagtttgcacgcct25<210>115<211>25<212>dna<213>人工序列<400>115gccagcaatccccagacaaaagcga25<210>116<211>25<212>dna<213>人工序列<400>116cgccctgagtgcttgcggcagcgtg25<210>117<211>47<212>dna<213>人工序列<400>117cgcgcggatcttccagagattgggcccccccgcttgtaaatgcccac47<210>118<211>47<212>dna<213>人工序列<400>118aagcttgacgtccaggtgcctcttaagagagagcgctaatggccgcc47<210>119<211>47<212>dna<213>人工序列<400>119cacgctagcggatccgagctcggtaccctgcggttagtcgctggttg47<210>120<211>47<212>dna<213>人工序列<400>120cgtgcggacggcaagctgctacagctgttcagtcgccaccgtttccc47<210>121<211>25<212>dna<213>人工序列<400>121tgaattagaactcggtacgcgcgga25<210>122<211>25<212>dna<213>人工序列<400>122tagagaataggaacttcgaactgca25<210>123<211>25<212>dna<213>人工序列<400>123aacttcgaagcagctccagcctaca25<210>124<211>25<212>dna<213>人工序列<400>124atgattacgccaagtttgcacgcct25<210>125<211>25<212>dna<213>人工序列<400>125gtgtttgataccgcggcgatccatg25<210>126<211>25<212>dna<213>人工序列<400>126cgccctgagtgcttgcggcagcgtg25<210>127<211>47<212>dna<213>人工序列<400>127cgcgcggatcttccagagattgggcccagctggctgaagaataaaca47<210>128<211>47<212>dna<213>人工序列<400>128aagcttgacgtccaggtgcctcttaaggtcagactcctaacttccat47<210>129<211>47<212>dna<213>人工序列<400>129cacgctagcggatccgagctcggtaccatgaaactctacaatctgaa47<210>130<211>47<212>dna<213>人工序列<400>130cgtgcggacggcaagctgctacagctgcatcatccacggctgcataa47<210>131<211>25<212>dna<213>人工序列<400>131tgaattagaactcggtacgcgcgga25<210>132<211>25<212>dna<213>人工序列<400>132tagagaataggaacttcgaactgca25<210>133<211>25<212>dna<213>人工序列<400>133aacttcgaagcagctccagcctaca25<210>134<211>25<212>dna<213>人工序列<400>134atgattacgccaagtttgcacgcct25<210>135<211>25<212>dna<213>人工序列<400>135tcatcagatgctgttcaataccgat25<210>136<211>25<212>dna<213>人工序列<400>136cgccctgagtgcttgcggcagcgtg25<210>137<211>47<212>dna<213>人工序列<400>137cgcgcggatcttccagagattgggcccgccgccgcctgtagcgataa47<210>138<211>47<212>dna<213>人工序列<400>138aagcttgacgtccaggtgcctcttaagtgcctcactcctgccgtgaa47<210>139<211>47<212>dna<213>人工序列<400>139cacgctagcggatccgagctcggtacctgactttcgccggacgtcag47<210>140<211>47<212>dna<213>人工序列<400>140cgtgcggacggcaagctgctacagctgcgtgcggttggcctggctgg47<210>141<211>25<212>dna<213>人工序列<400>141tgaattagaactcggtacgcgcgga25<210>142<211>25<212>dna<213>人工序列<400>142tagagaataggaacttcgaactgca25<210>143<211>25<212>dna<213>人工序列<400>143aacttcgaagcagctccagcctaca25<210>144<211>25<212>dna<213>人工序列<400>144atgattacgccaagtttgcacgcct25<210>145<211>25<212>dna<213>人工序列<400>145cagcttgcagtcggttaaccaggac25<210>146<211>25<212>dna<213>人工序列<400>146cgccctgagtgcttgcggcagcgtg25<210>147<211>47<212>dna<213>人工序列<400>147cgcgcggatcttccagagattgggcccttgcatttccttaaactgta47<210>148<211>47<212>dna<213>人工序列<400>148aagcttgacgtccaggtgcctcttaaggattcttcgcctttggtttg47<210>149<211>47<212>dna<213>人工序列<400>149cacgctagcggatccgagctcggtacctaccccttgcggggttgttt47<210>150<211>46<212>dna<213>人工序列<400>150cgtgcggacggcaagctgctacagcttccggcgtctcttccgaaag46<210>151<211>25<212>dna<213>人工序列<400>151tgaattagaactcggtacgcgcgga25<210>152<211>25<212>dna<213>人工序列<400>152tagagaataggaacttcgaactgca25<210>153<211>25<212>dna<213>人工序列<400>153aacttcgaagcagctccagcctaca25<210>154<211>25<212>dna<213>人工序列<400>154atgattacgccaagtttgcacgcct25<210>155<211>25<212>dna<213>人工序列<400>155gtcagcccggagagttcggcggcac25<210>156<211>25<212>dna<213>人工序列<400>156cgccctgagtgcttgcggcagcgtg25<210>157<211>36<212>dna<213>人工序列<400>157ccacagccaggatccgatgaacatgggaaaagcgtt36<210>158<211>41<212>dna<213>人工序列<400>158gcattatgcggccgcaagcttttaatctaccagcaccggat41<210>159<211>41<212>dna<213>人工序列<400>159gcgatcgctgacgtcggtaccttggttaaaaaagaaaatat41<210>160<211>41<212>dna<213>人工序列<400>160ggtttctttaccagactcgagtcaactcattctgtcgcgat41当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1