一种检测手足口病病原体核酸的试剂盒及其应用的制作方法
本发明涉及生物检测技术领域,具体涉及一种基于双扩增(rna恒温扩增+多生物素信号放大)技术检测手足口病病原体并确定是否为柯萨奇病毒a组16型或肠道病毒71型核酸的试剂盒及其应用。
背景技术:
手足口病是由部分肠道病毒感染而引起,以发热,咽喉痛,全身不适,手、足、口腔等部位皮疹或疱疹为主要特征的常见传染病。大多数手足口病患者症状轻微,并且具有自限性,但有相当一部分患者有致死性心肺功能疾病和神经系统并发症,个别重症患者病情进展迅速,甚至可导致死亡。我国手足口病疫情尤为严峻,其已成为严重影响我国居民生命和健康的公共卫生问题。引起手足口病的肠道病毒包括柯萨奇病毒a组的2、4、5、6、7、9、10、16型等,b组的1、2、3、4、5型等,肠道病毒71型及埃可病毒等,其中以ev71型和ca16型较为常见。
手足口病的实验室诊断方法有很多,目前临床上常用的方法包括病毒分离培养法、血清学分析法、病毒核酸检测方法等。病毒分离培养法是鉴定病毒感染性疾病的金标准,其关键在于根据不同时期、不同地区流行株的不同选择对病毒敏感性不同的细胞系进行病毒培养。然而该方法病毒培养过程繁琐,对病毒分离培养人员的操作技能要求较高,需要耗费大量的人力物力,无法满足流行地区大样本的检测,限制了它的临床应用。血清学分析法常用的包括中和抗体检测法、酶联免疫吸附法(elisa)、补体结合试验(cf)等。血清学分析法通过抗原抗体特异性结合反应对血清标本中病毒进行检测以作出诊断,该方法快速简便、对实验操作人员无特殊要求,对检测设备的要求也不高,适合在基层医院推广应用。但该方法存在观察时间较长、抗原漂移、出现交叉免疫反应等造成结果的不确定性,而且患者感染病毒后免疫应答产生抗体尚需一定时间,不适用于hfmd的早期诊断。病毒核酸检测相较于传统的血清学检测方法具有快速、特异、敏感的优点,在缩短检测窗口期、提高病原体检出率方面具有相当的优势。现在常用的手足口病病原体核酸检测都是基于rt-pcr的方法,这些方法需要复杂的rna提取过程,特殊的pcr扩增条件,专门的实验室和荧光定量pcr仪,而且在检测过程中极易产生污染。因此在rna恒温扩增技术的基础上,结合多生物素信号放大技术,建立单管联合检测肠道病毒通用型、柯萨奇病毒a组16型及肠道病毒71型核酸的方法具有非常重要的意义。
技术实现要素:
鉴于以上所述现有技术的缺点,本发明的目的在于提供一种基于rna恒温扩增-多生物素信号放大技术联合检测手足口病病原体并确定是否为柯萨奇病毒a组16型或肠道病毒71型核酸的试剂盒及其应用。该试剂盒通过将采集的样本经细胞裂解液裂解后释放病原体核酸,然后在逆转录酶和t7rna聚合酶的作用下,经过逆转录和转录过程,实现病原体核酸片段的扩增。扩增后的rna产物加入到包被有包被探针的微孔中进行杂交,同时加入的还有该包被探针对应的特异探针及放大探针。其中包被探针可以和每个指标的ces系列探针一端序列互补配对结合,ces系列探针的另一端可结合到rna产物上,使rna产物锚定到微孔中;每个指标的les系列探针的一端可结合到rna产物上,另一端与放大探针结合,实现信号放大过程。标记有多生物素的放大探针随后与链霉亲和素-hrp酶联物结合,最终形成包被探针-特异探针-rna扩增产物-特异探针-放大探针-链霉亲和素-hrp酶联物复合体,最后加入hrp酶化学发光底物,在化学发光仪上进行检测。实现病原体核酸的检测。因此,本发明没有复杂的rna提取过程,即使是一个水浴锅都能实现扩增反应,基于rna分子易降解的特点,本发明在实际检测中不易污染,具有灵敏度高、特异性强以及操作简单的优势,使常见的手足口病原体、柯萨奇病毒a组16型及肠道病毒71型核酸检测得到广泛应用成为一种可能。
为了实现上述目标,本发明采用如下技术方案:
第一方面,提供一种基于rna恒温扩增-多生物素信号放大技术联合检测肠道病毒通用型、柯萨奇病毒a组16型及肠道病毒71型核酸的试剂盒,包括:
1)扩增反应液:含40mmtris-hcl(ph8.0),12mmmgcl2,70mmkcl,15%dmso,5mmdtt,每种dntp各1mm,每种ntp各2mm,扩增引物各0.2μm;
本发明针对手足口病病原体核酸的三个检测指标,即肠道病毒通用型ev-u、肠道病毒71型(ev71)、柯萨奇病毒a组16型(ca16)和作为内参指标的人类18srrna基因,在rna恒温扩增中所选用的扩增对象为分布不同区域的一段保守序列,其中r引物的5’端有t7rna聚合酶启动子序列,其分布区域以及对应的两条扩增引物序列如表1所示;
表1扩增引物序列
2)rna恒温扩增中需要的三种酶:逆转录酶(如amv或m-mlv)、t7rna聚合酶和rnaseh;
3)细胞裂解液:可以裂解细胞,释放核酸;
4)放大探针fdp:一种标有生物素的核酸序列,该探针可以和特异探针les系列的一端结合,具体序列(5’-3’)为:agaaggcgtccgtctttgaggc-biotin-acccgatggataggtcggtgaa-biotin-taagcatcgtgccctttcgcag-biotin-accacgttcgcgttctcacatg;
5)特异探针:特异探针分为ces系列和les系列,本方法检测的三个指标以及内参指标所对应使用的ces系列和les系列均不相同;并且其可以同时有多条,这样可与待测核酸不同部位杂交结合,增加了检测的灵敏性;具体探针序列如表2所示;
表2特异探针序列
6)微孔板:每个微孔中包被包被探针,该包被探针可以和每个指标的ces系列探针一端序列互补配对结合,具体序列为:acatacatacgtacag;
7)hrp-链霉亲和素酶联物:标记有链霉亲和素的hrp酶联物,该酶联物可以和放大探针上生物素结合;
8)配制杂交液、洗液a(5×)、洗液b(5×)、封闭液、底物稀释液,杂交液:含0.1%sds的5×ssc;洗液a(5×):含0.5%sds的5×ssc;洗液b(5×):含0.5%sds的5×pbs;封闭液:含0.5%bsa、1×pbs;底物稀释液:50mmph8.5tris-hcl;
9)底物:鲁米诺化学发光底物(购至thermofisher,货号37075),遇到hrp酶催化后能够产生化学发光信号,被仪器检测到。
本发明提供利用上述基于rna恒温扩增-多生物素信号放大技术联合检测肠道病毒通用型、柯萨奇病毒a组16型及肠道病毒71型核酸的试剂盒检测肠道病毒通用型、柯萨奇病毒a组16型及肠道病毒71型核酸的方法,包括如下步骤:
(1)rna恒温扩增
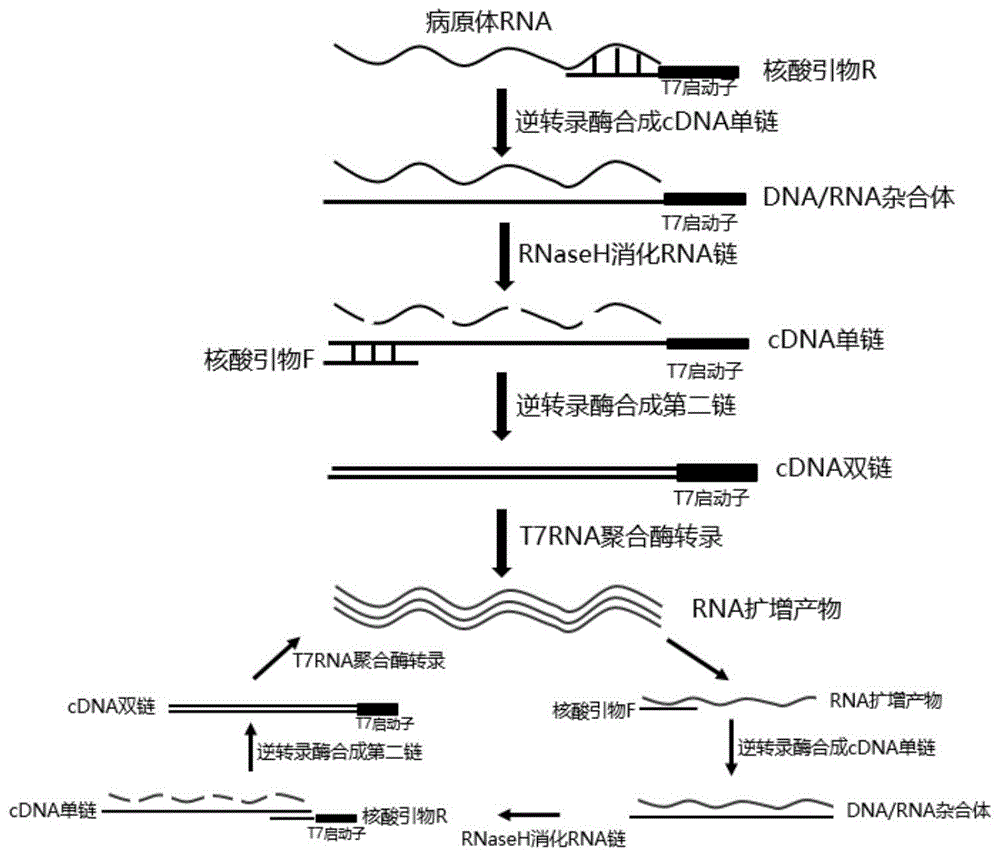
本发明检测指标有四个:肠道病毒通用型(ev-u)、肠道病毒71型(ev71)和柯萨奇病毒a组16型(ca16)以及内参人类18srrna基因。针对每个指标设计一对(f/r引物)扩增引物,其中r引物5’端带有t7rna聚合酶启动子。本发明在同一扩增管内实现各个指标核酸的扩增,具体如下:扩增时,在带有t7启动子的r引物和逆转录酶的作用下,将待测rna转化为rna:cdna杂合体;扩增酶中的rnaseh消化rna:cdna中的rna,得到单链cdna;在f引物和反转录酶的dna聚合酶功能的作用下合成第二链,形成带有t7启动子的双链dna;带有t7启动子的双链dna在t7rna聚合酶的作用下转录生成rna分子产物。转录的rna分子产物可以进入循环扩增过程,首先f引物会结合转录的rna分子产物,在逆转录酶的作用下将转录的rna转化为rna:cdna杂合体;扩增酶中的rnaseh消化rna:cdna中的rna,得到单链cdna;接着r引物会结合到单链cdna上,在反转录酶的dna聚合酶功能的作用下合成第二链,再次富集合成更多的带有t7启动子的双链dna分子,为t7rna聚合酶提供更多的转录模板,进而在t7rna聚合酶的作用下转录生成大量的rna分子产物(如图1)。
本发明设计人内参基因检测的目的是为了监测样本采集的有效性、扩增体系的有效性。当样本采集合格时样本中一定会含有人脱落细胞,在检测时一定会被检测到,样本检测阴性时内参应该为阳性,否则整个检测需要重新采样进行重测。
(2)多生物素信号放大
a,设计特异探针、放大探针和包被探针
特异探针:每个指标特异探针包括在两种:ces系列和les系列,每种探针可以设计多条。其中ces探针包含两个部分,一端可以和扩增的rna产物特异性结合,另一端可以和包被在微孔板中的包被探针集合,起到固定扩增产物rna的作用,这两个部分用4~5个t链接。每条les探针也包含两个部分,一端可以和扩增的rna产物特异性结合,另一端可以和放大探针结合,起到链接放大探针的作用,这两个部分用4~5个t链接。
放大探针:放大探针为含有多个生物素的探针,该探针可以和特异探针les的一端结合,探针上的生物素可以和hrp-链霉亲和素酶联物结合。
包被探针:包被探针被固定在微孔板中,可以和特异探针ces的一端结合,起到固定的作用。
所诉特异探针在设计时必须要求同种指标不同探针之间无交叉,ces系列同放大探针以及包被探针无交叉,以保证检测的特异性。
所诉特异探针的ces系列和les系列设计多条的目的是为了提高多生物素放大步骤的灵敏度,从而增加检测体系的灵敏度。
b,检测结果判断
扩增检测时同时设置阴性对照,即在检测样本的同时也扩增阴性质控物(细胞裂解液,购至美国signosis公司,货号cl-0001)。每管扩增产物会被均分到四个微孔板微孔中(布板图如图3),其中ev-u微孔在杂交时会加入肠道病毒通用型特异探针,ev71微孔在杂交时会加入肠道病毒71型特异探针,ca16微孔在杂交时会加入柯萨奇病毒a组16型,18s微孔在杂交时会加入内参人类18srrna基因特异探针。杂交洗涤后加入化学发光底物,并进行化学发光值测定,对样本检测指标的比值r进行计算,其中r=待测样本检测指标相对光单位(rlu)/(5×阴性质控物检测指标相对光单位(rlu)值);根据比值r对样本进行定性判断,比值r大于1.0则判断为阳性,小于等于1.0则为阴性。
结合上述原理,对本发明上述方法的工作过程介绍如下:
(1)核酸提取
采集疑似手足口患者的咽拭子样本,使用细胞裂解液裂解的方法释放病毒rna分子。
(2)rna恒温扩增
向17μl的含有肠道病毒通用型(ev-u)、肠道病毒71型(ev71)和柯萨奇病毒a组16型(ca16)和内参引物的扩增反应液中加入2μl核酸提取物,95℃加热两分钟,42℃预热2分钟,加入1μl扩增酶,42℃恒温扩增1小时。待测样本中若有对应指标核酸,扩增时会对指标rna分子进行大量扩增富集。
(3)多生物素信号放大
a,将rna恒温扩增产物与特异探针(包括ces系列探针与les系列探针)、放大探针及杂交液同时加入到微孔板中,50℃恒温孵育1小时。
扩增出的rna分子与特异探针(包括ces系列探针与les系列探针)互补配对结合。ces系列探针一端同rna分子杂交互补配对,另一端同微孔板中的包被探针结合,可以将rna分子固定在微孔板中;les系列探针一端同rna分子杂交互补配对,另一端可以与放大探针互补配对,形成ces探针-rna分子-les探针-放大探针复合物,该复合物被固定在微孔板上(如图2)。
b,用洗液a洗去未结合于微孔板上的rna分子、特异探针和放大探针。
c,加入封闭液,室温封闭1~2分钟,封闭非特异性位点。
d,将hrp-链霉亲和素酶联物加入微孔板孵育,该酶联物可以和放大探针上的生物素结合,最终形成ces探针-rna分子-les探针-放大探针-hrp-链霉亲和素酶联物,并被固定在微孔板上。
e,用洗液b洗去游离的hrp-链霉亲和素酶联物。
f,按照底物a:底物b:底物稀释液=1:1:8的比例配置底物,向每个微孔加入底物,并进行化学发光值测定。
g,计算待测样本各个标的比值r。
r=待测样本检测指标相对光单位(rlu)/(5×阴性质控物检测指标相对光单位(rlu)值)。
比值r大于1.0则判断为阳性,小于等于1.0则为阴性。
第二方面,提供上述基于rna恒温扩增-多生物素信号放大技术联合检测肠道病毒通用型、柯萨奇病毒a组16型及肠道病毒71型核酸的试剂盒在制备手足口病病原体并确定是否为柯萨奇病毒a组16型或肠道病毒71型检测试剂中的应用。
本发明和现有技术相比较,具有如下有益效果:
1、本发明通过rna恒温扩增的方法在同一管内能够同时扩增肠道病毒通用型、肠道病毒71型、柯萨奇病毒a组16型和人内参基因18srna四种指标。所扩增出的核酸产物为rna,rna在自然环境中容易降解,相比较于pcr的方法扩增出dna更容易起到防止污染的效果。rna恒温扩增是在42℃环境中进行,即使是一个水浴锅都能实现扩增反应,最大限度的降低了实验仪器的要求。
2、由于肠道病毒通用型序列变异较大,本发明在设计引物时必须保证能够扩增出所有常见的亚型流行株核酸,所选基因区域较为保守,可以对肠道病毒通用型常见流行株进行全覆盖。同时本发明在设计引物时同时经过多伦测试,保证了各单项引物扩增效率高、不同引物彼此之间无干扰,整体扩增效果好。由表10可知本发明试剂盒对16株不同来源的肠道病毒具有很好的检出能力。
3、本发明在设计时引入的特异探针ces系列和特异探针les系列有桥分子成分的作用,两种探针成功的将放大探针和rna核酸扩增片段串联结合到一块,实现指标rna核酸片段的特异检测。正是这种两套探针的使用,使得其中任何一套探针和指标核酸扩增片段杂交失败,都不能够被成功的固定在微孔板上,也就出现不了阳性检测结果,保证了检测的特异性。本发明试剂盒对表9所列的30种其它微生物的检测结果都为阴性,证明本发明试剂盒与其它微生物之间没有交叉反应。其中每套探针都可以设计两条以上,这样的设计又有利于提高试纸条的灵敏度。本发明试剂盒肠道病毒通用型检测孔对肠道病毒71型(ev71)(atccvr-1432)的最低检测限为5×10tcid50/ml、echo9(atccvr-39)的最低检测限为2.5×10tcid50/ml、coxb5(atccvr-185)的最低检测限为3.5×10tcid50/ml、ca16(atcc编号:vr-1022)的最低检测限为2×10tcid50/ml;ev71检测孔对肠道病毒71型(ev71)(atccvr-1432)的最低检测限为1×10tcid50/ml;ca16检测孔对ca16(atcc编号:vr-1022)的最低检测限为5tcid50/ml。对于681份诊断结果均与手足口病原体感染相关临床样本进行肠道病毒通用型、柯萨奇病毒a组16型或肠道病毒71型检测,本申请的试剂盒检测灵敏度、特异性高于某商品化检测手足口病原体荧光定量pcr试剂盒。
附图说明
图1rna恒温扩增原理图;
图2多生物素信号放大原理图;
图396孔微孔板布板示意图。
具体实施方式
通过以下详细说明结合附图可以进一步理解本发明的特点和优点。所提供的实施例仅是对本发明方法的说明,而不以任何方式限制本发明揭示的其余内容。实施例中所用的化学发光检测仪器为厦门天众达科技股份有限公司的化学发光免疫分析仪,型号tzd-cl-200g。
下列实施例中未注明具体条件的实验方法,通常按照常规条件,如《分子克隆:实验室指南》第3版(newyork:coldspringharborlaboratorypress,2005)中所述的条件进行。
将atcc来源的echo9(atcc编号:vr-39)、coxb5(atcc编号:vr-185)、ev71(atcc编号:vr-1432)、ca16(atcc编号:vr-1022)病毒原液进行梯度稀释液进行最低检测限确定,每个梯度的病毒稀释液重复3~5份,每份进行20次的重复检测,将具有90%~95%阳性检出率的病毒水平作为最低检测限,检出结果如下:
echo9最低检测限检测(ev-u检测孔)
表3.1不同滴度echo9的检测实验数据
表3.2echo9最低检测限实验数据
coxb5最低检测限检测(ev-u检测孔)
表4.1不同滴度coxb5的检测实验数据
表4.2coxb5最低检测限实验数据
ev71最低检测限检测(ev-u检测孔)
表5.1不同滴度ev71的检测实验数据
表5.2ev71最低检测限实验数据
ca16最低检测限检测(ev-u检测孔)
表6.1不同滴度ca16的检测实验数据
表6.2ca16最低检测限实验数据
ev71最低检测限检测(ev71检测孔)
表7.1不同滴度ev71的检测实验数据
表7.2ev71最低检测限实验数据
ca16最低检测限检测(ca16检测孔)
表8.1不同滴度ca16的检测实验数据
表8.2ca16最低检测限实验数据
最终确定本发明试剂盒的检测灵敏度为:
【实施例2】特异性验证
1,测试菌株信息及检测结果
将不同的微生物提取核酸后检测,验证本发明试剂盒引物及探针设计的特异性。相关病原体信息及检测结果如下:
表9特异性验证测试结果
2,结论
从以上数据可以看出,本发明试剂盒对这些微生物的检测结果都为阴性,证明本发明试剂盒与其他微生物之间没有交叉反应,体现试剂盒检测病原体的特异性强。
【实施例3】病原体检出力验证
使用本发明试剂盒对不同来源的肠道病毒(分别来源于美国菌种保藏中心atcc、武汉病毒所赠送、武汉大学赠送、湖北省疾控中心赠送、福建省立医院赠送)进行实验,以分析该试剂盒对不同地区来源肠道病毒的检出能力。结果如下:
表10不同病毒株检测结果
检测结果显示,本发明试剂盒对于肠道病毒通用型、ev71及ca16的不同来源株均具有很好的检出能力。
【实施例4】临床样本的验证
在湖北省预防医学科学院/疾病预防控制中心、湖北省妇幼保健院、福建省立医院共做样本681例,使用本发明试剂盒和某商品化荧光pcr试剂盒进行比对,检测结果如下:
1,肠道病毒通用型检测结果
四格表检测结果:
待考核试剂和测序方法检测结果(四格表)
采用测序的方法对不一致的37例样本进行复测,结果有27例肠道病毒阳性,包括25例本发明试剂盒检测阳性某商品化荧光pcr试剂盒检测阴性样本,2例本发明试剂盒检测阴性某商品化荧光pcr试剂盒检测阳性样本。
总体分析可知,本发明试剂盒检测样本是肠道病毒检出情况灵敏度更高,特异性更强。
2,肠道病毒71型检测结果
四格表检测结果:
待考核试剂和测序方法检测结果(四格表)
采用测序的方法对不一致的23例样本进行复测,结果有20例肠道病毒阳性,包括17例本发明试剂盒检测阳性某商品化荧光pcr试剂盒检测阴性样本,3例本发明试剂盒检测阴性某商品化荧光pcr试剂盒检测阳性样本。
总体分析可知,本发明试剂盒检测样本是肠道病毒检出情况灵敏度更高,特异性更强。
3,ca16检测结果
四格表检测结果:
待考核试剂和测序方法检测结果(四格表)
采用测序的方法对不一致的15例样本进行复测,结果有10例肠道病毒阳性,包括8例本发明试剂盒检测阳性某商品化荧光pcr试剂盒检测阴性样本,2例本发明试剂盒检测阴性某商品化荧光pcr试剂盒检测阳性样本。
总体分析可知,本发明试剂盒检测样本是肠道病毒检出情况灵敏度更高,特异性更强。
序列表
<110>武汉中帜生物科技股份有限公司
<120>一种检测手足口病病原体核酸的试剂盒及其应用
<160>30
<170>siposequencelisting1.0
<210>1
<211>21
<212>dna
<213>人工序列(artificialsequence)
<400>1
gttatccggctagctacttcg21
<210>2
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>2
taatacgactcactatagggagagctcagtagactcttcacacca45
<210>3
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>3
gaaagttccataggagatag20
<210>4
<211>43
<212>dna
<213>人工序列(artificialsequence)
<400>4
taatacgactcactatagggagaggacacagcgtgtctcaatc43
<210>5
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>5
ggaatttctttagccgtgct20
<210>6
<211>43
<212>dna
<213>人工序列(artificialsequence)
<400>6
taatacgactcactatagggagagccaagcaaacgaatctctg43
<210>7
<211>19
<212>dna
<213>人工序列(artificialsequence)
<400>7
agaaacggctaccacatcc19
<210>8
<211>41
<212>dna
<213>人工序列(artificialsequence)
<400>8
taatacgactcactatagggagacaccagacttgccctcca41
<210>9
<211>88
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(22)..(23)
<223>两个碱基之间用biotin连接
<220>
<221>misc_feature
<222>(44)..(45)
<223>两个碱基之间用biotin连接
<220>
<221>misc_feature
<222>(66)..(67)
<223>两个碱基之间用biotin连接
<400>9
agaaggcgtccgtctttgaggcacccgatggataggtcggtgaataagcatcgtgccctt60
tcgcagaccacgttcgcgttctcacatg88
<210>10
<211>40
<212>dna
<213>人工序列(artificialsequence)
<400>10
cggaaaacctagtaacaccattttctgtacgtatgtatgt40
<210>11
<211>40
<212>dna
<213>人工序列(artificialsequence)
<400>11
tgaaagttgcggagagcttcttttctgtacgtatgtatgt40
<210>12
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>12
gttcagcactcccccagtgtttttcatgagaacgcgaacgtggt44
<210>13
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>13
agatcaggccgatgagtcacttttcatgagaacgcgaacgtggt44
<210>14
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>14
cgcattccccacgggcgaccttttcatgagaacgcgaacgtggt44
<210>15
<211>40
<212>dna
<213>人工序列(artificialsequence)
<400>15
cgtgagcagagccctcactcttttctgtacgtatgtatgt40
<210>16
<211>40
<212>dna
<213>人工序列(artificialsequence)
<400>16
gctctaccagcacccacaggttttctgtacgtatgtatgt40
<210>17
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>17
ccagaacacacaggtgagcattttcatgagaacgcgaacgtggt44
<210>18
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>18
gtcatcgactggatacgggcttttcatgagaacgcgaacgtggt44
<210>19
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>19
aaggttccagcactccaagcttttcatgagaacgcgaacgtggt44
<210>20
<211>40
<212>dna
<213>人工序列(artificialsequence)
<400>20
atgcccaccacgggtacacattttctgtacgtatgtatgt40
<210>21
<211>40
<212>dna
<213>人工序列(artificialsequence)
<400>21
acagatggttatgttaattgttttctgtacgtatgtatgt40
<210>22
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>22
ggacattgatttaatgggatttttcatgagaacgcgaacgtggt44
<210>23
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>23
gctcagctgcggcggaaatgttttcatgagaacgcgaacgtggt44
<210>24
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>24
gagttatttacctacatgcgttttcatgagaacgcgaacgtggt44
<210>25
<211>36
<212>dna
<213>人工序列(artificialsequence)
<400>25
aaggaaggcagcaggcttttctgtacgtatgtatgt36
<210>26
<211>36
<212>dna
<213>人工序列(artificialsequence)
<400>26
gcgcaaattacccactttttctgtacgattgtatgt36
<210>27
<211>40
<212>dna
<213>人工序列(artificialsequence)
<400>27
cccgacccggggaggtttttcatgagaacgcgaacgtggt40
<210>28
<211>40
<212>dna
<213>人工序列(artificialsequence)
<400>28
agtgacgaaaaataacttttcatgagaacgcgaacgtggt40
<210>29
<211>40
<212>dna
<213>人工序列(artificialsequence)
<400>29
aatacaggactctttcttttcatgagaacgcgaacgtggt40
<210>30
<211>16
<212>dna
<213>人工序列(artificialsequence)
<400>30
acatacatacgtacag16
- 还没有人留言评论。精彩留言会获得点赞!