从多发性硬化患者分离的质粒编码的复制蛋白的晶体结构的制作方法
本发明涉及通过从多发性硬化患者分离的质粒编码的复制蛋白的晶体形式,从它们获得的晶体结构信息,制备这样的晶体形式的方法,其用于识别和/或设计所述复制蛋白的抑制剂的用途,以及用于识别、优化和设计应具有与复制蛋白相互作用的能力或抑制复制蛋白的能力的化合物的方法。
背景技术:
牛肉和牛奶的摄食被认为是发展人退行性和恶性疾病,例如结肠和乳腺癌症的一种危险因素(1-3)。实际上,流行病学的数据表明这些癌症与来自欧亚的欧洲野牛衍生的牛的牛产品的摄食存在关联(4-7)。牛肉和牛奶因子(bmmf)是环状的,单链的附加体dna(<3kb),其已经在牛肉和牛奶产品中检测出。bmmf被认为在人恶性和退行性疾病中发挥作用。bmmf编码在人细胞中活跃转录和翻译的复制起始蛋白(rep)。因此,bmmf可能代表了此类疾病可能的病原因子(8-10)。然而,也从具有多发性硬化的患者中分离出bmmf并且研究表明这些是此种疾病可能的感染原(10-13)。
典型地,bmmf编码自主质粒反式作用复制起始蛋白,称为“rep”。rep结合在dna上的复制起点处(称为on)并且在大部分情况下,包括一组重复的dna元件(称为重复子),其存在于大多数bmmf中(5)。各种质粒(包括环状编码rep的单链(cress)dna病毒)的复制,还需要rep与特定dna序列的结合(14)。在原核生物中,rep在保持质粒拷贝数中起核心作用,如对于在大肠杆菌(escherichiacoli)中的f质粒的报道(15)。这种调控对于复制质粒衍生的、噬菌体样的或病毒样的dna基因组也是至关重要的(16)。rep对于人中耐多药细菌的复制是必不可少的(17)并且研究已经表明rep在可传播性淀粉样蛋白病中发挥作用(18-20)。
已经很好地证明了rep的x射线晶体结构,并且已经描述了对于自主复制的结构基础(24-27)。rep由两个翼状螺旋结构域(称为wh1和wh2)组成,它们本质上是融合的n末端和c末端蛋白。rep在单体和二聚体形式之间转换,取决于特定功能和与dna的结合(28)。涉及两种结构域的大的结构变化补充了这些寡聚形式。结构转换需要rep上的某些α-螺旋和β-链再折叠和/或移位(26)。在二聚体形式中,rep起阻遏物作用,其中wh2结合至每个操纵元件dna重复,以及wh1发挥作用以形成二聚化界面。在单体形式中,rep起复制起始子作用,其中wh1经历大的结构移动,即二聚体分解,因此允许wh1结合至重复子末尾,而wh2结合至相反的重复子末尾。
最近,从患有多发性硬化患者的脑样品中分离出附加体环状dna(分离物msbi1.176,登录号lk931491.1)(11)。msbi1.176rep呈现出与sphinx-1.76编码的rep(genbankadr65123.1和hq444404.1)98%的氨基酸同一性,其分离自可传播性脑病相关病原体的培养物和脑制剂(21)。此外,有迹象表明,基于sphinx-1.76特异性抗体在神经细胞(gt1-细胞系)和小鼠cns、仓鼠cns和人胶质母细胞瘤的脑样品中检测到了sphinx.1.76编码的rep(22)。基于msbi1.176rep抗原的血清学示出了对于健康人血液供体的阳性免疫应答并且表明对于这些病原体可能的预暴露(23)。因此,解密bmmf在人恶性和退行性疾病中的功能变得越来越重要。
发明目的
对于解密bmmf在人恶性和退行性疾病中的功能,具有msbi1.176rep蛋白的晶体结构是非常有帮助的,然而到目前为止都失败了。此种晶体结构还有助于识别和/或设计所述复制蛋白的抑制剂,以及用于识别、优化和设计具有与复制蛋白相互作用的能力或抑制复制蛋白能力的化合物的方法。
因此,本发明的目的是提供msbi1.176rep蛋白的晶体结构坐标,获得msbi1.176rep蛋白晶体形式的方法,以及用于基于晶体结构和晶体结构坐标来设计、识别和优化作为msbi1.176rep蛋白抑制剂的化合物的方法。
具体实施方式
本发明的目的通过独立权利要求的教导实现。本发明的进一步有利的特征、方面和细节从本申请的从属权利要求、说明书、附图和实施例中是显而易见的。
因此,本发明涉及如在权利要求1中表征的msbi1.176rep蛋白的晶体形式。优选的实施方式是从属权利要求的主题。
msbi1rep源全长复制蛋白
genbank:cds63398.1
(在复制感受态附加体dnamsbi1.176中编码)
embl登录号dna序列:lk931491.1
来源:患多发性硬化的人脑
>msbi1.176推定的复制起始蛋白
msdlivkdnalmnasynlalveqrlillaiiearetgkginandpltvhassyinqfnverhtayqalkdackdlfarqfsyqekrergrinitsrwvsqigymddtatveiifapavvplitrleeqftqydieqisglssayavrmyellicwrstgktpiieldefrkrigvldteytrtdnlkmrvielalkqinehtditasyeqhkkgrvitgfsfkfkhkkqnsdktpknsdssprivkhsqiptnivkqpenakmsdlehrasrvtgeimrnrlsdrfkqgdesaidmmkriqseiitdaiadqweskleefgvvf
在产生本发明的实验期间,将msbi1.176wh1结构域(残基2-133)的x射线晶体结构解析至
在表1中总结对msbi1.176wh1蛋白结构进行的数据收集和细化统计:
表1.msbi1.176wh1蛋白结构的数据收集和细化统计。
a括号中的值是用于最高分辨率壳层(shell)。
数据库搜索密切相关的结构和序列显示msbi1.176wh1具有以下格外低的氨基酸同一性:分别与丁香假单胞菌(pseudomonassyringae)repawh1(drepa,pdbid1hkq)与大肠杆菌repe融合wh1-2(repe54,1rep)的28%和17%的氨基酸同一性(24,25)。与drepa相似,msbi1.176wh1也被折叠成为复制惰性二聚体,而repe54(wh1-2构建体)以单体起始形式被晶体化。msbi1.176wh1和drepawh1的叠加示出了这两个结构域是结构上相似的
发明人还观察到包括α1-α2-α5的msbi1.176wh1区域在取向上与drepa相似,具有典型的α1-α2弯曲因此形成了v型结构(图3c)。形成了wh2的接头的drepa的该区域还包含了疏水的七口袋,其通常包含一些亮氨酸残基(例如,drepa:leu12、leu19和leu26;repe:leu24、leu31和leu39)。msbi1.176wh1疏水口袋还包括三个亮氨酸残基或由三个亮氨酸残基组成,即,leu11、leu18和/或ile25,它们与drepa中的位置相似。并不奇怪,在msbi1.176wh1疏水口袋中不存在水分子(图3b)。
总体上,许多msbi1.176wh1结构特征相对于其他已知的二聚体rep结构是保守的(24-27)。然而,在不同的结构中已经观察到环的移动以及不同的α-螺旋和β-链。当与drepa的β2-β3-发夹相比时,在msbi1.176的情况下的β2-β3-发夹移位了大约
drepa结构域先前的建模分析表明,在α2、β2、β3和相邻的环上的六个碱性残基(drepa编号:lys74、arg81、arg91、arg93、lys62和arg78)可能接在dna主链的小沟(groove)之后(24)。在msbi1.176wh1结构中,在此区域中也发现了六个碱性残基。这些被认为是代表了包括或由以下组成的dna相互作用位点:lys69、lys73(位于α2)、lys85(β2)、arg90(β3)、arg78和/或arg96(在相邻的环上)。在dna相互作用位点中,优选累计正电荷的氨基酸以造成与dna的负电荷的静电相互作用。尽管对于msbi1.176wh1lys73a/b链、lys85a/b链和arg90a链侧链的电子密度是弱的,这些残基中的两个(arg78和arg96)位于等效的drepa位置并且被认为与dna分子相互作用(24)。尽管msbi1wh1在β2链中具有三个氨基酸的插入,其延伸了片层,但msbi1.176wh1的β2-β3片层取向的功能不明显。大概,当与等效的drepa残基arg81和a1rg91相比,这种插入使在β2-β3片层上的msbi1.176wh1的lys85(β2)和arg90(β3)优雅地移位了。从另一角度看,msbi1.176的β2-β3片层是相当令人满意的,而drepa的β2-β3发夹在相反的方向钩住(图2)。
msbi1.176wh1结构的阐明为更好地理解在不同组的bmmfdna中rep蛋白之间结构特征中可以如何变化迈出了关键的一步。从患有多发性硬化患者分离的此种msbi1.176wh1蛋白与原核生物的rep结构非常相似的发现具有重要的意义。总的来说,此种新的结构信息支持了可以抑制rep寡聚物性质的新药品靶标的开发和设计。根据本发明,使用x射线晶体学确定从多发性硬化人脑样品分离的在bmmf(msbi1.176)上编码的repwh1结构域,至分辨率
进一步,本发明能够建立用于识别抑制剂msbi1.176的方法,以及用于制备msbi1.176rep蛋白的晶体形式和它们的晶体结构信息的方法。数据使得基于使用此类结构数据的合理药品设计成为可能。
本发明还提供了识别和/或设计msbi1.176rep蛋白的抑制剂的可能性,以及涉及msbi1.176rep蛋白的晶体形式,从晶体形式获得的晶体结构信息,制备此类晶体形式的方法,其用于识别和/或设计msbi1.176rep蛋白活性的抑制剂的用途,以及通过这些方法识别的msbi1.176rep蛋白的这些抑制剂的诊断和/或药物用途。
术语“晶体形式”或“晶体结构”(其互换使用)是指有或没有结合至msbi1.176rep蛋白的表面活性剂和/或核酸(尤其是:dna)的情况下的msbi1.176rep蛋白的晶体形式。
如本文所用,术语“单元晶胞”是指只用平移操作就可以生成晶体的最小重复单元。单元晶胞是体积的最小单元,其包含晶体的所有结构和对称信息,并且通过平移可以在所有空间中再现图形。结构信息因此是图形(原子)加上所有周围的空间,并且对称信息意指镜像、滑动、轴线和反转中心。平移是指沿着晶胞边缘的晶胞边缘长度移动。不对称单元是可以只使用经晶体学对称性允许的对称操作来旋转和平移以生成一个单元晶胞的最小单元。不对称单元可以是多聚体蛋白的一个分子或一个亚单元,但也可以是多于一个。因此不对称单元是包含所有的结构信息并且通过采用对称操作可以再现单元晶胞的体积最小单元。单元晶胞的形状由聚集组成群的对称元素约束。对于空间群,七种晶格类型可能是:三斜晶系、单斜晶系、正交晶系、四方晶系、六方晶系、斜方晶系(rhombic)、立方晶系(cubic)。在晶体学中,空间群也被称作晶体学群或费多洛夫(fedorov)群。单元晶胞的边缘限定了一组单位向量轴a、b和c,其具有的单元晶胞的尺寸分别为它们的长度a、b和c。这些向量不需要成直角,并且两个轴之间的角记为:在bc轴之间的α,在ac轴之间的β,以及在ab轴之间的γ。不同形状的出现取决于发生在三个边缘(a、b和c)的长度和三个角(α、β以及γ)的值上的限制。
在本发明的另一种优选的实施方式中,msbi1.176rep蛋白的晶体形式包含或进一步包含疏水口袋,其中,疏水口袋包含氨基酸leu11、leu18和ile25。
msbi1.176rep蛋白的潜在抑制剂可以结合至dna相互作用位点,以便降低rep蛋白的活性。因此,用于设计、识别和/或优化可能具有结合至msbi1.176rep蛋白能力的化合物的方法可以基于晶体结构坐标。
msbi1.176rep蛋白的潜在抑制剂可以结合至rep寡聚化界面,调整基于结构的功能和蛋白活性/定位/稳定性/修饰。
msbi1.176rep蛋白的潜在抑制剂可以结合至还未知的蛋白相互作用界面,调整基于结构的功能和蛋白活性/定位/稳定性/修饰。
msbi1.176rep蛋白的潜在抑制剂可以结合至结构上相关的原核rep(例如,来自鲍曼不动杆菌(acinetobacterbaumannii)种)以基于dna结合、寡聚化和/或额外的蛋白相互作用界面的空间位阻来降低来自不同来源的此类rep蛋白的活性。
如本文所公开的msbi1.176rep蛋白的晶体形式可以通过以下结晶化方法获得。因此本发明涉及用于结晶化msbi1.176rep蛋白的方法,其包括步骤:
(a)制备在结晶化试剂中的重组制备的msbi1.176wh1结构域的溶液,该结晶化试剂优选包含至少一种无机盐和至少一种沉淀剂;
(b)通过蒸气扩散结晶化所述msbi1.176wh1结构域。
以0.01mm至2m之间、优选0.1mm至1m、更优选0.1m至0.5m之间、更优选约0.2m的量使用至少一种无机盐。如果使用多于一种无机盐,上文提及的浓度是指所有的无机盐一起的浓度,并且不是在无机盐混合物中使用的每种单一盐的浓度。
优选地,无机盐选自包括以下的组或由以下组成的组:氯化铵、硫酸铵、乙酸铵、氟化铵、溴化铵、碘化铵、氮化铵、氯化钙、乙酸钙、乙酸镁、甲酸镁、硝酸镁、乙酸钾、溴化钾、氟化钾、氯化钾、碘化钾、硝酸钾、乙酸钠、氢氧化钠、溴化钠、氟化钠、碘化钠、硝酸钠、硫酸钠、氯化钠、氯化锌、硫酸锌、乙酸锌。优选的无机盐是乙酸镁。
理论上,至少一种沉淀剂与蛋白溶质争夺水,因此导致蛋白的过饱和。晶体通常仅由过饱和状态生长,并且因此它们可以由沉淀物生长。盐、聚合物和有机溶剂是适合的沉淀剂,以沉淀剂或沉淀剂的混合物相对于缓冲沉淀溶液的总重量计,以以下的量使用:按重量计5%至按重量计50%之间、优选按重量计10%至按重量计40%之间、和更优选在按重量计15%至35%之间,以及最优选按重量计20%至按重量计30%之间。
在步骤(b)的缓冲沉淀溶液中使用的沉淀剂,可以优选选自包括以下的组或由以下组成的组:2-甲基-2,4戊二醇、甘油、聚乙二醇(peg)、丙氧化季戊四醇(pentaerythritolpropoxylate)、乙氧化季戊四醇(pentaerythritolethoxylate)、聚丙烯酸钠、己二醇、异丙醇、乙醇、叔丁醇、二氧六环、乙烯亚胺聚合物、乙二醇(etyleneglycol)、丙二醇、聚丙烯酸、聚乙烯吡咯烷酮、2-乙氧基乙醇或其混合物。作为沉淀剂最优选的是peg,其优选具有peg200至peg20,000范围的分子量,更优选具有peg1,000至peg18,000范围的分子量,还更优选具有peg3,000至peg15,000范围的分子量。peg是非常优选的沉淀剂,其优选以按重量计缓冲沉淀溶液的20-30%的量使用。
在最优选的实际方式中,结晶化缓冲液包含0.2m乙酸镁和20%peg3350。
在优选的实施方式中,使用悬滴或坐滴方法结晶化。“悬滴蒸气扩散”技术是用于结晶化大分子的最流行方法。蒸气扩散的原理是直接的。使由样品和结晶化试剂的混合物组成的液滴与试剂的液体贮液蒸气平衡。典型地,液滴包含比贮液更低的试剂浓度。为了达到平衡,水蒸气离开液滴,并且最终进入贮液。由于水离开液滴,样品发生了在过饱和方向上的增加。由于水离开液滴进入贮液,样品和试剂二者浓度增加。当液滴中的试剂浓度与贮液中的试剂浓度大约相同时,达到平衡。
本发明的进一步重要的方面涉及msbi1.176rep蛋白的晶体形式用于对于识别、优化和/或设计msbi1.176rep蛋白抑制剂的计算机预测模型的用途。知道氨基酸原子在dna相互作用位点中的确切位置,提供了以下的可能性:设计适合的抑制剂、识别合适的抑制剂,例如来自化合物文库的,或通过提高抑制潜力来优化已知的适合的抑制剂。可以用本领域熟知的标准的基于计算机的方法和软件程序进行设计、识别和优化适合的抑制剂。各种商购的软件程序可用于在基于计算机系统中进行分析并比较数据。本领域的技术人员将容易识别哪些对于进行计算机分析可用的算法或执行软件包可以利用于或适应于基于计算机系统的用途。靶标结构基序或靶标基序是指是指任何合理选择的序列或序列的组合,其中,一个或多个序列是基于靶标基序折叠形成的三维构型或电子密度图选择的。存在本领域已知的各种靶标基序。可以将用于输入和输出方式的各种结构格式用于本发明的基于计算机的系统中输入和输出信息。可以将各种比较方式用于将靶标序列或靶标基序与数据存储手段进行比较,以识别结构基序或解释部分源自原子坐标/x射线衍射数据的电子密度图。本领域的技术人员可以容易意识到可以使用的公开可用的计算机建模程序的任何一种。可以用于查看、分析、设计和/或建模蛋白的适合的软件包括alchemytm、labvisiontm、sybyltm、molcaddtm、leapfrogtm、matchmaker20tm、genefoldtm和siteltm(获得自triposinc.,圣路易斯,密苏里州)、quantatm、macromodeltm和grasptm、univisiontm、chem3dtm以及proteinexperttm。
因此在进一步的方面,本发明涉及用于通过以下设计、识别和优化msbi1.176rep蛋白抑制剂的方法:采用晶体形式和晶体形式相关的结构坐标或至少一种dna相互作用位点,以便借助于基于计算机的方法或软件程序来设计、识别和优化抑制剂。
可以将原子坐标/x射线衍射数据用于创建可以随后用于设计化合物的分子模型的物理模型,该化合物应该具有抑制以下和/或与以下相互作用的能力或特性:确定的dna相互作用位点或者其他结构或功能结构域或亚结构域,诸如疏水口袋和/或dna相互作用位点附近的口袋。替代地,络合物的原子坐标/x射线衍射数据可以表示为计算机可读介质上的原子模型输出数据,其可以用于计算机建模系统来计算预期抑制以下和/或与以下相互作用的不同分子:确定的位点,或者其他结构或功能结构域或亚结构域。例如,数据的计算机分析允许计算出msbi1.176rep蛋白和化合物的三维相互作用,以确定化合物结合至特定的一个或多个结构域或一个或多个亚结构域,或者改变特定的一个或多个结构域或一个或多个亚结构域的构型。然后可以用适当的方式对由物理或计算机模型的分析确定的化合物进行合成和生物学活性的测试。
如果抑制剂是从化合物文库中识别的或如果已知的抑制剂是通过理论化学修饰来优化的,期望测试实际化合物以便确定抑制效应并继续优化过程。因此,优选用于识别和/或优化化合物的上文方法进一步包括以下步骤
(a)获得识别的或优化的化合物;以及
(b)将识别的或优化的化合物与msbi1.176rep蛋白接触,以便确定对msbi1.176rep蛋白的抑制作用。
没有必要使用表1中列出的所有结构坐标。因此可以使用的只有疏水口袋和/或dna相互作用位点的结构坐标。
在本申请中,使用x射线晶体学确定从多发性硬化人脑样品分离的bmmf(msbi1.176)上编码的repwh1结构域,至分辨率
将以下的缩写用于本文提到的一般和改变的氨基酸。
氨基酸
ala丙氨酸
arg精氨酸
asn天冬酰胺
asp天冬氨酸(天冬氨酸盐)
cys半胱氨酸
gin谷氨酰胺
glu谷氨酸(谷氨酸盐)
gly甘氨酸
his组氨酸
ile异亮氨酸
leu亮氨酸
lys赖氨酸
met甲硫氨酸
phe苯丙氨酸
pro脯氨酸
ser丝氨酸
thr苏氨酸
trp色氨酸
tyr酪氨酸
val缬氨酸
附图说明
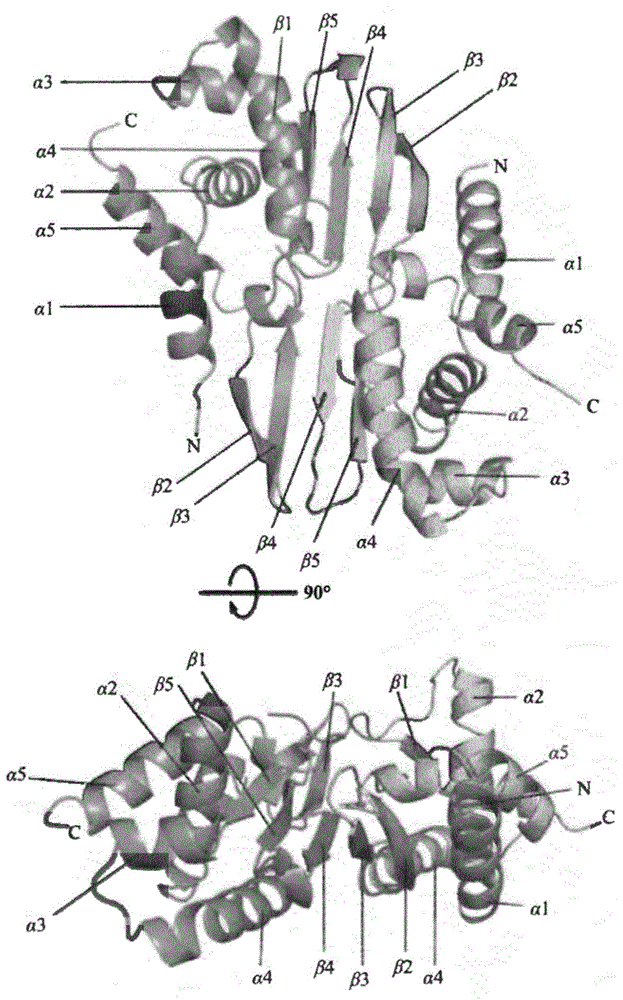
图1.msbi1.176wh1二聚体的x射线晶体结构。
将msbi1.176wh1原聚体a链着色为青色以及b链为橙色。一个原聚体包括五个α-螺旋(α1-α5)和五个β-链(β1-β5)。二聚体界面涉及两条链(β4-β3)。
图2.与紧密匹配的原核生物repawh1的结构比较。
msbi1.176wh1和repawh1具有28%的氨基酸同一性。与repa(灰色)的叠加示出了这两种wh1二聚体是高度相似的,其具有
图3.msbi1.176wh1的结构相似性的msbi1.176wh1。
(a)五个β-片层(β1-β5-β4-β3-β2)示出了在msbi1.176wh1(青色和橙色)和repawhi(灰色)中的主链相互作用。β链通过许多主链氢键(虚线)固定,与repa相似,包括二聚体界面(β4-β3)。
(b)包含α1-α2-α5的区域与在repa中的那个取向相似。这个区域产生了v型结构并且α5是wh2结构域的接头区。疏水口袋还包含三个亮氨酸残基,即leu11,leu18和ile25,它们在repa中位置相似(数据未示出)。
图4.msbi1.176wh1的β2-β3发夹在具有pdb代码1hkq(repa)和2z9o(repe)的原核rep蛋白上的叠加。
当与等效repa发夹相比时,msbi1.176wh1的β2-β3发夹移位了大约
包括以下实施例以展示本发明优选的实施方式。本领域技术人员应当理解,在以下实施例中公开的技术代表由发明人发现的技术在本发明的实践中发挥良好作用,并且因此可以被认为构建其实践的优选模式。鉴于此描述,本发明的各种方面的的修改的和可替代的实施方式对于本领域的技术人员是显而易见的。
因此,本说明书仅应被解释为说明性的并且旨在向本领域的技术人员教导实施本发明的一般方法。应当理解,将在本文示出和描述的发明形式视为实施方式的实施例。元素和材料可以替代本文说明和描述的那些,部分和过程可以调换,并且本发明的某些特征可以独立利用,在受益于本发明的说明书之后,所有这些对于本领域技术人员将是显而易见的。
实施例
实施例1:获得msbi1wh1晶体的方法
从患有多发性硬化患者的脑样品中分离msbi1.176dna(lk931491.1)(11)。如人诺如病毒突出结构域的先前描述(29),在e.coli中表达msbi1wh1结构域(残基1-135)并纯化。简要地,将密码子优化的wh1克隆至修饰的表达载体pmal-c2x(geneart)中,并转化至bl21细胞中用于蛋白表达。转化的细胞在补充有100μg/ml氨苄青霉素的lb培养基中于37℃生长4小时。在22℃下用在od600为0.7下iptg(0.75mm)诱导表达18h。将细胞通过在6000rpm离心15min收集并在冰上通过超声处理破碎。有his标签的融合mbsm蛋白首先从ni柱(qiagen)中纯化,在具有10mm咪唑的凝胶过滤缓冲液(gfb:25mmtris-hcl和300mmnacl)中透析,并用hrv-3c蛋白酶(novagen)于4℃消化过夜。随后将裂解的msbi1wh1再次应用至ni柱中以分离并收集裂解的蛋白,并在gfb中于4℃透析过夜。将msbi1wh1蛋白通过尺寸排阻色谱法进一步纯化,浓缩至5mg/ml并于4℃储存在gfb中。msbi1wh1晶体使用悬滴蒸气扩散方法在蛋白样品和母液(0.2m乙酸镁和20%peg3350)的1:1混合物中于18℃生长~6-10天。在数据收集之前,将msbi1wh1晶体转移至包含具有40%peg3350母液的冷冻保护剂中,随后在液氮中快速冻结。
实施例2:x射线衍射
msbi1wh1结构域的x射线衍射数据是在欧洲同步辐射装置(europeansynchrotronradiationfacility,esrf)的光束线id23-1和id30b上收集的。对于使用原生硫的单波长异常衍射(s-sad)实验,在配备有dectris像素阵列检测器pilatus-6m的光束线id23-1上以
使用s-sad收集了一些数据集用于进一步加工(33)。使用如在hkl2map(34)中执行的shelxc/d/e管道进行s-sad定相规程。在shelxc中进行了用于确定子结构的一千个试验。使用
实施例3:dna结合的建模
repa结构域先前的建模分析表明,在α2、β2、β3以及相邻的环上的六个碱性残基(repa编号:lys74、arg81、arg91、arg93、lys62和arg78)可能接在dna主链的小沟之后(24)。在msbi1.176wh1结构中,在此区域中也发现了六个碱性残基,即lys69、lys73(二者都位于α4上)、lys85(β2)、arg90(β3)、arg78和arg96(二者在相邻的环上)。尽管msbi1.176wh1lys73a/b链、lys85a/b链以及arg90a链侧链的电子密度是弱的,这些残基中的两个(arg78和arg96)位于等效repa位置并且被认为与dna分子相互作用(24)。尽管msbi1.176wh1在β2链上具有三个氨基酸的插入,其延伸了片层,但msbi1.176wh1的β2-β3片层取向的功能不明显。大概,当与等效的repa残基arg81和arg91相比,这种插入使在β2-β3片层上的msbi1.176wh1的lys85(β2)和arg90(β3)优雅地移位了。从另一角度看,msbi1.176的β2-β3片层是相当令人满意的,而repa的β2-β3发夹在相反的方向钩住。
使用clustalw(genetyx)生成msbi1.176(lk931491.1)和repa(pdbid1hkq)的上述氨基酸序列比对。使用晶体结构示出和确定二级结构元素。同一的和同源的残基分别在填充的背景和方框中突出显示。大概,dna分子将沿着二聚体界面并且可能与此区域中的六个碱性残基,即lys69(α4)、lys73(α4)、arg78、lys85(β2)、arg90(β3)和及arg96相互作用。将被认为参与dna相互作用的repa的碱性氨基酸残基用倒三角形
参考文献
1.chands,etal.(2011)redandprocessedmeatandcolorectalcancerincidence:meta-analysisofprospectivestudies.plosone6(6):e20456.
2.corpetde(2011)redmeatandcoloncancer:shouldwebecomevegetarians,orcanwemakemeatsafer?meatscience89(3):310-316.
3.huxleyrr,etal.(2009)theimpactofdietaryandlifestyleriskfactorsonriskofcolorectalcancer:aquantitativeoverviewoftheepidemiologicalevidence.internationaljournalofcancer125(1):171-180.
4.zurhausenh&devilliersem(2015)dairycattleserumandmilkfactorscontributingtotheriskofcolonandbreastcancers.internationaljournalofcancer137(4):959-967.
5.zurhausenh,bundt,&devilliersem(2017)infectiousagentsinbovineredmeatandmilkandtheirpotentialroleincancerandotherchronicdiseases.currenttopicsinmicrobiologyandimmunology407:83-116.
6.zurhausenh(2012)redmeatconsumptionandcancer:reasonstosuspectinvolvementofbovineinfectiousfactorsincolorectalcancer.internationaljournalofcancer130(11):2475-2483.
7.zurhausenh(2015)riskfactors:whatdobreastandcrccancersandmshaveincommon?natrevclinoncol12(10):569-570.
8.funkm,etal.(2014)isolationofprotein-associatedcirculardnafromhealthycattleserum.genomeannouncements2(4).
9.falidak,eilebrechts,gunstk,zurhausenh,&devilliersem(2017)isolationoftwovirus-likecirculardnasfromcommerciallyavailablemilksamples.genomeannouncements5(17).
10.zurhausenh,bundt,&devillierse-m(inpress)specificnutritionalinfectionsearlyinlifeasriskfactorsforhumancolonandbreastcancersseveraldecadeslater.internationaljournalofcancer.
11.whitleyc,etal.(2014)novelreplication-competentcirculardnamoleculesfromhealthycattleserumandmilkandmultiplesclerosis-affectedhumanbraintissue.genomeannouncements2(4).
12.lambertoi,gunstk,mullerh,zurhausenh,&devilliersem(2014)mycovirus-likednavirussequencesfromcattleserumandhumanbrainandserumsamplesfrommultiplesclerosispatients.genomeannouncements2(4).
13.gunstk,zurhausenh,&devilliersem(2014)isolationofbacterialplasmid-relatedreplication-associatedcirculardnafromaserumsampleofamultiplesclerosispatient.genomeannouncements2(4).
14.kornberga,andbaker,t.(1992)dnareplication.universitysciencebooks2nded.
15.klinebc(1985)areviewofmini-fplasmidmaintenance.plasmid14(1):1-16.
16.ruiz-masoja,etal.(2015)plasmidrolling-circlereplication.microbiologyspectrum3(1):plas-0035-2014.
17.schumacherma,etal.(2014)mechanismofstaphylococcalmultiresistanceplasmidreplicationoriginassemblybytherepaprotein.proceedingsofthenationalacademyofsciencesoftheunitedstatesofamerica111(25):9121-9126.
18.molina-garcial,gasset-rosaf,alamomm,delaespinasm,&giraldor(2018)addressingintracellularamyloidosisinbacteriawithrepa-wh1,aprion-likeprotein.methodsinmolecularbiology1779:289-312.
19.giraldor,etal.(2016)repa-wh1prionoid:cluesfrombacteriaonfactorsgoverningphasetransitionsinamyloidogenesis.prion10(1):41-49.
20.giraldor,moreno-diazdelaespinas,fernandez-tresguerresme,&gasset-rosaf(2011)repa-wh1prionoid:asyntheticamyloidproteinopathyinaminimalisthost.prion5(2):60-64.
21.manuelidisl(2011)nucleaseresistantcirculardnascopurifywithinfectivityinscrapieandcjd.journalofneurovirology17(2):131-145.
22.yehyh,gunasekharanv,&manuelidisl(2017)aprokaryoticviralsequenceisexpressedandconservedinmammalianbrain.proceedingsofthenationalacademyofsciencesoftheunitedstatesofamerica114(27):7118-7123.
23.eilebrechts,etal.(2018)expressionandreplicationofvirus-likecirculardnainhumancells.scientificreports8(1):2851.
24.giraldor,fernandez-torneroc,evanspr,diaz-orejasr,&romeroa(2003)aconformationalswitchbetweentranscriptionalrepressionandreplicationinitiationintherepadimerizationdomain.naturestructuralbiology10(7):565-571.
25.komorih,etal.(1999)crystalstructureofaprokaryoticreplicationinitiatorproteinboundtodnaat2.6aresolution.theembojournal18(17):4597-4607.
26.nakamuraa,wadac,&mikik(2007)structuralbasisforregulationofbifunctionalrolesinreplicationinitiatorprotein.proceedingsofthenationalacademyofsciencesoftheunitedstatesofamerica104(47):18484-18489.
27.swanmk,bastiad,&daviesc(2006)crystalstructureofpiinitiatorprotein-iteroncomplexofplasmidr6k:implicationsforinitiationofplasmiddnareplication.proceedingsofthenationalacademyofsciencesoftheunitedstatesofamerica103(49):18481-18486.
28.forestkt&filutowiczms(2003)remodelingofreplicationinitiatorproteins.naturestructuralbiology10(7):496-498.
29.hansmangs,etal.(2011)crystalstructuresofgii.10andgii.12norovirusprotrudingdomainsincomplexwithhisto-bloodgroupantigensrevealdetailsforapotentialsiteofvulnerability.journalofvirology85(13):6687-6701.
30.bourenkovgp&popovan(2010)optimizationofdatacollectiontakingradiationdamageintoaccount.actacrystallographica.sectiond,biologicalcrystallography66(pt4):409-419.
31.gabadinhoj,etal.(2010)mxcube:asynchrotronbeamlinecontrolenvironmentcustomizedformacromolecularcrystallographyexperiments.journalofsynchrotronradiation17(5):700-707.
32.kabschw(2010)xds.actacryst.d66:125-132.
33.liuq,etal.(2012)structuresfromanomalousdiffractionofnativebiologicalmacromolecules.science336(6084):1033-1037.
34.sheldrickgm(2010)experimentalphasingwithshelxc/d/e:combiningchaintracingwithdensitymodification.actacrystallographica.sectiond,biologicalcrystallography66(pt4):479-485.
35.langerg,cohensx,lamzinvs,&perrakisa(2008)automatedmacromolecularmodelbuildingforx-raycrystallographyusingarp/warpversion7.natureprotocols3(7):1171-1179.
36.emsleyplb,scottwg,cowtank.(2010)featuresanddevelopmentofcoot.actacrystallographicasectiond:biologicalcrystallography.66(4):486-501.
37.adamspd,etal.(2010)phenix:acomprehensivepython-basedsystemformacromolecularstructuresolution.actacrystallographicasectiond66(2):213-221。
序列表
<110>德国癌症研究公共权益基金会(deutscheskrebsforschungszentrum)
<120>从多发性硬化患者分离的质粒编码的复制蛋白的晶体结构
<130>k3469pct
<150>ep18206532.6
<151>2018-11-15
<160>2
<170>bissap1.3.6
<210>1
<211>324
<212>prt
<213>sphinx
<220>
<223>msbi1.76蛋白
<400>1
metseraspleuilevallysaspasnalaleumetasnalasertyr
151015
asnleualaleuvalgluglnargleuileleuleualaileileglu
202530
alaarggluthrglylysglyileasnalaasnaspproleuthrval
354045
hisalasersertyrileasnglnpheasnvalgluarghisthrala
505560
tyrglnalaleulysaspalacyslysaspleuphealaargglnphe
65707580
sertyrglnglulysarggluargglyargileasnilethrserarg
859095
trpvalserglnileglytyrmetaspaspthralathrvalgluile
100105110
ilephealaproalavalvalproleuilethrargleugluglugln
115120125
phethrglntyraspilegluglnileserglyleuserseralatyr
130135140
alavalargmettyrgluleuleuilecystrpargserthrglylys
145150155160
thrproileilegluleuaspgluphearglysargileglyvalleu
165170175
aspthrglutyrthrargthraspasnleulysmetargvalileglu
180185190
leualaleulysglnileasngluhisthraspilethralasertyr
195200205
gluglnhislyslysglyargvalilethrglypheserphelysphe
210215220
lyshislyslysglnasnserasplysthrprolysasnseraspser
225230235240
serproargilevallyshisserglnileprothrasnilevallys
245250255
glnprogluasnalalysmetseraspleugluhisargalaserarg
260265270
valthrglygluilemetargasnargleuseraspargphelysgln
275280285
glyaspgluseralaileaspmetmetlysargileglnsergluile
290295300
ilethraspalailealaaspglntrpgluserlysleuglugluphe
305310315320
glyvalvalphe
<210>2
<211>131
<212>prt
<213>sphinx
<220>
<223>repa结构域
<400>2
metvalaspasnlysvalthrglnserasnlysleuilegluserser
151015
histhrleuthrleuasnglulysargleuvalleucysalaalaser
202530
leuileaspserarglysproleuprolysaspglytyrleuthrile
354045
argalaaspthrphealagluvalpheglyileaspvallyshisala
505560
tyralaalaleuaspaspalaalathrlysleupheasnargaspile
65707580
argargtyrvallysglylysvalvalgluargmetargtrpvalphe
859095
hisvallystyrarggluglyglycysvalgluleuglypheserpro
100105110
thrileileprohisleuthrmetleuhislysgluphethrsertyr
115120125
glnleulys
130
- 还没有人留言评论。精彩留言会获得点赞!