荧光生物传感器,其制备方法及其用途与流程
本发明涉及荧光传感器制备技术领域,具体涉及荧光生物传感器,其制备方法及其用途。
背景技术:
在过去的几十年,生物分子模板的荧光无机金属纳米颗粒具有获得了大量的关注,在这些生物大分子中,DNA因其独特的纳米结构,优良的可编程特性,包括杂环氮原子的丰富功能基团,氨基组和磷酸基团,它可以与特定的金属离子结合,并提供金属纳米粒子的成核位点,成为了最佳的候选者。在过去的十年中,DNA为模板不同的荧光金属纳米颗粒已经得到了深入的关注,如金纳米粒子和银纳米粒子。最近几年,一种新的荧光纳米材料–DNA模板的铜纳米粒子类(DNA-Cu NPs)作为荧光光探针,因其具有良好的生物相容性、成本低,易于制备,毒性低,优异的荧光性能等优点,已经逐渐的应用在构建荧光生物传感上。
聚胸腺嘧啶单链(poly T)为模板的制备发荧光的Cu NPs最早由王课题组最早发现。随后利用poly T为模板的作为发荧光的Cu NPs探针,运用在生物传感器上检测各种不同的生物物质不断被报道。与此同时增强Cu NPs的荧光效率成为关注的课题。尽管不久前,卢的小组设计了一种由多聚T环和一个随机的双链序列DNA序列为茎新型发夹DNA结构,作为Cu NPs形成的模板,以此来提高其荧光效率。但是我们仍然不知道什么状态下的聚T构象可以直接影响Cu NPs的荧光信号。因此,基于聚T单链DNA模板,设计可控和高效的荧光Cu NPs仍然是一个挑战。
技术实现要素:
针对以上现有技术问题,本发明的目的在于提供一种基于poly T为基础的新型的“糖果”(CS)构型的DNA结构,可以增强Cu NPs的荧光效率,并运用此构造设计了一种无标记的荧光恢复的生物传感器用于检测特殊序列的DNA片段(目标DNA)。根据不同浓度的目标DNA,构建线性关系,实现了对目标DNA基因灵敏性、特异性的检测。具体技术方案如下:
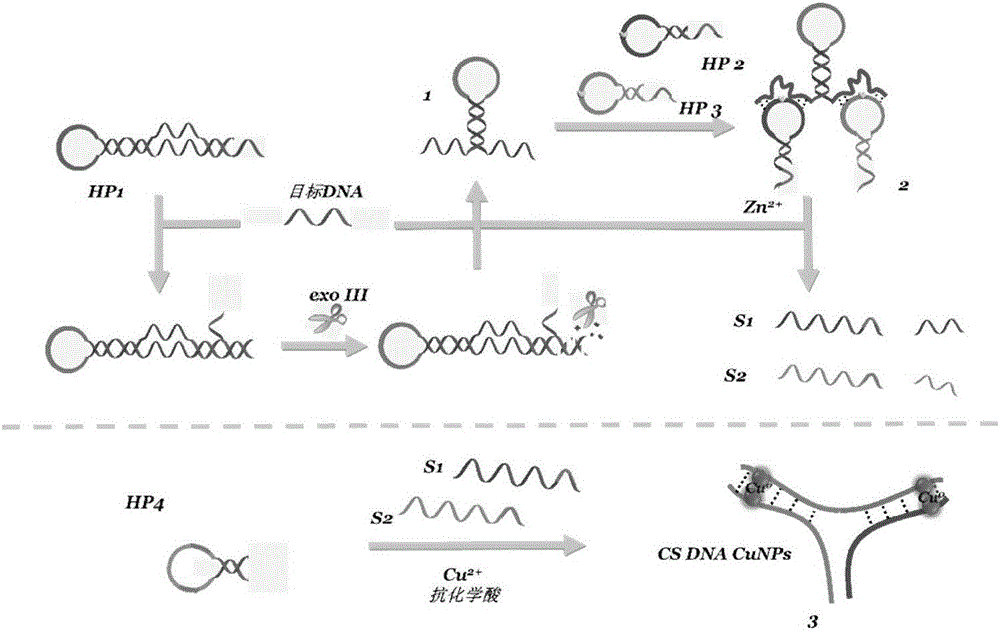
荧光生物传感器的制备方法,包括如下步骤:
(1)DNA用缓冲溶液稀释到并储存;
(2)发夹DNA包括HP1,HP2,HP3和HP4使用前加热,冷却到室温,使之形成发夹结构;
(3)发夹HP1和目标DNA在缓冲溶液中混合,然后在反应的溶液中加入exo III,溶液中的DNA位点被exo III切割完成后,使exo III失活;
(4)释放出DNAzyme,释放的DNAzyme与HP3和HP2杂交,加入Zn2+溶液,Zn2+切割发夹DNA上的rA位点。
进一步包括如下步骤:
(5)S1和S2被释放与HP4杂交;
(6)点亮Cu NPs。
进一步地,步骤(1)中,所有的DNA全部用10mMMOPS(3-(N-吗啉基)丙磺酸)PH=7.6的缓冲溶液,稀释到100μM,储存在4℃中至少24小时后可用。
进一步地,步骤(2)中,HP1,HP2,HP3和HP4使用前加热到90℃,10分钟,避免聚集,然后保持恒定摇速2h,让其慢慢冷却到室温,使之形成发夹结构。
进一步地,步骤(3)中,包括如下步骤:
(3-1)在切割酶(exo III)辅助的目标放大体系中:1μM的发夹HP1和目标DNA在缓冲溶液中,室温下混合30分钟;
(3-2)在反应的溶液中加入12U exo III,加热到37℃,35分钟;
(3-3)溶液中的DNA位点被exo III切割完成;
(3-4)将反应溶液加热到85℃,时间为15分钟,使exo III失活。
进一步地,步骤(4)中,释放出DNAzyme(1),释放的DNAzyme会与HP3和HP2杂交,加入1nM的Zn2+溶液,Zn2+切割发夹DNA上的rA位点,时间40分钟。
进一步地,步骤(5)中,S1和S2将被释放与HP4杂交,形成双通“糖果”结构形成;和/或,步骤(6)中,加入16μL,100mM的抗化学酸钠和8μL,10mΜ的硫酸铜,点亮Cu NPs,终体积是260μL。
进一步地,步骤(1)之前还包括如下步骤:CS DNA Cu NPs和poly T Cu NPs的制备:
a.1DNA和2DNA用10mM MOPS缓冲溶液相互混合在离心管A里,各自的终浓度分别是500Nm;
b.将混合溶液放在室温下,20分钟;
c.DNA用10mM MOPS缓冲溶液稀释在离心管B里,终浓度分别是1μM;
d.在离心管A和B中均加入16μL100mM的抗坏血酸和8μL 10mΜ的CuSO4,终体积均为260μL。
荧光生物传感器,采用上述制备方法制备得到。
上述荧光生物传感器的用途,作为荧光探针用于检测目标DNA。
与目前现有技术相比,本发明发现了一种新型的“糖果”(CS)造型的DNA结构,相较于传统的聚胸腺嘧啶单链(poly T)为模板的制备发荧光的铜纳米粒子(Cu NPs),该方法增强了Cu NPs的荧光信号。基于此事实,我们设计了一种新型的无标记的荧光恢复的生物传感器,该传感器利用了双循环自动放大手段,能够特异性的检测一段特殊序列的DNA。
提供基于聚胸腺嘧啶单链(poly T)DNA为基础的“糖果”(CS)DNA结构,相较于传统的poly T为模板的制备Cu NPs,CS DNA制备的Cu NPs的荧光信号明显增强。可应用于目标DNA的检测。本发明公开了一种荧光生物传感器的制备方法及其应用,基于CS DNA结构,我们设计了一种新型的无标记的荧光恢复的生物传感器,该传感器利用了外切酶(exo III)和金属离子切割的双循环自动放大手段,特异性的检测特殊序列的DNA。该传感器具有很好的灵敏度,而且具有高的选择性,价格低廉和操作简单的特点。
附图说明
图1A为实施例1制备CS DNA的Cu NPs与携带poly T为模板的Cu NPs的荧光对比示意图;
图1B为基于CS DNA可以增强Cu NPs的荧光的现象设计的检测目标DNA的生物传感器;
图2A为CS DNA Cu NPs和poly T Cu NPs的荧光激发和发射光谱图;
图2B1为CS DNA Cu NPs和poly T Cu NPs的高分辨透射电镜;
图2B2为各自的粒子大小分部统计数;
图2C为CS DNA Cu NPs和poly T Cu NPs的紫外吸收光谱图;
图2D为CS DNA Cu NPs和poly T Cu NPs的圆二色谱图;
图2E为CS DNA Cu NPs和poly T Cu NPs的的凝胶电泳;
图3A为对CS DNA Cu NPs头尾DNA互补的碱基配对数对荧光强度的影响的研究;
图3B对CS DNA Cu NPs和poly T Cu NPs在不同PH的缓冲溶液中荧光强度的研究;
图3C对CS DNA Cu NPs和poly T Cu NPs在不同温度培养下的荧光强度的研究;
图4A为基于CS DNA Cu NPs设计的传感器的可行性图;
图4B为该传感器对目标DNA的检测的线性原图;
图4C为该传感器对目标DNA的检测的线性方程图;
图5为荧光强度图。
具体实施方式
下面根据附图对本发明进行详细描述,其为本发明多种实施方式中的一种优选实施例。
在一个优选实施例中,荧光生物传感器的制备方法,包括以下步骤:
(1)、将1DNA溶液和2DNA溶液杂交形成CS DNA与3DNA(携带poly T),分别加入硫酸铜和抗化学酸以此点亮Cu NPs,将两者的荧光强度进行对比;
(2)、将步骤(1)制备的CS DNA用于荧光生物传感器的设计中:发夹DNA(HP1)的尾端可以和目标DNA碱基互补配对,加入剪切酶exoIII可以特异性剪切互补配对的发夹DNA,从而释放目标DNA和DNA模拟酶1,释放的目标DNA可以不断进行循环作用。在发夹DNA(HP2和HP3)的加入下,模拟酶的自由茎端可以和HP2和HP3配对形成结构2,Zn2+的加入可以对存在HP2和HP3上的rA碱基进行切割,从而释放出S2和S3。此体系引入发夹HP4DNA后,S2和S3的存在可以打开发夹HP4DNA,从而形成双通的“糖果”DNA结构,最终加入硫酸铜和抗化学酸可以点亮Cu NPs。Cu NPs作为荧光探针,可制备无标记的荧光生物传感器用于检测目标DNA。
步骤(1)中CS DNA Cu NPs和poly T Cu NPs的制备:首先,1DNA和2DNA用10mM MOPS缓冲溶液相互混合在离心管A里,各自的终浓度分别是500nM,将混合溶液放在室温下,20分钟。3DNA用10mM MOPS缓冲溶液稀释在离心管B里,终浓度分别是1μM。然后在离心管A和B中均加入16μL100mM的抗坏血酸和8μL 10mΜ的CuSO4,终体积均为260μL。
步骤(2)具体为:(1)所有的DNA全部用10mM MOPS(3-(N-吗啉基)丙磺酸)PH=7.6的缓冲溶液,稀释到100μM,储存在4℃中至少24小时后可用。实验过程中所用的稀释液都为MOPS缓冲溶液。(2)HP1,HP2,HP3和HP4使用前加热到90℃,10分钟,避免聚集。然后保持恒定摇速2h,让其慢慢冷却到室温,使之形成发夹结构。(3)在切割酶(exo III)辅助的目标放大体系中:1μM的发夹HP1和目标DNA在缓冲溶液中,室温下混合30分钟。然后在反应的溶液中加入12U exo III,加热到37℃,35分钟。溶液中的DNA位点被exo III切割完成后,将反应溶液加热到85℃,时间为15分钟,使exo III失活。(4)以上的步骤完成后,释放出DNAzyme(1),释放的DNAzyme会与HP3和HP2杂交,加入1nM的Zn2+溶液,Zn2+切割发夹DNA上的rA位点,时间40分钟。(5)以上的步骤完成后,S1和S2将被释放与HP4杂交,形成双通“糖果”结构形成。(6)最后,加入16μL,100mM的抗化学酸钠和8μL,10mΜ的硫酸铜,点亮Cu NPs,终体积是260μL。
检测目标DNA的应用,应用方法具体为:发现一种改进传统的poly T为模板的新型CS DNA为模板制备Cu NPs,并利用该信号为荧光探针制备的荧光生物传感器,不同浓度的目标DNA的加入致使荧光信号强度不同,构建线性关系,实现了对荧光生物传感器灵敏性、特异性的检测。
在另一个优选实施例中,可以采用如下方案:荧光生物传感器的制备方法,包括以下步骤:所有的DNA全部用10mMMOPS(3-(N-吗啉基)丙磺酸)PH=7.6的缓冲溶液,稀释到100μM,储存在4℃中至少24小时后可用。实验过程中所用的稀释液都为MOPS缓冲溶液。HP1,HP2,HP3和HP4使用前加热到90℃,10分钟,避免聚集。然后保持恒定摇速2h,让其慢慢冷却到室温,使之形成发夹结构。在切割酶(exo III)辅助的目标放大体系中:1μM的发夹HP1和目标DNA在缓冲溶液中,室温下混合30分钟。然后在反应的溶液中加入12U exo III,加热到37℃,35分钟。溶液中的DNA位点被exo III切割完成后,将反应溶液加热到85℃,时间为15分钟,使exo III失活。以上的步骤完成后,释放出DNAzyme(1),释放的DNAzyme会与HP3和HP2杂交,加入1nM的Zn2+溶液,Zn2+切割发夹DNA上的rA位点,时间40分钟。以上的步骤完成后,S1和S2将被释放与HP4杂交,形成双通“糖果”结构形成。最后,加入16μL,100mM的抗化学酸钠和8μL,10mΜ的硫酸铜,点亮Cu NPs,终体积是260μL。
上面结合附图对本发明进行了示例性描述,显然本发明具体实现并不受上述方式的限制,只要采用了本发明的方法构思和技术方案进行的各种改进,或未经改进直接应用于其它场合的,均在本发明的保护范围之内。
- 还没有人留言评论。精彩留言会获得点赞!