二代测序短序列快速比对分析方法及装置与流程
本发明属于生物信息工程领域,涉及生物信息技术和计算机应用技术,具体地说,涉及DNA序列二代测序的短序列快速比对分析方法。
背景技术:
DNA测序在解读物种生命的基因序列编码中,起着最基础和最广泛的作用。早在发现DNA双螺旋之初,就有人报道过DAN的测序技术,只是流程过于复杂。随后不久,在1977年Sanger发明了末端终止测序法,具有里程碑的意义。迄今随着生物信息技术科学的发展,Sanger测序法已经无法满足研究的需要,于是成本更低,通量更高,速度更快的第二代测序技术应运而生。它的核心思想是边合成边测序。DNA序列比对是将测序得到的DNA短序列(reads)与参考基因组进行比对分析,常用以分析物种相似性和同源性,也可以通过计算机技术进行基因信息的挖掘和分析。
目前,针对二代测序序列比对的技术主要有两个方向的发展:一是基于设计hash算法映射构建hash表库思路,这种技术优点比对速度快,准确率极高,但占用内存和CUP较高。该技术实现的软件代表有SOAP,MAQ等。二是基于BWT转换算法构建后缀树查询数据结构的技术,该技术先对基因组序列循环移位,然后排序,利用BWT压缩并建立索引。在比对时,利用查找和回溯来定位序列的位置信息。该技术需要事先构建索引文件分步骤操作,比对速度也不及接近常数平均时间的hash表结构。它通常能够实现所有测序数据的常规比对,但相对hash数据结构,树结构要占用庞大的内存资源,并且查询比对的速度远不及平均常数时间的hash表库。
技术实现要素:
有鉴于此,本发明提供一种二代测序短序列快速比对分析方法及装置,用以解决测序数据的比对效率低以及内存占用高的问题。
一方面,本发明实施例提出一种二代测序短序列快速比对分析方法,包括:
S1、获取测序得到的DNA短序列,并采用第一hash算法和第二hash算法分别映射编码所述DNA短序列,分别得到第一索引和第二索引;
S2、基于预设的index查询库、所述第一索引和第二索引将所述DNA短序列和参考基因组进行比对,其中,所述index查询库由单元结构体数组构成,每个单元结构体包含有value值和index2值,所述value值为对应的组成所述参考基因组的K-mer片段的位置信息摘要的压缩,所述index2为所述K-mer片段的第二hash算法的映射结果,存储每个所述单元结构体数组索引偏移量为对应的K-mer片段的第一hash算法的映射结果index1,K为片段序列长度;
S3、根据比对的结果,若比对上,则获取与对应的DNA短序列比对上的K-mer片段的value值,并根据所述与对应的DNA短序列比对上的K-mer片段的value值确定出所述对应的DNA短序列所在染色体号和所在染色体上的位点。
优选地,所述第一hash算法为XDDHash算法,所述第二hash算法为MWFHash算法。
优选地,所述S2,包括:
对于每一个DNA短序列,查找首地址为该DNA短序列的第一索引的内存块,若查找到首地址为该DNA短序列的第一索引的内存块,则判断该内存块的数据存储位置是否为1个;
若为1个,则确定出该DNA短序列与该内存块的数据存储位置处存储的结构体对应的K-mer片段比对上。
优选地,所述方法还包括:
若不为1个,则从所述首地址开始,采用移位线性探测的方法获取当前地址处存储的结构体的index2值,判断该index2值是否与该DNA短序列的第二索引相同;
若该index2值与该DNA短序列的第二索引相同,则确定出该DNA短序列与该当前地址处存储的结构体对应的K-mer片段比对上。
优选地,所述index查询库的装填因子为0.607。
优选地,在所述S2之前,所述方法还包括:
S40、依据先验数据模型,对所述参考基因组的24条染色体按照历史比对上的DNA序列的数量从多到少进行排序;
S41、根据排序的结果,按照从前到后的顺序,对当前染色体进行滑窗式截取,得到连续的K-mer片段,并标记各片段所在的位置信息;
S42、去除包含简并碱基的K-mer片段和重复的K-mer片段;
S43、对于得到的每一个K-mer片段,对该K-mer片段分别进行第一hash算法映射和第二hash算法映射,分别得到index1值和index2值,并计算该K-mer片段的位置信息摘要的压缩value值,生成包含所述value值和index2值的结构体;
S44、将index1值作为索引构建所述index查询库。
优选地,在所述DNA短序列和参考基因组进行比对时,采用多线程技术实现正链和反链短序列的并行比对。
另一方面,本发明实施例提出一种二代测序短序列快速比对分析装置,包括:
映射单元,用于获取测序得到的DNA短序列,并采用第一hash算法和第二hash算法分别映射编码所述DNA短序列,分别得到第一索引和第二索引;
比对单元,用于基于预设的index查询库、所述第一索引和第二索引将所述DNA短序列和参考基因组进行比对,其中,所述index查询库由单元结构体数组构成,每个单元结构体包含有value值和index2值,所述value值为对应的组成所述参考基因组的K-mer片段的位置信息摘要的压缩,所述index2为所述K-mer片段的第二hash算法的映射结果,存储每个所述单元结构体的数组索引偏移量为对应的K-mer片段的第一hash算法的映射结果index1,K为片段序列长度;
计算单元,用于根据比对的结果,若比对上,则获取与对应的DNA短序列比对上的K-mer片段的value值,并根据所述与对应的DNA短序列比对上的K-mer片段的value值确定出所述对应的DNA短序列所在染色体号和所在染色体上的位点。
本发明具有如下有益效果:
1、采用了hash算法,比对速度快,接近常数时间O(1),提高项目产品时间效率;
2、采用二次hash映射技术,无需直接保存reads序列,节省大量内存资源,提高项目产品资源效益;
3、采用先验数据模型重排hash索引,预先比对出现频率高的reads序列,加速比对;
4、采用多线技术比对并行执行比对任务,缩短了比对时间。
附图说明
图1为本发明二代测序短序列快速比对分析方法一实施例的流程示意图;
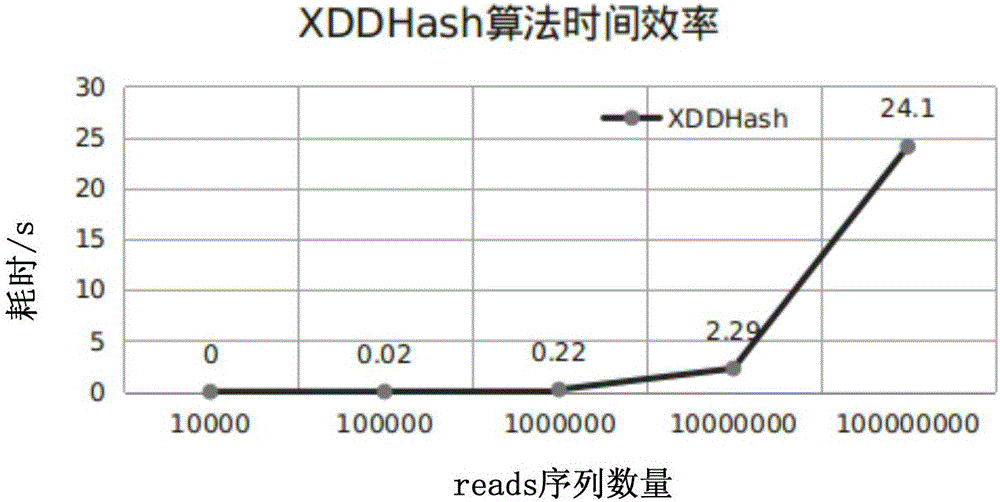
图2为本发明用来做一次hash函数映射的XDDHash算法的时间性能图;
图3为本发明用来做一次hash函数映射的XDDHash算法的冲突量性能图;
图4为本发明用来做二次hash函数映射的MWFHash算法的时间性能图;
图5为本发明用来做二次hash函数映射的MWFHash算法的冲突量性能图;
图6为本发明构建的hash表的装填因子和平均探测次数的关系图;
图7为本发明方法在设计成软件程序产品后,比对样本的数据量和花费时间的关系图;
图8为本发明二代测序短序列快速比对分析装置一实施例的结构示意图。
具体实施方式
为使本发明实施例的目的、技术方案和优点更加清楚,下面将结合本发明实施例中的附图,对本发明实施例中的技术方案进行清楚地描述,显然,所描述的实施例是本发明一部分实施例,而不是全部的实施例。基于本发明中的实施例,本领域普通技术人员在没有做出创造性劳动前提下所获得的所有其他实施例,都属于本发明保护的范围。
参看图1,本实施例公开一种二代测序短序列快速比对分析方法,包括:
S1、获取测序得到的DNA短序列,并采用第一hash算法和第二hash算法分别映射编码所述DNA短序列,分别得到第一索引和第二索引;
S2、基于预设的index查询库、所述第一索引和第二索引将所述DNA短序列和参考基因组进行比对,其中,所述index查询库由单元结构体数组构成,每个单元结构体包含有value值和index2值,所述value值为对应的组成所述参考基因组的K-mer片段的位置信息摘要的压缩,所述index2为所述K-mer片段的第二hash算法的映射结果,存储每个所述单元结构体的数组索引偏移量为对应的K-mer片段的第一hash算法的映射结果index1,K为片段序列长度;
S3、根据比对的结果,若比对上,则获取与对应的DNA短序列比对上的K-mer片段的value值,并根据所述与对应的DNA短序列比对上的K-mer片段的value值确定出所述对应的DNA短序列所在染色体号和所在染色体上的位点。
查询时序列位置信息的获取如下:
内存索引index获取:通过第一hash算法和第二hash算法,将样本中序列信息转换为第一索引和第二索引,取得内存中value值。
序列位置获取:
DNA短序列所在染色体号K-mer_number=value%26(求模运算,即求余);
DNA短序列所在染色体上位点K-mer_location=value/26(求整除)。
本发明实施例中,采用二次hash算法,一次hash的映射结果用来查询定位,二次hash的映射结果用来冲突筛选定位,能够加快比对速度,且无须直接保存庞大的reads序列信息即可统计出比对结果,降低对内存资源的浪费。
可选地,在本发明二代测序短序列快速比对分析方法的另一实施例中,所述第一hash算法为XDDHash算法,所述第二hash算法为MWFHash算法。
一次映射的XDDhash压缩算法,用来索引关键字信息摘要。其算法伪函数如下:
hash[i+1]=(hash[i]*seed+str[i])^0x00000000FFFFFFFF(i=0,1,2,…,N-2)
其中,hash[0]设定为63,乘法因子种子seed取33,str[i]为reads序列的第i+1个碱基字符的ASCⅡ值,N为待查询比对reads序列的长度。P^0x00000000FFFFFFFF表示对P取其低32位二进制值。做循环递归运算直到求得hash[N-1]即为第一索引。
经理论和程序检测的实践验证,该算法具有较好的性能:冲突量少,压缩映射速度快。其性能统计见说明书附图图2和3,其中图2为用来做一次hash函数映射的XDDHash算法的时间性能图,反映XDDHash算法随着reads数量变化耗时关系图,图3为用来做一次hash函数映射的XDDHash算法的冲突量性能图,反映XDDHash算法随着reads数量变化产生的冲突量的关系图。
二次映射的MWFhash压缩算法,用来分离索引冲突值,对奇偶位采用不同的移位因子算法如下,
奇数位索引算法(i为奇数值时):
hash[i+1]=(hash[i]*seedodd+i*str[i])^0x00000000FFFFFFFF
偶数位索引算法(i为偶数值时):
hash[i+1]=(hash[i]*seedeven+i*str[i])^0x00000000FFFFFFFF
其中,hash[0]设定为0,奇数移位因子种子seedodd取值5,偶数移位因子种子seedeven取值7。做循环递归运算得到hash[N-1]值,即为第二索引。
经理论和程序检测的实践验证,该算法具有较好的性能:冲突量少,压缩映射速度快。其性能统计见说明书附图图4和5,图4为用来做二次hash函数映射的MWFHash算法的时间性能图,反映MWFHash算法随着reads数量变化所耗时的关系图,图5为用来做二次hash函数映射的MWFHash算法的冲突量性能图,反映MWFHash算法随着reads数量变化产生的冲突量的关系图。
本发明实施例中,在hash算法编码映射reads序列时,直接编码取其低32位二进制值简化了算法时间复杂度。
可选地,在本发明二代测序短序列快速比对分析方法的另一实施例中,所述S2,包括:
对于每一个DNA短序列,查找首地址为该DNA短序列的第一索引的内存块,若查找到首地址为该DNA短序列的第一索引的内存块,则判断该内存块的数据存储位置是否为1个;
若为1个,则确定出该DNA短序列与该内存块的数据存储位置处存储的结构体对应的K-mer片段比对上。
可选地,在本发明二代测序短序列快速比对分析方法的另一实施例中,还包括:
若不为1个,则从所述首地址开始,采用移位线性探测的方法获取当前地址处存储的结构体的index2值,判断该index2值是否与该DNA短序列的第二索引相同;
若该index2值与该DNA短序列的第二索引相同,则确定出该DNA短序列与该当前地址处存储的结构体对应的K-mer片段比对上。
本发明实施例中,依据样本短序列hash映射值的特点,利用改进过的线性探测的方法解决hash值的冲突问题。具体是说,在索引向下移位探测时,只需要索引自加运算即可,降低了比对搜索时的时间复杂度。
可选地,在本发明二代测序短序列快速比对分析方法的另一实施例中,所述index查询库的装填因子为0.607。
所述index查询库实质是hash表,Hash表装填因子K的选取:
装填因子:hash表中实际存放的记录数与hash表大小的比值,是衡量hash表性能的一个重要指标。其值越大,hash值冲突数越多,导致hash表的查询速度降低。其值过小,浪费内存资源。我们基于XDDHash算法和MWFHash算法设计程序测试统计装填因子和平均探测次数(次数越多,耗时越多)之间的关系图见图6所示,它反映所构建表的查询比对的效率性能,本发明设计的装填因子K为0.607,可以观察到其平均查找次数仅在3次左右,其接近常数时间查找。
可选地,在本发明二代测序短序列快速比对分析方法的另一实施例中,在所述S2之前,还包括:
S40、依据先验数据模型,对所述参考基因组的24条染色体按照历史比对上的DNA序列的数量从多到少进行排序;
S41、根据排序的结果,按照从前到后的顺序,对当前染色体进行滑窗式截取,得到连续的K-mer片段,并标记各片段所在的位置信息;
S42、去除包含简并碱基的K-mer片段和重复的K-mer片段;
在具体实施例中,包含简并碱基的K-mer片段在参考基因组hg19代表未知序列,构建index库时不可用,去掉后也可以节省内存占用。
S43、对于得到的每一个K-mer片段,对该K-mer片段分别进行第一hash算法映射和第二hash算法映射,分别得到index1值和index2值,并计算该K-mer片段的位置信息摘要的压缩value值,生成包含所述value值和index2值的结构体;
S44、将index1值作为索引构建所述index查询库。
value值是K-mer片段序列的位置信息摘要的压缩,压缩函数如下:
Value=K-mer_number*26+K-mer_location,
其中,K-mer_number为K-mer片段序列所在染色体号,K-mer_location为K-mer片段序列所在染色体上的位点。
Index2值和value值存储于hash结构体中:
Typedef struct Node{
unsigned int value;
unsigned int index2;
}HashType
这样构建的hash表的伪表达式可以简单写为:
Hash[index1]=HashType element;
就实现在内存index1处存储HashType类型的值。
本发明实施例中,依据先验数据模型,优先排序出现频率高的短序列,以提高查询速度,这个具体方法是说,在处理有重复冲突的hash索引时,优先查找实际测序样本中出现率高的reads序列,减少索引移位搜索次数,避开不必要的时间开销。
可选地,在本发明二代测序短序列快速比对分析方法的另一实施例中,在所述DNA短序列和参考基因组进行比对时,采用多线程技术实现正链和反链短序列的并行比对。
在具体应用中,可以实时监视存储测序得到的DNA序列的下机目录,只要有新下机样本数据,就进行比对分析出结果数据。
实施例1基于二代测序短序列快速比对分析方法应用于无创产前检测的数据分析
本发明实现无创产前检测的模块流程首先是利用perl语言和shell命令预处理人类参考基因组hg19.fa,处理成index查询库所需的格式,包括三列:
第一列:36-mer DNA序列片段;
第二列:片段所在染色体号;
第三列:片段所在染色体上的位点。
然后其核心方法是基于C语言算法和数据结构来实现。
本发明设计过程和运行需要硬件和软件环境:Linux系统;3个核心以上;35内存以上;Linux平台下C库;Gcc编译器;Gdb调试软件。
输入样本文件格式:
Illumina测序平台NIPT原始测序数据,为fastq格式,每4行为一条read信息:
第一行:以@开头,标示一条序列,包括测序仪名字,标记位置,单/双端测序以及过滤情况和引物接头等相关信息描述;
第二行:reads序列;
第三行:以‘+’开头,后面是序列标示符、描述信息,或者什么也不加;
第四行:是reads质量信息,和第二行的序列相对应。
运行以及输出结果
(1)利用linux下命令nohup和&转后台,可以一直扫描下机目录,只要有新批次样本数据出现就可以出结果。运行命令:
nohup./hashtb_pth.c&;
(2)输入样本文件数据:
我们将7.4M大小样本文件Human_A1600169-R169_good_1.fq数据和Human_A1600171-R171_good_1.fq存入下机目录。输入文件:
Human_A1600169-R169_good_11.fq、Human_A1600169-R169_good_12.fq、Human_A1600169-R169_good_13.fq、Human_A1600169-R169_good_14.fq、Human_A1600169-R169_good_15.fq、Human_A1600169-R169_good_1.fq和Human_A1600171-R171_good_1.fq,其中,前6个是同一样本数据的copy,用以检测程序的稳定性,最后一个用来检测程序处理差异样本的能力。
(3)比对结果
结果目录中产生的文件:
Human_A1600169-R169_good_1、Human_A1600169-R169_good_11、Human_A1600169-R169_good_12、Human_A1600169-R169_good_13、Human_A1600169-R169_good_14、Human_A1600169-R169_good_15和Human_A1600171-R171_good_1,
样本Human_A1600169-R169_good_1.fq的6个copy的结果数据完全一致,反映基于本发明方法实现的软件在即使连续处理多个样本数据时是稳定可靠的。
由结果数据可以简单计算出:
样本唯一比对率为:72.772%,
样本比对率为:74.443%。
对差异样本样本Human_A1600171-R171_good_1.fq结果数据后4行的结果做简单计算:
样本唯一比对率:71.531%,
样本比对率:73.487%。
结果和Human_A1600169-R169_good_1.fq不同,说明软件程序能够即时连续处理同一批次目录下的不同样本数据。
(4)比对用时
处理7个样本用时:77.850秒。
统计样本reads量和比对时间之间的关系图,见图7,图7为本发明方法在设计成软件程序产品后,比对样本的数据量和花费时间的关系图,这是由真实样本实例测试得出,它综合反映了本发明方法由C编程语言实现后的效果。它是采用无创产前基因检测样本的原始数据做的测试。
(5)结果验证和比较
对照结果我们采用主流可靠的BWA软件来实现:
我们利用BWA对样本Human_A1600169-R169_good_1.fq比对。流程如下:
1.利用参考基因组构建index库,运行
bwa index-a bwtsw hg19.fa,
其中,hg19.fa为参考基因组,由于参考基因组hg19.fa大于2GB,-a参数采用bwtsw。
2.样本序列和参考序列index库比对,运行
bwa aln-f Human_A1600169-R169_good_1.sai hg19.faHuman_A1600169-R169_good_1.fq
生成sai文件,包含有SA coordinates信息摘要。
3.将sai文件转换为sam输出,运行
bwa samse-f Human_A1600169-R169_good_1.sam hg19.fa
Human_A1600169-R169_good_1.sai
Human_A1600169-R169_good_1.fastq,
其中,sam文件包含比对结果的各种信息。
4.利用perl语言提取比对结果数据并统计
样本唯一比对率:72.772%,
样本比对率:74.443%。
本软件BWA和结果一致,但bwa整个流程需要半小时并占用大量资源,同时bwa不能即时连续处理多个样本,单样本处理也不能直接给出分析NIPT所需的统计结果,要另外写脚本统计分析。
针对无创产前检测项目二者比较(单个样本):
参看图8,本实施例公开一种二代测序短序列快速比对分析装置,包括:
映射单元1,用于获取测序得到的DNA短序列,并采用第一hash算法和第二hash算法分别映射编码所述DNA短序列,分别得到第一索引和第二索引;
比对单元,用于基于预设的index查询库、所述第一索引和第二索引将所述DNA短序列和参考基因组进行比对,其中,所述index查询库由单元结构体数组构成,每个单元结构体包含有value值和index2值,所述value值为对应的组成所述参考基因组的K-mer片段的位置信息摘要的压缩,所述index2为所述K-mer片段的第二hash算法的映射结果,存储每个所述单元结构体的数组索引偏移量为对应的K-mer片段的第一hash算法的映射结果index1,K为片段序列长度;
计算单元3,用于根据比对的结果,若比对上,则获取与对应的DNA短序列比对上的K-mer片段的value值,并根据所述与对应的DNA短序列比对上的K-mer片段的value值确定出所述对应的DNA短序列所在染色体号和所在染色体上的位点。
虽然结合附图描述了本发明的实施方式,但是本领域技术人员可以在不脱离本发明的精神和范围的情况下做出各种修改和变型,这样的修改和变型均落入由所附权利要求所限定的范围之内。
- 还没有人留言评论。精彩留言会获得点赞!