基于代价敏感LSTM网络的蛋白质域检测方法与流程
本发明涉及的是一种生物医学领域的技术,具体是一种基于代价敏感LSTM网络的蛋白质域检测方法。
背景技术:
蛋白质结构域是蛋白质三级结构上的单元,是蛋白质结构和功能的基本单位。结构域是具有高度保守性的蛋白质区域,在具有多个域的蛋白质中,结构域被一些松散的边界区域分开。蛋白质域的预测是蛋白质结构研究的重要组成,是蛋白质三级结构预测的预备步骤。近年来数据采集技术的飞速发展产生了海量的生物数据,而采用实验方法在蛋白质分子上分割蛋白质域需要耗费大量的成本和时间,因此需要计算性的方法来预测蛋白质域,弥补生物数据产生能力和理解速度之间的差距。
当前预测蛋白质域的方法主要可以分为两类:基于模板的方法和从头预测方法。基于模板的方法在蛋白质结构数据库中搜索蛋白质序列的同源模板,得到若干个具有相似模板的片段,组合这些片段来获得结构域和域边界的信息。基于模板的方法具有较高的准确度,但依赖于模板,在缺乏相似模板时不再有效。从头预测方法利用结构域内氨基酸残基和域边界区域氨基酸残基的不同统计和物理特征,提取这些特征,并采用机器学习方法对氨基酸残基进行二元分类,将它们分为域内残基和域边界残基。从头预测方法仅从蛋白质的一级序列中预测蛋白质域,适用于任何蛋白质,且可以用来发现新的蛋白质结构。本发明即是属于从头预测方法。
在蛋白质域的预测中,对蛋白质序列上每个氨基酸残基的预测不是孤立进行的,要考虑周围区域残基的影响。特别地,由于蛋白质的三维空间结构,两端在序列位置上相距较远的残基可能经过折叠在空间上相邻并相互作用和影响,即存在长程相关性。已有的从头预测方法采用的机器学习模型主要有支持向量机、前馈神经网络、双向循环神经网络,它们都不能对蛋白质序列的长程相关性进行有效建模。
由于支持向量机和前馈神经网络不具有记忆性,在序列上的每个节点被处理完后,它的状态将被丢弃,不能处理上下文信息。一种常用的方法是采用移动窗口来选取周围的一段残基区域,将它们的特征共同作为模型的输入。这种方法在模型的输入层或前处理中引入了残基周围区域的影响,但它对距离相关性的处理能力依赖于所选取窗口的大小。如果想通过扩大窗口来描述长程相关性,则可能使得模型训练时间很长或产生过拟合。
循环神经网络引入了步的概念,通过隐藏层节点间的连接,对前面的信息进行记忆并应用于当前输出的计算中,在网络结构中加入了对序列相关性的处理,从而对序列数据进行建模。但是它在处理长程相关性时存在梯度消失问题,序列节点之间的感知力会随着节点间距离的增大而降低,并不能真正实现长程信息的记忆。
经过对现有技术的检索发现,中国专利文献号CN105740646A,公开(公告)日2016.07.06,公开了一种用于蛋白质二级结构预测的BP神经网络训练及预测方法,首先从PDB中选取一组α螺旋、β折叠和卷曲三类结构占正常比例的训练样本集,对蛋白质的氨基酸序列信息编码后作为网络输入,对应氨基酸的二级结构作为网络输出;基于梯度方法上进行优化,引入附带动量项和自适应学习率的学习规则,避免“振荡现象”和陷入局部极小值;在输入层采用六位输入编码方式和滑动窗口技术,隐藏层结构根据经验公式和滑动窗口大小进行设置,输出层依据DSSP算法对蛋白质二级结构的分类进行输出预测。但该技术无法解决蛋白质序列的长程相关性,对距离相关性的处理能力依赖于滑动窗口的大小,且在滑动窗口范围内,氨基酸残基间的感知力随距离的增大呈指数衰减;没有对于样本集不平衡的情况进行处理。
技术实现要素:
本发明针对现有技术存在的上述不足,提出一种基于代价敏感LSTM网络的蛋白质域检测方法,采用双向LSTM网络作为检测模型,是在蛋白质域检测中第一次对蛋白质的长程相关性进行有效建模;并在蛋白质域检测中首次引入代价敏感的损失函数来训练模型,使得模型对于类别错分的代价敏感,适应于蛋白质域正负样本不平衡的特征。与已存在的蛋白质域从头检测方法相比,本发明对于蛋白质序列进行了更为准确有效的建模,具有更高的检测准确率。
本发明是通过以下技术方案实现的:
本发明通过建立双向LSTM网络作为检测模型,然后采用代价敏感的损失函数进行网络训练,并在网络训练过程中自适应地调整损失函数的代价权重;再用经训练得到的双向LSTM网络检测蛋白质序列的结构域,即原始输出,最后采用平滑、删除、合并操作对训练后的双向LSTM网络的原始输出进行后处理,得到蛋白质域结果。
所述的双向LSTM网络包括输入层、隐藏层和输出层。
所述的建立双向LSTM网络是指:设置输入单元数为提取的特征维数,输出单元数为1,网络的隐藏层由两个传播方向相反的隐藏层叠加构成,采用随机梯度下降算法更新网络权重。
所述的代价敏感的损失函数为:L(y,z)=-wzlny-(1-z)ln(1-y),其中:y为网络输出;z为样本类标,z=0表示域内残基,z=1表示域边界残基;w为代价权重。据此损失函数计算偏导得到网络输出层的校正误差为:
所述的自适应地调整损失函数的代价权重是指:当给定一个任意的初始代价权重值w0,在每轮迭代后计算并更新代价权重为:w′=w+cr,其中:m为训练集样本数;TP、FP、FN分别为真阳性、假阳性、假阴性结果;c为常数,用于调节更新的幅度,以防止权重更新过慢或震荡变化。
所述的平滑,即采用窗口对模型原始输出值进行平均处理:其中:i为氨基酸残基在蛋白质序列中的位置,2l+1为窗口大小,边界值以0补齐。
所述的删除,即当平滑后的值为y’,域边界区域定义为输出值大于截断值yc的一段连续残基,其中:n为域边界数,y’(i)‐yc表示了边界检测的置信度,定义域边界区域面积当截断得到的域边界区域面积小于阈值,即S(k)<smin,则认为该域边界为假阳性结果,予以删除。
所述的合并,即当两个检测的域边界位置间的距离很接近,就将这两个域边界区域合并为一个,具体为:分别为域边界区域的起始和结束位置,则所检测的域边界位置为合并后的域边界位置为:
本发明涉及一种实现上述方法的系统,包括:双向LSTM网络模块、代价敏感损失函数模块、自适应调整权重模块,其中:自适应调整权重模块与代价敏感损失函数模块相连并传输代价权重w信息,代价敏感损失函数模块与双向LSTM网络模块相连并传输网络输出层的校正误差dk信息,双向LSTM网络模块输出网络输出值y信息。
技术效果
与现有技术相比,本发明的技术效果包括:
1)采用了双向LSTM网络作为机器学习模型,对于蛋白质序列的长程相关性进行了有效建模;
2)在模型中训练中采用了代价敏感的损失函数,并能够自适应地调整代价权重,提高了模型对于不平衡数据集的适应性;
3)对于模型原始输出进行平滑、删除、合并等后处理操作,减少了假阳性结果,提高了整体检测准确度。
附图说明
图1是代价敏感的双向LSTM网络的训练流程图;
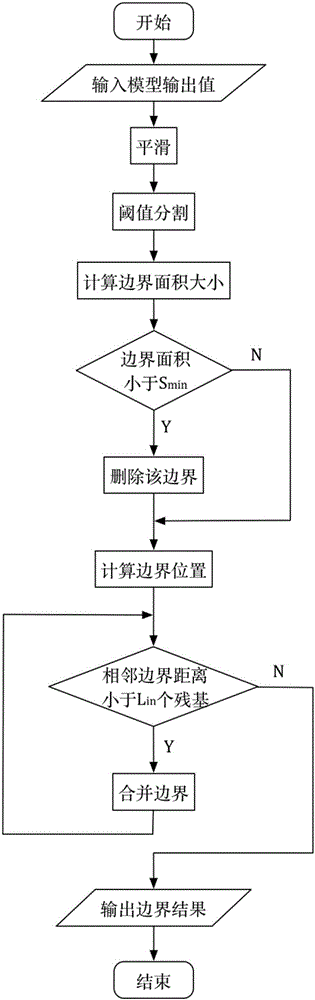
图2是模型后处理部分流程图。
具体实施方式
本实施例首先建立包括输入层、隐藏层和输出层的双向LSTM网络作为检测模型,并设置双向LSTM网络作为检测模型的输入层单元数为提取的氨基酸残基的特征维数,输出层单元数为1,输出值用作二元分类。
如图1所示,为本实施例的代价敏感的双向LSTM网络的训练流程。采用随机梯度下降算法进行训练如下:
步骤1.随机排列训练集中的所有样本。
步骤2.对于每个训练样本,通过前向传播,计算得到代价敏感的损失函数和偏导值:
L(y,z)=-wzlny-(1-z)ln(1-y)
再通过后向传播计算梯度并更新网络中的权值。
在步骤2中,训练双向LSTM网络的步骤如下:
步骤2.1、初始化损失函数以及w′=w+cr的代价权重w0。
步骤2.2、随机排列训练集样本。
步骤2.3、顺序选取训练集样本,前向传播得到网络中各层单元的输出值。
步骤2.4、由网络实际输出和期望输出值计算损失函数L(y,z)=-wzlny-(1-z)ln(1-y)及偏导值。
步骤2.5、后向传播,得到损失函数关于隐藏层单元输入的偏导值。
步骤2.6、计算损失函数关于网络权重的偏导值并更新网络权重。
步骤2.7、选取下一个训练样本,重复步骤2.3~2.6,直到遍历训练集所有样本。
步骤2.8、对验证集进行前向传播并计算损失函数值。
步骤2.9、与训练过程中验证集的最小损失函数值进行比较,并累计损失值不再降低的连续迭代次数。
步骤2.10、判断损失值不再降低的连续迭代次数是否达到设定的最大次数。若小于设定的最大次数,则计算并更新损失函数的代价权重,重复步骤2.2~2.10;否则,结束训练。
步骤3.每轮遍历后,通过前向传播计算验证集样本的损失函数平均值,判断是否满足停止条件。若达到停止条件,则停止训练;若未达到停止条件,则计算并更新代价权重,重复上述步骤。
在步骤3中,后处理的实现步骤如下:
步骤3.1、采用滑动窗口对模型原始输出进行平滑滤噪。
步骤3.2、用阈值对平滑后的值进行分割,高于阈值的判定为域边界残基,低于阈值的判定为域内残基。
步骤3.3、对于分割得到的每个域边界区域,计算域边界面积若域边界面积小于Smin,则认为该域边界是一个假阳性结果,予以删除;否则,予以保留。
步骤3.4、对余下的每个域边界区域计算域边界位置若相邻域边界位置间的距离较小,则合并这两个边界,并计算合并后的域边界,直至相邻域边界间距离均符合要求。
如图2所示,为模型后处理部分流程图。用经训练得到的双向LSTM网络检测蛋白质序列的结构域,对模型的原始输出进行后处理,得到检测的域边界,从而利用域边界分割蛋白质序列得到检测的蛋白质域。具体步骤如下:
第一步、对模型原始输出进行一定残基长度窗口的平滑滤噪。
第二步、用阈值对平滑后的值进行分割,高于阈值的判定为域边界残基,低于阈值的判定为域内残基。
第三步、对于分割得到的每个域边界区域,计算域边界面积大小。若域边界面积小于Smin,则认为该域边界是一个假阳性结果,予以删除;否则,予以保留。
第四步、对余下的每个域边界区域,根据前述定义计算域边界位置。若相邻域边界位置间的距离小于Lin个残基,则合并这两个边界。
第五步、根据合并后的边界得到检测的域边界结果。
若检测的域边界在定义边界的±20个残基内,则认为该检测结果是正确的,否则认为是一个错误的(假阳性)结果。
实施例2
本实施例采用与实施例1相似的操作步骤,以国际蛋白质结构竞赛CASP11提供的蛋白质序列T0780为例,该序列与模型训练集间的一致性小于25%。T0780的蛋白质序列为:
MKKNSLYIISSLFFACVLFVYATATNFQNSTSARQVKTETYTNTVTNVPIDIRYNSDKYFISGFASEVSVVLTGANRLSLASEMQESTRKFKVTADLTDAGVGTIEVPLSIEDLPNGLTAVATPQKITVKIGKKAQKDKVKIVPEIDPSQIDSRVQIENVMVSDKEVSITSDQETLDRIDKIIAVLPTSERITGNYSGSVPLQAIDRNGVVLPAVITPFDTIMKVTTKPVAPSSSTSNSSTSSSSETSSSTKATSSKTN
CASP11中定义的蛋白质的结构域为1‐134,135‐259,即域边界位置为134。
设置双向LSTM网络为提取20维PSSM、3维检测的二级结构、1维检测的可溶解性、1维检测的无序信息共25维特征作模型输入。
用经训练得到的双向LSTM网络检测的域边界位置为138,则分割得到蛋白质域为1‐138,139‐259。
从结果可以看出,本方法有效并且准确地检测了蛋白质域。其检测的蛋白质域数正确,域边界正确且与定义边界仅相距4个残基长度。
上述具体实施可由本领域技术人员在不背离本发明原理和宗旨的前提下以不同的方式对其进行局部调整,本发明的保护范围以权利要求书为准且不由上述具体实施所限,在其范围内的各个实现方案均受本发明之约束。
- 还没有人留言评论。精彩留言会获得点赞!