L3和/或L5源作为寄生虫病疫苗或诊断的用途的制作方法
本申请为申请日为2010年11月12日、申请号为201080061107.7、名称为“l3和/或l5源作为寄生虫病疫苗或诊断的用途”的中国发明专利申请的分案申请。
本发明涉及一种用于制备治疗、预防和/或诊断寄生虫病的药剂或药物的l3和/或l5源。
背景技术:
利什曼病包括若干种由属于利什曼原虫(leishmania)属的细胞内原生寄生虫引起的疾病,利什曼原虫主要感染各种哺乳动物(包括人和犬)的巨噬细胞。疾病谱在很大程度上取决于寄生虫的种类和人宿主的免疫能力状态,范围从自愈的皮肤利什曼病(cl)到致死的内脏利什曼病(vl)或黑热病(kalaazar)(18)。由婴儿利什曼原虫(leishmaniainfantum)和恰氏利什曼原虫(l.chagasi)引起的犬内脏皮肤利什曼病(vcl)是发现于地中海盆地、中东和拉丁美洲周围国家的重要新兴动物传染病(16);作为这些寄生虫的主要储主,犬在通过白蛉(phebotominesandflies)(44)传播给人中起着重要的作用。感染的结果由宿主免疫系统和不同寄生虫种类间的相互作用决定,但是利什曼病的发病机理仍是不清楚的且关于涉及对人和犬中利什曼原虫的免疫反应的机制的认识仍是有限的。一般而言,保护性免疫与典型的细胞介导的免疫反应相关,该典型的细胞介导的免疫反应通过t细胞产生的细胞因子诱导巨噬细胞活化。另一方面,不可治愈的疾病与强烈的体液反应的产生相关(15、24)。
基于未加工的寄生虫成分或确定的寄生虫抗原,对于第二代疫苗的开发的研究,集中于不同表面或分泌的寄生虫分子(secretedparasitemolecules)的鉴定,所述分泌的寄生虫分子已利用不同佐药以几种试验模式作为候选疫苗被测试(1、17、20、43、45、46、49、50)。来自感染的动物或人的血清的表达库的筛选能够促进作为候选疫苗的一些抗原的选择(参见(9))。在表达库中,在感染的小鼠或患者细胞中主要引出th1-型免疫反应的表达库,不考虑细胞位置,与不同动物模型中的保护性反应的产生有关(48、51、52)。另一方面,一些分离的抗原为细胞内保守蛋白,在用实验方法感染的小鼠中,所述细胞内保守蛋白主要刺激遭受vl或th2-介导的体液反应的人或犬中的体液反应(3、33、35、37、39)。在患有利什曼病的犬中对利什曼病所引起的不充足的体液反应被认为会导致免疫病理(immunopathology),主要由于免疫复合物(immunecomplexes)的副作用如葡萄膜炎(uveitis)(13)、中枢神经系统病变(centralnervoussystemlesions)(14)或肾炎(21、22、30、31)。最近还显示了人体内igg免疫复合物的存在和vl与不能解除感染相关,表明了免疫复合物对感染的宿主可能是不利的(27)。
尽管不作为优良的疫苗候选被首先考虑,在感染过程中引起强烈体液反应的蛋白质与保护性反应的诱导相关。例如,寄生虫的微管蛋白和组蛋白h2b被来源于免疫供体的t-细胞克隆识别(36)。另外,通过免疫小鼠的t细胞,rk39引起增殖并产生ifn-γ(23)。还显示了在小鼠内脏利什曼病模型中,利用寄生虫h2b、h3和h4基因的基因免疫会引起保护(62)。而且,激活的c激酶(lack)受体的免疫(29),一些寄生虫半胱氨酸蛋白酶(38、41)或形成th1促进佐药管理的组蛋白的寄生虫核小体(11、19)产生了与小鼠模型中抗皮肤利什曼病的保护相关的免疫反应。
在利什曼原虫进化的保守抗原中,几种迹象表明,在利什曼原虫感染过程中,核糖体蛋白质为免疫相关的分子。一些情况下,在感染过程中,核糖体成分能够通过调节细胞活性和细胞因子的释放的能力导致宿主免疫系统功能紊乱。因此,将硕大利什曼原虫(l.major)核糖体蛋白质s3a注入balb/c小鼠中,引起了b-细胞克隆的多克隆扩增(polyclonalexpansion)并抑制了t-细胞的增殖(10)。而且,在小鼠模型中,通过il-10和th2细胞因子的诱导,利用dna疫苗编码用于公认的60s核糖体蛋白质l31的基因免疫加重了病情(41、63)。另外,在患有利什曼病的犬和人中,一些寄生虫核糖体蛋白质如寄生虫酸性p蛋白与强烈体液反应的发生相关(参见(39))。然而,还指出了几种试验的核糖体蛋白质并不能诱导产生免疫性的保护性反应,或者获得的免疫性保护反应是次优的(55、41)。
目前,虽然做了许多努力,但仍没有针对寄生虫病(如利什曼原虫病)有价值的疫苗。因此,仍然迫切需要这种疫苗。
技术实现要素:
在本发明中,我们意外地发现两种硕大利什曼原虫的核糖体蛋白质l3和l5是抗原性的。这意味着这两种蛋白质的每一种都能够被感染利什曼病的个体的血清识别。另外,在皮肤利什曼病的小鼠模型中,发现了igg1同型的抗l3和抗l5抗体。在balb/c小鼠中两种蛋白质的免疫,在cpg-odn的存在下,诱导抗两种蛋白质的th1反应。作为疫苗接种的结果,能够保护小鼠抵抗硕大利什曼原虫攻击后cl的发展。我们进一步论证了硕大利什曼原虫蛋白质还能够赋予保护抵抗巴西利什曼原虫的感染。我们还论证了l3和l5核糖体蛋白质至少在不同的利什曼原虫物种间是高度保守的。将这种组合物用于药物组合物,特别是作为诊断、疫苗或治疗用途是非常吸引人的。下面进一步描述本发明。
用途
在本发明的第一方面,提供了l3和/或l5源与任选的佐药在制备用于治疗或预防受试对象的寄生虫病的药物或药剂中的用途。
l3和l5蛋白为核糖体蛋白质。核糖体蛋白质是很保守的胞浆蛋白质。因此,l3和/或l5源可以从任何的真核生物中制得,所述真核生物为植物或动物,可以选自哺乳动物、爬行动物、鱼类、昆虫类或任何其他具有染色体的生物,如原生动物。l3和/或l5源优选从与疾病紧密相关的生物中获得,优选从进化树中引起寄生虫病的生物中获得。因此,特别优选用于预防和/或治疗寄生虫病的l3和/或l5源为原生动物如疟原虫(plasmodium)和特别是锥虫(trypanosomatid)科中的成员,更特别是锥虫原生动物利什曼原虫的不同物种。存在超过20种已知的利什曼原虫,包括利什曼原虫亚属的物种,包括复杂的硕大利什曼原虫,包括硕大利什曼原虫,复杂的杜氏利什曼原虫(l.donovani),包括恰氏利什曼原虫、杜氏利什曼原虫和婴儿利什曼原虫,复杂的墨西哥利什曼原虫(l.mexicana),包括亚马孙利什曼原虫(l.amazonensis)和墨西哥利什曼原虫,以及维纳尼亚亚种(subspeciesviannia),包括复杂的巴西利什曼原虫(l.braziliensis),包括巴西利什曼原虫和秘鲁利什曼原虫(l.peruviana)和复杂的圭亚利什曼原虫(l.guyanensis),包括圭亚利什曼原虫和巴拿马利什曼原虫(l.panamensis)。特别优选的疟原虫为恶性疟原虫(plasmodiumfalciparum)和间日疟原虫(plasmodiumvivax)。在优选的实施方式中,l3和/或l5源从利什曼原虫物种中获得,优选硕大利什曼原虫、婴儿利什曼原虫、杜氏利什曼原虫、墨西哥利什曼原虫、恰氏利什曼原虫和/或巴西利什曼原虫。更优选l3源从利什曼原虫物种中获得,优选硕大利什曼原虫、婴儿利什曼原虫和/或墨西哥利什曼原虫。更优选l5源从利什曼原虫物种中获得,优选硕大利什曼原虫、婴儿利什曼原虫、巴西利什曼原虫和/或墨西哥利什曼原虫。在实施例2中,我们证实了在至少三个不同的利什曼原虫物种中存在高度保守的l3和l5同系物(一致性至少为90%,见表1)。在另一优选实施方式中,l3和/或l5源从疟原虫物种中获得。本领域技术人员将理解,l3和/或l5源优选还可以通过混合来自本文限定的几种不同生物的l3和/或l5源而制得。由于l3和/或l5源在治疗对象(treatedsubject)中表现出诱导免疫保护性反应,本文已证实将l3和/或l5源用于疫苗具有诱人的免疫性能。
术语“l3和/或l5源”可以被“l3源和/或l5源”替换。l3和/或l5源优选包括l3和/或l5蛋白、l3和/或l5衍生的肽或蛋白片段和/或编码l3和/或l5蛋白或衍生的肽或蛋白片段的核酸。优选的l3蛋白如seqidno:1所示。该优选的l3蛋白源于硕大利什曼原虫且优选由seqidno:2编码。另一优选的l3蛋白如seqidno:48所示。该优选的l3蛋白源于婴儿利什曼原虫且优选由seqidno:49编码。另一优选的l3蛋白如seqidno:50所示。该优选的l3蛋白源于墨西哥利什曼原虫且优选由seqidno:51编码。
优选的l5蛋白如seqidno:3所示。该优选的l5蛋白源于硕大利什曼原虫且优选由seqidno:4编码。另一优选的l5蛋白如seqidno:52所示。该优选的l5蛋白源于婴儿利什曼原虫且优选由seqidno:53编码。另一优选的l5蛋白如seqidno:54所示。该优选的l5蛋白源于墨西哥利什曼原虫且优选由seqidno:55编码。另一优选的l5蛋白如seqidno:56所示。该优选的l5蛋白源于巴西利什曼原虫且优选由seqidno:65编码。
贯穿本申请,每次提及的特定核苷酸序列seqidno(以seqidno:2或4或49或51或53或55或65为例)可以被包括与seqidno:2或4或49或51或53或55或65具有至少60%的序列一致性或相似性的核苷酸序列的核苷酸序列替代。
贯穿本申请,每次提及的特定氨基酸序列seqidno(以seqidno:1或3或48或50或52或54或56为例)可以被包括与氨基酸序列seqidno:1或3或48或50或52或54或56具有至少60%的序列一致性或相似性的氨基酸序列的多肽替代。
因此,在优选的实施方式中,l3源为包括与氨基酸序列seqidno:1或48或50具有至少60%的序列一致性或相似性的氨基酸序列的多肽和/或由与seqidno:2或49或51具有至少60%的一致性的核苷酸序列编码的多肽。
因此,在优选的实施方式中,l5源为包括与氨基酸序列seqidno:3或52或54或56具有至少60%的序列一致性或相似性的氨基酸序列的多肽和/或由与seqidno:4或53或55或65具有至少60%的一致性的核苷酸序列编码的多肽。
因此,在优选的实施方式中,l3源为包括与核苷酸序列seqidno:2或49或51具有至少60%的序列一致性或相似性的核苷酸序列的核酸和/或编码与seqidno:1或48或50具有至少60%的一致性的氨基酸序列的核酸。
因此,在优选的实施方式中,l5源为包括与核苷酸序列seqidno:4或53或55或65具有至少60%的序列一致性或相似性的核苷酸序列的核酸和/或编码与seqidno:3或52或54或56具有至少60%的一致性的氨基酸序列的核酸。
优选地,当编码的蛋白质或多肽、蛋白片段、肽至少在一定程度上仍能至少引起从seqidno:1或3或48或50或52或54或56获得的免疫反应时,与特定限定的氨基酸或核苷酸序列具有至少60%的一致性或相似性的所述氨基酸序列或核苷酸序列被本发明涵盖且据说是功能性的。至少在一定程度上优选指至少50%、至少60%、70%、80%、至少90%或100%。引起免疫反应定义如下:当能够引起治疗对象的免疫反应,优选指能够促进或引起th1免疫反应对给定的抗原l3和/或l5和/或能够预防和/或延缓真皮或粘膜病变的发展和/或引起重要的真皮和/或粘膜病变中和/或耳朵中和/或引流淋巴结(draininglymphnode)(dln)(优选引流任意这些感染的部位(真皮、粘膜部位、耳朵))中以及内部器官(如肝脏、脾脏、骨髓、肾脏、大脑等)中寄生虫量下降的时候,化合物是功能性的。涵盖于本发明的氨基酸序列可以包括一、二、三、四、五或更多的取代和/或插入和/或缺失和/或添加的n-或c-端氨基酸或化学基团,以增强稳定性、溶解性和免疫原性。
本文定义的l3和/或l5蛋白片段或l3和/或l5衍生的肽优选为包括与l3和/或l5蛋白对应的至少2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、26、27、28、29、30或更多个连续的氨基酸的片段并能够引起前文定义的免疫反应。因此,在优选实施方式中,本文定义的l3和/或l5蛋白片段或l3和/或l5衍生的肽优选为包括seqidno:1或3或48或50或52或54或56的至少2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、26、27、28、29、30或更多个连续的氨基酸的片段。
作为优选的实施方式,优选的l3蛋白片段或优选的l3-衍生的肽包括或由l3蛋白的c-端部的最后的40、39、38、37、36、35、34、33、32、31、30、29、28、27、26、25、24、23、22、21、20个连续的氨基酸组成。甚至更优选地,包括或由seqidno:1或48或50的最后的40、39、38、37、36、35、34、33、32、31、30、29、28、27、26、25、24、23、22、21、20个连续的氨基酸组成。甚至更优选地,包括或由seqidno:1的氨基酸394-419组成。作为另一优选实施方式,优选的l5蛋白片段或优选的l5-衍生的肽包括或由l5蛋白的n-端部的起始的40、39、38、37、36、35、34、33、32、31、30、29、28、27、26、25、24、23、22、21、20个连续的氨基酸组成。甚至更优选地,包括或由seqidno:3或52或54或56的起始的40、39、38、37、36、35、34、33、32、31、30、29、28、27、26、25、24、23、22、21、20个连续的氨基酸组成。甚至更优选地,包括或由seqidno:3的氨基酸1-23组成。作为另一优选实施方式,优选的l5蛋白片段或优选的l5-衍生的肽包括或由l5蛋白的c-端部的最后的40、39、38、37、36、35、34、33、32、31、30、29、28、27、26、25、24、23、22、21、20、19、18、17、16、15、14、13、12、11、10、9、8、7、6个连续的氨基酸组成。甚至更优选地,包括或由seqidno:3或52或54或56的最后的40、39、38、37、36、35、34、33、32、31、30、29、28、27、26、25、24、23、22、21、20、19、18、17、16、15、14、13、12、11、10、9、8、7、6个连续的氨基酸组成。甚至更优选地,包括或由seqidno:3的氨基酸322-328组成。在另一优选实施方式中,l3和l5源含有蛋白质,所述蛋白质含有至少一种l3蛋白的蛋白片段以及至少一种l5蛋白的蛋白片段。更优选地,将l3蛋白片段和l5蛋白片段融合在一起以形成嵌合蛋白(chimericprotein)。因此,本发明还涵盖一种核酸或编码该l3和l5源的核酸。
在优选的实施方式中,l3和/或l5源包括至少一种l3和/或l5蛋白和/或至少一种l3和/或l5的蛋白片段。在更优选的实施方式中,l3和/或l5源包括至少两个l3和/或l5蛋白和/或至少两个l3和/或l5的蛋白片段。该实施方式涉及蛋白质-基源,优选蛋白质基疫苗。
提出在优选实施方式中本文限定的l3和/或l5源不能理解为涵盖wo2009/090175中限定的核糖体蛋白质提取物(rpe)。利用存在于受试对象体内时引起寄生虫病的寄生虫细胞通过进行以下步骤获得rpe:
a.将寄生虫细胞与裂解缓冲液混合,
b.离心获得的混合物以获得胞浆提取物,
c.从获得的胞浆提取物中制备rpe。
因此在优选的实施方式中,l3和/或l5源不是以上定义的rpe。
在另一优选实施方式中,l3和/或l5源包括至少一种编码l3和/或l5的核酸和/或至少一种编码l3和/或l5的蛋白片段的核酸。在更优选的实施方式中,l3和/或l5源包括至少两个编码l3和/或l5蛋白的核酸和/或至少两个编码l3和/或l5蛋白片段的核酸。该实施方式涉及核酸-基源,优选核酸-基疫苗。
l3和/或l5源可以为蛋白质、蛋白质的消化物和/或其片段,可以为纯化的形式或者可以包含在未加工的组合物中,优选为生物源的组合物,如细菌溶菌产物、酵母菌溶菌产物、真菌溶菌产物,超声波降解(sonicate)或固定(fixate)。替换地,l3和/或l5源可以为化学合成的或体外酶促反应的产物。l3和/或l5源蛋白或其片段还可以为从rna或dna模版编码所述蛋白或其片段的核酸。rna或dna分子可以为‘裸’dna,优选包含在囊泡或脂质体中,或包含在载体中。所述载体可以为本领域已知的任意(重组)dna或rna载体,且优选为质粒;其中,将编码潜在抗原的基因可操作地连接于赋予编码信使的表达和翻译的调控序列上。所述载体还可以为任意dna或rna病毒,例如但不限于腺病毒、腺-相关病毒(aav)、逆转录病毒、慢病毒、修饰的痘苗安卡拉病毒(modifiedvacciniaankaravirus)(mva)或禽痘病毒或任何其他能够赋予多肽表达至被选择的受试对象的病毒性载体。dna载体可以为非整合的,例如附加型复制载体(episomallyreplicatingvectors),或可以为通过随机整合或通过同源重组整合到宿主基因组的载体。
根据本发明,dna分子包括编码l3和/或l5蛋白或其片段的基因,任选地嵌入载体如病毒或质粒中,可以整合到受试对象的基因组中。在本发明优选的实施方式中,所述宿主可以为微生物。优选该重组微生物为分枝杆菌(mycobacterium),种类如结核分枝杆菌(m.tuberculosis)、耻垢分枝杆菌(m.smegmatis)(yue.y.etal、(2007),j.virol.meth.,141:41-48,cayabiyaby.etal,(2006),j.virol.,80:1645-1652)或牛分枝杆菌(m.bovis)且最优选牛分枝杆菌卡介苗(bacilluscalmetteguerin)(bcg)或耻垢分枝杆菌,根据本发明能够传递多肽或其片段到宿主中。重组bcg和重组的方法为本领域所熟知;例如,wo2004094469。可以将该重组微生物制成活重组体和/或活减毒疫苗,如jacobsetal.1987,nature,327(6122):532-5)。所述载体还可以包含在细菌源的宿主中,例如但不限于活减毒的和/或重组的志贺氏杆菌(shigella)或沙门氏菌(salmonellabacteria)。
本发明可以使用任何已知的佐药。本领域技术人员知道几种合适的佐药。佐药最优选选自以下列出的佐药:阳离子(抗菌的)肽、皂角苷(saponine)和toll-样受体(tlr)配体例如但不限于聚(i:c)、cpg基序、lps、脂质a、脂肽pam3cys和细菌鞭毛蛋白或其部分,及它们的具有化学修饰的衍生物。在根据本发明的方法和组合物中使用的其他优选的佐药为:活的或死的bcg的混合物、带有所述潜在抗原或其部分的免疫球蛋白复合物、ic31(来自www.intercell.com;wo03047602中)、qs21/mpl(us2003095974)、dda/mpl(wo2005004911)、da/tdb(wo2005004911;holten-andersenetal,2004infectimmun.2004mar;72(3):1608-17.)和可溶的lag3(cd223)(来自www.immunotep.com;us2002192195)。另外,其他优选的佐药包括短小棒状杆菌(corynebacteriumparyum)或痤疮丙酸杆菌(propionobacteriumacnes)的使用(64、65、66)。
特别优选的佐药为已知通过toll-样受体作用的佐药。能够激活先天免疫系统的佐药能通过toll样受体(tlr’s)(包括tlr’s1–10)和/或通过rig-1(维甲酸诱导基因-1)蛋白和/或通过内皮素受体(endothelinreceptor)很好地被激活。本领域很好地记载了能够激活tlr受体的化合物及其变形和衍生物。tlr1可以被细菌脂蛋白及其乙酰化形式激活,tlr2还可以被革兰氏阳性细菌的糖脂、lps、lpa、lta、菌毛、外膜蛋白、来自细菌或来自宿主的热休克蛋白(heatshockproteins)、以及分枝杆菌的脂阿拉伯甘露聚糖(mycobacteriallipoarabinomannans)激活。tlr3可以被dsrna,尤其为病毒源的dsrna,或被化合物聚(i:c)激活。tlr4可以被革兰氏阴性lps、lta、来自宿主或来自细菌源的热休克蛋白、病毒衣壳或包膜蛋白、紫杉醇(taxol)或其衍生物、含有低聚糖和纤维连接蛋白的透明质酸激活。tlr5可以被细菌的鞭毛或鞭毛蛋白激活。tlr6可以被分枝杆菌的脂蛋白和b族链球菌不耐热的可溶性因子(gbs-f)或葡萄球菌调节蛋白(staphylococcusmodulins)激活。tlr7可以被喹啉咪唑(imidazoquinolines)及衍生物激活。tlr9可以被未甲基化的cpgdna或染色质-igg复合物激活。特别地,tlr3、tlr4、tlr7和tlr9在调节对病毒感染的先天免疫反应中发挥着重要的作用,并将能够激活这些受体的化合物特别优选用于本发明。特别优选的佐药包括但不限于合成的化合物,包括dsrna、聚(i:c)、触发tlr3和tlr9受体的未甲基化的cpgdna、ic31、tlr9激动剂、imsavac、tlr4激动剂。在另一优选的实施方式中,所述佐药物理地连接至前文定义的l3和/或l5源上。佐药和共刺激化合物(costimulatorycompounds)或官能团与含有肽的hlai类和hlaii类表位的物理连接通过同时发生的抗原递呈细胞(特别是吸收、消化并显示抗原的树突细胞)的刺激提供了增强的免疫反应。其他优选的免疫修饰化合物为t细胞粘附抑制剂,更优选为内皮素受体如bq-788(67)的抑制剂。bq-788为n-顺-2,6-二甲基哌啶羰基-l-γ-甲基亮氨酰-d-1-甲氧基羰基色氨酰基-d-正亮氨酸(n-cis-2,6-dimethylpiperidinocarbonyl-l-gamma-methylleucyl-d-1-methoxy-carbonyltryptophanyl-d-norleucine)。然而,任意bq-788的衍生物或改性的bq-788化合物也涵盖在本发明的范围内。
其他佐药包括mpl-se(glaxosmithklinebiologicals,比利时)或em005(idri美国)。
在优选的实施方式中,佐药为th1-促进佐药(如含有cpgodn基序的佐药)。如文献(68)所述,th1-促进佐药是当与抗原(此处为l3和/或l5源)一起使用时,能够促进或引起对给定抗原的th1免疫反应的佐药,当与抗原一起培养时,在治疗对象的脾细胞的上层清液中检测到。作为对照,在同一受试对象的脾细胞群中评测th1免疫反应的促进或引起,所述受试对象未用抗原和佐药处理,或者同一群仅用抗原处理。优选用通过与抗原一起培养治疗对象的脾细胞和/或通过诱导产生抗原特异性igg2a免疫球蛋白检测到的ifnγ的诱导定义引起或促进th1免疫反应。优选通过实施例中描述的脾细胞的elisa而进行该细胞因子诱导的评测。优选通过实施例1中描述的elisa或蛋白质印迹(westernblot)进行igg2a诱导的评测。用l3和/或l5源和佐药刺激脾细胞的ifnγ和/或igg2a的诱导优选指佐药适合用作th1-促进佐药。
替换地或者与以上给出的引起或促进th1免疫反应的第一定义结合,引起或促进th1免疫反应可以进一步定义为不存在(或诱导的不存在)th2免疫反应。th2免疫反应的特征在于当与未处理的脾细胞相比时,il-4、il-10诱导的显著增强和/或显著igg1免疫球蛋白的产生。优选通过实施例中描述的脾细胞的elisa进行il-4和/或il-10的诱导的评测。优选通过实施例1中描述的elisa或蛋白质印迹进行igg1诱导的评测。
替换地或者与以上给出的引起或促进th1免疫反应的两个第一定义结合,引起或促进th1免疫反应可以进一步定义为针对指定抗原(l3和/或l5源的情况),发生ifnγ/il-10比例和/或ifnγ/il-4比例的增大和/或igg1/igg2a比例的减小。在优选的实施方式中,这些比例中任意2个以上的变化表明佐药具有th1性能。优选通过实施例中描述的脾细胞的elisa进行各个提到的细胞因子诱导的评测。优选通过实施例1中描述的elisa或蛋白质印迹进行免疫球蛋白igg1或igg2a诱导的评测。
在优选的实施方式中,th1-促进佐药是,或包括,或由寡脱氧核苷酸(oligodeoxynucleotide)组成。更优选地,寡脱氧核苷酸(odn)包括或由cpg组成,其中,c是未甲基化的(cpgodn):3’嘌呤-cpg-5’嘧啶。优选的寡脱氧核苷酸是,或包括,或由硫代磷酸-修饰的odn序列组成。使用具有这种修饰的寡脱氧核苷酸是有利的,因为使用的寡脱氧核苷酸因此比未修饰的寡脱氧核苷酸更稳定且因此它们在血流中不易降解。优选的th1-促进佐药由或包括至少一种cpg基序、至少两种或至少三种组成。优选的免疫刺激odn(immunostimulatoryodn)(5’至3’)序列为tcaacgttga(seqidno:5)和gctagcgttagcgt(seqidno:6)。本领域技术人员不会受到本文明确描述的序列的限制。他/她可以设计其他序列并随后测试它们如前文定义的它们的th-1促进性能。该优选限定的佐药cpgodn极具吸引力,因为在实施例中证实了lrpe与该th1-促进佐药的混合接种(co-inoculation)诱导了抵抗balb/c和c57bl/6小鼠菌株中抗硕大利什曼原虫寄生虫的攻击的保护。在两个模型中,保护与特定ifn-γ的产生有关。在balb/c中,还检测到了il-4和il-10产生的限制。
本发明提供了l3和/或l5源的适当的用途如1)预防(预防剂),2)暴露/感染后(post-exposure/infection)或3)治疗/医疗的疫苗。本发明的一个优点是可以制备用于治疗寄生虫病的广谱药制剂即跨物种特异性的药的制备。在许多寄生虫病中,疫苗针对特定的物种,只对特定的物种有作用。其中这种寄生虫病的一个实例为利什曼病。目前,该病已被药物控制,但是药物治疗并不能防止该病的传播且在很多情况下并不是很有效。在优选的实施方式中,寄生虫病为利什曼病或疟疾。更优选地,寄生虫病由利什曼原虫或疟原虫物种引起。在进一步优选的实施方式中,寄生虫病由除l3和/或l5源来源的物种外的不同物种引起。具体地,由利什曼原虫属的一种物种引起的利什曼病可以通过使用基于来源于另一利什曼原虫物种的l3和/或l5源的组合物来治疗,如实验部分(见实施例2)所示,几个利什曼原虫物种的l3和l5同系物是高度保守的(至少90%的一致性,见表1)。另外,我们还在实验部分(见实施例1和3)证实了硕大利什曼原虫的l3和l5蛋白能够保护抵抗硕大利什曼原虫或巴西利什曼原虫感染。在一个实施方式中,由硕大利什曼原虫引起的利什曼病成功地被含有来自婴儿利什曼原虫、恰氏利什曼原虫、亚马孙利什曼原虫或巴西利什曼原虫的l3和/或l5源的组合物治疗。在另一实施方式中,由恰氏利什曼原虫或亚马孙利什曼原虫引起的利什曼病成功地被含有来自婴儿利什曼原虫的l3和/或l5源的组合物治疗。在另一实施方式中,由硕大利什曼原虫或巴西利什曼原虫引起的利什曼病成功地被含有来自硕大利什曼原虫的l3和/或l5源的组合物治疗。替换地,其他寄生虫病如疟疾可以成功地被基于其他物种的l3和/或l5源(例如基于婴儿利什曼原虫或硕大利什曼原虫或墨西哥利什曼原虫或巴西利什曼原虫的l3和/或l5源)的组合物治疗。
在本发明的上下文中,受试对象指人或动物。涵盖在本发明范围内的动物包括哺乳动物,优选为犬。
在优选的实施方式中,本文定义的药物(或药制剂或制药组合物或药剂)用于增强人或动物免疫系统抗感染和/或疾病(更优选为寄生虫感染和/或寄生虫病)的能力。具体地,它可给人或动物受试对象服用。本文定义的药物优选肠道外给药,例如通过静脉、皮下、腹腔、肌肉、动脉或病灶(intralesional)途径注射或注入。优选的给药模式为皮下注射。本发明并不限于l3和/或l5源给药的特定模式。优选的给药模式为使用胶囊或片剂的口服给药。替换地,l3和/或l5源可以通过导管或泵、或栓剂局部给药。替换地,l3和/或l5源可以整体给药。l3和/或l5源或包括l3和/或l5源的组合物的配制取决于预期的给药模式和(治疗)应用。药物载体可以为任何适于递送l3和/或l5源给受试对象的可兼容的、无毒的物质。如可以使用无菌水、或惰性固体或赋形剂作为载体,通常补充制药可接受的佐药、缓冲剂、分散剂等。组合物可以为液体的,如稳定的l3和/或l5源或包括l3和/或l5源的组合物的悬浮液,也可以为固体的和/或干燥的形式:如粉末。对于口服和直肠给药,l3和/或l5源可以以固体制剂如胶囊、片剂、栓剂和粉末的形式给药,或者以液体制剂如酏剂、糖浆剂、乳剂、软膏和悬浮液的形式给药。另一形式可以为半固体或半液体形式,其中,l3和/或l5在固体支持物(如块)中或上以液体形式存在。
可以通过本领域熟知的传统技术将药物与制药可接受的介质或递送载体组合。例如,可以将l3和/或l5源和任选的佐药溶于磷酸盐缓冲液(pbs)中。制备肠道外给药的组合物的方法为本领域所熟知且在多种资源中描述得更详细,包括例如,remington'spharmaceuticalsciences,ed.argennaro,第20版,2000,williams&wilkins,pa,美国。优选药物以治疗有效剂量给药,治疗有效剂量即可增强人或动物免疫系统抗本文定义的感染和/或疾病的能力的剂量。优选地,本发明药物制剂的治疗有效剂量能够引起本文定义的免疫反应:当能够引起治疗对象免疫反应时,优选指当能够对给定的抗原l3和/或l5促进或引起th1免疫反应和/或能够预防和/或延缓真皮或粘膜病变的发展和/或引起真皮和/或粘膜病变中和/或耳朵中和/或引流淋巴结(dln)(优选引流任意感染的部位(真皮、粘膜部位、耳朵))中以及内部器官(如肝脏、脾脏、骨髓、肾脏、大脑等)中寄生虫量的显著下降时,剂量是治疗有效的。真皮病变存在的评测如实施例1所描述(见图4:脚掌肿胀(footpadswelling))。寄生虫量的评测如实施例所描述(见图4)。在使用本发明组合物的最初一次疫苗接种然后一次连续用寄生虫感染并等待约6个星期的时间段之后,本发明药物的治疗有效剂量将优选预防真皮病变的发展和/或将优选诱导耳朵中寄生虫量下降约3个数量级和/或dln中约相似的数量级。在优选的实施方式中,本文定义的药物为疫苗。在更优选的实施方式中,将至少12μg的l3和/或l5源用作疫苗。使用的l3和/或l5源的量指的是使用的l3和l5的总量。在甚至更优选的实施方式中,应当将至少12-20μg的l3和/或l5源用于提供免疫反应,任选地与至少50μg的佐药结合,例如优选为th1促进佐药如cpgodn。本文定义的疫苗可以为预防的或治疗的疫苗。l3和/或l5源和任选的佐药(优选th1促进佐药)溶解的量可以在100-500微升范围内变动。
组合物
在另外的方面,提供了一种包括l3和/或l5源和任选的佐药(优选th1促进佐药)的组合物。本文已定义l3和/或l5源和佐药。在优选的实施方式中,组合物由l3和/或l5源和th1促进佐药组成。优选的th1促进佐药为cpgodn。优选的组合物包括或由l3和/或l5源和任选的佐药(优选th1促进佐药)溶于pbs中组成。在更优选的实施方式中,本发明还涵盖将l3和/或l5源和佐药(优选th1促进佐药)连续给药。因此,两种成分不需要完全地存在于一种单一的组合物中,只要将它们都供给受试对象即可。
该组合物进一步包括制药可接受的佐药和/或载体。
优选将该组合物用作药物。所述药物优选为疫苗。本文已扩展定义了药物、佐药和疫苗。
如本文已定义的,组合物可以为液体、固体或半液体或半固体形式。
在优选的实施方式中,为了改善治疗或预防处理的特异性,与l3和/或l5源按顺序或同时使用其他化合物。例如使用其他化合物是有利的,将进一步增强治疗对象的免疫反应。更优选地,该化合物不与l3和/或l5源一起存在于单一组合物中。例如,所述化合物选自由来自引起寄生虫病(19)如利什曼病的寄生虫的其他蛋白质源组成的组中。所述蛋白质源与前文定义的l3和/或l5源具有相同的含义。优选上下文中的蛋白质为组蛋白如h2a、h2b、h3、h4,其他核糖体蛋白如li2a(lip)、lip2b(lip’)、lip0、l2、l7、l8、l16、s6、l19和s4。
优选的h2a蛋白如seqidno:7所示。优选的编码h2a的核酸如seqidno:8所示。优选的h2b蛋白如seqidno:9所示。优选的编码h2b的核酸如seqidno:10所示。优选的h3蛋白如seqidno:11所示。优选的编码h3的核酸如seqidno:12所示。优选的h4蛋白如seqidno:13所示。优选的编码h4的核酸如seqidno:14所示。优选的li2a(也称为lip2a蛋白)如seqidno:15或16所示。优选的编码li2a的核酸如seqidno:17所示。seqidno:17为基因组序列。本领域技术人员可以从该基因组序列衍生出优选的编码序列。该优选的编码序列对应于编码mrna的核酸,编码如seqidno:15或16所示的li2a蛋白的mrna:一个来自seqidno:17的核苷酸791-1111且一个来自1662-1982。
优选的lip2b蛋白如seqidno:18所示。优选的编码lip2b的核酸如seqidno:19所示。优选的lip0蛋白如seqidno:20所示。优选的编码lip0的核酸如seqidno:21所示。
seqidno:8、10、12、14、19和21为基因组序列。本领域技术人员可以从这些基因组序列衍生出其他优选的编码序列。这些其他优选的编码序列对应于编码mrna的核酸,mrna编码各自的蛋白质:这些核酸序列分别如seqidno:68、69、70、71、72和73所示。
优选的l2蛋白如seqidno:22所示。优选的编码l2的核酸如seqidno:23所示。优选的l7蛋白如seqidno:24所示。优选的编码l7的核酸如seqidno:25所示。优选的l8蛋白如seqidno:26所示。优选的编码l8的核酸如seqidno:27所示。优选的l16蛋白如seqidno:28所示。优选的编码l16的核酸如seqidno:29所示。优选的l19蛋白如seqidno:30所示。优选的编码l19的核酸如seqidno:31所示。优选的s4蛋白如seqidno:32所示。优选的编码s4的核酸如seqidno:33所示。优选的s6蛋白如seqidno:34所示。优选的编码s6的核酸如seqidno:35所示。
另一实例是使用含有几种寄生虫抗原的聚蛋白(63,65)。聚蛋白的例子为ep1141305中限定的蛋白q。编码蛋白q的核酸分子如seqidno:36所示。对应的编码的蛋白q如seqidno:37所示。蛋白q或其部分或其片段或蛋白q源或蛋白q的片段源可以与l3和/或l5源组合使用。另一聚蛋白的例子为leish-110f(69)。leish-110f或其部分或其片段或leish-110f源或leish-110f的片段源可以与l3和/或l5源组合使用。
下段中,可以用来与l3和/或l5蛋白源组合的蛋白质以组蛋白源为例。同样适用于以上定义的其他蛋白质,优选除l3和/或l5外的其他核糖体蛋白。优选的化合物包括组蛋白或其片段,或编码所述组蛋白或所述组蛋白片段的核酸分子。更优选地,组蛋白为ep1687023中限定的h2a、h2b、h3和/或h4。组蛋白h2a、h2b、h3和h4为很保守的核蛋白且其序列为本领域所熟知,见参考文献39。优选地,组蛋白从在进化树中与引起疾病的生物邻近的生物中获得。因此,特别优选用于治疗寄生虫病如利什曼病的组蛋白源为原生动物且特别是锥虫科中的成员,更特别是锥虫原生动物利什曼原虫的不同物种。
其他优选的化合物包括其他核糖体蛋白或其片段或编码所述蛋白或其片段的核酸分子。其他核糖体蛋白的实例包括l19和s4。
其他优选的化合物包括wo2009/090175中限定的核糖体蛋白提取物。
各化合物或各化合物源可以与l3和/或l5源组合使用。组合使用可以按顺序或同时。
在优选的实施方式中,l3和/或l5源与s4和/或s6源组合使用。更优选地,l3、l5、s4和s6源组合使用。在另一更优选的实施方式中,l3、l5和s4源组合使用。在另一更优选的实施方式中,l3、l5和s6源组合使用。我们证实了(见实施例5)与单独使用l3或l5或s4相比,将l3、l5和s4源组合使用提供了协同保护作用。在上下文中,优选的s4蛋白如seqidno:32所示。优选的编码s4的核酸如seqidno:33所示。优选的s6蛋白如seqidno:34所示。优选的编码s6的核酸如seqidno:35所示。在“s4和/或s6源”中使用的术语“源”与在“l3和/或l5源”中使用的术语“源”具有相同的含义。
因此,在优选的实施方式中,s4源为多肽,所述多肽包括与氨基酸序列seqidno:32具有至少60%的序列一致性或相似性的氨基酸序列和/或由与seqidno:33具有至少60%的一致性的核苷酸序列编码的氨基酸序列。
因此,在优选的实施方式中,s6源为多肽,所述多肽包括与氨基酸序列seqidno:34具有至少60%的序列一致性或相似性的氨基酸序列和/或由与seqidno:35具有至少60%的一致性的核苷酸序列编码的氨基酸序列。
因此,在优选的实施方式中,s4源为核酸,所述核酸包括与核苷酸序列seqidno:33具有至少60%的序列一致性或相似性的核苷酸序列和/或编码与seqidno:32具有至少60%的一致性的氨基酸序列的核苷酸序列。
取决于使用的源的类型(蛋白质基或核酸基),本领域技术人员将知道哪种类型的制剂是合适的。源可以这样给药(裸蛋白或核酸)。替换地,核酸基源可以利用本文定义的核酸构建体(nucleicacidconstruct)给药。
s6源
在另一方面提供了一种s6源或组合物,所述组合物包括或由s6源但不包括l3和/或l5和/或s4源组成。我们证实了(见实施例4)单独使用s6能够提供保护抵抗利什曼原虫感染。
在“s6源”中使用的术语“源”与在“l3和/或l5源”中使用的术语“源”具有相同的含义。
本文定义的包括l3和/或l5源的各个用途或方法或组合物的类型也适用于s6源。
因此,在优选的实施方式中,s6源为多肽,所述多肽包括与氨基酸序列seqidno:34具有至少60%的序列一致性或相似性的氨基酸序列和/或由与seqidno:35具有至少60%的一致性的核苷酸序列编码的氨基酸序列。
因此,在优选的实施方式中,s6源为核酸,所述核酸包括与核苷酸序列seqidno:35具有至少60%的序列一致性或相似性的核苷酸序列和/或编码与seqidno:34具有至少60%的一致性的氨基酸序列的核苷酸序列。
方法
在另一方面,本发明提供了一种预防和/或治疗寄生虫病和/或延缓其发展和/或能够引起本文定义的免疫反应的方法:当能够引起治疗对象的免疫反应,优选指能够对给定的抗原l3和/或l5和/或对给定的l3和/或l5源促进或引起th1免疫反应和/或能够预防和/或延缓真皮或粘膜病变的发展和/或引起真皮和/或粘膜病变中和/或耳朵中和/或引流淋巴结(dln)(优选引流任意感染的部位(真皮、粘膜部位、耳朵))中以及内部器官(如肝脏、脾脏、骨髓、肾脏、大脑等)中寄生虫量的显著下降的时候,方法是有疗效的。在该方法中,本发明的疫苗发挥治疗疫苗的作用。典型地,在感染和患病之间存在一定的时间。在这种情况下,疫苗会发挥药理学免疫产品(pharmacologicalimmuneproduct)的作用,所述药理学免疫产品会通过引发宿主中与感染的病理学作用相抵消的免疫反应来预防和/或治疗疾病和/或延缓其发展。治疗疫苗与预防疫苗的不同在于治疗疫苗将在已感染或患病的患者中诱导保护。本发明既涵盖治疗也涵盖预防疫苗。在该方法中,任选的s4和/或s6源可以与l3和/或l5源组合使用。
用途
在另一方面,提供了l3和/或l5源在诊断受试对象寄生虫病中的用途。寄生虫病、l3和/或l5源和受试对象前文已定义。在这种情况下,任选的s4和/或s6源可以与l3和/或l5源组合使用。
本发明的一个优点是可以获得特定和早期寄生虫病的广谱诊断。寄生虫病的一个实例为利什曼病。在优选的实施方式中,寄生虫病为利什曼病或疟疾。更优选地,寄生虫病由利什曼原虫或疟原虫物种引起。在进一步优选的实施方式中,寄生虫病由除l3和/或l5源来源的物种外的不同物种引起。具体地,由利什曼原虫属的一个物种引起的利什曼病可以通过使用基于来源于另一利什曼原虫物种的l3和/或l5源的组合物来诊断。在一个实施方式中,由硕大利什曼原虫引起的利什曼病成功地被含有来自硕大利什曼原虫、婴儿利什曼原虫、巴西利什曼原虫或墨西哥利什曼原虫的l3和/或l5源的组合物诊断。替换地,其他寄生虫病如疟疾可以成功地被基于另一物种的l3和/或l5源(例如基于婴儿利什曼原虫、硕大利什曼原虫、巴西利什曼原虫或墨西哥利什曼原虫的l3和/或l5源)的组合物诊断。
原则上,任何受试对象都可以利用本发明进行诊断。诊断方法可以根据需要时常用于受试对象。优选地,诊断的受试对象为疑似存在感染引起所述寄生虫病的所述寄生虫的风险。疑似存在感染所述寄生虫的风险的受试对象可以生活在病区(endemicarea)或去过病区。病区包括从阿尔及利亚到沙特阿拉伯、肯尼亚、苏丹、埃塞俄比亚的北非。还包括南欧:地中海国家西班牙、法国、希腊等。还包括中美洲(所有国家)和南美洲:巴西、委内瑞拉、秘鲁、玻利维亚、哥伦比亚、阿根廷北部、巴拉圭、乌拉圭,亚洲中部至西南部:印度、伊朗、伊拉克、蒙古、阿富汗、尼泊尔、孟加拉国。
在本发明的上下文中,本文定义的用途优选为体外(invitro)或离体(exvivo)的用途。优选指所述用途在来自于所述受试对象的样品上进行。优选的样品包括血液、血清、血浆、唾液、脑脊液或尿液。更优选地,所述样品为从受试对象获得的血液或血清样品。
在优选的实施方式中,在表现出所述寄生虫病的症状之前诊断出来,称为症状前诊断或无症状诊断受试对象。在该上下文中,“症状前”优选指在表现出第一症状的至少1天、至少2天、至少3天、至少4天、至少5天、至少6天、至少7天、至少8天、至少9天、至少10天、至少15天、至少20天、至少25天、至少30天或更多天前。与寄生虫病有关的第一症状或第一临床表现如利什曼病可以选自发热、脾肿大、肝肿大、淋巴结肿大、结膜炎、皮炎钩甲(onychogriphosis)、角膜结膜炎、冷漠(apathy)和极度瘦弱(cachexia)。他们中的大多数可以通过身体的外部检查进行简单的检测。结膜炎、皮炎钩甲、角膜结膜炎是皮肤变化(cutaneousalteration)的形式。
优选的与利什曼病有关的第一症状是淋巴结肿大。可以通过身体的外部检查如触诊(palpation)进行检测。
在另一优选的实施方式中,在表现出所述寄生虫病的一些症状之前诊断出来,称为寡症状(oligosymptomatic)受试对象的诊断。在该上下文中,“寡症状”优选指具有最多三种以上定义的症状的受试对象。
在另一优选的实施方式中,在表现出所述寄生虫病的所有症状之前诊断出来,称为有症状的受试对象的诊断。在该上下文中,“有症状的”优选指具有至少四种以上定义症状(包括以上定义的皮肤变化形式)的受试对象。
本领域技术人员可以理解的是诊断的最重要类型是无症状受试对象的诊断,因为这将有助于防止疾病的进一步传播且可以更有效地帮助并治疗无症状的受试对象,如果他们被诊断处于该阶段。
在这种用途中,l3和/或l5和/或s4和/或s6源可以为用于检测下文阐释的样品中抗体存在的l3和/或l5和/或s4和/或s6蛋白或蛋白片段。
替换地,l3和/或l5和/或s4和/或s6源可以为用于检测下文阐释的样品中l3和/或l5和/或s4和/或s6核酸存在的核酸分子。
方法
在另一方面,提供了一种利用l3和/或l5源诊断受试对象寄生虫病的方法,该方法包括检验从受试对象获得的样品中是否存在识别l3和/或l5源的抗体。任选地,可以将s4和/或s6源与l3和/或l5源组合使用。本发明优选的方法为关于优选体外或离体进行的本发明的优选用途。前文已定义。
在优选的方法中,l3和/或l5源存在于组合物中。在优选的实施方式中,另一种化合物也存在于所述组合物中。替换地,没有其他的化合物存在于所述组合物中。
在优选的实施方式中,为了改善方法的特异性,其他化合物与l3和/或l5源按顺序或同时使用。例如,使用将能够辨别无症状、寡症状或有症状受试对象和接种了疫苗的受试对象的其他化合物是有利的。更优选地,所述化合物不与l3和/或l5源一起存在于单一的组合物中。在上下文中,可以使用名为组合物部分中鉴别的每种蛋白质。例如,所述化合物选自由引起寄生虫病(19)如利什曼病的其他蛋白质源组成的组中。所述蛋白质源与前文定义的l3和/或l5源具有相同的含义。优选上下文中的蛋白质为组蛋白如h2a、h2b、h3、h4,另一核糖体蛋白如li2a(lip)、lip2b(lip’)、lip0、l2、l7、l8、l16、s6、l19和s4。
另一实例为使用含有几种寄生虫抗原的聚蛋白(59,61)。聚蛋白的例子为ep1141305中限定的蛋白q。蛋白q或其部分或其片段或蛋白q源或蛋白q的片段源可以与l3和/或l5源组合使用。
优选的抗原包括组蛋白或其片段或编码所述组蛋白的核酸分子。更优选地,组蛋白为ep1687023中限定的h2a、h2b、h3和/或h4。组蛋白h2a、h2b、h3和h4为很保守的核蛋白且其序列为本领域所熟知,见参考文献39。优选地,组蛋白从在进化树中与引起疾病的生物邻近的生物中获得。因此,特别优选用于治疗寄生虫病如利什曼病的组蛋白源为原生动物且特别是锥虫科中的成员,例如疟原虫如恶性疟原虫,更特别是锥虫原生动物利什曼原虫的不同物种。
在更优选的诊断方法中,当存在可检测量的识别l3和/或l5源的抗体(优选为蛋白质或肽或蛋白部分)时和/或当所述抗体存在的量增加时,诊断寄生虫病。在受控制的或健康的受试对象中,一般不能检测所述抗体。
利用本领域技术人员所熟知的方法如elisa进行所述抗体存在的检测。优选的检测方式如实施例1所述。
识别l3和/或l5源的抗体(优选为蛋白质或肽或蛋白部分)优选指至少存在一种能够识别存在于l3和/或l5源中的至少一种化合物的抗体。所述化合物可以为l3和/或l5蛋白或l3和/或l5蛋白片段或蛋白部分或肽。同样适用于识别s4和/或s6源的抗体。
在另一方法中,利用另一核酸分子检测l3和/或l5核酸分子。l3和/或l5核酸分子优选为前文定义的编码l3和/或l5分子的核酸分子或其部分。另一核酸分子优选为引物,所述引物被设计为能够检测pcr反应或rna印迹中l3和/或l5核酸分子的存在。同样适用于能够检测s4和/或s6核酸分子存在的引物。检测l3和/或l5核酸分子存在的引物优选包括或由以下序列组成:
通过pcr特异性检测l3的引物序列
正义链,5’-aacacgaaggagggcaaggtc-3’(lml3序列的核苷酸418-438)(seqidno:38)
反义链,5’-cttcttcgcggcctttgccttg-3’(lml3序列的核苷酸1242-1263的反向和互补)(seqidno:39)
通过pcr特异性检测l5的引物序列
正义链,5’-tgcacgctggcaaattgggtac-3’(lml5序列的核苷酸10-31)(seqidno:40)
反义链,5’-cttcttcgtgcgcacagcag-3’(lml5序列的核苷酸464-483的反向和互补)(seqidno:41)
通过rna印迹特异性检测l3的引物序列
5’-cttcttcgcggcctttgccttg-3’(lml3序列的核苷酸1242-1263的反向和互补)(seqidno:42)
通过rna印迹特异性检测l5的引物序列
5’-cttcttcgtgcgcacagcag-3’(lml5序列的核苷酸464-483的反向和互补)(seqidno:43)。l3和/或l5核酸分子的表达水平的检测或增强优选定义为与所述核酸分子在对照受试对象中的表达水平相比,所述核酸分子的表达水平的可检测的变化。通常,对照受试对象不包括该l3和/或l5核酸分子。优选地,l3和/或l5核酸分子表达水平的增强指利用pcr核苷酸序列的表达水平的至少5%的增加。
分析
在另一方面,提供了一种用于诊断受试对象寄生虫病的分析设备,其中,所述设备包括l3和/或l5源。任选地,s4和/或s6源与l3和/或l5源组合也存在于该分析设备中。可以通过任何本领域技术人员熟知的标准方法检测特异性识别所述源的抗体的存在(如参见通过引用的方式并入本文的harlow和lane,antibodies:alaboratorymanual,coldspringharborlaboratory,1988)。适当的方法包括亲和层析联合电泳(ace)分析和(酶联免疫吸附分析)elisa。优选地,所述分析包括elisa。以下将更广泛地描述多种分析方法。
在优选的实施方式中,分析涉及固定于以结合至固体支撑(solidsupport)上的l3和/或l5源的使用并除去样品中的抗体。然后可以利用与抗体/l3和/或l5源复合物结合的且具有可检测的信息基团的检测试剂检测所述结合的抗体。适当的检测试剂包括与抗体/l3和/或l5源复合物结合的抗体以及用信息基团标记的游离多肽(如半竞争性分析中)。替换地,可以使用竞争性分析,其中,与l3和/或l5源结合的抗体用信息基团标记且在带有样品的源温育后,将所述抗体结合于固定的l3和/或l5源。样品的成分抑制标记抗体与所述l3和/或l5源结合的程度表示样品与固定的l3和/或l5源的反应度。
固体支撑可以为本领域普通技术人员熟知的可以粘附l3和/或l5源的任何材料。例如,支撑可以为微量滴定板(microtiterplate)或硝化纤维或其他适当的膜中的试验孔(testwell)。替换地,支撑可以为小珠(bead)或圆盘(disc),如玻璃、玻璃纤维、乳胶或塑料材料如聚苯乙烯或聚氯乙烯。支撑还可以为磁性粒子或光纤传感器(fiberopticsensor),例如美国专利5,359,681中公开的那些材料。
可以利用各种本领域已知的技术将l3和/或l5源结合至所述固体支撑。在本发明的上下文中,术语“结合”既指非共价连接(如吸附),也指共价连接(可以是在抗原和支撑上的功能基团之间的直接连接或者可以是通过交联剂的方式的连接)。优选通过吸附结合至微量滴定板或膜中的孔。在这种情况下,可以通过将适当缓冲液中的l3和/或l5源与固体支撑接触适当的时间完成吸附。接触时间随着温度而变化,但是典型的在1小时和1天之间。通常,与塑料的微量滴定板(如聚苯乙烯或聚氯乙烯)的孔接触的l3和/或l5源的量在10ng-1g范围内,优选为100ng,足以结合充足量的l3和/或l5源。此外,当给出了一定数量或量的l3和/或l5源时,就确定了使用的l3和/或l5源的总量。
通常可以通过支撑与双官能团试剂的第一反应实现l3和/或l5源与固体支撑的共价连接,所述双官能团试剂将与支撑和功能基团(如多肽上的羟基或氨基)反应。例如,可以利用苯醌或通过支撑上的醛基与多肽上的氨基和活性氢的缩合将l3和/或l5源结合至具有适当聚合物涂层的支撑上(如参见pierceimmunotechnologycatalogandhandbook(1991)ata12-a13)。
在特定的实施方式中,分析为酶联免疫吸附分析(elisa)。该分析可以通过首先将已固定在固体支撑(通常为微量滴定板的孔)上的l3和/或l5源与样品接触,使得样品内l3和/或l5源特异的抗体与固定的l3和/或l5源结合进行。然后从固定源上除去未结合的样品并添加能够与固定的抗体-l3和/或l5源复合物结合的检测试剂。然后利用适于特异性检测试剂的方法测定仍然结合至固体支撑上的检测试剂的量。
一旦将l3和/或l5源固定在支撑上,通常将所述支撑上剩余的蛋白质结合位点封闭。可以使用本领域普通技术人员熟知的任何适当的封闭剂,如牛血清白蛋白(bsa)或吐温20(sigmachemical公司,圣路易斯,mo)。然后将固定的l3和/或l5源与样品一起温育,并使抗体(如果样品中存在)与所述源结合。可以在温育前用适当的稀释剂(如磷酸盐缓冲液(pbs))稀释样品。通常,合适的接触时间(即温育时间)为允许充分检测样品中抗体存在的时段。优选地,所述接触时间为足以获得结合程度至少为结合与未结合抗体间平衡时获得的95%。本领域普通技术人员可以认识的是,可以容易地通过分析时段内产生的结合程度而测得达到平衡所需的时间。在室温下,通常充足的温育时间约为30min。
然后可以通过用适当的缓冲液清洗固体支撑除去未结合的样品,如然后可以将含有0.1%吐温20的pbs检测试剂添加至固体支撑。适当的检测试剂为任何与固定的抗体-l3和/或l5源复合物结合的化合物且可以通过任何各种本领域熟知的方式检测。优选地,所述检测试剂含有与信息基团结合的结合剂(例如,如蛋白a、蛋白g、免疫球蛋白、凝集素或游离的抗原)。优选的信息基团包括酶(如辣根过氧化物酶)、底物、辅因子、抑制因子、染料、放射性核素(radionucleides)、发光基团、荧光基团和生物素。可以利用本领域普通技术人员熟知的标准方法实现结合剂与信息基团的结合。可以商购的常规结合剂与来自一些源(如zymedlaboratories,旧金山,caandpierce,罗克福德,il)的信息基团结合。
然后将检测试剂与固定的抗体l3和/或l5源复合物温育一段充足的时间以检测结合的抗体。通常可以从厂家的说明书或通过分析时段内产生的结合程度确定合适的时间。然后除去未结合的检测试剂并利用信息基团检测结合的检测试剂。
用来检测信息基团的方法取决于信息基团的性质。对于放射性基团,闪烁计数(scintillationcounting)或自动射线照相方法通常是合适的。可以使用光谱法检测染料、发光基团和荧光基团。可以利用联接至不同信息基团(通常放射性或荧光基团或酶)的抗生物素蛋白检测生物素。一般可以通过添加底物(通常为特定的时段),然后通过光谱或其他反应产物的分析方法检测酶信息基团。
为了确定样品中寄生虫病如利什曼病特异性抗体的存在或不存在,一般将检测到的来自仍结合至固体支撑上的信息基团的信号与相应的预测定的临界值(cut-offvalue)比较。在一种优选的实施方式中,临界值优选为当固定的l3和/或l5源与来自未感染的受试对象的样品温育时获得的信号的平均值。通常,产生在预测定的临界值以上为三个标准偏差的信号的样品被认为是阳性的(即与l3和/或l5源反应)。在替换的优选的实施方式中,根据sackettetal,clinicalepidemiology:abasicscienceforclinicalmedicine,p.106-7(littlebrownandco.,1985)的方法,利用接收器操作曲线(receiveroperatorcurve)测定所述临界值。简单地说,在该实施方式中,可以从多对对应于各个可能的诊断测试结果的临界值的真阳性率(即敏感性)和假阳性率(100%-特异性)的曲线(plot)中确定所述临界值。
曲线上最接近左上角(即围绕最大区域的值)的临界值是最准确的临界值,且产生比通过该方法确定的临界值高的信号的样品可以认为是阳性的。替换地,所述临界值可以沿曲线移动至左边,以减小假阳性率,或至右边,以减小假阴性率。
在相关的实施方式中,以穿流或带状测试形式(flow-throughorstriptestformat)进行分析,其中,将l3和/或l5源固定在膜如硝化纤维上。在穿流测试中,当样品穿过膜时,样品中的抗体与固定的l3和/或l5源结合。然后当含有检测试剂的溶液穿过膜时,检测试剂(如蛋白a胶体金(proteina-colloidalgold))与抗体-l3和/或l5源复合物结合。然后可以如上所述的进行结合的检测试剂的检测。在带状测试形式中,将结合有源的膜的一端浸入含有样品的溶液中。样品沿膜移动穿过含有检测试剂的区域至固定有多肽的区域。l3和/或l5源中检测试剂的浓度表示样品中存在引起寄生虫病如利什曼病的寄生虫的抗原的特异性抗体。通常地,那个位点上检测试剂的浓度会形成图案,如线条,是可见的。不存在该图案表示阴性结果。通常,如上所述,当样品含有抗体的量足以在elisa中产生阳性信号时,选择膜上固定的l3和/或l5源的量以产生视觉可辨别的图案。优选地,膜上固定的l3和/或l5源的量在25ng-500ng范围内。通常可以用很少量(如一滴)受试对象的血清或血液进行这样的测试。
如果在该分析设备中s4和/或s6源与l3和/或l5源组合使用,公开的用于l3和/或l5源的分析设备的各个部件也可以用于s4和/或s6源。
任何受试对象或医师可以在办公室/家中使用该设备,每当有需要时,重复使用该设备。
一般在分析中使用额外的分子作为阳性或阴性对照。典型的阳性对照可以为识别测试样品中已知存在的分子的抗体。典型的阴性对照可以为识别测试样品中已知存在的分子的抗体。
一般定义
在本发明的上下文中,蛋白质或蛋白片段由氨基酸序列表示。
在本发明的上下文中,核酸分子由编码蛋白质或多肽或蛋白片段的核酸或核苷酸序列表示。核酸分子可以包括调控区域。
应当理解的是,本文通过给出的序列号(seqidno)限定的各个核酸分子或蛋白质或蛋白片段并不限于所公开的特定序列。本文限定的各个基因序列或核苷酸序列编码给出的蛋白质或多肽或蛋白片段或自己本身是蛋白质或蛋白片段。贯穿本申请,每次提及的特定核苷酸序列seqidno(以seqidno:2或4为例),可以将其替换为:
i.含有与seqidno:2或4或49或51或53或55或65(示例)具有至少60%的序列一致性或相似性的核苷酸序列的核苷酸序列。
ii.与(i)的序列的核酸分子杂交的核苷酸序列的互补链;
iii.由于遗传密码的简并,与(iii)的核酸分子的序列不同的核苷酸序列。
iv.编码与由核苷酸序列seqidno:2或4或49或51或53或55或65编码的氨基酸序列具有至少60%的氨基酸一致性或相似性的氨基酸序列的核苷酸序列。
贯穿本申请,每次提及的特定氨基酸序列seqidno(以seqidno:1或3或48或50或52或54或56为例),可以将其替换为:
含有与氨基酸序列seqidno:1或3或48或50或52或54或56具有至少60%的序列一致性或相似性的氨基酸序列的多肽。
本文通过借助与给定的核苷酸序列或氨基酸序列的一致性或相似性百分比(至少60%)描述的各个核苷酸序列或氨基酸序列在优选的实施方式中分别与给定的核苷酸或氨基酸序列具有至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%的一致性或相似性或者分别与给定的核苷酸或氨基酸序列具有更多的一致性或相似性。在优选的实施方式中,通过比对前文限定的序列的全长确定序列一致性或相似性。
本文将“序列一致性”定义为两个或更多的氨基酸(多肽或蛋白质)序列间的关系或两个或更多的核酸(多核苷酸)序列间的关系,通过比对序列而确定。在优选的实施方式中,基于两个给出的seqidno的全长或其部分计算序列一致性。其部分优选指两个seqidno的至少50%、60%、70%、80%、90%或100%。本领域中,“一致性”还指氨基酸或核酸序列间序列相关性的程度,根据具体情况,通过这些序列的条带(strings)间的匹配确定。
通过将氨基酸序列及其一个多肽的保守氨基酸取代与第二个多肽序列比对确定两个氨基酸序列间的“相似性”。可以通过已知的方法容易地计算“一致性”和“相似性”,包括但不限于这些文献中描述的方法:computationalmolecularbiology,lesk,a.m.,ed.,牛津大学出版社,纽约,1988;biocomputing:informaticsandgenomeprojects,smith,d.w.,ed.,学术出版社,纽约,1993;computeranalysisofsequencedata,parti,griffin,a.m.,andgriffin,h.g.,eds.,胡马纳出版社,新泽西,1994;sequenceanalysisinmolecularbiology,vonheine,g.,学术出版社,1987;andsequenceanalysisprimer,gribskov,m.anddevereux,j.,eds.,mstockton出版社,纽约,1991;以及carillo,h.,andlipman,d.,siamj.appliedmath.,48:1073(1988)。
设计确定一致性的优选方法以在测试的序列间产生最大的匹配。确定一致性和相似性的方法编纂在公开可用的计算机程序中。优选的确定两个序列一致性和相似性的计算机程序方法包括例如gcg程序包(devereux、j.,etal.,nucleicacidsresearch12(1):387(1984))、bestfit、blastp、blastn和fasta(altschul、s.f.etal.,j.mol.biol.215:403-410(1990)。blastx程序是ncbi和其他来源(blastmanual、altschul,s.,etal.,ncbinlmnihbethesda,md20894;altschul,s.,etal.,j.mol.biol.215:403-410(1990))公开可获得的。还可以使用众所周知的smithwaterman算法来确定一致性。
优选的多肽序列比对参数包括如下:规则系统(algorithm):needlemanandwunsch,j.mol.biol.48:443-453(1970);比对矩阵(comparisonmatrix):blossum62来自hentikoffandhentikoff,proc.natl.acad.sci.usa.89:10915-10919(1992);间隙罚分(gappenalty):12和间隙长度罚分(gaplengthpenalty):4。就像来自位于麦迪逊,威斯康星州(madison,wi)的遗传学电脑集团的“ogap”程序一样,带有这些参数的有用的程序是公开可获得的。前述的参数为氨基酸比对(闭口间隙(endgaps)不伴随罚分(penalty))的默认参数。
优选的核酸比对参数包括如下:规则系统:needlemanandwunsch,j.mol.biol.48:443-453(1970);比对矩阵:匹配(matches)=+10,失配(mismatch)=0;间隙罚分:50;间隙长度罚分:3。可获得如来自麦迪逊,威斯康星州(madison,wis)的遗传学电脑集团的间隙程序。以上给出的是核酸比对的默认参数。
任选地,本领域技术人员清楚的是,在确定氨基酸相似性程度时,本领域技术人员还可以考虑称为“保守的”氨基酸取代。保守的氨基酸取代指的是具有相似的侧链的残基的可替换性。例如,一组具有脂肪族侧链的氨基酸是甘氨酸、丙氨酸、缬氨酸、亮氨酸和异亮氨酸;一组具有脂肪族-羟基的侧链的氨基酸是丝氨酸和苏氨酸;一组具有含酰胺侧链的氨基酸是天冬酰胺和谷氨酰胺;一组具有芳香族侧链的氨基酸是苯丙氨酸、酪氨酸和色氨酸;一组具有碱性侧链的氨基酸是赖氨酸、精氨酸和组氨酸;以及一组具有含硫侧链的氨基酸是半胱氨酸和蛋氨酸。优选的保守氨基酸取代组为:缬氨酸-亮氨酸-异亮氨酸、苯丙氨酸-酪氨酸、赖氨酸-精氨酸、丙氨酸-缬氨酸、天冬酰胺-谷氨酰胺。本文公开的氨基酸序列取代变体为公开的序列中至少一个残基缺失且不同的残基在其位置插入的氨基酸序列。优选地,氨基酸的变化是保守的。优选的各个自然发生的氨基酸的保守取代如下:ala到ser;arg到lys;asn到gln或his;asp到glu;cys到ser或ala;gln到asn;glu到asp;gly到pro;his到asn或gln;ile到leu或val;leu到ile或val;lys到arg;gln或glu;met到leu或ile;phe到met、leu或tyr;ser到thr;thr到ser;trp到tyr;tyr到trp或phe;以及val到ile或leu。
核酸构建体
核酸构建体包括编码本文定义的蛋白质或蛋白片段的核苷酸序列。包括编码本文定义的给定的蛋白质或蛋白片段的核酸分子的核酸构建体将确保给定的核酸分子、以及相应的蛋白质或蛋白片段在治疗对象中的表达。在更优选的实施方式中,核酸构建体包括1个以上的核酸分子,各个核酸分子编码给定的蛋白质或蛋白片段。在甚至更优选的实施方式中,核酸构建体包括2个、3个、4个核酸分子,各个核酸分子编码给定的蛋白质或蛋白片段。在优选的实施方式中,核酸构建体包括表达盒,所述表达盒包括各种所需的核酸分子。各个核酸分子可操作地与存在的其他核酸分子相连。最优选地,适当的启动子可操作地与所述表达盒相连,以确保受试对象中核酸分子的表达。
在本文和其权利要求中,动词“包括”及其词形变化(conjugations)以其非限制性的意义使用,意指含有词后的那些项,但是没有排除未明确提到的项。另外,动词“组成”可以由“基本上由…组成”替换,意指除了明确鉴定的成分外,本文定义的产物或组合物或l3或l5源可以包括其他的成分,所述其他的成分不会改变本发明的独特特征。
另外,关于用不定冠词“a”或“an”修饰的元件不排除存在一个以上的可能性,除非上下文中明确要求存在一个且仅一个元件。因此不定冠词“a”或“an”通常指“至少一个”。
将本说明书中引用参考的所有专利和文献通过引用其全部内容的方式并入本文。
以下通过实施例进一步阐述本发明,实施例不应当理解为是对本发明范围的限制。
附图说明
图1.重组硕大利什曼原虫rlml3和rlml5蛋白质的表达和纯化。诱导后(泳道1)、通过ni-nta琼脂糖柱传代后(泳道2)以及纯化的重组rlml3和rlml5(泳道3)的pqelml3(板l3)或pqelml5(板l5)载体转染的大肠杆菌溶菌产物的考马斯亮蓝染色的(coomassie-blue-stained)13%聚丙烯酰胺凝胶。
图2a-2c.l3和l5蛋白的抗原性
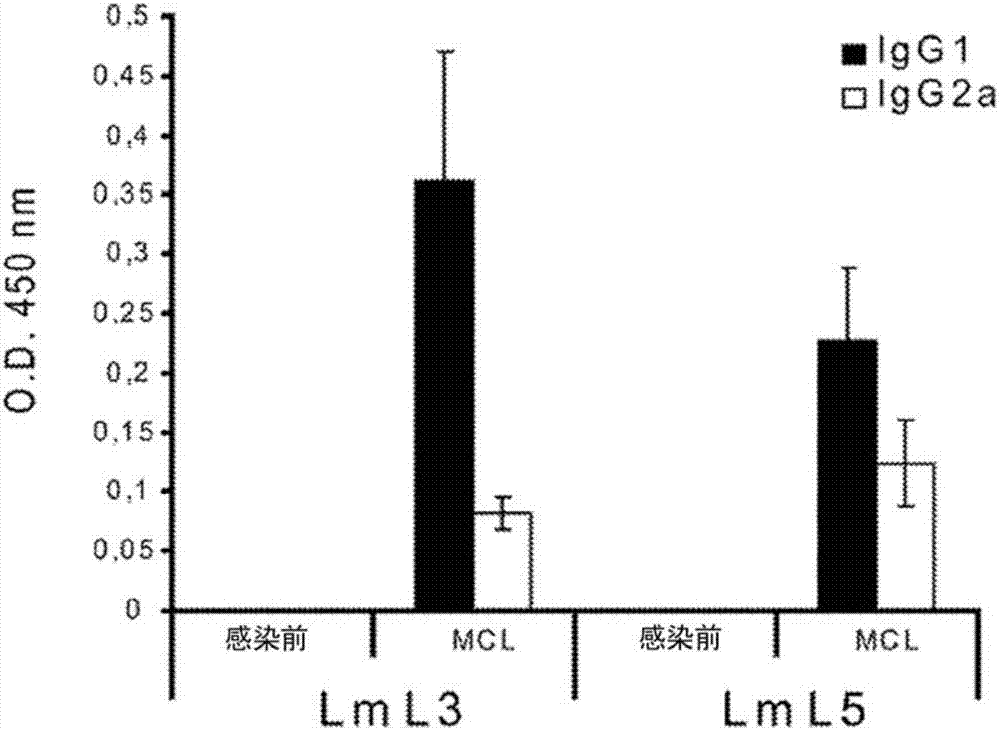
(a)用5×104的硕大利什曼原虫稳定期的前鞭毛体s.c.感染12只balb/c小鼠的左脚掌并在攻击后8周获得血清。通过elisa分别确定患有皮肤利什曼病(mcl)的小鼠血清的igg1和igg2a抗体抗rlml3和rlml5的相关性(relativities)。还表征了感染前相同小鼠血清缺乏相关性。患有有症状cvl犬的血清和抗rlml3(b)和rlml5(c)重组蛋白质的对照组血清的elisa相关性。
图3a-3f.balb/c小鼠中通过接种疫苗诱导的细胞因子产物。将balb/c小鼠(每组6只)用10μg的rlml3+50μg的cpg-odn(l3+cpg)或10μg的rlml5+50μg的cpg-odn(l5+cpg)、用50μg的cpg-odn(cpg)或用pbs(生理盐水)s.c.免疫三次。接种疫苗后4周获得脾脏细胞并于存在rlml3(a、c和e)、rlml5(b、d和f)和仅培养基中体外培养48h。通过elisa评估培养物上层清液中ifn-γ(a和b)、il-4(c和d)和il-10(e和f)的水平。各条代表来自单个小鼠的数据的平均值±sd。
图4a-4b.攻击后balb/c接种疫苗的小鼠中硕大利什曼原虫感染的过程。(a将给出的脚掌肿胀作为感染和未感染的对侧脚掌间厚度的差异。(b在攻击后8周,通过有限稀释各自确定感染了的腿的腿弯部(poplyteal)引流淋巴结和脾脏中有活性的寄生虫的量。(*,p<0.01)
图5a-5c.l3+cpgodn接种疫苗的小鼠中由感染引起的细胞免疫反应。寄生虫攻击后8周,制备脾脏细胞培养物。将细胞不刺激(培养基)或在37℃下于5%co2中,分别用lrp(利什曼原虫核糖体蛋白质)(12μgml-1)、sla(溶解的利什曼原虫抗原)(12μgml-1)、mrp(鼠核糖体蛋白质)(12μgml-1)或l3(6μgml-1)刺激48h。通过捕获酶联免疫吸附方法(captureenzyme-linkedimmunosorbentassay)测量培养物上层清液中ifn-γ(a,)il-4(b)和il-10(c)水平。各条代表每组6只单个小鼠中细胞因子水平的平均值加上标准偏差。
图6a-6c.l5+cpgodn接种疫苗的小鼠中由感染引起的细胞免疫反应。寄生虫攻击后8周,制备脾脏细胞培养物。将细胞不刺激(培养基)或在37℃下于5%co2中,分别用lrp(12μgml-1)、sla(12μgml-1)、mrp(12μgml-1)或l5(6μgml-1)刺激48h。通过捕获酶联免疫吸附方法测量培养物上层清液中ifn-γ(a)il-4(c)和il-10(b)水平。各条代表每组6只单个小鼠中测定的细胞因子水平的平均值加上标准偏差。
图7a-7b.攻击后balb/c接种疫苗的小鼠中巴西利什曼原虫感染的过程。(a)感染耳朵的炎性病变。病变尺寸(毫米)用在5只小鼠上进行一个实验获得的平均值±sd表示。用rlml5+cpg-odn或rlml3+cpg-odn接种疫苗的以及两对照组的小鼠间的*p<0.05。(b)在感染后5周,测定耳朵真皮中寄生虫载量。结果用每组5个耳朵的平均值±sd表示。rlml5+cpg-odn和两对照小鼠组间*p<0.05显著下降。
图8a-8b.lml3和lml5蛋白的核糖体位置。将1μg的rlml3蛋白(a)、1μg的rlml5蛋白(b)和10μg的硕大利什曼原虫lrp提取物(a和b)在线性10-13%梯度sds-page凝胶上进行电泳。考马斯亮蓝染色的凝胶如左边板所示(a和b)。将相等的凝胶与用rlml3(板α-lml3)(a)免疫的小鼠的血清和五犬内脏利什曼病血清(板α-lml5)(b)的亲和纯化的抗lml5抗体部分杂交并探测。
图9a-9b.在cpg的存在下用核糖体蛋白质s6、l2、l7、l8和l6接种疫苗的小鼠中感染参数的分析。(a)每周检测感染组的病变发展直至感染后8周。感染后8周,s6加cpgodn和对照(生理盐水和佐药)组间脚掌肿胀的差异是统计上显著的(*p<0.05)。(b)感染后8周,分析dlns和脾脏中寄生虫载量测定。s6加cpgodn接种免疫组和对照(生理盐水和佐药)组的脾脏中寄生虫负载的差异是统计上显著的(*p<0.05)。为了简明,仅显示了生理盐水组和s6加cpgodn接种免疫组的sd。
图10a-10b.s6加cpgodn接种疫苗引起的免疫反应。(a)在最后一次剂量给药后,从用s6加cpgodn、cpgodn和生理盐水免疫了4周的小鼠中获得血清样品。通过elisa分别测试血清以确定抗-s6特异性igg、igg1和igg2a抗体的存在。s6加cpgodn组和对照(生理盐水和cpg)组间统计上显著的差异(*p<0.05)。(b)将脾脏细胞不刺激(培养基)或在37℃下于5%co2中用s6刺激48h。通过捕获酶联免疫吸附方法测量培养物上层清液中ifn-γ、il-4和il-10水平。各条代表每组4只单个小鼠中测定的细胞因子水平的平均值加上标准偏差。s6加cpgodn组和对照(生理盐水和cpg)组间统计上显著的差异(*p<0.05)。
图11a-11b.在cpg的不存在或存在下用l3、l5和s4单独或混合制剂免疫的小鼠中病变的发展。每周检测病变直至感染后6周。(a)对照小鼠组和用不含佐药的重组蛋白质接种疫苗的小鼠组。(b)对照小鼠组和用重组蛋白质加cpgodn接种疫苗的小鼠组。感染后6周,l3加cpgodn、l5加cpgodn、l3加l5加cpgodn或l3加l5加s6加cpgodn以及对照(生理盐水和佐药)组间脚掌肿胀的差异是统计上显著的(*p<0.05)。
图12a-12b.接种疫苗的小鼠中寄生虫载量。感染后7周,确定脾脏和dln中的寄生虫负载。(a)对照小鼠组和用不含佐药的重组蛋白质接种疫苗的小鼠组。(b)对照小鼠组和用重组蛋白质加cpgodn接种疫苗的小鼠组。l3加cpgodn、l5加cpgodn、l3加l5加cpgodn或l3加l5加s6加cpgodn以及对照(生理盐水和佐药)组间脾脏寄生虫载量的差异是统计上显著的(*p<0.05)。l3加l5加cpgodn或l3加l5加s6加cpgodn以及对照(生理盐水和佐药)组间腿弯部(popliteal)寄生虫载量的差异是统计上显著的(*p<0.05)。
图13.提出的嵌合结构的图示。显示了为克隆设计的引物和选择的酶切位点。
具体实施方式
实施例
实施例1:硕大利什曼原虫l3和l5蛋白的克隆以及这些蛋白赋予的对硕大利什曼原虫感染的防护作用
材料和方法
小鼠菌株和寄生虫
雌性balb/c小鼠(6-8周大)购于harlaninterfaunaibéricas.a.(巴塞罗那,西班牙)。
硕大利什曼原虫寄生虫(whom/ir/-/173)通过在balb/c小鼠中传代而保持在易传染的状态(virulentstate)。通过在补充有20%的胎牛血清的schneider’s培养基(gibco,brl,格兰德岛,纽约,美国)中于26℃下培养而获得硕大利什曼原虫无鞭毛体并转化成前鞭毛体。
cpg-odn
硫代磷酸修饰的cpg-odn(5’-tcaacgttga-3’和5’-gctagcgttagcgt-3’)(seqidno:5、6)通过等基因(isogen)(荷兰)合成。
编码硕大利什曼原虫核糖体蛋白l3和l5的dna序列的克隆。
从硕大利什曼原虫基因组数据库(www.genedb.org/genedb/leish)利用酿酒酵母(s.cerevisiae)l3和l5蛋白序列作为探针(56)获得编码硕大利什曼原虫l3和l5蛋白的开放阅读框(orf)。对于rlml3和rlml5蛋白的表达,使用硕大利什曼原虫(mhom/il/80(friedlin))的dna作为模板pcr扩增其编码区(cr)。将扩增的dnas克隆至pbluescript质粒(stratagene,lajolla加拿大)并在克隆至pqe30表达载体(qiagen,希尔登,德国)前进行测序。
lml3cr克隆使用的引物为:正义链,5'-cgggatccatgtctcactgcaagttcgag-3’(lml3cr的位置1-20(lmjf34.2880))(seqidno:44);反义链,5’-aactgcagttacttcttcgcggcctttg-3’(与lml3cr的位置1241-1260反向和互补(lmjf34.2880))(seqidno:45)。包括bamhi和psti限制酶切位点(画有下划线)用于克隆的目的。
lml5cr克隆使用的引物为:正义链,5'-cgggatccatgtgcacgctggcaaattg-3’(lml5cr的位置1-20(lmjf35.1890))(seqidno:46);反义链,5'-cccaagcttttacttgccgaggcgctcgc-3’(与lml5cr的位置968-987反向和互补(lmjf35.1890.319))(seqidno:47)。包括bamhi和hindiii限制酶切位点(画有下划线)用于克隆的目的。
蛋白质的纯化
rlml3和rlml5蛋白在用pqe-lml3或pqe-lml5质粒转化的大肠杆菌中过表达并在变性的条件下于ni-次氮基三乙酸(ni-nta)琼脂糖柱(qiagen)上纯化。结合到ni-nta琼脂糖上后,重组蛋白质在所述亲和柱上重折叠(57)。所述重组蛋白质穿过多粘菌素-琼脂糖(sigma,圣路易斯,密苏里州)。用定量显色鲎变形细胞法(quantitativechromogeniclimulusamebocyteassay)qcl-1000(biowhittaker,walkersville,马里兰州)测定残余的内毒素含量(<12pg/μg的重组蛋白质)。
免疫、寄生虫攻击和寄生虫的定量
用各自与25μg的cpg-odn混合的10μg的rlml3或10μg的rlml5在两个独立的balb/c小鼠组(每组六只)的右脚掌(rightfootpad)上皮下(s.c.)接种。作为对照,分别仅用25μg的cpg-odn或用具有磷酸盐缓冲液(pbs)的cpg-odn接种两个另外的小鼠组。2和4周后,每组使用用于启动(priming)的相同剂量进行提升(boosted)。最后一次接种后4周,通过在左(未处理)脚掌s.c.接种5×104稳定期的硕大利什曼原虫(whom/ir/-/173)的前鞭毛体进行寄生虫攻击。用度量卡尺(metriccaliper)测定脚掌肿胀并计算左脚掌的厚度减去右脚掌的厚度。通过所述的有限稀释测定法(limitingdilutionassay)(70)确定耳朵、引流淋巴结(dln)和脾脏中的寄生虫数量。
利什曼原虫抗原和小鼠核糖体蛋白。
对于硕大利什曼原虫lrp的制备,收获109个前鞭毛体,在预冷的pbs中洗涤两次并悬浮于1ml的np40裂解缓冲液(10mmtrishcl、ph8.0、150mmnacl、1.5mmmgcl2和0.5%np40)中并用移液器吸取来回10次。裂解后,在4℃下将样品以3000×g微量离心(microfuged)2min,以使细胞核团成小球(pelletthenuclei)。在4℃下将上层清液以13000×g微量离心15min并如[57]所述地从细胞溶质上层清液中制得核糖体。简单地说,在4℃下将细胞溶质置于beckmantl100.3转子中以90000rpm高速离心30min。将未加工的核糖体小球(pellet)重新悬浮于缓冲液a(20mmtris-hcl、ph7.4、500mmacnh4、100mmmgcl2、5mmβ-巯基乙醇)中并在4℃下置于tl100.3转子中以90000rpm离心通过缓冲液a中的不连续蔗糖梯度(20/40%)。将洗过的核糖体小球溶解于pbs中并用超声波处理直至核糖体rna完全降解。利用相同的方法从5×107raw264.7鼠科动物的巨噬细胞中制得小鼠核糖体蛋白提取物(mrp)。
如(70)所述地制备硕大利什曼原虫的总蛋白(可溶的利什曼原虫抗原[sla])。简单地说,将硕大利什曼原虫前鞭毛体(1010)在pbs中洗涤两次,重新悬浮于500ml的pbs中并通过三次冷冻和解冻的循环进行裂解。细胞裂解后,通过利用微型离心机以12000×g离心15min从不溶的部分分离可溶的抗原。将上清液等分(aliquoted)并贮存于-70℃。
上层清液中细胞因子的测量
利用商用elisa试剂盒(diaclone,
在小鼠和犬中抗-l3和抗-l5抗体反应的检测
从埃斯特雷马杜拉区(extremaduraregion)(西班牙)的20只天生被婴儿利什曼原虫感染的有临床症状的犬中收集犬血清样品。在西班牙克瑞斯的埃斯特雷马杜拉大学兽医院寄生虫系(thedepartmentofparasitologyoftheveterinaryschool,extremadurauniversity,cáceres,spain)临床分析评价感染的动物。当通过间接免疫荧光法测试时,所有的血清是呈阳性的,并通过直接观察确定腿弯部和前肩胛骨淋巴结中寄生虫无鞭毛体形式的存在。从寄生虫系(埃斯特雷马杜拉大学)饲养的8只健康动物中获得对照组血清。
感染后8周,收集用实验方法感染5×104个稳定期硕大利什曼原虫(whom/ir/-/173)前鞭毛体的12只balb/c小鼠的血清。作为对照,收集相同小鼠感染前的血清。
在室温下用100μl各个重组核糖体蛋白(pbs中2μgml-1)包被标准elisa板过夜。在pbs-tween20(0.5%)-酪蛋白(5%)中以1/200的稀释度分析犬和鼠的血清样品。使用购于北欧免疫学实验室(nordicimmunologicallaboratories)(蒂尔堡,荷兰)的第二抗体辣根过氧化酶偶联的抗-犬-igg(1/1000)、抗-鼠-igg1(1/1000)和抗-鼠-igg2a(1/500)。使用邻苯二胺氢氯化物(ortophenylediaminedihydrochloride)(opd)(dako,a/s,葛洛斯楚普,丹麦)作为elisa分析的过氧化物酶底物。15min后,添加100μl的h2so41m终止反应并读取450nm下的吸光度。
统计分析
通过学生t测试进行统计分析。当p<0.05时,认为差异是显著的。
结果和讨论
lml3和lml5蛋白的鉴定、克隆和表达
为了鉴定硕大利什曼原虫l3和l5的编码区,我们进行了blastp检索,利用酿酒酵母l3和l5的氨基酸序列(分别为yor063w和ypl131w)作为探针[58]。将注释为推定lml3和lml5蛋白的两个不同的词条(lmjf34.2880和lmjf35.1890)用blast分值的显著值显示(rescued)。基于数据库序列数据,设计pcr引物以扩增lml3和lml5cr,包括克隆目的的不同限制性内切酶酶切位点。对扩增的dnas在pbluescript中进行亚克隆并测序。硕大利什曼原虫推定的l3蛋白拥有分子量为47.5kda的419氨基酸和11.67的预测等电点。硕大利什曼原虫推定的l5蛋白拥有分子量为36.6kda的328氨基酸和10.69的预测等电点。硕大利什曼原虫l3和l5推导氨基酸序列与它们的酿酒酵母对应物的比较揭示了高度的同源性:l3蛋白57%的一致性,73.5%的相似性;l5蛋白51.2%的一致性,66.3%的相似性(见比对图)。本文中的比对图显示了利什曼原虫l3和l5蛋白含有一些具有高度相似性的区域。还明显地观察到这两种寄生虫的蛋白质比酵母的蛋白质长,在lml3的羧基端和lml5的两端存在额外的氨基酸残基。属于保守蛋白家族的利什曼原虫蛋白质不寻常的一级结构似乎与免疫相关,因为在感染过程中引起的抗这些蛋白质的体液和细胞反应能够特异性地抗寄生虫,而不与宿主对应物的同源蛋白交叉反应(39、36、58)。
在pqe30表达载体中将lml3和lml5cr亚克隆。推导重组蛋白质的氨基酸序列如比对图的c和d部分所示。两种蛋白质均存在包括用于亲和层析纯化的6个组氨酸的n-端标记。随后,两种蛋白质在大肠杆菌培养物中过表达并被纯化。如图1所示,根据它们n-端区12个氨基酸额外的组氨酸-标记延伸(stretch)的存在,纯化的rlml3和rlml5蛋白质显示明显的分子量分别为48kda和38kda。因为在考马斯亮蓝染色sds-page凝胶(图1)中两个纯化的重组蛋白质均观察到了单一的条带,所以确定了蛋白质的纯度。
通过犬内脏利什曼病(cvl)血清和来自感染了硕大利什曼原虫的balb/c小鼠(mcl)的血清识别lml3和lml5。
为了确定vl影响的犬体内硕大利什曼原虫l3和l5蛋白质的抗原性,在利用20只婴儿利什曼原虫感染的犬的血清的elisa分析中,使用重组的rlml3和rlml5蛋白质作为抗原。作为对照,分析从8只健康犬获得的血清对两个重组蛋白质的反应。cvl血清75%(15/20)识别rlml3蛋白质(图2b)且90%(18/20)识别反应活性值(reactivityvalues)高于阻断值(cutoffvalue)的rlml5蛋白质(图2c)。抗rlml3和rlml5间的吸光值的光谱不同,因为cvl血清抗rlml5的反应活性高(平均值=0.61±0.36)于rlml3获得的反应活性(平均值=0.28±0.10)。可以推论,犬天生利什曼病的免疫系统中,暴露的两种寄生虫蛋白质是lml5蛋白比lml3蛋白与免疫原更相关。虽然此处使用的血清量有限,但是获得的数据可以作为可以与其他抗原组合使用两种重组寄生虫蛋白质的指示,用于促进cvl的血清学诊断测试。
接下来我们利用由于感染硕大利什曼原虫而患有皮肤利什曼病(mcl)的balb/c小鼠血清分析了lml3和lml5蛋白质的抗原性。为了那个目的,通过elisa分析了抗两种重组蛋白质的igg1和igg2a抗体的存在。两种蛋白质被mcl血清识别,mcl血清为igg1同型的主要抗它们而引起的抗体(图2a)。感染前相同小鼠中未观察到抗它们的反应活性(图2a)。由于分别将igg1和igg2a抗体的诱导用作th2-型和th1-型免疫反应的标志(8),我们可以推断在balb/c小鼠感染硕大利什曼原虫的过程中,诱导了th2-样体液反应来抗这些抗原。
在cpgodn存在下用rlml3和rlml5重组核糖体蛋白免疫balb/c小鼠诱导了抗它们的th1-型反应。
由于在感染的balb/c小鼠中引起了抗它们的th2介导的体液反应,我们分析了在th1诱导佐药(cpg-odn)存在下的rlml3和rlml5的免疫效果。为了那个目的,将6只小鼠的组各自用结合cpg-odn的rlml3和rlml5进行免疫。作为对照,将6只小鼠的组仅用cpg-odn和pbs(用作辅料的缓冲液)进行免疫。三次剂量后,分析由免疫带来的细胞反应。获得脾脏细胞并在相应rlml3和rlml5抗原的存在和不存在下培养。在rlml3抗原刺激后,用rlml3+cpg-odn免疫了的小鼠的脾脏细胞产生高水平的ifn-γ(图3a)。在rlml5抗原刺激后,在用rlml5+cpg-odn免疫了的小鼠的脾脏细胞上层清液中检测相似的ifn-γ水平(图3b)。相反,在对rlml3或rlml5刺激的反应中,用佐药或辅料免疫了的小鼠的脾脏细胞产生低水平的ifn-γ(图3ab)。从分别用rlml3+cpg-odn(图3c)或rlml5+cpg-odn(图3d)免疫了的小鼠获得的脾脏细胞的rlml3或rlml5刺激后,对il-4产物,检测到该细胞因子非常低的水平。最后,在任何组中都没有检测到il-10特异性产物(图3ef)。因此,可以推断cpg-odn佐药使抗重组抗原对th1反应的免疫反应偏斜(skews)。
rlml3+cpg-odn和rlml5+cpg-odn的疫苗接种保护balb/c小鼠抗硕大利什曼原虫的攻击。
我们分析了给药两种重组蛋白质是否能够诱导保护易受感染的balb/c小鼠抗硕大利什曼原虫感染。rlml3+cpg-odn或rlml5+cpg-odn接种疫苗的小鼠的脚掌肿胀明显小于pbs或cpg-odn对照组的脚掌肿胀(图4a)。另外,在rlml3+cpg-odn或rlml5+cpg-odn免疫的小鼠的引流淋巴结细胞中,观察到了大约2-log寄生虫载量(parasiteburden)的下降。最后,在脾脏中不能检测到寄生虫,然而在两个对照组小鼠的脾脏中都检测到了寄生虫(图3b)。可以推断用寄生虫lml3和lml5蛋白(表示为重组蛋白质)与cpg-odn混合免疫了的小鼠被保护抗硕大利什曼原虫感染。接种疫苗的小鼠真皮病状不存在或很低。在这些小鼠中,寄生虫的存在受腿弯部引流淋巴结的限制。观察到的防护与通过用编码利什曼原虫核小体组蛋白(19)、如dna疫苗一样给药的p0蛋白(54)和与cpg-odn结合的lrp提取物(55)的质粒dna混合物的小鼠的免疫而获得的防护相似。因此,我们已确定了两种核糖体成分的特征,所述两种核糖体成分的免疫可以促进抗利什曼病的有效分子定义的疫苗的更合理的发展。
与防护相关的免疫参数的分析
为了确定与防护相关的免疫参数,攻击后8周,分析接种疫苗的小鼠组和对照小鼠组中由sla、lrp、mrp和相应的重组蛋白质驱动的细胞因子产物(ifn-γ,il-4和il-10)。攻击后8周,l3和l5接种疫苗的小鼠比对照组小鼠的脾脏细胞产生更多的sla、lrp和重组抗原特异性ifn-γ(图5a为l3的且图6a为l5的)。因为脾脏细胞与mrp的培养物的刺激不导致该细胞因子的产生,发现ifn-γ产物特异性地由利什曼原虫核糖体蛋白质诱导(图5a为l3的且图6a为l5的)。另外,与对照组小鼠(cpg和生理盐水)相比,从防护小鼠获得的脾脏细胞上层清液中发现了更低水平的sla和lrp特异性il-10(图5b为l3的且图6b为l5的)和il-4(图5c为l3的且图6c为l5的)。另外,在防护的小鼠中,il-10和il-4的mrp-特异性l3和l5依赖的产物很低。因此,可以推断感染后,防护的表型与能够控制由寄生虫引起的il-4和il-10反应的l3和l5th1反应的诱导相关。
lml3和lml5序列数据的比对图。硕大利什曼原虫l3(a)和l5(b)蛋白与它们的酿酒酵母同源基因的氨基酸序列比对图。保守氨基酸加有阴影。显示了lml3和lml5氨基酸数量。从它们相应的dna序列预测硕大利什曼原虫氨基酸序列。(c和d)重组lml3(rlml3)(c)和lml5(rlml5)(d)蛋白的氨基酸序列或预测的氨基酸序列在细菌中表达。位于它们n-端的额外组氨酸-标记序列加粗并画下划线表示。显示了lml3和lml5蛋白的氨基酸数量。
a
b
c
d
实施例2:l3和l5同系物的分析
l3和l5同系物的比对图
我们分析了不同利什曼原虫物种间l3和l5核糖体蛋白质保守的程度。为了那个目的,通过基因组数据库(www.genedb.org)的生物信息学(insilico)分析并与硕大利什曼原虫同源基因的氨基酸序列(seqidno:1为l3的且seqidno:3为l5的)(比较的比对图在下页,部分a和b))比较显示(rescued)了婴儿利什曼原虫和墨西哥利什曼原虫的l3蛋白(seqidno:48and50)以及婴儿利什曼原虫(克隆jpcm5[mcan/es/98/llm-877])、巴西利什曼原虫(mhom/br/75/m2904)和墨西哥利什曼原虫(mhom/gt/2001/u1103)(seqidno:52、56和54)的l5蛋白的氨基酸序列。硕大利什曼原虫l5蛋白中除了n-端延伸的存在,还观察到了不同物种间的高度保守。表1a-b分别显示了l3和l5同源基因间的一致性和相似性百分比。
硕大利什曼原虫l3和l5在核糖体中的位置
为了论证该工作使用的重组lml3和lml5对应于位于寄生虫核糖体中的蛋白质,我们使用在含有重组蛋白质和lrp提取物的蛋白印迹中对重组蛋白质特异性的抗体。图8a显示了从用重组蛋白质免疫了的小鼠中获得的抗-lml3抗体识别了在lrp提取物中具有预期分子量(47,5kda)的单个条带。对于l5蛋白观察到了的相似的结果(图8b)。当我们如前所述使用通过rlml5-shepharose4b柱中的亲和层析从婴儿利什曼原虫天生感染犬血清中纯化的抗-lml5抗体时,在lrp提取物中观察到了36,6kda的单个条带(71)。作为阳性对照,在两种情况下,通过特异性抗体识别相应的重组蛋白质。
利什曼原虫l3和l5序列比较。硕大利什曼原虫、婴儿利什曼原虫、婴儿利什曼原虫和墨西哥利什曼原虫l3(a)(seqidno:1、48和50)以及硕大利什曼原虫、巴西利什曼原虫、婴儿利什曼原虫和墨西哥利什曼原虫l5(b)(seqidno:3、56、52和54)的氨基酸序列比对图。利用clustalw(dnastar软件)的默认设置比对婴儿利什曼原虫(genedbidentifierlinj32_v3.3320)和墨西哥利什曼原虫(genedbidentifierlmxm33.2900)的lml3蛋白、推定的l3蛋白序列,以及巴西利什曼原虫(genedbidentifierlbrm34_v2.1790)、婴儿利什曼原虫(genedbidentifierlinj35_v3.1870)和墨西哥利什曼原虫(genedbidentifierlmxm34.1880)的lml5蛋白和推定的l5蛋白序列。氨基酸取代加有阴影。
a
b
表1
a
b
显示了一致性和相似性值(括号内)。
实施例3:用rlml3+cpg-odn和rlml5+cpg-odn保护balb/c小鼠抗巴西利什曼原虫攻击的接种免疫
在由感染巴西利什曼原虫引起的皮肤利什曼病的发展中,我们分析了硕大利什曼原虫重组蛋白质rlml3和rlml5与th1诱导佐药(cpg-odn)结合的免疫的效果。为了那个目的,5只小鼠的组各自用10μg的rlml3或10μg的rlml5与50μg的cpg-odn(25μg的cpg-1[5’-tcaacgttga-3’]加25μg的cpg-2[5’-gctagcgttagcgt-3’)](seqidno:5和6))结合进行免疫。作为对照,5只小鼠的组仅用50μg的cpg-odn或用pbs(用作辅料的缓冲液)进行免疫。在小鼠耳朵真皮(左耳)处接种。2和4周后,将各个组用与启动时使用的相同剂量进行提升。通过将105稳定的巴西利什曼原虫(mhom/br/01/ba788)前鞭毛体与两对长须罗蛉(lutzomyaintermedia)沙蝇唾腺结合注射到右(未处理)耳中进行寄生虫攻击。在该感染模式中,寄生虫攻击balb/c小鼠在感染耳朵处产生炎性病变后,所述炎性病变稳定变化并在大约5周时达最大限度。此后,病变尺寸复原且在感染后约9周时观察到了耳朵完全结疤(72)。我们分析了4个小鼠组中皮肤病变的发展,直到寄生虫攻击后5周,用度量卡尺测量病变的厚度。如图7a所示,与cpg-odn对照组的pbs相比,rlml3+cpg-odn或rlml5+cpg-odn接种疫苗的小鼠的耳朵病变明显更低。另外,感染后5周,分析感染的耳朵中的寄生虫载量。通过所述的有限稀释测定法确定寄生虫量(73)。当与对照组相比时,在接种疫苗的小鼠中观察到了寄生虫载量的下降。发现rlml5+cpg-odn组相对于两个对照组的差异是显著的(p<0.05,t分布检验(t-studenttest))。而且,5只小鼠(rlml5+cpg-odn组)中的4只和rlml3+cpg-odn组5只小鼠中的1只的耳朵中未检测到寄生虫(图7b)。因此,可以推断用表示为重组蛋白质并与cpg-odn结合的硕大利什曼原虫l3和l5核糖体蛋白质接种疫苗的小鼠被保护抗不同巴西利什曼原虫的攻击。
实施例4:硕大利什曼原虫s6赋予的抗硕大利什曼原虫感染的部分防护
在由前面的seqidno:5和6定义的cpgodn(50μg)的存在下,各自用10μg的s6(seqidno:34)、l2(seqidno:22)、l7(seqidno:24)、l8(seqidno:26)和l16(seqidno:28)免疫5个小鼠组(每组n=4)。作为对照,用佐药免疫小鼠组且用辅料(pbs-生理盐水)免疫其他组。间隔2周地给药三个剂量。所有的免疫均在右脚掌上进行。最后一次剂量后一个月,左脚掌皮下注射105硕大利什曼原虫稳定的前鞭毛体使小鼠感染。通过测量脚掌肿胀直至攻击后8周而评价真皮病变的发生(图9a)。虽然用s6加cpgodn接种疫苗的小鼠的脚掌肿胀明显低于对照组以及用其他四种蛋白质免疫的小鼠中观察到的脚掌肿胀,所有组的小鼠均发生了炎性病变。另外,分析了小鼠引流淋巴结(dln)和脾脏中的寄生虫载量。分别与生理盐水和cpgodn组相比,用s6加cpgodn免疫的动物脾脏中的寄生虫数量表现出2-log下降(图9b)。然而,s6加cpgodn组的dln中发现的寄生虫载量与对照组中观察到的寄生虫载量相似。
从该分析可以推断,在cpgodn佐药的存在下,用s6重组蛋白质的接种免疫诱导了免疫状态(immunestate),所述免疫状态导致balb/c小鼠中部分保护抗由硕大利什曼原虫感染引起的cl:感染位点中炎性病变的存在和脾脏中的寄生虫载量低于对照组。另一方面,相对于对照组,用其他四种抗原(l5、l7、l8和l16)的接种免疫不会导致cl发展的显著变化。
为了分析由接种免疫诱导的免疫反应,将s6+cpgodn接种免疫的小鼠引起的体液和细胞反应与用佐药和疫苗稀释液免疫的小鼠进行比较。图10显示了疫苗制剂诱导了抗s6蛋白的th1/th2混合反应,由于在接种免疫的小鼠血清中检测到了抗-s6特异性igg2a和igg1抗体(见图10a)。另外,虽然s6体外刺激接种免疫小鼠脾脏细胞后产生了ifnγ,还在培养物上层清液中观察到了可检测的il-4细胞因子的存在(见图10b)。
可以推断接种免疫诱导了主要的抗s6蛋白的th1反应,还观察到了抗该蛋白的th2反应的轻微刺激(s6-特异性il-4和s6-特异性igg1抗体的可检测水平)。
实施例5:基于l3、l5和s4硕大利什曼原虫重组蛋白质的疫苗
为了更详细地分析l3、l5和s4核糖体蛋白质诱导的防护,进行了新的免疫-感染实验。分析中包括12个小鼠组(每组n=4)。在所有情况中,于小鼠右脚掌皮下免疫3次(间隔2周)。然后分析各组。
■疫苗辅料:生理盐水。
■疫苗佐药:cpg-odn。每个剂量:50μg的cpgodn(25μgcpg-odn-1[5’-tcaacgttga-3’](seqidno:5)和25μg的cpg-odn-2[5’-gctagcgttagcgt-3’](seqidno:6)。
■l3(seqidno:1)。每个剂量:10μg的重组蛋白质。
■l3+cpgodn。每个剂量:10μg的重组蛋白质和50μg的cpgodn。
■l5(seqidno:3)。每个剂量:10μg的重组蛋白质。
■l5+cpgodn。每个剂量:10μg的重组蛋白质和50μg的cpgodn。
■s4(seqidno:32)。每个剂量:10μg的重组蛋白质。
■s4+cpgodn。每个剂量:10μg的重组蛋白质和50μg的cpgodn。
■l3+l5。每个剂量:10μg总重组蛋白质;5μg各个蛋白质。
■l3+l5+cpgodn。每个剂量:10μg的总重组蛋白质和50μg的cpgodn。
■l3+l5+s4。每个剂量:10μg总重组蛋白质;3.3μg各个蛋白质。
■l3+l5+s4+cpgodn。每个剂量:10μg的总重组蛋白质和50μg的cpgodn。
简单地说,在cpgodn的存在和不存在下,分析3种抗原。另外,在cpgodn的存在和不存在下,分析l3+l5和l3+l5+s4的结合。最后一次剂量后一个月,左脚掌皮下注射105硕大利什曼原虫稳定的前鞭毛体使小鼠感染。
通过测量脚掌肿胀直至攻击后6周而评价真皮病变的发展。图11a显示了两个对照组(生理盐水和cpgodn)以及用不含佐药的抗原接种免疫的5个组。未观察到对照组和用不含佐药的蛋白质接种免疫的5个组之间炎性病变的差异。图11b显示了两个对照组(生理盐水和cpgodn)以及用与佐药结合的抗原接种免疫的5个组。这种情况中,在佐药的存在下,除了s4+cpgodn组,所有用重组蛋白质接种免疫的小鼠组均表现出脚掌肿胀的下降。在l5+cpgodn、l3+l5+cpgodn和l3+l5+s4+cpgodn组中观察到了更低的炎性病变。
感染后7周,分析了所有小鼠组dln和脾脏中的寄生虫载量。未发现对照组和用不含佐药的核糖体蛋白质免疫的小鼠中包括的小鼠之间统计上的差异(图12a)。另一方面,除了s4加cpgodn组,核糖体蛋白质加cpgodn免疫的小鼠均表现出脾脏中寄生虫载量的显著下降(图12b)。置于腿弯部淋巴结中的寄生虫负载,我们仅发现用2个(l3+l5)或3个(l3+l5+s4)核糖体蛋白质加cpgodn的结合接种免疫的组中的显著下降。l3加cpgodn和l5加cpgodn接种免疫的小鼠还表现出比对照组更低的寄生虫载量。然而,由于发现不同动物间高度的可变性(variability),结果并不是统计上有意义的。为了分析疫苗对局部寄生虫载量的影响,应该用大量的小鼠重复这些分析,因为在之前的分析中,我们发现了l3加cpgodn和l5加cpgodn接种免疫的以及对照组的小鼠间统计上的差异。
关于基于单个抗原或不同抗原的共同给药的疫苗的使用,我们的结果表明,组合的疫苗比由单个抗原组成的疫苗诱导更高程度的防护。
实施例6:用于结合4种已被表征的防护性核糖体抗原(硕大利什曼原虫的l3、l5、s4和s6)的重组分子构建的克隆步骤的设计。
基于前面的结果,我们计划制备基于硕大利什曼原虫l3(seqidno:1)、l5(seqidno:3)、s4(seqidno:32)和s6(seqidno:34)蛋白的新重组产物。
首先,将编码四种蛋白质的dna嵌入物(dnainserts)克隆至真核生物的表达载体(pcdna-3;stratagene)中。该载体使得利什曼原虫蛋白质在哺乳动物细胞中表达,可以用于测试dna疫苗。
其次,我们通过不同的四种抗原的结合设计了克隆不同嵌合蛋白质的步骤(图13)。首先将基因嵌合体克隆至pbluescript(分析质粒)中,然后将dna嵌入物克隆至两种不同的表达质粒中:
■pqe30;原核生物的表达质粒。
■pcdna3,真核生物的表达质粒。
该克隆策略使得我们通过提出的抗原结合同时拥有在大肠杆菌中表达的dna疫苗和重组蛋白质。
使用以下引物:
lml3正义链5’-cgggatccatgtctcactgcaagttcgag-3’(seqidno:57)
反义链5’-gcgatatctcccttcttcgcggcctttgcc-3’(seqidno:58)
lms4正义链5’-gcgatatcgggatggccaagaagcacctcaag-3’(seqidno:59)
反义链5’-cggaattctcccttgcgggccctgcggg-3’(seqidno:60)
lms6正义链5’-cggaattcgggatgaagctcaacatcgcgtac-3’(seqidno:61)
反义链5’-gcgatatctcccttcttctggaatgctgccac-3’(seqidno:62)
lml5正义链5’-gcgatatcgggatgtgcacgctggcaaattg3’(seqidno:63)
反义链5’-ggggtaccggatccttacttgccgaggcgctcgc-3’(seqidno:64)
产生的嵌合蛋白如由seqidno:67组成的氨基酸序列所示。该嵌合蛋白由seqidno:66组成的核酸分子所示的核酸编码。
参考文献
1.aguilar-be,i.,r.dasilvazardo,e.paraguaidesouza,g.p.borja-cabrera,m.rosado-vallado,m.mut-martin,r.garcia-missmdel,c.b.palatnikdesousa,ande.dumonteil.2005.cross-protectiveefficacyofaprophylacticleishmaniadonovanidnavaccineagainstvisceralandcutaneousmurineleishmaniasis.infectimmun73:812-9.
2.anderson,c.f.,m.oukka,v.j.kuchroo,andd.sacks.2007.cd4(+)cd25(-)foxp3(-)th1cellsarethesourceofil-10-mediatedimmunesuppressioninchroniccutaneousleishmaniasis.jexpmed204:285-97.
3.badaro,r.,d.benson,m.c.eulalio,m.freire,s.cunha,e.m.netto,d.pedral-sampaio,c.madureira,j.m.burns,r.l.houghton,j.r.david,ands.g.reed.1996.rk39:aclonedantigenofleishmaniachagasithatpredictsactivevisceralleishmaniasis.jinfectdis173:758-61.
4.belkaid,y.,k.f.hoffmann,s.mendez,s.kamhawi,m.c.udey,t.a.wynn,andd.l.sacks.2001.theroleofinterleukin(il)-10inthepersistenceofleishmaniamajorintheskinafterhealingandthetherapeuticpotentialofantiil-10receptorantibodyforsterilecure.jexpmed194:1497-506.
5.belkaid,y.,s.kamhawi,g.modi,j.valenzuela,n.noben-trauth,e.rowton,j.ribeiro,andd.l.sacks.1998.developmentofanaturalmodelofcutaneousleishmaniasis:powerfuleffectsofvectorsalivaandsalivapreexposureonthelong-termoutcomeofleishmaniamajorinfectioninthemouseeardermis.jexpmed188:1941-53.
6.buffet,p.a.,a.sulahian,y.j.garin,n.nassar,andf.derouin.1995.culturemicrotitration:asensitivemethodforquantifyingleishmaniainfantumintissuesofinfectedmice.antimicrobagentschemother39:2167-8.
7.campos-neto,a.2005.whataboutth1/th2incutaneousleishmaniasisvaccinediscovery?brazjmedbiolres38:979-84.
8.coffman,r.l.1993.mechanismsofhelpert-cellregulationofb-cellactivity.annnyacadsci681:25-8.
9.coler,r.n.,ands.g.reed.2005.second-generationvaccinesagainstleishmaniasis.trendsparasitol21:244-9.
10.cordeiro-da-silva,a.,m.c.borges,e.guilvard,anda.ouaissi.2001.dualroleoftheleishmaniamajorribosomalproteins3ahomologueinregulationoft-andb-cellactivation.infectimmun69:6588-96.
11.chenik,m.,h.louzir,h.ksontini,a.dilou,i.abdmouleh,andk.dellagi.2006.vaccinationwiththedivergentportionoftheproteinhistoneh2bofleishmaniaprotectssusceptiblebalb/cmiceagainstavirulentchallengewithleishmaniamajor.vaccine24:2521-9.
12.chiaramonte,m.g.,m.hesse,a.w.cheever,andt.a.wynn.2000.cpgoligonucleotidescanprophylacticallyimmunizeagainstth2-mediatedschistosomeegg-inducedpathologybyanil-12-independentmechanism.jimmunol164:973-85.
13.garcia-alonso,m.,a.blanco,d.reina,f.j.serrano,c.alonso,andc.g.nieto.1996.immunopathologyoftheuveitisincanineleishmaniasis.parasiteimmunol18:617-23.
14.garcia-alonso,m.,c.g.nieto,a.blanco,j.m.requena,c.alonso,andi.navarrete.1996.presenceofantibodiesintheaqueoushumourandcerebrospinalfluidduringleishmaniainfectionsindogs.pathologicalfeaturesatthecentralnervoussystem.parasiteimmunol18:539-46.
15.gradoni,l.2001.anupdateonantileishmanialvaccinecandidatesandprospectsforacanineleishmaniavaccine.vetparasitol100:87-103.
16.gramiccia,m.,andl.gradoni.2005.thecurrentstatusofzoonoticleishmaniasesandapproachestodiseasecontrol.intjparasitol35:1169-80.
17.handman,e.,a.h.noormohammadi,j.m.curtis,t.baldwin,anda.sjolander.2000.therapyofmurinecutaneousleishmaniasisbydnavaccination.vaccine18:3011-7.
18.herwaldt,b.l.1999.leishmaniasis.lancet354:1191-9.
19.iborra,s.,m.soto,j.carrion,c.alonso,andj.m.requena.2004.vaccinationwithaplasmiddnacocktailencodingthenucleosomalhistonesof
leishmaniaconfersprotectionagainstmurinecutaneousleishmaniosis.vaccine22:3865-76.
20.jaafari,m.r.,a.ghafarian,a.farrokh-gisour,a.samiei,m.t.kheiri,f.mahboudi,f.barkhordari,a.khamesipour,andw.r.mcmaster.2006.immuneresponseandprotectionassayofrecombinantmajorsurfaceglycoproteinofleishmania(rgp63)reconstitutedwithliposomesinbalb/cmice.vaccine24:5708-17.
21.lopez,r.,r.lucena,m.novales,p.j.ginel,e.martin,andj.m.molleda.1996.circulatingimmunecomplexesandrenalfunctionincanineleishmaniasis.zentralblveterinarmedb43:469-74.
22.mancianti,f.,a.poli,anda.bionda.1989.analysisofrenalimmune-depositsincanineleishmaniasis.preliminaryresults.parassitologia31:213-30.
23.martins,d.r.,s.m.jeronimo,j.e.donelson,andm.e.wilson.2006.
leishmaniachagasit-cellantigensidentifiedthroughadoublelibraryscreen.infectimmun74:6940-8.
24.mcmahon-pratt,d.,andj.alexander.2004.doestheleishmaniamajorparadigmofpathogenesisandprotectionholdfornewworldcutaneousleishmaniasesorthevisceraldisease?immunolrev201:206-24.
25.mendez,s.,y.belkaid,r.a.seder,andd.sacks.2002.optimizationofdnavaccinationagainstcutaneousleishmaniasis.vaccine20:3702-8.
26.mendez,s.,s.gurunathan,s.kamhawi,y.belkaid,m.a.moga,y.a.skeiky,a.campos-neto,s.reed,r.a.seder,andd.sacks.2001.thepotencyanddurabilityofdna-andprotein-basedvaccinesagainstleishmaniamajorevaluatedusinglow-dose,intradermalchallenge.jimmunol166:5122-8.
27.miles,s.a.,s.m.conrad,r.g.alves,s.m.jeronimo,andd.m.mosser.2005.aroleforiggimmunecomplexesduringinfectionwiththeintracellularpathogenleishmania.jexpmed201:747-54.
28.moore,k.w.,r.dewaalmalefyt,r.l.coffman,anda.o'garra.2001.interleukin-10andtheinterleukin-10receptor.annurevimmunol19:683-765.
29.mougneau,e.,f.altare,a.e.wakil,s.zheng,t.coppola,z.e.wang,r.waldmann,r.m.locksley,andn.glaichenhaus.1995.expressioncloningofaprotectiveleishmaniaantigen.science268:563-6.
30.nieto,c.g.,r.barrera,m.a.habela,i.navarrete,c.molina,a.jimenez,andj.l.serrera.1992.changesintheplasmaconcentrationsoflipidsandlipoproteinfractionsindogsinfectedwithleishmaniainfantum.vetparasitol44:175-82.
31.nieto,c.g.,i.navarrete,m.a.habela,f.serrano,ande.redondo.1992.pathologicalchangesinkidneysofdogswithnaturalleishmaniainfection.vetparasitol45:33-47.
32.noben-trauth,n.,r.lira,h.nagase,w.e.paul,andd.l.sacks.2003.therelativecontributionofil-4receptorsignalingandil-10tosusceptibilitytoleishmaniamajor.jimmunol170:5152-8.
33.pateraki,e.,r.portocala,h.labrousse,andj.l.guesdon.1983.antiactinandantitubulinantibodiesincaninevisceralleishmaniasis.infectimmun42:496-500.
34.peters,n.,andd.sacks.2006.immuneprivilegeinsitesofchronicinfection:leishmaniaandregulatorytcells.immunolrev213:159-79.
35.pollock,k.g.,k.s.mcneil,j.c.mottram,r.e.lyons,j.m.brewer,p.scott,g.h.coombs,andj.alexander.2003.theleishmaniamexicanacysteineprotease,cpb2.8,inducespotentth2responses.jimmunol170:1746-53.
36.probst,p.,e.stromberg,h.w.ghalib,m.mozel,r.badaro,s.g.reed,andj.r.webb.2001.identificationandcharacterizationoftcell-stimulatingantigensfromleishmaniabycd4tcellexpressioncloning.jimmunol166:498-505.
37.rafati,s.,a.nakhaee,t.taheri,a.ghashghaii,a.h.salmanian,m.jimenez,m.mohebali,s.masina,andn.fasel.2003.expressionofcysteineproteinasetypeiandiiofleishmaniainfantumandtheirrecognitionbyseraduringcanineandhumanvisceralleishmaniasis.expparasitol103:143-51.
38.rafati,s.,a.h.salmanian,t.taheri,m.vafa,andn.fasel.2001.aprotectivecocktailvaccineagainstmurinecutaneousleishmaniasiswithdnaencodingcysteineproteinasesofleishmaniamajor.vaccine19:3369-75.
39.requena,j.m.,c.alonso,andm.soto.2000.evolutionarilyconservedproteinsasprominentimmunogensduringleishmaniainfections.parasitoltoday16:246-50.
40.rhee,e.g.,s.mendez,j.a.shah,c.y.wu,j.r.kirman,t.n.turon,d.f.davey,h.davis,d.m.klinman,r.n.coler,d.l.sacks,andr.a.seder.2002.vaccinationwithheat-killedleishmaniaantigenorrecombinantleishmanialproteinandcpgoligodeoxynucleotidesinduceslong-termmemorycd4+andcd8+tcellresponsesandprotectionagainstleishmaniamajorinfection.jexpmed195:1565-73.
41.roberts,m.t.,c.b.stober,a.n.mckenzie,andj.m.blackwell.2005.interleukin-4(il-4)andil-10colludeinvaccinefailurefornovelexacerbatoryantigensinmurineleishmaniamajorinfection.infectimmun73:7620-8.
42.rodriguez-gabriel,m.a.,m.remacha,andj.p.ballesta.2000.thernainteractingdomainbutnottheproteininteractingdomainishighlyconservedinribosomalproteinp0.jbiolchem275:2130-6.
43.rosa,r.,c.marques,o.r.rodrigues,andg.m.santos-gomes.2007.immunizationwithleishmaniainfantumreleasedproteinsconferspartialprotectionagainstparasiteinfectionwithapredominantth1specificimmuneresponse.vaccine25:4525-32.
44.santos-gomes,g.m.-,r.rosa,c.leandro,s.cortes,p.romao,andh.silveira.2002.cytokineexpressionduringtheoutcomeofcanineexperimentalinfectionbyleishmaniainfantum.vetimmunolimmunopathol88:21-30.
45.santos,w.r.,v.m.delima,e.p.desouza,r.r.bernardo,m.palatnik,andc.b.palatnikdesousa.2002.saponins,il12andbcgadjuvantinthefml-vaccineformulationagainstmurinevisceralleishmaniasis.vaccine21:30-43.
46.saraiva,e.m.,a.defigueiredobarbosa,f.n.santos,g.p.borja-cabrera,d.nico,l.o.souza,c.deoliveiramendes-aguiar,e.p.desouza,p.fampa,l.e.parra,i.menz,j.g.dias,jr.,s.m.deoliveira,andc.b.palatnik-de-sousa.2006.thefml-vaccine(leishmune)againstcaninevisceralleishmaniasis:atransmissionblockingvaccine.vaccine24:2423-31.
47.serezani,c.h.,a.r.franco,m.wajc,j.k.umadayokoyama-yasunaka,g.wunderlich,m.m.borges,ands.r.uliana.2002.evaluationofthemurineimmuneresponsetoleishmaniameta1antigendeliveredasrecombinantproteinordnavaccine.vaccine20:3755-63.
48.skeiky,y.a.,j.a.guderian,d.r.benson,o.bacelar,e.m.carvalho,m.kubin,r.badaro,g.trinchieri,ands.g.reed.1995.arecombinantleishmaniaantigenthatstimulateshumanperipheralbloodmononuclearcellstoexpressath1-typecytokineprofileandtoproduceinterleukin12.jexpmed181:1527-37.
49.stager,s.,d.f.smith,andp.m.kaye.2000.immunizationwitharecombinantstage-regulatedsurfaceproteinfromleishmaniadonovaniinducesprotectionagainstvisceralleishmaniasis.jimmunol165:7064-71.
50.tonui,w.k.,j.s.mejia,l.hochberg,m.l.mbow,j.r.ryan,a.s.chan,s.k.martin,andr.g.titus.2004.immunizationwithleishmaniamajorexogenousantigensprotectssusceptiblebalb/cmiceagainstchallengeinfectionwithl.major.infectimmun72:5654-61.
51.webb,j.r.,a.campos-neto,y.a.skeiky,ands.g.reed.1997.molecularcharacterizationoftheheat-induciblelmsti1proteinofleishmaniamajor.molbiochemparasitol89:179-93.
52.webb,j.r.,d.kaufmann,a.campos-neto,ands.g.reed.1996.molecularcloningofanovelproteinantigenofleishmaniamajorthatelicitsapotentimmuneresponseinexperimentalmurineleishmaniasis.jimmunol157:5034-41.
53.zimmermann,s.,o.egeter,s.hausmann,g.b.lipford,m.rocken,h.wagner,andk.heeg.1998.cpgoligodeoxynucleotidestriggerprotectiveandcurativeth1responsesinlethalmurineleishmaniasis.jimmunol160:3627-30.
54.s.iborra,m.soto,j.carrion,a.nieto,e.fernandez,c.alonso,j.m.requena,theleishmaniainfantumacidicribosomalproteinp0administeredasadnavaccineconfersprotectiveimmunitytoleishmaniamajorinfectioninbalb/cmice,infectimmun71(2003)6562-6572.
55.s.iborra,n.parody,d.r.abanades,p.bonay,d.prates,f.o.novais,m.barral-netto,c.alonso,m.soto,vaccinationwiththeleishmaniamajorribosomalproteinspluscpgoligodeoxynucleotidesinducesprotectionagainstexperimentalcutaneousleishmaniasisinmice,microbesinfect10(2008)1133-1141.
56.w.h.mager,r.j.planta,j.g.ballesta,j.c.lee,k.mizuta,k.suzuki,j.r.warner,j.woolford,anewnomenclatureforthecytoplasmicribosomalproteinsofsaccharomycescerevisiae,nucleicacidsresearch25(1997)4872-4875.
57.p.y.shi,n.maizels,a.m.weiner,recoveryofsoluble,activerecombinantproteinfrominclusionbodies,biotechniques23(1997)1036-1038.
58.n.santarem,r.silvestre,j.tavares,m.silva,s.cabral,j.maciel,a.cordeiro-da-silva,immuneresponseregulationbyleishmaniasecretedandnonsecretedantigens,jbiomedbiotechnol2007(2007)85154.
59.boarino,a.,a.scalone,l.gradoni,e.ferroglio,f.vitale,r.zanatta,m.g.giuffrida,ands.rosati.2005.developmentofrecombinantchimericantigenexpressingimmunodominantbepitopesofleishmaniainfantumforserodiagnosisofvisceralleishmaniasis.clindiagnlabimmunol12:647-53.
60.porrozzi,r.,m.v.santosdacosta,a.teva,a.falqueto,a.l.ferreira,c.d.dossantos,a.p.fernandes,r.t.gazzinelli,a.campos-neto,andg.grimaldi,jr.2007.comparativeevaluationofenzyme-linkedimmunosorbentassaysbasedoncrudeandrecombinantleishmanialantigensforserodiagnosisofsymptomaticandasymptomaticleishmaniainfantumvisceralinfectionsindogs.clinvaccineimmunol14:544-8.
61.soto,m.,j.m.requena,l.quijada,andc.alonso.1998.multicomponentchimericantigenforserodiagnosisofcaninevisceralleishmaniasis.jclinmicrobiol36:58-63.
62.melbyp.c.g.b.,ogdenh.a.,floresw.,zhaoc.,geldmacher,n.m.,biedigers.k.,ahuja,j.,urangaandm.melendez(2000),identificationofvaccinecandidatesforexperimentalvisceralleishmaniasisbyimmunizationwithsequentialfractionsofacdnalibrary.infect.immun.,68:5595-5602.
63.stoberc.b.u.g.,langem.t.,robertsb,gilmartinr.,francisr.,almeidac.s.,peacoks.,mccannandj.m.blackwell,(2006),fromgenometovaccinesforleishmaniasis:screening100novelvaccinecandidatesagainstmurineleishmaniasismajorinfection.vaccine.,24:2602-2616.
64.aebischert.,etal,(2000)infectionandimmunity.,68:1328-1336.
65.pootjetal,(2009),vaccine,27:4439-4446.
66.ferreiraj.h.etal,(2008),vaccine,26:67-685.
67.buckanovichr.j.,etal,(1994),proc.natl.acad.sci.usa,91:4892.
68.liun.,etal(2003),natureimmunology,687-693).
69.bertholets,gotoy,carterl,bhatiaa,howardrf,carterd,colerrn,vedvickts,reedsg.vaccine.2009nov23;27(50):7036-45.
70.s.iborra,j.carrion,c.anderson,c.alonso,d.sacks,m.soto,vaccinationwiththeleishmaniainfantumacidicribosomalp0proteinpluscpgoligodeoxynucleotidesinducesprotectionagainstcutaneousleishmaniasisinc57bl/6micebutdoesnotpreventprogressivediseaseinbalb/cmice,infectimmun73(2005)5842-5852
71.m.soto,j.m.requena,l.quijada,m.j.perez,c.g.nieto,f.guzman,m.e.patarroyo,c.alonso,antigenicityoftheleishmaniainfantumhistonesh2bandh4duringcanineviscerocutaneousleishmaniasis,clinexpimmunol115(1999)342-349.
72.t.r.demoura,f.o.novais,f.oliveira,j.clarencio,a.noronha,a.barral,c.brodskyn,c.i.deoliveira,towardanovelexperimentalmodelofinfectiontostudyamericancutaneousleishmaniasiscausedbyleishmaniabraziliensis,infectimmun73(2005)5827-5834.
73.s.iborra,j.carrion,c.anderson,c.alonso,d.sacks,m.soto,vaccinationwiththeleishmaniainfantumacidicribosomalp0proteinpluscpgoligodeoxynucleotidesinducesprotectionagainstcutaneousleishmaniasisinc57bl/6micebutdoesnotpreventprogressivediseaseinbalb/cmice,infectimmun73(2005)5842-5852.
sequencelisting
<110>热体实验室有限公司
<120>l3和/或l5源作为寄生虫病疫苗或诊断的用途
<130>i46363neo
<150>ep09175929.0
<151>2009-11-13
<150>us61/261,020
<151>2009-11-13
<160>73
<170>patentinversion3.3
<210>1
<211>419
<212>prt
<213>leishmaniamajor
<400>1
metserhiscyslysphegluhisproarghisglyhisleuglyphe
151015
leuproarglysargserargglnileargglyargalaargalaphe
202530
prolysaspaspalathrglnlysprohisleuthrserphemetval
354045
phelysalaglymetthrhisilevalargaspvalaspargprogly
505560
serlysvalasnlyslysgluvalvalgluprovalthrileleuglu
65707580
alaproprometvalilevalglyilevalglytyrargglnthrpro
859095
valglyleulysthrileglythrvaltrpalahishisthrserval
100105110
glupheargargargtyrtyrlysasntrplysglnseralaglnleu
115120125
alapheserargglnlysglnphealaasnthrlysgluglylysval
130135140
alaglualaargthrleuasnalaphealalyslysalaservalile
145150155160
argvalilealahisthrglnleuarglysleuargasnhisargval
165170175
glyvallyslysalahisvalglngluileglnvalasnglyglyser
180185190
valalaalalysilealaleualalysserleuleuglulysgluval
195200205
argvalaspservalpheglnglnserglualacysaspvalcysser
210215220
valthrlysglyhisglythrgluglyvalvallysargtrpglyval
225230235240
alacysleuproarglysthrhisargglyleuarglysvalalacys
245250255
ileglyalatrphisproalaargvalmettyrthrvalalaargala
260265270
glyglnhisglytyrhishisargthrglnleuasnlyslysiletyr
275280285
glnileglyargservalalavalgluproasnglnalathrthrthr
290295300
tyraspleuthralalysthrilethrprometglyglyphevalgly
305310315320
tyrglythrvalargasnasptyrvalmetleulysglyservalser
325330335
glyproargargargvalmetthrleuargargprometalaprogln
340345350
thrserargglnleulysglulysilevalleulyspheileaspthr
355360365
serserlysileglyhisglyargpheglnthrlyslysglulysasn
370375380
glntrppheglyproleulyslysaspargileargargglugluarg
385390395400
leuarglysgluargalaalaargalavalgluarglysalalysala
405410415
alalyslys
<210>2
<211>1260
<212>dna
<213>leishmaniamajor
<400>2
atgtctcactgcaagttcgagcacccccgccacggccatctcggcttcctgccgcgcaag60
cgctcgcgccagatccgcggccgtgcgcgcgcgttccccaaggacgacgcgacgcagaag120
ccccacctgacgagcttcatggtgttcaaggccggtatgacgcacattgtgcgtgatgtc180
gatcgccctggatcgaaggtgaacaagaaggaagtggtggagccggtgacgatcctggag240
gcgccgccgatggtgattgtcggcattgtgggctaccgccaaacgccggttggcctgaag300
acgatcggcaccgtgtgggcgcaccacacgagcgtcgagttccgccgccgctactacaag360
aactggaagcagtctgcgcaactggccttctcccgccagaagcagtttgcgaacacgaag420
gagggcaaggtcgccgaggcgcgcacgctgaacgcgttcgcgaagaaggcgtccgtcatc480
cgcgtgatcgcgcacacgcagctgcgcaagcttcgcaaccaccgcgtgggcgtgaagaag540
gcgcacgtgcaggagatccaggtcaacggcggcagcgttgcggcgaagatcgcgctggcc600
aagtccctgctggagaaggaggtgcgcgtcgactccgtgttccagcagtccgaggcgtgc660
gacgtgtgctccgtcacgaaaggccacggtacggagggcgtggtgaagcgctggggcgtt720
gcctgcctgccacgcaagacgcaccgcggtctgcgcaaggttgcgtgcatcggcgcgtgg780
caccctgcccgcgtcatgtacactgtcgcgcgcgccggtcagcacggttaccaccaccgc840
acgcagctgaacaagaagatctaccagatcggccgctccgttgctgtggagccgaaccag900
gcgacgacgacctacgatctgacagccaagacgatcacgcccatgggtggcttcgtcggc960
tacggtacggtgcgcaacgactacgtgatgctgaagggctccgtgtctggcccgcgccgc1020
cgtgtgatgacgctgcgccgcccgatggcgccgcagacgtcgcgccagctgaaggagaag1080
atcgtgctgaagttcatcgacacgagctcgaagatcggccacggccgcttccagacgaag1140
aaggagaagaaccagtggttcggcccgctcaagaaggaccgcatccgccgcgaggagcgc1200
ctgcgcaaggagcgcgctgcccgcgccgtggagcgcaaggcaaaggccgcgaagaagtaa1260
<210>3
<211>328
<212>prt
<213>leishmaniamajor
<400>3
metcysthrleualaasntrpvalargalaileilelyslyshisser
151015
thrleualahisthrleuglumetprophevallysvalvallysasn
202530
lysalatyrphelysargpheglnvallystyrargargargargglu
354045
glylysthrasptyrhisalaargargglnmetvalleuglnasplys
505560
thrlyspheglyserprolystyrargleuvalvalargilethrasn
65707580
lysaspileilealaglnilevalglnalalysilevalglyaspglu
859095
valvalmetalaalatyralahisgluleuproalapheglyileglu
100105110
hisglyleuthrasntyralaalaalatyralathrglyleuleuleu
115120125
alaargargthrleualalysleuglyilealaasplyspheglngly
130135140
alalysglualaaspglysertyrseralavalargthrlyslysasp
145150155160
aspgluglyaspaspglugluargpheprophelysalaileleuasp
165170175
valglyleualaargthrthrthrglyalaargvalpheglyvalleu
180185190
lysglyalavalaspglyglymetalavalprohisargproasnarg
195200205
pheproglytyrasnlysglulysserserleuaspalalysvalhis
210215220
argaspargilepheglylyshisvalalaasptyrleulysglnval
225230235240
lysgluglualaserserasnproaspglulyscysvalglnpheser
245250255
lystyrmetalaalalysvalleuprogluserilegluglymettyr
260265270
lyslysalahisalaalaileargalaaspproserlysserleupro
275280285
lyslysalalyslysgluglyvalalahislyssertyrlysthrlys
290295300
lysleuserglyalaglulysargalaalaalalysalalysvalala
305310315320
alailearggluargleuglylys
325
<210>4
<211>987
<212>dna
<213>leishmaniamajor
<400>4
atgtgcacgctggcaaattgggtacgcgctatcatcaagaaacactcaacactcgcccac60
acactcgagatgccgttcgtcaaggtcgtgaagaacaaggcgtacttcaagcgcttccag120
gtgaagtaccgccgtcgccgcgagggcaagacggactaccacgcgcgccggcagatggtg180
ctgcaggacaagacgaagttcggctcgcccaagtaccgccttgttgtgcgcatcacgaac240
aaggacatcattgcgcagatcgtgcaggcgaagatcgtcggcgacgaggtggtgatggcc300
gcgtacgcgcacgagctgcctgcgttcggcattgagcacggcctgacaaactacgctgct360
gcgtacgcgactggtctgctgctggcgcgccgcacgctggcgaagctgggcatcgcggac420
aagttccagggcgcgaaggaggcggacggctcgtactctgctgtgcgcacgaagaaggac480
gacgagggcgacgacgaggagcgctttccgttcaaggcgatcctggacgtcggccttgcg540
cgcacgacgaccggcgcccgcgtgttcggcgtgctgaagggcgcggtggacggcggtatg600
gctgtgccgcaccgccccaaccgcttccccggctacaacaaggagaagagctcgctggac660
gcgaaggtgcaccgcgaccgcatctttggcaagcacgtggcggactacctgaagcaggtg720
aaggaggaggcgagctcgaaccctgacgagaagtgcgtgcagttctcgaagtacatggcc780
gcgaaggttttgccggagagcatcgagggcatgtacaagaaggcgcacgcggcgatccgc840
gcggacccgtcgaagtcgctgccgaagaaggcgaagaaggagggcgtcgcgcacaagagc900
tacaagacgaagaagctgagcggcgcggagaagagggccgccgcgaaggcgaaggtcgcg960
gccatccgcgagcgcctcggcaagtaa987
<210>5
<211>10
<212>dna
<213>artificial
<220>
<223>adjuvant
<400>5
tcaacgttga10
<210>6
<211>14
<212>dna
<213>artificial
<220>
<223>adjuvant
<400>6
gctagcgttagcgt14
<210>7
<211>132
<212>prt
<213>leishmaniainfantum
<400>7
metalathrproargseralalyslysalavalarglysserglyser
151015
lysseralalyscysglyleuilepheprovalglyargvalglygly
202530
metmetargargglyglntyralaargargileglyalaserglyala
354045
valtyrleualaalavalleuglutyrleuthralagluleuleuglu
505560
leuservallysalaalaalaglnserglylyslysargcysargleu
65707580
asnproargthrvalmetleualaalaarghisaspaspaspilegly
859095
thrleuleulysasnvalthrleuserhisserglyvalvalproasn
100105110
ileserlysalametalalyslyslysglyglylyslysglylysala
115120125
thrproserala
130
<210>8
<211>691
<212>dna
<213>leishmaniainfantum
<400>8
gcctcatccgtcatccgtcatctttgtgctacagctttactctcactcccctccaaccta60
cccatcgcagccatggctactcctcgcagcgccaagaaggccgtccgcaagagcggctcc120
aagtccgcgaaatgtggtctgatcttcccggtgggccgcgtcggcgggatgatgcgccgc180
ggccagtacgctcgccgcatcggtgcctctggcgccgtgtacctggccgccgtgctggag240
tacctgacggcggagctgctggagctgtccgtgaaggcggccgcgcagagcgggaagaag300
cggtgccgcctgaacccgcgcaccgtgatgctggccgcgcgccacgacgacgacatcggc360
acgcttctgaagaacgtgaccttgtctcacagcggcgttgtgccgaacatcagcaaggcg420
atggcaaagaagaagggcggcaagaagggcaaggcgacaccgagcgcgtaagtcctccgg480
cctgacagcgcacacgcgccgctgtattgtgcgcgtgcgcgcgggtcccgactggggccg540
gcgatgaggcgcatcatacctccatagagaccctatcttttgttttatggcttctcagat600
gaccacttggttcttcctgcctttgtttggtttgtttctctcctcccctccgccgagggt660
acgagtcagggtaggctcggacaaaaaaaaa691
<210>9
<211>111
<212>prt
<213>leishmaniainfantum
<400>9
metalaserserargseralaproarglysalaserhisalahislys
151015
serhisarglysprolysargsertrpasnvaltyrvalglyargser
202530
leulysalaileasnalaglnmetsermetserhisargthrmetser
354045
ilevalasnsertyrvalasnaspvalmetgluargilecysmetglu
505560
alaalaserilevalargalaasnlyslysargthrleuglyalaarg
65707580
gluvalglnthralavalargilevalleuproalagluleualalys
859095
hisalametalagluglythrlysalavalserseralaserala
100105110
<210>10
<211>894
<212>dna
<213>leishmaniainfantum
<400>10
ccaagccagccaaatccttcgcactttcacgctgtccctcctttccaaccaacccacatc60
accatggcctcttctcgctctgctccccgcaaggcttcccacgcgcacaagtcgcaccgc120
aagccgaagcgctcgtggaacgtgtacgtgggccgctcgctgaaggcgatcaacgcccag180
atgtcgatgtcgcaccgcacgatgagcatcgtgaactcgtacgtgaacgacgtgatggag240
cgcatctgcatggaggccgcgtcgatcgttcgcgcgaacaagaagcgcacgttgggtgcg300
cgcgaggtgcagacggcggtgcgcattgtgctgccggcggagctcgcgaagcacgccatg360
gctgagggcacgaaggccgtgtcgagcgcgtcggcttgagcggctcagttagagggtttg420
tccacgcctcggccgtgtgtccggggtgtggggtaccctcaactcccctctccccgccta480
cgccgtgggttttcatagagatttattgtttctttttcgattctctttccttgaaggtga540
tgtctcgtcctttgctggagtgcgtgccgggttcgcgggcggtagaaagcagcggcggag600
gaggcagcggcggcgcgagacggtgaaggggaggagaggcgggccgaaagcacagatgcg660
cttctccgtctctttctcccttctctgcattcgccctcgctgctcctctctgatgccctc720
gtacctcgtggtgcgcgcgtctcccgctcgccgtccgcgccacgctgcacagaggcgtgc780
acggtttgtcttctatctcagaacgagtgacacacacgttttcttgttcccccctccccc840
cttcgtcatcgcttcttcgttttcgttgtcgtctcgacgcccaaaaaaaaaaaa894
<210>11
<211>129
<212>prt
<213>leishmaniainfantum
<400>11
metserargthrlysgluthralaargalalysargthrilethrser
151015
lyslysserlyslysalaproserglyalaserglyvallysargser
202530
hisargargtrpargproglythrcysalailearggluilearglys
354045
pheglnlysserthrserleuleuileglncysalapropheglnarg
505560
leuvalargglyvalgluargglnlysgluglyleuargpheglnser
65707580
seralailemetalaleuglnglualathrglualatyrilevalser
859095
leumetalaaspthrasnleualacysilehisalalysargvalthr
100105110
ileglnprolysaspileglnleualaleuargleuargglygluarg
115120125
his
<210>12
<211>587
<212>dna
<213>leishmaniainfantum
<400>12
gtttcactaccgccatccaaccccctgccactcccacccccaccgcaccaccatgtcccg60
caccaaggagaccgcccgcgcgaagcgcaccatcacgtcgaagaagagcaagaaggcgcc120
gagcggggcgtccggcgtgaagaggtcgcatcgccgctggcgcccgggcacctgcgcgat180
ccgcgagatccgcaagttccagaagagtacgagcctgctgatccagtgcgcgccgttcca240
gcgcctggtgcgaggtgtcgagcggcagaaggagggcctgcgcttccagagcagcgctat300
catggcgctgcaggaggcgacggaggcgtacattgtgtcgctgatggcggacacgaacct360
cgcctgcatccacgcgaagcgcgtgacgatccagccgaaggacatccagctggcgctgcg420
cctgcgcggtgagcgccactagggcgggcccgctctcccccccctcatagataccatgtt480
tttgtttcctttcttttcgccttccctaagtcgtgcacgctgccctgccgcggcagccga540
gagagtgagagggtcattgaacctctagagcccgccaaaaaaaaaaa587
<210>13
<211>100
<212>prt
<213>leishmaniainfantum
<400>13
metalalysglylysargserthraspalalysglyserglnargarg
151015
glnlyslysvalleuargaspasnileargglyilethrargglycys
202530
valargargmetalaargargglyglyvallysargileserthrglu
354045
valtyrglugluvalargargvalleulysalatyrvalgluaspile
505560
valargcysserthralatyrthrglutyralaarglyslysthrval
65707580
thralacysaspvalvalthralaleuarglysglnglyhisileleu
859095
tyrglytyrala
100
<210>14
<211>531
<212>dna
<213>leishmaniainfantum
<400>14
gctccctttcttgcctcctctcccccccacgcctcctcccttcacatatccaccatggcc60
aagggcaagcgttccactgatgccaagggcagccagaggcgccagaagaaggtgctgcgc120
gacaacatccgcggcatcactcgcggctgcgtccgccgcatggcgcgccgcggtggcgtg180
aagcgcatctcgaccgaggtgtacgaagaggtgcgccgtgtgctgaaggcctacgtggag240
gacattgtgcgctgcagcacggcctacaccgagtacgcgcgcaagaagaccgtgacggcg300
tgcgatgttgtgaccgcgctgcgcaagcaaggccacatcctgtacggctacgcgtaaatg360
ctcgcagagccgctgcacactcatagatacaccttctttgttcatgccgtcgtttcgttg420
gctttcttggttttcgacttcccttccccccactatggcttttctttcgtctcgtgctgg480
cacccttccctactcatcgctgtttgctgaaggcagtacagaacgaagcgg531
<210>15
<211>106
<212>prt
<213>leishmaniainfantum
<400>15
metglntyrleualaalatyralaleuvalalaleuserglylysthr
151015
proserlysalaaspvalglnalavalleulysalaalaglyvalala
202530
valaspalaserargvalaspalavalpheglngluvalgluglylys
354045
serpheaspalaleuvalalagluglyargthrlysleuvalglyser
505560
glyseralaalaproalaglyalavalserthralaglyalaglyala
65707580
glyalavalalaglualalyslysglugluproglugluglugluala
859095
aspaspaspmetglypheglyleupheasp
100105
<210>16
<211>106
<212>prt
<213>leishmaniainfantum
<400>16
metglntyrleualaalatyralaleuvalalaleuserglylysthr
151015
proserlysalaaspvalglnalavalleulysalaalaglyvalala
202530
valaspalaserargvalaspalavalpheglngluvalgluglylys
354045
serpheaspalaleuvalalagluglyargthrlysleuvalglyser
505560
glyseralaalaproalaglyalavalserthralaglyalaglyala
65707580
glyalavalalaglualalyslysglugluproglugluglugluala
859095
aspaspaspmetglypheglyleupheasp
100105
<210>17
<211>2850
<212>dna
<213>leishmaniainfantum
<400>17
gtcgacggcgacatggctagagcagtttgtgcagctcccgctgagcgacaagaaacacga60
tctgccgcgtcgccacggaggtgtttctccatgcctttgcccagtgcatcaatgagtccg120
ggatctttgtcgcaccaccagcaaaagaggagcagtgcctactcgcggccttaggaacgg180
caatgcgaccttttgcacagtaccccgaggagacgatcgcccaggcaaatgcgtttctgc240
atcaaggagggcttccgcatgtcccgttcgcagctgaggcggtggagcagcaggttatga300
atctccagcggctgcattaatcgccgcctgccagacaccggggaggtcctctgtttctgt360
ttttgcgtgttgcgtctttctctttatgtttgctcctttgtgtctgtcggttaagagctc420
ctcccttgcccagaaaacaggagtaaccgagtacgccgcagcgcctgcgccacacgttgt480
ccatggaacccctcccctcctcgctcctcccttctccactccctccttctgggtgctgca540
tgtgtgtgtgcatgtgtgtgtaactttgcctcggtgtggctggcacgctgcgccccctcc600
ccccccccccccaaaaaaaaaaaacagcatcatcagtgggctgacctggatacatctcct660
cctctccttgtgttccccatcccctcttcgctcttcctctatgcacctcgcccactgcgc720
gcatcacgcacgcatcatcgcggctacggaacacgcgacccccaccccacataggttttt780
tcaacgagaaatgcagtaccttgccgcgtacgccctcgtggcgctgtctggcaagacgcc840
gtcgaaggcggacgttcaggctgtcctgaaggccgccggcgttgccgtggatgcctcccg900
cgtggatgccgtcttccaggaggtggagggcaagagcttcgatgcgctggtggccgaggg960
ccgcacgaagctggtgggctctggctctgccgctcctgctggcgctgtctccactgctgg1020
tgccggcgctggcgcggtggccgaggcgaagaaggaggagcccgaggaggaggaggccga1080
tgatgacatgggcttcggtctctttgactaagcagccccgcactgcgctgcaggcgcctc1140
tgccgaagattctcacgcgggcctgctctcattgttgtgatgcatcgtttctttctttgc1200
ttgtgacttcggttcgtcttttgatttcgagtggaaagactctgcaaatcgaacaacccg1260
tgcgagatgagctgggagcgtaggcgaggtggctgctcgcgaggctgtaacgaaaaaaaa1320
aagacagcagcggcgccctcggcacaaacacagcgagccctcccctcccccgcttcgtcc1380
ctcctcgagaagagagagacaaagaatctccacagacgctgtacgagaggcaccggcctc1440
gtcatcgagagaagcaaccgcgctttcgtgccgtgacccgctgaccttcgataaccgaga1500
gagggtgtcttctcttctcaaagtgggttcattgcgaagtgctgctctactgtccctcct1560
gctcgtcttcccccagttctcgtttcgtctcttttttgttcgttccatgcactttctctc1620
atactgtttttgcctcttgtcgtacaagaggtgtatcaaacatgcagtacctcgccgcgt1680
acgccctcgtggcgctgtctggcaagacgccgtcgaaggcggacgttcaggctgtcctga1740
aggccgccggcgttgccgtggatgcctcccgcgtggatgccgtcttccaggaggtggagg1800
gcaagagcttcgatgcgctggtggccgagggtcgcacgaagctggtgggctctggctctg1860
ccgctcctgctggcgctgtctccactgctggtgccggcgctggcgcggtggccgaggcga1920
agaaggaggagcccgaggaggaggaggccgatgatgacatgggcttcggtctctttgact1980
aagcagcctccgtgagtggtctactgtcgttactttttgacttgtttcatttgacttgtt2040
ttcttcccgtaagcaaagaacagagtaagcaagcatccatgcacgtcgaagcgatgctac2100
gaaccggtctccctgctgcccatatcccctcagcgacggcgaccctccctcctttccacg2160
tgctgacctcatctcctccaatctttcaccttctcttctgtctctcttcttgggttccct2220
cttgaccgatcctgcatcgaccatgaccaggtaattgctatggctcttcaagagaatagg2280
gactcctacaaggtcaaagctttttccttgtccgagtttgtgcggcagtgttccgactgc2340
caccagctgtgcctcgagaagacggcccgcgttgtggaaaacagcagtcgtgccccgcgg2400
ggatgggaagccctattcaactccgttgcggaggagcggcggtctgaggacttggacctc2460
tgcgagtcgcgatgccggtacatgcaggccacctgcccgtcacaggagaaggtccagcag2520
tacgagcaggctctcgccgccgcgttactcaagccgaaaaagagcacgcccccaaacccg2580
tctgtcttgcaagacgcttcccggtgcggcacgcggtcgaggttttctcgaacgtcctgt2640
tgcccgtggcgcacaaacccagaaggtatccttcccaaccaaagacctgaaacgggaggc2700
acaccgacgccggtcccacgccggacggccgacggaggcgaccgaaggtgctcgagggga2760
ccaagacatggacaacgatatttcgcgccgctggagcaagcttcgccaagagcgcttcgg2820
ctcaaaggcagagcacgttgctccgtcgac2850
<210>18
<211>111
<212>prt
<213>leishmaniainfantum
<400>18
metserthrlystyrleualaalatyralaleualaserleuserlys
151015
alaserproserglnalaaspvalglualailecyslysalavalhis
202530
ileaspvalaspglnalathrleualaphevalmetgluservalthr
354045
glyargaspvalalathrleuilealagluglyalaalalysmetser
505560
alametproalaalaserserglyalaalaalaglyvalthralaser
65707580
alaalaglyaspalaalaproalaalaalaalaalalyslysaspglu
859095
proglugluglualaaspaspaspmetglypheglyleupheasp
100105110
<210>19
<211>2936
<212>dna
<213>leishmaniainfantum
<400>19
gtcgacgccgcacctccgtctctccccctcctcgcccgccatgcgtcgtatgtcgatgct60
tccggtgggggggtgcagtgaacgtagtcgatgatccatgccgcgtcatctctctataag120
tacgtggcacatgcacctacacccaccaccaccgttacccatccacccctggcaccgcat180
gcacccgccactcgcggcttttctctctttttgatctcacccatcccaccttgcccagta240
ttgttcctctgcgactccccgagtgttctcacgcacgcctcctcccacgatcgcttactt300
tcgaatcttcgcttcaccatgtccaccaagtacctcgccgcgtacgctctggcctccctg360
agcaaggcgtccccgtctcaggcggacgtggaggctatctgcaaggccgtccacatcgac420
gtcgaccaggccaccctcgcctttgtgatggagagcgttacgggacgcgacgtggccacc480
ctgatcgcggagggcgccgcgaagatgagcgcgatgccggcggccagctctggtgccgct540
gctggcgtcactgcttccgctgcgggtgatgcggccccggctgccgccgccgcgaagaag600
gacgagcccgaggaggaggccgacgacgacatgggcttcggtctgttcgactaagcccat660
cgacgcgttgtttgcgtgtgtgcgtgtatgtaagggtggatgcggggggcttgctgtttc720
ggtaaagtttcttctggcgtggccccgcgtgcgcccagtcgttcccttgagggcatgaca780
gaagcacacgaaaccggcatgcgcggctccgtgactgaagaggggggcgcccgtctctct840
ttctgcgcgtgtgtcactcattttttttttgccgtcttaactcacaggaaaacgccgccg900
agatggtgttgcggtgtctgatgtagggaggccaatcagggatgaggatggcacggttgg960
tgctcacaacagcggaggcagcccttcaagacgcgtacgcggtcctctctgtctttctca1020
cgtgcatccaccatcgcgcccaagcacgcgcccagcgaaggcgcacacgcgacgaacaga1080
tgcgcggcggatagcggaatggaggaagaggaaggcgggtgggcgcacgcactgaaagct1140
gcgagataggcgagcaatgaacgcgttgatgggtcgatcccccttcctcgtgcagccctg1200
tcgtccatgcggggaggaggtggtgtcagcagcgcagcgaaaaggatggaagcgagggag1260
agagagagagagagacgtgcataatctcgcgggcgggggcggggaggagtgatgatggtg1320
ctgatggacgggcggagcacggatggcgagcagtcggcgcatgcctgtctctcttggcag1380
tgcagcacccggtggagagtctccctcctccaccctgccccgcccttcccacattgctgg1440
tccgccatgtgtgcacgcctgtgcctgtatgcctttacacgaacctttcctactgttcct1500
cacacgcccacaactctatcgaacgtctcctccccccccccctcgacgcatccgtgctaa1560
ccacacaacgcgcacacacgcacgcacgcaatacacacacacgtatatctgcatatacgc1620
atgccacccaccgctgctcgggtgtcgacgtggacgtaaagctttctgtctcgttttacg1680
ccagtcgcacactctccaccatgtccaccaagtacctcgccgcgtacgctctggcctccc1740
tgagcaaggcgtccccgtctcaggcggacgtggaggctatctgcaaggccgtccacatcg1800
acgtcgaccaggccaccctcgcctttgtgatggagagcgttacgggacgcgacgtggcca1860
ccctgatcgcggagggcgccgcgaagatgagcgcgatgccggcggccagctctggtgccg1920
ctgctggcgtcactgcttccgctgcgggtgatgcggccccggctgccgccgccgcgaaga1980
aggacgagcccgaggaggaggccgacgacgacatgggcttcggtctgttcgactaagccc2040
atcgacgcgttggcagctgtgacatgtgacacggcggcggtcctgcttctctcctgccca2100
ccttcatcttctcggaagccgagccgcttatcatctctctgtcttctcgcgcgcgcggct2160
gccgcctctctgtgagcatgcgtgcgtgtacgtcgtgtcgtcccatagcgagctgctggc2220
gcgcgccgcgagagagagagagagagcacgaggcgcgggggaagtggaggcgaatgggga2280
atagagtgggggccctgcgcacgggaaaagccttctcccggaaagtgataccgacacatg2340
cgtatgggaggagggggtgggagaggatgcgagacgtcgttgtctgcatcctctccaacc2400
ccacccccgcgtgtgtgcctgcacttatttttcgtttttcgataatttcttcgtcactta2460
ccagcatctggcatatatgacatacctgcagacacacaagacaggcgcgcagcacacgac2520
gacgggcgccggtgcggtgccgacagacatgttcactgtgcctaacctgtgtatgcgtgt2580
gtgtgtgtgtgtgtgtgtgtgtgcatgggtgggtgaatacgtctctctcacggcggtggt2640
gcgacaacgcagagcgatagggagaggagaagggggcaggagggggcggcactcctcgtg2700
cgcccgcttcccgcgtcgccgacctccactcccttgctcactcactctcgataccagtct2760
cgtggcggagaggaggcgggagtcactctggatatcgccctgtgagagcggcccaacacg2820
ctcacatgagccccaccgctctccatctcatctcctcacggccatcacacatacgcacgc2880
gccactgcctctccttcaacttcctcccacatgattgcttcgacgccactgtcgac2936
<210>20
<211>323
<212>prt
<213>leishmaniainfantum
<400>20
metproserilethrthralalysargglutyrglugluargleuval
151015
aspcysleuthrlystyrsercysvalleuphevalglymetaspasn
202530
valargserglnglnvalhisaspvalglyargalaleuargalalys
354045
alagluphemetmetglylyslysthrleuglnglylysilevalglu
505560
lysargalaglnalalysaspalaserproglualalyshispheasn
65707580
aspglncysgluglutyrasnleuleuserglyasnthrglyleuile
859095
phethrasnasnalavalglngluilethrservalleuaspalahis
100105110
argvallysargalaalaargvalglyalaileserprocysaspval
115120125
ilevalalaalaglyserthrglymetgluprothrglnthrserphe
130135140
pheglnalaleuasnilealathrlysilealalysglymetvalglu
145150155160
ilevalthrglulyslysvalleuservalglyasplysvalaspasn
165170175
serthralathrleuleuglnlysleuasnileserprophetyrtyr
180185190
glnvalasnvalleuservaltrpaspargglyvalleuphethrarg
195200205
gluaspleumetmetthrgluaspmetvalglulysmetleumetglu
210215220
glyleuserasnvalalaalametalaleuglyalaglyileprothr
225230235240
serserthrileglyprometleuvalaspalaphelysasnleuleu
245250255
alavalservalalathrsertyrglupheglugluhisasnglylys
260265270
gluleuargglualaalaileasnglyleuleualaglysercysser
275280285
alaalaalagluproalaalaalaalaproalaalaproseralaala
290295300
alalysglugluproglugluseraspgluaspasppheglymetgly
305310315320
glyleuphe
<210>21
<211>3790
<212>dna
<213>leishmaniainfantum
<400>21
atgcgcgcgcgcgcgcgagagagcatgtatccctgcgtgccttcaatggagacttgacac60
ccctcttctctgctctctgctttctgctccgtcccctaattaccttgactgccttttact120
tgttccctttctatttcctcgggttttggcaaccttccttatgcgcccaacacccacaac180
atacccacccacaaatcgttgcttcacggcctcccctcgtgctttgcagctccctttagc240
aacgatgccgtctatcaccactgccaagcgcgagtacgaggagcgcctcgtcgactgcct300
gaccaagtacagctgcgtgctgttcgtgggcatggacaacgtccgctcgcagcaggtgca360
cgatgtcggccgtgcgctgcgcgcgaaggccgagttcatgatgggcaagaagacgctgca420
gggcaagatcgtggagaagcgcgcgcaagccaaggacgcgagccccgaggcgaagcactt480
caacgatcagtgtgaggagtacaacctgctgagcggcaacaccggcctcatcttcacgaa540
caacgctgtccaggagatcacgtctgtgcttgacgcgcaccgcgtgaagcgcgcggcgcg600
tgtcggagcgatttccccgtgtgacgtgattgtcgctgctggcagcaccggcatggagcc660
gacccagacgtccttcttccaggcgctgaacattgcgacgaagattgccaagggtatggt720
ggagatcgtgacggagaagaaggtgctgagcgtcggcgacaaggtggacaactcgacggc780
gacgctgctgcaaaagctgaacatcagcccgttctactaccaggtgaatgtgctgtccgt840
gtgggaccgcggtgtgctgttcacccgcgaggacctgatgatgacggaggacatggtgga900
gaagatgctgatggaaggcctgagcaacgttgcggcgatggcgctgggtgctggcatccc960
gacgtcttcgacgattggcccgatgctggtggacgccttcaagaacctgctggctgtctc1020
tgtggcgacctcgtacgagttcgaggagcacaacggcaaggagctgcgcgaggccgcgat1080
caacggcctgctggccggctcttgctcggctgctgcggagcccgccgctgccgcgccggc1140
cgcccctagcgccgctgccaaggaggagccggaggagagcgacgaggacgacttcggcat1200
gggcggtctcttctaagcgactcgccatctcttagcctccttgtggtgcgcttgaggtgc1260
tctcgctctgcttctccttgcagtgttggctgactctagcgggtatgtgtcgtcgcatta1320
cacccacctctcccacccctttgctctacgcgctcgcatgcgcaatccgtgaatcatcga1380
gggaagtctctctgggtggcagtgggtaagcttgtgaggaaagaggtgtgtgtgtgagcg1440
ggcaggtacgtcggaccacttaaacaaacaaacacacacacacacggaaagactcacgta1500
cagcatccgtccggcgcaacagcaacgtccgccgcgcgaagcagagcgcgtgcgctcatt1560
gtaccgctgtgaacggaggagggggggactcttcgcttttttctttttcttttttttgtt1620
tcggtagtttattcttcattttccgtctcaactcaaaaaacagcacaaaaacgcggaaac1680
gcagcatgagtggcgccgttgcaatcggggacggtggcggcgcaacgcgtcgtggcaact1740
gcgcatgggttgctatctgatggatggttgcactgctgctcgaacacaggtggacctccc1800
ccccccccgcaacgacgacgtccggtcgagtcgcgggcgtgtggccgtgagcacagggta1860
gcctttctttgcgtcgcacagcacctatcgtcgtcgtcggcactcctcatcacatctccc1920
tcgtgtcgcacgaaggtgtgctgtctgtgaggacgcttccgtgtgagtaggtgcgtgcaa1980
acatgcgtgcatcggcaccggatcgcggtcgggtaggttccacgctcctggagggtcgca2040
agtgtcttgctgctccaggtgactgatgaccaaggccatatcctcacgcaacaccttcac2100
tgctgccgcgctgctttcctccagcacgaagcgagcacaggggcacgggtgggggcggca2160
agcgagtagcctctgaggttgtgcgtaggcgacacgtcgtgtgccagtgggcactgcgca2220
ccttttcagtgttgtgtgtggaacacagggtcggcgcacgctgtcttcggtgatgctttc2280
tcattatgagccgcttgccgagcgtgcgcgcgacccccggcccctcctcacctcctcgcg2340
cggagttaacgcgtgcacgctgtgtcccctgtgtaaagacagcttcccccacccccttgt2400
caactccctctcggtccgtctttctcgcgttcattctctcttcttcgtgaacgaaacacg2460
accactcgcctcgcatattccgcgtgcccaatatcccactcactcccttacacatgcatt2520
gtccgtgccacaacccggcgcacacttcggcacacgaaaaacaccttccccgaccccacg2580
acagatagccaaggctattgcaagtctcacaagatgccgtctatcaccactgccaagcgc2640
gagtacgaggagcgcctcgtcgactgcctgaccaagtacagctgcgtgctgttcgtgggc2700
atggacaacgtccgctcgcagcaggtgcacgatgtcggccgtgcgctgcgcgcgaaggcc2760
gagttcatgatgggcaagaagacgctgcagggcaagatcgtggagaagcgcgcgcaagcc2820
aaggacgcgagccccgaggcgaagcacttcaacgatcagtgtgaggagtacaacctgctg2880
agcggcaacaccggcctcatcttcacgaacaacgctgtccaggagatcacgtctgtgctt2940
gacgcgcaccgcgtgaagcgcgcggcgcgtgtcggagcgatttccccgtgtgacgtgatt3000
gtcgctgctggcagcaccggcatggagccgacccagacgtccttcttccaggcgctgaac3060
attgcgacgaagattgccaagggtatggtggagatcgtgacggagaagaaggtgctgagc3120
gtcggcgacaaggtggacaactcgacggcgacgctgctgcaaaagctgaacatcagcccg3180
ttctactaccaggtgaatgtgctgtccgtgtgggaccgcggtgtgctgttcacccgcgag3240
gacctgatgatgacggaggacatggtggagaagatgctgatggaaggcctgagcaacgtt3300
gcggcgatggcgctgggtgctggcatcccgacgtcttcgacgattggcccgatgctggtg3360
gacgccttcaagaacctgctggctgtctctgtggcgacctcgtacgagttcgaggagcac3420
aacggcaaggagctgcgcgaggccgcgatcaacggcctgctggccggctcttgctcggct3480
gctgcggagcccgccgctgccgcgccggccgcccctagcgccgctgccaaggaggagccg3540
gaggagagcgacgaggacgacttcggcatgggcggtctcttctaagcgactcgccatctc3600
ccactgagcaccgtcgagtgttcgtgtgttcgcagggtggacagcggcgagcgtgtgatg3660
cccttggatcatcaggaagcaactctctccctttctctctgtgttcttcgtttcttcttt3720
cattagttttggatcgccgtgcgctgcgcatcgctcagttctcatttatatcaataacaa3780
caacgaagac3790
<210>22
<211>260
<212>prt
<213>leishmaniamajor
<400>22
metglylysthrvalleusercysarglysglyasnglyservaltyr
151015
glnvalhisglyhislysargleuglyproalalysleuargileleu
202530
asptyralagluarghisglytyrmetargglyvalvallysserile
354045
gluhisglualaglyargglyalaalaleualaargvalgluphearg
505560
hisprotyrlyspheargargvallysgluleumetvalalaproglu
65707580
glymetphethrglyglnservalphecysglyglnlysalaproleu
859095
alaileglyasnvalleuproleuglyglnilethrgluglycysile
100105110
valcysasnvalglualalysproglyaspargglythrleualaarg
115120125
alaserglyasptyrcysileileileserhisasnhisgluthrgly
130135140
argthrargleulysleuproserglyglnlyslysservalproser
145150155160
thrserargalametileglyileileserglyglyglyargileglu
165170175
lysprovalleulysalaglyasnserphetyrargpheargglylys
180185190
argasncystrpprolysvalargglyvalalaargasnprovalglu
195200205
hisprohisglyglyglyasnhisglnhisileglyhisproserthr
210215220
valserarghisserproproglyglnlysvalglyleuilealaala
225230235240
argargthrglyargileargglyglylysalavallysglyalatrp
245250255
hisprogluglu
260
<210>23
<211>783
<212>dna
<213>leishmaniamajor
<400>23
atgggtaagactgtgctgagctgccgtaagggcaacggctccgtgtaccaggtgcacggc60
cacaagcgccttggccccgccaagctgcgcattctggactacgccgagcgccacggctac120
atgcgcggtgtggtgaagtcgatcgagcacgaggctggccgcggtgcggcgctggcgcgc180
gtggagttccgccacccgtacaagttccgccgcgtgaaggagctgatggtggcgccggag240
ggcatgttcaccggccagtcggtgttctgcggccagaaggccccgctcgcgatcggcaac300
gtgctgccccttggccagatcacggagggctgcattgtgtgcaacgtggaggcgaagccc360
ggtgaccgcggcacgctggcgcgcgcgtccggcgactactgcatcatcatctcgcacaac420
cacgagacaggccgcacgcgcctgaagctgccgagcgggcagaagaagtccgtgccgagc480
acgagccgcgcgatgatcggcatcatcagcggcggtggccgcatcgagaagcccgtgctg540
aaggccggtaactcgttctaccgcttccgcggcaagcgcaactgctggcccaaggtgcgt600
ggtgttgcccgcaacccggtggagcacccgcacggtggtggtaaccatcagcacattggc660
cacccgtcgacggtgtcgcgccactcgccgccgggccagaaggtgggtctgatcgctgcc720
cgtcgcaccggccgtattcgcggtggtaaggctgtcaagggcgcgtggcacccggaggag780
taa783
<210>24
<211>252
<212>prt
<213>leishmaniamajor
<400>24
metalathrhisservaltyrglyasnalaseraspmetproalaval
151015
proalaprogluseralailelysargalaalaphelysglnglngln
202530
thrgluserphelyslysalavalvalalaarglysalaalalysala
354045
alaleulyslysthralatyrleuargalaarglystyrserargglu
505560
tyrargglyalaglulyslysleuvalthrleuargargglnalaala
65707580
serhisglyasntyrtyrleuglualalysprolysvalalavalval
859095
thrargileargglyilealalysvalasnprolysglnarglysile
100105110
leuglnleuleuargleuargglnilepheasnthrvalphevallys
115120125
metasnlysprometgluasnmetleuargalavalgluprotyrile
130135140
alatyrglytyrproserleualathrvalargalametvaltyrlys
145150155160
argglytyrleulysileasnglyglnargvallysilethraspasn
165170175
glnmetilelysasplystyrasnasnvalaspilevalcysalaglu
180185190
aspmetvalasnglniletyrthrcysglylyshispheargthrval
195200205
thrhisglymettrpprophelysleualaproproalaglyglymet
210215220
argglnlysargarghisphevalgluglyglyasptyrglyasnarg
225230235240
aspthrleuileasnargpheleualaargmetile
245250
<210>25
<211>759
<212>dna
<213>leishmaniamajor
<400>25
atggccacacactcagtttacggcaacgcatccgacatgcccgctgtccctgcccctgag60
tccgcgatcaagcgtgctgcgttcaagcagcagcagacggagagcttcaagaaggccgtg120
gtggccagaaaggctgccaaggctgccctgaagaagaccgcctacctgcgtgcccgcaaa180
tactcccgcgagtaccgcggtgcggagaagaagctggtgacgctgcgccgccaggccgcc240
tctcacggtaactactacctggaggcgaagccgaaggttgccgtggtgactcgcatccgc300
ggtatcgccaaggtgaacccgaagcagcgcaagattcttcagttgctgcgcctgcgccag360
atcttcaacacggtgtttgtgaagatgaacaagccgatggagaacatgctgcgtgcggtg420
gagccctacatcgcgtacggctacccgtccctggccaccgtccgcgcgatggtgtacaag480
cgcggctacctgaagatcaacggccagcgcgtgaagatcaccgacaaccagatgatcaag540
gataagtacaacaacgtggacattgtgtgtgccgaggatatggtgaaccagatctacacc600
tgcggcaagcacttccgcacggtgacgcacggcatgtggcccttcaagctggcccctccg660
gccggtggcatgcgccagaagcgccgtcacttcgtggagggtggcgactatggtaaccgc720
gacaccttgatcaaccgcttcctcgcccgcatgatctga759
<210>26
<211>264
<212>prt
<213>leishmaniamajor
<400>26
metproglylysgluvallyslysvalthrglnproalalysalaala
151015
serprotyrlyslysproalavalalaserhisphealaalaargpro
202530
lysasnpheglyileglyglnaspvalprotyralaargaspleuser
354045
argphemetargtrpprothrphevalthrmetglnarglyslysarg
505560
valleuglnargargleulysvalproproalaleuasnglnphethr
65707580
lysvalleuaspargalaserargasnglualaleulysleuilelys
859095
lystyralaprogluthrarglysalaargarggluargleuglnlys
100105110
valalagluglulyslyslysaspprolyslysthrvalserthrlys
115120125
alaproleualavalvalthrglyleuglngluvalthrargalaile
130135140
glulyslysglnalaargmetvalvalilealaasnasnvalasppro
145150155160
valgluleuvalleutrpmetproasnleucysargalaasnlysile
165170175
protyralailevallysaspmetalaargleuglyaspalailegly
180185190
arglysthralathrcysvalalaleuthraspvalasnalagluasp
195200205
glualathrleulysasnleuileargservalasnalaargpheleu
210215220
serargseraspvalileargargglntrpglyglyleuglnleuser
225230235240
leuargserargalagluleuarglyslyshisalaargasnalagly
245250255
valaspalaalaalaileilegln
260
<210>27
<211>795
<212>dna
<213>leishmaniamajor
<400>27
atgcccggcaaggaagtgaagaaggtgacgcagcccgcgaaggccgcgtctccgtacaag60
aagcccgccgttgcgtcgcatttcgcggcccgcccgaagaacttcggtattggccaggat120
gtgccgtacgcgcgtgacctgtcccgcttcatgcggtggccgacgttcgtgacgatgcag180
cgcaagaagcgcgtgctgcagcgccgcctgaaggtgccgccggcgctgaaccagttcacg240
aaggtgctggaccgcgcgagccgaaacgaggcgctgaagctgattaagaagtacgcgccg300
gagacccgcaaggctcgccgcgagcgcctgcagaaggttgccgaggagaagaagaaggac360
ccgaagaagacggtatcgacgaaggctcccctggctgttgtgaccggtctgcaggaggtg420
acgcgcgcgatcgagaagaagcaggctcgcatggttgtgatcgcgaacaacgtggaccct480
gtggagctcgtgctgtggatgccgaacctgtgccgcgcgaacaagatcccgtatgccatc540
gtgaaggacatggcgcgcctgggcgatgcgatcgggcggaagacggcgacgtgcgttgcg600
ctcaccgacgtgaacgccgaggatgaggcgacgctgaagaacctgatccgctccgtgaac660
gctcgcttcttgtcccgctcggacgtgatccgccgccagtggggtggtctgcagctgtct720
ctgcgatcccgcgcggagctgcgcaagaagcatgcccgcaacgctggtgtggacgccgcg780
gccatcatccagtaa795
<210>28
<211>222
<212>prt
<213>leishmaniamajor
<400>28
metalapheproserarglysaspalapheargalaglnarglysgly
151015
alalyslyshisargprogluileilevalileaspleulysasphis
202530
valleuglyargalaalaalavalvalalalysglnleuleuleugly
354045
lyslysilethrvalvalargcysgluglnleuasnilealaglythr
505560
gluileargasnlysilelystyrleuglntyrleuarglysarglys
65707580
leuthrasnprothrlysglyprophehishisargalaproserasp
859095
valphevalargthrvalargsermetleuproargtyrthrlysarg
100105110
glymetlysalaleuasnserleuvalalatyrgluglyilepropro
115120125
asnvalvalargthrglyglyargvalvalileproargalaglnarg
130135140
hisvalcystyrargsergluargprotyrthrvalleuglyasnmet
145150155160
cyslyshisvalglytrplystyrseraspvalvalalaasnleuglu
165170175
lysalaargvalglulysalaserarghishisglulysglnalalys
180185190
leuargaspalatrplysseralaarglysglualaleualalysmet
195200205
prolyshisasnvalgluvalleulyslyspheglytyrala
210215220
<210>29
<211>669
<212>dna
<213>leishmaniamajor
<400>29
atggcctttcctagccgcaaggatgcgttccgcgcgcagcgcaagggcgccaagaagcac60
cgccccgagatcatcgtgatcgacctgaaggatcacgtgcttggtcgcgcggcggctgtg120
gttgccaagcagctgctcctgggtaagaagatcaccgtggtgcgctgcgagcagctcaac180
attgccggtacggagatccgcaacaagatcaagtacctgcagtacctgcgcaagcggaag240
ctgacgaaccccacaaagggtcccttccaccaccgtgccccgtccgacgtgtttgtccgc300
actgtgcgcagcatgctgccccggtacacgaagcgcggcatgaaggcgcttaactcgctg360
gtggcctacgagggaattccgcccaacgtggtgcgcacgggcgggcgcgtggtgatcccg420
cgcgcccagcgccatgtgtgctaccgctcggagcgtccttacacagtgctcggcaacatg480
tgcaagcacgtgggctggaagtacagcgacgtcgtcgccaatctcgagaaggctcgcgtg540
gagaaggcgtcccgccaccacgaaaagcaggcgaagcttcgcgacgcgtggaagtcggcc600
cgcaaggaggcgctcgccaagatgcccaagcacaacgtggaggtgctgaagaagtttggc660
tacgcgtag669
<210>30
<211>267
<212>prt
<213>leishmaniamajor
<400>30
metthrproleuserleuserserserarghisserphelysglnasn
151015
gluthrglnasnmetvalserleulysleuglnalaargleualaser
202530
serileleuglycysglyargalaargvaltrpleuaspproasnglu
354045
alavalgluileglnasnalaasnserarglysservalarglysleu
505560
ilelysaspglypheileilearglysprovallysvalhisserarg
65707580
alaargtrparglysmetlysglualalysaspmetglyarghisasn
859095
glyvalglyargarggluglyserargglualaargmetproserlys
100105110
gluleutrpmetargargleuargileleuargargleuleuarglys
115120125
tyrargalaasplyslysileasparghisvaltyrargaspleutyr
130135140
metargalalysglyasnvalpheargasnlysargasnleuvalglu
145150155160
hisilehislysilelysasnglulyslyslysgluargglnleuala
165170175
gluglnleualaalalyshisleuargaspgluglnasnargasnlys
180185190
alaarglysglngluleulyslysargglulysgluarggluargala
195200205
argargaspaspalaalaalaalaalaglnlyslyslysalaaspala
210215220
alalyslysseralaalaproalaalalysseralaalaproalaala
225230235240
lysalaalaalaproalathrlysalaalaalaalaalaproalathr
245250255
lysglyalaalaprovallyslysserlyslys
260265
<210>31
<211>804
<212>dna
<213>leishmaniamajor
<400>31
atgacccctctctccctctcttcctcccgccacagttttaagcagaacgaaacgcagaac60
atggtgtctctgaagctgcaggctcgccttgcgtcgagcatcctcggctgcggccgcgcc120
cgcgtgtggctggaccccaacgaggcggtggagatccagaacgcgaactcgcgcaagagc180
gtgcgcaagctgatcaaggatggcttcatcatccgcaagccggtgaaggtgcactcgcgc240
gcgcggtggcgtaaaatgaaggaggcgaaggacatggggcgccacaacggcgttgggcgc300
cgcgagggtagccgcgaggcccgcatgccgagcaaggagttgtggatgcgccgcctgcgc360
attctgcgccgcctgctgcgcaagtaccgcgcggacaagaagattgaccgccacgtgtac420
cgcgacctgtacatgcgcgcgaagggtaacgtgttccgcaacaagcgcaaccttgtggag480
cacatccacaagatcaagaatgagaagaagaaggagcgccagctggcggagcagctcgcg540
gcgaagcacctgcgcgacgagcagaaccgcaacaaggctcgcaagcaggagctgaagaag600
cgcgagaaggagcgcgagcgcgcgaggcgcgacgacgctgctgccgctgcgcagaagaag660
aaggcggacgccgcgaagaagtccgccgcgcctgctgcgaagtccgccgcgcctgccgcg720
aaggctgctgcccccgccacgaaggccgctgctgctgcccccgccacgaagggtgctgcg780
ccggtgaagaagtcgaagaagtaa804
<210>32
<211>273
<212>prt
<213>leishmaniamajor
<400>32
metalalyslyshisleulysargleutyralaprolysasptrpmet
151015
leuserlysleuthrglyvalphealaproargproargproglypro
202530
hislysleuargglucysleuproleuleuvalileileargasnarg
354045
leulystyralaleuasnalaarggluglyglumetileleuarggln
505560
glyleuvalhisvalaspasnhisproargargaspglylystyrpro
65707580
alaglyphemetaspvalvalgluileprolysthrglyaspargphe
859095
argleumettyraspvallysglyargphealaleuvalasnleuser
100105110
glualaglualaglnilelysleumetlysvalvalasnleutyrthr
115120125
alathrglyargvalprovalalavalthrhisaspglyhisargile
130135140
argtyrproaspprohisthrserileglyaspthrilevaltyrasn
145150155160
vallysglulyslyscysvalaspleuilelysasnargglnglylys
165170175
alavalilevalthrglyglyalaasnargglyargileglygluile
180185190
vallysvalglucyshisproglyalapheasnilealahisleulys
195200205
aspalaserglyalagluphealathrargalaalaasnilepheval
210215220
ileglylysaspleuasnasnleuglnvalthrvalprolysglngln
225230235240
glyleuargmetasnvalileglngluargglugluargleuileala
245250255
alaglualaarglysasnalaproalaargglyalaargargalaarg
260265270
lys
<210>33
<211>822
<212>dna
<213>leishmaniamajor
<400>33
atggccaagaagcacctcaagcgcttgtatgcgcccaaggactggatgctgagcaagctg60
accggcgtgttcgcgccgcgtccgcgtccgggtccgcacaagctgcgcgagtgcctgccg120
ctgctggtgatcatccgcaaccggctgaagtacgcgctgaacgcgcgcgagggtgagatg180
atcctgcgccagggtctggtgcacgtggacaaccacccgcgccgcgacggcaagtatccc240
gccggtttcatggacgtggtcgagatcccgaagacgggcgaccgcttccgcctgatgtac300
gacgtcaagggccgcttcgcgttggtgaacctgtccgaggcggaggcgcagatcaagctg360
atgaaggttgtgaacctgtacacggccaccggccgcgtgccggtcgctgtgacgcacgac420
ggccaccgcatccgctacccggacccgcacacctccattggtgacaccatcgtgtacaac480
gtcaaggagaagaagtgcgtggacctgatcaagaaccgccagggcaaggccgtgatcgtg540
accggtggcgccaaccgcggccgcatcggcgagatcgtgaaggtggagtgccaccccggt600
gcgttcaacattgcgcacctgaaggacgcgtccggcgccgagttcgccacccgcgccgcg660
aacatcttcgtgatcggcaaggacctgaacaacctgcaggtaacggtgccgaagcagcag720
ggcctgcgcatgaacgtgatccaggagcgcgaggagcgcctgatcgcggcggaggcccgc780
aagaacgcgccggctcgtggtgcccgcagggcccgcaagtga822
<210>34
<211>249
<212>prt
<213>leishmaniamajor
<400>34
metlysleuasnilealatyrproargasnglythrvallysglnphe
151015
gluileseraspgluvalleuargargvalglnleuglnasptyrarg
202530
leuglyasngluvalaspglyalailepheglysergluphelysgly
354045
tyrilepheargleuargglyglyserasplysaspglyphepromet
505560
valproglyvalleualaserserargvalserleuleuvallysarg
65707580
glyalaileglypheasnthrpheargglytyrglnglygluargarg
859095
arglysasnvalargglycysvalleualaseraspilealaleuval
100105110
asnvalthrileserlysvalglyaspglnproilegluglyvalthr
115120125
aspthrthralaproargargleuglyprolysargalaserlysile
130135140
arglysleupheasnleuserargthrgluaspvalarglystyrval
145150155160
valargargargvalvallysserglylyslysaspargleulysala
165170175
prolysileglnargleuilethrproargvallysalaargargala
180185190
lyslysalalysaspalailealalysvalargalaseralaalaglu
195200205
argargglutyrleuargleuilealaserasnargargalaleuarg
210215220
glnargasphisserlyslyshisthrarglysvalhisalaglnarg
225230235240
alagluvalalaalapheglnlyslys
245
<210>35
<211>750
<212>dna
<213>leishmaniamajor
<400>35
atgaagctcaacatcgcgtacccccgcaacgggacggtgaagcagttcgagatctcggac60
gaggtgctccgccgcgtgcagctgcaggactaccgcctcggcaacgaggtggacggcgcc120
atctttggtagcgagttcaagggctacatcttccgcctgcgcggtggctcggacaaggat180
ggtttcccgatggtccctggcgtgcttgcctccagccgtgtgtcgctgctggtgaagcgc240
ggtgcgatcggcttcaacaccttccgcggctaccagggtgagcgccgccgcaagaacgtt300
cgcggctgcgtgctcgcgagcgacattgcgctggtgaacgtgaccatctccaaggtcggt360
gaccagccgatcgagggtgtgacggacaccacggctccccgccgtctgggtccgaagcgc420
gcgagcaagatccgcaagctcttcaacctgtcccgcaccgaagacgtgcggaagtacgtt480
gttcgccgccgcgtcgtgaagagcggcaagaaggaccggctgaaggccccgaagatccag540
cgtctgatcacgccgagggtcaaggcccgccgtgccaagaaggccaaggacgccatcgcc600
aaggtgcgcgcgtctgccgctgagcgccgtgagtacctgcgccttatcgcctcgaaccgc660
cgtgcgctgcgccagcgtgaccactccaagaagcacacccggaaggtgcacgcccagcgc720
gctgaggtggcagcattccagaagaagtaa750
<210>36
<211>1436
<212>dna
<213>artificial
<220>
<223>proteinq
<400>36
atgagaggatctcaccatcaccatcaccatacggatccgcatgcgagctcgaacaacaac60
aacaataacaataacaacaacctcgggatcgagggaaggcctttagctactcctcgcagc120
gccaagaaggccgtccgcaagagcggctccaagtccgcgaaatgtggtctgatcttcccg180
gtgggccgcgtcggcgggatgatgcgccgcggccagtacgctcgccgcatcggtgcctct240
ggcgcccccaggatttcagaattctccgtgaaggcggccgcgcagagcgggaagaagcgg300
tgccgcctgaacccgcgcaccgtgatgctggccgcgcgccacgacgacgacatcggcacg360
cttctgaagaacgtgaccttgtctcacagcggcgttgtgccgaacatcagcaaggcgatg420
gcaaagaagaagggcggcaagaagggcaaggcgacaccgagcgcgcccgaattcggatcc480
tctagacccatgtccaccaagtacctcgccgcgtacgctctggcctccctgagcaaggcg540
tccccgtctcaggcggacgtggaggctatctgcaaggccgtccacatcgacgtcgaccag600
gccaccctcgcctttgtgatggagagcgttacgggacgcgacgtggccaccctgatcgcg660
gagggcgccgcgaagatgagcgcgatgccggcggccagctctggtgccgctgctggcgtc720
actgcttccgctgcgggtgatgcggctccggctgccgccgccgcgaagaaggacgagccc780
gaggaggaggccgacgacgacatgggcccctctagagtcgaccccatgcagtacctcgcc840
gcgtacgccctcgtggcgctgtctggcaagacgccgtcgaaggcggacgttcaggctgtc900
ctgaaggccgccggcgttgccgtggatgcctcccgcgtggatgccgtcttccaggaggtg960
gagggcaagagcttcgatgcgctggtggccgagggccgcacgaagctggtgggctctggc1020
tctgccgctcctgctggcgctgtctccactgctggtgccgccgctggcgcggtggccgag1080
gcgaagaaggaggagcccgaggaggaggaggccgatgatgacatgggccccgtcgacctg1140
cagcccgccgctgccgcgccggccgcccctagcgccgctgccaaggaggagccggaggag1200
agcgacgaggacgacttcggcatgggcggtctcttctaagcgactcgccatctcttagcc1260
tccttgtggtgcgcttgaggtgctctcgctctgcttctccttgcagtgttggctgactct1320
ggcgggtatgtgccgtcgcattacacccacctctcccacccctttgccctacgcgctcgc1380
atgcgcaatccgtgaatcatcgagggaagtctctctgggtggcagtgggtaagctt1436
<210>37
<211>412
<212>prt
<213>artificial
<220>
<223>proteinq
<400>37
metargglyserhishishishishishisthraspprohisalaser
151015
serasnasnasnasnasnasnasnasnasnasnleuglyileglugly
202530
argproleualathrproargseralalyslysalavalarglysser
354045
glyserlysseralalyscysglyleuilepheprovalglyargval
505560
glyglymetmetargargglyglntyralaargargileglyalaser
65707580
glyalaproargileserglupheservallysalaalaalaglnser
859095
glylyslysargcysargleuasnproargthrvalmetleualaala
100105110
arghisaspaspaspileglythrleuleulysasnvalthrleuser
115120125
hisserglyvalvalproasnileserlysalametalalyslyslys
130135140
glyglylyslysglylysalathrproseralaproglupheglyser
145150155160
serargprometserthrlystyrleualaalatyralaleualaser
165170175
leuserlysalaserproserglnalaaspvalglualailecyslys
180185190
alavalhisileaspvalaspglnalathrleualaphevalmetglu
195200205
servalthrglyargaspvalalathrleuilealagluglyalaala
210215220
lysmetseralametproalaalaserserglyalaalaalaglyval
225230235240
thralaseralaalaglyaspalaalaproalaalaalaalaalalys
245250255
lysaspgluproglugluglualaaspaspaspmetglyproserarg
260265270
valaspprometglntyrleualaalatyralaleuvalalaleuser
275280285
glylysthrproserlysalaaspvalglnalavalleulysalaala
290295300
glyvalalavalaspalaserargvalaspalavalpheglngluval
305310315320
gluglylysserpheaspalaleuvalalagluglyargthrlysleu
325330335
valglyserglyseralaalaproalaglyalavalserthralagly
340345350
alaalaalaglyalavalalaglualalyslysglugluprogluglu
355360365
gluglualaaspaspaspmetglyprovalaspleuglnproalaala
370375380
alaalaproalaalaproseralaalaalalysglugluprogluglu
385390395400
seraspgluaspasppheglymetglyglyleuphe
405410
<210>38
<211>21
<212>dna
<213>artificial
<220>
<223>primer
<400>38
aacacgaaggagggcaaggtc21
<210>39
<211>22
<212>dna
<213>artificial
<220>
<223>primer
<400>39
cttcttcgcggcctttgccttg22
<210>40
<211>22
<212>dna
<213>artificial
<220>
<223>primer
<400>40
tgcacgctggcaaattgggtac22
<210>41
<211>20
<212>dna
<213>artificial
<220>
<223>primer
<400>41
cttcttcgtgcgcacagcag20
<210>42
<211>22
<212>dna
<213>artificial
<220>
<223>primer
<400>42
cttcttcgcggcctttgccttg22
<210>43
<211>20
<212>dna
<213>artificial
<220>
<223>primer
<400>43
cttcttcgtgcgcacagcag20
<210>44
<211>29
<212>dna
<213>artificial
<220>
<223>primer
<400>44
cgggatccatgtctcactgcaagttcgag29
<210>45
<211>28
<212>dna
<213>artificial
<220>
<223>primer
<400>45
aactgcagttacttcttcgcggcctttg28
<210>46
<211>28
<212>dna
<213>artificial
<220>
<223>primer
<400>46
cgggatccatgtgcacgctggcaaattg28
<210>47
<211>29
<212>dna
<213>artificial
<220>
<223>primer
<400>47
cccaagcttttacttgccgaggcgctcgc29
<210>48
<211>419
<212>prt
<213>leishmaniainfantum
<400>48
metserhiscyslysphegluhisproarghisglyhisleuglyphe
151015
leuproarglysargserargglnileargglyargalaargalaphe
202530
prolysaspaspalathrglnlysprohisleuthrserphemetval
354045
phelysalaglymetthrhisilevalargaspvalaspargprogly
505560
serlysvalasnlyslysgluvalvalgluprovalthrileleuglu
65707580
alaproprometvalilevalglyilevalglytyrargglnthrpro
859095
valglyleulysthrileglythrvaltrpalahishisthrserval
100105110
glupheargargargtyrtyrlysasntrplysglnseralaglnleu
115120125
alapheserargglnlysglnphealaasnthrlysgluglylysval
130135140
alaglualaargthrleuasnalaphealalyslysalaservalile
145150155160
argvalilealahisthrglnleuarglysleuargasnhisargval
165170175
glyvallyslysalahisvalglngluileglnvalasnglyglyser
180185190
valalaalalysilealaleualalysserleuleuglulysgluval
195200205
argvalaspservalpheglnglnserglualacysaspvalcysser
210215220
valthrlysglyhisglythrgluglyvalvallysargtrpglyval
225230235240
alacysleuproarglysthrhisargglyleuarglysvalalacys
245250255
ileglyalatrphisproalaargvalmettyrthrvalalaargala
260265270
glyglnhisglytyrhishisargthrglnleuasnlyslysiletyr
275280285
glnileglyargservalalavalgluproasnglnalathrthrthr
290295300
tyraspleuthralalysthrilethrprometglyglyphevalgly
305310315320
tyrglythrvalargasnasptyrvalmetleulysglyservalser
325330335
glyproargargargvalmetthrleuargargprometalaprogln
340345350
thrserarghisleulysglulysilevalleulyspheileaspthr
355360365
serserlysileglyhisglyargpheglnthrlyslysglulysasn
370375380
glntrppheglyproleulyslysaspargileargargglugluarg
385390395400
leuarglysgluargalaalaargalavalgluarglysalalysval
405410415
alalyslys
<210>49
<211>1260
<212>dna
<213>leishmaniainfantum
<400>49
atgtctcactgcaagttcgagcacccccgccacggccatctcggcttcctgccgcgcaag60
cgctcgcgccagatccgcggccgcgcgcgcgcgttccccaaggacgacgcgacgcagaag120
ccccacctgacgagtttcatggtgttcaaggccggcatgacgcacattgtgcgtgatgtc180
gatcgccctggatcgaaggtgaacaagaaggaggtggtggagccggtgacgattctggag240
gcgccgccgatggtgattgtcggcattgtgggctaccgccaaacgccggtcggcctgaag300
acgatcggcaccgtgtgggcgcaccacacgagcgtcgagttccgccgccgctactacaag360
aactggaagcagtctgcgcaactggccttctcccgccagaagcagtttgcgaacacgaag420
gagggcaaggtcgccgaggcgcgcacgctgaacgcgttcgcgaagaaggcgtccgtcatc480
cgcgtgatcgcgcacacgcagctgcgcaagcttcgcaaccaccgcgtgggcgtgaagaag540
gcgcacgtgcaggagatccaggtcaacggcggcagcgttgcggcgaagatcgcgctggcc600
aagtccctgctggagaaggaggtgcgcgtggactccgtgttccagcagtcggaggcgtgc660
gacgtgtgctccgtgacgaaaggccacggtacggagggcgtggtgaagcgctggggcgtt720
gcctgcctgccacgcaagacgcaccgcggtctgcgcaaggttgcgtgcatcggcgcgtgg780
caccctgcccgcgtcatgtacactgtcgcgcgcgccggtcagcacggttaccaccaccgc840
acgcagctgaacaagaagatctaccagatcggccgctccgttgctgtggaaccgaaccag900
gcgacgacgacctacgatctgacggccaagacgatcacacccatgggtggcttcgtcggc960
tacggcacggtgcgcaacgactacgtgatgctgaagggctccgtgtctggtccgcgccgc1020
cgtgtgatgacgctccgccgcccgatggcaccgcagacgtcgcgccacctgaaggagaag1080
atcgtgctgaagttcatcgacactagctcgaagattggccacggccgcttccagacgaag1140
aaggagaagaaccagtggttcggcccgctcaagaaggaccgcatccgccgcgaggagcgc1200
ctgcgcaaggagcgcgctgcccgcgccgtggagcgcaaggcaaaggtcgcgaagaagtaa1260
<210>50
<211>419
<212>prt
<213>leishmaniamexicana
<400>50
metserhiscyslysphegluhisproarghisglyhisleuglyphe
151015
leuproarglysargserargglnileargglyargalaargalaphe
202530
prolysaspaspalathrglnlysprohisleuthrserphemetval
354045
phelysalaglymetthrhisilevalargaspvalaspargprogly
505560
serlysvalasnlyslysgluvalvalgluprovalthrileleuglu
65707580
alaproprometvalilevalglyilevalglytyrargglnthrpro
859095
valglyleulysthrileglythrvaltrpalahishisthrserval
100105110
glupheargargargtyrtyrlysasntrplysglnseralaglnleu
115120125
alapheserargglnlysglnphealaasnthrlysgluglyargile
130135140
alaglualaargthrleuasnalaphealalyslysalaservalile
145150155160
argvalilealahisthrglnleuarglysleuargasnhisargval
165170175
glyvallyslysalahisvalglngluileglnileasnglyglyasn
180185190
valalaalalysilealaleualalysserleuleuglulysgluval
195200205
argvalaspservalpheglnglnserglualacysaspvalcysser
210215220
valthrlysglyhisglythrgluglyvalvallysargtrpglyval
225230235240
alacysleuproarglysthrhisargglyleuarglysvalalacys
245250255
ileglyalatrphisproalaargvalmettyrthrvalalaargala
260265270
glyglnhisglytyrhishisargthrglnleuasnlyslysiletyr
275280285
glnileglyargservalalavalgluproasnglnalathrthrthr
290295300
tyraspleuthralalysthrilethrprometglyglyphevalgly
305310315320
tyrglythrvalargasnasptyrvalmetleulysglyservalser
325330335
glyproargargargvalmetthrleuargargprometalaprogln
340345350
thrserargglnleulysglulysilevalleulyspheileaspthr
355360365
serserlysileglyhisglyargpheglnthrlyslysglulysser
370375380
glntrppheglyproleulyslysaspargileargargglugluarg
385390395400
leuarglysgluargalaalaargalavalgluarglysalalysala
405410415
alalyslys
<210>51
<211>1260
<212>dna
<213>leishmaniamexicana
<400>51
atgtctcactgcaagttcgagcacccccgccacggccatctcggcttcctgccgcgcaag60
cgctcgcgccagatccgcggccgcgcgcgcgcgttccccaaggacgacgcgacgcagaag120
ccccacctaacgagcttcatggtgttcaaggctggcatgacgcacattgtgcgtgatgtc180
gaccgccctggatcgaaggtgaacaagaaggaggtggtggagccggtgacgatcctggag240
gcgccgccgatggtgattgtcggcattgtgggctaccgccaaacgccggttggcctgaag300
acgatcggcaccgtgtgggcgcaccacacgagcgtggagttccgccgccgctactacaag360
aactggaagcagtctgcgcagctggccttctcccgccagaagcagttcgcgaacacgaag420
gagggcaggatcgctgaggcgcgcacgctgaacgcgttcgcgaagaaggcgtccgtcatc480
cgcgtgatcgcgcacacgcagctgcgcaagcttcgcaaccaccgcgtgggcgtgaagaag540
gcgcacgtgcaggagatccagatcaacggcggcaacgttgcggcgaagatcgcgctggcc600
aagtccctgctggagaaggaggtgcgcgtcgactccgtgttccagcagtcggaggcgtgc660
gacgtgtgctccgtgacgaaaggtcacggtacggagggcgtggtgaagcgctggggcgtt720
gcctgcctgccgcgcaagacacaccgcggcctgcgcaaggtggcgtgcatcggcgcgtgg780
caccctgcccgcgtcatgtacactgtcgcgcgcgccggtcagcacggctaccaccaccgc840
acgcagctgaacaagaagatctaccagatcggccgctccgttgctgtggagccgaaccag900
gcgacgacgacctacgatctgacggccaagacgatcacgcccatgggcggcttcgtcggc960
tacggtacggtgcgcaacgactacgtgatgctgaagggctccgtgtctggcccgcgccgc1020
cgtgtgatgacgctgcgacgcccgatggcgccgcagacgtcgcgccagctgaaggagaag1080
atcgtgctgaagttcatcgacacgagctcgaagatcggccacggccgcttccagacgaag1140
aaggagaagagccagtggttcggcccgctcaagaaggaccgcatccgccgcgaggagcgc1200
ctgcgcaaggagcgcgctgcccgcgccgtggagcgcaaggcaaaggccgcgaagaagtaa1260
<210>52
<211>305
<212>prt
<213>leishmaniainfantum
<400>52
metprophevallysvalvallysasnlysalatyrphelysargphe
151015
glnvallystyrargargargarggluglylysthrasptyrhisala
202530
argargglnmetvalleuglnasplysthrlyspheglyserprolys
354045
tyrargleuvalvalargilethrasnlysaspileilealaglnile
505560
valglnalalysilevalglyaspgluvalvalmetalaalatyrala
65707580
hisgluleuproalapheglyilegluhisglyleuthrasntyrala
859095
alaalatyralathrglyleuleuleualaargargthrleualalys
100105110
leuglyilealaasplyspheglnglyalalysglualaaspglyser
115120125
tyrseralavalargthrlyslysaspaspgluglyaspaspgluglu
130135140
argpheprophelysalaileleuaspvalglyleualaargthrthr
145150155160
thrglyalaargvalpheglyvalleulysglyalavalaspglygly
165170175
metalavalprohisargproasnargpheproglytyrasnlysglu
180185190
lysserserleuaspalalysvalhisargaspargilepheglylys
195200205
hisvalalaglutyrleulysglnvallysgluglualaserserasn
210215220
proaspglulyscysvalglnpheserargtyrmetalaalalysval
225230235240
leuprogluserilegluglymettyrlyslysalahisalaalaile
245250255
argalaaspproserlysserleuprolyslysalalyslysglugly
260265270
valalahislyssertyrlysthrlyslysleuserglyalaglulys
275280285
argalaalaalalysalalysvalalaalailearggluargleugly
290295300
lys
305
<210>53
<211>918
<212>dna
<213>leishmaniainfantum
<400>53
atgccgttcgtcaaggtcgtgaagaacaaggcgtacttcaagcgcttccaggtgaagtac60
cgccgtcgccgcgagggcaagacggactaccacgcgcgccgccagatggtgctgcaggac120
aagacgaagttcggctcgcccaagtaccgccttgttgtgcgcatcacgaacaaggacatc180
attgcgcagatcgtgcaggcgaagatcgtcggcgacgaggtggtgatggccgcgtacgcg240
cacgagctgcctgcgttcggcattgagcacggcctgacaaactacgctgctgcgtacgcg300
actggtctgctgctggcgcgccgcacgctggcgaagctgggcatcgcggacaagttccag360
ggcgcgaaggaggcggacggctcgtactctgctgtgcgcacgaagaaggacgacgagggc420
gacgacgaggagcgcttcccgttcaaggcgatcctggacgtcgggcttgcgcgcacgacg480
accggcgcccgcgtgttcggcgtgctgaagggcgcggtggacggcggtatggctgtgccg540
caccgccccaaccgcttccccggctacaacaaggagaagagctcgctggacgcgaaggtg600
caccgcgaccgcatctttggcaagcacgtggcggagtacctgaagcaggtgaaggaggag660
gcgagctcgaaccccgacgagaagtgcgtgcagttctcgaggtacatggccgcgaaggtt720
ttgccggagagcatcgagggcatgtacaagaaggcgcacgcggcgatccgcgcggacccg780
tcgaagtcgctgccgaagaaggcgaagaaggagggcgtcgcgcacaagagctacaagacg840
aagaagctgagcggcgcggagaagagggccgccgcgaaggcgaaggtcgcggccatccgc900
gagcgcctcggcaagtaa918
<210>54
<211>305
<212>prt
<213>leishmaniamexicana
<400>54
metprophevallysvalvallysasnlysalatyrphelysargphe
151015
glnvallystyrargargargarggluglylysthrasptyrhisala
202530
argargglnmetvalleuglnasplysthrlyspheglyserprolys
354045
tyrargleuvalvalargilethrasnlysaspileilealaglnile
505560
valglnalalysilevalglyaspgluvalvalmetalaalatyrala
65707580
hisgluleuproalapheglyilegluhisglyleuthrasntyrala
859095
alaalatyralathrglyleuleuleualaargargthrleualalys
100105110
leuglyilealaasplyspheglnglyalalysglualaaspglyser
115120125
tyrseralavalargthrlyslysaspaspgluglyaspaspgluglu
130135140
argpheprophelysalaileleuaspvalglyleualaargthrthr
145150155160
thrglyalaargvalpheglyvalleulysglyalavalaspglygly
165170175
metalavalprohisargproasnargpheproglytyrasnlysglu
180185190
lysserserleuaspalalysvalhisargaspargilepheglylys
195200205
hisvalalaglutyrleulysglnvallysgluglualaserserasn
210215220
proaspglulyscysvalglnpheserlystyrmetalaalalysval
225230235240
leuprogluserilegluglymettyrlyslysalahisalaalaile
245250255
argalaaspproserlysserleuprolyslysalalyslysgluser
260265270
valalahislyssertyrlysthrlyslysleuserglyalaglulys
275280285
argalaalaalalysalalysvalalaalailearggluargleugly
290295300
lys
305
<210>55
<211>918
<212>dna
<213>leishmaniamexicana
<400>55
atgccgttcgtcaaggtcgtgaagaacaaggcgtacttcaagcgctttcaggtgaagtac60
cgccgtcgccgcgagggcaagacggactaccacgcgcgccgccagatggtgctgcaggac120
aagacgaagtttggctcgcccaagtaccgccttgttgtgcgcatcacgaacaaggacatc180
attgcgcagatcgtgcaggcgaagatcgtcggcgacgaggtggtgatggccgcgtacgcg240
cacgagctgcctgcgttcgggattgagcacggcctgacaaactacgccgctgcgtacgcg300
actgggctgctgctggcgcgccgcacgctggcgaagctgggcatcgcggacaagttccag360
ggcgcgaaggaggcggacggctcgtactctgctgtgcgcacgaagaaggacgacgagggc420
gacgacgaggagcgcttcccgttcaaggcgatcctggacgtcggcctcgcgcgcacgacg480
accggtgcccgcgtgttcggcgtgctgaagggcgcggtggacggcggtatggctgtgccg540
caccgccccaaccgcttccccggctacaacaaggagaagagctcactggacgcgaaggtg600
caccgcgaccgcatctttggcaagcacgttgcggagtacctgaagcaggtgaaggaggag660
gcgagctcgaaccccgacgagaagtgcgtgcagttctcgaagtacatggccgcgaaggtt720
ttgccggagagcatcgagggcatgtacaagaaggcgcacgcggcgatccgcgcggacccg780
tcgaagtcgctgccgaagaaggcgaagaaggagagcgtcgcgcacaagagctacaagacg840
aagaagctgagcggcgcggagaagagggccgccgcgaaggcgaaggtcgcggccatccgc900
gagcgcctcggcaagtaa918
<210>56
<211>305
<212>prt
<213>leishmaniabraziliensis
<400>56
metprophevallysvalvallysasnlysalatyrphelysargphe
151015
glnvallystyrargargargarggluglylysthrasptyrhisala
202530
argargglnmetvalleuglnasplysthrlyspheglyserprolys
354045
tyrargleuvalvalargthrthrasnlysaspileilealaglnile
505560
valglnalalysilealaglyaspgluvalleumetalaalatyrala
65707580
hisgluleuproalapheglyilegluhisglyleuthrasntyrala
859095
alaalatyralathrglyleuleuleualaargargthrleualalys
100105110
leuglyilealaasplyspheglnglyalalysglualaaspglyser
115120125
tyrseralavalargthrlyslysaspaspglnglyaspaspgluala
130135140
argpheprophelysalaileleuaspvalglyleualaargthrthr
145150155160
thrglyalaargvalpheglyvalleulysglyalavalaspglygly
165170175
ileservalprohisargproasnargpheproglytyrserlysglu
180185190
lysseralaleuaspalalysvalhisargaspargilepheglylys
195200205
hisvalalaglutyrleulysglnvallysgluglualaserserasn
210215220
proaspglulyscysvalglnpheserlystyrmetglualalysval
225230235240
alaprogluserileglucysmettyrlyslysalahisalaalaile
245250255
argalaaspproserlysserleuprolyslysalalyslysglugly
260265270
alalyshislyssertyrlysthrlyslysmetserglyalaglulys
275280285
argalaalaalalysalalysvalalaalailearggluargleugly
290295300
lys
305
<210>57
<211>29
<212>dna
<213>artificial
<220>
<223>primer
<400>57
cgggatccatgtctcactgcaagttcgag29
<210>58
<211>30
<212>dna
<213>artificial
<220>
<223>primer
<400>58
gcgatatctcccttcttcgcggcctttgcc30
<210>59
<211>32
<212>dna
<213>artificial
<220>
<223>primer
<400>59
gcgatatcgggatggccaagaagcacctcaag32
<210>60
<211>28
<212>dna
<213>artificial
<220>
<223>primer
<400>60
cggaattctcccttgcgggccctgcggg28
<210>61
<211>32
<212>dna
<213>artificial
<220>
<223>primer
<400>61
cggaattcgggatgaagctcaacatcgcgtac32
<210>62
<211>32
<212>dna
<213>artificial
<220>
<223>primer
<400>62
gcgatatctcccttcttctggaatgctgccac32
<210>63
<211>31
<212>dna
<213>artificial
<220>
<223>primer
<400>63
gcgatatcgggatgtgcacgctggcaaattg31
<210>64
<211>34
<212>dna
<213>artificial
<220>
<223>primer
<400>64
ggggtaccggatccttacttgccgaggcgctcgc34
<210>65
<211>918
<212>dna
<213>leishmaniabraziliensis
<400>65
atgccgttcgtcaaggtcgtgaagaacaaggcgtacttcaagcgcttccaggtgaagtac60
cgccgtcgccgcgagggcaagacggactaccacgcacgccgccagatggtgctgcaggac120
aagacgaagtttggctcacccaagtaccgccttgttgtgcgcacgacgaacaaggacatc180
attgcgcagatcgtgcaggcgaagatcgccggcgacgaggtgctgatggctgcgtacgcg240
cacgagctgcctgcgttcgggattgagcacggcctgacgaactacgctgcggcgtacgcg300
acgggcctgctgctggcgcgccgcacgctggcgaagttgggcatcgcggacaagttccag360
ggcgcgaaggaggcggacggctcgtactctgctgtgcgcacgaagaaggacgaccagggc420
gacgacgaggcgcgcttcccgttcaaggcgatcctggacgttggtcttgcgcgcacgacg480
acgggtgcccgcgtgttcggcgtgctgaagggcgctgtggacggcggcatctcggtgccg540
caccgccccaaccgcttccccggctacagcaaggagaagagcgccctggacgcgaaggtg600
caccgtgaccgcatcttcggcaagcacgttgcggagtacctgaagcaggtgaaggaggag660
gcgagctcgaaccctgacgagaagtgcgtgcagttctcgaagtacatggaggcgaaggtt720
gcgccagagagcatcgagtgcatgtacaagaaggcacacgcggcgatccgcgcggacccg780
tcgaagtcgctgccgaagaaggcgaagaaggagggcgccaagcacaagagctacaagacg840
aagaagatgagcggcgcggagaagagggccgccgcgaaggcgaaggtcgccgccattcgc900
gagcgccttggcaagtga918
<210>66
<211>3846
<212>dna
<213>artificial
<220>
<223>chimericdnaencodingl3-s4-s6-l5
<400>66
atgtctcactgcaagttcgagcacccccgccacggccatctcggcttcctgccgcgcaag60
cgctcgcgccagatccgcggccgtgcgcgcgcgttccccaaggacgacgcgacgcagaag120
ccccacctgacgagcttcatggtgttcaaggccggtatgacgcacattgtgcgtgatgtc180
gatcgccctggatcgaaggtgaacaagaaggaagtggtggagccggtgacgatcctggag240
gcgccgccgatggtgattgtcggcattgtgggctaccgccaaacgccggttggcctgaag300
acgatcggcaccgtgtgggcgcaccacacgagcgtcgagttccgccgccgctactacaag360
aactggaagcagtctgcgcaactggccttctcccgccagaagcagtttgcgaacacgaag420
gagggcaaggtcgccgaggcgcgcacgctgaacgcgttcgcgaagaaggcgtccgtcatc480
cgcgtgatcgcgcacacgcagctgcgcaagcttcgcaaccaccgcgtgggcgtgaagaag540
gcgcacgtgcaggagatccaggtcaacggcggcagcgttgcggcgaagatcgcgctggcc600
aagtccctgctggagaaggaggtgcgcgtcgactccgtgttccagcagtccgaggcgtgc660
gacgtgtgctccgtcacgaaaggccacggtacggagggcgtggtgaagcgctggggcgtt720
gcctgcctgccacgcaagacgcaccgcggtctgcgcaaggttgcgtgcatcggcgcgtgg780
caccctgcccgcgtcatgtacactgtcgcgcgcgccggtcagcacggttaccaccaccgc840
acgcagctgaacaagaagatctaccagatcggccgctccgttgctgtggagccgaaccag900
gcgacgacgacctacgatctgacagccaagacgatcacgcccatgggtggcttcgtcggc960
tacggtacggtgcgcaacgactacgtgatgctgaagggctccgtgtctggcccgcgccgc1020
cgtgtgatgacgctgcgccgcccgatggcgccgcagacgtcgcgccagctgaaggagaag1080
atcgtgctgaagttcatcgacacgagctcgaagatcggccacggccgcttccagacgaag1140
aaggagaagaaccagtggttcggcccgctcaagaaggaccgcatccgccgcgaggagcgc1200
ctgcgcaaggagcgcgctgcccgcgccgtggagcgcaaggcaaaggccgcgaagaaggga1260
gatatcgggatggccaagaagcacctcaagcgcttgtatgcgcccaaggactggatgctg1320
agcaagctgaccggcgtgttcgcgccgcgtccgcgtccgggtccgcacaagctgcgcgag1380
tgcctgccgctgctggtgatcatccgcaaccggctgaagtacgcgctgaacgcgcgcgag1440
ggtgagatgatcctgcgccagggtctggtgcacgtggacaaccacccgcgccgcgacggc1500
aagtatcccgccggtttcatggacgtggtcgagatcccgaagacgggcgaccgcttccgc1560
ctgatgtacgacgtcaagggccgcttcgcgttggtgaacctgtccgaggcggaggcgcag1620
atcaagctgatgaaggttgtgaacctgtacacggccaccggccgcgtgccggtcgctgtg1680
acgcacgacggccaccgcatccgctacccggacccgcacacctccattggtgacaccatc1740
gtgtacaacgtcaaggagaagaagtgcgtggacctgatcaagaaccgccagggcaaggcc1800
gtgatcgtgaccggtggcgccaaccgcggccgcatcggcgagatcgtgaaggtggagtgc1860
caccccggtgcgttcaacattgcgcacctgaaggacgcgtccggcgccgagttcgccacc1920
cgcgccgcgaacatcttcgtgatcggcaaggacctgaacaacctgcaggtaacggtgccg1980
aagcagcagggcctgcgcatgaacgtgatccaggagcgcgaggagcgcctgatcgcggcg2040
gaggcccgcaagaacgcgccggctcgtggtgcccgcagggcccgcaagggagaattcggg2100
atgaagctcaacatcgcgtacccccgcaacgggacggtgaagcagttcgagatctcggac2160
gaggtgctccgccgcgtgcagctgcaggactaccgcctcggcaacgaggtggacggcgcc2220
atctttggtagcgagttcaagggctacatcttccgcctgcgcggtggctcggacaaggat2280
ggtttcccgatggtccctggcgtgcttgcctccagccgtgtgtcgctgctggtgaagcgc2340
ggtgcgatcggcttcaacaccttccgcggctaccagggtgagcgccgccgcaagaacgtt2400
cgcggctgcgtgctcgcgagcgacattgcgctggtgaacgtgaccatctccaaggtcggt2460
gaccagccgatcgagggtgtgacggacaccacggctccccgccgtctgggtccgaagcgc2520
gcgagcaagatccgcaagctcttcaacctgtcccgcaccgaagacgtgcggaagtacgtt2580
gttcgccgccgcgtcgtgaagagcggcaagaaggaccggctgaaggccccgaagatccag2640
cgtctgatcacgccgagggtcaaggcccgccgtgccaagaaggccaaggacgccatcgcc2700
aaggtgcgcgcgtctgccgctgagcgccgtgagtacctgcgccttatcgcctcgaaccgc2760
cgtgcgctgcgccagcgtgaccactccaagaagcacacccggaaggtgcacgcccagcgc2820
gctgaggtggcagcattccagaagaagggagatatcgggatgtgcacgctggcaaattgg2880
gtacgcgctatcatcaagaaacactcaacactcgcccacacactcgagatgccgttcgtc2940
aaggtcgtgaagaacaaggcgtacttcaagcgcttccaggtgaagtaccgccgtcgccgc3000
gagggcaagacggactaccacgcgcgccggcagatggtgctgcaggacaagacgaagttc3060
ggctcgcccaagtaccgccttgttgtgcgcatcacgaacaaggacatcattgcgcagatc3120
gtgcaggcgaagatcgtcggcgacgaggtggtgatggccgcgtacgcgcacgagctgcct3180
gcgttcggcattgagcacggcctgacaaactacgctgctgcgtacgcgactggtctgctg3240
ctggcgcgccgcacgctggcgaagctgggcatcgcggacaagttccagggcgcgaaggag3300
gcggacggctcgtactctgctgtgcgcacgaagaaggacgacgagggcgacgacgaggag3360
cgctttccgttcaaggcgatcctggacgtcggccttgcgcgcacgacgaccggcgcccgc3420
gtgttcggcgtgctgaagggcgcggtggacggcggtatggctgtgccgcaccgccccaac3480
cgcttccccggctacaacaaggagaagagctcgctggacgcgaaggtgcaccgcgaccgc3540
atctttggcaagcacgtggcggactacctgaagcaggtgaaggaggaggcgagctcgaac3600
cctgacgagaagtgcgtgcagttctcgaagtacatggccgcgaaggttttgccggagagc3660
atcgagggcatgtacaagaaggcgcacgcggcgatccgcgcggacccgtcgaagtcgctg3720
ccgaagaaggcgaagaaggagggcgtcgcgcacaagagctacaagacgaagaagctgagc3780
ggcgcggagaagagggccgccgcgaaggcgaaggtcgcggccatccgcgagcgcctcggc3840
aagtaa3846
<210>67
<211>1281
<212>prt
<213>artificial
<220>
<223>chimericproteinl3-s4-s6-l5
<400>67
metserhiscyslysphegluhisproarghisglyhisleuglyphe
151015
leuproarglysargserargglnileargglyargalaargalaphe
202530
prolysaspaspalathrglnlysprohisleuthrserphemetval
354045
phelysalaglymetthrhisilevalargaspvalaspargprogly
505560
serlysvalasnlyslysgluvalvalgluprovalthrileleuglu
65707580
alaproprometvalilevalglyilevalglytyrargglnthrpro
859095
valglyleulysthrileglythrvaltrpalahishisthrserval
100105110
glupheargargargtyrtyrlysasntrplysglnseralaglnleu
115120125
alapheserargglnlysglnphealaasnthrlysgluglylysval
130135140
alaglualaargthrleuasnalaphealalyslysalaservalile
145150155160
argvalilealahisthrglnleuarglysleuargasnhisargval
165170175
glyvallyslysalahisvalglngluileglnvalasnglyglyser
180185190
valalaalalysilealaleualalysserleuleuglulysgluval
195200205
argvalaspservalpheglnglnserglualacysaspvalcysser
210215220
valthrlysglyhisglythrgluglyvalvallysargtrpglyval
225230235240
alacysleuproarglysthrhisargglyleuarglysvalalacys
245250255
ileglyalatrphisproalaargvalmettyrthrvalalaargala
260265270
glyglnhisglytyrhishisargthrglnleuasnlyslysiletyr
275280285
glnileglyargservalalavalgluproasnglnalathrthrthr
290295300
tyraspleuthralalysthrilethrprometglyglyphevalgly
305310315320
tyrglythrvalargasnasptyrvalmetleulysglyservalser
325330335
glyproargargargvalmetthrleuargargprometalaprogln
340345350
thrserargglnleulysglulysilevalleulyspheileaspthr
355360365
serserlysileglyhisglyargpheglnthrlyslysglulysasn
370375380
glntrppheglyproleulyslysaspargileargargglugluarg
385390395400
leuarglysgluargalaalaargalavalgluarglysalalysala
405410415
alalyslysglyaspileglymetalalyslyshisleulysargleu
420425430
tyralaprolysasptrpmetleuserlysleuthrglyvalpheala
435440445
proargproargproglyprohislysleuargglucysleuproleu
450455460
leuvalileileargasnargleulystyralaleuasnalaargglu
465470475480
glyglumetileleuargglnglyleuvalhisvalaspasnhispro
485490495
argargaspglylystyrproalaglyphemetaspvalvalgluile
500505510
prolysthrglyaspargpheargleumettyraspvallysglyarg
515520525
phealaleuvalasnleuserglualaglualaglnilelysleumet
530535540
lysvalvalasnleutyrthralathrglyargvalprovalalaval
545550555560
thrhisaspglyhisargileargtyrproaspprohisthrserile
565570575
glyaspthrilevaltyrasnvallysglulyslyscysvalaspleu
580585590
ilelysasnargglnglylysalavalilevalthrglyglyalaasn
595600605
argglyargileglygluilevallysvalglucyshisproglyala
610615620
pheasnilealahisleulysaspalaserglyalagluphealathr
625630635640
argalaalaasnilephevalileglylysaspleuasnasnleugln
645650655
valthrvalprolysglnglnglyleuargmetasnvalileglnglu
660665670
argglugluargleuilealaalaglualaarglysasnalaproala
675680685
argglyalaargargalaarglysglyglupheglymetlysleuasn
690695700
ilealatyrproargasnglythrvallysglnphegluileserasp
705710715720
gluvalleuargargvalglnleuglnasptyrargleuglyasnglu
725730735
valaspglyalailepheglysergluphelysglytyrilephearg
740745750
leuargglyglyserasplysaspglypheprometvalproglyval
755760765
leualaserserargvalserleuleuvallysargglyalailegly
770775780
pheasnthrpheargglytyrglnglygluargargarglysasnval
785790795800
argglycysvalleualaseraspilealaleuvalasnvalthrile
805810815
serlysvalglyaspglnproilegluglyvalthraspthrthrala
820825830
proargargleuglyprolysargalaserlysilearglysleuphe
835840845
asnleuserargthrgluaspvalarglystyrvalvalargargarg
850855860
valvallysserglylyslysaspargleulysalaprolysilegln
865870875880
argleuilethrproargvallysalaargargalalyslysalalys
885890895
aspalailealalysvalargalaseralaalagluargargglutyr
900905910
leuargleuilealaserasnargargalaleuargglnargasphis
915920925
serlyslyshisthrarglysvalhisalaglnargalagluvalala
930935940
alapheglnlyslysglyaspileglymetcysthrleualaasntrp
945950955960
valargalaileilelyslyshisserthrleualahisthrleuglu
965970975
metprophevallysvalvallysasnlysalatyrphelysargphe
980985990
glnvallystyrargargargarggluglylysthrasptyrhisala
99510001005
argargglnmetvalleuglnasplysthrlyspheglyserpro
101010151020
lystyrargleuvalvalargilethrasnlysaspileileala
102510301035
glnilevalglnalalysilevalglyaspgluvalvalmetala
104010451050
alatyralahisgluleuproalapheglyilegluhisglyleu
105510601065
thrasntyralaalaalatyralathrglyleuleuleualaarg
107010751080
argthrleualalysleuglyilealaasplyspheglnglyala
108510901095
lysglualaaspglysertyrseralavalargthrlyslysasp
110011051110
aspgluglyaspaspglugluargpheprophelysalaileleu
111511201125
aspvalglyleualaargthrthrthrglyalaargvalphegly
113011351140
valleulysglyalavalaspglyglymetalavalprohisarg
114511501155
proasnargpheproglytyrasnlysglulysserserleuasp
116011651170
alalysvalhisargaspargilepheglylyshisvalalaasp
117511801185
tyrleulysglnvallysgluglualaserserasnproaspglu
119011951200
lyscysvalglnpheserlystyrmetalaalalysvalleupro
120512101215
gluserilegluglymettyrlyslysalahisalaalailearg
122012251230
alaaspproserlysserleuprolyslysalalyslysglugly
123512401245
valalahislyssertyrlysthrlyslysleuserglyalaglu
125012551260
lysargalaalaalalysalalysvalalaalailearggluarg
126512701275
leuglylys
1280
<210>68
<211>399
<212>dna
<213>leishmaniainfantum
<400>68
atggctactcctcgcagcgccaagaaggccgtccgcaagagcggctccaagtccgcgaaa60
tgtggtctgatcttcccggtgggccgcgtcggcgggatgatgcgccgcggccagtacgct120
cgccgcatcggtgcctctggcgccgtgtacctggccgccgtgctggagtacctgacggcg180
gagctgctggagctgtccgtgaaggcggccgcgcagagcgggaagaagcggtgccgcctg240
aacccgcgcaccgtgatgctggccgcgcgccacgacgacgacatcggcacgcttctgaag300
aacgtgaccttgtctcacagcggcgttgtgccgaacatcagcaaggcgatggcaaagaag360
aagggcggcaagaagggcaaggcgacaccgagcgcgtaa399
<210>69
<211>336
<212>dna
<213>leishmaniainfantum
<400>69
atggcctcttctcgctctgctccccgcaaggcttcccacgcgcacaagtcgcaccgcaag60
ccgaagcgctcgtggaacgtgtacgtgggccgctcgctgaaggcgatcaacgcccagatg120
tcgatgtcgcaccgcacgatgagcatcgtgaactcgtacgtgaacgacgtgatggagcgc180
atctgcatggaggccgcgtcgatcgttcgcgcgaacaagaagcgcacgttgggtgcgcgc240
gaggtgcagacggcggtgcgcattgtgctgccggcggagctcgcgaagcacgccatggct300
gagggcacgaaggccgtgtcgagcgcgtcggcttga336
<210>70
<211>390
<212>dna
<213>leishmaniainfantum
<400>70
atgtcccgcaccaaggagaccgcccgcgcgaagcgcaccatcacgtcgaagaagagcaag60
aaggcgccgagcggggcgtccggcgtgaagaggtcgcatcgccgctggcgcccgggcacc120
tgcgcgatccgcgagatccgcaagttccagaagagtacgagcctgctgatccagtgcgcg180
ccgttccagcgcctggtgcgaggtgtcgagcggcagaaggagggcctgcgcttccagagc240
agcgctatcatggcgctgcaggaggcgacggaggcgtacattgtgtcgctgatggcggac300
acgaacctcgcctgcatccacgcgaagcgcgtgacgatccagccgaaggacatccagctg360
gcgctgcgcctgcgcggtgagcgccactag390
<210>71
<211>303
<212>dna
<213>leishmaniainfantum
<400>71
atggccaagggcaagcgttccactgatgccaagggcagccagaggcgccagaagaaggtg60
ctgcgcgacaacatccgcggcatcactcgcggctgcgtccgccgcatggcgcgccgcggt120
ggcgtgaagcgcatctcgaccgaggtgtacgaagaggtgcgccgtgtgctgaaggcctac180
gtggaggacattgtgcgctgcagcacggcctacaccgagtacgcgcgcaagaagaccgtg240
acggcgtgcgatgttgtgaccgcgctgcgcaagcaaggccacatcctgtacggctacgcg300
taa303
<210>72
<211>336
<212>dna
<213>leishmaniainfantum
<400>72
atgtccaccaagtacctcgccgcgtacgctctggcctccctgagcaaggcgtccccgtct60
caggcggacgtggaggctatctgcaaggccgtccacatcgacgtcgaccaggccaccctc120
gcctttgtgatggagagcgttacgggacgcgacgtggccaccctgatcgcggagggcgcc180
gcgaagatgagcgcgatgccggcggccagctctggtgccgctgctggcgtcactgcttcc240
gctgcgggtgatgcggccccggctgccgccgccgcgaagaaggacgagcccgaggaggag300
gccgacgacgacatgggcttcggtctgttcgactaa336
<210>73
<211>972
<212>dna
<213>leishmaniainfantum
<400>73
atgccgtctatcaccactgccaagcgcgagtacgaggagcgcctcgtcgactgcctgacc60
aagtacagctgcgtgctgttcgtgggcatggacaacgtccgctcgcagcaggtgcacgat120
gtcggccgtgcgctgcgcgcgaaggccgagttcatgatgggcaagaagacgctgcagggc180
aagatcgtggagaagcgcgcgcaagccaaggacgcgagccccgaggcgaagcacttcaac240
gatcagtgtgaggagtacaacctgctgagcggcaacaccggcctcatcttcacgaacaac300
gctgtccaggagatcacgtctgtgcttgacgcgcaccgcgtgaagcgcgcggcgcgtgtc360
ggagcgatttccccgtgtgacgtgattgtcgctgctggcagcaccggcatggagccgacc420
cagacgtccttcttccaggcgctgaacattgcgacgaagattgccaagggtatggtggag480
atcgtgacggagaagaaggtgctgagcgtcggcgacaaggtggacaactcgacggcgacg540
ctgctgcaaaagctgaacatcagcccgttctactaccaggtgaatgtgctgtccgtgtgg600
gaccgcggtgtgctgttcacccgcgaggacctgatgatgacggaggacatggtggagaag660
atgctgatggaaggcctgagcaacgttgcggcgatggcgctgggtgctggcatcccgacg720
tcttcgacgattggcccgatgctggtggacgccttcaagaacctgctggctgtctctgtg780
gcgacctcgtacgagttcgaggagcacaacggcaaggagctgcgcgaggccgcgatcaac840
ggcctgctggccggctcttgctcggctgctgcggagcccgccgctgccgcgccggccgcc900
cctagcgccgctgccaaggaggagccggaggagagcgacgaggacgacttcggcatgggc960
ggtctcttctaa972
- 还没有人留言评论。精彩留言会获得点赞!