基因变异检测方法、装置、终端设备和可读存储介质与流程
本技术涉及数据处理,具体涉及一种基因变异检测方法、装置、终端设备和计算机可读存储介质。
背景技术:
1、基因变异检测,又称为高通量变异检测,其基本原理是对样本dna随机打断后进行高通量测序并获得大量片段reads,再通过生物信息学分析软件将测得的reads比对到参考基因组,得到每一条reads在参考基因组上的位置信息。
2、临床高通量检测的目的是对患者基因发生的变异进行检测,并将变异进行注释及报告解读用于指导临床诊断和治疗,这些变异包括点突变(snv)、插入缺失突变(indel)、结构变异(sv)和拷贝数变化(cnv)等。而主要结构变异(sv)是最重要的一种变异。据统计每个人类基因组都有超过20000个结构变异,基因组结构变异可能导致的疾病已经超过1000种,例如渐冻症、精神分裂症以及癌症等。作为肿瘤基因组的主要遗传标志,结构变异影响肿瘤相关功能基因的表达,并对肿瘤的发生发展有驱动力量。因此,准确检测肿瘤结构变异是诊断和治疗癌症非常重要的一步,也是当前研究的重点。通过基因结构变异推断肿瘤发育,识别肿瘤靶向基因,从而为肿瘤的诊断和治疗提供重要依据。
3、基因组发生结构变异可分为大片段(高于50bp)插入(inversion)、缺失(insertion)、倒位(deletion)、易位(translocation)和重复(duplication)。由于高通量测序产生的是短reads,而结构变异片段普遍较长,因此对于高通量测序数据结构变异的生物信息学分析,一种是利用发生结构变异断点的信息来进行判断,即一条reads的一部分比对到基因组的某一位置,另一部分比对到另一位置,称之为split reads(即覆盖了断裂点的单端reads)。在比对中如果不能完全比对到完整的基因,比对软件则尝试使用更为宽松的局部比对尝试匹配其他的基因,如果比对上则会保留这部分序列并进行cigar值上的标注。这种机制称为soft-clipped,这类reads也在后续所称sc reads。另一种是discordantpaired reads(不一致序列),即pair reads关系异常,一条read比对到其中一个基因,另一条read比对到另一个基因。
4、对比pair reads判断sv的方法,基于split reads方法可以精准到单个碱基,但由于一般情况下reads数更少,同时对实验打断与数据噪声的容错更低,只使用split reads的方法判断sv有时会导致精准度不够与假阳过多等问题。此外,针对以上结构变异类型还有基于de novo组装并测序的方法,对于组装的准确率,质量要求相对较高。
5、现有的sv检测软件主要基于split reads以及discordant pair reads。但由于缺乏“黄金标准”,不同软件的内部过滤条件,检测方法以及各类参数均有差异。现有的针对以上结构变异的不同检测软件可检测的结构变异的类型有差异。虽然以上软件均可以完成结构变异的部分检测,但鲜有一款软件可以保证灵敏度与准确性的完成以上五种结构变异检测,比如lumpy软件主要检测的类型为deletions,duplications,inversions,translocations,对于insertion检测存在检测局限性;manta软件可检测的类型有insertions,deletions,translocations,inversions,duplications五种类型,但该软件经过标准品测试,检测灵敏性偏低,存在检测不稳定性导致漏检问题出现。
技术实现思路
1、有鉴于此,本技术实施例中提供了一种基因变异检测方法、装置、终端设备和计算机可读存储介质,以克服现有技术基因变异检测灵敏度以及准确性差的问题。
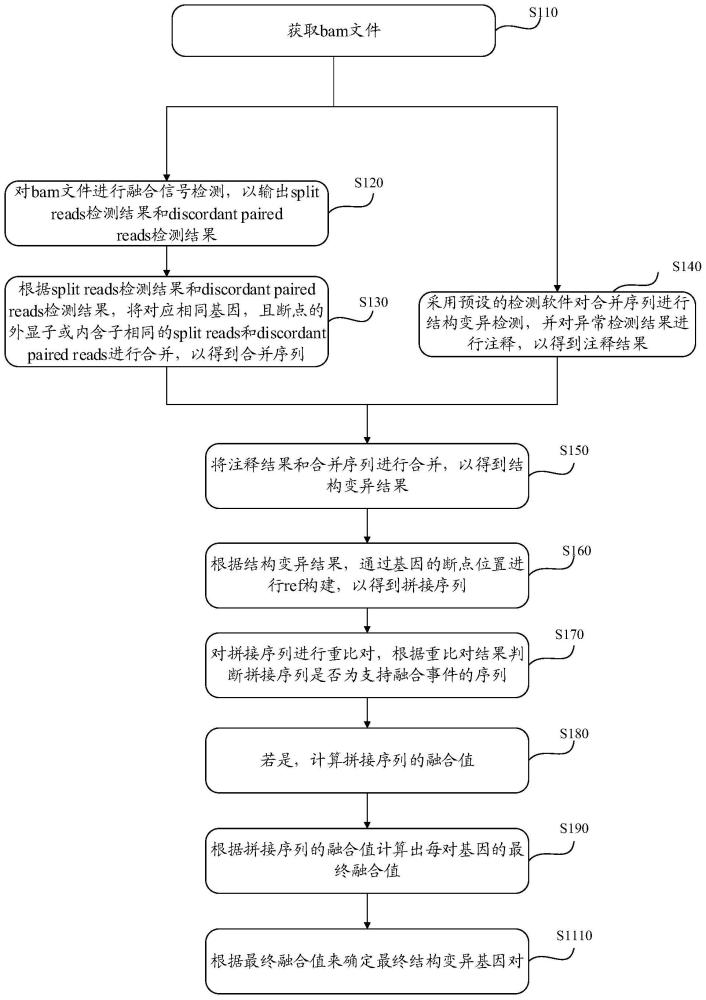
2、第一方面,本技术实施例提供了一种基因变异检测方法,该方法包括:获取bam文件;其中所述bam文件是对测序样本进行高通量测序得到的测序文件,进行参考基因组比对获得的;
3、对bam文件进行融合信号检测,以输出split reads检测结果和discordantpaired reads检测结果;
4、根据所述split reads检测结果和所述discordant paired reads检测结果,将对应相同基因,且断点的外显子或内含子相同的split reads和discordant paired reads进行合并,以得到合并序列;
5、采用预设的检测软件对所述合并序列进行结构变异检测,并对异常检测结果进行注释,以得到注释结果;
6、将所述注释结果和所述合并序列进行合并,以得到结构变异结果;
7、根据所述结构变异结果,通过基因的断点位置进行ref构建,以得到拼接序列;
8、对所述拼接序列进行重比对,根据重比对结果判断所述拼接序列是否为支持融合事件的序列;
9、若是,计算拼接序列的融合值;
10、根据所述拼接序列的融合值计算出每对基因的最终融合值;
11、根据所述最终融合值来确定最终结构变异基因对。
12、第二方面,本技术实施例提供了一种基因变异检测装置,该装置包括:
13、bam文件获取模块,用于获取bam文件;其中所述bam文件是对测序样本进行高通量测序得到的测序文件,进行参考基因组比对获得的;
14、信号检测模块,用于对bam文件进行融合信号检测,以输出split reads检测结果和discordant paired reads检测结果;
15、第一合并模块,用于根据所述split reads检测结果和所述discordant pairedreads检测结果,将对应相同基因,且断点的外显子或内含子相同的split reads和discordant paired reads进行合并,以得到合并序列;
16、变异检测模块,用于采用预设的检测软件对所述bam文件进行结构变异检测,并对变异检测结果进行注释,以得到注释结果;
17、第二合并模块,用于将所述注释结果和所述合并序列进行合并,以得到结构变异结果;
18、ref构建模块,用于根据所述结构变异结果,通过基因的断点位置进行ref构建,以得到拼接序列;
19、判断模块,用于对所述拼接序列进行重比对,根据重比对结果判断所述拼接序列是否为支持融合事件的序列;
20、融合值计算模块,用于当所述拼接序列为支持融合事件的序列时,计算拼接序列的融合值;
21、最终融合值计算模块,用于根据所述拼接序列的融合值计算出每对基因的最终融合值;
22、变异基因对确定模块,用于根据所述最终融合值来确定最终结构变异基因对。
23、第三方面,本技术实施例提供了一种终端设备,包括:存储器;一个或多个处理器,与所述存储器耦接;一个或多个应用程序,其中,一个或多个应用程序被存储在存储器中并被配置为由一个或多个处理器执行,一个或多个应用程序配置用于执行上述第一方面提供的基因变异检测方法。
24、第四方面,本技术实施例提供了一种计算机可读取存储介质,计算机可读取存储介质中存储有程序代码,程序代码可被处理器调用执行上述第一方面提供的基因变异检测方法。
25、本技术实施例提供的基因变异检测方法、装置、终端设备和计算机可读存储介质,获取bam文件;其中bam文件是对测序样本进行高通量测序得到的测序文件,进行参考基因组比对获得的;对bam文件进行融合信号检测,以输出split reads检测结果和discordantpaired reads检测结果;根据split reads检测结果和discordant paired reads检测结果,将对应相同基因,且断点的外显子或内含子相同的split reads和discordant pairedreads进行合并,以得到合并序列;采用预设的检测软件对bam文件进行结构变异检测,并对变异检测结果进行注释,以得到注释结果;将注释结果和合并序列进行合并,以得到结构变异结果;根据结构变异结果,通过基因的断点位置进行ref构建,以得到拼接序列;对拼接序列进行重比对,根据重比对结果判断拼接序列是否为支持融合事件的序列;若是,计算拼接序列的融合值;根据拼接序列的融合值计算出每对基因的最终融合值;根据最终融合值来确定最终结构变异基因对。
26、本技术实施例中的基因变异检测方法,基于split reads和discordant pairedreads,并集合了预设的检测软件,能完成了多种变异类型的结构变异检测,从而实现了dna数据的全面结构变异检测;所有变异类型的检测均保证了灵敏度与准确性。另外,通过序列拼接-重比对的验证方式,增加了最终输出的可信度。
- 还没有人留言评论。精彩留言会获得点赞!