一种识别mRNA非翻译区药物作用靶点的方法、装置及计算机可读存储介质与流程
本发明属于生物信息,具体涉及一种识别mrna非翻译区药物作用靶点的方法、装置及计算机可读存储介质。
背景技术:
1、靶点是新药研发的基础,传统的新药开发靶点一般为蛋白质。然而据统计,在约2万种人类蛋白质中,疾病相关的蛋白质大约只有700种,对许多疾病来说,其相关蛋白是没有小分子药物作用结构的。最新研究表明,除了蛋白质以外,rna也可能是药物开发的可行靶标,特别是mrna的非翻译区。这预示着药物作用靶点的搜索空间大大增加。但是相对于构象稳定的蛋白质靶点来说,mrna的非翻译区在结构上具有很大的不稳定性,这使得制药企业投资开发靶向rna的药物的失败风险较大。此外,rna的功能调控机理非常复杂,即使找到mrna非翻译区上一些区域的稳定构象,也很难确定这些区域具有功能调控作用。如果rna序列不具有功能调控作用,则会导致即使药物小分子能结合上去,也不能起到治疗疾病的作用。
2、因此,在发现候选药物作用靶点后,还必须通过结构生物学方法、组学测序分析、序列标签数据库搜寻等方法对靶点mrna进行功能验证,以确定该靶点是否在疾病发生过程中起关键作用。
3、由于mrna靶点在人体中的作用机制未知,在药物研发的前期阶段,往往需要进行大量的mrna结构和功能的实验探测该靶点是否满足药物作用的条件,如具有稳定的三维空间结构,与药物小分子结合后是否会影响细胞功能。对于成千上成万编码不同功能蛋白的mrna序列,通过实验探测靶点作用机制的筛选方法犹如大海捞针,具有周期长,成本高,靶点功能与预期不符等局限性和风险。
技术实现思路
1、本发明所要解决的技术问题是如何识别mrna非翻译区的药物作用靶点。
2、为了解决上述技术问题,本发明首先提供了预测或识别mrna非翻译区药物作用靶点的方法,所述方法可包括训练预测mrna非翻译区药物作用靶点的模型,并使用所述模型预测待测mrna非翻译区序列是否含有药物作用靶点的步骤。
3、所述模型的训练可包括如下步骤:
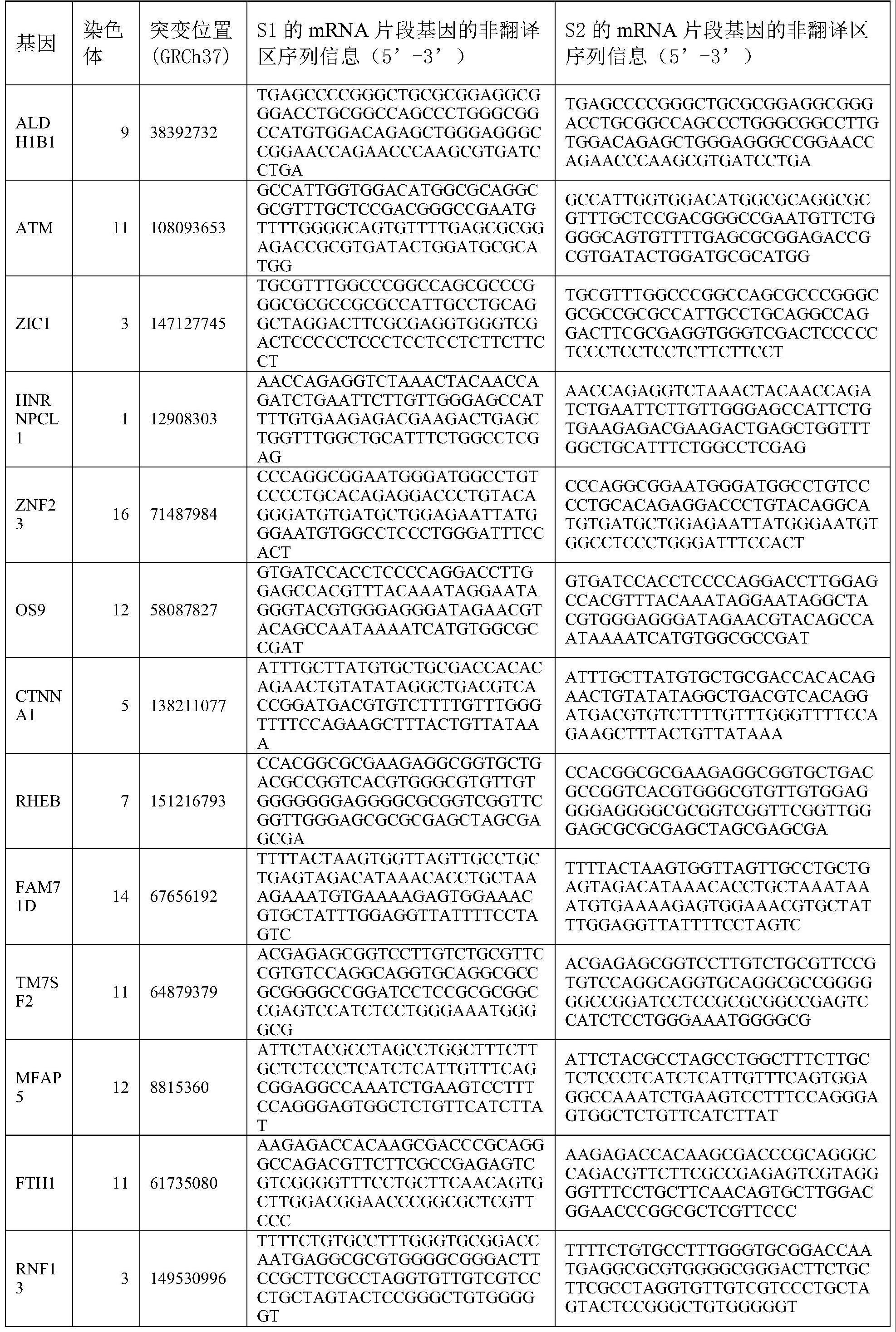
4、a1)数据获取:合成人类基因组中致病基因的包含野生型mrna非翻译区的mrna基因序列得到s1文库,所述s1文库中基因序列编码的mrna为s1数据集;合成人类基因组中致病基因的包含突变mrna非翻译区的mrna序列得到s2文库,所述s2文库中基因序列编码的mrna为s2数据集;分别将所述s1文库和s2文库转化细胞系进行表达分别获得名称为s1-te的s1文库mrna序列编码蛋白的蛋白表达水平数据集和名称为s2-te的s2文库中mrna序列编码蛋白的蛋白表达水平数据集。
5、a2)模型训练:使用卷积层训练所述s1数据集,s1-te和s2数据集,s2-te获得mrna非翻译区靶点序列特征模式;使用周期激活函数s提取所述s1-te和s2-te中mrna非翻译区靶点序列的位置特征值,所述位置特征值含有所述mrna非翻译区靶点序列的周期特征;将所述序列特征模式与所述位置特征值进行融合获得mrna非翻译区靶点序列的融合特征得到所述模型的参数。
6、a3)mrna非翻译区靶点预测:使用所述模型的参数扫描待测mrna非翻译区序列以确定待测mrna非翻译区序列是否含有药物作用靶点。
7、上述方法中使用所述模型预测待测mrna非翻译区序列是否含有药物作用靶点可包括如下步骤:将待测mrna非翻译区序列数据输入所述模型,使用所述模型的参数对待测mrna非翻译区序列进行扫描获得待测mrna非翻译区序列的融合特征评分,根据所述融合特征评分确定待测mrna非翻译区序列是否含有药物作用靶点。
8、上述方法中,所述s1数据集和s2数据集中的mrna非翻译区序列可为经过随机截断操作获得的序列。
9、所述随机截断操作可包括如下步骤:将s1和s2数据集中长度100nt的mrna非翻译区序列进行随机截取获得截取后mrna非翻译区序列。所述截取后mrna非翻译区序列的起始位点位于所述(原始)mrna非翻译区序列5’端的第1-10个核苷酸,所述截取后mrna非翻译区序列的结束位点位于所述(原始)mrna非翻译区序列5’端的第90-100个核苷酸。
10、为了解决上述技术问题,本发明还提供了预测mrna非翻译区药物作用靶点的装置,所述装置可包括模型训练模块和模型预测模块。所述模型训练模块可包括如下模块:
11、b1)数据获取模块:用于获取人类基因组中致病基因的包含野生型mrna非翻译区的mrna基因序列的s1文库和人类基因组中致病基因的包含突变mrna非翻译区的mrna基因序列的s2文库;以及将所述s1文库和s2文库转化细胞系进行表达分别获得s1文库mrna基因序列编码蛋白的蛋白表达水平数据集s1-te和s2文库中mrna基因序列编码蛋白的蛋白表达水平数据集s2-te;所述s1文库中基因序列编码的mrna为s1数据集,所述s2文库中基因序列编码的mrna为s2数据集;
12、b2)模型训练模块:用于使用卷积层训练所述s1数据集、s2数据集、s1-te和s2-te获得mrna非翻译区靶点序列特征模式;使用周期激活函数s提取所述s1-te和s2-te中mrna非翻译区靶点序列的位置特征值;将所述序列特征模式与所述位置特征值进行融合获得mrna非翻译区靶点序列的融合特征得到所述模型的参数;
13、c3)药物作用靶点的评分模块:用于使用所述模型的参数扫描待测mrna非翻译区序列获得待测mrna非翻译区序列的融合特征评分,根据所述融合特征评分确定待测mrna非翻译区序列是否含有药物作用靶点。
14、上述装置中,所述s1数据集和s2数据集中的mrna非翻译区序列可为经过随机截断操作获得的序列。
15、所述随机截断操作可包括如下步骤:将s1和s2数据集中长度100nt的mrna非翻译区序列进行随机截取获得截取后mrna非翻译区序列。所述截取后mrna非翻译区序列的起始位点位于所述(原始)mrna非翻译区序列5’端的第1-10个核苷酸,所述截取后mrna非翻译区序列的结束位点位于所述(原始)mrna非翻译区序列5’端的第90-100个核苷酸。
16、为了解决上述技术问题,本发明还提供了预测mrna非翻译区药物作用靶点的计算机可读存储介质,所述计算机可读存储介质可使计算机运行如上文所述方法的步骤。
17、上文所述方法在开发预防和/或治疗疾病相关药物中的应用也属于本发明的保护范围。
18、上文所述装置和/或上文所述计算机可读存储介质在开发预防和/或治疗疾病相关药物中的应用也属于本发明的保护范围。
19、为了解决上述技术问题,本发明还提供了mrna非翻译区靶点基因序列特征模式在开发预防和/或治疗疾病相关药物中的应用。所述mrna非翻译区靶点基因序列特征模式可为146种dna片段。所述146种dna片段的核苷酸序列分别可为序列表中序列1-序列40、5’-atntaa-3’、5’-ttttctct-3’、5’-tttacaccg-3’、5’-tgatgtgnc-3’、5’-atggnntga-3’、5’-atgnatg-3’、5’-gngggnag-3’、5’-atgatg-3’、5’-ttgcnggt-3’、5’-atntttnt-3’、5’-cnnatggng-3’、5’-tntagtatg-3’、5’-atgnnnatg-3’、5’-tacaac-3’、5’-agtgtna-3’、5’-tgtctgcna-3’、5’-tccgcgcc-3’、5’-atgtagcnt-3’、5’-ctgggttt-3’、5’-atnnnatg-3’、5’-tntctncnc-3’、5’-atgnnntag-3’、5’-atgtnacna-3’、5’-tgatgtgnc-3’、5’-atggnntga-3’、5’-atgnatg-3’、5’-gngggnag-3’、5’-atgatg-3’、5’-ttgcnggt-3’、5’-atntttnt-3’、5’-cnnatggng-3’、5’-tntagtatg-3’、5’-atgnnnatg-3’、5’-tacaac-3’、5’-agtgtna-3’、5’-tgtctgcna-3’、5’-tccgcgcc-3’、5’-atgtagcnt-3’、5’-ctgggttt-3’、5’-atnnnatg-3’、5’-tntctncnc-3’、5’-atgnnntag-3’、5’-atgtnacna-3’、5’-ccnagngatc-3’、5’-agtattgngg-3’、5’-cangatggtg-3’、5’-ctgancactg-3’、5’-gggggggccc-3’、5’-gtnacnatgg-3’、5’-ggtgcnangc-3’、5’-gcnntgacnc-3’、5’-tgnnntggcg-3’、5’-tgttctganc-3’、5’-atgnngnatg-3’、5’-annatgcnag-3’、5’-tatnggatag-3’、5’-gatganntga-3’、5’-gtnacnatgg-3’、5’-ggtgcnangc-3’、5’-gcnntgacnc-3’、5’-tgnnntggcg-3’、5’-tgttctganc-3’、5’-atgnngnatg-3’、5’-annatgcnag-3’、5’-tatnggatag-3’、5’-gatganntga-3’、5’-ggagnggnaga-3’、5’-agcctngatnc-3’、5’-gtacgancnna-3’、5’-atggagcnnat-3’、5’-attgnnnnatg-3’、5’-cnatgtnnncc-3’、5’-cnnnaagtatg-3’、5’-tcnnngncata-3’、5’-antactagnaa-3’、5’-gccctntnatc-3’、5’-tgncnanntaa-3’、5’-cgagtaanncg-3’、5’-ggantgnccna-3’、5’-atgnntncccc-3’、5’-tttnnaggagc-3’、5’-tgggncagnnc-3’、5’-anatgncnngt-3’、5’-ananaaatant-3’、5’-anatgnncccc-3’、5’-atcncaananc-3’、5’-tnttggcnaca-3’、5’-atggagcnnat-3’、5’-attgnnnnatg-3’、5’-tcannatggct-3’、5’-cnatgtnnncc-3’、5’-cnnnaagtatg-3’、5’-tcnnngncata-3’、5’-antactagnaa-3’、5’-gccctntnatc-3’、5’-tgncnanntaa-3’、5’-cgagtaanncg-3’、5’-ggantgnccna-3’、5’-atgnntncccc-3’、5’-tttnnaggagc-3’、5’-tgggncagnnc-3’、5’-anatgncnngt-3’、5’-ananaaatant-3’、5’-anatgnncccc-3’、5’-atcncaananc-3’和5’-tnttggcnaca-3’。
20、检测上述的mrna非翻译区靶点基因序列的装置在开发预防和/或治疗疾病相关药物中的应用也属于本发明的保护范围。
21、所述装置可包括检测所述mrna非翻译区靶点基因序列的模块。
22、为了解决目前靶点发现和验证方法的缺陷,本发明提供了一种通过大量mrna功能表达数据进行数据建模、预测来加速mrna靶点发现的技术方案。
23、本发明的主要目的在于提供一种基于深度学习模型的药物靶点确定方法和装置,提高基于mrna组学确定药物靶点准确性和多样性。其实施方案主要通过体外培养细胞,采集细胞中的mrna翻译水平数据作为数据标签进行模型训练,模型训练的目标是拟合序列对应的翻译水平标签,使得模型具备预测mrna翻译水平的能力。并将训练得到的卷积特征与位置周期特征在mrna非编码序列上进行连续扫描,根据特征扫描得到融合特征评分来选取mrna上的功能靶点区域。本发明方案能缩减mrna靶点发现的周期,并提供更多样性的侯选mrna功能靶点区域。
24、本发明方案总体上是通过记录和分析不同mrna非翻译区对应的功能表达数据,并识别序列突变造成的差异性大小,以确定该序列区域具有功能调控的作用。具体地,本技术方案分为以下步骤进行。
25、一)功能表达数据获取
26、a)合成人类致病基因对应的所有mrna片段3782条,将含有非翻译区基因片段的所有mrna片段转染进k562细胞系中进行体外培养;
27、b)对上述致病基因的mrna片段的非翻译区进行随机突变,将突变后非翻译区mrna片段加上编码区的完整基因片段转染进k562细胞系中进行体外培养;
28、c)对以上k562细胞系进行核糖体测序,得到各种不同的mrna非翻译区序列进行翻译功能调控对应的蛋白编码区结合的核糖体载量,以平均核糖体载量作为翻译效率数值,该数值反应了mrna非翻译区序列对应的编码蛋白表达水平。
29、二)训练深度学习模型进行序列卷积特征和周期特征提取
30、如上图(图1)所示,在本发明中药物靶点识别所采用的深度学习模型是指基于深度卷积滤波器与周期激活函数共同预测mrna功能性靶点的模型。模型的权重参数是基于对一)中所述功能数据的采集、清洗、转换、训练获取。
31、图中特征编码器e有以下重要组成部分:
32、1)序列随机截断操作
33、2)卷积层c
34、3)周期激活函数s
35、与现有技术相比具有的有益效果:
36、相比于传统的rna靶点发现方法,本发明方案具有以下优点:
37、1)高通量:相对于传统的rna靶点功能机制发现需要一个个靶点序列进行细胞实验和测序验证的流程来说,本发明通过学习大量序列-功能配对数据,得到泛化的功能特征编码器权重,可以批量对所有人体mrna非翻译区序列的功能位点进行预测,并将预测得到靶点集合进一步验证,实现了在更短周期内更高通量发现新rna功能靶点。
38、举例如下,本发明使序列特征编码中的某个样式片段如下图(图4)进行rna特征序列激活搜索,在所有人体mrna非翻译区中找到37个符合要求的功能位点。
39、接下来,合成对上述37个功能位点的mrna非翻译区正常及突变序列,进行体外k562细胞系培养(参见三中具体实施方式),获得37对功能表达水平数据样本点。下图为突变引起表达水平变化的箱线图。从图(图5)中可以看出,该功能位点对蛋白翻译水平有显著负向调控作用。
40、相比于传统的rna靶点功能机制发现过程,需要发现、验证新功能靶点需要非常漫长的迭代试验,而对于本发明方案而言,只需搜索-预测-体外细胞培养验证即可快速验证新发现的rna功能靶点。
41、2)可以对任意长度的rna序列进行连续扫描。如果采用经典的序列卷积神经网络的建模方法,其输入要求是一段固定长度的序列截取窗口。而本发明通过提取神经网络中的卷积层特征作为特征滤波器,可以对任意长度的序列进行特征扫描。人类中的rna非翻译区片段长度分布包含几十个碱基到数千个碱基,采用固定长度的序列显然不合适,因此能对全长rna非翻译区域进行连续扫描是本发明的另一个优点。
- 还没有人留言评论。精彩留言会获得点赞!