一种干扰生物学通路的活性结构预测方法
本发明涉及药物开发和环境复合体系中活性结构预测领域,更具体的说涉及一种基于明确生物学通路和化合物联系,预测干扰特定生物学通路的活性结构的方法。
背景技术:
1、核糖核酸(rna)作为遗传讯息中间载体,除了参与蛋白质合成还参与基因表达调控等一系列重要生命过程,二代测序和基因芯片技术发展使得rna转录组数据井喷,极大的促进了医学、生命科学、环境科学等多个领域的发展。现存大量细胞(in vitro)、个体(invivo)在不同化合物(例如药物和污染物)处理下rna转录组数据,例如ctd(comparativetoxicogenomics database)与connectivitymap数据库。通常来说,主要活性结构相似的化合物会导致相似的生物效应或作用机制,因此其转录组学结果也会相应具有一致性。而转录组数据能有效提供多维且平行性良好的的生物特征指纹,因此这给予科学家开发一个类似反向定量构效关系(quantitative structure-activity relationship,qsar)应用的机会,即通过输入rna信息(差异表达基因或生物学通路)预测导致rna变化的潜在活性结构。而获得对特定生物靶点和药物功能有重要贡献的活性结构将有效的促进新药的设计和开发,极大的节约时间和金钱成本。此外,通过转录组结果识别环境混合体系关键毒物的活性结构也将对关键毒物鉴别、优控物质推荐、人体和生态健康风险评估提供支持。但是已有转录组学训练集质量和预测方法不足,严重阻碍了活性结构预测模型的开发和应用。
技术实现思路
1、1.要解决的问题
2、针对现有rna信息和活性结构之间联系不足的问题,本发明的目的是提供一种干扰生物学通路的活性结构预测方法。
3、2.技术方案
4、为了解决上述问题,本发明所采用的技术方案如下:
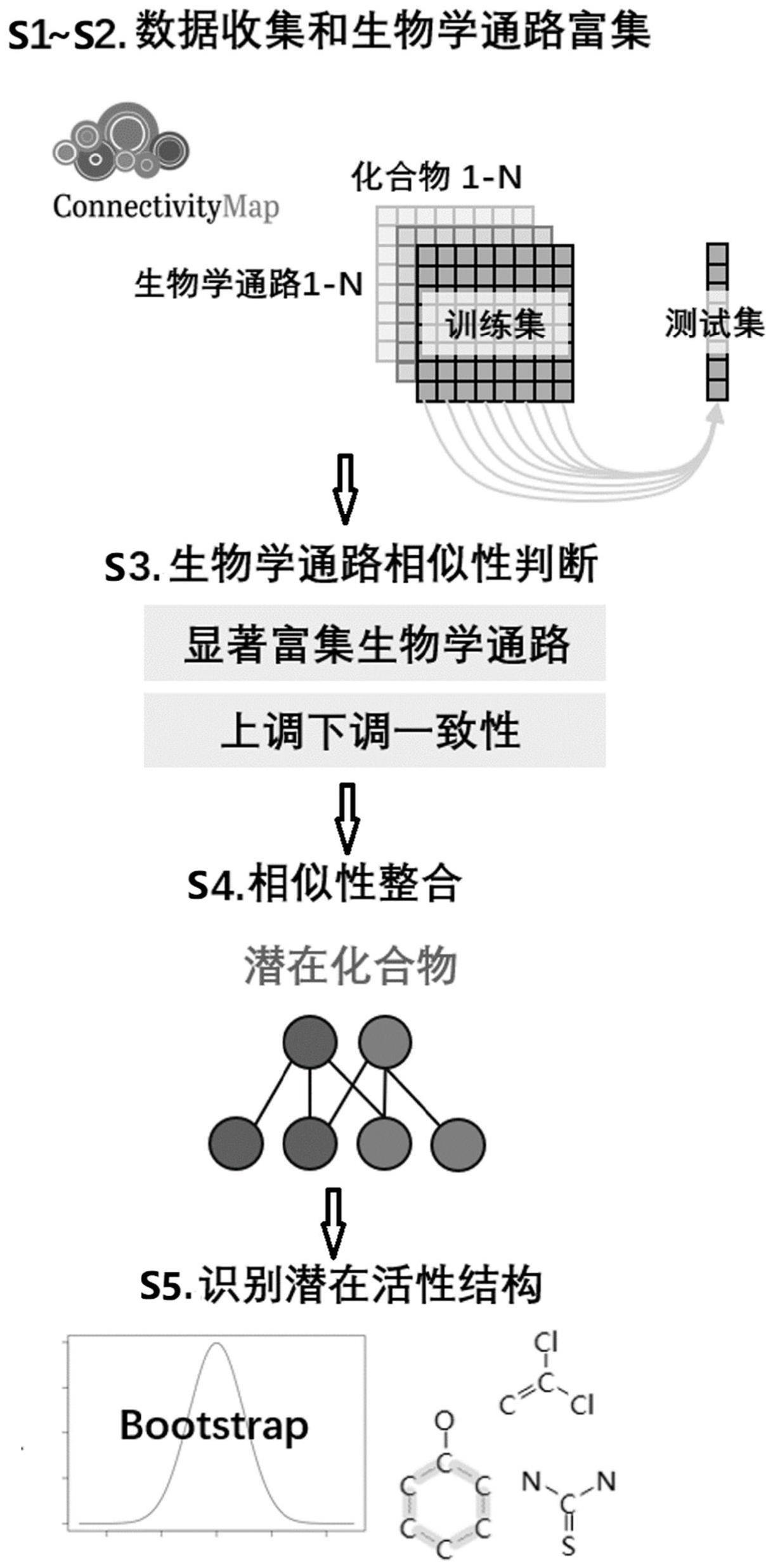
5、一种干扰生物学通路的活性结构预测方法,包括以下步骤:
6、1)数据的收集:
7、收集人种转录组基因表达数据;
8、把基因名称转换为entrze id;
9、对数据进行生物标签的划分;所述生物标签包括细胞系、暴露时间、暴露浓度、数据质量;
10、对数据进行化学标签的划分;所述化学标签包括smiles描述的分子结构和toxprint描述的分子结构;
11、其中,对于收集得到的人种转录组基因表达数据,按需要基于基因表达强度和数据平行性去除低质量数据,得到保留数据;原则上,对于connectivitymap使用的基因芯片技术,采用qc_pass等于1,tas值大于1.5的筛选标准;
12、对于目前转录组更常用的二代测序技术,使用fastqc软件进行测序质量质控,筛选标准主要包括序列测序质量统计10%分位数大于20,每条序列的测序质量统计峰值大于20,碱基分布中g/c比例小于20%,序列平均gc含量分布小于30%,序列的n含量小于20%,重复序列占总数小于50%;
13、2)生物学通路富集:
14、将步骤1)中收集并保留的转录组基因表达数据划分为训练集和测试集;其中,所述测试集的数据量占比不低于10%;
15、对步骤1)中收集并保留的转录组基因表达数据,使用基因集富集分析对生物学通路进行富集,所述基因集富集分析得到的p gsea值和nes值;
16、区分出显著富集的生物学通路;
17、3)生物学通路相似性判断:
18、通过累计超几何分布,判断训练集和测试集之间显著富集的生物学通路的交叉程度;
19、通过累计伯努利分布,判断训练集和测试集之间均显著富集生物学通路调控趋势的一致性;
20、4)相似性结果的整合:
21、对累计超几何分布的phypergeometric值进行错误发现率的校正,得到pfrd1值;
22、对累计伯努利分布的pbernoulli值进行错误发现率的校正,得到pfrd2值;
23、若pfrd1值、pfrd2值均满足小于0.05,则认为训练集数据与测试集数据在生物学通路干扰上相似;
24、基于训练集中生物学通路和化合物的联系进一步识别到一批导致测试集生物学通路干扰的潜在化合物;
25、5)活性结构计算:
26、按潜在化合物数量,通过自举法从训练集中获取数据样本,计算潜在化合物每个分子描述符在高斯分布中的累积概率pdistribution,pdistribution值满足小于0.05的分子描述符所描述的结构,即被认为是测试集中干扰特定生物学通路的活性结构。
27、进一步地,步骤2)中,通过entrze id和基因相对表达强度进行生物学通路的富集。
28、进一步地,步骤2)中,所述通路富集条件包括:
29、选择生物学通路数据库,每条通路所富集的基因个数为4~1000,物种设置为人种类;
30、其中,所述生物学通路数据库包括kegg、reactome和wikipathway中的任意一个或多个。
31、进一步地,基因集富集分析所得的p gsea值小于0.05的生物学通路,则被认为是显著富集;
32、所述p gsea值作为生物学通路干扰的特征进行使用。
33、进一步地,所述生物学通路的整体表现包括上调和下调;
34、所述基因集富集分析所得的nes正值表示上调;
35、所述基因集富集分析所得的nes负值表示下调。
36、进一步地,步骤3)中,所述累计超几何分布结果用phypergeometric值表示;
37、其中,累计超几何分布的公式如下:
38、
39、n为背景通路数量;
40、n为测试集显著富集到的生物学通路数量,即pgsea值<0.05;
41、m为训练集和测试集共同显著富集到的通路数量;
42、m为训练集显著富集到的通路数量。
43、进一步地,所述累计伯努利分布结果用pbernoulli值表示;
44、pbernoulli值越小表明训练集和测试集均显著富集的生物学通路调控趋势的一致性越高,进而说明两者在生物学通路干扰上越相似;
45、其中,累计伯努利的公式如下:
46、
47、m为测试集和训练集共同显著富集到的通路数量;
48、k为训练集和测试集均显著富集到的生物学通路中调控趋势一致的通路数量;
49、p参数设置为0.5。
50、进一步地,步骤4)中,基于训练集中生物学通路和化合物的联系进一步识别到一批导致测试集生物学通路干扰的潜在化合物,使用少数服从多数的原则对于同一化合物、同一浓度的多条记录进行相似性判断,若判断相似和不相似的数量一致也被认为是不相似;而对于同一化合物、不同浓度的记录差异,保留最相似的结果。
51、进一步地,步骤5)中,按潜在化合物的数量,通过自举法从训练集放回、不重复抽样,计算每次抽样中各分子描述符出现次数并拟合为高斯分布;
52、其中,所述抽样次数不低于1000次;
53、统计潜在化合物中各分子描述符出现次数并计算各分子描述符在高斯分布中的累积概率p;
54、去除掉在抽样中标准差小于1的分子描述符后,累计概率pdistribution值小于0.05的分子描述符所代表的结构即被认为造成测试集生物学通路干扰的活性结构。
55、进一步地,步骤5)中,使用r语言pnorm函数拟合为高斯分布。
56、3.有益效果
57、相比于现有技术,本发明的有益效果为:
58、(1)本发明的一种干扰特定生物学通路的活性结构预测方法,以已有转录组数据作为媒介,基于训练集和测试集生物学通路在交叉程度和调控趋势的一致性,通过已知结构化合物(活性结构)预测未知的活性结构,在新型药物开发、环境复合体系关键毒物鉴别等领域具有极大应用价值。
59、(2)本发明的一种干扰特定生物学通路的活性结构预测方法,相较于目前化合物和基因的联系,通过gsea生物学通路富集在不损失信息量的前提下整合了rna变化趋势和强度,构建了更为可信和有效的训练集。此外,本方法明确设置了暴露浓度、暴露时间、细胞系的标签,通过对数据的科学筛选进一步增强了数据质量。
60、(3)本发明的一种干扰特定生物学通路的活性结构预测方法,利用累计超几何分布和累计伯努利分布分别评估训练集和测试集之间在生物学通路交叉程度和上调下调趋势的一致性,通过训练集数据明确的化学标签,进而从两个不同的维度有效识别到了一批可能造成测试集生物学通路干扰的潜在化合物。该方法充分的考虑了转录组所反映的特征和信息,填补了基于生物学通路进行活性结构预测的空白。
61、(4)本发明的一种干扰特定生物学通路的活性结构预测方法,使用自举法抽样获得训练集中各分子描述符背景分布,通过评估曲线下累积概率的方式识别活性结构。本方法将获得的潜在化合物集合设置为一个小概率且独特的事件而不是与背景的差异判断,因此相较于显著性检验判断活性结构的方法大大降低了假阳性的概率,提升了活性结构的预测结果。
- 还没有人留言评论。精彩留言会获得点赞!