基于深度搜索的生物逆合成预测方法、装置和电子设备与流程
本发明涉及生物逆合成,尤其涉及基于深度搜索的生物逆合成预测方法、装置和电子设备。
背景技术:
1、天然产物(natural products)是临床药物开发的主要来源,1981-2020这三十年间获批上市的小分子药物中超过60%是天然产物及其衍生物或类似物。由于从自然界直接提取的方法往往会对生物多样性造成破坏,而天然产物结构中多样的环体系和手性中心导致其化学合成难度较大,因此,异源生物合成是获得复杂天然产物的重要手段。解析天然产物的生物合成途径是实现异源生物合成中最基础的一步,然而现有生物代谢反应数据库中仅涉及到约3万个天然产物,远小于目前已知的天然产物数目。大量天然产物的生物合成途径目前仍然未知,这极大地限制了复杂天然产物的生物合成。
2、自从corey在1960年第一次提出逆合成分析后,该方法成为了设计有机合成路线的重要工具,也是有机合成路线设计的最简单、最基础的方法,帮助化学家对于一些复杂分子完成目标分子的逐步拆解,推导至更为简单、更容易合成的中间体或可购买化合物(available compound,ac),从而获得一系列可靠的分子合成路线。corey研究组使用了这样的分析技术帮助完成了包括像美登素、阿霉素、赤霉素这类复杂的天然产物的合成,同时也发展了首个可以辅助有机合成路径设计的软件ocss(organic chemical synthesissimulation,有机化学合成的模拟),开创了计算机辅助有机分子合成的新纪元。此后,世界各地实验室在化学全合成、生物合成以及仿生合成领域均取得了重要成果。同时,化学合成路线设计的标准化流程也使得计算机参与成为可能。
3、目前,生物逆合成方法大致可以分为两类:基于知识的方法和基于规则的方法。基于知识的方法根据现有的反应数据库列举可能的生物合成路径,并通过化学相似性和底盘等评分函数对建议的路线进行排序,基于知识的生物逆合成方法仅对数据库中的天然产物有用,当复杂的天然产物的合成反应不在数据库中时,该方法通常就不适用了;基于模板或规则的方法将查询的分子与广义的反应规则的集合相匹配,基于规则的方法需要由专家来手动汇总生物反应规则,或从反应数据库中自动提取。因此这种方法也无法预测规则数据库以外的反应,另外制定专家认可的规则复杂且耗时。虽然已有基于数据库和反应规则的生物合成路径预测工具,但由于本身已知的酶反应数量不足,且由于不同酶的催化杂泛性和专一性也不同,导致现有反应规则或模板无法很好地反映酶的催化功能。因此,对于以酶催化反应为主的生物合成来说,现有基于模板的方法给出的反应路径在实际中常常无法通过相应的酶来催化,并且对于许多生源合成步骤较长,结构较复杂的天然产物,并没有相似度较高的反应模板与之匹配。同时单步反应的预测误差在多步的反应路径预测中会不断积累,因此对于天然产物生物合成路径预测来说,存在单步反应的预测精度低的现象,现有技术中无法预测数据库以外的生物逆合成路径,以及现有的搜索算法对逆合成路径的搜索效率较差。
4、申请公开号为cn114360659a的中国专利,公开了一种结合与或树与单步反应规则预测的生物逆合成方法及系统,通过基于单步反应规则预测模型预测能生成产物分子的代谢反应规则,并对与或树进行扩展,可以实现合成路径的生成,但其公开的单步反应规则预测模型存在不能预测数据库以外的生物逆合成路径的问题,且缺乏对路径搜索策略的优化,无法提升对逆合成路径的搜索效率。
技术实现思路
1、本发明提供了基于深度搜索的生物逆合成预测方法、装置和电子设备,以解决现有技术中无法预测数据库以外的生物逆合成路径的问题以及逆合成路径的搜索效率慢的问题。
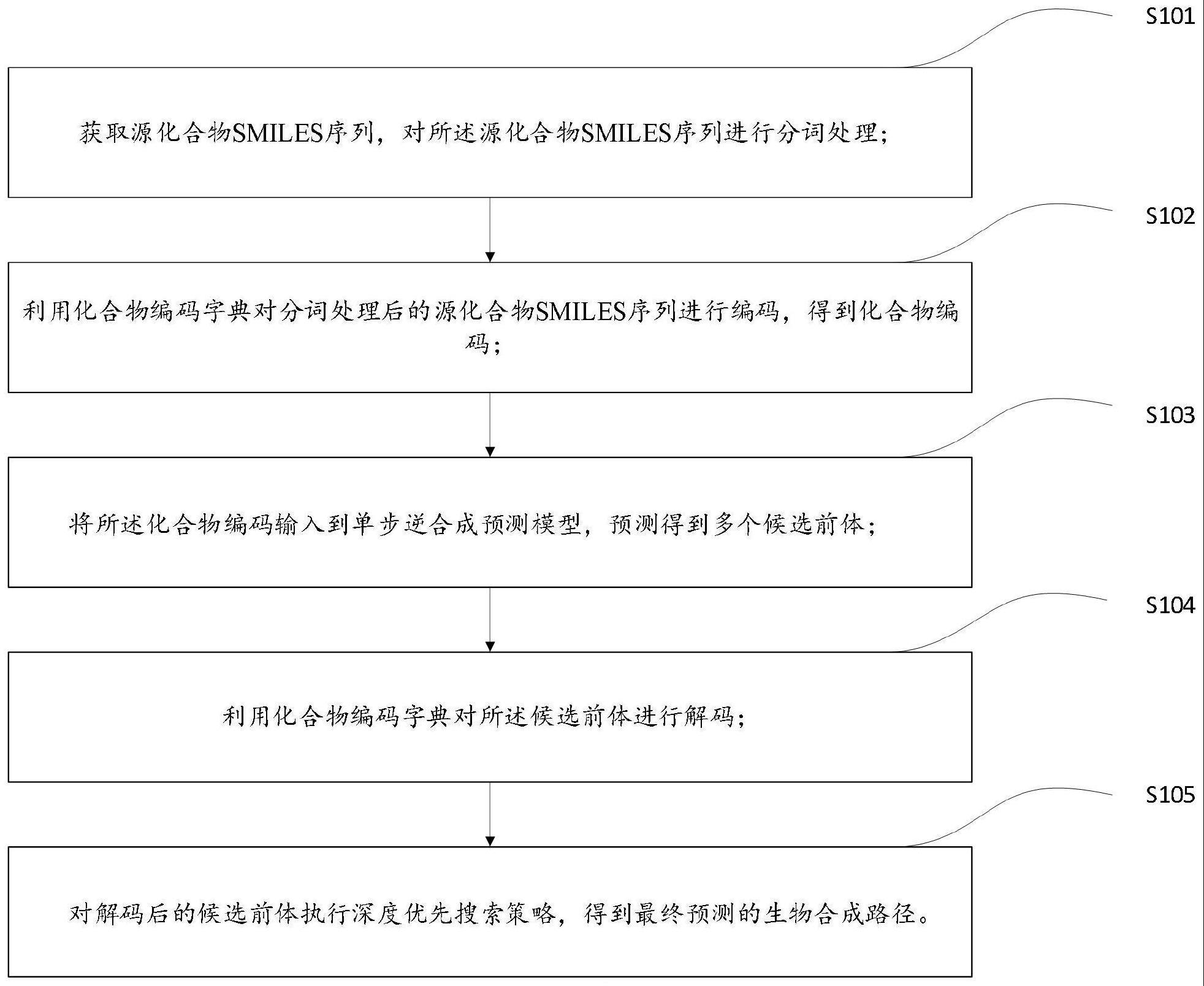
2、本说明书实施例提供了基于深度搜索的生物逆合成预测方法,包括:
3、获取源化合物smiles(simplified molecular input line entry system,一种用于输入和表示分子反应的线性符号)序列,对所述源化合物smiles序列进行分词处理;
4、利用化合物编码字典对分词处理后的源化合物smiles序列进行编码,得到化合物编码;
5、将所述化合物编码输入到单步逆合成预测模型,预测得到多个候选前体;
6、利用化合物编码字典对所述候选前体进行解码;
7、对解码后的候选前体执行深度优先搜索策略,得到最终预测的生物合成路径。
8、优选的,在获取源化合物smiles序列之前,包括:
9、建立化合物编码字典;
10、利用自然语言处理技术中的transformer模型构建单步逆合成预测模型;
11、对深度优先搜索策略中的参数进行设定。
12、优选的,所述建立化合物编码字典,包括:
13、采集化合物smiles序列数据,通过正则表达式对所述化合物smiles序列进行分词处理;
14、对分词处理后的化合物smiles序列数据建立相应的编码,得到化合物编码字典。
15、优选的,所述对深度优先搜索策略中的参数进行设定,包括:
16、设置搜索算法的超参数;
17、设置搜索的最大路径长度;
18、定义有向无环图,并初始化所述有向无环图;
19、定义当前路径列表。
20、优选的,所述对解码后的候选前体执行深度优先搜索策略,得到最终预测的生物合成路径,包括:
21、利用编程语言中的rdkit库对解码后的候选前体进行分子正确性检查,得到合理逆合成预测结果;
22、计算分子正确性检查合格的候选前体与所述源化合物的smiles序列的分子相似度、结果分子的合成难度;
23、根据分子相似度、结果分子的合成难度对分子正确性检查合格的候选前体进行排序,得到优先级序列;
24、根据所述优先级序列依次对所述候选前体中的结果分子是否是终止底物,和/或是否已存入所述有向无环图进行判断;
25、基于判断结果确定最终预测的生物合成路径。
26、优选的,所述基于判断结果确定最终预测的生物合成路径,包括:
27、当所述结果分子是终止底物,和/或已存入有向无环图时,将所述结果分子放入当前路径列表中,得到更新的路径列表;将所述更新的路径列表复制存入有向无环图中,并将所述结果分子从所述更新的路径列表中删除,所述有向无环图为最终预测的生物合成路径。
28、优选的,所述基于判断结果确定最终预测的生物合成路径,还包括:
29、当所述结果分子不是终止底物,和/或未存入有向无环图时,将所述结果分子放入所述当前路径列表中,对所述当前路径列表的长度是否大于等于搜索的最大路径长度进行判断;
30、当所述当前路径列表的长度大于等于搜索的最大路径长度时,结束扩展合成路径;
31、当所述当前路径列表的长度小于搜索的最大路径长度时,将当前路径列表对应的候选前体循环输入到所述单步逆合成预测模型,直至所述结果分子是终止底物,和/或已存入有向无环图。
32、本说明书实施例还提供基于深度搜索的生物逆合成预测装置,其特征在于,包括:
33、序列处理模块,用于获取源化合物smiles序列,对所述源化合物smiles序列进行分词处理;
34、分词编码模块,用于利用化合物编码字典对分词处理后的源化合物smiles序列进行编码,得到化合物编码;
35、逆合成预测模块,用于将所述化合物编码输入到单步逆合成预测模型,预测得到多个候选前体;
36、解码模块,用于利用化合物编码字典对所述候选前体进行解码;
37、策略执行模块,用于对解码后的候选前体执行深度优先搜索策略,得到最终预测的生物合成路径。
38、一种电子设备,其中,该电子设备包括:
39、处理器以及存储计算机可执行指令的存储器,所述可执行指令在被执行时使所述处理器执行上述任一项所述的方法。
40、一种计算机可读存储介质,其中,所述计算机可读存储介质存储一个或多个指令,所述一个或多个指令当被处理器执行时,实现上述任一项所述的方法。
41、本发明采用基于深度学习的无规则生物逆合成方法能够预测数据库以外的生物反应,大大提高了预测完整生物逆合成路径的可能性,本发明以深度优先搜索策略为基础,保证了返回的路径结果是特定深度和宽度搜索树的全部解,无需多次迭代,优化后的搜索策略,避免了对重复节点的扩展,大大提高了搜索效率,相比现有搜索策略,耗时缩短五倍。
- 还没有人留言评论。精彩留言会获得点赞!