一种基于对比学习的生物合成表征方法及模型与流程
本发明涉及生物合成反应的表征领域,具体涉及一种基于对比学习的生物合成表征方法及模型。
背景技术:
1、催化剂能够加速化学反应过程并可以重复利用,提高了化学生产的效率,酶是一种大分子生物催化剂,具有区域选择性、立体选择性、高效性、可分解和反应条件温和等特点,参与到各种生物合成反应中并应用于多种药物的生产。酶的功能标注通常包含氧化还原酶、脱氢酶、水解酶、转移酶等类型,但同一种酶通常不止有一个功能类型,因此酶的功能标注往往需要耗费大量的人力物力。因此,出现了使用深度学习的方法对酶的功能进行标注,但是,当前的方法主要关注酶的单个功能的功能标注,缺乏通用的生物合成反应表征模型,无法满足实际应用中多种功能预测的需求。
2、公开号为cn116844646a的专利文献公开了一种基于深度对比学习的酶功能预测方法,包括:步骤1、将蛋白酶序列输入蛋白质语言模型esm-2进行预处理,得到特征提取后的酶序列信息,并保存;步骤2、计算步骤1中预处理后各类酶的聚类中心之间的欧氏距离,并存储为成对距离矩阵;步骤3、基于步骤2中的成对距离矩阵为神经网络的训练选取三元组,即随机选取一个样本,称为锚,选取一个同类别的样本,称为正样本,选取一个不同类别的样本,称为负样本;步骤4、把按步骤3中的策略选取的三元组输入基于对比学习的并行卷积神经网络,训练并保存酶功能预测模型;步骤5、将需要预测的蛋白酶序列经过步骤1处理后输入步骤4中保存好的模型,以预测其类别。但是该发明仅仅涉及到提高酶功能预测的正确率,并未涉及到酶的多功能预测。
3、公开号为cn116705146a的专利文献公开了兼顾分子结构与序列挖掘的多视角酶功能预测方法,包括:使用biovec生物序列处理方法酶的氨基酸序列进行初始特征提取,将每个酶的氨基酸序列表示为向量,作为酶的初始序列特征fs1;从每种酶的pdb文件中按顺序提取氨基酸序列采用one-hot进行编码,再从氨基酸序列中提取碳原子的三维坐标(x,y,z),将(n*24)维的矩阵作为酶的初始结构特征ft1;针对酶的初始序列特征fs1采用smote数据过采样处理,得到特征fs2;基于特征fs2,采用bbcnet神经网络提取深度序列特征fs3,该网络包含四个模块:bba残差模块、bio-cs注意力模块和全连接模块;针对酶的初始结构特征ft1采用pointnet++点云网络提取深度结构特征ft2,该步骤包含sampling layer、grouping layer、pointnet layer和结构特征最终提取模块;使用tsk模糊系统进行5折交叉试验,分别训练深度序列特征fs3和深度结构特征ft2,学习每个视角的独立信息;使用多视角tsk模糊系统进行5折交叉试验,重新训练深度序列特征fs3和深度结构特征ft2,通过之前学到的信息,利用信息熵调整不同视角之间的重要性,对样本进行分类测试。但是该发明同时考虑了酶的结构特征和序列特征,导致构建的模型复杂,算法繁琐。
技术实现思路
1、本发明的目的是提供一种基于对比学习的生物合成表征方法及模型,建立了包含酶的表征模型和小分子表征模型的生物合成表征模型,通过酶的表征模型预测酶的功能,通过小分子表征模型分析化学反应中底物小分子的特性,从而为酶的功能预测的完备性提供支撑,能够实现多种酶的多种功能的通用性预测。
2、为实现上述发明目的,本发明提供的技术方案如下:
3、第一方面,本发明实施例提供的一种基于对比学习的生物合成表征方法,包括以下步骤:
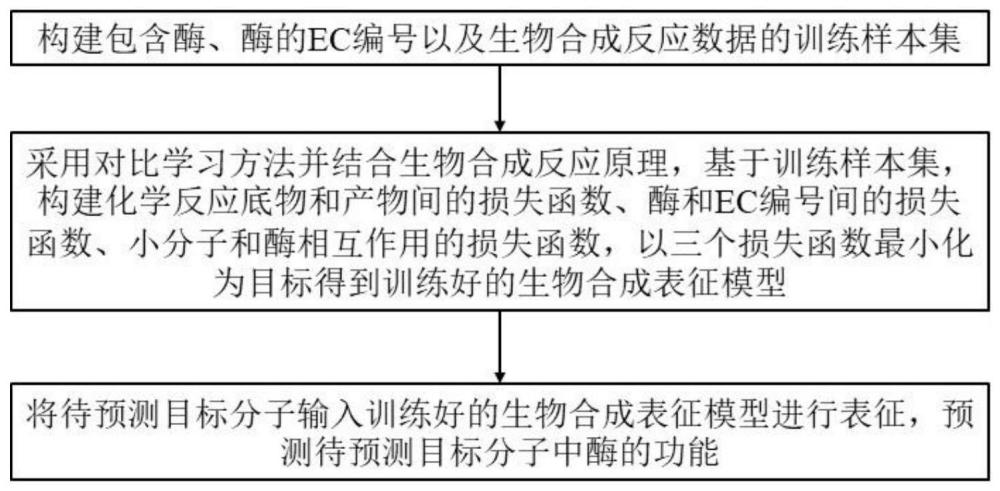
4、步骤1:构建包含酶、酶的ec编号以及生物合成反应数据的训练样本集;
5、步骤2:采用对比学习方法并结合生物合成反应原理,基于训练样本集,构建化学反应底物和产物间的损失函数、酶和ec编号间的损失函数、小分子和酶相互作用的损失函数,以三个损失函数最小化为目标得到训练好的生物合成表征模型;
6、步骤3:将待预测目标分子输入训练好的生物合成表征模型进行表征,预测待预测目标分子中酶的功能。
7、本发明通过建立包含酶的表征模型和小分子表征模型的生物合成表征模型,采用对比学习的方法,在训练过程中,使化学反应底物和产物间的损失函数、酶和ec编号间的损失函数、小分子和酶相互作用的损失函数最小化,得到通用的生物合成表征模型。在实际应用时,通过酶的表征模型预测酶的功能,通过小分子表征模型分析底物小分子的特性,从而为酶的功能预测的完备性提供支撑,实现对多种酶的多种功能的预测。
8、进一步的,步骤1中,酶和酶的ec编号来自于uniprot数据库中提供的蛋白质生物功能信息。
9、进一步的,步骤1中,所述生物合成反应数据来自于ecreact数据集提供的酶促反应数据,其中包含酶和酶催化的化学反应数据,所述酶催化的化学反应数据中包含化学反应的底物、产物和非产物,且底物、产物和非产物均为小分子。
10、进一步的,步骤2中,所述生物合成表征模型包含酶的表征模型和小分子表征模型:
11、所述酶的表征模型由esm-1b模型和第一多层感知机组成,其中,esm-1b模型用于提取酶的表征向量,第一多层感知机用于将酶的表征向量映射为酶表征;
12、所述小分子表征模型由图神经网络和第二多层感知机组成,其中,图神经网络用于提取小分子的浅层表征,第二多层感知机用于将小分子的浅层表征映射为小分子的表征输出向量。
13、进一步的,步骤2中,酶和ec编号间的损失函数最小化的过程,包括:
14、将训练样本集输入酶的表征模型中,分别通过esm-1b模型和第一多层感知机,得到酶表征;
15、基于酶表征,采用对比学习方法,根据正样本的表征缩小相同ec编号的酶表征之间的距离,根据负样本的表征增大不同ec编号的酶表征之间的距离,实现酶和ec编号间的损失函数最小化。
16、进一步的,步骤2中,小分子表征模型中的图神经网络为预训练的图神经网络,预训练的过程,包括:
17、基于uspto数据集构建包含化学反应中产物、非产物和底物的预训练样本;
18、将预训练样本输入到图神经网络中进行表征,得到第一产物表征,第一底物表征,第一非产物表征;
19、使用对比学习方法,以缩小第一底物表征和第一产物表征之间的欧式距离,增大第一底物表征和第一非产物表征之间的欧式距离为目标,完成对图神经网络的预训练。
20、进一步的,步骤2中,化学反应底物和产物间的损失函数最小化的过程,包括:
21、预训练的图神经网络对训练样本集中的小分子进行表征,得到小分子的浅层表征,所述小分子的浅层表征包含第二产物表征、第二底物表征和第二非产物表征;
22、基于小分子的浅层表征,采用对比学习方法,缩小第二底物表征和第二产物表征之间的欧式距离,增大第二底物表征和第二非产物表征之间的欧式距离,实现化学反应底物和产物间的损失函数最小化。
23、进一步的,步骤2中,小分子和酶相互作用的损失函数最小化的过程,包括:
24、使用transe的方法,将酶的表征模型输出的酶表征和小分子表征模型输出的小分子的表征输出向量中的第三底物表征相加,得到酶催化表征,所述小分子的表征输出向量包含第三产物表征、第三底物表征和第三非产物表征;
25、利用对比学习方法,缩小酶催化表征和第三产物表征之间的欧式距离,增大酶催化表征和第三非产物表征之间的欧式距离,实现小分子和酶相互作用的损失函数最小化。
26、进一步的,步骤2中,正样本选取与训练样本集中酶的ec编号相同的氨基酸序列,依次输入esm-1b模型和第一多层感知机中,得到正样本的表征;负样本选取与训练样本集中酶的ec编号不同的氨基酸序列,输入到esm-1b模型中,得到负样本的表征。
27、第二方面,为实现上述发明目的,本发明实施例还提供了一种基于对比学习的生物合成表征模型,包括训练样本集构建单元、表征模型训练单元、表征模型应用单元;
28、所述训练样本集构建单元用于构建包含酶、酶的ec编号以及生物合成反应数据的训练样本集;
29、所述表征模型训练单元用于采用对比学习方法并结合生物合成反应原理,基于训练样本集,构建化学反应底物和产物间的损失函数、酶和ec编号间的损失函数、小分子和酶相互作用的损失函数,以三个损失函数最小化为目标得到训练好的生物合成表征模型;
30、所述表征模型应用单元用于将待预测目标分子输入训练好的生物合成表征模型进行表征,预测待预测目标分子中酶的功能。
31、本发明的有益效果如下:
32、(1)本发明构建了包含酶的表征模型和小分子表征模型的生物合成表征模型,能够同时对酶和酶促反应中的底物小分子进行表征,即酶的表征模型用于酶的功能预测,小分子表征模型对底物小分子的特性进行表征,小分子的底物特性又进一步为酶的功能预测提供支撑,确保了酶功能预测的完备性;
33、(2)本发明提出的生物合成表征模型在采用酶的表征模型进行酶功能预测的同时,还提出了用于表征酶促反应相关的化学反应中底物小分子的小分子表征模型,保证了本发明构建的模型能够预测任意酶的多种功能,即能够应用于预测酶的ec编号或酶的催化常数等多种相关的下游任务。
- 还没有人留言评论。精彩留言会获得点赞!