鉴别干酪乳酸杆菌群的乳酸菌的种间关系与种内关系的方法与流程
本发明系关于使用引物对扩增特定的目标基因,并藉由定序和比对来鉴别干酪乳酸菌群(Lactobacillus casei group)的乳酸菌(1actic acid bacteria)的分类的方法。
背景技术:
干酪乳酸杆菌群中的菌种,包括干酪乳酸菌(Lactobacilluscasei)、副干酪乳酸菌(L.paracasei)与鼠李糖乳酸菌(L.rhamnosus)等,为公认安全(generally recognized as safe,GRAS)的微生物,且已广泛用为市售的益生菌或用作酸奶产品的发酵菌。然而,该类微生物属于分类学上所定义的近缘物种(closely related species),其菌种间的基因型(16S rRNA基因序列的平均相似度>98.9%)与表现型非常相似而不易正确鉴别。此外,益生菌的功能特性具有菌株品系专一性(strain specific)。因此联合国粮农组织/世界卫生组织(FAO/WHO)于2002年规定益生菌学名鉴定必需提升至菌株层次(strain level)。
脉冲式胶体电泳(pulsed-field gel electrophoresis,PFGE)普遍被认定是微生物同种不同菌株分型的黄金标准法(gold standard method),尽管所述方法能达到种内区分(intraspecies discrimination)的目的,但其操作繁琐耗时并存在着再现性的问题。
多基因座序列分型(multilocus sequence typing,MLST)是近年高度发展的菌株分类技术,其原理为针对6~7个约450bp的持家基因(housekeeping genes)进行定序,再依照这些持家基因序列上的碱基变异进而组合成特定的序列型(sequence type,ST)。此技术具有良好的鉴别力和实验再现性,其所建立的序列数据库又可于不同实验室间互相比对,因此MLST可用来加强PFGE的准确性甚至取代PFGE。
Cai等人(″Genotypic and Phenotypic Characterization of Lactobvacillus casei Strains Isolated from Different Ecological Niches Suggests Frequent Recombination and Niche Specificity.″Microbiology,153,pp.2655-2665,2007)和Diancourt等人(″Multilocus Sequence Typing of Latobacillus casei Reveals a Clonal Population Structure with Low Levels of Homologous Recombination.″Applied and Environmental Microbiology,Oct.2007,pp.6601-6611)即采用MLST技术进行分析,其分别将40株和52株干酪乳酸菌区分成36种和31种单套型(haplotypes)。目前亦已有相当多发表文献选择蛋白质编码基因作为干酪乳酸菌群的鉴别目标,例如recA、yycH、dnaK、hsp60、tuf、rpoA与pheS等基因,其中又以recA、yycH与pheS基因的区分性最佳。然而这些鉴别目标大多只适用于种间层次鉴别,在同种的不同菌株间则无法做有效的分型鉴别。尚且,每株菌需采用6至7个基因座的定序信息才能完成菌株分型鉴别,故需耗费较多的时间且分析成本亦较高。
Alekseeva等人(WO2013/100810 A2)以MazEF和RelBE部分基因序列来分析常被人类食用的乳酸菌,包括嗜酸乳酸菌(L.acidophilus)、瑞士乳酸菌(L.helveticus)、胚芽乳酸菌(L.plantarum)、副干酪乳酸菌和鼠李糖乳酸菌等菌种,藉由序列比对方式进行菌种和菌株的间的鉴别。Alekseeva等人的结果显示,副干酪乳酸菌和鼠李糖乳酸菌中的MazEF基因的序列相似度为约83%,故具有明显的区分效果。但Alekseeva等人未将另一类缘关系相近的菌种纳入比对(即干酪乳酸菌),且Alekseeva等人并未使用鼠李糖乳酸菌的标准菌种来做比对,故其的结果无法确认MazEF基因是否可用于区别干酪乳酸菌、副干酪乳酸菌和鼠李糖乳酸菌的种间关系。在菌株品系分型部分,Alekseeva等人发现使用MazEF基因序列可将7株的副干酪乳酸菌区分成3种型别,并可将5株的鼠李糖乳酸菌区分成3种型别。Alekseeva等人亦发现藉由relE1基因的序列比对将5株的鼠李糖乳酸菌区分成3种型别。
因此在本技术领域中,仍需要发展一种能够简便、快速并可同时且准确鉴定乳酸菌的种间关系和种内关系的方法。
技术实现要素:
本发明的一目的系在于提供一种可用于扩增乳酸菌的mutL(DNA错配修复蛋白质(DNA mismatch repair protein))基因或dnaJ(热休克蛋白质(heat shock protein))基因的部分序列的引物对。
本发明的另一目的系在于提供一种包含可用于扩增干酪乳酸菌群的乳酸菌的mutL基因或dnaJ基因的引物的组合物。
本发明的另一目的系在于提供一种藉由上述引物对或组合物来鉴定干酪乳酸菌群的乳酸菌的菌种的方法。
本发明的另一目的系在于提供一种由上述藉由上述引物对或组合物来鉴定一干酪乳酸菌群的乳酸菌种与同种而不同乳酸菌株是否为不同单套型的方法。
本发明的另一目的系在于提供一种包含上述引物对或组合物的试剂盒。
本发明在以下部分中详细描述。本发明的其他特征、目的和优点可易见于本发明的实施方式和申请专利范围中。
附图说明
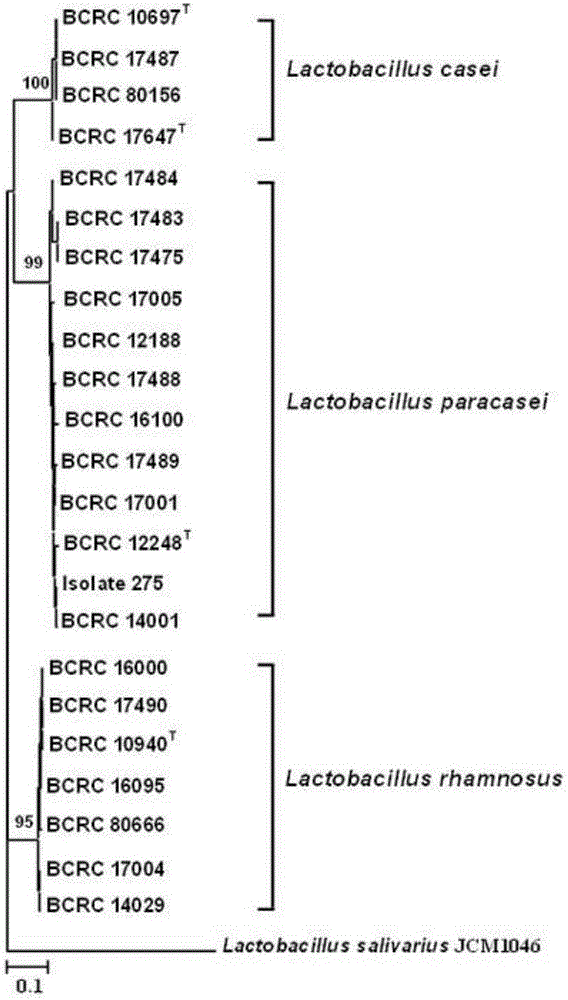
图1显示藉由mutL基因部分序列的定序结果所建构干酪乳酸菌群相关菌种的类缘树(phylogenetic tree)。
图2显示藉由dnaJ基因部分序列的定序结果所建构干酪乳酸菌群相关菌种的类缘树。
具体实施方式
本发明可藉由下述实施方式中所揭示的各种发明态样、实施例和表列的相关叙述所了解。除非在本文中另作定义,否则与本发明关联使用的术语(包含技术和科学术语)应具有本发明所属技术领域中具有通常知识者所了解的含义。且当可了解,除非本文中提供的定义另作说明,在任何潜在歧义的情况,术语的定义应与所述普遍使用的术语(如词典中所定义)一致。可进一步了解者,本案所使用的术语仅系用作描述特定实施态样的目的,而非用于限定。
必须注意的是,除非有清楚的相反指示,于说明书或申请专利范围使用的单数格式″一″、″一种″和″所述″亦包含复数表示。因此,除非上下文另有需要,单数术语应包含复数而复数术语亦包含单数。
本文中的范围以″自一′约′特定数值和/或至另一′约′特定数值″表示。当范围藉上述方式表示时,其包含自一特定数值和/或至另一特定数值的范围。同样地,当数值可藉由术语″约″以表示近似值,将可了解其为一特定值的另一个态样。可进一步了解,当提和有关其它端点和其他端点本身而言,每一范围的两端点皆为有意义的。根据本发明,″约″ 可表示±20%,优选为±10%,更优选为±5%。
于本文中,术语″经分离″或″分离″意谓使物质自其原始环境(例如,若天然存在则为天然环境)中移出。术语″经分离″或″分离″并不一定指物质系经纯化者。
于文中,″一致″或″一致性″百分比系指两个或两个以上聚核苷酸序列比较和比对时,所述两个或两个以上序列的同位点相同的程度;而″相似″或″相似度″百分比系指两个或两个以上聚核苷酸序列比较和比对时,所述两个或两个以上序列中碱基相同的程度。
于本文中,″标准菌株″系指模式菌株,可作为菌株参考比对,以判定是否为同种名的菌种。
于本文中,″引物″系指长度约15至45个核苷酸残基,且能够与指定聚核苷酸模板特异性杂交且为互补聚核苷酸的合成提供起始点的寡核苷酸。当将寡核苷酸引物置于诱导合成的条件下(亦即在核苷酸、互补聚核苷酸模板,和诸如DNA聚合酶的聚合用试剂存在下)时发生此合成。而″简并引物″系指引物中的一或多个密码子被简并密码子取代。
于本文中,″单套型(haplotype)″系指扩增序列的单一核苷酸多态性(single nucleotide polymorphisms(SNPs)的组合图谱。
本发明的目的一在于提供一种可用于分别扩增乳酸菌的mutL基因和dnaJ基因的部分序列的引物对。于本发明的优选实施态样,所述引物为简并引物。
于本文中,可用于扩增mutL基因的部分序列的引物对的第一引物包含5’-ATTGAYCARCTATCKGCMAC-3’(SEQ ID NO:1)的核苷酸序列,其中Y=C或T;R=A或G;K=G或T;且M=A或C,而第二引物包含5’-GAGCCRTAACCGGCAATMA-3’(SEQ ID NO:2)的核苷酸序列,其中R=A或G;且M=A或C。
于本文中,可用于扩增dnaJ基因的部分序列的第一引物包含5’-TCTGAAYTTVGTYTGRCTTTG-3’(SEQ ID NO:3)的核苷酸序列,其中Y=C或T;V=G、A或C;且R=A或G,而第二引物包含5’-GTGGTGCVCARGCKAATC-3’(SEQ ID NO:4)的核苷酸序列,其中V=G、A或C;R=A或G;且K=G或T。
本发明的引物可使用任何本技术领域所熟知的方法来获得。例如,化学合成法。
本发明的另一目的在于提供一种用于扩增mutL基因的部分序列的组合物,其包含第一和/或第二引物和溶剂,其中所述第一引物包含5’-ATTGAYCARCTATCKGCMAC-3’ (SEQ ID NO:1)的核苷酸序列,其中Y=C或T;R=A或G;K=G或T;且M=A或C,而第二引物包含5’-GAGCCRTAACCGGCAATMA-3’(SEQ ID NO:2)的核苷酸序列,其中R=A或G;且M=A或C。
本发明的另一目的在于提供一种用于扩增dnaJ基因的部分序列的组合物,其包含第一和/或第二引物和溶剂,其中所述第一引物包含5’-TCTGAAYTTVGTYTGRCTTTG-3’(SEQ ID NO:3)的核苷酸序列,其中Y=C或T;V=G、A或C;且R=A或G,而第二引物包含5’-GTGGTGCVCARGCKAATC-3’(SEQ ID NO:4)的核苷酸序列,其中V=G、A或C;R=A或G;且K=G或T。
于本发明中的一个实施态样中,所述组合物中的引物为第一引物。于本发明的另一实施态样中,所述组合物中的引物为第二引物。于本发明的进一步实施态样中,所述组合物中的引物包含第一引物和第二引物。
本发明组合物中的溶液可为任何可溶解寡核苷酸序列液体,例如(但不限于)水、或磷酸盐缓冲液。
本发明的另一目的在于提供一种鉴定干酪乳酸菌群的乳酸菌的菌种的方法,其中包含:
(a)获得所述待鉴定乳酸菌的DNA序列;
(b)使用上述的引物对或上述的组合物,扩增获自步骤(a)的所述DNA序列中mutL基因和/或dnaJ基因的部分序列;
(c)将所述经扩增的部分序列定序;和
(d)将所述经定序的序列与复数种的干酪乳酸菌群标准菌株的对应基因序列分别进行比对,获得各别的一致性和/或相似度,其中一致性和/或相似度最高者,则表示所述待鉴定乳酸菌与所述干酪乳酸菌群标准菌株为同一菌种。
于本文中,″干酪乳酸菌群的乳酸菌″系指所述已经鉴定并分类为干酪乳酸菌群中的一个菌种。
本发明步骤(a)中,获得所述待鉴定乳酸菌的DNA序列的方法可为任何本技术领域所熟知的方法。例如(但不限于),以DNeasy Blood&Tissue kit(Qiagen,U.S.A.)萃取并纯化或使用FavorPrep Blood/Cultured Cell Genomic DNA Extraction Mini Kit(Favorgen Biotech,Taiwan)。
本发明步骤(b)中,″扩增″系指复制聚核苷酸序列且从而扩大为较多数目的聚核苷酸分子所用的任何手段,例如藉由反转录、聚合酶链反应和连接酶链反应。
于本发明的实施态样中,步骤(b)系使用包含5’-ATTGAYCARCTATCKGCMAC-3’(SEQ ID NO:1)的核苷酸序列,其中Y=C或T;R=A或G;K=G或T;且M=A或C的第一引物/组合物,与包含5’-GAGCCRTAACCGGCAATMA-3’(SEQ ID NO:2)的核苷酸序列其中R=A或G;且M=A或C的第二引物/组合物,来扩增mutL基因的部分序列。于本发明的另一实施态样中,步骤(b)系使用包含5’-TCTGAAYTTVGTYTGRCTTTG-3’(SEQ ID NO:3)的核苷酸序列,其中Y=C或T;V=G、A或C;且R=A或G的第一引物/组合物,与包含5’-GTGGTGCVCARGCKAATC-3’(SEQ ID NO:4)的核苷酸序列,其中V=G、A或C;R=A或G;且K=G或T的第二引物/组合物,来扩增dnaJ基因的部分序列。于本发明的进一步实施态样中,mutL基因的部分序列与dnaJ基因的部分序列皆被扩增。
本发明步骤(c)中,所述复数种的干酪乳酸菌群标准菌株包含(但不限定为)一种以上选自下列的菌种:干酪乳酸菌BCRC 10697T(ATCC393)、副干酪乳酸菌BCRC 12248T(ATCC25302)、和鼠李糖乳酸菌BCRC 10940T(ATCC 12116)。
核苷酸序列的定序可使用任何本技术领域所熟知的技术手段和软件。例如(但不限于),使用BigDye Terminator v3.1Cycle Squencing Kit(Applied Biosystems,CA,USA)、或PRISM 3730Sequencer(Applied Biosystems)。
本发明步骤(d)中,一致性/相似度可使用如以下所述序列比较算法的一或藉由目视检查所量测。
对于序列比较,通常一个序列充当测试序列与的比较的参考序列。当使用序列比较算法时,将测试和参考序列输入计算机中。接着序列比较算法基于指定的程序参数,计算测试序列相对于参考序列的序列一致性/相似度百分比。
可例如藉由Smith和Waterman,Adv.Appl.Math.2:482,1981的局部同源性算法,藉由Needleman和Wunsch,J.Mol.Biol.48:443,1970的同源性比对算法,藉由Pearson和Lipman,Proc.Natl.Acad.Sci.USA 85:2444,1988的相似性搜寻法,藉由此等算法的计算 机化工具(Wisconsin Genetics Software Package,Genetics Computer Group,575Science Dr.,Madison,WI中的GAP、BESTFIT、FASTA和TFASTA)或藉由目视检查来进行序列的最佳比对以作比较。
适用算法的一实例为PILEUP。PILEUP使用渐进性成对比对产生一组相关序列的多重序列比对以显示关系和序列一致性百分比。其亦绘制显示用于产生比对的类缘树(phylogenetic tress)。类缘树亦可使用临近连接法(neighbor-joining,NJ)来建构(Saitou and Nei,Mol Biol Evol,4:406-425,1987)。PILEUP使用Feng和Doolittle,J.Mol.Evol.35:351-360,1987的渐进性比对方法的简化形式。所用方法类似于由Higgins和Sharp,CABIOS 5:151-153,1989描述的方法。适用于产生序列的多重比对的另一算法为Clustal W程序(Thompson等人,Nucleic Acids Research 22:4673-4680,1994)和Clustal X(1.83)程序(Thompson等人,Nucleic Acids Res 25:4876-4882,1997)。
适用于测定序列一致性和序列相似性百分比的算法的另一实例为BLAST算法,其描述于Altschul等人,J.Mol.Biol.215:403-410,1990中。用于进行BLAST分析的软件可经由国家生物技术信息中心(National Center for Biotechnology Information)公开获得。亦可利用MatGAT 2.02软件(Campanella等人,BMC Bioinformatics,4:29,2003)或Vector NTI v 9.0(Invitrogen,San Diego,CA,USA)计算序列一致性/相似性。
于获得所述待鉴定乳酸菌的序列与每一个复数种的干酪乳酸菌群标准菌的对应基因序列分别比对的各别一致性和/或相似度后,将待鉴定乳酸菌与具有最高一致性和/或相似度的干酪乳酸菌群标准菌株归类为同一种。
本发明的另一目的在于提供一种鉴定一干酪乳酸菌群的乳酸菌种与其他同种的复数个干酪乳酸菌群菌株是否为不同单套型的方法,其中包含:
(a)获得所述待鉴定乳酸菌种的DNA序列;
(b)使用上述的引物对或上述的组合物,扩增获自步骤(a)的所述DNA序列中mutL基因和/或dnaJ基因的部分序列;
(c)将所述经扩增的部分序列定序;和
(d)将所述经定序的序列与所述其他同种的复数个干酪乳酸菌群菌株的对应基因序列分别进行单套型分析,若所分析的序列彼此不相同时,即鉴定所述经分析的菌株为不 同的单套体。
本方法的步骤(a)、(b)和(c)可依如上所述的方式进行;且步骤(d)中的单套型分析可使用DnaSP v.5.0(Librado等人,Bioinformatic,25:1451-1452,2009)软件或BioNumerics软件(Applied Maths,U.S.A)来进行。
于本发明的一个实施态样中,所述其他同种的复数个干酪乳酸菌群菌株为不同的副干酪乳酸菌株,其包含(但不限定为)一种以上选自下列的菌株:副干酪乳酸菌BCRC12248T、副干酪乳酸菌BCRC 12188、副干酪乳酸菌BCRC 14001、副干酪乳酸菌BCRC16100、副干酪乳酸菌BCRC 17001、副干酪乳酸菌BCRC 17005、副干酪乳酸菌BCRC17475、副干酪乳酸菌BCRC 17483、副干酪乳酸菌BCRC 17484、副干酪乳酸菌BCRC17488、副干酪乳酸菌BCRC 17489、副干酪乳酸菌BCRC 12193、副干酪乳酸菌BCRC18853、副干酪乳酸菌BCRC 18854、副干酪乳酸菌BCRC 14023、副干酪乳酸菌BCRC16094和副干酪乳酸菌BCRC 17002。
于本发明的一另个实施态样种,所述其他同种的复数个干酪乳酸菌群菌株为不同的鼠李糖乳酸菌株,其包含(但不限定为)一种以上选自下列的菌株:鼠李糖乳酸菌BCRC10940T、鼠李糖乳酸菌BCRC 14029、鼠李糖乳酸菌BCRC 16000、鼠李糖乳酸菌BCRC16095、鼠李糖乳酸菌BCRC 17004、鼠李糖乳酸菌BCRC 17490、鼠李糖乳酸菌BCRC80663和鼠李糖乳酸菌BCRC 80666菌株。
本发明的另一目的在于提供一种试剂盒,其包含上述的引物对或上述的组合物,其中所述第一引物/组合物与第二引物/组合物系分别装于不同的容器或同一个容器中。
本发明的试剂盒中可进一步包含可供比对或分析的至少一种干酪乳酸菌群株的mutL基因和/或dnaJ基因的序列信息。所述序列信息可包含于任意型态的计算机可读取媒体中,例如(但不限定为)光盘或优盘(USB闪存盘)。
所述至少一种乳酸菌株可为(但不限定为)干酪乳酸菌BCRC 10697T、干酪乳酸菌BCRC 17487、干酪乳酸菌BCRC 17942、干酪乳酸菌BCRC 80156、干酪乳酸菌BCRC17647、副干酪乳酸菌BCRC 12248T、副干酪乳酸菌BCRC 12188、副干酪乳酸菌BCRC14001、副干酪乳酸菌BCRC 16100、副干酪乳酸菌BCRC 17001、副干酪乳酸菌BCRC17005、副干酪乳酸菌BCRC 17475、副干酪乳酸菌BCRC 17483、副干酪乳酸菌BCRC 17484、副干酪乳酸菌BCRC 17488、副干酪乳酸菌BCRC 17489、副干酪乳酸菌BCRC12193、副干酪乳酸菌BCRC 18853、副干酪乳酸菌BCRC 18854、副干酪乳酸菌BCRC14023、副干酪乳酸菌BCRC 16094、鼠李糖乳酸菌BCRC 10940T、鼠李糖乳酸菌BCRC14029、鼠李糖乳酸菌BCRC 16000、鼠李糖乳酸菌BCRC 16095、鼠李糖乳酸菌BCRC17004、鼠李糖乳酸菌BCRC 17490、鼠李糖乳酸菌BCRC 80663、鼠李糖乳酸菌BCRC80666或其任意的组合。
本发明的试剂盒中尚可包含一使用说明,说明如何使用所述试剂盒进行前述的方法,来达到鉴定干酪乳酸菌群的乳酸菌的种间关系和/或种内关系的目的。
本文所述的所有公开案、专利和专利文献均以全文引用的方式并入本文中。
提供以下实例以辅助熟习此项技术者实施本发明。即使如此,不应将所述实例视为本发明的限制,因为本发明所属技术领域中具有通常知识者在不背离本发明的精神或范畴的情况下对本文所讨论的实施例进行的修改和变化,而仍属于本发明的范围。
实施例
1.乳酸菌菌株
所有乳酸菌参考菌株(标准菌株和分离株)皆由台湾新竹市财团法人食品工业发展研究所的生物资源保存和研究中心(Bioresources Collection and Research Center(BCRC))所提供(表1)。
表1.试验菌株名称和其编号
T:表示所述菌株为标准菌株
2.菌株培养条件
以MRS琼脂(Oxoid,Basingstoke,UK)当作标准固体培养基,于30C下进行厌氧培养。将菌株培养于MRS琼脂平板上,于30℃下培养48小时后,刮取少许新鲜菌体,放入1.5mL微量离心管中,以DNeasy Blood&Tissue kit(Qiagen,U.S.A.)萃取并纯化DNA。将DNA溶于适量的去离子水。取少量的DNA溶液,以1%琼脂凝胶进行电泳来确认DNA质量,其余则置入-20℃保存备用。chnol.23:1260-1268)。
实例一、以mutL基因作为鉴定物
1.简并引物(degenerate primers)的设计
由GenBank下载干酪乳酸菌(原命名为:Lactobacillus casei subsp.casei ATCC393)、副干酪乳酸菌(原命名为:Lactobacillus casei ATCC 334)和鼠李糖乳酸菌ATCC8530的mutL基因序列,其登录编号分别为AP012544、CP000423和CP003094。针对dnsJ基因序列中保留度较大的区域设计出的简并引物对为:Lcagp-mutlF3 (5’-ATTGAYCARCTATCKGCMAC-3’(SEQ ID NO:1)和Lcagp-mutlR4(5’-GAGCCRTAACCGGCAATMA-3’(SEQ ID NO:2),其中Y可为C或T残基,R可为A或G残基,K可为G或T残基,且M可为A或C残基。
2.PCR扩增mutL基因片段与定序
将受测干酪乳酸菌、副干酪乳酸菌和鼠李糖乳酸菌的mutL基因的部分片段(约809bp)使用简并引物对进行PCR扩增。PCR反应条件如下:94℃反应5分钟、94℃反应1分钟、60℃反应1分钟和72℃反应1分钟,共进行30个循环,最后再以72℃反应7分钟。扩增出的PCR产物再以QIAquick PCR Purification Kit(Qiagen,U.S.A.)纯化,最后利用BigDye Terminator v3.1Cycle Squencing Kit(Applied Biosystems,CA,USA)和PRISM 3730Sequencer(Applied Biosystems)进行定序。
3.种间关系分析
将DNA序列先经由Clustal X(1.83)程序(Thompson等人,1997)进行排序,再利用MatGAT 2.02软件(Campanella等人,2003)计算种间序列一致性。表2提供由所有受测干酪乳酸菌、副干酪乳酸菌和鼠李糖乳酸菌的各菌株中所扩增mutL基因部分序列的一致性。利用邻近连接法(neighbor-joining,NJ)建构类缘树(phylogenetic tress)(Saitou and Nei,″The neighbor-joining method:a new method for reconstructing phylogenetic trees.″Mol Biol Evol;1987;4:406-425),不同菌种会明显区分为不同群集(图1)。
由表2和图1的结果可发现,经扩增mutL基因的序列一致性可用于判定干酪乳酸菌群的种间关系。例如,将待测菌的经扩增mutL基因序列与干酪乳酸菌、副干酪乳酸菌和鼠李糖乳酸菌的标准菌株的对应mutL基因序列分别进行比对,其间一致性最高的即可被归类为所述菌种。
4.种内关系分析
利用DnaSP v.5.0(Librado and Rozas,2009)软件进行单套型(haplotype)分析。其结果显示所有受测的副干酪乳酸菌BCRC 12248T、BCRC 12193、BCRC 17482、BCRC16100、BCRC 17005、BCRC 18853、BCRC 18854、BCRC 17484、BCRC 17488、BCRC17489、BCRC 14023、BCRC 16094、BCRC 17002和BCRC 17457菌株皆被分为14种不同的单套型;且所有受测的鼠李糖乳酸菌BCRC 10940T、BCRC 80666、BCRC16095、BCRC 17490、BCRC 16000、BCRC 80663和BCRC 14029菌株亦皆被分为7种不同的单套型。故经扩增mutL基因的序列可用于判定副干酪乳酸菌株和鼠李糖乳酸菌株的种内关系。例如,将已确认为副干酪乳酸菌或鼠李糖乳酸菌的待测菌的经扩增mutL基因序列与其他副干酪乳酸菌或鼠李糖乳酸菌的不同菌株的对应mutL基因序列分别进行分析,确认其是否为相同的单套型。
实例二、以dnaJ基因作为鉴定物
1.简并引物(degenerate primers)的设计
由GenBank下载干酪乳酸菌(原命名为:Lactobacillus casei subsp.casei ATCC393)、副干酪乳酸菌(原命名为:Lactobacillus paracasei subsp.paracasei JCM 8130)和鼠李糖乳酸菌ATCC 8530的dnaJ基因序列,其登录编号分别为AP012344、AP012541和CP003094。针对dnsJ基因序列中保留度较大的区域设计出的简并引物对为:LcasDnaj-F1(5’-TCTGAAYTTVGTYTGRCTTTG-3’;SEQ ID NO:3)和LcasDnaj-R1(5’-GTGGTGCVCARGCKAATC-3’;SEQ ID NO:4),其中Y表C或T残基,V表G、A或C残基,R表A或G残基,且K表G或T残基。
2.PCR扩增dnaJ基因片段与定序
将受测干酪乳酸菌、副干酪乳酸菌和鼠李糖乳酸菌的dnaJ基因的部分片段(约620bp)使用简并引物对进行PCR扩增。PCR反应条件如下:94℃反应5分钟、94℃反应1分钟、60℃反应1分钟和72℃反应1分钟,共进行30个循环,最后再以72℃反应7分钟。扩增出的PCR产物再以QIAquick PCR Purification Kit(Qiagen,U.S.A.)纯化,最后利用BigDye Terminator v3.1Cycle Squencing Kit(Applied Biosystems,CA,USA)和PRISM 3730Sequencer(Applied Biosystems)进行定序。
3.种间关系分析
将DNA序列先经由Clustal X(1.83)程序(Thompson等人,1997)进行排序,再利用MatGAT 2.02软件(Campanella等人,2003)计算种间序列一致性。表3提供由所有受测干酪乳酸菌、副干酪乳酸菌和鼠李糖乳酸菌的各菌株中所扩增dnad基因部分序列的一致性。利用邻近连接法建构类缘树(Saitou和Nei,1987),不同菌种会明显区分为不同群集(图2)。
由表3和图2的结果可发现,经扩增dnad基因的序列一致性可用于判定干酪乳酸菌群的种间关系。例如,将待测菌的经扩增dnad基因序列与干酪乳酸菌、副干酪乳酸菌和鼠李糖乳酸菌的标准菌株的对应dnad基因序列分别进行比对,其中一致性最高的即可被归类为所述菌种。
4.种内关系分析
利用DnaSP v.5.0(Librado and Rozas,2009)软件进行单套型(haplotype)分析。其结果显示所有受测的副干酪乳酸菌BCRC 12248T、BCRC 12188、BCRC 14001、BCRC 16100、BCRC 17001、BCRC 17005、BCRC 17475、BCRC 17483、BCRC17484、BCRC 17488、BCRC 17489和分离株275菌株皆被分为12种不同的单套型;且所有受测的鼠李糖乳酸菌BCRC 10940T、BCRC 14029、BCRC 16000、BCRC16095、BCRC 17004、BCRC 17490和BCRC 80666菌株亦皆被分为7种不同的单套型。故经扩增dnad基因的序列可用于判定副干酪乳酸菌株和鼠李糖乳酸菌株的种内关系。例如,将已确认为副干酪乳酸菌或鼠李糖乳酸菌的待测菌的经扩增dnad基因序列与其他副干酪乳酸菌或鼠李糖乳酸菌的不同菌株的对应dnad基因序列分别进行分析,确认其是否为相同的单套型。
- 还没有人留言评论。精彩留言会获得点赞!