一种海参养殖水体长石莼、硬石莼和萱藻孢子/配子的实时定量PCR检测方法与流程
本发明涉及海参养殖水体青苔爆发的防控与预警领域,具体地说是一种海参养殖水体长石莼、硬石莼和萱藻孢子/配子的动态监测方法。
背景技术:
“青苔”是对养殖水体中易爆发的大型藻类生物的俗称。近年来海参池塘养殖过程中,大型青苔藻类爆发有增加的趋势。大型藻类爆发,与有益微藻(硅藻等)竞争光、营养盐等限制性资源,导致饵料生物生长缓慢乃至缺乏,限制海参的生长;藻类爆发可能伴随着毒素的分泌,对海参造成生理胁迫和化学毒害;青苔的丝状结构会使海参困在其中,影响海参的运动和摄食;而且,藻类死后沉积在池底,在微生物作用下分解并消耗大量溶解氧,引起缺氧和底质腐败,并导致海参的缺氧死亡,给海参养殖产业带来巨大经济损失。
目前常用的防治办法有人工捞除,加入除草剂清除等。然而藻类一旦爆发,人工捞除后会伴随残余藻类组织或丝状体的再次大量增殖,耗费大量人工和物力财力,效果并不明显。除草剂的加入对于水体中的有益藻类、养殖生物(如海参)都会带来严重的刺激作用,破坏水体的生态平衡。因此,在爆发前期和非爆发期对大型藻类孢子/配子数量进行监控和预测,具有重要的现实意义。
然而,因孢子体和配子体等生殖细胞个体较小(<10μm),在光镜下不易与其他单细胞原生生物(如自养型及混合营养型鞭毛虫)相区别,仅通过传统形态学观察难以确定其数量或生物量,故无法实现对环境样品中大型青苔藻类孢子体与配子体的数量检测与动态监测。
技术实现要素:
本发明的目的是提供一种基于基因层面的高灵敏度快速有效的分子生物学监测海参养殖池塘长石莼、硬石莼和萱藻(青苔常见种类)孢子/配子量动态的方法。
为实现上述目的,本发明采用的技术方案如下:
一种海参养殖水体长石莼、硬石莼和萱藻孢子/配子的实时定量PCR检测方法,包括以下步骤:
1)设计ITS区特异性引物:在长石莼(Ulva linza)、硬石莼(Ulva rigida)和萱藻(Scytosiphon lomentaria)18S rDNA序列右端和ITS序列区域分别查找涵盖部分ITS区或ITS区全长的物种特异性引物;分别选取序列相似性高于90%的同属或同科物种作为参考,进行特异性位点查找;使用Intergrated DNA Technologies(http://sg.idtdna.com/site)网站对所得特异性引物的GC含量,Tm值,引物二聚体形成,发卡结构产生等参数进行在线评估,筛选得出高质量引物对;
2)引物特异性验证:利用NCBI(National Center for Biotechnology Information)的BLASTn功能和Primer-BLAST在线软件对步骤1)中所得引物的特异性进行验证;以实验室对照藻类和养殖水样DNA为模板,扩增其ITS序列,验证引物特异性和普适性;
3)定量PCR条件优化:利用包含目标片段的标准线性DNA构建标准曲线,回归系数达到0.98以上,扩增效率为大于90%;
5)定量PCR法实时监测海参养殖水体大型藻类孢子/配子的数量。
步骤2)扩增程序为:94℃预变性5min;94℃变性60s,55~57℃退火45s,72℃延伸60s,循环30~35次;72℃延伸10min。
所述养殖水样DNA的获取是将不同时期采集的养殖水样经200μm孔径的筛绢预过滤后,缓慢抽滤到0.22μm孔径的醋酸纤维素膜上(直径47mm;Millipore,美国),膜样使用土壤提取试剂盒(Mpbio,美国)提取DNA。
定量PCR标准曲线的构建是将含有藻类ITS片段的标准DNA按1:102~1:108倍比稀释,进行定量PCR实验,根据所得结果中的临界循环值CT绘制参考标准曲线;使用微量紫外分光光度计测量不同稀释倍数下质粒的DNA浓度,并利用公式:copies(/μL)=(6.02×1023×(DNA浓度(ng/μL)×10-9))/(DNA长度×660)计算标准DNA浓度;标准曲线以质粒DNA中ITS拷贝数(log10)为横坐标,CT值为纵坐标进行绘制;qPCR实验的扩增效率根据质粒标准曲线斜率(k)计算,计算公式为:E=(10-1/k-1)×100%。
所述的定量PCR条件优化扩增程序为:95℃预变性7min;95℃变性30s,56~58℃退火30s,72℃延伸45s,72~80℃采集信号,40个循环。样品扩增产物溶解曲线显示单峰,且扩增产物的凝胶电泳检测为目标单一条带。
本发明所具有的优点:
1.本发明采用的实时定量PCR检测方法,基于DNA层面,灵敏度高,且方法简单易行、经济廉价,在实践中有很好的应用潜力。
2.本发明针对ITS区设计引物,特异性强,能够有效区分不同种属藻类,既可以监测藻类孢子/配子数量变化,又可以掌握海藻的组成动态。
3.本发明提供的实时定量PCR检测方法不仅适用于海参养殖水体,也可用于鱼类和虾类等养殖水体藻类的监测,在青苔防控和预警方面具有一定的指导意义。
附图说明
图1示出本发明实施案例中长石莼、硬石莼和萱藻ITS区域特异性引物示意图。
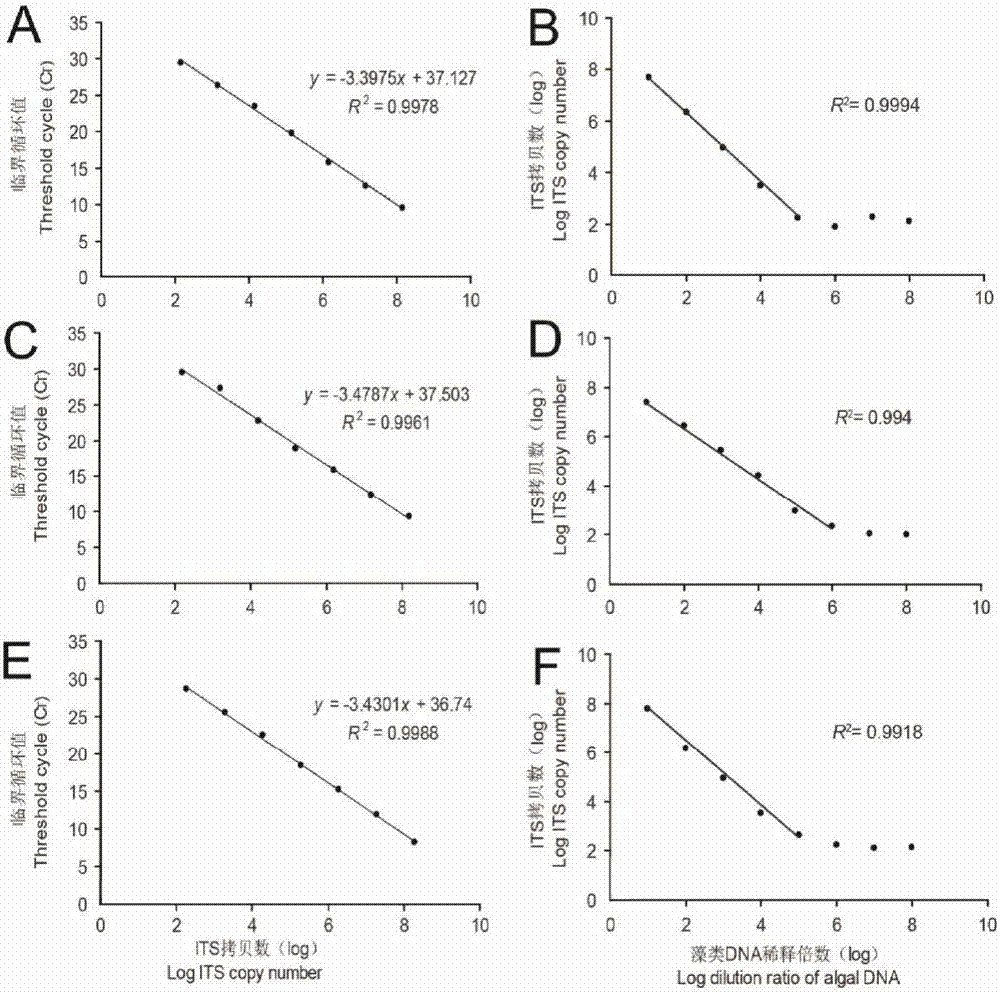
图2示出本发明实施案例中三种藻类标准曲线(A,C,E)和不同稀释倍数藻类rDNA所含ITS拷贝数(B,D,F)(A,B.长石莼;C,D.硬石莼;E,F.萱藻)。
图3示出本发明实施案例中海参养殖水体萱藻ITS拷贝数动态(黑色和灰色表示每个月份两组生物学重复)
具体实施方式
本发明的实时定量PCR检测方法步骤如下:
步骤1)设计特异性引物:在Genbank数据库中获得的长石莼、硬石莼和萱藻18S rDNA序列右端和ITS序列区域分别查找涵盖部分ITS区或ITS区全长的物种特异性引物;分别选取序列相似性高于90%的同属或同科物种作为参考,进行特异性位点查找;使用Intergrated DNA Technologies(http://sg.idtdna.com/site)网站对所得特异性引物的GC含量,Tm值,引物二聚体形成,发卡结构产生等参数进行在线评估,筛选得出高质量引物对;
2)引物特异性验证:利用NCBI的BLASTn功能和Primer-BLAST在线软件对步骤1)中所得引物的特异性进行验证;以实验室对照藻类和养殖水样DNA为模板,验证引物特异性和适用性;养殖水样DNA的获取是将不同时期采集的养殖水样经200μm孔径的筛绢预过滤后,缓慢抽滤到0.22μm孔径的醋酸纤维素膜上(直径47mm;Millipore,美国),膜样使用土壤提取试剂盒(Mpbio,美国)提取DNA;所有扩增产物经纯化后克隆测序以确定其专一性;
3)定量PCR条件优化:以实验室对照藻类和养殖水样DNA为模板,利用步骤2)验证的ITS特异性引物进行定量PCR实验;扩增程序为:95℃预变性7min;95℃变性30s,56~58℃退火30s,72℃延伸45s,72~80℃采集信号,40个循环;利用无菌去离子水作为阴性对照,利用包含目标片段的标准线性质粒构建标准曲线,回归系数大于0.98,扩增效率大于90%;样品扩增产物溶解曲线显示单峰,且扩增产物的凝胶电泳检测为目标单一条带;
4)定量PCR法实时监测海参养殖水体大型藻类孢子/配子的数量:利用步骤2)验证的ITS特异性引物和步骤3)优化的定量PCR条件,监测不同时期养殖水体大型藻类孢子/配子数量的动态变化。
下面结合具体实施例说明:
实施例1
海参养殖水体不同季节藻类孢子/配子数量检测
采集烟台市云溪村东方海洋海参养殖基地水体样品,经200μm孔径的筛绢预过滤后,缓慢抽滤到0.22μm孔径的醋酸纤维素膜上(直径47mm;Millipore,美国),膜样使用土壤提取试剂盒(Mpbio,美国)提取DNA。
在Genbank数据库中获得长石莼、硬石莼和萱藻18S rDNA序列右端和ITS序列区域分别查找涵盖部分ITS区或ITS区全长的物种特异性引物。分别选取序列相似性高于90%的同属或同科物种作为参考,进行特异性位点查找。使用Intergrated DNA Technologies(http://sg.idtdna.com/site)网站对所得特异性引物的GC含量,Tm值,引物二聚体形成,发卡结构产生等参数进行在线评估,筛选得出高质量引物对(表1和图1);利用NCBI的BLASTn功能和Primer-BLAST在线软件对所得引物的特异性进行验证。以实验室对照藻类萱藻,长石莼,硬石莼,曲石莼(Ulva flexuosa)和扁石莼(Ulva compressa)基因组DNA和养殖水样DNA为模板,验证引物特异性和适用性(表2)。所有扩增产物经纯化后克隆测序以确定其专一性。
以对照藻类萱藻,长石莼,硬石莼DNA为模板,利用ITS特异性引物进行定量PCR实验。扩增程序为:95℃预变性7min;95℃变性30s,56~58℃退火30s,72℃延伸45s,72~80℃采集信号,40个循环。利用无菌去离子水作为阴性对照,利用包含目标片段的标准线性质粒构建标准曲线,回归系数大于0.98,扩增效率大于90%(图2)。样品扩增产物与标准DNA扩增产物的溶解曲线重合,且扩增产物的凝胶电泳检测为目标单一条带。
利用上述验证的三种藻类特异性ITS引物和优化的定量PCR条件,检测海参养殖水体2014年12月、2015年3月(“青苔”爆发)、6月、9月及12月三种藻类孢子/配子的数量变化。结果显示,两种石莼类藻类孢子/配子低于检测限;2014年12月份萱藻ITS rDNA拷贝数约有1.0×107拷贝/mL,2015年3月份达到最高,约2.5×107拷贝/mL;自2015年6月起萱藻拷贝数降低至5.0×106拷贝/mL,此后均低于4.0×106拷贝/mL(图3)。由此可得,该海参养殖水体青苔爆发的优势种为萱藻,爆发月份(2015年3月)前一时期(2014年12月)水体中藻类孢子量高于其他非爆发月份。
实施例2
海参养殖水体同一季节有无青苔爆发情况下的藻类孢子/配子数量比较
大型藻类的分类鉴定,ITS区特异性引物的设计和验证以及定量PCR条件优化方法同实施案例1。
利用三种藻类(长石莼、硬石莼和萱藻)ITS区特异性引物和优化的定量PCR条件,比较海参养殖水体2014年12月和2015年12月以及2015年3月(青苔爆发)和2016年3月(未爆发青苔)间的藻类孢子/配子数量。结果显示,长石莼和硬石莼的孢子/配子数量低于检测限;2014年12月萱藻ITS rDNA拷贝数约有1.0×107拷贝/mL,而2015年12月萱藻ITS rDNA拷贝数仅为约3.0×106拷贝/mL;2015年3月青苔爆发时期萱藻ITS rDNA拷贝数高达2.5×107拷贝/mL,而2016年3月未爆发青苔,萱藻ITS rDNA拷贝数仅为约3.0×106拷贝/mL(图3)。
表1示出本发明实施案例中长石莼、硬石莼和萱藻ITS区域特异性PCR引物参数。
表2示出本发明实施案例中特异性引物扩增对照藻类和养殖水体DNA结果。
表1长石莼、硬石莼和萱藻ITS区域特异性PCR引物参数
表2特异性引物扩增对照藻类和养殖水体DNA结果
- 还没有人留言评论。精彩留言会获得点赞!