体内DNA无缝组装方法与流程
本发明属于分子生物学领域,更具体地,本发明涉及体内dna无缝组装方法。
背景技术:
dna组装技术是合成生物学实现各种目标的重要环节及关键步骤。根据组装时所处的环境,dna组装可分为体内组装和体外组装。体外组装最常见的就是用限制性内切酶和连接酶来完成的方法。但是这种方法对于多基因或者大片段的组装是不行的,因为受到限制性内切酶识别位点的限制。最近这些年发展起来的体外组装技术归类主要有:①连接酶链式反应与pcr的结合(lcr-pcr);②重叠多片段短核苷酸与pcr的结合,该方法类似于重叠延伸pcr(oe-pcr);③利用同尾酶酶切连接技术,包括biobrick、bglbrick和ibrick等;④利用iis限制性内切酶或其他酶辅助的dna组装技术,如goldengate组装法、master连接法、user克隆等;⑤配对选择组装法(psa);⑥利用特殊的重组酶来完成的dna组装,如gateway、in-fusion等;⑦依赖dna聚合酶的3’-5’核酸外切酶活性来完成的dna组装技术,如slic、gibson恒温组装法等。
体内dna组装方法相对要少很多,根据不同的宿主目前主要有这么几类:①以酵母为宿主的方法:主要是利用酵母自身体内所具有的重组功能,可以实现多个dna片段在酵母体内一步完成组装。gibson体内组装法、dnaassembler、体内迭代法等都是利用了酵母体内的重组功能。但是酵母可能对大肠杆菌复制子进行修饰,导致组装后的载体无法工作。②以枯草芽孢杆菌为宿主的方法:在枯草芽孢杆菌体内进行dna组装主要是日本科学家itaya实验室建立起来的,他们使用bacillussubtilis168基因组(4.2mb)作为克隆载体(bgm),首先将光合细菌synechocystispcc6803染色体上间隔排列的多段序列lps(每段约5kb)克隆到lpa质粒,然后通过同源重组将lps序列整合到bgm上,然后再次进行同源重组,将synechocystispcc6803染色体上大约30kb的序列整合到bgm上,随后再将另外一个lpa质粒上的lps序列整合到bgm上,然后将紧邻synechocystispcc6803染色体上另外一段约30kb的序列整合到bgm上,依次迭代,将光合细菌synechocystispcc68033.5mb的染色体克隆到bgm上。但是该方法需要100kb以上的dna片段作为模板,其应用很受限。随后,他们改变了策略,首先通过pcr将染色体上相互重叠的序列(约4-6kb)克隆到质粒pcisp401和pcisp402上,然后再通过pcr从质粒上将要整合的序列克隆下来,并在两侧带上了同源臂,然后再通过同源重组将各个片段依次拼接在bgm上,通过这样的方法,将小鼠线粒体基因组(16.3kb)和水稻叶绿体基因组(134.5kb)组装成功。③以大肠杆菌为宿主的方法和酵母及枯草芽孢杆菌不同,线性dna序列并不容易直接转化进e.coli,因为atp依赖的外切酶recbcd能够消化掉线性dna片段。在e.coli中,通常用λred重组酶系统和recet重组系统来进行同源重组。λred重组酶系统更加适合线性和环状dna之间的重组,而recet重组系统更加适合两个线性dna片段之间的重组。对于dna片段整合到e.coli染色体上,随着dna片段长度的增加,整合的效率会大幅度降低,kuhlman等人设计了一种系统,利用red重组系统和i-scei内切酶,可以最大将7kb的dna片段一次整合到e.coli染色体上。但该方法需要使用三个质粒,两次转化,用的时间也较长。
综上,本领域中还需要进一步研究更为简便,易于操作的方法,来提高dna体内组装效率。
技术实现要素:
本发明的目的在于提供一种体内dna无缝组装方法。
在本发明的第一方面,提供一种dna无缝组装方法,所述方法包括:
(1)扩增获得基因a的序列,其5’端加上其上游同源序列;在其3’端加上一段与基因b的5’端相同的序列片段1;
(2)扩增获得teta的序列,在其5’端加上(5’→3’)i-scei识别序列及一段与基因b的5’端相同的序列片段2,以及i-scei识别序列;在其3’端加上(5’→3’)i-scei识别序列及与基因b的5’端相同的序列片段3;
(3)扩增获得基因b的序列,其3’端加上其下游同源序列;
(4)将前述步骤(1)~(3)获得的扩增产物混合作为模板,进行pcr,获得“基因a-teta-基因b”序列;
(5)在宿主细胞中诱导表达red重组系统,将步骤(4)获得的“基因a-teta-基因b”序列转入宿主细胞,使“基因a-teta-基因b”序列整合到染色体上;
(6)诱导表达red重组系统以及该系统中的i-scei限制性内切酶,获得基因a和基因b发生无缝组装的融合基因。
在一个优选例中,步骤(6)之后,还包括:(7)将步骤(6)获得的基因a和基因b发生无缝组装的融合基因与其它1个或多个基因无缝组装的步骤(重复步骤(1)~(6),以基因b替换基因a,以其它基因替换基因b)。
在另一优选例中,所述的red重组系统是同时含有能用不同诱导剂诱导表达red重组系统和i-scei酶的质粒;较佳地包括:帮助质粒ptkred。
在另一优选例中,步骤(1)中,所述的与基因b的5’端相同的序列片段1的长度至少为25bp,较佳地25~40bp。
在另一优选例中,步骤(2)中,所述的与基因b的5’端相同的序列片段2的长度至少为25bp;较佳地25~40bp。
在另一优选例中,步骤(2)中,所述的与基因b的5’端相同的序列片段3的长度至少为25bp;较佳地25~40bp。
在另一优选例中,所述的上游同源序列的长度至少为25bp;较佳地40~1000bp。
在另一优选例中,所述的下游同源序列的长度至少为25bp;较佳地40~1000bp。
在另一优选例中,所述的i-scei识别序列为:tagggataacagggtaat或其反向互补序列。
在另一优选例中,所述的teta的序列包括:启动子和teta基因序列。
在另一优选例中,所述的启动子的序列如seqidno:1。
在另一优选例中,所述的teta基因序列如seqidno:2所示。
在另一优选例中,所述的宿主细胞是原核细胞;较佳地,为大肠杆菌细胞或沙门氏菌细胞。
在另一优选例中,所述的基因a为crte,所述的基因b为crtb,所述的其它基因为crti。
在本发明的另一方面,提供一种用于进行dna无缝组装的试剂盒,所述试剂盒中包括:teta基因或含有teta基因序列的质粒;较佳地,所述的teta基因包括启动子;更佳地,所述的启动子的序列如seqidno:1所示。
在一个优选例中,所述试剂盒中还包括:同时含有能用不同诱导剂诱导表达red重组系统和i-scei酶的质粒;较佳地包括:帮助质粒ptkred。
在另一优选例中,所述试剂盒中还包括:四环素类抗生素。
本发明的其它方面由于本文的公开内容,对本领域的技术人员而言是显而易见的。
附图说明
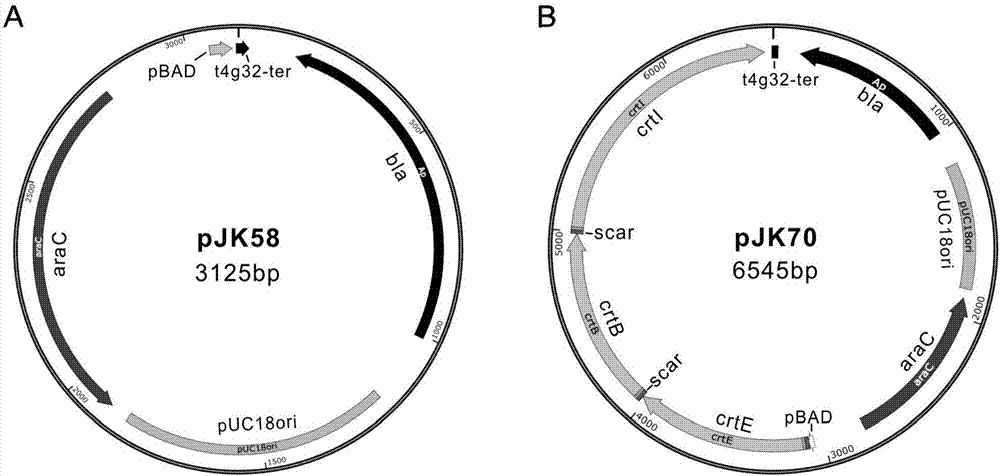
图1a、本发明改造的质粒pjk58的图谱。
图1b、本发明改造的质粒pjk70的图谱。
图2、本发明的dna无缝组装的序列拼接的原理示意图。图中,teta区段包含了位于编码序列5’端的启动子序列(在teta启动子的5’端加上与基因b的5’端相同的序列片段2)。
图3、本发明的dna无缝组装实施过程的原理示意图。
图4、无缝组装(crte、crtb和crti组装)在细胞内完成之后,细胞在bma培养基上进行负筛的表型。
图5、dna无缝组装(crte、crtb和crti组装)后获得的组装部位序列(crte、crtb和crti组装)的测序结果,清楚显示出组装过程没有引入任何附加序列。
图6、把组装好的序列(crte、crtb和crti组装)克隆到载体pjk58上,转化到大肠杆菌中,使用阿拉伯糖诱导表达,检测番茄红素的表达情况,结果进一步表明组装是成功的。
具体实施方式
本发明人经过深入的研究,揭示了一种简便、易于操作的dna无缝连接方法。本发明的方法可以实现多个dna片段之间的无缝组装,或可以将多个dna片段无缝组装到染色体上。采用本发明的方法,可以把任意长度的dna片段整合到细胞(如e.coli)染色体上。
如本文所用,所述的“dna”、“基因”包括了“dna片段”、“基因片段”。
如本文所用,鉴于dna的互补特点,所述的“基因x的5’端相同的序列”,涵盖与基因x的5’端序列完全一致的序列,或经转录或扩增后与之完全一致的序列。
本发明的方法的具体原理如图2~图3所示。
根据图2,首先,通过扩增获得基因a的序列,其5’端加上其上游同源序列;在其3’端加上一段与基因b的5’端相同的序列片段;然后,扩增获得teta的序列,在其5’端加上(5’→3’)i-scei识别序列及基因b的5’端序列片段;在其3’端加上(5’→3’)i-scei识别序列与基因b的5’端相同的序列片段;第三,扩增获得基因b的序列,其3’端加上其下游同源序列;第四,将前述步骤(1)~(3)获得的扩增产物混合作为模板,进行pcr获得“基因a-teta-基因b”序列。
根据图3,首先诱导表达red重组系统,对前述获得的“基因a-teta-基因b”序列整合到宿主染色体上,使用teta正向选择,获得阳性克隆并测序确认。随后同时诱导表达red重组系统和i-scei限制性内切酶,i-scei酶识别酶切位点并对序列进行剪切,再藉由red重组系统进行同源重组,使用teta负筛方法获得基因a和基因b发生无缝组装的宿主细胞并测序验证,从而获得基因a和基因b发生无缝组装的融合基因。
如上方法,可以实现基因a与基因b的无缝组装。
本发明中,以teta基因作为实现基因a与基因b无缝连接的中间基因。teta基因编码一种跨膜泵蛋白,能从细胞内将四环素类抗生素排出。作为本发明的优选方式,本发明人通过优化比较,对teta使用了高效启动子序列,teta序列也进行了优化选择,使得负筛效率可以达到100%。因此,作为本发明的优选方式,应用seqidno:1所示的序列作为驱动teta基因表达的启动子,应用seqidno:2所示的序列作为teta基因编码序列。
本发明中,使用red重组系统进行序列重组。red同源重组技术是一项可在染色体水平上进行遗传操作技术,只需较短的同源序列,而不需要特定的限制性酶切位点,即可在生物体内完成重组过程。
本发明中,使用i-scei限制性内切酶进行序列剪切。i-scei识别位点含有18个碱基,所以在染色体上含有此识别位点的概率非常低,这保证了该方法能够用于基因序列在染色体上无缝组装。
本发明方法,只需要一个帮助质粒,一次转化,通过使用teta负筛来找到最终的阳性克隆。
基于本发明的新方法,本发明还提供了一种用于进行dna无缝组装的试剂盒,所述试剂盒中包括在本发明的方法中所应用的部分或全部的试剂。
作为本发明的优选方式,所述的试剂盒中包括选自以下的试剂:teta基因或含有teta基因序列的质粒,所述的teta基因包括启动子和tetadna序列;ptkred质粒,四环素类抗生素。
所述的试剂盒中还可包括使用说明书,其中描述了本发明的进行dna无缝组装的方法,以便于本领域技术人员应用。
下面结合具体实施例,进一步阐述本发明。应理解,这些实施例仅用于说明本发明而不用于限制本发明的范围。下列实施例中未注明具体条件的实验方法,通常按照常规条件如j.萨姆布鲁克等编著,分子克隆实验指南,第三版,科学出版社,2002中所述的条件,或按照制造厂商所建议的条件。
材料与方法
菌种
采用大肠杆菌e.colik-12mg1655。
质粒
采用ptkred(购买自addgene)、pjk58(本实验室构建)、pjk70(本实验室构建)和pucteta(本实验室构建)。
pjk58的构建方法:bla基因通过pcr从puc18质粒上克隆得到,前向引物包含了t4-g32终止子序列,pucori复制子从puc18质粒上pcr克隆得到,随后进行overlappcr,使得前面得到的两段序列融合成一条序列。arac基因序列及阿拉伯糖诱导型启动子序列pbad从ptkred载体上pcr克隆获得,然后对得到的两条序列进行酶切连接,构建成质粒pjk58(见图1a)。
pjk70:以pjk58为基础,采用ibrick的方法,把crte、crtb和crti组装到pjk58上形成了pjk70(见图1b)。
pucteta:在puc18上连接teta基因及所需的启动子序列,连接入hindiii和ecori之间,形成了新的质粒pucteta。
lb培养基(/l)
蛋白质(tryptone):10g;
酵母菌(yeastextract):5g;
nacl:10g;
调节ph值到7.0,然后121℃湿热灭菌。
bma培养基配方:
瓶a:agar12g;蛋白胨4g;酵母抽提物4g;加ddh2o到400ml;
瓶b:nacl8g;nah2po48g;加ddh2o到400ml。
各自灭菌20分钟,冷却到适当温度后,在瓶a中加入500μlfusaricacid(即9.6mgfusaricacid溶于dimethylformamide),在瓶b中加入4ml20mmzncl2。然后混合瓶a和瓶b。混合好之后开始倒平板,避光,在36小时内使用这些平板。
pcr试剂使用全式金的fastpfudna聚合酶及配套的试剂。
实施例1、拼接crte、crtb和crti三个基因的引物及方法设计
本实施例中,以无缝拼接crte、crtb和crti三个基因为例。以teta基因负筛技术来选择阳性克隆。先以crte作为基因a,crtb作为基因b。
本实施例中,所涉及的各个基因的dna序列如下:
(1)teta基因使用的启动子序列(seqidno:1):
atcgttgacggctagctcagtcctaggtacagtgctagctactagagaaagaggagaaatactag
(2)teta基因序列(seqidno:2):
atgaatagttcgacaaagatcgcattggtaattacgttactcgatgccatggggattggccttatcatgccagtcttgccaacgttattacgtgaatttattgcttcggaagatatcgctaaccactttggcgtattgcttgcactttatgcgttaatgcaggttatctttgctccttggcttggaaaaatgtctgaccgatttggtcggcgcccagtgctgttgttgtcattaataggcgcatcgctggattacttattgctggctttttcaagtgcgctttggatgctgtatttaggccgtttgctttcagggatcacaggagctactggggctgtcgcggcatcggtcattgccgataccacctcagcttctcaacgcgtgaagtggttcggttggttaggggcaagttttgggcttggtttaatagcggggcctattattggtggttttgcaggagagatttcaccgcatagtcccttttttatcgctgcgttgctaaatattgtcactttccttgtggttatgttttggttccgtgaaaccaaaaatacacgtgataatacagataccgaagtaggggttgagacgcaatcaaattcggtgtacatcactttatttaaaacgatgcccattttgttgattatttatttttcagcgcaattgataggccaaattcccgcaacggtgtgggtgctatttaccgaaaatcgttttggatggaatagcatgatggttggcttttcattagcgggtcttggtcttttacactcagtattccaagcctttgtggcaggaagaatagccactaaatggggcgaaaaaacggcagtactgctcggatttattgcagatagtagtgcatttgcctttttagcgtttatatctgaaggttggttagttttccctgttttaattttattggctggtggtgggatcgctttacctgcattacagggagtgatgtctatccaaacaaagagtcatcagcaaggtgctttacagggattattggtgagccttaccaatgcaaccggtgttattggcccattactgtttgctgttatttataatcattcactaccaatttgggatggctggatttggattattggtttagcgttttactgtattattatcctgctatcaatgaccttcatgttgacccctcaagctcaggggagtaaacaggagacaagtgcttag(seqidno:2)
(3)crte基因序列(seqidno:3)
atgacggtctgcgcaaaaaaacacgttcatctcactcgcgatgctgcggagcagttactggctgatattgatcgacgccttgatcagttattgcccgtggagggagaacgggatgttgtgggtgccgcgatgcgtgaaggtgcgctggcaccgggaaaacgtattcgccccatgttgctgttgctgaccgcccgcgatctgggttgcgctgtcagccatgacggattactggatttggcctgtgcggtggaaatggtccacgcggcttcgctgatccttgacgatatgccctgcatggacgatgcgaagctgcggcgcggacgccctaccattcattctcattacggagagcatgtggcaatactggcggcggttgccttgctgagtaaagcctttggcgtaattgccgatgcagatggcctcacgccgctggcaaaaaatcgggcggtttctgaactgtcaaacgccatcggcatgcaaggattggttcagggtcagttcaaggatctgtctgaaggggataagccgcgcagcgctgaagctattttgatgacgaatcactttaaaaccagcacgctgttttgtgcctccatgcagatggcctcgattgttgcgaatgcctccagcgaagcgcgtgattgcctgcatcgtttttcacttgatcttggtcaggcatttcaactgctggacgatttgaccgatggcatgaccgacaccggtaaggatagcaatcaggacgccggtaaatcgacgctggtcaatctgttaggcccgagggcggttgaagaacgtctgagacaacatcttcagcttgccagtgagcatctctctgcggcctgccaacacgggcacgccactcaacattttattcaggcctggtttgacaaaaaactcgctgccgtcagttaa
(4)crtb基因序列(seqidno:4)
atgaataatccgtcgttactcaatcatgcggtcgaaacgatggcagttggctcgaaaagttttgcgacagcctcaaagttatttgatgcaaaaacccggcgcagcgtactgatgctctacgcctggtgccgccattgtgacgatgttattgacgatcagacgctgggctttcaggcccggcagcctgccttacaaacgcccgaacaacgtctgatgcaacttgagatgaaaacgcgccaggcctatgcaggatcgcagatgcacgaaccggcgtttgcggcttttcaggaagtggctatggctcatgatatcgccccggcttacgcgtttgatcatctggaaggcttcgccatggatgtacgcgaagcgcaatacagccaactggatgatacgctgcgctattgctatcacgttgcaggcgttgtcggcttgatgatggcgcaaatcatgggcgtgcgggataacgccacgctggaccgcgcctgtgaccttgggctggcatttcagttgaccaatattgctcgcgatattgtggacgatgcgcatgcgggccgctgttatctgccggcaagctggctggagcatgaaggtctgaacaaagagaattatgcggcacctgaaaaccgtcaggcgctgagccgtatcgcccgtcgtttggtgcaggaagcagaaccttactatttgtctgccacagccggcctggcagggttgcccctgcgttccgcctgggcaatcgctacggcgaagcaggtttaccggaaaataggtgtcaaagttgaacaggccggtcagcaagcctgggatcagcggcagtcaacgaccacgcccgaaaaattaacgctgctgctggccgcctctggtcaggcccttacttcccggatgcgggctcatcctccccgccctgcgcatctctggcagcgcccgctctag
(5)crti基因序列(seqidno:5)
atgaaaccaactacggtaattggtgcaggcttcggtggcctggcactggcaattcgtctacaagctgcggggatccccgtcttactgcttgaacaacgtgataaacccggcggtcgggcttatgtctacgaggatcaggggtttacctttgatgcaggcccgacggttatcaccgatcccagtgccattgaagaactgtttgcactggcaggaaaacagttaaaagagtatgtcgaactgctgccggttacgccgttttaccgcctgtgttgggagtcagggaaggtctttaattacgataacgatcaaacccggctcgaagcgcagattcagcagtttaatccccgcgatgtcgaaggttatcgtcagtttctggactattcacgcgcggtgtttaaagaaggctatctaaagctcggtactgtcccttttttatcgttcagagacatgcttcgcgccgcacctcaactggcgaaactgcaagcatggagaagcgtttacagtaaggttgccagttacatcgaagatgaacatctgcgccaggcgttttctttccactcgctgttggtgggcggcaatcccttcgccacctcatccatttatacgttgatacacgcgctggagcgtgagtggggcgtctggtttccgcgtggcggcaccggcgcattagttcaggggatgataaagctgtttcaggatctgggtggcgaagtcgtgttaaacgccagagtcagccatatggaaacgacaggaaacaagattgaagccgtgcatttagaggacggtcgcaggttcctgacgcaagccgtcgcgtcaaatgcagatgtggttcatacctatcgcgacctgttaagccagcaccctgccgcggttaagcagtccaacaaactgcaaactaagcgcatgagtaactctctgtttgtgctctattttggtttgaatcaccatcatgatcagctcgcgcatcacacggtttgtttcggcccgcgttaccgcgagctgattgacgaaatttttaatcatgatggcctcgcagaggacttctcactttatctgcacgcgccctgtgtcacggattcgtcactggcgcctgaaggttgcggcagttactatgtgttggcgccggtgccgcatttaggcaccgcgaacctcgactggacggttgaggggccaaaactacgcgaccgtatttttgcgtaccttgagcagcattacatgcctggcttacggagtcagctggtcacgcaccggatgtttacgccgtttgattttcgcgaccagcttaatgcctatcatggctcagccttttctgtggagcccgttcttacccagagcgcctggtttcggccgcataaccgcgataaaaccattactaatctctacctggtcggcgcaggcacgcatcccggcgcaggcattcctggcgtcatcggctcggcaaaagcgacagcaggtttgatgctggaggatctgatatga
本实施例中所使用的引物如下:
(1)第一轮中,用于连接crte与crtb的引物
用于扩增上游同源序列的引物(该引物扩增后,可以在crte的加上上游同源序列):
crte-h-f1:tgtcggcgtgtagtagtgaacctgttc(seqidno:6);
crte-h-r1:gcagaccgtcatctagtatttctcctcttttcttgccgctcc
cctgcatt(seqidno:7)。
用于扩增crte的引物(该引物扩增后,可以在crte的3’端加上一段crtb的5’端相同的序列片段):
crte-f:aatgcaggggagcggcaagaaaagaggagaaatactagatga
cggtctgcg(seqidno:8);
crte-r1:acgacggattattcatctagtatttctcctctttttaactgacg
gcagcgagttttttgt(seqidno:9);
crte-r2:gttatccctaaccgcatgattgagtaacgacggattattca
tctagtatttctcctcttt(seqidno:10);在crte-r1和crte-r2中下划线标示的序列用于在crte的3’端加上一段crtb的5’端相同的序列片段。
用于扩增teta及其启动子的引物(第一轮)(该引物扩增后,可以在teta的两端加上i-scei识别序列及crtb的5’端序列片段):
teta-f1:atgaataatccgtcgttactcaatcatgcggttagggataaca
gggtaatatcgttg(seqidno:11);其中,曲线下划线标示序列为crtb编码区的1-32区间,直线下划线标示序列为i-scei识别序列。
teta-r1:attattcatctagtatttctcctctttattaccctgttatccc
tactaagcacttgtctc(seqidno:12);其中,直线下划线标示序列为crtb的1-9区间的反向互补序列,曲线双下划线标示序列为i-scei识别序列。
用于扩增crtb的引物:
crtb-f1:ataacagggtaataaagaggagaaatactagatgaataat
ccgtcgttactcaatcatgc(seqidno:13);
crtb-r1:cttaaatgtgattcaacatcactggagaaagtcttctagag
cgggcgctgccag(seqidno:14);其中,下划线部分用于在crtb序列的3’端加上其下游同源序列。
用于扩增下游同源序列的引物:
crtb-h-f1:tctggcagcgcccgctctagaagactttctccagtgatgt
tgaatcacattt(seqidno:15);
crtb-h-r1:caatttcgccagacaagcagaatcaag(seqidno:16)。
(2)第二轮,用于将crti连接到crte-crtb所需的引物
第二轮用于扩增上游同源序列的引物:
teta-h-f:ctatgcaggatcgcagatgcacgaa(seqidno:17);
teta-h-r1:accgtagttggtttcatctagtatttctcctctttctagagcgggcgctgccagag(seqidno:18);
teta-h-r2:gccaccgaagcctgcaccaattaccgtagttggtttcatctagtatttctcctcttt(seqidno:19);
teta-h-r3:gtcaacgatattaccctgttatccctagccaccgaagcctgcaccaatt(seqidno:20);
用于扩增teta的引物(第二轮):
teta-f2:aattggtgcaggcttcggtggctagggataacagggtaatatcgttgacgg(seqidno:21);
teta-r2:attaccctgttatccctactaagcacttgtctc(seqidno:22)。
用于扩增crti序列:
crti-f1:gataacagggtaataaagaggagaaatactagatgaaaccaactacggtaattggtgcag(seqidno:23);
crti-f2:aaacaggagacaagtgcttagtagggataacagggtaataaagaggagaaatactagatg(seqidno:24);
crti-r1:gtgattcaacatcactggagaaagtctttcatatcagatcctccagcatcaaacct(seqidno:25);
用于扩增下游同源序列(第二轮):
crti-h-f1:aggtttgatgctggaggatctgatatgaaagactttctccagtgatgttgaatcacattt(seqidno:26);
反向引物使用crtb-h-r1。
(3)克隆到载体的引物
组装好的crte、crtb和crti基因克隆到载体pjk58所用的引物:
crtebi-f1:taccttggcaaacagctattatgggtattatgggtaaagaggagaaatactagatgacggtctgc(seqidno:27);
crtebi-r1:taggatacccataatacccataatagctgtttgccatcata
tcagatcctccagcatcaaacct(seqidno:28)。
实施例2、dna片段制备
根据图2所示,首先进行pcr,扩增得到所需要的dna序列。pcr共分为两轮。
1、第一轮
(1)、获得crte序列及其上游同源序列的融合序列
以e.coli染色体为模板,使用引物crte-h-f1和crte-h-r1扩增出crte的上游同源序列;
以pjk70为模板,使用crte-f和crte-r1、crte-r2扩增得到crte序列;
然后,通过overlap-pcr的方法,把crte序列及其上游同源序列融合在一起。
(2)、获得teta基因序列
以pucteta为模板,使用teta-f1和teta-r1扩增出teta基因。
(3)、获得crtb与其下游同源序列的融合序列
以pjk70为模板,使用crtb-f1和crtb-r1扩增出crtb序列;
以e.coli染色体为模板,使用crtb-h-f1和crtb-h-r1扩增出crtb下游同源序列;
然后,通过overlap-pcr的方法,把crtb与其下游同源序列融合在一起。
(4)、进一步融合
使用前述获得的三个pcr产物适当比例混合在一起作为模板,以crte-h-f1和crtb-h-r1分别作为前向和反向引物,采用touchdownpcr方法,将前面获得的三个pcr产物融合在一起,形成“上游同源序列+crte+teta+crtb+下游同源序列”dna序列片段。
2、第二轮
采用实施例1所列的第二轮的引物,在第一轮经过转化诱导组装筛选之后的基础上进行pcr,采用与前述类似的方法,形成“上游同源序列+teta+crti+下游同源序列”,以便进行第二轮的转化诱导组装筛选。
实施例2、转化
根据图3进行,首先将质粒ptkred转化入e.colik-12,然后使用iptg(终浓度1mm)诱导red重组系统表达。之后,制作电转化感受态,将实施例1第1轮制备的dna片段电转入,在含有四环素(终浓度10μg/ml)的平板上筛选阳性克隆,测序确定dna片段是否准确整合到e.coli染色体上设定位点。
对于正确的克隆,过夜培养,同时加入iptg(终浓度1mm)和阿拉伯糖(终浓度0.2%)诱导red重组系统和i-scei酶表达,随后取适量培养基,梯度稀释,在bma平板上进行负筛,对于长出来的克隆进行测序验证,确定crte和crtb基因无缝组装成功。
随后,使用测序正确的菌株作为宿主,使用第二轮pcr产生的dna片段,依照上述类似的方法,进行第二轮的转化诱导筛选测序验证,确定crti与crte-crtb无缝组装成功。
由于本发明人使用了优化选择的启动子和teta基因序列,在bma培养基上负筛的效率能够达到100%。负筛表型见图4。测序结果见图5。
上述结果说明,本发明的方法进行dna无缝组装是可行的,而且筛选效率是高的。
实施例3、诱导进一步验证
由于本发明人使用了crte、crtb和crti组装作为实例,这三个基因表达可以产生番茄红素,本发明人把组装好的序列克隆到载体pjk58上,转化到大肠杆菌中,使用阿拉伯糖诱导表达。
结果如图6所示,可以看到番茄红素得到了表达。
进一步地,本发明人对菌体进行裂解,并用乙酸乙酯进行抽提,进行lc-ms表征,结果显示产生番茄红素。这说明本发明人的组装方法是正确的,可以推广用的。
在本发明提及的所有文献都在本申请中引用作为参考,就如同每一篇文献被单独引用作为参考那样。此外应理解,在阅读了本发明的上述讲授内容之后,本领域技术人员可以对本发明作各种改动或修改,这些等价形式同样落于本申请所附权利要求书所限定的范围。
序列表
<110>华东理工大学
<120>体内dna无缝组装方法
<130>174587
<160>28
<170>patentinversion3.3
<210>1
<211>65
<212>dna
<213>人工序列
<220>
<221>misc_feature
<223>teta基因启动子
<400>1
atcgttgacggctagctcagtcctaggtacagtgctagctactagagaaagaggagaaat60
actag65
<210>2
<211>1206
<212>dna
<213>人工序列
<220>
<221>misc_feature
<223>teta基因序列
<400>2
atgaatagttcgacaaagatcgcattggtaattacgttactcgatgccatggggattggc60
cttatcatgccagtcttgccaacgttattacgtgaatttattgcttcggaagatatcgct120
aaccactttggcgtattgcttgcactttatgcgttaatgcaggttatctttgctccttgg180
cttggaaaaatgtctgaccgatttggtcggcgcccagtgctgttgttgtcattaataggc240
gcatcgctggattacttattgctggctttttcaagtgcgctttggatgctgtatttaggc300
cgtttgctttcagggatcacaggagctactggggctgtcgcggcatcggtcattgccgat360
accacctcagcttctcaacgcgtgaagtggttcggttggttaggggcaagttttgggctt420
ggtttaatagcggggcctattattggtggttttgcaggagagatttcaccgcatagtccc480
ttttttatcgctgcgttgctaaatattgtcactttccttgtggttatgttttggttccgt540
gaaaccaaaaatacacgtgataatacagataccgaagtaggggttgagacgcaatcaaat600
tcggtgtacatcactttatttaaaacgatgcccattttgttgattatttatttttcagcg660
caattgataggccaaattcccgcaacggtgtgggtgctatttaccgaaaatcgttttgga720
tggaatagcatgatggttggcttttcattagcgggtcttggtcttttacactcagtattc780
caagcctttgtggcaggaagaatagccactaaatggggcgaaaaaacggcagtactgctc840
ggatttattgcagatagtagtgcatttgcctttttagcgtttatatctgaaggttggtta900
gttttccctgttttaattttattggctggtggtgggatcgctttacctgcattacaggga960
gtgatgtctatccaaacaaagagtcatcagcaaggtgctttacagggattattggtgagc1020
cttaccaatgcaaccggtgttattggcccattactgtttgctgttatttataatcattca1080
ctaccaatttgggatggctggatttggattattggtttagcgttttactgtattattatc1140
ctgctatcaatgaccttcatgttgacccctcaagctcaggggagtaaacaggagacaagt1200
gcttag1206
<210>3
<211>909
<212>dna
<213>人工序列
<220>
<221>misc_feature
<223>crte基因序列
<400>3
atgacggtctgcgcaaaaaaacacgttcatctcactcgcgatgctgcggagcagttactg60
gctgatattgatcgacgccttgatcagttattgcccgtggagggagaacgggatgttgtg120
ggtgccgcgatgcgtgaaggtgcgctggcaccgggaaaacgtattcgccccatgttgctg180
ttgctgaccgcccgcgatctgggttgcgctgtcagccatgacggattactggatttggcc240
tgtgcggtggaaatggtccacgcggcttcgctgatccttgacgatatgccctgcatggac300
gatgcgaagctgcggcgcggacgccctaccattcattctcattacggagagcatgtggca360
atactggcggcggttgccttgctgagtaaagcctttggcgtaattgccgatgcagatggc420
ctcacgccgctggcaaaaaatcgggcggtttctgaactgtcaaacgccatcggcatgcaa480
ggattggttcagggtcagttcaaggatctgtctgaaggggataagccgcgcagcgctgaa540
gctattttgatgacgaatcactttaaaaccagcacgctgttttgtgcctccatgcagatg600
gcctcgattgttgcgaatgcctccagcgaagcgcgtgattgcctgcatcgtttttcactt660
gatcttggtcaggcatttcaactgctggacgatttgaccgatggcatgaccgacaccggt720
aaggatagcaatcaggacgccggtaaatcgacgctggtcaatctgttaggcccgagggcg780
gttgaagaacgtctgagacaacatcttcagcttgccagtgagcatctctctgcggcctgc840
caacacgggcacgccactcaacattttattcaggcctggtttgacaaaaaactcgctgcc900
gtcagttaa909
<210>4
<211>930
<212>dna
<213>人工序列
<220>
<221>misc_feature
<223>crtb基因序列
<400>4
atgaataatccgtcgttactcaatcatgcggtcgaaacgatggcagttggctcgaaaagt60
tttgcgacagcctcaaagttatttgatgcaaaaacccggcgcagcgtactgatgctctac120
gcctggtgccgccattgtgacgatgttattgacgatcagacgctgggctttcaggcccgg180
cagcctgccttacaaacgcccgaacaacgtctgatgcaacttgagatgaaaacgcgccag240
gcctatgcaggatcgcagatgcacgaaccggcgtttgcggcttttcaggaagtggctatg300
gctcatgatatcgccccggcttacgcgtttgatcatctggaaggcttcgccatggatgta360
cgcgaagcgcaatacagccaactggatgatacgctgcgctattgctatcacgttgcaggc420
gttgtcggcttgatgatggcgcaaatcatgggcgtgcgggataacgccacgctggaccgc480
gcctgtgaccttgggctggcatttcagttgaccaatattgctcgcgatattgtggacgat540
gcgcatgcgggccgctgttatctgccggcaagctggctggagcatgaaggtctgaacaaa600
gagaattatgcggcacctgaaaaccgtcaggcgctgagccgtatcgcccgtcgtttggtg660
caggaagcagaaccttactatttgtctgccacagccggcctggcagggttgcccctgcgt720
tccgcctgggcaatcgctacggcgaagcaggtttaccggaaaataggtgtcaaagttgaa780
caggccggtcagcaagcctgggatcagcggcagtcaacgaccacgcccgaaaaattaacg840
ctgctgctggccgcctctggtcaggcccttacttcccggatgcgggctcatcctccccgc900
cctgcgcatctctggcagcgcccgctctag930
<210>5
<211>1479
<212>dna
<213>人工序列
<220>
<221>misc_feature
<223>crti基因序列
<400>5
atgaaaccaactacggtaattggtgcaggcttcggtggcctggcactggcaattcgtcta60
caagctgcggggatccccgtcttactgcttgaacaacgtgataaacccggcggtcgggct120
tatgtctacgaggatcaggggtttacctttgatgcaggcccgacggttatcaccgatccc180
agtgccattgaagaactgtttgcactggcaggaaaacagttaaaagagtatgtcgaactg240
ctgccggttacgccgttttaccgcctgtgttgggagtcagggaaggtctttaattacgat300
aacgatcaaacccggctcgaagcgcagattcagcagtttaatccccgcgatgtcgaaggt360
tatcgtcagtttctggactattcacgcgcggtgtttaaagaaggctatctaaagctcggt420
actgtcccttttttatcgttcagagacatgcttcgcgccgcacctcaactggcgaaactg480
caagcatggagaagcgtttacagtaaggttgccagttacatcgaagatgaacatctgcgc540
caggcgttttctttccactcgctgttggtgggcggcaatcccttcgccacctcatccatt600
tatacgttgatacacgcgctggagcgtgagtggggcgtctggtttccgcgtggcggcacc660
ggcgcattagttcaggggatgataaagctgtttcaggatctgggtggcgaagtcgtgtta720
aacgccagagtcagccatatggaaacgacaggaaacaagattgaagccgtgcatttagag780
gacggtcgcaggttcctgacgcaagccgtcgcgtcaaatgcagatgtggttcatacctat840
cgcgacctgttaagccagcaccctgccgcggttaagcagtccaacaaactgcaaactaag900
cgcatgagtaactctctgtttgtgctctattttggtttgaatcaccatcatgatcagctc960
gcgcatcacacggtttgtttcggcccgcgttaccgcgagctgattgacgaaatttttaat1020
catgatggcctcgcagaggacttctcactttatctgcacgcgccctgtgtcacggattcg1080
tcactggcgcctgaaggttgcggcagttactatgtgttggcgccggtgccgcatttaggc1140
accgcgaacctcgactggacggttgaggggccaaaactacgcgaccgtatttttgcgtac1200
cttgagcagcattacatgcctggcttacggagtcagctggtcacgcaccggatgtttacg1260
ccgtttgattttcgcgaccagcttaatgcctatcatggctcagccttttctgtggagccc1320
gttcttacccagagcgcctggtttcggccgcataaccgcgataaaaccattactaatctc1380
tacctggtcggcgcaggcacgcatcccggcgcaggcattcctggcgtcatcggctcggca1440
aaagcgacagcaggtttgatgctggaggatctgatatga1479
<210>6
<211>27
<212>dna
<213>人工序列
<220>
<221>misc_feature
<223>引物
<400>6
tgtcggcgtgtagtagtgaacctgttc27
<210>7
<211>50
<212>dna
<213>人工序列
<220>
<221>misc_feature
<223>引物
<400>7
gcagaccgtcatctagtatttctcctcttttcttgccgctcccctgcatt50
<210>8
<211>51
<212>dna
<213>人工序列
<220>
<221>misc_feature
<223>引物
<400>8
aatgcaggggagcggcaagaaaagaggagaaatactagatgacggtctgcg51
<210>9
<211>60
<212>dna
<213>人工序列
<220>
<221>misc_feature
<223>引物
<400>9
acgacggattattcatctagtatttctcctctttttaactgacggcagcgagttttttgt60
<210>10
<211>60
<212>dna
<213>人工序列
<220>
<221>misc_feature
<223>引物
<400>10
gttatccctaaccgcatgattgagtaacgacggattattcatctagtatttctcctcttt60
<210>11
<211>57
<212>dna
<213>人工序列
<220>
<221>misc_feature
<223>引物
<400>11
atgaataatccgtcgttactcaatcatgcggttagggataacagggtaatatcgttg57
<210>12
<211>60
<212>dna
<213>人工序列
<220>
<221>misc_feature
<223>引物
<400>12
attattcatctagtatttctcctctttattaccctgttatccctactaagcacttgtctc60
<210>13
<211>60
<212>dna
<213>人工序列
<220>
<221>misc_feature
<223>引物
<400>13
ataacagggtaataaagaggagaaatactagatgaataatccgtcgttactcaatcatgc60
<210>14
<211>54
<212>dna
<213>人工序列
<220>
<221>misc_feature
<223>引物
<400>14
cttaaatgtgattcaacatcactggagaaagtcttctagagcgggcgctgccag54
<210>15
<211>52
<212>dna
<213>人工序列
<220>
<221>misc_feature
<223>引物
<400>15
tctggcagcgcccgctctagaagactttctccagtgatgttgaatcacattt52
<210>16
<211>27
<212>dna
<213>人工序列
<220>
<221>misc_feature
<223>引物
<400>16
caatttcgccagacaagcagaatcaag27
<210>17
<211>25
<212>dna
<213>人工序列
<220>
<221>misc_feature
<223>引物
<400>17
ctatgcaggatcgcagatgcacgaa25
<210>18
<211>56
<212>dna
<213>人工序列
<220>
<221>misc_feature
<223>引物
<400>18
accgtagttggtttcatctagtatttctcctctttctagagcgggcgctgccagag56
<210>19
<211>57
<212>dna
<213>人工序列
<220>
<221>misc_feature
<223>引物
<400>19
gccaccgaagcctgcaccaattaccgtagttggtttcatctagtatttctcctcttt57
<210>20
<211>49
<212>dna
<213>人工序列
<220>
<221>misc_feature
<223>引物
<400>20
gtcaacgatattaccctgttatccctagccaccgaagcctgcaccaatt49
<210>21
<211>51
<212>dna
<213>人工序列
<220>
<221>misc_feature
<223>引物
<400>21
aattggtgcaggcttcggtggctagggataacagggtaatatcgttgacgg51
<210>22
<211>33
<212>dna
<213>人工序列
<220>
<221>misc_feature
<223>引物
<400>22
attaccctgttatccctactaagcacttgtctc33
<210>23
<211>60
<212>dna
<213>人工序列
<220>
<221>misc_feature
<223>引物
<400>23
gataacagggtaataaagaggagaaatactagatgaaaccaactacggtaattggtgcag60
<210>24
<211>60
<212>dna
<213>人工序列
<220>
<221>misc_feature
<223>引物
<400>24
aaacaggagacaagtgcttagtagggataacagggtaataaagaggagaaatactagatg60
<210>25
<211>56
<212>dna
<213>人工序列
<220>
<221>misc_feature
<223>引物
<400>25
gtgattcaacatcactggagaaagtctttcatatcagatcctccagcatcaaacct56
<210>26
<211>60
<212>dna
<213>人工序列
<220>
<221>misc_feature
<223>引物
<400>26
aggtttgatgctggaggatctgatatgaaagactttctccagtgatgttgaatcacattt60
<210>27
<211>65
<212>dna
<213>人工序列
<220>
<221>misc_feature
<223>引物
<400>27
taccttggcaaacagctattatgggtattatgggtaaagaggagaaatactagatgacgg60
tctgc65
<210>28
<211>64
<212>dna
<213>人工序列
<220>
<221>misc_feature
<223>引物
<400>28
taggatacccataatacccataatagctgtttgccatcatatcagatcctccagcatcaa60
acct64
- 还没有人留言评论。精彩留言会获得点赞!