果胶酸裂解酶PL1W及其基因和应用的制作方法
本发明属于农业生物技术领域,具体涉及一种果胶酸裂解酶pl1w及其基因和应用。
背景技术:
根据不同的作用底物类型,果胶酶可分为原果胶酶(protopectinase)、果胶酯酶(pectinesterase)、果胶解聚酶(pectindepolymerase)三种。原果胶酶主要作用于原果胶,其可将不溶性的果胶水解为水溶性的果胶物质。果胶酯酶主要作用于酯化程度高的果胶,其可以脱去果胶主链中的甲酯基团,产生果胶酸和甲醇。果胶解旋酶作用于果胶酸和果胶质酸,可将主链糖或侧链糖水解成寡糖或者单糖。根据不同的水解机制,果胶酶又可分为果胶水解酶(pectinhydrolase)和果胶裂解酶(pectinlyase)两类。果胶裂解酶的解旋不需要水分子参与,而是发生反式消去反应,消去一个h原子,从而断裂α-1,4-糖苷键并产生一个不饱和的双键。
根据糖苷酶数据库cazy(carbohydrate-activeenzymesdatabase)中酶氨基酸序列和结构的划分可将果胶酶分为糖苷水解酶(glycosidehydrolases;ghs)、多糖裂解酶(polysaccharidelyases;pls)、糖酯酶(carbohydrateesterases;ces)三类。对于糖苷水解酶来说,gh28家族包括了几乎全部的果胶解旋酶,如内切多聚半乳糖醛酸酶、外切多聚半乳糖醛酸酶、鼠李半乳糖醛酸酶、木糖基半乳糖醛酸酶、阿拉伯聚糖酶等。多糖裂解酶家族中,pl1家族中有3种果胶裂解酶,果胶酸裂解酶(ec4.2.2.2)、外切果胶酸裂解酶(ec4.2.2.9)、果胶裂解(ec4.2.2.10);pl2家族中有2种,果胶酸裂解酶(ec4.2.2.2)、外切果胶酸裂解酶(ec4.2.2.9);pl3家族中只有1种果胶酸裂解酶(ec4.2.2.2);pl9家族中有1种,果胶酸裂解酶(ec4.2.2.2)、外切果胶酸裂解酶(ec4.2.2.9);pl11家族中2种,鼠李糖半乳糖醛酸裂解酶(ec4.2.2.-);外切型鼠李糖半乳糖醛酸裂解酶(ec4.2.2.-)。
近几年来,随着人们对果胶结构的不断深入研究和新的认识,裂解酶对降解果胶的独特作用被重新认识和应用。果胶裂解酶是果胶酶中唯一一种能够降解高酯化度果胶且不产生甲醇的酶类,根据酶作用底物环境的酸碱性,可分为酸性果胶裂解酶和碱性果胶裂解酶两类。其广泛地应用于食品工业、纺织工业、造纸业、含果胶类物质的污水处理等。
技术实现要素:
本发明的目的在于提供一种果胶裂解酶pl1w。
本发明的再一目的在于提供一种果胶裂解酶pl1w基因。
本发明的再一目的在于提供包含果胶裂解酶pl1w基因的重组表达载体。
本发明的再一目的在于提供包含果胶裂解酶pl1w基因的重组菌株。
本发明的再一目的在于提供上述果胶裂解酶pl1w的制备方法。
本发明的再一目的在于提供上述果胶裂解酶pl1w的应用。
根据本发明具体实施方式的果胶酸裂解酶pl1w,其氨基酸序列如seqidno.1所示:
其中,该酶全长249个氨基酸,n端16个氨基酸为信号肽序列“mhsiltlivtaglvta”。
因此,成熟的果胶酸裂解酶的理论分子量为25.7kda,其氨基酸序列如seqidno.2所示:
本发明还提供了编码上述果胶酸裂解酶pl1w的基因,该酶的全基因序列如seqidno.3所示:
本发明通过pcr的方法分离克隆了这个果胶酸裂解酶基因,dna全序列分析结果表明,果胶酸裂解酶结构基因全长840bp,含有1个内含子,371~411bp为其内含子序列,cdna长747bp,其cdna序列如seqidno.4所示:
其中,信号肽的碱基序列为:
“caaaccaacacgaacctccaaaccaagttccccacggccaccagcacg”
因此,成熟基因的编码序列为seqidno.5所示:
本发明还提供了包含上述果胶酸裂解酶基因的重组载体,优选为ppic9-pl1w。将本发明的果胶酸裂解酶基因插入到表达载体合适的限制性酶切位点之间,使其核苷酸序列可操作的与表达调控序列相连接。作为本发明的一个最优选的实施方案,优选为将果胶酸裂解酶基因插入到质粒ppic9上的ecori和noti限制性酶切位点之间,使该核苷酸序列位于aoxl启动子的下游并受其调控,得到重组酵母表达质粒ppic9-pl1w。
本发明还提供了包含上述果胶酸裂解酶基因的重组菌株,优选为重组菌株gs115/pl1w。
本发明还提供了一种制备果胶酸裂解酶的方法,包括以下步骤:
1)用上述重组载体转化宿主细胞,得重组菌株;
2)培养重组菌株,诱导重组果胶酸裂解酶的表达;
3)回收并纯化所表达的果胶酸裂解酶。
其中,优选所述宿主细胞为毕赤酵母(pichiapastoris)细胞,优选将重组酵母表达质粒转化毕赤酵母细胞(pichicpastoris)gs115,得到重组菌株gs115/pl1w。本发明还提供了上述果胶酸裂解酶的应用,运用基因工程手段来产业化生产果胶酸裂解酶。
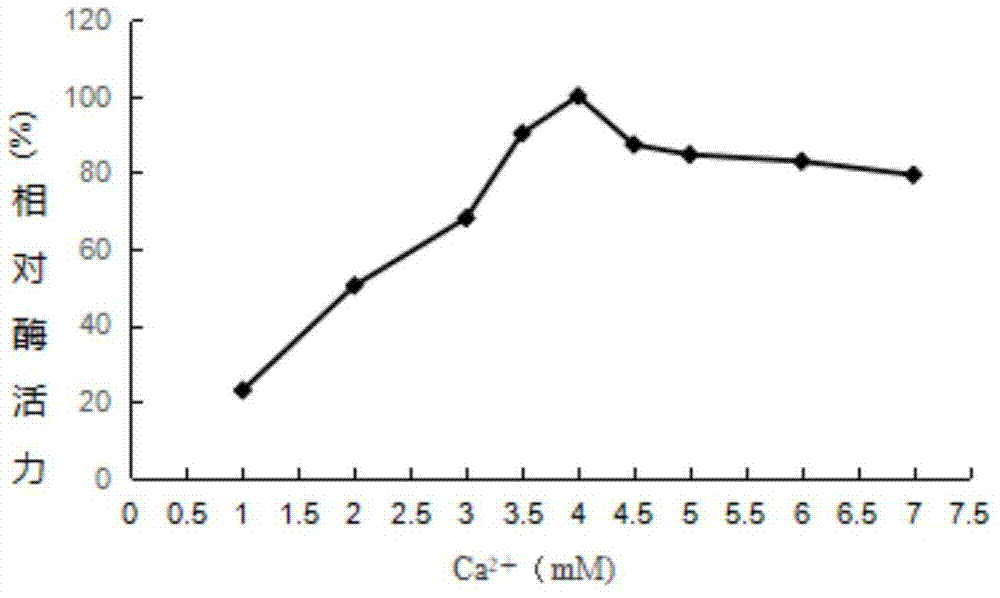
本发明的果胶酸裂解酶pl1w具有ca2+依赖性,当反应体系中ca2+浓度达到4mm时,酶活力达到最高值,在ca2+浓度超过4mm时酶活力呈现下降趋势,ca2+浓度过高会对酶的催化有抑制作用。果胶酸裂解酶pl1w的最适ph为10.0,在ph8~10范围内30℃温度下处理1h,酶活仍有90%以上的酶活;其最适温度为55℃,在30℃时依然具有约63%的酶活力,45℃处理处理60min剩余67%以上的酶活。
本发明的新的果胶酸裂解酶基因,可应用于饲料、食品、医药等工业。根据本发明的技术方案可以实现利用基因工程手段生产性质优良适合工业应用的果胶酸裂解酶。
附图说明
图1显示ca2+对果胶酸裂解酶pl1w酶活力的影响情况;
图2显示果胶酸裂解酶pl1w的最适ph;
图3显示果胶酸裂解酶pl1w的ph稳定性情况;
图4显示果胶酸裂解酶pl1w的最适温度;
图5显示果胶酸裂解酶pl1w的温度稳定性情况。
具体实施方式
试验材料和试剂
1、菌株及载体:毕赤酵母(pichiapastorisgs115)及毕赤酵母表达载体ppic9;
2、培养基:
(1)ypd酵母培养基:酵母粉10g/l,蛋白胨20g/l,葡萄糖20g/l,115℃,灭菌处理30min。
(2)lb大肠杆菌培养基:酵母提取物5g/l,蛋白胨10g/l,nacl10g/l,ph7.0。
(3)bmgy培养基:酵母提取物10g/l,蛋白胨20g/l,ynb13.4g/l,生物素4mg/l,1%甘油。
(4)bmmy培养基:酵母提取物10g/l,蛋白胨20g/l,ynb13.4g/l,生物素4mg/l,0.5%甲醇(v/v)。
(5)md固体培养基:琼脂糖20g/l,葡萄糖20g/l,ynb13.4g/l,生物素4mg/l。
实施例1克隆果胶酸裂解酶编码基因pl1w
1.提取humicolay1基因组dna
将液体培养3天的菌,12,000rpm离心10min,收集的菌丝体加入已高温灭菌的研钵中,用液氮迅速研磨至粉末,然后将研磨好的菌体转移至一个新的、装有15mlctab裂解液50ml离心管中,轻柔上下倒置混匀,置于65℃水浴锅保温3h,每隔20min,上下倒置轻柔混匀一次,以便充分裂解菌体。4℃、12,000rpm离心10min,吸取上清至新的离心管中,加入等体积的氯仿抽提,室温放置5min。4℃、12,000rpm离心10min。取上清再加入等体积的酚/氯仿抽提,室温放置5min。4℃、12,000rpm离心10min。以便尽量除去杂蛋白,再取上清加入等体积异丙醇,于室温静置5min后,4℃下l0000rpm离心l0min。弃上清,沉淀用70%的乙醇洗涤两次,真空干燥,加入适量ddh2o溶解,置于-20℃备用。
以humicolay1总dna为模板,以pl1w-f和pl1w-r为引物进行pcr扩增如表1所示。pcr反应参数为:95℃5min;94℃30sec,60℃30sec,72℃30sec,30个循环,得到一约800bp片段,将该片段回收测序,得到果胶酸裂解酶pl1w的基因组序列。
表1本发明的引物
2.果胶酸裂解酶cdna的获得
提取humicolay1总rna,利用oligo(dt)20和反转录酶得到cdna的一条链,然后以pl1w-f和pl1w-r,扩增该单链cdna,获得果胶酸裂解酶的cdna序列,扩增得到产物回收测序,得到果胶酸裂解酶pl1w的cdna序列。
通过对果胶酸裂解酶pl1w的基因组序列和cdna序列比对后发现,基因组序中含有1个内含子,cdna长747bp,编码248个氨基酸和一个终止密码子,n端16个氨基酸为其信号肽序列,经比对证明从humicolay1中分离克隆得到的编码果胶酸裂解酶的基因为新基因。
实施例2构建果胶酸裂解酶工程菌株的构建
1.表达载体的构建及表达
以测序正确的果胶酸裂解酶pl1w的cdna为模板,以带有ecori和noti限制性酶切位点的引物pl1w-f和pl1w-r对pl1w的成熟蛋白的编码区进行扩增,并利用ecori和noti酶切pcr产物,连接进入表达载体ppic9,果胶酸裂解酶pl1w成熟蛋白的序列插入到上述表达载体的信号肽序列的下游,与信号肽形成正确的阅读框架,构建成酵母表达载体ppic9-pl1w,转化大肠杆菌感受态细胞trans1。挑取阳性转化子进行dna测序,测序表明序列正确的转化子用于大量制备重组质粒。用限制性内切酶bglii进行线性化表达质粒载体dna,电击转化酵母gs115感受态细胞,30℃培养2~3天,挑取在md平板上生长的转化子进行进一步的表达实验。
2.筛选果胶酸裂解酶高活性转化子
用灭过菌的牙签从长有转化子的md板上挑取单菌落,按照编号先点到md平板上,将md平板置于30℃培养箱中培养2~3天,至菌落长出。按编号从md平板上挑取转化子接种于装有3mlbmgy培养基的离心管中,30℃、220rpm摇床培养48h;将摇床培养48h的菌液4500rmp离心10min,去上清,离心管中再加入1ml含有0.5%甲醇的bmmy培养基,在30℃、220rpm诱导培养;诱导培养48h后,12000rmp离心10min,取上清用于酶活性检测,从中筛选出高果胶酸裂解酶活性的转化子。
实施例3重组果胶酸裂解酶的制备
1.果胶酸裂解酶pl1w在毕赤酵母中摇瓶水平的大量表达
筛选出酶活较高的转化子,接种ypd培养基经48h活化后,按4%的接种量转接于300mlbmgy液体培养基的1l三角瓶中,30℃,220rpm摇床振荡培养48h;5,000rpm离心5min,轻柔弃上清,再向菌体加入200ml含有0.5%甲醇的bmmy液体培养基,30℃,220rpm诱导培养72h。诱导培养期间,间隔24h补加一次甲醇溶液以补偿甲醇的损失,使甲醇浓度保持在0.5%左右;12,000rpm离心10min,收集上清发酵液,检测酶活性并进行sds-page蛋白电泳分析。
2.果胶酸裂解酶pl1w的纯化
收集摇瓶表达的重组果胶酸裂解酶pl1w上清液,通过5kda膜包进行浓缩,同时用低盐缓冲液置换其中的培养基,然后用3kda超滤管进一步的浓缩。浓缩能稀释到一定倍数的重组果胶酸裂解酶pl1w,通过离子交换层析进行纯化。具体地,取pl1w浓缩液2.0ml经预先用10mm甘氨酸-氢氧化钠(ph10.0)平衡过的histraptmqhp阴离子柱,然后用0~1m的nacl进行线性梯度洗脱,对分步收集的洗脱液检测酶活性和进行蛋白电泳的检测。
实施例4果胶酸裂解酶pl1w的性质分析
采用hcl终止法对果胶酸裂解酶pl1w进行活性分析。具体方法如下:
在ph10.0,55℃条件下,3.0ml的反应体系包括l00μl适当的稀释酶液,2.9ml底物,反应30min,加入1.0ml2mhcl终止反应,混匀后。冷却后235nm测定od值。果胶酸裂解酶活性单位定义:在一定条件下,每分钟作用果胶生成lμmol双键所需的酶量为1个活性单位(u)。
1.ca2+对果胶酸裂解酶pl1w酶活力的影响
经纯化的果胶酸裂解酶pl1w在ph10.0、55℃条件下,在反应体系中分别添加ca2+的浓度为1.0mm、2.0mm、3.0mm、3.5mm、4.0mm、4.5mm、5.0mm、6.0mm、7.0mm,测定的ca2+依赖性适性结果。
如图1所示,当反应体系中ca2+浓度达到4.0mm时,酶活力达到最高值,当ca2+浓度超过4.0mm时酶活力呈现下降趋势,因此,ca2+浓度过高会对酶的催化有轻微的抑制作用。
2.果胶酸裂解酶pl1w的最适ph及ph稳定性测定
纯化的果胶酸裂解酶pl1w在不同的ph下进行酶促反应以测定其最适ph。所用缓冲液为ph1.0~3.0甘氨酸-盐酸缓冲液,ph3.0~8.0的柠檬酸-磷酸氢二钠缓冲液,ph8.0~9.0tris-盐酸缓冲液,ph9.0~12.0甘氨酸-氢氧化钠缓冲液。所用底物为0.2%的多聚半乳糖醛酸pga(polygalacturonicacid)。
如图2所示,果胶酸裂解酶pl1w在55℃温度下,其最适ph为10.0,呈碱性。
将酶液在不同ph值的缓冲液中于37℃下孵育1h,再测定酶活性以研究酶的ph稳定性。所用缓冲液为ph1.0~3.0甘氨酸-盐酸缓冲液,ph3.0~8.0柠檬酸-磷酸氢二钠缓冲液,ph8.0~9.0tris-盐酸缓冲液,ph9.0~12.0甘氨酸-氢氧化钠缓冲液。
如图3所示,结果表明在ph8.0~10.0范围内37℃处理1h,果胶酸裂解酶仍有90%以上的酶活。
3.果胶酸裂解酶pl1w反应最适温度及热稳定性
纯化的果胶酸裂解酶pl1w在ph10.0条件下,测定不同温度(40℃~80℃)下的酶活性。
如图4所示,果胶裂解酶的最适反应温度为55℃,在50℃时依然具有79%以上的酶活力。
耐温性测定为果胶酸裂解酶pl1w在不同温度下处理不同时间,再在55℃下进行酶活性测定。
如图5所示,果胶酸裂解酶pl1w在55℃下处理60min,剩余酶活在18%以上;果胶裂解酶pl1w在60℃下处理15min,依然能够保持27%的酶活力。
序列表
<110>中国农业科学院饲料研究所
<120>果胶酸裂解酶pl1w及其基因和应用
<160>5
<170>siposequencelisting1.0
<210>1
<211>248
<212>prt
<213>腐质霉y1(humicolay1)
<400>1
methisserileleuthrleuilevalthralaglyleuvalthrala
151015
glnthrasnthrasnleuglnthrlyspheprothralathrserthr
202530
thrglyileproalaalaargthrilealaalaglythrserpheasp
354045
glyglnmetleuglntrpaspargserproserthrcyslysaspgln
505560
thrgluglyglyglulysaspalaleuphevalleugluasnglyala
65707580
thrleuserasnvalilevalglyproglyalaglygluglyiletyr
859095
cyslysglysercysthrleuasnasnvaltrptrpthraspvalcys
100105110
gluaspalaalathrpheleuglnserserglythrsertyrvalasn
115120125
glyglyglyalaargasnalaalaasplysvalpheglnhisasngly
130135140
glyglythrvalservallysasnpheleuserserlysasnglylys
145150155160
iletyrargsercysglyasncyslysthrglnvalalaarglysser
165170175
ilephegluasnileargalaileaspserleuservalvalalagly
180185190
valasnalaasnpheglyaspserthrthrilelysasnsercysleu
195200205
leulysglyargthrcysaspilephegluglyasnasnserglylys
210215220
gluprothrlysileglythrglyproserglythralacysvalval
225230235240
argalavalglnthrserglycys
245
<210>2
<211>232
<212>prt
<213>腐质霉y1(humicolay1)
<400>2
glnthrasnthrasnleuglnthrlyspheprothralathrserthr
151015
thrglyileproalaalaargthrilealaalaglythrserpheasp
202530
glyglnmetleuglntrpaspargserproserthrcyslysaspgln
354045
thrgluglyglyglulysaspalaleuphevalleugluasnglyala
505560
thrleuserasnvalilevalglyproglyalaglygluglyiletyr
65707580
cyslysglysercysthrleuasnasnvaltrptrpthraspvalcys
859095
gluaspalaalathrpheleuglnserserglythrsertyrvalasn
100105110
glyglyglyalaargasnalaalaasplysvalpheglnhisasngly
115120125
glyglythrvalservallysasnpheleuserserlysasnglylys
130135140
iletyrargsercysglyasncyslysthrglnvalalaarglysser
145150155160
ilephegluasnileargalaileaspserleuservalvalalagly
165170175
valasnalaasnpheglyaspserthrthrilelysasnsercysleu
180185190
leulysglyargthrcysaspilephegluglyasnasnserglylys
195200205
gluprothrlysileglythrglyproserglythralacysvalval
210215220
argalavalglnthrserglycys
225230
<210>3
<211>839
<212>dna
<213>腐质霉y1(humicolay1)
<400>3
atgcattccatcctgaccctcatcgtcactgccggccttgtgacggcccaaaccaacacg60
aacctccaaaccaagttccccacggccaccagcacgactggtattcccgcagcgcgcaca120
attgctgctggaactagctttgatggccaaatgctccagtgggaccgcagcccgtcgacc180
tgcaaagatcaaaccgagggcggcgagaaggatgccctgttcgtcctcgagaatggtgct240
actttgagcaacgtgattgttggccccggggctggtgagggtatctattgcaaggggtcg300
tgcactctgaataacgtgtaagtctctctcaacagaatcttagaacgtgaaactctctcc360
cctgattcgcatgcgtgagtatactgggcctaaccaattgtttccaatagatggtggacc420
gacgtctgcgaagacgccgcaaccttcctccaatcctcgggcacgtcgtacgtcaatggc480
ggcggcgcccgcaacgcagccgacaaggtcttccagcacaacggcggcggcaccgtctcg540
gtcaagaatttcctgtcctccaagaacggcaagatctaccgctcctgcggcaactgcaag600
actcaggtcgcgcgcaagtccatcttcgagaacatccgggccatcgactccctctcggtg660
gttgcgggcgtgaatgccaactttggtgactcgacgacgatcaagaactcgtgcttgctc720
aaggggcggacgtgcgatatctttgaggggaacaatagcggaaaggagccgactaagatt780
ggaacgggccgtcgggaacggcgtgcgtcgtgcgggcggtgcagacttcggggtgctga839
<210>4
<211>745
<212>dna
<213>腐质霉y1(humicolay1)
<400>4
atgcattccatcctgaccctcatcgtcactgccggccttgtgacggcccaaaccaacacg60
aacctccaaaccaagttccccacggccaccagcacgactgtattcccgcagcgcgcacaa120
ttgctgctggaactagctttgatggccaaatgctccagtgggaccgcagcccgtcgacct180
gcaaagatcaaaccgagggcggcgagaaggatgccctgttcgtcctcgagaatggtgcta240
ctttgagcaacgtgattgttggccccggggctggtgagggtatctattgcaaggggtcgt300
gcactctgaataacgtatggtggaccgacgtctgcgaagacgccgcaaccttcctccaat360
cctcgggcacgtcgtacgtcaatggcggcggcgcccgcaacgcagccgacaaggtcttcc420
agcacaacggcggcggcacgtctcggtcaagaatttcctgtcctccaagaacggcaagat480
ctaccgctcctgcggcaactgcaagactcaggtcgcgcgcaagtccatcttcgagaacat540
ccgggccatcgactccctctcggtggttgcgggcgtgaatgccaactttggtgactcgac600
gacgatcaagaactcgtgcttgctcaaggggcggacgtgcgatatctttgaggggaacaa660
tagcggaaaggagccgactaagattggaacgggcccgtcgggaacggcgtgcgtcgtgcg720
ggcggtgcagacttcggggtgctga745
<210>5
<211>698
<212>dna
<213>腐质霉y1(humicolay1)
<400>5
caaaccaacacgaacctccaaaccaagttccccacggccaccagcacgactggtattccc60
gcagcgcgcacaattgctgctggaactagctttgatggccaaatgctccagtgggaccgc120
agcccgtcgacctgcaaagatcaaaccgagggcggcgagaaggatgccctgttcgtcctc180
gagaatggtgctactttgagcaacgtgattgttggccccggggctggtgagggtatctat240
tgcaaggggtcgtgcactctgaataacgtatggtggaccgacgtctgcgaagacgccgca300
accttcctccaatcctcgggcacgtcgtacgtcaatggcggcggcgcccgcaacgcagcc360
gacaaggtcttccagcacaacggcggcggcccgtctcggtcaagaatttcctgtcctcca420
agaacggcaagatctaccgctcctgcggcaactgcaagactcaggtcgcgcgcaagtcca480
tcttcgagaacatccgggccatcgactccctctcggtggttgcgggcgtgaatgccaact540
ttggtgactcgacgacgatcaagaactcgtgcttgctcaaggggcggacgtgcgatatct600
ttgaggggaacaatagcggaaaggagccgactaagattggaacgggcccgtcgggaacgg660
cgtgcgtcgtgcgggcggtgcagacttcggggtgctga698
- 还没有人留言评论。精彩留言会获得点赞!