测定土壤样品DNA提取率的方法与流程
本发明属于土壤生物化学领域,具体涉及一种适用于所有土壤样品dna提取率测定的方法。
背景技术:
土壤微生物(细菌、真菌和古菌等)是土壤生态系统中一类非常重要的生命体。这类微生物种类繁多、数量庞大、功能多样,在土壤有机质的降解、土壤养分的转化与循环、作物的生长与发育、温室气体的排放、污染物的固定和降解等方面起着重要作用。然而,土壤中绝大多数微生物是不可培养的,利用传统的分离培养方法只能得到少数(0.1%-10%)微生物的信息,不能完全反应土壤环境中微生物群落结构的组成、多样性和功能特征。当前,基于土壤微生物dna的分子生物学技术,如实时荧光定量核酸扩增检测系统(qpcr)、扩增子测序(16srrna和its)、宏基因组测序、稳定性同位素核酸探针技术(dna-sip)等正在迅猛发展,已经在土壤微生物领域得到广泛应用,很大程度上解决了土壤微生物不可分离培养这样一个瓶颈,在解析土壤微生物丰度、多样性和功能上起着至关重要的作用。
但是,这些分子生物学技术还存在一些问题,前期土壤dna的提取效果好坏对下游的扩增和测序等步骤影响较大,最终获取的微生物信息(丰度、多样性和功能数据)可能存在一些偏倚。研究表明,土壤dna的提取方法和土壤样品性质均会影响土壤dna的提取效果。不同的提取方法(试剂盒、细胞裂解时间和力度不同等)会造成提取dna的数量和质量有所差异。通常,为了解决这个偏倚,可以通过采用统一的提取方法对所有待测的样品进行统一处理;然而,土壤具有高度异质性,不同类型的土壤样品组分、ph、有机质含量、矿物含量差异很大,对dna的吸附能力不同,导致dna的提取效果也存在差异。黏粒土壤对dna的吸附作用大于沙质土壤,而不同类型的有机质对dna的吸附能力也大不相同,这些吸附能力与土壤的比表面积、有机官能团和离子交换性能密切相关。土壤对dna吸附能力的差异决定了土壤dna的提取效果,进一步影响下游分析的准确度。
通常,土壤样品提取所得的dna数量可用qpcr的目的基因拷贝数表示(可作为土壤样品微生物丰度的指标之一)。不同土壤样品对dna吸附性能差异越小,提取所得的dna数量(提取率)差异也就越小,可比性越强;不同土壤样品对dna的吸附性能差异越大,说明不同土壤类型对目的基因的吸附能力差异很大,结果可比性就越低;同时,样品提取的dna数量还会影响扩增子和宏基因组测序的结果,若提取的dna数量偏少,一些本身数量偏少但是在土壤生态系统中具有重要作用的稀有微生物就不容易被检测出来,导致最终得到的微生物信息存在偏倚。目前,人们只能测定提取之后土壤dna的浓度,而土壤样品原始的dna浓度没法测定,因此就无法计算到底有多少dna在浸提过程中被土壤样品所吸附。
技术实现要素:
本发明要解决的技术问题是提供一种快速、高效且适用于所有土壤样品dna提取率测定的方法。
为了解决上述技术问题,本发明提供aliivibriofischeri菌株dna的用途:利用aliivibriofischeri菌株dna测定土壤样品的dna提取率。
本发明还同时提供了一种测定土壤样品dna提取率的方法,包括如下步骤:
1)、培养aliivibriofischeriatcc7744t菌株(简称a.fischeri),提取其dna,作为测定土壤样品dna提取率的内标;
2)、设计aliivibriofischeri中16srrna基因片段的特异性引物(并检验其特异性);
3)、先在土壤样品中加入内标的dna,随后立即提取dna(即,在研磨均质和细胞裂解步骤之前,在土壤样品中加入内标的dna);
4)、采用qpcr测定添加到土壤中内标的基因数量和提取之后土壤样品中内标的基因数量,计算dna提取率。
作为本发明的测定土壤样品dna提取率的方法的改进:
所述步骤2)中的特异性引物为:
正向引物af-f:5'-gcggaaacgacttaactgaacc-3'
反向引物af-r:5'-gaaggtccccctctttggtc-3'。
作为本发明的测定土壤样品dna提取率的方法的进一步改进:
内标dna的添加量为土壤样品总dna的0.1%-1%。
备注:添加的内标为dna,非细胞;内标添加的时间在研磨均质和细胞裂解步骤之前。
作为本发明的测定土壤样品dna提取率的方法的进一步改进,所述步骤4)中:
测定内标基因数的方法为:荧光定量pcr(qpcr);
dna提取率的计算公式为:dna提取率=提取后内标的基因拷贝数/加入的基因拷贝数。
本发明采用在土壤样品dna提取之前,添加一种内标(一种特殊无害菌株的dna)的方法,然后采用qpcr测定添加前和添加后内标的基因拷贝数,最终计算内标dna的提取率来代表土壤dna样品的提取率。
本发明探讨不同土壤样品之间的dna提取率是否存在差异,评估下游数据的可靠性,为改进土壤dna的提取方法提供参考;同时也可以用于解析不同类型土壤对dna的吸附差异和潜在机理。
具体而言,在本发明中:
步骤1),a.fischeri菌株只生长在海洋生态系统中,土壤生态系统中不存在此菌,dna浸提过程中不存在土壤本体a.fischeri对提取率的干扰。该菌可在正规的生物科技公司购买,例如美国的atcc公司。该菌的培养方法十分简单,可在室温(25℃)下采用含有3%氯化钠胰蛋白胨大豆琼脂培养基(tsa)进行培养。a.fischeri的dna提取的试剂盒为强力
步骤2),引物特异性检验表明,该引物成功扩增a.fischeri的16srrna基因片段,而无法扩增土壤中其他微生物16srrna的基因片段,证明该引物具有特异性。同时,该菌dna能被土壤微生物的通用引物515f和806r扩增,证明该菌作为内标且用于测定dna提取率的可行性。
步骤3),在dna提取之前,在土壤样品中加入a.fischeri的dna,立即提取dna。
该步骤中a.fischeridna的添加量视土壤样品dna的浓度而定,一般为土壤样品总dna的0.1%-1%左右。内标dna浓度的确定,建议事先提取几个土壤样品的dna,用qubit3.0测定其浓度,然后计算内标dna的添加量,也可以直接估算土壤样品的dna浓度确定内标dna的添加量。土壤样品的dna提取试剂盒为强力
步骤4),采用qpcr测定内标添加前的基因数量和dna浸提之后的内标基因数量。此步骤中的qpcr需要不同浓度梯度的标准样品制备标线,标准样品可通过a.fischeridna的pcr扩增、跑胶、切胶、纯化并稀释获取。qpcr的试剂和扩增条件如下:
表1、qpcr扩增体系
表2、qpcr扩增条件
最终的dna提取率计算公式为:
dna提取率=提取后内标的基因拷贝数/添加的内标基因拷贝数。
与现有技术相比,本发明具有如下技术优势:
1)、本发明操作快速简单,发明中的a.fischeri获取的渠道多,可直接通过购买的方式获得。培养方法十分简便,培养基制备简单,且在室温中即可培养。提取后的内标dna可长期保存,随时待用。
2)、本发明中的a.fischeri生物危害等级为ⅰ,危害等级最低,不会引起健康工作者感染致病,而其他方法采用的内标危害指数高。
3)、本发明采用的内标为海洋生态系统中的微生物a.fischeri,在土壤中不存在。因此,dna提取率不会受到土壤中本体微生物的干扰,可适用于所有土壤样品dna提取率的测定。有学者曾采用大肠杆菌、恶臭假单胞菌等作为内标,但是这些菌在土壤中本身就存在,计算的提取率不精准。
4)、有研究将a.fischeri用于扩增子测序的定量测序,获取土壤中各类微生物的绝对丰度。而本发明可以配合定量测序,在获得土壤中各类微生物的绝对丰度的同时,还可以比较不同类型土壤dna的提取率。
综上所述,本发明提供了一个快速、高效且能适用于所有土壤样品dna提取率测定的方法。此方法可以在扩增和测序之前,判断不同类型土壤的dna提取效率是否存在较大差异,为下游步骤获取的微生物信息的准确性提供参考依据,也为今后改进dna提取方法、降低dna提取过程中导致的偏倚提供参考;另一方面,还可以根据dna提取率的不同,解析不同类型土壤对dna的吸附差异特征和潜在机理,从而辅助评估微生物dna与土壤样品的相互作用和行为特征。
附图说明
下面结合附图对本发明的具体实施方式作进一步详细说明。
图1为a.fischeri引物的特异性检验;a为内标特异性引物的pcr验证结果,b为细菌通用引物的pcr结果;图标“黄壤”、“黑炭300”、“黑炭700”为培养前的样品,“黄壤+黑炭300”为培养后的样品,“a.fischeri”为内标dna。
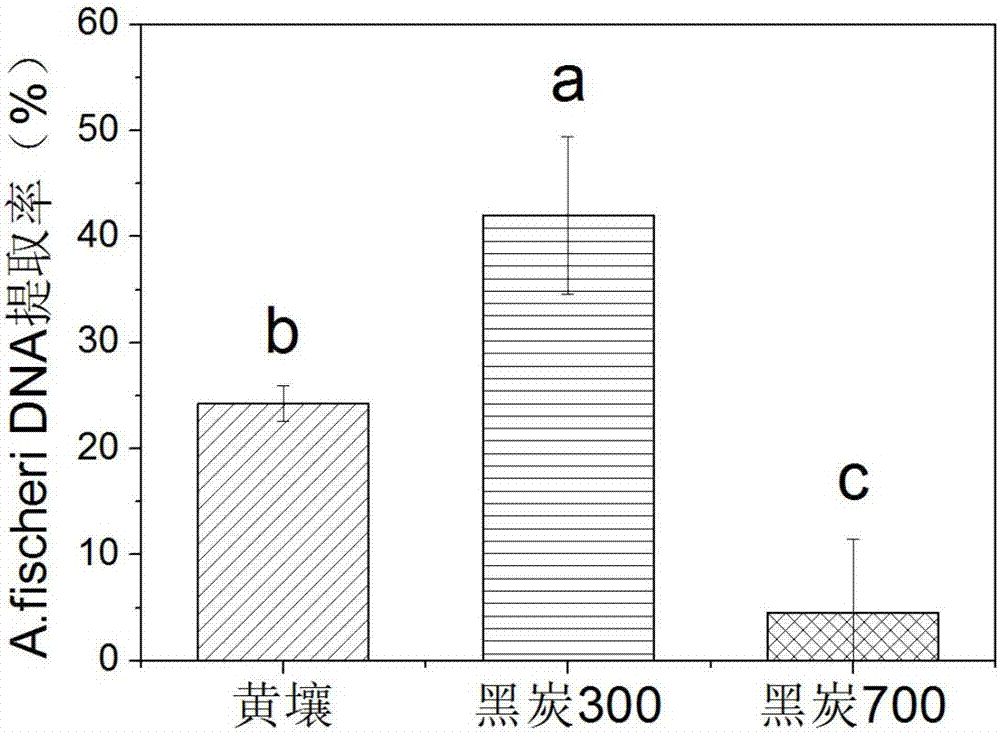
图2为培养前土壤和两种有机碳的dna提取率对比图。
图3为培养后的对照土壤和两种有机碳改良土壤的dna提取率对比图。
图4为排除特殊有机质干扰qpcr过程的结果图。
具体实施方式
下面结合具体实施例对本发明做进一步描述,但本发明的保护范围并不仅限于此。
实施例1、一种测定所有土壤样品dna提取率的方法,依次进行以下步骤:
a)、购买a.fischeriatcc7744t菌株,在浓度为3%nacl的胰胨大豆琼脂培养基上室温(25℃)培养,用强力
b)、采用一种矿质土壤(黄壤)、一种300℃热解所得的黑炭(黑炭300)和一种700℃热解所得的黑炭(黑炭700)作为测试材料。其中,黑炭300富含脂肪族碳结构,而黑炭700富含芳香族碳结构,两者碳结构差异巨大,可视为两种不同类型的土壤有机质。同时,将黑炭300和黑炭700分别加入到矿质土壤黄壤中(黑炭300与黄壤的重量比为1:20,黑炭700与黄壤的重量比为1:20),进行培养,同时将不添加黑炭的黄壤作为对照,培养时间为150天,培养温度为30℃。最终获得六种测试样品:培养前的黄壤、黑炭300、黑炭700和培养后的黄壤、黄壤+黑炭300、黄壤+黑炭700。
c)、采用步骤b)中培养前的黄壤、黑炭300、黑炭700、培养后的黄壤+黑炭300和内标dna,用pcr检验内标引物的特异性。经过pcr后跑胶,若内标dna有明显的条带,而其他样品没有条带,说明该引物只能扩增a.fischeri的基因片段,具有特异性。
qpcr的体系和扩增条件如下:
qpcr扩增体系
*qpcrmix为phusionflashhigh-fidelitypcrmastermix。
qpcr扩增条件:
94℃2min-1个循环;
98℃30s(变性),60℃30s(梯度退火-0.5℃/每个循环),72℃30s(延伸)-40个循环;
72℃1min-1个循环。
当上述pcr体系中:
前引物(正向引物)af-f:5'-gcggaaacgacttaactgaacc-3'
后引物(反向引物)af-r:5'-gaaggtccccctctttggtc-3';
结果为:内标dna有明显的相应条带,而其他样品则没有此条带。
d)、称取一定量(为0.2g)的步骤b)中三种培养前的样品加入到dna试剂管powerbeadtubes中,加入1μl的内标dna(浓度为4ngμl-1),提取dna,具体提取方法参见强力
备注说明:可先用qubit3.0测定三种培养前的样品的dna浓度,分别为5ngul-1(黄壤)、2.5ngul-1(黑炭300)、1ngul-1(黑炭700)。
另外,称取一定量(为0.5g)的步骤b)中三种培养后的样品加入到试剂管powerbeadtubes中,分别加入1.25ul、1.5ul和1ul的内标dna到黄壤、黄壤+黑炭300和黄壤+黑炭700中,提取dna,具体提取方法参见强力
同理,可先用qubit3.0测定三种培养后的样品的dna浓度,分别为10ngul-1(黄壤)、12ngul-1(黄壤+黑炭300)和8ngul-1(黄壤+黑炭700)。
e)、采用qpcr测定步骤d)中添加内标前样品的16srrna基因拷贝数,同时测定dna提取之后的样品的内标16srrna基因拷贝数。测定内标基因数的方法为:荧光定量pcr(qpcr);qpcr的引物为af-f和af-r,qpcr的试剂和条件同上述步骤c),计算六种样品的dna提取率。
dna提取率的计算公式为:dna提取率=提取后内标的基因拷贝数/加入的基因拷贝数。
f)、提取步骤b)中黑炭700的dna(不添加内标),采用spike-recovery方法,在黑炭700的dna中,添加已知基因拷贝数的内标,设置不添加内标作为对照,进行qpcr扩展,测定内标基因拷贝数。此步骤旨在观测后续的扩增过程是不是对dna提取率产生影响,若qpcr所得的内标基因拷贝数与添加前的拷贝数没有显著差异,说明所得的dna提取率只受到dna提取过程中样品吸附的影响,而与qpcr扩增中特殊有机质干扰降低最终的基因拷贝数无关。
实际结果为:qpcr所得的内标基因拷贝数与添加前的拷贝数没有显著差异(p>0.05)(图4);因此说明使用本发明所得的dna提取率只受到dna提取过程中样品吸附作用的影响,而与qpcr扩增中特殊有机质干扰降低基因拷贝数无关。
具体如下:
一、内标引物特异性检验结果如图1所述:
本发明设计的a.fischeri特异性引物能够扩增a.fischeri16srrna基因片段,即在图1-a的“a.fischeri”处理有明显的条带。但是,此引物无法扩增“黄壤”、“黄壤+黑炭300”、“黑炭300”和“黑炭700”四个样品中任何微生物的dna,即四个处理均没有条带显示,证明本发明的引物对内标具有特异性。另一方面,本发明发现细菌的通用引物515f和806r能将所有样品(包括内标的dna样品)的16srrna基因片段扩增出来,证明该内标与土壤中的其他微生物一样,均可以被细菌通用引物扩增,可用于一切关于扩增和测序的下游步骤。
注:黄壤+黑炭700的结果与黄壤+黑炭300的结果一致。
二、培养前土壤和两种有机碳的dna提取率如图2所示:
通常,无论是矿物土壤还是有机质土壤,对dna有一定的吸附作用,吸附作用越强烈,浸提出来的dna就越少。当浸提出来的dna量太少时,就会影响下游qpcr或高通量测序结果的准确性。目前,人们往往忽视了土壤样品对dna的吸附导致最终获得的微生物信息有误差。通常情况,人们默认不同土壤样品之间,对dna的吸附能力(即最终得到的dna提取率)相同或差异较小,因而得到的qpcr和高通量测序的数据有可比性。但是,图2表明,黄壤dna的提取率是24.2%,黑炭300的dna提取率为42.0%,而黑炭700的dna提取率仅为4.6%,远远低于黄壤和黑炭300。该结果表明,有大量的dna被黑炭700所吸附,而这些被吸附的dna有可能导致后续的qpcr和高通量测序中损失一定的微生物信息量。
三、培养后土壤和两种有机碳改良的土壤dna提取率如图3所示:
与图2相似,培养后的三种土壤样品dna的提取率也存在差异,添加量黑炭700的土壤dna提取率显著小于对照和添加黑炭300的土壤。这些结果表明,不同类型的土壤样品dna提取率存在差异,可能导致下游所得的微生物信息存在偏倚,需要引起人们的重视。基于不同样品dna提取率存在较大差异,dna提取方法的改进将是未来研究的重点,寻找一个充分提取出土壤样品dna的高效提取方法有待进一步探索。
另一方面,在获得不同类型样品dna提取率的基础上,本发明可以深入分析不同样品对dna吸附的机理。从图2和图3可得,黑炭700的dna提取率很低,说明对dna的吸附能力很强。黑炭700是一种具有高的芳香结构的材料,亲水能力强,比表面积大,而dna属于亲水分子,更容易吸附在黑炭700上面;而黑炭300和黄壤含有多数脂肪族有机碳,比表面积小,疏水官能团多,因此对dna的吸附能力较小。该发明还可以根据dna提取率的不同,解析不同类型土壤对dna的吸附差异特征和潜在机理,从而辅助评估微生物dna与土壤样品的相互作用和行为特征。
四、排除特殊有机质干扰qpcr过程导致dna提取率下降的可能性验证如图4所示:
研究表明,样品中若含有一些特殊有机质组分,可能会干扰pcr过程,导致dna的扩增受阻,目的基因无法或部分被扩增出来。由图2和图3所示,黑炭700的dna提取率很低,可能还因为黑炭700含有特殊的有机质干扰了qpcr的过程,导致最终获得的基因拷贝数偏低,而不是黑炭700对dna的吸附。为了验证该假设,本发明提取了黑炭700的dna,然后在dna中加入已知基因拷贝数的内标dna,再进行qpcr扩增。结果表明,qpcr扩增后的基因拷贝数与添加之前的基因拷贝数没有显著差异,表明qpcr过程中特殊有机质对基因扩增没有影响,黑炭700的dna提取率偏低只是因为黑炭700的强吸附性能。
综上所述,本发明既能够测定所有土壤样品dna的提取率,为后续qpcr和高通量测序的结果准确性提供参考,也可以用来探讨不同土壤样品对dna的吸附性能和吸附机理。
最后,还需要注意的是,以上列举的仅是本发明的若干个具体实施例。显然,本发明不限于以上实施例,还可以有许多变形。本领域的普通技术人员能从本发明公开的内容直接导出或联想到的所有变形,均应认为是本发明的保护范围。
序列表
<110>浙江大学
<120>测定土壤样品dna提取率的方法
<160>2
<170>siposequencelisting1.0
<210>1
<211>22
<212>dna
<213>人工序列(artificialsequence)
<400>1
gcggaaacgacttaactgaacc22
<210>2
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>2
gaaggtccccctctttggtc20
- 还没有人留言评论。精彩留言会获得点赞!