分子编程工具的制作方法
本申请要求2016年2月17日提交的美国临时申请号62/296,310、2016年2月24日提交的美国临时申请号62/299,206、2016年12月2日提交的美国临时申请号62/429,149和2016年12月9日提交的美国临时申请号62/432,017在35u.s.c.§119(e)下的权益,上述文献各自通过引用完整地并入本文。
联邦政府资助的研究
本发明是在nationalinstitutesofhealth授予的第eb018659号拨款、nationalsciencefoundation授予的第1317291、2014188、1540214和1334109号拨款和u.s.departmentofdefense,officeofnavalresearch授予的n00014-14-1-0610、n00014-13-1-0593和n00014-16-1-2410拨款下借助政府资助完成的。政府对本发明享有一定的权利。
背景
脱氧核糖核酸(dna)的信息编码特性和编程的watson-crick碱基配对互补性的相对便利性导致dna在最近20年中被用作许多不同应用的底层支持1–9。此外,dna已被编程以自组装成规定的2d和3d形状的纳米结构10–18,允许生物分子的精确空间图案化19–26和甚至无机分子的成形27。这些合成系统已经在合成生物学、材料科学和生物成像的领域中显示出它们的价值,其中研究人员致力于dna纳米结构在生物分子核磁共振(nmr)结构测定28、条件细胞通路调节22、29、30和dna纳米结构作为药物递送的载体31、32中的应用。
概述
在一些方面,本文提供了基于核酸的分子工具,其使得能够记录和重构分子景观。dna通过编码基因组中不同分子功能的信息来模拟生命。它还用作多种合成反应网络的模板底物,例如聚合酶链反应(pcr)(randallksaiki等人science,239(4839):487–491、1988)、滚环扩增(rca)(paulmlizardi等人naturegenetics,19(3):225–232,1998)和链置换线路(circuit)(luluqian和erikwinfree.science,332(6034):1196–1201,201;davidyuzhang和georgseelig.naturechemistry,3(2):103–113,2011)。本公开尤其介绍了引物交换反应(per)的概念,其使用催化dna发夹物种,在一些实施方案中,以逐步方式模拟单链dna(ssdna)的等温合成。本文提供的数据显示引物交换反应级联能够用于按照规定的途径生长dna的新生链,并且还能够用于构建例如线性扩增特异性引物信号的合成端粒酶。本文提供的其它数据证明了处理和响应溶液中rna信号的几种功能系统的实施,包括无标记生物传感器,时间记录器,逻辑线路和将靶信号检测转换为操作独立的rna序列的功能性dna酶(dnazyme)的纳米装置。例如,本公开的方法可用于在等温原位环境中合成任意ssdna,并为新一代分子装置提供基础。
因此,一些方面提供了动态dna线路的分子基元,在本文中称为“引物交换”,其尤其为原位合成高度稳健的动态构建体提供了基础。在引物交换反应(per)期间,使用链置换聚合酶和起催化作用的部分配对分子(例如发夹分子)将离散的核苷酸序列(结构域)添加(合成)至“生长的”核酸链(参见,例如图1a-1b)。per代表了分子编程的新范例,具有催化活性,模块性,稳健性,基本燃料物种(dntp),原位操作和单分子转录物记录。这种用任意用户指定的序列等温和自主合成单链dna的方法可以适用于原位和体内使用。
本公开内容的其它方面提供了分子(例如dna)马达系统,其使用作为轨道单个位点的核酸分子将化学能(例如与dna杂交相关的焓或与从复合物中释放dna分子相关的熵)转换成机械功,所述轨道单个位点提供物理支持,并且在某些情况下,为不断增长的核酸信息记录规定分子运动的方向或路径(参见,例如图23a-24b)。如本文所提供的,分子马达系统使得仅在需要时能够在原位上可编程地生成分子成分。也就是说,可以在进行反应时主动地制造分子。该技术使得能够在分子水平上检测分子环境和条件。该技术还通过与单个轨道位点分子相互作用产生的检查记录提供分子景观的定量和重构。在特定环境/景观中作为从一个轨道位点分子移动到另一个轨道位点分子的核酸的生长链被称为“分子爬行者”(参见,例如图28a-28b)和“分子步行者”(参见,例如图29a-29b)。
本公开的其它方面提供了一种自主的自下而上工具,其通过记录配对的核酸分子内的距离信息(称为记录)来记录靶分子之间的纳米级(或微米级)距离。在反应过程中产生的核酸记录的长度直接对应于测量的距离(例如生物分子之间的距离)。另外,每个核酸记录可以编码靶分子的身份作为其序列的一部分。该分子标尺系统(参见,例如图33a-34)能够记录固相或原位靶分子溶液中的距离。
因此,本公开的一些方面提供引物交换反应(per)系统,包含:(a)起始催化分子,其包含(i)未配对的3'立足点(toehold)结构域和(ii)位于立足点结构域的5'的配对结构域;(b)与未配对的3'立足点结构域互补的起始引物;和(c)具有链置换活性的聚合酶。在一些实施方案中,起始催化分子具有发夹结构且由此,还包含位于3'立足点结构域相对侧末端的环状结构域;其为来自配对结构域的5'(参见,例如图1a)。在一些实施方案中,该系统还包含脱氧核糖核苷酸三磷酸(dntp)。
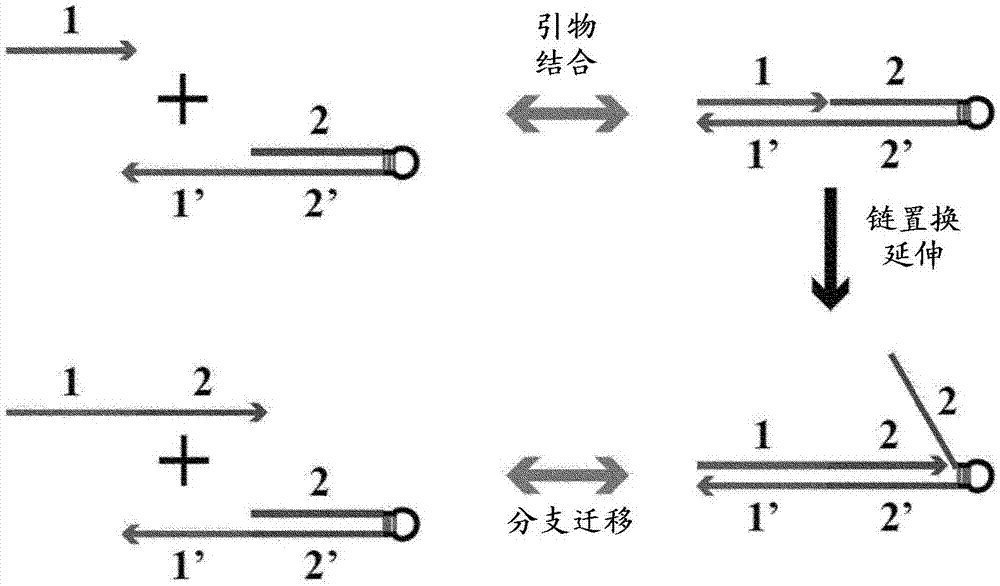
“结构域”是指个核苷酸或核苷酸碱基对的离散的、连续的序列,这分别取决于结构域是否是未配对的(单链个核苷酸)或配对的(双链核苷酸碱基对)。在一些实施方案中,为了定义分子内(在相同的分子物种内)和分子间(在两个分开的分子物种之间)互补性,将结构域描述为具有多个亚结构域。如果一个结构域含有核苷酸,其与另一个结构域的核苷酸发生碱基对(通过watson-crick核苷酸碱基配对而杂交/结合),则一个结构域(或一个亚结构域)与另一个结构域“互补”,使得这两个结构域形成配对(双链)或部分配对的分子物种/结构。互补结构域不需要完全(100%)互补以形成配对结构,但在一些实施方案中,提供了完美的互补性。因此,与特定结构域“互补”的引物与该结构域结合,例如,结合足以在聚合酶存在下引发聚合的时间。图1a,例如,显示引物‘1’与催化发夹分子的结构域‘1”结合。类似地,图1b显示了与催化发夹分子的结构域‘2”结合的引物的引物结构域‘2’。
在一些实施方案中描述了不具有发夹环的催化分子(不是由连续的核苷酸段(stretch)形成),因为它是包括与“模板链”配对(杂交/结合)的“置换链”的双链体(duplex)。图24(第三个“双链体”图)显示通过置换链‘1’(顶部,较短链)与模板链‘1’+1”(底部,较长链)结合形成的双链体催化分子的实例。
应当理解,本文提供的任何一种催化分子可包含位于分子末端与立足点结构域相对的“连接结构域”。连接结构域可以是发夹环(环结构域),如图1a-1c中的实施例所示,或连接结构域可包括彼此共价交联的互补核苷酸(例如至少1、2、3、4、5、6、7、8、9或10个共价交联的核苷酸碱基对)。在一些实施方案中,连接结构域简单地是稳定的配对结构域,例如,具有至少10个核苷酸的长度(例如,使得该结构域在整个per条件下保持配对)。
本公开内容的一些方面提供了引物交换反应(per)系统,其包含:(a)起始催化发夹分子,其包含(i)未配对的3'立足点结构域;(ii)由分子内核苷酸形成的配对的茎结构域,所述分子内核苷酸在该分子的3'亚结构域和该分子的5'亚结构域之间碱基配对,和(iii)环状结构域;(b)与未配对的3'立足点结构域互补的起始引物;(c)具有链置换活性的聚合酶。发夹分子通常通过分子内核苷酸碱基配对形成,其是指核酸的相同连续核酸链的结构域之间的结合。例如,图1a显示通过5'(上游)结构域‘2’与3'(下游)结构域‘2”的结合形成的发夹分子。应当理解,5'结构域‘2’以类似于双链体催化分子的置换链的方式起作用,并且3'结构域‘2”与立足点结构域‘1”一起以类似于双链体催化分子的模板链的方式起作用。
本公开的其它方面提供了引物交换反应(per)系统,其包括:(a)起始催化分子,其包含(i)未配对的3'立足点结构域和(ii)位于立足点结构域的5'的配对结构域,所述配对结构域通过置换链与含有立足点结构域的模板链之间的核苷酸碱基配对形成;(b)第二催化分子,其包含(i)未配对的3'立足点结构域和(ii)位于立足点结构域5'的配对结构域,所述配对结构域通过第二催化分子的置换链与含有立足点结构域的模板链之间的核苷酸碱基配对形成,其中第二催化分子的3'立足点结构域与起始催化分子的置换链互补;和(c)与起始催化分子的未配对的3'立足点结构域互补的起始引物。在一些实施方案中,per系统还包含具有链置换活性的聚合酶。在一些实施方案中,per系统还包含dntp。
在一些实施方案中,per系统包含:(a)起始催化发夹分子,其包含(i)未配对的3'立足点结构域;(ii)由分子的5'亚结构域与分子的3'亚结构域之间的分子内核苷酸碱基配对形成的配对的茎结构域;和(iii)环状结构域;(b)第二催化发夹分子,其包含(i)未配对的3'立足点结构域;(ii)由第二发夹分子的3'亚结构域与第二发夹分子的5'亚结构域之间的分子内核苷酸碱基配对形成的配对的茎结构域;和(iii)环状结构域,其中第二发夹分子的3'立足点结构域与起始发夹分子的5'亚结构域互补;和(c)与起始发夹分子的未配对的3'立足点结构域互补的起始引物。
本公开的其它方面提供引物交换反应(per)方法,包括:在反应缓冲液中合并:(a)起始催化分子,其包含(i)未配对的3'立足点结构域和(ii)位于立足点结构域的5'的配对结构域,所述配对结构域由置换链与含有立足点结构域的模板链之间的核苷酸碱基配对形成;(b)第二催化分子,其包含(i)未配对的3'立足点结构域和(ii)位于立足点结构域的5'的配对结构域,所述配对结构域通过置换链与包含第二催化分子的立足点结构域的模板链之间的核苷酸碱基配对形成,其中第二催化分子的3'立足点结构域与起始催化分子的置换链互补;(c)与起始催化分子的未配对的3'立足点结构域互补的引物;(d)具有链置换活性的聚合酶;和(e)脱氧核糖核苷酸三磷酸(dntp),从而形成反应混合物;并且在导致核酸聚合、链置换和退火的条件下将反应混合物孵育足以产生单链核酸记录的时间。
在一些实施方案中,per方法包括:在反应缓冲液中合并:(a)起始催化发夹分子,其包含(i)未配对的3'立足点结构域;(ii)通过分子的3'亚结构域与分子的5'亚结构域之间的核苷酸碱基配对形成的配对的茎结构域;和(iii)发夹环状结构域,(b)第二催化发夹分子,其包含(i)未配对的3'立足点结构域;(ii)通过第二发夹分子的3'亚结构域与第二发夹分子的5'结构域之间的分子内核苷酸碱基配对形成的配对的茎结构域,和(iii)环状结构域,其中第二发夹分子的3'立足点结构域与起始发夹分子的5'亚结构域互补;(c)与起始发夹分子的未配对的3'立足点结构域互补的引物;(d)具有链置换活性的聚合酶;和(e)脱氧核糖核苷酸三磷酸(dntp),从而形成反应混合物;在导致核酸聚合、链置换和退火的条件下将反应混合物孵育足以产生单链核酸记录的时间。
在一些实施方案中,per方法包括(a)在具有链置换活性的聚合酶和脱氧核糖核苷酸三磷酸(dntp)存在下使输入引物与催化分子接触,其中催化分子包含(i)未配对的3'立足点结构域和(ii)位于立足点结构域的5'的配对结构域,所述配对结构域通过置换链与含有立足点结构域的模板链之间的核苷酸碱基配对形成,其中输入引物与发夹分子的3'立足点结构域互补;(b)通过催化分子的配对结构域使引物延伸,从而置换置换链并形成延伸的输出引物;(c)通过置换链和模板链之间的核苷酸碱基配对置换来自发夹分子的延伸的输出引物;并且(d)在具有链置换活性和dntp的聚合酶存在下,使(c)的置换的延伸的输出引物与第二催化分子接触,其中第二催化分子包含(i)未配对的3'立足点结构域和(ii)位于立足点结构域的5'的配对结构域,所述配对结构域通过置换链与含有第二催化分子的立足点结构域的模板链之间的核苷酸碱基配对形成,并且其中延伸的输出引物与第二催化分子的3'立足点结构域互补。在一些实施方案中,催化分子是催化发夹分子。
本文还提供了组合物,其包含(a)催化发夹分子,其包含(i)未配对的3'立足点结构域;(ii)通过分子的3'亚结构域与分子的5'亚结构域之间的分子内核苷酸碱基配对形成的配对的茎结构域;和(iii)连接结构域,其中(a)(i)和(a)(ii)的结构域形成串联重复序列;(b)至少一个其它催化发夹分子,其包含(i)3'立足点结构域;(ii)通过分子的3'亚结构域与分子的5'亚结构域之间的分子内核苷酸碱基配对形成的配对的茎结构域;和(iii)连接结构域,其中(b)(i)和(b)(ii)的结构域形成被信号序列中断的串联重复序列,并且其中(b)(i)的3'立足点结构域不可逆地结合于保护链(protectorstrand);和(c)核酸引物,其包含与(a)的催化发夹分子的3'立足点结构域互补并且与(b)的催化发夹分子的3'立足点结构域互补的结构域。如果两种核酸能够在per条件下彼此结合并彼此解离,则认为它们彼此“不可逆地结合”。“串联重复序列”的实例描述于图21a-21b中(参见a*-a*)。在图21a-21b中还描绘了“由信号序列中断的串联重复序列”的实例(参见a*-s*-a*)。在一些实施方案中,信号序列可以是按照与串核重复结构域中核苷酸的顺序不同的顺序排列的至少1个核苷酸或至少2个核苷酸(例如1-20个核苷酸或至少2、3、4、5、6、7、8、9或10个核苷酸)。在一些实施方案中,保护链与(b)的至少一个其它催化发夹分子的3'立足点结构域任选地通过环状结构域连接(参见,例如图42a)。
本公开的一些方面提供生产单链核酸的方法,包括:在反应缓冲液中合并:(a)起始催化分子,其包含(i)未配对的3'立足点结构域和(ii)位于立足点结构域的5'的配对结构域,所述配对结构域通过置换链与含有立足点结构域的模板链之间的核苷酸碱基配对形成;(b)多个不同催化分子,每个发夹分子包含:(i)未配对的3'立足点结构域和(ii)位于立足点结构域的5'的配对结构域,所述配对结构域通过置换链与含有立足点结构域的模板链之间的核苷酸碱基配对形成,其中每个催化分子的3'立足点结构域与多个催化分子中的一个其它催化分子的置换链互补;(c)与起始催化分子的3'立足点结构域互补的起始引物;(d)具有链置换活性的聚合酶;和(e)脱氧核糖核苷酸三磷酸(dntp),由此形成反应混合物;并且在导致核酸聚合、链置换和退火的条件下,将反应混合物孵育足以产生比起始引物长的单链核酸记录的时间。
在一些实施方案中,产生单链核酸的方法包括:在反应缓冲液中合并:(a)起始发夹分子,其包含(i)未配对的3'立足点结构域;(ii)通过分子的3'亚结构域与分子的5'亚结构域之间的分子内核苷酸碱基配对形成的配对的茎结构域;和(iii)发夹环状结构域;(b)多个不同的发夹分子,每个发夹分子包含(i)未配对的3'立足点结构域;(ii)由多个发夹分子中的发夹分子的3'亚结构域与多个发夹分子中的发夹分子的5'亚结构域之间的分子内核苷酸碱基配对形成的配对的茎结构域;和(iii)环状结构域,其中每个发夹分子的3'立足点结构域与多个发夹分子中的一个其它发夹分子的5'亚结构域互补;(c)与起始发夹分子的3'立足点结构域互补的起始引物;(d)具有链置换活性的聚合酶;和(e)脱氧核糖核苷酸三磷酸(dntp),从而形成反应混合物;在导致核酸聚合、链置换和退火的条件下将反应混合物孵育足以产生比起始引物长的单链核酸记录的时间。
本公开的其它方面提供了测定分子事件之间的时间的方法,包括:在反应缓冲液中合并:(a)起始催化分子,其包含(i)未配对的3'立足点结构域和(ii)位于立足点结构域的5'的配对结构域,所述配对结构域通过置换链与含有立足点结构域的模板链之间的核苷酸碱基配对形成;(b)多个不同的催化分子,每个催化分子包含(i)未配对的3'立足点结构域(ii)位于立足点结构域的5'的配对结构域,所述配对结构域通过置换链与含有立足点结构域的模板链之间的核苷酸碱基配对形成,其中每个催化分子的3'立足点结构域与多个催化分子中的一个其它催化分子的置换链互补;(c)与起始催化分子的未配对的3'立足点结构域互补的起始引物;(d)具有链置换活性的聚合酶;和(e)脱氧核糖核苷酸三磷酸(dntp),从而形成反应混合物;将反应混合物暴露于第一分子事件;在导致核酸聚合、链置换和退火的条件下将反应混合物孵育足以产生单链核酸记录的时间;并将反应混合物暴露于第二分子事件。
在一些实施方案中,测定分子事件之间的时间的方法包括:在反应缓冲液中合并:(a)起始催化发夹分子,其包含(i)未配对的3'立足点结构域;(ii)通过分子的3'亚结构域与分子的5'亚结构域之间分子内核苷酸碱基配对形成的配对的茎结构域;和(iii)发夹环状结构域;(b)多个不同的催化发夹分子,每个发夹分子包含(i)未配对的3'立足点结构域;(ii)由发夹分子的3'亚结构域与发夹分子的5'亚结构域之间的分子内核苷酸碱基配对形成的配对的茎结构域,和(iii)环状结构域,其中每个发夹分子的3'立足点结构域与多个发夹分子中的一个其它发夹分子的5'亚结构域互补;(c)与起始发夹分子的未配对的3'立足点结构域互补的起始引物;(d)具有链置换活性的聚合酶;和(e)脱氧核糖核苷酸三磷酸(dntp),从而形成反应混合物;将反应混合物暴露于第一分子事件;在导致核酸聚合、链置换和退火的条件下将反应混合物孵育足以产生单链核酸记录的时间;并将反应混合物暴露于第二分子事件。
本公开的其它方面提供了分子马达系统,其包含:(a)起始核酸分子,其包含(i)未配对的3'立足点结构域和(ii)位于立足点结构域5'的配对结构域,所述配对结构域通过置换链与含有立足点结构域的模板链之间的核苷酸碱基配对形成;(b)第二核酸分子,其包含(i)未配对的3'立足点结构域和(ii)位于立足点结构域5'的配对结构域,其由置换链与含有立足点结构域的模板链之间的核苷酸碱基配对形成,其中第二核酸分子的未配对的3'立足点结构域与起始核酸分子的置换链的互补;和(c)与位于起始核酸分子的未配对的3'立足点结构域中的核苷酸互补的引物。
本公开的其它方面提供了分子记录系统,其包括:(a)起始发夹分子,其包含(i)未配对的3'立足点结构域;(ii)通过分子的3'亚结构域和分子的5'亚结构域的分子间核苷酸碱基配对形成的配对的茎结构域;和(iii)环状结构域,其中起始发夹分子与靶生物分子连接;(b)第二发夹分子,其包含(i)未配对的3'立足点结构域;(ii)分子的3'亚结构域与分子的5'亚结构域之间的分子内核苷酸碱基配对形成的配对的茎结构域;和(iii)环状结构域,其中第二发夹分子与靶生物分子连接,并且其中起始发夹分子的5'亚结构域与第二发夹分子的5'亚结构域互补;(c)两种引物,其中之一与起始发夹分子的未配对的3'立足点结构域互补,而其中另一个与第二个发夹分子的未配对的3'立足点结构域互补;(d)多个催化发夹分子,每个分子包含(i)未配对的3'立足点结构域;(ii)通过分子的3'亚结构域与分子的5'亚结构域之间的分子内核苷酸碱基配对形成的配对的茎结构域;和(iii)环状结构域,其中多个发夹分子中的每个发夹分子的5'亚结构域与多个发夹分子中的一个其它发夹分子的5'亚结构域互补,其中多个发夹分子之一的3'立足点结构域与起始发夹分子的5'亚结构域互补,并且其中多个发夹分子中的另一个的3'立足点结构域与第二发夹分子的5'亚结构域互补;和(e)具有链置换活性的聚合酶。
本公开的其它方面提供了记录靶生物分子之间的距离的方法,包括在反应缓冲液中合并:(a)起始发夹分子,其包含(i)未配对的3'立足点结构域;(ii)通过分子的3'亚结构域与分子的5'亚结构域之间的分子内核苷酸碱基配对形成的配对的茎结构域;和(iii)环状结构域,其中起始发夹分子与靶生物分子连接;(b)第二发夹分子,其包含(i)未配对的3'立足点结构域;(ii)通过分子的3'亚结构域与分子的5'亚结构域之间的分子内核苷酸碱基配对形成的配对的茎结构域;和(iii)环状结构域,其中第二发夹分子与靶生物分子连接,并且其中起始发夹分子的5'亚结构域与第二发夹分子的5'亚结构域互补,(c)两种引物,其中之一与起始发夹分子的未配对的3'立足点结构域互补,而其中另一个与第二发夹分子的未配对的3'立足点结构域互补;(d)多个发夹分子,每个分子包含(i)未配对的3'立足点结构域;(ii)通过分子的3'亚结构域与分子的5'亚结构域之间的分子内核苷酸碱基配对形成的配对的茎结构域;和(iii)环状结构域,其中多个发夹分子中的每个发夹分子的5'亚结构域与多个发夹分子中的一个其它发夹分子的5'亚结构域互补,其中多个发夹分子之一的3'立足点结构域与起始发夹分子的5'亚结构域互补,并且其中多个发夹分子中的另一个的3'立足点结构域与第二发夹分子的5'亚结构域互补;和(e)具有链置换活性的聚合酶和脱氧核糖核苷酸三磷酸(dntp),从而形成反应混合物;在导致核酸聚合、链置换和退火的条件下将反应混合物孵育足以产生双链核酸记录的时间。
附图简要说明
图1a显示了引物交换反应(per)的实例或多步骤per的单步骤(循环)的图。发夹起催化作用以将2结构域附加到具有1结构域的引物上。图1b还显示了per或多步骤per的单个步骤。图1c显示了引物交换反应(per)的实例的另一个图:(i)引物交换反应,其使用催化发夹产生形式ab的转录物。(ii)使用发夹的实例反应实施方案,其使用g-c对作为终止序列。本文描述了其它终止序列。(iii)引物交换反应循环。首先,引物a与催化发夹结合(步骤1)。然后,用链置换聚合酶将b结构域连接(concatenate)到a上,直到它到达终止序列(步骤2)。分支迁移置换了合成的b结构域(步骤3)。ab转录物与发夹解离,而发夹再循环(步骤4)。(iv)page变性凝胶,其描绘单个引物交换反应的反应时间序列和发夹浓度梯度。对于两个实验,将引物浓度固定在100nm,并将反应与10μm的datp、dttp和dctp在37℃一起孵育。泳道11和13对应于相同的条件。
图2a显示了per级联的实例。(i)将几种per发夹和其它试剂组合用于引物序列的自主逐步生长的示意图。(ii)由发夹a、b、c、d和e模拟的五个延伸步骤的反应图。(iii)变性凝胶,其证明存在的发夹物种的不同子集的差异延伸。将反应在37℃孵育4小时,其中引物浓度为100nm,发夹为10nm,datp、dttp和dctp各为10μm。(iv)用于折纸结构的40个订书钉链的平行合成的示意图和折叠的折纸边界重叠的这些结构中的三个的afm图像。图2b显示了状态转换图的实例。发夹起催化作用以将3结构域附加到具有1和2结构域的引物上。
图3a和3b显示根据规定的途径的per延伸链的图像。
图4显示了证明per能够用于构建合成端粒酶的图像。使用一个发夹,实施复制特定10个核苷酸序列的系统。泳道(a)显示没有任何发夹的引物,泳道(b)-(d)显示使用不同浓度的发夹的结果。将反应在37℃孵育4小时。
图5显示证实多步骤per的图像。
图6显示几种应用的实例,其中per可用作工具,包括复杂纳米结构的触发组装物(小图1),测量时间的分子时钟和控制随着时间过去的信号的计时器(小图2),以及随着时间过去区分响应信号和记录信号的系统(小图3)。
图7显示了几种应用的实例,其中per可用于复杂纳米结构的触发组装。顶图显示了长链固定长度的触发组装。中图组显示了折叠成任意2d和3d形状的长链。底图显示了也能够折叠成形状的树枝状主链结构的组装。
图8a-8b显示了用于按比例放大长支架链的组装的两种实例方法。图8a描绘了增加催化发夹的数量,而图8b描绘了增加每个发夹内的复制区域的长度。
图9a-9c显示了使用per触发核酸纳米结构组装的三种实例方法。图9a显示了常规dna折纸纳米结构的触发组装的实例。图9b显示了单链拼块(tile)纳米结构的触发组装的实例。图9c显示了单链dna折纸纳米结构的触发组装的实例。
图10a-10c显示用per的树枝状组装实例机制。图10a显示了除了发夹之外,使用单链燃料(核酸)以指数生长动力学触发合成树枝状聚合物。图10b显示了树枝状结构的实例。图10c显示了使用订书钉链(短寡核苷酸)折叠成特定2d和3d形状的树枝状主链。
图11显示了几种应用的实例,其中per可以用作分子时钟来测量时间和作为计时器来随时间的变化控制信号。顶图显示用作分子时钟以测量经过的时间的per的示意图。中图显示用作可编程时序启动的分子计时器的per的示意图。底图显示了用于多重时序记录和启动的per的示意图。
图12a-12f显示了信号检测模块的实例。图12a显示了条件状态转换的抽象。图12b显示了涉及不可逆引物交换步骤的检测。图12c显示了用特定波长的光激活发夹和使发夹失活的可逆交联反应57。图12d显示使用保护链检测的mrna信号,所述保护链在没有信号的情况下封闭发夹上的结合位点。在一些情况下,该输入也可以是ssdna或另一种单链核酸。在一些实施方案中,该触发信号是通过分开的per反应合成的链。图12e显示了通过用pcr附加立足点区域实现的双链dna(dsdna)的检测45。图12f显示了识别小分子和蛋白质靶的适体以解除对发夹的封闭。
图13a-13c显示分子时钟的实例。图13a显示了触发向端粒化(telomerization)状态转换的输入,以及停止此操作的第二输入。图13b显示了每个状态转换使用的一个发夹,如图12a-12f所示。图13c显示了输出的长度,其表示两个输入之间经过的时间。
图14a-14c显示分子计时器的实例。图14a显示了使用延迟线路的计时器,其中引物在启动之前经历固定数量的状态转换。图14b显示了每个状态转换的一个发夹。图14c描绘了输出曲线。
图15a-15d显示使用per记录蛋白质合成脉冲的实例。图15a显示了输出1和2之间的程序性延迟,为立足点开关激活的时序控制提供了基础。图15b显示了分子实施的实例。图15c显示了由被编程为激活然后使立足点开关失活的线路产生的输出序列。图15d显示了输出的轨迹。
图16a-16i显示per逻辑的实例。逻辑运算and(图16a-16c)、or(图16d-16f)和not(图16g-16i)能够用per反应图实现。
图17a-17c显示使用per的逻辑计算的实例。图17a显示了实例数字逻辑线路。图17b显示了涉及6个状态的系统。图17c显示了涉及集成到线路中的信号检测(图12a-12f)和逻辑(图16a-16i)模块的分子实施。
图18显示了两种应用的实例,其中per可用于实现随着时间过去区分响应信号和记录信号的系统。顶图显示了发展自组装的实例,其中原位的不同结构生长遵循发展途径。底图显示了分子自动收报机带的实例,其记录了信号随着时间过去的时间轨迹。
图19a-19c显示使用per的发展自组装的实例。图19a显示了基于随着时间过去遇到的信号(a-e)进行状态区分的引物。图19b显示了触发形状的原位合成的区分状态。图19c显示了如图12a-12f中实现的状态转换。
图20a-20d显示使用per的分子自动收报机带(tickettape)构造的实例。图20a显示了自动收报机带是一个端粒化线路,其中一个转换用作时钟,且然后是每个信号类型一个。图20b显示了产生图20c中的转录物的信号轨迹,其中编码有信号历史。图20d显示了每个信号需要一个组成型活性端粒化发夹和多个检测器的系统。
图21a-21d显示单信号发夹系统。(图21a)将结构域复制到引物上的发夹用于通过连续地将结构域附加到转录物序列来对反应计时。(图21b)将重复的结构域a前面的一对额外碱基(s结构域)复制到转录物记录上的发夹用作信号。(图21c)假想的信号浓度曲线。(图21d)含有信号结构域s的转录物的示意图,其中根据时变信号浓度将结构域s插入重复的a结构域内。
图22显示了衔接子标记的实例。在记录产生来自起始引物序列的转录物后,引入衔接子序列并使用一对夹板序列将衔接子序列连接到转录物的任一末端。
图23a-23b显示来自实验b和c的信号浓度的统计推断。(图23a)实验b采用500pm的恒定信号浓度(顶部),而实验c采用阶梯信号函数,1小时后稳定在200pm。(图23b)基于具有总共179个信号的300个转录物(实验b,顶部)和具有127个总信号的1,000个转录物(实验c,底部),推断两个实验的信号浓度。实线表示后验分布的均值(基于烧入(burn-in)后的100个样品),而灰色表示两个推断的95%可信区域。将两个实验标准化,使得实验b的后验分布的按时间平均的平均值等于500pm。
图24显示单一催化发夹分子(左图)、拴系催化发夹分子(中图)和双链体催化分子(右图)。
图25显示用于分子靶检测的实例per系统。
图26a-26e显示per纳米装置的实例。(图26a)检测mirna靶并抑制独立基因的纳米装置的示意图。(图26b)合成的核苷酸iso-dg和iso-dc,它们用作四字母代码合成的终止序列。(图26c)纳米装置的系统设置和反应图。致癌mir-19a序列通过由a、b和c发夹合成的10-23dna酶序列(dz-twt)触发twist基因(twt)片段的切割。(图26d)附加有dna酶(dz-twt)的mirna靶(mir-19a)的序列断裂。由序列上方的线指示新生引物链的结合区域,而用虚线描绘dna酶的催化结构域。(图26e)page变性凝胶验证了在孵育溶液中给出不同发夹的mirna和twistmrna片段状态。将反应在37℃孵育4小时。
图27a-27e显示使用per的无标记生物传感器的实例。(图27a)实施具有单个per发夹的合成端粒酶的示意图。(图27b)page变性凝胶,显示在不同发夹浓度下的端粒化。将引物与给定的发夹浓度在37℃孵育4小时。(图27c)无标记生物传感器的示意图,其中mirna靶激活荧光端粒链的合成。(图27d)生物传感器的系统成分和反应图。门控发夹(a)和端粒酶发夹(b)被设计用于检测mirna信号并连接人端粒序列ttaggg的重复序列,硫磺素(tht)嵌入其中。(图27e)天然page凝胶,显示在10nmmirna信号存在下的条件端粒化。靶检测可以通过琥珀色滤镜单元用蓝光透射仪进行可视化(vis),并且在typhoon扫描仪上也可以对反应的荧光进行可视化(fam)。
图28a-28e显示用per实现逻辑计算的实例。(图28a)用于评估具有rna输入的逻辑表达式的操作示意图。(图28b)mir-19aortwt门反应成分和page变性凝胶,其描绘了响应于不同rna输入的转录物产生。通过查找由虚线表示的特定长度的转录物来读取真的输出。还显示了(图28c)mir-19aandtwt、(图28d)notmir-21和(图28e)(mir-19aortwt)and(notmir-21)的反应设置和page变性凝胶结果。
图29a-29d显示使用per的事件记录的实例。(图29a)事件记录器示意图,其产生了响应于时变rna信号的per转录物。(图29b)用于活化门控发夹的立足点交换机制。(图29c)记录器的系统成分和反应示意图。4个门控发夹(a、b、c和d)用于编程不同序列合成,其响应于证明两种rna靶(twistmrna(twt)片段和mir-19a)的不同次序。(图29d)page变性凝胶显示在37℃对于记录在1和3小时时不同rna信号峰掺入(spike)5小时反应的不同长度转录物。另外的细节参见方法部分。
图30a-30b显示了如何在体内实施per的实例。提出了两种体内per级联策略。(图30a)在第一种中,通过转化或转染将成分直接引入细胞中。一些细胞将接收所有成分,从而实现成功的per合成。(图30b)第二种策略编码质粒或基因组形式的发夹per成分,并且发夹表达为rna构建体,用于编程rna寡核苷酸的实时合成。
图31a显示了分子爬行者的基本操作机制。顶部的行描述了对单个位点的单位操作。中间的行描绘了两个相邻位点之间爬行的一个步骤。底部的行描绘了三位点轨道的起始和最终状态。生成的记录可以通过多种机制释放。一些链物种末端的圆形箭头表示用于保护免于聚合酶延伸的修饰(例如反向dt)。图31b显示了分子爬行者在2d空间中的运动。锚定在基板上的探针位置被描绘为简化的单线,为清楚起见省略了辅助结构域。爬行者能够沿着不同的路径在空间中移动,创建多种记录,由产品旁边的数字字符串表示。
图32a显示了分子步行者的基本操作机制。顶部的行描述了对单个位点的单位操作。中间的行描绘了两个相邻位点之间步行的一个步骤。底部的行描绘了三位点轨道的起始和最终状态。生成的记录可以通过多种机制释放。一些链物种末端的圆形箭头表示用于保护免于聚合酶延伸的修饰(例如反向dt)。图32b显示了分子步行者在2d空间中的运动。锚定在基板上的探针位置被描绘为简化的单线,为清楚起见省略了辅助结构域。步行者可以沿着不同的路径在空间中移动,创建多种记录,由产品旁边的数字串表示。
图33a显示检查分子靶并产生反映位点数量的记录的分子马达。图33b显示了应用于生物系统以计算效价的分子马达。长度分布反映了靶的效价。图33c显示了作为模型系统的核孔复合物和作为参考数据的超分辨率图像。左图显示了宽视野视图,中图显示了具有6个亚单位的单个核孔复合物。右图显示线性化圆形密度投影的直方图和拟合,其显示亚单位之间约30nm的距离。比例尺,300nm(左图)和30nm(中图)。图33d显示了在每个位点上编码的独特信息,爬行者复制,记录和报告独特信息。图33e显示了检查给定分子景观的几个分子马达,每个分子马达遵循不同的路径,并产生反映空间信息的独特记录。集体分析(collectiveanalysis)允许重构分子景观。
图34a显示记录的dna分子内距离。dna记录的阴影末端表示靶的独特条形码。图34b显示了使用分子标尺系统的一轮dna记录产生。
图35a显示记录的大小随着dna靶的定位而变化。图35b显示了对dna纳米结构的npc模拟物,产生了28个不同的记录,相当于四个距离之一d1;d2;d3或d4。
图36a-36h显示分子标尺系统的详细机制。
图37a-37c显示了记录双链dna棒两端之间距离的分子标尺的实例。
图38a-38b显示分子距离记录实验的结果。
图39a-39b表示来自实施例21(图38a)中描述的分子标尺距离记录实验的条带的定量,并随着距离(以dna核苷酸的形式)变化绘制它们(图39a)。峰值相当于预期距离。图39b定量来自图38b中描述的分子标尺距离记录实验的条带,并将随着距离(以纳米的形式)变化绘制它们。峰值对应于预期距离。请注意,最短距离dna棒(5.4nm)仅产生与其距离对应的单个记录,因此不会在该图上绘制成图。
图40显示蛋白质“指纹图谱”方法的实例。
图41a-41d显示4-成分分子记录系统的实例。
图42a-42d显示3-成分分子记录系统的实例。
图43a-43c显示有关分子马达运动的数据。图43a显示了在dna纳米结构平台(上图)上测试的轨道设计的示意图以及在马达完成记录反应之后轨道的分子细节(下图)。图43b表明,在回收和pcr扩增后,产生的记录在变性凝胶电泳下出现在预期的长度范围(118nt)。图43c是轨道位点和马达的原子力显微术(afm)可视化。在添加马达之前(左图),着色为灰色的探针位点显示为点;由于每个探针由两个松散的单链环(通常为3t)锚定,afm尖端的扫描捕获轨道位置的模糊图像。两个黑点是参考点。在记录反应之后(右图),现在轨道位点通过马达连接并保持在一起,它们看起来让人想起三脚架的特征。每个折纸矩形的大小约为80nm×100nm。
图44a-44f显示另一种分子爬行者机制。图44a描绘了含有两个引物结合位点结构域“a”和“b”的探针。图44b显示了超过发夹探针的游离引物,其结合探针并通过置换聚合酶延伸。图44c显示了用于减慢或终止进一步聚合的几种方法(探针上的甲基化rna碱基,“b*”和“b”之间末端核苷酸的错配,探针上的iso-dc核苷酸和每个“a*”引物5'端上相应的iso-dg核苷酸,或类似物)。图44d描绘了通过偶尔结合低浓度游离“b”结构域链终止新的串联(concatenated)条形码链,导致聚合酶复制新的串联链并从所有探针释放它。因此,探针再生以供进一步使用。图44e显示了最终结果:串联条形码拷贝的某种程度的随机拼凑。图44f显示了实例探针序列,其中表示了结构域。从上到下的序列对应于seqidno:2-4。
描述
本文提供的工具使得能够记录分子结构和可溶信号以及分子结构的程序性组装。例如,本公开提供(a)用于单链dna的等温和自主合成的组合物和方法,其可用于原位改造复杂结构的触发组装或用作体内治疗/诊断,(b)用于测定经过时间的分子时钟和在时间延迟之后控制信号的计时器,以及(c)环境响应的纳米机器,其响应于环境信号而区分并随时间记录这些信号。本公开内容提供了转化应用的基础,例如用于cryo-em成像的标记物的原位生长,污染物的长期环境监测,条件基因调控,以及毒素的原位触发包封。
引物交换反应
本公开的该方面的基础在于引物交换反应(per),其描绘在图1a-1b中。这种用于从短引物序列合成核酸的酶促方法以等温方式、典型地例如在37℃操作,并且仅需要dntp作为燃料。可以合并反应以形成使长核酸序列生长的per级联,并且这些合成反应能够依赖于存在的多重环境信号进行。这使得能够将信号信息、例如存在的特异性mrna物种时空记录成为实时合成的转录物。此外,在一些实施方案中,因为合成的转录物能够具有任意的序列,所以合成的链自身能够在细胞中完成程序性功能行为作为对特定条件的响应。
基本引物交换反应在三个一般步骤中发生。首先,引物(结构域1)可逆地结合催化分子(例如发夹分子),这有利于延伸。然后,链置换聚合酶延伸引物以复制催化分子中的茎序列(结构域2’),直至达到终止序列(或终止聚合的其它分子)。在延伸终止后,分子的置换的茎区域能够与其在分子上的相对链重新杂交,以将引物序列置换为可以自发地与催化分子解离并且可以与溶液中另一同源催化分子自由相互作用的点。该引物交换反应能够以特定的可编程方式将序列附加到生长的链上。这些模块化反应单位能够与其它此类反应组合以产生具有特定功能的分子程序。这些反应均以等温方式进行,并由溶液中的dntp提供动力。通过使一个分子(例如发夹)-催化的反应的输出引物序列作为另一个分子的输入引物,能够容易地将引物交换反应连接在一起,并且这些反应关系可以用状态转换图抽象来表示,如图2所示(第一栏)。引物的状态由在其3’末端的结构域指示,并且图中的每个有向边缘用单个发夹物种实施。例如,状态1和状态2之间的边缘表示催化以具有2结构域序列的1结构域结束的链的延伸的发夹的存在。这种抽象描述了系统的功能,可以直接编译成发夹和引物结构域。
这种具有可编程性的模型可以进一步通过使催化分子有条件地暴露作为对于环境触发剂的响应来界定,所述环境触发剂允许进行引物交换反应以便对局部环境做出动态地响应。例如,如下所述,这使得能够进行信号记录和加工应用。
通过使一个发夹催化反应的输出引物序列作为另一个发夹催化反应的输入引物,引物交换反应(per)能够容易地连接在一起形成反应级联(图2a)。图2a(i)显示了用于合成五结构域序列的分子成分。一个发夹用于附加每个结构域,反应过程如图2a(ii)所示。图2a(iii)显示了用溶液中存在的五种发夹的不同子集进行的合成反应的结果。图2a(iv)描绘了使用80种不同的催化发夹和40种引物合成40种dna折纸订书钉链能够在一锅法反应中进行。当与支架链合并并退火时,这些订书钉链形成dna折纸结构,其能够聚集在一起形成长结构。
在一些实施方案中,引物交换反应(per)系统,通过将途径外(off-pathway)泄露反应减少几个数量级来驱动稳健系统。驱动per系统的链置换聚合级联动态地合成新信息,并且活化能高,以引发泄漏聚合反应。在一些实施方案中,可以使用这些低泄漏系统来构建稳健且可扩展的分子系统。此外,本文提供的per系统能够使溶液中的一组核酸链爬上自由能景观,允许大的可编程行为空间和对无酶(例如聚合酶)系统中在热力学上不可能进行的操作的可实现性。能量通过聚合反应引入系统,因此廉价的dntp作为燃料不断驱动per系统。在一些实施方案中,dntp浓度足以使引物交换系统长时间运行,并调节浓度或补充dntp进一步延长了这个操作时间。此外,per系统原位合成链。例如,位于固定细胞内的引物能够在合适位置直接聚合,从而克服了将大寡核苷酸递送到拥挤的细胞环境中的挑战。这种能力开辟了广阔的应用空间,具有在现场组装(生长)大型结构的许多可能性。更进一步,在一些实施方案中,per系统具有单分子分辨率,在溶液中每个引物分子自动生长一个转录物。每个转录物指示它在状态转换图中随时间经过的有序状态组(历史),其中能够在凝胶上读出的过程长度信息和能够通过测序获得的更精确的信息。例如,可以改造这些转录物,以随时间记录当环境信号存在于溶液中时的转录物。
在一些实施方案中,可以将各个per合成反应编程为有条件地有活性(图23a-23d和图26a-26d),从而使得能够实现大量且多样化的可能的可编程行为,以及原位记录环境信号的机会。图23a-23d显示了致癌mirna靶mir-19a如何能够触发dna酶dz-twt的合成,dz-twt已被证明可促进小鼠细胞中的细胞凋亡2。通过可视化dz-twt靶的片段twt的切割来验证dna酶行为(图23e)。图26a-26d显示了引物交换反应如何能够用于记录溶液中遇到相同的两个靶mir-19a和twt的顺序。
总之,本文提供的引物交换反应代表了分子编程的新范例,其具有催化活性,模块性,稳健性,基本燃料物种(dntp),原位操作和单分子转录物记录。
引物交换组合物和系统
催化核酸分子(“催化分子”)通常包括未配对的(单链)3'立足点结构域和位于3'立足点结构域的5'(和在一些实施方案中,直接与3'立足点结构域相邻)的配对(双链)结构域。“催化发夹分子”还包括环状结构域。例如,可以通过改变催化分子(例如催化分子的一个或多个结构域)的长度、组成和浓度来控制引物交换反应的动力学。
催化发夹(参见图1a作为示例性实例)包括与连接至发夹环状结构域(环样结构)的发夹茎结构域(例如通过亚结构域“2”与亚结构域“2'”的分子内结合形成)连接的3'立足点结构域(“1'”)。图24(“双链体”)显示了没有环状结构域的催化分子的实例。催化分子(例如催化发夹分子)的长度可以变化。在一些实施方案中,催化分子具有25-300个核苷酸的长度。例如,催化分子可具有25-250、25-200、25-150、25-100、25-50、50-300、50-250、50-200、50-150或50-100个核苷酸的长度。在一些实施方案中,催化分子具有30-50、40-60、50-70、60-80、70-90、80-100、100-125、100-150或100-200个核苷酸的长度。在一些实施方案中,催化分子具有35、36、37、38、39、40、41、42、43、44、45、46、47、48、49、50、51、52、53、54、55、56、57、58、59、60、61、62、63、64、65、66、67、68、69、70、71、72、73、74、75、76、77、78、79、80、81、82、83、84、85、86、87、88、89、90、91、92、93、94、95、96、97、98、99或100个核苷酸的长度。在一些实施方案中,催化分子长于300个核苷酸或短于25个核苷酸。
“立足点结构域”是指位于催化分子3'末端的未配对的核苷酸序列,其与引物(或引物的引物结构域)的核苷酸序列互补(并结合)。立足点结构域的长度可以变化。在一些实施方案中,立足点结构域具有5-40个核苷酸的长度。例如,立足点结构域可以具有2-35、2-30、2-25、2-20、2-15、2-10、5-35、5-30、5-25、5-20、5-15、5-10、10-40、10-35、10-30、10-25、10-20、10-15、15-40、15-35、15-30、15-25、15-20、20-40、20-35、20-30、20-25、25-40、25-35、25-30、30-40、30-35或35-40个核苷酸的长度。在一些实施方案中,立足点结构域具有5、10、15、20、25、30、35或40个核苷酸的长度。在一些实施方案中,立足点结构域具有10、11、12、13、14、15、16、17、18、19、20、21、22、23、24或25个核苷酸的长度。在一些实施方案中,立足点结构域长于40个核苷酸或短于5个核苷酸。
起始引物(或引物结构域)结合催化分子的3'未配对(单链)立足点结构域以引发引物交换反应。在该反应中(参见图1a作为示例性实例),起始输入引物(“1”)结合催化分子的立足点结构域(“1'”),并通过分支迁移方法经存在于反应溶液中的链置换聚合酶延伸引物置换催化分子的茎结构域的亚结构域之一(“2”)。总体效果是发夹茎结构域的亚结构域之一(“2”)被延伸的(新合成的)引物结构域代替。
催化分子的“配对结构域”或“茎结构域”是指位于催化分子的未配对立足点结构域的5'(和,在一些实施方案中,与之直接相邻)的配对的核苷酸序列(例如watson-crick核碱基配对)。催化分子的配对结构域由置换链与含有立足点结构域的模板链之间的核苷酸碱基配对形成。催化发夹分子的配对茎结构域通过催化发夹分子的两个亚结构域的分子内碱基配对(同一分子内的核苷酸之间的碱基配对)形成:例如位于与位于催化发夹5'末端的亚结构域结合(杂交)的立足点结构域5'的内部/中心亚结构域。配对结构域的长度可以改变。在一些实施方案中,配对结构域具有5-40个核苷酸的长度。例如,配对结构域的长度可以具有5-35、5-30、5-25、5-20、5-15、5-10、10-40、10-35、10-30、10-25、10-20、10-15、15-40、15-35、15-30、15-25、15-20、20-40、20-35、20-30、20-25、25-40、25-35、25-30、30-40、30-35或35-40个核苷酸的长度。在一些实施方案中,配对结构域具有5、10、15、20、25、30、35或40个核苷酸。在一些实施方案中,配对结构域具有的长度10、11、12、13、14、15、16、17、18、19、20、21、22、23、24或25个核苷酸的长度。在一些实施方案中,配对结构域长于40个核苷酸或短于5个核苷酸。
虽然配对结构域通常由催化分子的两个亚结构域的分子内碱基配对形成,但应理解该配对结构域可含有至少一个错误配对(例如a与c或g的配对或t与c或g的配对)。在一些实施方案中,茎结构域具有1-5个错配的核苷酸碱基对。例如,配对结构域可以具有1、2、3、4或5个错配的核苷酸碱基对。
在一些实施方案中,通过置换聚合酶的引物(与引物结合位点结合)延伸通过催化分子中终止聚合的分子或修饰的存在而终止。因此,在一些实施方案中,本公开的催化分子包含终止聚合的分子或修饰。终止聚合的分子或修饰(“终止物”)典型地位于催化分子的配对结构域(例如茎结构域)中,使得聚合终止引物通过配对结构域的延伸。对于以发夹形式排列的催化分子,终止聚合的分子或修饰可位于配对的茎结构域和环状结构域之间。在一些实施方案中,终止聚合的分子是合成的非dna接头,例如,三乙二醇间隔物,例如intspacer9(isp9)或spacer18(integrateddnatechnologies(idt))。应当理解,终止通过聚合酶的聚合的任何非天然接头可以如本文提供的那样进行使用。此类分子和修饰的其它非限制性实施例包括三碳键(/ispc3/)(idt),acryditetm(idt),腺苷酸化,叠氮化物,地高辛(nhs酯),胆固醇基-teg(idt),i-linkertm(idt)和3-氰基乙烯基咔唑(cnvk)及其变体。典型地,短接头(例如isp9)导致更快的反应时间,但并非总是如此。
在一些实施方案中,终止聚合的分子是单个或配对的非天然核苷酸序列,例如iso-dg和iso-dc(idt),它们分别是胞嘧啶和鸟嘌呤的化学变体。iso-dc将与iso-dg(但不与dg)碱基配对(氢键)。类似地,iso-dg将与iso-dc碱基配对但不与dc碱基配对。通过(在终止物位置)在发夹的相对侧将这些核苷酸并入核苷酸对中,聚合酶将停止,因为在该位置它在溶液中没有互补的核苷酸可添加。
在一些实施方案中,通过将反应中的dntp浓度(例如从200μm)降低至100μm、10μm、1μm或以下,改善了“终止物”修饰的性能效率。
包含终止聚合的分子或修饰通常在催化分子的双链结构域(例如发夹结构的茎结构域)中产生“凸起”,因为分子或修饰不配对。因此,在一些实施方案中,催化分子被设计为包括与分子或修饰相对的单一核苷酸(例如胸腺嘧啶),至少两个相同的核苷酸(例如胸腺嘧啶二聚体(tt)或三聚体(ttt))或非天然修饰。
催化发夹的“环状结构域”主要是指在茎结构域的末端(与其相邻)形成环状结构的未配对的单核苷酸序列。环状结构域的长度可以变化。在一个实施方案中,环状结构域的长度为3-200个核苷酸。例如,环状结构域的长度可以是3-175、3-150、3-125、3-100、3-75、3-50、3-25、4-175、4-150、4-125、4-100、4-75、4-50、4-25、5-175、5-150、5-125、5-100、5-75、5-50或5-25个核苷酸。在一些实施方案中,环状结构域具有3-10、3-15、32-10、3-25、3-30、3-35、3-40、3-35、3-40、3-45、3-50、4-10、4-15、4-10、4-25、4-30、4-35、4-40、4-35、4-40、4-45或4-50个核苷酸的长度。在一些实施方案中,环状结构域具有3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、26、27、28、29、30、31、32、33、34、35、36、37、38、39、40、41、42、43、45、46、47、48、49或50个核苷酸的长度。在一些实施方案中,环状结构域长于300个核苷酸。
在一些实施方案中,催化分子不含发夹环状结构域。例如,催化分子可以单纯地是包含与配对结构域相邻的3'未配对立足点结构域的双链体(参见,例如图24“双链体”),类似于茎结构域(没有相邻的环状结构域)。不包括环状结构域的催化分子可以通过在一段(stretch)(例如10个或更多个)核苷酸碱基对之间的交联或核苷酸碱基互补性稳定在与3'立足点结构域相对的末端。
除催化发夹外,引物交换反应系统包括称为输入引物或输出引物的引物。“引物”是这样的核酸,当与另一核酸结合时其是在聚合酶存在下聚合的起点。如本文所用,引物通常是核酸(例如单链核酸)具有与催化分子的立足点结构域互补的核苷酸序列(结构域)(参见例如图1a左上的分子“1”;还参见图1b左上的分子“1+2”)。“输入引物”是与催化分子结合以引发引物交换反应的引物。“输出引物”是在引物交换反应的每个步骤结束时从催化分子释放的延伸产物。然后,在从催化分子解离后,输出引物可以在引物交换反应的另一(后续)步骤中用作输入引物。
图1a中描绘了引物交换反应的完整“步骤”。输入引物(“1”)与催化分子的立足点结构域(“1'”)结合以开始引物交换反应。在含有聚合酶(例如链置换聚合酶)和dntp的反应溶液中与催化分子结合后,起始引物延伸通过配对结构域,置换配对结构域的亚结构域(“2”)。然后,置换的亚结构域(“2”)与延伸引物(“1+2”)竞争与其互补的亚结构域(“2'”)的结合(再退火),从而置换延伸的输出引物“1+2”。这完成了引物交换反应的一个步骤。然后,置换的输出引物“1+2”可以继续在反应的下一步骤中用作输入引物。
例如,如图1b所示,在引物交换反应的另一步骤中,置换的输出引物“1+2”用作输入引物,通过其引物结构域“2”与另一个催化分子的立足点结构域“2'”结合,从而引发引物交换反应中的另一步骤。在含有聚合酶和dntp的反应溶液中与催化分子结合后,输入引物“1+2”延伸通过配对结构域,置换配对结构域的亚结构域(“3”)。然后,置换的亚结构域(“3”)与延伸引物(“1+2+3”)竞争与其互补的亚结构域(“3'”)的结合(再退火),从而置换延伸的输出引物“1+2+3”。这完成了引物交换反应的另一步骤。然后,置换的输出引物“1+2+3”可以继续在反应的下一步骤中用作输入引物。
在一些实施方案中,引物或引物结构域(结合催化分子的立足点结构域的核苷酸序列)具有10-50个核苷酸的长度。例如,引物或引物结构域可以具有10-45、10-40、10-35、10-30、10-25、10-20、10-15、15-50、15-45、15-40、15-35、15-30、15-25、15-20、20-50、20-45、20-40、20-35、20-30、20-25、25-50、25-45、25-40、25-35、25-30、30-50、30-45、30-40、30-35、35-50、35-45、35-40、40-50、40-45或45-50个核苷酸的长度。在一些实施方案中,引物或引物结构域具有的长度10、15、20、25、30、35、40、45或50个核苷酸。在一些实施方案中,引物或引物结构域具有15、16、17、18、19、20、21、22、23、24或25个核苷酸的长度。在一些实施方案中,引物或引物结构域长于50个核苷酸或短于10个核苷酸。应当理解,引物的全长取决于、至少部分取决于附加(聚合)序列的数量和长度,这取决于反应中存在的催化分子的数量和长度。
如本文提供的引物可以与可检测分子(例如发射可检测信号、例如荧光或化学发光信号的分子)连接(标记)。在一些实施方案中,标记是荧光团。与荧光团或其它荧光/化学发光分子连接的引物简称为“荧光引物”。可用于本文的荧光团的实例包括但不限于羟基香豆素,甲氧基香豆素,alexafluor,氨基香豆素,cy2,fam,alexafluor405,alexafluor488,荧光素fitc,alexafluor430,alexafluor532,hex,cy3,tritc,alexafluor546,alexafluor555,r-藻红蛋白(pe),罗丹明red-x,tamara,cy3.5581,rox,alexafluor568,red613,texasred,alexafluor594,alexafluor633,别藻蓝蛋白,alexafluor647,cy5,alexafluor660,cy5.5,trured,alexafluor680,cy7和cy7.5。本公开涵盖发射可检测信号的其它荧光团和分子。
在一些实施方案中,可检测分子连接至非引物的催化分子。
在一些实施方案中,例如,如图27d所示,将可检测的分子并入正在生长的核酸链中。因此,在一些实施方案中,可检测的分子是硫磺素(tht),其嵌入人端粒序列ttaggg的多联体重复序列中。本文涵盖其它嵌入的可检测分子。
在一些实施方案中,引物与生物分子连接。生物分子包括例如核酸(例如dna或rna)和蛋白质。生物分子可以是治疗性、预防性、诊断性或成像分子。在一些实施方案中,生物分子是与疾病相关的或与药物相关的生物分子,例如癌症相关基因或蛋白质或fda批准的或潜在的药物靶。在一些实施方案中,生物分子是酶,抗原,受体,配体,膜蛋白,分泌蛋白或转录因子。
在一些实施方案中,催化分子连接至生物分子。
引物交换反应需要使用聚合酶。在一些实施方案中,聚合酶是dna聚合酶(dnap),例如具有dna链置换活性的dna聚合酶(链置换聚合酶)。“链置换”描述了置换合成期间遇到的下游dna的能力。可以如本文提供的那样使用的具有dna链置换活性的聚合酶的实例包括但不限于phi29dna聚合酶(例如neb#m0269),bstdna聚合酶,大片段(例如neb#m0275)或bsudna聚合酶,大片段(例如neb#m0330)。可以使用具有链置换活性的其它聚合酶。在一些实施方案中,聚合酶是rna聚合酶。
在一些实施方案中,聚合酶是phi29dna聚合酶。在这样的实施方案中,反应条件可以如下:补充有纯化的牛血清白蛋白(bsa)的1x反应缓冲液(例如50mmtris-hcl、10mmmgcl2、10mm(nh4)2so4、4mmdtt),ph7.5,在30℃孵育。
在一些实施方案中,聚合酶是bstdna聚合酶,大片段。在此类实施方案中,反应条件可以如下:1x反应缓冲液(例如20mmtris-hcl、10mm(nh4)2so4、10mmkcl、2mmmgso4、1%
在一些实施方案中,聚合酶是bsudna聚合酶。在此类实施方案中,反应条件可以如下:1x反应缓冲液(例如50mmnacl、10mmtris-hcl、10mmmgcl2、1mmdtt),ph7.9,在37℃孵育。
引物交换反应系统中引物、催化分子和dntp的浓度可以根据例如特定应用和该特定应用所需的动力学而变化。
引物交换反应中引物的浓度可以为例如10nm至1000nm。在一些实施方案中,引物交换反应中的引物浓度为10-20、10-30、10-40、10-50、10-60、10-70、10-80、10-90、10-100、10-125、10-150、10-200、25-50、25-75、25-100、25-150、25-200、50-75、50-100、50-150或50-200nm。在一些实施方案中,引物交换反应中的引物浓度为100-200、100-300、100-400、100-500、100-600、100-70、100-800、100-900或100-1000nm。在一些实施方案中,引物交换反应中的引物浓度为10、15、20、25、30、35、40、45、50、55、60、65、70、75、80、85、90、95、100、105、110、115、120、125、130、135、140、145、150、155、160、165、170、175、180、185、190、195或200nm。在一些实施方案中,引物交换反应中的引物浓度为100、200、300、400、500、600、700、800、900或1000nm。引物交换反应中的引物浓度可以低于10nm或高于1000nm。
引物交换反应中的催化分子(例如催化发夹)浓度可以为例如5nm至1000nm。在一些实施方案中,引物交换反应中的催化分子浓度为5-10、5-20、5-30、5-40、5-50、5-60、5-70、5-80、5-90、5-100、5-125、5-150、5-200、10-50、10-75、10-100、10-150、10-200、25-75、25-100、25-125或25-200nm。在一些实施方案中,引物交换反应中的催化分子浓度为10-200、10-300、10-400、10-500、10-600、10-70、10-800、10-900或10-100nm。在一些实施方案中,引物交换反应中的催化分子浓度为10、15、20、25、30、35、40、45、50、55、60、65、70、75、80、85、90、95、100、105、110、115、120、125、130、135、140、145、150、155、160、165、170、175、180、185、190、195或200nm。在一些实施方案中,引物交换反应中的催化分子浓度为10、20、30、40、50、60、70、80、90或100nm。引物交换反应中的催化分子浓度可以低于5nm或高于1000nm。
引物交换反应中引物与催化分子之比可以为2:1至100:1。在一些实施方案中,引物与催化分子之比为2:1、3:1、4:1、5:1、6:1、:1、8:1、9:1、10:1、11:1、12:1、13:1、14:1、15:1、16:1、17:1、18:1、19:1或20:1。在一些实施方案中,引物与催化分子之比为30:1、40:1、50:1、60:1、70:1、80:1或90:1。
引物交换反应中不同催化分子的数量是非限制性的。引物交换反应可以包含1-1010个不同的催化分子(例如,每个具有特定的立足点结构域序列)。在一些实施方案中,引物交换反应包含1-10、1-102、1-103、1-104、1-105、1-106、1-107、1-108、1-109、1-1010个或以上的不同催化分子。在一些实施方案中,引物交换反应包含1-5、1-10、1-15、1-20、1-25、1-30、1-35、1-40、1-45、1-50、1-55、1-60、1-65、1-70、1-75、1-80、1-85、1-90、1-95、1-100、5-10、5-15、5-20、5-25、5-30、5-35、5-40、5-45、5-50、5-55、5-60、5-65、5-70、5-75、5-80、5-85、5-90、5-95、5-100、10-15、10-20、10-25、10-30、10-35、10-40、10-45、10-50、10-55、10-60、10-65、10-70、10-75、10-80、10-85、10-90、10-95或10-100个不同催化分子。在一些实施方案中,引物交换反应包含1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、28、19、20、21、22、23、24或25个不同催化分子。例如,如果它们的立足点结构域彼此不同,则催化分子彼此可以不同。
例如,可以通过改变温度、时间、缓冲液/盐条件和脱氧核糖核苷酸三磷酸(dntp)浓度来控制引物交换反应的动力学。与大多数酶一样,聚合酶对许多缓冲条件敏感,包括离子强度、ph和存在的金属离子类型(例如钠离子vs.镁离子)。因此,进行引物交换反应的温度可以在例如4℃至65℃(例如4℃、25℃、37℃、42℃或65℃)变化。在一些实施方案中,进行引物交换反应的温度为4-25℃、4-30℃、4-35℃、4-40℃、4-45℃、4-50℃、4-55℃、4-60℃、10-25℃、10-30℃、10-35℃、10-40℃、10-45℃、10-50℃、10-55℃、10-60℃、25-30℃、25-35℃、25-40℃、25-45℃、25-50℃、25-55℃、25-60℃、25-65℃、35-40℃、35-45℃、35-50℃、35-55℃、35-60℃或35-65℃。在一些实施方案中,引物交换反应在室温下进行,而在其它实施方案中,引物交换反应在37℃进行。
可以进行引物交换反应(孵育)30分钟(min)至24小时(hr)。在一些实施方案中,使引物交换反应进行10min、35min、40min、45min、50min、55min、60min、1hr、2hr、3hr、4hr、5hr、6hr、7hr、8hr、9hr、10hr、11hr、12hr、18hr或24hr。
脱氧核糖核苷酸(dntp)是驱动引物交换反应的“燃料”。因此,在一些实施方案中,引物交换反应的动力学在很大程度上取决于反应中dntp的浓度。引物交换反应中dntp的浓度可以为例如2-1000μm。在一些实施方案中,引物交换反应中的dntp浓度为2-10μm、2-15μm、2-20μm、2-25μm、2-30μm、2-35μm、2-40μm、2-45μm、2-50μm、2-55μm、2-60μm、2-65μm、2-70μm、2-75μm、2-80μm、2-85μm、2-90μm、2-95μm、2-100μm、2-110μm、2-120μm、2-130μm、2-140μm、2-150μm、2-160μm、2-170μm、2-180μm、2-190μm、2-200μm、2-250μm、2-300μm、2-350μm、2-400μm、2-450μm、2-500μm、2-600μm、2-700μm、2-800μm、2-900μm或2-1000μm。例如,引物交换反应中的dntp浓度可以为2μm、5μm、10μm、15μm、20μm、25μm、30μm、35μm、40μm、45μm、50μm、55μm,60μm,65μm,70μm,75μm,80μm,85μm,90μm,95μm、100μm、105μm、110μm、115μm、120μm、125μm、130μm、135μm、140μm、145μm、150μm、155μm、160μm、165μm、170μm、175μm、180μm、185μm、190μm、195μm或200μm。在一些实施方案中,引物交换反应中的dntp浓度为10-20μm、10-30μm、10-40μm、10-50μm、10-60μm、10-70μm、10-80μm、10-90μm或10-100μm。
在一些实施方案中,使用dntp变体。例如,per系统可以使用热启动/cleanampdntp,硫代磷酸酯dntp或荧光dntp。可以使用其它dntp变体。因为一些修饰的dntp不利于正常(未修饰的)dna-dna结合,所以发夹回复置换(backdisplacement)过程可随其使用而增加。类似地,在一些实施方案中,可以使用由不同类型的核酸组成的发夹(例如lna,rna或散布的修饰碱基,例如甲基dc或supertidt修饰),以通过形成比合成的引物相对于催化分子更强的键来提高per的速度。
在一些实施方案中,催化分子与生物分子如荧光团或蛋白质共价连接。在一些实施方案中,催化分子含有生物素修饰,因此它们可以通过生物素-链霉抗生物素蛋白键拴系到表面。在一些实施方案中,催化分子含有例如在亚结构域之一中的叠氮化物修饰的修饰,其允许它们通过点击化学与其它分子例如炔烃共价连接。本公开涵盖其它化学和生物学连接。
应当理解,本公开的核酸不是天然存在的。因此,核酸可称为“改造的核酸”。“改造的核酸”是一种核酸(例如至少两个核苷酸共价连接在一起,并且在一些情况下,含有磷酸二酯键,称为磷酸二酯“主链”),其不是天然存在的。改造的核酸包括重组核酸和合成核酸。“重组核酸”是通过连接核酸(例如分离的核酸、合成核酸或其组合)构建的分子,并且在一些实施方案中,可以在活细胞中复制。“合成核酸”是扩增或通过化学或通过其它方式合成的分子。合成核酸包括化学修饰或以其它方式修饰的那些,但可以与天然存在的核酸分子碱基配对(也称为“结合”,例如瞬时或稳定地结合)。重组和合成核酸还包括由前述任一种的复制产生的那些分子。
虽然改造的核酸作为整体不是天然存在的,但它可以包括野生型核苷酸序列。在一些实施方案中,改造的核酸包含从不同生物获得的核苷酸序列(例如从不同物种获得)。例如,在一些实施方案中,改造的核酸包括鼠核苷酸序列,细菌核苷酸序列,人核苷酸序列,病毒核苷酸序列或前述序列中任何两种或更多种的组合。在一些实施方案中,改造的核酸含有一个或多个随机碱基。
在一些实施方案中,本公开改造的核酸可包含除磷酸二酯主链之外的主链。例如,在一些实施方案中,改造的核酸可包含磷酰胺,硫代磷酸酯,二硫代磷酸酯,o-甲基磷酰胺键,肽核酸或任何两种或更多种前述键的组合。如所指定的,改造的核酸可以是单链(ss)或双链(ds),或者改造的核酸可以包含单链和双链序列二者的部分。在一些实施方案中,改造的核酸含有三链序列的部分或其它非watson-crick碱基配对,例如g-四联体,g-四链体和i-基序。改造的核酸可包含dna(例如基因组dna,cdna或基因组dna和cdna的组合),rna或杂合分子,例如,其中核酸含有脱氧核糖核苷酸和核糖核苷酸(例如人工或天然)的任何组合,和包括尿嘧啶,腺嘌呤,胸腺嘧啶,胞嘧啶,鸟嘌呤,肌苷,黄嘌呤,次黄嘌呤,异胞嘧啶和异鸟嘌呤的两种或更多种碱基的任何组合。
本公开改造的核酸可以使用标准分子生物学方法生产(参见,例如greenandsambrook,molecularcloning,alaboratorymanual,2012,coldspringharborpress)。在一些实施方案中,使用gibson
本文提及dna折纸(origami)“支架(scaffold)”和“订书钉(staple)”链。这些是本领域已知的术语60。支架链典型地是长的单链核酸(例如dna),其长度大于200个核苷酸。订书钉链典型地是长度为200个核苷酸或更少的短单链核酸(寡核苷酸)。应当理解,在定义dna折纸系统的成分中,200个核苷酸的长度(长于或短于)本身并不重要,而是相对长度通常是重要的。dna支架链比多个较短的订书钉链更长,使得使用较短的短链(通过核苷酸碱基互补性)将较长的支架链折叠成任意形状(例如2d或3d结构)。
引物交换方法和应用
在胚胎发育过程中,动力学途径与环境信号相结合,产生复杂和功能性结构如器官和四肢的组装。本公开内容正是至少部分地基于动力学、信号传导和结构之间的这种紧密关系,以及生物系统中令人难以置信的可编程性和复杂性。迄今为止,在很大程度上不可能动态地创建形状以响应甚至接近能够用退火方案形成的结构的复杂性的环境信号。本文提供的引物交换技术弥补了这一差距。其中per可用作工具的几种应用的实例在图6、7、11和18中描述并在下面描述。
触发的结构生长和折叠
在一些实施方案中,引物交换反应(per)用于合成长的、固定长度的核酸链(图7a)。核酸链的长度取决于几个因素,包括反应中催化分子的长度和数量。每个催化反应(包括引物结合,引物的链置换延伸和分支迁移;参见图1)向生长的核酸链添加结构域。因为所有引物和催化分子成分相对较小,所以可以在其中完全合成的核酸支架的直接递送可能是不可行的拥挤的环境、例如固定细胞中的那些环境中改造链合成。
规定的1d生长(组装)的每个步骤添加新的引物序列(参见,例如图2的下图),因此引物交换反应能够用于通过任意数量的退火方案基于互补核酸结构域编程折叠形状(图7b)9、27、38、51–53。因为引物交换是等温操作的并且其动力学可以调节以在宽范围的温度和盐条件下操作,所以该方法可以用于触发复杂结构的组装。例如,这些复杂结构可用作cryo-em(电子显微镜)和其它类型成像的标记物,或用作蛋白质和其它生物分子的图案化的支架。
除了通过将支架链(例如长度大于100个核苷酸的长单链核酸)折叠成特定形状来组装结构之外,通过设计具有指数生长动力学的结构,可以使用引物交换反应以时间有效的方式构建大结构。例如,可以通过组装树枝状支架主链来生产大结构(图7c)。该主链在一些实施方案中可以折叠成不同的2d和3d形状。使用图10a-10c(参见实施例5)中描述的分支方法,例如,类似尺寸的结构可以在比线性支架合成方法短得多的时间段内组装,其中时间与主链的总长度成次线性比例。这允许在原位以时间有效得多的方式构建大型形状。
分子时钟和计时器
本公开的引物交换反应系统是模块化技术,其也可以用于改造合成系统,该系统能够测量任意分子事件(例如暴露于物理或化学信号)之间的时间并且将它们原位编码在dna中(作为“分子时钟”)。per能够用于直接在其天然环境中,并且在一些实施方案中,在没有任何可能破坏动力学的荧光标记或条形码以及同时检测多个信号的可能性的情况下测量信号。在一些实施方案中,per系统可以用于通过在感测事件之间的时间期间连续地将碱基附加到链的末端来记录时间。这些链的长度分布可以用作两个分子信号之间的时间量的指示物,并且该分布可以例如直接在变性凝胶上读出。因为可以用发夹浓度控制反应速度,所以可能测量跨越许多不同时间尺度的时间(例如几分钟到几小时或更长)。
除了测量信号之间的时间之外,可以使用引物交换反应,在一些实施方案中,随时间测量信号(称为“分子计时器”)(图11)。通过编程一组状态转换并修改每个状态转换的催化发夹的浓度,计时器可用于在输入信号发生后的特定时间间隔释放输出信号(输出引物与催化发夹解离)。这种类型的控制对于可编程材料和合成基因调控是有用的。此外,这种时间延迟可以跨多个时间段编程。
基于per的分子时钟和计时器系统可以在一些实施方案中按比例放大(多重检测),以同时检测和启动多个信号。这些系统以几种方式执行复杂的逻辑运算并影响基因调控网络,增加了合成生物学家可用的无数工具。另外,例如,信号和基因的时序控制可用于对生物分子进行体外实验,以研究它们的功能特性。
环境响应记录器
生物从单个细胞发育而来,从基因组程序中自组装,分子信号指导专门发育途径的遍历。受这些令人难以置信的有效和稳健的发育途径的启发,通过本公开的引物交换反应系统提供合成的发展自组装,由此由于信号浓度的时间和空间变化而形成不同的结构。使用上述结构合成框架和信号检测和启动能力,编程发展自组装,在一些实施方案中,由此结构响应于它们随时间遇到的不同环境信号而生长和改变形状。这些结构将遵循规定的动力学途径,同时动态记录分子事件并组装成特定结构,用于成像、系统生物学和生物信号处理中的应用。这些反应等温地进行,并且可以改变反应动力学以匹配系统的所需操作温度、速度和离子浓度。这种方法的成功实施引入了合成自组装领域的深刻范式转变,因为它具有适应不同环境条件的能力,并且能够在很少或没有人为干预的情况下长时间运行。例如,本文提供的引物交换反应系统可用于图案化和指导结构的生长作为用于成像的标记物、构建用于图案化生物分子的支架乃至合成形成在特定靶分子周围以捕获它的结构。
检测到的进入核酸链中的信号的per编码也能够扩展到实现能够长时间记录多个信号的“自动收报机带”。因为遍历反应步骤的每个分子的整个历史记录在转录物中,并且这些分子具有特定且一致的动力学性质,所以这些自动收报机带转录物能够用于随时间计算溶液中的任意信号的精确时间轨迹。这些系统可用于在原位和体内随时间跟踪系统动态,并提供能够在任意时间内记录任意信号的无与伦比的技术。
图41a显示了检测靶链x的分子记录系统的实施例,其包含以下四个成分的:信号发夹(si)、封闭信号发夹的引物结合位点的保护链a*、引物a和时钟发夹(cl)。“保护链”是指与靶和包含信号序列的催化分子的3'立足点结构域二者结合的单链核酸。在一些实施方案中,保护链长于3'立足点结构域(例如长至少1、2、3、4、5、6、7、8、9或10个核苷酸)。图41b显示了如何通过时钟发夹(c1)进行单个时钟并入,其被编程以将另外的结构域串联到以a结束的引物序列上。因为在整个实验中该时钟以恒定浓度包含,所以在生长的引物链中并入a的速率在时间上保持大致恒定。这意味着最终转录物中a结构域的数量和位置反映了分子事件之间经过的时间量,类似于图13中描述的分子时钟拟合。如图41c所示,例如,在存在靶的情况下,信号发夹变得有活性。当靶x与保护链上暴露的单链区结合并通过立足点交换反应将该保护链从信号发夹上置换时,就会发生这种情况。这暴露了发夹上的引物结合位点a*,使得信号发夹可以将序列sa附加到以结构域a结束的引物上。序列s表示对靶x特异的条形码序列,其用于指示发生该反应(信号并入)而不是时钟反应(时钟并入)。因为立足点交换系统达到可预测的平衡状态,所以在给定时间点的信号并入速率反映了此时靶x的浓度。因此,信号与时钟并入的相对速率能够用于随实验时间拟合靶b的浓度曲线。如图41d所示,例如,当不存在靶时,在转录物中没有信号条形码,因此所有的并入都是时钟。然而,当存在靶时,信号并入(sa)被插入到重复结构域b的串联中,并且它们的时变频率适合于恢复靶b的时变浓度值。
图42a描绘了检测靶链b的分子记录系统的实例,其包括以下三个成分:折叠成最初封闭引物结合位点a*的构型的信号发夹(si)、引物a和时钟发夹(cl)。图42b显示了如何通过时钟发夹(cl)进行单个时钟并入的实例,其被编程以将另外的结构域串联到以a结束的引物序列上。因为在整个实验中该时钟以恒定浓度包含,所以在生长的引物链中并入a的速率在时间上保持大致恒定。因此,最终转录物中a结构域的数目和位置反映了分子事件之间经过的时间量,类似于图13中描述的分子时钟拟合。如图42c所示,例如,在存在靶的情况下,信号发夹变得有活性。当靶x与信号发夹上暴露的单链环区结合并通过立足点交换反应置换封闭引物结合位点a*的链时,就会发生这种情况。这允许信号发夹将序列sa附加到以结构域a结束的引物上。序列s表示对靶x特异的条形码序列,其用于指示发生该反应(信号并入)而不是时钟反应(时钟并入)。因为立足点交换系统达到可预测的平衡状态,所以在给定时间点的信号并入速率反映了此时靶x的浓度。因此,信号与时钟并入的相对速率能够用于随实验时间拟合靶x的浓度曲线。如图42d所示,例如,当不存在靶时,在转录物中没有信号条形码,因此所有并入都是时钟。然而,当存在靶时,信号并入(sa)被插入到重复结构域a的串联中,并且它们的时变频率适合于恢复靶x的时变浓度值。通常,具有靶特异性信号序列条形码的附加信号发夹能够用于随时间对多个不同靶的多重监测。图20中显示了自动收报机带的双信号实例。
另外的应用
cryo-em标记物原位触发合成。对蛋白质的大的、结构上合理的dna纳米结构的触发生长可以用作使用cryo-em进行结构测定的标记物。利用已知形状的不对称标记物,蛋白质结构的类平均(classaveraging)和3d重构的过程应该更容易。这种方法特别有用,例如,用于绘制以其他方式难以成像的蛋白质的结构。
污染物质的长期环境监测。引物交换反应由dntp驱动,可以运行很长一段时间。长时间测量溶液中污染物水平的分子记录器可以通过将引物交换反应物拴系在暴露于环境的室的侧面来实现。
ph、温度计的构建。ph和温度的变化对聚合酶和反应动力学具有可预测的影响,因此per系统能够用于跟踪它们所暴露的温度或ph。因为ph和温度影响聚合酶动力学,所以可以使用外部时钟(例如所提出的光门控机制)来获得精确的时间轨迹。
瞬时材料的程序性老化。例如,使用可编程的蛋白质合成脉冲,引物交换系统能够检测特定的环境信号,然后首先产生使溶液聚合的试剂,然后在一组量的时间后产生完全破坏聚合的底物的试剂。
按照时间表进行的药物递送。通过实施反应循环,可以用引物交换反应编程启动周期的振荡。该重复状态可以激活特定信号的周期性释放,该信号随后可以通过立足点开关转换为蛋白质信号。由于引物交换反应和立足点开关的模块化,该系统的输出可以是在特定间隔后并且仅响应于特定的环境信号产生治疗性蛋白质。
用于环境控制的条件基因合成。用rna实现和/或在基因组中编码的引物交换反应能够用于通过合成功能性rna调节剂或mrna转录物来记录和调节基因表达。在一些实施方案中,per用于条件基因调控。
毒素的原位触发包封。因为结构的生长是由引物触发的并且可以以环境信号的存在为条件,所以还提供了信号检测方法,其中特定致病标记物的检测紧接着是围绕和灭活毒素的结构的触发生长。
原位信号放大。例如,长端粒多联体的生长可用于原位信号放大以用于成像。多个端粒化反应可以并行进行以复用信号放大。参见,例如图25。
蛋白质检测。通过使用有条件地暴露必需的引物结合位点的适体序列,还可以调整条件引物交换反应以检测除氨基酸之外的蛋白质。这使得能够实现大范围的分子行为,因为合成可以与许多不同的分子实体连接以用于记录和编程行为。
体内应用
在一些实施方案中,在体内实施per级联使得能够在细胞内可编程地动态合成核酸。这些系统能够在单细胞水平上响应,产生根据特定细胞环境合成的转录物群。这些转录物可以是随时间检测的信号的记录,或者它们可以是功能性rna转录物,其使细胞具有针对特定细胞条件的程序性响应。
作为实例,利用真核细胞中的rnaper,诸如核定位信号(nls)序列的定位标记物可以在其离开细胞核之前动态地附加到mrna序列以将合成的蛋白质引导回细胞核中。该空间可编程性可用于蛋白质的特异性激活或失活。
通常,per级联在很大程度上与序列无关,因此任意序列可以从头合成或附加到现存的寡核苷酸上。每种细胞类型都需要链置换聚合酶,理想情况下是细胞中已提供的rna聚合酶,以限制对细胞过程的破坏。用于实现发夹终止序列的任何蛋白质也应该被并入。一个实例是使用dcas9蛋白,可能进化为具有更强的结合,因为它已成功地引入原核生物和真核生物。
可以通过使用动态合成的输出序列来监测实时合成以激活或抑制荧光报告基因。例如,可以合成dna酶以切割gfp蛋白的mrna。或者,可以合成指导rna以抑制报告基因。如果不需要立即恢复在转录物中编码的序列信息,则可以稍后对在细胞内产生的记录进行测序以提取时序记录信息。
因为per发夹具有催化作用,所以反应速率可以在体外调节几个数量级。在细胞中,可以根据必要的合成速率调整质粒拷贝数等因子以控制浓度。在一些情况下,可首先在体外转录/翻译系统中评估性能,例如purexpress长度分辨率和/或细胞提取物(如果可用于所需细胞类型)。
作为一个实例,合成的链可以通过切割mrna,产生背景特异性指导rna,合成反义转录物或通过激活sirna途径来抑制基因。mrna转录物也可以以模块化方式合成或延伸,以根据需要产生具有针对每种背景定制的结构域的新蛋白质。可以随时间检测和记录许多类型的调节核酸和蛋白质。
可以动态合成序列并响应不同的环境条件的具有高度可编程的体内合成平台在合成生物学中具有主要应用。靶蛋白可配备有引物,其记录定位于细胞不同部分的发夹,以阐明关于目标靶的时空信息。通常,体内per带来了一种全新的动态和响应性核酸合成方法,其高度可编程性使得该技术可适用于许多不同的环境的可能性。
分子马达
本文还提供了在分子水平上检查分子环境的分子马达系统。为此,单位操作应以局部规模进行,后续操作应沿相邻位点顺序进行。单位交互应具有明确定义的距离依赖性行为。单位步骤的反应速率取决于爬行者和模板的不同部分的长度以及位点之间的距离。“分子爬行者”是一种蛇样分子物种,它在轨道周围漫游,当它从第一个轨道位点生长到最终轨道位点时跨越整个轨道(参见,例如图31a)。当爬行者在位点之间移动时,它从位点复制信息并将信息记录在其生长的主体内(核酸)。“分子步行者”在位点之间移动,在每个步骤之后离开前一位点(参见,例如图32a)。当沿着轨道行进时,步行者生长它的主体,复制并保留来自轨道位点的信息。
对单个位点的单位操作的机制显示在图31a和32a的顶部的行中。在两个系统中,通过引物(输入信号;图31a中的‘a’和图32a中的‘1’)与其位点的互补引物结合区域的结合来引发反应。下一步骤是聚合酶沿着模板延伸引物,直到聚合酶撞到“聚合酶终止物”点(终止聚合的分子)。在聚合酶系统中提供dna碱基单体(dntp)以添加到新合成的部分中。例如,终止点可以通过以下两种方式之一进行编码。可以加入非核苷酸化学间隔物(例如三乙二醇间隔物)作为终止物,或者可以在系统中使用碱基的子集,并且将排除的碱基用作终止物。终止聚合的其它分子在本文其它地方描述。例如,如果使用模板中具有a、t和c的三字母代码,则在系统中提供它们的互补碱基单体a、t和g,并且g嵌入在模板的末端。聚合酶不能延伸新链,因为该系统没有互补碱基单体c。当聚合酶完成新结构域的合成并到达终止物点时,它离开模板(与模板解离)。然后,由于新合成的结构域与模板共享相同的序列,因此可以进行随机游走分支迁移过程。如果原始模板置换新合成的结构域,则暴露出用于下一反应的新引物。
在单位操作之后,在两个分子马达系统中,第一位点具有新合成的结构域,其现在能够充当下一位点的引物。由于分子马达分子(例如爬行者或步行者)仍然锚定在第一位点——分子中相关部分的长度将被设计为满足这种条件——新的引物仅在邻近的位点上局部起作用。两个系统之间的下一位点的移动机制是不同的。在分子爬行者系统中,新引物通过互补性结合下一位点的引物结合区域,并重复单位操作。结果是沿着第二位点(图31a的中间行)具有延伸的主体的爬行者。注意,应该保护引物结合结构域(例如第二位点的结构域b*)以防止引物延伸,以防止爬行者在轨道中间自发释放;这种保护通过并入不可延伸的碱基(例如,在链的3'末端的反向dt)来实现。
在分子步行者系统中,步行者分子在当前和下一位点之间经历竞争性分支迁移过程(图32a的中间行)。如果第二位点置换第一位点的相应部分,则可以将步行者运输到第二位点;引物的长度应该设计成使得一个引物与其互补序列的结合(例如,结构域1和1*之间)足够弱以从前一位点释放步行者,而两个连续引物与它们的互补序列的结合(例如,结构域1-2和2*-1*之间)足够强以将步行者保持在下一位点上。然而,由于步行者仍然含有与前一位点的引物结合区域互补的结构域,步行者的运动是可逆的(能够向后步行)。这可以是需要重新访问多个位点的情况下的特征,例如在迷宫求解中。注意,在步行者离开位点后,轨道位点会恢复其原始形式,从而可以重复使用。
在沿着三个相邻位点(图28a和29a的底行)重复步骤之后,蛇样爬行者现在跨越整个轨道,而步行者已经遍历到最终位点。用于回收记录和历史的爬行者的释放可以通过多种方法实现。在一个实例中,在记录反应结束时,可以添加“反向引物”以合成爬行者的互补拷贝,从而从模板轨道置换掉爬行者。例如,在蛇样爬行者的情况下具有结构域“d*”的引物,以及在步行者的情况下具有结构域“5*”的引物,可以启动这样的反向复制过程。作为另一实例,可以就在最终位点之后嵌入以自主方式指导反向复制过程的特殊类型的轨道位点。在相应情况下,含有结构域“d*”和“5*”的引物可以在特殊位点并入,以在马达接近时启动反向复制过程。作为又一实例,可以使用马达的基于热介导去杂交的更简单的机构。虽然系统范围的热量可使平台结构或某些成分变性,但是当例如与具有特异性引物的pcr扩增组合时,可以选择性检测靶信号。
在一些实施方案中,可以使用如本文提供的重复使用的探针生成可变长串联的条形码串,使得能够并行操作茎编码模板复制、释放和下游结合的相对慢的步骤。如图44a所示,除了结构域“a”的引物结合位点之外,探针还包括附加在探针3'末端的另一个引物结合位点(结构域“b”)。此外,在一些实施方案中,“终止物配对”是非天然碱基配对iso-dc:iso-dg(integrateddnatechnologies),并且未延伸引物在最后两个碱基之间含有硫代磷酸酯键。这些修饰在复制发夹模板“b-t”后使聚合酶停止(终止物配对),并使发夹探针和延伸引物的平衡状态发生偏倚,使得延伸的引物更可能是单链的并且发夹是封闭的。结构域“t”表示随机条形码或其它探针特异性信息。超过发夹探针的游离引物最初像通常一样结合探针并通过置换聚合酶延伸(图44b)。新结构域“b*”不是复制在apr中的回文结构域,而是能够结合在任何其它探针的游离3'端上结构域“b”。
在一些实施方案中,调整结构域长度或序列以限制或防止自身结合。当“b*”与另一个探针结合时,所显示的几种方法中的任何一种(探针上的甲基化rna碱基,“b*”和“b”之间末端核苷酸的错配,探针上的iso-dc核苷酸和每个“a*”引物5'端上相应的iso-dg核苷酸,或类似物)能够用于减慢或终止进一步聚合。例如,在甲基化rna核苷酸的情况下,发现如bst(integrateddnatechnologies)或bsm(thermofisher)的聚合酶从进一步延伸减慢。在错配核苷酸的情况下,这允许dna末端的紧密但不完美的匹配,减慢或阻止进一步聚合,但仍允许“粗心”连接酶(例如t4dna连接酶)连接上这种连接。如果减慢/终止物失效,则反应像该步骤之前的爬行者设计的那样继续进行。与爬行者设计一样,新的串联条形码链可以通过偶尔结合低浓度、游离的“b”结构域链而终止,这导致聚合酶复制新的串联链,从所有探针释放它,因此重新生成探针以供进一步使用(图44d)。与爬行者一样,最终结果是串联条形码拷贝的一定程度的随机拼凑,首先与它们各自的探针结合,但后来释放并以可能不同的模式重复(图44e)。像以前一样,可以通过pcr、杂交、下一代测序或其它工具分析链含量。图44f提供了探针序列的实施例,其中表示了相应的结构域。
在一些实施方案中,分子马达系统使用分子仪器对大量分子级靶进行平行“自下而上”检查。可以沿着给定靶的分子景观重复产生分子记录,每个分子记录用独特的dna条形码标记,而不干扰或破坏靶本身,并且随后通过高通量测序读取以用于以靶为基础的图像的计算重构。通过相同规模的样品和“仪器”,该技术解决了与基于显微镜的技术相关的许多挑战。例如,可以记录动态过程的高空间分辨率;与其靶标大小相同的分子仪器将具有“超锐利的分子视觉”。作为另一实例,每个靶(而不是靶物种)具有独特的条形码,分子马达系统提供高度多路复用以跟踪每个分子的空间和时间分布。作为又一实例,分子马达能够以大规模并行方式记录过程,从而使得能够实现超高通量分子成像。作为又一实例,分子马达能够原位接近分子靶,而没有自顶向下显微镜的结构或环境约束,并且避免了苛刻和破坏性的样品处理。分子马达系统以大规模并行的方式原位识别、跟踪和记录单个分子的景观,然后读取和重新组装信息的潜力,使得具有高精度的分子结构图像的计算重新创建成为可能。分子马达进一步使得能够定量空间内的分子靶标,这些分子靶标是不能通过显微镜分辨的,并且例如对来自各个网络的生物结构、真实的连通性和动态数据的超高分辨率可视化。分子马达系统通过提供非常有用的分子景观并行和多重检测工具改变基础生物学研究、药物发现和诊断。
在一些实施方案中,用于分子马达的轨道位点(例如爬行者分子和步行者分子)包括(i)未配对的3'立足点结构域和(ii)位于立足点结构域的5'的配对结构域,其通过置换链的核苷酸与含有立足点结构域的模板链的核苷酸之间的碱基配对形成。具有这种配置的轨道位点的实例如图31a和图32a所示。
参考图31a,未配对的3'立足点结构域(类似于上面讨论的per系统的立足点结构域)由‘a*’表示。含有未配对的立足点结构域的核酸链称为“模板链”。这是引物与其退火以引发聚合的链。模板链结合(配对、杂交)的相对链被称为“置换链”。置换链的亚结构域‘1’和‘b’分别与模板链的亚结构域‘1*’和‘b*’配对,形成位于立足点结构域5'的配对结构域。在通过引物与模板链的立足点结构域结合而引发的聚合期间,置换链最初被延伸产物置换。然而,随后,置换链置换延伸产物并再次结合模板链(称为分支迁移的过程)(顶部的行)。现在含有来自起始分子位点信息的延伸产物随后游离以起引物的作用并与另一个分子位点的立足点结构域结合,开始另一个延伸/分支迁移过程的循环(中间的行)。对于每个循环,将来自每个分子位点的信息记录添加到正在生长的核酸聚合物链,称为爬行者分子(底部的行,‘1+2+3’)。
参考图32a,未配对的3'立足点结构域由‘1*’+‘2*’表示。含有未配对的起始结构域的核酸链是引物‘1’与其退火以引发聚合的模板。模板链结合(配对、杂交)的相对链是置换链。置换链的亚结构域‘3’与模板链的亚结构域‘3*’配对,形成位于立足点结构域5'的配对结构域。在通过引物与模板链的立足点结构域结合而引发的聚合期间,置换链最初被延伸产物置换。然而,随后,置换链置换延伸产物并再次结合模板链(顶部的行)。现在含有来自起始分子位点信息的延伸产物随后游离以起引物的作用并与另一个分子位点的立足点结构域结合(在这种情况下,与立足点结构域的亚结构域‘3*’结合),开始另一个延伸/分支迁移过程的循环(中间的行)。对于每个循环,将来自每个分子马达的信息记录(分子马达之间的每个相互作用)添加到正在生长的核酸聚合物链,称为步行者分子(底部的行,‘1+2+3+4+5’)。
如上所述,用于分子马达的轨道位点通常包括未配对的(单链)3'立足点结构域和位于3'立足点结构域5'的配对(双链)茎结构域。在一些实施方案中,配对结构域直接与立足点结构域相邻。
轨道位点分子的长度可以变化。在一些实施方案中,分子马达具有25-300个核苷酸的长度。例如,轨道位点分子可以具有25-250、25-200、25-150、25-100、25-50、50-300、50-250、50-200、50-150或50-100个核苷酸的长度。在一些实施方案中,轨道位点分子具有30-50、40-60、50-70、60-80、70-90、80-100、100-125、100-150或100-200个核苷酸的长度。在一些实施方案中,轨道位点分子具有35、36、37、38、39、40、41、42、43、44、45、46、47、48、49、50、51、52、53、54、55、56、57、58、59、60、61、62、63、64、65、66、67、68、69、70、71、72、73、74、75、76、77、78、79、80、81、82、83、84、85、86、87、88、89、90、91、92、93、94、95、96、97、98、99或100个核苷酸的长度。在一些实施方案中,轨道位点分子长于300个核苷酸或短于25个核苷酸。
上文讨论的“立足点结构域”是指位于轨道位点分子3'末端的未配对的核苷酸序列,其与引物(或引物结构域)的核苷酸序列互补(并结合)。立足点结构域的长度可以变化。在一些实施方案中,立足点结构域具有5-40个核苷酸的长度。例如,立足点结构域可以具有2-35、2-30、2-25、2-20、2-15、2-10、5-35、5-30、5-25、5-20、5-15、5-10、10-40、10-35、10-30、10-25、10-20、10-15、15-40、15-35、15-30、15-25、15-20、20-40、20-35、20-30、20-25、25-40、25-35、25-30、30-40、30-35或35-40个核苷酸的长度。在一些实施方案中,立足点结构域具有5、10、15、20、25、30、35或40个核苷酸的长度。在一些实施方案中,立足点结构域具有10、11、12、13、14、15、16、17、18、19、20、21、22、23、24或25个核苷酸的长度。在一些实施方案中,立足点结构域长于40个核苷酸或短于5个核苷酸。
在一些实施方案中,立足点结构域具有亚结构域(例如两个亚结构域),例如,如图32a所示。在此类实施方案中,引物通常结合大部分3'亚结构域(‘1*’)。也就是说,在分子马达反应中使用的引物不一定跨越立足点结构域的整个长度——它可以仅结合到立足点结构域的一个亚结构域(的一部分)。
轨道位点分子的“配对结构域”是指位于轨道位点的未配对立足点结构域附近(和5')的配对的核苷酸序列(例如watson-crick核碱基配对)。通过模板链的结构域和置换链的结构域之间的碱基配对形成轨道位点分子的配对结构域。配对结构域的长度可以改变。在一些实施方案中,配对结构域具有5-40个核苷酸的长度。例如,配对结构域可以具有5-35、5-30、5-25、5-20、5-15、5-10、10-40、10-35、10-30、10-25、10-20、10-15、15-40、15-35、15-30、15-25、15-20、20-40、20-35、20-30、20-25、25-40、25-35、25-30、30-40、30-35或35-40个核苷酸的长度。在一些实施方案中,配对结构域具有5、10、15、20、25、30、35或40个核苷酸的长度。在一些实施方案中,配对结构域具有10、11、12、13、14、15、16、17、18、19、20、21、22、23、24或25个核苷酸的长度。在一些实施方案中,配对结构域长于40个核苷酸或短于5个核苷酸。
在一些实施方案中,通过置换聚合酶延伸引物(与引物结合位点结合)通过轨道位点中的终止聚合的分子或修饰的存在而终止。因此,在一些实施方案中,本公开的轨道位点分子马达包含终止聚合的分子或修饰。终止聚合的分子或修饰(“终止物”)典型地位于轨道位点的模板链的配对结构域中,使得聚合终止引物通过配对结构域的延伸。在一些实施方案中,终止聚合的分子是合成的非dna接头,例如,三甘醇间隔物,例如intspacer9(isp9)或spacer18(integrateddnatechnologies(idt))。应当理解,终止通过聚合酶的聚合的任何非天然接头可以如本文提供的那样进行使用。此类分子和修饰的其它非限制性实施例包括三碳键(/ispc3/)(idt),acryditetm(idt),腺苷酸化,叠氮化物,地高辛(nhs酯),胆固醇基-teg(idt),i-linkertm(idt)和3-氰基乙烯基咔唑(cnvk)及其变体。典型地,短接头(例如isp9)导致更快的反应时间,但并非总是如此。
在一些实施方案中,终止聚合的分子是单个或配对的非天然核苷酸序列,例如iso-dg和iso-dc(idt),它们分别是胞嘧啶和鸟嘌呤的化学变体。iso-dc将与iso-dg(但不与dg)碱基配对(氢键)。类似地,iso-dg将与iso-dc碱基配对但不与dc碱基配对。通过(在终止物位置)在发夹的相对侧将这些核苷酸并入核苷酸对中,聚合酶将停止,因为在该位置它在溶液中没有互补的核苷酸可添加。
在一些实施方案中,通过将反应中的dntp浓度(例如从200μm)降低至100μm、10μm、1μm或以下,改善了“终止物”修饰的性能效率。
包含终止聚合的分子或修饰通常在催化分子的双链结构域(例如发夹结构的茎结构域)中产生“凸起”,因为分子或修饰不配对。因此,在一些实施方案中,分子马达的轨道位点包括与分子或修饰相对的单一核苷酸(例如胸腺嘧啶),至少两个相同的核苷酸(例如胸腺嘧啶二聚体(tt)或三聚体(ttt))或非天然修饰。
除了轨道位点分子之外,分子马达系统包括称为输入引物或输出引物的引物,其被延伸成为爬行者分子和/或步行者分子。引物在本文其它地方描述。
图31a中描绘了分子马达反应的完整“步骤”。输入引物(‘a’)与轨道位点分子的立足点结构域(‘a*’)结合以开始反应。在含有聚合酶(例如链置换聚合酶)和dntp的反应溶液中与轨道位点分子结合后,起始引物延伸通过配对结构域,置换配对结构域的置换链(亚结构域‘1+b’)。然后,置换的链与延伸的引物竞争与其在模板链上的互补结合结构域的结合(再退火),从而置换延伸的输出引物。这完成了反应的一个步骤。然后,置换的输出引物“1+b”可以继续在反应的下一步骤中用作输入引物。
在一些实施方案中,引物或引物结构域(结合轨道位点分子的立足点结构域的核苷酸序列)具有10-50个核苷酸的长度。例如,引物或引物结构域可以具有10-45、10-40、10-35、10-30、10-25、10-20、10-15、15-50、15-45、15-40、15-35、15-30、15-25、15-20、20-50、20-45、20-40、20-35、20-30、20-25、25-50、25-45、25-40、25-35、25-30、30-50、30-45、30-40、30-35、35-50、35-45、35-40、40-50、40-45或45-50个核苷酸的长度。在一些实施方案中,引物或引物结构域具有10、15、20、25、30、35、40、45或50个核苷酸的长度。在一些实施方案中,引物或引物结构域具有15、16、17、18、19、20、21、22、23、24或25个核苷酸的长度。在一些实施方案中,引物或引物结构域长于50个核苷酸或短于10个核苷酸。应当理解,引物的全长取决于、至少部分取决于附加(聚合)序列的数量和长度,这取决于反应中存在的催化分子的数量和长度。
如本文提供的引物可以与可检测分子(例如发射可检测信号、例如荧光或化学发光信号的分子)连接(标记)。在一些实施方案中,标记是荧光团。与荧光团或其它荧光/化学发光分子连接的引物简称为“荧光引物”。可用于本文的荧光团的实例包括但不限于羟基香豆素,甲氧基香豆素,alexafluor,氨基香豆素,cy2,fam,alexafluor405,alexafluor488,荧光素fitc,alexafluor430,alexafluor532,hex,cy3,tritc,alexafluor546,alexafluor555,r-藻红蛋白(pe),罗丹明red-x,tamara,cy3.5581,rox,alexafluor568,red613,texasred,alexafluor594,alexafluor633,别藻蓝蛋白,alexafluor647,cy5,alexafluor660,cy5.5,trured,alexafluor680,cy7和cy7.5。本公开涵盖发射可检测信号的其它荧光团和分子。
在一些实施方案中,可检测分子连接至非引物的轨道位点分子。
在一些实施方案中,轨道位点分子连接至生物分子。生物分子包括例如核酸(例如dna或rna)和蛋白质。生物分子可以是治疗性、预防性、诊断性或成像分子。在一些实施方案中,生物分子是与疾病相关的或与药物相关的生物分子,例如癌症相关基因或蛋白质或fda批准的或潜在的药物靶。在一些实施方案中,生物分子是酶,抗原,受体,配体,膜蛋白,分泌蛋白或转录因子。
分子马达系统需要使用聚合酶。在一些实施方案中,聚合酶是dna聚合酶(dnap),例如具有dna链置换活性的dna聚合酶(链置换聚合酶)。“链置换”描述了置换合成期间遇到的下游dna的能力。可以如本文提供的那样使用的具有dna链置换活性的聚合酶的实例包括但不限于phi29dna聚合酶(例如neb#m0269),bstdna聚合酶,大片段(例如neb#m0275)或bsudna聚合酶,大片段(例如neb#m0330)。可以使用具有链置换活性的其它聚合酶。在一些实施方案中,聚合酶是rna聚合酶。
在一些实施方案中,聚合酶是phi29dna聚合酶。在这样的实施方案中,反应条件可以如下:补充有纯化的牛血清白蛋白(bsa)的1x反应缓冲液(例如50mmtris-hcl、10mmmgcl2、10mm(nh4)2so4、4mmdtt),ph7.5,在30℃孵育。
在一些实施方案中,聚合酶是bstdna聚合酶,大片段。在这样的实施方案中,反应条件可以如下:1x反应缓冲液(例如20mmtris-hcl、10mm(nh4)2so4、10mmkcl、2mmmgso4、1%
在一些实施方案中,聚合酶是bsudna聚合酶。在此类实施方案中,反应条件可以如下:1x反应缓冲液(例如50mmnacl、10mmtris-hcl、10mmmgcl2、1mmdtt),ph7.9,在37℃孵育。
引物、分子轨道和dntp在反应系统中的浓度可以根据例如特定应用和特定应用所需动力学的不同而变化。
引物在反应中的浓度可以为例如10nm至1000nm。在一些实施方案中,引物在反应中的浓度为10-20、10-30、10-40、10-50、10-60、10-70、10-80、10-90、10-100、10-125、10-150、10-200、25-50、25-75、25-100、25-150、25-200、50-75、50-100、50-150或50-200nm。在一些实施方案中,引物在反应中的浓度为100-200、100-300、100-400、100-500、100-600、100-70、100-800、100-900或100-1000nm。在一些实施方案中,引物在反应中的浓度为10、15、20、25、30、35、40、45、50、55、60、65、70、75、80、85、90、95、100、105、110、115、120、125、130、135、140、145、150、155、160、165、170、175、180、185、190、195或200nm。在一些实施方案中,引物在反应中的浓度为100、200、300、400、500、600、700、800、900或1000nm。引物在反应中的浓度可以低于10nm或高于1000nm。
反应中的轨道位点分子浓度可以为例如5nm至1000nm。在一些实施方案中,反应中的轨道位点分子浓度可以为5-10、5-20、5-30、5-40、5-50、5-60、5-70、5-80、5-90、5-100、5-125、5-150、5-200、10-50、10-75、10-100、10-150、10-200、25-75、25-100、25-125或25-200nm。在一些实施方案中,反应中的轨道位点分子浓度可以为10-200、10-300、10-400、10-500、10-600、10-70、10-800、10-900或10-100nm。在一些实施方案中,反应中的轨道位点分子浓度可以为10、15、20、25、30、35、40、45、50、55、60、65、70、75、80、85、90、95、100、105、110、115、120、125、130、135、140、145、150、155、160、165、170、175、180、185、190、195或200nm。在一些实施方案中,反应中的轨道位点分子浓度可以为10、20、30、40、50、60、70、80、90或100nm。反应中的轨道位点分子浓度可以低于5nm或高于1000nm。
反应中的引物与轨道位点分子之比可以为2:1至100:1。在一些实施方案中,引物与分子马达之比为2:1、3:1、4:1、5:1、6:1、:1、8:1、9:1、10:1、11:1、12:1、13:1、14:1、15:1、16:1、17:1、18:1、19:1或20:1。在一些实施方案中,引物与轨道位点分子之比为30:1、40:1、50:1、60:1、70:1、80:1或90:1。
不同轨道位点分子在反应中的数量是非限制性的。反应可以包含1-1010个不同的轨道位点分子(例如,各自具有特异性立足点结构域序列)。在一些实施方案中,反应包含1-10、1-102、1-103、1-104、1-105、1-106、1-107、1-108、1-109、1-1010个或以上的不同的轨道位点分子。在一些实施方案中,反应包含1-5、1-10、1-15、1-20、1-25、1-30、1-35、1-40、1-45、1-50、1-55、1-60、1-65、1-70、1-75、1-80、1-85、1-90、1-95、1-100、5-10、5-15、5-20、5-25、5-30、5-35、5-40、5-45、5-50、5-55、5-60、5-65、5-70、5-75、5-80、5-85、5-90、5-95、5-100、10-15、10-20、10-25、10-30、10-35、10-40、10-45、10-50、10-55、10-60、10-65、10-70、10-75、10-80、10-85、10-90、10-95或10-100个不同的轨道位点分子。在一些实施方案中,反应包含1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、28、19、20、21、22、23、24或25个不同的轨道位点分子。例如,如果它们的立足点结构域彼此不同,则轨道位点分子彼此不同。
例如,可以通过改变温度、时间、缓冲液/盐条件和脱氧核糖核苷酸三磷酸(dntp)浓度来控制反应的动力学。与大多数酶一样,聚合酶对许多缓冲条件敏感,包括离子强度、ph和存在的金属离子类型(例如钠离子vs.镁离子)。因此,进行反应的温度可以在例如4℃至65℃(例如4℃、25℃、37℃、42℃或65℃)变化。在一些实施方案中,进行反应的温度为4-25℃、4-30℃、4-35℃、4-40℃、4-45℃、4-50℃、4-55℃、4-60℃、10-25℃、10-30℃、10-35℃、10-40℃、10-45℃、10-50℃、10-55℃、10-60℃、25-30℃、25-35℃、25-40℃、25-45℃、25-50℃、25-55℃、25-60℃、25-65℃、35-40℃、35-45℃、35-50℃、35-55℃、35-60℃或35-65℃。在一些实施方案中,反应在室温下进行,而在另外的实施方案中,反应在37℃进行。
可以进行反应(孵育)30分钟(min)至24小时(hr)。在一些实施方案中,使反应进行10min、35min、40min、45min、50min、55min、60min、1hr、2hr、3hr、4hr、5hr、6hr、7hr、8hr、9hr、10hr、11hr、12hr、18hr或24hr。
反应中dntp的浓度可以为,例如,2-1000μm。在一些实施方案中,反应中的dntp浓度为2-10μm、2-15μm、2-20μm、2-25μm、2-30μm、2-35μm、2-40μm、2-45μm、2-50μm、2-55μm、2-60μm、2-65μm、2-70μm、2-75μm、2-80μm、2-85μm、2-90μm、2-95μm、2-100μm、2-110μm、2-120μm、2-130μm、2-140μm、2-150μm、2-160μm、2-170μm、2-180μm、2-190μm、2-200μm、2-250μm、2-300μm、2-350μm、2-400μm、2-450μm、2-500μm、2-600μm、2-700μm、2-800μm、2-900μm或2-1000μm。例如,反应中的dntp浓度可以为2μm、5μm、10μm、15μm、20μm、25μm、30μm、35μm、40μm、45μm、50μm、55μm、60μm、65μm、70μm、75μm、80μm、85μm、90μm、95μm、100μm、105μm、110μm、115μm、120μm、125μm、130μm、135μm、140μm、145μm、150μm、155μm、160μm、165μm、170μm、175μm、180μm、185μm、190μm、195μm或200μm。在一些实施方案中,反应中的dntp浓度为10-20μm、10-30μm、10-40μm、10-50μm、10-60μm、10-70μm、10-80μm、10-90μm或10-100μm。
在一些实施方案中,可以使用dntp变体。例如,分子马达系统可以热启动/cleanampdntp,硫代磷酸酯dntp或荧光dntp。可以使用其它dntp变体。
在一些实施方案中,基于单分子水平的单个蛋白质分子或纯化的蛋白质样品的鉴定,分子马达可用于蛋白质“指纹图谱识别”。例如,蛋白质的序列信息可以转换为核酸的序列信息,然后可以由分子爬行者记录和报告。
在一些实施方案中,通过以下方法(图40),实现蛋白质指纹识别。(1)蛋白质分子附着在表面上并变性和拉伸。蛋白质分子可以通过n-末端或c-末端特异性化学偶联方法固定在表面上。表面结合的蛋白质分子可以被常见的变性剂如尿素或十二烷基硫酸钠变性,并且可以通过施加外力(例如通过在远离表面的端部附加磁珠并施加磁场或通过使用带电粒子和类似的方式的电场)来拉伸。(2)氨基酸残基由含有独特标识符的dna链特异性标记。dna和氨基酸残基之间的化学偶联可以通过氨基酸特异性化学修饰方法完成。例如,赖氨酸残基可以用nhs-酯化学修饰,半胱氨酸残基可以选择性地与马来酰亚胺基团相互作用。与氨基酸结合的dna链可以仅编码靶氨基酸的身份信息,或者还包括特定位点的独特分子信息(例如通过随机序列)。(3)分子爬行者检查标记的位点并记录序列信息。分子爬行者基于允许下游分子成分以编程方式原位合成的原理操作,从靶分子复制信息。成群的分子爬行者在空间中漫游,以大规模并行的方式检查和报告分子景观的定量和几何信息。多个爬行者可以对同一靶标执行操作并生成部分冗余记录。(4)记录被释放和收集。记录可以通过原位合成互补链或通过热介导的去杂交来释放。(5)通过聚合酶链反应和凝胶电泳或通过下一代测序分析回收的记录。(6)来自记录的冗余和部分重叠的序列信息用于重构标记的氨基酸的序列。标记的氨基酸代表靶蛋白的完整氨基酸序列的子集。然后将该子序列信息与来自全基因组测序的所有遗传鉴定的蛋白质编码序列的文库进行比较和匹配,从而揭示该蛋白质的身份。
分子标尺
生物学研究需要能够报告关于分子相互作用的定量、灵敏和系统级信息的工具。这些工具必须处理这种相互作用的绝对数量和种类,同时还能够在单分子水平上长时间报告相互作用。本文还提供了称为“分子标尺”的分子记录系统,其通过记录dna分子内的距离信息来记录靶分子之间的纳米级距离(图31a-31b)。产生的dna记录的长度直接对应于所测量的距离。此外,每个dna记录编码靶分子的身份作为其序列的一部分。一轮的记录生产过程如下(参见图34b):(a)靶接种有dna记录的前体。(b)前体借助于链置换聚合酶和催化分子(例如催化发夹)生长,直到它们遇到伴侣核酸。伴侣杂交并延伸至记录中,每个使用另一个作为模板。(c)释放记录并靶被重新开始,使得能够进行进一步多轮的记录。多轮记录导致记录与多个邻居的距离以及距离的变化/差异。
在自主产生的dna记录(一元编码)长度中编码距离,并且对于每个靶配对产生多个记录。分子标尺能够保持改变距离的轨道并且报告距离分布(超分辨率拟合)。通过凝胶电泳报告所产生的记录的读出值,使得它适合几乎任意的研究实验室。
分子标尺能够记录靶分子在溶液中或固相或原位的距离。分子标尺几乎使得任何实验室能够使用传统且廉价的试剂和仪器进行分子距离测量。由于输出是dna分子形式,其可以简洁地编码大量信息,因此该技术具有大规模多重检测能力。根据特定需要,可以通过快速和常规测定(例如凝胶电泳)读取输出,或者可以利用下一代测序的高通量和单分子功能。这些特征使得本文提供的分子标尺技术例如能够测定蛋白质相互作用,研究蛋白质复合物组装并揭示染色体构象。
在一些实施方案中,分子标记包含多个与结合到靶生物分子的催化分子相互作用的游离(未结合)催化分子(如上所述)。这些相互作用由分子标尺反应中存在的引物引发,并且由置换的延伸产物介导(在一个循环中的输出引物在随后循环中起输入引物的作用)。分子标尺系统的实例如图36a-36h所示。
每个靶生物分子(灰色圆圈和黑色圆圈)与催化分子(在该实例中为发夹)连接(图36a)。该反应还包括与催化分子之一的立足点结构域结合的引物,和与另一个催化分子的立足点结构域结合的另一引物(图36b)。在链置换聚合酶和dntp存在下,引物与立足点结构域的结合引发每个分子的延伸过程。引物延伸通过配对结构域,破坏配对结构域中的碱基配对。置换的链与延伸产物竞争以重新形成原始配对结构域(图36c),然后置换的延伸产物用作与反应中存在的另外的游离催化分子结合的引物(图36d)。将置换的延伸产物与游离催化分子结合(图36e)通过游离催化分子的配对结构域引发聚合,随后置换进一步延伸的产物(现在含有来自与生物分子结合的催化分子的信息(dna),和来自自由催化分子的信息(dna))(图36f)。该过程在反应中继续与其它自由催化分子一起进行,每个循环向生长与生物分子连接的催化分子的聚合物添加新信息,直至两个聚合物相遇并相互结合(图36g)。此时聚合反应通过聚合物的长度进行,以形成所有分子标尺相互作用的双链核酸记录(图36h)。核酸记录的长度对应于两种生物分子之间的距离。
图37a显示了记录双链dna棒两端之间距离的分子标尺的另一实例。dna序列结构域用字母表示,有时用下标表示(例如s,b,a1等)。“*”符号表示结构域的互补序列(例如,r*是r的互补序列)。记录自主进行,但可以在概念上理解为分四步进行。(1)接种:前体与dna棒杂交。(2)激活:前体借助于链置换聚合酶(例如bst大片段)和dntp复制条形码序列(在该实例中为a1或a2)和激活结构域域(rrr或r*r*r*)。它们被“终止序列”(此处为s或s*)终止。终止序列是一个或多个dna碱基/核苷酸(例如ggg)的段(stretch),但在溶液中没有提供相应的核苷酸(例如dgtp)。因此,它们能够终止聚合酶进一步复制核苷酸。可以使用终止聚合的其它分子。(3)生长:激活结构域(此处为rrr和r*r*r*)允许前体与催化发夹相互作用以重复添加序列结构域(此处为r和r*)。(4a)释放:当前体生长到足够长度时,它们可以彼此相遇并通过互补结构域(这里是r和r*)杂交。然后聚合酶可以延伸它们,每个使用另一个作为模板,直到它们被释放到溶液中。释放的记录是dna棒末端之间距离的量度。(4b)重新开始:dna棒现在可以接种进一步的前体并产生更多记录,导致反复测量距离。
图37b和37c显示生长步骤(图37a中的步骤3)的实例的更多细节。在生长步骤中,将短序列结构域(r或r*)重复并自主地添加到激活的前体分子的末端。激活的前体(图37a中的a1rrr和a2r*r*r*)与催化生长发夹的立足点杂交。借助于链置换聚合酶和dntp,通过单个结构域(r或r*)延伸序列。它们通过终止序列(x)终止进一步延伸。此时,新复制的结构域可以从催化发夹中置换。短立足点结构域(rrr或r*r*r*)自发地从复合物中解离出来。解离的激活前体分子现在可以再次通过其末端三个结构域参与生长步骤(rrr或r*r*r*)。
分子标尺是上述催化分子(例如催化发夹分子)的变化形式。分子标尺(例如与靶生物分子结合的核酸分子)的长度可以变化。在一些实施方案中,分子标尺具有25-300个核苷酸的长度。例如,分子标尺可以具有25-250、25-200、25-150、25-100、25-50、50-300、50-250、50-200、50-150或50-100个核苷酸的长度。在一些实施方案中,分子标尺具有30-50、40-60、50-70、60-80、70-90、80-100、100-125、100-150或100-200个核苷酸的长度。在一些实施方案中,分子标尺具有35、36、37、38、39、40、41、42、43、44、45、46、47、48、49、50、51、52、53、54、55、56、57、58、59、60、61、62、63、64、65、66、67、68、69、70、71、72、73、74、75、76、77、78、79、80、81、82、83、84、85、86、87、88、89、90、91、92、93、94、95、96、97、98、99或100个核苷酸的长度。在一些实施方案中,分子标尺长于300个核苷酸或短于25个核苷酸。
上面讨论的“立足点结构域”是指位于分子标尺3'末端的未配对的核苷酸序列,并且与引物(引物的)的核苷酸序列互补(并结合)。立足点结构域的长度可以变化。在一些实施方案中,立足点结构域具有5-40个核苷酸的长度。例如,立足点结构域可以具有2-35、2-30、2-25、2-20、2-15、2-10、5-35、5-30、5-25、5-20、5-15、5-10、10-40、10-35、10-30、10-25、10-20、10-15、15-40、15-35、15-30、15-25、15-20、20-40、20-35、20-30、20-25、25-40、25-35、25-30、30-40、30-35或35-40个核苷酸的长度。在一些实施方案中,立足点结构域具有5、10、15、20、25、30、35或40个核苷酸的长度。在一些实施方案中,立足点结构域具有10、11、12、13、14、15、16、17、18、19、20、21、22、23、24或25个核苷酸的长度。在一些实施方案中,立足点结构域长于40个核苷酸或短于5个核苷酸。
分子标尺的“配对结构域”是指位于分子标尺的未配对立足点结构域附近(和5')的配对的核苷酸序列(例如watson-crick核碱基配对)。通过模板链的结构域和置换链的结构域之间的碱基配对或通过相同核酸链结构域之间的分子内碱基配对(形成发夹结构)形成分子标尺的配对结构域。配对结构域的长度可以改变。在一些实施方案中,配对结构域具有5-40个核苷酸的长度。例如,配对结构域可以具有5-35、5-30、5-25、5-20、5-15、5-10、10-40、10-35、10-30、10-25、10-20、10-15、15-40、15-35、15-30、15-25、15-20、20-40、20-35、20-30、20-25、25-40、25-35、25-30、30-40、30-35或35-40个核苷酸长度。在一些实施方案中,配对结构域具有5、10、15、20、25、30、35或40个核苷酸的长度。在一些实施方案中,配对结构域具有10、11、12、13、14、15、16、17、18、19、20、21、22、23、24或25个核苷酸的长度。在一些实施方案中,配对结构域长于40个核苷酸或短于5个核苷酸。
在一些实施方案中,通过链置换聚合酶延伸引物(与引物结合位点结合)通过分子标尺中终止聚合的分子或修饰的存在而终止。因此,在一些实施方案中,本公开的分子标尺包含终止聚合的分子或修饰。终止聚合的分子或修饰(“终止物”)典型地位于分子标尺的模板链上的配对结构域中,使得聚合终止引物通过配对结构域的延伸。在一些实施方案中,终止聚合的分子是合成的非dna接头,例如,三乙二醇间隔基,例如intspacer9(isp9)或spacer18(integrateddnatechnologies(idt))。应当理解,终止通过聚合酶的聚合的任何非天然接头可以如本文提供的那样进行使用。此类分子和修饰的其它非限制性实施例包括三碳键(/ispc3/)(idt),acryditetm(idt),腺苷酸化,叠氮化物,地高辛(nhs酯),胆固醇基-teg(idt),i-linkertm(idt)和3-氰基乙烯基咔唑(cnvk)及其变体。通常,短接头(例如isp9)导致更快的反应时间,但并非总是如此。
在一些实施方案中,终止聚合的分子是单个或配对的非天然核苷酸序列,例如iso-dg和iso-dc(idt),它们分别是胞嘧啶和鸟嘌呤的化学变体。iso-dc将与iso-dg(但不与dg)碱基配对(氢键)。类似地,iso-dg将与iso-dc碱基配对但不与dc碱基配对。通过(在终止物位置)在发夹的相对侧将这些核苷酸并入核苷酸对中,聚合酶将停止,因为在该位置它在溶液中没有互补的核苷酸可添加。
在一些实施方案中,通过将反应中的dntp浓度(例如从200μm)降低至100μm、10μm、1μm或以下,改善了“终止物”修饰的性能效率。
包含终止聚合的分子或修饰通常在催化分子的双链结构域(例如发夹结构的茎结构域)中产生“凸起”,因为分子或修饰不配对。因此,在一些实施方案中,分子标尺被设计为包括与分子或修饰相对的单一核苷酸(例如胸腺嘧啶),至少两个相同的核苷酸(例如胸腺嘧啶二聚体(tt)或三聚体(ttt))或非天然修饰。
分子标尺反应系统还包括称为输入引物或输出引物的引物。引物在本文其它部分描述。
分子标尺反应的完整“步骤”描绘在图37a-37c中。
在一些实施方案中,引物或引物结构域(结合分子标尺立足点结构域的核苷酸序列)具有10-50个核苷酸的长度。例如,引物或引物结构域可以具有10-45、10-40、10-35、10-30、10-25、10-20、10-15、15-50、15-45、15-40、15-35、15-30、15-25、15-20、20-50、20-45、20-40、20-35、20-30、20-25、25-50、25-45、25-40、25-35、25-30、30-50、30-45、30-40、30-35、35-50、35-45、35-40、40-50、40-45或45-50个核苷酸的长度。在一些实施方案中,引物或引物结构域具有10、15、20、25、30、35、40、45或50个核苷酸的长度。在一些实施方案中,引物或引物结构域具有15、16、17、18、19、20、21、22、23、24或25个核苷酸的长度。在一些实施方案中,引物或引物结构域长于50个核苷酸或短于10个核苷酸。应当理解,引物的全长取决于、至少部分取决于附加(聚合)序列的数量和长度,这取决于存在于反应中的游离(不与生物分子如蛋白质连接)催化分子的数量和长度以及与催化分子连接的靶生物分子之间的距离。
如本文提供的引物可以与可检测分子(例如发射可检测信号、例如荧光或化学发光信号的分子)连接(标记)。在一些实施方案中,标记是荧光团。与荧光团或其它荧光/化学发光分子连接的引物简称为“荧光引物”。可用于本文的荧光团的实例包括但不限于羟基香豆素,甲氧基香豆素,alexafluor,氨基香豆素,cy2,fam,alexafluor405,alexafluor488,荧光素fitc,alexafluor430,alexafluor532,hex,cy3,tritc,alexafluor546,alexafluor555,r-藻红蛋白(pe),罗丹明red-x,tamara,cy3.5581,rox,alexafluor568,red613,texasred,alexafluor594,alexafluor633,别藻蓝蛋白,alexafluor647,cy5,alexafluor660,cy5.5,trured,alexafluor680,cy7和cy7.5。本公开涵盖发射可检测信号的其它荧光团和分子。
在一些实施方案中,可检测分子连接至非引物的分子标尺。
在一些实施方案中,分子标尺连接至生物分子。生物分子包括例如核酸(例如dna或rna)和蛋白质。生物分子可以是治疗性、预防性、诊断性或成像分子。在一些实施方案中,生物分子是与疾病相关的或与药物相关的生物分子,例如癌症相关基因或蛋白质或fda批准的或潜在的药物靶。在一些实施方案中,生物分子是酶,抗原,受体,配体,膜蛋白,分泌蛋白或转录因子。
分子标尺反应需要使用聚合酶。在一些实施方案中,聚合酶是dna聚合酶(dnap),例如具有dna链置换活性的dna聚合酶(链置换聚合酶)。“链置换”描述了置换合成期间遇到的下游dna的能力。可以如本文提供的那样使用的具有dna链置换活性的聚合酶的实例包括但不限于phi29dna聚合酶(例如neb#m0269),bstdna聚合酶,大片段(例如neb#m0275)或bsudna聚合酶,大片段(例如neb#m0330)。可以使用具有链置换活性的其它聚合酶。在一些实施方案中,聚合酶是rna聚合酶。
在一些实施方案中,聚合酶是phi29dna聚合酶。在这样的实施方案中,反应条件可以如下:补充有纯化的牛血清白蛋白(bsa)的1x反应缓冲液(例如50mmtris-hcl、10mmmgcl2、10mm(nh4)2so4、4mmdtt),ph7.5,在30℃孵育。
在一些实施方案中,聚合酶是bstdna聚合酶,大片段。在这样的实施方案中,反应条件可以如下:1×反应缓冲液(例如20mmtris-hcl、10mm(nh4)2so4、10mmkcl、2mmmgso4、1%
在一些实施方案中,聚合酶是bsudna聚合酶。在此类实施方案中,反应条件可以如下:1×反应缓冲液(例如50mmnacl、10mmtris-hcl、10mmmgcl2、1mmdtt),ph7.9,在37℃孵育。
反应系统中引物、催化分子和dntp的浓度可以根据例如特定应用和该特定应用所需的动力学而变化。
引物在反应中的浓度可以为例如10nm至1000nm。在一些实施方案中,反应中引物的浓度为10-20、10-30、10-40、10-50、10-60、10-70、10-80、10-90、10-100、10-125、10-150、10-200、25-50、25-75、25-100、25-150、25-200、50-75、50-100、50-150或50-200nm。在一些实施方案中,反应中引物的浓度为100-200、100-300、100-400、100-500、100-600、100-70、100-800、100-900或100-1000nm。在一些实施方案中,反应中引物的浓度为10、15、20、25、30、35、40、45、50、55、60、65、70、75、80、85、90、95、100、105、110、115、120、125、130、135、140、145、150、155、160、165、170、175、180、185、190、195或200nm。在一些实施方案中,反应中引物的浓度为100、200、300、400、500、600、700、800、900或1000nm。反应中引物的浓度可以低于10nm或高于1000nm。
反应中分子标尺和/或催化分子的浓度可以为例如5nm至1000nm。在一些实施方案中,反应中分子标尺和/或催化分子的浓度为5-10、5-20、5-30、5-40、5-50、5-60、5-70、5-80、5-90、5-100、5-125、5-150、5-200、10-50、10-75、10-100、10-150、10-200、25-75、25-100、25-125或25-200nm。在一些实施方案中,反应中分子标尺和/或催化分子的浓度为10-200、10-300、10-400、10-500、10-600、10-70、10-800、10-900或10-100nm。在一些实施方案中,反应中分子标尺和/或催化分子的浓度为10、15、20、25、30、35、40、45、50、55、60、65、70、75、80、85、90、95、100、105、110、115、120、125、130、135、140、145、150、155、160、165、170、175、180、185、190、195或200nm。在一些实施方案中,反应中分子标尺和/或催化分子的浓度为10、20、30、40、50、60、70、80、90或100nm。反应中分子标尺和/或催化分子的浓度可以低于5nm或高于1000nm。
反应中分子标尺和/或催化分子之比可以为2:1至100:1。在一些实施方案中,分子标尺和/或催化分子之比可以为2:1、3:1、4:1、5:1、6:1、:1、8:1、9:1、10:1、11:1、12:1、13:1、14:1、15:1、16:1、17:1、18:1、19:1或20:1。在一些实施方案中,分子标尺和/或催化分子之比可以为30:1、40:1、50:1、60:1、70:1、80:1或90:1。
不同分子标尺和/或催化分子在反应中的数量是非限制性的。反应可以包含1-1010个不同的分子标尺和/或催化分子(例如,各自具有特异性立足点结构域序列)。在一些实施方案中,分子标尺反应包含1-10、1-102、1-103、1-104、1-105、1-106、1-107、1-108、1-109、1-1010个或以上的不同的分子标尺和/或催化分子。在一些实施方案中,反应包含1-5、1-10、1-15、1-20、1-25、1-30、1-35、1-40、1-45、1-50、1-55、1-60、1-65、1-70、1-75、1-80、1-85、1-90、1-95、1-100、5-10、5-15、5-20、5-25、5-30、5-35、5-40、5-45、5-50、5-55、5-60、5-65、5-70、5-75、5-80、5-85、5-90、5-95、5-100、10-15、10-20、10-25、10-30、10-35、10-40、10-45、10-50、10-55、10-60、10-65、10-70、10-75、10-80、10-85、10-90、10-95或10-100个不同的分子标尺和/或催化分子。在一些实施方案中,反应包含1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、28、19、20、21、22、23、24或25个不同的分子标尺和/或催化分子。例如,如果它们的立足点结构域彼此不同,则分子标尺彼此不同。
例如,可以通过改变温度、时间、缓冲液/盐条件和脱氧核糖核苷酸三磷酸(dntp)浓度来控制反应的动力学。与大多数酶一样,聚合酶对许多缓冲条件敏感,包括离子强度、ph和存在的金属离子类型(例如钠离子对镁离子)。因此,进行反应的温度可以在例如4℃至65℃(例如4℃、25℃、37℃、42℃或65℃)变化。在一些实施方案中,进行反应的温度为4-25℃、4-30℃、4-35℃、4-40℃、4-45℃、4-50℃、4-55℃、4-60℃、10-25℃、10-30℃、10-35℃、10-40℃、10-45℃、10-50℃、10-55℃、10-60℃、25-30℃、25-35℃、25-40℃、25-45℃、25-50℃、25-55℃、25-60℃、25-65℃、35-40℃、35-45℃、35-50℃、35-55℃、35-60℃或35-65℃。在一些实施方案中,反应在室温下进行,而在其它实施方案中,反应在37℃进行。
可以进行反应(孵育)30分钟(min)至24小时(hr)。在一些实施方案中,使反应进行10min、35min、40min、45min、50min、55min、60min、1hr、2hr、3hr、4hr、5hr、6hr、7hr、8hr、9hr、10hr、11hr、12hr、18hr或24hr。
dntp在反应中的浓度可以为例如2-1000μm。在一些实施方案中,反应中的dntp浓度为2-10μm、2-15μm、2-20μm、2-25μm、2-30μm、2-35μm、2-40μm、2-45μm、2-50μm、2-55μm、2-60μm、2-65μm、2-70μm、2-75μm、2-80μm、2-85μm、2-90μm、2-95μm、2-100μm、2-110μm、2-120μm、2-130μm、2-140μm、2-150μm、2-160μm、2-170μm、2-180μm、2-190μm、2-200μm、2-250μm、2-300μm、2-350μm、2-400μm、2-450μm、2-500μm、2-600μm、2-700μm、2-800μm、2-900μm或2-1000μm。例如,反应中的dntp浓度可以为2μm、5μm、10μm、15μm、20μm、25μm、30μm、35μm、40μm、45μm、50μm、55μm、60μm、65μm、70μm、75μm、80μm、85μm、90μm、95μm、100μm、105μm、110μm、115μm、120μm、125μm、130μm、135μm、140μm、145μm、150μm、155μm、160μm、165μm、170μm、175μm、180μm、185μm、190μm、195μm或200μm。在一些实施方案中,反应中的dntp浓度为10-20μm、10-30μm、10-40μm、10-50μm、10-60μm、10-70μm、10-80μm、10-90μm或10-100μm。
在一些实施方案中,使用dntp变体。例如,分子标尺系统可以使用热启动/cleanampdntp,硫代磷酸酯dntp或荧光dntp。可以使用其它dntp变体。
本公开还提供由如下编号段落涵盖的实施方案:
1.引物交换反应(per)系统,包含:
(a)起始催化发夹分子,其包含(i)未配对的3'立足点结构域;(ii)分子的3'亚结构域与分子的5'亚结构域之间的分子内核苷酸碱基配对形成的配对的茎结构域;和(iii)连接结构域;
(b)与3'立足点结构域互补并且结合的起始引物;和
(c)具有链置换活性的聚合酶。
2.段落1的per系统,其中连接结构域是环状结构域。
3.段落1或2的per系统,还包含(d)第二催化发夹分子,包含(i)未配对的3'立足点结构域;(ii)通过第二发夹分子的3'亚结构域与第二发夹分子的5'亚结构域之间分子内核苷酸碱基配对形成的配对的茎结构域;和(iii)环状结构域,其中第二催化发夹分子的3'立足点结构域与起始发夹分子的5'亚结构域互补。
4.段落3的per系统,还包含第二引物,其包含与位于第二发夹分子的未配对的3'立足点结构域中的核苷酸互补的核苷酸。
5.段落1-4任一项的per系统,还包含(e)第三催化发夹分子,包含(i)未配对的3'立足点结构域;(ii)通过第三发夹分子的3'亚结构域与第三发夹分子的5'亚结构域之间分子内核苷酸碱基配对形成的配对的茎结构域;和(iii)环状结构域,其中第三催化发夹分子的3'立足点结构域与第二发夹分子的5'亚结构域互补。
6.段落5的per系统,还包含与第三发夹分子的未配对的3'立足点结构域互补的第三引物。
7.段落5或6的per系统,还包含多个催化发夹分子,每个发夹分子包含:(i)未配对的3'立足点结构域;(ii)通过多个发夹分子中的发夹分子的3'亚结构域与多个发夹分子中的发夹分子的5'亚结构域之间分子内核苷酸碱基配对形成的配对的茎结构域;和(iii)环状结构域,其中每个发夹分子的3'立足点结构域与多个发夹分子中的一个其它发夹分子的5'亚结构域互补。
8.段落7的per系统,还包含多个引物,每个引物与多个发夹分子之一的未配对的3'立足点结构域互补。
9.段落1-8任一项的per系统,其中引物连接至可检测分子。
10.段落1-9任一项的per系统,其中引物连接至生物分子。
11.段落10的per系统,其中生物分子是蛋白质。
12.段落1-11任一项的per系统,其中聚合酶是phi29dna聚合酶、bstdna聚合酶或bsudna聚合酶。
13.段落1-12任一项的per系统,还包含脱氧核糖核苷酸三磷酸(dntp)。
14.引物交换反应(per)系统,包含:
(a)起始催化发夹分子,包含(i)未配对的3'立足点结构域;(ii)通过分子的5'亚结构域与分子的3'亚结构域之间分子内核苷酸碱基配对形成的配对的茎结构域;和(iii)环状结构域;
(b)第二催化发夹分子,包含(i)未配对的3'立足点结构域;(ii)通过第二发夹分子的3'亚结构域与第二发夹分子的5'亚结构域之间分子内核苷酸碱基配对形成的配对的茎结构域;和(iii)连接结构域,其中第二催化发夹分子的3'立足点结构域与起始发夹分子的5'亚结构域互补;和
(c)与起始发夹分子的未配对的3'立足点结构域互补的起始引物。
15.段落14的per系统,其中连接结构域是环状结构域。
16.段落14或15的per系统,还包含与第二发夹分子的未配对的3'立足点结构域互补的第二引物。
17.段落14或16的per系统,还包含具有链置换活性的聚合酶。
18.段落14-17任一项的per系统,还包含脱氧核糖核苷酸三磷酸(dntp)。
19.引物交换反应方法,包括:
在反应缓冲液中合并:
(a)起始催化发夹分子,包含(i)未配对的3'立足点结构域;(ii)通过分子的5'亚结构域与分子的3'亚结构域之间分子内核苷酸碱基配对形成的配对的茎结构域;和(iii)连接结构域;
(b)第二催化发夹分子,包含(i)未配对的3'立足点结构域;(ii)通过第二发夹分子的3'亚结构域与第二发夹分子的5'亚结构域之间分子内核苷酸碱基配对形成的配对的茎结构域;和(iii)环状结构域,其中第二催化发夹分子的3'立足点结构域与起始发夹分子的5'亚结构域互补;
(c)与起始发夹分子的未配对的3'立足点结构域互补的引物;
(d)具有链置换活性的聚合酶;和
(e)脱氧核糖核苷酸三磷酸(dntp),由此形成反应混合物;和
在导致核酸聚合、链置换和退火的条件下将反应混合物孵育足以产生单链核酸记录的时间。
20.段落19的引物交换反应,其中连接结构域是环状结构域。
21.引物交换反应,包括:
(a)在具有链置换活性的聚合酶和脱氧核糖核苷酸三磷酸(dntp)的存在下使输入引物接触催化分子,其中催化分子包含(i)未配对的3'立足点结构域和(ii)位于立足点结构域的5'并且通过置换链与含有立足点结构域的模板链之间的核苷酸碱基配对形成的配对结构域,其中输入引物与催化分子的3'立足点结构域互补;
(b)使引物通过催化分子的配对结构域延伸,由此将置换链置换并且形成延伸的输出引物;
(c)通过置换链与模板链之间的分子内核苷酸碱基配对置换从发夹分子延伸的输出引物;和
(d)在具有链置换活性的聚合酶和dntp的存在下使(c)的置换的延伸的输出引物接触第二催化分子,其中第二催化分子包含(i)未配对的3'立足点结构域和(ii)位于立足点结构域的5'并且通过置换链与含有立足点结构域的模板链之间的核苷酸碱基配对形成的配对结构域,且其中延伸的输出引物与第二催化分子的3'立足点结构域互补。
22.生产单链核酸的方法,包括:
在反应缓冲液中合并:
(a)起始催化发夹分子,包含(i)未配对的3'立足点结构域;(ii)通过分子的5'亚结构域与分子的3'亚结构域之间分子内核苷酸碱基配对形成的配对的茎结构域;和(iii)发夹结构域;
(b)多个不同发夹分子,每个发夹分子包含:(i)未配对的3'立足点结构域;(ii)通过多个发夹分子中的发夹分子的3'亚结构域与多个发夹分子中的发夹分子的5'亚结构域之间分子内核苷酸碱基配对形成的配对的茎结构域;和(iii)环状结构域,其中每个发夹分子的3'立足点结构域与多个发夹分子中的一个其它发夹分子的5'亚结构域互补;
(c)与起始发夹分子的3'立足点结构域互补的起始引物;
(d)具有链置换活性的聚合酶;和
(e)脱氧核糖核苷酸三磷酸(dntp),由此形成反应混合物;和
在导致核酸聚合、链置换和退火的条件下将反应混合物孵育足以产生比起始引物更长的单链核酸记录的时间。
23.段落22的方法,其中(e)中产生的单链核酸包含与多个发夹分子的茎结构域互补的核苷酸结构域的多联体。
24.段落22或23的方法,其中(e)中产生的单链核酸包含自我互补性结构域。
25.段落24的方法,还包含在导致(e)中产生的单链核酸通过自我互补性结构域之间的分子内核苷酸碱基配对而折叠的条件下孵育反应混合物。
26.段落22-25任一项的方法,还包含在导致(e)中通过单链核酸结构域与核酸订书钉链之间的分子内核苷酸碱基配对产生的单链核酸折叠的条件下孵育反应混合物。
27.包含段落1-13任一项的per系统的细胞。
28.段落27任一项的细胞,其中该细胞是原核细胞或真核细胞。
29.段落28任一项的细胞,其中该细胞是哺乳动物细胞。
30.包含至少两种核酸的载体,所述核酸各自编码催化发夹分子,其包含(i)未配对的3'立足点结构域;(ii)催化发夹分子的3'亚结构域与同一催化发夹分子的5'亚结构域之间的分子内核苷酸碱基配对形成的配对的茎结构域;和(iii)环状结构域。
31.段落30的载体,其中该载体编码至少3种、至少4种或至少5种核酸,它们各自编码催化发夹分子,其包含:(i)未配对的3'立足点结构域;(ii)催化发夹分子的3'亚结构域与同一催化发夹分子的5'亚结构域之间的分子内核苷酸碱基配对形成的配对的茎结构域;和(iii)环状结构域。
32.包含段落30或31的载体的细胞。
33.段落32任一项的细胞,还包含与催化分子的3'立足点结构域互补和结合的起始引物。
34.段落32或33任一项的细胞,还包含具有链置换活性的聚合酶。
35.组合物,包含:
(a)催化发夹分子,其包含:(i)未配对的3'立足点结构域;(ii)分子的3'亚结构域与分子的5'亚结构域之间的分子内核苷酸碱基配对形成的配对的茎结构域;和(iii)连接结构域,其中(a)(i)和(a)(ii)的结构域形成串联重复序列;
(b)至少一种其它催化发夹分子,其包含:(i)3'立足点结构域;(ii)分子的3'亚结构域与分子的5'亚结构域之间的分子内核苷酸碱基配对形成的配对的茎结构域;和(iii)连接结构域,其中(b)(i)和(b)(ii)的结构域形成被信号序列中断的串联重复序列,并且其中(b)(i)的3'立足点结构域不可逆地与保护链结合;和
(c)核酸引物,其包含与(a)的催化发夹分子的3'立足点结构域互补并且与(b)的催化发夹分子的3'立足点结构域互补的结构域。
36.段落35的组合物,其中(a)(iii)和/或(b)(iii)的连接结构域是环状结构域。
37.段落35的组合物,其中(a)(iii)和/或(b)(iii)的连接结构域包含至少一种共价交联的核苷酸。
38.段落35的组合物,其中连接结构域是具有至少10个核苷酸长度的稳定配对结构域。
39.段落35-38任一项的组合物,其中信号序列具有2-20个核苷酸的长度。
40.段落35-39任一项的组合物,其中信号序列对于靶分子特异性地标注条形码。
41.段落35-40任一项的组合物,其中引物包含实验特异性条形码。
42.段落35-41任一项的组合物,其中保护链任选地通过环状结构域连接至(b)的至少一个其它催化发夹分子的3'立足点结构域。
43.段落35-42任一项的组合物,还包含靶分子。
44.段落43的组合物,其中靶分子是核酸。
45.段落44的组合物,其中核酸是单链核酸或双链核酸。
46.段落43的组合物,其中靶分子是来自具有小于900道尔顿的分子量的蛋白质、肽、脂质、碳水化合物、脂肪和小分子的生物分子。
47.段落43-46任一项的组合物,其中保护链能够结合靶分子。
48.段落47的组合物,其中靶分子包含或连接至与保护链互补的核苷酸序列。
49.段落35-48任一项的组合物,还包含具有链置换活性的聚合酶。
50.检测靶分子的方法,包括:
在包含靶分子、链置换聚合酶和脱氧核糖核苷酸三磷酸(dntp)的反应缓冲液中合并:
(a)催化发夹分子,其包含:(i)未配对的3'立足点结构域;(ii)分子的3'亚结构域与分子的5'亚结构域之间的分子内核苷酸碱基配对形成的配对的茎结构域;和(iii)连接结构域,其中(a)(i)和(a)(ii)的结构域形成串联重复序列;
(b)至少一个其它催化发夹分子,其包含:(i)3'立足点结构域;(ii)通过分子的3'亚结构域与分子的5'亚结构域之间的分子内核苷酸碱基配对形成的配对的茎结构域;和(iii)连接结构域,其中(b)(i)和(b)(ii)的结构域形成被信号序列中断的串联重复序列,并且其中(b)(i)的3'立足点结构域不可逆地与能够结合靶分子的保护链结合;和
(c)核酸引物,其包含与(a)的催化发夹分子的3'立足点结构域互补并且与(b)的催化发夹分子的3'立足点结构域互补的结构域,
在导致核酸聚合、链置换和退火的条件下将反应混合物孵育足以产生比起始引物更长且包含信号序列中至少一个的单链核酸记录的时间。
51.测定分子事件之间的时间的方法,包括:
在反应缓冲液中合并:
(a)起始催化发夹分子,包含(i)未配对的3'立足点结构域;(ii)通过分子的5'亚结构域与分子的3'亚结构域之间分子内核苷酸碱基配对形成的配对的茎结构域;和(iii)发夹结构域;
(b)多个不同催化发夹分子,每个发夹分子包含:(i)未配对的3'立足点结构域;(ii)通过发夹分子的3'亚结构域与发夹分子的5'亚结构域之间分子内核苷酸碱基配对形成的配对的茎结构域;和(iii)环状结构域,其中每个发夹分子的3'立足点结构域与多个发夹分子中的一个其它发夹分子的5'亚结构域互补;
(c)与起始发夹分子的未配对的3'立足点结构域互补的起始引物;
(d)具有链置换活性的聚合酶;和
(e)脱氧核糖核苷酸三磷酸(dntp),由此形成反应混合物;
使反应混合物暴露于第一个分子事件;
在导致核酸聚合、链置换和退火的条件下将反应混合物孵育足以产生单链核酸记录的时间;和
使反应混合物暴露于第二个分子事件。
52.段落51的方法,其中第一个分子事件启动dna聚合。
53.段落51或52的方法,其中第二个分子事件终止dna聚合。
54.段落51-53任一项的方法,还包含基于产生的单链核酸的长度确定第一个分子事件与第二个分子事件之间的间隔。
55.引物交换反应(per)系统,包含:
(a)起始催化分子,其包含未配对的3'立足点结构域和位于立足点结构域的5'并且通过置换链与含有立足点结构域的模板链之间的核苷酸碱基配对形成的配对结构域;
(b)与未配对的3'立足点结构域互补的起始引物;和
(c)具有链置换活性的聚合酶。
56.段落55的per系统,还包含第二催化分子,其包含未配对的3'立足点结构域和位于立足点结构域的5'并且通过置换链与含有立足点结构域的模板链之间的核苷酸碱基配对形成的配对结构域,其中第二催化分子的3'立足点结构域与起始催化分子的配对结构域的置换链互补。
57.分子马达系统,包含:
(a)起始核酸分子,其包含:(i)未配对的3'立足点结构域和(ii)位于立足点结构域的5'并且通过置换链与含有立足点结构域的模板链之间的核苷酸碱基配对形成的配对结构域;
(b)第二核酸分子,其包含:(i)未配对的3'立足点结构域和(ii)位于立足点结构域的5'并且通过置换链与含有立足点结构域的模板链之间的核苷酸碱基配对形成的配对结构域;
其中第二核酸分子的未配对的3'立足点结构域与起始核酸分子的置换链互补;和
(c)与位于起始核酸分子的未配对的3'立足点结构域中的核苷酸互补的引物。
58.段落57的分子马达系统,还包含具有链置换活性的聚合酶。
59.段落57或58的分子马达系统,其中起始核酸分子的配对结构域包含终止聚合的分子。
60.段落57-59任一项的分子马达系统,其中第二核酸分子的配对结构域包含终止聚合的分子。
61.段落57-59任一项的分子马达系统,其中第二核酸分子的3'立足点结构域包含终止聚合的3'分子。
62.段落57-61任一项的分子马达系统,还包含多个核酸分子,每个分子包含:(i)未配对的3'立足点结构域和(ii)位于立足点结构域的5'并且通过置换链与含有立足点结构域的模板链之间的核苷酸碱基配对形成的配对结构域,其中多个核酸分子中的一个分子的未配对的3'立足点结构域与多个分子中的一个其它核酸分子的置换链互补。
63.段落57-62任一项的分子马达系统,其中核酸分子的至少一个连接至靶生物分子。
64.段落63的分子马达系统,其中核酸分子各自连接至不同的靶生物分子。
65.记录靶生物分子之间距离的方法,包括:
在反应缓冲液中合并:
(a)起始核酸分子,其包含(i)未配对的3'立足点结构域和(ii)位于立足点结构域的5'并且通过置换链与含有立足点结构域的模板链之间的核苷酸碱基配对形成的配对结构域,其中起始核酸分子连接至靶生物分子;
(b)第二核酸分子,其包含:(i)未配对的3'立足点结构域和(ii)位于立足点结构域的5'的配对结构域,所述配对结构域通过置换链与含有立足点结构域的模板链之间的核苷酸碱基配对形成,其中第二核酸分子的未配对的3'立足点结构域与起始核酸分子的置换链互补,并且其中第二核酸分子连接至靶生物分子;
(c)与起始核酸分子的未配对的3'立足点结构域互补的引物;
(d)具有链置换活性的聚合酶;和
(e)脱氧核糖核苷酸三磷酸(dntp),由此形成反应混合物;和
在导致核酸聚合、链置换和退火的条件下将反应混合物孵育足以产生单链核酸记录的时间。
66.段落65的方法,其中反应混合物还包含多个核酸分子,每个分子包含:(i)未配对的3'立足点结构域和(ii)位于立足点结构域的5'的配对结构域,所述配对结构域通过置换链与含有立足点结构域的模板链之间的核苷酸碱基配对形成,其中多个核酸分子中的一个核酸分子的未配对的3'立足点结构域与多个核酸分子中的一个其它核酸分子的置换链互补,并且其中多个核酸分子中的每个核酸分子连接至靶生物分子。
67.分子记录系统,包含:
(a)起始发夹分子,其包含(i)未配对的3'立足点结构域;(ii)通过分子的3'亚结构域与分子的5'亚结构域之间分子内核苷酸碱基配对形成的配对的茎结构域;和(iii)环状结构域,其中起始发夹分子连接至靶生物分子;
(b)第二发夹分子,其包含(i)未配对的3'立足点结构域;(ii)通过分子的3'亚结构域与分子的5'亚结构域之间分子内核苷酸碱基配对形成的配对的茎结构域;和(iii)环状结构域,其中第二发夹分子连接至靶生物分子,并且其中起始发夹分子的5'亚结构域与第二发夹分子的5'亚结构域互补;
(c)两种引物,其中之一与起始发夹分子的未配对的3'立足点结构域互补,并且其中另一种与第二发夹分子的未配对的3'立足点结构域互补;
(d)多个催化发夹分子,每个分子包含:(i)未配对的3'立足点结构域;(ii)通过分子的3'亚结构域与分子的5'亚结构域之间分子内核苷酸碱基配对形成的配对的茎结构域;和(iii)环状结构域,其中多个催化发夹分子中的每一个发夹分子的5'亚结构域与多个发夹分子中的一个其它发夹分子的5'亚结构域互补,其中多个发夹分子之一的3'立足点结构域与起始发夹分子的5'亚结构域互补,并且其中多个发夹分子中的另一个的3'立足点结构域与第二发夹分子的5'亚结构域互补;和
(e)具有链置换活性的聚合酶。
68.记录靶生物分子之间距离的方法,包括:
在反应缓冲液中合并:
(a)起始发夹分子,其包含(i)未配对的3'立足点结构域和(ii)通过分子的3'亚结构域与分子的5'亚结构域之间分子内核苷酸碱基配对形成的配对的茎结构域;和(iii)环状结构域,其中起始发夹分子连接至靶生物分子;
(b)第二发夹分子,其包含:(i)未配对的3'立足点结构域和(ii)通过分子的3'亚结构域与分子的5'亚结构域之间分子内核苷酸碱基配对形成的配对的茎结构域;和(iii)环状结构域,其中第二发夹分子连接至靶生物分子,并且其中起始发夹分子的5'亚结构域与第二发夹分子的5'亚结构域互补;
(c)两种引物,其中之一与起始发夹分子的未配对的3'立足点结构域互补,并且其中另一种与第二发夹分子的未配对的3'立足点结构域互补;
(d)多个发夹分子,每个分子包含:(i)未配对的3'立足点结构域;(ii)通过分子的3'亚结构域与分子的5'亚结构域之间分子内核苷酸碱基配对形成的配对的茎结构域;和(iii)环状结构域,其中多个发夹分子中的每一个发夹分子的5'亚结构域与多个发夹分子中的一个其它发夹分子的5'亚结构域互补,其中多个发夹分子之一的3'立足点结构域与起始发夹分子的5'亚结构域互补,并且其中多个发夹分子中的另一个的3'立足点结构域与第二发夹分子的5'亚结构域互补;和
(e)具有链置换活性的聚合酶和脱氧核糖核苷酸三磷酸(dntp),由此形成反应混合物;和
在导致核酸聚合、链置换和退火的条件下将反应混合物孵育足以产生双链核酸记录的时间。
实施例
下面的实施例尤其证明了利用per分子基元的几种系统的设计和实施,其提供了原位等温合成任意ssdna序列的一般框架。数据显示引物交换反应(per)如何链接在一起形成合成固定长度寡核苷酸的反应级联。如下所述,已成功实现了一种纳米装置,其响应于感测致癌mirna标记而合成功能性dna酶,所述致癌mirna标记为无标记生物传感器,其能够使用合成端粒酶检测和扩增mirna靶的信号检测,能够检测正交rna输入的逻辑线路,以及分子记录两个输入信号顺序的记录器。在许多情况下,系统的逻辑能够通过将相同的分子输入和发夹重新配置成不同的途径来实现。
引物交换反应不需要热循环以促进新生引物链的催化延伸,并且由dntp提供燃料,dntp便宜且易于分子生物学家获得。此外,反应速率可以通过调整溶液中发夹和镁离子的浓度来调整。per的环境响应性在纳米器件、生物传感器、逻辑线路和记录器中得到证明,并且为使用dna动态合成作为信号处理和驱动平台提供了一种强有力的新方法。此外,限定(prescribe)单链输出序列的能力提供了与dna酶一起直接与任何数量的现存纳米器件接口的机会,例如立足点开关(alexanderagreen等人cell,159(4):925–939,2014)或dna链置换线路(davidyuzhang和georgseelig.naturechemistry,3(2):103–113,2011)。此外,per的环境响应性可以进一步扩展到通过适体和动态基因合成的蛋白质检测。per线路的可编程记录、处理和驱动功能为分子编程应用提供了新的范例。
例如,特别感兴趣的是per途径能够实现分子记录应用。因为长聚合物能够根据环境的当前状态在不同途径下生长,所以per途径可以用于产生分子“自动收报机带”,其在长时间内记录环境信号信息。然后可以对这些富含信息的dna聚合物带进行测序以恢复分子事件报告。这种类型的记录行为对动态生物系统的研究具有深远的影响,例如,通过记录关于不同分子物种和事件的信息而不会以显著的方式干扰系统。
实施例1
该实施例证明引物交换反应能够用于根据规定的途径延伸核酸链。第一反应在37℃孵育2小时,使用浓度为100nm的cy5标记的引物和浓度为10nm的发夹(图3a)。第二反应在37℃孵育4小时,使用fam标记的引物孵育1小时,然后引入cy5标记引物。引物以100nm的浓度使用,发夹使用浓度为10nm。通过在80℃孵育20分钟来使聚合酶热失活停止反应。也可以通过聚合酶的热失活或通过引入螯合剂(例如edta)来停止反应。
在另一反应中,将引物和发夹在不同条件下一起孵育以验证和表征基本单引物交换反应(图1c(iv))。该系统包括两个成分,图1c(ii)中描绘的荧光团标记的引物和发夹,并且变性凝胶电泳用于跟踪基本引物交换反应的进展。泳道1显示对应于没有发夹的引物的条带,泳道2-11描绘了单引物交换反应以10分钟的间隔用100:1的引物与发夹比进行的时间进程。泳道12-22显示用不同量的起始发夹孵育相同的反应90分钟。这些数据确认了发夹在溶液中的催化再循环,并证明了发夹浓度的多功能性,可用于调节per系统的速率。
在设计引物与发夹的结合亲和力时,需要考虑几个因素。首先,引物应足够短以自发在最后一步中从发夹上解离,既保持生长链主要是单链状态又促进任何后续引物交换反应。另一方面,引物应制成足够长以使发夹结合足够的时间以通过聚合酶延伸。使用聚合酶、温度、盐和缓冲条件的特定组合,该长度范围可以是例如7-10个碱基对。
实施例2.
引物交换反应可以链接在一起形成per级联,其在规定的反应途径后生长固定长度的链。我们实施的第一个per途径是由具有dntp、聚合酶、镁和引物的溶液中的一组催化发夹物种介导的5个延伸步骤的级联(图2a(i))。当所有5个发夹和引物存在于溶液中时,该途径进行5个延伸步骤(图2a(ii))。发夹a催化具有结构域b的引物结构域a的延伸。然后,发夹b催化具有结构域c的结构域b的延伸。并且发夹c通过e分别催化具有d的c、具有e的d、和具有f的e的延伸。
当与发夹的不同子集混合时,变性凝胶电泳验证了引物链的有序伸长(图2a(iii))。泳道1显示了仅有引物和没有发夹一起孵育的反应。泳道2显示与发夹b至e一起孵育的引物,除了引发生长的发夹外,所有发夹都是发夹a。该对照显示没有可检测的泄漏。泳道3显示了第一次延伸步骤,其中引物和发夹a一起孵育。当引物与前两个发夹a和b一起孵育时,泳道4显示两个延伸。当分别与发夹a至c、a至d和a至e一起孵育时,泳道5、6和7显示引物的三个、四个和五个延伸步骤。在sybrgold染色后通过凝胶成像进一步验证产物条带的长度。尽管发夹以引物浓度的1/10存在,但通过将所有引物几乎完全转变为最后状态来验证所有发夹的催化转换。
为了观察是否有可能进一步放大per合成的规模,dna折纸结构的订书钉链是在一锅法反应中合成的。由40个订书钉链固定在一起的由3个字母的代码支架组成的结构被设计成折叠成紧凑的矩形形状,其沿其短端聚集以产生折纸结构链。设计总共80个发夹以从40个引物合成40个订书钉链(图2a(iv)),每个在两个反应步骤中。通过与聚合酶在37℃孵育1小时,80个反应全部平行进行。在热灭活后,将支架链直接引入per混合物中并退火1小时。然后通过原子力显微镜观察折纸结构以验证恰当的结构形成。
在本实施例中进一步描述了具有低泄漏和高转变率的引物交换反应的实施例。使用引物交换反应来构建合成的端粒酶,允许连续复制任意序列,导致其在短短几个小时内延伸至多数百个碱基(图4)。合成的端粒酶构建体(图3)使用cy5标记的引物,终浓度为100nm,发夹浓度为10nm-100nm。将bst大片段聚合酶与1×thermopol缓冲液(20mmtris-hcl、10mm(nh4)2so4、10mmkcl、2mmmgso4和0.1%
还编程了更复杂的状态转换,例如通过给定数量的延伸步骤延伸链(图5)。多步骤反应(图4)使用100nm的引物浓度,10nm的发夹浓度和10μmdatp、dctp和dttp。所有其他反应和成像条件与端粒酶构建体保持相同。没有进行预孵育清理步骤。
这两个实验均使用bstdna聚合酶,大片段,其在37℃的孵育温度具有足够的链置换活性,以促进引物交换反应。结构域1至6(图5)由由c、a和t碱基组成的三字母代码组成,因此g碱基可用作终止聚合酶的终止序列。引物序列长度各为8-9个碱基对,设计得足够长,以便它们能够足够稳定地结合到发夹上以便复制,但也足够短以在复制后自发地从发夹上解离。对于较高温度反应,引物序列长度可以更长,或对于较低温度反应,引物序列长度可以更短。
实施例3.长聚合物的触发组装
在一个实验中,发夹序列被设计为每个引物交换反应步骤复制10个碱基,步骤为5个步骤(图5),导致总共添加50个碱基。每个序列添加使用一个发夹,使用引物交换反应在5个连续延伸反应的每一个中将9个核苷酸的引物序列延伸10个核苷酸。将反应在37℃孵育4小时。使用两种互补方法可以增加延伸(图8a-8b)。首先,可以设计正交引物序列(例如至少30个引物序列)以增加反应图中的步骤数。复制的每个发夹上的碱基数也可以增加。可以将每个发夹编程为允许在下一个引物序列之前复制任意序列,尽管在某些情况下,聚合酶的持续合成能力和后链置换过程的时间增加对该区域的长度施加一些实际限制。
在另一实验中,引物交换反应条件是使得每次延伸至少复制30个碱基。因此,合成了长度为至少900个碱基的支架。使用分阶段组装策略,其中依次引入每个发夹以延伸对接到表面的引物链,其间具有洗涤步骤。因为在任何给定的时间点溶液中没有其他发夹,所以发生虚假引发事件的可能性最小。因此,该方法可以容易地放大到更大的长度的规模。然后在原位组装支架后引入钉书钉链,以避免途径外结合事件。还使用了一种自主策略,其中在支架合成期间所有发夹都包括在溶液中。
方法。所有dna链在商业上合成并悬浮在1×te缓冲液(10mmtris-hcl和1mmedta)中,在-20℃长期储存。使用nupack软件包设计dna序列,以及开发的软件以优化具有所需结合能的引物。引物最初是hplc纯化的,并且发夹有序未经纯化,其3'末端具有反向dt修饰,以防止聚合酶延伸。对于反应,dna与bst链置换聚合酶(或phi29,bsu大片段或klenow(外-))和1×thermopol缓冲液(20mmtris-hcl、10mm(nh4)2so4、10mmkcl、2mmmgso4和0.1%
实施例4.2d和3d结构的触发组装和折叠
在该实施例中,长的固定长度的链用作支架以折叠任意的2d和3d形状。对于传统的dna折纸方法,长链用作支架并折叠成形状。使用光栅化策略,其中支架链来回穿过结构区域(图9a)。通过这种策略,支架链被引导通过2d或3d形状,然后与订书钉链拴系在一起,所述订书钉链与应该定位的支架上的结构域互补。为了组装具有足够大的特征以通过显微镜观察的结构,使用实施例3中生产的900bp支架。使用大约20个订书钉链(每个具有42-45个碱基对的长度)。
用引物交换组装规定形状结构的另一种方法是合成其自组装成块结构的dna单链拼块(sst)结构(图9b)。通过这种方法,许多小dna链基于它们的互补区域协同组装成复杂的形状。每条链编码在单个发夹中,因此不同发夹物种的数量等于不同sst单体的数量。或者,所有sst序列在单链支架上合成,然后后来通过限制酶切割开。该策略允许使用更少的使用实施例3中的拷贝区的发夹,尽管由于所有sst序列在一条链上的共定位,长链可能与其自身杂交并减慢延伸。
用于组装结构的另一种方法是使用合成的链作为单链dna折纸的支架(图9c)。通过这种方法,链被设计成以未打结的方式折叠到自身上以形成形状。折叠不需要订书钉链,因为结合相互作用被编程为分子内。在一些情况下,所有结合区域以这样的方式插入引物区域,使得结合区域全部在发夹的区域内,该区域在下一个引物序列之前被复制,使得它们不干扰延伸反应。
方法。孵育反应的方法与实施例3中的相同。在一些情况下,在引物交换反应完成后补充额外的镁以促进结构折叠和稳定性,并且使用浓度范围为10mm至100mm的额外阳离子。琼脂糖凝胶电泳(2%,用sybrgold染料染色)用于使体积结构形成可视化。切割出对应于完全形成的结构的凝胶条带,并在具有相同盐条件的缓冲液中洗脱,以用透射电子显微镜(tem)和/或原子力显微镜(afm)成像进行验证。dna-paint超分辨率成像也可用于直接可视化和评估生长的各个结构的空间构象。将所有引物交换反应在37℃孵育,并在该温度或室温折叠结构以确保与固定细胞条件的相容性。
实施例5.分支结构的有效生长
在该实施例中,使用引物交换反应组装分支结构。因为新引物的延伸应该仅在它们结合到结构时发生一次,所以发夹应该识别这种共定位以便于复制。这可以通过使用由两个序列组成的引发序列来实现(图10a)。两个单独的引物区域(图10a中的a1和a2)本身应该与发夹结合太弱以足够长地结合以引发引发反应。然而,当共定位时,它们应具有与先前与引物交换发夹一起使用的8-9碱基对引物相似的动力学,使得它们足够短以自发地从发夹中解离但足够长以成功启动链置换引发。因为8-9个碱基对对于正常引物交换反应中的引物长度而言是足够的,所以使用范围为4-6个碱基对的a1和a2引物区域。
方法。用于孵育和评价结构的方法保持与实施例4中相同。
实施例6.纳米装置
作为引物交换反应应用的多功能性的证明,我们实施了一种纳米装置,其检测致癌mir-19a信号并随后合成功能性dna酶,其被编程以切割独立的rna转录物(图26a)。对于靶检测,我们设计了发夹以结合致癌微rnamir-19a和mir-19b的共有3'区域。响应于检测,per途径合成dna酶(dz-twt),已显示其体内切割twist基因的全长mrna并促进细胞凋亡。
对于该应用,使用一对合成的核苷酸iso-dg和iso-dc作为所有发夹上的终止序列(图26b)。这使我们能够利用合成序列中的所有四个dna碱基。作为per途径的输出构建体的实例,我们选择使用已经在体内验证的dz-twtdna酶。
dz-twt是10-23个dna酶类的一部分,它具有15nt催化结构域,可切割两条臂之间的特定嘌呤-嘧啶键,其设计为与同源rna序列互补,并可重新编程以切割任意rna序列。为了实现系统逻辑,设计了三个per发夹以仅在检测到靶微rna时合成dz-twt序列(图26c)。当靶标存在时,a发夹将结构域a的模拟合成到靶链上(步骤1)。b和c发夹随后可以分别对域b和c的附加进行模拟(步骤2-3)。完成的abc序列形成dz-twt序列,其可以与溶液中的24bptwtrna片段结合,形成催化切割特定碱基处twtmrna的环(步骤4-5)。然后dna酶在与切割的片段解离后可以再循环(步骤6)。
附着于靶的完整dna酶的序列示于图26d中。我们将dz-twt序列分成三个结构域以不同的颜色显示。发夹上的引物结合位点的长度(分别为发夹a、b和c上的19s*、as*和bs*)被调整为结合新生链的8-9bp以确保拷贝后新生链可以与发夹解离。
为了评估纳米装置功能,我们包括不同的发夹子集和具有和不具有cy5标记的mirna靶的fam标记的twt片段(图26e)。泳道1和2显示了在没有任何发夹存在的情况下孵育时的twt片段和mir-19a条带。泳道3显示,在最后两个发夹b和c的存在下mirna没有延伸,但不是第一个发夹a。泳道4显示当mirna仅与a发夹和twt片段孵育时延伸的第一步。泳道5显示了靶与前两个发夹a和b的孵育,这导致两个步骤延长。当靶与所有三个发夹混合时(泳道6),将完整的dz-twt序列附加到其上并且能够成功切割twt片段。但是当所有发夹在没有靶的情况下混合时,不会发生twt片段的切割(泳道7)。dna酶构建体的催化转换(turnover)通过仅用10nmmirna靶基本上完全切割20nmtwtrna来指示。
实施例7.无标记生物传感器
通过纳米装置,我们能够响应任意输入序列合成任意生物学相关的dna序列。这种将一个序列转换成另一个序列的per途径的可编程性为环境响应合成系统提供了强大的模块化框架,我们进一步探索了其他几个应用。我们开始实施单发夹系统,其合成长链重复序列结构域,我们称之为合成端粒酶,然后将此构建体用作无标记生物传感器的信号放大形式,在检测到某种特异性mirna输入时生长荧光多联体(图27c)。
合成的端粒酶系统包含单个引物序列(具有结构域a)和发夹,其将重复结构域a附加到催化到生长链上(图27a)。对于该证明,我们选择10bp序列atctcttatt(seqidno:1)作为重复结构域,其中发夹结合区对应于该序列的最后9bp。跟之前一样,引物用cy5荧光团标记用于凝胶可视化,并且发夹在其3'末端装配有反向dt以防止其延伸。
我们使用与先前证实相似的实验设置验证了构建体,并显示了如何通过将发夹浓度改变几个数量级来调整反应速率(图27b)。在与设计用于消耗外来dgtp的专门发夹物种孵育15分钟后,将发夹与引物一起孵育。泳道1显示没有添加发夹的引物条带,泳道2到6显示发夹浓度范围为1到100nm所生长的端粒。通过调整系统中发夹的浓度可以很容易地调整反应速率,这为我们提供了具有可控制的大动态反应速率范围。我们还发现镁浓度可用于调节端粒化速率,提供另一种调节反应动力学的方法。我们随后设计了一种实施无标记生物传感器的策略,该生物传感器可以仅响应于特定mirna信号的存在而有条件地生长这种类型的端粒输出(图27a)。通过设计结合特定荧光染料的序列,我们能够使这些合成的端粒发荧光,允许在蓝光下直接观察结果。通过硫磺素t(tht)染料的结合实现荧光,一旦插入到通过人端粒序列ttaggg的重复形成的四链体基序中,已经显示荧光变得更加强。我们选择的靶是致癌mirnamir-19a。
实施检测物-端粒酶系统需要三个成分(图27d):引物(p)、门控发夹(a)和端粒酶发夹(b)。为了将靶检测转换成人端粒序列的合成,设计了立足点交换反应,使得per发夹(a)的引物结合位点仅在同源mir-19a信号存在下暴露。mirna靶可以结合到与a发夹结合的保护链上的短暴露结构域上,并且分支通过剩余的互补序列迁移。一旦互补的mir-19a*结构域被完全置换,保护链就可以自发地从受保护的per发夹上解离,从而暴露a发夹上的引物结合位点a*(步骤1)。一旦暴露,该per发夹便于将b结构域附加到引物的a结构域上(步骤2)。b结构域对应于人端粒序列ttaggg,其随后通过组成型活性端粒酶发夹b进行端粒化(步骤3)。最后,这些端粒形成四链体结构,tht染料嵌入其中并变成发荧光(步骤4)。
为了评估条件性端粒化反应,我们孵育了具有不同成分子集的反应,并在天然page凝胶上显现了结果(图27e)。泳道1显示仅有mirna的反应,并且没有分子程序成分。使用引物p和端粒酶发夹b但没有门控发夹a,mirna靶不与任何寡核苷酸结合,并且没有端粒化(泳道2)。使用引物p和门控发夹a,但没有端粒酶发夹b,mirna与保护链结合,如其条带的移位所示(泳道3)。只有当p、a和b中的所有三个与mirna靶一起存在时,才发生端粒化(泳道4)。在不含mirna靶的对照反应中,元件p、a和b几乎不产生背景端粒化(泳道5)。
作为更简单的读出方法,可以使用safeimager2.0transilluminator通过琥珀色滤镜单元(vis)显示反应的荧光。这提供了一种安全、成本有效且节省时间的信号读出方式。还可以在荧光扫描仪上在fam通道(fam)下观察管(参见方法部分以获得完整细节)。
实施例8.逻辑计算
通过逻辑表达式评估对靶序列进行信号处理已成为编程复杂动态分子行为的有价值框架。下面,我们将展示per如何能够用于实现任意序列的and、or和not逻辑,只需编程哪些引物序列根据靶链的存在(或不存在)进行追加。基本策略是用门控发夹平衡rna靶,引入引物,并在孵育后通过长度在凝胶上读出结果(图28a)。
用于两个rna输入的or门可以用传感器和记录器应用中引入的两个门控发夹实现(图28b)。每个靶可以激活两个发夹中的一个,并且当两个发夹中的一个或两个被激活时,b结构域能够附加到引物的a结构域上。结果用变性凝胶验证,当没有任一靶存在时,其没有显示延伸产物(泳道1)。然而,如果存在一个或两个靶,则引物延伸一个结构域(泳道2-4)。为了评估两个靶是否都存在(and逻辑),以逐步的方式检查它们的存在(图28c)。如果两个靶都不存在(泳道5)或仅存在twt靶(泳道6),则引物结构域a不会延伸。如果仅存在mir-19a靶,则将引物延伸一个结构域为ab(泳道7)。然而,如果两个靶都存在,则引物延伸至完整的abc序列,表明and表达的成功评估(泳道8)。
实现not逻辑需要在靶接受和拒绝之间分开时间尺度(图28d)。这可以通过以比评价为真(bc)的发夹高得多的浓度包括靶依赖性沉降反应来实现。因此,如果存在靶(在该情况下是mir-21),那么b引物会变成漏斗成为无活性(灰色)状态。然而,如果不存在靶,则以慢速率将所有b引物转变为bc。通过分别比较没有靶(泳道9)和有靶(泳道10)的孵育结果来验证门控。
最后,我们证明了几种这些类型的门可以连接在一起来计算表达式(mir-19aortwt)and(notmir-21)(图28e)。该计算是通过将图28b中的mir-19aortwt门的结果级联到图28d的notmir-21门来实现的。通过凝胶电泳如前验证结果。当没有输入或仅存在mir-21靶时,引物仍然主要处于其未延伸状态(泳道13和14)。当仅存在twt或仅存在mir-19a时,表达式评估为真,如完全延伸的abc产物(泳道15和16)所示。然而,当除了mir-21之外还存在这些中的一种时,引物变成漏斗成为无活性状态(泳道17和18)。同时具有mir-19a和twt但没有mir-21再次导致结果为真(泳道19),但是当存在所有三个靶时,表达式再次评估为假(泳道20)。这些线路成分证实了per方法的模块性。通过匹配合成的和结合的引物结构域,可以容易地将一个逻辑评估的结果相互级联,并且这种类型的重构在理论上可以扩展到给定任何单链rna或dna输入的任何引物结构域。
实施例9.事件记录器
通过前述系统,我们展示了per如何提供模块化框架来实现环境响应行为,因为输出序列可以完全独立于输入序列。为了进一步证明per的可编程性及其在分子信号处理中的应用,我们创建了一种时间记录系统,能够编码两个rna目标被见证成动态合成的转录物的顺序(图29a)。
该应用中使用的四个发夹中的每一个利用与无标记生物传感器相同的立足点交换方法进行靶检测,其中门控per发夹的结合位点仅在存在同源信号的情况下有条件地暴露,在这种情况下是mir-19a(图29b)。门控发夹a至d模拟引物的动态延伸(图29c),其中发夹a和d仅在存在mir-19a信号时被激活,并且发夹b和c在twist片段存在下被激活(图29c)。
根据引入两个rna信号的顺序,引物将经历两个延伸途径中的一个。如果首先引入mir-19a,那么初始引物通过发夹a用b延伸。然后,如果引入twt靶,则发夹b可以将c附加到引物上。另一方面,如果首先引入twt,则将d添加到具有暴露的c发夹的引物序列中,并且如果稍后遇到mir-19a信号,则发夹d可以附加e。
因为发夹被设计成每次添加复制不同数量的核苷酸,所以基于在溶液中引入信号的顺序来实现序列长度的区分。这些结果可以在page变性凝胶上读出(图29d)。通过在引物和发夹a至d孵育5小时的1小时或3小时内引入信号,评估记录行为。泳道1显示没有引入靶的系统,并且由于所有四个发夹的连续保护而没有显示延伸。泳道2和3显示当分别在1小时和3小时引入mir-19a(mir)时由发夹a模拟的10bp步骤的单一延伸。在1或3小时引入twt导致暴露的c发夹(泳道4和5)单次延伸14bp。首先引入mirna然后引入twt导致两个10bp延伸以形成abc序列。最后,首先引入twt靶然后mirna导致两个14bp延伸步骤以形成ade,从而验证我们在合成dna的长度中读出两个rna信号的时序关系的能力。
通过per进行动态和环境响应的链合成使得能够实现这种类型的可编程时间记录,并且代表了per技术创建分子事件转录物的关键优势。我们在每个步骤都具有完整的序列可编程性,使得能够检测任意靶序列并随后将信号信息转换为序列身份和长度,作为分子记忆的一种形式。
实施例10.体内实施per
因为per提供了耦合独立核酸序列的读取和写入的新方法,所以在体内实施它使得细胞内的高度可编程的基因调节和记录平台成为可能。per级联已设计为在37℃(标准细胞培养温度)操作,尽管可调节引物长度以允许per在不同温度下操作。该反应已在各种镁浓度(2mm至22mm)下得到验证,这与生物样品相容。
本文提供了至少两种在体内实施per的方法,每种方法具有不同的应用(图30)。第一种方法是用per发夹直接转化或转染靶细胞群(图30a)。引物与发夹一起引入,或者可以是已经在每个细胞内转录的序列。由于该方法不靶向每个细胞,因此应用可主要用于记录。例如,per可用于记录单细胞水平的多重信号信息,并且可以回收合成的转录物并测序。由于发夹的3'末端通常具有反向dt碱基,在某些情况下,它们还具有某种防止细胞内降解的保护作用。
第二种方法将per发夹序列整合到质粒或细胞的基因组中,使得它们可以转录成rna发夹以与rnaper一起使用(图30b)。如果选择成功并入成分的细胞,则该表达方法具有产生具有细胞内per合成的完整群体的益处。因为覆盖范围要大得多,并且由于per级联成分传递了几代,所以相对于第一种方法,细胞成分的记录可以在更长的时间范围内发生。任何程序性行为也有更大的覆盖范围。多聚t尾巴可用于发夹的3'末端,因为它们已经被证明是不希望的发夹延伸的有效抑制剂。
可靠的终止序列将阻止链置换rna聚合酶继续通过它,这是新合成的链从发夹中置换所必需的。在一些实施方案中,序列基序(例如四重(四重的)和三重结构)可以使用在终止连接处折叠结构以抑制聚合。在其它实施方案中,可以使用结合所有发夹茎中共有的特定序列基序以阻碍聚合的强结合蛋白(例如dcas9,其rna结合柄是可直接包括作为所有发夹的茎环部分的发夹结构)。
实施例11.用于测定时间的分子时钟
为了最大化per多路复用能力,针对各种单链核酸(例如dna和mrna、双链dna和小分子和蛋白质)编译了大量操作信号检测模块(图12a-12f)。例如,只有当同源信号(例如钙离子浓度)存在于溶液中时,发夹的引物结合区才暴露,无论是用保护链还是用交联剂。因为每个发夹限定了引物链状态之间的转换,所以当不存在所需信号时抑制发夹有效地使引物停留在其当前状态。为了检测单链dna和rna,使用修饰的立足点交换反应58、59,其中保护链(例如图12a中的序列x2’x1’1)对发夹浓度过量浓度以降低泄漏的可能性。通过使用不对称pcr方法将同源立足点对连接到双链体的末端来完成检测双链dna。通过使用适体序列仅在信号不存在时封闭发夹的引物结合区域,该方法进一步扩展到检测蛋白质和小分子。最后,通过使用cnvk交联剂检测紫外照射57。所有这些信号检测机制都是可逆的,这意味着信号不会无限期地从它们可能正在运行的溶液中隔离(sequestered),并且信号浓度变化通过发夹结合位点暴露的变化反映出来。
为了测定时间,通过在序列延伸步骤中跟踪分布,将端粒酶子线路用作时钟(图13)。因为检测到的信号不会改变系统的动力学特性,例如溶液中的链在检测信号之间经历的引物交换延伸步骤的“开启”率、端粒酶步骤的数量表示这些端粒酶子线路是有活性的时间量。总体而言,该系统充当合成秒表,通过将端粒酶与两个输入模块连接来编码检测两个信号之间所经过的时间量。此外,可以通过改变发夹浓度来调整测量时间的时间尺度,这有效地改变了反应的速度,以及溶液中dntp的量。
方法。反应孵育条件与实施例4中的相同。dntp最初以100μm浓度使用,发夹浓度在10nm至1μm之间变化,端粒酶发夹的浓度低于其他的浓度。改变dntp浓度以匹配所需的动力学,使得dntp浓度足够低以促进高保真聚合,但足够高以至于不能通过系统的操作被基本上消耗。改变输入信号的浓度以优化信号检测模块并找到特异性和产量之间的正确折衷。将引物在100nm孵育并如上所述用cy5染料标记,以便能够在变性聚丙烯酰胺(page)凝胶上进行筛选。检测到的分子事件之间的适当梯形合成可以通过page凝胶上的条带长度分布来显现,并与在相同反应条件下孵育特定时间量的基准反应进行比较。因为凝胶的分辨率可以使较长序列的精确分布难以拟合,所以使用下一代测序进一步验证它们。
实施例12.用于程序可控的时序启动的分子计时器
本实施例中描述的延迟线路涉及信号检测事件和启动之间的一组连续延伸步骤,其被编程为在一定时间段内发生(图14a-14c)。通过使引物在启动之前穿过几个反应状态,通过改变反应的数量或发夹的浓度以及因此改变反应的速度来产生和调节时间延迟。这种模块化允许在大范围的时间延迟之后启动(例如,在几分钟到几天的时间尺度上启动)。
由于能够在引物交换发夹上复制任意序列,引物交换反应系统能够输出许多任意单链信号作为输出。该单链信号可用于与溶液中的链置换线路相互作用或与互补rna分子杂交。在一些情况下,该输出激活立足点开关58,其激活蛋白质合成。或者,有毒信号的感测触发具有一个输出的立足点开关,然后在设定的时间量之后用另一个开关停用该相同的开关。这允许响应于环境信号的输出信号的脉冲,而不需要无限期地激活输出(图15a-15d)。蛋白质信号的这种可编程的时间控制水平难以用先前的技术放大规模,并且对于工程功能合成系统或在受控的体外环境中研究基本生物学机制(例如反应动力学)是有用的。
方法。引物交换培养条件保持与实施例6中相同。通过直接测量随时间的输出来评估计时器线路延迟,以验证启动中的适当延迟,例如使用荧光分光光度计测量荧光团标记的链通过输出信号随时间从猝灭剂标记的互补链的置换。测量延迟的另一个选择是随着时间的推移使用产生gfp的立足点开关的激活来监测荧光随时间的变化。
实施例13.多重时序记录和启动
引物交换反应提供模块化框架以响应于不同组的信号而启动,例如,仅当感测到两个分子中的一个(or逻辑)或仅在两个分子被感测(and逻辑)时释放输出(图16a-16i)。在一些情况下,它们还被编程为检测信号是否不存在(not逻辑),如果在程序性延迟之后未检测到输入。这些门连接在一起以对大量环境信号执行逻辑并以上述方式启动(图17a-17c)。这些数字逻辑线路响应于在不同时间检测到的不同组的信号,并为与转录和翻译网络相互作用的复杂信号处理系统提供基础。
如实施例6和7所述,将集成系统中的每个模块首先分别进行测试。随后,它们以逐步的方式组合,以确保系统中的每个子线路保持其功能并便于解决任何问题所需的调试。
方法。方法保持与实施例6和7中的方法相同。
实施例14.通过发展自组装的区分
通过组合实施例6-8中开发的信号检测模块和实施例3-5中开发的触发结构合成,可以基于环境信号改造形状形成的区分(图19a-19c)。每个信号检测器模块需要单个发夹,其引物结合区仅在存在同源信号的情况下暴露。区分树的终末状态用作结构形成的触发器。
方法。反应孵育和结构评估的方法保持与上述实施例中的相同。使用cy5标记的引物,通过变性聚丙烯酰胺凝胶电泳(变性page,典型地为tbe-尿素)验证发展自组装的第一步的适当长度的链分布。随后,首先用非变性琼脂糖凝胶电泳评估在引物上生长的区分形状,然后使用tem或afm成像进行可视化。结构也可以用dna-paint超分辨率方法成像8、9。
实施例15.用于环境记录的分子自动收报机带
per级联的动态合成反应可用于记录随时间的分子信号信息。这种情况下的一个强有力的实施例是使用分子自动收报机带系统,其将信号依赖性合成与连续per端粒化反应耦合以记录关于溶液中何时存在特定信号的信息(图20a-20d)。首先,实现了单信号跟踪系统,该系统可用于阐明单个信号在两小时时间段上的动力学。下面提供对实验及其结果的描述。
分子实施方式。单信号系统的分子实现由两个per发夹组成,一个用作分子“时钟”并且在所有实验中以相同的恒定浓度存在,并且一个用作“信号”并且模拟复制在重复引物序列之前的一些额外碱基(图21a-21d)。每个不同的实验使用在重复结构域之前具有实验特异性条形码序列的引物,因此可以将所有实验合并在一起并在单次测序运行中一起提交。
实验概述
记录。进行了三次记录实验。在实验a中,在整个2小时的孵育期间没有引入信号。这用作对照实验以验证发生的正确时钟反应。在整个2小时的孵育过程中,实验b具有500pm的恒定信号发夹浓度。该反应用作拟合最后一个实验的基准,因为相对于时钟速率的信号可用于校准浓度曲线。实验c在没有信号的情况下运行一小时,然后在中途引入200pm的信号,使浓度曲线成为阶梯函数。
记录在37℃进行2小时。用5μl10×thermopol缓冲液,40单位bst大片段dna聚合酶,5μl100mmmgso4,5μl100μm联合datp/dttp/dctp溶液,5μl1μm适当引物,5μl50nm时钟发夹,5μl5nm信号溶液(仅实验b)在冰上制备反应物,加水至50μl。对于反应c,在孵育1小时后加入1μl10nm信号溶液。孵育后,将反应物移至冰上并用10μl0.5medta终止反应。然后通过nebmonarchpcr&dnacleanupkit进行合并反应,以纯化记录的转录物。
衔接子标记。然后在其5'和3'末端用衔接子序列标记纯化的转录物(图22)。将连接链和记录与过量的夹板链混合,并用1×t4dna连接酶缓冲液中的t4dna连接酶连接,在23℃孵育1小时。连接后,再次用dna净化柱纯化转录物。
凝胶提取。纯化的衔接子标记的寡核苷酸在保持在65℃的15%page变性凝胶(1×tbe)上运行。凝胶在200v电泳15分钟并用1×sybrgold染料染色。在观察sybrgold通道中的序列分布后,在typhoonfla9000扫描仪上进行衔接子标记序列的凝胶提取。将提取的凝胶块放入1.5ml锥形管中并粉碎,然后与50μl1×te混合并离心。在-80℃和90℃连续孵育各10分钟后,将成分放入freeze'nsqueezedna凝胶提取柱中,并以15,000g离心1分钟。将提取的转录物通过一个额外的dna清除柱,然后测序。
转录物测序。
序列分析。以fastq格式接收序列,并如下解析为时钟(0)和信号(1)记录。首先,仅回收可被鉴定的具有有效实验条形码的初始引物序列和3'衔接子标记的开始的序列,因为这表明完整记录被测序并且可以应用于所进行的实验之一。接下来,每次出现的对应于信号发夹的序列被替换为‘1’,然后由时钟发夹编码的每次出现的重复时钟结构域被转换为‘0’,排除了引物上的初始重复结构域。只分析了具有完美二进制字符串0和1的记录。
结果
使用解析的二进制序列拟合浓度曲线,并且该优化的结果可以在图23a-23b中看到。
在该实施例中,我们证明我们不仅能够在分子自动收报机带的环境中恢复信号的浓度信息,我们还可以恢复这些信号随着时间如何变化的动力学信息。这种类型的时序记录可能对我们以多重和定量方式研究生物现象的能力产生深远的影响。此外,我们的测序数据可以轻松检测到200pm的信号,这表明该记录技术的另一个潜在应用:高灵敏度的靶标检测。如果我们可以进一步将可检测限度扩展到更低的浓度,则该技术可以应用于浓度非常低的标记物的多重检测,例如血清或其它体液中的mirna。
作为另一实例,通过组合实施例6-8中实现的时钟和路径重构系统,可以记录关于环境信号的信息,然后高精度地随时间读出。这是通过使用编码成连续延伸链的信号检测事件来实现的。一个端粒化反应用作时钟,并且附加反应随时间记录不同的信号(图20a-20d)。通过考虑实际信号浓度导致的时钟和信号序列结合随时间的分布,每个信号在绝对时间内的浓度轨迹拟合于该数据。为了拟合这些数据,通过运行受控实验来测量每种并入类型的反应速率,其中只有时钟在运行和其中信号浓度变化且只有一个信号检测器模块在运行。由于线路运行的时间和信号浓度的量是已知的,长度分布用于确定每个单独反应的开启率kon。然后使用这些率来确定信号随时间的相对浓度,其基于自动收报机带转录物中的并入数和已知的反应速率。
引物序列在其5'末端含有由随机碱基序列组成的独特分子标识符(umi)。这些用于识别测序数据中的重复读数,并通过尽可能多地保留关于环境信号的时间依赖性的定量信息来减少数据分析中的偏差。
方法。反应条件与实施例6-8中的相同。每个cy5标记的引物也用15个核苷酸的独特分子标识符(umi)标记,以便通过pcr反应扩增转录物并用下一代测序方法读出。使用15个核苷酸的umi,存在1,073,741,824种可能性,因此即使具有核苷酸偏差,在来自测序的多达2000万个读数中具有相同umi的读数也不太可能来自不同的引物。使用专门的脚本分析数据,以便将数据解析,排序和拟合到时间轨迹。
为了记录影响聚合酶动力学的信号,例如钙浓度峰值,可以使用用于基准测试的外部时钟。例如,交联的发夹可以用于检测特定波长的紫外照射,其可以在特定的时间间隔之后脉冲并且使用图12d中引入的检测器记录。
实施例16.1d形式的分子马达运动
用于测试爬行者式基本操作的1d轨道建立在结构良好定义的刚性dna纳米结构上。使用简单的dna折纸矩形或dna块系统52,53。以下参数被修改:(1)孵育时间,(2)引物、聚合酶和核苷酸单体的浓度,(3)不同结构域(引物、信息编码位点等)的结合强度,(4)轨道上相邻位点之间的空间间隔,以及(5)释放条件。使用1-2小时的时间尺度,这允许接近完成反应。还测试了孵育时间的多种变化,包括数秒至数分钟,以更好地理解反应的动力学。测试引物浓度为100nm,dntp浓度为10-100nm。通过改变结构域的长度和gc含量,以及通过引入额外的辅助成分(例如凸起)来改变不同结构域的结合强度,以在需要时使反应平衡发生偏倚。通过组装轨道锚定点之间具有不同距离的纳米结构来产生具有不同空间间隔的轨道。首先测试一个简单的双位点轨道,以表征基本的距离依赖性性能。也测试了如上所述的不同释放策略,例如在孵育结束时手动添加“反向引物”,在最终轨道位点附近嵌入释放信号位点或热介导的解离。
另外,测试轨道的不同总长度以测量长行程距离上的任何性能降级。还测试了不同的轨道组成。完全寻址的轨道,具有顺序指令(例如引物结合位点a*、b*和c*,按顺序,如图31a所示)沿着规定的路径引导爬行者。具有重复引物的轨道(例如,考虑所有引物对统一为图31a中的a-a*)允许爬行者选择起点和进行的方向。
为了表征轨道结构的组装,使用直接纳米级成像方法,例如原子力显微镜和超分辨率成像。对于爬行者的操作,在初始研究阶段,主要进行基于凝胶电泳的定性表征。凝胶电泳允许通过按大小分类dna分子来快速分析反应产物。比较给定步骤之前和之后的反应混合物允许评估反应物向产物的转变。所有完整反应都是可见的条带,其长度对应于连接在一起的三个单个记录的长度(具有辅助部分)。对于不正确的产物,取决于反应规模和产量,添加使用聚合酶链式反应(pcr)的产物扩增步骤,然后进行凝胶表征。测量凝胶条带的密度使得能够估计反应产量。在小规模产品形成的情况下,定量实时pcr(qpcr)允许通过随时间分析扩增迹线并外推回到初始状态来估计产物量。使用随机dna序列的独特鉴定分子,结合单分子分析工具,例如新一代测序,通过检测单个轨道上多个记录的产生,用于显示爬行者的催化和重复记录行为。
实施例17.2d形式的分子马达运动
本实施例证明了以下特性:(1)爬行者在2d空间中的全角运动,(2)爬行者在周围漫游并“选择”遵循的路径的能力,(3)爬行者共同收集有关2d轨道的信息的能力。
2d轨道建立在结构良好定义的刚性dna纳米结构上。通过组装具有不同位置的轨道锚定点的纳米结构来创建具有不同空间布置的轨道。具有角度的简单三位点轨道(例如“l”形状)用于测试爬行者在交接点处的行为。两种类型的分子马达系统(爬行者和步行者)都经过测试。调整一些参数,例如孵育时间和浓度,以适应组合数量的可能路径。类似地,测试不同的轨道组成。完全寻址的轨道,带有顺序指令,引导爬行者沿规定的2d路径移动。在具有重复(a-a*,然后a-a*等)或交替(a-a*,然后b-b*,然后是a-a*等)引物的轨道的情况下,分子可以自由选择在交接点处遵循的路径(图31b和32b)。图31b和32b的右侧分别示出了两个系统的分子马达的实例轨迹。爬行者遵循的不同轨道路径反映为生成的记录的不同长度和身份。
为了表征轨道结构的组装,使用直接纳米级成像方法,例如原子力显微镜和超分辨率成像。为了表征结果并证实爬行者的关键能力,使用了多种表征方法。首先,凝胶电泳显示具有正确长度的记录的形成以及在具有冗余引物的轨道的情况下形成多种类型的记录。对于完全规定的轨道,凝胶条带似乎具有对应于预定数量的轨道位点的长度。对于具有冗余引物的轨道,允许爬行者自由地漫游,可以看到具有跨越不同长度的长度的凝胶条带的分布;在爬行者的情况下,观察到的最大长度对应于轨道位点的数量。与1d轨道一样,根据反应规模和记录产生产量,任选地添加记录的pcr扩增,然后进行凝胶表征。其次,pcr用于选择性地放大特定种类的记录并检测指定记录的形成。例如,与第一和最后一个轨道位点的引物/引物结合区互补的pcr引物用于选择性扩增完整记录。第三,使用下一代测序方法直接检查所生成记录的身份。这些表征方法的组合允许确认分子爬行者通过多种路径重复访问所有轨道点。
实施例18.定量信息检查
分子马达可以在给定轨道周围漫游,并复制和“记录”来自轨道位点的信息。为了这种能力的一般性,推定未知的轨道,每个位点用冗余引物标记(所有位点具有相同的引物)。根据产生的记录的长度,获得关于轨道尺寸和分子所采取的步骤数量的定量信息。
首先在具有确定数量的轨道位置的dna纳米结构轨道上测试分子马达的计数能力。例如,在1d布置上具有3个点和5个点的轨道被测试用于表征和优化性能(图33a)。还测试了更复杂的2d轨道,例如圆圈,以检查在广义轨道上成功计数行为的能力。从具有冗余引物的轨道,爬行者生成具有长度分布的记录,其中最大长度指示轨道位点的最大可能数量(参见图33b中的示意图),因为一旦访问位点,则位点不能被重新访问,直至从轨道上释放完整的爬行者。步行者生成具有长度分布的记录,其反映出步行者所采取的步骤数量。接下来,分子马达系统在生物系统中实施(图28b)。例如,测试了特定分子复合物的靶向。使用偶联有其附着了“轨道”位点的dna锚的抗体靶向核孔复合物用于收集定量信息(参见例如图33c,用于测试平台)。核孔复合物的尺寸作为测试系统是相关且理想的,因为相邻亚单位之间的距离为约30nm。正交系统用于获得定量信息,例如超分辨率的直接成像(图31c)或基于随机成像的动力学轨迹的分析计数,称为qpaint(可靠地应用于图31c中所示的相同系统),作为用于比较读数和不同方法的相对性能的参考。
实施例19.身份信息检查
当分子马达复制并报告来自轨道的信息时,如果轨道位点含有例如独特dna序列的独特信息,则爬行者生成的记录将含有每个位点的身份信息。因此,除了定量信息之外,还可以获得独特的身份信息(图33d)。
为了显示含有位点的身份信息的记录,使用多种表征方法。首先,凝胶电泳结合特定种类记录的选择性pcr扩增揭示了特定记录的产生。其次,基于下一代测序方法直接检查序列信息,揭示了所产生记录的分子身份。
实施例20.分子景观重构
分子马达的性质:(1)记录给定分子靶标的身份信息和(2)通过多种路径沿同一靶标重复记录,使得马达能够检查和报告靶标的景观。通过多个爬行者的动作收集的集体信息用于分析靶位点的几何排列,并重构分子景观。
沿着给定分子景观的信息收集在具有确定的轨道位点排列的dna纳米结构轨道上进行测试(图33e)。爬行者可以在每轮检查和记录中采用不同的路径。每轮后生成的记录反映了相邻分子位点的邻近(proximity)信息的集合。通过下一代测序方法直接检查记录,使得能够将每条记录分配到沿着景观的可能路径。一些邻近信息与来自不同记录的信息重叠。在交接点周围生成的记录包含来自多对轨道位点的邻近信息。编译所有邻近信息使得能够收集相邻点和交接点的信息,从而允许重构分子景观。
实施例21.分子距离记录
四个不同长度的dna棒(10nm)(rod38、rod49、rod59、rod70)和阴性对照用于本实施例中描述的分子记录实验。实验结果如图38a所示。数字(例如rod38)表明碱基对中双链区的长度。dna棒与在1×thermopol缓冲液(3mmmg2+)中的前体(100nm)、催化发夹(1μm)、dats(10μm)、dttp(10μm)和bst大片段dna聚合酶(0.26u/μl)在37℃独立孵育处理4h。反应产物在65℃在15%page凝胶(0.5×tbe,8m脲)上电泳并用typhoon凝胶扫描仪可视化。结果显示产生的记录对应于测量的距离。一些凝胶条带用其相应的dna记录标记。
将三种不同长度(5.4nm、12.6nm和19.7nm)的dna棒(浓度100pm)分别与在thermopol缓冲液(补充有5mmmgso4)中的前体(100pm)、催化发夹(200nm)、datp(100um)、dttp(100μm)和dctp(100μm)和bst大片段dna聚合酶(0.4u/ul)于37℃孵育30min。然后用exoidna核酸外切酶(0.1u/μl)处理溶液以消化未反应和未完成的单链产物。产生的记录通过pcr扩增(5个主要cy5染料标记的引物-250nm,dntp-200μm,ventexo-0.5u/μl,thermopol缓冲液-1×,18个循环,标准pcr方案,退火温度46℃)。pcr反应的结果在12%page凝胶(0.5×tbe,8m脲,200v,65℃)上电泳,并用typhoon凝胶扫描仪可视化。结果如图38b所示。产生的记录对应于测量的距离。一些凝胶条带用其相应的dna记录标记,以帮助解释凝胶。
实施例22.有关dna纳米结构的分子标尺技术的验证
dna纳米结构用于精确定位已知的固定距离处的dna靶(图35a)且实验表明分子标尺可以记录这些距离。
实施例23.对复杂2d几何形状的单一分子、多重和高通量测定
核孔复合物(npc)的结构模拟物通过将八个条形码的dna靶放置在dna纳米结构上的正八边形的顶点上精确产生(图35b)。然后,分子标尺用于记录距离。使用下一代序列技术读出距离和相应的编码条形码。然后使用分子标尺来记录npc模拟物的低拷贝数(104-106)来对应于典型生物学实验中的原位条件。
实施例24.将分子标尺应用于固定的u2os细胞中的核孔复合物
固定的u2os细胞(人骨骨肉瘤细胞)中的核孔蛋白nup98用定制dna缀合的单克隆抗体(#2598,cellsignaling)标记。超分辨率研究表明,这种标记导致八个簇呈八边形排列,相邻簇之间的距离约为30nm。分子标尺用于记录簇之间的距离,其分辨率低于4nm。凝胶电泳读出值显示预期的四个不同的对角线距离及其变异值。ngs读出值揭示了单分子距离,使得能够研究npc的异质性。
方法
dna合成和纯化。所有的寡核苷酸都是从idt订购的,未经过纯化处理或hplc纯化的。订购了用无rnase的hplc进行纯化的纯化的rna分子。通过qiagenminelutepcr纯化柱运行100μl的100μm未纯化的寡核苷酸从而在内部纯化一些未纯化的寡核苷酸,并按照试剂盒说明书进行洗涤。将柱结合的寡核苷酸洗脱至15μl,并使用nanodrop及其寡核苷酸分析仪(www.idtdna.com/site/order/oligoentry)消光系数测量浓度。订购了预先悬浮于100μm的1×te缓冲液中的寡核苷酸,并且推定所有稀释液都具有这些浓度,但minelute纯化的寡核苷酸除外。将所有寡核苷酸都在1×te中稀释至10μm的工作浓度,将dna的储备和工作溶液储存在-20℃并将rna储存在-80℃。
per孵育。将所有per实验通常与1xthermopol缓冲液(20mmtris-hcl、10mm(nh4)2so4、10mmkcl、2mmmgso4和0.1%
凝胶电泳。大多数实验使用15%tbe-ureapage变性凝胶,其在65℃在200v下电泳35分钟并用cy5和fam通道扫描。凝胶也用sybrgold染色几分钟,然后用sybrgold通道成像。一些实验使用不同的凝胶条件。
afm。使用nanoscopev机器进行afm成像。
序列设计。大多数序列使用与命令行nupack可执行文件配对的内部优化代码设计。nupack互联网应用程序也用于分析构建体。
实施例25.爬行者系统
对分子马达系统之一(爬行者)进行一系列测试以确认基本操作(图43a-43c)。设计了沿dna纳米结构平台上的三角形排列的三点轨道。图43a的上图描绘了该设计的示意图,图43a的下图显示了在三个靶位点上爬行之后的爬行者的分子细节。完成爬行过程后,爬行者将成为长度为118nt的完整记录。当通过pcr扩增并在变性凝胶上电泳时,最终记录出现在预期的长度范围内(图43b)。还使用原子力显微镜(afm)使爬行者可视化。图43c的左图显示了在添加引发爬行反应的引物之前的靶探针,其中探针显示为点。在约1小时的记录反应之后,爬行者将三个轨道位点连接在一起,如图43c的右图所示,因此在afm图像中相应地出现。这些测试结果证明了爬行系统每个步骤的基本操作:(1)引物结合,(2)通过聚合酶的引物延伸,(3)通过模板的置换,(4)与邻近位点的相互作用,(5)通过聚合酶的额外延伸,(6)记录的自主释放。
参考文献,各自以其完整形式并入。
1.b.yurke,a.j.turberfield,a.p.mills,jr.,f.c.simmel和j.l.neumann.adna-fueledmolecularmachinemadeofdna.nature,406:605–608,2000.
2.r.m.dirks和n.a.pierce.triggeredamplificationbyhybridizationchainreaction.pnas,101:15275–15278,2004.
3.g.seelig,d.soloveichik,d.y.zhang和e.winfree.enzyme-freenucleicacidlogiccircuits.science,314(5805):1585–1588,2006.
4.l.qian和e.winfree.scalingupdigitalcircuitcomputationwithdnastranddisplacementcascades.science,332:1196–1201,2011.
5.w.b.sherman和n.c.seeman.apreciselycontrolleddnabipedwalkingdevice.nanoletters,4:1203–1207,2004.
6.p.yin,h.m.t.choi,c.r.calvert和n.a.pierce.programmingbiomolecularself-assemblypathways.nature,451:318–322,2008.
7.t.omabegho,r.sha和n.seeman.abipedaldnabrownianmotorwithcoordinatedlegs.science,2009.
8.ralfjungmann,christiansteinhauer,maxscheible,antonkuzyk,philiptinnefeld和friedrichcsimmel.singlemoleculekineticsandsuper-resolutionmicroscopybyfluorescenceimagingoftransientbindingondnaorigami.nanoletters,10(11):4756–4761,2010.
9.ralfjungmann,maiers
10.j.chen和n.c.seeman.thesynthesisfromdnaofamoleculewiththeconnectivityofacube.nature,350:631–633,1991.
11.p.w.k.rothemund.foldingdnatocreatenanoscaleshapesandpatterns.nature,440(7082):297–302,2006.
12.y.he,t.ye,m.su,c.zhang,a.e.ribbe,w.jiang和c.d.mao.hierarchicalself-assemblyofdnaintosymmetricsupramolecularpolyhedra.nature,452:198–201,2008.
13.y.ke,j.sharma,m.liu,k.jahn,y.liu和h.yan.scaffoldeddnaorigamiofadnatetrahedronmolecularcontainer.nano.lett.,9:2445–2447,2009.
14.e.s.andersen,m.dong,m.m.nielsen,k.jahn,r.subramani,w.mamdouh,m.m.golas,b.sander,h.stark,c.l.p.oliveira,j.s.pedersen,v.birkedal,f.besenbacher,k.v.gothelf和j.kjems.self-assemblyofananoscalednaboxwithacontrollablelid.nature,459:73–76,2009.
15.s.douglas,h.dietz,t.liedl,b.
16.h.dietz,s.douglas,andw.shih.foldingdnaintotwistedandcurvednanoscaleshapes.science,325:725–730,2009.
17.t.liedl,b.
18.d.han,s.pal,j.nangreave,z.deng,y.liu,andh.yan.dnaorigamiwithcomplexcurvaturesinthree-dimensionalspace.science,332:342–346,2011.
19.s.h.park,p.yin,y.liu,j.h.reif,t.h.labean和h.yan.programmablednaself-assembliesfornanoscaleorganizationofligandsandproteins.nanolett.,5:729–733,2005.
20.f.aldaye,a.palmer和h.sleiman.assemblingmaterialswithdnaastheguide.science,321:1795–1799,2008.
21.h.yan,s.h.park,g.finkelstein,j.h.reif,andt.h.labean.dna-templatedself-assemblyofproteinarraysandhighlyconductivenanowires.science,301(5641):1882–1884,2003.
22.c.j.delebecque,a.b.lindner,p.a.silver,andf.a.aldaye.organizationofintracellularreactionswithrationallydesignedrnaassemblies.science,333:470–474,2011.
23.a.kuzyk,r.schreiber,z.fan,g.pardatscher,e.-m.roller,a.hogele,f.c.simmel,a.o.govorov,andtliedl.dna-basedself-assemblyofchiralplasmonicnanostructureswithtailoredopticalresponse.nature,483:311–314,2012.
24.h.m.t.choi,trinhchang,j.y.,padillal.a.,s.e.j.e.,fraser和n.a.pierce.programmableinsituamplificationformultiplexedimagingofmrnaexpression.naturebiotechnol,28:1208,2010.
25.c.lin,r.jungmann,a.m.leifer,c.li,d.levner,g.m.church,w.m.shih和p.yin.submicrometregeometricallyencodedfluorescentbarcodesself-assembledfromdna.naturechemistry,4:832–839,2012.
26.jinglinfu,minghuiliu,yanliu,nealwwoodbury和haoyan.interenzymesubstratediffusionforanenzymecascadeorganizedonspatiallyaddressablednananostructures.journaloftheamericanchemicalsociety,134(12):5516–5519,2012.
27.weisun,etienneboulais,yerahakobyan,weiliwang,amyguan,markbathe和pengyin.castinginorganicstructureswithdnamolds.science,346(6210):1258361,2014.
28.m.j.berardi,w.m.shih,s.c.harrison和j.j.chou.mitochondrialuncouplingprotein2structuredeterminedbynmrmolecularfragmentsearching.nature,476:109–113,2011.
29.s.venkataraman,r.m.dirks,c.t.ueda和n.a.pierce.selectivecelldeathmediatedbysmallconditionalrnas.procnatlacadsciusa,107:16777,2010.
30.lisamhochrein,maayanschwarzkopf,monashahgholi,pengyin,andnilesapierce.conditionaldicersubstrateformationviashapeandsequencetransductionwithsmallconditionalrnas.journaloftheamericanchemicalsociety,135(46):17322–17330,2013.
31.s.m.douglas,i.bachelet和g.m.church.alogic-gatednanorobotfortargetedtransportofmolecularpayloads.science,335:831–834,2012.
32.qiaojiang,chensong,jeanettenangreave,xiaoweiliu,linlin,dengliqiu,zhen-gangwang,guozhangzou,xingjieliang,haoyan等人dnaorigamiasacarrierforcircumventionofdrugresistance.journaloftheamericanchemicalsociety,134(32):13396–13403,2012.
33.t.j.fu和n.c.seeman.dnadouble-crossovermolecules.biochemistry,32:3211–3220,1993.
34.e.winfree,f.liu,l.a.wenzler和n.c.seeman.designandself-assemblyoftwo-dimensionaldnacrystals.nature,394:539–544,1998.
35.s.douglas,a.h.marblestone,s.teerapittayanon,a.vazquez,g.m.church和w.shih.rapidprototypingof3ddnaorigamishapeswithcadnano.nucleicacidsresearch,37:5001–5006,2009.
36.carlosernestocastro,fabiankilchherr,do-nyunkim,enriquelinshiao,tobiaswauer,philippwortmann,markbathe和hendrikdietz.aprimertoscaffoldeddnaorigami.naturemethods,8(3):221–229,2011.
37.p.wrothemund,a.ekani-nkodo,n.papadakis,a.kumar,d.k.fygenson和winfreee.designandcharacterizationofprogrammablednananotubes.jacs,126:16344–163452,2005.
38.p.yin,r.hariadi,s.sahu,h.m.t.choi,s.h.park,t.h.labean,andj.h.reif.programmingmoleculartubecircumferences.science,321:824–826,2008.
39.j.p.zheng,j.birktoft,y.chen,t.wang,r.j.sha,p.constantinou,s.ginell,c.d.mao,andn.seeman.frommoleculartomacroscopicviatherationaldesignofaself-assembled3ddnacrystal.nature,461:74–77,2009.
40.p.w.k.rothemund,n.papadakis和e.winfree.algorithmicself-assemblyofdnasierpinskitriangles.plosbiology,2:2041–2053,2004.
41.a.chworos,isevercan,a.y.a.y.koyfman,p.weinkam,e.eminoroudjev,h.g.hansma和ljaeger.buildingprogrammablejigsawpuzzleswithrna.science,306:2068–2072,2004.
42.i.severcan,gearyc.,chworosa.,vossn.,jacovettye.和jaegerl.apolyhedronmadeoftrnas.naturechemistry,2:772–779,2010.
43.d.soloveichik,g.seelig和e.winfree.dnaasauniversalsubstrateforchemicalkinetics.proceedingsofthenationalacademyofsciencesoftheunitedstatesofamerica,107:5393–5398,2010.
44.davidyuzhang,sherryxichen,andpengyin.optimizingthespecificityofnucleicacidhybridization.naturechemistry,4(3):208–214,2012.
45.sherryxichen,davidyuzhang,andgeorgseelig.conditionallyfluorescentmolecularprobesfordetectingsinglebasechangesindouble-strandeddna.naturechemistry,5(9):782–789,2013.
46.j.kim,j.j.hopfield和e.winfree.neuralnetworkcomputationbyinvitrotranscriptionalcircuits.in18thneuralinformationprocessingsystems(nips),2004.
47.luluqian,erikwinfree和jehoshuabruck.neuralnetworkcomputationwithdnastranddisplacementcascades.nature,475(7356):368–372,2011.
48.jongminkim和erikwinfree.syntheticinvitrotranscriptionaloscillators.molecularsystemsbiology,7(1):465,2011.
49.joshuaiglaser,bradleymzamft,adamhmarblestone,jeffreyrmoffitt,keithtyo,edwardsboyden,georgechurch和konradpkording.statisticalanalysisofmolecularsignalrecording.ploscomput.biol,9:e1003145,2013.
50.christhachuk,erikwinfree和davidsoloveichik.leaklessdnastranddisplacementsystems.indnacomputingandmolecularprogramming,pages133–153.springer,2015.
51.ryosukeiinuma,yonggangke,ralfjungmann,thomasschlichthaerle,johannesbwoehrstein,andpengyin.polyhedraself-assembledfromdnatripodsandcharacterizedwith3ddna-paint.science,344(6179):65–69,2014.
52.d.wei,m.dai和p.yin.complexshapesself-assembledfrommodularmolecularcomponents.nature,485:623–626,2012.
53.y.ke,l.l.ong,w.m.shih和p.yin.three-dimensionalstructuresself-assembledfromdnabricks.science,2012.
54.d.han和p.yin.replicablesingle-strandeddnaorigami.inpreparation,2015.
55.johnpsadowski,colbyrcalvert,davidyuzhang,nilesapierce和pengyin.developmentalself-assemblyofadnatetrahedron.acsnano,8(4):3251–3259,2014.
56.simonfredriksson,matsgullberg,jonasjarvius,charlottaolsson,kristianpietras,sigrúnmargrétgústafsdóttir,arne
57.yoshinagayoshimura和kenzofujimoto.ultrafastreversiblephoto-cross-linkingreaction:towardinsitudnamanipulation.organicletters,10(15):3227–3230,2008.
58.alexanderagreen,pamelaasilver,jamesjcollins和pengyin.toeholdswitches:de-novo-designedregulatorsofgeneexpression.cell,159(4):925–939,2014.
58.davidyuzhang和erikwinfree.controlofdnastranddisplacementkineticsusingtoeholdexchange.jamchemsoc,131(47):17303-14,2009.
59.国际公开号wo2012/058488a1(国际申请号pct/us2011/058178,2011年10月27日提交).
60.rothemund,paulw.k.foldingdnatocreatenanoscaleshapesandpatterns.nature440,297-302,2006.
本文公开的全部参考文献、专利和专利申请在其引述的各自主题方面通过引用并入,在一些情况中,其可以涵盖整个文件。
如在本说明书和权利要求中所用,除非明确相反指出,否则不定冠词“一个/种(a)”和“一个/种(an)”应理解为“至少一个/种”。
还应当理解,除非明确相反指出,否则在本文所要求保护的包括不止一个步骤或动作的任何方法中,方法的步骤或动作的顺序不一定限于其中引述该方法的步骤或动作的顺序。
在权利要求中以及上述说明书中,所有转换性短语如“包含”、“包含”、“携带”、“具有”、“含有”、“涉及”、“持有”、“包括”等应理解为是开放式的,即意味着包括但不限于此。只有“由...组成”和“基本上由...组成”的转换性短语分别是“美国专利局专利审查程序手册”第2111.03节中规定的封闭或半封闭的连接词。
序列表
<110>哈佛学院院长及董事
<120>分子编程工具
<130>h0498.70589wo00
<140>notyetassigned
<141>2017-02-16
<150>us62/432,017
<151>2016-12-09
<150>us62/429,149
<151>2016-12-02
<150>us62/299,206
<151>2016-02-24
<150>us62/296,310
<151>2016-02-17
<160>7
<170>patentinversion3.5
<210>1
<211>10
<212>dna
<213>artificialsequence
<220>
<223>syntheticpolynucleotide
<400>1
atctcttatt10
<210>2
<211>69
<212>dna
<213>artificialsequence
<220>
<223>syntheticpolynucleotide
<220>
<221>misc_feature
<222>(15)..(15)
<223>gisiisodg
<220>
<221>misc_feature
<222>(24)..(24)
<223>tisibiodt
<220>
<221>misc_feature
<222>(32)..(32)
<223>modifiedbyamethylgroup
<220>
<221>misc_feature
<222>(33)..(33)
<223>cisime-isodc
<220>
<221>misc_feature
<222>(60)..(60)
<223>cisime-isodc
<220>
<221>misc_feature
<222>(69)..(69)
<223>tis3invdt
<400>2
acatactcatctcggcgctggtttttccagcgccgagatgagtatgtagttagagatgtc60
acgagatgt69
<210>3
<211>69
<212>dna
<213>artificialsequence
<220>
<223>syntheticpolynucleotide
<220>
<221>misc_feature
<222>(15)..(15)
<223>gisiisodg
<220>
<221>misc_feature
<222>(24)..(24)
<223>tisibiodt
<220>
<221>misc_feature
<222>(32)..(32)
<223>modifiedbyamethylgroup
<220>
<221>misc_feature
<222>(33)..(33)
<223>cisime-isodc
<220>
<221>misc_feature
<222>(69)..(69)
<223>tis3invdt
<400>3
acatactcatctccgcgctggtttttccagcgcggagatgagtatgtagttagagatgtg60
acgagatgt69
<210>4
<211>69
<212>dna
<213>artificialsequence
<220>
<223>syntheticpolynucleotide
<220>
<221>misc_feature
<222>(15)..(15)
<223>gisiisodg
<220>
<221>misc_feature
<222>(24)..(24)
<223>tisibiodt
<220>
<221>misc_feature
<222>(32)..(32)
<223>modifiedbyamethylgroup
<220>
<221>misc_feature
<222>(33)..(33)
<223>cisime-isodc
<220>
<221>misc_feature
<222>(59)..(59)
<223>modifiedbyamethylgroup
<220>
<221>misc_feature
<222>(60)..(60)
<223>modifiedbyamethylgroup
<220>
<221>misc_feature
<222>(61)..(61)
<223>modifiedbyamethylgroup
<220>
<221>misc_feature
<222>(69)..(69)
<223>tis3invdt
<400>4
acatactcatctcggcgctggtttttccagcgccgagatgagtatgtagttagagatgtg60
acgagatgt69
<210>5
<211>10
<212>dna
<213>artificialsequence
<220>
<223>syntheticpolynucleotide
<400>5
ttctcttatt10
<210>6
<211>43
<212>dna
<213>artificialsequence
<220>
<223>syntheticpolynucleotide
<400>6
actaaattcagggccttttggccctgaatttagtaataagaga43
<210>7
<211>57
<212>dna
<213>artificialsequence
<220>
<223>syntheticpolynucleotide
<400>7
ugugcaaaucuaugcaaaacugaatagtgggaggctagctacaacgagcggacattt57
- 还没有人留言评论。精彩留言会获得点赞!