植物内生菌中的代谢产物产生的制作方法
本发明涉及janthitrem化合物的生物合成。具体地,本发明涉及编码负责合成janthitrem的酶的基因以及相关的构建体、载体和方法。
背景技术:
植物内生菌存在于活植物的组织中,并提供新化合物和基因的特别多样的来源,其可以为社会、特别是为农业提供重要的益处。植物内生菌通常与其宿主形成共生关系,植物内生菌通常通过产生防御化合物赋予宿主增加的适应性。同时,宿主植物为植物内生菌提供受保护的环境和营养物的益处。该植物为植物内生菌提供营养物,以及通过种子的传播途径。植物内生菌保护宿主免受生物(例如昆虫和哺乳动物的食草动物)和非生物胁迫(例如干旱)。
发现重要的禾本科牧草多年生黑麦草和高羊茅草通常与真菌性植物内生菌有关。有益和有害的农艺性质都是由这种关联产生的,包括改善的对水和营养胁迫的耐受性以及对昆虫害虫的抗性。昆虫抗性由植物内生菌产生的特定代谢产物提供,特别是黑麦草碱生物碱和波胺(peramine)。由植物内生菌产生的其他代谢产物(黑麦草神经毒素(lolitrem)和麦角生物碱)对食草动物有毒并减少食草动物的摄食。这些化合物可以在植物中积累到很高的水平,在植物中它们可以作为对抗一系列昆虫害虫的有效摄食忌避剂(feedingdeterrent)。
janthitrem是一类吲哚二萜并由植物内生菌亚群产生。在1980年,黑麦草蹒跚综合征(ryegrassstaggerssyndrome)的爆发使得首次识别janthitrem生物碱(gallagher等人,1980)。最近的发现突出了janthitrem的多样性;来自澳大利亚和新西兰的微紫青霉菌(p.janthinellum)分离株产生广泛的janthitrem(janthitremb、c、d、e、f和g)。
janthitrem是一类与黑麦草神经毒素b结构相似的吲哚二萜(图1)。环氧-janthitrem是一组五种化合物:与环氧-janthitremi一起分离的三种其他结构被指定为环氧-janthitremii[10-脱乙酰基-10,34-(3-甲基丁-2-烯基缩醛)];环氧-janthitremiii[10-脱乙酰基-34-o-(3-甲基丁-2-烯基)];和环氧-janthitremiv[34-o-(3-甲基丁-2-烯基)],基于lc-ms分析,其中每一种都是环氧-janthitremi的衍生物。环氧-janthitremi是由多年生黑麦草植物内生菌产生的主要的janthitrem生物碱。
多年生黑麦草牧场中的janthitrem的存在针对各种重要的牧草害虫提供了卓越的保护。最近的发现表明,janthitrem在本质上可能是震颤源的,类似于黑麦草神经毒素b。已知黑麦草神经毒素b是黑麦草蹒跚病的主要致病剂。这种情况下,以植物内生菌感染的牧草为食的动物会出现共济失调、震颤和对外部刺激的过敏。像黑麦草神经毒素b一样,janthitremb可以诱发震颤反应。最近来自微紫青霉菌的janthitrema和b的生物活性研究发现这两种化合物对小鼠具有震颤作用并且对porina(wiseanacervinata)幼虫具有抗昆虫活性(babu,2009)。此外,当纯化时,已观察到
尽管有这些有用的性质,但与由植物内生菌合成的其他生物碱组相比,janthitrem生物碱还未被很好了解。越来越需要进一步理解janthitrem及其生物合成,因为这将提供有助于操纵janthitrem产生的信息。
本发明的一个目的是克服或至少减轻与现有技术相关的一个或多个困难或缺陷。
技术实现要素:
一方面,本发明提供了基本上纯化或分离的核酸或核酸片段,其编码参与植物内生菌中janthitrem生物合成的基因。
“核酸”是指能够具有遗传信息的核苷酸链。该术语通常是指基因或其功能活性片段或变体和/或生物体基因组中影响其表型的其他序列。术语“核酸”包括单链或双链的dna(例如cdna或基因组dna)和rna(例如mrna或microrna),任选地含有合成的、非天然的或改变的核苷酸碱基,合成的核酸和它们的组合。
本发明的核酸可以是全长基因或其部分,并且在本说明书中也称为“核酸片段”和“核苷酸序列”。为方便起见,表述“核酸或核酸片段”用于涵盖所有这些。
“基本上纯化的”是指核酸不含这样的基因,即在本发明的核酸所源自的生物体的天然存在的基因组中位于所述核酸侧翼的基因。因此,该术语包括,例如,掺入载体中的核酸;掺入自主复制的质粒或病毒中的核酸;或掺入原核生物或真核生物的基因组dna中的核酸;或作为独立于其他序列的独立分子存在的核酸(例如通过pcr或限制性核酸内切酶消化产生的cdna或基因组或cdna片段)。它还包括核酸,其是编码另外的多肽序列的杂合基因的一部分。优选地,基本上纯化的核酸纯度是90%,更优选95%,甚至更优选98%。
术语“分离的”是指将物质从其原始环境(例如,如果它是天然存在的则为自然环境)中移出。例如,活植物中的天然存在的核酸是未分离的,而从自然体系中的一些或所有共存物质分离出的相同核酸是分离的。此类核酸可以是载体的一部分和/或此类核酸可以是组合物的一部分,并且仍然是分离的,因为这样的载体或组合物不是其自然环境的一部分。
在本发明该方面的一个优选实施方案中,产生janthitrem的植物内生菌是
在本发明的第二方面,提供了基本上纯化或分离的核酸或核酸片段,其编码janthitrem生物合成多肽,或与编码janthitrem生物合成多肽的序列互补或反义,所述核酸或核酸片段包括选自以下的核苷酸序列:(a)图7、10、13和16中所示的序列(序列idno1、2、5、6、9、10、13和14);(b)(a)中所述序列的互补序列;(c)与(a)和(b)中所述序列反义的序列;和(d)(a)、(b)和(c)中所述序列的功能活性片段和变体,其与(a)、(b)和(c)中所述序列的相关部分具有至少约80%的同一性并且大小为至少20个核苷酸。
本发明包括本发明核酸的功能活性片段和变体。关于核酸的“功能活性”是指片段或变体(例如类似物、衍生物或突变体)能够调节janthitrem生物合成。此类变体包括天然存在的等位基因变体和非天然存在的变体。考虑了一个或多个核苷酸的插入、缺失、置换和衍生化,只要这些修饰不会导致片段或变体的功能活性丧失。优选地,功能活性片段或变体与所述片段或变体对应的上述序列的相关部分具有至少约80%的同一性,更优选至少约90%的同一性,甚至更优选至少约95%的同一性,最优选至少约98%的同一性。这些功能活性变体和片段包括例如具有保守核酸变化的功能活性变体和片段。
优选地,片段的大小为至少20个核苷酸,更优选至少50个核苷酸,更优选至少100个核苷酸,更优选至少200个核苷酸,更优选至少500个核苷酸。
“保守核酸变化”是指由于遗传密码的简并性而导致编码蛋白中氨基酸保守的核酸置换。此类功能活性变体和片段还包括,例如,具有导致相应氨基酸序列中一个或多个残基的保守氨基酸置换的核酸变化的那些功能活性变体和片段。
“保守氨基酸置换”是指由同一类别中的另一个氨基酸置换,所述类别如下:
非极性:ala、val、leu、ile、pro、met、phe、trp
不带电的极性:gly、ser、thr、cys、tyr、asn、gln
酸性:asp、glu
碱性:lys、arg、his
其他保守氨基酸置换也可以如下进行:
芳香族:phe、tyr、his
质子供体:asn、gln、lys、arg、his、trp
质子受体:glu、asp、thr、ser、tyr、asn、gln
在本发明的另一方面,提供了包含本发明的核酸的遗传构建体。在优选的实施方案中,遗传构建体可以包括嵌合序列,其包含本发明的核酸和编码janthitrem生物合成的介体或调节子的基因。优选地,编码janthitrem生物合成的介体或调节子的基因是外源的,即它不天然地与本发明的核酸组合存在。
如本文所用的术语“遗传构建体”是指人工组装或分离的核酸分子,其包括目的基因。优选地,遗传构建体是重组核酸分子。通常,构建体可以包括一个或多个目的基因、在某些情况下也可以是目的基因的标记物基因和合适的调节序列。优选地,标记物基因是外源的,即它不天然地与本发明的核酸组合存在。
应当理解,在构建体中包含调节序列是任选的,例如,在要使用宿主细胞的调节序列的情况下可能不需要这样的序列。术语构建体包括载体,但不应视为限于此。
“嵌合序列”是指通过重组方式通过表达融合基因产生的杂交体,所述融合基因包括两种或更多种连接的最初编码单独蛋白的核酸或其功能活性片段或变体。
“融合基因”是指两种或更多种核酸以允许融合蛋白表达的方式连接,优选作为翻译融合。这通常涉及从编码第一蛋白的核酸序列中除去终止密码子,然后在框内添加第二蛋白的核酸序列。然后融合基因由细胞表达为单一蛋白。可以将蛋白改造成包括两种原始蛋白的全部序列,或其中一种或两种的功能活性片段或变体。
在优选的实施方案中,本发明的遗传构建体可以是载体。
载体可以是任何合适的类型,可以是病毒的或非病毒的。载体可以是表达载体。这些载体包括染色体、非染色体和合成的核酸序列,例如,植物病毒的衍生物;细菌质粒;来自根癌土壤杆菌(agrobacteriumtumefaciens)的ti质粒的衍生物;来自毛根土壤杆菌(agrobacteriumrhizogenes)的ri质粒的衍生物;噬菌体dna;酵母人工染色体;细菌人工染色体;二元细菌人工染色体;衍生自质粒和噬菌体dna的组合的载体。然而,可以使用任何其他载体,只要它在靶细胞中是可复制的或整合的或可存活的即可。
在本发明该方面的一个优选实施方案中,遗传构建体可以进一步包括启动子和终止子;所述启动子、基因和终止子是可操作地连接的。
“启动子”是指足以指导可操作地连接的核酸序列转录的核酸序列。
“可操作地连接的”是指核酸和调节序列(例如启动子)是以允许所述核酸在适当条件下表达的方式连接的,例如当适当的分子(如转录激活蛋白)与调节序列结合时。优选地,可操作地连接的启动子位于相关核酸的上游。
“上游”是指沿核酸的3′→5′方向。
启动子和终止子可以是任何合适的类型,并且可以是靶细胞的内源的,或者可以是外源的,只要它们在靶细胞中有功能。优选地,启动子和/或终止子是外源的,即它不天然地与本发明的核酸组合存在。
可用于本发明遗传构建体中的多种终止子也是本领域技术人员公知的。终止子可以来自与启动子序列相同的基因或不同的基因。特别合适的终止子是多腺苷酸化信号,例如(camv)35spolya和来自胭脂碱合酶(nos)基因和章鱼碱合酶(ocs)基因的其他终止子。
除了启动子、基因和终止子之外,遗传构建体可以包括以不同组合表达核酸所必需的其他元件,例如载体骨架、复制起点(ori)、多克隆位点、间隔子序列、增强子、内含子(如玉米泛素ubi内含子)、抗生素抗性基因和其他选择性标记物基因[如新霉素磷酸转移酶(nptii)基因、潮霉素磷酸转移酶(hph)基因、膦丝菌素乙酰转移酶(bar或pat)基因]和报告子基因[如β-葡糖醛酸酶(gus)基因(gusa)和绿色荧光蛋白(gfp)基因(gfp)]。遗传构建体还可含有翻译起始的核糖体结合位点。遗传构建体还可以包括用于扩增表达的合适序列。
本领域技术人员将理解,遗传构建体的各种组分是可操作地连接的,从而导致所述核酸的表达。用于可操作地连接本发明的遗传构建体的组分的技术是本领域技术人员公知的。这些技术包括使用接头,例如合成接头,例如包括一个或多个限制酶位点。
优选地,遗传构建体基本上是纯化的或分离的。
“基本上纯化的”是指遗传构建体不含这样的基因,即在本发明核酸或启动子所源自的生物体的天然存在的基因组中位于所述核酸或启动子的侧翼的基因。因此,该术语包括例如掺入载体中的遗传构建体;掺入自主复制的质粒或病毒中的遗传构建体;或掺入原核生物或真核生物的基因组dna中的遗传构建体;或作为独立于其他序列的独立分子存在的遗传构建体(例如通过pcr或限制性核酸内切酶消化产生的cdna或基因组或cdna片段)。它还包括遗传构建体,其是编码另外的多肽序列的杂合基因的一部分。
优选地,基本上纯化的遗传构建体纯度为至少约90%,更优选纯度为至少约95%,甚至更优选纯度为至少约98%。
术语“分离的”是指将物质从其原始环境(例如如果它是天然存在的则为自然环境)中移出。例如,存在于活植物中的天然存在的核酸是未分离的,而从自然体系中的一些或所有共存物质分离出的相同核酸是分离的。此类核酸可以是载体的一部分和/或此类核酸可以是组合物的一部分,并且仍然是分离的,因为这样的载体或组合物不是其自然环境的一部分。
作为使用选择性标记物基因以提供用于选择转化宿主细胞的表型性状的替代方案,转化细胞中遗传构建体的存在可以通过本领域公知的其他技术确定,例如pcr(聚合酶链反应)、southern印迹杂交分析、组织化学测定(例如gus测定)、薄层色谱(tlc)、northern和western印迹杂交分析。
可以通过任何合适的技术将本发明的遗传构建体引入植物或真菌中。将本发明的遗传构建体掺入植物细胞或真菌细胞(例如通过转导、转染、转化或基因靶向)的技术是本领域技术人员公知的。这些技术包括土壤杆菌属(agrobacterium)介导的引入,根瘤菌(rhizobium)介导的引入,对组织、细胞和原生质体的电穿孔,原生质体融合,注入生殖器官,注入未成熟胚和高速投射引入细胞、组织、愈伤组织、未成熟胚和成熟胚,生物弹道转化,whiskers转化及其组合。技术的选择在很大程度上取决于待转化的植物或真菌的类型,并且可以由合适的本领域技术人员容易地确定。对于原生质体的转化,特别优选peg介导的转化。对于真菌转化,特别优选peg介导的转化和原生质体的电穿孔和土壤杆菌属介导的菌丝外植体的引入。
如下所述,可以选择掺入本发明遗传构建体的细胞,然后使用本领域公知的技术在合适的培养基中培养以再生转化的植物或真菌。培养条件,例如温度、ph等,对于本领域技术人员而言是显而易见的。可以使用本领域公知的方法,使所得植物进行有性或无性繁殖,以产生连续世代的转化植物或真菌。
因此,在本发明的另一方面,提供了转基因植物细胞、植物、植物种子或其他植物部分,或转基因真菌、真菌细胞或其他真菌部分,其能够比未转化的对照植物细胞、植物、植物种子或其他植物部分,或未转化的真菌、真菌细胞或其他真菌部分产生更大量的janthitrem。
在优选的实施方案中,转基因植物细胞、植物、植物种子或其他植物部分或转基因真菌、真菌细胞或其他真菌部分相对于未转化的对照,产生的janthitrem的量增加至少约10%,更优选至少约20%,更优选至少约30%,更优选至少约40%。
例如,相对于未转化的对照,janthitrem的量可以增加大约10%至300%,更优选大约20%至200%,更优选大约30%至100%,更优选大约40%至80%。
优选地,转基因植物细胞、植物、植物种子或其他植物部分或转基因真菌、真菌细胞或其他真菌部分包括本发明的核酸、遗传构建体或载体。优选地,通过本发明的方法产生转基因植物细胞、植物、植物种子或其他植物部分或转基因真菌、真菌细胞或其他真菌部分。
本发明还提供了衍生自本发明的植物或真菌细胞并且包括本发明的核酸、遗传构建体或载体的转基因植物、植物种子或其他植物部分或转基因真菌、真菌细胞或其他真菌部分。
本发明还提供了衍生自本发明的植物或真菌并且包括本发明的核酸、遗传构建体或载体的转基因植物、植物种子或其他植物部分或转基因真菌、真菌细胞或其他真菌部分。
“植物细胞”是指由半透膜界定并含有质体的任何自繁殖细胞。如果需要进一步繁殖,这种细胞也需要细胞壁。如本文所用,植物细胞包括但不限于藻类、蓝细菌、种子悬浮培养物、胚、分生组织区、愈伤组织、叶、根、芽、配子体、孢子体、花粉和小孢子。
“真菌细胞”是指真菌的任何细胞。术语“真菌”是指全真菌、真菌器官和组织(例如子囊、菌丝、假菌丝、假根、菌核、担孢子梗(sterigmata)、孢子、分生孢子座、孢子囊、菌丝束(synnemata)、分生孢子、子囊座、闭囊壳、菌丝体、子囊壳、担子等)、孢子、真菌细胞及其后代。真菌可以作为单个细胞存在,也可以构成称为菌丝体的多细胞体,其由称为菌丝的细丝组成。大多数真菌细胞是多核的并且具有细胞壁,其主要由几丁质组成。
“转基因”是指包含dna序列的任何细胞,所述dna序列通过人工插入细胞并成为从该细胞发育的生物体的基因组的一部分。
另一方面,本发明提供了一种修饰植物内生菌中janthitrem生物合成的方法,所述方法包括向所述植物内生菌中引入有效量的如上所述的核酸或核酸片段或构建体。本发明还提供了包含如上所述的核酸或核酸片段或构建体的植物内生菌(例如用如上所述的核酸或核酸片段或构建体的内生菌转化的植物内生菌)。可以通过如上所述的任何合适的方法将核酸、核酸片段或构建体引入植物内生菌中。
另一方面,本发明提供了一种用如上所述的植物内生菌接种的植物,所述植物包含用所述植物内生菌稳定地感染的无植物内生菌的宿主植物。优选地,植物是其中植物内生菌不是天然存在的植物。
优选地,通过选自接种、育种、杂交(crossing)、杂化(hybridization)及其组合的方法,用植物内生菌感染植物。
在优选的实施方案中,植物可以通过等基因接种来感染。这具有以下优点:可以在不存在宿主特异性遗传效应的情况下评估植物内生菌的表型效应。更具体地,可以在植物种质中进行多次植物内生菌接种,并且在转移到土壤之前在培养物中再生小植株。
在植物中高度相容和稳定的相对的交配型植物内生菌的鉴定提供了用于多年生黑麦草的植物内生菌的分子育种的手段。优选地,植物可以通过超接种来感染。
相对的交配型植物内生菌菌株之间的菌丝融合提供了将有利性状递送到宿主植物中的手段,优选通过超接种。这样的菌株优选选自:如下植物内生菌菌株,其表现出高接种频率和与多种种质(优选优良多年生黑麦草和/或高羊茅宿主种质)具有高度相容性的有利特性,以及如下植物内生菌,其表现出低接种频率和低相容性,但具有非常有利的生物碱毒素谱。
一般认为植物内生菌分类群和寄主草之间的相互作用将是物种特异性的。申请人惊奇地发现来自高羊茅的植物内生菌可用于向黑麦草(例如多年生黑麦草)提供有利的性状。
另一方面,本发明提供了植物、植物种子或其他植物部分,其源自本发明的植物并且被本发明的植物内生菌稳定地感染。
优选地,植物细胞、植物、植物种子或其他植物部分是草,更优选饲料草、草皮草或生物能草,例如黑麦草属和羊茅属的那些,包括多年生黑麦草(l.perenne)和l.arundinaceum。
“植物细胞”是指由半透膜界定并含有质体的任何自繁殖细胞。如果需要进一步繁殖,这种细胞也需要细胞壁。如本文所用,植物细胞包括但不限于种子悬浮培养物、胚、分生组织区、愈伤组织、叶、根、芽、配子体、孢子体、花粉和小孢子。
在另一方面,本发明提供了如上所述的植物内生菌用于产生用所述植物内生菌稳定地感染的植物的用途。
在另一方面,本发明提供了一种基本上纯化或分离的多肽,其参与植物内生菌中的janthitrem生物合成。
在一个优选的实施方案中,本发明提供了基本上纯化或分离的janthitrem生物合成多肽,其包括选自以下的氨基酸序列:(a)图8、11、14和17中所示的序列(序列idno3、4、7、8、11、12、15和16);和(b)(a)中所述序列的功能活性片段和变体,其与(a)中所述序列的相关部分具有至少约80%的同一性并且大小为至少20个氨基酸。
本发明包括本发明的多肽的功能活性片段和变体。在本文中,“功能活性”是指片段或变体具有片段或变体从中衍生的相应蛋白的一种或多种生物学性质。考虑了一个或多个氨基酸的插入、缺失、置换和衍生化,只要这些修饰不会导致片段或变体的功能活性丧失。优选地,片段或变体与片段或变体对应的上述序列的相关部分具有至少约80%的同一性,更优选至少约90%的同一性,更优选至少约95%的同一性,最优选至少约98%的同一性。这些功能活性变体和片段包括,例如,在相应的氨基酸序列中具有一个或多个残基的保守氨基酸置换的功能活性变体和片段。
“保守氨基酸置换”是指由同一类别中的另一个氨基酸置换,所述类别如下:
非极性:ala、val、leu、lle、pro、met、phe、trp
不带电的极性:gly、ser、thr、cys、tyr、asn、gln
酸性:asp、glu
碱性:lys、arg、his
其他保守氨基酸置换也可以如下进行:
芳香族:phe、tyr、his
质子供体:asn、gln、lys、arg、his、trp
质子受体:glu、asp、thr、ser、tyr、asn、gln
优选地,片段的大小为至少10个氨基酸,更优选至少20个氨基酸,更优选至少50个氨基酸,更优选至少100个氨基酸,更优选至少200个氨基酸。
在本发明该方面的另一个实施方案中,提供了由本发明的核酸或核酸片段重组产生的多肽。用于重组产生多肽的技术是本领域技术人员已知的。
本发明的核苷酸序列和推导出的氨基酸序列的可用性促进了cdna表达文库的免疫筛选。可以合成代表本发明氨基酸序列部分的合成肽。这些肽可用于免疫动物以产生对包括氨基酸序列的肽和/或蛋白具有特异性的多克隆或单克隆抗体。然后可以将这些抗体用于筛选cdna表达文库以分离目的全长cdna克隆。
现在将参考所附实施例和附图更全面地描述本发明。然而,应该理解,以下描述仅是说明性的,不应以任何方式视为对上述本发明的一般性的限制。
附图简要说明
在附图中:
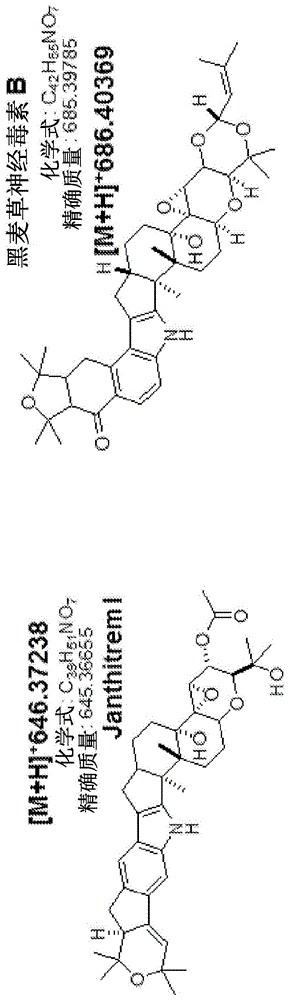
图1:环氧-janthitremi和黑麦草神经毒素b。环氧-janthitremi是类似于蕈青霉素(paxilline)的吲哚二萜,其显示出与黑麦草神经毒素b的结构相似性。来自tapper等人2004的11,12-环氧janthitremg(环氧-janthitremi)的化学式(c39h51no7)和精确质量(645.3665)。
图2:与参照
图3:基因组调查序列分析用于确定植物内生菌菌株中负责波胺、麦角瓦灵和黑麦草神经毒素b生物合成的基因的存在/不存在情况,所述植物内生菌菌株代表观察到与多年生黑麦草形成联合的四个分类群中的每一个(
图4:nea12pacbio重叠群3的长度为247475bp,在四个簇中具有13个预测和已知基因。簇1(ltmg、ltms、ltmm、ltmk),簇2(ltmp、ltmq、ltmf、ltmc、ltmb),簇3(pp01、pp02)和簇4(jtmd和jtmo)。浅灰色箭头显示预测和已知基因及其取向。还显示了pks假基因、具有mule结构域的转座酶(pp03)、helitron解旋酶样转座元件(te)和三个富含at的区域的位置。pp=预测的蛋白;te=转座元件;ψ=假基因。
图5:将来自4个分类群
图6:在植物中表达nea12基因组pacbio重叠群3基因。将来自lptg-3(ar37)和
图6的图解
图7:pp01基因的核苷酸序列(序列idno1)。预测的pp01蛋白的编码序列以灰色突出显示(序列idno2)。通过比对来自图6中所述植物中(alto-nea12)转录组数据的rna读数,然后从nea12pacbio重叠群3中提取dna序列来鉴定pp01基因的完整核苷酸序列。在alto-nea12转录组数据集的分析中未观察到以小写字母显示的核苷酸。
图8:预测pp01是长度为387个氨基酸的细胞色素p450单加氧酶。这里显示的是来自lptg-3菌株nea12(序列idno3)和明尼苏达被毛孢(hirsutellaminnesotensis)(kjz77225氨基酸3-379)(序列idno4)的pp01的预测氨基酸序列的比对。蛋白同一性:258/387(66.7%);蛋白相似性:304/387(78.6%);空位:10/387(2.6%)。使用embossneedle比对序列。
图9:通过来自lptg-3(nea12)的pp01的预测氨基酸序列和ncbi数据库中前6个blastp命中(hit)的最大似然分析产生的自助(bootstrap)共有树。使用具有默认参数的clustalw进行完整预测的蛋白序列的多重比对。为了构建树形拓扑,使用最大似然(ml),如使用默认参数和500次自助重复(replicate)、在mega6中执行的。来自500次自助重复的自助值大于70%的分支在各分支邻近标记。每个树图中提供了每个蛋白序列的genbank登录号。pp01表现出与细胞色素p450单加氧酶的序列相似性:kjz77225.1[68%;明尼苏达被毛孢3608];eql02233.1[57%;冬虫夏草菌(ophiocordycepssinensis)co18];knd87478.1[53%;tolypocladiumophioglossoidescbs100239];oaq66296.1[50%;厚垣普可尼亚菌(pochoniachlamydosporia)170];kom22171.1[55%;偏侧蛇虫草菌(ophiocordycepsunilateralis)];xp_013947710.1[48%;深绿木霉菌(trichodermaatroviride)imi206040]。
图10.pp02基因的核苷酸序列(序列idno5)。预测的pp02蛋白的编码序列以灰色突出显示(序列idno6)。起始密码子(atg)和终止密码子(tga)序列以粗体显示。未翻译的5′和3′序列以小写字母显示。通过比对图6中所述植物中(alto-nea12)转录组数据的rna读数,然后从nea12pacbio重叠群3中提取dna序列来鉴定pp02基因的完整核苷酸序列。
图11.预测pp02是长度为315个氨基酸的膜结合的o-酰基转移酶(mboat)蛋白。这里显示的是来自lptg-3菌株nea12(序列idno7)和大孢树粉孢菌(oidiodendronmaius)zn(kim95229)(序列idno8)的pp02的预测氨基酸序列的比对。蛋白同一性:110/412(26.7%);蛋白相似性:165/412(40.0%);空位:118/412(28.6%)。在预测的mboat结构域内(以粗体显示),两个序列的蛋白同一性为42%(37/89),蛋白相似性为61%(54/89)。使用embossneedle比对序列。
图12.通过来自lptg-3(nea12)的pp02的预测氨基酸序列和ncbi数据库中前5个blastp命中的最大似然分析产生的自助共有树。使用具有默认参数的clustalw进行完整预测的蛋白序列的多重比对。为了构建树形拓扑,使用最大似然(ml),如使用默认参数和500次自助重复、在mega6中执行的。来自500次自助重复的自助值大于70%的分支在各分支邻近标记。每个树图中提供了每个蛋白序列的genbank登录号。pp02表现出与mboat蛋白的序列相似性:kim95229.1[33%;大孢树粉孢菌zn];kzl85868.1[30%;colletotrichumincanum];ccx05903.1[30%;烧土火丝菌(pyronemaomphalodes)cbs100304];kzp09605.1[29%;fibulorhizoctoniasp.cbs109695];xp_007593790.1[31%;colletotrichumfioriniaepj7]。
图13.jtmd基因的核苷酸序列(序列idno9)。预测的jtmd蛋白的编码序列以灰色突出显示(序列idno10)。起始密码子(atg)和终止密码子(tga)序列以粗体显示。未翻译的5′和3′序列以小写字母显示。通过比对图6中所述植物中(alto-nea12)转录组数据的rna读数,然后从nea12pacbio重叠群3中提取dna序列来鉴定jtmd基因的完整核苷酸序列。
图14.预测jtmd是长度为420个氨基酸的芳香族异戊二烯基转移酶(序列idno11)。jtmd与来自偏侧蛇虫草菌(kom22681.1)的预测蛋白(序列idno12)具有最高的同源性。蛋白同一性:264/420(62.9%);蛋白相似性:334/420(79.5%);空位:26/420(6.2%)。使用embossneedle比对序列。
图15.通过来自lptg-3(nea12)的jtmd的预测氨基酸序列和ncbi数据库中前11个blastp命中的最大似然分析产生的自助共有树。使用具有默认参数的clustalw进行完整预测的蛋白序列的多重比对。为了构建树形拓扑,使用最大似然(ml),如使用默认参数和500次自助重复、在mega6中执行的。来自500次自助重复的自助值大于70%的分支在各分支邻近标记。每个树图中提供了每个蛋白序列的genbank登录号。jtmd表现出与芳香族异戊烯基转移酶的氨基酸序列同一性:kom22681.1[67%;偏侧蛇虫草菌];agz20478.1[49%;微紫青霉菌];aak11526.2[46%;p.paxilli];kos22745.1[50%;e.weberi];cej54109.1[47%;巴西青霉(p.brasilianum)];bau61555.1[31%;简单青霉(p.simplicissimum)];agz20194.1[31%;皮落青霉(p.crustosum)];kzf25225.1[33%;xylonaheveaetc161];kgo76903.1[30%;意大利青霉菌(p.italicum)];kjk61458.1[31%;寄生曲霉(aspergillusparasiticus)su-1];cap53937.2[31%;黄曲霉(aspergillusflavus)]。
图16.jtmo基因的核苷酸序列(序列idno13)。预测的jtmo蛋白的编码序列以灰色突出显示(序列idno.14)。起始密码子(atg)和终止密码子(tga)序列以粗体显示。未翻译的5′和3′序列以小写字母显示。通过比对来自图6中描述的植物中(alto-nea12)转录组数据的rna读数,然后从nea12pacbio重叠群3中提取dna序列来鉴定jtmo基因的完整核苷酸序列。
图17.预测jtmo是长度为479个氨基酸的fad结合氧化还原酶(序列idno15)。jtmo与来自escovopsisweberi(kos22754.1)的预测蛋白(6-羟基-d-烟碱氧化酶)(序列idno16)具有最高的同源性。蛋白同一性:271/481(56.3%);蛋白相似性:344/481(71.5%);空位:39/481(8.1%)。使用embossneedle比对序列。
图18.通过来自lptg-3(nea12)和lptg-4(e1)的jtmo的预测氨基酸序列和ncbi数据库中前6个blastp命中的最大似然分析产生的自助共有树。使用具有默认参数的clustalw进行完整预测的蛋白序列的多重比对。为了构建树形拓扑,使用最大似然(ml),如使用默认参数和500次自助重复、在mega6中执行的。来自500次自助重复的自助值大于70%的分支在各分支邻近标记。每个树图中提供了每个蛋白序列的genbank登录号。jtmo表现出与fad结合氧化还原酶的氨基酸序列相似性:kos22754.1[56%;escovopsisweberi];agz20488.1[52%;微紫青霉菌];ado29935.1[49%;p.paxilli];bau61564.1[43%;简单青霉];agz20199.1[43%;皮落青霉];eon68203.1[coniosporiumapollinis]。
图19.在正电离模式(esi+)中从0-20min收集的多年生黑麦草-lptg-3联合中观察到的代谢产物的lc-esi-ftms提取离子色谱图。
图20.环氧-janthitrem生物合成的建议途径。建议的方案遵循吲哚-二萜生物合成途径,说明了得到环氧-janthitremi(11,12-环氧janthitremg)及其变体(环氧-janthitremsii-iv)的简约途径。通过lc-ms/ms观察所有代谢产物(图19)。
图21.jtmd的核苷酸序列(序列idno17)。突出显示选择用于产生rnai沉默载体的基因序列:选择用于盒2、3和4的基因序列分别以斜体(序列idno18)显示,下划线(序列idno19)显示和粗体(序列idno20)显示。
图22.基因沉默载体的示意图。为了产生入门克隆,使用bp克隆酶(invitrogen,usa)将基因盒[候选基因序列的反向重复,由147bp间隔子(来自m.grisea的角质酶基因内含子)隔开并含有attb1和attb2位点]克隆到pdonr221载体中。通过修饰在trpcp(构巢曲霉(aspergillusnidulans)色氨酸生物合成启动子)和trpct(构巢曲霉色氨酸生物合成终止子)控制下的含有hph基因(选择性标记物)的ppzp200的t-dna区域和在gpdp(构巢曲霉甘油醛-3-磷酸脱氢酶启动子)和trpct(构巢曲霉色氨酸生物合成终止子)的控制下的第一阅读框a[rfa-a]盒(gateway),构建具有gatewaytm功能的目的载体(pend0002)。最终的rna沉默载体由入门载体和pend002载体之间的lr克隆酶反应产生。
图23.真菌原生质体再生。a.没有潮霉素选择的真菌原生质体的再生,原生质体活力的评估。b.用潮霉素选择的rna沉默载体转化的真菌原生质体的再生(箭头表示单个菌落)。c.通过潮霉素选择回收携带rna沉默载体的e1菌株。
优选实施方案的详细说明
鉴定lptg-3植物内生菌菌株nea12中用于janthitrem生物合成的基因
全基因组序列分析用于鉴定nea12基因组中用于janthitrem生物合成的候选基因。来自标准植物内生菌(se)菌株的蛋白序列ltme和ltmj用作查询序列以检索源自nea12基因组的预测蛋白数据库。使用这种方法,blastp检索在预测的nea12蛋白的文库中产生了13个推定的ltme蛋白同源物和26个推定的ltmj蛋白同源物。
预计nea12基因组具有与se菌株一样的预测的ltme和ltmj蛋白同源物。然而,用于janthitrem产生的候选物将是lptg-3和lptg-4基因组所独有的。由于se不产生janthitrem,进行了进一步的分析以将候选物的数量减少至仅存在于lptg-3和lptg-4植物内生菌中的那些。将13个推定的ltme蛋白同源物和26个推定的ltmj蛋白同源物中的每一个用作预测的se蛋白数据库的blastx查询。在该分析中鉴定出单个ltmenea12同源物(g30.t1)(表1),因此是用于进一步研究的最佳可能候选物。预测的基因g30.t1的蛋白序列与来自微紫青霉菌(jand;49%)和p.paxilli(paxd;46%)的芳香族异戊烯基转移酶具有同源性(表2)。这些基因分别与吲哚二萜shearininek和蕈青霉素的合成有关。因此,基因g30.t1此后称为jtmd。
鉴定lptg-3基因组中的janthitrem生物合成基因簇
使用pacbiosequel测序平台(pacbio)对nea12基因组进行测序。使用jtmd基因序列作为查询鉴定含有推定的lptg-3janthitrem生物合成基因簇的重叠群。然后使用已知的ltm基因的基因序列(young等人,2005,2006)和jtmd,使用augustus(stankeandmorgenstern,2005)基因预测和手动注释的组合注释含有jtmd的nea12pacbio重叠群3(247475kb)的基因含量(表2)。
nea12pacbio重叠群3含有13个预测和已知基因(图4)。簇1(ltmg、ltms、ltmm、ltmk)和簇2(ltmp、ltmq、ltmf、ltmc、ltmb)分别位于c.55243-67332bp和c.6838bp-16951bp(表2)。与
pks假基因定义了与lptg-3和
将来自4个分类群的
位于pacbio重叠群3内的基因的转录物表达
通过将由两个多年生黑麦草-植物内生菌转录组研究产生的读数与nea12pacbio重叠群3比对,使用多年生黑麦草-植物内生菌联合转录组数据的rna-seq分析来确定lptg-3(nea12)、lptg-4(e1)和
除了先前定义的簇1和簇2基因之外,还表达了被提出参与janthitrem生物合成的基因pp01、pp02、jtmd和jtmo。由于簇3和簇4基因不存在于
nea12pacbio重叠群3上四种基因簇的详细描述
簇1(ltm1)和簇2(ltm2)
簇3基因
簇3(116033bp-119536bp)含有2种基因,即预测的基因pp01(预测的蛋白1)——推定的细胞色素p450单加氧酶,和pp02——预测是膜结合的o-酰基转移酶(mboat)蛋白(表2)。
pp01
pp01基因的核苷酸序列示于图7中。pp01显示与来自明尼苏达被毛孢——大豆胞囊线虫(大豆异皮线虫(heteroderaglycines))的内寄生真菌——的推定的细胞色素p450单加氧酶的同源性(图8;kjz77225.1)。pp01可能在janthitrem生物合成中起作用,然而,pp01在迄今为止表征的任何其他吲哚-二萜基因簇中不具有同源物。例如,pp01与先前描述的参与吲哚-二萜生物合成的细胞色素p450单加氧酶(例如ltmp、ltmq/paxq/atmq、ltmk)不具有序列相似性(图9)。来自e1(lptg-4)的pp01的预测蛋白序列与nea12(lptg-3)的pp01的预测蛋白序列具有1个氨基酸差异(在氨基酸42d>g处)。
pp02
pp02基因的核苷酸序列示于图10中。预测pp02是膜结合的o-酰基转移酶(mboat)蛋白(图11;图12)。来自e1(lptg-4)的pp02的预测蛋白序列与nea12(lptg-3)的pp02的预测蛋白序列是相同的。虽然膜相关,但是pp02不是基于tmhmm预测分析的跨膜蛋白。
簇4
簇4(150720bp-175051bp)含有2种基因,即芳香族异戊二烯基转移酶jtmd,以及预测编码fad结合氧化还原酶的jtmo。
jtmd
jtmd基因的核苷酸序列示于图13中。预测为芳香族异戊二烯基转移酶的jtmd与来自偏侧蛇虫草菌的预测蛋白具有最高的同源性(63%;图14)。jtmd的预测蛋白序列也与芳香族异戊二烯基转移酶具有同源性,例如来自微紫青霉菌(jand;49%)和p.paxilli(paxd;46%)的那些芳香族异戊二烯基转移酶(图15;nicholson等人,2015)。这些基因分别与吲哚二萜shearininek和蕈青霉素的合成有关。来自nea12(lptg-3)的jtmd的预测蛋白序列与e1(lptg-4)的jtmd的预测蛋白序列相同。
jtmo
jtmo基因的核苷酸序列示于图16中。jtmo与来自escovopsisweberi的预测蛋白(6-羟基-d-烟碱氧化酶)具有最高的同源性(59%;图17)。jtmo也与jano具有同源性,所述jano预测为fad结合氧化还原酶,与微紫青霉菌中的shearinine合成相关(52%;nicholson等人,2015)。已经鉴定了其他吲哚-二萜基因簇中具有类似预测功能的基因(图18)。jtmo蛋白产物可能在随后的吲哚-二萜核心的修饰中起作用。nea12(lptg-3)和e1(lptg-4)中jtmo的预测蛋白序列具有97.9%的同一性。与nea12的jtmo预测蛋白相比,e1jtmo预测蛋白具有9个氨基酸缺失(aa12-20)和一个氨基酸改变(氨基酸326处的t>a)。
jtmo
jtmo基因的核苷酸序列示于图16中。jtmo与来自escovopsisweberi的预测蛋白(6-羟基-d-烟碱氧化酶)具有最高的同源性(59%;图17)。jtmo也与jano具有同源性,所述jano预测为fad结合氧化还原酶,与微紫青霉菌中的shearinine合成相关(52%;nicholson等人,2015)。已经鉴定了其他吲哚-二萜基因簇中具有类似预测功能的基因(图18)。jtmo蛋白产物可能在随后的吲哚-二萜核心的修饰中起作用。nea12(lptg-3)和e1(lptg-4)中jtmo的预测蛋白序列具有97.9%的同一性。与nea12的jtmo预测蛋白相比,e1jtmo预测蛋白具有9个氨基酸缺失(aa12-20)和一个氨基酸改变(氨基酸326处的t>a)。
以前没有在
提出的用于janthitrem生产的生物合成途径
此处描述的工作为
迄今鉴定的所有吲哚-二萜基因簇都具有用于合成雀稗灵的核心基因组,以及编码多功能细胞色素p450单加氧酶、fad依赖性单加氧酶和异戊二烯基转移酶的一系列其他基因,所述酶催化分子骨架上的各种区域特异性和立体特异性氧化,以产生多种吲哚-二萜产物。
采用稳健的液相色谱-质谱(lc-ms)法来靶向与吲哚-二萜生物碱的生物合成相关的关键代谢产物。在植物中从多年生黑麦草-lptg-3联合体中分离的这些代谢产物的提取离子色谱图示于图19中。观察到的精确质量和裂解谱(通过lc-ms/ms分析)如表3所示。
虽然申请人不希望受到理论的限制,但基于这些代谢产物的鉴定和片段化,我们已经提出了环氧-janthitrem的生物合成的框架(图20)。在此处,我们提出janthitrem生物合成可能源自通过ltmp和ltmq的雀稗灵到β-paxitriol的合成。jtmd和jtmo是janthitrem的初始生物合成所必需的,其次是pp01和pp02。ltmf和ltmk是环氧-janthitremii和iv的合成所必需的。
环氧-janthitremi生物合成所需的候选基因的功能分析
jtmd基因的rnai沉默
载体构建
选择jtmd内的三个候选基因序列(95bp、129bp和432bp)用于设计rnai沉默载体(图21)。为了产生入门克隆,将基因盒克隆到pdonr221载体中。通过lr克隆酶反应在入门克隆和具有gatewaytm功能的目的载体(pend0002)之间产生rna沉默载体(图22)(hettiarachchige,2014)。
真菌原生质体的分离
在无菌条件下,通过经由漏斗内衬的miracloth层过滤来收集菌丝体,并用30ml无菌ddh2o洗涤3次。用10mlom缓冲液(1.2mmgso4·7h2o、10mmna2hpo4、100mmnah2po4·2h2o,ph5.8)洗涤菌丝体,并转移至无菌的250ml塑料容器中。加入新鲜制备的10mg/ml在om中的glucanex(30ml)(sigmaaldrich),并在30℃下温和振荡(80-100rpm)孵育18小时。在显微镜下检查glucanex/原生质体溶液(30-50μl)以确认成功消化。将原生质体在漏斗中通过miracloth过滤到15ml无菌玻璃离心管(gentaur,比利时)中并置于冰上。用2mlst缓冲液(0.6m山梨糖醇、100mmtris-hcl,ph8.0)小心地覆盖每个管并离心(beckmancoulter,
peg介导的真菌原生质体转化
在递送至真菌原生质体之前,通过限制酶消化和sanger测序验证三种rna沉默载体(图22)(数据未显示)。根据制造商的说明,使用pureyieldtmplasmidmidiprepsystem(promega)生产适合转化成真菌原生质体的高质量质粒dna。在冰上制备稀释的原生质体(1.25×108原生质体/ml)的等分试样(80μl)。向每个等分试样中加入2μl50mm亚精胺、5μl5mg/ml肝素(在stc缓冲液中制备的)、10μg质粒dna(1μg/μl,不超过20μl)和20μl70%(w/v)peg溶液[70%(w/v)peg4000、10mmtris-hclph8.0、10mmcacl2]。轻轻混合eppendorf管并在冰上孵育30min。加入1.5mlstc缓冲液后,将原生质体混合并离心(eppendorf,centrifuge5424r)(在4℃下5000rpm,持续5min)。除去上清液,将原生质体重新悬浮于再生培养基ii(rgii,500μl)(304g/l蔗糖、1g/lkh2po4、1g/lnh4no3、1g/lnacl、0.25g/l无水mgso4、0.13g/lcacl2·2h2o、1g/l酵母提取物、12g/l脱水马铃薯葡萄糖、1g/l蛋白胨、1g/l酪蛋白酸水解产物)中并孵育过夜(22℃,黑暗,45rpm)。
真菌原生质体再生
将过夜原生质体溶液(200μl)与800μl40%(w/v)peg溶液[40%(w/v)peg4000、1m山梨糖醇、50mmtris-hclph8.0、50mmcacl2]在室温下孵育15min。将含有100μl原生质体/peg混合物的熔融的(50℃)0.4%rgii(5ml)(304g/l蔗糖、1g/lkh2po4、1g/lnh4no3、1g/lnacl、0.25g/l无水mgso4、0.13g/lcacl2·2h2o、1g/l酵母提取物、12g/l脱水马铃薯葡萄糖肉汤、1g/l蛋白胨、1g/l酪蛋白酸水解物、4g/l琼脂糖)在含有100μg/ml潮霉素b的0.6%rgii琼脂糖培养皿(304g/l蔗糖、1g/lkh2po4、1g/lnh4no3、1g/lnacl、0.25g/l无水mgso4、0.13g/lcacl2·2h2o、1g/l酵母提取物、12g/l脱水马铃薯葡萄糖肉汤、1g/l蛋白胨、1g/l酪蛋白酸水解物、6g/l琼脂糖)上涂抹均匀。将代表性的rgii培养皿保持没有潮霉素覆盖,作为对照以评估植物内生菌活力。将所有培养皿在22℃下在黑暗中孵育4-6周直至观察到再生(图23)。
转化的真菌原生质体的鉴定
将单个再生的菌落转移到含有15%(w/v)马铃薯葡萄糖琼脂(pda)和100μg/ml潮霉素选择的培养皿上并孵育(22℃,黑暗,10-21天)。使潮霉素抗性菌落在250ml无菌培养容器中的含有100μg/ml潮霉素的pd肉汤(50ml)中生长(22℃,黑暗,150rpm,10-21天),并且在无菌条件下通过经由漏斗内衬的miracloth层过滤收集菌丝体,用30ml无菌m9磷酸盐缓冲液(1g/lnh4cl、11g/lna2hpo4·7h2o、3g/lkh2po4、5g/lnacl)洗涤。将洗过的菌丝体转移到eppendorf管中,冻干(24-48小时),并使用dneasy植物微型试剂盒(qiagen,德国)根据制造商的说明提取dna。通过聚合酶链反应(pcr)鉴定转化的个体中的潮霉素基因(hph;fwd5’-tgtcgtccatcacagtttgc-3’(序列idno21)、rev5’-gcgccgatggtttctacaaa-3’(序列idno22),和/或由rna沉默载体携带的候选jtmd基因片段[jtmd(95bp)fwd5’-gcctttcttcttgcctgtca-3’(序列idno23)、rev5’-gaccgcctgtgtgttttgaa-3’(序列idno24);jtmd(129bp)fwd5′-cacacagcccaagattgcat-3(序列idno25)′、rev5′-tggaagtctatcgccactgg-3′(序列idno26)、jtmd(432bp)fwd5′-ggagttcagtgcatgctcag-3′(序列idno27)、rev5′-ggcaagaagaaaggctcacc-3′(序列idno28)]。pcr组分和循环条件使用cfxconnecttm实时pcr检测系统(biorad)[2xfaststartsybrgreenmastermix(roche)、10μm正向和反向引物、2μl模板dna、无菌ddh2o(vt10μl);95℃10分钟,(95℃30秒,60℃60秒,72℃30秒)×40,60-95℃(0.5℃inc.)5分钟]。该测定包括适当的阳性和阴性对照dna。
最后,应该理解,在不脱离本文概述的本发明的精神的情况下,可以进行各种改变、修饰和/或添加。
参考文献
babu,j.v.(2009)bioactivechemicalsofimportanceinendophyte-infectedgrasses.phdthesis,universityofwaikato,newzeaiand.
cogan,n.o.i.,shinozuka,h.,sawbridge,t.i.,spangenberg,g.c.,forster,j.w.(2012)developmentofatranscriptomeatlasforperennialryegrass(loliumperennel.).in‘abstracts7thinternationalsympowiumonmolecularbreedingofforageandturf’.july2012,saltlakecity,ut,usa.p.25.
gallagher,r.t.,latch,g.c.,keogh,r.g.(1980)thejanthitrems:fluorescenttremorgenictoxinsproducedbypenicilliumjanthinellumisolatesfromryegrasspastures.appliedandenvironmentalmicrobiology39:272-273.
hennessy,l.(2015).epoxy-janthitrems,effects0ftemperatureoninplantaexpressionandtheirbioactivityagainstporinalarvae.mscthesis.universityofwaikato,newzealand.
nicholson,m.j.,eaton,c.j.,
saikia,s.,nicholson,m.j.,young,c.,parker,e.j.,scott,b.(2008)thegeneticbasisforindole-diterpenechemicaldiversityinfilamentousfungi.mycologicalresearch112(2):184-199.
sawbridge,t.i.(2016)genomicandtranscriptomicanalysisofperennialryegruss/
stanke,m.andmorgenstern,b.(2005)augustus:awebserverforgenepredictionineukaryotesthatallowsuser-definedconstraints.nucleicacidsres.33:465-467.
young,c.a.,felitti,s.,shields,k.,spangenberg,g.,johnson,r.d.,bryan,g.t.,saikia,s.,scott,b.(2006)acomplexgenecluwterforindole-diterpenebiosynthesisinthegrassendophyteneotyphodiumlolii.fungalgeneticandbiology43:679-693.
young,c.a.,bryant,m.k.,christensen,m.j.,tapper,b.a.,bryan,g.t.,scott,b.(2005)molecularcloningandgeneticanalysisofasymbiosis-expressedgeneclusterforlolitrembiosynthesisfromamutualisticendophyteofperennialryegrass.moleculargeneticsandgenomics274(1):13-29.
序列表
<110>维多利亚农业服务控股公司
<120>植物内生菌中的代谢产物产生
<130>pc.dpi.0110
<150>2016903172
<151>2016-08-12
<160>28
<170>patentinversion3.5
<210>1
<211>1484
<212>dna
<213>lptg-3
<400>1
atgccgtccattccgagtctcgcatcgggcctccaggcatggctcatcagtggcgtcgtt60
ctcttgttggcggcggcggcagtgcttgtcggacagatggcggcgtcgcggccgcgcctt120
gacgaccgagcgcctcgtctcctgaagggggcgccgattctcggctgcctcgacttcttc180
cgctgccgaagcgaattcctgctcaaggggagggaccgggaccccagccggcagtttagc240
ttcttctacggaccctatcccattgtcgcattgtctggctctgcggcccggtcctttttc300
tacactgcgcgcggcctcgactttatcccagggtatatcttcaatttaatgctcgtcaaa360
acgggccgtgggcgagctgacacgctgtgaaatgctagctttctggccctcgttgccgca420
ggcccgagcatcgagcagctcttacccggcggcgactttcggaccctcttcgtctcgtct480
ttcaagcatttcatgcacaaaaagcagctcgccgccaacctcggttacctgacgaccgac540
gcagacgtggcccttggcgccatcgacacatcgttgcccgttgagccctttaagctgatg600
ctacacctcatctaccagctcagccaccgcgtcctggggaggcacgacatttccgacgac660
cccaagctgcttgccgacaccgtgtctgcgtttggactcctcgacgactcgtcggctctc720
gaggtcatgttcccccgtgtcccctggcccagcaaggtccgcaagatgctcgccggtgcc780
aagttgcaccgcgtgctctccaaaatcacgagcgaccgccgcaagactcgcagaaccaag840
agggatgccatgcagaccttgatggatcaaggccacccggatactatcgtgtctgccgta900
cgccctggtccccccgtctcccgctcttatacctcgacacttataccttgcgtacatgta960
gctcatcatcggcgccctcagtgcaggacttgccaatagcgccttcagtgccgcctggat1020
ccttgcctacctttccgtcaaccgcgagtggtacgcccggatacgagccgaagtcgacgc1080
tgctgttgccaagcaccgccgctcgcgggtggagtcggcgcccgaagtgctcttgcgcct1140
gtccatgggcgagtgggagtccgagttcccaatgatctgcacggccctacgcgagacgat1200
ccgtatgattctgcagctcacgtcaatacgtaaaaatattagcggcaaggatattcagat1260
tgccgaaactggcatcatcgcactttgtacttcgtacaaaaataaattaaatataaatat1320
gtgaactaattcaataaatttctttattgatcggactatatcttgatctttttaacttaa1380
ctatttttaagaattggggggaaggaaagttagctacttagtctttctttcttctttcct1440
taagtttcttaatactatattaacaccctattatagctacctag1484
<210>2
<211>1164
<212>dna
<213>lptg-3
<400>2
atgccgtccattccgagtctcgcatcgggcctccaggcatggctcatcagtggcgtcgtt60
ctcttgttggcggcggcggcagtgcttgtcggacagatggcggcgtcgcggccgcgcctt120
gacgaccgagcgcctcgtctcctgaagggggcgccgattctcggctgcctcgacttcttc180
cgctgccgaagcgaattcctgctcaaggggagggaccgggaccccagccggcagtttagc240
ttcttctacggaccctatcccattgtcgcattgtctggctctgcggcccggtcctttttc300
tacactgcgcgcggcctcgactttatcccaggctttctggccctcgttgccgcaggcccg360
agcatcgagcagctcttacccggcggcgactttcggaccctcttcgtctcgtctttcaag420
catttcatgcacaaaaagcagctcgccgccaacctcggttacctgacgaccgacgcagac480
gtggcccttggcgccatcgacacatcgttgcccgttgagccctttaagctgatgctacac540
ctcatctaccagctcagccaccgcgtcctggggaggcacgacatttccgacgaccccaag600
ctgcttgccgacaccgtgtctgcgtttggactcctcgacgactcgtcggctctcgaggtc660
atgttcccccgtgtcccctggcccagcaaggtccgcaagatgctcgccggtgccaagttg720
caccgcgtgctctccaaaatcacgagcgaccgccgcaagactcgcagaaccaagagggat780
gccatgcagaccttgatggatcaaggccacccggatactatcgtgtctgccctcatcatc840
ggcgccctcagtgcaggacttgccaatagcgccttcagtgccgcctggatccttgcctac900
ctttccgtcaaccgcgagtggtacgcccggatacgagccgaagtcgacgctgctgttgcc960
aagcaccgccgctcgcgggtggagtcggcgcccgaagtgctcttgcgcctgtccatgggc1020
gagtgggagtccgagttcccaatgatctgcacggccctacgcgagacgatccgtatgatt1080
ctgcagctcacgtcaatacgtaaaaatattagcggcaaggatattcagattgccgaaact1140
ggcatcatcgcactttctacctag1164
<210>3
<211>387
<212>prt
<213>lptg-3
<400>3
metproserileproserleualaserglyleuglnalatrpleuile
151015
serglyvalvalleuleuleualaalaalaalavalleuvalglygln
202530
metalaalaserargproargleuaspaspargalaproargleuleu
354045
lysglyalaproileleuglycysleuaspphepheargcysargser
505560
glupheleuleulysglyargaspargaspproserargglnpheser
65707580
phephetyrglyprotyrproilevalalaleuserglyseralaala
859095
argserphephetyrthralaargglyleuasppheileproglyphe
100105110
leualaleuvalalaalaglyproserilegluglnleuleuprogly
115120125
glyasppheargthrleuphevalserserphelyshisphemethis
130135140
lyslysglnleualaalaasnleuglytyrleuthrthraspalaasp
145150155160
valalaleuglyalaileaspthrserleuprovalgluprophelys
165170175
leumetleuhisleuiletyrglnleuserhisargvalleuglyarg
180185190
hisaspileseraspaspprolysleuleualaaspthrvalserala
195200205
pheglyleuleuaspaspserseralaleugluvalmetpheproarg
210215220
valprotrpproserlysvalarglysmetleualaglyalalysleu
225230235240
hisargvalleuserlysilethrseraspargarglysthrargarg
245250255
thrlysargaspalametglnthrleumetaspglnglyhisproasp
260265270
thrilevalseralaleuileileglyalaleuseralaglyleuala
275280285
asnseralapheseralaalatrpileleualatyrleuservalasn
290295300
argglutrptyralaargileargalagluvalaspalaalavalala
305310315320
lyshisargargserargvalgluseralaprogluvalleuleuarg
325330335
leusermetglyglutrpgluserglupheprometilecysthrala
340345350
leuarggluthrileargmetileleuglnleuthrserilearglys
355360365
asnileserglylysaspileglnilealagluthrglyileileala
370375380
leuserthr
385
<210>4
<211>377
<212>prt
<213>明尼苏达被毛孢(hirsutellaminnesotensis)
<400>4
serleuileproglyproglualatrpproalaservalleumetleu
151015
leuleuthrglyalaalaalailevalileglnmetvalalaserarg
202530
proserpheproserglyalaproargleuleulysglythrproile
354045
leuglycysleuaspphepheargserargserglupheleuleulys
505560
glyargaspasnaspproserargglnpheserphetyrtyrglypro
65707580
hisproilevalvalvalserglyserseralaargserphephetyr
859095
asnalaargglyleuaspleuglnalaglypheserthrleupheala
100105110
alaglyproserleuasphisleuhisthrglyaspileargthrile
115120125
pheilethrserphelyshisleumethislysaspargleuglnala
130135140
asnleuhishisleuvalasnaspalaaspvalalaleuglyglyleu
145150155160
aspvalserargprovalglupropheargvalmetleuhisleuile
165170175
tyrglnleuthrhisargthrleuglyserasnaspilealagluasn
180185190
prolysleuleualalysthrleugluserpheglyargleuaspasp
195200205
serseralametgluilemetpheprotrpvalprotrpproserlys
210215220
metlyslysmetvalalaglyalalysleuhisargthrpheserasn
225230235240
ilemetasnaspargargargthrglyargvalgluproaspalamet
245250255
glnthrleumetaspglnglyhisglnaspleuileileserilephe
260265270
ileileglyalaleuphealaglyleuileasnseralapheserala
275280285
alatrpileleualatyrleuthrasnasnproglutrptyralaarg
290295300
ileargasngluileaspalaalavalalalyshisargtyrserglu
305310315320
glngluseralaprolysvalleualaargleusermetaspasptrp
325330335
gluthrglupheprometvalaspleualaleuarggluthrilearg
340345350
valileleuglnglysersermetarglysasnvalserglyglnasp
355360365
ileproileglyaspthrglyglnile
370375
<210>5
<211>1844
<212>dna
<213>lptg-3
<400>5
cgagcatcaagccgcgtgccgagggaatgtcggacattggccgaaggcgtacatggaccg60
tatacatcaagccgcaccactgactgcagcctagaaacagatgggcatagttggatatgt120
tgcttcgttattacgtaccgccaatatcctcgatgtgcccctggtgttgctgctctgcag180
actcgctacggacctcggctggaccgcacccatgctataagccccatcaaggccgatccg240
gttctgcttcatatcctcttgcattaatcccgaagcccgacctttcgtgactgtgctctg300
gcggaacacagatttccggtttgcctactcagcggcaacagttggtaccccgtgacgatc360
cttggagcttgctgccgggataatcatggcatcaacccgggttcttgcgctcccaatgtt420
ccccatcttgatgactatttctctttacctcggatactacactcggcggcccattcgcct480
tgtctattgcggcataatattatgcgcctttcttagtgtcgtgcgctgcgcaccacagga540
cgaggtagggagtgattatgccttcgggatgagtacatacatatccaagcccctaccgtg600
ctgaagagtgttgacgcgtcgagtaaaatagtcggttgcgcgtgcctttgcaaggcattc660
cagatgctcgtcatcgagagagacatctacgccgactattacgagttggatgacaaggaa720
aagacgcccgtcagatacaggagcctatcgagttgggggaaatgggagtggtgccttgcg780
cactgcttctctgctcgagggattggctttagctgggccatccctcaccttcccgaggcc840
atgcccagcaacacgacgattcgggactacctgcgggcgagcgctcttaatctcggctgg900
ctctaccttgtccaggatctcggacgctcgctgctatctgcggacttgtttgctcacgag960
ggcgtcggagccagcgatacaaagggcggcgcccgttttctgaccgtctattcacttgga1020
atcggggccttgttgaacatcgacatgccataccgtgctgtatgcgccatgggcatggcc1080
agcgggtgcttctggaccaggccgcaccacaaccggcctgcagtcggccgctggcgggac1140
gcatggacgctgcgacgcttctggggccgcgtctggcaccagacgttccgcaaggtacgt1200
acatatgcctgcagcggccggccggtgaggagtgctaacgtctgtcccagccctggcagt1260
cgatcggtcaatggattgcctgggaagtcatgcgcgccctcaaaggctcccttgtctcga1320
gatacgtccaggtctacacttcttttctgctatcggccttgatgcacgttgccgctgcgc1380
ggatggccgatccgcaccgacgctcctgcgccgggacttggatcttctttctcatgcagg1440
ccaacggcatcgtggccgaggatgttgtgcagtgggctggtaagaagacggggatgcggg1500
agtcgtccagcctgacccgtttcctgggccgggcttgggtgctttgctggtttgcatgga1560
cggcgccgtggttctttggggatatcgccgacgttggcctgatccgcctcgagacgttcc1620
ctctgtcggtgactcggggtctgtggaatcggcagtggaagatgtgagacggtgtggaaa1680
gtgatgatgatgtattcagctatctaggctatccacacatgtggcacaacgagggcatag1740
gtattatcgtgcgctaggcacgtgacttggaaaagatatcccctcgcaggatgataaagg1800
tagaaaaaaggattgaattaaagctatcttctattatataaata1844
<210>6
<211>948
<212>dna
<213>lptg-3
<400>6
atgctcgtcatcgagagagacatctacgccgactattacgagttggatgacaaggaaaag60
acgcccgtcagatacaggagcctatcgagttgggggaaatgggagtggtgccttgcgcac120
tgcttctctgctcgagggattggctttagctgggccatccctcaccttcccgaggccatg180
cccagcaacacgacgattcgggactacctgcgggcgagcgctcttaatctcggctggctc240
taccttgtccaggatctcggacgctcgctgctatctgcggacttgtttgctcacgagggc300
gtcggagccagcgatacaaagggcggcgcccgttttctgaccgtctattcacttggaatc360
ggggccttgttgaacatcgacatgccataccgtgctgtatgcgccatgggcatggccagc420
gggtgcttctggaccaggccgcaccacaaccggcctgcagtcggccgctggcgggacgca480
tggacgctgcgacgcttctggggccgcgtctggcaccagacgttccgcaagccctggcag540
tcgatcggtcaatggattgcctgggaagtcatgcgcgccctcaaaggctcccttgtctcg600
agatacgtccaggtctacacttcttttctgctatcggccttgatgcacgttgccgctgcg660
cggatggccgatccgcaccgacgctcctgcgccgggacttggatcttctttctcatgcag720
gccaacggcatcgtggccgaggatgttgtgcagtgggctggtaagaagacggggatgcgg780
gagtcgtccagcctgacccgtttcctgggccgggcttgggtgctttgctggtttgcatgg840
acggcgccgtggttctttggggatatcgccgacgttggcctgatccgcctcgagacgttc900
cctctgtcggtgactcggggtctgtggaatcggcagtggaagatgtga948
<210>7
<211>315
<212>prt
<213>lptg-3
<400>7
metleuvalilegluargaspiletyralaasptyrtyrgluleuasp
151015
asplysglulysthrprovalargtyrargserleusersertrpgly
202530
lystrpglutrpcysleualahiscyspheseralaargglyilegly
354045
phesertrpalaileprohisleuproglualametproserasnthr
505560
thrileargasptyrleuargalaseralaleuasnleuglytrpleu
65707580
tyrleuvalglnaspleuglyargserleuleuseralaaspleuphe
859095
alahisgluglyvalglyalaseraspthrlysglyglyalaargphe
100105110
leuthrvaltyrserleuglyileglyalaleuleuasnileaspmet
115120125
protyrargalavalcysalametglymetalaserglycysphetrp
130135140
thrargprohishisasnargproalavalglyargtrpargaspala
145150155160
trpthrleuargargphetrpglyargvaltrphisglnthrphearg
165170175
lysprotrpglnserileglyglntrpilealatrpgluvalmetarg
180185190
alaleulysglyserleuvalserargtyrvalglnvaltyrthrser
195200205
pheleuleuseralaleumethisvalalaalaalaargmetalaasp
210215220
prohisargargsercysalaglythrtrpilephepheleumetgln
225230235240
alaasnglyilevalalagluaspvalvalglntrpalaglylyslys
245250255
thrglymetarggluserserserleuthrargpheleuglyargala
260265270
trpvalleucystrpphealatrpthralaprotrpphepheglyasp
275280285
ilealaaspvalglyleuileargleugluthrpheproleuserval
290295300
thrargglyleutrpasnargglntrplysmet
305310315
<210>8
<211>391
<212>prt
<213>大孢树粉孢菌(oidiodendronmaius)
<400>8
metgluargalaasnphethrleuleuvaltyrleuleuvalproval
151015
asnleupheilealaserglnthrprolysargpheargpheleutyr
202530
alavalpheglnleuglyleutyrphealailevalleuleuvalpro
354045
proglyservalproserserasptyrthrpheglythrthrphetyr
505560
servalleualaalapheasnphephephephecysaspprotyrglu
65707580
gluhistrpglnilealaproglugluasplysileaspglnglyarg
859095
aspargserargglnserserprovallystyrlysaspleuaspleu
100105110
argalaservalleutrpcysileserasnalaphealaleuarggly
115120125
ileglytrpasntrpargileprohisleuproproglyprothrarg
130135140
glyileserargvalprotyrleuileaspvalglythrthrleuleu
145150155160
lysleutyrleuleuhisasppheseralathrleuleuglulysval
165170175
thrleuglyglyglnleuproleugluasnileargleuaspleuarg
180185190
thrvalalavalvalserphealavalserserilethrleuileglu
195200205
pheglytyrglnileilecysphealaglyalaalathrglyleuphe
210215220
trpthrargpheglnaspasnhisprovalileglyservaltyrglu
225230235240
glytyrthrileglyargphetrpglyargvaltrphisglnasnmet
245250255
argargalaproglylystyrleualaglnlysvalleuhisvallys
260265270
argglyglyleuvalserargtyrvalglnsertyrthralaphephe
275280285
leuserglyvaltyrhistyrileglyalalysserserleuprohis
290295300
gluglnleuasnargthrcystrpphepheleuleuglnproasnleu
305310315320
metleuilegluaspphealaleutrppheglylysglulysleugly
325330335
leulysserproargtrpthrcysleuglytyrvaltrpthrpheval
340345350
metleuthrvalthralaalaglyphevalaspaspcysilearghis
355360365
glnleuvalproprothrseralapheserpheserleualaalaleu
370375380
leuileglnlystrpgluleu
385390
<210>9
<211>1672
<212>dna
<213>lptg-3
<400>9
ccgccaccgacataacgagccgttgtctataagagtccaatcgtgccaggacgcaacgtc60
caagtagccctgattcttgctcaacccgacgtggctcgctttactggcttagagacctca120
tctgacggccagtctctgattcgaaacccatttgcacgaccgaccaagatgggcacctgt180
tccactcgcgtaggcgagacgccatcgaaaccagcagatgtgacgccgcctgagccatgg240
caggccctagctcagggtctagggttcgccaatgagaacgagaggtactggtggtccaaa300
cttgcccccctggctggcaagatgatgaagtgggggcagtactcgacgccggagcagtac360
agagtcctggcattcatacacgcgtatattgtccctagctgtggcccaaggcctggggat420
ggcggtgatctgttctggaaggtgtttctcaactacgattgcacccctatccagctcagt480
ctcaactaccacgatgggaagatgacgcttcggacagctcatatacctatcagcaatatc540
tccggcacagcagaagaccccattaaccagaaggctgcgatagatgccatggtccgccag600
caacaggtcctgccgtcccaggacatgcgctggtttaaccactttgtatctaagctcttc660
ctggatcgagatacggcggccaccctcaaggccaaggtggacgagttccagatccggcag720
ggagttcagtgcatgctcagccacgactttcccgacaatcacatccagtgtaagctagcc780
tttgcctcccactggaagtctatcgccactggccttgataaggaggaagttatctgggat840
gcaatcttggggttaggggacgacgttattccgtataagccagtgctcgctatgctccag900
caatactcaacgtccaaaagtgccgcagctgcaggggcacatccgatctttttcgccatc960
gactcggtgctcaaagacgactatacaagctcacgtatcaagatctattttgttacccac1020
cgaactgcctttaacgtcatggttgacatctataccctaggtggtttgctaatggggcct1080
tgtattgaaaagggcacgcaggccttgaggacactctggaaggccgtgctcaatgttccc1140
gaggggtggccagacgataaggatctacccatcaaccccaatggctgtgcggcagtcatc1200
ttcaactttgaggtccggcctggcgccgagttcccggctcctaagatttacctcccagcc1260
cattactatggccgacccgacttggagattgctgatggaatggaccgctttttcctgagc1320
cagggatgggatggtatttaccctggctataagaaaaactacctcaagtgcttgtctgtt1380
cttgttgcccctggccttctttccctttgcccttggctaacagtaaggtagtatgaactc1440
ggacaaccagctcacggctgtgcaccacgacatttctttttcattcaaggggactaaccc1500
ctatgttaccgtctattataagcctgagttgcatttggaggctgagtaggcggtggacca1560
agcggtaaggttagatggggtgttgattccttaattgcttccctttatttttgttatttt1620
ttgttacgtagcagagttaatggcatctgtcctatgcaggtatttccagggg1672
<210>10
<211>1263
<212>dna
<213>lptg-3
<400>10
atgggcacctgttccactcgcgtaggcgagacgccatcgaaaccagcagatgtgacgccg60
cctgagccatggcaggccctagctcagggtctagggttcgccaatgagaacgagaggtac120
tggtggtccaaacttgcccccctggctggcaagatgatgaagtgggggcagtactcgacg180
ccggagcagtacagagtcctggcattcatacacgcgtatattgtccctagctgtggccca240
aggcctggggatggcggtgatctgttctggaaggtgtttctcaactacgattgcacccct300
atccagctcagtctcaactaccacgatgggaagatgacgcttcggacagctcatatacct360
atcagcaatatctccggcacagcagaagaccccattaaccagaaggctgcgatagatgcc420
atggtccgccagcaacaggtcctgccgtcccaggacatgcgctggtttaaccactttgta480
tctaagctcttcctggatcgagatacggcggccaccctcaaggccaaggtggacgagttc540
cagatccggcagggagttcagtgcatgctcagccacgactttcccgacaatcacatccag600
tgtaagctagcctttgcctcccactggaagtctatcgccactggccttgataaggaggaa660
gttatctgggatgcaatcttggggttaggggacgacgttattccgtataagccagtgctc720
gctatgctccagcaatactcaacgtccaaaagtgccgcagctgcaggggcacatccgatc780
tttttcgccatcgactcggtgctcaaagacgactatacaagctcacgtatcaagatctat840
tttgttacccaccgaactgcctttaacgtcatggttgacatctataccctaggtggtttg900
ctaatggggccttgtattgaaaagggcacgcaggccttgaggacactctggaaggccgtg960
ctcaatgttcccgaggggtggccagacgataaggatctacccatcaaccccaatggctgt1020
gcggcagtcatcttcaactttgaggtccggcctggcgccgagttcccggctcctaagatt1080
tacctcccagcccattactatggccgacccgacttggagattgctgatggaatggaccgc1140
tttttcctgagccagggatgggatggtatttaccctggctataagaaaaactacctcaag1200
tgcttgtctgttcttgttgcccctggccttctttccctttgcccttggctaacagtaagg1260
tag1263
<210>11
<211>420
<212>prt
<213>lptg-3
<400>11
metglythrcysserthrargvalglygluthrproserlysproala
151015
aspvalthrproprogluprotrpglnalaleualaglnglyleugly
202530
phealaasngluasngluargtyrtrptrpserlysleualaproleu
354045
alaglylysmetmetlystrpglyglntyrserthrprogluglntyr
505560
argvalleualapheilehisalatyrilevalprosercysglypro
65707580
argproglyaspglyglyaspleuphetrplysvalpheleuasntyr
859095
aspcysthrproileglnleuserleuasntyrhisaspglylysmet
100105110
thrleuargthralahisileproileserasnileserglythrala
115120125
gluaspproileasnglnlysalaalaileaspalametvalarggln
130135140
glnglnvalleuproserglnaspmetargtrppheasnhispheval
145150155160
serlysleupheleuaspargaspthralaalathrleulysalalys
165170175
valaspglupheglnileargglnglyvalglncysmetleuserhis
180185190
asppheproaspasnhisileglncyslysleualaphealaserhis
195200205
trplysserilealathrglyleuasplysglugluvaliletrpasp
210215220
alaileleuglyleuglyaspaspvalileprotyrlysprovalleu
225230235240
alametleuglnglntyrserthrserlysseralaalaalaalagly
245250255
alahisproilephephealaileaspservalleulysaspasptyr
260265270
thrserserargilelysiletyrphevalthrhisargthralaphe
275280285
asnvalmetvalaspiletyrthrleuglyglyleuleumetglypro
290295300
cysileglulysglythrglnalaleuargthrleutrplysalaval
305310315320
leuasnvalprogluglytrpproaspasplysaspleuproileasn
325330335
proasnglycysalaalavalilepheasnphegluvalargprogly
340345350
alaglupheproalaprolysiletyrleuproalahistyrtyrgly
355360365
argproaspleugluilealaaspglymetaspargphepheleuser
370375380
glnglytrpaspglyiletyrproglytyrlyslysasntyrleulys
385390395400
cysleuservalleuvalalaproglyleuleuserleucysprotrp
405410415
leuthrvalarg
420
<210>12
<211>394
<212>prt
<213>偏侧蛇虫草菌(ophiocordycepsunilateralis)
<400>12
metalaalaserprothrtyrgluasnglythrproserglnprotrp
151015
glnalaleualaglnglyleuglytyrvalasnglnaspgluglntyr
202530
trptrpserlysvalglyproleualaglnargleumetglutrpala
354045
argtyrserthrprogluargtyrargvalleualapheiletyrthr
505560
tyrilevalproalacysglyprolysproaspaspasnglyglnval
65707580
phetrplysthrtyrileasntyraspcysthrproileglnleuser
859095
leuasnphehisasplyslysvalthrpheargthralaasnileser
100105110
serseraspileserglythralalysaspproileasnglnglnala
115120125
alavalaspalametilelysglnlysargvalleuproserglnasn
130135140
metargtrppheasnhisphemetserlysleupheleugluproglu
145150155160
alaalaalaalaleulysalalysalaaspglupheglnileargasn
165170175
glyvalglncysmetleuserhisasppheproasnserglnvalgln
180185190
cyslysalaphephealaproasntrplysalaphealathrglyile
195200205
glumetlysaspvaliletrpaspalailemetalaleuglyaspasp
210215220
ileleuprotyrlysserglyleualaileleuaspargphethrthr
225230235240
seralaseralaalaalaalaglyalavalprovalcysphealaphe
245250255
aspservalleugluglyasptyrlysasnserargilelysiletyr
260265270
tyralathrleuargthralapheaspvalmetvalgluiletyrthr
275280285
leuglyglyleuleuthrglyproglumetglulysglyvalglnala
290295300
leuargmetleutrpasnalavalvalasnileproaspglytrppro
305310315320
aspaspthraspleuproalaasnprohisargphealaalavalleu
325330335
pheasnphegluilearghisglyalagluleuprovalproglnile
340345350
tyrileproalahistyrtyrglyargseraspleugluilealaasp
355360365
glyvalaspargphepheglnserglnglyleuaspalaasptyrpro
370375380
protyrlysgluasntyrilelyscysleu
385390
<210>13
<211>1992
<212>dna
<213>lptg-3
<400>13
tcctgaccatgttggctccgccaacggtgagactcacccttgttctgacacgcataccgc60
gagagtaatatttcatgtacatgaacaaagcgcacggtgcggtatcttgcgtttaattat120
tgctccatattgcaggctgcatatgcattacgggagatggttgcattcaatgccatatga180
tgccgaggagagcggccgatacgcgccccgcctgtgctgtcccggattgaagaaatgccc240
gaggaaatgggatctgttggctataatagcaagggaagtaagcgtgtatcatccccggaa300
ccatcaagcactaccgctttgaatcgcttctcttcttgacagcatgggagaccctcttcc360
cggcaacacgcgagactgtctctcccgcaacatgcgagactcctcaaccgagaagctccc420
cattctgtggcggaccgactcccccctcaaccagtacgatgaagcacggtgcagagtctt480
caacggcaggcggcccgagcatttcccacgcgcaatcgtccaggccacgacgctcgacca540
catcgtagcggctgtgaggctggccgtggagtccgccgcccctgtggccgtccgctcagg600
tggccacagtctctcctgctggaccatgcgccatgatgccatcctcattgatctcaagga660
ctttagctatctaagctacgatgaagaaacacaccaagtccaggcctctcccagtaccct720
aacgggagaattgctcgagtttcttgcccagaagcagcgattctttcccgtaggccactc780
agggggcattggcctaggtggctacctcctccaagctggaatcggactcaactgccgggt840
atgcgtgcttgctctgcctgccccatgcttgcctatccgtttctcttcatccactgacgg900
caacgtctagggctatgggtacgcatgcgagtctgtctctggaatcgacattgttaccgc960
cgatggctgcattaagcactgtgacaaagaagaaaacgctgatttgtattgggccgctcg1020
cggagctgggccgggtgagtccctctctgaaagccttccgcattaaagccgtggcaaatc1080
taactaaacagagttccctgccattgtcacacgcttctacctcgagactcgaccgatgcc1140
ggtttgcaaccggagcacgtacatctggccggcgaccatgtatgaccaggttttcccttg1200
gctcgaccgcgtgagtagctcgtgtccatgtccccagccaagctcacgaggtttcaagct1260
cttaactacgctggacgagaacgtcgaggttggcgtgttcggatttacagtcccccaact1320
caaccagccggggctacacgtgctcgcaacagcattcggagactcggatgaggatacccg1380
gcgaatgctcacacccttcatcgacacccaccccccaggagcgattcacgcccaggactt1440
tgtggcgactgacttcgctagcgactacgttctagataagacagtcctgccgcaaggtgc1500
tcgttacttcaccgatagcgtctttctcaagcctggcaccgacctagtggtggcttgtaa1560
ggacatgtttacaggactaaagcatccgcgcgcattggcatattggcagccgatgaagac1620
cgccactgcccgcacccttcccgacatggccatgagcatacatagcgaccattacgtatc1680
cctactaggaatctacgacgattccgcccaagacgatgagcagacgtcctggatcgtgga1740
ttatatgcgtaagctggagccatttgtcttgggcacgtttgtgggggatgcgcatgtgtt1800
ggaaagaccgtctaattactggtcagaggaggccaaagagcgagtgctccgtgttggaaa1860
gaagtgggatcctagtggaagaattcgggggatgctcctcagtgactcgtaggcagcgat1920
ttatttaaagggcgtgcttatcaggcaaacgcgtacgcgtaattagcacccctaaaggta1980
gctaaggtagct1992
<210>14
<211>1440
<212>dna
<213>lptg-3
<400>14
atgggagaccctcttcccggcaacacgcgagactgtctctcccgcaacatgcgagactcc60
tcaaccgagaagctccccattctgtggcggaccgactcccccctcaaccagtacgatgaa120
gcacggtgcagagtcttcaacggcaggcggcccgagcatttcccacgcgcaatcgtccag180
gccacgacgctcgaccacatcgtagcggctgtgaggctggccgtggagtccgccgcccct240
gtggccgtccgctcaggtggccacagtctctcctgctggaccatgcgccatgatgccatc300
ctcattgatctcaaggactttagctatctaagctacgatgaagaaacacaccaagtccag360
gcctctcccagtaccctaacgggagaattgctcgagtttcttgcccagaagcagcgattc420
tttcccgtaggccactcagggggcattggcctaggtggctacctcctccaagctggaatc480
ggactcaactgccggggctatgggtacgcatgcgagtctgtctctggaatcgacattgtt540
accgccgatggctgcattaagcactgtgacaaagaagaaaacgctgatttgtattgggcc600
gctcgcggagctgggccggagttccctgccattgtcacacgcttctacctcgagactcga660
ccgatgccggtttgcaaccggagcacgtacatctggccggcgaccatgtatgaccaggtt720
ttcccttggctcgaccgcgtgagtagctcgtgtccatgtccccagccaagctcacgaggt780
ttcaagctcttaactacgctggacgagaacgtcgaggttggcgtgttcggatttacagtc840
ccccaactcaaccagccggggctacacgtgctcgcaacagcattcggagactcggatgag900
gatacccggcgaatgctcacacccttcatcgacacccaccccccaggagcgattcacgcc960
caggactttgtggcgactgacttcgctagcgactacgttctagataagacagtcctgccg1020
caaggtgctcgttacttcaccgatagcgtctttctcaagcctggcaccgacctagtggtg1080
gcttgtaaggacatgtttacaggactaaagcatccgcgcgcattggcatattggcagccg1140
atgaagaccgccactgcccgcacccttcccgacatggccatgagcatacatagcgaccat1200
tacgtatccctactaggaatctacgacgattccgcccaagacgatgagcagacgtcctgg1260
atcgtggattatatgcgtaagctggagccatttgtcttgggcacgtttgtgggggatgcg1320
catgtgttggaaagaccgtctaattactggtcagaggaggccaaagagcgagtgctccgt1380
gttggaaagaagtgggatcctagtggaagaattcgggggatgctcctcagtgactcgtag1440
<210>15
<211>479
<212>prt
<213>lptg-3
<400>15
metglyaspproleuproglyasnthrargaspcysleuserargasn
151015
metargaspserserthrglulysleuproileleutrpargthrasp
202530
serproleuasnglntyraspglualaargcysargvalpheasngly
354045
argargprogluhispheproargalailevalglnalathrthrleu
505560
asphisilevalalaalavalargleualavalgluseralaalapro
65707580
valalavalargserglyglyhisserleusercystrpthrmetarg
859095
hisaspalaileleuileaspleulysaspphesertyrleusertyr
100105110
aspglugluthrhisglnvalglnalaserproserthrleuthrgly
115120125
gluleuleuglupheleualaglnlysglnargphepheprovalgly
130135140
hisserglyglyileglyleuglyglytyrleuleuglnalaglyile
145150155160
glyleuasncysargglytyrglytyralacysgluservalsergly
165170175
ileaspilevalthralaaspglycysilelyshiscysasplysglu
180185190
gluasnalaaspleutyrtrpalaalaargglyalaglyprogluphe
195200205
proalailevalthrargphetyrleugluthrargprometproval
210215220
cysasnargserthrtyriletrpproalathrmettyraspglnval
225230235240
pheprotrpleuaspargvalsersersercysprocysproglnpro
245250255
serserargglyphelysleuleuthrthrleuaspgluasnvalglu
260265270
valglyvalpheglyphethrvalproglnleuasnglnproglyleu
275280285
hisvalleualathralapheglyaspseraspgluaspthrargarg
290295300
metleuthrpropheileaspthrhisproproglyalailehisala
305310315320
glnaspphevalalathraspphealaserasptyrvalleuasplys
325330335
thrvalleuproglnglyalaargtyrphethraspservalpheleu
340345350
lysproglythraspleuvalvalalacyslysaspmetphethrgly
355360365
leulyshisproargalaleualatyrtrpglnprometlysthrala
370375380
thralaargthrleuproaspmetalametserilehisserasphis
385390395400
tyrvalserleuleuglyiletyraspaspseralaglnaspaspglu
405410415
glnthrsertrpilevalasptyrmetarglysleuglupropheval
420425430
leuglythrphevalglyaspalahisvalleugluargproserasn
435440445
tyrtrpsergluglualalysgluargvalleuargvalglylyslys
450455460
trpaspproserglyargileargglymetleuleuseraspser
465470475
<210>16
<211>444
<212>prt
<213>escovopsisweberi
<400>16
metalaaspleuproileiletrpargseraspthrgluseralaala
151015
lystyrgluglualaargcysargilepheasnileargargproglu
202530
hispheproargalailevallysalathrthrleugluhisileval
354045
alaalavallysleualaalagluglnglyvalargvalvalalaarg
505560
serglyglyhisglyleuseralatrpthrleuarghisasnalaile
65707580
leuileaspleuglnasnphelyshismetsertyraspgluglulys
859095
asnglualaglnvalserproserthrleualaglugluleuleuasp
100105110
pheleualagluarglysargphepheproalaglyhisthrglyasp
115120125
ileglyleuglyglytyrleuleuglnglyglyileglyleusercys
130135140
argglytyrglytyralacysglutyrvalthrglyvalaspvalval
145150155160
thralagluglyaspvalvalhisalaaspgluasngluasnalaasp
165170175
leutyrtrpalaalaargglyalaglyproglupheproalaileval
180185190
thrargphetyrleulysthrileproleuglnprovalalalysgly
195200205
cysargtyriletrpproalavalmettyraspalailephesertrp
210215220
ileasplysileseralaserleuaspgluhisvalaspproserval
225230235240
pheglyphemetileproglyileasnglnproglyleumetpheser
245250255
alaservalphealaglnthrglugluglualaargarglysleuala
260265270
proleuvalgluthrhisproproglyalametvalalagluaspphe
275280285
valaspserserilethrthrvaltyralaglyserargglnpheasn
290295300
proproglycysargtyrphethraspservalpheleulysprogly
305310315320
thraspvalvalglualacysarghismetphethrglnileprophe
325330335
proargglyleualatyrtrpglnproleuargileserproalaarg
340345350
lysglnproaspmetalaleuserileglnsergluhistyrvalser
355360365
leuleualavaltyraspasnglualagluaspglualaglnthrglu
370375380
trpvalilegluglyilearglysleugluproglnilehisglythr
385390395400
pheilealaaspalahisproglumetargthrserasntyrtrpser
405410415
gluglualathralaargleualaalavalglyserlystrpasppro
420425430
lysglyargilethrglyilevalvalargglnglu
435440
<210>17
<211>1263
<212>dna
<213>lptg-3
<400>17
ctaccttactgttagccaagggcaaagggaaagaaggccaggggcaacaagaacagacaa60
gcacttgaggtagtttttcttatagccagggtaaataccatcccatccctggctcaggaa120
aaagcggtccattccatcagcaatctccaagtcgggtcggccatagtaatgggctgggag180
gtaaatcttaggagccgggaactcggcgccaggccggacctcaaagttgaagatgactgc240
cgcacagccattggggttgatgggtagatccttatcgtctggccacccctcgggaacatt300
gagcacggccttccagagtgtcctcaaggcctgcgtgcccttttcaatacaaggccccat360
tagcaaaccacctagggtatagatgtcaaccatgacgttaaaggcagttcggtgggtaac420
aaaatagatcttgatacgtgagcttgtatagtcgtctttgagcaccgagtcgatggcgaa480
aaagatcggatgtgcccctgcagctgcggcacttttggacgttgagtattgctggagcat540
agcgagcactggcttatacggaataacgtcgtcccctaaccccaagattgcatcccagat600
aacttcctccttatcaaggccagtggcgatagacttccagtgggaggcaaaggctagctt660
acactggatgtgattgtcgggaaagtcgtggctgagcatgcactgaactccctgccggat720
ctggaactcgtccaccttggccttgagggtggccgccgtatctcgatccaggaagagctt780
agatacaaagtggttaaaccagcgcatgtcctgggacggcaggacctgttgctggcggac840
catggcatctatcgcagccttctggttaatggggtcttctgctgtgccggagatattgct900
gataggtatatgagctgtccgaagcgtcatcttcccatcgtggtagttgagactgagctg960
gataggggtgcaatcgtagttgagaaacaccttccagaacagatcaccgccatccccagg1020
ccttgggccacagctagggacaatatacgcgtgtatgaatgccaggactctgtactgctc1080
cggcgtcgagtactgcccccacttcatcatcttgccagccaggggggcaagtttggacca1140
ccagtacctctcgttctcattggcgaaccctagaccctgagctagggcctgccatggctc1200
aggcggcgtcacatctgctggtttcgatggcgtctcgcctacgcgagtggaacaggtgcc1260
cat1263
<210>18
<211>129
<212>dna
<213>lptg-3
<400>18
gcggtccattccatcagcaatctccaagtcgggtcggccatagtaatgggctgggaggta60
aatcttaggagccgggaactcggcgccaggccggacctcaaagttgaagatgactgccgc120
acagccatt129
<210>19
<211>95
<212>dna
<213>lptg-3
<400>19
cccaagattgcatcccagataacttcctccttatcaaggccagtggcgatagacttccag60
tgggaggcaaaggctagcttacactggatgtgatt95
<210>20
<211>561
<212>dna
<213>lptg-3
<400>20
gcggtccattccatcagcaatctccaagtcgggtcggccatagtaatgggctgggaggta60
aatcttaggagccgggaactcggcgccaggccggacctcaaagttgaagatgactgccgc120
acagccattccaagattgcatcccagataacttcctccttatcaaggccagtggcgatag180
acttccagtgggaggcaaaggctagcttacactggatgtgattgtcgggaaagtcgtggc240
tgagcatgcactgaactccctgccggatctggaactcgtccaccttggccttgagggtgg300
ccgccgtatctcgatccaggaagagcttagatacaaagtggttaaaccagcgcatgtcct360
gggacggcaggacctgttgctggcggaccatggcatctatcgcagccttctggttaatgg420
ggtcttctgctgtgccggagatattgctgataggtatatgagctgtccgaagcgtcatct480
tcccatcgtggtagttgagactgagctggataggggtgcaatcgtagttgagaaacacct540
tccagaacagatcaccgccat561
<210>21
<211>20
<212>dna
<213>人工序列
<220>
<223>引物
<400>21
tgtcgtccatcacagtttgc20
<210>22
<211>20
<212>dna
<213>人工序列
<220>
<223>引物
<400>22
gcgccgatggtttctacaaa20
<210>23
<211>20
<212>dna
<213>人工序列
<220>
<223>引物
<400>23
gcctttcttcttgcctgtca20
<210>24
<211>20
<212>dna
<213>人工序列
<220>
<223>引物
<400>24
gaccgcctgtgtgttttgaa20
<210>25
<211>20
<212>dna
<213>人工序列
<220>
<223>引物
<400>25
cacacagcccaagattgcat20
<210>26
<211>20
<212>dna
<213>人工序列
<220>
<223>引物
<400>26
tggaagtctatcgccactgg20
<210>27
<211>20
<212>dna
<213>人工序列
<220>
<223>引物
<400>27
ggagttcagtgcatgctcag20
<210>28
<211>20
<212>dna
<213>人工序列
<220>
<223>引物
<400>28
ggcaagaagaaaggctcacc20
- 还没有人留言评论。精彩留言会获得点赞!