一种具有增加的基因突变特异性的DNA聚合酶活性增强用PCR缓冲液组合物的制作方法
本发明涉及一种具有增加的基因突变特异性的dna聚合酶活性增强用pcr缓冲液组合物及其用途,更具体地说,本发明提供在特定氨基酸位置诱导突变以增加基因突变特异性的dna聚合酶活增强性用pcr缓冲液组合物、包含所述pcr缓冲液组合物及/或具有增加的基因突变特异性的dna聚合酶的用于检测基因突变或snp的pcr试剂盒、及利用所述试剂盒从一个以上的模板中体外(invitro)检测至少一种基因突变或snp的检测方法。
背景技术:
自从第一个人类基因组序列被发现,研究者的研究主要集中探索单核苷酸突变(单核苷酸多态性,snps)等个体间的遗传差异上。基因组中的单核苷酸突变与各种疾病的不同耐药性或易感体质相关,这已经越来越明确,因此成为主要关心对象。未来医学相关核苷酸变异的知识可适用于个体遗传供应的治疗方法,并可防止无效或导致副作用的药物治疗(shi,expertrev.mol.diagn.1,363-365(2001))。在时间和成本上有效率的核苷酸变异鉴定技术的开发将带来药物遗传学的进一步发展。
snps在人类基因组中占据主要的遗传变异,可以诱发个体间差异的90%以上。(kwok,annu.rev.genomicshum,genet.2,235-258(2001);kwokandchen,curr.issuesmol.biol.5,43-60(2003);twymanandprimrose,pharmacogenomics4,67-79(2003))。为了检测这些遗传变异和突变等其他核酸变异,可以使用各种方法。例如,可以通过在适当的杂交条件下将待分析的核酸样品与对于序列变体具有特异性的杂交引物杂交来实现对靶核酸的变体的鉴定(guoetal.,nat.biotechnol.15,331-335(1997))。
但是,发现这种杂交方法,尤其在测定时必需的灵敏度方面无法满足临床需要。因此,pcr已被广泛使用于分子生物学、snp及其他等位基因序列变体等突变检测的诊断检测方法中(saiki等人,science239,487-490(1988)),其中,考虑到变体的存在,在杂交前通过聚合酶链式反应(pcr)扩增待检测的靶核酸。用于这种测定的杂交引物,通常使用单链寡核苷酸。所述测定的修改的具体实例包括使用荧光杂交探针(livak,genet.73-52017-07-12anal.14,143-149(1999))。一般而言,已经尝试使snp和其他序列变异的测定方法自动化(gut,hum.mutat.17,475-492(2001))。
本领域已知的序列变异特异性杂交的对策通过所谓的基因突变特异性扩增来提供。在该检测方法中,已经在扩增过程中,使用变异特异性扩增引物,通常引物的3'-末端具有所谓的差异性末端核苷酸残基,残基仅对待检测靶核酸的一个特异性变异互补。在该方法中,通过pcr扩增后以dna产物的存在或不存在来测量核苷酸变体。基因突变特异性扩增的原理基于在基因突变特异性扩增引物末端形成典型(canonical)或非典型引物-模板复合物。在精确配对的3'引物末端,产生dna聚合酶引起的扩增,而在错配引物末端延伸被抑制。
例如,美国专利第5,595,890号公开了基因突变特异性扩增方法及其应用,如揭示了用于检测k-ras肿瘤基因中的临床相关点突变(pointmutation)的使用方法。另外,美国专利第5,521,301号公开了用于abo血型系统的基因型分析的等位基因-特异性扩增方法。相反,美国专利第5,639,611号公开了与检测导致镰状细胞贫血症的点突变有关的等位基因-特异性扩增的使用方法。然而,基因突变特异性扩增或等位基因特异性扩增具有低选择性的问题,因此需要更复杂的、时间和成本密集型的最优化步骤。
所述用于检测序列变异、多态性和集中于点突变的方法,当待检测的序列变异与相同核酸片段(或相同基因)的显性变异相比不充分时,尤其需要基因-特异性扩增(或基因突变特异性扩增)。
例如,当通过基因突变特异性扩增在体液如血液、血清或血浆中检测散发性肿瘤细胞时发生这种情况(美国专利第5,496,699号)。为此,dna首先从血液、血清或血浆等体液中分离出来,而dna由不充足的散发性肿瘤细胞和过量的非增殖细胞组成。因此,在过量的野生型dna存在的情况下,在k-ras基因中的肿瘤dna重要的突变应该从多个复制中被检测到。
现有技术中公开的所有用于基因突变特异性扩增的方法,尽管使用3'-差异性核苷酸残基,但即使靶核酸不完全与待检测的序列变体相匹配,在合适的dna聚合酶存在条件下,具有发生较低水平的引物延伸的缺点。特别地,当特定的序列变体通过包含另一个序列变体的过量的背景核酸而被检测出来,会导致假阳性结果。已知方法的另一个缺点是需要使用3'-末端差异性寡核苷酸残基。这种基于pcr的方法所存在的缺点,主要是由于用于充分区分错配碱基的方法中使用的聚合酶的不适用性而产生。因此,尚不可能通过pcr直接获得关于是否存在突变的明确信息。迄今为止,需要额外的时间和成本密集型的纯化和测定方法来明确诊断突变。因此,提高基因突变特异性或等位基因-特异性pcr扩增的选择性的新方法,会对通过pcr的直接基因突变或snp分析的可信度和强效性带来很大影响。
因此,需要持续研究具有增加的基因突变特异性的dna聚合酶及可使所述dna聚合酶发挥其功能的、混合多种物质的最佳反应缓冲液。
本发明人致力于开发能够提高基因突变-特异性pcr扩增选择性的新型dna聚合酶及用于提高其活性的反应缓冲液,结果证实对taq聚合酶特定位置处的氨基酸残基诱发突变时,基因突变特异性显著增加,当控制pcr缓冲液组合物中的kcl、(nh4)2so4和/或tmac(四甲基氯化铵,tetramethylammoniumchloride)的浓度时,所述具有增加的基因突变特异性的dna聚合酶的活性增强,从而完成本发明。
技术实现要素:
本发明为了解决所述技术课题而提出,提供一种具有增加的基因突变特异性的dna聚合酶活性增强用pcr缓冲液组合物。
本发明的另一个目的是提供包含所述pcr缓冲液组合物及/或具有增加的基因突变特异性的dna聚合酶的用于检测基因突变或snp的pcr试剂盒。
本发明的另一个目的是提供利用所述pcr试剂盒从一个以上的模板中体外(invitro)检测至少一种基因突变或snp的检测方法。
为了实现所述目的,本发明提供具有增加的基因突变特异性的dna聚合酶活性增强用pcr缓冲液组合物,所述组合物包含25至100mm的kcl;1至15mm的(nh4)2so4;最终ph为8.0至9.0。
根据本发明的一个优选实施例,所述kcl的浓度为60至90mm。
根据本发明的另一个优选实施例,所述(nh4)2so4的浓度为2至8mm。
根据本发明的又一个优选实施例,所述kcl的浓度为70至80mm,所述(nh4)2so4的浓度为4至6mm。
本发明提供具有增加的基因突变特异性的dna聚合酶活性增强用pcr缓冲液组合物,所述pcr缓冲液组合物进一步包含5至80mm的tmac(四甲基氯化铵,tetramethylammoniumchloride)。
根据本发明的一个优选实施例,所述kcl的浓度为40至90mm。
根据本发明的另一个优选实施例,所述(nh4)2so4的浓度为1至7mm。
根据本发明的又一个优选实施例,所述tmac(四甲基氯化铵,tetramethylammoniumchloride)的浓度为15至70mm,所述kcl的浓度为50至80mm,所述(nh4)2so4的浓度为1.5至6mm。
根据本发明的另一个优选实施例,所述pcr缓冲液组合物可以进一步包含tris·cl及mgcl2
本发明提供一种包含所述pcr缓冲液组合物的用于检测基因突变或snp的pcr试剂盒。
根据本发明的优选实施例,所述pcr试剂盒可进一步包含由seqidno:1的氨基酸序列(seqidno:6的碱基序列)组成的taq聚合酶的dna聚合酶,所述dna聚合酶可包含(a)seqidno:1的氨基酸序列中第507个氨基酸残基的取代;及(b)seqidno:1的氨基酸序列中的第536个氨基酸残基的取代、第660个氨基酸残基的取代或第536个及第660个即两个氨基酸残基的取代。
根据本发明的另一个优选实施例,所述第507个氨基酸残基的取代是谷氨酸(e)被赖氨酸(k)取代,所述第536个氨基酸残基的取代是精氨酸(r)被赖氨酸取代(k),所述第660个氨基酸残基的取代是精氨酸(r)被缬氨酸(v)取代。
根据本发明的另一个优选实施例,所述pcr试剂盒进一步包含:a)三磷酸核苷;b)与双链dna结合的定量试剂;c)聚合酶阻断抗体;d)一个或多个对照值或对照序列;e)一个或多个模板。
本发明还提供一种利用所述pcr试剂盒从一个以上的模板中体外(invitro)检测至少一种基因突变或snp的检测方法。
与现有技术相比,本发明具有如下的有益效果:
本发明提供一种可以使具有增加的基因突变特异性的dna聚合酶发挥其功能的最佳pcr缓冲液组合物,与具有增加的基因突变特异性的dna聚合酶一起使用,显著提高所述dna聚合酶的活性,可实现可靠的基因突变-特异性扩增。另外,包含本发明的pcr缓冲液组合物和/或具有增加的基因特异性的dna聚合酶的试剂盒可以有效地检测基因突变或snp,因此可以有效地利用于疾病的医学诊断和重组dna的研究。
附图说明
通过阅读参照以下附图对非限制性实施例所作的详细描述,本发明的其它特征、目的和优点将会变得更明显:
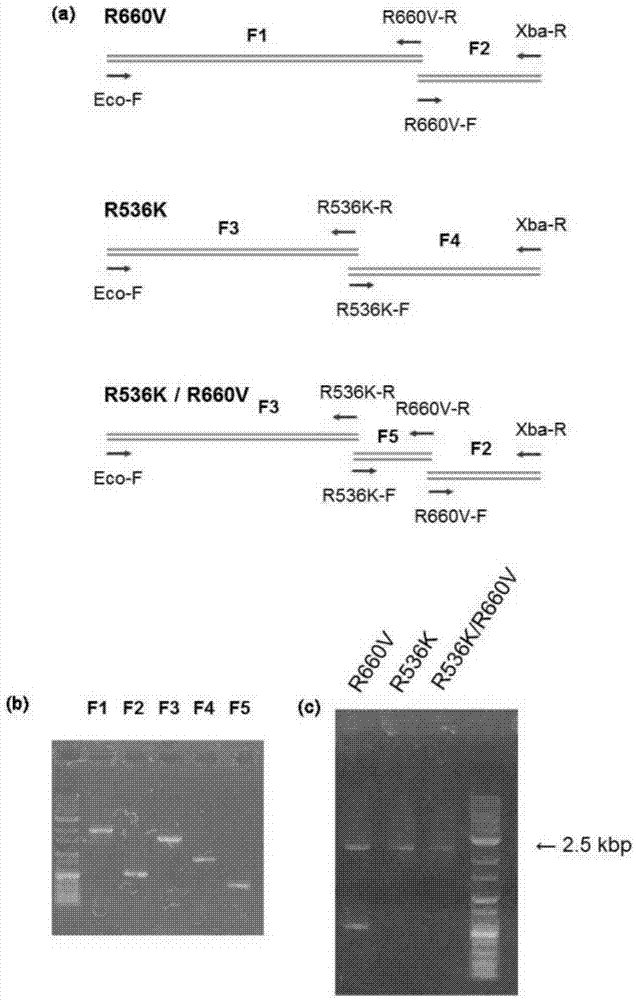
图1描述了分别含有r536k、r660v和r536k/r660v突变的taqdna聚合酶的制备过程,其中,(a)是片段pcr及重叠pcr的示意图,(b)显示在片段pcr中的扩增产物的电泳确认结果,(c)显示用重叠pcr扩增全长后通过电泳确认扩增产物的结果;
图2显示,为了提取凝胶,用限制性酶ecori/xbai分解后进行sap处理的puc19载体和纯化的图1(c)的重叠pcr产物的电泳确认结果;
图3是制备分别包含e507k、e507k/r536k、e507k/r660v及e507k/r536k/r660v突变的taqdna聚合酶过程中片段pcr和重叠pcr的示意图;
图4显示,为了提取凝胶,用限制性酶ecori/xbai分解后进行sap处理的puc19载体和纯化的图3的重叠pcr产物的电泳确认结果;
图5是显示通过收集口腔上皮细胞来制备pcr模板的过程的示意图;
图6显示了使用本发明的e507k/r536k、e507k/r660v和e507k/r536k/r660vtaq聚合酶对rs1408799进行的as-qpcr的结果;作为对照组,使用包括e507k突变的taq聚合酶;
图7显示了使用本发明的e507k/r536k、e507k/r660v和e507k/r536k/r660vtaq聚合酶对rs1015362进行的as-qpcr的结果;作为对照组,使用包括e507k突变的taq聚合酶;
图8显示了使用本发明的e507k/r536k、e507k/r660v和e507k/r536k/r660vtaq聚合酶对rs4911414进行的as-qpcr的结果;作为对照组,使用包括e507k突变的taq聚合酶;
图9显示了使用本发明的e507k、e507k/r536k、e507k/r660v和e507k/r536k/r660vtaq聚合酶,根据反应缓冲液中kcl浓度的变化,确认错配扩增的延迟效果的图表;
图10显示,为了确定反应缓冲液中最佳的kcl浓度,固定(nh4)2so4的浓度并变化kcl的浓度进行扩增后,通过电泳确认pcr产物的结果;
图11显示,为了确定反应缓冲液中最佳的(nh4)2so4浓度,固定kcl的浓度并变化(nh4)2so4的浓度进行扩增后,通过电泳确认pcr产物的结果;
图12显示,根据反应缓冲液中(nh4)2so4浓度的变化,确认错配扩增的延迟效果的图表;
图13显示,在反应缓冲液中固定kcl和(nh4)2so4的浓度,根据tmac浓度的变化,确认错配扩增的延迟效果的图表;
图14显示,在反应缓冲液中固定tmac和(nh4)2so4的浓度,根据kcl浓度的变化,确认错配扩增的延迟效果的图表。
具体实施方式
下面将对本发明进行详细说明。
如上所述,为了改善以往技术中揭示的基因突变-特异性扩增方法的缺点,需要持续开发具有增加的基因突变特异性的dna聚合酶及可使所述dna聚合酶发挥其功能的混合多种物质的最佳反应缓冲液,这些方法的开发,将对通过pcr的直接的基因突变或snp分析的可信度和强效性带来很大影响。本发明人致力于开发能够提高基因突变-特异性pcr扩增选择性的新型dna聚合酶及用于提高其活性的反应缓冲液,结果证实对taq聚合酶特定位置处的氨基酸残基诱发突变时,基因突变特异性显著增加,当控制pcr缓冲液组合物中的kcl、(nh4)2so4和/或tmac(四甲基氯化铵,tetramethylammoniumchloride)的浓度时,所述具有增加的基因突变特异性的dna聚合酶的活性增强,从而完成本发明。
本发明提供可使具有增加的基因突变特异性的dna聚合酶发挥其功能的最佳pcr缓冲液组合物,与具有增加的基因突变特异性的dna聚合酶一起使用,显著提高所述dna聚合酶的活性,可实现可靠的基因突变-特异性扩增。另外,包含本发明的pcr缓冲液组合物和/或具有增加的基因特异性的dna聚合酶的试剂盒可以有效地检测基因突变或snp,因此可以有效地利用于疾病的医学诊断和重组dna的研究。
下面,将对本文所用术语进行说明。
本文中“氨基酸”是指可以导入到肽、多肽或蛋白质中的任何单体单元。如本文所使用,术语“氨基酸”包括以下20种天然或遗传编码的α-氨基酸:丙氨酸(ala或a)、精氨酸(arg或r)、天冬酰胺(asn或n)、天冬氨酸(asp或d)、半胱氨酸(cys或c)、谷氨酰胺(gln或q)、谷氨酸(glu或e)、甘氨酸(gly或g)、组氨酸(his或h)、异亮氨酸(ile或i)、亮氨酸(leu或l)、赖氨酸(lys或k)、蛋氨酸(met或m)、苯丙氨酸(phe或f)、脯氨酸(pro或p)、丝氨酸(ser或s)、苏氨酸(thr或t)、色氨酸(trp或w)、酪氨酸(tyr或y)、缬氨酸(val或v)。
氨基酸通常是有机酸,可包含被取代或未被取代氨基、被取代或未被取代的羧基和至少一个侧链(chain)或基团(group),或这些基团的任何类似物。示例性的侧链包括巯基、硒基、磺酰基、烷基、芳基、酰基、酮基、叠氮基、羟基、肼基、氰基、卤基、酰肼、烯基、炔基、醚基、硼酸酯、硼酸基、二氧磷基、膦磺酸基、膦基、杂环、烯酮、亚胺、醛基、酯基、硫代酸、羟胺或这些基团的任何组合。
其他示例性氨基酸包括但不限于:含有可光活化交联剂的氨基酸、金属结合氨基酸、自旋标记氨基酸、荧光氨基酸、含金属的氨基酸、具有新型官能团的氨基酸、与其他分子共价或非共价相互作用的氨基酸、可光笼化和/或可光异构化氨基酸、放射性氨基酸、含有生物素或生物素类似物的氨基酸、糖基化氨基酸、其他碳水化合物修饰的氨基酸、含有聚乙二醇或聚醚的氨基酸、重原子取代氨基酸、可化学分解和/或可光分解的氨基酸、碳连接的含糖氨基酸、氧化还原活性氨基酸、含硫羧酸的氨基酸及含有至少一个毒性部分的氨基酸。
在本发明的dna聚合酶中,术语“突变体”指相对于天然存在的或未修饰的dna聚合酶包含一个或多个氨基酸取代的重组多肽。
术语“热稳定性聚合酶”(指对热稳定的酶)指具有足够的热抵抗性,保持足够的活性以实现随后的多核苷酸延伸反应,并且在实现双链核酸变性所需的时间过程中升温处理时不会不可逆地变性(失活)。如本文所用,它适用于pcr等反应中的循环温度。这里的不可逆变性是指永久性的酶活性的完全丧失。对于热稳定性聚合酶,酶活性是指用合适的方式催化核苷酸组合以在模板核酸链上形成互补的多核苷酸延伸产物。嗜热性细菌衍生的热稳定性dna聚合酶包括例如:来自海栖热袍菌(thermatogamaritime)、水生栖热菌(thermusacquaticus)、嗜热栖热菌(thermusthermophilus)、黄栖热菌(thermusflavus)、丝状栖热菌(thermusfiliformus)、栖热菌种sps17、栖热菌种z05、热坚栖热菌(thermuscaldophilus)、热坚芽孢杆菌(bacilluscaldotenax)、新阿波罗栖热袍菌(thermotoganeapolitana)、非洲栖热腔菌(thermosiphoafricanus)的dna聚合酶。
术语“热活性”是指在rt-pcr和/或pcr反应中的逆转录或退火/延伸阶段通常使用的温度(即,45~80℃)下保持其催化性能的酶。热稳定性酶是当用核酸变性所需的高温处理时不会不可逆地失活或变性的酶。而热活性酶可能是热稳定的或可能不是热稳定的。热活性dna聚合酶可以包括但不限于依赖于嗜热性物种或嗜温性物种的dna或rna。
术语“宿主细胞”是指用于细胞培养基中培养的来自高等植物或动物的单细胞原核生物和真核生物有机体(例如细菌、酵母和放线菌)以及单细胞。
术语“载体(vector)”是能够被复制,并能够将基因等将外源dna传递到受体细胞的dna分子如质粒、噬菌体和人工染色体等。“质粒”、“载体”或“质粒载体”在本文中可互换使用。
术语“核苷酸(nucleotide)”是指以单链(singlestrand)或双链(doublestrand)形式存在的脱氧核糖核酸(deoxyribonucleicacid;dna)或核糖核酸(ribonucleicacid;rna),并且除非另有特别说明,可以包括天然核苷酸的类似物。
术语“核酸”或“多核苷酸”是指dna或rna聚合物,或其类似物的聚合物。核酸可以是例如染色体或染色体片段、载体(例如表达载体)、表达盒、裸露的dna或rna聚合物、聚合酶链式反应(pcr)产物、寡核苷酸、探针或引物,或包含所述物质。核酸可以是例如单链、双链或三链,但不限于任何特定的长度。除非另有说明,特定的核酸序列除了包含指定的任何序列之外还包括互补序列或可以对其进行编码。
术语“引物”是指当多核苷酸在延伸启动条件下时可以充当模板方向上的核酸合成起始点的多核苷酸。引物也可用于多种其他寡核苷酸介导的合成过程,包括从头合成(denovo)rna和作为试管内转录相关过程的启动子。引物通常是单链寡核苷酸(例如,寡脱氧核糖核苷酸)。引物的适当长度通常在6至40个核苷酸范围内,更典型的长度在15至35个核苷酸范围内并取决于预期用途。短引物分子通常需要较低的温度以与模板形成足够稳定的杂交复合物。引物不需要反映模板的正确序列,但引物必须有足够的互补性才能与需延长的模板进行杂交。在特定实施方案中,术语“引物对”是指包括与待扩增的核酸序列的5'末端互补杂交的5'正义引物,与待扩增序列的3'末端杂交的3'-反义引物的引物组。如有必要,可以通过掺入可通过光谱、光化学、生物化学、免疫化学或化学手段检测到的标签来标记引物。例如,有用的标记包括:32p、荧光染料、电子密度试剂、酶(通常用于elisa分析)、生物素或半抗原、以及可以使用抗血清或单克隆抗体的蛋白质。
术语“5'-核酸水解酶(核酸酶,nuclease)探针”是指包含至少一个用于进行5'-核酸水解酶反应的发光标记物部分以检测靶核酸的寡核苷酸。在一些实施方案中,例如,5'-核酸水解酶探针仅包含单个发光部分(例如荧光染料等)。在某些实施方案中,5'-核酸水解酶探针包含自我互补区域,使得探针可以在选择条件下形成发夹结构。在一些实施方案中,5'-核酸水解酶探针包含两个或更多个标记部分,两个标记中的一个从寡核苷酸分离或分解,辐射强度增加而被释放。在某些实施方案中,5'-核酸水解酶探针用两种不同的荧光染料标记,例如5'-末端报道染料和3'-末端淬灭染料或部分标记。在一些实施方案中,5'-核酸酶探针在末端上再加标记或在除末端以外的一个或多个位置加标记。当探针完好时,能量转移通常发生在两种荧光材料之间,使得来自报道染料的荧光发射被淬灭一定部分以上。在聚合酶链式反应的延伸步骤期间,例如,与模板核酸结合的5'-核酸水解酶探针具有使得报道染料的荧光发射不再被淬灭的活性,例如被taq聚合酶或其他聚合酶的5'至3'-核酸水解酶活性分解。在一些实施方案中,5'-核酸水解酶探针可以用两种或更多种不同的报道染料和3'-末端淬灭染料或部分标记。
术语“fret”或“荧光共振能量转移”或“forester共振能量转移”是指两个或更多个发色团,供体发色团和受体发色团(称为淬灭剂)之间的能量转移。供体通常通过发射适当波长的光而激发,将能量转移给受体。受体通常以重新发射不同波长的光的形式将移动的能量再发射出去。当受体是“暗”淬灭剂时,以光以外的形式将移动的能量进行分散。一个特定的荧光物质是否充当供体或受体取决于fret对其他成员的性质。常用的供体-受体对包括fam-tamra对。常用的淬灭剂是dabcyl和tamra。常用的暗淬灭剂包括:blackholequencherstm(bhq),(biosearchtechnologies,inc.,novato,cal.),iowablacktm(integrateddnatech.,inc.,coralville,iowa),
当提及核酸碱基、三磷酸核苷或核苷酸时,术语“常规的”或“天然的”是指天然发生于所述多核苷酸(即,对于dna是datp、dgtp、dctp、dttp),而且ditp和7-deaza-dgtp经常用来代替dgtp,在测序等试管内dna合成反应中可以用来代替datp。
提及核酸碱基、核苷、或核苷酸时,术语“非常规”或“修饰”包含在特定多核苷酸中天然发生的常规的碱基、核苷、或核苷酸的修饰、衍生物或类似物。与常规的dntp相比,特定的非常规的核苷酸在核糖的2'位置被修饰。因此,rna的天然发生的核苷酸即使为核糖核苷酸(即,atp、gtp、ctputp、统称rntp),核苷酸在糖2’位置具有羟基基团,相对来说dntp不存在,因此如本文所用,核糖核苷酸是非常规的作为dna聚合酶底物的核苷酸。如本文所用,非常规核苷酸包括但不限于用作核酸测序终止子的化合物。示例性终止子化合物包括但不限于具有2',3'-二脱氧结构的化合物,被称为双脱氧核苷三磷酸。双脱氧核苷三磷酸ddatp、ddttp、ddctp和ddgtp,被统称为ddntp。终止子化合物的其他例子包括核糖核苷酸的2'-po4类似物。其他非常规核苷酸包括但不限于硫代dntp([[α]-s]dntp)、5'-[α]-硼(borano)-dntp、[α]-甲基-膦酸dntp、核糖核苷三磷酸(rntp)。非常规的碱基可用放射性同位素如32p、33p或35s;荧光标记;化学发光标记;生物发光标记;半抗原标签如生物素;或酶标签如链霉抗生物素蛋白或抗生物素蛋白来标记。荧光标记可以包括带负电荷的染料如荧光素家族的染料、或带中性电荷的染料如罗丹明家族的染料、或带正电荷的染料如花青家族。荧光素家族的染料包括例如fam、hex、tet、joe、nan及zoe。罗丹明家族的染料包括texasred、rox、r110、r6g及tamra。用fam、hex、tet、joe、nan、zoe、rox、r110、r6g、texasred和tamra标记的各种染料或核苷酸通过perkin-elmer(波士顿,马萨诸塞州)、appliedbiosystems(福斯特市,加州)、invitrogen/molecularprobes(尤金,俄勒冈州)市场销售。花青家族染料包括cy2、cy3、cy5、及cy7,通过gehealthcareuklimited(amershamplace,littlechalfont,白金汉郡,英格兰)市场销售。
术语“错配的区分”,是指通过在核酸上将一个或多个核苷酸粘附(例如共价地),以模板依赖性方式延伸核酸(例如引物或其它寡核苷酸)时,用于从错配-包含序列区分完全互补的序列的生物催化(例如聚合酶、连接酶等酶)的能力。术语“错配的区分”,是指延伸的核酸(如引物或其它寡核苷酸)与核酸杂交的模板相比,从3'末端核酸具有错配的错配-包含(约互补)序列区分完全互补的序列的生物催化能力。在一些实施方案中,延伸的核酸对于完全互补序列在3'末端包含错配。在一些实施方案中,延伸的核酸对于完全互补序列在末端第二个(n-1)3'-位置和/或n-2位置上包含错配。
除非另有说明,本说明书中使用的所有技术及科学术语具有与本领域普通技术人员通常理解的相同的含义。
本发明涉及具有增加的基因突变特异性的dna聚合酶活性增强用pcr缓冲液组合物,所述组合物包含25至100mm的kci;1至15mm的(nh4)2so4;最终ph为8.0至9.0。
pcr中使用的聚合酶应使用混合各种物质的最佳反应缓冲液,以发挥其功能。反应缓冲液通常含有稳定ph的因子,作为辅助因子(cofactor)的金属离子和防止聚合酶变性的稳定化成分。
所述kcl可作为稳定酶的必要因子,帮助引物与靶dna配对(pairing)。在本发明中,通过调节反应缓冲液中kcl的浓度来确定最佳浓度,以确认最大程度延迟由错配引起的扩增,同时不降低由匹配引起的扩增效率的高阳离子浓度。
分别使用e507k、e507k/r536k、e507k/r660v及e507k/r536k/r660vtaq聚合酶,确认根据反应缓冲液中kcl浓度变化产生的错配扩增延迟效果,结果如图9所示,对于e507k/r536k/r660vtaq聚合酶,即使反应缓冲液中没有kcl,其错配扩增延迟效果也很优秀;对于e507k/r536和e507k/r660v,在50mm;作为对照组e507k,在100mm时,错配扩增延迟效果优秀。结果来看,对于kcl浓度的阈值,e507k/r536k/r660vtaq聚合酶最低,e507k/r536k和e507k/r660v比e507k低。
为了进一步推导适当的kci浓度,使用e507k/r536k/r660vtaq聚合酶,将反应缓冲液中的(nh4)2so4浓度固定在一定数值,采用多种kcl浓度进行扩增。通过电泳确认扩增产物的结果,如图10所示,可以确认适当的kci浓度为75mm。
因此,本发明的pcr反应缓冲液组合物中kcl浓度可以是25~100mm,优选为60~90mm,更优选为70~80mm,最佳为75mm。
当kcl浓度低于25mm时,对于一般靶标的扩增没有影响,但可使由匹配引物产生的扩增与由错配引物产生的扩增差异减小,当浓度高于100mm时,产生一般靶标的扩增效率下降的问题。
对于pcr反应缓冲液组合物,所述(nh4)2so4为酶活性的必要辅助因子,与tris一同用于提高聚合酶的活性。本发明的一个实施例中,以上述推导结果作为基础,将反应缓冲液中kcl浓度固定在75mm,将(nh4)2so4的浓度从2.5mm到25mm进行各种变化,以确认适当的(nh4)2so4的浓度。
其结果如图11所示,在2.5mm到15mm的(nh4)2so4的浓度下,确认扩增产物,且适当的(nh4)2so4的浓度为5mm。
进一步在(nh4)2so4的的浓度接近5mm(分别用2.5mm、5mm、10mm)的情况下,进行as-qpcr,结果如图12所示,虽然在10mm下ct值差异最大,但确认匹配扩增中ct稍微延迟,峰向下倾倒,因此确定适当的(nh4)2so4的浓度为5mm。
因此,本发明的pcr反应缓冲液组合物中(nh4)2so4的浓度可以是1~15mm,优选为2.5~8mm,更优选为4~6mm,最佳为5mm。
当(nh4)2so4浓度低于1mm时,对于一般靶标的扩增没有影响,但可使由匹配引物产生的扩增与由错配引物产生的扩增差异减小,当浓度高于15mm时,产生一般靶标的扩增效率下降的问题。
因此,本发明的最佳pcr缓冲液组合物可以包含70至80mm的kcl;4至6mm的(nh4)2so4;最终ph为8.0至9.0。
本发明的pcr缓冲液组合物可以进一步包含5至80mm的tmac(四甲基氯化铵,tetramethylammoniumchloride)。
tmac通常用于减少由错配引起的扩增或提高杂交反应的严谨性(stringency)。本发明的一个实施例中,以上述推导结果作为基础,将反应缓冲液中kcl浓度固定在75mm,(nh4)2so4的浓度固定在5mm,将tmac浓度从0mm到80mm进行各种变化,以确认适当的tmac浓度。
其结果如图13所示,确认了适当的tmac浓度,对于e507k/r536ktaq聚合酶为70mm;对于e507k/r536k/r660vtaq聚合酶为25mm。进一步将tmac浓度固定在25mm,(nh4)2so4的浓度固定在2.5mm,将kcl浓度分别调整为20、40、60、80mm,进行扩增,结果如图14所示,确认对于snprs1015362及rs4911414,适当的kcl浓度为60mm。
当tmac浓度超过80mm时,扩增效率降低,因此优选在所述范围内使用tmac。
因此,当本发明的pcr缓冲液组合物包含5~80mm的tmac时,kcl的浓度可以是40~90mm,优选50~80mm,(nh4)2so4的浓度可以是1~7mm,优选1.5~6mm。
因此,当包含tmac时,本发明的最佳pcr缓冲液组合物可以包含15至70mm的tmac;50至80mm的kcl;1.5至6mm的(nh4)2so4;最终ph为8.0至9.0。
本发明的pcr缓冲液组合物还可以进一步包括tris·cl及mgcl2,还可以进一步包括tween20及牛血清白蛋白(bsa)。
本发明提供包含所述pcr缓冲液组合物的用于检测基因突变或snp的pcr试剂盒。
本发明的pcr试剂盒可进一步包含由seqidno:1的氨基酸序列组成的taq聚合酶的dna聚合酶,所述dna聚合酶可包含(a)seqidno:1的氨基酸序列中第507个氨基酸残基的取代;及(b)seqidno:1的氨基酸序列中的第536个氨基酸残基的取代、第660个氨基酸残基的取代或第536个及第660个即两个氨基酸残基的取代。
根据本发明的另一个优选实施例,所述第507个氨基酸残基的取代是谷氨酸(e)被赖氨酸(k)取代,所述第536个氨基酸残基的取代是精氨酸(r)被赖氨酸取代(k),所述第660个氨基酸残基的取代是精氨酸(r)被缬氨酸(v)取代。
本发明的pcr试剂盒可包括一般技术人员在引物延伸过程中使用的已知任意试剂或其他要素。
根据本发明的另一个优选实施例,所述pcr试剂盒包括一个以上的匹配引物、一个以上的错配引物或同时包含一个以上的匹配引物和一个以上的错配引物,所述一个以上的匹配引物及一个以上的错配引物与靶序列杂交,所述错配引物,对于杂交靶序列,在其3'末端至第7个碱基位置包含非典型(non-canonical)核苷酸。
本发明的pcr试剂盒还可进一步包含:a)三磷酸核苷;b)与双链dna结合的定量试剂;c)聚合酶阻断抗体;d)一个或多个对照值或对照序列;e)一个或多个模板。
所述“taq聚合酶”,是根据嗜热性细菌水生栖热菌(thermusaquaticus)的名字来命名的耐热性dna聚合酶,最初从所述细菌中被分离出来。水生栖热菌是栖息在温泉和热水喷出口中的细菌,taq聚合酶被确认为是能够承受在pcr过程中所需的蛋白变性条件(高温)的酶。taq聚合酶的最佳活性温度是75~80℃,半衰期在92.5℃条件下为2小时以上,在95℃条件下为40分钟,97.5℃条件下为9分钟,在72℃条件下10秒内可复制1000个碱基对dna。这缺乏3→'5'核酸外切酶(exonuclease)校正活性,并且在9000个核苷酸中,约在1个中测量出错误率。例如,当使用耐热taq时,pcr可以在高温(60℃或更高)下进行。对于taq聚合酶,使用seqidno:1中所示的氨基酸序列作为基准序列。
在本发明中,seqidno:1的氨基酸序列中,第507个氨基酸残基的谷氨酸(e)被取代为赖氨酸(k)的taq聚合酶被命名为“e507k”(seqidno:2,碱基序列为seqidno:7);seqidno:1的氨基酸序列中,第507个氨基酸残基的谷氨酸(e)被取代为赖氨酸(k),第536个氨基酸残基的精氨酸(r)被取代为赖氨酸(k)的taq聚合酶被命名为“e507k/r536k”(seqidno:3,碱基序列为seqidno:8);seqidno:1的氨基酸序列中,第507个氨基酸残基的谷氨酸(e)被取代为赖氨酸(k),第660个氨基酸残基的精氨酸(r)被取代为缬氨酸(v)的taq聚合酶被命名为“e507k/r660v”(seqidno:4,碱基序列为seqidno:9);最后,seqidno:1的氨基酸序列中,第507个氨基酸残基的谷氨酸(e)被取代为赖氨酸(k),第536个氨基酸残基的精氨酸(r)被取代为赖氨酸(k),第660个氨基酸残基的精氨酸(r)被取代为缬氨酸(v)的taq聚合酶被命名为“e507k/r536k/r660v”(seqidno:5,碱基序列为seqidno:10)。
根据本发明的一个实施例,所述dna聚合酶区分匹配引物和错配引物,所述匹配引物和错配引物与靶序列杂交,所述错配引物,对于杂交靶序列,在其3'末端可包含非典型核苷酸。
所述错配引物是杂交寡核苷酸,必须充分互补以与靶序列杂交,但不反映靶序列的确切序列。
所述“典型核苷酸(canonicalnucleotide)”或“互补核苷酸”是指标准沃森-克里克(watson-crick)碱基对、a-u、a-t和g-c。
所述“非典型核苷酸(non-canonicalnucleotide)”或“非互补核苷酸”是指除沃森-克里克(watson-crick)碱基对以外的a-c、a-g、g-u、g-t、t-c、t-u、a、g-g、t-t、u-u、c-c、c-u。
根据本发明的一个优选实施例,包含匹配引物的靶序列的扩增,与包含错配引物的靶序列的扩增相比,所述dna聚合酶可表现出比更低的ct值。
例如,通过将一个或多个核苷酸共价连接至引物,dna聚合酶可以比以靶序列依赖性方式错配的引物更高效率地延伸匹配的引物。在这里,更高的效率可通过以下例子观察到,如rt-pcr中与错配引物相比,匹配引物的ct值更低。匹配引物和错配引物之间的ct值的差异为10或更高,优选10~20,或者不存在由错配引物引起的扩增子合成。
例如,第一反应中使用匹配的正向引物和反向引物,第二反应中,以相同的实验设置,使用错配的正向引物和匹配的反向引物,通过标准pcr形成的产物,第一个反应比第二个反应更大。
ct(阈值交叉循环)值表示定量pcr的dna定量方法,取决于绘制对数相循环数量上的荧光。基于dna的荧光检测的阈值设置为至少比背景稍高。荧光超过阈值的循环数称为ct或根据miqe准则称为cq(定量循环,quantificationcycle)。给定反应的ct值定义为荧光发射交叉固定极限值的循环次数。例如,sybrgreeni和荧光探针可用于模板dna定量的实时pcr。在pcr期间每个循环收集来自样品的荧光,并对照循环次数绘图。起始模板浓度与荧光信号的荧光信号最初显示时间成反比。模板浓度越高,信号越早出现(在较低的循环数出现)。
本发明还涉及编码上述dna聚合酶的核酸序列和包含该核酸序列的载体和宿主细胞。使用编码本发明的dna聚合酶的核酸可以制备各种载体。可以使用包括来源于可与宿主细胞互换的物种的复制子和控制序列的任何载体。本发明的载体可以是表达载体并且包括与编码本发明的dna聚合酶的核酸序列可操作地连接的控制转录和翻译的核酸区域。调控序列是指在特定宿主生物体中表达可操作地连接的编码序列所需的dna序列。例如,适用于原核生物的调控序列包括启动子、任何操作序列和alc核糖体结合位点。另外,载体可以含有“正调节因子(positiveretroregulatoryelement,pre)”以增强转录的mrna的半衰期。控制转录和翻译核酸区域通常适用于用于表达聚合酶的宿主细胞。本领域已知用于各种宿主细胞的各种类型的合适的表达载体和合适的调控序列。通常,转录和翻译调控序列可以包括例如启动子序列、核糖体结合位点、转录起始和终止序列、翻译起始和终止序列以及增强子或激活序列。在典型的实施方案中,调空序列包括启动子和转录起始和终止序列。载体通常还包含含有几个用于插入外源dna的限制性位点的多位点接头区域。在某些实施方案中,使用“融合标记”来促进纯化,并且如果需要,随后去除标签/标志(例如“组氨酸标签(his-tag)”)。然而,当从使用“加热步骤”的嗜温宿主(例如大肠杆菌)纯化热活性和/或热稳定性蛋白时,这些通常是不必要的。使用标准重组dna技术制备含有编码dna复制序列、调控序列、表型选择基因的合适载体和关注的突变聚合酶。如本领域已知,分离的质粒、病毒载体和dna片段被分解并剪切并以特定顺序连接在一起以产生期望的载体。
在本发明的一个优选实施例中,表达载体含有用于选择转化宿主细胞的选择标记基因。选择基因在本领域中是已知的,并且根据所使用的宿主细胞会有所不同。合适的选择基因可以包括编码氨苄青霉素和/或四环素抗性的基因,由此可以在存在这些抗生素的情况下转化培养这些载体的细胞。
在本发明的一个优选实施例中,编码本发明的dna聚合酶的核酸序列单独或与载体组合导入细胞中。导入或其等同表达是指核酸以适于随后的整合、扩增和/或表达的方式进入细胞。导入方法包括例如capo4沉淀、脂质体融合、
原核生物用作本发明初始克隆步骤的宿主细胞。为了快速制备大量的dna,为了制备用于生成位点-取向突变的单链dna模板,为了同时筛选许多突变体,为了对所产生的突变体进行dna测序,这是特别有用的。原核细胞的合适的宿主细胞包括大肠杆菌e.colik12菌株94(atccno.31,446)、大肠杆菌e.coli菌株w3110(atccno.27,325)、大肠杆菌e.colik12菌株dg116(atccno.53,606)、大肠杆菌e.colix1776(atccno.31,537)和大肠杆菌e.colib;大肠杆菌e.coli的许多不同的菌株如hb101、jm101、nm522、nm538、nm539,以及其他物种,包括芽孢杆菌科如枯草芽孢杆菌((bacillussubtilis)、其他肠杆菌科如鼠伤寒沙门氏杆菌(salmonellatyphimurium)、粘质沙雷氏菌(serratiamarcesans)、以及各种假单胞菌种(假单胞菌属,pseudomonassp.)的原核生物属可被作为宿主。典型地,用于转化大肠杆菌(e.coli)的质粒包括pbr322、puci8、puci9、pucil8、puc119及bluescriptm13。然而,也可以使用许多其他合适的载体。
本发明还提供制备dna聚合酶的方法,包括以下步骤:培养所述宿主细胞的步骤;从培养物及其培养上清液中分离dna聚合酶的步骤。
本发明的dna聚合酶通过在诱导或引起dna聚合酶表达的适当条件下,培养通过含有编码dna聚合酶的核酸序列的表达载体转化的宿主细胞来制备。在适合于蛋白质表达的条件下培养转化的宿主细胞的方法是本领域已知的。用于以含有λpl启动子的质粒载体制备聚合酶的合适宿主细胞是大肠杆菌菌株dg116(atccno.53606)。根据表达情况,可以收集并分离聚合酶。
一旦纯化,就可以测定本发明dna聚合酶的错配区分。例如,通过比较引物完全匹配的靶序列的扩增与引物3’末端的单碱基错配的靶序列的扩增来测量错配区别活性。扩增可以实时被检测,例如通过使用taqmantm探针。可以通过比较两种反应的ct来估计聚合酶区分两种靶序列的能力。
本发明提供利用所述pcr试剂盒从一个以上的模板中体外(invitro)检测至少一种基因突变或snp的检测方法。
术语“snp(单核苷酸多态性,singlenucleotidepolymorphisms)”是指在dna核苷酸序列中显示一个核苷酸序列(a、t、g或c)的差异的遗传变化或突变。
在本发明的体外(invitro)snp检测方法中,靶序列可以存在于测试样品中并包括dna、cdna或rna,优选包括基因组dna。测试样品可以是从细菌、细菌培养物或细胞培养物制备的一种细胞裂解物。另外,测试样品可以包含在动物中,优选包含在脊椎动物中,更优选包含在人受试者中。靶序列可以包含在基因组dna中,优选包含在个体的基因组dna中,更优选包含在细菌或脊椎动物,最优选包含在人受试者的基因组dna中。
本发明的snp检测方法可以包括利用双链特异性染料如sybrgreeni的熔解温度分析。
熔解温度曲线分析可以在包含软件(onboardsoftwaresds2.1)的abi5700/7000(96孔格式)或abi7900(384孔格式)等实时pcr设备中进行。或者,熔解温度曲线分析可以以终点分析来执行。
“结合双链dna的染料”或“双链特异性染料”,和未与双链dna结合时相比,与双链dna结合时具有高荧光的情况下可以被使用。这种染料的实例包括soyto-9、soyto-13、soyto-16、soyto-60、soyto-64、syto-82、溴乙烷(etbr)、sytoxorange、to-pro-1、sybrgreeni、to-pro-3或evagreen。除了etbr和evagreen(quiagen),这些染料已用于实时应用测试。
本发明的体外(invitro)snp检测方法可通过实时pcr、标准pcr后的琼脂糖凝胶分析、通过实时pcr的基因突变特异性扩增或等位基因特异性扩增、四引物扩增受阻突变系统pcr或等温扩增来进行。
所谓“标准pcr”是一般技术人员已知的用于扩增dna或cdna的单拷贝或多拷贝的技术。几乎所有的pcr都使用taq聚合酶或热稳定dna聚合酶如klentaq。dna聚合酶通过使用单链dna作为模板并使用寡核苷酸(引物)从核苷酸酶促装配新的dna链。通过pcr产生的扩增子,可以在琼脂糖凝胶上分析。
所述“实时pcr”可以在执行pcr时实时监控其过程。因此,在整个pcr过程中收集数据,而不是在pcr结束时收集。在实时pcr中,反应的特征是循环中首次检测到扩增的时间点,而不是固定循环次数后累积的目标量。主要使用基于染料的检测和基于探针的检测两种方法来执行定量pcr。
所述“等位基因特异性扩增(allelespecificamplification,asa)”是一种扩增技术,其pcr引物被设计为单个核苷酸残基可以区分其他模板。
所述“通过实时pcr的基因突变特异性扩增或等位基因特异性扩增”可以以高效方式检测基因突变或snp。与大多数检测基因突变或snp的其他方法不同,目标基因材料的预扩增不是必需的。asa根据匹配和错配的引物/靶序列复合物之间的区别,在单个反应中结合扩增和检测。反应过程中扩增dna的增加可以通过由诸如sybrgreeni的染料引起的荧光信号的增加来实时监测,所述染料在与双链dna结合时发光。通过实时pcr的遗传突变特异性扩增或等位基因特异性扩增,发生错配时出现荧光信号的延迟或信号不存在。在基因突变或snp检测中,这提供了关于是否存在基因突变或snp的信息。
所述“四引物扩增受阻突变系统pcr”在单管pcr反应中与对照片段一同扩增野生型和突变型等位基因。非等位基因特异性对照扩增子通过突变区侧面的两个通用(外部)引物扩增。两个等位基因特异性(内部)引物的设计方向与通用引物相反,与通用引物一同扩增野生型和突变型扩增子。结果,两个等位基因特异性扩增子由于突变的位置相对于通用(外部)引物是不对称的,因此具有不同的长度,可以通过标准凝胶电泳容易地分离。所述对照扩增子提供了假阴性和扩增失败的内部对照,并且两个等位基因特异性扩增子中的至少一个始终存在于四引物扩增受阻突变系统pcr中。
所述“等温扩增”是指核酸的扩增在较低温度下进行,而不依赖于热循环仪,优选温度不需要在扩增期间改变。等温扩增中使用的温度可以在室温(22~24℃)至约65℃之间,或者在约60~65℃、45~50℃、37℃~42℃或22~24℃的常温。等温扩增产物可通过凝胶电泳、elisa、elosa(酶联寡核苷酸试验,enzymelinkedoligosorbentassay)、实时pcr、ecl(改进的化学发光)、分析rna、dna和蛋白质或浊度的基于芯片的毛细管电泳装置生物分析仪(bioanalyzer)检测。
在本发明的一个实施例中,使用e507k/r536k、e507k/r660v或e507k/r536k/r660vtaq聚合酶,确认对于包含snp(rs1408799、rs1015362和/或rs4911414)的模板,其错配引物延伸能力是否减少。
结果,如图6至8所示,与e507ktaq聚合酶相比,可以证实e507k/r536k、e507k/r660v或e507k/r536k/r660vtaq聚合酶的由错配引物引起的扩增延迟,其效果在e507k/r536k/r660vtaq聚合酶中最为明显。
这证实了与常规taq聚合酶(e507k)相比,本发明的dna聚合酶具有更高的错配延伸选择性。因此,预计本发明的dna聚合酶可有效应用于疾病的医学诊断和重组dna的研究。
根据本发明的另一个优选实施例,所述pcr试剂盒包括一个以上的匹配引物、一个以上的错配引物或同时包含一个以上的匹配引物和一个以上的错配引物,所述一个以上的匹配引物及一个以上的错配引物与靶序列杂交,所述错配引物,对于杂交靶序列,在其3'末端至第7个碱基位置包含非典型(non-canonical)核苷酸。
本发明的pcr试剂盒可以进一步包含三磷酸核苷。
本发明的pcr试剂盒还包括a)一种或多种缓冲液;b)与双链dna结合的定量试剂;c)聚合酶阻断抗体;d)一个或多个对照值或对照序列;e)一个或多个模板;如图所示。
下面将通过实施例对本发明进行详细说明。这些实施例是对本发明的示例性说明,本领域具有一般知识的人应理解,本发明的权利范围并不局限于所述实施例。
实施例1诱发taq聚合酶突变
1-1.片段pcr
在本实施例中,按照以下方法制备seqidno:1的氨基酸序列中的第536个氨基酸残基的精氨酸被赖氨酸取代的taqdna聚合酶(以下称为“r536k”)、第660个氨基酸残基的精氨酸被缬氨酸取代的taqdna聚合酶(以下称为“r660v”)、和第536个氨基酸残基的精氨酸被赖氨酸取代以及第660个氨基酸残基的精氨酸被缬氨酸取代的taqdna聚合酶(以下称为“r536k/r660v”)。
首先,使用表1所示的突变特异性引物通过pcr扩增taqdna聚合酶片段(f1至f5),如图1(a)所示。反应条件如表2所示。
表1
表2
通过电泳确认pcr产物,结果如图1(b)所示,确认了各片段的条带,以此确认靶片段已被扩增。
1-2.重叠(overlap)pcr
将所述1-1中扩增的各片段作为模板,两端使用引物(eco-f和xba-r引物)扩增全长。反应条件如表3和表4所示。
表3
表4
结果,如图1(c)所示,确认了“r536k”、“r660v”、“r536k/r660v”tag聚合酶被扩增。
1-3.连接(ligation)
在表5所示的条件下,用限制性酶ecori/xbai将puc19在37℃下分解4小时并纯化dna,在表6所示的条件下,将纯化的dna在37℃用sap处理1小时以制备载体。
表5
表6
对于插入物(insert),将所述实施例1-2的重叠pcr产物纯化,并在表7所示条件下,用限制性酶ecori/xbai在37℃下分解3小时,然后与制备的载体一同进行凝胶提取(图2)。
表7
在表8所示的条件下,在室温(rt)下连接2小时后,转化大肠杆菌e.colidh5α并在含有氨苄青霉素的培养基上筛选。从获得的菌落制备的质粒进行测序,获得引入了所需突变的taqdna聚合酶突变体(“r536k”、“r660v”和“r536k/r660v”)。
表8
实施例2引入e507k突变
2-1.片段pcr
对所述实施例1中制备的“r536k”、“r660v”及“r536k/r660v”taqdna聚合酶的活性进行测试,结果确认活性下降(数据未图示),分别对“r536k”、“r660v”及“r536k/r660v”引入e507k突变(seqidno:1的氨基酸序列中,第507个氨基酸残基的谷氨酸被赖氨酸取代),作为对照组,在野生型(wt)taqdna聚合酶中也引入e507k突变。引入e507k突变的taqdna聚合酶制备方法与实施例1相同。
使用表9所示的突变特异性引物通过pcr扩增taqdna聚合酶片段(f6至f7),如图3所示。反应条件如表10所示。
表9
表10
*模板质粒:puc19-tag(wt)
puc19-tag(r536k)
puc19-tag(r660v)
puc19-tag(r536k/r660v)
2-2.重叠(overlap)pcr
将所述2-1中扩增的各片段作为模板,两端使用引物(eco-f和xba-r引物)扩增全长。反应条件如表11所示。
表11
2-3.连接(ligation)
在表5所示的条件下,用限制性酶ecori/xbai将puc19在37℃下分解4小时并纯化dna,在表6所示的条件下,将纯化的dna在37℃用sap处理1小时以制备载体。
对于插入物(insert),将所述实施例2-2的重叠pcr产物纯化,并在表7所示条件下,用限制性酶ecori/xbai在37℃下分解3小时,然后与制备的载体一同进行凝胶提取(图4)。
在表8所示的条件下,在室温(rt)下连接2小时后,转化大肠杆菌e.colidh5α或dh10β并在含有氨苄青霉素的培养基上筛选。从获得的菌落制备的质粒进行测序,获得引入了所需突变的taqdna聚合酶突变体(“e507k/r536k”、“e507k/r660v”和“e507k/r536k/r660v”)。
实施例3使用本发明的dna聚合酶进行qpcr
使用各自含有所述实施例2中获得的“e507k/r536k”、“e507k/r660v”和“e507k/r536k/r660v”突变的taq聚合酶,确认对于含有snp的模板其延伸错配引物能力是否减少。作为对照组,使用含有e507k突变的“e507k”taq聚合酶。
在本发明中使用的包含snp的模板为rs1408799、rs1015362和rs4911414,各模板的基因型和其特异性引物(idt,美国)的序列信息如表12和表13所示。
表12
表13
qpcr条件(appliedbiosystems7500fast)如下表14所示。
表14
探针进行双重标记,如下表15所示。
表15
使用购自noblebio的口腔上皮细胞收集试剂盒收集口腔上皮细胞,将其溶解于500μl裂解液(lysissolution)中,并以12,000×g离心3分钟。将上清液转移到新管中,每次使用1μl(图5)。
反应条件如表16表示,反应缓冲液的组成如表17表示。
表16
表17
除了表13所示的特异性引物以外,其他反应液以同样方式在两个试管中制备,通过添加各等位基因特异性引物进行qpcr。此时,合并各试管中检测到的荧光信号以在ab7500软件(v2.0.6)上计算和分析达到阈值(threshold))荧光值的循环(ct)值的差异。由错配引物引起的扩增中的ct值延迟的时间越长,可判断为基因突变特异性或等位基因特异性良好。
在rs1408799、rs1015362及rs4911414上进行as-qpcr的结果,如图6至8所示,与对照组e507k相比,可以证实包含e507k/r536k、e507k/r660v或e507k/r536k/r660v突变的taq聚合酶的情况下,错配引物引起的扩增延迟,其效果在e507k/r536k/r660v突变中最为明显。
这证实了与包含e507k突变的taq聚合酶相比,本发明的包含e507k/r536k、e507k/r660v或e507k/r536k/r660v突变的taqdna聚合酶具有更优秀的错配延伸选择性。因此,预计本发明的taqdna聚合酶可有效应用于疾病的医学诊断和重组dna的研究。
实施例4反应缓冲液的kcl浓度最佳化
在本实施例中,为了寻找使由匹配引起的扩增效率不降低而由错配引起的扩增最大限度地延迟的高阳离子浓度,调整pcr反应缓冲液中的kcl浓度来确定kcl的最佳浓度。
使用实施例2中获得的分别包含“e507k/r536k”、“e507k/r660v”或“e507k/r536k/r660v”突变的taq聚合酶,并与包括e507k突变的taq聚合酶比较kcl浓度阈值。
包含snp的模板使用rs1408799,模板的基因型为tt,引物使用表12中记载的rs1408799引物。qpcr条件(appliedbiosystems7500fast)以表14的条件进行,双重标记探针使用表15的1408799-fam,反应条件如表18所示,反应缓冲液组成如表19。
表18
表19
结果如图9所示,e507k/r536k/r660vtaq聚合酶的kci浓度阈值最低,e507k/r536k、e507k/r660v的kcl浓度阈值比e507k低。
以上述结果为基础,为了确定适当的kcl浓度,使用e507k/r536k/r660vtaq聚合酶进行进一步实验,引物使用表13中记载的rs1408799-t特异性引物。qpcr条件(appliedbiosystems7500fast)以表14的条件进行35个循环,反应条件如表20所示。
表20
对照组的反应缓冲液组成与表21相同,实验组的反应缓冲液的组成如表8,将(nh4)2so4浓度固定在2.5mm,对kcl浓度进行各种变化。
表21
用所述条件扩增后通过电泳确认pcr产物的结果,如图10所示,可以确认既可使由错配引起的扩增最大限度地延迟而又使由匹配引起的扩增效率不降低的适当的kcl浓度为75mm。
实施例5反应缓冲液的(nh4)2so4浓度最佳化
本实施例以所述实施例4的结果为基础,将反应缓冲液内kcl浓度固定在75mm,对(nh4)2so4浓度进行各种变化,以此确认最佳的(nh4)2so4浓度。引物使用表13中记载的rs1408799-t特异性引物,qpcr条件(appliedbiosystems7500fast)以表14的条件进行35个循环,反应条件如表20所示,对照组的反应缓冲液组成与表21相同。
其结果,如图11所示,适当的(nh4)2so4浓度为5mm。
以所述结果作为基础,将反应缓冲液中kcl浓度固定在75mm,将(nh4)2so4的浓度设定在5mm左右(分别为2.5mm、5mm及10mm),进一步确认由错配引起的扩增延迟效果。
引物使用表13中记载的rs1408799引物,双重标记探针使用表15的1408799-fam,反应条件如表22所示。
表22
其结果,如图12所示,(nh4)2so4的浓度在10mm下ct值差异最大,但确认由匹配引起的扩增ct稍微延迟,峰向下倾倒,因此确定适当的(nh4)2so4的浓度为5mm。
综合实施例4及实施例5的结果,确认最佳反应缓冲液组合包含50mm的tris·cl、2.5mm的mgcl2、75mm的kcl、5mm的(nh4)2so4、0.1%tween20及0.01%bsa。
实施例6反应缓冲液中添加tmac及其浓度最佳化
在本实施例中,在反应缓冲液中添加tmac,并确认其最佳浓度。以所述实施例4及实施例5的结果为基础,将反应缓冲液中kcl浓度固定在75mm,(nh4)2so4浓度固定在5mm,对tmac的浓度进行各种变化,以此确认最佳的tmac浓度。
使用e507k/r536k或e507k/r536k/r660vtaq聚合酶,包含snp的模板使用rs1408799,模板的基因型为tt,引物使用表12中记载的rs1408799引物。qpcr条件(appliedbiosystems7500fast)以表14的条件进行,双重标记探针使用表15的1408799-fam,反应条件如表22所示。
实验结果如图13所示,确认了适当的tmac浓度,对于e507k/r536ktaq聚合酶为60mm;对于e507k/r536k/r660vtaq聚合酶为25mm,并确认若tmac浓度过高,扩增效率降低。
实施例7反应缓冲液的kcl、(nh4)2so4及tmac浓度最佳化
在本实施例中,基于所述实施例6确认的结果,使用e507k/r536k/r660vtaq聚合酶确认反应缓冲液中最佳的kcl、(nh4)2so4及tmac浓度。
具体而言,将反应缓冲液中tmac浓度固定在25mm,(nh4)2so4浓度固定在2.5mm,将kcl浓度分别调整为20、40、60、80mm。对于两个snp即rs1015362及rs4911414进行实验,模板的基因型如表12所述,引物使用表13中记载的rs1015362及rs4911414引物,qpcr条件(appliedbiosystems7500fast)以表14的条件进行,双重标记探针使用表15的1408799-fam,反应条件如表22所示。
实验结果,如图14所示,对于两个snp,适当的kcl浓度为60mm,浓度为80mm时,可以观察到扩增效率降低。
通过以上结果,确认反应缓冲液中最佳的kcl浓度为60mm,(nh4)2so4浓度为2.5mm,tmac浓度为25mm。更具体来说,确认对于e507k/r536k聚合酶,使用浓度为75mm的kcl、5mm的(nh4)2so4、60mm的tmac最有效;对于e507k/r536k/r660v聚合酶,使用浓度为60mm的kcl、2.5mm的(nh4)2so4浓度、25mm的tmac最有效。
序列表
<110>基因凯斯特有限公司
<120>一种具有增加的基因突变特异性的dna聚合酶活性增强用pcr缓冲液组合物
<130>dag35927
<150>kr10-2017-0088376
<151>2017-07-12
<160>30
<170>siposequencelisting1.0
<210>1
<211>832
<212>prt
<213>人工序列(artificialsequence)
<400>1
metargglymetleuproleuphegluprolysglyargvalleuleu
151015
valaspglyhishisleualatyrargthrphehisalaleulysgly
202530
leuthrthrserargglygluprovalglnalavaltyrglypheala
354045
lysserleuleulysalaleulysgluaspglyaspalavalileval
505560
valpheaspalalysalaproserphearghisglualatyrglygly
65707580
tyrlysalaglyargalaprothrprogluasppheproargglnleu
859095
alaleuilelysgluleuvalaspleuleuglyleualaargleuglu
100105110
valproglytyrglualaaspaspvalleualaserleualalyslys
115120125
alaglulysgluglytyrgluvalargileleuthralaasplysasp
130135140
leutyrglnleuleuseraspargilehisvalleuhisproglugly
145150155160
tyrleuilethrproalatrpleutrpglulystyrglyleuargpro
165170175
aspglntrpalaasptyrargalaleuthrglyaspgluseraspasn
180185190
leuproglyvallysglyileglyglulysthralaarglysleuleu
195200205
gluglutrpglyserleuglualaleuleulysasnleuaspargleu
210215220
lysproalaileargglulysileleualahismetaspaspleulys
225230235240
leusertrpaspleualalysvalargthraspleuproleugluval
245250255
aspphealalysargarggluproasparggluargleuargalaphe
260265270
leugluargleuglupheglyserleuleuhisglupheglyleuleu
275280285
gluserprolysalaleugluglualaprotrpproproproglugly
290295300
alaphevalglyphevalleuserarglysglupromettrpalaasp
305310315320
leuleualaleualaalaalaargglyglyargvalhisargalapro
325330335
gluprotyrlysalaleuargaspleulysglualaargglyleuleu
340345350
alalysaspleuservalleualaleuarggluglyleuglyleupro
355360365
proglyaspaspprometleuleualatyrleuleuaspproserasn
370375380
thrthrprogluglyvalalaargargtyrglyglyglutrpthrglu
385390395400
glualaglygluargalaalaleusergluargleuphealaasnleu
405410415
trpglyargleugluglyglugluargleuleutrpleutyrargglu
420425430
valgluargproleuseralavalleualahismetglualathrgly
435440445
valargleuaspvalalatyrleuargalaleuserleugluvalala
450455460
glugluilealaargleuglualagluvalpheargleualaglyhis
465470475480
propheasnleuasnserargaspglnleugluargvalleupheasp
485490495
gluleuglyleuproalaileglylysthrglulysthrglylysarg
500505510
serthrseralaalavalleuglualaleuargglualahisproile
515520525
valglulysileleuglntyrarggluleuthrlysleulysserthr
530535540
tyrileaspproleuproaspleuilehisproargthrglyargleu
545550555560
histhrargpheasnglnthralathralathrglyargleuserser
565570575
seraspproasnleuglnasnileprovalargthrproleuglygln
580585590
argileargargalapheilealaglugluglytrpleuleuvalala
595600605
leuasptyrserglnilegluleuargvalleualahisleusergly
610615620
aspgluasnleuileargvalpheglngluglyargaspilehisthr
625630635640
gluthralasertrpmetpheglyvalproargglualavalasppro
645650655
leumetargargalaalalysthrileasnpheglyvalleutyrgly
660665670
metseralahisargleuserglngluleualaileprotyrgluglu
675680685
alaglnalapheilegluargtyrpheglnserpheprolysvalarg
690695700
alatrpileglulysthrleuglugluglyargargargglytyrval
705710715720
gluthrleupheglyargargargtyrvalproaspleuglualaarg
725730735
vallysservalargglualaalagluargmetalapheasnmetpro
740745750
valglnglythralaalaaspleumetlysleualametvallysleu
755760765
pheproargleugluglumetglyalaargmetleuleuglnvalhis
770775780
aspgluleuvalleuglualaprolysgluargalaglualavalala
785790795800
argleualalysgluvalmetgluglyvaltyrproleualavalpro
805810815
leugluvalgluvalglyileglygluasptrpleuseralalysglu
820825830
<210>2
<211>832
<212>prt
<213>人工序列(artificialsequence)
<400>2
metargglymetleuproleuphegluprolysglyargvalleuleu
151015
valaspglyhishisleualatyrargthrphehisalaleulysgly
202530
leuthrthrserargglygluprovalglnalavaltyrglypheala
354045
lysserleuleulysalaleulysgluaspglyaspalavalileval
505560
valpheaspalalysalaproserphearghisglualatyrglygly
65707580
tyrlysalaglyargalaprothrprogluasppheproargglnleu
859095
alaleuilelysgluleuvalaspleuleuglyleualaargleuglu
100105110
valproglytyrglualaaspaspvalleualaserleualalyslys
115120125
alaglulysgluglytyrgluvalargileleuthralaasplysasp
130135140
leutyrglnleuleuseraspargilehisvalleuhisproglugly
145150155160
tyrleuilethrproalatrpleutrpglulystyrglyleuargpro
165170175
aspglntrpalaasptyrargalaleuthrglyaspgluseraspasn
180185190
leuproglyvallysglyileglyglulysthralaarglysleuleu
195200205
gluglutrpglyserleuglualaleuleulysasnleuaspargleu
210215220
lysproalaileargglulysileleualahismetaspaspleulys
225230235240
leusertrpaspleualalysvalargthraspleuproleugluval
245250255
aspphealalysargarggluproasparggluargleuargalaphe
260265270
leugluargleuglupheglyserleuleuhisglupheglyleuleu
275280285
gluserprolysalaleugluglualaprotrpproproproglugly
290295300
alaphevalglyphevalleuserarglysglupromettrpalaasp
305310315320
leuleualaleualaalaalaargglyglyargvalhisargalapro
325330335
gluprotyrlysalaleuargaspleulysglualaargglyleuleu
340345350
alalysaspleuservalleualaleuarggluglyleuglyleupro
355360365
proglyaspaspprometleuleualatyrleuleuaspproserasn
370375380
thrthrprogluglyvalalaargargtyrglyglyglutrpthrglu
385390395400
glualaglygluargalaalaleusergluargleuphealaasnleu
405410415
trpglyargleugluglyglugluargleuleutrpleutyrargglu
420425430
valgluargproleuseralavalleualahismetglualathrgly
435440445
valargleuaspvalalatyrleuargalaleuserleugluvalala
450455460
glugluilealaargleuglualagluvalpheargleualaglyhis
465470475480
propheasnleuasnserargaspglnleugluargvalleupheasp
485490495
gluleuglyleuproalaileglylysthrlyslysthrglylysarg
500505510
serthrseralaalavalleuglualaleuargglualahisproile
515520525
valglulysileleuglntyrarggluleuthrlysleulysserthr
530535540
tyrileaspproleuproaspleuilehisproargthrglyargleu
545550555560
histhrargpheasnglnthralathralathrglyargleuserser
565570575
seraspproasnleuglnasnileprovalargthrproleuglygln
580585590
argileargargalapheilealaglugluglytrpleuleuvalala
595600605
leuasptyrserglnilegluleuargvalleualahisleusergly
610615620
aspgluasnleuileargvalpheglngluglyargaspilehisthr
625630635640
gluthralasertrpmetpheglyvalproargglualavalasppro
645650655
leumetargargalaalalysthrileasnpheglyvalleutyrgly
660665670
metseralahisargleuserglngluleualaileprotyrgluglu
675680685
alaglnalapheilegluargtyrpheglnserpheprolysvalarg
690695700
alatrpileglulysthrleuglugluglyargargargglytyrval
705710715720
gluthrleupheglyargargargtyrvalproaspleuglualaarg
725730735
vallysservalargglualaalagluargmetalapheasnmetpro
740745750
valglnglythralaalaaspleumetlysleualametvallysleu
755760765
pheproargleugluglumetglyalaargmetleuleuglnvalhis
770775780
aspgluleuvalleuglualaprolysgluargalaglualavalala
785790795800
argleualalysgluvalmetgluglyvaltyrproleualavalpro
805810815
leugluvalgluvalglyileglygluasptrpleuseralalysglu
820825830
<210>3
<211>832
<212>prt
<213>人工序列(artificialsequence)
<400>3
metargglymetleuproleuphegluprolysglyargvalleuleu
151015
valaspglyhishisleualatyrargthrphehisalaleulysgly
202530
leuthrthrserargglygluprovalglnalavaltyrglypheala
354045
lysserleuleulysalaleulysgluaspglyaspalavalileval
505560
valpheaspalalysalaproserphearghisglualatyrglygly
65707580
tyrlysalaglyargalaprothrprogluasppheproargglnleu
859095
alaleuilelysgluleuvalaspleuleuglyleualaargleuglu
100105110
valproglytyrglualaaspaspvalleualaserleualalyslys
115120125
alaglulysgluglytyrgluvalargileleuthralaasplysasp
130135140
leutyrglnleuleuseraspargilehisvalleuhisproglugly
145150155160
tyrleuilethrproalatrpleutrpglulystyrglyleuargpro
165170175
aspglntrpalaasptyrargalaleuthrglyaspgluseraspasn
180185190
leuproglyvallysglyileglyglulysthralaarglysleuleu
195200205
gluglutrpglyserleuglualaleuleulysasnleuaspargleu
210215220
lysproalaileargglulysileleualahismetaspaspleulys
225230235240
leusertrpaspleualalysvalargthraspleuproleugluval
245250255
aspphealalysargarggluproasparggluargleuargalaphe
260265270
leugluargleuglupheglyserleuleuhisglupheglyleuleu
275280285
gluserprolysalaleugluglualaprotrpproproproglugly
290295300
alaphevalglyphevalleuserarglysglupromettrpalaasp
305310315320
leuleualaleualaalaalaargglyglyargvalhisargalapro
325330335
gluprotyrlysalaleuargaspleulysglualaargglyleuleu
340345350
alalysaspleuservalleualaleuarggluglyleuglyleupro
355360365
proglyaspaspprometleuleualatyrleuleuaspproserasn
370375380
thrthrprogluglyvalalaargargtyrglyglyglutrpthrglu
385390395400
glualaglygluargalaalaleusergluargleuphealaasnleu
405410415
trpglyargleugluglyglugluargleuleutrpleutyrargglu
420425430
valgluargproleuseralavalleualahismetglualathrgly
435440445
valargleuaspvalalatyrleuargalaleuserleugluvalala
450455460
glugluilealaargleuglualagluvalpheargleualaglyhis
465470475480
propheasnleuasnserargaspglnleugluargvalleupheasp
485490495
gluleuglyleuproalaileglylysthrlyslysthrglylysarg
500505510
serthrseralaalavalleuglualaleuargglualahisproile
515520525
valglulysileleuglntyrlysgluleuthrlysleulysserthr
530535540
tyrileaspproleuproaspleuilehisproargthrglyargleu
545550555560
histhrargpheasnglnthralathralathrglyargleuserser
565570575
seraspproasnleuglnasnileprovalargthrproleuglygln
580585590
argileargargalapheilealaglugluglytrpleuleuvalala
595600605
leuasptyrserglnilegluleuargvalleualahisleusergly
610615620
aspgluasnleuileargvalpheglngluglyargaspilehisthr
625630635640
gluthralasertrpmetpheglyvalproargglualavalasppro
645650655
leumetargargalaalalysthrileasnpheglyvalleutyrgly
660665670
metseralahisargleuserglngluleualaileprotyrgluglu
675680685
alaglnalapheilegluargtyrpheglnserpheprolysvalarg
690695700
alatrpileglulysthrleuglugluglyargargargglytyrval
705710715720
gluthrleupheglyargargargtyrvalproaspleuglualaarg
725730735
vallysservalargglualaalagluargmetalapheasnmetpro
740745750
valglnglythralaalaaspleumetlysleualametvallysleu
755760765
pheproargleugluglumetglyalaargmetleuleuglnvalhis
770775780
aspgluleuvalleuglualaprolysgluargalaglualavalala
785790795800
argleualalysgluvalmetgluglyvaltyrproleualavalpro
805810815
leugluvalgluvalglyileglygluasptrpleuseralalysglu
820825830
<210>4
<211>832
<212>prt
<213>人工序列(artificialsequence)
<400>4
metargglymetleuproleuphegluprolysglyargvalleuleu
151015
valaspglyhishisleualatyrargthrphehisalaleulysgly
202530
leuthrthrserargglygluprovalglnalavaltyrglypheala
354045
lysserleuleulysalaleulysgluaspglyaspalavalileval
505560
valpheaspalalysalaproserphearghisglualatyrglygly
65707580
tyrlysalaglyargalaprothrprogluasppheproargglnleu
859095
alaleuilelysgluleuvalaspleuleuglyleualaargleuglu
100105110
valproglytyrglualaaspaspvalleualaserleualalyslys
115120125
alaglulysgluglytyrgluvalargileleuthralaasplysasp
130135140
leutyrglnleuleuseraspargilehisvalleuhisproglugly
145150155160
tyrleuilethrproalatrpleutrpglulystyrglyleuargpro
165170175
aspglntrpalaasptyrargalaleuthrglyaspgluseraspasn
180185190
leuproglyvallysglyileglyglulysthralaarglysleuleu
195200205
gluglutrpglyserleuglualaleuleulysasnleuaspargleu
210215220
lysproalaileargglulysileleualahismetaspaspleulys
225230235240
leusertrpaspleualalysvalargthraspleuproleugluval
245250255
aspphealalysargarggluproasparggluargleuargalaphe
260265270
leugluargleuglupheglyserleuleuhisglupheglyleuleu
275280285
gluserprolysalaleugluglualaprotrpproproproglugly
290295300
alaphevalglyphevalleuserarglysglupromettrpalaasp
305310315320
leuleualaleualaalaalaargglyglyargvalhisargalapro
325330335
gluprotyrlysalaleuargaspleulysglualaargglyleuleu
340345350
alalysaspleuservalleualaleuarggluglyleuglyleupro
355360365
proglyaspaspprometleuleualatyrleuleuaspproserasn
370375380
thrthrprogluglyvalalaargargtyrglyglyglutrpthrglu
385390395400
glualaglygluargalaalaleusergluargleuphealaasnleu
405410415
trpglyargleugluglyglugluargleuleutrpleutyrargglu
420425430
valgluargproleuseralavalleualahismetglualathrgly
435440445
valargleuaspvalalatyrleuargalaleuserleugluvalala
450455460
glugluilealaargleuglualagluvalpheargleualaglyhis
465470475480
propheasnleuasnserargaspglnleugluargvalleupheasp
485490495
gluleuglyleuproalaileglylysthrlyslysthrglylysarg
500505510
serthrseralaalavalleuglualaleuargglualahisproile
515520525
valglulysileleuglntyrarggluleuthrlysleulysserthr
530535540
tyrileaspproleuproaspleuilehisproargthrglyargleu
545550555560
histhrargpheasnglnthralathralathrglyargleuserser
565570575
seraspproasnleuglnasnileprovalargthrproleuglygln
580585590
argileargargalapheilealaglugluglytrpleuleuvalala
595600605
leuasptyrserglnilegluleuargvalleualahisleusergly
610615620
aspgluasnleuileargvalpheglngluglyargaspilehisthr
625630635640
gluthralasertrpmetpheglyvalproargglualavalasppro
645650655
leumetargvalalaalalysthrileasnpheglyvalleutyrgly
660665670
metseralahisargleuserglngluleualaileprotyrgluglu
675680685
alaglnalapheilegluargtyrpheglnserpheprolysvalarg
690695700
alatrpileglulysthrleuglugluglyargargargglytyrval
705710715720
gluthrleupheglyargargargtyrvalproaspleuglualaarg
725730735
vallysservalargglualaalagluargmetalapheasnmetpro
740745750
valglnglythralaalaaspleumetlysleualametvallysleu
755760765
pheproargleugluglumetglyalaargmetleuleuglnvalhis
770775780
aspgluleuvalleuglualaprolysgluargalaglualavalala
785790795800
argleualalysgluvalmetgluglyvaltyrproleualavalpro
805810815
leugluvalgluvalglyileglygluasptrpleuseralalysglu
820825830
<210>5
<211>832
<212>prt
<213>人工序列(artificialsequence)
<400>5
metargglymetleuproleuphegluprolysglyargvalleuleu
151015
valaspglyhishisleualatyrargthrphehisalaleulysgly
202530
leuthrthrserargglygluprovalglnalavaltyrglypheala
354045
lysserleuleulysalaleulysgluaspglyaspalavalileval
505560
valpheaspalalysalaproserphearghisglualatyrglygly
65707580
tyrlysalaglyargalaprothrprogluasppheproargglnleu
859095
alaleuilelysgluleuvalaspleuleuglyleualaargleuglu
100105110
valproglytyrglualaaspaspvalleualaserleualalyslys
115120125
alaglulysgluglytyrgluvalargileleuthralaasplysasp
130135140
leutyrglnleuleuseraspargilehisvalleuhisproglugly
145150155160
tyrleuilethrproalatrpleutrpglulystyrglyleuargpro
165170175
aspglntrpalaasptyrargalaleuthrglyaspgluseraspasn
180185190
leuproglyvallysglyileglyglulysthralaarglysleuleu
195200205
gluglutrpglyserleuglualaleuleulysasnleuaspargleu
210215220
lysproalaileargglulysileleualahismetaspaspleulys
225230235240
leusertrpaspleualalysvalargthraspleuproleugluval
245250255
aspphealalysargarggluproasparggluargleuargalaphe
260265270
leugluargleuglupheglyserleuleuhisglupheglyleuleu
275280285
gluserprolysalaleugluglualaprotrpproproproglugly
290295300
alaphevalglyphevalleuserarglysglupromettrpalaasp
305310315320
leuleualaleualaalaalaargglyglyargvalhisargalapro
325330335
gluprotyrlysalaleuargaspleulysglualaargglyleuleu
340345350
alalysaspleuservalleualaleuarggluglyleuglyleupro
355360365
proglyaspaspprometleuleualatyrleuleuaspproserasn
370375380
thrthrprogluglyvalalaargargtyrglyglyglutrpthrglu
385390395400
glualaglygluargalaalaleusergluargleuphealaasnleu
405410415
trpglyargleugluglyglugluargleuleutrpleutyrargglu
420425430
valgluargproleuseralavalleualahismetglualathrgly
435440445
valargleuaspvalalatyrleuargalaleuserleugluvalala
450455460
glugluilealaargleuglualagluvalpheargleualaglyhis
465470475480
propheasnleuasnserargaspglnleugluargvalleupheasp
485490495
gluleuglyleuproalaileglylysglulyslysthrglylysarg
500505510
serthrseralaalavalleuglualaleuargglualahisproile
515520525
valglulysileleuglntyrlysgluleuthrlysleulysserthr
530535540
tyrileaspproleuproaspleuilehisproargthrglyargleu
545550555560
histhrargpheasnglnthralathralathrglyargleuserser
565570575
seraspproasnleuglnasnileprovalargthrproleuglygln
580585590
argileargargalapheilealaglugluglytrpleuleuvalala
595600605
leuasptyrserglnilegluleuargvalleualahisleusergly
610615620
aspgluasnleuileargvalpheglngluglyargaspilehisthr
625630635640
gluthralasertrpmetpheglyvalproargglualavalasppro
645650655
leumetargvalalaalalysthrileasnpheglyvalleutyrgly
660665670
metseralahisargleuserglngluleualaileprotyrgluglu
675680685
alaglnalapheilegluargtyrpheglnserpheprolysvalarg
690695700
alatrpileglulysthrleuglugluglyargargargglytyrval
705710715720
gluthrleupheglyargargargtyrvalproaspleuglualaarg
725730735
vallysservalargglualaalagluargmetalapheasnmetpro
740745750
valglnglythralaalaaspleumetlysleualametvallysleu
755760765
pheproargleugluglumetglyalaargmetleuleuglnvalhis
770775780
aspgluleuvalleuglualaprolysgluargalaglualavalala
785790795800
argleualalysgluvalmetgluglyvaltyrproleualavalpro
805810815
leugluvalgluvalglyileglygluasptrpleuseralalysglu
820825830
<210>6
<211>2499
<212>dna
<213>人工序列(artificialsequence)
<400>6
atgagggggatgctgcccctctttgagcccaagggccgggtcctcctggtggacggccac60
cacctggcctaccgcaccttccacgccctgaagggcctcaccaccagccggggggagccg120
gtgcaggcggtctacggcttcgccaagagcctcctcaaggccctcaaggaggacggggac180
gcggtgatcgtggtctttgacgccaaggccccctccttccgccacgaggcctacgggggg240
tacaaggcgggccgggcccccacgccggaggactttccccggcaactcgccctcatcaag300
gagctggtggacctcctggggctggcgcgcctcgaggtcccgggctacgaggcggacgac360
gtcctggccagcctggccaagaaggcggaaaaggagggctacgaggtccgcatcctcacc420
gccgacaaagacctttaccagctcctttccgaccgcatccacgtcctccaccccgagggg480
tacctcatcaccccggcctggctttgggaaaagtacggcctgaggcccgaccagtgggcc540
gactaccgggccctgaccggggacgagtccgacaaccttcccggggtcaagggcatcggg600
gagaagacggcgaggaagcttctggaggagtgggggagcctggaagccctcctcaagaac660
ctggaccggctgaagcccgccatccgggagaagatcctggcccacatggacgatctgaag720
ctctcctgggacctggccaaggtgcgcaccgacctgcccctggaggtggacttcgccaaa780
aggcgggagcccgaccgggagaggcttagggcctttctggagaggcttgagtttggcagc840
ctcctccacgagttcggccttctggaaagccccaaggccctggaggaggccccctggccc900
ccgccggaaggggccttcgtgggctttgtgctttcccgcaaggagcccatgtgggccgat960
cttctggccctggccgccgccagggggggccgggtccaccgggcccccgagccttataaa1020
gccctcagggacctgaaggaggcgcgggggcttctcgccaaagacctgagcgttctggcc1080
ctgagggaaggccttggcctcccgcccggcgacgaccccatgctcctcgcctacctcctg1140
gacccttccaacaccacccccgagggggtggcccggcgctacggcggggagtggacggag1200
gaggcgggggagcgggccgccctttccgagaggctcttcgccaacctgtgggggaggctt1260
gagggggaggagaggctcctttggctttaccgggaggtggagaggcccctttccgctgtc1320
ctggcccacatggaggccacgggggtgcgcctggacgtggcctatctcagggccttgtcc1380
ctggaggtggccgaggagatcgcccgcctcgaggccgaggtcttccgcctggccggccac1440
cccttcaacctcaactcccgggaccagctggaaagggtcctctttgacgagctagggctt1500
cccgccatcggcaagacggagaagaccggcaagcgctccaccagcgccgccgtcctggag1560
gccctccgcgaggcccaccccatcgtggagaagatcctgcagtaccgggagctcaccaag1620
ctgaagagcacctacattgaccccttgccggacctcatccaccccaggacgggccgcctc1680
cacacccgcttcaaccagacggccacggccacgggcaggctaagtagctccgatcccaac1740
ctccagaacatccccgtccgcaccccgcttgggcagaggatccgccgggccttcatcgcc1800
gaggaggggtggctattggtggccctggactatagccagatagagctcagggtgctggcc1860
cacctctccggcgacgagaacctgatccgggtcttccaggaggggcgggacatccacacg1920
gagaccgccagctggatgttcggcgtcccccgggaggccgtggaccccctgatgcgccgg1980
gcggccaagaccatcaacttcggggtcctctacggcatgtcggcccaccgcctctcccag2040
gagctagccatcccttacgaggaggcccaggccttcattgagcgctactttcagagcttc2100
cccaaggtgcgggcctggattgagaagaccctggaggagggcaggaggcgggggtacgtg2160
gagaccctcttcggccgccgccgctacgtgccagacctagaggcccgggtgaagagcgtg2220
cgggaggcggccgagcgcatggccttcaacatgcccgtccagggcaccgccgccgacctc2280
atgaagctggctatggtgaagctcttccccaggctggaggaaatgggggccaggatgctc2340
cttcaggtccacgacgagctggtcctcgaggccccaaaagagagggcggaggccgtggcc2400
cggctggccaaggaggtcatggagggggtgtatcccctggccgtgcccctggaggtggag2460
gtggggataggggaggactggctctccgccaaggagtga2499
<210>7
<211>2499
<212>dna
<213>人工序列(artificialsequence)
<400>7
atgagggggatgctgcccctctttgagcccaagggccgggtcctcctggtggacggccac60
cacctggcctaccgcaccttccacgccctgaagggcctcaccaccagccggggggagccg120
gtgcaggcggtctacggcttcgccaagagcctcctcaaggccctcaaggaggacggggac180
gcggtgatcgtggtctttgacgccaaggccccctccttccgccacgaggcctacgggggg240
tacaaggcgggccgggcccccacgccggaggactttccccggcaactcgccctcatcaag300
gagctggtggacctcctggggctggcgcgcctcgaggtcccgggctacgaggcggacgac360
gtcctggccagcctggccaagaaggcggaaaaggagggctacgaggtccgcatcctcacc420
gccgacaaagacctttaccagctcctttccgaccgcatccacgtcctccaccccgagggg480
tacctcatcaccccggcctggctttgggaaaagtacggcctgaggcccgaccagtgggcc540
gactaccgggccctgaccggggacgagtccgacaaccttcccggggtcaagggcatcggg600
gagaagacggcgaggaagcttctggaggagtgggggagcctggaagccctcctcaagaac660
ctggaccggctgaagcccgccatccgggagaagatcctggcccacatggacgatctgaag720
ctctcctgggacctggccaaggtgcgcaccgacctgcccctggaggtggacttcgccaaa780
aggcgggagcccgaccgggagaggcttagggcctttctggagaggcttgagtttggcagc840
ctcctccacgagttcggccttctggaaagccccaaggccctggaggaggccccctggccc900
ccgccggaaggggccttcgtgggctttgtgctttcccgcaaggagcccatgtgggccgat960
cttctggccctggccgccgccagggggggccgggtccaccgggcccccgagccttataaa1020
gccctcagggacctgaaggaggcgcgggggcttctcgccaaagacctgagcgttctggcc1080
ctgagggaaggccttggcctcccgcccggcgacgaccccatgctcctcgcctacctcctg1140
gacccttccaacaccacccccgagggggtggcccggcgctacggcggggagtggacggag1200
gaggcgggggagcgggccgccctttccgagaggctcttcgccaacctgtgggggaggctt1260
gagggggaggagaggctcctttggctttaccgggaggtggagaggcccctttccgctgtc1320
ctggcccacatggaggccacgggggtgcgcctggacgtggcctatctcagggccttgtcc1380
ctggaggtggccgaggagatcgcccgcctcgaggccgaggtcttccgcctggccggccac1440
cccttcaacctcaactcccgggaccagctggaaagggtcctctttgacgagctagggctt1500
cccgccatcggcaagacgaaaaagaccggcaagcgctccaccagcgccgccgtcctggag1560
gccctccgcgaggcccaccccatcgtggagaagatcctgcagtaccgggagctcaccaag1620
ctgaagagcacctacattgaccccttgccggacctcatccaccccaggacgggccgcctc1680
cacacccgcttcaaccagacggccacggccacgggcaggctaagtagctccgatcccaac1740
ctccagaacatccccgtccgcaccccgcttgggcagaggatccgccgggccttcatcgcc1800
gaggaggggtggctattggtggccctggactatagccagatagagctcagggtgctggcc1860
cacctctccggcgacgagaacctgatccgggtcttccaggaggggcgggacatccacacg1920
gagaccgccagctggatgttcggcgtcccccgggaggccgtggaccccctgatgcgccgg1980
gcggccaagaccatcaacttcggggtcctctacggcatgtcggcccaccgcctctcccag2040
gagctagccatcccttacgaggaggcccaggccttcattgagcgctactttcagagcttc2100
cccaaggtgcgggcctggattgagaagaccctggaggagggcaggaggcgggggtacgtg2160
gagaccctcttcggccgccgccgctacgtgccagacctagaggcccgggtgaagagcgtg2220
cgggaggcggccgagcgcatggccttcaacatgcccgtccagggcaccgccgccgacctc2280
atgaagctggctatggtgaagctcttccccaggctggaggaaatgggggccaggatgctc2340
cttcaggtccacgacgagctggtcctcgaggccccaaaagagagggcggaggccgtggcc2400
cggctggccaaggaggtcatggagggggtgtatcccctggccgtgcccctggaggtggag2460
gtggggataggggaggactggctctccgccaaggagtga2499
<210>8
<211>2499
<212>dna
<213>人工序列(artificialsequence)
<400>8
atgagggggatgctgcccctctttgagcccaagggccgggtcctcctggtggacggccac60
cacctggcctaccgcaccttccacgccctgaagggcctcaccaccagccggggggagccg120
gtgcaggcggtctacggcttcgccaagagcctcctcaaggccctcaaggaggacggggac180
gcggtgatcgtggtctttgacgccaaggccccctccttccgccacgaggcctacgggggg240
tacaaggcgggccgggcccccacgccggaggactttccccggcaactcgccctcatcaag300
gagctggtggacctcctggggctggcgcgcctcgaggtcccgggctacgaggcggacgac360
gtcctggccagcctggccaagaaggcggaaaaggagggctacgaggtccgcatcctcacc420
gccgacaaagacctttaccagctcctttccgaccgcatccacgtcctccaccccgagggg480
tacctcatcaccccggcctggctttgggaaaagtacggcctgaggcccgaccagtgggcc540
gactaccgggccctgaccggggacgagtccgacaaccttcccggggtcaagggcatcggg600
gagaagacggcgaggaagcttctggaggagtgggggagcctggaagccctcctcaagaac660
ctggaccggctgaagcccgccatccgggagaagatcctggcccacatggacgatctgaag720
ctctcctgggacctggccaaggtgcgcaccgacctgcccctggaggtggacttcgccaaa780
aggcgggagcccgaccgggagaggcttagggcctttctggagaggcttgagtttggcagc840
ctcctccacgagttcggccttctggaaagccccaaggccctggaggaggccccctggccc900
ccgccggaaggggccttcgtgggctttgtgctttcccgcaaggagcccatgtgggccgat960
cttctggccctggccgccgccagggggggccgggtccaccgggcccccgagccttataaa1020
gccctcagggacctgaaggaggcgcgggggcttctcgccaaagacctgagcgttctggcc1080
ctgagggaaggccttggcctcccgcccggcgacgaccccatgctcctcgcctacctcctg1140
gacccttccaacaccacccccgagggggtggcccggcgctacggcggggagtggacggag1200
gaggcgggggagcgggccgccctttccgagaggctcttcgccaacctgtgggggaggctt1260
gagggggaggagaggctcctttggctttaccgggaggtggagaggcccctttccgctgtc1320
ctggcccacatggaggccacgggggtgcgcctggacgtggcctatctcagggccttgtcc1380
ctggaggtggccgaggagatcgcccgcctcgaggccgaggtcttccgcctggccggccac1440
cccttcaacctcaactcccgggaccagctggaaagggtcctctttgacgagctagggctt1500
cccgccatcggcaagacgaaaaagaccggcaagcgctccaccagcgccgccgtcctggag1560
gccctccgcgaggcccaccccatcgtggagaagatcctgcagtacaaggagctcaccaag1620
ctgaagagcacctacattgaccccttgccggacctcatccaccccaggacgggccgcctc1680
cacacccgcttcaaccagacggccacggccacgggcaggctaagtagctccgatcccaac1740
ctccagaacatccccgtccgcaccccgcttgggcagaggatccgccgggccttcatcgcc1800
gaggaggggtggctattggtggccctggactatagccagatagagctcagggtgctggcc1860
cacctctccggcgacgagaacctgatccgggtcttccaggaggggcgggacatccacacg1920
gagaccgccagctggatgttcggcgtcccccgggaggccgtggaccccctgatgcgccgg1980
gcggccaagaccatcaacttcggggtcctctacggcatgtcggcccaccgcctctcccag2040
gagctagccatcccttacgaggaggcccaggccttcattgagcgctactttcagagcttc2100
cccaaggtgcgggcctggattgagaagaccctggaggagggcaggaggcgggggtacgtg2160
gagaccctcttcggccgccgccgctacgtgccagacctagaggcccgggtgaagagcgtg2220
cgggaggcggccgagcgcatggccttcaacatgcccgtccagggcaccgccgccgacctc2280
atgaagctggctatggtgaagctcttccccaggctggaggaaatgggggccaggatgctc2340
cttcaggtccacgacgagctggtcctcgaggccccaaaagagagggcggaggccgtggcc2400
cggctggccaaggaggtcatggagggggtgtatcccctggccgtgcccctggaggtggag2460
gtggggataggggaggactggctctccgccaaggagtga2499
<210>9
<211>2499
<212>dna
<213>人工序列(artificialsequence)
<400>9
atgagggggatgctgcccctctttgagcccaagggccgggtcctcctggtggacggccac60
cacctggcctaccgcaccttccacgccctgaagggcctcaccaccagccggggggagccg120
gtgcaggcggtctacggcttcgccaagagcctcctcaaggccctcaaggaggacggggac180
gcggtgatcgtggtctttgacgccaaggccccctccttccgccacgaggcctacgggggg240
tacaaggcgggccgggcccccacgccggaggactttccccggcaactcgccctcatcaag300
gagctggtggacctcctggggctggcgcgcctcgaggtcccgggctacgaggcggacgac360
gtcctggccagcctggccaagaaggcggaaaaggagggctacgaggtccgcatcctcacc420
gccgacaaagacctttaccagctcctttccgaccgcatccacgtcctccaccccgagggg480
tacctcatcaccccggcctggctttgggaaaagtacggcctgaggcccgaccagtgggcc540
gactaccgggccctgaccggggacgagtccgacaaccttcccggggtcaagggcatcggg600
gagaagacggcgaggaagcttctggaggagtgggggagcctggaagccctcctcaagaac660
ctggaccggctgaagcccgccatccgggagaagatcctggcccacatggacgatctgaag720
ctctcctgggacctggccaaggtgcgcaccgacctgcccctggaggtggacttcgccaaa780
aggcgggagcccgaccgggagaggcttagggcctttctggagaggcttgagtttggcagc840
ctcctccacgagttcggccttctggaaagccccaaggccctggaggaggccccctggccc900
ccgccggaaggggccttcgtgggctttgtgctttcccgcaaggagcccatgtgggccgat960
cttctggccctggccgccgccagggggggccgggtccaccgggcccccgagccttataaa1020
gccctcagggacctgaaggaggcgcgggggcttctcgccaaagacctgagcgttctggcc1080
ctgagggaaggccttggcctcccgcccggcgacgaccccatgctcctcgcctacctcctg1140
gacccttccaacaccacccccgagggggtggcccggcgctacggcggggagtggacggag1200
gaggcgggggagcgggccgccctttccgagaggctcttcgccaacctgtgggggaggctt1260
gagggggaggagaggctcctttggctttaccgggaggtggagaggcccctttccgctgtc1320
ctggcccacatggaggccacgggggtgcgcctggacgtggcctatctcagggccttgtcc1380
ctggaggtggccgaggagatcgcccgcctcgaggccgaggtcttccgcctggccggccac1440
cccttcaacctcaactcccgggaccagctggaaagggtcctctttgacgagctagggctt1500
cccgccatcggcaagacgaaaaagaccggcaagcgctccaccagcgccgccgtcctggag1560
gccctccgcgaggcccaccccatcgtggagaagatcctgcagtaccgggagctcaccaag1620
ctgaagagcacctacattgaccccttgccggacctcatccaccccaggacgggccgcctc1680
cacacccgcttcaaccagacggccacggccacgggcaggctaagtagctccgatcccaac1740
ctccagaacatccccgtccgcaccccgcttgggcagaggatccgccgggccttcatcgcc1800
gaggaggggtggctattggtggccctggactatagccagatagagctcagggtgctggcc1860
cacctctccggcgacgagaacctgatccgggtcttccaggaggggcgggacatccacacg1920
gagaccgccagctggatgttcggcgtcccccgggaggccgtggaccccctgatgcgcgtg1980
gcggccaagaccatcaacttcggggtcctctacggcatgtcggcccaccgcctctcccag2040
gagctagccatcccttacgaggaggcccaggccttcattgagcgctactttcagagcttc2100
cccaaggtgcgggcctggattgagaagaccctggaggagggcaggaggcgggggtacgtg2160
gagaccctcttcggccgccgccgctacgtgccagacctagaggcccgggtgaagagcgtg2220
cgggaggcggccgagcgcatggccttcaacatgcccgtccagggcaccgccgccgacctc2280
atgaagctggctatggtgaagctcttccccaggctggaggaaatgggggccaggatgctc2340
cttcaggtccacgacgagctggtcctcgaggccccaaaagagagggcggaggccgtggcc2400
cggctggccaaggaggtcatggagggggtgtatcccctggccgtgcccctggaggtggag2460
gtggggataggggaggactggctctccgccaaggagtga2499
<210>10
<211>2499
<212>dna
<213>人工序列(artificialsequence)
<400>10
atgagggggatgctgcccctctttgagcccaagggccgggtcctcctggtggacggccac60
cacctggcctaccgcaccttccacgccctgaagggcctcaccaccagccggggggagccg120
gtgcaggcggtctacggcttcgccaagagcctcctcaaggccctcaaggaggacggggac180
gcggtgatcgtggtctttgacgccaaggccccctccttccgccacgaggcctacgggggg240
tacaaggcgggccgggcccccacgccggaggactttccccggcaactcgccctcatcaag300
gagctggtggacctcctggggctggcgcgcctcgaggtcccgggctacgaggcggacgac360
gtcctggccagcctggccaagaaggcggaaaaggagggctacgaggtccgcatcctcacc420
gccgacaaagacctttaccagctcctttccgaccgcatccacgtcctccaccccgagggg480
tacctcatcaccccggcctggctttgggaaaagtacggcctgaggcccgaccagtgggcc540
gactaccgggccctgaccggggacgagtccgacaaccttcccggggtcaagggcatcggg600
gagaagacggcgaggaagcttctggaggagtgggggagcctggaagccctcctcaagaac660
ctggaccggctgaagcccgccatccgggagaagatcctggcccacatggacgatctgaag720
ctctcctgggacctggccaaggtgcgcaccgacctgcccctggaggtggacttcgccaaa780
aggcgggagcccgaccgggagaggcttagggcctttctggagaggcttgagtttggcagc840
ctcctccacgagttcggccttctggaaagccccaaggccctggaggaggccccctggccc900
ccgccggaaggggccttcgtgggctttgtgctttcccgcaaggagcccatgtgggccgat960
cttctggccctggccgccgccagggggggccgggtccaccgggcccccgagccttataaa1020
gccctcagggacctgaaggaggcgcgggggcttctcgccaaagacctgagcgttctggcc1080
ctgagggaaggccttggcctcccgcccggcgacgaccccatgctcctcgcctacctcctg1140
gacccttccaacaccacccccgagggggtggcccggcgctacggcggggagtggacggag1200
gaggcgggggagcgggccgccctttccgagaggctcttcgccaacctgtgggggaggctt1260
gagggggaggagaggctcctttggctttaccgggaggtggagaggcccctttccgctgtc1320
ctggcccacatggaggccacgggggtgcgcctggacgtggcctatctcagggccttgtcc1380
ctggaggtggccgaggagatcgcccgcctcgaggccgaggtcttccgcctggccggccac1440
cccttcaacctcaactcccgggaccagctggaaagggtcctctttgacgagctagggctt1500
cccgccatcggcaagacgaaaaagaccggcaagcgctccaccagcgccgccgtcctggag1560
gccctccgcgaggcccaccccatcgtggagaagatcctgcagtacaaggagctcaccaag1620
ctgaagagcacctacattgaccccttgccggacctcatccaccccaggacgggccgcctc1680
cacacccgcttcaaccagacggccacggccacgggcaggctaagtagctccgatcccaac1740
ctccagaacatccccgtccgcaccccgcttgggcagaggatccgccgggccttcatcgcc1800
gaggaggggtggctattggtggccctggactatagccagatagagctcagggtgctggcc1860
cacctctccggcgacgagaacctgatccgggtcttccaggaggggcgggacatccacacg1920
gagaccgccagctggatgttcggcgtcccccgggaggccgtggaccccctgatgcgcgtg1980
gcggccaagaccatcaacttcggggtcctctacggcatgtcggcccaccgcctctcccag2040
gagctagccatcccttacgaggaggcccaggccttcattgagcgctactttcagagcttc2100
cccaaggtgcgggcctggattgagaagaccctggaggagggcaggaggcgggggtacgtg2160
gagaccctcttcggccgccgccgctacgtgccagacctagaggcccgggtgaagagcgtg2220
cgggaggcggccgagcgcatggccttcaacatgcccgtccagggcaccgccgccgacctc2280
atgaagctggctatggtgaagctcttccccaggctggaggaaatgggggccaggatgctc2340
cttcaggtccacgacgagctggtcctcgaggccccaaaagagagggcggaggccgtggcc2400
cggctggccaaggaggtcatggagggggtgtatcccctggccgtgcccctggaggtggag2460
gtggggataggggaggactggctctccgccaaggagtga2499
<210>11
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>11
ggggtacctcatcaccccgg20
<210>12
<211>27
<212>dna
<213>人工序列(artificialsequence)
<400>12
cttggtgagctccttgtactgcaggat27
<210>13
<211>27
<212>dna
<213>人工序列(artificialsequence)
<400>13
atcctgcagtacaaggagctcaccaag27
<210>14
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>14
gatggtcttggccgccacgcgcatcagggg30
<210>15
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>15
cccctgatgcgcgtggcggccaagaccatc30
<210>16
<211>32
<212>dna
<213>人工序列(artificialsequence)
<400>16
gctctagactatcactccttggcggagagcca32
<210>17
<211>27
<212>dna
<213>人工序列(artificialsequence)
<400>17
cttgccggtctttttcgtcttgccgat27
<210>18
<211>27
<212>dna
<213>人工序列(artificialsequence)
<400>18
atcggcaagacgaaaaagaccggcaag27
<210>19
<211>25
<212>dna
<213>人工序列(artificialsequence)
<400>19
ccagtgttaggttatttctaacttg25
<210>20
<211>19
<212>dna
<213>人工序列(artificialsequence)
<400>20
gctcggagcacatggtcaa19
<210>21
<211>19
<212>dna
<213>人工序列(artificialsequence)
<400>21
gctcggagcacatggtcag19
<210>22
<211>22
<212>dna
<213>人工序列(artificialsequence)
<400>22
tgaagagcaggaaagttcttca22
<210>23
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>23
actgtgtgtctgaaacagtg20
<210>24
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>24
actgtgtgtctgaaacagta20
<210>25
<211>26
<212>dna
<213>人工序列(artificialsequence)
<400>25
gtaagtctttgctgagaaattcattg26
<210>26
<211>26
<212>dna
<213>人工序列(artificialsequence)
<400>26
gtaagtctttgctgagaaattcattt26
<210>27
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>27
agtatccagggttaatgtgaaag23
<210>28
<211>25
<212>dna
<213>人工序列(artificialsequence)
<400>28
agatatttgtaaggtattctggcct25
<210>29
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>29
tgctgaacaaatagtcccgaccag24
<210>30
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>30
tttctctagttgcctttaagattt24
- 还没有人留言评论。精彩留言会获得点赞!