蛋白TaNADH-GoGAT在调控植物产量中的应用的制作方法
本发明涉及生物技术领域,具体涉及蛋白tanadh-gogat在调控植物产量中应用。
背景技术
小麦是三大谷物之一,几乎全作食用,仅约有六分之一作为饲料使用。两河流域是世界上最早栽培小麦的地区,中国是世界最早种植小麦的国家之一。2010年小麦是世界上总产量位居第二的粮食作物(6.51亿吨),仅次于玉米(8.44亿吨),约有40%的人口以小麦为主粮,小麦的营养丰富,小麦磨成面粉后可制作馒头、面包、饼干等食物。
小麦营养价值很高,所含的b族维生素和矿物质对人体健康很有益处。近年来随着人民生活水平的提高,社会对优质专用小麦的需求量越来越大,优质小麦种植面积迅速增加,随着加工企业专用粉产品升级和加工工艺不断现代化,现代制粉行业对大批量商品小麦的品质及稳定性提出了越来越高的要求,为生产出高蛋白质含量的小麦,不合理的施用化肥,不但导致土壤板结,肥料利用率低,而且流失污染严重,极大的影响了小麦的产量和质量,同时对环境危害较大。因此,通过基因工程等生物手段培育高产的优良小麦品种将具有重要的意义。
技术实现要素:
本发明的目的是提供一种蛋白质tanadh-gogat在调控植物产量中应用。
第一方面,本发明要求保护tanadh-gogat蛋白或其相关生物材料在调控植物产量中的应用。
所述相关生物材料可为能够表达所述tanadh-gogat蛋白的核酸分子或含有所述核酸分子的表达盒、重组载体、重组菌或转基因细胞系。
进一步地,所述tanadh-gogat蛋白或其编码基因在所述植物中的表达量和/或活性提高,所述植物产量提高;所述tanadh-gogat蛋白或其编码基因在所述植物中的表达量和/或活性降低,所述植物产量降低。
其中,所述植物产量可体现为如下中至少一种:
(a1)植物单株籽粒产量;
(a2)植物每株穗数;
(a3)植物单株生物量(干重);
(a4)植物单穗生物量(干重)。
第二方面,本发明要求保护一种培育产量提高的植物品种的方法。
本发明所提供的培育产量提高的植物品种的方法,可包括使受体植物中tanadh-gogat蛋白的表达量和/或活性提高的步骤。其中,所述产量提高可体现为单株籽粒产量提高和/或每株穗数增加和/或单株生物量提高和/或单穗生物量提高。
进一步,本发明提供了一种培育产量提高的转基因植物的方法。
本发明所提供的培育产量提高的转基因植物的方法,具体可包括如下步骤:向受体植物中导入能够表达tanadh-gogat蛋白的核酸分子,得到转基因植物;所述转基因植物与所述受体植物相比产量提高。其中,所述产量提高可体现为单株籽粒产量提高和/或每株穗数增加和/或单株生物量提高和/或单穗生物量提高。
第三方面,本发明要求保护一种培育产量降低的植物品种的方法。
本发明所提供的培育产量降低的植物品种的方法,可包括使受体植物中tanadh-gogat蛋白的表达量和/或活性降低的步骤。其中,所述产量降低可体现为单株籽粒产量降低和/或每株穗数减少和/或单株生物量降低和/或单穗生物量降低。
进一步,本发明提供了一种培育产量降低的转基因植物的方法。
本发明所提供的培育产量降低的转基因植物的方法,具体可包括如下步骤:对受体植物中tanadh-gogat蛋白的编码基因进行抑制表达,得到转基因植物;所述转基因植物与所述受体植物相比产量降低。其中,所述产量降低可体现为单株籽粒产量降低和/或每株穗数减少和/或单株生物量降低和/或单穗生物量降低。
在第二方面中,所述“向受体植物中导入能够表达所述tanadh-gogat蛋白的核酸分子”可通过向所述受体植物中导入含有所述tanadh-gogat蛋白的编码基因的重组表达载体实现。
所述重组表达载体可用现有的植物表达载体构建。所述植物表达载体包括双元农杆菌载体和可用于植物微弹轰击的载体等,如pcambia-1300-221、pgreen0029、pcambia3301、pcambia1300、pbi121、pbin19、pcambia2301、pcambia1301-ubin或其它衍生植物表达载体。所述植物表达载体还可包含外源基因的3’端非翻译区域,即包含聚腺苷酸信号和任何其它参与mrna加工或基因表达的dna片段。所述聚腺苷酸信号可引导聚腺苷酸加入到mrna前体的3’端。使用所述基因构建重组表达载体时,在其转录起始核苷酸前可加上任何一种增强型、组成型、组织特异型或诱导型启动子,例如花椰菜花叶病毒(camv)35s启动子、泛素基因ubiquitin启动子(pubi)、胁迫诱导型启动子rd29a等,它们可单独使用或与其它的植物启动子结合使用;此外,使用本发明的基因构建重组表达载体时,还可使用增强子,包括翻译增强子或转录增强子,这些增强子区域可以是atg起始密码子或邻接区域起始密码子等,但必需与编码序列的阅读框相同,以保证整个序列的正确翻译。所述翻译控制信号和起始密码子的来源是广泛的,可以是天然的,也可以是合成的。翻译起始区域可以来自转录起始区域或结构基因。为了便于对转基因植物细胞或植物进行鉴定及筛选,可对所用重组表达载体进行加工,如加入可在植物中表达的编码可产生颜色变化的酶或发光化合物的基因、具有抗性的抗生素标记物或是抗化学试剂标记基因等。也可不加任何选择性标记基因,直接以逆境筛选转化植株。
在本发明中,所述重组载体中启动所述tanadh-gogat蛋白的编码基因转录的启动子为ubi启动子。
更加具体的,所述重组表达载体为将所述tanadh-gogat蛋白的编码基因插入到pubi-163载体的多克隆位点处(hindiii和ecori)所得的重组质粒。
在第三方面中,所述“对受体植物中tanadh-gogat蛋白的编码基因进行抑制表达”可通过向所述受体植物中导入含有如式(i)所示的dna片段的干扰载体实现;
seq正向-x-seq反向(i)
所述seq正向的序列为seqidno.3的第1-426位;
所述seq反向的序列与所述seq正向的序列反向互补(具体为seqidno.3的第608-1033位);
所述x是所述seq正向与所述seq反向之间的间隔序列,在序列上,所述x与所述seq正向及所述seq反向均不互补。
在本发明中,式(i)中所述x具体如seqidno.3的第433-601位所示。
更加具体的,式(i)所示的dna片段的核苷酸序列如seqidno.3所示。
在本发明的具体实施方式中,所述干扰载体具体为将所述如式(i)所示的dna片段插入到pubi-163载体的多克隆位点处(bamhi和kpni)所得的重组质粒。
在上述方法中,将携带有所述tanadh-gogat蛋白的编码基因的所述重组表达载体或者携带有所述如式(i)所示的dna片段的所述的干扰载体导入所述受体植物,具体可为:通过使用ti质粒、ri质粒、植物病毒载体、直接dna转化、显微注射、电导、农杆菌介导等常规生物学方法转化植物细胞或组织,并将转化的植物组织培育成植株。
在第一方面、第二方面和第三方面中,所述tanadh-gogat蛋白均可为如下任一所示蛋白质:
(a1)氨基酸序列为seqidno.1的蛋白质;
(a2)将seqidno.1所示的氨基酸序列经过一个或几个氨基酸残基的取代和/或缺失和/或添加且具有相同功能的蛋白质;
(a3)与(a1)-(a2)中任一所限定的氨基酸序列具有99%以上、95%以上、90%以上、85%以上或者80%以上同源性且具有相同功能的蛋白质;
(a4)在(a1)-(a3)中任一所限定的蛋白质的n端和/或c端连接标签后得到的融合蛋白。
相应的,在第一方面、第二方面和第三方面中,所述“能够表达所述tanadh-gogat蛋白的核酸分子”均可为所述tanadh-gogat蛋白的编码基因。更进一步,所述tanadh-gogat蛋白的编码基因具体可如下任一所述的dna分子:
(b1)seqidno.2所示的dna分子;
(b2)在严格条件下与(b1)限定的dna分子杂交且编码所述tanadh-gogat蛋白的dna分子;
(b3)与(b1)或(b2)限定的dna序列具有99%以上、95%以上、90%以上、85%以上或者80%以上同源性且编码所述tanadh-gogat蛋白的dna分子。
上述严格条件可为用6×ssc,0.5%sds的溶液,在65℃下杂交,然后用2×ssc,0.1%sds和1×ssc,0.1%sds各洗膜一次。
在第一方面、第二方面和第三方面中,所述植物均可为单子叶植物,也可为双子叶植物。
进一步地,所述单子叶植物可为禾本科植物。
更进一步地,所述禾本科植物可为小麦。
在本发明的具体实施方式中,所述植物具体为小麦品种kn199(科农199)。
实验证明,本发明所提供的蛋白质tanadh-gogat调控植物产量:与野生型植物相比,tanadh-gogat超量表达转基因植物的单株籽粒产量、每株穗数和单株生物量均显著提高;tanadh-gogat减量表达转基因植物的单株籽粒产量、每株穗数和单株生物量均显著降低。因此,可以利用蛋白质tanadh-gogat调控植物产量。本发明对选育高产植物新材料的具有重要应用价值。
附图说明
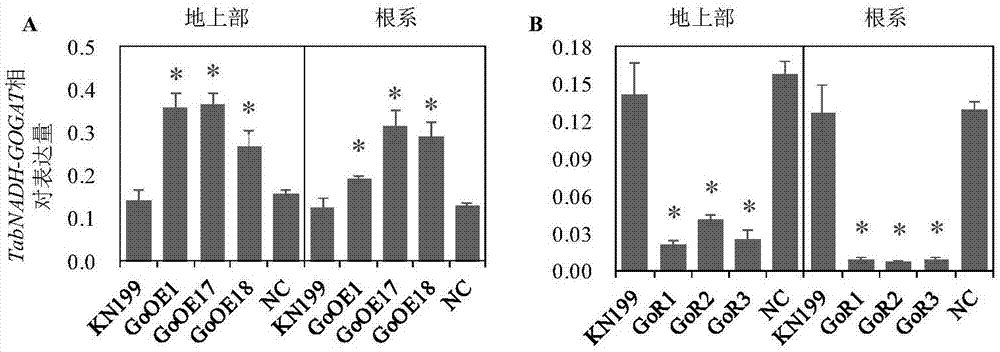
图1为tanadh-gogat转基因系的表达量鉴定。其中,a为tanadh-gogat超量表达鉴定。b为tanadh-gogat减量表达鉴定。nc代表空载对照。图中表达量是相对于taactin的表达量。图中数据为3个重复的平均值±s.e.。*代表差异达到p<0.05显著性。
图2为2015-2016生长季tanadh-gogat超量表达系的表型及产量相关性状的测定。其中,a为单株籽粒产量。b为穗粒数。c为千粒重。d为单株穗数。nc代表空载对照。图中数据是3个重复的平均值±s.e.。*代表差异达到p<0.05显著性。
图3为2016-2017生长季tanadh-gogat转基因系的表型及产量相关性状的测定。其中,a为植株生物量。b为籽粒产量。gooe1、gooe17和gooe18为超量表达系,gooe1-、gooe17-和gooe18-为对应的阴性系,gor1、gor2和gor3为减量表达系,gor1-、gor2-和gor3-为对应的阴性系。图中数据是3个重复的平均值±s.e.。*代表差异达到p<0.05显著性。
具体实施方式
下述实施例中所使用的实验方法如无特殊说明,均为常规方法。
下述实施例中所用的材料、试剂等,如无特殊说明,均可从商业途径得到。
小麦品种kn199(科农199):遗传所李俊明培育的品种,公众可从商业途径得到或从中国科学院遗传与发育生物学研究所获得。
pubi-163载体:记载于“shao,a.,ma,w.y.,zhao,x.q.,hu,m.y.,he,x.,teng,w.,li,h.,andtong,y.p.(2017).theauxinbiosynthetictryptophanaminotransferaserelatedtatar2.1-3aincreasesgrainyieldofwheat.plantphysiol.174,2274-2288.”一文,公众可从申请人处获得,仅可用于重复本发明实验使用。
实施例1、转tanadh-gogat基因小麦的构建
一、转tanadh-gogat基因植株的制备
(一)tanadh-gogat基因的获得
1、提取小麦品种kn199的总rna,反转录得到其基因组cdna。
2、以步骤1得到的cdna为模板,以如下引物为引物进行pcr扩增构建超量表达tanadh-gogat转基因系小麦所需序列:
tanadh-gogat-oe-f:5’-aagcttatgccgacggcgcaggggattgg-3’(下划线所示序列为hindiii酶切识别位点);
tanadh-gogat-oe-r:5’-gaattcctatgtaaggcctgcgacattttg-3’(下划线所示序列为ecori酶切识别位点)。
以如下引物为引物进行pcr扩增构建减量表达tanadh-gogat转基因系小麦所需序列:
tanadh-gogat-rnai-f1:5’-ggatccggattggtttgaagcacg-3’(下划线所示序列为bamhⅰ酶切识别位点);
tanadh-gogat-rnai-r1:5’-gaattcaggtgtgtggtagagcaacg-3’(下划线所示序列为ecorⅰ酶切识别位点);
tanadh-gogat-rnai-f2:5’-ggtaccggattggtttgaagcacg-3’(下划线所示序列为kpni酶切识别位点);
tanadh-gogat-rnai-r2:5’-aagcttaggtgtgtggtagagcaacg-3’(下划线所示序列为hindiii酶切识别位点)。
pcr体系:模板cdna4μl,kodplusdna聚合酶1μl,10×pcrbufferforkodplus4μl,dntps(2mmeach)4μl,25mm的mgso42μl,上下游引物各20mm,再用双蒸水将反应体系补足至40μl。
pcr反应程序:98℃2min;98℃30sec,58℃30sec,68℃45sec,38个循环。
用于超量表达tanadh-gogat基因的pcr产物(记为pcr产物1)的核苷酸序列为“5’-aagctt+seqidno.2+gaattc-3’”。其中,seqidno.2为tanadh-gogat基因的cdna序列(编码seqidno.1所示的tanadh-gogat蛋白)。
减量表达tanadh-gogat基因的pcr产物有两种,其一记为pcr产物2,另一记为pcr产物3;其中pcr产物2的核苷酸序列为“5’-ggatcc+seqidno.2的第17-442位+gaattc-3’”,pcr产物3的核苷酸序列为“5’-ggtacc+seqidno.2的第17-442位+aagctt-3’”。
(二)tanadh-gogat基因克隆载体的构建
用hindiii和ecori双酶切上述pcr产物1,得到基因片段;hindiii和ecori双酶切pubi-163载体,得到载体大片段,将基因片段与载体大片段连接,得到重组质粒,将其命名为pubi-163-tanadh-gogat,将重组质粒送测序,结果正确。pubi-163-tanadh-gogat中tanadh-gogat基因由ubi启动子启动。pubi-163-tanadh-gogat载体的结构描述:将seqidno.2所示dna片段替换pubi-163载体的酶切位点hindiii和ecori之间的小片段后所得的重组质粒。
用bamhⅰ和ecorⅰ双酶切上述pcr产物2,得到基因片段s1,用kpni和hindiii双酶切上述pcr产物3,得到基因片段s2,用ecorⅰ和kpni酶切pubi-163载体回收载体内含子片段s3,用bamhi和kpni酶切pubi-163载体回收载体骨架v1。然后将v1、s3分别与s1、s2连接构建成pubi-tanadh-gogat-rnai载体。测序并酶切验证。
pubi-tanadh-gogat-rnai载体的结构描述:将seqidno.3所示dna片段替换pubi-163载体的酶切位点bamhⅰ和kpni之间的小片段后所得的重组质粒。seqidno.3的第1-426位为茎环结构双链部分的正向序列,第427-432位为ecori的识别序列,第433-601位为内含子序列,第602-607位为hindiii的识别序列,第608-1033位为茎环结构双链部分的反向序列。
(三)转基因小麦的获得
将pubi-163-tanadh-gogat载体和pubi-tanadh-gogat-rnai载体用基因枪的方法分别转入野生型小麦kn199中,得到t0代tanadh-gogat超量表达和减量表达转基因小麦。提取t0代tanadh-gogat超量表达和减量表达转基因小麦叶片的基因组dna,以其作为模板,用各自的上游引物和下游引物进行pcr扩增,得到1200bp和800bp左右的片段,即为阳性t0代tanadh-gogat超量表达和减量表达转基因小麦。
用于鉴定tanadh-gogat超量表达转基因小麦的引物:
上游引物t-oepf:5’-tcggagtagaatactgtttcaaactacc-3’(载体引物);
下游引物t-oepr:5’-agcatctgaatggcatcgac-3’。
用于鉴定tanadh-gogat减量表达转基因小麦的引物:
上游引物t-rnaipf:5'-tcggagtagaatactgtttcaaactacc-3'(载体引物);
下游引物t-rnaipr:5'-aatggtgatcatccagctctc-3'(载体引物)。
将上述鉴定为阳性的t0代转tanadh-gogat超量表达和减量表达转基因小麦培养至t2代,t1-t2代按照t0代的鉴定方法进行鉴定,收获种子。
实验同时设置向野生型小麦品种kn199中导入pubi-163载体的空载对照(下文简称空载对照植株)。
二、转基因植株的检测
(一)dna水平检测
分别提取t2代tanadh-gogat超量表达小麦、tanadh-gogat减量表达小麦以及野生型小麦kn199的叶片的dna,分别以其为模板,以t-oepf和t-oepr(具体序列同上)为引物鉴定tanadh-gogat超量表达系,以t-rnaipf和t-rnaipr为引物(具体序列同上)鉴定tanadh-gogat减量表达系,同时以各自的载体为阳性对照(pc),以野生型小麦kn199号作为阴性对照(wt)。
pcr反应体系:dna模板(约20ng/μl)2μl;正向引物(10μm)0.5μl;反向引物(10μm)0.5μl;10×pcr扩增缓冲液2μl;dntpmixture1μl;taqdna聚合酶0.2μl;ddh2o补足至20μl。
pcr反应程序:94℃,3min;94℃30s,60℃30s,72℃40s,40个循环;72℃5min。
tanadh-gogat超量表达小麦的目的pcr扩增条带为1200bp左右,tanadh-gogat减量表达小麦的目的pcr扩增条带为800bp左右。野生型小麦kn199没有目的条带,三个t2代tanadh-gogat超量表达小麦株系(gooe1、gooe17和gooe18)和三个t2代tanadh-gogat减量表达小麦株系(gor1、gor2和gor3)经初步鉴定为阳性小麦。
(二)rna水平检测
1、分别提取t2代tanadh-gogat超量表达小麦、tanadh-gogat减量表达小麦以及野生型小麦kn199的茎和根的总rna,并反转录成cdna。
2、分别以步骤1得到的cdna为模板,以tanadh-gogatrtpf和tanadh-gogatrtpr为引物进行rt-pcr,扩增tanadh-gogat基因,同时以taactinpf和taactinpr为引物进行rt-pcr,扩增内参基因taactin。
引物如下:
上游引物tanadh-gogatrtpf:5’-gcggaagtgccataccaatac-3’;
下游引物tanadh-gogatrtpr:5’-aaagttaatgacatgctctggttctc-3’;
上游引物taactinpf:5'-accttcagttgcccagcaat-3';
下游引物taactinpr:5'-cagagtcgagcacaataccagttg-3'。
pcr体系:dna模板(约20ng/μl)2μl;上游引物(10μm)0.4μl;下游引物(10μm)0.4μl;2×mixture(lightcyclersybrgreenimaster,roche)10μl;ddh2o补足至20μl。
pcr程序:94℃,5min;94℃20s,60℃20s,72℃15s,45个循环。
定量分析:采用rochelightcycler480ⅱrealtimepcr仪分析其ct值。以taactin基因为内参,t2代tanadh-gogat转基因小麦和野生型小麦kn199中的tanadh-gogat基因用2-δct进行相对定量。
gooe1、gooe17和gooe18三个t2代tanadh-gogat超量表达小麦以及gor1、gor2和gor3三个t2代tanadh-gogat减量表达小麦中tanadh-gogat基因的检测结果如图1所示。
图1表明,与野生型小麦kn199相比,gooe1、gooe17和gooe18三个t2代tanadh-gogat超量表达小麦中tanadh-gogat基因的表达量显著增加。而gor1、gor2和gor3三个t2代tanadh-gogat减量表达小麦中tanadh-gogat基因的表达量显著。
经过步骤(一)的dna水平检测和步骤(二)的rna水平检测确定gooe1、gooe17和gooe18三个t2代tanadh-gogat超量表达小麦以及gor1、gor2和gor3三个t2代tanadh-gogat减量表达小麦构建成功。
实施例2、tanadh-gogat转基因小麦的产量性状鉴定
供试小麦:tanadh-gogat超量表达小麦gooe1、gooe17和gooe18,tanadh-gogat减量表达小麦gor1、gor2和gor3,野生型小麦kn199,以及空载对照植株(nc)和各转基因系分离出来的阴性对照系。
小麦产量相关性状的测定是在大田条件下,进行了两年大田试验(2015-2016生长季和2016-2017生长季),具体如下:
2015-2016生长季对纯合t3代tanadh-gogat转基因系表型进行鉴定。田间表型鉴定采用2m行长,行距30cm,株距10cm。成熟后单株收获测定表型。测定各供试小麦的如下指标:单株籽粒产量(克/株)、每株穗数、每穗穗粒数、千粒重(克)。结果显示,与受体亲本kn199相比,tanadh-gogat超量表达系(gooe1、gooe17和gooe18)的单株籽粒产量(克/株)和每株穗数显著增加,但是每穗穗粒数、千粒重(克)无显著变化。tanadh-gogat减量表达系(gor1、gor2和gor3)的单株籽粒产量(克/株)和每株穗数显著降低,但是每穗穗粒数、千粒重(克)无显著变化。见图2。
在2016-2017年对纯合的tanadh-gogat超量表达和减量表达转基因系t4代的表型进行了鉴定。每个转基因系种3个重复,每个重复1行。田间表型鉴定采用2m行长,行距30cm,株距5cm,成熟后每行选择10株代表性单株测定农艺性状。田测定各供试小麦的如下指标:籽粒产量(克/10株)、穗数(数量/10株)、植株生物量(克/10株)(干重)。另外还测定了每穗穗粒数、千粒重(克)。结果显示,与受体亲本kn199和阴性对照(各转基因系分离出来的阴性对照系)相比,tanadh-gogat超量表达转基因系的籽粒产量、植物生物量和穗数显著增加,千粒重和穗粒数等其他农艺性状无明显差异。与受体亲本kn199和阴性对照(各转基因系分离出来的阴性对照系)相比,tanadh-gogat减量表达转基因系的植株生物量、籽粒产量和穗数均显著降低,穗粒数也显著降低,千粒重没有显著性差异。见图3。
另外,在开花期到成熟期随机采集的单穗考种结果显示,在开花期,转基因系和受体亲本kn199的单穗地上部干重无显著差异;在成熟期减量表达系的单穗地上部干重显著低于kn199,超量表达系则显著高于kn199,说明超量表达tanadh-gogat促进了花后单穗干物质积累,减量表达tanadh-gogat抑制了地上部干物质积累。结合单株和单穗考种结果,穗数和单穗生物量的增加是超量表达系产量增加的主要原因,穗数减少和单穗生物量减少是减量表达系减产的主要原因。
<110>中国科学院遗传与发育生物学研究所
<120>蛋白tanadh-gogat在调控植物产量中的应用
<130>gncln181551
<160>3
<170>patentinversion3.5
<210>1
<211>2144
<212>prt
<213>triticumaestivuml.
<400>1
metprothralaglnglyileglyleulyshisalaalaproprogly
151015
glyargargalaargargserglnseralaalaalaproglyargser
202530
alaargglnalahisglyalametserleuaspglyglypheleugly
354045
glyalaglnargthrglugluargvalalaproargproproargala
505560
alaalaargaspalagluserileargprometserleuleuproglu
65707580
serserileglyleutyraspproalaphegluargaspsercysgly
859095
valglyphevalalagluleuserglyvalaspasnargalathrval
100105110
valaspalaileglnmetleugluargmetalahisargglyalacys
115120125
glycysglulysasnthrglyaspglyalaglyileleuvalalaleu
130135140
prohisthrphephearggluvalthrlysaspalaglyphegluleu
145150155160
proproproglyglutyralavalglymetvalpheleuprothrasp
165170175
glulysargarggluargserlysthrgluphethrlysvalalaglu
180185190
serleuglyhisserileleuglytrpargglnvalprothraspasn
195200205
seraspleuglyglnalaalaleuaspthrgluproalaileglugln
210215220
valpheleuthrlysserserlysserlysalaaspphegluglngln
225230235240
leupheileleuargargleuserilevalserileargalaalaleu
245250255
asnleuglnargglyglygluargaspphetyrmetcysserleuser
260265270
serargthrilevaltyrlysglyglnleumetproserglnleugln
275280285
glytyrtyrtyralaaspileglyhisgluasnphesersertyrmet
290295300
alaleuvalhisserargpheserthrasnthrpheprosertrpasp
305310315320
argalaglnprometargvalleuglyhisasnglygluileasnthr
325330335
leulysglyasnlysasntrpmetlysalaarggluglyleuleuglu
340345350
cysglulysleuglyleuserglnaspglumetserlysileleupro
355360365
ilevalaspalathrserseraspserglyalapheaspglyvalleu
370375380
gluleuleuileargglyglyargserleuproglualavalmetmet
385390395400
metileproglualatrpglnasnaspvalasnmetgluproasplys
405410415
lysalaleutyrglupheleuseralaleumetgluprotrpaspgly
420425430
proalaleuileserphethraspglyargtyrleuglyalathrleu
435440445
aspargasnglyleuargproglyargphetyrvalthrhissergly
450455460
argvalvalmetglysergluvalglyvalvalaspileproalagln
465470475480
aspvalleuarglysglyargleuasnproglymetmetleuleuval
485490495
asppheaspasnhisthrvalvalaspaspglualaleulysalagln
500505510
tyrserlysalahisprotyrglyglutrpleulysargglnlysmet
515520525
tyrleulysaspilevalgluservalprogluthraspargvalala
530535540
proserileserglyserilethrglnthrasngluasnlysglucys
545550555560
valglyileasnalailevalthrproleulysalapheglytyrthr
565570575
leuglualaleuglumetleuleuleuprometalalysaspglyval
580585590
glualaleuglysermetglyasnaspalaproleualavalmetser
595600605
asnargglulysleuthrpheglutyrphelysglnmetphealagln
610615620
valthrasnproproileaspproileargglulysilevalthrser
625630635640
metglucysmetileglyprogluglyaspleuleugluilethrglu
645650655
lysglncysasnargleualaleulysglyproleuvalsermetasp
660665670
glumetgluserilelyslysmetasntyrargglytrpargserlys
675680685
valleuaspilethrtyrprolysasnserglyarglysglyleuglu
690695700
gluthrleuaspargilecysalaglualaargglualaileargglu
705710715720
glytyrlysileleuvalleuseraspargglypheserserasparg
725730735
valalavalserserleuleualavalglyalavalhisglnhisleu
740745750
valalaasnleugluargthrargvalglyleuleuvalgluserala
755760765
gluproarggluvalhishisphecysthrleuvalglypheglyala
770775780
aspalailecysprotyrleualaileglualailetrpcysleugln
785790795800
thraspglylysileproprothraspserlysglugluleuvalglu
805810815
lystyrphetyralaseriletyrglymetmetlysvalleualalys
820825830
metglyileserthrleualasertyrlysglyalaglnilepheglu
835840845
alaleuglyleusersergluvalilehislyscysphegluglythr
850855860
proserargilegluglyalathrpheglumetleualaargaspala
865870875880
leuargleuhisgluleualapheproserargthrproproprogly
885890895
seralaaspalalysalaleuproasnproglyasptyrhistrparg
900905910
lysasnglygluvalhisleuasnaspproleualametalalysleu
915920925
glnglualaalalysvalasnserargglualatyrlysglutyrser
930935940
lysargileglngluleuasnlysalacysasnleuargglymetleu
945950955960
lyspheileaspserthrserlysileserleuaspgluvalglupro
965970975
alasergluilevallysargphecysthrglyalametsertyrgly
980985990
serileserleuglualahisthralaleualavalalametasnlys
99510001005
leuglyglylysserasnthrglygluglyglygluglnproser
101010151020
argmetgluproleuproaspglysermetasnprolysargser
102510301035
alailelysglnvalalaserglyargpheglyvalsersertyr
104010451050
tyrleuthrasnalaaspgluleuglnilelysmetalaglngly
105510601065
alalysproglygluglyglygluleuproglyhislysvalile
107010751080
glyaspilealavalthrarghisserthralaglyvalglyleu
108510901095
ileserproproprohishisaspiletyrserilegluaspleu
110011051110
alaglnleuilehisaspleulysasnserasnproglnalaarg
111511201125
ileservallysleuvalserglualaglyvalglyvalvalala
113011351140
serglyvalvallysglyhisalaasphisvalleuilesergly
114511501155
hisaspglyglythrglyalaserargtrpthrglyilelysasn
116011651170
alaglyleuprotrpgluleuglyleualagluthrhisglnthr
117511801185
leuvalalaasnglyleuargglyargalavalleuglnthrasp
119011951200
glyglnleulysthrglyargaspvalalavalalacysleuleu
120512101215
glyalagluglupheglypheserthralaproleuilethrleu
122012251230
glycysilemetmetarglyscyshisthrasnthrcysproval
123512401245
glyilealathrglnaspprovalleuargglulysphealagly
125012551260
gluprogluhisvalileasnphephephemetleualagluglu
126512701275
leuarggluilemetalaglnleuglyleuargthrileasnglu
128012851290
metvalglyargseraspmetleugluvalaspprogluvalval
129513001305
lysserasnglulysleugluasnileaspleuserleuileleu
131013151320
lysproalaalagluileargproglyalaalaglntyrcysval
132513301335
glulysglnasphisglyleuaspmetalaleuaspasnlysleu
134013451350
ilealaleuserargalaalaleuglulysgluvalargvalphe
135513601365
ilegluthrproilelysasnthrasnargalavalglythrthr
137013751380
leuserhisgluvalthrlysargtyrhismetlysglyleuasp
138513901395
proglythrilehisvallysleuthrglyseralaglyglnser
140014051410
pheglyalapheleucysproglyilethrleugluleuglugly
141514201425
aspserasnasptyrvalglylysglyleuserglyglylysile
143014351440
valvaltyrproproargasnserthrpheseralagluaspasn
144514501455
ilevalileglyasnvalalaleutyrglyalathrlysglyglu
146014651470
alatyrpheasnglymetalaalagluargphecysvalargasn
147514801485
serglyalaargthrvalvalgluglyileglyasphisglycys
149014951500
glutyrmetthrglyglythrvalvalileleuglylysthrgly
150515101515
argasnphealaalaglymetserglyglyilealatyrvaltyr
152015251530
aspvalaspglythrpheservalargcysasnasngluleuval
153515401545
aspleutyrhisvalgluglugluaspaspvalthrthrleulys
155015551560
metmetilegluglnhisargleuhisthrgluservalleuala
156515701575
lysaspileleuserlyspheaspthrleuleuprolyspheval
158015851590
lysvaltyrproargasptyrlysargvalleugluglumetlys
159516001605
alaglulysalaalaalaargprothrlysgluprolysvalala
161016151620
asnglyvalservalthrthrlyslysileglnthrglulysser
162516301635
serserargprothrargvalalaasnalalyslystyrarggly
164016451650
phevalthrtyrgluarggluglyvalsertyrargaspproasn
165516601665
gluargvallysasptrpasngluvalalailegluservalpro
167016751680
glyproleuleuasnthrglnseralaargcysmetaspcysgly
168516901695
thrprophecyshisglngluserserglyalaglycysproleu
170017051710
glyasnlysileproglupheasngluleuvalhisglnasnarg
171517201725
trpargglualaleuaspargleuleugluthrasnasnphepro
173017351740
gluphethrglyargvalcysproalaprocysgluglysercys
174517501755
valleuglyileilegluasnprovalserilelysserileglu
176017651770
cysserileileasplysglypheglugluglytrpmetvalpro
177517801785
argproproleuglnargthrglylyslysilealailevalgly
179017951800
serglyproalaglyleualaalaalaaspglnleuasnlysmet
180518101815
glyhisphevalthrvalphegluargseraspargileglygly
182018251830
leumetmettyrglyvalproasnmetlysthrasplysilegly
183518401845
valvalglnargargvalasnleumetalaglugluglyvalthr
185018551860
phevalvalasnalaasnvalglyseraspproleutyrserile
186518701875
gluargleuhissergluasnasnalavalileleualacysgly
188018851890
alathrlysproargaspleuserileproglyarggluleuala
189519001905
glyvalhisphealametglupheleuhisalaasnthrlysser
191019151920
leuleuaspserasnleugluaspglyargtyrileseralagln
192519301935
glylyslysvalvalvalileglyglyglyaspthrglythrasp
194019451950
cysileglythrservalarghisglycysserserilevalasn
195519601965
leugluleuleuthrlysproproserlysargalaseraspasn
197019751980
protrpproglntrpproargvalpheargvalasptyrglyhis
198519901995
glnglualaserthrlyspheglyasnaspproargthrtyrglu
200020052010
valleuthrlysargpheileglyaspgluaspglylysleulys
201520202025
alaleugluvalvalargvallystrpglulysvalaspglylys
203020352040
pheglnphelysgluilegluglyserglngluileilegluala
204520502055
aspleuvalleuleualametglypheleuglyproglugluasn
206020652070
ilealaasplysleuglyleuglulysaspasnargserasnphe
207520802085
lysalaglnpheglyhispheglythrservalaspglyvalphe
209020952100
alaalaglyaspcysargargglyglnserleuvalvaltrpala
210521102115
ilethrgluglyargglualaalaalaalavalasplystyrleu
212021252130
serargaspgluglnasnvalalaglyleuthr
21352140
<210>2
<211>6435
<212>dna
<213>triticumaestivuml.
<400>2
atgccgacggcgcaggggattggtttgaagcacgcggcgccgccgggcggccgcagggcc60
cgccgcagccagtccgcggccgcgccgggccgctccgcaaggcaggcgcacggcgccatg120
tccctggatggcgggttcctcggcggcgcgcagcgcaccgaggaacgcgtcgcgccacgc180
ccgcctcgggccgcggcgcgcgacgccgagtccatcaggcccatgtctctgctacccgag240
agcagcattgggctgtacgacccggcgttcgagcgtgactcgtgcggcgttggcttcgtc300
gccgagctgtcgggcgttgacaaccgggcgaccgtcgtcgatgccattcagatgcttgaa360
agaatggcacaccgaggtgcctgcggctgtgagaaaaacactggtgatggtgccggcatt420
ctcgttgctctaccacacaccttcttccgagaggtgacaaaggatgccggtttcgagtta480
ccgccaccaggtgagtatgctgttggaatggtcttcctgccaaccgatgagaagcgccgc540
gagaggagcaaaactgagtttacaaaggtcgctgagtctctaggacattcgatacttggg600
tggcgccaggttcccactgacaattcagacttgggccaagctgcgctcgatactgaacca660
gccattgaacaggttttcctcaccaagagttcaaaatcgaaggccgacttcgaacagcag720
ttgtttatcctgaggaggctctcaattgtatctatccgggccgcgctgaatcttcagcgt780
ggaggagagagagatttctacatgtgctctctatcttcaaggaccattgtttacaagggc840
caacttatgccatcccagctccaagggtactactatgcggatataggtcatgaaaacttc900
tccagttatatggctctggttcactcaaggttctccaccaacaccttccccagctgggac960
cgtgcacagccaatgcgtgtcttaggccacaatggagagatcaatactctcaaagggaac1020
aaaaactggatgaaagctcgtgagggtctcttggagtgtgagaagcttggcctatcacag1080
gatgaaatgtcaaagattcttccaatagtagatgccacttcttcagattcaggtgcattt1140
gatggtgttctcgagctccttatccgtggtggaagaagcctgccagaagctgtgatgatg1200
atgatccctgaggcgtggcagaatgatgtaaacatggaacctgataagaaagctctgtac1260
gagttcttgtcagcccttatggagccttgggatggacctgctctcatatcttttaccgat1320
ggccgctaccttggagctaccctggatcgcaatggccttaggcctggtcgattttatgta1380
acccacagtggacgtgtggtcatgggtagtgaagttggtgttgtggatattcctgcccaa1440
gatgtgttgagaaagggtcggcttaaccctggaatgatgttactcgttgacttcgacaac1500
catactgtagtagatgatgaagcactcaaggcacagtactctaaagctcacccgtatgga1560
gaatggctcaagcgacaaaagatgtacctcaaagacattgtagaatctgtcccagaaact1620
gacagagttgctccaagcatttctggttctattacgcaaacaaatgagaacaaggaatgt1680
gtaggcatcaatgcaattgtgactccactaaaggcgtttgggtacacattggaagcccta1740
gaaatgttgctgctgccaatggctaaagatggagtggaagctcttggatcaatgggaaat1800
gatgcacccctagcagtgatgtcaaacagagagaagctgacttttgagtacttcaagcag1860
atgtttgcacaagtaacaaaccctccaatcgatccaattagggagaagattgttacatct1920
atggaatgtatgattgggccagaaggagatttgctggaaataaccgaaaagcaatgcaac1980
cgccttgcacttaaaggtcctttggtgtcaatggatgaaatggaatctatcaagaagatg2040
aactaccgtggttggcgcagcaaggtgctcgacataacttatccgaagaattctggaagg2100
aagggcttggaagaaactttggatagaatttgtgctgaagcccgggaagctatacgcgag2160
ggatacaaaattttagttctttcagacagaggattttcttcagaccgtgttgctgtcagt2220
tccctcttagcagttggagcagtacatcaacaccttgttgcaaatcttgagaggacacgc2280
gtaggattattggttgagtctgctgaacctcgtgaagtgcaccatttctgtacactcgtt2340
ggatttggtgcagatgctatatgcccttatttggccattgaagcaatttggtgcttgcag2400
actgatgggaagattccccctaccgactcaaaggaggaacttgtcgaaaaatatttttat2460
gcttccatctatggaatgatgaaggttcttgcaaagatgggaatatccacccttgcatct2520
tacaaaggggcacagatttttgaagctcttggactttcttctgaagtgattcacaagtgt2580
tttgaaggcactcctagcagaattgagggtgcaacgttcgaaatgcttgcacgtgatgct2640
ctccgtcttcacgagttggcattcccatcaagaacacctccgcctggcagtgcagatgca2700
aaagcccttcccaatccaggggattaccactggaggaaaaatggtgaagtccatctaaat2760
gatcctcttgcaatggcaaaattacaagaagcagctaaagtaaacagccgggaagcatac2820
aaagaatattctaagcgaattcaagagcttaacaaggcgtgcaatctgcgtggtatgctg2880
aaatttatagatagcactagcaagatttctttggatgaggttgaacctgcaagtgagata2940
gtgaaacgcttttgtactggggcaatgagttatggatctatttcgttggaggcacatact3000
gcccttgctgtggccatgaacaaactgggaggcaaatctaacacaggggagggaggggag3060
cagccttctcgtatggagcctcttcctgatggttcgatgaatccaaaacgaagtgcaatc3120
aagcaagttgccagtggacgatttggagtttccagctattatctgactaatgcagatgag3180
ctgcagataaaaatggctcagggtgctaagcctggtgaaggtggtgagctaccaggtcac3240
aaggttatcggtgacattgcagttaccagacattctacagctggtgttgggcttattagt3300
ccacctcctcaccatgatatatattctatcgaggatcttgcacagcttatccatgacctt3360
aagaattcaaatcctcaagctcgaatcagcgtgaagctagtgtctgaggctggtgtygga3420
gttgtagctagcggtgttgtaaaaggacacgcagaccatgttcttatttctggccatgac3480
ggtggcaccggtgcatcaagatggacaggtatcaagaatgctggacttccatgggaacta3540
ggattggcygagacacatcaaactttagtggcaaatgggcttcgtggtcgagctgtccta3600
caaacagatggccaattaaaaactggtagagatgttgctgtggcgtgcttacttggtgca3660
gaggaatttggtttcagcactgctccactgatcacacttggctgcattatgatgcggaag3720
tgccataccaatacttgccctgttggtatagccactcaagatccagtgctccgagaaaaa3780
tttgctggagaaccagagcatgtcattaactttttcttcatgcttgctgaggagctaaga3840
gaaattatggctcagcttggcctccgtacaattaatgaaatggttggacgctctgatatg3900
cttgaagttgatccagaagtagttaagagcaatgagaaacttgaaaatattgatctctca3960
ttgatcttaaaaccagctgcagagattcgtcctggggctgctcagtactgtgttgaaaag4020
caagaccatggccttgacatggctttggataacaaacttatagctttatcaagggctgca4080
cttgaaaaggaagttcgtgttttcattgagactccaatcaagaacactaatcgggcagta4140
ggcaccacacttagccatgaagtcacaaaacgttatcacatgaagggattagatcctgga4200
accattcatgtgaaactcactggaagtgctggtcagagttttggtgcttttctctgtcct4260
ggaataacccttgagcttgaaggagacagcaatgattacgttggcaaaggattatctggt4320
ggaaagattgttgtgtacccacccaggaacagtacatttagtgcagaagacaatattgtc4380
attggtaatgtggccctgtatggtgctaccaagggagaagcatacttcaatggaatggca4440
gcagaaaggttttgtgttcgtaattctggtgctcgaacagtggttgaaggaattggtgat4500
catggatgcgagtacatgacagggggtactgtagtcatccttggtaaaacaggaagaaat4560
tttgctgctgggatgagtggaggcatcgcttatgtttatgatgttgatgggacattcagt4620
gtccgctgtaacaatgagttggttgatctatatcatgtggaggaagaggatgatgtaacc4680
actttgaaaatgatgatagagcaacatcgacttcacacagagagtgtcctggccaaagac4740
atactctctaaattcgacactcttcttccaaaatttgtaaaagtatacccaagggattat4800
aagagggttctagaagaaatgaaggcagaaaaagctgcagctaggcctacaaaggaacct4860
aaggtggcaaatggtgtttctgtgacaactaagaaaatacaaacagagaagtcatccagc4920
cgaccaacacgtgttgccaacgccaaaaagtaccggggttttgtaacatatgagcgagag4980
ggtgtttcttaccgtgatccaaatgagcgtgttaaagattggaacgaggttgccattgaa5040
tcagttccagggccactgttaaacacacagtctgctcgttgtatggattgtggcactcct5100
ttctgtcatcaggaaagctcaggtgctggctgtcctcttggaaataagatcccagagttc5160
aatgaattagttcaccagaatagatggcgtgaagcattggatcgcctactagagacaaac5220
aacttccctgaatttactggacgtgtgtgccctgctccttgtgaggggtcttgtgttctt5280
ggcattattgagaacccagtatcaatcaaaagcatagaatgctcgattatagacaaaggt5340
tttgaagagggatggatggtaccacgaccaccacttcaaagaacaggaaagaaaatcgct5400
atagttggcagtggtcctgctggtttggctgccgctgatcaactaaataaaatgggccat5460
tttgtaactgtatttgaacgttcggatcgtataggaggtcttatgatgtatggagtacca5520
aacatgaagacagacaagattggagttgttcagcgtcgtgtcaatttaatggccgaagag5580
ggtgtaacatttgtggtgaatgctaatgtagggagtgatcctttatactcaattgaacgt5640
ctccattctgagaacaatgcagttattttggcttgtggagctacaaaaccaagggacctc5700
agtattcctggccgtgagctagctggagttcattttgccatggaatttctccacgcaaat5760
accaaaagtttgcttgatagcaacctggaggatggaagatacatatctgcccagggtaag5820
aaggtggtggtcattggtggtggagacacaggcacagattgcatcggtacgtctgttagg5880
catggttgcagcagcattgtaaatctggagcttctcaccaagccaccaagcaagagagct5940
tctgacaacccctggccccagtggcctagagtcttccgagtggactatgggcaccaggaa6000
gcatctaccaaatttggaaatgatccaagaacttacgaagtcttaaccaagcgtttcatt6060
ggtgatgaagatggaaaattgaaggcccttgaggtggtgcgcgtgaagtgggagaaagta6120
gatggaaaattccagttcaaggagattgaaggatcacaagagatcatcgaggcagacctt6180
gtcctccttgccatgggattcctgggccctgaagagaacatcgctgacaaactgggttta6240
gagaaagacaaccgttccaacttcaaagctcaattcggacacttcgggaccagtgtggat6300
ggcgtttttgccgctggggattgcaggcgcgggcaatcgctggttgtttgggccatcacc6360
gaagggcgtgaagctgctgctgcagtagataagtacttgtcaagggatgaacaaaatgtc6420
gcaggccttacatag6435
<210>3
<211>1033
<212>dna
<213>artificialsequence
<400>3
ggattggtttgaagcacgcggcgccgccgggcggccgcagggcccgccgcagccagtccg60
cggccgcgccgggccgctccgcaaggcaggcgcacggcgccatgtccctggatggcgggt120
tcctcggcggcgcgcagcgcaccgaggaacgcgtcgcgccacgcccgcctcgggccgcgg180
cgcgcgacgccgagtccatcaggcccatgtctctgctacccgagagcagcattgggctgt240
acgacccggcgttcgagcgtgactcgtgcggcgttggcttcgtcgccgagctgtcgggcg300
ttgacaaccgggcgaccgtcgtcgatgccattcagatgcttgaaagaatggcacaccgag360
gtgcctgcggctgtgagaaaaacactggtgatggtgccggcattctcgttgctctaccac420
acacctgaattcaagcttacgtcctcccctgcgcggcgcgcaacaagggacgacgacggc480
acccagatacaaaaaaaaatggtgatcatccagctctctcaagaaaatatcaagttcttc540
agagttcagattacacacactctagcttgaactagtaggcgtgcttgatcttgatcttac600
caagcttaggtgtgtggtagagcaacgagaatgccggcaccatcaccagtgtttttctca660
cagccgcaggcacctcggtgtgccattctttcaagcatctgaatggcatcgacgacggtc720
gcccggttgtcaacgcccgacagctcggcgacgaagccaacgccgcacgagtcacgctcg780
aacgccgggtcgtacagcccaatgctgctctcgggtagcagagacatgggcctgatggac840
tcggcgtcgcgcgccgcggcccgaggcgggcgtggcgcgacgcgttcctcggtgcgctgc900
gcgccgccgaggaacccgccatccagggacatggcgccgtgcgcctgccttgcggagcgg960
cccggcgcggccgcggactggctgcggcgggccctgcggccgcccggcggcgccgcgtgc1020
ttcaaaccaatcc1033
- 还没有人留言评论。精彩留言会获得点赞!