一种快速检测三种小麦根茎部病害的方法与流程
本发明属于植物保护领域,具体涉及一种快速检测三种小麦根茎部病害的方法。
背景技术:
小麦是一种重要的粮食作物,在中国的粮食安全和农业现代化发展中都占有重要地位。山东省是中国小麦第二大主产区,种植面积及产量均居全国前列。但是,近年来,小麦根茎部病害的流行,对山东省小麦的品质和产量造成了很大的损失。小麦纹枯病、小麦根腐病和小麦茎基腐病是三种重要的小麦根茎部病害。小麦纹枯病的主要病原是禾谷丝核菌(Rhizoctonia cerealis)。小麦根腐病的主要病原是麦根腐平脐蠕孢菌(Bipolaris sorokiniana)。小麦茎基腐病的病原是镰孢属(Fusarium spp.)真菌。这些病害的病原菌能在土壤中长期存活,在小麦苗期时即可侵染为害。一旦条件合适,病症容易扩展导致蔓延成灾。这些病害常常混合发生,早期症状相似,不易区分,防治较为困难。因此,建立快速检测技术对于小麦根茎部病害的防治具有重要的指导意义。
分子生物学的发展和 PCR 技术的诞生为植物病原菌的检测、鉴定和量化提供了新的方法和手段。多重PCR(multiplex PCR)是指在同一反应体系中加入两对或两对以上引物,当体系中存在与各引物对特异互补的模板时,就会出现相应的条带。这一概念是Chamberlian于1988年首先提出。多重PCR具有快速、特异、灵敏、低成本等优点,现已被广泛应用于植物病原菌的检测与鉴别。陈思等利用多重PCR技术,在同一体系中对小麦秆锈病、小麦叶锈病和小麦白粉病进行了准确地区分;高士刚等建立了同步检测黄瓜靶斑病原菌、黄瓜炭疽病菌和黄瓜细菌性角斑病菌的多重PCR方法。
目前,虽然已经有相关文献报道过这三种病害(小麦纹枯病、小麦根腐病和小麦茎基腐病)的同时检测,但本发明所采用的引物都是自主设计的,并利用正交设计的方法建立和优化了检测体系,能同时检测以上三种小麦根茎部病害,具有重要的实践意义。
技术实现要素:
本发明提供了一种快速检测三种小麦根茎部病害的方法,利用多重PCR技术可以在小麦苗期实现快速检测。
本发明是通过以下技术方案实现的:
本发明提供了一种快速检测三种小麦根茎部病害的方法,包括以下步骤:
ST1 取样,田间取苗期植株;
ST2 提取基因组DNA,采用改良的CTAB法提取基因组DNA;
ST3 进行多重 PCR 反应,添加根据禾谷丝核菌rDNA-ITS、麦根腐平脐蠕孢菌rDNA-ITS、镰孢菌(Fusarium spp.)EF-1α基因序列设计的引物各一对,添加Taq DNA聚合酶、d NTP,进行多重 PCR 反应,PCR扩增程序为:预变性 94℃,5 min;94℃变性30s,53.0-63℃时退火30s,72℃延伸 1min,35个循环,72℃延伸 5 min;
ST4 进行DNA电泳并分析。
优选的,步骤ST3所述设计的引物为SEQ ID NO.1-SEQ ID NO.6 。
优选的,步骤ST3所述退火30s,温度为55℃。
优选的,步骤ST3中WKF-S18/WKR-S8浓度为0.24-0.48µmol/L。
优选的,步骤ST3中RBF-S2/RBR-S1浓度为0.16-0.48µmol/L。
优选的,步骤ST3中EF-SF9/EF-SR8浓度为0.24-0.40µmol/L。
优选的,步骤ST3中Taq DNA聚合酶为1.5-2.5U。
优选的,步骤ST3中dNTP浓度为0.15-0.25mmol/L。
更优选的,步骤ST3中WKF-S18/WKR-S8为0.36µmol/L,RBF-S2/RBR-S1为0.48µmol/L,EF-SF9/EF-SR8为0.24µmol/L,Taq DNA聚合酶为2.5 U, d NTP为0.15mmol/L。
更优选的,同时扩增小麦纹枯病菌、小麦根腐病菌和小麦茎基腐病菌的基因组DNA,获得174、418和 600 bp 的特异条带,三种病菌的检测灵敏度都是100 pg 。
本发明的有益效果是:本发明设计小麦纹枯病菌、小麦根腐病菌和小麦茎基腐病菌的特异性引物,建立并采用正交设计的方法优化这三种病害的 PCR 检测体系,在小麦苗期时对样本进行多重PCR检测,确定病原菌种类,为小麦根茎部病害的早期诊断和有效防治提供技术支持。在优化体系和条件下能够同时得到扩增长度为174、418、600bp 3条特异性片段,未扩增出全蚀病菌、链格孢、根霉菌等其它病原菌模板特异性片段,对小麦纹枯病菌、小麦根腐病菌和小麦茎基腐病菌的检测灵敏度都是100 pg。该PCR检测方法具有特异、敏感、高效等优点,可应用于田间小麦样本根茎部病害的快速检测,具有较高的实际应用价值。
附图说明
图1为不同温度下多重PCR扩增结果,其中M为DNA marker DL2000,泳道1-8温度依次为:53.0、53.7、54.9、56.8、59.1、61.0、62.2、63.0℃。
图2为正交设计多重PCR扩增电泳图,其中M为DNA marker DL2000;泳道 1—18为表4中正交试验1-18个水平处理。
图3为小麦纹枯病菌(A)、蠕孢菌(B)、镰孢菌(C)单一PCR灵敏度检测结果,其中,M:DL2000 的 marker;泳道 1:阳性对照;泳道 2:阴性对照;泳道 3:10 ng;泳道 4:1 ng;泳道 5:100 pg;泳道 6:10 pg;泳道 7:1 pg;泳道 8:100 fg;泳道 9:10 fg 。
图4为混合DNA多重PCR灵敏度检测结果,其中,M:DL2000 的 marker;泳道 1:阳性对照;泳道 2:阴性对照;泳道 3:10 ng;泳道 4:1 ng;泳道 5:100 pg;泳道 6:10 pg;泳道 7:1 pg;泳道 8:100 fg;泳道 9:10 fg 。
图5为多重PCR特异性检测结果,其中:M:DNA marker DL2000;泳道1:阳性对照;泳道2:阴性对照;泳道3:禾顶囊壳小麦变种;泳道4:链格孢;泳道5:小孢根霉;泳道6:立枯丝核菌;泳道7:大丽花轮枝孢;泳道8:寄生疫霉烟草致病型;泳道9:桃褐腐病菌;泳道10:齐整小核菌。
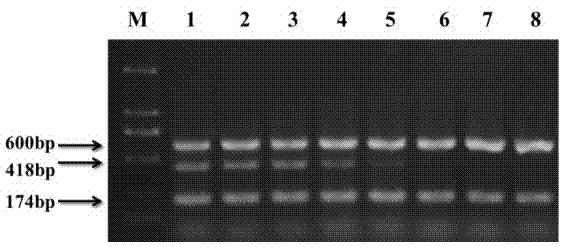
图6为各地区多重PCR检测结果,其中:M:DNA marker DL2000;泳道1:阳性对照;泳道2:阴性对照;泳道3-8:德州地区小麦样本;泳道9-14:聊城地区小麦样本;泳道15:青岛地区小麦样本;泳道16:潍坊地区小麦样本;泳道17-20:泰安地区小麦样本。
具体实施方式
实施例1 快速检测三种小麦根茎部病害的方法的建立和验证
No.1 材料与方法
No.1.1 材料
供试菌株的种名、编号及宿主见表1。所用供试病原真菌均由山东农业大学真菌与真菌资源利用实验室提供。
表1 供试菌株
小麦样本:2018年3月于山东省德州市、泰安市、潍坊市、临沂市以及青岛市采集田间小麦植株。
No.1.2 方法
No.1.2.1 基因组DNA的提取
病菌基因组DNA及小麦病组织DNA的提取均采用改良的CTAB法。
No.1.2.2 多重 PCR 引物组合
根据禾谷丝核菌(Rhizoctonia cerealis)rDNA-ITS、麦根腐平脐蠕孢菌(Bipolaris sorokiniana)rDNA-ITS以及镰孢菌(Fusarium spp.)EF-1α基因序列,利用Primer5软件设计多对引物,并从中筛选特异性较好的引物,供试引物组合见表2。引物由上海生工合成。
表2 试验引物
No.1.2.3 多重 PCR 反应体系优化
No.1.2.3.1 退火温度对多重 PCR 反应的影响
选择在53℃到 63℃之间设计梯度 PCR 程序,共 8 个梯度,依次为:53.0、53.7、54.9、56.8、59.1、61.0、62.2、63.0℃,通过梯度实验确定多重 PCR 反应的最佳退火温度 。
No.1.2.3.2 正交试验优化多重 PCR 反应条件
针对影响多重 PCR 体系的重要因素即三组引物浓度、Taq DNA聚合酶、d NTP ,按照五因素三水平L18(37)正交实验设计正交表(表 3,表 4)。反应体系总体积为 25 µL,包括 10×PCR buffer 2.5µL,模板 DNA 1µL。其它各成分按照表 4添加,最后用 dd H2O 补足。反应结束后,扩增产物取 5 µL样品在 2.0%琼脂糖凝胶于1×TAE 中电泳,并用 UVP 凝胶成像系统照相。
按照多重 PCR 扩增条带的有无及强弱对各病原菌扩增的敏感度评价打分,分值越高其扩增效果越好。
表 3 Multiplex PCR 正交试验因素水平表
表4 Multiplex PCR 正交试验设计 L18(37)
No.1.2.4单一PCR和多重PCR灵敏度检测
分别设置 3 种病原菌基因组 DNA和三者混合DNA 模板浓度为 10 ng、1 ng、100 pg、10 pg、1 pg、100 fg、10 fg,在优化后的多重 PCR 体系程序下扩增,检测多重 PCR 的检测灵敏度。
No.1.2.5多重 PCR 反应特异性检测
分别以全蚀病菌、链格孢、根霉菌等其它植物病原真菌基因组 DNA 为模板,进行多重 PCR 检测,测试多重 PCR 的特异性。
No.1.2.6 田间小麦样本的快速检测
田间采集小麦植株,依照 1.2.1 中的基因组 DNA 提取方法,提取小麦根茎部DNA,并采用建立的多重 PCR 反应体系对小麦样本进行检测。
No.2 结果
No.2.1 多重 PCR 反应体系的建立
No.2.1.1 退火温度优化
在53℃至 63℃间设8 个温度梯度进行PCR扩增,如图1所示,退火温度在53.0、53.7、54.9、56.8℃时扩增效果较好,都能扩增到三条清晰的条带。但是,考虑到温度过低容易引起非特异扩增,所以,最终确定最佳退火温度为 55℃。PCR扩增程序为:预变性 94℃,5 min;94℃变性30s,55℃退火 30s,72℃延伸 1min,35个循环,72℃延伸 5 min。
No.2.1.2 正交优化多重 PCR 反应体系
正交试验的结果见图 2,图中 1—18 依次为表 4 中的处理编号。从图 2 可见,不同的引物浓度、聚合酶和d NTP 对多重 PCR 反应有一定影响。综合评价特异扩增条带的清晰程度和丰富程度,依次打分(最优 9 分,最差 1 分)。图 2 中 1—18 个处理的打分依次为:6.5、7.5、8、6、8、8.5、6、8、9、6、8、8.5、7.5、8、9、6、9、9。正交设计直观分析结果如表 5;方差分析结果见表6。
表5 正交设计直观分析
正交试验直观分析结果表明,多重 PCR 反应最佳条件为:WKF-S18/WKR-S8 0.36μmol/L,RBF-S2/RBR-S1 0.48μmol/L,EF-SF9/EF-SR8 0.24μmol/L,Taq DNA聚合酶2.5 U, d NTP 0.15mmol/L,据此优化条件进行多重 PCR 扩增,可得到三个清晰的特异条带。
表6 正交设计方差分析
正交试验方差分析结果表明,引物RBF-S2/RBR-S1浓度的变化对试验结果的影响显著;而引物WKF-S18/WKR-S8、EF-SF9/EF-SR8的浓度,Taq DNA聚合酶量和 d NTP 的浓度变化对试验结果无显著影响。
No.2.2单一PCR和多重 PCR 检测的灵敏度
试验结果表明,在 25 μL 的多重 PCR 体系中,扩增小麦纹枯病菌和小麦根腐病菌单个模板都能检测到 10 pg 基因组DNA (图 3A 3B) ,扩增小麦茎基腐病菌单个模板能够检测到 100 pg 基因组DNA (图 3C) ,同时扩增3个模板能分别检测到100pg的小麦纹枯病菌、100pg的小麦根腐病菌和100pg的小麦茎基腐病菌。(图4)。
No.2.3 多重 PCR 反应特异性检测
按照正交试验优化后的反应条件,同时扩增小麦纹枯病菌、小麦根腐病菌和小麦茎基腐病菌的基因组DNA,获得174、418和 600 bp 的特异条带(图 5),而小麦其它病原菌或其它常见植物病原菌都没有该特异条带。
No.2.4 田间小麦样本的快速检测
结果显示(图 6),德州地区可以检测到小麦纹枯病菌、小麦根腐病菌和小麦茎基腐病菌;聊城地区可以检测到小麦纹枯病菌和小麦茎基腐病菌;青岛地区和潍坊地区检测到的都是小麦纹枯病菌;泰安地区可以检测到小麦纹枯病菌和小麦茎基腐病菌。
No.2.5 总结
本发明建立了一种可同时检测小麦纹枯病、小麦根腐病和小麦茎基腐病三种小麦根茎部病害的PCR检测体系。根据禾谷丝核菌(Rhizoctonia cerealis)rDNA-ITS、麦根腐平脐蠕孢菌(Bipolaris sorokiniana)rDNA-ITS以及镰孢菌(Fusarium spp.)EF-1α基因序列设计并合成引物。研究多重 PCR 的影响因素,优化 PCR 退火温度,采用正交试验方法,以 3个引物组、Taq DNA 聚合酶、d NTP 五因素三水平优化多重 PCR 体系,并进行特异性、灵敏度分析。结果表明,在优化体系和条件下能够同时得到扩增长度为174、418、600bp 3条特异性片段,未扩增出全蚀病菌、链格孢、根霉菌等其它病原菌模板特异性片段,对小麦纹枯病菌、小麦根腐病菌和小麦茎基腐病菌的检测灵敏度都是100 pg。该PCR检测方法具有特异、敏感、高效等优点,可应用于田间小麦样本根茎部病害的快速检测,具有较高的实际应用价值。
以上所述仅是本专利的优选实施方式,应当指出,对于本技术领域的普通技术人员来说,在不脱离本专利技术原理的前提下,还可以做出若干改进和替换,这些改进和替换也应视为本专利的保护范围。
序列表
<110> 山东农业大学
<120> 一种快速检测三种小麦根茎部病害的方法
<160> 6
<170> SIPOSequenceListing 1.0
<210> 1
<211> 26
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 1
gcaagtcttt tgttaattca actggc 26
<210> 2
<211> 18
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 2
ttcatccatg aatccggc 18
<210> 3
<211> 19
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 3
acttgttgtt tcctgggcg 19
<210> 4
<211> 22
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 4
gtagagtctt gatggattac cg 22
<210> 5
<211> 25
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 5
ggagacaaga ctcaccttaa cgtcg 25
<210> 6
<211> 25
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 6
gtgacaacat accaatgacg gtgac 25
- 还没有人留言评论。精彩留言会获得点赞!