萝卜肉质根相关性状的QTLs及其定位方法与流程
本发明属于分子生物学
技术领域:
,具体为萝卜肉质根相关性状的qtls及其定位方法。
背景技术:
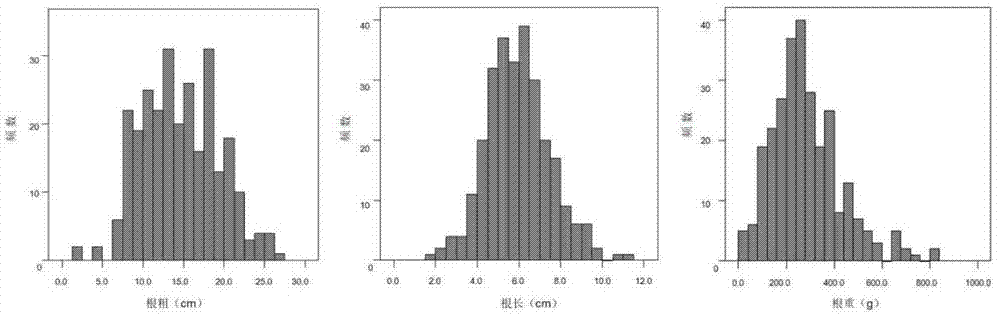
:萝卜(raphanussativusl.,2n=2×=18)是世界范围内重要的根类蔬菜。其肉质根含有多种对人类健康有益的成分。肉质根的发育对于决定萝卜产品器官的产量和品质至关重要。与其他根类植物类似,萝卜肉质根的发育同样受到遗传、环境和生理因素等诸多因子调控。目前,对于萝卜肉质根发育的遗传和分子机理受到关注。肉质根田间性状调查费时费力,使得萝卜肉质根的遗传改良受到限制。基于dna序列开发分子标记如aflp、caps、scar和ssr等是对植物无损害的收集肉质根性状信息的方法。一些控制萝卜肉质根发育相关性状的qtls通过aflp、ssr、caps和sts等分离标记被初步鉴定出来。如3个控制肉质根根形的qtls被鉴定出来,它们位于lg3、lg8和lg9连锁群上,lod值介于2.42~3.22之间,共解释42.4%的表型变异率;2个控制肉质根直径的qtls位于lg4和lg8,其中位于lg8上qtl的lod值为4.51,可解释23.8%的表型变异率;控制萝卜肉质根根重的qtls在两个自交系‘rattail’和日本萝卜‘harufuku’构建的ril群体被鉴定出来,它们可解释14.6~33.9%的表型变异率。通过qtls位点的鉴定及分子标记辅助选育(mas)可提高品种选育的效率。但是萝卜肉质根根长qtls定位尚未被研究,而且以往的萝卜连锁图谱并没有与萝卜基因组物理图谱联系起来,不便于肉质根相关性状的控制基因的发掘。技术实现要素:为解决现有技术中萝卜肉质根根长相关qtls位点尚未研究,且以往的萝卜连锁图谱并没有与萝卜基因组物理图谱联系的缺陷,本发明提供了萝卜肉质根相关性状的qtls及其定位方法,实现的目的为,本发明通过将不能形成肉质根的长荚萝卜‘pp12q-4-2’与可以正常形成肉质根的栽培萝卜‘36-2’作为亲本,将萝卜的连锁图谱与其基因组物理图谱整合,定位到控制萝卜肉质根性状包括根长、根粗和根重的qtls位点,进而提高品种的选育效率。为了实现上述目的,本发明提供以下技术方案:本发明发现的萝卜肉质根相关形状的13个qtls,分别位于染色体chr1、chr2、chr4、chr5、chr6、chr7和chr8;其中2个qtls控制根长,分别为qtrl1-1:侧翼标记为1ssr-27和1ssr-17和qtrl8-1:侧翼标记为41_14295和rs8-2;6个qtls控制根粗,分别为qtrt2-1:侧翼标记为rs2-22和2ssr-15、qtrt3-1:侧翼标记为3ssr-13和rs3-15、qtrt4-1:侧翼标记为rs4-12和53_16593、qtrt6-1:侧翼标记为rs6-7和rs6-15、qtrt8-1:侧翼标记为12_6264和rs8-2和qtrt9-1:侧翼标记为9ssr-14和9ssr-10;5个qtls控制根重,分别为qtrw3-1:侧翼标记为rs3-12和3ssr-19、qtrw3-2:侧翼标记为rs3-21和pr104、qtrw4-1:侧翼标记为rss2126和rs4-36、qtrw8-1:侧翼标记为rs8-44和8ssr-42和qtrw8-2:侧翼标记为151_26694和rs8-2;以上所述侧翼标记序列如seqidno.1~seqidno.48。本发明还提供了定位如上所述qtls的方法,包括如下步骤,(1)以两个萝卜高代自交系‘pp12q-4-2’和‘36-2’作为亲本材料进行子代f1、f2分离群体的构建;(2)对父母本、子代f1和f2分离群体单株肉质根相关性状包括根长、根粗和根重进行表型数据的采集,统计分析获得其表型变异及分布,并对肉质根性状进行相关性检验,采用shapiro-wilk对肉质根性状的正态分布进行检验;(3)采用ctab法分别提取父母本、子代f1和f2的dna;(4)多态性ssr引物筛选及f2群体基因型鉴定:利用父母本和f1筛选多态性ssr引物,选取扩增效果好的多态性ssr引物对f2分离群体进行dna扩增,确定每个等位位点的基因型;(5)利用joinmap4.0软件进行遗传图谱的构建:按照joinmap4.0软件要求的方法分别统计与父本一致、与母本一致以及杂合条带,采用joinmap4.0软件作图,利用kosambi作图将重组交换率转化为染色体图上基因遗传距离单位cm;(6)利用mapqtl4.0软件进行qtls分析,将lod值≥2的qtls位点保留,利用区间作图法im对这些qtls进行分析,对于im分析检测到的qtls,将最高lod值所在位置的标记或与其紧密连锁的标记作为协同因子,再对im检测到的qtls进行多座位qtl模型检测,作为最终的定位结果,以连锁群上lod值最高的位置作为qtl的位置,同时分析每个qtl对萝卜肉质根性状的遗传参数和贡献率。所述步骤(4)中筛选ssr引物扩增的15μlpcr反应体系包括:2μl,质量浓度为40ng·μl-1的dna;ssr引物的正向、反向引物各0.25μl,正向、反向引物的质量浓度均为10ng·μl-1;质量浓度为2.5u·μl-1的taq酶0.2ul;dntp0.2μl;25mm的mg2+1.2μl;10×taqbuffer1.5μl;ddh209.4μl;pcr反应程序:94℃预变性5min;94℃变性30s,58℃退火30s,72℃延伸45s,30个循环;72℃保温8min,扩增产物用质量浓度为8.0%非变性聚丙烯酰胺凝胶,160v恒功率电泳分离1h,银染显色后在胶片观察灯下进行带型统计分析;所述dna在多态性引物筛选时指父、母本和f1的dna,在扩增分离群体时,指的是f2各个单株的dna。本发明利用不能形成肉质根的长荚萝卜‘pp12q-4-2’与可以正常形成肉质根的栽培萝卜‘36-2’作为亲本,其中根重、根长和根粗均呈连续分布,说明这些性状均是由多基因控制的。进一步的,所述步骤(4)中最终筛选出在父母本中呈现多态性的ssr引物,分别包括记为1ssr-27、1ssr-17、41_14295、rs8-2、rs2-22、2ssr-15、3ssr-13的这些引物,所述引物的基因序列如seqidno.49~seqidno.62所示,这些引物同样用于遗传图谱的构建,构建的遗传图谱包含萝卜的9个染色体chr1-chr9。本发明采用上述技术方案,包括以下有益效果:以不能形成肉质根的长荚萝卜‘pp12q-4-2’与可以正常形成肉质根的栽培萝卜‘36-2’作为亲本,首次将萝卜连锁图谱与其基因组物理图谱联系起来,且首次定位到控制萝卜根长的2个qtls位点,从‘36-2’中找到可以将‘pp12q-4-2’不产生肉质根的位点代替的位点,进而提高品种的选育效率。附图说明图1为萝卜三个肉质根性状在f2群体的分布;图2为萝卜遗传图谱的第一条染色体及肉质根性状相关qtl的分布;图3为萝卜遗传图谱的第二条染色体及肉质根性状相关qtls的分布;图4为萝卜遗传图谱的第三条染色体及肉质根性状相关qtls的分布;图5为萝卜遗传图谱的第四条染色体及肉质根性状相关qtls的分布;图6为萝卜遗传图谱的第五条染色体及肉质根性状相关qtls的分布;图7为萝卜遗传图谱的第六条染色体及肉质根性状相关qtls的分布;图8为萝卜遗传图谱的第七条染色体及肉质根性状相关qtls的分布;图9为萝卜遗传图谱的第八条染色体及肉质根性状相关qtls的分布;图10为萝卜遗传图谱的第九条染色体及肉质根性状相关qtls的分布。具体实施方式下面通过具体的实施例对本发明做进一步的详细描述。实施例一:本发明提供的一种萝卜肉质根相关性状的qtls定位的方法,该方法包括如下步骤,(1)以两个萝卜高代自交系‘pp12q-4-2’和‘36-2’作为亲本材料进行子代f1、f2分离群体的构建:母本‘pp12q-4-2’为栽培型长荚萝卜,根部细长,不膨大;父本‘36-2’为栽培型萝卜,肉质根为长圆柱型,白皮白肉。两个亲本材料肉质根的表型有显著差异。f1通过两个亲本材料的人工杂交授粉获得,通过对f1严格的人工蕾期自交授粉获得f2。父母本(各30株)、f1(30株)和f2(330株)于2013年秋季播种于中国农业科学院蔬菜花卉研究所大棚,采用常规方法进行栽培管理。(2)各世代植株根长、根粗和根重进行表型数据的采集:播种后75d,当父本‘36-2’的肉质根完全成熟时,对父母本、f1和f2分离群体肉质根根长、根粗和根重进行表型数据的采集。根长采用精度为0.1cm的直尺进行测量,测量部位为主根部分的头部到尾部的距离;根粗采用精度为0.1mm的电子游标卡尺进行测量,测量部位为肉质根的最大直径处;根重用精确度为0.01g的电子称进行称量。去除一些死亡、病害和生长不正常的植物,共有父母本各25株、f123株、f2278株用于后续的试验。利用spss13.0统计软件对亲本、子代f1及f2群体各单株的根长、根粗和根重的调查结果进行统计分析并获得其表型变异及分布,并对3个性状之间进行相关性检验,采用shapiro-wilk对性状的正态分布进行检验。(3)采用ctab法分别提取父母本、子代f1和f2的dna;(4)多态性ssr引物筛选及f2群体单株基因型鉴定:首先利用父母本和f1进行多态性ssr引物筛选,选取扩增效果好的多态性ssr引物进行f2分离群体单株dna的pcr扩增。(5)利用joinmap4.0软件进行遗传图谱的构建:按照joinmap4.0软件要求的方法统计与父本一致、与母本一致、杂合条带,采用joinmap4.0软件作图,利用kosambi作图将重组交换率转化为染色体图上基因距离单位cm:与父本‘36-2’一致的带型记为a,与母本‘pp12q-4-2’一致的带型记为b,杂合的带型记为h,未扩增出的或模糊不清的带型记u。所述步骤(6)中最终筛选出与萝卜肉质根性状相关的13个qtls位点,分别位于染色体chr1、chr2、chr4、chr5、chr6、chr7和chr8;(6)利用mapqtl4.0软件进行qtls分析,将lod值≥2的qtls位点保留,利用区间作图法im对这些qtls进行分析,对于im分析检测到的qtls,将最高lod值所在位置的标记或与其紧密连锁的标记作为协同因子,再对im检测到的qtls进行多座位qtl模型检测,作为最终的定位结果,以连锁群上lod值最高的位置作为qtl的位置,同时分析每个qtl对萝卜肉质根性状的遗传参数和贡献率。所述步骤(4)中筛选ssr引物扩增的15μlpcr反应体系包括:2μl,质量浓度为40ng·μl-1的dna;ssr引物的正向、反向引物各0.25μl,正向、反向引物的质量浓度均为10ng·μl-1;质量浓度为2.5u·μl-1的taq酶0.2ul;dntp0.2μl;25mm的mg2+1.2μl;10×taqbuffer1.5μl;ddh209.4μl;pcr反应程序:94℃预变性5min;94℃变性30s,58℃退火30s,72℃延伸45s,30个循环;72℃保温8min,扩增产物用质量浓度为8.0%非变性聚丙烯酰胺凝胶,160v恒功率电泳分离1h,银染显色后在胶片观察灯下进行带型统计分析;所述dna在多态性引物筛选时指父母本和f1的dna,在扩增分离群体时,指的是f2各个单株的dna。所述步骤(4)中最终筛选出在父母本中呈现多态性的ssr引物,包括分别记为1ssr-27、1ssr-17、41_14295、rs8-2、rs2-22、2ssr-15、3ssr-13的这些引物,所述引物的基因序列如seqidno.49~seqidno.62所示,这些引物同样用于遗传图谱的构建,构建的遗传图谱包含萝卜的9个染色体chr1-chr9。在构建遗传图谱时,如还需要其他引物,则采用的都是现有技术的引物。本发明方法得到的结果:一、肉质根性状表型分布及相关性分析萝卜肉质根的根长、根粗和根重表型数据见表1。‘36-2’根长、根粗和根重的平均值均高于‘pp12q-4-2’。f1的根长介于双亲之间,根粗和根重均大于亲本‘36-2’,表现为超亲遗传。f2群体在这三个根部性状中均表现为较大的表型分离。一些植株在根长、根粗和根重的表现高于‘36-2’,另一些植株的根长、根粗和根重表现低于‘pp12q-4-2’。说明f2群体植株在两个方向均表现为超亲分离(表1和图1)。表1亲本、f1和f2群体的表型差异f2群体的根部性状表现为连续分布,表明这些性状为多个基因控制的数量性状,可以进行后续的qtls分析。经shapiro-wilk对f2群体表型分布进行检验,结果表明,根粗性状在f2群体中表现为正态分布(p=0.146),而根长和根重性状不符合正态分布(p<0.05)。3个肉质根性状之间存在相关性(表2)。其中根重与根长、根粗均呈现极显著正相关,相关系数分别为0.701和0.542;根粗和根长呈显著相关,相关系数为0.153。表2肉质根性状之间的相关性分析肉质根性状taproottraits根粗taprootthickness根长taprootlength根重taprootweight根粗taprootthickness1根长taprootlength0.153*1根重taprootweight0.701**0.542**1*significantatp<0.05;**significantatp<0.01二、肉质根性状的遗传图谱与qtls定位本发明最终筛选得到在亲本间呈现多态性的ssr引物,多态性比率为13.43%。利用joinmap4.0软件构建的遗传图谱共将多态性ssr标记定位到9个连锁群,连锁图谱的总长为904.1cm,两标记之间的平均长度为7.93cm。这9条连锁群的长度介于51.6~166.2cm之间。每个连锁群上的标记数在6~20。这9个连锁群对应萝卜的9条染色体上,chr1~chr9(图2-10)。利用区间作图法(im)并结合多座位qtl模型(mqm)检测,以lod≥2为阈值,共检测到13个与萝卜肉质根性状相关的qtls,分布于chr1、chr2、chr4、chr5、chr6、chr7和chr8上(图2-10和表3)。lod值介于2.26~4.11,可解释4.0~9.2%的表型变异率。表3控制萝卜肉质根性状的qtls位置及效应2个控制根长的qtls,qtrl1-1和qtrl8-1被鉴定出来。它们分别位于chr5和chr1,lod值分别为3.16和2.36,共解释11.4%的表型变异率,加性效应分别为-0.89和0.94,显性效应分别为1.70和1.56。说明qtrl1-1具有增加根长,而qtrl8-1具有减少根长的效应。6个控制根粗的qtls,qtrt2-1、qtrt3-1、qtrt4-1、qtrt6-1、qtrt8-1和qtrt9-1被鉴定出来,lod值介于2.26~3.31,共解释30.9%的表型变异率。4个qtls包括qtrt3-1、qtrt4-1、qtrt8-1和qtrt9-1的加性效应均为正值,可以提高根粗;其它两个qtls,qtrt2-1和qtrt6-1,加性效应为负值,起到降低根粗的效应。5个与根重相关的qtls,qtrw3-1、qtrw3-2、qtrw4-1、qtrw8-1和qtrw8-2被鉴定出来。它们的lod值介于2.43~4.11,可解释4.5%~9.2%的表型变异率。这5个qtls的显性效应均为正值,说明利用‘36-2’可以置换‘pp12q-4-2’中影响根重的位点,进而提高品种的选育效率。qtrw3-2、qtrw4-1、qtrw8-1和qtrw8-2的加性效应均为正值,说明利用这些位点可以提高根重。以上所述侧翼标记序列如seqidno.1~seqidno.48所示。肉质根的生长和变粗在决定其根形、产量和品质中起到重要的作用,而目前关于萝卜肉质根发育的遗传研究还相对较少。本发明的优点在于,以不能形成肉质根的长荚萝卜‘pp12q-4-2’与可以正常形成肉质根的栽培萝卜‘36-2’作为亲本,首次将萝卜连锁图谱与其基因组物理图谱联系起来,且首次定位到控制萝卜肉质根根长的2个qtls位点,从‘36-2’中找到可以将‘pp12q-4-2’不产生肉质根代替的位点,进而提高品种的选育效率。以上所述仅为本发明的优选实施例而已,并不用于限制本发明,对于本领域的技术人员来说,本发明可以有各种更改和变化。凡在本发明的精神和原则之内,所作的任何修改、等同替换、改进等,均应包含在本发明的保护范围之内。序列表<110>中国农业科学院蔬菜花卉研究所<120>一种萝卜肉质根相关性状的qtls定位的方法<160>62<170>siposequencelisting1.0<210>1<211>20<212>dna<213>1ssr-27侧翼标记正向引物序列(2ambystomalateralexambystomajeffersonianum)<400>1catctgtggctcactttcag20<210>2<211>20<212>dna<213>1ssr-27侧翼标记反向引物序列(2ambystomalateralexambystomajeffersonianum)<400>2cgccgctagaagaatatacc20<210>3<211>20<212>dna<213>1ssr-17侧翼标记正向引物序列(2ambystomalateralexambystomajeffersonianum)<400>3cgctattaagcttggttggt20<210>4<211>20<212>dna<213>1ssr-17侧翼标记反向引物序列(2ambystomalateralexambystomajeffersonianum)<400>4ctgaagtcgccagtcttctt20<210>5<211>21<212>dna<213>41_14295侧翼标记正向引物序列(2ambystomalateralexambystomajeffersonianum)<400>5tgcctcttctcttgactttgt21<210>6<211>20<212>dna<213>41_14295侧翼标记反向引物序列(2ambystomalateralexambystomajeffersonianum)<400>6ctacattgatctcccccaat20<210>7<211>20<212>dna<213>rs8-2侧翼标记正向引物序列(2ambystomalateralexambystomajeffersonianum)<400>7ttgcctttgattgtgttttt20<210>8<211>22<212>dna<213>rs8-2侧翼标记反向引物序列(2ambystomalateralexambystomajeffersonianum)<400>8gtttctatcggtgtacagacct22<210>9<211>20<212>dna<213>rs2-22侧翼标记正向引物序列(2ambystomalateralexambystomajeffersonianum)<400>9aattgggatccacttcactg20<210>10<211>22<212>dna<213>rs2-22侧翼标记反向引物序列(2ambystomalateralexambystomajeffersonianum)<400>10tggaaccaatatgattaaagcc22<210>11<211>20<212>dna<213>2ssr-15侧翼标记正向引物序列(2ambystomalateralexambystomajeffersonianum)<400>11aagaagctgaaatcactccc20<210>12<211>21<212>dna<213>2ssr-15侧翼标记反向引物序列(2ambystomalateralexambystomajeffersonianum)<400>12agttaaggaagctgagatgga21<210>13<211>20<212>dna<213>3ssr-13侧翼标记正向引物序列(2ambystomalateralexambystomajeffersonianum)<400>13gagtttgaaggactcagcgt20<210>14<211>20<212>dna<213>3ssr-13侧翼标记反向引物序列(2ambystomalateralexambystomajeffersonianum)<400>14gaaataaggcgaagaactgc20<210>15<211>20<212>dna<213>rs3-15侧翼标记正向引物序列(2ambystomalateralexambystomajeffersonianum)<400>15tacgcttgttccgatgagta20<210>16<211>20<212>dna<213>rs3-15侧翼标记反向引物序列(2ambystomalateralexambystomajeffersonianum)<400>16gacaccaccgtgtgttttta20<210>17<211>20<212>dna<213>rs4-12侧翼标记正向引物序列(2ambystomalateralexambystomajeffersonianum)<400>17acggttcagtcaaaaggaga20<210>18<211>20<212>dna<213>rs4-12侧翼标记反向引物序列(2ambystomalateralexambystomajeffersonianum)<400>18ggtgacgatgagctctgttt20<210>19<211>22<212>dna<213>53_16593侧翼标记正向引物序列(2ambystomalateralexambystomajeffersonianum)<400>19gtcgaaatcctttcatagatcc22<210>20<211>22<212>dna<213>53_16593侧翼标记反向引物序列(2ambystomalateralexambystomajeffersonianum)<400>20tgtgacttaattcttcaaacgg22<210>21<211>20<212>dna<213>rs6-7侧翼标记正向引物序列(2ambystomalateralexambystomajeffersonianum)<400>21ggaacacagctactgcattg20<210>22<211>20<212>dna<213>rs6-7侧翼标记反向引物序列(2ambystomalateralexambystomajeffersonianum)<400>22ttaggcaatgcttgattttg20<210>23<211>20<212>dna<213>rs6-15侧翼标记正向引物序列(2ambystomalateralexambystomajeffersonianum)<400>23gtgatttaaaaatcccgcac20<210>24<211>20<212>dna<213>rs6-15侧翼标记反向引物序列(2ambystomalateralexambystomajeffersonianum)<400>24acctcgagtgtcttcaccat20<210>25<211>20<212>dna<213>12_6264侧翼标记正向引物序列(2ambystomalateralexambystomajeffersonianum)<400>25ccctccctccagacatatta20<210>26<211>20<212>dna<213>12_6264侧翼标记反向引物序列(2ambystomalateralexambystomajeffersonianum)<400>26tgcaagtctaacatcggaaa20<210>27<211>20<212>dna<213>9ssr-14侧翼标记正向引物序列(2ambystomalateralexambystomajeffersonianum)<400>27caaccacaaaactccaaaca20<210>28<211>20<212>dna<213>9ssr-14侧翼标记反向引物序列(2ambystomalateralexambystomajeffersonianum)<400>28gtggaacactattgcgatca20<210>29<211>20<212>dna<213>9ssr-10侧翼标记正向引物序列(2ambystomalateralexambystomajeffersonianum)<400>29cttctgtccacggattacct20<210>30<211>21<212>dna<213>9ssr-10侧翼标记反向引物序列(2ambystomalateralexambystomajeffersonianum)<400>30tgaggatgatgtttctgaatg21<210>31<211>20<212>dna<213>rs3-12侧翼标记正向引物序列(2ambystomalateralexambystomajeffersonianum)<400>31aaaatttcgccattatcgag20<210>32<211>20<212>dna<213>rs3-12侧翼标记反向引物序列(2ambystomalateralexambystomajeffersonianum)<400>32tctctgatttgtcccctttc20<210>33<211>20<212>dna<213>3ssr-19侧翼标记正向引物序列(2ambystomalateralexambystomajeffersonianum)<400>33gaagggattaacgagctcaa20<210>34<211>20<212>dna<213>3ssr-19侧翼标记反向引物序列(2ambystomalateralexambystomajeffersonianum)<400>34tcatagctttcatgtggcat20<210>35<211>21<212>dna<213>rs3-21侧翼标记正向引物序列(2ambystomalateralexambystomajeffersonianum)<400>35cgctttaaagggaagactttt21<210>36<211>20<212>dna<213>rs3-21侧翼标记反向引物序列(2ambystomalateralexambystomajeffersonianum)<400>36ttctatcaccaaatggagcc20<210>37<211>20<212>dna<213>pr104侧翼标记正向引物序列(2ambystomalateralexambystomajeffersonianum)<400>37gggtcccaatgctagttgtg20<210>38<211>20<212>dna<213>pr104侧翼标记反向引物序列(2ambystomalateralexambystomajeffersonianum)<400>38caatgccagaaggacaaggt20<210>39<211>20<212>dna<213>rss2126侧翼标记正向引物序列(2ambystomalateralexambystomajeffersonianum)<400>39taaaaaccgttcctttcccc20<210>40<211>20<212>dna<213>rss2126侧翼标记反向引物序列(2ambystomalateralexambystomajeffersonianum)<400>40tgcttccgagtgctttacct20<210>41<211>21<212>dna<213>rs4-36侧翼标记正向引物序列(2ambystomalateralexambystomajeffersonianum)<400>41ggttttaatttaaccggcttt21<210>42<211>21<212>dna<213>rs4-36侧翼标记反向引物序列(2ambystomalateralexambystomajeffersonianum)<400>42ccgtaaaccaaacaacacttt21<210>43<211>20<212>dna<213>rs8-44侧翼标记正向引物序列(2ambystomalateralexambystomajeffersonianum)<400>43tcgggagactttgatttgtt20<210>44<211>20<212>dna<213>rs8-44侧翼标记反向引物序列(2ambystomalateralexambystomajeffersonianum)<400>44cactgtatcagagctggctg20<210>45<211>20<212>dna<213>8ssr-42侧翼标记正向引物序列(2ambystomalateralexambystomajeffersonianum)<400>45ggtgtgggacgacttctatc20<210>46<211>20<212>dna<213>8ssr-42侧翼标记反向引物序列(2ambystomalateralexambystomajeffersonianum)<400>46ctttcgagagaaaggcaaac20<210>47<211>21<212>dna<213>151_26694侧翼标记正向引物序列(2ambystomalateralexambystomajeffersonianum)<400>47gaacttcagtcctgcaagaaa21<210>48<211>20<212>dna<213>151_26694侧翼标记反向引物序列(2ambystomalateralexambystomajeffersonianum)<400>48tgatggtatttggatcagca20<210>49<211>20<212>dna<213>1ssr-27正向引物序列(2ambystomalateralexambystomajeffersonianum)<400>49catctgtggctcactttcag20<210>50<211>20<212>dna<213>1ssr-27反向引物序列(2ambystomalateralexambystomajeffersonianum)<400>50cgccgctagaagaatatacc20<210>51<211>20<212>dna<213>1ssr-17正向引物序列(2ambystomalateralexambystomajeffersonianum)<400>51cgctattaagcttggttggt20<210>52<211>20<212>dna<213>1ssr-17反向引物序列(2ambystomalateralexambystomajeffersonianum)<400>52ctgaagtcgccagtcttctt20<210>53<211>21<212>dna<213>41_14295正向引物序列(2ambystomalateralexambystomajeffersonianum)<400>53tgcctcttctcttgactttgt21<210>54<211>20<212>dna<213>41_14295反向引物序列(2ambystomalateralexambystomajeffersonianum)<400>54ctacattgatctcccccaat20<210>55<211>20<212>dna<213>rs8-2正向引物序列(2ambystomalateralexambystomajeffersonianum)<400>55ttgcctttgattgtgttttt20<210>56<211>22<212>dna<213>rs8-2反向引物序列(2ambystomalateralexambystomajeffersonianum)<400>56gtttctatcggtgtacagacct22<210>57<211>20<212>dna<213>rs2-22正向引物序列(2ambystomalateralexambystomajeffersonianum)<400>57aattgggatccacttcactg20<210>58<211>22<212>dna<213>rs2-22反向引物序列(2ambystomalateralexambystomajeffersonianum)<400>58tggaaccaatatgattaaagcc22<210>59<211>20<212>dna<213>2ssr-15正向引物序列(2ambystomalateralexambystomajeffersonianum)<400>59aagaagctgaaatcactccc20<210>60<211>21<212>dna<213>2ssr-15反向引物序列(2ambystomalateralexambystomajeffersonianum)<400>60agttaaggaagctgagatgga21<210>61<211>20<212>dna<213>3ssr-13正向引物序列(2ambystomalateralexambystomajeffersonianum)<400>61gagtttgaaggactcagcgt20<210>62<211>20<212>dna<213>3ssr-13反向引物序列(2ambystomalateralexambystomajeffersonianum)<400>62gaaataaggcgaagaactgc20当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1