一种构建酿酒酵母裂解工程菌的重组载体及其应用的制作方法
本发明属于生物技术领域,特别提供一种构建酿酒酵母快速裂解工程菌的重组载体和供体dna,以及通过其获得的酿酒酵母工程菌在酵母细胞内活性物质的提取、基因工程酵母表达系统表达产物的提取以及酵母细胞胞内表达重组蛋白(酶)快速检测的应用。
背景技术:
酿酒酵母(saccharomycescerevisiae)是一种广泛研究的单细胞真核微生物,具有复杂的细胞壁结构。酿酒酵母细胞壁厚度约100~300nm,类似“三明治”结构,主要由β-d-葡聚糖(β-d-glucan)、α-d-甘露聚糖(α-d-mannose)和少量的几丁质(chitin)组成。其中葡聚糖是最主要的结构成分,它与原生质体膜相连,起到维持细胞形态和正常渗透压的作用。甘露聚糖与蛋白质共价键相连形成甘露糖蛋白(mannoprotein),甘露糖蛋白主要分布在细胞壁外层,可以过滤大分子物质。而几丁质只占细胞干重的1~2%,其还原端与β-1,3-葡聚糖支链的还原端以β-1,2键或β-1,4键的形式相连。
目前酿酒酵母破壁的方法有很多,根据作用细胞壁的结构部位不同,可分为破坏葡聚糖结构、破坏甘露糖结构、破坏蛋白质结构和破坏多处结构等。采用的方法包括酸碱破壁、微波破壁、超声波法破壁、单酶法和复合酶法等。虽然这些方法不断的改进,但仍存在操作繁琐、破壁效率低、时间较长和对仪器要求较高等问题。
酿酒酵母在生物研究领域应用越来越广泛,但现有的破壁方法严重限制了外源基因在酿酒酵母中的克隆、表达及活性鉴定,因此,建立简单快捷并可广泛推广的破壁方法、以有效实现酿酒酵母细胞来源的核酸、蛋白、酶、多肽等各种活性物质的提取制备以及基因工程酵母表达产物的活性鉴定和提取、生产尤为重要。
sed1基因存在于酿酒酵母基因组中,其翻译的蛋白sed1p经过糖基化形成甘露糖蛋白,甘露糖蛋白与葡聚糖通过共价键结合最终锚定在细胞壁外侧(shimoih,etal.sed1pisamajorcellwallproteinofsaccharomycescerevisiaeinthestationaryphaseandisinvolvedinlyticenzymeresistance.jbacteriol,1998,180:3381-7.)。因为甘露糖蛋白分布在细胞壁的外侧,所以部分研究学者将外源蛋白与sed1p蛋白融合表达,使外源蛋白展示在细胞表面(kurodak,etal.enhancementofdisplayefficiencyinyeastdisplaysys-tembyvectorengineeringandgenedisruption.applmicrobiolbiotechnol,2009,82:713–9.)。sed1p蛋白可以维持细胞壁和线粒体基因组的稳定性,同时敲除sed1基因不但不会影响细胞的正常生长,反而还会提高酵母细胞对乳酸的耐受性(phadnisnetal.roleoftheputativestructuralproteinsed1pinmitochondrialgenomemaintenance.jmoliol,2004,342(4):1115-29;toshihiros,etal.disruptionofmultiplegeneswhosedeletioncauseslactic-acidresistanceimproveslactic-acidresistanceandproductivityinsaccharomycescerevisiae.jbioscibioeng,2013,115(5):467-474)。
技术实现要素:
为了克服现有技术的缺点与不足,本发明的目的在于提供一种构建酿酒酵母裂解工程菌的重组载体。
本发明的另一目的在于提供一种酿酒酵母裂解工程菌的构建方法。
本发明的另一目的在于提供通过上述构建方法构建得到的酿酒酵母裂解工程菌。
本发明的另一目的在于提供通过上述酿酒酵母裂解工程菌的应用。
本发明的再一目的在于提供一种酿酒酵母裂解方法。
本发明提供一种可构建高效、快速地对酿酒酵母细胞壁破壁的方法。
本发明提供一种便捷、灵敏度高、耗时短、成本低的破壁步骤,易于实现酿酒酵母快速裂解。
sed1基因的表达与酿酒酵母的细胞壁结构有关,其表达的蛋白经糖基化之后转运至细胞壁,通过磷脂酰肌醇(phosphatidylinositol)与细胞周质空间共价结合,同时与周围的葡聚糖形成β-1,6、β-1,3糖苷键使sed1p糖蛋白锚定在细胞壁上。sed1p糖蛋白属于甘露糖蛋白,其含量占细胞壁总蛋白的30%,是主要的细胞壁蛋白。敲除sed1基因后细胞壁的结构发生变化,降低细胞壁的坚固性,特别的增强其对裂解酶zymolyase的灵敏性,而细胞生长并不受影响。
本发明的目的通过下述技术方案实现:
一种构建酿酒酵母裂解工程菌的重组载体,该重组载体包括自5′到3′顺次连接cas9表达框和gsed1表达框;
所述的cas9表达框核苷酸序列,可为cas9表达框的任意序列,优选为序列表中seqidno.1核苷酸序列。
所述的gsed1表达框核苷酸序列,可为gsed1表达框的任意序列,优选为序列表seqidno.2核苷酸序列。
用于构建所述重组载体的出发载体可为任意一种酿酒酵母载体,优选p426(addgene公司的商业化质粒p426-snr52p-grna.can1.y-sup4t)和p414(addgene公司的商业化质粒p414-tef1p-cas9-cyc1t)载体。其中,以p426为出发载体,构建的重组载体为p426-cas9-gsed1。
一种酿酒酵母裂解工程菌的构建方法,包括如下步骤:
将上述重组载体和供体dna同时转化到酿酒酵母感受态细胞中,实现基因组中sed1基因的敲除,获得酿酒酵母突变株,即酿酒酵母裂解工程菌。
所述的供体dna核苷酸序列,可为供体dna的任意序列,优选为序列表seqidno.3核苷酸序列。
所述的酿酒酵母为酿酒酵母by4741,酿酒酵母bj5464,酿酒酵母cen.pk2-1ca。
所述的酿酒酵母突变株为酿酒酵母by4741δsed1,酿酒酵母bj5464δsed1,酿酒酵母cen.pk2-1caδsed1。
一种酿酒酵母裂解工程菌,通过上述构建方法构建得到。
一种酿酒酵母裂解方法,本发明提供一种酿酒酵母快速裂解释放胞内活性物质如核酸、蛋白、多肽等的方法,及其在重组蛋白(酶)快速筛查方面的应用。包括酿酒酵母重组菌菌落pcr鉴定、质粒提取、蛋白释放以及胞内蛋白(酶)快速筛查等。具体来讲,包括以下步骤:
(1)将外源基因表达载体转化到上述酿酒酵母突变株中,得到重组突变菌株;
(2)培养重组突变菌株,加入裂解酶zymolyase孵育,酿酒酵母细胞裂解,胞内物质释放到胞外;
(3)对释放到胞外的胞内物质可以进行酶活检测、菌落pcr、质粒提取、蛋白鉴定和胞内蛋白(酶)高通量筛选。
所述裂解酶zymolyase浓度为1~10u/ml,孵育时间为1~6h。
优选的,所述的裂解酶zymolyase浓度为5~10u/ml。
所述酿酒酵母裂解工程菌或酿酒酵母裂解方法在酶活检测、菌落pcr、质粒提取、蛋白鉴定和胞内蛋白(酶)高通量筛选中的应用。
本发明相对于现有技术具有如下的优点及效果:
(1)操作对象为酿酒酵母,它是生物领域使用最广泛的单细胞真核生物,具有广泛适用性。
(2)裂解时间短,2h可将细胞完全裂解,大大缩短了以往酿酒酵母细胞裂解的时间。
(3)裂解过程中无需添加额外试剂(如β-巯基乙醇等)和玻璃珠,操作简洁、成本廉价。
(4)操作简便,不需繁琐的步骤和配制复杂的裂解液,只需一种简单的缓冲液即可,大大简化了实验流程。
(5)应用较广,裂解后的细胞可进行酶活检测、菌落pcr、质粒提取、蛋白鉴定和胞内蛋白(酶)高通量筛选等,具有较广的应用前景。
(6)本发明的重组载体可以敲除酿酒酵母基因组中的sed1基因,得到的突变菌株在裂解酶zymolyase作用下能使细胞快速裂解,释放细胞内物质至胞外。因此,可以利用本发明的酿酒酵母裂解方法进行酶活检测、菌落pcr、质粒提取、蛋白鉴定和胞内蛋白(酶)高通量筛选等,具有简易、快捷、廉价的优点。
附图说明
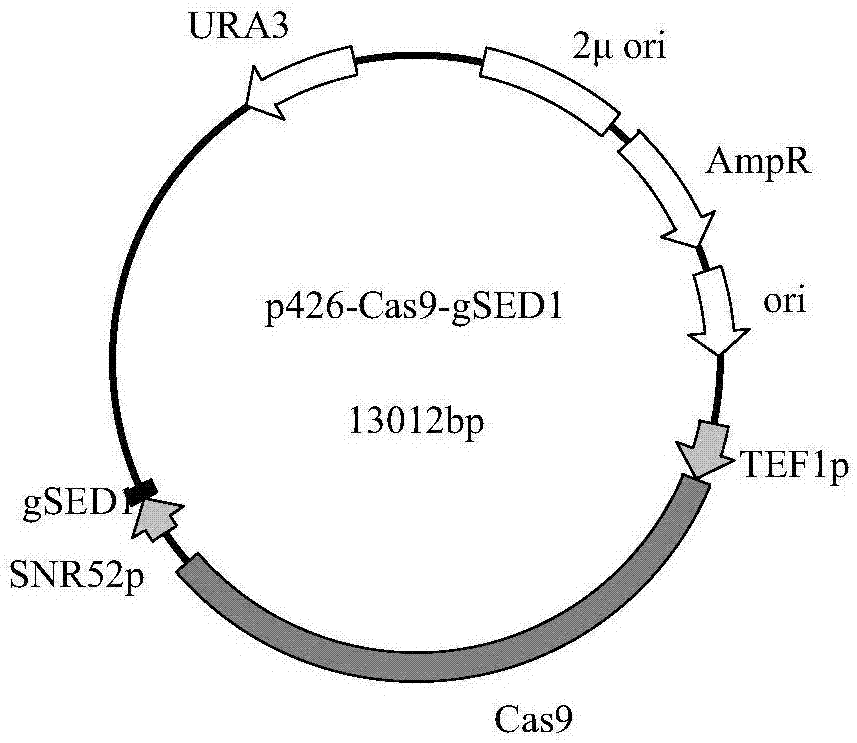
图1是重组载体p426-cas9-gsed1图谱。
图2是酿酒酵母突变菌株和野生菌株对zymolyase的敏感度;其中,pk2表示cen.pk2-1ca。
图3是裂解体系应用于酿酒酵母菌落pcr的结果图。
图4是裂解体系应用于蛋白质鉴定的结果图。
图5是裂解体系应用于酶活检测的结果图。
图6是裂解体系应用于酶活高通量筛选的结果图。
具体实施方式
下面结合实施例及附图对本发明作进一步详细的描述,但本发明的实施方式不限于此。
下列实施例中未注明具体实验条件的试验方法,通常按照常规实验条件或按照制造厂所建议的实验条件。所使用的材料、试剂等,如无特别说明,均为从商业途径得到的试剂和材料。
实施例中所用的载体yep352-tef2p-cyc1t,其构建过程如下:
(1)以酿酒酵母by4741基因组为模板,分别以p15/p16和p17/p18为引物对,分别扩增得到cyc1终止子片段和tef2启动子片段。将yep352载体(addgene公司的商业化质粒)和cyc1片段用sali/psti双酶切,得到的线性片段进行t4连接酶连接,获得载体yep352-cyc1t。
(2)将上述获得的yep352-cyc1t载体和tef2片段用bamhi/xbai双酶切,得到的线性片段进行t4连接酶连接,获得载体yep352-tef2p-cyc1t。
p15:5′-tttacagtcgacgggccgcatcatgtaatta-3′;
p16:5′-tttacactgcaggcaaattaaagccttcgag-3′;
p17:5′-tttacaggatccattacccataaggttgtttga-3′;
p18:5′-gagcgatctagagtttagttaattatagttcgt-3′。
实施例1酿酒酵母重组载体和供体dna的构建
构建酿酒酵母重组载体p426-cas9-gsed1和供体dna,其具体构建方法如下:
1.重组载体p426-gsed1的构建
以p426载体为模板,分别以p1/p2和p3/p4为引物对,分别扩增得到sed1-1和sed1-2片段。利用
p1:5′-taataatggtttcttagtatga-3′;
p2:5′-ccgatgtcacttcctcctctgatcatttatctttcactgc-3′;
p3:5′-agaggaggaagtgacatcgggttttagagctagaaata-3′;
p4:5′-actaagaaaccattattatcat-3′。
2.重组载体p426-cas9-gsed1的构建
以p414载体为模板,以p5/p6为引物扩增含有启动子tef1p、基因cas9和终止子cyc1t的片段。以p7/p8为引物扩增重组载体p426-gsed1,得到载体p426-gsed1的线性化片段。将上述扩增得到的片段,利用
p5:5′-ctaaagggaacaaaagctggcatagcttcaaaatgtttcta-3′;
p6:5′-atacattatcttttcaaagagcaaattaaagccttcgagcgtcc-3′;
p7:5′-gctcgaaggctttaatttgctctttgaaaagataatgtatgat-3′;
p8:5′-agaaacattttgaagctatgccagcttttgttccctttagt-3′。
3.合成可以阻断sed1表达的供体dna
引物p9(5′-tactttggcccaattttccaacagtacatctgcttcttaaaccgatgtcacttcctcct-3′),p10(5′-tgtgatagttactgagccagaggaagtggagatggaagaggaggaagtgacatcggttt-3′)互为模板扩增得到供体dna。
4.酵母基因组sed1位点编辑验证
将p426-cas9-gsed1和供体dna同时转化到s.cerevisiaeby4741(matahis3δ1leu2δ0met15δ0ura3δ0,atcc4040002),bj5464(matαura3-52trp1leu2δ1his3δ200pep4::his3prb1δ1.6rcan1gal,atcc208288)和cen.pk2-1ca(matatrp1-289leu2-3leu2-112ura3-52his3δ1matatrp1-289leu2-3leu2-112ura3-52his3δ1,atccmya-1108tm)的感受态细胞中,在cas9蛋白协助下,sed1基因转录的grna序列进行定位,同时供体dna重组至编辑位点来阻断sed1基因的正常表达,实现基因组中sed1基因的敲除,获得突变株s.cerevisiaeby4741δsed1,bj5464δsed1,cen.pk2-1caδsed1。
随机挑选5个酿酒酵母转化子,以p11(5′-ccctcttttgaactgtcata-3′)/p12(5′-gtagttggtgggaaagctga-3′)为引物进行菌落pcr扩增,并将pcr产物送测。测序结果显示,5个转化子的目的基因sed1位点均发生突变,即成功地敲除了sed1基因。
实施例2酿酒酵母快速裂解方法
1.酿酒酵母的复苏活化
(1)将s.cerevisiaeby4741,bj5464,cen.pk2-1ca的突变菌株和野生菌株分别从-80℃冰箱划线至ypd平板培养基上,在30℃条件下培养48h。
(2)挑取单菌落,分别接种至ypd液体培养基中,在30℃条件下培养至稳定期。
2.酿酒酵母悬浮液的制备:
(1)将培养的菌液3000×g,离心5min。
(2)获得的菌体用缓冲液tris-hcl(ph7.5)洗涤2次。
(3)用缓冲液tris-hcl(ph7.5)重悬,使其od600至0.6~0.7,得到酿酒酵母悬浮液。
3.与裂解酶zymolyase孵育
分别取200μl酿酒酵母悬浮液加入到96孔板中,加入不同浓度(1u/ml、5u/ml、10u/ml)zymolyase,在30℃,250rpm振荡培养6h,每1h测得od600,设置3个平行组,计算细胞残余率od600(%)。
其中,t表示不同时间点1h,2h,3h...
0表示0h。
4.实验结果(图2)表明,除了1u/mlzymolyase对细胞的作用无明显效应外,在5u/ml和10u/mlzymolyase浓度下,od600(%)随着时间的增加逐渐的下降,说明zymolyase对菌株的效应浓度为5u/ml和10u/ml。同时by4741δsed1,bj5464δsed1和cen.pk2-1caδsed1突变菌株比其野生菌株裂解得都要快,其在5u/mlzymolyase孵育6h后裂解效率分别提高了12.23%,12.46%和16.64%,同样在10u/mlzymolyas条件下其效率分别有19.24%,9.47%和11.58%的提高,说明突变菌株对zymolyase更敏感,更易于细胞的快速裂解。另外,用10u/mlzymolyase处理突变菌株6h后,bj5464δsed1和cen.pk2-1caδsed1od600(%)分别降到了29.19%和37.04%,细胞几乎完全被裂解。
综上所述,与野生菌株相比,突变菌株对zymolyase更敏感,更易被裂解,且5u/mlzymolyase裂解酶便可达到裂解细胞的目的,节约了成本。另外操作过程简单,不需额外有机试剂的添加和烦琐的物理处理步骤,是一种理想的细胞快速裂解方法。
实施例3酿酒酵母快速裂解方法在菌落pcr中的应用
1.酿酒酵母菌落pcr
(1)挑取按照实施例6获得的s.cerevisiaeby4741重组野生菌株和重组突变菌株的单菌落,在sd(ura)板上划patch,30℃培养。
(2)patch板上挑取少许菌体至20μltris-hcl(ph7.5)缓冲液里,震荡使其均匀。
(3)分别加入1uzymolyase,30℃,孵育30min,60℃,失活5min。
(4)14,000rpm,离心2min,取0.5μl上清为模板进行pcr,引物为p13/p14。
(5)琼脂糖凝胶电泳进行鉴定。
2.实验结果表明,重组突变菌株和重组野生菌株都扩增出了目的条带,但重组突变菌株扩增的条带比重组野生菌株的要亮(见图3),说明突变菌株裂解的更充分,释放更多的目的基因到上清。该方法比传统的反复冻融进行菌落更省时,只需要35min便可达到裂解细胞的目的,而反复冻融的方法往往需要2h以上。并且该方法操作简便,不需要额外的实验材料如液氮等,所以也大大节约了成本。因此利用该体系进行酵母菌落pcr是一种较理想的方法。
实施例4酿酒酵母快速裂解方法在质粒提取中的应用
1.菌株的培养
(1)从-80℃复苏活化按照实施例6获得的s.cerevisiaeby4741重组突变菌株和重组野生菌株,30℃培养。
(2)挑取单菌落接入到sd(ura)液体培养基中,30℃培养36h。
2.细胞的裂解
(1)将菌液3000×g,5min离心收集菌体,每管收集菌体5od。
(2)将重组突变菌株用0.5mltris-hcl(ph7.5)裂解缓冲液重悬至2ml离心管中,然后加入25uzymolyase裂解酶,在30℃,250rpm条件下裂解细胞0.5h。
(3)重组野生菌株按照omega和sangon酵母质粒提取试剂盒的方法将细胞裂解。
3.纯化处理
(1)重组突变菌株裂解后加入1μl21μg/μlrnasea缓冲液,去除rna。余下纯化步骤参考sangon试剂盒说明书进行。
(2)重组野生菌株按照omega和sangon酵母质粒提取试剂盒的方法进行纯化质粒。
4.对提取到的质粒进行鉴定
将提取到的质粒(1μl)分别转化到20μle.colidh5α感受态细胞中,统计转化子个数。
5.通过对各质粒提取方法的比较(表1)可以看出,本发明建立的酵母质粒提取方法与sangon试剂盒相比,大大缩短了操作所需时间(从3.5~4h缩短至约1h);而跟omega试剂盒相比,操作过程中无需添加β-巯基乙醇和玻璃珠,大大简化了操作步骤,操作时间缩短了约0.5h。此外本发明涉及的质粒提取方法,通过转化实验也验证质粒质量均优于商业化sangon和omega试剂盒所得质粒。
表1与市面常用酵母质粒提取试剂盒操作步骤及质粒提取质量比较
实施例5酿酒酵母快速裂解方法在蛋白质鉴定中的应用
1.菌株的培养
(1)从-80℃复苏活化按照实施例6获得的s.cerevisiaeby4741重组突变菌株和重组野生菌株,30℃培养。
(2)挑取单菌落接入到sd(ura)液体培养基中,30℃,200rpm培养36h。
2.细胞的裂解
(1)3000×g,5min离心收集菌体,每管收集菌体10od。
(2)用10mmtris-hcl(ph7.5)缓冲液洗涤两次,最终用1ml缓冲液进行重悬。
(3)加入50uzymolyase,30℃,250rpm细胞裂解3h,对照组加入tris-hcl(ph7.5)缓冲液。
(4)为了优化裂解的时间,采用同样的裂解方法裂解细胞,并每小时进行取样。
3.蛋白质的鉴定
3000×g,5min离心取上清,sds-page鉴定。
4.实验结果表明,与对照实验结果相比突变菌株和野生菌株均在70kd左右出现目的条带,但目的条带比理论计算值要大,原因在于目的蛋白在真核细胞表达时被修饰例如糖基化,使实际检测的条带稍大。从图4a可以看出目的蛋白gus成功地在酵母细胞中表达。并且突变菌株的目的条带比野生的要亮一些,表明突变菌株裂解得更充分,将更多的目的蛋白释放到胞外。图4b表明通过优化细胞裂解时间,突变菌株只在1h内便可检测到目的条带,而野生菌株则需要2h的时间,说明突变菌株更易被裂解酶裂解,释放胞内目的蛋白至胞外。
上述实验结果表明,本发明的细胞快速裂解方法可以应用于目的蛋白的鉴定,且此方法简便易行,成本较低,时间较短,无需复杂的裂解缓冲液和昂贵的仪器,具有广泛的应用价值。
实施例6酿酒酵母快速裂解方法在酶活检测中的应用
1.构建目的基因表达载体
以e.colidh5α基因组dna为模板,以p13(5′-taattaactaaactctagaaatgttacgtcctgtagaaaccc-3′),p14(5′-acatgatgcggcccgtcgactcattgtttgcctccctgct-3′)为引物扩增β-葡萄糖苷酸酶基因(gus)片段。
将载体yep352-tef2p-cyc1t用xbai/sali双酶切,纯化得到该载体的线性化片段。利用
将上述构建的表达载体转化到s.cerevisiaeby4741,bj5464,cen.pk2-1ca突变菌株和野生菌株的感受态细胞中,得到重组突变菌株和重组野生菌株。
2.菌株的培养
挑取单菌落接入到sd(ura)液体培养基中,30℃,200rpm培养至稳定期。
3.细胞的裂解
按照实施案例2方法进行细胞的快速裂解。
4.gus酶活的检测
(1)将90μlpbs(ph7.0)缓冲液加入到96孔板中,然后加入底物1g/lp-nitrophenyl-β,d-galactopyranoside(pnpg)80μl。
(2)取10μl裂解液加入上述鉴定体系中,37℃,反应10min。
(3)加入20μl1mnaoh终止反应。
(4)用酶标仪测定od405值。
(5)测定不同浓度(0、25、50、75、100、150、200、250、300、400、500μm)产物对硝基苯酚(pnp)的od405值,绘制标准曲线。
(6)根据标准曲线计算gus酶活,即在ph7.0,37℃条件下,每小时内底物释放1μmol对硝基苯酚所需的酶量为1个活力单位,记作u。
5.实验结果(图5)表明,1u/mlzymolyase对细胞几乎无作用,释放到胞外的酶较低,其结果与od600(%)变化相一致。在5u/ml和10u/mlzymolyase孵育下,s.cerevisiaeby4741,bj5464,cen.pk2-1ca大量细胞被裂解,6h后其酶活达到了最高值(约30u/ml),说明细胞得到了充分地裂解。在5u/mlzymolyase作用4h后s.cerevisiaeby4741δsed1的酶活达到了最大值,是野生菌株的2倍以上,而其野生菌株6h时的酶活比突变菌株还要低,说明突变菌株更易裂解,更易释放目的蛋白酶到胞外。虽然s.cerevisiaebj5464,cen.pk2-1ca的突变菌株与野生菌株酶活差异不显著,但是整体来看突变菌株的酶活比野生菌株的要高,而且5h时也可以达到最高酶活。另外,在10u/mlzymolyase条件下,突变菌株的酶活也是比野生菌株的要高,而且在较短的时间内可以得到较高的酶活,4h时的酶活已达到较高值,说明增加裂解酶的浓度可以在较短的时间内释放更多的酶活,缩短了实验周期。
由上述实验结果来看,在5u/mlzymolyase条件下突变菌株可得到充分地裂解,释放胞内目的蛋白酶至胞外,更易检测酶活。因此本发明的酵母快速裂解方法可以很好地运用于胞内目的蛋白酶的检测,且步骤简便,无需有机试剂的添加,保证了目的蛋白酶的活性,是一种较好的酵母胞内蛋白酶的测定方法。
实施例7酿酒酵母快速裂解方法在高通量筛选中的应用
1.菌株的培养
(1)将实施例6获得的目的基因表达载体yep352-tef2p-gus-cyc1t转化到s.cerevisiaeby4741突变菌株和野生菌株的感受态细胞中,30℃培养。
(2)用牙签挑取单菌落均匀地转接到sd(dura)固体培养基上,继续30℃培养24h。
(3)将长出来的单菌落影印到硝酸纤维素膜上,有菌落的一面朝上,在sd(dura)固体培养基上继续培养12h。
2.酶活的高通量筛选
(1)用含有20u/mlzymolyasetris-hcl(ph7.5)缓冲液浸湿滤纸,将硝酸纤维素膜放置到浸湿后的滤纸上,有菌落的一面朝上,30℃裂解2h。
(2)用含有底物pnpg的缓冲液将滤纸浸湿,将硝酸纤维素膜放置到浸湿后的滤纸上,有菌落的一面朝上,37℃培养30min。
(3)培养后将硝酸纤维素膜转移至用0.1mnaoh浸湿后的滤纸上,30℃培养约1h至黄色出现。
(4)拍照,记录结果。
3.由图6可以看出,与对照组s.cerevisiaeby4741野生菌相比,突变菌株用上述方法进行酶活检测后,其菌落周围均出现了黄色圈,显色率得到了100%,说明胞内的gus目的蛋白酶得到了充分地释放,与底物反应后生成了黄色的产物。而野生菌的菌落周围几乎看不到黄色圈,表明突变菌株可以应用于胞内酶的高通量筛选。
由上述结果可以看出,本发明的酶活筛选过程只需要硝酸纤维素膜和滤纸,不需要大型昂贵的筛选仪器如流式细胞仪等,大大较低了成本,同时简化了操作步骤。另外本发明的酶活高通量筛选方法只需要3.5h,且阳性率达到了100%,表明此筛选方法时间短,效率高。因此本发明的筛选方法是一种较理想的酶活高通量筛选方法。
上述实施例为本发明较佳的实施方式,但本发明的实施方式并不受上述实施例的限制,其他的任何未背离本发明的精神实质与原理下所作的改变、修饰、替代、组合、简化,均应为等效的置换方式,都包含在本发明的保护范围之内。
序列表
<110>华南理工大学
<120>一种构建酿酒酵母裂解工程菌的重组载体及其应用
<160>21
<170>siposequencelisting1.0
<210>1
<211>4830
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>cas9表达框核苷酸序列
<400>1
catagcttcaaaatgtttctactccttttttactcttccagattttctcggactccgcgc60
atcgccgtaccacttcaaaacacccaagcacagcatactaaatttcccctctttcttcct120
ctagggtgtcgttaattacccgtactaaaggtttggaaaagaaaaaagagaccgcctcgt180
ttctttttcttcgtcgaaaaaggcaataaaaatttttatcacgtttctttttcttgaaaa240
ttttttttttgatttttttctctttcgatgacctcccattgatatttaagttaataaacg300
gtcttcaatttctcaagtttcagtttcatttttcttgttctattacaactttttttactt360
cttgctcattagaaagaaagcatagcaatctaatctaagttttctagaactagtggatcc420
cccgggaaaaatggacaagaagtactccattgggctcgatatcggcacaaacagcgtcgg480
ttgggccgtcattacggacgagtacaaggtgccgagcaaaaaattcaaagttctgggcaa540
taccgatcgccacagcataaagaagaacctcattggcgccctcctgttcgactccgggga600
gacggccgaagccacgcggctcaaaagaacagcacggcgcagatatacccgcagaaagaa660
tcggatctgctacctgcaggagatctttagtaatgagatggctaaggtggatgactcttt720
cttccataggctggaggagtcctttttggtggaggaggataaaaagcacgagcgccaccc780
aatctttggcaatatcgtggacgaggtggcgtaccatgaaaagtacccaaccatatatca840
tctgaggaagaagcttgtagacagtactgataaggctgacttgcggttgatctatctcgc900
gctggcgcatatgatcaaatttcggggacacttcctcatcgagggggacctgaacccaga960
caacagcgatgtcgacaaactctttatccaactggttcagacttacaatcagcttttcga1020
agagaacccgatcaacgcatccggagttgacgccaaagcaatcctgagcgctaggctgtc1080
caaatcccggcggctcgaaaacctcatcgcacagctccctggggagaagaagaacggcct1140
gtttggtaatcttatcgccctgtcactcgggctgacccccaactttaaatctaacttcga1200
cctggccgaagatgccaagcttcaactgagcaaagacacctacgatgatgatctcgacaa1260
tctgctggcccagatcggcgaccagtacgcagacctttttttggcggcaaagaacctgtc1320
agacgccattctgctgagtgatattctgcgagtgaacacggagatcaccaaagctccgct1380
gagcgctagtatgatcaagcgctatgatgagcaccaccaagacttgactttgctgaaggc1440
ccttgtcagacagcaactgcctgagaagtacaaggaaattttcttcgatcagtctaaaaa1500
tggctacgccggatacattgacggcggagcaagccaggaggaattttacaaatttattaa1560
gcccatcttggaaaaaatggacggcaccgaggagctgctggtaaagcttaacagagaaga1620
tctgttgcgcaaacagcgcactttcgacaatggaagcatcccccaccagattcacctggg1680
cgaactgcacgctatcctcaggcggcaagaggatttctacccctttttgaaagataacag1740
ggaaaagattgagaaaatcctcacatttcggataccctactatgtaggccccctcgcccg1800
gggaaattccagattcgcgtggatgactcgcaaatcagaagagaccatcactccctggaa1860
cttcgaggaagtcgtggataagggggcctctgcccagtccttcatcgaaaggatgactaa1920
ctttgataaaaatctgcctaacgaaaaggtgcttcctaaacactctctgctgtacgagta1980
cttcacagtttataacgagctcaccaaggtcaaatacgtcacagaagggatgagaaagcc2040
agcattcctgtctggagagcagaagaaagctatcgtggacctcctcttcaagacgaaccg2100
gaaagttaccgtgaaacagctcaaagaagactatttcaaaaagattgaatgtttcgactc2160
tgttgaaatcagcggagtggaggatcgcttcaacgcatccctgggaacgtatcacgatct2220
cctgaaaatcattaaagacaaggacttcctggacaatgaggagaacgaggacattcttga2280
ggacattgtcctcacccttacgttgtttgaagatagggagatgattgaagaacgcttgaa2340
aacttacgctcatctcttcgacgacaaagtcatgaaacagctcaagaggcgccgatatac2400
aggatgggggcggctgtcaagaaaactgatcaatgggatccgagacaagcagagtggaaa2460
gacaatcctggattttcttaagtccgatggatttgccaaccggaacttcatgcagttgat2520
ccatgatgactctctcacctttaaggaggacatccagaaagcacaagtttctggccaggg2580
ggacagtcttcacgagcacatcgctaatcttgcaggtagcccagctatcaaaaagggaat2640
actgcagaccgttaaggtcgtggatgaactcgtcaaagtaatgggaaggcataagcccga2700
gaatatcgttatcgagatggcccgagagaaccaaactacccagaagggacagaagaacag2760
tagggaaaggatgaagaggattgaagagggtataaaagaactggggtcccaaatccttaa2820
ggaacacccagttgaaaacacccagcttcagaatgagaagctctacctgtactacctgca2880
gaacggcagggacatgtacgtggatcaggaactggacatcaatcggctctccgactacga2940
cgtggatcatatcgtgccccagtcttttctcaaagatgattctattgataataaagtgtt3000
gacaagatccgataaaaatagagggaagagtgataacgtcccctcagaagaagttgtcaa3060
gaaaatgaaaaattattggcggcagctgctgaacgccaaactgatcacacaacggaagtt3120
cgataatctgactaaggctgaacgaggtggcctgtctgagttggataaagccggcttcat3180
caaaaggcagcttgttgagacacgccagatcaccaagcacgtggcccaaattctcgattc3240
acgcatgaacaccaagtacgatgaaaatgacaaactgattcgagaggtgaaagttattac3300
tctgaagtctaagctggtctcagatttcagaaaggactttcagttttataaggtgagaga3360
gatcaacaattaccaccatgcgcatgatgcctacctgaatgcagtggtaggcactgcact3420
tatcaaaaaatatcccaagcttgaatctgaatttgtttacggagactataaagtgtacga3480
tgttaggaaaatgatcgcaaagtctgagcaggaaataggcaaggccaccgctaagtactt3540
cttttacagcaatattatgaattttttcaagaccgagattacactggccaatggagagat3600
tcggaagcgaccacttatcgaaacaaacggagaaacaggagaaatcgtgtgggacaaggg3660
tagggatttcgcgacagtccggaaggtcctgtccatgccgcaggtgaacatcgttaaaaa3720
gaccgaagtacagaccggaggcttctccaaggaaagtatcctcccgaaaaggaacagcga3780
caagctgatcgcacgcaaaaaagattgggaccccaagaaatacggcggattcgattctcc3840
tacagtcgcttacagtgtactggttgtggccaaagtggagaaagggaagtctaaaaaact3900
caaaagcgtcaaggaactgctgggcatcacaatcatggagcgatcaagcttcgaaaaaaa3960
ccccatcgactttctcgaggcgaaaggatataaagaggtcaaaaaagacctcatcattaa4020
gcttcccaagtactctctctttgagcttgaaaacggccggaaacgaatgctcgctagtgc4080
gggcgagctgcagaaaggtaacgagctggcactgccctctaaatacgttaatttcttgta4140
tctggccagccactatgaaaagctcaaagggtctcccgaagataatgagcagaagcagct4200
gttcgtggaacaacacaaacactaccttgatgagatcatcgagcaaataagcgaattctc4260
caaaagagtgatcctcgccgacgctaacctcgataaggtgctttctgcttacaataagca4320
cagggataagcccatcagggagcaggcagaaaacattatccacttgtttactctgaccaa4380
cttgggcgcgcctgcagccttcaagtacttcgacaccaccatagacagaaagcggtacac4440
ctctacaaaggaggtcctggacgccacactgattcatcagtcaattacggggctctatga4500
aacaagaatcgacctctctcagctcggtggagacagcagggctgaccccaagaagaagag4560
gaaggtgtgatctcttctcgagtcatgtaattagttatgtcacgcttacattcacgccct4620
ccccccacatccgctctaaccgaaaaggaaggagttagacaacctgaagtctaggtccct4680
atttatttttttatagttatgttagtattaagaacgttatttatatttcaaatttttctt4740
ttttttctgtacagacgcgtgtacgcatgtaacattatactgaaaaccttgcttgagaag4800
gttttgggacgctcgaaggctttaatttgc4830
<210>2
<211>388
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>gsed1表达框核苷酸序列
<400>2
tctttgaaaagataatgtatgattatgctttcactcatatttatacagaaacttgatgtt60
ttctttcgagtatatacaaggtgattacatgtacgtttgaagtacaactctagattttgt120
agtgccctcttgggctagcggtaaaggtgcgcattttttcacaccctacaatgttctgtt180
caaaagattttggtcaaacgctgtagaagtgaaagttggtgcgcatgtttcggcgttcga240
aacttctccgcagtgaaagataaatgatcagaggaggaagtgacatcgggttttagagct300
agaaatagcaagttaaaataaggctagtccgttatcaacttgaaaaagtggcaccgagtc360
ggtggtgctttttttgttttttatgtct388
<210>3
<211>97
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>供体dna核苷酸序列
<400>3
tactttggcccaattttccaacagtacatctgcttcttaaaccgatgtcacttcctcctc60
ttccatctccacttcctctggctcagtaactatcaca97
<210>4
<211>22
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>p1
<400>4
taataatggtttcttagtatga22
<210>5
<211>40
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>p2
<400>5
ccgatgtcacttcctcctctgatcatttatctttcactgc40
<210>6
<211>38
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>p3
<400>6
agaggaggaagtgacatcgggttttagagctagaaata38
<210>7
<211>22
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>p4
<400>7
actaagaaaccattattatcat22
<210>8
<211>41
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>p5
<400>8
ctaaagggaacaaaagctggcatagcttcaaaatgtttcta41
<210>9
<211>44
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>p6
<400>9
atacattatcttttcaaagagcaaattaaagccttcgagcgtcc44
<210>10
<211>43
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>p7
<400>10
gctcgaaggctttaatttgctctttgaaaagataatgtatgat43
<210>11
<211>41
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>p8
<400>11
agaaacattttgaagctatgccagcttttgttccctttagt41
<210>12
<211>59
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>p9
<400>12
tactttggcccaattttccaacagtacatctgcttcttaaaccgatgtcacttcctcct59
<210>13
<211>59
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>p10
<400>13
tgtgatagttactgagccagaggaagtggagatggaagaggaggaagtgacatcggttt59
<210>14
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>p11
<400>14
ccctcttttgaactgtcata20
<210>15
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>p12
<400>15
gtagttggtgggaaagctga20
<210>16
<211>42
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>p13
<400>16
taattaactaaactctagaaatgttacgtcctgtagaaaccc42
<210>17
<211>40
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>p14
<400>17
acatgatgcggcccgtcgactcattgtttgcctccctgct40
<210>18
<211>31
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>p15
<400>18
tttacagtcgacgggccgcatcatgtaatta31
<210>19
<211>31
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>p16
<400>19
tttacactgcaggcaaattaaagccttcgag31
<210>20
<211>33
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>p17
<400>20
tttacaggatccattacccataaggttgtttga33
<210>21
<211>33
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>p18
<400>21
gagcgatctagagtttagttaattatagttcgt33
- 还没有人留言评论。精彩留言会获得点赞!