用于小麦赤霉病抗性检测的KASP引物组及其应用的制作方法
本申请涉及小麦育种领域,特别是一种用于小麦赤霉病抗性检测的kasp引物组及其应用。
背景技术:
小麦赤霉病(fusariumheadblight,fhb)是由镰孢属(fusariumspp.)引起的一种世界性小麦穗部病害,不仅造成产量损失,且其引起的真菌毒素还严重危害人畜健康。随着全球性气候变暖、耕作制度以及耕作方式的改变,小麦赤霉病正向黄淮麦区、北方麦区、西南麦区和西北麦区扩展,且大流行频率不断增加,2000至2012年,我国有9个年份赤霉病的发生面积超过3.3×106hm2。改变耕作制度和栽培技术难以解决赤霉病的侵染和蔓延,依赖化学防治尽管对控制赤霉病的大发生和大流行取得了一定成效,但不可避免地造成成本增加和环境污染,有悖于绿色发展理念,因此培育抗病品种成为减轻赤霉病危害的主要途径。
国内外对赤霉病的抗性遗传进行了大量研究,发现多个赤霉病抗性qtl,并命名7个抗赤霉病基因。由于抗性效应及稳定性等方面原因,目前广泛应用的仅有位于3bs染色体上的fhb1位点,为了应对生产需求,新的赤霉病抗性基因亟待挖掘。长江中下游麦区是我国最早开展赤霉病抗性育种的区域,其代表性系列品种宁麦、扬麦、镇麦等均具有较高的赤霉病抗性,部分品种含有fhb1,系谱研究表明其fhb1多源于宁麦9号。
单核苷酸多态性(snp)标记,是继限制性片段长度多态性(rflp)和简单重复序列(ssr)之后的第3代分子标记,具有密度高、代表性强、遗传稳定性好和自动化程度高等优点,因此在遗传研究中具有广阔的应用潜力。在大量测序数据积累的基础上,芯片技术得到快速发展,相继开发出小麦illuminaiselect9k、illuminaiselect90k、affymetrixaxiom660k等芯片,并广泛应用于小麦遗传连锁图谱的构建、群体结构和连锁不平衡分析等研究。竞争性等位基因特异性pcr(kompetitiveallelespecificpcr,kasp)标记是针对基因组dna样品,通过引物末端碱基的特异匹配来对snp以及indel位点进行精准的双等位基因分型的pcr技术,目前广泛应用在水稻、小麦、大豆等作物的分子标记辅助选择中。
扬麦158是90年代长江中下游麦区的主推品种,同时也是重要的亲本材料,其赤霉病抗性达到中抗水平,但经鉴定其中不含fhb1,目前,针对扬麦158系的赤霉病抗性检测引物尚未见报道。
技术实现要素:
针对上述问题,本身提供一组针对扬麦158的小麦赤霉病抗性检测的kasp引物组及其应用,
小麦赤霉病抗性检测的kasp引物组,包括核苷酸序列如seqidno.1所示的前引物allelefam、核苷酸序列如seqidno.2所示的前引物allelehex、核苷酸序列如seqidno.3所示的后引物common。
上述kasp引物组在检测小麦赤霉病抗性中的应用,其具体步骤如下:利用上述kasp引物组配制成pcr检测体系并进行pcr扩增,再利用多功能酶标仪检测pcr扩增产物并以klustercallersoftware软件对5a染色体48.2cm处的标记excalibur_c6567_743进行基因分型,该位点等位变异为t/g,含有等位基因g的小麦赤霉病抗性显著高于含有等位基因t的小麦。扬麦158含有的优势等位变异为g,与前引物allelehex结合,t为非优势等位变异,可与前引物allelefam结合,从而利用kasp引物组将二者区分。
所述pcr检测体系(5μl):包括2.5μldna(30ngμl-1),kaspmix2.5μl(lgcgenomics,hoddeston,uk)和0.07μlkaspassaymix;
其中,kaspassaymix配制方法如下:每100μlkaspassaymix包含浓度为100μm的前引物allelefam(seqidno.1)12μl、浓度为100μm的前引物allelehex(seqidno.2)12μl、浓度为100μm的后引物common(seqidno.3)30μl、加入ddh2o补足至100μl。
pcr扩增程序:94℃热激活15min;94℃变性20s,61~55℃退火和延伸60s,10个循环(touch-down,每循环降低0.6℃);94℃变性20s,55℃退火和延伸60s,26个循环。
本发明同时还提供了一种用于检测小麦赤霉病抗性的基因盒,包括:2.5μldna(30ngμl-1),kaspmix2.5μl(lgcgenomics,hoddeston,uk)和0.07μlkaspassaymix;
其中,kaspassaymix配制方法如下:每100μlkaspassaymix包含浓度为100μm的allelefam12μl、浓度为100μm的allelehex12μl、浓度为100μm的后引物30μl、加入ddh2o补足至100μl。
本发明利用illuminaiselect90k芯片对宁麦9号×扬麦158的282份重组自交系进行基因组扫描,构建遗传图谱,结合3个环境的表型鉴定数据进行qtl定位,最终在5a染色体上检测到一个来源于扬麦158的稳定抗性位点,进一步转化为kasp标记,并验证其对赤霉病抗性影响显著,可应用于育种选择。
附图说明
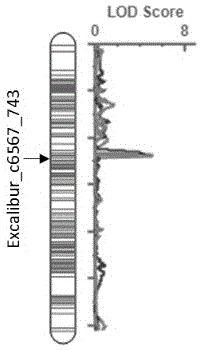
图1为遗传图谱示意图。
图2为实施例1kasp标记扩增检测结果示意图。
图3为实施例2基因分型检测结果示意图。
具体实施方式
遗传定位使用的宁麦9号×扬麦158的282份重组自交系群体由江苏省农业科学院粮食作物研究所保存。
标记验证使用的46份品种材料由江苏省农业科学院粮食作物研究所收集并保存。
赤霉菌由江苏省农业科学院植物保护研究所提供。
实施例1扬麦158的稳定抗性位点筛选
1、筛选方法
本实施例以源自于宁麦9号×扬麦158的282份重组自交系(rils)为材料,2015-2016生长季将所有rils及其亲本种植于江苏省农科院院内试验基地,单行区,行长2.0m,行距0.3m,每行50粒,2次重复。单花滴注的赤霉菌孢子液10μl,孢子浓度为5×105ml-1。在始花期,每个株系接种10穗,套袋保湿72h后,在弥雾保湿条件下继续生长,接种后21d调查病小穗数,作为赤霉病抗性评价指标。2016-2017生长季在江苏省农科院院内试验基地(玄武)及六合试验基地(六合)两个地点采用同样方法进行重复鉴定。利用excel2016对表型数据进行初步统计处理,用ibmspss19.0进行相关分析及方差分析。
采用ctab法从试验材料植株幼嫩叶片中分离dna(具体方法参见文献:doyle,j.j.(1987).arapiddnaisolationprocedureforsmallamountsoffreshleaftissue.phytochembull19,11-15.),利用illumina90k基因芯片(参见文献:wang,s.,wong,d.,forrest,k.,allen,a.,chao,s.,huang,b.e.,maccaferri,m.,salvi,s.,milner,s.g.,andcattivelli,l.(2014).characterizationofpolyploidwheatgenomicdiversityusingahigh-density90000singlenucleotidepolymorphismarray.plantbiotechnologyjournal12,787-796.)对试验材料进行基因组扫描,构建遗传连锁图,综合基因型与表型数据,使用icimapping4.1的完备区间作图方法(icim)进行qtl定位,统计在多环境中稳定检测到的位点,并进行标记转化,在品种材料中进行验证。
2、筛选结果
通过上述筛选方法检测到一个来源于扬麦158的稳定抗性位点,位于5a染色体48.2cm处(图1箭头标记处),excalibur_c6567_743为其紧密连锁标记(如图1所示),其等位变异为t/g,扬麦158含有的优势等位变异为g。
为了能够进一步验证并利用此标记,申请人其转化为kasp标记,包括两条前引物(allelefam、allelehex)与一条后引物(common),序列分别如下,
allelefam(seqidno.1):gaaggtgaccaagttcatgctcgactaatttaaacttggagctgtgat;
allelehex(seqidno.2):gaaggtcggagtcaacggattgactaatttaaacttggagctgtgag;
common(seqidno.3):gttcatacctacacccgtactgacaa。
并进行pcr扩增反应,反应体系及扩增程序如下:
pcr体系(5μl):包括2.5μl样品dna(30ngμl-1),kaspmix2.5μl(lgcgenomics,hoddeston,uk)和0.07μlkaspassaymix;
其中,kaspassaymix配制方法如下:每100μlkaspassaymix包含浓度为100μm的allelefam12μl、浓度为100μm的allelehex12μl、浓度为100μm的后引物30μl、加入ddh2o补足至100μl。
pcr程序:94℃热激活15min;94℃变性20s,61~55℃退火和延伸60s,10个循环(touch-down,每循环降低0.6℃);94℃变性20s,55℃退火和延伸60s,26个循环。
利用多功能酶标仪(pherastar,mglabtech,germany)检测pcr扩增产物,并使用klustercallersoftware(lgcgenomics,beverly,usa)进行基因分型(参见文献:wuj,lius,wangq,zengq,muj,huangs,yus,hand,kangz(2018)rapididentificationofanadultplantstriperustresistancegeneinhexaploidwheatbyhigh-throughputsnparraygenotypingofpooledextremes.theoreticalandappliedgenetics131:43-58)。
从282份rils中随机选取46份材料按如上方法进行kasp标记扩增检测,检测结果如图2所示。图2中,红色为携带优势等位变异g(+),蓝色为携带非优势等位变异t(-),且分型结果良好,材料分型与芯片检测数据完全一致,说明kasp标记开发成功,可进一步应用于育种材料检测。
实施例2kasp引物组基因组分型鉴定
利用实施例1获得的kasp引物组对46份品种材料(表1)进行基因分型(检测结果见图3及表1所示),并结合表型鉴定数据进行统计分析(t检验),统计软件采用spss19.0,结果如表2所示。
表1小麦材料分型检测结果1
表1中,“+”表示携带优势等位变异g,“-”表示携带非优势等位变异t。
表2小麦材料分型鉴定结果
表2中,t、p代表统计参数。
t检验显示携带优势等位变异g的材料赤霉病小穗数显著低于含非优势等位变异t的材料,表明含有等位基因g的小麦赤霉病抗性显著高于含有等位基因t的小麦,证明该kasp引物组可有效的帮助进行赤霉病筛选。
序列表
<110>江苏省农业科学院
<120>用于小麦赤霉病抗性检测的kasp引物组及其应用
<160>3
<170>siposequencelisting1.0
<210>1
<211>48
<212>dna
<213>人工序列(artificialsequence)
<400>1
gaaggtgaccaagttcatgctcgactaatttaaacttggagctgtgat48
<210>2
<211>47
<212>dna
<213>人工序列(artificialsequence)
<400>2
gaaggtcggagtcaacggattgactaatttaaacttggagctgtgag47
<210>3
<211>26
<212>dna
<213>人工序列(artificialsequence)
<400>3
gttcatacctacacccgtactgacaa26
- 还没有人留言评论。精彩留言会获得点赞!