单体链霉亲和素和高斯荧光素酶的融合蛋白及其应用的制作方法
本发明涉及抗体技术领域,具体涉及一种单体链霉亲和素和高斯荧光素酶的融合蛋白及其应用。
背景技术:
链霉亲和素(streptavidin,sa)是chaiet在1963年发现的,是streptomycesavidinii菌在培养过程中分泌的产物。一条sa肽链中有159个氨基酸残基,分子量为16.5kda。完整的sa蛋白由4条sa多肽链组成,形成一个四聚体结构。每条多肽链都可以特异性地结合一个生物素(biotin)分子,因此,1分子sa可以结合4分子的生物素。两者之间的相互作用以非共价结合形式存在,亲和力达到10-15/m,比抗原抗体的结合常数高若干数量级,是自然界中存在的最稳定的蛋白与小分子间相互作用。两者之间的强结合导致生物素和sa经常被用于开发标记和检测的工具。应用时通常将生物素标记于待检测的底物,用sa结合于标记在底物上的生物素,再用sa的抗体结合于sa,同时抗体上预先连接有发光基团,可被激发发光,最终通过检测发光而检测到底物。
sa和生物素之间1:4的结合比例可能引起生物素修饰的底物的聚集,在某些应用下带来不必要的副效果。因此,有研究人员将链霉亲和素和根霉亲和素(另一种与链霉亲和素结构类似,可以高效结合生物素的蛋白)的序列进行杂合,通过基因工程的手段开发出一种由一条肽链形成的单体链霉亲和素(monosa),可以1:1的比例结合生物素。monosa对生物素仍保持了较高的结合活性,达到10-9/m,同时避免了底物聚集带来的副作用。
荧光素酶是生物体内催化荧光素或脂肪醛氧化发光的一类酶的总称。通常发现于低等动物体内。目前常用的荧光素酶有萤火虫荧光素酶、海肾荧光素酶及高斯荧光素酶。萤火虫荧光素酶发光需要atp和mg2+的辅助,海肾荧光素酶虽然不依赖于atp和mg2+等辅助因子,但发光强度较弱,应用时需要更灵敏的检测。
高斯荧光素酶(gaussialuciferase,gluc)很好地弥补了萤火虫荧光素酶和海肾荧光素酶的缺点,发光时既不需要atp和mg2+等辅助因子,也有高于海肾荧光素酶100倍的发光效率。高斯荧光素酶是由一种海洋挠趾类动物分泌的荧光素酶,是目前发现的分子量最小的荧光素酶,并且具有信号肽,可以分泌到胞外,便于活性检测。在氧气存在的条件下,高斯荧光素酶可自发催化底物腔肠素氧化发光。发光强度是目前发现的荧光素酶中最高的,易于检测。由于以上优点,高斯荧光素酶在活细胞检测、蛋白与蛋白相互作用、蛋白定位、小干扰rna沉默技术、高通量药物筛选等领域中得到了广泛的应用。
目前,利用monosa进行生物素标记的识别主要通过monosa和anti-monosa抗体的联用。即用monosa特异性识别生物素,然后用带有发光基团的anti-monosa抗体识别monosa,最终通过检测抗体上的发光基团的发光来检测生物素。这种方法过程繁琐,耗时长。且如果使用的是sa和anti-sa抗体还有可能造成底物聚集。
技术实现要素:
本发明提供一种单体链霉亲和素和高斯荧光素酶的融合蛋白及其应用,该融合蛋白既可以特异性结合生物素也具备高强度自发光的能力,可以方便快速的检测生物素标记的底物。
根据第一方面,一种实施例中提供一种单体链霉亲和素和高斯荧光素酶的融合蛋白,该融合蛋白包括单体链霉亲和素全长蛋白和高斯荧光素酶全长蛋白或持续发光突变体,上述单体链霉亲和素全长蛋白与上述高斯荧光素酶全长蛋白或持续发光突变体通过柔性肽连接。
在优选实施例中,上述单体链霉亲和素全长蛋白如seqidno:1所示。
在优选实施例中,上述高斯荧光素酶全长蛋白序列如seqidno:2所示。
在优选实施例中,上述持续发光突变体序列如seqidno:3所示。
在优选实施例中,上述柔性肽序列如seqidno:4所示。
在优选实施例中,上述融合蛋白还包括:信号肽,该信号肽用于引导上述融合蛋白从表达细胞内向外分泌。
在优选实施例中,上述信号肽位于上述融合蛋白的n端。
在优选实施例中,上述信号肽序列如seqidno:5所示。
在优选实施例中,上述融合蛋白还包括:融合标签,该融合标签用于亲和纯化上述融合蛋白。
在优选实施例中,上述融合标签是6×组氨酸标签。
在优选实施例中,上述融合蛋白序列如seqidno:6或seqidno:7所示。
根据第二方面,一种实施例中提供一种分离的核酸,该分离的核酸编码第一方面的融合蛋白。
在优选实施例中,上述分离的核酸包括单体链霉亲和素全长蛋白编码序列和高斯荧光素酶全长蛋白编码序列或持续发光突变体编码序列,上述单体链霉亲和素全长蛋白编码序列与上述高斯荧光素酶全长蛋白编码序列或持续发光突变体编码序列通过连接序列连接。
在优选实施例中,上述单体链霉亲和素全长蛋白编码序列如seqidno:8所示。
在优选实施例中,上述高斯荧光素酶全长蛋白编码序列如seqidno:9所示。
在优选实施例中,上述持续发光突变体编码序列如seqidno:10所示。
在优选实施例中,上述连接序列如seqidno:11所示。
在优选实施例中,上述分离的核酸还包括:信号肽编码序列,该信号肽编码序列编码信号肽,该信号肽用于引导上述融合蛋白从表达细胞内向外分泌。
在优选实施例中,上述信号肽编码序列位于上述分离的核酸的5’端。
在优选实施例中,上述信号肽编码序列如seqidno:12所示。
在优选实施例中,上述分离的核酸还包括:融合标签编码序列,该融合标签编码序列编码融合标签,该融合标签用于亲和纯化上述融合蛋白。
在优选实施例中,上述融合标签编码序列是编码6×组氨酸标签的序列。
在优选实施例中,上述分离的核酸序列如seqidno:13或seqidno:14所示。
根据第三方面,一种实施例中提供一种表达载体,该表达载体包括编码第一方面的融合蛋白的核酸序列,以及载体骨架序列。
根据第四方面,一种实施例中提供一种重组宿主细胞,该重组宿主细胞内包含第三方面的表达载体。
根据第五方面,一种实施例中提供第一方面的融合蛋白、第二方面的分离的核酸、第三方面的表达载体或第四方面的重组宿主细胞在生物素分子检测中的用途。
本发明通过基因工程技术,将monosa和高斯荧光素酶基因进行融合表达,形成融合蛋白。该融合蛋白既可以特异性结合生物素也具备高强度自发光的能力,可以方便快速的检测生物素标记的底物。相比于sa和anti-sa抗体联用检测生物素,这种融合蛋白不仅方便快速,还不会引起底物的聚集,而且只需一次表达纯化即可得到融合蛋白,生产工艺简单,有利于规模生产。该融合蛋白在dna杂交、免疫检测、生化诊断等领域具有广阔的应用前景。
附图说明
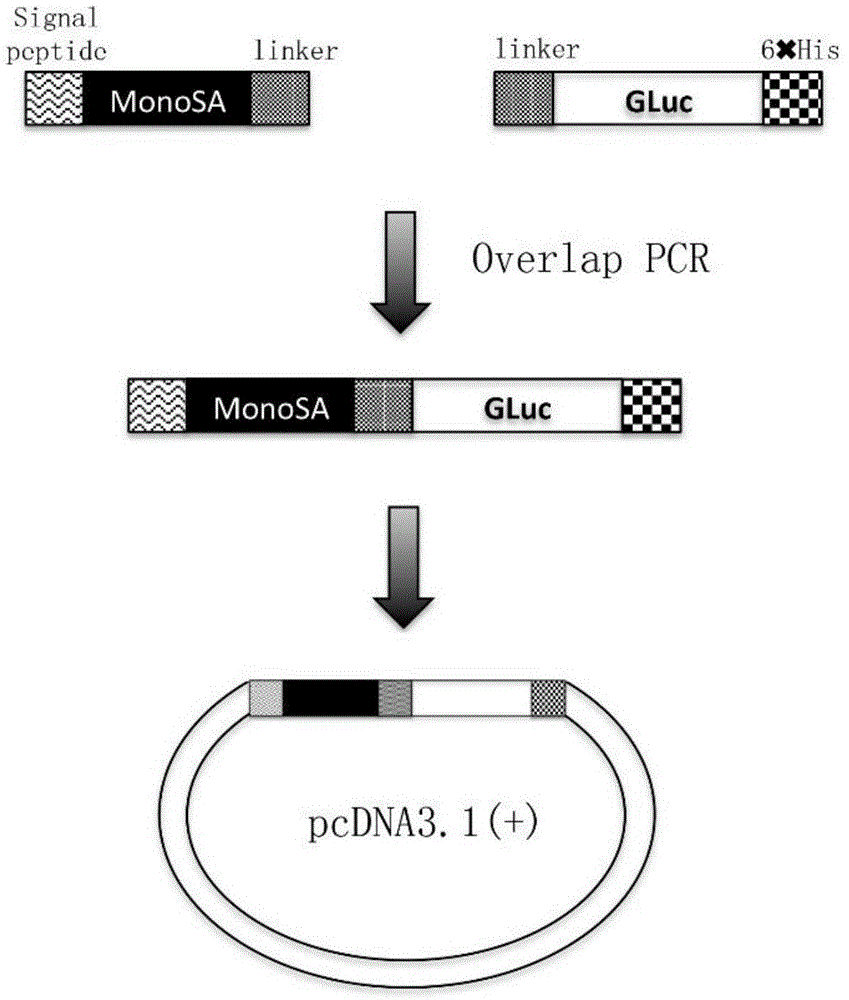
图1为本发明实施例中融合载体的构建示意图,其中monosa表示单体链霉亲和素全长蛋白编码序列,gluc表示高斯荧光素酶全长蛋白编码序列或持续发光突变体编码序列,linker表示连接序列,signalpeptide表示信号肽编码序列,overlappcr表示搭桥pcr。
图2为本发明实施例中融合蛋白sds-page检测结果图,其中1泳道:monosa-gluc融合蛋白,2泳道:marker。
图3为本发明实施例中野生型monosa-gluc融合蛋白和突变体monosa-g2l融合蛋白的生物发光检测结果图,其中monosa-gluc表示野生型融合蛋白,monosa-g2l表示突变体融合蛋白,gluc表示高斯荧光素酶,横坐标表示时间(time),纵坐标表示发光强度(clvalues)。
图4为本发明实施例中elisa法测定野生型monosa-gluc融合蛋白和突变体monosa-g2l融合蛋白对生物素的结合力结果图,其中,monosa-gluc表示野生型融合蛋白,monosa-g2l表示突变体融合蛋白,横坐标表示浓度(concentration)值,纵坐标表示od450nm吸光值。
具体实施方式
下面通过具体实施方式结合附图对本发明作进一步详细说明。在以下的实施方式中,很多细节描述是为了使得本发明能被更好的理解。然而,本领域技术人员可以毫不费力的认识到,其中部分特征在不同情况下是可以省略的,或者可以由其他材料、方法所替代。
另外,说明书中所描述的特点、操作或者特征可以以任意适当的方式结合形成各种实施方式。同时,方法描述中的各步骤或者动作也可以按照本领域技术人员所能显而易见的方式进行顺序调换或调整。因此,说明书和附图中的各种顺序只是为了清楚描述某一个实施例,并不意味着是必须的顺序,除非另有说明其中某个顺序是必须遵循的。
本发明目的在于提供一种单体链霉亲和素和高斯荧光素酶的融合蛋白。该蛋白既能够特异性结合生物素又能够催化底物腔肠素发光,达到方便快速检测任何带有生物素(biotin)标记的分子的融合蛋白的目的,该蛋白具有制备方法简单,易于生产且不容易造成底物聚集的特点。
本发明属于基因工程领域,具体涉及高斯荧光素酶和单体链霉亲和素(monosa)的融合蛋白的制备和用途。本发明利用基因工程技术将高斯荧光素酶全长蛋白(或持续发光突变体g2l)和单体链霉亲和素(monosa)全长蛋白通过柔性肽(linker)连接得到融合蛋白。在其c端连接多聚组氨酸标签,通过cho细胞进行表达及镍柱纯化,得到的融合蛋白一方面能够特异性结合生物素,另一方面能够氧化底物腔肠素发光。该自发光可被酶标仪等仪器检测到,并且在一定条件下可以被肉眼观察到,可以方便地检测任何带有生物素标记的分子,而不需要任何抗体及其他发光分子的参与。
利用pcr及双酶切技术将编码高斯荧光素酶全长基因(或持续发光突变体g2l基因)和单体链霉亲和素(monosa)全长基因构建进真核重组表达载体,并且在目的基因n端添加信号肽,c端添加6×组氨酸(6×his)标签,同时在高斯荧光素酶和单体链霉亲和素两个基因中间加入柔性肽的编码基因。通过脂质体法将构建的表达载体质粒转化进cho细胞,进行瞬时表达。蛋白在细胞中表达后,在信号肽的作用下分泌到细胞培养液中,并在分泌过程中切去信号肽。发酵完成后,离心收集细胞培养液。将收集的培养液过ni柱纯化后得到融合蛋白。该融合蛋白应具有与生物素特异性结合能力,同时具备催化底物腔肠素氧化发光的能力。
本发明提供一种单体链霉亲和素和高斯荧光素酶的融合蛋白,该融合蛋白包括单体链霉亲和素全长蛋白和高斯荧光素酶全长蛋白或持续发光突变体,单体链霉亲和素全长蛋白与高斯荧光素酶全长蛋白或持续发光突变体通过柔性肽连接。
本发明的融合蛋白中,单体链霉亲和素全长蛋白和高斯荧光素酶全长蛋白或持续发光突变体的连接方式(即二者的前后顺序)可以调整,例如,单体链霉亲和素全长蛋白在融合蛋白的n端,高斯荧光素酶全长蛋白或持续发光突变体在融合蛋白的c端;或者高斯荧光素酶全长蛋白或持续发光突变体在融合蛋白的n端,单体链霉亲和素全长蛋白在融合蛋白的c端。在优选实施例中,单体链霉亲和素全长蛋白在融合蛋白的n端,高斯荧光素酶全长蛋白或持续发光突变体在融合蛋白的c端。
本发明的融合蛋白中,持续发光突变体是指高斯荧光素酶全长蛋白的持续发光突变体,可以是指任何相对于野生型高斯荧光素酶全长蛋白发生一个或多个氨基酸突变但仍然保持高斯荧光素酶全长蛋白发光特性的突变体。在优选实施例中,该持续发光突变体是指持续发光突变体g2l,详见下文。
在优选实施例中,单体链霉亲和素全长蛋白如下seqidno:1所示:
efasaeagitgtwynqhgstftvtagadgnltgqyenraqgtgcqnspytltgryngtklewrvewnnstenchsrtewrgqyqggaearintqwnltyeggsgpateqgqdtftkvkpsaas(seqidno:1)。
在优选实施例中,高斯荧光素酶全长蛋白序列如下seqidno:2所示:
kptennedfnivavasnfattdldadrgklpgkklplevlkemeanarkagctrgcliclshikctpkmkkfipgrchtyegdkesaqggigeaivdipeipgfkdlepmeqfiaqvdlcvdcttgclkglanvqcsdllkkwlpqrcatfaskiqgqvdkikgaggd(seqidno:2)。
在优选实施例中,持续发光突变体是g2l突变体,其序列如下seqidno:3所示:
kptennedfnivavasnfattdldadrgklpgkklplevlkeleanarkagctrgcliclshikctpkmkkfipgrchtyegdkesaqggigeaivdipeipgfkdlepleqfiaqvdlcvdcttgclkglanvqcsdllkkwlpqrcatfaskiqgqvdkikgaggd(seqidno:3)。其中,带下划线的“l”分别是指相对于野生型高斯荧光素酶全长蛋白序列的突变氨基酸位点,即发生突变m43l和m110l。
需要说明的是,本发明的融合蛋白除了包括采用seqidno:1和seqidno:2,或seqidno:1和seqidno:3所示的序列连接而成以外,还包括虽然序列有一定差别但具有相同功能的序列形式。例如,对于单体链霉亲和素全长蛋白而言,可以是与seqidno:1序列有一些差别但是也具有相应的单体链霉亲和素全长蛋白亲和活性的序列,如在seqidno:1序列两端增加或减少若干氨基酸但保持相同亲和活性的序列;对于高斯荧光素酶全长蛋白序列而言,可以是与seqidno:2有一些差别但是也具有高斯荧光素酶全长蛋白序列活性的序列,如在seqidno:2序列两端增加或减少若干氨基酸但保持相同高斯荧光素酶全长蛋白序列活性的序列;对于持续发光突变体序列而言,可以是与seqidno:3有一些差别但是也具有持续发光突变体序列活性的序列,如在seqidno:3序列两端增加或减少若干氨基酸但保持相同持续发光突变体序列活性的序列。
本发明的融合蛋白中,具有连接作用的柔性肽可以选自如下序列:
ggggsggggs(seqidno:4);
gggggs(seqidno:15);
gqgqgqgqgqg(seqidno:16);
gstsgsgkssekgkg(seqidno:17);
vpgvgvpgvg(seqidno:18);
sapgtpsr(seqidno:19);
egkssgsgseskef(seqidno:20);
gsggsg(seqidno:21);
gsggsggg(seqidno:22)。
在优选实施例中,柔性肽序列如下seqidno:4所示:
ggggsggggs(seqidno:4)。
在优选实施例中,融合蛋白还包括:信号肽,该信号肽用于引导融合蛋白从表达细胞内向外分泌。
信号肽可以位于本发明的融合蛋白的n端或c端,在不影响本发明的融合蛋白中的亲和活性和发光活性的情况下,在优选实施例中,信号肽位于上述融合蛋白的n端。
在优选实施例中,信号肽序列如下seqidno:5所示:
mgvkvlfaliciavaea(seqidno:5)。
本发明中,可用于纯化融合蛋白的方法,包括但不局限于:镍柱纯化、离子交换层析(q柱、p柱)、疏水层析、肝素柱层析、硫酸铵沉淀法等。
在优选实施例中,本发明的融合蛋白还包括:融合标签,该融合标签用于亲和纯化该融合蛋白。具体而言,融合标签与各种纯化方法中使用的纯化装置(如纯化柱)亲和结合,然后通过适当方法获取纯化的融合蛋白。融合标签可以位于本发明的融合蛋白的n端或c端,优选位于c端,不影响本发明的融合蛋白中的亲和活性和发光活性。
融合标签可以是6×组氨酸标签(6×histag),以及gst、mbp、sumo等其他融合标签,根据融合标签不同,选用融合标签相对应的亲和纯化方法。在优选实施例中,融合标签是6×组氨酸标签。
在一个最优选的实施例中,本发明的融合蛋白序列如下seqidno:6或seqidno:7所示:
mgvkvlfaliciavaeaefasaeagitgtwynqhgstftvtagadgnltgqyenraqgtgcqnspytltgryngtklewrvewnnstenchsrtewrgqyqggaearintqwnltyeggsgpateqgqdtftkvkpsaasggggsggggskptennedfnivavasnfattdldadrgklpgkklplevlkemeanarkagctrgcliclshikctpkmkkfipgrchtyegdkesaqggigeaivdipeipgfkdlepmeqfiaqvdlcvdcttgclkglanvqcsdllkkwlpqrcatfaskiqgqvdkikgaggdhhhhhh(seqidno:6);
mgvkvlfaliciavaeaefasaeagitgtwynqhgstftvtagadgnltgqyenraqgtgcqnspytltgryngtklewrvewnnstenchsrtewrgqyqggaearintqwnltyeggsgpateqgqdtftkvkpsaasggggsggggskptennedfnivavasnfattdldadrgklpgkklplevlkeleanarkagctrgcliclshikctpkmkkfipgrchtyegdkesaqggigeaivdipeipgfkdlepleqfiaqvdlcvdcttgclkglanvqcsdllkkwlpqrcatfaskiqgqvdkikgaggdhhhhhh(seqidno:7)。
其中,带下划线的序列(mgvkvlfaliciavaea)表示信号肽;带下划线的序列(ggggsggggs)表示柔性肽;带下划线的序列(hhhhhh)表示6×组氨酸标签序列。
本发明的一种实施例提供一种分离的核酸,该分离的核酸编码第一方面的融合蛋白。
在优选实施例中,分离的核酸包括单体链霉亲和素全长蛋白编码序列和高斯荧光素酶全长蛋白编码序列或持续发光突变体编码序列,单体链霉亲和素全长蛋白编码序列与高斯荧光素酶全长蛋白编码序列或持续发光突变体编码序列通过连接序列连接。
需要说明的是,由于密码子的简并性,编码同一个多肽或蛋白的核苷酸序列可以不同,因此本发明的分离的核酸只要能够编码本发明的融合蛋白即可。对于序列没有特别限定。
在优选实施例中,单体链霉亲和素全长蛋白编码序列如下seqidno:8所示:
gagtttgcttccgccgaggctggcatcaccggcacatggtacaaccagcacggaagcaccttcacagtgaccgctggagctgatggaaacctgacaggacagtatgagaatagggctcagggcaccggctgccagaactctccctacacactgaccggcaggtataatggcaccaagctggagtggcgggtggagtggaacaatagcacagagaattgtcattctaggaccgagtggaggggacagtaccagggaggagctgaggctaggatcaacacacagtggaatctgacctatgagggaggatccggaccagctaccgagcagggccaggataccttcaccaaggtgaagccttccgccgctagc(seqidno:8)。
在优选实施例中,高斯荧光素酶全长蛋白编码序列如下seqidno:9所示:
aagccaacagagaacaatgaggatttcaacatcgtggctgtggccagcaattttgctaccacagacctggatgccgacagaggcaagctgccaggcaagaagctgcccctggaggtgctgaaggagatggaggctaacgctaggaaggctggatgtaccaggggatgcctgatctgtctgtctcacatcaagtgcacacctaagatgaagaagttcatcccaggccgctgtcatacctacgagggcgataaggagtccgctcagggaggaatcggagaggccatcgtggatatccccgagatccctggcttcaaggacctggagcccatggagcagtttatcgctcaggtggatctgtgcgtggactgtaccacaggctgcctgaagggcctggccaatgtgcagtgttccgacctgctgaagaagtggctgcctcagaggtgcgctacctttgccagcaagatccagggccaggtggataagatcaagggagctggaggcgac(seqidno:9)。
在优选实施例中,持续发光突变体编码序列如下seqidno:10所示:
aagccaacagagaacaatgaggatttcaacatcgtggctgtggccagcaattttgctaccacagacctggatgccgacagaggcaagctgccaggcaagaagctgcccctggaggtgctgaaggagctcgaggctaacgctaggaaggctggatgtaccaggggatgcctgatctgtctgtctcacatcaagtgcacacctaagatgaagaagttcatcccaggccgctgtcatacctacgagggcgataaggagtccgctcagggaggaatcggagaggccatcgtggatatccccgagatccctggcttcaaggacctggagcccctcgagcagtttatcgctcaggtggatctgtgcgtggactgtaccacaggctgcctgaagggcctggccaatgtgcagtgttccgacctgctgaagaagtggctgcctcagaggtgcgctacctttgccagcaagatccagggccaggtggataagatcaagggagctggaggcgac(seqidno:10)。其中,带下划线的密码子表示相对于高斯荧光素酶全长蛋白编码序列的突变碱基。
在优选实施例中,连接序列如下seqidno:11所示:
ggaggaggaggatctggaggaggaggatcc(seqidno:11)。
在优选实施例中,分离的核酸还包括:信号肽编码序列,该信号肽编码序列编码信号肽,该信号肽用于引导融合蛋白从表达细胞内向外分泌。
由于本发明的融合蛋白中信号肽可以位于融合蛋白的n端或c端,因此信号肽编码序列可以位于分离的核酸的5’端或3’端。在不影响本发明的融合蛋白中的亲和活性和发光活性的情况下,信号肽位于上述融合蛋白的n端,因此在优选实施例中,信号肽编码序列位于分离的核酸的5’端。
在优选实施例中,信号肽编码序列如下seqidno:12所示:
atgggcgtgaaggtgctgttcgccctgatctgcatcgccgtggctgaggcc(seqidno:12)。
在优选实施例中,分离的核酸还包括融合标签编码序列,该融合标签编码序列用于编码融合标签,该融合标签用于亲和纯化上述融合蛋白。
在优选实施例中,融合标签编码序列是编码6×组氨酸标签的序列。
融合标签编码序列可以位于分离的核酸的3’端或5’端,在优选实施例中,融合标签编码序列位于分离的核酸的3’端,不影响融合蛋白的亲和活性和发光活性部分的表达。
在一个最优选的实施例中,本发明的分离的核酸序列如下seqidno:13或seqidno:14所示:
atgggcgtgaaggtgctgttcgccctgatctgcatcgccgtggctgaggccgagtttgcttccgccgaggctggcatcaccggcacatggtacaaccagcacggaagcaccttcacagtgaccgctggagctgatggaaacctgacaggacagtatgagaatagggctcagggcaccggctgccagaactctccctacacactgaccggcaggtataatggcaccaagctggagtggcgggtggagtggaacaatagcacagagaattgtcattctaggaccgagtggaggggacagtaccagggaggagctgaggctaggatcaacacacagtggaatctgacctatgagggaggatccggaccagctaccgagcagggccaggataccttcaccaaggtgaagccttccgccgctagcggaggaggaggatctggaggaggaggatccaagccaacagagaacaatgaggatttcaacatcgtggctgtggccagcaattttgctaccacagacctggatgccgacagaggcaagctgccaggcaagaagctgcccctggaggtgctgaaggagatggaggctaacgctaggaaggctggatgtaccaggggatgcctgatctgtctgtctcacatcaagtgcacacctaagatgaagaagttcatcccaggccgctgtcatacctacgagggcgataaggagtccgctcagggaggaatcggagaggccatcgtggatatccccgagatccctggcttcaaggacctggagcccatggagcagtttatcgctcaggtggatctgtgcgtggactgtaccacaggctgcctgaagggcctggccaatgtgcagtgttccgacctgctgaagaagtggctgcctcagaggtgcgctacctttgccagcaagatccagggccaggtggataagatcaagggagctggaggcgaccaccatcaccatcaccat(seqidno:13);
atgggcgtgaaggtgctgttcgccctgatctgcatcgccgtggctgaggccgagtttgcttccgccgaggctggcatcaccggcacatggtacaaccagcacggaagcaccttcacagtgaccgctggagctgatggaaacctgacaggacagtatgagaatagggctcagggcaccggctgccagaactctccctacacactgaccggcaggtataatggcaccaagctggagtggcgggtggagtggaacaatagcacagagaattgtcattctaggaccgagtggaggggacagtaccagggaggagctgaggctaggatcaacacacagtggaatctgacctatgagggaggatccggaccagctaccgagcagggccaggataccttcaccaaggtgaagccttccgccgctagcggaggaggaggatctggaggaggaggatccaagccaacagagaacaatgaggatttcaacatcgtggctgtggccagcaattttgctaccacagacctggatgccgacagaggcaagctgccaggcaagaagctgcccctggaggtgctgaaggagctcgaggctaacgctaggaaggctggatgtaccaggggatgcctgatctgtctgtctcacatcaagtgcacacctaagatgaagaagttcatcccaggccgctgtcatacctacgagggcgataaggagtccgctcagggaggaatcggagaggccatcgtggatatccccgagatccctggcttcaaggacctggagcccctcgagcagtttatcgctcaggtggatctgtgcgtggactgtaccacaggctgcctgaagggcctggccaatgtgcagtgttccgacctgctgaagaagtggctgcctcagaggtgcgctacctttgccagcaagatccagggccaggtggataagatcaagggagctggaggcgaccaccatcaccatcaccat(seqidno:14)。
其中,seqidno:13或seqidno:14序列中,从5’端至3’端带下划线的序列分别是信号肽编码序列,柔性肽编码序列(连接序列),以及用于编码6×组氨酸标签的序列。
本发明的一种实施例提供一种表达载体,该表达载体包括编码本发明的融合蛋白的核酸序列,以及载体骨架序列。其中,载体骨架序列可以是任何合适的表达载体序列,例如,在一个实施例中,载体骨架序列是pcdna3.1(+)。
本发明的一种实施例提供一种重组宿主细胞,该重组宿主细胞内包含本发明的表达载体。宿主细胞可以是任何能够表达本发明的融合蛋白的宿主细胞,例如,在一个实施例中,是cho细胞。
本发明还提供融合蛋白、分离的核酸、表达载体或重组宿主细胞在生物素分子检测中的用途。
本发明的用途在于检测生物素分子,该生物素分子可以是独立存在的游离的形式,也可以是任何带有生物素标记的分子,例如生物素标记的蛋白。在一些实施例中,生物素标记于待检测的底物上,通过检测底物上的生物素分子可以检测底物。本发明在dna杂交、免疫检测、生化诊断等领域具有广泛用途。
本发明的融合蛋白能够方便、特异性地识别生物素分子,并具有催化底物氧化发光的功能,便于检测。相对于现有技术中的高斯荧光素酶与链霉亲和素的偶联蛋白,本发明的融合蛋白具有工艺简单,便于生产的优点。
以下通过实施例详细说明本发明的技术方案,应当理解,实施例仅是示例性的,不能理解为对本发明保护范围的限制。
实施例1:融合表达载体的构建
如图1所示,以monosa基因和高斯荧光素酶基因为模板,设计合成引物,进行pcr扩增,分别得到monosa和高斯荧光素酶基因扩增产物。以此pcr产物为基础,进行第二轮搭桥pcr,得到monosa和高斯荧光素酶融合基因扩增产物(序列如seqidno:13)。将pcr扩增产物用1%琼脂糖凝胶进行电泳检测,并用胶回收试剂盒对产物进行回收。回收到的产物和载体质粒pcdna3.1(+)分别用ecori和hindiii进行双酶切反应,反应结束后进行用1%琼脂糖凝胶进行电泳检测,并用胶回收试剂盒对产物进行回收。将pcr酶切回收产物和质粒酶切回收产物按摩尔比3:1进行混合,并用t4dna连接酶连接。连接产物转化top10感受态,涂布于含100ng/μl氨苄西林的lb平板。次日,于平板挑取单克隆,扩大培养后抽提质粒,pcr鉴定为阳性后,测序确保序列正确。
突变体g2l融合表达载体在成功制备的野生型融合蛋白表达载体基础上,通过设计含突变基因的引物进行质粒pcr(其中,monosa和持续发光突变体融合基因序列如seqidno:14所示),然后将pcr产物用dpni酶处理,37℃反应2h。产物转化top10感受态细胞,涂布于含100ng/μl氨苄西林的lb平板。次日,于平板挑取单克隆,扩大培养后抽提质粒,测序确保突变位点正确。
实施例2:融合表达载体的转染、表达和纯化
将25μg构建好的pcdna3.1-monosa-gluc融合表达质粒与1ml细胞培养基混合均匀。另取920μl细胞培养基与80μllipo2000转染试剂(invitrogen)混匀。将稀释好的表达载体和稀释好的转染试剂混匀,室温放置20分钟。准备25ml浓度为2×106/ml的细胞,将混好的pcdna3.1-monosa-gluc融合表达质粒及转染试剂混合物加入cho细胞,轻轻摇匀。37℃,8%co2条件下培养3天。
收集细胞培养液,6000rpm离心10min,收集上清。将上清过滤后流转镍柱,pbs平衡10ml。后用缓冲液(20mmtris,ph8.0,150mmnacl,20mm咪唑)冲洗10ml以去除杂蛋白,最后用缓冲液(20mmtris,ph8.0,150mmnacl,500mm咪唑)洗脱。用12%sds-page检测洗脱得到的蛋白,monosa-gluc融合蛋白的sds-page检测结果如图2所示。显示monosa-gluc融合蛋白表达成功。
pcdna3.1-monosa-g2l融合表达质粒参照上述同样操作。
实施例3:融合蛋白荧光素酶生物发光检测
将monosa-gluc融合蛋白和monosa-g2l融合蛋白分别用底物稀释液(50mmtris,ph8.0,100mmnacl)稀释至3nm,取10μl加入96孔酶标板。再加入用相同溶液稀释至10μm的底物腔肠素90μl,用酶标仪自发光模块读取发光强度,结果如图3所示,野生型monosa-gluc融合蛋白发光强度与同浓度的gluc蛋白相当。突变体融合蛋白monosa-g2l融合蛋白发光强度与gluc相当,但明显表现出持续发光特性,30s后发光没有明显减弱。
实施例4:融合蛋白与生物素结合能力的elisa测试实验
准备溶液:
pbs:138mmnacl,2.6mmkcl(溶于10mm磷酸钾溶液,ph7.4);
pbst:pbs加0.05%tween20;
封闭液:pbs加40μg/mlbsa;
稀释溶液:pbs加0.15μg/mlbsa。
(1)取一个96孔酶标板,每孔加入10ng/μg的生物素标记的bsa100μl,37℃包被1小时。
(2)倒掉包被液,用100μlpbst轻摇清洗3次,每次5分钟。
(3)每孔加入100μl封闭液,37℃封闭1小时。
(4)倒掉封闭液,用100μlpbst轻摇清洗3次,每次5分钟。
(5)用稀释液将monosa-gluc融合蛋白和monosa-g2l融合蛋白分别稀释至500nm,再按5倍的梯度稀释至100nm,20nm,4nm,0.8nm,0.16nm和0.032nm,将各个梯度的稀释液依次加入96孔板,每个梯度做3个重复孔。以稀释液不加融合蛋白为阴性对照。37℃结合1小时。
(6)倒掉板中溶液,用100μlpbst轻摇清洗3次,每次5分钟。
(7)将辣根过氧化物酶标记的sa抗体用稀释液稀释后加入96孔板,每孔100μl,37℃结合1小时。
(8)倒掉板中溶液,用100μlpbst轻摇清洗3次,每次5分钟。再用100μlpbs轻摇清洗3次,每次5分钟。
(9)用dmb发光试剂盒(生工生物公司)进行发光显色。按试剂盒说明书进行操作,显色后用酶标仪读取450nm的吸光值。
(10)将各浓度吸光值进行统计,取3个重复孔的平均数,做出浓度与吸光值的对应曲线,计算结合力常数。
结果如图4所示,显示野生型monosa-gluc融合蛋白和持续型发光突变体monosa-g2l对生物素的结合力均在10-9数量级。
以上应用了具体个例对本发明进行阐述,只是用于帮助理解本发明,并不用以限制本发明。对于本发明所属技术领域的技术人员,依据本发明的思想,还可以做出若干简单推演、变形或替换。
sequencelisting
<110>深圳华大生命科学研究院
<120>单体链霉亲和素和高斯荧光素酶的融合蛋白及其应用
<130>18i27211
<160>22
<170>patentinversion3.3
<210>1
<211>123
<212>prt
<213>人工序列
<400>1
gluphealaseralaglualaglyilethrglythrtrptyrasngln
151015
hisglyserthrphethrvalthralaglyalaaspglyasnleuthr
202530
glyglntyrgluasnargalaglnglythrglycysglnasnserpro
354045
tyrthrleuthrglyargtyrasnglythrlysleuglutrpargval
505560
glutrpasnasnserthrgluasncyshisserargthrglutrparg
65707580
glyglntyrglnglyglyalaglualaargileasnthrglntrpasn
859095
leuthrtyrgluglyglyserglyproalathrgluglnglyglnasp
100105110
thrphethrlysvallysproseralaalaser
115120
<210>2
<211>168
<212>prt
<213>人工序列
<400>2
lysprothrgluasnasngluasppheasnilevalalavalalaser
151015
asnphealathrthraspleuaspalaaspargglylysleuprogly
202530
lyslysleuproleugluvalleulysglumetglualaasnalaarg
354045
lysalaglycysthrargglycysleuilecysleuserhisilelys
505560
cysthrprolysmetlyslyspheileproglyargcyshisthrtyr
65707580
gluglyasplysgluseralaglnglyglyileglyglualaileval
859095
aspileprogluileproglyphelysaspleugluprometglugln
100105110
pheilealaglnvalaspleucysvalaspcysthrthrglycysleu
115120125
lysglyleualaasnvalglncysseraspleuleulyslystrpleu
130135140
proglnargcysalathrphealaserlysileglnglyglnvalasp
145150155160
lysilelysglyalaglyglyasp
165
<210>3
<211>168
<212>prt
<213>人工序列
<400>3
lysprothrgluasnasngluasppheasnilevalalavalalaser
151015
asnphealathrthraspleuaspalaaspargglylysleuprogly
202530
lyslysleuproleugluvalleulysgluleuglualaasnalaarg
354045
lysalaglycysthrargglycysleuilecysleuserhisilelys
505560
cysthrprolysmetlyslyspheileproglyargcyshisthrtyr
65707580
gluglyasplysgluseralaglnglyglyileglyglualaileval
859095
aspileprogluileproglyphelysaspleugluproleuglugln
100105110
pheilealaglnvalaspleucysvalaspcysthrthrglycysleu
115120125
lysglyleualaasnvalglncysseraspleuleulyslystrpleu
130135140
proglnargcysalathrphealaserlysileglnglyglnvalasp
145150155160
lysilelysglyalaglyglyasp
165
<210>4
<211>10
<212>prt
<213>人工序列
<400>4
glyglyglyglyserglyglyglyglyser
1510
<210>5
<211>17
<212>prt
<213>人工序列
<400>5
metglyvallysvalleuphealaleuilecysilealavalalaglu
151015
ala
<210>6
<211>324
<212>prt
<213>人工序列
<400>6
metglyvallysvalleuphealaleuilecysilealavalalaglu
151015
alagluphealaseralaglualaglyilethrglythrtrptyrasn
202530
glnhisglyserthrphethrvalthralaglyalaaspglyasnleu
354045
thrglyglntyrgluasnargalaglnglythrglycysglnasnser
505560
protyrthrleuthrglyargtyrasnglythrlysleuglutrparg
65707580
valglutrpasnasnserthrgluasncyshisserargthrglutrp
859095
argglyglntyrglnglyglyalaglualaargileasnthrglntrp
100105110
asnleuthrtyrgluglyglyserglyproalathrgluglnglygln
115120125
aspthrphethrlysvallysproseralaalaserglyglyglygly
130135140
serglyglyglyglyserlysprothrgluasnasngluasppheasn
145150155160
ilevalalavalalaserasnphealathrthraspleuaspalaasp
165170175
argglylysleuproglylyslysleuproleugluvalleulysglu
180185190
metglualaasnalaarglysalaglycysthrargglycysleuile
195200205
cysleuserhisilelyscysthrprolysmetlyslyspheilepro
210215220
glyargcyshisthrtyrgluglyasplysgluseralaglnglygly
225230235240
ileglyglualailevalaspileprogluileproglyphelysasp
245250255
leugluprometgluglnpheilealaglnvalaspleucysvalasp
260265270
cysthrthrglycysleulysglyleualaasnvalglncysserasp
275280285
leuleulyslystrpleuproglnargcysalathrphealaserlys
290295300
ileglnglyglnvalasplysilelysglyalaglyglyasphishis
305310315320
hishishishis
<210>7
<211>324
<212>prt
<213>人工序列
<400>7
metglyvallysvalleuphealaleuilecysilealavalalaglu
151015
alagluphealaseralaglualaglyilethrglythrtrptyrasn
202530
glnhisglyserthrphethrvalthralaglyalaaspglyasnleu
354045
thrglyglntyrgluasnargalaglnglythrglycysglnasnser
505560
protyrthrleuthrglyargtyrasnglythrlysleuglutrparg
65707580
valglutrpasnasnserthrgluasncyshisserargthrglutrp
859095
argglyglntyrglnglyglyalaglualaargileasnthrglntrp
100105110
asnleuthrtyrgluglyglyserglyproalathrgluglnglygln
115120125
aspthrphethrlysvallysproseralaalaserglyglyglygly
130135140
serglyglyglyglyserlysprothrgluasnasngluasppheasn
145150155160
ilevalalavalalaserasnphealathrthraspleuaspalaasp
165170175
argglylysleuproglylyslysleuproleugluvalleulysglu
180185190
leuglualaasnalaarglysalaglycysthrargglycysleuile
195200205
cysleuserhisilelyscysthrprolysmetlyslyspheilepro
210215220
glyargcyshisthrtyrgluglyasplysgluseralaglnglygly
225230235240
ileglyglualailevalaspileprogluileproglyphelysasp
245250255
leugluproleugluglnpheilealaglnvalaspleucysvalasp
260265270
cysthrthrglycysleulysglyleualaasnvalglncysserasp
275280285
leuleulyslystrpleuproglnargcysalathrphealaserlys
290295300
ileglnglyglnvalasplysilelysglyalaglyglyasphishis
305310315320
hishishishis
<210>8
<211>369
<212>dna
<213>人工序列
<400>8
gagtttgcttccgccgaggctggcatcaccggcacatggtacaaccagcacggaagcacc60
ttcacagtgaccgctggagctgatggaaacctgacaggacagtatgagaatagggctcag120
ggcaccggctgccagaactctccctacacactgaccggcaggtataatggcaccaagctg180
gagtggcgggtggagtggaacaatagcacagagaattgtcattctaggaccgagtggagg240
ggacagtaccagggaggagctgaggctaggatcaacacacagtggaatctgacctatgag300
ggaggatccggaccagctaccgagcagggccaggataccttcaccaaggtgaagccttcc360
gccgctagc369
<210>9
<211>504
<212>dna
<213>人工序列
<400>9
aagccaacagagaacaatgaggatttcaacatcgtggctgtggccagcaattttgctacc60
acagacctggatgccgacagaggcaagctgccaggcaagaagctgcccctggaggtgctg120
aaggagatggaggctaacgctaggaaggctggatgtaccaggggatgcctgatctgtctg180
tctcacatcaagtgcacacctaagatgaagaagttcatcccaggccgctgtcatacctac240
gagggcgataaggagtccgctcagggaggaatcggagaggccatcgtggatatccccgag300
atccctggcttcaaggacctggagcccatggagcagtttatcgctcaggtggatctgtgc360
gtggactgtaccacaggctgcctgaagggcctggccaatgtgcagtgttccgacctgctg420
aagaagtggctgcctcagaggtgcgctacctttgccagcaagatccagggccaggtggat480
aagatcaagggagctggaggcgac504
<210>10
<211>504
<212>dna
<213>人工序列
<400>10
aagccaacagagaacaatgaggatttcaacatcgtggctgtggccagcaattttgctacc60
acagacctggatgccgacagaggcaagctgccaggcaagaagctgcccctggaggtgctg120
aaggagctcgaggctaacgctaggaaggctggatgtaccaggggatgcctgatctgtctg180
tctcacatcaagtgcacacctaagatgaagaagttcatcccaggccgctgtcatacctac240
gagggcgataaggagtccgctcagggaggaatcggagaggccatcgtggatatccccgag300
atccctggcttcaaggacctggagcccctcgagcagtttatcgctcaggtggatctgtgc360
gtggactgtaccacaggctgcctgaagggcctggccaatgtgcagtgttccgacctgctg420
aagaagtggctgcctcagaggtgcgctacctttgccagcaagatccagggccaggtggat480
aagatcaagggagctggaggcgac504
<210>11
<211>30
<212>dna
<213>人工序列
<400>11
ggaggaggaggatctggaggaggaggatcc30
<210>12
<211>51
<212>dna
<213>人工序列
<400>12
atgggcgtgaaggtgctgttcgccctgatctgcatcgccgtggctgaggcc51
<210>13
<211>972
<212>dna
<213>人工序列
<400>13
atgggcgtgaaggtgctgttcgccctgatctgcatcgccgtggctgaggccgagtttgct60
tccgccgaggctggcatcaccggcacatggtacaaccagcacggaagcaccttcacagtg120
accgctggagctgatggaaacctgacaggacagtatgagaatagggctcagggcaccggc180
tgccagaactctccctacacactgaccggcaggtataatggcaccaagctggagtggcgg240
gtggagtggaacaatagcacagagaattgtcattctaggaccgagtggaggggacagtac300
cagggaggagctgaggctaggatcaacacacagtggaatctgacctatgagggaggatcc360
ggaccagctaccgagcagggccaggataccttcaccaaggtgaagccttccgccgctagc420
ggaggaggaggatctggaggaggaggatccaagccaacagagaacaatgaggatttcaac480
atcgtggctgtggccagcaattttgctaccacagacctggatgccgacagaggcaagctg540
ccaggcaagaagctgcccctggaggtgctgaaggagatggaggctaacgctaggaaggct600
ggatgtaccaggggatgcctgatctgtctgtctcacatcaagtgcacacctaagatgaag660
aagttcatcccaggccgctgtcatacctacgagggcgataaggagtccgctcagggagga720
atcggagaggccatcgtggatatccccgagatccctggcttcaaggacctggagcccatg780
gagcagtttatcgctcaggtggatctgtgcgtggactgtaccacaggctgcctgaagggc840
ctggccaatgtgcagtgttccgacctgctgaagaagtggctgcctcagaggtgcgctacc900
tttgccagcaagatccagggccaggtggataagatcaagggagctggaggcgaccaccat960
caccatcaccat972
<210>14
<211>972
<212>dna
<213>人工序列
<400>14
atgggcgtgaaggtgctgttcgccctgatctgcatcgccgtggctgaggccgagtttgct60
tccgccgaggctggcatcaccggcacatggtacaaccagcacggaagcaccttcacagtg120
accgctggagctgatggaaacctgacaggacagtatgagaatagggctcagggcaccggc180
tgccagaactctccctacacactgaccggcaggtataatggcaccaagctggagtggcgg240
gtggagtggaacaatagcacagagaattgtcattctaggaccgagtggaggggacagtac300
cagggaggagctgaggctaggatcaacacacagtggaatctgacctatgagggaggatcc360
ggaccagctaccgagcagggccaggataccttcaccaaggtgaagccttccgccgctagc420
ggaggaggaggatctggaggaggaggatccaagccaacagagaacaatgaggatttcaac480
atcgtggctgtggccagcaattttgctaccacagacctggatgccgacagaggcaagctg540
ccaggcaagaagctgcccctggaggtgctgaaggagctcgaggctaacgctaggaaggct600
ggatgtaccaggggatgcctgatctgtctgtctcacatcaagtgcacacctaagatgaag660
aagttcatcccaggccgctgtcatacctacgagggcgataaggagtccgctcagggagga720
atcggagaggccatcgtggatatccccgagatccctggcttcaaggacctggagcccctc780
gagcagtttatcgctcaggtggatctgtgcgtggactgtaccacaggctgcctgaagggc840
ctggccaatgtgcagtgttccgacctgctgaagaagtggctgcctcagaggtgcgctacc900
tttgccagcaagatccagggccaggtggataagatcaagggagctggaggcgaccaccat960
caccatcaccat972
<210>15
<211>6
<212>prt
<213>人工序列
<400>15
glyglyglyglyglyser
15
<210>16
<211>11
<212>prt
<213>人工序列
<400>16
glyglnglyglnglyglnglyglnglyglngly
1510
<210>17
<211>15
<212>prt
<213>人工序列
<400>17
glyserthrserglyserglylysserserglulysglylysgly
151015
<210>18
<211>10
<212>prt
<213>人工序列
<400>18
valproglyvalglyvalproglyvalgly
1510
<210>19
<211>8
<212>prt
<213>人工序列
<400>19
seralaproglythrproserarg
15
<210>20
<211>14
<212>prt
<213>人工序列
<400>20
gluglylysserserglyserglysergluserlysgluphe
1510
<210>21
<211>6
<212>prt
<213>人工序列
<400>21
glyserglyglysergly
15
<210>22
<211>8
<212>prt
<213>人工序列
<400>22
glyserglyglyserglyglygly
15
- 还没有人留言评论。精彩留言会获得点赞!