包含细菌甘露聚糖酶的洗涤剂组合物的制作方法
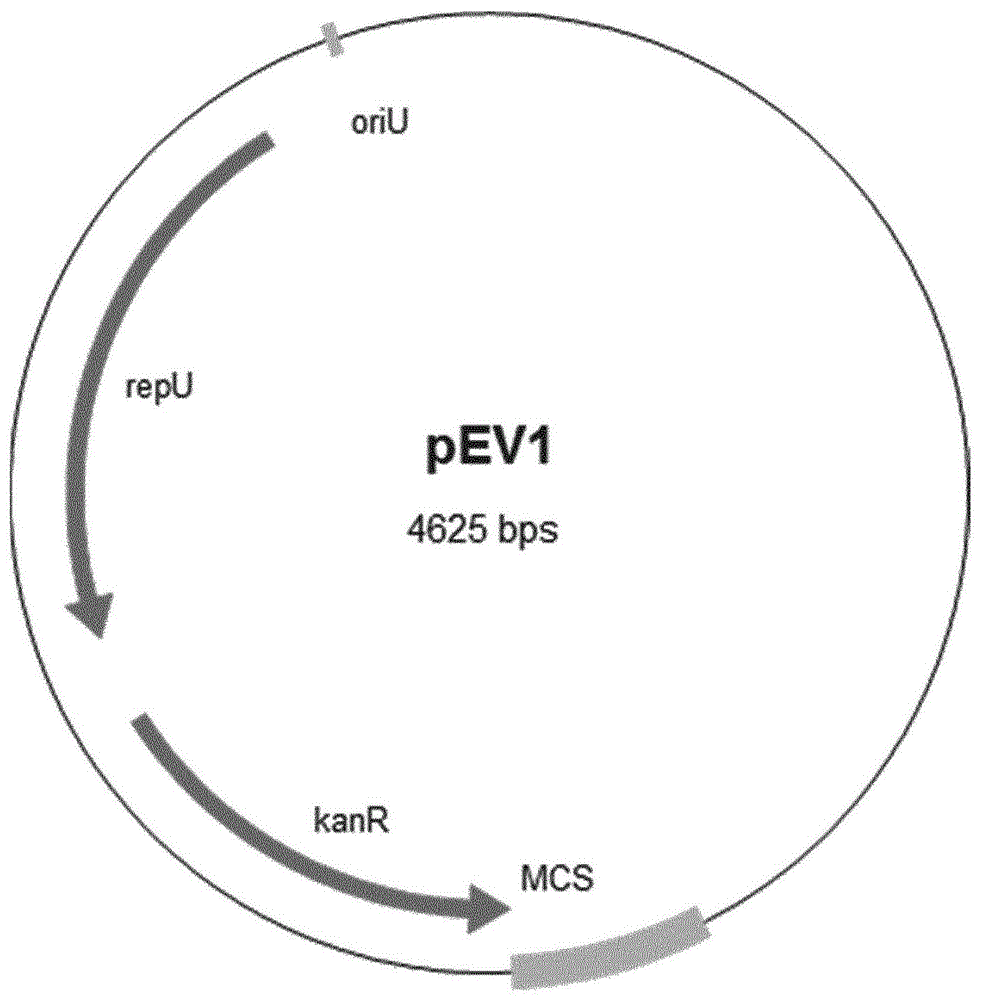
本发明涉及包含细菌甘露聚糖酶的新的洗涤剂组合物。所述包含细菌甘露聚糖酶的洗涤剂组合物可用于希望甘露聚糖降解或改性的洗衣和清洁应用中。本发明还涉及所述洗涤剂组合物在洗衣和清洁应用中的用途以及降解甘露聚糖的方法。
背景技术:
:甘露聚糖是在多种植物中发现的含有甘露糖的多糖。甘露聚糖在水性环境中溶解性差,并且它们的物理化学性质产生粘性分散体。此外,甘露聚糖具有高度的水结合能力。所有这些特征在包括酿造、烘焙、动物营养以及洗衣和清洁应用在内的若干行业中都引起问题。在基于植物的饮食中,存在不同的β-甘露聚糖,并且取决于它们的量和性质,它们可以损害营养物消化、微生物定殖和生长性能。甘露聚糖的酶促降解降低了高水溶性甘露聚糖的消化物粘度,并导致甘露寡糖的产生,甘露寡糖可形成豆科中存在的水不溶性线性甘露聚糖。甘露聚糖酶增加所有单胃动物的平均日增重、饲料效率、重量均匀度和存活率。对于动物饲料应用,例如用谷物食料喂养单胃动物,甘露聚糖是造成肠内容物粘稠的因素,因此它对饲料消化性和动物生长速率产生不利影响。对于反刍动物,甘露聚糖代表了纤维摄入的重要组分,甘露聚糖的更完全消化将促进更高的饲料转化效率。对于洗衣和清洁应用,包含甘露聚糖酶的洗涤剂组合物可用于降解甘露聚糖。然而,提供在不同的储存和使用条件下稳定同时仍显示出良好的甘露聚糖降解活性的甘露聚糖酶是困难的。本发明的一个目的是提供洗涤剂组合物,其包含当应用于这些洗涤剂组合物中时表现出甘露聚糖酶活性的新的酶。本发明的另一个目的是提供对含甘露聚糖的污渍的去污性能增加的洗涤剂组合物。技术实现要素:根据本发明的第一方面,提供了一种洗涤剂组合物,其包含至少一种酶,所述酶的氨基酸序列与seqidno:16(man7)的氨基酸序列具有至少74%的序列同一性、和/或与seqidno:12(man6)的氨基酸序列具有至少93%的序列同一性、和/或与seqidno:20(man14)的氨基酸序列具有至少79%的序列同一性。在本发明的一个实施方式中,所述至少一种酶的氨基酸序列与seqidno:16具有至少75%、至少76%、至少77%、至少78%、至少79%、至少80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%的序列同一性。在本发明的一个实施方式中,所述至少一种酶的氨基酸序列与seqidno:12具有至少94%、至少95%、至少96%、至少97%、至少98%或至少99%的序列同一性。在本发明的一个进一步的实施方式中,所述至少一种酶具有甘露聚糖降解活性。包含在本发明的洗涤剂组合物中的甘露聚糖酶适用于在各种化学环境中、优选在洗涤剂组合物中降解和改性含甘露聚糖的材料。在本发明的一个实施方式中,所述洗涤剂组合物还包含一种或多种其他酶,所述其他酶选自蛋白酶、脂肪酶、角质酶、淀粉酶、糖酶、纤维素酶、果胶酶、果胶酸裂解酶、甘露聚糖酶、阿拉伯糖酶、半乳聚糖酶、木聚糖酶、氧化酶、黄原胶酶、漆酶和/或过氧化物酶,优选选自蛋白酶、淀粉酶、纤维素酶和脂肪酶。在本发明的一个进一步的实施方式中,所述洗涤剂组合物为棒剂、均质片剂、具有两个或更多个层的片剂、具有一个或多个隔室的小袋剂、常规或致密粉末剂、颗粒剂、糊剂、凝胶剂或者常规、致密或浓缩液体剂的形式。在一个实施方式中,所述洗涤剂组合物可以是衣物洗涤剂组合物,优选为液体或固体衣物洗涤剂组合物。本发明还涉及如本文公开的洗涤剂组合物在降解甘露聚糖中的用途。在一个进一步的实施方式中,本发明涉及如本文公开的洗涤剂组合物在洗衣过程中的用途。本发明还涉及一种从表面去除污渍的方法,所述方法包括使所述表面与如本文公开的洗涤剂组合物相接触。本发明还涉及一种降解甘露聚糖的方法,所述方法包括将如本文公开的洗涤剂组合物应用于甘露聚糖,优选地,其中所述甘露聚糖在纺织品的表面上。附图说明图1显示了用于在芽孢杆菌中复制的载体pev1的示意图。图2示意性地显示了用于转化里氏木霉(trichodermareesei)原生质体以过量产生重组甘露聚糖酶蛋白(man6、man7和man14)的表达盒。所述甘露聚糖酶基因在里氏木霉(t.reesei)cel7a/cbh1启动子(pcbh1)的控制下,并通过使用里氏木霉cel7a/cbh1终止子序列(tab/71)确保转录的终止。包含amds基因作为转化标记。图3显示了在40mmbritton-robinson缓冲液ph7中使用10分钟反应时间测定的重组man6、man7和man14(芽孢杆菌(bacillus)产生的)甘露聚糖酶的温度曲线,使用天青蛋白交联的角豆半乳甘露聚糖作为底物。所有测量至少一式两份进行。数据点是单独测量的平均值。图4描述了在ph4至ph11的40mmbritton-robinson缓冲液中,ph对重组man6、man7和man14(芽孢杆菌产生的)甘露聚糖酶蛋白的活性的效应。反应温度为50℃,反应时间为10分钟。使用天青蛋白交联的角豆半乳甘露聚糖作为底物。所有测量至少一式两份进行。数据点是单独测量的平均值。图5显示了细菌甘露聚糖酶的sdspage分析。图6描述了在4.4g/l商业重垢液体洗涤剂a存在下,在40℃、16°dh、60min、ph约8.3和按活性单位计量配给的酶下,man6和man7(在芽孢杆菌和木霉(trichoderma)中产生)按光亮度增加(4个污渍的δl*之和)的去污性能。商业制品4.0l用于比较。图7描述了在4.4g/l商业重垢液体洗涤剂a存在下,在40℃、16°dh、60min、ph约8.3和按活性酶蛋白(aep)配量的酶下,man6和man7(在芽孢杆菌中产生)按光亮度增加(4个污渍的δl*之和)的去污性能。商业制品4.0l用于比较。图8描述了在3.8g/l商业护色洗涤剂粉末存在下,在40℃、16°dh、60min、ph约10和按活性单位配量的酶下,man6和man7(在芽孢杆菌中产生)按光亮度增加(4个污渍的δl*之和)的去污性能。商业制品4.0l用于比较。图9描述了在3.8g/l商业护色洗涤剂粉末存在下,在40℃、16°dh、60min、ph约10和按活性酶蛋白配量的酶下,man6和man7(在芽孢杆菌中产生)按光亮度增加(4个污渍的δl*之和)的去污性能。商业制品4.0l用于比较。图10描述了在4.2g/l商业漂白洗涤剂粉末存在下,在40℃、16°dh、60min、ph约10和按活性酶蛋白配量的酶下,man6和man7(在芽孢杆菌中产生)按光亮度增加(3个污渍的δl*之和)的去污性能。商业制品4.0l用于比较。图11在5g/l商业重垢液体洗涤剂b存在下,在40℃、16°dh、60min、ph约8.3和按活性单位配量的酶下,man14(在芽孢杆菌中产生)按光亮度增加(2个污渍的δl*之和)的去污性能。商业制品4.0l用于比较。图12在5g/l商业重垢液体洗涤剂b存在下,在40℃、16°dh、60min、ph约8.3和按活性酶蛋白配量的酶下,man14(在芽孢杆菌中产生)按光亮度增加(2个污渍的δl*之和)的去污性能。商业制品4.0l用于比较。图13描述了man6和man7(在芽孢杆菌中产生)在37℃下液体洗涤剂(omocolor)中的稳定性。商业制品4.0l用于比较。图14描述了man6(在芽孢杆菌中产生)在商业重垢液体洗涤剂a中的稳定性。商业制品4.0l用于比较。序列表seqidno:1寡核苷酸引物man6_1的序列seqidno:2寡核苷酸引物man6_2的序列seqidno:3寡核苷酸引物man7_1的序列seqidno:4寡核苷酸引物man7_2的序列seqidno:5寡核苷酸引物man14_1的序列seqidno:6寡核苷酸引物man14_2的序列seqidno:7寡核苷酸引物vec_1的序列seqidno:8寡核苷酸引物vec_2的序列seqidno:9克劳氏芽孢杆菌(bacillusclausii)man6的核苷酸序列seqidno:10克劳氏芽孢杆菌man6的核苷酸序列,没有信号肽编码序列并具有对里氏木霉的密码子优化seqidno:11克劳氏芽孢杆菌man6的推断的氨基酸序列seqidno:12克劳氏芽孢杆菌man6的推断的氨基酸序列,没有信号肽seqidno:13解半纤维素芽胞杆菌(bacillushemicellulosilyticus)man7的核苷酸序列seqidno:14解半纤维素芽胞杆菌man7的核苷酸序列,没有信号肽编码序列并具有对里氏木霉的密码子优化seqidno:15解半纤维素芽孢杆菌man7的推断的氨基酸序列seqidno:16解半纤维素芽孢杆菌man7的推断的氨基酸序列,没有信号肽seqidno:17土壤枝芽孢杆菌(virgibacillussoli)man14的核苷酸序列seqidno:18土壤枝芽孢杆菌man14的核苷酸序列,没有信号肽编码序列并具有对里氏木霉的密码子优化seqidno:19土壤枝芽孢杆菌man14的推断的氨基酸序列seqidno:20土壤枝芽孢杆菌man14的推断的氨基酸序列,没有信号肽seqidno:21寡核苷酸引物bman1的序列seqidno:22寡核苷酸引物bman2的序列seqidno:23寡核苷酸引物bman3的序列seqidno:24寡核苷酸引物bman4的序列seqidno:25短小芽孢杆菌(bacilluspumilus)man31的核苷酸序列seqidno:26短小芽孢杆菌man31的推断的氨基酸序列seqidno:27解淀粉芽孢杆菌(bacillusamyloliquefaciens)man32的核苷酸序列seqidno:28解淀粉芽孢杆菌man32的推断的氨基酸序列seqidno:29木聚糖兼性芽胞杆菌(amphibacillusxylanus)man33的核苷酸序列seqidno:30木聚糖兼性芽胞杆菌man33的推断的氨基酸序列seqidno:31多粘类芽孢杆菌(paenibacilluspolymyxa)man34的核苷酸序列seqidno:32多粘类芽孢杆菌man34的推断的氨基酸序列seqidno:33解半纤维素芽胞杆菌man35的核苷酸序列seqidno:34解半纤维素芽胞杆菌man35的推断的氨基酸序列seqidno:35嗜碱芽孢杆菌(bacillusalcalophilus)man36的核苷酸序列seqidno:36嗜碱芽孢杆菌man36的推断的氨基酸序列seqidno:37芽孢杆菌菌种(bacillussp.)man37的核苷酸序列seqidno:38芽孢杆菌菌种man37的推断的氨基酸序列seqidno:39环状芽孢杆菌(bacilluscirculans)man38的核苷酸序列seqidno:40环状芽孢杆菌man38的推断的氨基酸序列seqidno:41类芽孢杆菌菌种(paenibacillussp.)man39的核苷酸序列seqidno:42类芽孢杆菌菌种man39的推断的氨基酸序列seqidno:43环状芽孢杆菌man40的核苷酸序列seqidno:44环状芽孢杆菌man40的推断的氨基酸序列seqidno:45尼氏芽孢杆菌(bacillusnealsonii)man41的核苷酸序列seqidno:46尼氏芽孢杆菌man41的推断的氨基酸序列seqidno:47环状芽孢杆菌man42的核苷酸序列seqidno:48环状芽孢杆菌man42的核苷酸序列具体实施方式用于本文中时,“分离的”是指自然界中不存在的形式的或环境中的物质。分离的物质的非限制性例子包括(1)任何非天然存在的物质,(2)从与其天然相关的一种或多种或所有天然存在的成分中至少部分去除的任何物质,包括任何酶、变体、核酸、蛋白、肽或辅因子;(3)相对于自然界中发现的物质被人工修饰的任何物质;或(4)通过相对于与其天然相关的其他组分增加或减少所述物质的量进行修饰的任何物质(例如,宿主细胞中的重组体产生,编码该物质的基因的一个或多个拷贝,以及使用与编码该物质的基因天然相关的启动子的替代启动子)。在一个实施方式中,分离的是本发明的多肽、酶、多核苷酸、宿主细胞或组合物。用于本文中时,术语“包含”涵盖“包括”、“含有”和“理解”的更广泛含义,以及“由...组成”和“仅由...组成”的较窄表达。用于本文中时,“片段”是指缺失一个或多个氨基酸或核苷酸的蛋白质或多核苷酸。在dna的背景下,片段包括任何长度的单链和双链dna。片段可以是活性片段,其具有蛋白质或多核苷酸的生物学功能,例如酶活性或调节活性。片段也可以是无活性片段,即它不具有天然蛋白质或多核苷酸的一种或多种生物效应。用于本文中时,“变体”是指插入或缺失一个或多个核苷酸/氨基酸或经化学修饰的序列(核苷酸或氨基酸)的片段。用于本文中时,“肽”和“多肽”是包含多个连续聚合的氨基酸残基的氨基酸序列。出于本发明的目的,肽是包含至多20个氨基酸残基的分子,并且多肽包含多于20个氨基酸残基。肽或多肽可包括修饰的氨基酸残基、不由密码子编码的天然存在的氨基酸残基和非天然存在的氨基酸残基。用于本文中时,“蛋白质”可以指任何大小的肽或多肽。蛋白质可以是酶、蛋白、抗体、膜蛋白、肽激素、调节剂或任何其他蛋白。术语“多核苷酸”表示从5'至3'末端读取的脱氧核糖核苷酸或核糖核苷酸碱基的单链或双链聚合物。多核苷酸包括rna和dna,并且可以从天然来源分离、在体外合成、或由天然分子和合成分子的组合制备。用于本文中时,多核苷酸背景中的“修饰”、“修饰的”和类似术语是指在多核苷酸的编码或非编码区中的修饰,所述编码或非编码区例如调节序列、5'非翻译区、3'非翻译区、上调遗传元件、下调遗传元件、增强子、抑制子、启动子、外显子或内含子区域。在一些实施方式中,修饰可以仅是结构性的,对多核苷酸的生物效应、作用或功能没有影响。在其他实施方式中,修饰是提供多核苷酸的生物效应、作用或功能方面变化的结构修饰。这样的修饰可以增强、抑制或改变多核苷酸的生物功能。用于本文中时,“同一性”是指在两个比对序列中有残基存在的位置数量上,这两个序列之间的氨基酸残基精确匹配的百分比。当一个序列具有的残基在另一个序列中没有相应残基时,比对程序允许比对中的间隙,并且该位置不计入同一性计算的分母中。同一性是使用embl-ebi网站(www.ebi.ac.uk/tools/psa/emboss_needle/)上的成对序列比对(pairwisesequencealignment)工具embossneedle确定的值。用于本文中时,“宿主细胞”是指容易受到用包含多核苷酸的核酸构建体或表达载体转化、转染、转导、配合、杂交等的任何细胞类型。术语“宿主细胞”包括由于复制期间发生的突变而不相同的任何后代。宿主细胞的非限制性例子是真菌细胞,来自子囊菌门(ascomycota)、盘菌亚门(pezizomycotina)的丝状真菌细胞;优选选自粪壳菌纲(sordariomycetes)、肉座菌亚纲(hypocreomycetidae)、肉座菌目(hypocreales)和小囊菌目(microascales)以及曲霉属(aspergillus)、金孢霉属(chrysosporium)、毁丝霉属(myceliophthora)和腐质霉属(humicola)的成员;更优选选自肉座菌科(hypocreacea)、丛赤壳科(nectriaceae)、麦角菌科(clavicipitaceae)、小囊菌科(microascaceae),以及木霉属(trichoderma)(肉座菌属(hypocrea)的无性型)、镰刀菌属(fusarium)、赤霉属(gibberella)、丛赤壳属(nectria)、葡萄状穗霉属(stachybotrys)、麦角菌属(claviceps)、绿僵菌属(metarhizium)、villosiclava、线虫草属(ophiocordyceps)、头孢霉属(cephalosporium)和赛多孢子菌属(scedosporium);更优选选自里氏木霉(红褐肉座菌(hypocreajecorina))、桔绿木霉(t.citrinoviridae)、长枝木霉(t.longibrachiatum)、绿木霉(t.virens)、哈茨木霉(t.harzianum)、棘孢木霉(t.asperellum)、深绿木霉(t.atroviridae)、近里氏木霉(t.parareesei)、尖孢镰刀菌(fusariumoxysporum)、禾谷镰刀菌(f.gramineanum)、假禾谷镰刀菌(f.pseudograminearum)、f.venenatum、藤仓赤霉(gibberellafujikuroi)、串珠赤霉(g.moniliformis)、玉蜀黍赤霉(g.zeaea)、鞭毛藻丛赤壳菌(血赤壳菌)(nectria(haematonectria)haematococca)、黑葡萄穗霉(stachybotryschartarum)、s.chlorohalonata、黑麦麦角菌(clavicepspurpurea)、黄绿绿僵菌(metarhiziumacridum)、金龟子绿僵菌(m.anisopliae)、villosiclavavirens、冬虫夏草菌(ophiocordycepssinensis)、产黄枝顶孢霉(头孢霉)(acremonium((cephalosporium))chrysogenum)和尖端赛多孢子菌(scedosporiumapiospermum)以及黑曲霉(aspergillusniger)、泡盛曲霉(a.awamori)、米曲霉(a.oryzae)、chrysosporiumlucknowense、嗜热毁丝霉(myceliophthorathermophila)、特异腐质霉(humicolainsolens)和灰腐质霉(humicolagrisea);最优选是里氏木霉。宿主细胞的非限制性例子是细菌细胞,优选革兰氏阳性杆菌(例如枯草芽孢杆菌(b.subtilis)、地衣芽孢杆菌(b.licheniformis)、巨大芽孢杆菌(b.megaterium)、解淀粉芽孢杆菌(b.amyloliquefaciens)、短小芽孢杆菌(b.pumilus))、放线菌(例如链霉菌属(链霉属)菌种)和酵母(例如酿酒酵母(saccharomycescerevisiae)、巴斯德毕赤氏酵母(pichiapastoris)、解脂耶氏酵母(yarrowialipolytica))。在一个示例性实施方式中,宿主细胞是真菌细胞,优选丝状真菌细胞,例如木霉属或里氏木霉。在一个示例性实施方式中,宿主细胞是细菌细胞,优选革兰氏阳性芽孢杆菌细胞,例如枯草芽孢杆菌、地衣芽孢杆菌、巨大芽孢杆菌、解淀粉芽孢杆菌、短小芽孢杆菌。“重组细胞”或“重组宿主细胞”是指经遗传修饰或改变以包含对所述细胞或宿主细胞不是天然的核酸序列的细胞或宿主细胞。在一个实施方式中,遗传修饰包括将所述多核苷酸整合到宿主细胞的基因组中。在另一个实施方式中,与宿主细胞相比,所述多核苷酸是外源的。用于本文中时,“表达”包括参与宿主细胞中产生多肽的任何步骤,包括但不限于转录、翻译、翻译后修饰和分泌。表达之后可以收获、即回收宿主细胞或表达产物。术语“表达载体”表示线性或环状的dna分子,其包含编码目的多肽的区段,所述区段与提供其转录的其他区段可操作地连接。这样的其他区段可包括启动子和终止子序列,并且可任选包括一个或多个复制起点、一个或多个选择性标记、增强子、多腺苷酸化信号、载体等。表达载体通常源自于质粒或病毒dna,或可含有这两者的元件。表达载体可以是方便进行重组dna程序的任何表达载体,并且载体的选择往往取决于所述载体将要导入的宿主细胞。因此,所述载体可以是自主复制载体,即作为染色体外实体存在、其复制独立于染色体复制的载体,例如质粒。或者,所述载体可以是当被导入宿主细胞时整合到宿主细胞基因组中并与其所整合的染色体一起复制的载体。本文中关于多肽或蛋白质表达的术语“重组性表达的”或“重组表达的”根据本领域的标准定义来定义。本文所用的关于特定微生物来源的术语“获得自”和“可获得的”是指多核苷酸和/或多肽由特定来源产生(同源表达),或由其中已插入来自所述来源的基因的细胞产生(异源表达)。术语“酶组合物”是指来自单一微生物物种的常规酶促发酵产物,可能是分离和纯化的,这样的制剂通常包含许多不同的酶活性;或单组分酶的混合物,优选通过使用常规重组技术从细菌或者真菌物种得来的酶,所述酶已经分别发酵并且可能分离和纯化,并且其可以来源于不同物种,优选真菌或细菌物种,或微生物的发酵产物,所述微生物充当用于表达重组甘露聚糖酶的宿主细胞,但该微生物同时产生其他酶。当提及dna区段时,术语“可操作地连接”表示所述区段被安排成使得所述区段为了它们的预期目的一齐起作用,例如在启动子中启动转录并通过编码区段进行到终止子。术语“启动子”表示含有dna序列的基因的一部分,其提供rna聚合酶的结合和转录的起始。启动子序列通常、但不总是存在于基因的5'非编码区中。术语“分泌信号序列”表示一种编码多肽(“分泌肽”)的dna序列,所述多肽作为较大的多肽的组分,引导该较大的多肽通过合成它的宿主细胞的分泌途径。分泌信号序列可以是天然的,或者它可以用来自另一来源的分泌信号序列或载体序列替换。取决于宿主细胞,可以切割所述较大的肽以在运输通过分泌途径期间除去所述分泌肽。术语“核心区”表示酶的结构域,其可以经过或可以不经过修饰或改变,但保持其原始活性;本领域已知的催化结构域保持功能性。术语“接头”或“间隔区”是指包含至少两个氨基酸的多肽,其可存在于多结构域蛋白、例如包含酶核心和结合结构域例如碳水化合物结合模块(cbm)的酶或任何其他酶杂合体的结构域之间,或在表达为融合多肽的两种蛋白或多肽、例如包含两种核心酶的融合蛋白之间。例如,通过将编码酶核心的dna序列、编码接头的dna序列和编码cbm的dna序列依次融合到一个开放阅读框中并表达该构建体来提供酶核心与cbm的融合蛋白。除非另有说明,术语“洗涤剂组合物”包括颗粒或粉末形式的通用或重垢洗涤剂,尤其是清洁洗涤剂;液体、凝胶或糊剂形式的通用洗涤剂,尤其是所谓的重垢液体(hdl)类型;液体精细织物洗涤剂;手洗洗碗剂或轻垢洗碗剂,尤其是高发泡型洗碗剂;机器洗碗剂,包括用于家庭和机构用途的各种片剂、颗粒、液体和漂洗辅助类型;液体清洁和消毒剂,汽车或地毯香波,浴室清洁剂;金属清洁剂;以及清洁助剂,例如漂白添加剂和“污渍棒”或预处理类型。术语“洗涤剂组合物”和“洗涤剂制剂”用于指意欲用于洗涤介质中以清洁脏污物体的混合物。在一些实施方案中,该术语用于指洗涤织物和/或衣服(例如“衣物洗涤剂”)。在另外的实施方式中,该术语是指其他洗涤剂,例如用于清洁餐具、刀具等的那些洗涤剂(例如,“洗碗洗涤剂”)。并未意欲将本发明限于任何特定的洗涤剂制剂或组合物。意欲除了本发明的变体之外,该术语还包括可以含有例如表面活性剂、助洗剂、螯合剂、漂白剂体系或漂白剂组分、聚合物、织物调理剂、泡沫促进剂、抑泡剂、染料,香料、褐变抑制剂、光学增亮剂、杀菌剂、杀真菌剂、污垢悬浮剂、抗腐蚀剂、酶抑制剂或稳定剂、酶活化剂、转移酶、水解酶、氧化还原酶、上蓝剂和荧光染料、抗氧化剂和增溶剂的洗涤剂。术语“纺织品”是指任何纺织材料,包括纱线、纱线中间体、纤维、非织造材料、天然材料、合成材料和任何其他纺织材料、由这些材料制成的织物和由织物制成的产品(例如,服装和其他制品)。纺织品或织物可以是针织物、机织物、牛仔布、无纺布、毡、纱线和毛巾布的形式。纺织品可以是纤维素基的,例如天然纤维素,包括棉、亚麻、黄麻、苎麻、剑麻或椰纤维,或人造纤维素质(例如源自木浆),包括粘胶/人造丝、苎麻、醋酸纤维素纤维(tricell)、莱赛尔(lyocell)或其掺合物。纺织品或织物也可以是非纤维素基的,例如天然聚酰胺,包括羊毛、驼毛、羊绒、马海毛、兔毛和丝,或合成聚合物,例如尼龙、芳纶、聚酯、丙烯腈系纤维、聚丙烯和氨纶纤维(spandex)/弹性纤维,或其掺合物,以及纤维素基纤维和非纤维素基纤维的掺合物。掺合物的例子是棉和/或人造丝/粘胶与一种或多种伴侣材料的掺合物,所述伴侣材料例如羊毛、合成纤维(例如聚酰胺纤维、丙烯酸纤维、聚酯纤维、聚乙烯醇纤维、聚氯乙烯纤维、聚氨酯纤维、聚脲纤维、芳族聚酰胺纤维)以及含纤维素的纤维(例如人造丝/粘胶、苎麻、亚麻、黄麻、醋酸纤维素纤维、莱赛尔纤维)。织物可以是常规可洗的洗衣物,例如沾污的家用洗衣物。当使用术语织物或服装时,其意欲也包括更广义的术语纺织品。术语“稳定性”包括储存稳定性和使用期间的稳定性,例如在洗涤过程期间的稳定性(洗涤稳定性),并反映本发明的蛋白酶变体随时间变化的稳定性,例如当蛋白酶保持在溶液中、特别是在洗涤剂溶液中时保留了多少活性。稳定性受许多因素例如ph、温度、洗涤剂组成例如助洗剂的量、表面活性剂等的影响。蛋白酶稳定性可以使用如本文中的材料和方法部分所述的“稳定性测定”来测量。术语“稳定性改善”或“稳定性增加”在本文中定义为相对于亲本蛋白酶的稳定性,变体蛋白酶显示出在溶液中的稳定性增加。术语“稳定性改善”和“稳定性增加”包括“化学稳定性改善”、“洗涤剂稳定性”或“洗涤剂稳定性改善”。洗涤剂组合物本发明涉及包含细菌甘露聚糖酶的新的洗涤剂组合物。所述包含细菌甘露聚糖酶的洗涤剂组合物可用于希望甘露聚糖降解或改性的洗衣和清洁应用中。本发明还涉及所述洗涤剂组合物在洗衣和清洁应用中的用途以及降解甘露聚糖的方法。用于本文中时,术语“甘露聚糖”是指由通过β-1,4-键连接在一起的甘露糖主链与通过α-1,6-键附着于所述主链上的半乳糖侧链组成的多糖。甘露聚糖包含植物基材料,例如瓜尔胶和刺槐豆胶。葡甘露聚糖是具有或多或少规则交替的β-1,4连接的甘露糖和葡萄糖的主链的多糖,半乳甘露聚糖和半乳葡甘露聚糖是具有α-1,6连接的半乳糖侧链的甘露聚糖和葡甘露聚糖。用于本文中时,术语“甘露聚糖酶”或“半乳甘露聚糖酶”表示根据本领域定义为甘露聚糖内切-1,4-β-甘露糖苷酶并具有替代名称β-甘露聚糖酶和内切-1,4-甘露聚糖酶并催化甘露聚糖、半乳甘露聚糖、葡甘露聚糖和半乳葡甘露聚糖中的1,4-β-d-甘露糖苷键水解的甘露聚糖酶。甘露聚糖酶根据酶命名法分类为ec3.2.1.78。如本文所用的“甘露聚糖酶活性”是指多肽的甘露聚糖降解活性。如本文所用的降解或修饰意指通过甘露聚糖酶从甘露聚糖水解甘露糖单元。本发明多肽的甘露聚糖降解活性可以根据本领域已知的标准试验程序测试。实施例7提供了用于确定甘露聚糖酶活性的标准方法的示例。根据第一方面,本发明的洗涤剂组合物包含至少一种酶,所述酶的氨基酸序列与seqidno:16(man7)的氨基酸序列具有至少74%的序列同一性,和/或与seqidno:12(man6)的氨基酸序列具有93%的序列同一性,和/或与seqidno:20(man14)的氨基酸序列具有79%的序列同一性。在本发明的一个实施方式中,所述至少一种酶的氨基酸序列与seqidno:16的氨基酸序列具有至少75%、至少76%、至少77%、至少78%、至少79%、至少80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%的序列同一性。在本发明的一个实施方式中,所述至少一种酶的氨基酸序列与seqidno:12的氨基酸序列具有至少94%、至少95%、至少96%、至少97%、至少98%或至少99%的序列同一性。在本发明的一个进一步的实施方式中,所述至少一种酶具有甘露聚糖降解活性。包含在本发明的洗涤剂组合物中的所述甘露聚糖酶适用于在各种化学环境中、优选在洗涤剂组合物中降解和改性含甘露聚糖的材料。在本发明的一个实施方式中,所述洗涤剂组合物还包含一种或多种其他酶,所述其他酶选自蛋白酶、脂肪酶、角质酶、淀粉酶、糖酶、纤维素酶、果胶酶、果胶酸裂解酶、甘露聚糖酶、阿拉伯糖酶、半乳聚糖酶、木聚糖酶、氧化酶、黄原胶酶、漆酶和/或过氧化物酶,优选选自蛋白酶、淀粉酶、纤维素酶和脂肪酶。一般而言,所选择的酶的性质应与所选择的洗涤剂相容(即,ph最适、与其他酶性和非酶性成分的相容性等),并且所述酶应以有效量存在。纤维素酶合适的纤维素酶包括细菌或真菌来源的纤维素酶。化学修饰的或蛋白质工程的突变体包括在内。合适的纤维素酶包括来自芽孢杆菌属、假单胞菌属、腐质霉属、镰刀菌属、梭孢壳属、枝顶孢属的纤维素酶,例如,在us4,435,307、us5,648,263、us5,691,178、us5,776,757和wo89/09259中公开的由特异腐质霉、嗜热毁丝霉和尖孢镰刀菌产生的真菌纤维素酶。特别合适的纤维素酶是具有护色益处的碱性或中性纤维素酶。这样的纤维素酶的例子是在ep0495257、ep0531372、wo96/11262、wo96/29397、wo98/08940中描述的纤维素酶。其他例子是纤维素酶变体,例如在wo94/07998、ep0531315、us5,457,046、us5,686,593、us5,763,254、wo95/24471、wo98/12307和pct/dk98/00299中描述的那些。表现出内切-β-1,4-葡聚糖酶活性的纤维素酶(ec3.2.1.4)的例子是在wo02/099091中描述的那些。纤维素酶的其他例子包括wo96/29397中描述的45族纤维素酶,尤其是其在wo02/099091的seqidno:8中对应于以下位置的一个或多个位置处具有取代、插入和/或缺失的变体:第2位、第4位、第7位、第8位、第10位、第13位、第15位、第19位、第20位、第21位、第25位、第26位、第29位、第32位、第33位、第34位、第35位、第37位、第40位、第42位、第42a位、第43位、第44位、第48位、第53位、第54位、第55位、第58位、第59位、第63位、第64位、第65位、第66位、第67位、第70位、第72位、第76位、第79位、第80位、第82位、第84位、第86位、第88位、第90位、第91位、第93位、第95位、第95d位、第95h位、第95j位、第97位、第100位、第101位、第102位、第103位、第113位、第114位、第117位、第119位、第121位、第133位、第136位、第137位、第138位、第139位、第140a位、第141位、第143a位、第145位、第146位、第147位、第150e位、第150j位、第151位、第152位、第153位、第154位、第155位、第156位、第157位、第158位、第159位、第160c位、第160e位、第160k位、第161位、第162位、第164位、第165位、第168位、第170位、第171位、第172位、第173位、第175位、第176位、第178位、第181位、第183位、第184位、第185位、第186位、第188位、第191位、第192位、第195位、第196位、第200位和/或第20位,优选选自p19a、g20k、q44k、n48e、q119h或q146r。可商购的纤维素酶包括cellucleantm、celluzymetm和carezymetm(novozymesa/s),clazinasetm和puradaxhatm(genencorinternationalinc.),以及kac-500(b)tm(kaocorporation)。蛋白酶合适的蛋白酶包括细菌、真菌、植物、病毒或动物来源、例如植物或微生物来源的蛋白酶。微生物来源是优选的。化学修饰的或蛋白质工程的突变体包括在内。它可以是碱性蛋白酶,例如丝氨酸蛋白酶或金属蛋白酶。丝氨酸蛋白酶可以例如是s1家族,例如胰蛋白酶,或s8家族,例如枯草杆菌蛋白酶。金属蛋白酶可以是来自例如家族m4的嗜热菌蛋白酶,或其他金属蛋白酶,例如来自m5、m7或m8家族的那些。术语“枯草杆菌酶”是指根据siezen等人,proteinengng.4(1991)719-737和siezen等人proteinscience6(1997)501-523的丝氨酸蛋白酶亚组。丝氨酸蛋白酶是以在活性位点具有丝氨酸为特征的蛋白酶亚组,其与底物形成共价加合物。枯草属酶可分为6个分支,即枯草杆菌蛋白酶家族、热酶(thermitase)家族、蛋白酶k家族、lantibiotic肽酶家族、kexin家族和pyrolysin家族。枯草杆菌酶的例子是us8962042和wo09/021867中描述的源自于芽孢杆菌例如缓慢芽孢杆菌(bacilluslentus)、微小芽胞杆菌、解淀粉芽胞杆菌、短小芽孢杆菌和吉氏芽孢杆菌(bacillusgibsonii)的那些,以及wo89/06279中描述的缓慢枯草杆菌蛋白酶(subtilisinlentus)、novo枯草杆菌蛋白酶(subtilisinnovo)、carlsberg枯草杆菌蛋白酶(subtilisincarlsberg)、地衣芽孢杆菌(bacilluslicheniformis)、枯草杆菌蛋白酶bpn'、枯草杆菌蛋白酶309、枯草杆菌蛋白酶147和枯草杆菌蛋白酶168,以及wo93/18140中描述的蛋白酶pd138。其他有用的蛋白酶可以是wo92/175177、wo01/016285、wo02/026024和wo02/016547中描述的那些。胰蛋白酶样蛋白酶的例子是胰蛋白酶(例如猪或牛来源的)和在wo89/06270、wo94/25583和wo05/040372中描述的镰刀菌蛋白酶,以及wo05/052161和wo05/052146中描述的源自于纤维菌属(cellumonas)的胰凝乳蛋白酶。进一步优选的蛋白酶是如例如wo95/23221中所述的来自缓慢芽胞杆菌dsm5483的碱性蛋白酶,以及wo92/21760、wo95/23221、ep1921147和ep1921148中描述的其变体。金属蛋白酶的例子是如wo07/044993(genencorint.)中所述的中性金属蛋白酶,例如源自于解淀粉芽孢杆菌的那些。有用的蛋白酶的例子是下列文献中描述变体:wo92/19729、wo96/034946、wo98/20115、wo98/20116、wo99/011768、wo01/44452、wo03/006602、wo04/03186、wo04/041979、wo07/006305、wo11/036263、wo11/036264,尤其是在以下一个或多个使用bpn'编号的位置处具有取代的变体:第3位、第4位、第9位、第15位、第27位、第36位、第57位、第68位、第76位、第87位、第95位、第96位、第97位、第98位、第99位、第100位、第101位、第102位、第103位、第104位、第106位、第118位、第120位、第123位、第128位、第129位、第130位、第160位、第167位、第170位、第194位、第195位、第199位、第205位、第206位、第217位、第218位、第222位、第224位、第232位、第235位、第236位、第245位、第248位、第252位和第274位。更优选的是,所述枯草杆菌酶变体可以包含下列突变:s3t、v4i、s9r、a15t、k27r、*36d、v68a、n76d、n87s,r、*97e、a98s、s99g,d,a、s99ad、s101g,m,rs103a、v104i,y,n、s106a、g118v,r、h120d,n、n123s、s128l、p129q、s130a、g160d、y167a、r170s、a194p、g195e、v199m、v205i、l217d、n218d、m222s、a232v、k235l、q236h、q245r、n252k、t274a(使用bpn'编号)。合适的可商购蛋白酶包括:以商品名duralasetm、durazymtm、ultra、ultra、ultra、ultra、和(novozymesa/s)出售的那些,以商品名purafectprefer-enztm、purafectpurafectpurafecteffectenztm、和(danisco/dupont)、axapemtm(gist-brocasesn.v.)出售的那些,blap(序列示于us5352604的图29中)及其变体(henkelag)和来自kao的kap(嗜碱芽孢杆菌(bacillusalkalophilus)枯草杆菌蛋白酶)。脂肪酶和角质酶合适的脂肪酶和角质酶包括细菌或真菌来源的那些。化学修饰的或蛋白质工程的突变酶包括在内。实例包括来自嗜热丝孢菌(thermomyces)的脂肪酶,例如如ep258068和ep305216中所述的疏棉状嗜热丝孢菌(t.lanuginosus)(以前称为柔毛腐质霉(humicolalanuginosa)),来自腐质霉、例如特异腐质霉(h.insolens)的角质酶(wo96/13580),来自假单胞菌(pseudomonas)菌株(其中一些现在更名为伯克氏菌(burkholderia))、例如产碱伯克氏菌(p.alcaligenes)或类产碱伯克氏菌(p.pseudoalcaligenes)(ep218272)、洋葱伯克氏菌(p.cepacia)(ep331376),伯克氏菌菌种(p.sp.)菌株sd705(wo95/06720&wo96/27002)、p.wisconsinensis(wo96/12012)的脂肪酶,gdsl-型gdsl链霉菌(streptomyces)脂肪酶(wo10/065455),来自稻梨孢菌(magnaporthegrisea)的角质酶(wo10/107560),来自门多萨假单胞菌(pseudomonasmendocina)的角质酶(us5,389,536),来自褐色喜热裂孢菌(thermobifidafusca)的脂肪酶(wo11/084412),嗜热脂肪土芽孢杆菌(geobacillusstearothermophilus)脂肪酶(wo11/084417),来自枯草芽孢杆菌的脂肪酶(wo11/084599),以及来自灰色链霉菌(streptomycesgriseus)(wo11/150157)和始旋链霉菌(s.pristinaespiralis)(wo12/137147)的脂肪酶。其他例子是脂肪酶变体,例如在ep407225、wo92/05249、wo94/01541、wo94/25578、wo95/14783、wo95/30744、wo95/35381、wo95/22615、wo96/00292、wo97/04079、wo97/07202、wo00/34450、wo00/60063、wo01/92502、wo07/87508和wo09/109500中描述的那些。优选的商业脂肪酶产品包括lipolasetm,lipextm;lipolextm和lipocleantm(novozymesas),lumafast(来源于genencor)和lipomax(来源于gist-brocades)。还有其他例子是脂肪酶,有时称为酰基转移酶或过水解酶,例如与南极假丝酵母(candidaantarctica)脂肪酶a具有同源性的酰基转移酶(wo10/111143),来自耻垢分枝杆菌(mycobacteriumsmegmatis)的酰基转移酶(wo05/56782),来自ce7家族的过水解酶(wo09/67279),以及耻垢分枝杆菌过水解酶的变体,特别是来自huntsmantextileeffectspteltd的商品gentlepowerbleach中使用的s54v变体(wo10/100028)。淀粉酶可以与本发明的枯草杆菌酶变体一起使用的合适的淀粉酶可以是α-淀粉酶或葡糖淀粉酶,并且可以是细菌或真菌来源的。化学修饰的或蛋白质工程的突变体包括在内。淀粉酶包括,例如,从芽孢杆菌例如在gb1,296,839中有更详细描述的地衣芽孢杆菌特殊菌株获得的α-淀粉酶。合适的淀粉酶包括wo95/10603中具有seqidno:3的淀粉酶或与seqidno:3具有90%的序列同一性的其变体。优选的变体描述于wo94/02597、wo94/18314、wo97/43424以及wo99/019467的seqidno:4中,例如在一个或多个以下位置具有取代的变体:第15位、第23位、第105位、第106位、第124位、第128位、第133位、第154位、第156位、第178位、第179位、第181位、第188位、第190位、第197位、第201位、第202位、第207位、第208位、第209位、第211位、第243位、第264位、第304位、第305位、第391位、第408位和第444位。不同的合适淀粉酶包括wo02/010355中的具有seqidno:6的淀粉酶或与该淀粉酶具有90%的序列同一性的该淀粉酶的变体。优选的变体是在181和182位具有缺失并且在193位具有取代的那些。其他合适的淀粉酶是包含wo2006/066594的seqidno:6中显示的源自于解淀粉芽孢杆菌的α-淀粉酶的残基1-33和wo2006/066594的seqidno:4中所示的地衣芽孢杆菌α-淀粉酶的残基36-483的杂合α-淀粉酶,或具有90%的序列同一性的其变体。该杂合α-淀粉酶的优选变体是在一个或多个以下位置具有取代、缺失或插入的变体:g48、t49、g107、h156、a181、n190、m197、1201、a209和q264。包含wo2006/066594的seqidno:6中显示的源自于解淀粉芽孢杆菌的α-淀粉酶的残基1-33和wo2006/066594的seqidno:4的残基36-483的杂合α-淀粉酶的最优选变体是具有下列取代的那些:m197t;h156y+a181t+n190f+a209v+q264s;或g48a+t49i+g107a+h156y+a181t+n190f+i201f+a209v+q264s。其他合适的淀粉酶是wo99/019467中具有seqidno:6的淀粉酶或与seqidno:6具有90%的序列同一性的其变体。seqidno:6的优选变体是在一个或多个以下位置具有取代、缺失或插入的变体:r181、g182、h183、g184、n195、i206、e212、e216和k269。特别优选的淀粉酶是在r181和g182位或h183和g184位具有缺失的淀粉酶。可以使用的其他淀粉酶是具有wo96/023873的seqidno:1、seqidno:3、seqidno:2或seqidno:7的那些或与seqidno:1、seqidno:2、seqidno:3或seqidno:7具有90%的序列同一性的其变体。seqidno:1、seqidno:2、seqidno:3或seqidno:7的优选变体是在一个或多个以下位置具有取代、缺失或插入的变体:第140位、第181位、第182位、第183位、第184位、第195位、第206位、第212位、第243位、第260位、第269位、第304位或第476位。更优选的变体是在第181位和第182位或者第183位和第184位具有缺失的那些。seqidno:1、seqidno:2或seqidno:7的最优选淀粉酶变体是在第183位和第184位具有缺失并且在第140位、第195位、第206位、第243位、第260位、第304位和第476位中的一个或多个具有取代的那些。其他可以使用的淀粉酶是具有wo08/153815的seqidno:2、wo01/66712的seqidno:10的淀粉酶或与wo08/153815的seqidno:2具有90%的序列同一性或与wo01/66712中的seqidno:10具有90%的序列同一性的其变体。wo01/66712中seqidno:10的优选变体是在一个或多个以下位置具有取代、缺失或插入的变体:第176位、第177位、第178位、第179位、第190位、第201位、第207位、第211位和第264位。其他合适的淀粉酶是具有wo09/061380的seqidno:2的淀粉酶或与seqidno:2具有90%的序列同一性的其变体。seqidno:2的优选变体是具有c-末端截短和/或在一个或多个以下位置具有取代、缺失或插入的那些:q87、q98、s125、n128、t131、t165、k178、r180、s181、t182、g183、m201、f202、n225、s243、n272、n282、y305、r309、d319、q320、q359、k444和g475。seqidno:2的更优选变体是在一个或多个以下位置:q87e,r、q98r、s125a、n128c、t131i、t165i、k178l、t182g、m201l、f202y、n225e,r、n272e,r、s243q,a,e,d、y305r、r309a、q320r、q359e、k444e和g475k具有取代和/或在r180和/或s181或t182和/或g183位具有缺失的那些。seqidno:2的最优选的淀粉酶变体是具有以下取代的那些:n128c+k178l+t182g+y305r+g475k;n128c+k178l+t182g+f202y+y305r+d319t+g475k;s125a+n128c+k178l+t182g+y305r+g475k;或s125a+n128c+t131i+t165i+k178l+t182g+y305r+g475k,其中所述变体是c末端截短的并且任选还包含243位的取代和/或180位和/或181位的缺失。其他合适的淀粉酶是wo01/66712中具有seqidno:12的α-淀粉酶或与seqidno:12具有至少90%序列同一性的变体。优选的淀粉酶变体是在wo01/66712中seqidno:12的一个或多个以下位置具有取代、缺失或插入的变体:r28、r118、n174;r181、g182、d183、g184、g186、w189、n195、m202、y298、n299、k302、s303、n306、r310、n314、r320、h324、e345、y396、r400、w439、r444、n445、k446、q449、r458、n471、n484。特别优选的淀粉酶包括具有d183和g184的缺失并具有取代r118k、n195f、r320k和r458k的变体,以及在选自下列的一个或多个位置另外具有取代的变体:m9、g149、g182、g186、m202、t257、y295、n299、m323、e345和a339,最优选的是在所有这些位置另外具有取代的变体。其他例子是淀粉酶变体,例如在wo2011/098531、wo2013/001078和wo2013/001087中描述的那些。可商购的淀粉酶是duramyltm、termamyltm、fungamyltm、stainzymetm、stainzymeplustm、natalasetm、liquozymex和bantm(来自novozymesa/s),以及rapidasetm、purastartm/effectenztm、powerase和preferenzs100(来自genencorinternationalinc./dupont)。过氧化物酶/氧化酶合适的过氧化物酶/氧化酶包括植物、细菌或真菌来源的那些。化学修饰的或蛋白质工程的突变体包括在内。有用的过氧化物酶的例子包括来自鬼伞属(coprinus)、例如来自灰盖鬼伞(c.cinereus)的过氧化物酶,及它们的变体,如wo93/24618、wo95/10602和wo98/15257中描述的那些。可商购的过氧化物酶包括guardzymetm(novozymesa/s)。所述洗涤剂酶可以通过添加分别含有一种或多种酶的添加剂或通过添加包含所有这些酶的组合添加剂而包含在洗涤剂组合物中。本发明的洗涤剂添加剂,即分别的添加剂或组合的添加剂,可以配制为例如颗粒、液体、浆液等。优选的洗涤剂添加剂制剂是颗粒、特别是无尘颗粒,液体、特别是稳定的液体,或浆液。无尘颗粒可以,例如,如us4,106,991和4,661,452中所公开的那样生产,并且可以任选通过本领域已知的方法进行涂覆。蜡质涂料的例子是平均摩尔量为1000至20000的聚(环氧乙烷)产品(聚乙二醇,peg);具有16至50个环氧乙烷单元的乙氧基化壬基酚;其中醇含有12至20个碳原子并且其中有15至80个环氧乙烷单元的乙氧基化脂肪醇;脂肪醇;脂肪酸;以及脂肪酸的甘油一酯和二酯和三酯。适用于流化床技术应用的成膜涂料的例子在gb1483591中给出。例如,可以通过根据已建立的方法添加多元醇例如丙二醇、糖或糖醇、乳酸或硼酸来稳定液体酶制剂。受保护的酶可以根据ep238,216中公开的方法制备。用于固体衣物洗涤剂的组合物,例如,按组合物的总量计,可包含0.000001%-5%、例如0.000005%-2%、例如0.00001%-1%、例如0.00001%-0.1%的酶蛋白。用于洗衣液的组合物,例如,按组合物的总量计,可包含0.000001%-3%、例如0.000005-1%、例如0.00001%-0.01%的酶蛋白。用于自动洗碗机的组合物,例如,按组合物的总量计,可包含0.000001%-5%、例如0.000005%-2%、例如0.00001%-1%、例如0.00001%-0.1%的酶蛋白。本发明的洗涤剂组合物的酶可以使用常规稳定剂来稳定,所述常规稳定剂是例如多元醇例如丙二醇或甘油、糖或糖醇、乳酸、硼酸、或硼酸衍生物例如芳族硼酸酯、或苯基硼酸衍生物例如4-甲酰基苯基硼酸,并且所述组合物可以如例如wo92/19709和wo92/19708中所述进行配制。在一个实施方式中,本发明涉及洗涤剂组合物,其包含本发明的酶与一种或多种另外的清洁组合物组分的组合。另外的组分的选择在技术人员的技能范围内,并且包括常规成分,包括下面列出的示例性的非限制性组分。对于纺织品护理,组分的选择可包括考虑待清洁的纺织品类型、脏污的类型和/或程度、进行清洁的温度,以及洗涤剂产品的配方。虽然下面提到的组分根据特定功能通过普通标题进行分类,但是这不应被解释为限制,因为组分可以包含本领域技术人员将领会的另外的功能。1.表面活性剂所述洗涤剂组合物可包含一种或多种表面活性剂,其可以是阴离子和/或阳离子和/或非离子和/或半极性和/或两性离子的,或其混合物。在一个特定实施方式中,所述洗涤剂组合物包含一种或多种非离子表面活性剂和一种或多种阴离子表面活性剂的混合物。所述表面活性剂通常以约0.1至60重量%的水平存在,例如以约1%至约40%、或约3%至约20%、或约3%至约10%。所述表面活性剂基于所需的清洁应用进行选择,并包括本领域已知的任何常规表面活性剂。可以利用本领域已知的用于洗涤剂的任何表面活性剂。当其中包含时,所述洗涤剂将通常含有约1至约40重量%、例如约5%至约30%、包括约5%至约15%、或约20%至约25%的阴离子表面活性剂。阴离子表面活性剂的非限制性例子包括硫酸盐和磺酸盐,特别是直链烷基苯磺酸盐(las)、las的异构体、支链烷基苯磺酸盐(babs)、苯基链烷磺酸盐、α-烯烃磺酸盐(aos)、烯烃磺酸盐、链烯磺酸盐、链烷-2,3-二基双(硫酸盐)、羟基链烷磺酸盐和二磺酸盐、烷基硫酸盐(as)例如十二烷基硫酸钠(sds)、脂肪醇硫酸盐(fas)、伯醇硫酸盐(pas)、醇醚硫酸盐(aes或aeos或fes,也称为醇乙氧基硫酸盐或脂肪醇醚硫酸盐)、仲链烷磺酸盐(sas)、石蜡磺酸盐(ps)、酯磺酸盐、磺化脂肪酸甘油酯、α-磺基脂肪酸甲酯(α-sfme或ses)包括甲酯磺酸盐(mes)、烷基-或烯基琥珀酸、十二烯基/十四烯基琥珀酸(dtsa)、氨基酸的脂肪酸衍生物、磺基琥珀酸或皂的二酯和单酯,及其组合。当其中包含时,所述洗涤剂将通常含有约0至约10重量%的阳离子表面活性剂。阳离子表面活性剂的非限制性例子包括烷基二甲基乙醇胺季铵盐(admeaq)、鲸蜡基三甲基溴化铵(ctab)、二甲基二硬脂基氯化铵(dsdmac)和烷基苄基二甲基铵、烷基季铵化合物、烷氧基化季铵(aqa)化合物及其组合。当其中包含时,所述洗涤剂将通常含有约0.2至约40重量%的非离子表面活性剂,例如约0.5%至约30%,特别是约1%至约20%,约3%至约10%,例如约3%至约5%,或约8%至约12%。非离子表面活性剂的非限制性例子包括醇乙氧基化物(ae或aeo)、醇丙氧基化物、丙氧基化脂肪醇(pfa)、烷氧基化脂肪酸烷基酯例如乙氧基化和/或丙氧基化脂肪酸烷基酯、烷基酚乙氧基化物(ape)、壬基酚乙氧基化物(npe)、烷基多糖苷(apg)、烷氧基化胺、脂肪酸单乙醇酰胺(fam)、脂肪酸二乙醇酰胺(fada)、乙氧基化脂肪酸单乙醇酰胺(efam)、丙氧基化脂肪酸单乙醇酰胺(pfam)、多羟基烷基脂肪酸酰胺、或葡糖胺的n-酰基n-烷基衍生物(葡糖酰胺,ga,或脂肪酸葡糖酰胺,faga),以及商品名span和tween的现有产品,及其组合。当其中包含时,所述洗涤剂将通常含有约0至约10重量%的半极性表面活性剂。半极性表面活性剂的非限制性例子包括氧化胺(ao)例如烷基二甲基氧化胺、n-(椰油烷基)-n,n-二甲基氧化胺和n-(牛油烷基)-n、n-双(2-羟乙基)氧化胺、脂肪酸烷醇酰胺和乙氧基化脂肪酸链烷醇酰胺,及其组合。当其中包含时,所述洗涤剂将通常含有约0至约10重量%的两性离子表面活性剂。两性离子表面活性剂的非限制性例子包括甜菜碱、烷基二甲基甜菜碱、磺基甜菜碱及其组合。2.水溶助剂水溶助剂是能将疏水性化合物溶解在水溶液中(或相反地,将极性物质溶解在非极性环境中)的化合物。通常,水溶助剂兼具亲水和疏水特性(从表面活性剂中已知的所谓两亲性质);然而,水溶助剂的分子结构通常不利于自发的自聚集。水溶助剂不显示临界浓度,即高于该浓度发生自聚集,如发现表面活性剂和脂质形成胶束、片层或其他界限分明的中间相。相反,许多水溶助剂显示出连续型聚集过程,其中聚集体的尺寸随着浓度增加而增长。然而,许多水溶助剂改变含有极性和非极性特性物质的体系、包括水、油、表面活性剂和聚合物的混合物的相行为、稳定性和胶体性质。传统上在跨越从制药、个人护理、食品到技术应用的行业中都使用水溶助剂。在洗涤剂组合物中使用水溶助剂允许例如更浓缩的表面活性剂制剂(如在通过除去水使液体洗涤剂紧缩的过程中)而不会引起不希望的现象,例如相分离或高粘度。所述洗涤剂可含有0-5重量%、例如约0.5至约5%、或约3%至约5%的水溶助剂。可以利用本领域已知的用于洗涤剂的任何水溶助剂。水溶助剂的非限制性例子包括苯磺酸钠、对甲苯磺酸钠(sts)、二甲苯磺酸钠(sxs)、异丙苯磺酸钠(scs)、甲基异丙苯磺酸钠、氧化胺、醇和聚二醇醚、羟基萘甲酸钠、羟基萘磺酸钠、乙基己基硫酸钠及其组合。3.助洗剂和共助洗剂所述洗涤剂组合物可含有约0-65重量%、例如约5%至约45%的洗涤剂助洗剂或共助洗剂,或其混合物。在餐具洗涤剂中,助洗剂的水平通常为40-65%,特别是50-65%。助洗剂和/或共助洗剂特别可以是与ca和mg形成水溶性络合物的螯合剂。可以利用本领域已知用于洗衣洗涤剂的任何助洗剂和/或共助洗剂。助洗剂的非限制性例子包括沸石、二磷酸盐(焦磷酸盐)、三磷酸盐例如三磷酸钠(stp或stpp)、碳酸盐例如碳酸钠、可溶性硅酸盐例如偏硅酸钠、层状硅酸盐(例如来自hoechst的sks-6)、乙醇胺例如2-氨基乙-1-醇(mea)、二乙醇胺(dea、也称为亚氨基二乙醇)、三乙醇胺(tea,也称为2,2',2"-硝基三乙醇)和羧甲基菊粉(cmi),及其组合。所述洗涤剂组合物也可以含有约0-20重量%、例如约5%至约10%的洗涤剂共助洗剂,或其混合物。洗涤剂组合物可以只包含共助洗剂,或与助洗剂、例如沸石助洗剂组合。共助洗剂的非限制性例子包括聚丙烯酸酯的均聚物或其共聚物,例如聚(丙烯酸)(paa)或共聚(丙烯酸/马来酸)(paa/pma)。其他非限制性例子包括柠檬酸盐、螯合剂如氨基羧酸盐、氨基多羧酸盐和膦酸盐,以及烷基或烯基琥珀酸。另外的具体例子包括2,2',2"-次氮基三乙酸(nta)、乙二胺四乙酸(edta)、二亚乙基三胺五乙酸(dtpa)、亚氨基二琥珀酸(ids)、乙二胺-n,n-二琥珀酸(edds)、甲基甘氨酸二乙酸(mgda)、谷氨酸-n,n-二乙酸(glda)、1-羟基乙烷-1,1-二膦酸(hedp)、乙二胺四(亚甲基膦酸)(edtmpa)、二亚乙基三胺五(亚甲基膦酸)(dtpmpa或dtmpa)、n-(2-羟乙基)亚氨基二乙酸(edg)、天冬氨酸-n-单乙酸(asma)、天冬氨酸-n,n-二乙酸(asda)、天冬氨酸-n-单丙酸(asmp)、亚氨基二琥珀酸(ida)、n-(2-磺基甲基)-天冬氨酸(smas)、n-(2-磺基乙基)-天冬氨酸(seas)、n-(2-磺基甲基)-谷氨酸(smgl)、n-(2-磺基乙基)-谷氨酸(segl)、n-甲基亚氨基二乙酸(mida)、α-丙氨酸-n,n-二乙酸(α-alda)、丝氨酸-n,n-二乙酸(seda)、异丝氨酸-n,n-二乙酸(isda)、苯丙氨酸-n,n-二乙酸(phda)、邻氨基苯甲酸-n,n-二乙酸(anda)、磺胺酸-n,n-二乙酸(slda)、牛磺酸-n,n-二乙酸(tuda)和磺基甲基-n,n-二乙酸(smda)、n-(2-羟乙基)-乙二胺-n,n',n-三乙酸酯(hedta)、二乙醇甘氨酸(deg)、二亚乙基三胺五(亚甲基膦酸)(dtpmp)、氨基三(亚甲基膦酸)(atmp)及其组合和盐。其他示例性助洗剂和/或共助洗剂描述于例如wo09/102854、us5977053中。4.漂白体系洗涤剂可含有0-50重量%、例如约0.1%至约25%的漂白体系。可以利用本领域已知用于洗衣洗涤剂的任何漂白体系。合适的漂白体系组分包括漂白催化剂、光漂白剂、漂白活化剂、过氧化氢源例如过碳酸钠和过硼酸钠、预制的过酸及其混合物。合适的预制过酸包括但不限于过氧羧酸和盐、过碳酸和盐、过亚氨酸和盐、过氧单硫酸和盐例如oxone(r),及其混合物。漂白体系的非限制性例子包括基于过氧化物的漂白体系,其可包含例如无机盐,包括碱金属盐,例如过硼酸(通常为单或四水合物)、过碳酸、过硫酸、过磷酸、过硅酸的钠盐,与形成过酸的漂白活化剂的组合。术语漂白活化剂在本文中是指与过氧漂白剂如过氧化氢反应形成过酸的化合物。由此形成的过酸构成活化的漂白剂。在此使用的合适的漂白活化剂包括属于酯、酰胺、酰亚胺或酸酐类别的那些。合适的例子是四乙酰基乙二胺(taed)、4-[(3,5,5-三甲基己酰基)氧基]苯磺酸钠(isonobs)、二过氧十二烷酸、4-(十二烷酰氧基)苯磺酸盐(lobs)、4-(癸酰氧基)苯磺酸盐、4-(癸酰氧基)苯甲酸酯(dobs)、4-(壬酰氧基)-苯磺酸盐(nobs)和/或wo98/17767中公开的那些。在ep624154中公开了一种特定的目标漂白活化剂,特别优选该家族是乙酰基柠檬酸三乙酯(atc)。atc或短链甘油三酯如三醋精具有环境友好的优点,因为它最终降解成柠檬酸和醇。此外,乙酰基柠檬酸三乙酯和三醋精在储存时在产品中具有良好的水解稳定性,并且它是有效的漂白活化剂。最后,atc为洗衣添加剂提供了良好的助洗能力。或者,所述漂白体系可包含例如酰胺、酰亚胺或砜类型的过氧酸。所述漂白体系也可以包含过酸,例如6-(邻苯二甲酰亚氨基)过氧己酸(pap)。所述漂白体系也可包含漂白催化剂。在一些实施方式中,所述漂白组分可以是选自具有下式的有机催化剂及其混合物的有机催化剂:(iii);其中每个r1独立地是含有9至24个碳的支链烷基基团或含有11至24个碳的直链烷基基团,优选每个r1独立地是含有9至18个碳的支链烷基基团或含有11至18个碳的直链烷基基团,更优选每个r1独立地选自2-丙基庚基、2-丁基辛基、2-戊基壬基、2-己基癸基、正十二烷基、正十四烷基、正十六烷基、正十八烷基、异壬基、异癸基、异十三烷基和异十五烷基。其他示例性漂白体系在例如wo2007/087258、wo2007/087244、wo2007/087259和wo2007/087242中描述。合适的光漂白剂可以是例如磺化的锌酞菁。5.聚合物所述洗涤剂可含有0-10重量%、例如0.5-5%、2-5%、0.5-2%或0.2-1%的聚合物。可以利用本领域已知的用于洗涤剂的任何聚合物。聚合物可以如同上文所述的共助洗剂那样起作用,或者可以提供抗再沉积、纤维保护、污垢释放、染料转移抑制、油脂清洁和/或消泡性质。一些聚合物可多于一种上述性质和/或多于一种下述基序。示例性聚合物包括(羧甲基)纤维素(cmc)、聚乙烯醇(pva)、聚(乙烯基吡咯烷酮)(pvp)、聚(乙二醇)或聚(环氧乙烷)(peg)、乙氧基化聚(乙烯亚胺)、羧甲基菊粉(cmi)和聚羧酸盐如paa、paa/pma、聚天冬氨酸,以及甲基丙烯酸月桂酯/丙烯酸共聚物、疏水改性的cmc(hm-cmc)和有机硅、对苯二甲酸与低聚二醇的共聚物、聚(对苯二甲酸乙二醇酯)与聚(对苯二甲酸乙氧酯)的共聚物(pet-poet)、pvp、聚(乙烯基咪唑)(pvi)、聚(乙烯基吡啶-n-氧化物)(pvpo或pvpno)和聚乙烯吡咯烷酮-乙烯基咪唑(pvpvi)。其他示例性聚合物包括磺化多羧酸酯、聚环氧乙烷和聚环氧丙烷(peo-ppo)和二季铵乙氧基硫酸酯。其他示例性聚合物公开在例如wo2006/130575中。还考虑了上述聚合物的盐。6.织物调色剂本发明的洗涤剂组合物也可以包含织物调色剂,例如染料或颜料,其当配制在洗涤剂组合物中时,当织物与包含所述洗涤剂组合物的洗涤液相接触时可沉积在所述织物上并由此通过吸收/反射可见光而改变所述织物的色调。荧光增白剂发射至少一些可见光。相反,织物调色剂在它们吸收至少一部分可见光谱时改变表面的色调。合适的织物调色剂包含染料和染料-粘土结合物,并且也可包含颜料。合适的染料包括小分子染料和聚合物染料。合适的小分子染料包括选自落入直接蓝、直接红、直接紫、酸性蓝、酸性红、酸性紫、碱性蓝、碱性紫和碱性红的颜色指数(c.i.)分类内的染料或其混合物的小分子染料,例如,如wo2005/03274、wo2005/03275、wo2005/03276和ep1876226(在此通过引用并入)中所述。所述洗涤剂组合物优选包含约0.00003wt%至约0.2wt%、约0.00008wt%至约0.05wt%、或甚至约0.0001wt%至约0.04wt%的织物调色剂。所述组合物可包含0.0001wt%至0.2wt%的织物调色剂,当所述组合物是单位剂量小袋的形式时,这可能是特别优选的。合适的调色剂也公开于例如wo2007/087257和wo2007/087243中。7.辅助材料也可以利用本领域已知的用于洗衣洗涤剂的任何洗涤剂组分。其他任选的洗涤剂组分包括抗腐蚀剂、抗收缩剂、抗污垢再沉积剂、抗皱剂、杀菌剂、粘合剂、腐蚀抑制剂、崩解剂、染料、酶稳定剂(包括硼酸、硼酸盐、cmc、和/或多元醇例如丙二醇)、织物调理剂包括粘土、填料/加工助剂、荧光增白剂/光学增亮剂、泡沫促进剂、泡沫(泡沫)调节剂、香料、污垢悬浮剂、软化剂、抑泡剂、褐变抑制剂和芯吸剂,单独地或组合地。可以利用本领域已知用于洗衣洗涤剂的任何成分。这样的成分的选择完全在技术人员的技能范围内。分散剂:本发明的洗涤剂组合物也可含有分散剂。特别是粉末状洗涤剂可包含分散剂。合适的水溶性有机材料包括均聚或共聚酸或它们的盐,其中多羧酸包含至少两个被不多于两个碳原子彼此分开的羧基基团。合适的分散剂例如描述于marceldekker,inc.的粉末状洗涤剂(powdereddetergents),表面活性剂科学系列第71卷(surfactantscienceseriesvolume71)中。染料转移抑制剂:本发明的洗涤剂组合物也可包含一种或多种染料转移抑制剂。合适的聚合染料转移抑制剂包括但不限于聚乙烯吡咯烷酮聚合物、多胺n-氧化物聚合物、n-乙烯基吡咯烷酮与n-乙烯基咪唑的共聚物、聚乙烯基唑烷酮和聚乙烯基咪唑,或其混合物。当存在于主题组合物中时,所述染料转移抑制剂的存在水平可以为组合物重量的约0.0001%至约10%、约0.01%至约5%或甚至约0.1%至约3%。荧光增白剂:本发明的洗涤剂组合物将优选还含有可以着色被清洁的物品的其他组分,例如荧光增白剂或光学增亮剂。当存在时,所述增亮剂优选为约0.01%至约0.5%的水平。适用于衣物洗涤剂组合物的任何荧光增白剂均可用于本发明的组合物中。最常用的荧光增白剂是属于二氨基芪-磺酸衍生物、二芳基吡唑啉衍生物和双苯基-二苯乙烯基衍生物的那些。二氨基芪-磺酸衍生物类型的荧光增白剂的例子包括以下的钠盐:4,4'-双-(2-二乙醇氨基-4-苯胺基-s-三嗪-6-基氨基)芪-2,2'-二磺酸酯;4,4'-双-(2,4-二苯胺基-s-三嗪-6-基氨基)芪-2,2'-二磺酸酯;4,4'-双-(2-苯胺基-4-(n-甲基-n-2-羟基-乙氨基)-s-三嗪-6-基氨基)芪-2,2'-二磺酸酯,4,4'-双-(4-苯基-2,1,3-三唑-2-基)芪-2,2'-二磺酸酯;4,4'-双-(2-苯胺基-4-(1-甲基-2-羟基-乙氨基)-s-三嗪-6-基氨基)芪-2,2'-二磺酸酯和2-(芪基-4"-萘基-1.,2':4,5)-1,2,3-三唑-2"-磺酸酯。优选的荧光增白剂是tinopaldms和tinopalcbs,可得自瑞士basel的ciba-geigyag。tinopaldms是4,4'-双-(2-吗啉代-4-苯胺基-s-三嗪-6-基氨基)芪二磺酸酯的二钠盐。tinopalcbs是2,2'-双-(苯基-苯乙烯基)二磺酸酯的二钠盐。还优选荧光增白剂是可商购的parawhitekx,由印度mumbai的paramountmineralsandchemicals提供。适用于本发明的其他荧光剂包括1-3-二芳基吡唑啉和7-烷基氨基香豆素。合适的荧光增白剂水平包括从约0.01、0.05、约0.1或甚至约0.2wt%的较低水平至0.5或甚至0.75wt%的较高水平。污垢释放聚合物:本发明的洗涤剂组合物也可包含一种或多种污垢释放聚合物,其有助于从织物例如棉和聚酯基织物上除去污垢,特别是从聚酯基织物上除去疏水污垢。所述污垢释放聚合物可以是例如非离子或阴离子型对苯二甲酸酯基聚合物、聚乙烯基己内酰胺和相关共聚物、乙烯基接枝共聚物、聚酯聚酰胺,参见例如marceldekker,inc.的粉末状洗涤剂(powdereddetergents),表面活性剂科学系列71卷(surfactantscienceseries71)中的第7章。另一种类型的污垢释放聚合物是两亲性烷氧基化油脂清洁聚合物,其包含核心结构和连接于该核心结构的多个烷氧基化物基团。所述核心结构可包含聚亚烷基亚胺结构或聚链烷醇胺结构,如wo2009/087523中详细描述的(在此通过引用并入)。此外,无规接枝共聚物是合适的污垢释放聚合物。合适的接枝共聚物在wo2007/138054、wo2006/108856和wo2006/113314(在此通过引用并入)中有更详细的描述。其他污垢释放聚合物是取代的多糖结构,尤其是取代的纤维素结构,例如改性纤维素衍生物,例如ep1867808或wo2003/040279中描述的那些(两者均在此通过引用并入)。合适的纤维素性聚合物包括纤维素、纤维素醚、纤维素酯、纤维素酰胺及其混合物。合适的纤维素聚合物包括阴离子改性纤维素、非离子改性纤维素、阳离子改性纤维素、两性离子改性纤维素,及其混合物。合适的纤维素性聚合物包括甲基纤维素、羧甲基纤维素、乙基纤维素、羟乙基纤维素、羟丙基甲基纤维素、酯羧甲基纤维素,及其混合物。抗再沉积剂:本发明的洗涤剂组合物也可包含一种或多种抗再沉积剂,例如羧甲基纤维素(cmc)、聚乙烯醇(pva)、聚乙烯吡咯烷酮(pvp)、聚氧乙烯和/或聚乙二醇(peg)、丙烯酸的均聚物、丙烯酸与马来酸的共聚物以及乙氧基化聚乙烯亚胺。上面在污垢释放聚合物下描述的纤维素基聚合物也可以起抗再沉积剂的作用。其他合适的辅助材料包括但不限于,用于液体洗涤剂和/或结构弹性化剂的抗收缩剂、抗皱剂、杀菌剂、粘合剂、载剂、染料、酶稳定剂、织物柔软剂、填料、泡沫调节剂、水溶助剂、香料、颜料、抑泡剂、溶剂和结构剂。在本发明的一个进一步的实施方式中,所述洗涤剂组合物为棒剂、均质片剂、具有两个或更多个层的片剂、具有一个或多个隔室的小袋剂、常规或致密粉末剂、颗粒剂、糊剂、凝胶剂或者常规、致密或浓缩液体剂。在一个实施方式中,所述洗涤剂组合物可以是衣物洗涤剂组合物,优选为液体或固体衣物洗涤剂组合物。存在许多洗涤剂制剂形式例如层(相同或不同的相)、小袋,以及用于机器配料单元的形式。小袋可以构造为单隔室或多隔室。它可以是适合于保持所述组合物、例如在接触水之前不允许从小袋中释放组合物的的任何形式、形状和材料。所述小袋由包围内部容积的水溶性薄膜制成。所述内部容积可以划分成小袋的隔室。优选的膜是聚合材料,优选形成为膜或片的聚合物。优选的其聚合物、共聚物或衍生物是所选择的聚丙烯酸酯,以及水溶性丙烯酸酯共聚物、甲基纤维素、羧甲基纤维素、糊精钠、乙基纤维素、羟乙基纤维素、羟丙基甲基纤维素、麦芽糊精、聚甲基丙烯酸酯,最优选聚乙烯醇共聚物和羟丙基甲基纤维素(hpmc)。优选地,所述膜例如pva中的聚合物水平为至少约60%。优选的平均分子量将通常为约20,000至约150,000。膜也可以是包含可水解降解和水溶性聚合物掺合物例如聚丙交酯和聚乙烯醇(已知商品名为m8630,由美国美印第安纳州gary的chriscraftin.prod.出售)加上增塑剂如甘油、乙烯甘油、丙二醇、山梨糖醇及其混合物的掺合组合物。所述小袋可包含由水溶性薄膜分开的固体衣物洗涤剂组合物或部分组分和/或液体清洁组合物或部分组分。液体组分的隔室的组成可以与含有固体的隔室不同。参考:(us2009/0011970a1)。洗涤剂成分可以通过水溶性小袋中的隔室或片剂中的不同层彼此物理分离。从而可以避免组分之间的负储存相互作用。各隔室的不同溶解规律也可以产生选定的组分在洗涤溶液中的延迟溶解。不是按单位配量的液体或凝胶洗涤剂可以是含水的,通常含有至少20重量%和至多95%的水,例如至多约70%的水,至多约65%的水,至多约50%的水,至多约45%的水,至多约35%的水。其他类型的液体,包括但不限于,烷醇、胺、二醇、醚和多元醇,可以包含在含水液体或凝胶中。含水液体或凝胶洗涤剂可含有0-30%的有机溶剂。液体或凝胶洗涤剂可以是不含水的。本发明的洗涤剂组合物可以提供成洗衣皂条的形式,并用于手洗衣物、织物和/或纺织品。术语洗衣皂条包括洗衣条、肥皂条、组合条(combobars)、合成洗涤剂条和洗涤剂条。条的类型通常在它们含有的表面活性剂的类型方面有所不同,并且术语洗衣皂条包括含有来自脂肪酸的皂和/或合成皂的那些皂条。洗衣皂条具有在室温下是固体而不是液体、凝胶或粉末的物理形式。术语“固体”定义为不随时间显著变化的物理形式,即,如果将固体物体(例如洗衣皂条)放置在容器内,则固体物体不会变化以填充其所处的容器。所述条是通常呈条形式的固体,但也可以是其他固体形状,例如圆形或椭圆形。所述洗衣皂条可含有一种或多种其他酶、蛋白酶抑制剂例如肽醛(或亚硫酸氢盐加合物或半缩醛加合物)、硼酸、硼酸盐、硼砂和/或苯基硼酸衍生物例如4-甲酰基苯基硼酸、一种或多种皂或合成表面活性剂、多元醇例如甘油、ph控制化合物如脂肪酸、柠檬酸、乙酸和/或甲酸、和/或一价阳离子和有机阴离子的盐,其中所述一价阳离子可以是例如na+、k+或nh4+,以及所述有机阴离子可以是例如甲酸盐、乙酸盐、柠檬酸盐或乳酸盐,使得一价阳离子和有机阴离子的盐可以是例如甲酸钠。所述洗衣皂条也可含有络合剂如edta和hedp、香料和/或不同类型的填料、表面活性剂例如阴离子合成表面活性剂、助洗剂、聚合污垢释放剂、洗涤剂螯合剂、稳定剂、填料、染料、着色剂、染料转移抑制剂、烷氧基化聚碳酸酯、抑泡剂、结构剂、粘合剂、浸出剂、漂白活化剂、粘土去污剂、抗再沉积剂、聚合分散剂、增亮剂、织物柔软剂、香料和/或本领域已知的其他化合物。所述洗衣皂条可以在常规的洗衣皂条制造设备中加工,所述制造设备例如但不限于:混合器,压条机例如两级真空压条机,挤出机,切断机,徽标冲压机,冷却隧道和包装机。本发明不限于通过任何单一的方法制备洗衣皂条。本发明的预混物可以在过程的不同阶段添加到到皂中。例如,可以制备含有皂、酶、任选的一种或多种附加的酶、蛋白酶抑制剂以及一价阳离子和有机阴离子的盐的预混物,然后将所述混合物压条。所述酶和任选的附加酶可以与蛋白酶抑制剂同时例如以液体形式加入。除混合步骤和压条步骤之外,所述过程还可包括研磨、挤出、切断、冲压、冷却和/或包裹的步骤。本发明还涉及如本文公开的洗涤剂组合物的不同用途,例如用于降解甘露聚糖和用于洗衣过程。本发明还涉及一种从表面除去污渍的方法,所述方法包括使所述表面与如本文公开的洗涤剂组合物相接触。本发明还涉及一种降解甘露聚糖的方法,所述方法包括将如本文公开的洗涤剂组合物应用于甘露聚糖,优选地,其中所述甘露聚糖在纺织品的表面上。通过降解附着在纺织品或织物上的甘露聚糖,与甘露聚糖结合的污物或污垢被释放并且不能再次与甘露聚糖或甘露聚糖污渍结合。在本发明的一个实施方式中,包含在本发明的洗涤剂组合物中的甘露聚糖酶在45℃至65℃的温度范围内具有至少30%的相对活性。提供在高于环境温度的温度下和在酸性ph中保持活性的甘露聚糖酶对于需要在这样的条件下降解甘露聚糖的应用是有利的。在一个实施方式中,包含在本发明洗涤剂组合物中的甘露聚糖酶随机水解内切-β-1,4-甘露糖苷键。在一个实施方式中,包含在本发明洗涤剂组合物中的甘露聚糖酶可从细菌来源获得或衍生。在一个实施方式中,包含在本发明洗涤剂组合物中的甘露聚糖酶与至少一种其他多肽融合,从而形成融合多肽。除甘露聚糖酶的活性外,所述融合多肽或其他多肽还可具有其他催化或结合活性。在一个实施方式中,所述其他多肽包含碳水化合物结合模块或由其组成,所述碳水化合物结合模块任选地是源自于与甘露聚糖酶相同或不同的生物体的另一种蛋白或酶的片段。甘露聚糖酶可以通过接头与所述其他多肽连接。实施例提供以下实施例以说明本发明的各个方面。它们不意欲限制本发明,本发明由所附权利要求限定。实施例1:筛选为了鉴定新的β-1,4-甘露聚糖酶,筛选了公共数据库(ncbi,ebi)和选定的专有和公共基因组。本工作中使用的所有专有和公共基因组示于表1。将所有的命中进行分组,最后基于相互之间的系统发育距离选择15个细菌来源基因在芽孢杆菌中进行克隆(表2)。表1:用于筛选β-1,4-甘露聚糖酶的专有和公共基因组列表表2:选择用于在芽孢杆菌中克隆的基因列表实施例2:细菌甘露聚糖酶在芽孢杆菌中的克隆除非另有说明,否则包括dna操作和转化在内的分子生物学方法如sambrook和russell(2001)以及harwood和cutting(1990)中所述进行。使用pfxaccuprime聚合酶(invitrogen)通过pcr扩增基因man6、man7和man14。根据制造商的说明书进行pcr。以下pcr条件用于构建表达质粒:94℃初始变性120秒,然后在94℃下进行15秒的35个循环,在下面50/55℃之一下退火30秒,在68℃下延伸110/290秒,在68℃下最后延伸10分钟。为了扩增man7,使用解半纤维素芽孢杆菌jcm9152的基因组dna。man6和man14作为没有密码子优化的合成基因订购(eurofinsmwg,德国)。用于克隆的引物的序列示于表3中。用于杂交的突出端有下划线。表3:用于扩增man6、man7和man14的引物列表通过使用高保真dna组装主混合物(neb,frankfurt),将基因克隆到标准载体pev1pev1(图1)中,pub110衍生物包括来自地衣芽孢杆菌的启动子papre和来自解淀粉芽孢杆菌的木聚糖酶信号肽。载体:插入片段比例1:3用于克隆。片段的总量是在总体积20μl中0.2pmol。将样品在50℃下温育40分钟。为了构建目的,通过如zhang&zhang2011中所述,在枯草芽孢杆菌sck6中的诱导感受态来转化表达质粒。将转化的细胞在补充有10mg/l卡那霉素的lb(luria-bertani)平板上铺板。将平板在37℃温育20小时。挑取出突起的集落,用qiaprep微量制备试剂盒(qiagen,hilden)分离质粒。根据制造商对革兰氏阳性质粒制剂的推荐进行分离程序。通过sanger测序(gatc,德国)对插入片段进行测序,并展现对应于甘露聚糖酶man6、man7和man14的成熟部分的dna序列。使用clustalw序列比对(thompson等人1994)进行序列比较。最后,通过电穿孔将表达质粒转化到合适的芽孢杆菌生产菌株中。芽孢杆菌生产菌株在含有20g/l胰蛋白胨、10g/l酵母提取物、10gnacl和2m蔗糖的电穿孔培养基中生长,并在od(600nm)为0.4时收获10ml。用含有0.272m蔗糖、1mmmgcl2和7mmkh2po4的电穿孔缓冲液洗涤细胞,最后重悬于250μl电穿孔缓冲液中。使用以下条件进行电穿孔:1.2kv,150ω,50μf。之后添加1ml电穿孔培养基,并将细胞在37℃温育3小时。将细胞在补充有20mg/l卡那霉素的lb平板上铺板,并在37℃温育18小时。如上所述验证克隆并用于产生用于分析测试的材料。因此,以在蛋白质诱导条件下标准表达来接种菌株,并在37℃温育30小时。收获上清液并用于分析和应用测试。基因和酶特征如表4和5所示。表4.对来自克劳氏芽胞杆菌ksm-k16、解半纤维素芽胞杆菌jcm9152和土壤枝芽孢杆菌pl205的gh5家族甘露聚糖酶编码基因的概述基因包括sp在内的长度(bp)seqidnoman69759man7147313man14144917表5.对来自克劳氏芽胞杆菌ksm-k16、解半纤维素芽胞杆菌jcm9152和土壤枝芽孢杆菌pl205的gh5家族甘露聚糖酶编码基因的推断氨基酸序列的概述实施例3:细菌甘露聚糖酶man6和man7在里氏木霉中的pcr克隆标准分子生物学方法用于dna的分离和酶处理(例如,质粒dna的分离,dna的消化以产生dna片段)、大肠杆菌转化、测序等。使用的基本方法或是如酶、试剂或试剂盒制造商所述,或是如标准分子生物学手册、例如sambrook和russell(2001)中所述。基因组dna的分离如raeder和broda(1985)详细描述的那样进行。还分别克隆了来自克劳氏芽孢杆菌和解半纤维素芽孢杆菌的man6和man7,以在里氏木霉中表达。使用具有里氏木霉密码子优化的合成基因对所述基因进行pcr克隆。使用pcr除去编码man6和man7的信号肽的dna序列,并创建新的克隆位点。引物的序列示于表6(seqidno:21-24)。表6.用作pcr引物以扩增解半纤维素芽孢杆菌和克劳氏芽孢杆菌甘露聚糖酶基因的寡核苷酸(a括号中的“s”=有义链,“as”=反义链。用表6中所述的引物并使用合成的dna作为反应中的模板,通过pcr扩增所述基因。克劳氏芽胞杆菌man6和解半纤维素芽胞杆菌man7的pcr混合物各含有1x用于phusionhf聚合酶的hf缓冲液(neb/bionordika,芬兰)、0.2mmdntp混合物(thermofisherscientific,芬兰)、每种引物1μm、3%dmso(thermofisherscientific)、1单位的phusion高保真聚合酶(neb/bionordika,芬兰)和50ng相应的质粒dna。以下pcr反应的条件如下:98℃初始变性30秒,然后在98℃下进行10秒的28个循环,在下面45/50/55/60℃之一下退火30秒,在72℃下延伸45秒,在72℃下最后延伸7分钟。表6中描述的引物组合产生具有预期大小的特定dna产物。用genjet凝胶提取试剂盒(thermofisherscientific)根据制造商的说明从琼脂糖凝胶中分离pcr产物,用nrui和bamhi限制酶(thermofisherscientific)消化,并克隆到用nrui和bamhi切割的表达载体中。将连接混合物转化到大肠杆菌xl1-blue(ahdiagnostics)中并在含有50-100μg/ml氨苄青霉素的lb(luria-bertani)平板上铺板。从所述平板收集几个大肠杆菌菌落,用genjet质粒微量制备试剂盒(thermofisherscientific)分离dna。使用限制性消化筛选阳性克隆。对编码克劳氏芽胞杆菌man6和解半纤维素芽胞杆菌man7gh5甘露聚糖酶且没有自身信号肽编码序列的基因进行测序,并将质粒分别命名为palk4274和palk4273(详见实施例6)。实施例4:合成的细菌甘露聚糖酶man14的克隆标准分子生物学方法用于dna的分离和酶处理(例如,质粒dna的分离,dna的消化以产生dna片段)、大肠杆菌转化、测序等。使用的基本方法或是如酶、试剂或试剂盒制造商所述,或是如标准分子生物学手册、例如sambrook和russell(2001)中所述。基因组dna的分离如raeder和broda(1985)详细描述的那样进行。还将来自土壤枝芽孢杆菌的甘露聚糖酶基因man14克隆用于木霉表达。来自土壤枝芽孢杆菌的gh5家族甘露聚糖酶man14的编码基因作为具有里氏木霉密码子优化的合成构建体从genscript订购。将从genscript获得的包含man14基因的质粒dna重悬于无菌水中,用nrui和bamhi限制酶(thermofisherscientific)根据制造商的说明书消化,并克隆到用nrui和bamhi切割的表达载体中。将连接混合物转化到大肠杆菌xl1-blue(ahdiagnostics)中并在含有50-100μg/ml氨苄青霉素的lb(luria-bertani)平板上铺板。从所述平板收集几个大肠杆菌菌落,用genjet质粒微量制备试剂盒(thermofisherscientific)分离dna。阳性克隆使用限制性消化来筛选,并且它们显示出含有预期大小的插入片段。对土壤枝芽孢杆菌属man14与表达质粒的融合位点进行测序,并将所述质粒命名为palk4414(详情参见实施例6)。实施例5:芽孢杆菌中重组细菌gh5甘露聚糖酶蛋白的产生构建表达质粒用于产生来自克劳氏芽孢杆菌、解半纤维素芽孢杆菌和土壤枝芽孢杆菌的重组gh5甘露聚糖酶(man6、man7和man14)蛋白。构建的表达质粒列于表7中。将没有自声信号序列的重组gh5基因(man6、man7和man14)与地衣芽孢杆菌papre启动子和解淀粉芽孢杆菌木聚糖酶信号肽融合。通过强终止子确保转录终止,并使用卡那霉素抗性标记选择转化体。转化如实施例2中所述进行。表7.被构建成在适当的芽孢杆菌表达菌株中产生来自克劳氏芽孢杆菌、解半纤维素芽孢杆菌和土壤枝芽孢杆菌的man6、man7和man14重组蛋白的表达质粒甘露聚糖酶(gh5)蛋白表达质粒man6pev1man6man7pev1man7man14pev1man14从摇瓶培养的培养上清液中分析转化体的gh5产生。将所述转化体从lb平板接种到含有2%葡萄糖、6%玉米浆干粉、1.3%(nh4)2hpo4、0.05%mgso4×7h2o和0.5%cacl2的摇瓶中。将ph调节至ph7.5。在转化体在37℃、180rpm下生长30小时后,从培养物上清液中分析它们的gh5蛋白质产量。通过sds-page与随后的考马斯(coomassie)染色分析重组蛋白的异源产生。选择最佳生产转化体在实验室规模的生物反应器中培养。将所述转化体在生物反应器中在蛋白质诱导条件下37℃培养并继续饲养直至达到合适的收率。通过离心或过滤回收上清液用于应用测试。实施例6:在里氏木霉中产生重组细菌gh5甘露聚糖酶蛋白构建表达质粒用于在里氏木霉中产生来自克劳氏芽孢杆菌、解半纤维素芽孢杆菌和土壤枝芽孢杆菌的重组gh5甘露聚糖酶(man6、man7和man14)蛋白(参见实施例3和4)。构建的表达质粒列于表8中。用里氏木霉cel6a/cbh2cbm载体和接头将没有自身信号序列的重组gh5基因(man6、man7和man14)与里氏木霉cel7a/cbh1启动子融合,然后与kex2蛋白酶识别位点融合。通过里氏木霉cel7a/cbh1终止子确保转录终止,并如paloheimo等人所述(2003年),使用构巢曲霉(a.nidulans)amds标记基因用于选择转化体。在noti消化后从载体骨架分离线性表达盒(图2)并转化到里氏木霉原生质体中。使用的宿主菌株不产生四种主要的里氏木霉纤维素酶(cbhi,cbhii,egi,egii)中的任何一种。转化如penttila等人(1987)中所述加以karhunen等人所描述的修改(1993)进行,选择乙酰胺酶作为唯一的氮源(amds标记基因)。在选择平板上通过单个分生孢子纯化转化体,然后将它们在pd上形成孢子。表8.被构建成在里氏木霉中产生来自克劳氏芽孢杆菌、解半纤维素芽孢杆菌和土壤枝芽孢杆菌的man6、man7和man14重组蛋白的表达质粒。表达盒的总体结构如图2所述。露聚糖酶(gh5)蛋白表达质粒表达盒(aman6palk42747.0kbnotiman7palk42737.5kbnotiman14palk44147.6kbnoti(a通过使用noti消化从载体骨架分离用于里氏木霉转化的表达盒。从摇瓶培养的培养上清液分析转化体的甘露聚糖酶产生。将转化体从pd斜面接种到含有50ml用5%kh2po4缓冲的复合乳糖基纤维素酶诱导培养基(joutsjoki等人,1993)的摇瓶中。转化体在30℃、250rpm下生长7天后,从培养物上清液中分析它们的gh5蛋白产生。通过sds-page与随后的考马斯(coomassie)染色分析重组蛋白的异源产生。选择最佳生产转化体在实验室规模的生物反应器中培养。在典型的喜中温真菌培养温度和微酸性条件下,在蛋白诱导条件下,在生物反应器中分批或通过另外的饲养类型的方法培养所述转化体。连续培养直至培养基糖耗尽或直至达到合适的收率。通过离心或过滤回收上清液用于应用测试。实施例7:通过dns方法测定半乳甘露聚糖酶活性甘露聚糖酶活性(mnu)按50℃和ph7.0下5分钟内从半乳甘露聚糖(0.3w/w%)释放的还原糖测量。使用二硝基水杨酸通过分光光度法确定释放的还原性碳水化合物的量。如下制备所述测定中使用的底物(0.3w/w%):0.6g刺槐豆胶(sigmag-0753)溶于使用加热磁力搅拌器为约80℃的50mm柠檬酸钠缓冲液ph7(或柠檬酸盐磷酸盐缓冲液ph7)中,并加热至沸点。将溶液在冷室(2-8℃)中冷却并使其在连续搅拌下溶解过夜,通过离心除去不溶残渣。之后,通过缓冲液将溶液填充至200ml。将底物冷冻保存并在沸水浴中加热融化至约80℃,冷却至室温并在使用前小心混合。通过将50g的3,5-二硝基水杨酸(sigmad-550)溶解在约4升水中来制备所述测定中使用的dns试剂。在连续磁力搅拌下,逐渐添加80.0g的naoh并使其溶解。在连续搅拌下分小份添加1500g量的罗谢尔盐(rochellesalt)(酒石酸盐钾钠,merck8087)。将可能已经小心温热至最高温度45℃的溶液冷却至室温并填充至5000ml。之后,将其通过whatman1号滤纸过滤并在室温下储存在深色瓶中。首先通过向两个试管中的每一个添加1.8ml底物溶液来开始反应,并使其在50℃下平衡5分钟,之后将200μl适当稀释的酶溶液添加到其中一个试管,用涡旋混合器充分混合并在50℃温育正好5分钟。酶空白不需要平衡或温育。通过向两个试管中添加3.0mldns试剂并混合来终止反应。将200μl样品溶液添加到酶空白管中。将这两个管置于沸水浴中。煮沸正好5分钟后,将所述试管置于冷却水浴中并使其冷却至室温。在540nm处针对酶空白测量样品的吸光度,并从校准曲线读取活性并乘以稀释因子。合适的稀释样品产生0.15-0.4的吸光度差异。通过将360mg甘露糖(sigmam-6020,储存在干燥器中)溶解在测定缓冲液中并稀释成含有3、6、10和14μmol/ml甘露糖的溶液,从20mm甘露糖原液制备标准曲线。除了在50℃下温育之外,标准品的处理与样品相同。在540nm处针对试剂空白(含有缓冲液而不是甘露糖标准稀释液)测量吸光度。为每个测定系列构建校准曲线。一个甘露聚糖酶单位(mnu)定义为在测定条件下,在1秒内产生的还原性碳水化合物具有相当于来自半乳甘露聚糖的1nmol甘露糖的还原能力的酶量(1mnu=1nkat)。实施例8:man6甘露聚糖酶的纯化通过在4℃下以4000g离心10分钟,从发酵培养基中除去细胞和固体。10ml上清液用于蛋白质纯化。将样品通过0.44μmpvdf膜(millex-hv,merckmilliporeltd,carrigtwohill,irl)过滤。将滤液加载到在20mmhepesph7中平衡的hiprep26/10脱盐柱(gehealthcare,uppsala,瑞典)上。然后将脱盐的样品加载到用20mmhepesph7预平衡的5mlhitrapqhp柱(gehealthcare,uppsala,瑞典)上。样品加载后,用相同的缓冲液20ml洗涤柱子。用在15个cv中的线性盐梯度20mmhepes、500mmnaclph7洗脱蛋白质。收集5ml的级分并在sds-page上分析。将含有靶蛋白的级分合并,并使用vivaspin20、10kdamwco超滤装置(gehealthcare)浓缩至2ml。所述浓缩的样品使用用20mmmes,200mmnaclph6.5平衡的superdex7526/60凝胶过滤柱进一步分级。收集2ml的级分并通过sds-page分析。将含有纯甘露聚糖酶的级分合并。其他甘露聚糖酶使用相同的方案纯化,但在脱盐和离子交换步骤中改变缓冲液组成。缓冲液组合物示于表9中。表9.用于离子交换色谱的缓冲液甘露聚糖酶用于离子交换色谱的缓冲液man620mmhepesph7man720mmhepesph7man1420mmmesph6纯化的样品纯度超过95%。使用uv吸光度280nm测量来确定纯化样品的酶含量。通过使用expasy服务器http://web.expasy.org/protparam/,根据所述酶的氨基酸序列计算每种甘露聚糖酶的激发系数(gasteigere等人2005)。纯化样品的酶活性(mnu)如实施例7中所述按还原糖的释放进行测量。通过将纯化样品的mnu活性除以纯化酶的量,来计算甘露聚糖酶的比活性(mnu/mg)。获得的值用于计算实施例10和11中使用的酶剂量。甘露聚糖酶的ph曲线使用来自megazyme的β-甘露糖酶片剂测定天青蛋白交联的角豆半乳甘露聚糖(t-mnz11/14),对所建议的方案进行少许修改,确定纯化的甘露聚糖酶的ph曲线。已经用每种纯化的酶检查了所述测定的线性。所述测定在40mmbritton-robinson缓冲液中进行,所述缓冲液被调节至ph值在4和11之间。将所述酶溶液稀释到测定缓冲液中,并将500μl的酶溶液在50℃水浴中平衡5分钟,然后加入一个底物片剂。10分钟后,通过添加10ml2%trisph12终止反应。将反应试管在室温下放置5分钟,搅拌并将液体通过whatman1号滤纸过滤。通过测量595nm处的吸光度来定量从底物释放的蓝色染料。将每种ph下的酶活性报告为相对活性,其中将在最适ph下的活性设定为100%。ph曲线示于图3中。甘露聚糖酶的相对活性(%)通过将样品的甘露聚糖酶活性除以参比样品的甘露聚糖酶活性来计算。在ph曲线的情况下,参比样品是在最适ph下的样品。在温度曲线的情况下,参比样品是在最适温度下的样品。甘露聚糖酶的温度曲线使用来自megazyme的β-甘露糖酶片剂测定天青蛋白交联的角豆半乳甘露聚糖(t-mnz11/14),对所建议的方案进行少许修改,确定纯化的甘露聚糖酶的最适温度。所述测定在40mmbritton-robinson缓冲液ph7中、在30-90℃之间变化的温度下进行10分钟。将酶活性报告为相对活性,其中将最适温度下的活性设定为100%。温度曲线示于图4中。甘露聚糖酶的温度和ph特征man6的分子量在30-35kda之间。所述酶在ph7下的最适温度是50℃至70℃。所述酶在50℃下的最适ph在至少ph6至ph9的ph范围内。最适温度和最适ph使用10分钟反应时间和天青蛋白交联的角豆半乳甘露聚糖作为底物确定。man7的分子量在50-55kda之间。所述酶在ph7下的最适温度是50℃至70℃。所述酶在50℃下的最适ph在至少ph7至ph10的ph范围内。最适温度和最适ph使用10分钟反应时间和天青蛋白交联的角豆半乳甘露聚糖作为底物确定。man14的分子量在30-40kda之间。所述酶在ph7下的最适温度是50℃至70℃。所述酶在50℃下的最适ph在至少ph7至ph8的ph范围内。最适温度和最适ph使用10分钟反应时间和天青蛋白交联的角豆半乳甘露聚糖作为底物确定。实施例9:man6和man7甘露聚糖酶与没有漂白剂的商业洗涤剂一起的去污性能在芽孢杆菌(如实施例5中所述)和木霉(如实施例6中所述)中产生的man6和man7甘露聚糖酶,测试它们与没有漂白剂的商业洗涤剂一起在40℃和水硬度16°dh下除去甘露聚糖酶敏感的标准污渍的能力,并与商业甘露聚糖酶制剂4.0l(novozymes)比较。使用来自测试材料中心b.v.(centerfortestmaterialb.v.)(荷兰)的以下人工染污的测试布:在棉上甘露聚糖酶敏感的巧克力布丁(e-165),在棉上(cs-73)和聚酯/棉(pc-s-73)上刺槐豆胶与颜料,以及在棉上瓜尔胶与炭黑(cs-43)。将织物切成6cmx6cm的样片,并将各2块用于测试。含有除甘露聚糖酶以外的所有其他酶的商业重垢液体洗涤剂a以每升洗液4.4g的浓度使用,而不含酶的商业护色洗涤剂粉末以3.8g/l使用。我们在硬度为16°dh的合成自来水中制备的含洗涤剂的洗液。将蛋白酶16l(0.5w/w%)和淀粉酶12l(0.4w/w%)加入与商业护色洗涤剂粉末一起使用的硬水中,所述液体洗涤剂已含有淀粉酶和蛋白酶。所述护色洗涤剂粉末的洗液的ph值为约10,所述液体洗涤剂的ph值为约8.3。甘露聚糖酶的剂量在洗涤剂重量的0-0.2/0.25%范围内,但是为了评价,所述剂量计算为每毫升洗液的酶活性单位(mnu)或每升洗液的毫克活性酶蛋白(aep)。如实施例7中所述测量活性。通过将酶活性除以实施例8中定义的比活性来计算每种制剂的aep含量。对照样品含有洗涤剂溶液但没有甘露聚糖酶。对于硬度为16°dh的合成自来水,在去离子水(milli-q或等效物)中制备以下原液:钙硬度为1000°d的原液:cacl2×2h2o(1.02382.1000,merckkgaa,德国)26.22g/l镁硬度为200°d的原液:mgso4×7h2o(1.05886.1000,merckkgaa,德国)8.79g/lh2onahco3原液:nahco3(1.06329.1000merckkgaa,德国)29.6g/l将13.3mlcacl2溶液、13.3mlmgso4溶液和10.0ml新制备的nahco3溶液以给出的顺序加入容量瓶中,用去离子水补足至1升并混合。通过络合滴定法确定水的硬度,发现是正确的。如下在atlaslp-2水洗色牢度计(launder-ometer)中进行去污处理。水洗色牢度计首先被预热到40℃。然后将洗涤剂、250ml硬度为16°dh的合成自来水和稀释的酶(<1.0ml)加入1.2升容器中。添加污渍并将所述水洗色牢度计在40℃下以42rpm的转速运行60分钟。之后,将样品在流水下小心冲洗并在防日光的栅格上在室内空气中干燥过夜。通过用konicaminoltacm-3610a分光光度计使用l*a*b*色空间坐标(光源d65/100,420nm截止),按反射率值测量颜色来评价去污效果。污渍的褪色,表明甘露聚糖酶性能(去污效率),以δl*(deltal*)计算,其意味着经酶处理的织物的亮度值l*减去用没有甘露聚糖酶的洗液(对照)处理的织物的亮度值l*。最终结果(总去污效果)显示为各污渍的δl*之和。各污渍的色值是2个样片的平均值。用商业液体洗涤剂获得的结果示于图6-7中。与商业甘露聚糖酶制剂4.0l相比,当以活性单位或以活性酶蛋白计量配给时,本发明的甘露聚糖酶与液体洗涤剂一起具有相似的(man6)或明显更好的(man7)去污性能。不管表达宿主是芽孢杆菌还是木霉,用man6和man7都获得了相似的性能(图6)。用商业护色洗涤剂粉末获得的结果(图8-9)表明,与商业甘露聚糖酶制剂4.0l相比,当以活性单位或以活性酶蛋白计量配给时,本发明的甘露聚糖酶具有更好的去污性能。.实施例10:man6和man7甘露聚糖酶与含有漂白剂的洗涤剂一起的去污性能在芽孢杆菌(如实施例5中所述)和木霉(如实施例6中所述)中产生的man6和man7甘露聚糖酶,测试它们商业漂白洗涤剂一起在40℃和水硬度16°dh下除去甘露聚糖酶敏感的标准污渍的能力,并与商业甘露聚糖酶制剂4.0l(novozymes)比较。测试系统类似于实施例9中所述,不同在于使用来自材料中心b.v.(荷兰)的3种不同的人工染污的测试布:在棉上甘露聚糖酶敏感的巧克力布丁(e-165),在棉上刺槐豆胶与颜料(cs-73),以及在棉上瓜尔胶与炭黑(cs-43)。商业护色洗涤剂粉末以每升洗液4.2g的浓度使用,洗液的ph值为约9.5。因为洗涤剂不含任何酶,所以将蛋白酶16l(0.5w/w%)和淀粉酶12l(0.4w/w%)添加到试验中使用的硬水中。测量所述样片在处理后的颜色,并如实施例9中所述,将结果计算为各3个污渍的δl*的总和。用商业含漂白剂的洗涤剂获得的结果(图10)表明,当以活性酶蛋白计量配给时,本发明的甘露聚糖酶(man7)与商品甘露聚糖酶4.0l相比具有明显更好的去污性能。与商业细菌甘露聚糖酶相比,用man6获得了至少具有相似的性能。实施例11:man14甘露聚糖酶与商业液体洗涤剂一起的去污性能测试芽孢杆菌中产生的man14甘露聚糖酶(如实施例5中所述)与商业重垢液体洗涤剂b一起在40℃和水硬度为16°dh时去除甘露聚糖酶敏感性标准污渍的能力,并与商业甘露聚糖酶制剂4.0l(novozymes)进行比较。测试系统类似于实施例9中所述,除了使用来自材料中心b.v.(荷兰)的两种不同的人工染污测试布:在棉上甘露聚糖酶敏感的巧克力布丁(e-165),和在棉上刺槐豆胶与颜料(cs-73)。商业重垢液体洗涤剂b以每升洗液5克的浓度使用,并且洗液的ph值为约8.3。将蛋白酶16l(0.5w/w%)和淀粉酶12l(0.4w/w%)添加到用于测试的硬水中,因为所述洗涤剂不含任何酶。测量所述样片在处理后的颜色,并如实施例9中所述,将结果计算为各2个污渍的δl*之和。用含洗涤剂的商业液体获得的结果(图11-12)表明,当按活性单位或按活性酶蛋白配量时,man14在液体洗涤剂中具有与商业产品相当的良好性能。实施例12:man6和man7甘露聚糖酶在商业液体洗涤剂中的稳定性在从当地超市获得的omocolor液中测试芽孢杆菌中产生的man6和man7甘露聚糖酶制剂的稳定性,并与商业甘露聚糖酶制剂4.0l进行比较。在洗涤剂中添加0.5w/w%的甘露聚糖酶制剂,并将样品在带盖的塑料管中于37℃温育5周。除了使用30分钟温育时间外,通过实施例7中描述的活性测定以一定间隔测量活性。结果计算为残余活性(%),其通过将在某个时间点取得的样品的活性除以样品的初始活性而获得。在芽孢杆菌和木霉属二者中产生的man7的稳定性以及在木霉属中产生的man6还在含有蛋白酶但不含甘露聚糖酶的商业液体重垢洗涤剂a中相对于4.0l测试。在该试验中,使用1%-(w/w)的甘露聚糖酶,并将样品在37℃下温育12周。在omocolor中的结果(图13)表明,与4.0l相比,man6明显更好并且man7相似。与另一种商业液体洗涤剂a一起,man7和man6并且尤其man6均比4.0l更稳定,如图14所示。在相同条件下的另一个测试中获得的结果显示,不管表达宿主是芽孢杆菌还是木霉,man6都具有相似的稳定性(数据未显示)。稳定性实验的结果表明,即使当在高温如37℃下储存时,本发明的甘露聚糖酶在洗涤剂中稳定长达数周。与商业细菌甘露聚糖酶相比,本发明的甘露聚糖酶(man6和man7)在液体洗涤剂中的的稳定性改善。实施例13:本发明的液体洗涤剂组合物的洗涤性能本发明的液体洗涤剂组合物的洗涤性能通过使用可从cft(测试材料中心)b.v.,vlaardingen,荷兰(“cft”);material-undprufanstalttestmaterialienag[联邦材料和测试机构,测试材料]st.gallen,瑞士(“empa”);和warwickequestltdunit55,consettbusinesspark,consett,countydurham(“equest”)获得的标准化污渍确定。使用具有以下组成的液体洗剂作为基础配方(所有值均为重量百分比):化学名活性物质原料[%]活性物质洗涤剂配方[%]去矿质水100余量烷基苯磺酸962-7阴离子表面活性剂706-10c12-c18脂肪酸钠盐301-4非离子表面活性剂1004-7膦酸盐400.1-2柠檬酸1001-3naoh501-4硼酸1000.1-2消泡剂1000.01-1甘油1001-3酶1000.1-2防腐剂1000.05-1乙醇930.5-2光学增亮剂900.01-1香料1000.1-1染料1000.001-0.1所述洗涤剂组合物的ph值在8.2-8.6之间。基于该基础配方,通过添加如下所示的相应酶来制备液体洗涤剂组合物1和2:组合物1:根据seqidno:12的酶(man6)组合物2:根据seqidno:16的酶(man7)根据aise方法如下进行洗涤:3.5kg清洁压载布,4块sbl布,miele洗衣机,20℃和40℃短程序。所有甘露聚糖酶均基于总蛋白质含量以相同的量添加。所述液体洗涤剂的配量比为每升洗液4.0克。洗涤程序在20℃和40℃的温度下进行60分钟,水的水硬度在15.5°和16.5°(德国硬度)之间。得到的结果是用所述洗涤剂得到的减退单位和用含有可商购的参比甘露聚糖酶(mannaway4.0l,得自novozymes)的洗涤剂得到的减退单位之间的差值。因此,正值表明与包含所述参比甘露聚糖酶的相同洗涤剂组合物相比,包含本发明甘露聚糖酶的洗涤剂组合物的洗涤性能得到改善。在所述洗涤测试中,测试了大范围的污渍。通过光度法确定污渍的白度,即增亮,作为洗涤性能的指示。使用minoltacm508d光谱仪装置,其使用随所述装置一起提供的白度标准预先校准。得到的结果是用所述洗涤剂得到的减退单位和用含有所述酶组合的洗涤剂得到的减退单位之间的差值。因此,正值表明由于洗涤剂中存在的所述酶组合而洗涤性能改善。洗涤剂组合物中本发明的甘露聚糖酶对各种含有甘露聚糖的污渍显示出改善的性能。实施例14:本发明的粉末洗涤剂组合物的洗涤性能本发明的粉末洗涤剂组合物的洗涤性能通过使用可从cft(测试材料中心)b.v.,vlaardingen,荷兰(“cft”);material-undprufanstalttestmaterialienag[联邦材料和测试机构,测试材料]st.gallen,瑞士(“empa”);和warwickequestltdunit55,consettbusinesspark,consett,countydurham(“equest”)获得的标准化污渍确定。使用具有以下组成的固体洗剂作为基础配方(所有值均为重量百分比):化学名称活性物质原料[%]活性物质洗涤剂配方[%]去矿质水1001-4烷基苯磺酸979-13非离子表面活性剂1004-7过碳酸盐889-13taed921-5膦酸盐600.1-3聚丙烯酸酯451-4硅酸钠405-10碳酸钠10018-22羧甲基纤维素691-4污垢释放聚合物1000.1-1光学增亮剂700.1-1消泡剂t.q.0.01-1硫酸钠10022-30酶1000.1-1香料1000.1-1naoh1000.1-1其余-1-4基于该基础配方,通过添加如下所示的相应的酶来制备固体洗涤剂组合物3和4:组合物3:根据seqidno:12的酶(man6)组合物4:根据seqidno:16的酶(man7)根据aise方法如下进行洗涤:3.5kg清洁压载布,4块sbl布,miele洗衣机,20℃和40℃短程序。所有甘露聚糖酶均基于总蛋白质含量以相同的量添加。所述粉末洗涤剂的配量比为每升洗液3.8克。洗涤程序在20℃和40℃的温度下进行60分钟,水的水硬度在15.5°和16.5°(德国硬度)之间。通过光度法确定污渍的白度,即增亮,作为洗涤性能的指示。使用minoltacm508d光谱仪装置,其使用随所述装置一起提供的白度标准预先校准。得到的结果是用所述洗涤剂得到的减退单位和用含有参比甘露聚糖酶(mannaway4.0l,得自novozymes)的洗涤剂得到的减退单位之间的差值。因此,正值表明所述变体在洗涤剂中的洗涤性能改善。本发明的甘露聚糖酶对几种污渍显示出改善的性能。因此,显然本发明的甘露聚糖酶与mannaway相比显示出改善的洗涤性能。在不限制专利权利要求的范围和解释的情况下,本文中公开的一个或多个方面或实施方式的某些技术效果在下面列出:技术效果是甘露聚糖的降解或改性。另一个技术效果是提供具有良好储存稳定性的甘露聚糖酶。前面的描述已经作为本发明的特定实现和实施方式的非限制性示例提供了发明人当前对于实施本发明所设想的最佳模式的完整且提供信息的描述。然而,对于本领域技术人员来说很清楚,本发明不局限于上面提供的实施方式的细节,而是可以在不偏离本发明特征的情况下使用等同手段在其他实施方式中实现。此外,可以有利地使用本发明的上述公开方面和实施方式的一些特征而无需相应地使用其他特征。因此,前面的描述应该被认为仅仅是对本发明原理的说明,而不是对其的限制。因而,本发明的范围仅受所附的专利权利要求的限制。在一个实施方式中,与至少一种组分所源自的相应天然组分相比,本发明组合物的所述至少一种组分具有不同的化学、结构或物理特征。在一个实施方式中,所述特征是大小均一、均匀分散、不同的同种型、不同的密码子简并、不同的翻译后修饰、不同的甲基化、不同的三级或四级结构、不同的酶活性、不同的亲和力、不同的结合活性和不同的免疫原性中的至少一种。sequencelisting<110>henkelag&co.kgaa<120>detergentcompositionscomprisingbacterialmannanases<130>pt034268wo<160>48<170>patentinversion3.5<210>1<211>39<212>dna<213>artificialsequence<220><223>primer<400>1caaccgcctctgcagcttatgcacaaaacggatttcacg39<210>2<211>39<212>dna<213>artificialsequence<220><223>primer<400>2cggtatatctctgtcttaatcactcttaagcccattttc39<210>3<211>37<212>dna<213>artificialsequence<220><223>primer<400>3caaccgcctctgcagcttctgatggtcatagccaaac37<210>4<211>36<212>dna<213>artificialsequence<220><223>primer<400>4cggtatatctctgtcttattggattgttacatgatc36<210>5<211>40<212>dna<213>artificialsequence<220><223>primer<400>5caaccgcctctgcagctgcaagcgggttttatgtaaacgg40<210>6<211>39<212>dna<213>artificialsequence<220><223>primer<400>6cggtatatctctgtcttatttaatggtaacgttatcaac39<210>7<211>17<212>dna<213>artificialsequence<220><223>primer<400>7agctgcagaggcggttg17<210>8<211>21<212>dna<213>artificialsequence<220><223>primer<400>8gacagagatataccgacagtg21<210>9<211>975<212>dna<213>bacillusclausii<400>9atgaagagggaggacatggatcaaatgaaaagaaagcggttacaattgtttggaacacta60gtggtattggttttgttcgtgtacggtagcggttcggcatatgcacaaaacggatttcac120gtatccggtacagagttgttggacaaaaatggcgatccttatgttatgcgtggcgtcaac180catggacactcttggtttaagcaagatctggaggaagcaatccctgccatagcagaaaca240ggggcgaacacggtgagaatggtcttatccaatggacagcaatgggaaaaagatgatgcc300tctgagcttgcccgtgtgctggctgccacagaaacatatggattgacaactgtgctggaa360gtccatgacgctacaggaagtgacgatcctgctgatttagagaaagcagtcgattattgg420atcgaaatggctgatgttctcaaggggacagaagaccgagtaatcattaacgttgccaat480gaatggtatgggtcgtggaggagcgacgtttgggcagaagcatacgcacaagcgatcccg540cgcttgcgcagcgctggcctctcccatacattaatggttgatgcggcaggttggggccag600taccctgcctccatccacgagcggggagccgatgtgtttgcgtccgatccattaaaaaac660acgatgttttcgatccatatgtacgaatatgcaggagctgatagggcgacaattgcctat720aacattgatcgtgtgcttgctgaaaatcttgctgtggtgatcggtgaatttggccatagg780catcatgatggcgatgtcgatgaagatgcgattttggcctatacagcagagcggcaagtg840ggctggctggcctggtcatggtatggcaacagcgggggtgttgaatacttggatttagct900gaaggcccatcaggcccattgacgagttggggcaaacgaattgtttatggtgaaaatggg960cttaagagtgattaa975<210>10<211>870<212>dna<213>bacillusclausii<400>10cagaacggcttccacgtctccggcacggagctcctggacaagaacggcgacccttacgtc60atgcgcggcgtcaaccacggccacagctggttcaagcaggacctcgaggaagccatccct120gctatcgctgagacgggcgctaacacggtccgcatggtcctgagcaacggccagcagtgg180gagaaggacgacgctagcgagctggctcgcgtcctcgctgctacggagacgtacggcctc240accacggtcctggaggtccacgacgctacgggctcggacgaccccgccgacctcgagaag300gccgtcgactactggatcgagatggctgacgtcctgaagggcaccgaggaccgcgtcatc360atcaacgtcgccaacgagtggtacggctcctggcgcagcgacgtctgggccgaggcctac420gctcaggctatccctcgcctccgctcggccggcctctcccacacgctcatggtcgacgct480gccggctggggccagtaccctgcttccatccacgagcgcggcgctgacgtctttgcttcg540gaccccctgaagaacaccatgttctccatccacatgtacgagtacgctggcgctgaccgc600gctaccatcgcctacaacatcgaccgcgtcctggctgagaacctggctgtcgtcatcggc660gagtttggccaccgccaccacgacggcgacgtcgacgaggacgctatcctggcttacacc720gccgagcgccaggtcggctggctggcttggtcgtggtacggcaactcgggcggcgtcgag780tacctggacctggctgagggcccttcgggccctctcacgagctggggcaagcgcatcgtc840tacggcgagaacggcctgaagagcgactaa870<210>11<211>324<212>prt<213>bacillusclausii<400>11metlysarggluaspmetaspglnmetlysarglysargleuglnleu151015pheglythrleuvalvalleuvalleuphevaltyrglyserglyser202530alatyralaglnasnglyphehisvalserglythrgluleuleuasp354045lysasnglyaspprotyrvalmetargglyvalasnhisglyhisser505560trpphelysglnaspleugluglualaileproalailealagluthr65707580glyalaasnthrvalargmetvalleuserasnglyglnglntrpglu859095lysaspaspalasergluleualaargvalleualaalathrgluthr100105110tyrglyleuthrthrvalleugluvalhisaspalathrglyserasp115120125aspproalaaspleuglulysalavalasptyrtrpileglumetala130135140aspvalleulysglythrgluaspargvalileileasnvalalaasn145150155160glutrptyrglysertrpargseraspvaltrpalaglualatyrala165170175glnalaileproargleuargseralaglyleuserhisthrleumet180185190valaspalaalaglytrpglyglntyrproalaserilehisgluarg195200205glyalaaspvalphealaseraspproleulysasnthrmetpheser210215220ilehismettyrglutyralaglyalaaspargalathrilealatyr225230235240asnileaspargvalleualagluasnleualavalvalileglyglu245250255pheglyhisarghishisaspglyaspvalaspgluaspalaileleu260265270alatyrthralagluargglnvalglytrpleualatrpsertrptyr275280285glyasnserglyglyvalglutyrleuaspleualagluglyproser290295300glyproleuthrsertrpglylysargilevaltyrglygluasngly305310315320leulysserasp<210>12<211>289<212>prt<213>bacillusclausii<400>12glnasnglyphehisvalserglythrgluleuleuasplysasngly151015aspprotyrvalmetargglyvalasnhisglyhissertrpphelys202530glnaspleugluglualaileproalailealagluthrglyalaasn354045thrvalargmetvalleuserasnglyglnglntrpglulysaspasp505560alasergluleualaargvalleualaalathrgluthrtyrglyleu65707580thrthrvalleugluvalhisaspalathrglyseraspaspproala859095aspleuglulysalavalasptyrtrpileglumetalaaspvalleu100105110lysglythrgluaspargvalileileasnvalalaasnglutrptyr115120125glysertrpargseraspvaltrpalaglualatyralaglnalaile130135140proargleuargseralaglyleuserhisthrleumetvalaspala145150155160alaglytrpglyglntyrproalaserilehisgluargglyalaasp165170175valphealaseraspproleulysasnthrmetpheserilehismet180185190tyrglutyralaglyalaaspargalathrilealatyrasnileasp195200205argvalleualagluasnleualavalvalileglyglupheglyhis210215220arghishisaspglyaspvalaspgluaspalaileleualatyrthr225230235240alagluargglnvalglytrpleualatrpsertrptyrglyasnser245250255glyglyvalglutyrleuaspleualagluglyproserglyproleu260265270thrsertrpglylysargilevaltyrglygluasnglyleulysser275280285asp<210>13<211>1473<212>dna<213>bacillushemicellulosilyticus<400>13atgagaaatttcggtaagttaattgtcagttcttgtcttctattcagtttttttcttttt60gcctctgatggtcatagccaaacacattctggtttttatatcgaaggttcaaccctttat120gacgccaacggagagccctttgtaatgagaggtatcaatcatggacatgcctggtataaa180catgattctaacgtcgctataccagctattgctaatcaaggagcaaatacaattcgtatt240gttctgtcagatggtggtcaatgggcaaaagatgatataaacacattaaatcaagtgctc300gatttagcagaggaacatgagatgattgctgttgttgaggttcacgatgcaacaggatct360aattctatggctgacttaaatcgtgctgtcgattattggattgaaatgaaagacgcttta420attggaaaagaagatcgcgtcataattaacattgccaatgaatggtatggagcatgggac480ggacaaggctgggcaaatggctataaggaggttattccacgtttacgaaatgctggcttc540actcatacattaatggtagatgcagctggttggggacaataccctcaatcgattcatgat600tatggtcaagaggtatttaatgctgatcctttagcaaatacgatgttttccatccatatg660tatgaatatgctggcggaaatgcttcaatggtacaatctaatatcgatggtgtcgtcgat720caagggttagctcttgtaataggagaatttgggcatatgcatacggacggagatgttgat780gaagcaacgatattgagctactcgcaacaaagaggagtcggttggctagcttggtcttgg840aaaggcaatgggactcaatgggaatatctagatttatcttatgattggcaaggaacaaac900ttaacttcttggggaaataccattgtccacgggcctaatggattactcgaaacatccatt960ccaagctcgattttccataccgctccaaacaatggagatccccctcctcataacggtaat1020gaaacgatcttatatgatttcgaacatggcactcaaggctggtcaggttcttcacttctt1080ggaggaccttggacgacgaatgaatggagtacaaatggtaaccattcattaaaggccgat1140attttcttatcagctaactccaaacatgaattagcaaaagttgaaaatcgaaatttatca1200ggctactctactttacaagccactgtccgccatgcacattggggaaatgttggtaattta1260acggcgagaatgtatgtaaaaacgggctcaaactatagctggtttaatggtgatcctatc1320ccagtaaactcagcaaatggtacgactgtcactttgcctctttcatctattccaaaccta1380aatgacgtaaaagaaattggcgttgaatttattggagcttcaaatagcaatggacaaacc1440gccatttatttagatcatgtaacaatccaataa1473<210>14<211>1395<212>dna<213>bacillushemicellulosilyticus<400>14cagacccactcgggcttctacatcgagggctcgacgctctacgacgctaacggcgagcct60tttgtcatgcgcggcatcaaccacggccacgcctggtacaagcacgactccaacgtcgct120atccctgctatcgctaaccagggcgctaacaccatccgcatcgtcctcagcgacggtggc180cagtgggccaaggacgacatcaacacgctgaaccaggtcctcgacctggccgaggagcac240gagatgatcgctgtcgtcgaggtccacgacgctaccggctccaacagcatggccgacctc300aaccgcgccgtcgactactggatcgagatgaaggacgccctgatcggcaaggaagaccgc360gtcatcatcaacatcgctaacgagtggtacggcgcttgggacggccagggctgggccaac420ggctacaaggaagtcatccctcgcctgcgcaacgctggcttcacccacaccctcatggtc480gacgctgccggctggggccagtaccctcagagcatccacgactacggccaagaggtcttc540aacgccgaccctctggccaacaccatgttctccatccacatgtacgagtacgctggcggc600aacgcctccatggtccagagcaacatcgacggcgtcgtcgaccagggcctcgctctggtc660atcggcgagttcggccacatgcacacggacggcgacgtcgacgaggctaccatcctgagc720tactcgcagcagcgcggcgtcggctggctggcctggtcgtggaagggcaacggcacccag780tgggagtacctcgacctgagctacgactggcagggcaccaacctcacgtcgtggggcaac840acgatcgtccacggccctaacggcctcctggagacgtccatcccttccagcatctttcac900accgctcctaacaacggcgaccctcctccccacaacggcaacgagacgatcctgtacgac960ttcgagcacggcacgcagggctggtcgggctcgtccctgctgggcggcccttggaccacc1020aacgagtggtcgaccaacggcaaccactccctcaaggccgacatcttcctgtccgccaac1080agcaagcacgagctcgccaaggtcgagaaccgcaacctcagcggctactcgacgctgcag1140gctaccgtccgccacgctcactggggcaacgtcggcaacctgacggctcgcatgtacgtc1200aagacgggcagcaactactcgtggttcaacggcgaccccatccctgtcaactcggctaac1260ggcaccaccgtcaccctccctctgagctcgatccccaacctcaacgacgtcaaggagatc1320ggcgtcgagttcatcggcgctagcaacagcaacggccagaccgccatctacctggaccac1380gtcacgatccagtaa1395<210>15<211>490<212>prt<213>bacillushemicellulosilyticus<400>15metargasnpheglylysleuilevalsersercysleuleupheser151015phepheleuphealaseraspglyhisserglnthrhisserglyphe202530tyrilegluglyserthrleutyraspalaasnglyglupropheval354045metargglyileasnhisglyhisalatrptyrlyshisaspserasn505560valalaileproalailealaasnglnglyalaasnthrileargile65707580valleuseraspglyglyglntrpalalysaspaspileasnthrleu859095asnglnvalleuaspleualaglugluhisglumetilealavalval100105110gluvalhisaspalathrglyserasnsermetalaaspleuasnarg115120125alavalasptyrtrpileglumetlysaspalaleuileglylysglu130135140aspargvalileileasnilealaasnglutrptyrglyalatrpasp145150155160glyglnglytrpalaasnglytyrlysgluvalileproargleuarg165170175asnalaglyphethrhisthrleumetvalaspalaalaglytrpgly180185190glntyrproglnserilehisasptyrglyglngluvalpheasnala195200205aspproleualaasnthrmetpheserilehismettyrglutyrala210215220glyglyasnalasermetvalglnserasnileaspglyvalvalasp225230235240glnglyleualaleuvalileglyglupheglyhismethisthrasp245250255glyaspvalaspglualathrileleusertyrserglnglnarggly260265270valglytrpleualatrpsertrplysglyasnglythrglntrpglu275280285tyrleuaspleusertyrasptrpglnglythrasnleuthrsertrp290295300glyasnthrilevalhisglyproasnglyleuleugluthrserile305310315320proserserilephehisthralaproasnasnglyasppropropro325330335hisasnglyasngluthrileleutyraspphegluhisglythrgln340345350glytrpserglyserserleuleuglyglyprotrpthrthrasnglu355360365trpserthrasnglyasnhisserleulysalaaspilepheleuser370375380alaasnserlyshisgluleualalysvalgluasnargasnleuser385390395400glytyrserthrleuglnalathrvalarghisalahistrpglyasn405410415valglyasnleuthralaargmettyrvallysthrglyserasntyr420425430sertrppheasnglyaspproileprovalasnseralaasnglythr435440445thrvalthrleuproleuserserileproasnleuasnaspvallys450455460gluileglyvalglupheileglyalaserasnserasnglyglnthr465470475480alailetyrleuasphisvalthrilegln485490<210>16<211>464<212>prt<213>bacillushemicellulosilyticus<400>16glnthrhisserglyphetyrilegluglyserthrleutyraspala151015asnglygluprophevalmetargglyileasnhisglyhisalatrp202530tyrlyshisaspserasnvalalaileproalailealaasnglngly354045alaasnthrileargilevalleuseraspglyglyglntrpalalys505560aspaspileasnthrleuasnglnvalleuaspleualaglugluhis65707580glumetilealavalvalgluvalhisaspalathrglyserasnser859095metalaaspleuasnargalavalasptyrtrpileglumetlysasp100105110alaleuileglylysgluaspargvalileileasnilealaasnglu115120125trptyrglyalatrpaspglyglnglytrpalaasnglytyrlysglu130135140valileproargleuargasnalaglyphethrhisthrleumetval145150155160aspalaalaglytrpglyglntyrproglnserilehisasptyrgly165170175glngluvalpheasnalaaspproleualaasnthrmetpheserile180185190hismettyrglutyralaglyglyasnalasermetvalglnserasn195200205ileaspglyvalvalaspglnglyleualaleuvalileglygluphe210215220glyhismethisthraspglyaspvalaspglualathrileleuser225230235240tyrserglnglnargglyvalglytrpleualatrpsertrplysgly245250255asnglythrglntrpglutyrleuaspleusertyrasptrpglngly260265270thrasnleuthrsertrpglyasnthrilevalhisglyproasngly275280285leuleugluthrserileproserserilephehisthralaproasn290295300asnglyaspproproprohisasnglyasngluthrileleutyrasp305310315320phegluhisglythrglnglytrpserglyserserleuleuglygly325330335protrpthrthrasnglutrpserthrasnglyasnhisserleulys340345350alaaspilepheleuseralaasnserlyshisgluleualalysval355360365gluasnargasnleuserglytyrserthrleuglnalathrvalarg370375380hisalahistrpglyasnvalglyasnleuthralaargmettyrval385390395400lysthrglyserasntyrsertrppheasnglyaspproileproval405410415asnseralaasnglythrthrvalthrleuproleuserserilepro420425430asnleuasnaspvallysgluileglyvalglupheileglyalaser435440445asnserasnglyglnthralailetyrleuasphisvalthrilegln450455460<210>17<211>1449<212>dna<213>virgibacillussoli<400>17atgttattctctacttcactgtttacttctacttcaaaagcgaatgcagcaagcgggttt60tatgtaaacggaaacacactctatgacgcaacaggtaccccttttgtgataagaggaatc120aatcatgctcactcttggtttaaagacgacacagcaaccgcaatacctgccattgcagca180actggggcgaatactattagaatcgtattatcggatggcagccaatatagtcgggatgat240attgatggcgtgaggaatctaatatcattggctgaggaaaataatctaattgctatgtta300gaggtccacgatgctactggaaaagatgatatcagctcattagatagtgcggcagattat360tggattagtataaaagaagcacttatcggcaaggaagacaaagtcctaataaacatcgca420aatgaatggtacggtacttgggatggggctagttgggcggatggctacaaacaagtgatt480cccaaattaagaaatgcaggacttaaccacacactaatagtagactctgctggctggggg540caatttccggagtccattcacaattacggaaaagaagtattcaatgctgaccccctacaa600aatacaatgttctctattcatatgtatgaatatgctggtggggacgcttctactgtcaaa660gcaaatattgacggtgtattaaatcaaggtctagccgtaatcattggagaatttggacat720aggcatacagacggagatgtagatgaagcaacaattatgaattattcccaagagaaaaat780gttggctggctcgcatggtcgtggaaaggtaatggcatggaatgggattatttagactta840tcctatgattgggccggaaataacctaaccgactggggaaataccattgtaaatagtaca900aacggcttaaaagctacatctgaaataagtccagtatttggagatggagatgacggtgta960ggcgacggtggtcctggggattctaacggaactgaaactacgctttataacttcgaaacc1020gggacagaaggatggagcggcgaaaatatagaaactggaccttggtcagtgaatgagtgg1080gcagcaaaaggtaaccactctttaaaagctgatgttaatttgggtgataactctgaacat1140tatctatacctaactcaaaacctaaattttagcggaaagtcacaactcacagcgactgta1200aagcatgctgattggggaaacttcggggatgaaataaatgcaaagttatatgtaaaaaca1260gaatcagattggcaatggtttgatggaggaattgaaaagatcaattcttcaattggaact1320attataaccttagatttatcatcgctctcaaacccaagtgatattaaagaagttggtgtt1380cagtttacgggttcttcaaatagttatggcctaacagctttatatgttgataacgttacc1440attaaataa1449<210>18<211>1401<212>dna<213>virgibacillussoli<400>18gcctcgggcttctacgtcaacggcaacactctctacgacgccacgggcaccccatttgtc60atccgcggcatcaaccacgctcactcgtggttcaaggacgacactgccaccgctatccct120gctatcgctgctacgggcgccaacacgatccgcatcgtcctcagcgacggctcgcagtac180tcccgcgacgacatcgacggcgtccgcaacctcatctccctggccgaggagaacaacctc240atcgccatgctggaggtccacgacgctaccggcaaggacgacatcagctcgctggacagc300gccgccgactactggatctcgatcaaggaagccctcatcggcaaggaagacaaggtcctg360atcaacatcgccaacgagtggtacggcacctgggacggcgctagctgggctgacggctac420aagcaggtcatccctaagctccgcaacgccggcctcaaccacacgctcatcgtcgactcg480gctggctggggccagttcccggagagcatccacaactacggcaaggaagtcttcaacgcc540gaccccctgcagaacacgatgttctcgatccacatgtacgagtacgccggcggcgacgct600tccacggtcaaggccaacatcgacggcgtcctcaaccagggcctggctgtcatcatcggc660gagtttggccaccgccacaccgacggcgacgtcgacgaggccaccatcatgaactacagc720caggagaagaacgtcggctggctggcttggagctggaagggcaacggcatggagtgggac780tacctcgacctgagctacgactgggccggcaacaacctcaccgactggggcaacacgatc840gtcaactcgaccaacggcctgaaggccacctcggagatcagccctgtctttggcgacggc900gacgacggcgtcggcgacggtggccccggcgacagcaacggcaccgagacgacgctgtac960aactttgagacgggcaccgagggctggagcggcgagaacatcgagacgggcccttggtcg1020gtcaacgagtgggctgccaagggcaaccactccctcaaggccgacgtcaacctgggcgac1080aacagcgagcactacctctacctgacgcagaacctcaacttctccggcaagtcgcagctg1140acggctaccgtcaagcacgctgactggggcaacttcggcgacgagatcaacgccaagctc1200tacgtcaagaccgagagcgactggcagtggttcgacggtggcatcgagaagatcaactcc1260agcatcggcaccatcatcacgctcgacctgtcgtccctgtcgaacccgtccgacatcaag1320gaagtcggcgtccagttcactggctcgtctaactcttacggcctcactgctctttacgtc1380gacaacgtcactatcaagtag1401<210>19<211>482<212>prt<213>virgibacillussoli<400>19metleupheserthrserleuphethrserthrserlysalaasnala151015alaserglyphetyrvalasnglyasnthrleutyraspalathrgly202530thrprophevalileargglyileasnhisalahissertrpphelys354045aspaspthralathralaileproalailealaalathrglyalaasn505560thrileargilevalleuseraspglyserglntyrserargaspasp65707580ileaspglyvalargasnleuileserleualaglugluasnasnleu859095ilealametleugluvalhisaspalathrglylysaspaspileser100105110serleuaspseralaalaasptyrtrpileserilelysglualaleu115120125ileglylysgluasplysvalleuileasnilealaasnglutrptyr130135140glythrtrpaspglyalasertrpalaaspglytyrlysglnvalile145150155160prolysleuargasnalaglyleuasnhisthrleuilevalaspser165170175alaglytrpglyglnpheprogluserilehisasntyrglylysglu180185190valpheasnalaaspproleuglnasnthrmetpheserilehismet195200205tyrglutyralaglyglyaspalaserthrvallysalaasnileasp210215220glyvalleuasnglnglyleualavalileileglyglupheglyhis225230235240arghisthraspglyaspvalaspglualathrilemetasntyrser245250255glnglulysasnvalglytrpleualatrpsertrplysglyasngly260265270metglutrpasptyrleuaspleusertyrasptrpalaglyasnasn275280285leuthrasptrpglyasnthrilevalasnserthrasnglyleulys290295300alathrsergluileserprovalpheglyaspglyaspaspglyval305310315320glyaspglyglyproglyaspserasnglythrgluthrthrleutyr325330335asnphegluthrglythrgluglytrpserglygluasnilegluthr340345350glyprotrpservalasnglutrpalaalalysglyasnhisserleu355360365lysalaaspvalasnleuglyaspasnsergluhistyrleutyrleu370375380thrglnasnleuasnpheserglylysserglnleuthralathrval385390395400lyshisalaasptrpglyasnpheglyaspgluileasnalalysleu405410415tyrvallysthrgluserasptrpglntrppheaspglyglyileglu420425430lysileasnserserileglythrileilethrleuaspleuserser435440445leuserasnproseraspilelysgluvalglyvalglnphethrgly450455460serserasnsertyrglyleuthralaleutyrvalaspasnvalthr465470475480ilelys<210>20<211>466<212>prt<213>virgibacillussoli<400>20alaserglyphetyrvalasnglyasnthrleutyraspalathrgly151015thrprophevalileargglyileasnhisalahissertrpphelys202530aspaspthralathralaileproalailealaalathrglyalaasn354045thrileargilevalleuseraspglyserglntyrserargaspasp505560ileaspglyvalargasnleuileserleualaglugluasnasnleu65707580ilealametleugluvalhisaspalathrglylysaspaspileser859095serleuaspseralaalaasptyrtrpileserilelysglualaleu100105110ileglylysgluasplysvalleuileasnilealaasnglutrptyr115120125glythrtrpaspglyalasertrpalaaspglytyrlysglnvalile130135140prolysleuargasnalaglyleuasnhisthrleuilevalaspser145150155160alaglytrpglyglnpheprogluserilehisasntyrglylysglu165170175valpheasnalaaspproleuglnasnthrmetpheserilehismet180185190tyrglutyralaglyglyaspalaserthrvallysalaasnileasp195200205glyvalleuasnglnglyleualavalileileglyglupheglyhis210215220arghisthraspglyaspvalaspglualathrilemetasntyrser225230235240glnglulysasnvalglytrpleualatrpsertrplysglyasngly245250255metglutrpasptyrleuaspleusertyrasptrpalaglyasnasn260265270leuthrasptrpglyasnthrilevalasnserthrasnglyleulys275280285alathrsergluileserprovalpheglyaspglyaspaspglyval290295300glyaspglyglyproglyaspserasnglythrgluthrthrleutyr305310315320asnphegluthrglythrgluglytrpserglygluasnilegluthr325330335glyprotrpservalasnglutrpalaalalysglyasnhisserleu340345350lysalaaspvalasnleuglyaspasnsergluhistyrleutyrleu355360365thrglnasnleuasnpheserglylysserglnleuthralathrval370375380lyshisalaasptrpglyasnpheglyaspgluileasnalalysleu385390395400tyrvallysthrgluserasptrpglntrppheaspglyglyileglu405410415lysileasnserserileglythrileilethrleuaspleuserser420425430leuserasnproseraspilelysgluvalglyvalglnphethrgly435440445serserasnsertyrglyleuthralaleutyrvalaspasnvalthr450455460ilelys465<210>21<211>60<212>dna<213>artificialsequence<220><223>primer<400>21agtcaatcgcgacaagcgccagacccactcgggcttctacatcgagggctcgacgctcta60<210>22<211>46<212>dna<213>artificialsequence<220><223>primer<400>22cgcgccggatccttactggatcgtgacgtggtccaggtagatggcg46<210>23<211>60<212>dna<213>artificialsequence<220><223>primer<400>23agtcaatcgcgacaagcgccagaacggcttccacgtctccggcacggagctcctggacaa60<210>24<211>51<212>dna<213>artificialsequence<220><223>primer<400>24cgcgccggatccttagtcgctcttcaggccgttctcgccgtagacgatgcg51<210>25<211>1846<212>dna<213>bacilluspumilus<400>25atgaaaaaatgggttcaacgggtggcttgttttatgctgctgatcactttatgggcgggt60tggttcactctgaccgtaaaggcctcctcctatgtgcaaacatctggtacacattttgta120ttgaacaaccacccattttactttgctggcacaaataattattatttccattacaaatca180aaaaagatggtagatgctgtttttgacgatatgaaggcaatggatttaaaggttattcgt240atttggggatttcacgatggtacccctcaagaaaactcagtcttacaatctcgtccaggt300gtttatgaagaatccggttttcaaaaactagactatgcgatttataaagcagggcaggaa360ggaatcaagctggtcataccgctcgtgaacaattgggatgactttggcgggatgaatcaa420tatgtgaagtggtttcaggcaggatcacatgatcacttttatacagattctcggattaaa480acagcttacaaaaactatgtgcgctatgtattagagagaaccaatacgtactcaggtgtt540caatataaagatgaccctgctattatgacatgggagctcgccaatgagccgcgcgctcag600tcagacccttcgggagatatactagtaaactgggcagatgaaatgagtgcatggatcaaa660tcaattgactcgaatcatcttgttgctgtaggagacgaagggttctttcgcatgacaggt720catgatgattggttttacagtggaggagaaggtgttgattgggatcgtttgactgctctc780cctcatattgattatggaacctatcatttatacccggatcactggaatcagtctgctgca840tggggagtgaaatggatcaaagatcatatcacccgaggaaacgcaatcggaaaacctgtt900gtattagaagagtttggctatcaaaatcaagcagcccgtcctgatgtatatgatagctgg960ctgaagacaattgaacagctcggaggcgcaggtagccaattttggattttaacaagcatt1020caagacgatgattccctctacccggattatgatggttttcgagttttaaaggagagccgg1080gaggcaggaattattcgtgaacacgccaaaagaatgaatgaaaagaactgatgaagaatg1140cctgtttataaggaacttcatttgcataaaaaaattggatatggtatagtttttatggaa1200atgctaacgattaccgagacaagagtggggaaacccgctcttttgtattgaacaggcaat1260ttttgtctcgacattattcatccgttttctgctccccctgctcacaataaagcagggttt1320ttatgcagaatgattgataagagcgtttatcgaaagcacaaggaggaagagaatgagcaa1380aaaagtagtggatatcgtaagcgacatggtgcagccaattttagatggcttacagcttga1440actcgttgatgttgaatttgtcaaagagggtcaaaactggttccttcgcgtatttattga1500ctctgataaaggcgtcgatatcgaggagtgtgccaaagtgagcgaagccttgagcgaaaa1560gcttgatgaggcagatccaattagccaaaactactttcttgaagtgtcctctcctggagc1620ggagcgcccattaaagaaaaaagctgattttgaaaaagcacttggaaaaaatgttttcat1680gaaaacatacgaaccaattgatggtgaaaaggcatttgaaggtgagcttacaagctttga1740tggtgagattgcaacagtgacagtgaagatcaagacaagaaagaaagagatcaatattcc1800atacgaaaaaattgctaacgcaagattagcagtttcgttcaattaa1846<210>26<211>376<212>prt<213>bacilluspumilus<400>26metlyslystrpvalglnargvalalacysphemetleuleuilethr151015leutrpalaglytrpphethrleuthrvallysalasersertyrval202530glnthrserglythrhisphevalleuasnasnhisprophetyrphe354045alaglythrasnasntyrtyrphehistyrlysserlyslysmetval505560aspalavalpheaspaspmetlysalametaspleulysvalilearg65707580iletrpglyphehisaspglythrproglngluasnservalleugln859095serargproglyvaltyrglugluserglypheglnlysleuasptyr100105110alailetyrlysalaglyglngluglyilelysleuvalileproleu115120125valasnasntrpaspasppheglyglymetasnglntyrvallystrp130135140pheglnalaglyserhisasphisphetyrthraspserargilelys145150155160thralatyrlysasntyrvalargtyrvalleugluargthrasnthr165170175tyrserglyvalglntyrlysaspaspproalailemetthrtrpglu180185190leualaasngluproargalaglnseraspproserglyaspileleu195200205valasntrpalaaspglumetseralatrpilelysserileaspser210215220asnhisleuvalalavalglyaspgluglyphepheargmetthrgly225230235240hisaspasptrpphetyrserglyglygluglyvalasptrpasparg245250255leuthralaleuprohisileasptyrglythrtyrhisleutyrpro260265270asphistrpasnglnseralaalatrpglyvallystrpilelysasp275280285hisilethrargglyasnalaileglylysprovalvalleugluglu290295300pheglytyrglnasnglnalaalaargproaspvaltyraspsertrp305310315320leulysthrilegluglnleuglyglyalaglyserglnphetrpile325330335leuthrserileglnaspaspaspserleutyrproasptyraspgly340345350pheargvalleulysgluserargglualaglyileilearggluhis355360365alalysargmetasnglulysasn370375<210>27<211>1083<212>dna<213>bacillusamyloliquefaciens<400>27atgctcaaaaagttcgcagtctgtctgtctatcattttattactcatctcagccgcccgt60ccgatatcggctcacaccgtttaccctgtcaatcccaatgcccagcagacgacaaaagac120gtcatgaactggctggcgcatttgcccaaccgttcagaaaacagggtcatgtccggtgca180ttcggcgggtacagcgatgtcaccttttcaatgacggaggaaaaccgcttgaaaaacgcg240acgggacagtctcccgccatctacggctgtgattatgggagagggtggctggaaacatcg300gatatcaccgattctatcgactacagctgcaacagcagcctcatttcgtactggaaaagc360ggcggcctccctcaggtcagcctgcatctcgcaaatccggcctttccatcaggacactat420aaaacggccatttcaaacagccagtataaaaatatcctgaacccttcaactgttgaagga480cggcggcttgaggccttgctcagcaaaatcgccgacggccttactcagctgaaaaatcaa540ggcgtcaccgttctgttcaggccgctgcatgagatgaacggtgaatggttctggtggggg600ctgacaggctacaaccaaaaagacactgagagaatctcgctgtacaaagagctttacaag660aagatataccgctatatgacagagacaagaggattggataatcttttgtgggtgtattcg720cctgatgccaacagagacttcaaaacagacttctacccaggctcatcttatgtggatatt780accggactggatgcttacttcaccgacccgtatgcgatatcaggctatgatgaaatgctg840tctctgaaaaaaccgtttgcctttgccgagaccggtccgtccggtaatatcggaagcttt900gattacgctgtttttatcaatgcgatcaggcaaaagtatcccgagacaacctactttttg960acatgggatgaacaattaagcccggcagccaatcaaggcgcgcaaagcctttatcaaaac1020agctggacgctgaacaagggcgaaatgtggaatggcggaaccttgacgccgatcgcggaa1080taa1083<210>28<211>360<212>prt<213>bacillusamyloliquefaciens<400>28metleulyslysphealavalcysleuserileileleuleuleuile151015seralaalaargproileseralahisthrvaltyrprovalasnpro202530asnalaglnglnthrthrlysaspvalmetasntrpleualahisleu354045proasnargsergluasnargvalmetserglyalapheglyglytyr505560seraspvalthrphesermetthrglugluasnargleulysasnala65707580thrglyglnserproalailetyrglycysasptyrglyargglytrp859095leugluthrseraspilethraspserileasptyrsercysasnser100105110serleuilesertyrtrplysserglyglyleuproglnvalserleu115120125hisleualaasnproalapheproserglyhistyrlysthralaile130135140serasnserglntyrlysasnileleuasnproserthrvalglugly145150155160argargleuglualaleuleuserlysilealaaspglyleuthrgln165170175leulysasnglnglyvalthrvalleupheargproleuhisglumet180185190asnglyglutrpphetrptrpglyleuthrglytyrasnglnlysasp195200205thrgluargileserleutyrlysgluleutyrlyslysiletyrarg210215220tyrmetthrgluthrargglyleuaspasnleuleutrpvaltyrser225230235240proaspalaasnargaspphelysthraspphetyrproglyserser245250255tyrvalaspilethrglyleuaspalatyrphethraspprotyrala260265270ileserglytyraspglumetleuserleulyslysprophealaphe275280285alagluthrglyproserglyasnileglyserpheasptyralaval290295300pheileasnalaileargglnlystyrprogluthrthrtyrpheleu305310315320thrtrpaspgluglnleuserproalaalaasnglnglyalaglnser325330335leutyrglnasnsertrpthrleuasnlysglyglumettrpasngly340345350glythrleuthrproilealaglu355360<210>29<211>1494<212>dna<213>amphibacillusxylanus<400>29gtgaagttaactaaactaaaactattgagtagtgtattttttgttgtattaactgtgtta60atgttgtttgtccctgggaatattgtgaatgtaaaagctgctaacggcttttatgtaagc120gattccaatctgtatgatgcaaatggaaatcaatttgttatgcgtggggttaatcatgcc180cattcatggtataaggacacgtataccgaggcaattcctgcaattgcggctacaggagcg240aatactatccgaattgtattatctgatggagggcaataccaaaaagatgatataaacata300gtcagaaatttgattgaaaccgcagaagccaataatttagtcgctgtacttgaggttcat360gatgctactgggtcggattcattatcggatttgaaccgggctgtagattattggattgaa420attaaagatgcgttaattggtaaagaagatacggtgatcataaacattgtcaatgaatgg480tatggcacttgggatggtcgtctctgggcagatggttataaacaggcgataccgagatta540agagatgctggattaacacatacgttgatgattgatgcagcaggttgggggcaatttcct600agctcgatccatcaatatggtagagaagtatttaatgcagatcgtttagggaatacaatg660ttttcgattcatatgtatgaatatgctggcggtgatgatcaaatggttagagataatatt720aacggtgtgatcaatcaagacttagctctagtgattggtgaatttggtcattatcacaca780gatggcgatgttgatgaagatacgattttgagttacgcggagcagacaggtgttggttgg840ttagcatggtcatggaaaggcaatggaactgagtgggagtatcttgatctatcaaatgat900tggggaggaaattatttaacatcttggggtgacaggattgtaaatggagcaaatggatta960agagaaacgagtcaaattgcttctgttttttcaggaaacaatggcgggactcctggaaat1020ggtgaggaagagactcctggtgatgtaagtcatttcgcaaacttcgagaatggtactgaa1080ggttgggaagcaagcaatgtatctggtggaccttgggcaacaaatgaatggagtgctagt1140ggttcatatgctttaaaagccgatgcgcaattagcatctggaagagaacactatttatat1200cgaatcggtccctttaatttatctgggtcaacattaaacgcaacggtaaggggtgctaat1260tgggggaattatggatctggtatcgacgtgaagctatacgttaagtacggagatggctgg1320acgtggagagatagtggtgtacagacaattagagcgggagaatctattgatctatcacta1380gatttatcaaatgttgatcgctcaaacattagagaagttggtatccagtttattggtgga1440aatcattcatctggaaaaaccgctttttatgttgatcatgtttattcacattag1494<210>30<211>497<212>prt<213>amphibacillusxylanus<400>30vallysleuthrlysleulysleuleuserservalphephevalval151015leuthrvalleumetleuphevalproglyasnilevalasnvallys202530alaalaasnglyphetyrvalseraspserasnleutyraspalaasn354045glyasnglnphevalmetargglyvalasnhisalahissertrptyr505560lysaspthrtyrthrglualaileproalailealaalathrglyala65707580asnthrileargilevalleuseraspglyglyglntyrglnlysasp859095aspileasnilevalargasnleuilegluthralaglualaasnasn100105110leuvalalavalleugluvalhisaspalathrglyseraspserleu115120125seraspleuasnargalavalasptyrtrpilegluilelysaspala130135140leuileglylysgluaspthrvalileileasnilevalasnglutrp145150155160tyrglythrtrpaspglyargleutrpalaaspglytyrlysglnala165170175ileproargleuargaspalaglyleuthrhisthrleumetileasp180185190alaalaglytrpglyglnpheproserserilehisglntyrglyarg195200205gluvalpheasnalaaspargleuglyasnthrmetpheserilehis210215220mettyrglutyralaglyglyaspaspglnmetvalargaspasnile225230235240asnglyvalileasnglnaspleualaleuvalileglygluphegly245250255histyrhisthraspglyaspvalaspgluaspthrileleusertyr260265270alagluglnthrglyvalglytrpleualatrpsertrplysglyasn275280285glythrglutrpglutyrleuaspleuserasnasptrpglyglyasn290295300tyrleuthrsertrpglyaspargilevalasnglyalaasnglyleu305310315320arggluthrserglnilealaservalpheserglyasnasnglygly325330335thrproglyasnglygluglugluthrproglyaspvalserhisphe340345350alaasnphegluasnglythrgluglytrpglualaserasnvalser355360365glyglyprotrpalathrasnglutrpseralaserglysertyrala370375380leulysalaaspalaglnleualaserglyarggluhistyrleutyr385390395400argileglypropheasnleuserglyserthrleuasnalathrval405410415argglyalaasntrpglyasntyrglyserglyileaspvallysleu420425430tyrvallystyrglyaspglytrpthrtrpargaspserglyvalgln435440445thrileargalaglygluserileaspleuserleuaspleuserasn450455460valaspargserasnilearggluvalglyileglnpheileglygly465470475480asnhisserserglylysthralaphetyrvalasphisvaltyrser485490495his<210>31<211>1767<212>dna<213>paenibacilluspolymyxa<400>31atgaaaaaactactgtcttgtctcatttcgctgtcaatgcttgtgtatatcttaccgaca60atgatagtgtccgctaacaatgatggcgtaacgaaccttgctcttgattcaacacctagt120gcgcaaagtgatattatttctgatgctgtctacaaaatcacagctcagcattcaggaaaa180agccttgaggttgaaggcggttctaaagatgacggcgcgaatgttcaacaatggacagat240aacgggaaagaacagcagaaatggagagttgtggacgtcggtggcggatattacaagctc300atcagtcaatctagcggaaaagcactggatgtggcaggtggtaatacacatgatggtgcc360aatgtgcaacagtggacggacaacggaaatgctcagcaaaagtggaagatcatcgatgta420ggaggaggctattataagttgatctcacaaagctctggaaaggcactcgacgtcgttggt480ggttatacgcacgacggggccaatgtgcagcaatgggcagacaatggatctgctcaacag540cgctggcgtttcacacaaattgatacaaccacggatacgacgccgccaacagcaccaacg600aatttacaatcatcatcgaaaacaagtacctctgtaacattgacttggaccacaagcatt660gataatgtaggtgtgacaggctatgtcatttataatggaacagatttggtcgggacttct720acaactacatcttatattgttacaggattaacagcgaacacttcctataacttcactgtc780aaagcgaaggatgccgctgggaatatttcagaaccatcaaatgtcttgaaagtcacaacg840agttcagattcttctcaaaacacaggtttttatgtgaagggcacaacattatatgatgga900aacggtaatccatttgtgatgagaggaatcaatcatgcatacacatggtataaagggcaa960gaatcagtagcaattcctgcgattgcgaaaacgggtgcaaacaccatccggattgtctta1020tctgacggacagcagtggacaaaagatgatttaagcgcgcttcaaaatttgattacactc1080agtgagcaaaacaaacttgtagtgattttagaggtgcacgacggtactggcaatgacaat1140gccgcagttttaaataaaattgctgattattggattgaaatgaagtcagctttaattggg1200aaggaaaatacagttattttaaacatcgcaaatgaatggtttggtacatgggatggaaac1260ggctgggcgcagggctacaaatcagtcataccaaagctgcgaaatgcgggcatcaaaaac1320acgattatggtggatgcggctggatggggacaatatccaaaatcgatttttgattacgga1380acgcaagtgttcgatgcagatccgctcaagaatacgatgttttccattcatatgtatgaa1440tacgcaggcggcaacgcagaaacagtgaaaagtaatatcgacaacgtcctgaataaaaat1500cttgcactcatcattggagaatttggaattaaacatacaaacggagatgttgatgaagca1560acgatcatgtcatacgcacagcaaaaaggtgttgggtatcttggctggtcatggaaagga1620aatggttcaggtcttgaatatttagatatgagtaacgattgggctggcagcagttataca1680gagcaaggacatgccattatcgaaggaccaaatggcattcgtgcaacatcaaaattatca1740accatttacagcaatgggaaacaataa1767<210>32<211>588<212>prt<213>paenibacilluspolymyxa<400>32metlyslysleuleusercysleuileserleusermetleuvaltyr151015ileleuprothrmetilevalseralaasnasnaspglyvalthrasn202530leualaleuaspserthrproseralaglnseraspileileserasp354045alavaltyrlysilethralaglnhisserglylysserleugluval505560gluglyglyserlysaspaspglyalaasnvalglnglntrpthrasp65707580asnglylysgluglnglnlystrpargvalvalaspvalglyglygly859095tyrtyrlysleuileserglnserserglylysalaleuaspvalala100105110glyglyasnthrhisaspglyalaasnvalglnglntrpthraspasn115120125glyasnalaglnglnlystrplysileileaspvalglyglyglytyr130135140tyrlysleuileserglnserserglylysalaleuaspvalvalgly145150155160glytyrthrhisaspglyalaasnvalglnglntrpalaaspasngly165170175seralaglnglnargtrpargphethrglnileaspthrthrthrasp180185190thrthrproprothralaprothrasnleuglnserserserlysthr195200205serthrservalthrleuthrtrpthrthrserileaspasnvalgly210215220valthrglytyrvaliletyrasnglythraspleuvalglythrser225230235240thrthrthrsertyrilevalthrglyleuthralaasnthrsertyr245250255asnphethrvallysalalysaspalaalaglyasnileserglupro260265270serasnvalleulysvalthrthrserseraspserserglnasnthr275280285glyphetyrvallysglythrthrleutyraspglyasnglyasnpro290295300phevalmetargglyileasnhisalatyrthrtrptyrlysglygln305310315320gluservalalaileproalailealalysthrglyalaasnthrile325330335argilevalleuseraspglyglnglntrpthrlysaspaspleuser340345350alaleuglnasnleuilethrleusergluglnasnlysleuvalval355360365ileleugluvalhisaspglythrglyasnaspasnalaalavalleu370375380asnlysilealaasptyrtrpileglumetlysseralaleuilegly385390395400lysgluasnthrvalileleuasnilealaasnglutrppheglythr405410415trpaspglyasnglytrpalaglnglytyrlysservalileprolys420425430leuargasnalaglyilelysasnthrilemetvalaspalaalagly435440445trpglyglntyrprolysserilepheasptyrglythrglnvalphe450455460aspalaaspproleulysasnthrmetpheserilehismettyrglu465470475480tyralaglyglyasnalagluthrvallysserasnileaspasnval485490495leuasnlysasnleualaleuileileglyglupheglyilelyshis500505510thrasnglyaspvalaspglualathrilemetsertyralaglngln515520525lysglyvalglytyrleuglytrpsertrplysglyasnglysergly530535540leuglutyrleuaspmetserasnasptrpalaglysersertyrthr545550555560gluglnglyhisalaileilegluglyproasnglyileargalathr565570575serlysleuserthriletyrserasnglylysgln580585<210>33<211>1470<212>dna<213>bacillushemicellulosilyticus<400>33atggatatattaagaaagtgtgtacttgtactattggccttactattgttgttacctacg60acatcaacggcattttctgaaagcgcttctactaatgagagagtgctaaatttatctgat120ccgaatgcgacacgctatacgaaggaattgtttgcgtttcttcaagacgtgagtggtgag180caagtgttgttcgggcaacagcatgcaacagatgaagggttgactctgacaggtgaagga240aatcgaattggttcaactgagtcggaggtgaagaatgcagtaggtgattatccagctgtt300tttgggtgggatacgaacagcttggatggtcgtgaaaagccaggtacagatgtggaaagt360caagagcaacgaattttaaatacagcagaatcgatgaaagtggcacatgaattaggaggg420atcatcacattaagtatgcatccggataactttgttaccggtcattactatggcgatacg480gatggtaacgtcgttcaagaaatattgccaggtggctccaagcacaatgaatttaacgct540tggctagataatattgctgccctagcacatgaattagttgatgataatggagagcctatt600ccggttatcttccgtccattccatgagcaaacaggttcgtggttttggtggggtgcgagc660acaacaactcctgagcaatacaaagcgatttttcgatatacagtcgaatacttaagagat720gcaaagggtgttcataactttttatatggattctcccctggtgcgggtcctgctggcgat780ctagatcgatatttagaaacgtacccaggtgataattatgtcgatatcttaggtattgat840aattatgatagtaagtcaaatgcggggtcagacgcttggttatctggaatggtaaaagat900ttagcgatgatctcgaaattagcagaggaaagagggaaggtatcagcctttactgaattt960ggatacagcgctgaagggatgagtcaaacgggtgatgcgttagattggtatacacgtgtg1020ttaaatgcgataaaagcagatgaagatgcgcgaaacatatcctacatgctaacgtgggct1080aactttgggtggcctaataatatatttgttccgtatcgtgatgtgaatggggatttaggt1140ggagatcatgagttattacctgactttgtacagttttatgaagatgaatactcagcattt1200cgtgaagatataaatgaaagtgtttacaatcgtaatgagagttatattgttgcggatcat1260gagccatttatgtatgttgtttcccctacgacaggtacatatataacaggctcgtctgtt1320gtcttacgagcgaaagtagttaacgatgaggatccgtccgttacgtatcaagtggcgggt1380tctgaagaagtctatgagatgactttagatgaaaatgggtattactctgctgattatatt1440cctactgctcctaagaatggagctctgtag1470<210>34<211>489<212>prt<213>bacillushemicellulosilyticus<400>34metaspileleuarglyscysvalleuvalleuleualaleuleuleu151015leuleuprothrthrserthralaphesergluseralaserthrasn202530gluargvalleuasnleuseraspproasnalathrargtyrthrlys354045gluleuphealapheleuglnaspvalserglygluglnvalleuphe505560glyglnglnhisalathraspgluglyleuthrleuthrglyglugly65707580asnargileglyserthrglusergluvallysasnalavalglyasp859095tyrproalavalpheglytrpaspthrasnserleuaspglyargglu100105110lysproglythraspvalgluserglngluglnargileleuasnthr115120125alaglusermetlysvalalahisgluleuglyglyileilethrleu130135140sermethisproaspasnphevalthrglyhistyrtyrglyaspthr145150155160aspglyasnvalvalglngluileleuproglyglyserlyshisasn165170175glupheasnalatrpleuaspasnilealaalaleualahisgluleu180185190valaspaspasnglygluproileprovalilepheargprophehis195200205gluglnthrglysertrpphetrptrpglyalaserthrthrthrpro210215220gluglntyrlysalailepheargtyrthrvalglutyrleuargasp225230235240alalysglyvalhisasnpheleutyrglypheserproglyalagly245250255proalaglyaspleuaspargtyrleugluthrtyrproglyaspasn260265270tyrvalaspileleuglyileaspasntyraspserlysserasnala275280285glyseraspalatrpleuserglymetvallysaspleualametile290295300serlysleualaglugluargglylysvalseralaphethrgluphe305310315320glytyrseralagluglymetserglnthrglyaspalaleuasptrp325330335tyrthrargvalleuasnalailelysalaaspgluaspalaargasn340345350ilesertyrmetleuthrtrpalaasnpheglytrpproasnasnile355360365phevalprotyrargaspvalasnglyaspleuglyglyasphisglu370375380leuleuproaspphevalglnphetyrgluaspglutyrseralaphe385390395400arggluaspileasngluservaltyrasnargasnglusertyrile405410415valalaasphisgluprophemettyrvalvalserprothrthrgly420425430thrtyrilethrglyserservalvalleuargalalysvalvalasn435440445aspgluaspproservalthrtyrglnvalalaglyserglugluval450455460tyrglumetthrleuaspgluasnglytyrtyrseralaasptyrile465470475480prothralaprolysasnglyalaleu485<210>35<211>1110<212>dna<213>bacillusalcalophilus<400>35atgagaagtatgaagcttttatttgctatgtttattttagttttttcctcttttactttt60aacttagtagttgcgcaagctagtggacatggacaaatgcataaagtaccttgggcaccc120caagctgaagcacctggaaaaacggctgagaatggagtctgggataaagttagaaataat180cctggaaaagccaatcctccagcaggaaaagtcaatggtttttatatagatggaacaacc240ttatatgatgcaaatggtaagccatttgtgatgcgcggaattaaccacgctcattcctgg300tacaagcctcacatagaaaccgcgatggaggcaattgctgatactggagcaaactccatt360cgtgtagttctctcagatggacaacagtggaccaaagatgatgttgacgaagtagcaaaa420attatatctttagcagaaaaacattctttagttgctgttcttgaggtacatgatgcactc480ggaacagatgatattgaaccattacttaaaacagtcgattactggattgagatcaaagat540gctttaatcggaaaagaggacaaagtaattattaacatttctaatgaatggtttggttct600tggagcagtgaaggttgggcagaaggatataaaaaagcaattcctttactaagagaggcg660ggtctaaaacatacacttatggttgacgcagctgggtggggacaatttcctagatctatt720catgaaaaaggattagacgtttttaactcagacccattaaagaatacaatgttttccatt780catatgtatgaatgggcagcgggtaatcctcaacaagtaaaagacaatattgacggtgtt840cttgaaaagaatttagctgtagtaattggtgagttcggtcatcatcactacggaagagat900gttgctgttgatacgatcttaagtcattcagagaagtatgatgtaggttggcttgcctgg960tcttggcacggaaatagtggtggtgtagagtatcttgacttagcaacagatttttcaggg1020acgcaactaactgaatggggagaaagaattgtgtacggtccgaatggtttaaaagaaact1080tctgaaatcgttagtgtatacaaaaaataa1110<210>36<211>369<212>prt<213>bacillusalcalophilus<400>36metargsermetlysleuleuphealametpheileleuvalpheser151015serphethrpheasnleuvalvalalaglnalaserglyhisglygln202530methislysvalprotrpalaproglnalaglualaproglylysthr354045alagluasnglyvaltrpasplysvalargasnasnproglylysala505560asnproproalaglylysvalasnglyphetyrileaspglythrthr65707580leutyraspalaasnglylysprophevalmetargglyileasnhis859095alahissertrptyrlysprohisilegluthralametglualaile100105110alaaspthrglyalaasnserileargvalvalleuseraspglygln115120125glntrpthrlysaspaspvalaspgluvalalalysileileserleu130135140alaglulyshisserleuvalalavalleugluvalhisaspalaleu145150155160glythraspaspilegluproleuleulysthrvalasptyrtrpile165170175gluilelysaspalaleuileglylysgluasplysvalileileasn180185190ileserasnglutrppheglysertrpsersergluglytrpalaglu195200205glytyrlyslysalaileproleuleuargglualaglyleulyshis210215220thrleumetvalaspalaalaglytrpglyglnpheproargserile225230235240hisglulysglyleuaspvalpheasnseraspproleulysasnthr245250255metpheserilehismettyrglutrpalaalaglyasnproglngln260265270vallysaspasnileaspglyvalleuglulysasnleualavalval275280285ileglyglupheglyhishishistyrglyargaspvalalavalasp290295300thrileleuserhisserglulystyraspvalglytrpleualatrp305310315320sertrphisglyasnserglyglyvalglutyrleuaspleualathr325330335asppheserglythrglnleuthrglutrpglygluargilevaltyr340345350glyproasnglyleulysgluthrsergluilevalservaltyrlys355360365lys<210>37<211>1482<212>dna<213>bacillussp.<400>37atgaaaaaaaagttatcacagatttatcatttaattatttgcacacttataataagtgtg60ggaataatggggattacaacgtccccatcagaagcaagttcaggcttttatgttgatggc120aatacgttatatgacgcaaacgggcaaccatttgtcatgaaaggcattaaccatggacat180gcttggtataaagacaccgcttcaacagctattcctgccattgcagagcaaggcgcgaac240acgatacgtattgttttatcagatggcggtcaatgggaaaaagacgacattgacaccgtt300cgtgaagttattgagcttgcggagcaaaataaaatggtggctgtcgttgaagttcatgat360gccacgggccgtgattcacgcagtgatttagatcgggcagtcgattattggatagagatg420aaagatgcacttatcggcaaagaggatactgtcattattaacattgcaaacgaatggtat480ggcagttgggatggcgccgcttgggctgatggctacattgatgtcattccgaagcttcgc540gatgccggcttaacacacaccttaatggttgatgcagcaggatgggggcaatatccgcaa600tctattcatgattacggacaagatgtgtttaatgcagatccgttaaaaaatacgatattc660tccatccatatgtatgagtatgctggtggtgatgctaacactgttagatcaaatattgat720agagtcatagatcaagaccttgctctcgtaataggtgagttcggtcatagacacactgat780ggcgatgttgatgaagatacaatccttagttattctgaagaaactggcacaggatggctc840gcttggtcttggaaaggcaacagtgccgaatgggattatttagacctttcagaagattgg900gctggtaaccatttaactgattggggaaataggattgtccacggggcaaatggcttgcag960gaaacctccaaaccatccaccgtatttacagatgataacggtggtgcccctgaaccgcca1020actactactaccttgtatgactttgaaggaagcacacaagggtggcatggaagcaacgtg1080atgggtggcccttggtccgtaacagaatggggtgcgtcaggcaactactctttaaagggc1140gatgtcaatttaagctcaaattcttcacatgaactgtatagtgaacaaagtcgtaatcta1200cacggatactctcagctaaatgcaaccgttcgccatgccaattggggaaatcccggtaat1260ggcatgaatgcaagactttacgtgaaaacgggctctgattatacatggtatagcggtcct1320tttacacgtatcaatagctccaactcaggtacaacgttatcttttgatttaaacaacatc1380gaaaatagtcatcatgttagggaaataggtgtgcaattttcagctgcagataatagcagc1440ggtcaaactgctctatacgttgataatgttactttaagataa1482<210>38<211>493<212>prt<213>bacillussp.<400>38metlyslyslysleuserglniletyrhisleuileilecysthrleu151015ileileservalglyilemetglyilethrthrserprosergluala202530serserglyphetyrvalaspglyasnthrleutyraspalaasngly354045glnprophevalmetlysglyileasnhisglyhisalatrptyrlys505560aspthralaserthralaileproalailealagluglnglyalaasn65707580thrileargilevalleuseraspglyglyglntrpglulysaspasp859095ileaspthrvalarggluvalilegluleualagluglnasnlysmet100105110valalavalvalgluvalhisaspalathrglyargaspserargser115120125aspleuaspargalavalasptyrtrpileglumetlysaspalaleu130135140ileglylysgluaspthrvalileileasnilealaasnglutrptyr145150155160glysertrpaspglyalaalatrpalaaspglytyrileaspvalile165170175prolysleuargaspalaglyleuthrhisthrleumetvalaspala180185190alaglytrpglyglntyrproglnserilehisasptyrglyglnasp195200205valpheasnalaaspproleulysasnthrilepheserilehismet210215220tyrglutyralaglyglyaspalaasnthrvalargserasnileasp225230235240argvalileaspglnaspleualaleuvalileglyglupheglyhis245250255arghisthraspglyaspvalaspgluaspthrileleusertyrser260265270glugluthrglythrglytrpleualatrpsertrplysglyasnser275280285alaglutrpasptyrleuaspleusergluasptrpalaglyasnhis290295300leuthrasptrpglyasnargilevalhisglyalaasnglyleugln305310315320gluthrserlysproserthrvalphethraspaspasnglyglyala325330335progluproprothrthrthrthrleutyraspphegluglyserthr340345350glnglytrphisglyserasnvalmetglyglyprotrpservalthr355360365glutrpglyalaserglyasntyrserleulysglyaspvalasnleu370375380serserasnserserhisgluleutyrsergluglnserargasnleu385390395400hisglytyrserglnleuasnalathrvalarghisalaasntrpgly405410415asnproglyasnglymetasnalaargleutyrvallysthrglyser420425430asptyrthrtrptyrserglyprophethrargileasnserserasn435440445serglythrthrleuserpheaspleuasnasnilegluasnserhis450455460hisvalarggluileglyvalglnpheseralaalaaspasnserser465470475480glyglnthralaleutyrvalaspasnvalthrleuarg485490<210>39<211>1551<212>dna<213>bacilluscirculans<400>39atggggtggtttttagtgattttacgcaagtggttgattgcttttgtcgcatttttactg60atgttctcgtggactggacaacttacgaacaaagcacatgctgcaagcggattttatgta120agcggtaccaaattattggatgctacaggacaaccatttgtgatgcgaggagtcaatcat180gcgcacacatggtataaagatcaactatccaccgcaataccagccattgctaaaacaggt240gccaacacgatacgtattgtactggcgaatggacacaaatggacgcttgatgatgtaaac300accgtcaacaatattctcaccctctgtgaacaaaacaaactaattgccgttttggaagta360catgacgctacaggaagcgatagtctttccgatttagacaacgccgttaattactggatt420ggtattaaaagcgcgttgatcggcaaggaagaccgtgtaatcattaatatagctaacgag480tggtacggaacatgggatggagtcgcctgggctaatggttataagcaagccatacccaaa540ctgcgtaatgctggtctaactcatacgctgattgttgactccgctggatggggacaatat600ccagattcggtcaaaaattatgggacagaagtactgaatgcagacccgttaaaaaacaca660gtattctctatccatatgtatgaatatgctgggggcaatgcaagtaccgtcaaatccaat720attgacggtgtgctgaacaagaatcttgcactgattatcggcgaatttggtggacaacat780acaaacggtgatgtggatgaagccaccattatgagttattcccaagagaagggagtcggc840tggttggcttggtcctggaagggaaatagcagtgatttggcttatctcgatatgacaaat900gattgggctggtaactccctcacctcgttcggtaataccgtagtgaatggcagtaacggc960attaaagcaacttctgtgttatccggcatttttggaggtgttacgccaacctcaagccct1020acttctacacctacatctacgccaacctcaactcctactcctacgccaagtccgaccccg1080agtccaggtaataacgggacgatcttatatgatttcgaaacaggaactcaaggctggtcg1140ggaaacaatatttcgggaggcccatgggtcaccaatgaatggaaagcaacgggagcgcaa1200actctcaaagccgatgtctccttacaatccaattccacgcatagtctatatataacctct1260aatcaaaatctgtctggaaaaagcagtctgaaagcaacggttaagcatgcgaactggggc1320aatatcggcaacgggatttatgcaaaactatacgtaaagaccgggtccgggtggacatgg1380tacgattccggagagaatctgattcagtcaaacgacggtaccattttgacactatccctc1440agcggcatttcgaatttgtcctcagtcaaagaaattggggtagaattccgcgcctcctca1500aacagtagtggccaatcagctatttatgtagatagtgttagtctgcaataa1551<210>40<211>516<212>prt<213>bacilluscirculans<400>40metglytrppheleuvalileleuarglystrpleuilealapheval151015alapheleuleumetphesertrpthrglyglnleuthrasnlysala202530hisalaalaserglyphetyrvalserglythrlysleuleuaspala354045thrglyglnprophevalmetargglyvalasnhisalahisthrtrp505560tyrlysaspglnleuserthralaileproalailealalysthrgly65707580alaasnthrileargilevalleualaasnglyhislystrpthrleu859095aspaspvalasnthrvalasnasnileleuthrleucysgluglnasn100105110lysleuilealavalleugluvalhisaspalathrglyseraspser115120125leuseraspleuaspasnalavalasntyrtrpileglyilelysser130135140alaleuileglylysgluaspargvalileileasnilealaasnglu145150155160trptyrglythrtrpaspglyvalalatrpalaasnglytyrlysgln165170175alaileprolysleuargasnalaglyleuthrhisthrleuileval180185190aspseralaglytrpglyglntyrproaspservallysasntyrgly195200205thrgluvalleuasnalaaspproleulysasnthrvalpheserile210215220hismettyrglutyralaglyglyasnalaserthrvallysserasn225230235240ileaspglyvalleuasnlysasnleualaleuileileglygluphe245250255glyglyglnhisthrasnglyaspvalaspglualathrilemetser260265270tyrserglnglulysglyvalglytrpleualatrpsertrplysgly275280285asnserseraspleualatyrleuaspmetthrasnasptrpalagly290295300asnserleuthrserpheglyasnthrvalvalasnglyserasngly305310315320ilelysalathrservalleuserglyilepheglyglyvalthrpro325330335thrserserprothrserthrprothrserthrprothrserthrpro340345350thrprothrproserprothrproserproglyasnasnglythrile355360365leutyraspphegluthrglythrglnglytrpserglyasnasnile370375380serglyglyprotrpvalthrasnglutrplysalathrglyalagln385390395400thrleulysalaaspvalserleuglnserasnserthrhisserleu405410415tyrilethrserasnglnasnleuserglylysserserleulysala420425430thrvallyshisalaasntrpglyasnileglyasnglyiletyrala435440445lysleutyrvallysthrglyserglytrpthrtrptyraspsergly450455460gluasnleuileglnserasnaspglythrileleuthrleuserleu465470475480serglyileserasnleuserservallysgluileglyvalgluphe485490495argalaserserasnserserglyglnseralailetyrvalaspser500505510valserleugln515<210>41<211>984<212>dna<213>paenibacillussp.<400>41atgagacaacttttagcaaaaggtattttagctgcactggtcatgatgttagcgatgtat60ggattggggaatctctcttctaaagcttcggctgcaacaggtttttatgtaagcggtacc120actctatatgattctactggtaaaccttttgtaatgcgcggtgtcaatcattcgcatacc180tggttcaaaaatgatctaaatgcagccatccctgctattgccaaaacaggtgcaaataca240gtacgtatcgttttatctaatggtgttcagtatactagagatgatgtaaactcagtcaaa300aatattatttccctggttaaccaaaacaaaatgattgctgttcttgaggtgcatgatgct360accggtaaagacgattacgcttctcttgatgccgctgtaaactactggatcagcatcaaa420gatgccttgattggcaaggaagatcgagtcattgttaatattgccaatgaatggtacggt480acatggaatggcagtgcttgggcagatggttataagcaggctattcccaaactaagaaat540gcaggcatcaaaaacactttaatcgttgatgccgccggctggggacaatgtcctcaatcg600atcgttgattacgggcaaagtgtatttgcagcagattcgcttaaaaatacaattttctct660attcacatgtatgaatatgcaggcggtacagatgcgatcgtcaaaagcaatatggaaaat720gtactgaacaaaggacttcctttgatcatcggtgaatttggcgggcagcatacaaacggc780gatgtagatgaacatgcaattatgcgttatggtcagcaaaaaggtgtaggttggctggca840tggtcgtggtatggcaacaatagtgaactcagttatctggatttggctacaggtcccgcc900ggtagtctgacaagtatcggcaatacgattgtaaatgatccatatggtatcaaagctacc960tcgaaaaaagcgggtatcttctaa984<210>42<211>327<212>prt<213>paenibacillussp.<400>42metargglnleuleualalysglyileleualaalaleuvalmetmet151015leualamettyrglyleuglyasnleuserserlysalaseralaala202530thrglyphetyrvalserglythrthrleutyraspserthrglylys354045prophevalmetargglyvalasnhisserhisthrtrpphelysasn505560aspleuasnalaalaileproalailealalysthrglyalaasnthr65707580valargilevalleuserasnglyvalglntyrthrargaspaspval859095asnservallysasnileileserleuvalasnglnasnlysmetile100105110alavalleugluvalhisaspalathrglylysaspasptyralaser115120125leuaspalaalavalasntyrtrpileserilelysaspalaleuile130135140glylysgluaspargvalilevalasnilealaasnglutrptyrgly145150155160thrtrpasnglyseralatrpalaaspglytyrlysglnalailepro165170175lysleuargasnalaglyilelysasnthrleuilevalaspalaala180185190glytrpglyglncysproglnserilevalasptyrglyglnserval195200205phealaalaaspserleulysasnthrilepheserilehismettyr210215220glutyralaglyglythraspalailevallysserasnmetgluasn225230235240valleuasnlysglyleuproleuileileglyglupheglyglygln245250255histhrasnglyaspvalaspgluhisalailemetargtyrglygln260265270glnlysglyvalglytrpleualatrpsertrptyrglyasnasnser275280285gluleusertyrleuaspleualathrglyproalaglyserleuthr290295300serileglyasnthrilevalasnaspprotyrglyilelysalathr305310315320serlyslysalaglyilephe325<210>43<211>981<212>dna<213>bacilluscirculans<400>43atggccaagttgcaaaagggtacaatcttaacagtcattgcagcactgatgtttgtcatt60ttggggagcgcggcgcccaaagccgcagcagctacaggtttttacgtgaatggaggcaaa120ttgtacgattctacgggtaaaccattttacatgaggggtatcaatcatgggcactcctgg180tttaaaaatgatttgaacacggctatccctgcgatcgcaaaaacgggtgccaatacggta240cgaattgttttatcaaacggtacacaatacaccaaggatgatctgaattccgtaaaaaac300atcattaatgtcgtaaatgcaaacaagatgattgctgtgcttgaagtacacgatgccact360gggaaagatgacttcaactcgttggatgcagcggtcaactactggataagcatcaaagaa420gcactgatcgggaaggaagatcgggttattgtaaacattgcaaacgagtggtacggaaca480tggaacggaagcgcgtgggctgacgggtacaaaaaagctattccgaaattaagagatgcg540ggtattaaaaataccttgattgtagatgcagcaggctggggtcagtaccctcaatcgatc600gtcgattacggacaaagcgtattcgccgcggattcacagaaaaatacggcgttttccatt660cacatgtatgagtatgcaggcaaggatgcggccaccgtcaaatccaatatggaaaatgtg720ctgaataaggggctggccttaatcattggtgagttcggaggatatcacaccaatggagat780gtcgatgaatatgcaatcatgaaatatggtctggaaaaaggggtaggatggcttgcatgg840tcttggtacggtaatagctctggattaaactatcttgatttggcaacaggacctaacggc900agtttgacgagctatggtaatacggttgtcaatgatacttacggaattaaaaatacgtcc960caaaaagcgggaatcttttaa981<210>44<211>326<212>prt<213>bacilluscirculans<400>44metalalysleuglnlysglythrileleuthrvalilealaalaleu151015metphevalileleuglyseralaalaprolysalaalaalaalathr202530glyphetyrvalasnglyglylysleutyraspserthrglylyspro354045phetyrmetargglyileasnhisglyhissertrpphelysasnasp505560leuasnthralaileproalailealalysthrglyalaasnthrval65707580argilevalleuserasnglythrglntyrthrlysaspaspleuasn859095servallysasnileileasnvalvalasnalaasnlysmetileala100105110valleugluvalhisaspalathrglylysaspasppheasnserleu115120125aspalaalavalasntyrtrpileserilelysglualaleuilegly130135140lysgluaspargvalilevalasnilealaasnglutrptyrglythr145150155160trpasnglyseralatrpalaaspglytyrlyslysalaileprolys165170175leuargaspalaglyilelysasnthrleuilevalaspalaalagly180185190trpglyglntyrproglnserilevalasptyrglyglnservalphe195200205alaalaaspserglnlysasnthralapheserilehismettyrglu210215220tyralaglylysaspalaalathrvallysserasnmetgluasnval225230235240leuasnlysglyleualaleuileileglyglupheglyglytyrhis245250255thrasnglyaspvalaspglutyralailemetlystyrglyleuglu260265270lysglyvalglytrpleualatrpsertrptyrglyasnsersergly275280285leuasntyrleuaspleualathrglyproasnglyserleuthrser290295300tyrglyasnthrvalvalasnaspthrtyrglyilelysasnthrser305310315320glnlysalaglyilephe325<210>45<211>1110<212>dna<213>bacillusnealsonii<400>45atggttgtgaaaaaattatcaagttttattctaattttactgttagttacttctgctttg60tttattactgattcaaaagcaagtgctgcttcgggattttatgtaagcggtaccacttta120tatgatgcaacgggtaaaccgtttactatgagaggtgtaaatcatgctcattcttggttt180aaagaagattcagcagctgctattccagcaatagcagcaactggagcaaacacagtaaga240attgttttatctgatggtggacaatacaccaaagatgatattaatactgttaaaagcctt300ttgtcattggcagaaaaaataaacttgcattctggagtcatgacgcacagaaaagacgat360gtggaatctttaaatcgtgcagtcgattattggatcagcttaaaagacacattgataggc420aaagaagataaagtgataataaacattgcgaatgaatggtatggtacttgggatggtgcg480gcatgggcagctggttataaacaagctattccaaagttacggaatgcaggcttaaatcat540actctaataattgattctgctggatggggacaatacccagcttccattcataattatgga600aaagaggtatttaatgcggatccattgaaaaatacaatgttctccatacatatgtatgag660tacgctggtggggatgcagcaactgttaagtcaaatattgatggtgtcttaaaccaagga720ttagctttaataataggagagtttggacaaaaacatacaaatggagatgtagatgaagca780accatcatgagttattcacagcaaaaaaatatcggttggcttgcatggtcttggaaagga840aatagcacagattggagctatctggatttaagcaacgattggtctggtaacagtttaact900gattggggtaatacggttgttaatggggcaaatgggttaaaagccacttcaaaactaagc960ggagtattcggtagctcagcaggaacaaataatatattgtatgattttgaaagcggtaat1020caaaactggactggatcaaatatcgcgggtggaccttggaacgaattcaagcttgatatc1080attcaggacgagcctcagactccagcgtaa1110<210>46<211>369<212>prt<213>bacillusnealsonii<400>46metvalvallyslysleuserserpheileleuileleuleuleuval151015thrseralaleupheilethraspserlysalaseralaalasergly202530phetyrvalserglythrthrleutyraspalathrglylysprophe354045thrmetargglyvalasnhisalahissertrpphelysgluaspser505560alaalaalaileproalailealaalathrglyalaasnthrvalarg65707580ilevalleuseraspglyglyglntyrthrlysaspaspileasnthr859095vallysserleuleuserleualaglulysileasnleuhissergly100105110valmetthrhisarglysaspaspvalgluserleuasnargalaval115120125asptyrtrpileserleulysaspthrleuileglylysgluasplys130135140valileileasnilealaasnglutrptyrglythrtrpaspglyala145150155160alatrpalaalaglytyrlysglnalaileprolysleuargasnala165170175glyleuasnhisthrleuileileaspseralaglytrpglyglntyr180185190proalaserilehisasntyrglylysgluvalpheasnalaasppro195200205leulysasnthrmetpheserilehismettyrglutyralaglygly210215220aspalaalathrvallysserasnileaspglyvalleuasnglngly225230235240leualaleuileileglyglupheglyglnlyshisthrasnglyasp245250255valaspglualathrilemetsertyrserglnglnlysasnilegly260265270trpleualatrpsertrplysglyasnserthrasptrpsertyrleu275280285aspleuserasnasptrpserglyasnserleuthrasptrpglyasn290295300thrvalvalasnglyalaasnglyleulysalathrserlysleuser305310315320glyvalpheglyserseralaglythrasnasnileleutyraspphe325330335gluserglyasnglnasntrpthrglyserasnilealaglyglypro340345350trpasngluphelysleuaspileileglnaspgluproglnthrpro355360365ala<210>47<211>984<212>dna<213>bacilluscirculans<400>47atgatgttgatatggatgcagggatggaagtctattctagtcgcgatcttggcgtgtgtg60tcagtaggcggtgggcttcctagtccagaagcagccacaggattttatgtaaacggtacc120aagctgtatgattcaacgggcaaggcctttgtgatgaggggtgtaaatcatccccacacc180tggtacaagaatgatctgaacgcggctattccggctatcgcgcaaacgggagccaatacc240gtacgagtcgtcttgtcgaacgggtcgcaatggaccaaggatgacctgaactccgtcaac300agtatcatctcgctggtgtcgcagcatcaaatgatagccgttctggaggtgcatgatgcg360acaggcaaagatgagtatgcttcccttgaagcggccgtcgactattggatcagcatcaaa420ggggcattgatcggaaaagaagaccgcgtcatcgtcaatattgctaatgaatggtatgga480aattggaacagcagcggatgggccgatggttataagcaggccattcccaaattaagaaac540gcgggcattaagaatacgttgatcgttgatgcagcgggatgggggcaatacccgcaatcc600atcgtggatgagggggccgcggtatttgcttccgatcaactgaagaatacggtattctcc660atccatatgtatgagtatgccggtaaggatgccgctacggtgaaaacgaatatggacgat720gttttaaacaaaggattgcctttaatcattggggagttcggcggctatcatcaaggtgcc780gatgtcgatgagattgctattatgaagtacggacagcagaaggaagtgggctggctggct840tggtcctggtacggaaacagcccggagctgaacgatttggatctggctgcagggccaagc900ggaaacctgaccggctggggaaacacggtggttcatggaaccgacgggattcagcaaacc960tccaagaaagcgggcatttattaa984<210>48<211>327<212>prt<213>bacilluscirculans<400>48metmetleuiletrpmetglnglytrplysserileleuvalalaile151015leualacysvalservalglyglyglyleuproserproglualaala202530thrglyphetyrvalasnglythrlysleutyraspserthrglylys354045alaphevalmetargglyvalasnhisprohisthrtrptyrlysasn505560aspleuasnalaalaileproalailealaglnthrglyalaasnthr65707580valargvalvalleuserasnglyserglntrpthrlysaspaspleu859095asnservalasnserileileserleuvalserglnhisglnmetile100105110alavalleugluvalhisaspalathrglylysaspglutyralaser115120125leuglualaalavalasptyrtrpileserilelysglyalaleuile130135140glylysgluaspargvalilevalasnilealaasnglutrptyrgly145150155160asntrpasnserserglytrpalaaspglytyrlysglnalailepro165170175lysleuargasnalaglyilelysasnthrleuilevalaspalaala180185190glytrpglyglntyrproglnserilevalaspgluglyalaalaval195200205phealaseraspglnleulysasnthrvalpheserilehismettyr210215220glutyralaglylysaspalaalathrvallysthrasnmetaspasp225230235240valleuasnlysglyleuproleuileileglyglupheglyglytyr245250255hisglnglyalaaspvalaspgluilealailemetlystyrglygln260265270glnlysgluvalglytrpleualatrpsertrptyrglyasnserpro275280285gluleuasnaspleuaspleualaalaglyproserglyasnleuthr290295300glytrpglyasnthrvalvalhisglythraspglyileglnglnthr305310315320serlyslysalaglyiletyr325当前第1页1 2 3
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1