固氮酶多肽在植物细胞中的表达的制作方法
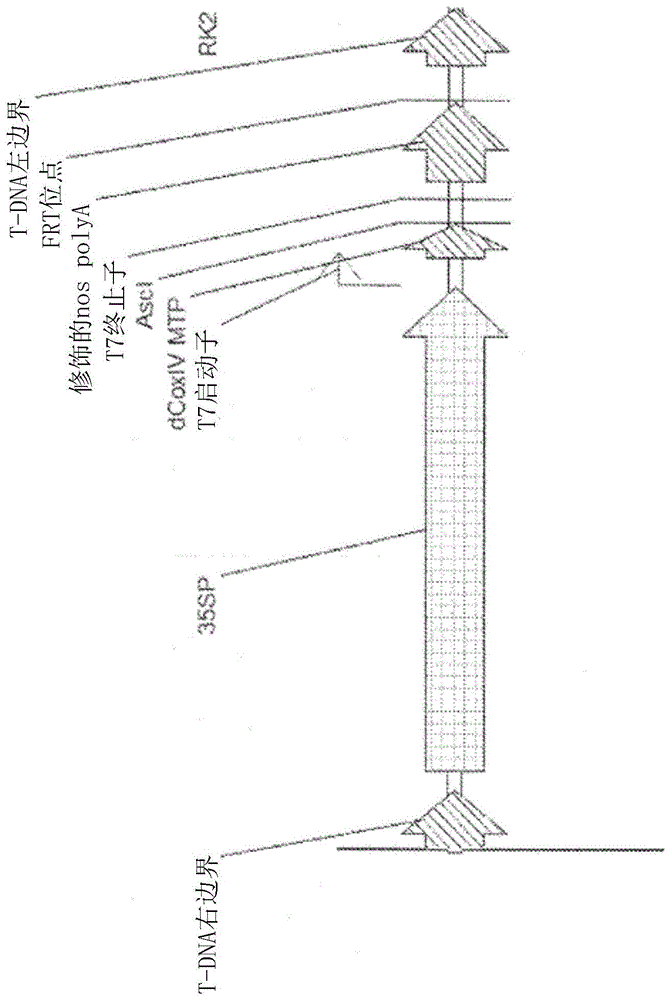
本发明涉及在植物细胞线粒体中产生固氮酶多肽的方法和手段。
背景技术:
:在酶复合物、固氮酶催化下,固氮细菌通过生物固氮(bnf)由n2气体产生氨。然而现代农业的需求远远超出了这种固定氮源,因此工业生产的氮肥广泛用于农业中(smil,2002)。然而,肥料生产和施用都是污染的原因(good和beatty,2011)并且被认为是不可持续的(rockstrom等人,2009)。世界范围内施用的大部分肥料不被作物吸收(cui等人,2013;debruijn,2015),导致肥料流失、杂草滋生和水道富营养化(good和beatty,2011)。由此产生的藻华降低了氧含量,对当地和整个珊瑚礁的近海造成环境破坏(de'ath等人,2012;glibert等人,2014;sutton等人,2008)。此外,尽管在许多发达国家中过度施肥是一个问题,但在某些地区,其可用性限制了作物产量(mueller等人,2012)。生产化肥本身需要大量的能源投入,估计每年需要1000亿美元。显然,需要采取策略减少工业生产的氮依赖性。为此,具有生物固氮作用的工程植物的概念长期以来引起了相当大的兴趣(merrick和dixon,1984),并且一直是最近评论的焦点(debruijn,2015;oldroyd和dixon,2014)。可能的方法包括i)将固氮生物(diazotrophs)的共生关系从豆类延伸到谷物(santi等人,2013年),ii)重建(re-engineering)内共生微生物使其能够具有固氮作用(geddes等人,2015年),iii)用基因工程将固氮酶植入植物细胞(curatti和rubio,2014年)。由于技术上的困难,所有这些方法都是宏伟的和推测性的。固氮酶是一种能够在固氮细菌(diazotrophicbacteria)中产生生物固氮作用的酶复合物,它需要一个多基因组装途径来实现其生物合成和功能(hu和ribbe,2013;rubo和ludden,2008;seefeldt等人,2009进行了广泛的综述)。典型的铁钼固氮酶(iron-molybdenumnitrogenase)的组件(component)包括称为nifd和nifk的催化蛋白和电子供体nifh。大约12种其他蛋白参与固氮细菌固氮酶的组装,包括复合物的成熟、支撑和辅助因子插入,具体为nifm、nifs、nifu、nife、nifn、nifx、nifv、nifj、nify、niff、nifz和nifq。遗传病变、固氮生物与非固氮原核生物之间的互补分析和系统发育分析(dossantos等人,2012;temme等人,2012;wang等人,2013)导致nif蛋白的子集(nifd、nifk、nifb、nife和nifn)被认为是核心组件,而其他被认为是优化活性所需的并且被认为是辅助的。固氮酶的组装和功能也需要特定的生化条件。最重要的是,固氮酶对氧极其敏感(robson和postgate,1980)。此外,金属蛋白酶催化中心的生物合成和功能需要大量atp、还原剂、容易获得的fe、mo、s-腺苷甲硫氨酸和高柠檬酸(hu和ribbe,2013;rubo和ludden,2008)。所有这些因素导致了在植物细胞中产生功能性固氮酶复合物的技术困难。技术实现要素:鉴于观察到在植物细胞中产生nifd困难,本发明人确定了在植物细胞中具有等量nifd和nifk的重要性。多肽的详细分析显示它们可以表达为融合蛋白并维持功能,和具有野生型c端的nifk组件的重要性。因此,一方面,本发明提供了包括线粒体和融合多肽的植物细胞,融合多肽包括(i)具有c端的线粒体靶向肽(mtp),(ii)具有n端和c端的nifd多肽(nd),(iii)寡肽接头,和(iv)具有n端的nifk多肽(nk),其中mtp的c端与nd的n端翻译融合,并且其中接头与nd的c端和nk的n端翻译融合。在一个实施方案中,融合多肽的c端是nk的c端。在一个优选的实施方案中,nk的c端与野生型nifk多肽的c端相同,即nk缺乏任何人工添加的c端延伸区。具有野生型c端在保持nifk活性方面是重要的。在另一个实施方案中,接头具有足够的长度以允许nd和nk在植物细胞或细菌细胞中以功能构型缔合。在一个实施方案中,接头长度为8至50个氨基酸。优选地,接头长度为至少约20个氨基酸、至少约25个氨基酸,或至少约30个氨基酸。更优选地,接头长度为25至35个氨基酸。最优选地,接头长度为约30个氨基酸。在本文中,“约30”是指27、28、29、30、31、32或33个氨基酸。在一个实施方案中,当作为两种单独的多肽存在时,融合多肽中的nifd多肽和nifk多肽具有与nifd多肽和nifk多肽相同的功能。优选地,融合多肽的生化活性与野生型nifd多肽和野生型nifk多肽相同。本发明人还确定了植物细胞中具有等量nife和nifn的重要性。对多肽的详细分析表明它们可以表达为融合蛋白并维持功能,以及需要存在接头以使nif多肽具有功能。因此,另一方面,本发明提供了包括线粒体和融合多肽的植物细胞,融合多肽包括(i)具有c端的线粒体靶向肽(mtp),(ii)具有n端和c端的nife多肽(ne),(iii)寡肽接头,和(iv)具有n端的nifn多肽(nn),其中mtp的c端与ne的n端翻译融合,并且其中接头与ne的c端和nn的n端翻译融合。在上述方面的一个实施方案中,接头具有足够的长度以允许ne和nn在植物细胞或细菌细胞中以功能构型缔合。例如,在一个实施方案中,接头的长度为至少约至少约或约至约或约至约或约或约在一个实施方案中,寡肽接头的长度为至少约20个氨基酸、或至少约30个氨基酸、或至少约40个氨基酸、或约20个氨基酸至约70个氨基酸、或约30个氨基酸至约70个氨基酸、或约30个氨基酸至约60个氨基酸、或约30个氨基酸至约50个氨基酸、或约25个氨基酸、或约30个氨基酸、或约35个氨基酸、或约40个氨基酸、或约45个氨基酸、或约46个氨基酸、或约50个氨基酸、或约55个氨基酸。在一个实施方案中,当作为两种单独的多肽存在时,融合多肽中的nife多肽和nifn多肽具有与nife多肽和nifn多肽相同的功能。优选地,融合多肽的生化活性与野生型nife多肽和野生型nifn多肽相同。在一个实施方案中,mtp包括用于基质作用蛋白酶(mpp)的蛋白酶切割位点,使得融合多肽能够被mpp切割以产生n端切割肽,和包括(ii)至(iv)的加工融合多肽(cf)。在一个实施方案中,cf包括mtp的一些但非全部的c端氨基酸,优选mtp的c端氨基酸的5至45个氨基酸。在一个实施方案中,mpp切割从融合多肽中除去5至50个氨基酸以产生cf。在优选的实施方案中,cf包括来自mtp的c端的约5至约11个氨基酸残基,例如来自mtp的c端的6或7个氨基酸、7或8个氨基酸、8或9个氨基酸、9或10个氨基酸、或11或12个氨基酸。在上述两个方面的实施方案中,植物细胞还包括一种或多种nf融合多肽(nf),每个nf包括(i)具有c端的线粒体靶向肽(mtp)和(ii)具有n端的nif多肽(np),其中mtp的c端与nf翻译融合至np的n端,其中每个mtp独立地相同或不同,并且每个np独立地相同或不同,并且其中线粒体包括一种或多种nf和/或其加工产物(cf),并且其中每个cf,如果存在的话,通过切割其mtp内相应的nf产生。优选地,至少一种nf多肽是nifh。在优选的实施方案中,每个cf独立的包括来自mtp的c端的约5至约11个氨基酸残基,例如来自mtp的c端的6或7个氨基酸、7或8个氨基酸、8或9个氨基酸、9或10个氨基酸、或11或12个氨基酸。在一个实施方案中,将编码融合多肽的外源多核苷酸整合到细胞基因组中。本发明人还展示了所有16种肺炎克雷伯菌(klebsiellapneumoniae)生物合成和功能性固氮酶(nif)蛋白都可以在植物细胞中,例如在本生烟(nicotianabenthamiana)叶片中作为线粒体靶向肽(mtp)-nif融合多肽单独表达。本发明人已经证明这些融合正确地靶向至线粒体基质(mm),线粒体基质是具有生物化学和遗传特性的亚细胞定位,具有支持固氮酶功能的潜力。采用westernblot方法检测nifj、nifh、nifd、nifk、nify、nife、nifn、nifx、nifu、nifs、nifv、nifm、niff、nifb和nifq多肽。然而nifd是参与催化的核心组件,其丰度最低。利用固氮酶nifd-nifk异二聚体的晶体结构设计了提高表达水平的nifd-nifk翻译融合蛋白。最后,成功共表达了4种nif融合多肽(nifb、nifs、nifh、nify),证明了固氮酶的多种组件可以靶向至线粒体。这些结果证实了在细胞内环境中重组固氮酶组件以支持氮气还原为氨气的可行性。在所有的nif蛋白中,固氮酶催化复合物的必要组件nifd是最难表达的。低水平的nifd蛋白与高水平的nifdrna相反,提示翻译速率或蛋白稳定性限制了nifd蛋白的丰度。考虑到nifd在催化中的关键重要性,其需要在细菌中高度表达(poza-carrion等人,2014),并且优选与nifk等摩尔比,本发明人将这两个关键组件融合在一起,并且发现通过该策略可以提高nifd的丰度。nifd-nifk融合还具有在表达中由单个盒连接的优点,允许两个亚基以理想的1∶1比率翻译,模拟天然异源四聚体的化学计量。此外,接头本身被设计成允许两个亚基具有足够的柔性以形成催化所需的正确的α2β2异源四聚体结构。本发明人预期nifd-nifk融合多肽将至少在功能上替代单个nifd和nifk表达,可能具有比先前证明的更高的效力。因此,一方面,本发明提供了包括线粒体和编码nifd融合多肽(ndf)的外源多核苷酸的植物细胞,ndf包括(i)具有c端的线粒体靶向肽(mtp)和(ii)具有n端的nifd多肽(nd),其中mtp的c端与nd的n端翻译融合,其中线粒体包括ndf和/或其加工的nifd产物(cdf),并且cdf,如果存在的话,则通过切割mtp内的ndf产生。在一个实施方案中,mtp包括用于基质作用蛋白酶(mpp)的蛋白酶切割位点,使得ndf能够被mpp切割以产生n端切割肽和cdf。在一个实施方案中,cdf包括来自mtp的c端的约5至约11个氨基酸残基,例如来自mtp的c端的6或7个氨基酸、7或8个氨基酸、8或9个氨基酸、9或10个氨基酸、或11或12个氨基酸。在一个方面,本发明提供了包括一种或多种nf融合多肽(nf)的植物细胞,每种nf包括(i)具有c端的线粒体靶向肽(mtp)和(ii)具有n端的nif多肽(np),np选自由nife、niff、nifj、nifm、nifn、nifq、nifs、nifu、nifv、nifw、nifx、nify和nifz组成的组,其中mtp的c端与np的n端翻译融合,其中每个mtp独立地相同或不同并且每个np独立地相同或不同。并且其中线粒体包括一种或多种nf和/或其加工产物(cf),并且其中每种cf,如果存在的话,通过在其mtp内切割相应的nf产生。在一个实施方案中,nf多肽还包括选自由nifd、nifh和nifk组成的组的一种或多种或优选全部的nif多肽。在优选的实施方案中,nifk和/或nifn的c端分别与野生型nifk多肽或野生型nifn多肽的c端相同,即nifk和/或nifn,优选两者,缺少任何人工添加的c端延伸区。在优选的实施方案中,每个cf独立的包括来自mtp的c端的约5至约11个氨基酸残基,例如来自mtp的c端的6或7个氨基酸、7或8个氨基酸、8或9个氨基酸、9或10个氨基酸、或11或12个氨基酸。另一方面,本发明提供了植物细胞,其包括线粒体、编码第一nif融合多肽(nf)的第一外源多核苷酸和编码第二nf的第二外源多核苷酸,每种nf包括(i)具有c端的线粒体靶向肽(mtp)和(ii)具有n端的nif多肽(np),其中mtp的c端与np的n端翻译融合,其中每个mtp独立地相同或不同并且每个np独立地相同或不同。并且其中线粒体包括(a)第一nf和/或其加工产物(第一cf)和(b)第二nf和/或其加工产物(第二cf),并且其中每种cf,如果存在的话,通过在其mtp内切割相应的nf产生。在优选的实施方案中,第一nf和第二nf选自由nife、niff、nifj、nifm、nifn、nifq、nifs、nifu、nifv、nifw、nifx、nify和nifz组成的组。在优选的实施方案中,nifk(如果存在)和/或nifn的c端分别与野生型nifk多肽或野生型nifn多肽的c端相同,即nifk和/或nifn,优选两者,缺少任何人工添加的c端延伸区。在优选的实施方案中,每个cf独立的包括来自mtp的c端的约5至约11个氨基酸残基,例如来自mtp的c端的6或7个氨基酸、7或8个氨基酸、8或9个氨基酸、9或10个氨基酸、或11或12个氨基酸。在一个实施方案中,植物细胞还包括一种或多种编码一种或多种nf的外源多核苷酸,每种nf包括(i)具有c端的线粒体靶向肽(mtp)和(ii)具有n端的nif多肽(np),其中mtp的c端与np的n端翻译融合,其中每个mtp独立地相同或不同并且每个np独立地相同或不同。并且其中线粒体包括一种或多种nf和/或其加工产物(cf),并且其中每种cf,如果存在的话,通过在其mtp内切割相应的nf产生。在优选的实施方案中,一种或多种nf选自由nife、niff、nifj、nifm、nifn、nifq、nifs、nifu、nifv、nifw、nifx、nify和nifz组成的组。在优选的实施方案中,nifk(如果存在)和/或nifn的c端分别与野生型nifk多肽或野生型nifn多肽的c端相同,即nifk和/或nifn,优选两者,缺少任何人工添加的c端延伸区。在一个或另一个实施方案中,至少一种mtp、或多于一种mtp、或每个mtp包括用于基质作用蛋白酶(mpp)的蛋白酶切割位点,使得包括mpp的蛋白酶切割位点的每种nf能够被mpp切割以产生n端切割肽和相应的cf。在优选的实施方案中,每个cf独立的包括来自mtp的c端的约5至约11个氨基酸残基,例如来自mtp的c端的6或7个氨基酸、7或8个氨基酸、8或9个氨基酸、9或10个氨基酸、或11或12个氨基酸。在另一方面,本发明提供了植物细胞,其包括线粒体和(a)编码nifd融合多肽(ndf)的第一外源多核苷酸,ndf包括(i)具有c端的第一线粒体靶向肽(mtp1)和(ii)具有n端的nifd多肽(nd),其中mtp1的c端与nd的n端翻译融合,(b)编码nifh融合多肽(nhf)的第二外源多核苷酸,nhf包括(i)具有c端的第二线粒体靶向肽(mtp2)和(ii)具有n端的nifh多肽(nh),其中mtp2的c端与nh的n端翻译融合,和(c)编码nifk融合多肽(nkf)的第三外源多核苷酸,nkf包括(i)具有c端的第三线粒体靶向肽(mtp3)和(ii)具有n端的nifk多肽(nk),其中第三mtp的c端与nk的n端翻译融合,其中mtp1、mtp2和mtp3各自独立地相同或不同,和其中植物细胞包括的ndf和/或其加工产物(cdf)的水平高于相应植物细胞中的ndf和/或cdf的水平,该相应植物细胞包括第一外源多核苷酸并且缺少第二和第三外源多核苷酸,并且cdf,如果存在的话,则通过切割mtp1内的ndf产生。在一个优选的实施方案中,植物细胞还包括一种或多种编码一种或多种nf的外源多核苷酸,nf选自由nife、niff、nifj、nifm、nifn、nifq、nifs、nifu、nifv、nifw、nifx、nify和nifz组成的组。在优选的实施方案中,nifk和/或nifn(如果存在)的c端分别与野生型nifk多肽或野生型nifn多肽的c端相同,即nifk和/或nifn,优选两者,缺少任何人工添加的c端延伸区。在一个实施方案中,cdf存在于线粒体中。在另一个实施方案中,线粒体还包括nhf(chf)和nkf(ckf)之一或两者的加工产物,其中chf和ckf,如果存在的话,分别通过切割mtp2和mtp3内的nhf和nkf而产生。在优选的实施方案中,chf和/或ckf独立地包括来自mtp的c端的约5至约11个氨基酸残基,例如来自mtp的c端的6或7个氨基酸、7或8个氨基酸、8或9个氨基酸、9或10个氨基酸、或11或12个氨基酸。在另一个实施方案中,植物细胞的特征在于以下的一个或多个或全部:(i)mtp1包括用于mpp的蛋白酶切割位点,使得ndf能够被mpp切割以产生n端切割肽和cdf,(ii)mtp2包括用于mpp的蛋白酶切割位点,使得nhf能够被mpp切割以产生n端切割肽和加工的nifk产物(ckf)和(iii)mtp3包括用于mpp的蛋白酶切割位点,使得nkf能够被mpp切割以产生n端切割肽和加工的nifk产物(ckf),其中线粒体包括cdf、chf和ckf中的一种或多种或全部。在优选的实施方案中,cdf、chf和/或ckf各自独立地包括来自mtp的c端的约5至约11个氨基酸残基,例如来自mtp的c端的6或7个氨基酸、7或8个氨基酸、8或9个氨基酸、9或10个氨基酸、或11或12个氨基酸。在另一方面,本发明提供一种植物细胞,其包括线粒体、编码nifd多肽(nd)的第一外源多核苷酸和编码nifk多肽(nk)的第二外源多核苷酸,其中nd和nk中的任一者或两者在其n端翻译融合至具有c端的线粒体靶向肽(mtp),其中如果nd和nk两者都翻译融合至mtp,则每个mtp独立地相同或不同,并且其中第二外源多核苷酸共价连接或不共价连接至第一外源多核苷酸,并且其中植物细胞包括大约相同的nd水平和nk水平。在一个优选的实施方案中,nifk的c端与野生型nifk多肽的c端相同,即nifk缺乏任何人工添加的c端延伸区。在优选的实施方案中,nd和/或nk的切割产生切割产物多肽,其具有来自mtp的c端的约5至约11个氨基酸残基,例如来自mtp的c端的6或7个氨基酸、7或8个氨基酸、8或9个氨基酸、9或10个氨基酸、或11或12个氨基酸。在一个实施方案中,nd水平和nk水平包括mtp-nif融合多肽及其加工产物。另一方面,本发明提供了包括线粒体和编码nifh融合多肽(nhf)的外源多核苷酸的植物细胞,nhf包括(i)具有c端的线粒体靶向肽(mtp)和(ii)具有n端的nifh多肽(nh),其中mtp的c端与nh的n端翻译融合,其中线粒体包括nhf和/或其加工的nifh产品(chf),并且其中chf,如果存在的话,则通过切割mtp内的nhf产生。在一个实施方案中,mtp包括用于基质作用蛋白酶(mpp)的蛋白酶切割位点,使得nhf能够被mpp切割以产生n端切割肽和chf。在一个实施方案中,chf包括来自mtp的c端的约5至约11个氨基酸残基,例如来自mtp的c端的6或7个氨基酸、7或8个氨基酸、8或9个氨基酸、9或10个氨基酸、或11或12个氨基酸。在另一方面,本发明提供了包括线粒体和编码nif融合多肽(nf)的外源多核苷酸的植物细胞,nf包括(i)具有c端的线粒体靶向肽(mtp)和(ii)具有n端的nif多肽(np),其中mtp的c端与np的n端翻译融合,其中线粒体包括nf和/或其加工的np产物(cf),并且其中cf,如果存在的话,通过mtp内nf的切割产生,并且其中nf和/或cf,优选nf和cf两者相对于相应的野生型nif多肽具有天然c端。在一个优选的实施方案中,np选自由nifd多肽、nifk多肽和nifn多肽组成的组。优选地,nifk和nifn都有。另一方面,本发明提供了包括线粒体和编码nifd融合多肽(ndf)的第一外源多核苷酸的植物细胞,ndf包括(i)具有c端的第一线粒体靶向肽(mtp1)和(ii)具有n端的nifd多肽(nd),其中mtp1的c端与nd的n端翻译融合,以及以下一个或多个或全部(a)编码nifh融合多肽(nhf)的第二外源多核苷酸,nhf包括(i)具有c端的第二线粒体靶向肽(mtp2)和(ii)具有n端的nifh多肽(nh),其中mtp2的c端与nh的n端翻译融合,(b)编码nifk融合多肽(nkf)的第三外源多核苷酸,nkf包括(i)具有c端的第三线粒体靶向肽(mtp3)和(ii)具有n端的nifk多肽(nk),其中mtp3的c端与nk的n端翻译融合,优选地,其中nkf相对于野生型nifk多肽具有天然c端,并且(c)编码除ndf、nhf和nkf之外的nif融合多肽(nf)的外源第四多核苷酸,nf包括(i)具有c端的第四线粒体靶向肽(mtp4)和(ii)具有n端的nif多肽(np),其中mtp4的c端与np的n端翻译融合,其中mtp1、mtp2、mtp3和mtp4各自独立地相同或不同。在一个实施方案中,第四外源多核苷酸编码选自由nife、niff、nifj、nifm、nifn、nifq、nifs、nifu、nifv、nifw、nifx、nify和nifz组成的组中的一种或多种np。在优选的实施方案中,nifk和/或nifn(如果存在)的c端分别与野生型nifk多肽或野生型nifn多肽的c端相同,即nifk和/或nifn,优选两者,缺少任何人工添加的c端延伸区。在一个实施方案中,每个nf能够易位到线粒体中。在上述任一方面的一个实施方案中,线粒体包括一种、至少两种、至少三种、至少四种或所有选自由(i)nifd、nifh、nifk、nifb、nife和nifn,或(ii)nifd、nifh、nifk和nifs组成的组的nif多肽。在一个或另一个实施方案中,通过植物细胞中的基质作用蛋白酶(mpp)在mtp内切割一种或多种或全部nif融合多肽,优选其中至少nifd融合多肽或nifh融合多肽被mpp切割。在一个或另一个实施方案中,选自由nnd、ndf、cdf、nh、nhf、chf、nk、nkf、ckf、nf、cfnb、ne、nn和ns组成的组中一种或多种多肽能够与至少1种、优选至少2种,或至少3种,或至少4种,或至少5种,或至少6种,或至少7种其他nif多肽结合形成nif蛋白复合物(npc),优选使得npc具有固氮酶活性。在一个或另一个实施方案中,cdf、ckf、chf或cf的大小分别大于nd、nk、nh或np,优选大5至50个氨基酸残基,和/或其中cdf、ckf、chf或cf的大小分别小于ndf、nkf、nhf或nf,优选小5至45个氨基酸残基。在优选的实施方案中,cdf、ckf、chf或cf各自独立地包括来自mtp的c端的约5至约11个氨基酸残基,例如来自mtp的c端的6或7个氨基酸、7或8个氨基酸、8或9个氨基酸、9或10个氨基酸、或11或12个氨基酸。在一个或另一个实施方案中,mtp包括至少10个氨基酸,优选10至80个氨基酸。在一个或另一个实施方案中,至少一种、或多种、或所有mtp包括线粒体蛋白前体的mtp,或其变异体,优选植物mtp。在一个或另一个实施方案中,外源多核苷酸整合到细胞基因组中。在一个或另一个实施方案中,细胞不是原生质体。在一个或另一个实施方案中,细胞是拟南芥原生质体以外的细胞。在另一方面,本发明提供包括根据本发明的细胞的转基因植物,其中转基因植物对于外源多核苷酸是转基因的和/或转基因植物对于编码融合多肽的外源多核苷酸是转基因的。在一个实施方案中,一种或多种或所有外源多核苷酸在植物的根中表达,优选在植物的根中比在植物的叶片中以更高水平表达。在一个或另一个实施方案中,转基因植物相对于相应的野生型植物具有改变的表型,其相对于相应的野生型植物在产量、生物质、生长速率、活力、源自生物固氮的氮增益、氮利用效率、非生物胁迫抗性和/或对营养缺乏的抗性方面有所增加。在另一个实施方案中,转基因植物相对于相应的野生型植物具有相同的生长速率和/或表型。在一个或另一个实施方案中,转基因植物是谷类植物,例如小麦、稻、玉米、黑小麦、燕麦或大麦,优选小麦。在一个实施方案中,转基因植物对于外源多核苷酸是纯合的或杂合的。在一个或另一个实施方案中,转基因植物在田地中培育。在另一方面,本发明提供了在田地中培育的根据本发明的至少100株植物的群体。另一方面,本发明提供了nifd融合多肽(ndf),该ndf包括(i)具有c端的线粒体靶向肽(mtp)和(ii)具有n端的nifd多肽(nd),其中mtp的c端与nd的n端翻译融合。另一方面,本发明提供了nifh融合多肽(nhf),该nhf包括(i)具有c端的线粒体靶向肽(mtp)和(ii)具有n端的nifh多肽(nh),其中mtp的c端与nh的n端翻译融合。在另一方面,本发明提供了nif融合多肽(nf),该nf包括(i)具有c端的线粒体靶向肽(mtp)和(ii)具有n端的nif多肽(np),其中mtp的c端与np的n端翻译融合,并且其中nf相对于相应的野生型nif多肽具有天然c端。在一个优选的实施方案中,np选自由nifd多肽、nifk多肽和nifn多肽组成的组。在另一方面,本发明提供了nif融合多肽(nf)的组合,其包括nifk融合多肽(nkf)和nifn融合多肽(nnf),每个nf包括(i)具有c端的线粒体靶向肽(mtp),和(ii)nif多肽(np),其具有:n端,其中mtp的c端与np的n端翻译融合,其中每个nf的mtp独立地相同或不同,并且其中nkf和nnf中的每一个相对于相应的野生型nif多肽具有天然c端。再一方面,本发明提供了融合多肽,其包括:(i)具有c端的线粒体靶向肽(mtp),(ii)具有n端和c端的nifd多肽(nd),(iii)寡肽接头,和(ii)具有n端的nifk多肽(nk),其中mtp的c端与nd的n端翻译融合,并且其中接头与nd的c端和nk的n端翻译融合。在一个实施方案中,接头具有足够的长度以允许nd和nk在植物细胞或细菌细胞中以功能构型缔合。在一个实施方案中,接头长度为8至50个氨基酸。优选地,接头长度为至少约20个氨基酸、至少约25个氨基酸、或至少约30个氨基酸。在一个实施方案中,融合多肽的c端是nk的c端。另一方面,本发明提供了融合多肽,其包括:(i)具有c端的线粒体靶向肽(mtp),(ii)具有n端和c端的nife多肽(ne),(iii)寡肽接头,和(iv)具有n端的nifn多肽(nn),其中mtp的c端与ne的n端翻译融合,并且其中接头与ne的c端和nn的n端翻译融合。在一个实施方案中,接头具有足够的长度以允许ne和nn在植物细胞或细菌细胞中以功能构型缔合。例如,在一个实施方案中,接头的长度为至少约至少约或约至约或约至约或约或约在上述方面的另一个实施方案中,寡肽接头的长度为至少约20个氨基酸、或至少约30个氨基酸、或至少约40个氨基酸、或约20个氨基酸至约70个氨基酸、或约30个氨基酸至约70个氨基酸、或约30个氨基酸至约60个氨基酸、或约30个氨基酸至约50个氨基酸、或约25个氨基酸、或约30个氨基酸、或约35个氨基酸、或约40个氨基酸、或约45个氨基酸、或约46个氨基酸、或约50个氨基酸、或约55个氨基酸。在一个实施方案中,上述任一方面的融合多肽能够被基质作用蛋白酶(mpp)分割以产生一种或多种加工的nif多肽产物。在另一个实施方案中,融合多肽存在于植物细胞或细菌细胞中,优选存在于植物细胞的线粒体中。在另一方面,本发明提供了通过在融合多肽的mtp内切割而由根据本发明的融合多肽产生的加工的nif多肽。在一个实施方案中,通过用mpp在植物细胞的线粒体基质中切割融合多肽来产生加工的nif多肽。在一个或另一个实施方案中,加工的nif多肽存在于植物细胞或细菌细胞中,优选存在于植物线粒体中,更优选存在于植物细胞线粒体的线粒体基质(mm)中。在一个或另一个实施方案中,融合多肽或加工的nif多肽具有与相应的野生型nif多肽相同的生物化学活性,优选具有与相应的野生型nif多肽大约相同水平的生物化学活性,任选地在细菌细胞中测定。在一个或另一个实施方案中,融合多肽或加工的nif多肽与至少1种,优选至少2种,或至少3种,或至少4种,或至少5种,或至少6种,或至少7种其他nif多肽缔合以形成nif蛋白复合物(npc),优选使得npc具有固氮酶活性。另一方面,本发明提供了编码根据本发明的一种或多种或所有的融合多肽或加工的nif多肽的多核苷酸。在一个实施方案中,nf是nifd融合多肽(ndf),并且多核苷酸还包括一个或多个核苷酸序列,其编码以下的一种或多种或全部:(i)nifh融合多肽(nhf),nhf包括具有c端的第二线粒体靶向肽(mtp2)和具有n端的nifh多肽(nh),使得mtp2的c端与nh的n端翻译融合,(ii)nifk融合多肽(nkf),nkf包括具有c端的第三线粒体靶向肽(mtp3)和具有n端的nifk多肽(nk),使得mtp3的c端与nk的n端翻译融合,和(iii)除ndf、nhf和nkf的nif融合多肽(nf),nf包括具有c端的第四线粒体靶向肽(mtp4)和具有n端的nif多肽(np),使得mtp4的c端与np的n端翻译融合,其中mtp1、mtp2、mtp3和mtp4各自独立地相同或不同。在一个或另一个实施方案中,多核苷酸经密码子修饰以在植物细胞中表达。在一个或另一个实施方案中,多核苷酸包括可操作地连接到多核苷酸的启动子或其内部编码融合多肽的每个序列,和/或可操作地连接到多核苷酸的翻译调节元件。在一个实施方案中,启动子促进多核苷酸在植物的根、叶片和/或茎中的表达,优选启动子促进多核苷酸在相对于植物种子的植物的根、叶或茎中的一个或多个或全部中表达。在一个或另一个实施方案中,多核苷酸存在于植物细胞或细菌细胞中,优选整合到植物细胞的基因组中。另一方面,本发明提供了包括编码第一nif融合多肽(nf)的第一多核苷酸和编码第二nf的第二多核苷酸的核酸构建体,每种nf包括(i)具有c端的线粒体靶向肽(mtp)和(ii)具有n端的nif多肽(np),其中mtp的c端与np的n端翻译融合,并且其中每个mtp独立地相同或不同。在一个实施方案中,核酸构建体进一步包括一种或多种编码一种或多种nf的多核苷酸,每种nf包括(i)具有c端的线粒体靶向肽(mtp)和(ii)具有n端的nif多肽(np),其中mtp的c端与np的n端翻译融合,其中每个mtp独立地相同或不同并且每个np独立地相同或不同。另一方面,本发明提供了核酸构建体,其编码nifd融合多肽(ndf)的第一多核苷酸,ndf包括(i)具有c端的第一线粒体靶向肽(mtp1)和(ii)具有n端的nifd多肽(nd),其中mtp1的c端与nd的n端翻译融合,以及一种、或多种、或所有(a)编码nifh融合多肽(nhf)的第二多核苷酸,nhf包括(i)具有c端的第二线粒体靶向肽(mtp2)和(ii)具有n端的nifh多肽(nh),其中mtp2的c端与nh的n端翻译融合,(b)编码nifk融合多肽(nkf)的第三多核苷酸,nkf包括(i)具有c端的第三线粒体靶向肽(mtp3)和(ii)具有n端的nifk多肽(nk),其中mtp3的c端与nk的n端翻译融合,优选地,其中nkf相对于野生型nifk多肽具有天然c端,并且(c)编码除ndf、nhf和nkf外的nif融合多肽(nf)的第四多核苷酸,nf包括(i)具有c端的第四线粒体靶向肽(mtp4)和(ii)具有n端的nif多肽(np),其中mtp4的c端与np的n端翻译融合,其中mtp1、mtp2、mtp3和mtp4各自独立地相同或不同。在一个实施方案中,每个nf能够易位到植物细胞的线粒体中。在另一方面,本发明提供嵌合载体(chimericvector),其包括或编码根据本发明的多核苷酸,或根据本发明的核酸构建体。在一个实施方案中,多核苷酸或其中编码融合多肽的每个序列与启动子和任选的转录终止序列可操作地连接。在一个实施方案中,启动子促进一种,或多种,或所有多核苷酸在植物的根、叶和/或茎中的表达,优选多核苷酸相对于植物种子优先在植物的一种,或多种,或所有根、叶或茎中表达。另一方面,本发明提供一种细胞,其包括一种,或多种,或所有根据本发明的融合多肽或加工的nif多肽,一种,或多种,或所有根据本发明的多核苷酸,根据本发明的核酸构建体,和/或根据本发明的载体(vector)。在一个实施方案中,细胞是植物细胞或细菌细胞。在另一个实施方案中,植物细胞是谷类植物细胞,例如小麦细胞、稻细胞、玉米细胞、黑小麦细胞、燕麦细胞或大麦细胞,优选小麦细胞。另一方面,本发明提供了生产根据本发明的融合多肽或加工的nif多肽的方法,该方法包括在细胞中表达根据本发明的多核苷酸。另一方面,本发明提供了生产根据本发明的细胞的方法,该方法包括将根据本发明的多核苷酸,根据本发明核酸构建体和/或根据本发明载体(vector)引入细胞的步骤。在另一方面,本发明提供了生产根据本发明的转基因植物的方法,该方法包括以下步骤:i)将根据本发明的多核苷酸,根据本发明的核酸构建体和/或根据本发明的载体(vector)引入植物细胞中,ii)从细胞再生转基因植物,和iii)任选地从植物收获种子,和/或iv)任选地从转基因植物产生一个或多个子代植物,由此产生转基因植物。在另一方面,本发明提供了一种生产植物的方法,其具有根据本发明整合到其基因组中的多核苷酸,或根据本发明的任何一种核酸构建体中定义的多核苷酸,该方法包括以下步骤:i)使两种亲本植物杂交,其中至少一种植物包括该多核苷酸,ii)从杂交中筛选一个或多个子代植物存在或不存在多核苷酸,以及iii)选择包括该多核苷酸的子代植物,由此产生植物。在一个实施方案中,多核苷酸编码促进植物固氮酶活性的多肽。在一个或另一个实施方案中,至少一种亲本植物是四倍体或六倍体小麦植物。在一个或另一个实施方案中,步骤ii)包括分析包括来自一个或多个子代植物dna样品中的多核苷酸。在一个或另一个实施方案中,步骤iii)包括i)选择多核苷酸纯合的子代植物,和/或ii)分析植物或其一个或多个子代植物的多核苷酸的存在和/或表达或如上所述的改变的表型。在一个或另一个实施方案中,该方法进一步包括iv)将步骤i)的杂交子代和与缺乏该多核苷酸的第一亲本植物具有相同基因型的植物回交足够次数以产生具有第一亲本的大部分基因型但包括该多核苷酸的植物,和iv)选择包括多核苷酸和/或具有如上定义的改变的表型的子代植物。在一个或另一个实施方案中,该方法还包括分析植物或子代植物的至少一个其他遗传标志的步骤。在另一方面,本发明提供了使用根据本发明的方法生产的植物。在另一方面,本发明提供了根据本发明的多核苷酸,根据本发明的核酸构建体和/或根据本发明的载体(vector)用于产生重组细胞和/或转基因植物的用途。在一个实施方案中,当与缺乏外源多核苷酸、核酸构建体和/或载体(vector)的相应植物相比时,转基因植物具有如上定义的改变的表型。在另一方面,本发明提供了用于鉴定植物的方法,该植物包括根据本发明的多核苷酸,或根据本发明的任何一种核酸构建体中定义的多核苷酸,该方法包括以下步骤:i)从植物获得核酸样本,以及ii)筛选样品中是否存在多核苷酸。在一个实施方案中,当与缺乏外源多核苷酸的相应植物相比时,多核苷酸的存在表明植物具有如上定义的改变的表型。在一个或另一个实施方案中,方法鉴定根据本发明的植物。在一个或另一个实施方案中,方法还包括在步骤i)之前从种子生产植物。在另一方面,本发明提供了根据本发明的植物的植物部分。在一个实施方案中,植物部分是种子,其包括根据本发明的多核苷酸,或根据本发明的任何一种核酸构建体中定义的多核苷酸。在另一方面,本发明提供了生产植物部分的方法,方法包括:a)培育根据本发明的植物,以及b)收获植物部分。在另一方面,本发明提供了从种子生产获得面粉、全麦面粉、淀粉、油、种子粉或其他产品的方法,方法包括:a)获得根据本发明的种子,以及b)提取面粉、全麦面粉、淀粉、油或其他产品,或生产种子粉。在另一方面,本发明提供由根据本发明的植物和/或植物部分生产的产品。在一个实施方案中,该部分是种子。在一个或另一个实施方案中,产品是食品或饮料产品。优选地,i)食品选自由以下组成的组:面粉、淀粉、油、膨松或未膨松的面包、意大利面、面条、动物饲料、早餐谷类食品、零食食品、蛋糕、麦芽、甜点和含有基于面粉酱汁的食品,或ii)饮料产品是果汁、啤酒或麦芽。这些产品的生产方法通常为本领域技术人员已知。在另一个实施方案中,产品是非食品。非食品的实例包括但不限于膜、涂层、粘合剂、建筑材料和包装材料。这些产品的生产方法通常为本领域技术人员已知。在另一方面,本发明提供了制备根据本发明的食品的方法,该方法包括将种子或面粉、全麦面粉或来自种子的淀粉与另一种食品配料混合。另一方面,本发明提供了制备麦芽的方法,包括根据本发明使种子发芽的步骤。在另一方面,本发明提供了根据本发明的植物或其部分作为动物饲料,或生产用于动物消费的饲料或用于人类消费的食物的用途。另一方面,本发明提供了一种组合物,其包括根据本发明的融合多肽或加工的nif多肽、根据本发明的多核苷酸、根据本发明的核酸构建体、根据本发明的载体(vector)或根据本发明的细胞,以及一种或多种可接受的载体(carrier)。另一方面,本发明提供了在植物细胞中重建固氮酶蛋白复合物的方法,该方法包括将两种或多种根据本发明的多核苷酸、两种或多种根据本发明的核酸构建体和/或根据本发明的载体(vector)引入细胞,并培养植物细胞足够的时间以表达多核苷酸或载体(vector)。在另一个方面,本发明提供了增加植物的产量、生物质、生长速率、活力、源自生物固氮的氮增益、氮利用效率、非生物胁迫抗性和/或对营养缺乏的抗性的方法,包括将两种或更多种根据本发明的多核苷酸、两种或更多种根据本发明的核酸构建体,和/或根据本发明的载体(vector)引入植物或植物细胞。本发明不限于本文描述的特定实施方案的范围,这些实施方案仅用于示例性目的。如本文所述,功能等同的产品、组合物和方法显然在本发明的范围内。在整个说明书中,除非另有特别说明或上下文另有要求,提及单个步骤、物质组成、步骤组或物质组成组应被认为包括一个和多个(即一个或更多个)那些步骤、物质组成、步骤组或物质组成组。以下通过以下非限制性实施例并参考附图描述本发明。附图说明图1—pcw440的t-dna遗传图谱。标记物:35sp,全长camv35s启动子,显示转录方向;t7启动子,t7rna聚合酶启动子;dcoxivmtp,dcoxiv线粒体靶向肽;asci,用于插入与mtp框内融合的asci的限制性酶切位点;t7终止子,t7rna聚合酶的转录终止区;修饰的nospolya,nos3'聚腺苷酸化信号。图2—细菌和本生烟叶片中nifh、nifd、nifk和nify融合多肽的westernblot分析照片,每个多肽被融合到dcoxivmtp和包括ha或flag表位的c端延伸区。泳道内容物:1.对照叶片提取物(仅p19压渗)和多肽分子量标志物;实心箭头=72kda,紧接着的下方箭头(未实心)为55、40、33、25和17kda;泳道2至5,在细菌bl21-gold(泳道2)或两种不同的叶片压渗物(泳道3、4)中表达的pcw446dcoxiv-nifh-ha;泳道5至7,在细菌(泳道5)或两种不同的叶片压渗物(泳道6、7)中表达的pcw447-dcoxiv-nifd-flag;泳道8至10,在细菌(泳道8)或两种不同的叶片压渗物(泳道9、10)中表达的pcw448-dcoxiv-nifk-ha;泳道11至13,在细菌(泳道11)或两种不同的叶片压渗物(泳道12、13)中表达的pcw449-dcoxiv-nify-ha;泳道14至15,在叶片压渗物中表达的四种载体(vector)pcw446、pcw447、pcw448和pcw449的组合。泳道15还包括表达定位于细胞质的lbh的载体(vector)pcw444的压渗物。印迹(blot)a用抗ha和抗flag两种第一抗体探测;印迹b仅用抗ha探测。印记a的较长曝光时间(泳道14)揭示了nifh、nifk和nify多肽但不是nifd的信号。图3—用于在本生烟叶片中瞬时表达pfaγ::gfp融合多肽的结构示意图。野生型pfaγ氨基酸序列如上所示,突变氨基酸序列(mfaγ)如下所示。箭头表示mpp切割的预测点。35spr:camv35s启动子;t7p:t7rna聚合酶启动子;mtp:pfaγ或mfaγ区;gfp:gfp多肽;t7t:t7rna聚合酶转录终止子;nos:nos基因的3'转录终止子/聚腺苷酸化区。图4—在对表达编码pfaγ::niff::ha或pfaγ::nifz::flag融合多肽的构建体的本生烟或大肠杆菌细胞的蛋白提取物进行sds-page后,用ha(左组)或flag(右组)抗体探测的westernblot的照片。泳道上方的+和-符号分别表示应用于泳道的本生烟或大肠杆菌蛋白提取物的存在或不存在;用于细菌提取物的提取物稀释因子如括号中所示。图5—用于在本生烟叶片细胞中表达pfaγ::nifh::ha的基因构建体的示意图。核苷酸序列中加下划线的残基表示胰蛋白酶的蛋白水解切割位点(羧基侧)。箭头表示线粒体加工肽酶(mpp)的切割点。肽istqvvristqvvr(seqidno:44)和avqgaptmr(seqidno:45)通过质谱分析检测。图6—在对表达编码pfaγ::nif::ha或pfaγ::nif::flag融合多肽的构建体的本生烟细胞的蛋白提取物进行sds-page后,用ha(左上组)或flag抗体(右上组)探测的westernblot的照片。泳道上方的字母(k、b、e、s等)表示包括在由基因构建体编码的融合多肽中的nif多肽。pfaγ::nifj::flag的印迹顶部附近的模糊条带由星号(*)表示。分子量标志(kda)的大小显示在左侧。下组显示考马斯亮蓝染色后相应的凝胶。图7—含有编码pfaγ::nifd::flag的相同构建体的本生烟叶片或大肠杆菌蛋白提取物的westernblot照片。用抗flag表位的抗体探测印迹。显示了第一泳道中标志物的分子量。大肠杆菌提取物的稀释因子如括号中所示。图8—含有编码pfaγ::nifd::ha或mfaγ::nifd::ha或pfaγ::nifk::ha和pfaγ::gfp的构建体的大肠杆菌或本生烟叶片的蛋白提取物分别在第2和第3泳道中作为阳性和阴性对照的westernblot照片。在每个泳道上标明使用的构建体和编码的nif多肽(d或k)。用抗ha表位的抗体探测印迹。显示了第一泳道中标志物的分子量。下组显示考马斯亮蓝染色的凝胶。考马斯亮蓝染色凝胶中的主要条带是来自本生烟叶片样品的核酮糖二磷酸羧化酶-加氧酶(rubisco)。图9—nifk和nifh多肽的共表达提高了植物原位nifd融合多肽水平。将构建体引入叶片细胞以共表达nifd、nifk和nifh融合多肽后产生的多肽的westernblot照片。泳道1,pra24+pra10+pra25;泳道2,pra22+pra10+pra25;泳道3,pra19+pra10+pra25;泳道4,pra19+pra25;泳道5,pra10+pra25;泳道6,pra10;泳道7,pra11;泳道8,pra25;泳道9,pra22;泳道10,pra24;泳道11,pra19;泳道12,分子量标志物,右边数字表示kda。参见表4的构建体和编码的多肽的列表,也在印迹的顶部示出。泳道1中的星号表示nifd特异性条带的位置。图10—将构建体组合引入叶片细胞以表达nifd::flag、nifk(无c端延伸区)、nifs::ha和nifh::ha融合多肽后产生的多肽的westernblot照片。上组用抗flag抗体探测,下组用抗ha抗体探测。泳道1和12显示具有指定大小(kda)的分子量标志物。泳道2中的单个星号表示nifd特异性条带位置,双星号表示较小断裂nifd::flag多肽或内部翻译起始多肽的位置。括号中的数字表示相对于背景flag条带的nifd特异性条带数量。nifk::ha、nifs::ha和nif::h融合多肽的位置示于下组。构建体和编码的多肽的组合显示在每个泳道的顶部。缩写:d-flag(pra07;pfaγ::nifd::flag),h-ha(pra10;pfaγ::nifh::ha),k(pra25;pfaγ::nifk),k-ha(pra11,pfaγ::nifk::ha),s-ha(pra16;pfaγ::nifs::ha),d-ha(pra19,pfaγ::nifd::ha)。图11—将构建体单独或组合引入叶片细胞以表达nifd::flag、nifk(无c端延伸区)或nifk::ha和nifh::ha融合多肽后产生的多肽的westernblot照片。引入的构建体显示在每个泳道的上方,编码的融合多肽显示在每个泳道的下方。切割印迹,用抗ha抗体(泳道1至7)或抗flag抗体(泳道9至12)探测。星号表示泳道11和12中的nifd::flag特异性条带。westernblot需要更长的曝光时间以观察pra19和pra07(泳道2和3)的模糊nifd条带。图12—nifd::接头::nifk异二聚体的模型,分别以绿色和蓝色显示nifd和nifk亚基。空间填充模型显示接头为红色,将nifd的c端连接到nifk的n端。图13—nifd::接头::nifk融合多肽作为二聚体与两个nifh多肽络合的同源模型,使用用于nifd(绿色)和nifk(蓝色)的肺炎克雷伯菌氨基酸序列,包括用于nifh亚基(紫色)的维涅兰德固氮菌氨基酸序列。设计的接头以红色范德华原子表示显示。左右图像绕垂直轴相对于彼此旋转90度。图14—上组:使用抗体检测由pra01(泳道2,gfp)、pra11(泳道3,pfaγ::nifk::ha)、pra19(泳道4,pfaγ::nifd::ha)和pra20(泳道5,pfaγ::nifd-接头(flag)-nifk::ha)产生的多肽ha表位的westernblot照片。分子量标志物(泳道1,kda)的大小显示在左侧。下组显示考马斯亮蓝染色的凝胶。图15—多表达盒载体(multi-cassettevector)pkt100和pkt-hc的结构示意图。lb:t-dna的左边界;rb:右边界;p1-5/t1-5:表达盒1-5的启动子/终止子。ps/ts:含有选择基因的表达盒的启动子/终止子;mar:基质相关区域。限制性酶切位点的位置用箭头标出。图16—上组:对来自已接受基因构建体的本生烟叶片样品的蛋白提取物进行凝胶电泳后,用抗ha和抗gfp抗体联合探测的westernblot照片。泳道1,多肽分子量标志物(kda)。引入的载体(vector):泳道2,pra01(pfaγ::gfp);泳道3-7,表达来自表达盒1-5的pfaγ::nifh::ha的载体(vector),以及pra01。下组:同膜丽春红(ponceau)染色。指示了pfaγ::nifh::ha条带和pfaγ::gfp双条带。图17-a.上组:对来自已接受基因构建体的本生烟叶片样品的蛋白提取物进行凝胶电泳后,用抗ha抗体探测的westernblot照片。泳道1和泳道3,多肽分子量标志物(kda)。引入的载体(vector):泳道2,pra01(pfaγ::gfp);泳道4,hc13。下组:同膜丽春红(ponceau)染色。指示了条带的同一性。b.用抗myc抗体探测相同的westernblot。图18.上组:在压渗pra01(pfaγ::gfp,g标记的泳道)、pra16(pfaγ::nifs::ha,s标记的泳道)和pra19(人类密码子优化的pfaγ::nifd::ha,d标记的泳道)的组合4天后,来自本生烟叶片的蛋白提取物的westernblot的照片。用抗ha和抗gfp抗体进行westernblot探测。为了溶解nifs和gfp而不出现过饱和,曝光受到限制。nifd::ha检测需要更长的印迹曝光时间。泳道1指示分子量标志物(kda)的大小。下组:在以pra01(pfaγ::gfp,g标记的泳道)、pra22(人密码子优化的mfaγ::nifd::ha,d-mmtp标记的泳道)、pra19(人密码子优化的pfaγ::nifd::ha,d-ha标记的泳道)、pra26(人密码子优化的δfaγ::nifd::ha,d-终止(d-stop)标记的泳道)和pra24(拟南芥密码子优化的pfaγ::nifd::ha,d-alt标记的泳道)的组合压渗4天后,来自本生烟叶片的蛋白提取物的westernblot的照片。用抗ha和抗gfp抗体探测印迹。nifd-ha检测需要更长的印迹曝光时间。图19—以共表达p19的构建体和表达nifd::ha(有或无pra25)的sn6、sn7或sn8构建体(实施例20)压渗后,对本生烟叶片细胞的蛋白提取物进行sda-page后,使用抗ha抗体进行westernblot的照片。unpro/pro:faγ51::nifd的未加工或加工形式。degrprod:大约48kda大小的nifd特异性降解产物。序列表的关键字seqidno:1-细胞色素c氧化酶亚基iv(coxivmtp)的天然mtp的氨基酸序列。seqidno:2-细胞色素c氧化酶亚基iv(dcoxiv)的衍生mtp的氨基酸序列。seqidno:3-参与mtp的导入和加工的dcoxiv的基序xrxxxssx中保守的精氨酸和丝氨酸残基。seqidno:4-pcw440的t-dna区域的核苷酸序列,具有组件t-dna右边界(核苷酸1-164)、侧接hindiii位点和xhoi位点的35s启动子(核苷酸219-1564)、ctcgag(xhoi)、t7启动子(核苷酸1571-1587)、以atg开始的dcoxiv编码序列(核苷酸1650-1742)、asci位点(核苷酸1743-1750)、t7终止子(核苷酸1810-1856)、nos3'终止子(核苷酸1861-2084)和t-dna左边界(核苷酸2186-2346)。seqidno:5-野生型肺炎克雷伯菌nifh的氨基酸序列。seqidno:6-野生型肺炎克雷伯菌nifd的氨基酸序列。seqidno:7-野生型肺炎克雷伯菌nifk的氨基酸序列。seqidno:8-野生型肺炎克雷伯菌nify的氨基酸序列。seqidno:9-野生型肺炎克雷伯菌nifb的氨基酸序列。seqidno:10-野生型肺炎克雷伯菌nife的氨基酸序列。seqidno:11-野生型肺炎克雷伯菌nifn的氨基酸序列。seqidno:12-野生型肺炎克雷伯菌nifq的氨基酸序列。seqidno:13-野生型肺炎克雷伯菌nifs的氨基酸序列。seqidno:14-野生型肺炎克雷伯菌nifu的氨基酸序列。seqidno:15-野生型肺炎克雷伯菌nifx的氨基酸序列。seqidno:16-野生型肺炎克雷伯菌niff的氨基酸序列。seqidno:17-野生型肺炎克雷伯菌nifz的氨基酸序列。seqidno:18-野生型肺炎克雷伯菌nifj的氨基酸序列。seqidno:19-野生型肺炎克雷伯菌nifm的氨基酸序列。seqidno:20-野生型肺炎克雷伯菌nifv的氨基酸序列。seqidno:21-包括ha表位(氨基酸7-15)的c端延伸区(17aa)的氨基酸序列。seqidno:22-包括flag表位的c端延伸区(12aa)的氨基酸序列。seqidno:23-pcw446编码的dcoxiv::nifh::ha融合多肽的氨基酸序列。氨基酸1-31对应于dcoxivmtp,氨基酸32-34是在asci位点克隆的结果,氨基酸35-326是肺炎克雷伯菌nifh氨基酸(去除起始密码子met),以及氨基酸327-343包括ha表位。seqidno:24-pcw447编码的dcoxiv::nifd::flag融合多肽的氨基酸序列。氨基酸1-31对应于dcoxivmtp,氨基酸32-34是在asci位点克隆的结果,氨基酸35-515是肺炎克雷伯菌nifd氨基酸(去除n端met),以及氨基酸516-527包括flag表位。seqidno:25-pcw448编码的dcoxiv::nifk::ha融合多肽的氨基酸序列。氨基酸1-31对应于dcoxivmtp,氨基酸32-34是在asci位点克隆的结果,氨基酸35-553是肺炎克雷伯菌nifk氨基酸,以及氨基酸554-570包括ha表位。seqidno:26-pcw449编码的dcoxiv::nifk::ha融合多肽的氨基酸序列。氨基酸1-31对应于dcoxivmtp,氨基酸32-34是在asci位点克隆的结果,氨基酸35-253是肺炎克雷伯菌nify氨基酸(无起始密码子met),以及氨基酸254-270包括ha表位。seqidno:27-编码nifh::ha、asci-nifh-ha-asci的asci片段的核苷酸序列。seqidno:28-编码nifd::flag、asci-nifd-flag-asci的asci片段的核苷酸序列。seqidno:29-编码nifk::ha、asci-nifk-ha-asci的asci片段的核苷酸序列。seqidno:30-编码nify::ha、asci-nify-ha-asci的asci片段的核苷酸序列。seqidno:31-pcw452编码的dcoxiv::nifb::ha融合多肽的氨基酸序列。氨基酸1-31对应于dcoxivmtp,氨基酸32-34是在asci位点克隆的结果,氨基酸35-501是肺炎克雷伯菌nifb氨基酸(无起始密码子met),以及氨基酸502-518包括ha表位。seqidno:32-pcw454编码的dcoxiv::nife::ha融合多肽的氨基酸序列。氨基酸1-31对应于dcoxivmtp,氨基酸32-34是在asci位点克隆的结果,氨基酸35-490是肺炎克雷伯菌nife氨基酸(无起始密码子met),以及氨基酸491-507包括ha表位。seqidno:33-pcw455编码的dcoxiv::nifn::flag融合多肽的氨基酸序列。氨基酸1-31对应于dcoxivmtp,氨基酸32-34是在asci位点克隆的结果,氨基酸35-493是肺炎克雷伯菌nifn氨基酸(无起始密码子met),以及氨基酸494-505包括flag表位。seqidno:34-pcw456编码的dcoxiv::nifq::ha融合多肽的氨基酸序列。氨基酸1-31对应于dcoxivmtp,氨基酸32-34是在asci位点克隆的结果,氨基酸35-200是克雷伯氏菌属(klebsiellasp.)nifq氨基酸(无起始密码子met),以及氨基酸201-217包括ha表位。seqidno:35-pcw450编码的dcoxiv::nifs::ha融合多肽的氨基酸序列。氨基酸1-31对应于dcoxivmtp,氨基酸32-34是在asci位点克隆的结果,氨基酸35-433是肺炎克雷伯菌nifs氨基酸(无起始密码子met),以及氨基酸434-450包括ha表位。seqidno:36-pcw451编码的dcoxiv::nifu::flag融合多肽的氨基酸序列。氨基酸1-31对应于dcoxivmtp,氨基酸32-34是在asci位点克隆的结果,氨基酸35-493是肺炎克雷伯菌nifu氨基酸(无起始密码子met),以及氨基酸494-505包括flag表位。seqidno:37-pcw453编码的dcoxiv::nifx::flag融合多肽的氨基酸序列。氨基酸1-31对应于dcoxivmtp,氨基酸32-34是在asci位点克隆的结果,氨基酸35-189是肺炎克雷伯菌nifx氨基酸(无起始密码子met),以及氨基酸190-201包括flag表位。seqidno:38-包括pfaγmtp(氨基酸1-77)和氨基酸三联体gap(78-80)的n端延伸区的氨基酸序列作为克隆策略的结果添加到pra00中。mpp切割发生在氨基酸残基42和43之间。seqidno:39-在载体(vector)pra21中编码、包括mfaγ序列(氨基酸1-77)和氨基酸三联体gap(78-80)的修饰的n端延伸区的氨基酸序列作为克隆策略的结果添加到pra21中。相对于seqidno:38,在氨基酸12-18、24-33和39-45处的丙氨酸氨基酸替换被设计为用来消除mpp的切割。seqidno:40-pra05编码的pfaγ::niff::ha融合多肽的氨基酸序列。氨基酸1-77对应于pfaγmtp,氨基酸78-80(gap)是在asci位点克隆肺炎克雷伯菌nifx的结果,氨基酸81-256是肺炎克雷伯菌niff氨基酸(seqidno:16)以及氨基酸257-267包括ha表位。seqidno:41-pra04编码的pfaγ::nifz::flag融合多肽的氨基酸序列。氨基酸1-77对应于pfaγmtp,氨基酸78-80(gap)是在asci位点克隆的结果,氨基酸81-228是肺炎克雷伯菌nifz氨基酸(seqidno:17),以及氨基酸229-238包括flag表位。seqidno:42-pra10编码的pfaγ::nifh::ha融合多肽的氨基酸序列。氨基酸1-77对应于pfaγmtp,氨基酸78-80(gap)是在asci位点克隆的结果,氨基酸81-372是肺炎克雷伯菌nifh氨基酸(seqidno:5,无起始子met),以及氨基酸373-389包括ha表位。seqidno:43-未加工的pfaγ中的胰蛋白酶消化肽段的氨基酸序列。seqidno:44-mpp加工后的pfaγ中的胰蛋白酶消化肽段的氨基酸序列。seqidno:45-mpp加工后的pfaγ中的胰蛋白酶消化肽段的氨基酸序列。seqidno:46-pra03编码的pfaγ::nifb::ha融合多肽的氨基酸序列。氨基酸1-77对应于pfaγmtp,氨基酸78-80(gap)是在asci位点克隆的结果,氨基酸81-547是肺炎克雷伯菌nifb氨基酸(seqidno:9,无起始子met),以及氨基酸548-564包括ha表位。seqidno:47-pra07编码的pfaγ::nifd::flag融合多肽的氨基酸序列。氨基酸1-77对应于pfaγmtp,氨基酸78-80(gap)是在asci位点克隆的结果,氨基酸81-561是肺炎克雷伯菌nifd氨基酸(seqidno:6,无起始子met和后续met),以及氨基酸562-573包括flag表位。seqidno:48-pra09编码的pfaγ::nife::ha融合多肽的氨基酸序列。氨基酸1-77对应于pfaγmtp,氨基酸78-80(gap)是在asci位点克隆的结果,氨基酸81-536是肺炎克雷伯菌nife氨基酸(seqidno:10,无起始子met),以及氨基酸537-553包括ha表位。seqidno:49-pra06编码的pfaγ::nifj::flag融合多肽的氨基酸序列。氨基酸1-77对应于pfaγmtp,氨基酸78-80(gap)是在asci位点克隆的结果,氨基酸81-1251是肺炎克雷伯菌nifj氨基酸(seqidno:18),以及氨基酸1252-1261包括flag表位。seqidno:50-pra11编码的pfaγ::nifk::ha融合多肽的氨基酸序列。氨基酸1-77对应于pfaγmtp,氨基酸78-80(gap)是在asci位点克隆的结果,氨基酸81-599是肺炎克雷伯菌nifk氨基酸(seqidno:7,无起始子met),以及氨基酸600-616包括ha表位。seqidno:51-pra18编码的pfaγ::nifm::ha融合多肽的氨基酸序列。氨基酸1-77对应于pfaγmtp,氨基酸78-80(gap)是在asci位点克隆的结果,氨基酸81-346是肺炎克雷伯菌nifm氨基酸(seqidno:19),以及氨基酸347-357包括ha表位。seqidno:52-pra13编码的pfaγ::nifn::flag融合多肽的氨基酸序列。氨基酸1-77对应于pfaγmtp,氨基酸78-80(gap)是在asci位点克隆的结果,氨基酸81-539是肺炎克雷伯菌nifn氨基酸(seqidno:11,无起始子met),以及氨基酸540-551包括flag表位。seqidno:53-pra08编码的pfaγ::nifq::ha融合多肽的氨基酸序列。氨基酸1-77对应于pfaγmtp,氨基酸78-80(gap)是在asci位点克隆的结果,氨基酸81-246是肺炎克雷伯菌nifq氨基酸(seqidno:12,无起始子met),以及氨基酸247-263包括ha表位。seqidno:54-pra16编码的pfaγ::nifs::ha融合多肽的氨基酸序列。氨基酸1-77对应于pfaγmtp,氨基酸78-80(gap)是在asci位点克隆的结果,氨基酸81-478是肺炎克雷伯菌nifs氨基酸(seqidno:13,无起始子met),以及氨基酸479-496包括ha表位。seqidno:55-pra15编码的pfaγ::nifu::flag融合多肽的氨基酸序列。氨基酸1-77对应于pfaγmtp,氨基酸78-80(gap)是在asci位点克隆的结果,氨基酸81-353是肺炎克雷伯菌nifu氨基酸(seqidno:14,无起始子met),以及氨基酸354-365包括flag表位。seqidno:56-pra17编码的pfaγ::nifv::flag融合多肽的氨基酸序列。氨基酸1-77对应于pfaγmtp,氨基酸78-80(gap)是在asci位点克隆的结果,氨基酸81-461是肺炎克雷伯菌nifv氨基酸(seqidno:20),以及氨基酸462-471包括flag表位。seqidno:57-pra14编码的pfaγ::nifx::flag融合多肽的氨基酸序列。氨基酸1-77对应于pfaγmtp,氨基酸78-80(gap)是在asci位点克隆的结果,氨基酸81-235是肺炎克雷伯菌nifx氨基酸(seqidno:15,无起始子met),以及氨基酸236-247包括flag表位。seqidno:58-pra12编码的pfaγ::nify::ha融合多肽的氨基酸序列。氨基酸1-77对应于pfaγmtp,氨基酸78-80(gap)是在asci位点克隆的结果,氨基酸81-299是肺炎克雷伯菌nify氨基酸(seqidno:8,无起始子met),以及氨基酸300-316包括ha表位。seqidno:59-pra19编码的pfaγ::nifd::ha融合多肽的氨基酸序列。氨基酸1-77对应于pfaγmtp,氨基酸78-80(gap)是在asci位点克隆的结果,氨基酸81-561是肺炎克雷伯菌nifd氨基酸(seqidno:6,无起始子met和后续met),以及氨基酸562-574包括ha表位。seqidno:60-pfaγ::nifk融合多肽(pra25)的氨基酸序列,缺少任意c端延伸区。氨基酸1-77对应于pfaγmtp,氨基酸78-80(gap)是在asci位点克隆的结果,氨基酸81-599是肺炎克雷伯菌nifk氨基酸(seqidno:7,无起始子met)。seqidno:61-来自红褐肉座菌(hypocreajecorina)外切纤维素酶ii的已知非结构化接头区的11个残基片段的氨基酸序列(登录号:aag39980.1)。seqidno:62-8残基flag表位的氨基酸序列。seqidno:63-接头的氨基酸序列。接头长度为30个残基,由来自红褐肉座菌外切纤维素酶ii的已知非结构化接头区的11个残基组成(登录号:aag39980.1,atpppgstttr;seqidno:61),其中最后的精氨酸被丙氨酸替换,然后是8个残基的flag表位(dykddk;seqidno:62),最后是11个残基的非结构化接头序列的另一个拷贝,其中精氨酸被丙氨酸替换。seqidno:64-pfaγ::nifd-接头-nifk融合多肽(pra02)的氨基酸序列,缺少nifk的任意c端延伸区。氨基酸1-77对应于pfaγmtp,氨基酸78-80(gap)是在asci位点克隆的结果,氨基酸81-561是肺炎克雷伯菌nifd氨基酸(seqidno:6,无起始子met和后续met),氨基酸562-592是30个氨基酸接头,以及氨基酸593-1110是肺炎克雷伯菌nifk氨基酸(seqidno:7,无起始子met)。seqidno:65-pfaγ::nifd-接头-nifk::ha融合多肽(pra20)的氨基酸序列,缺少nifk的任意c端延伸区。氨基酸1-77对应于pfaγmtp,氨基酸78-80(gap)是在asci位点克隆的结果,氨基酸81-561是肺炎克雷伯菌nifd氨基酸(seqidno:6,无起始子met和后续met),氨基酸562-592是30氨基酸接头,氨基酸593-1110是肺炎克雷伯菌nifk氨基酸(seqidno:7,无起始子met),以及氨基酸1111-1119包括ha表位。seqidno:66-包括myc表位的c端延伸区的氨基酸序列,对应于seqidno:67的氨基酸347-356。seqidno:67-pfaγ::nifm::myc融合多肽的氨基酸序列。seqidno:68-pra19中编码pfaγ::nifd::ha融合多肽的核苷酸序列,起始于翻译起始密码子。seqidno:69-来自肺炎克雷伯菌的nifk多肽c端最后四个氨基酸残基的氨基酸序列。seqidno:70.编码pfaγ-c多肽的dna片段的核苷酸序列。seqidno:71.pfaγ-c多肽的氨基酸序列。seqidno:72.寡核苷酸引物nifd-f。seqidno:73.寡核苷酸引物nifd-r。seqidno:74.野生型肺炎克雷伯菌nifw的氨基酸序列。seqidno:75.mtp-faγ51多肽的氨基酸序列。seqidno:76.faγ-scar9多肽的氨基酸序列。seqidno:77.cpn60mtp的氨基酸序列。seqidno:78.cpn60/无gg接头mtp的氨基酸序列。seqidno:79.超氧化物歧化酶(sod)mtp(at3g10920)的氨基酸序列。seqidno:80.超氧化物歧化酶加倍的(2sod)mtp(at3g10920)的氨基酸序列。seqidno:81.超氧化物歧化酶修饰的(sod)mtp(at3g10920)的氨基酸序列。seqidno:82.超氧化物歧化酶修饰的(2sodmod)加倍的mtp(at3g10920)的氨基酸序列。seqidno:83.l29mtp的氨基酸序列(at1g07830)。seqidno:84.粗糙脉孢菌(neurosporacrassa)f0atp酶亚基9(su9)mtp的氨基酸序列。seqidno:85.gatp酶γ亚基(faγ51)mtp的氨基酸序列。seqidno:86.coxivtwinstrep(abm97483)mtp的氨基酸序列。seqidno:87.coxiv10×his(abm97483)mtp的氨基酸序列。seqidno:88.超氧化物歧化酶(sod)mtp的预测的scar的氨基酸序列(seqidno:80)。seqidno:89.超氧化物歧化酶加倍的(2sod)mtp的预测的scar的氨基酸序列(seqidno:81)。seqidno:90.l29mtp的预测的scar的氨基酸序列(seqidno:84)。seqidno:91.粗糙脉孢菌f0atp酶亚基9(su9)mtp的预测的scar的氨基酸序列(seqidno:85)。seqidno:92.gatp酶γ亚基(faγ51)mtp的预测的scar的氨基酸序列(seqidno:86)。seqidno:93.coxivtwinstrepmtp的预测的scar的氨基酸序列(seqidno:87)。seqidno:94.coxiv10×hismtp的预测的scar的氨基酸序列(seqidno:88)。seqidno:95.变异体nifd的氨基酸序列。seqidno:96.变异体nifs的氨基酸序列。seqidno:97.nifk9氨基酸c端延伸区。seqidno:98.寡核苷酸引物。seqidno:99.寡核苷酸引物。seqidno:100.寡核苷酸引物。seqidno:101.寡核苷酸引物。seqidno:102.寡核苷酸引物bo1。seqidno:103.寡核苷酸引物bo2。seqidno:104.接头的氨基酸序列。seqidno:105.寡核苷酸引物pra31dk-fw。seqidno:106.寡核苷酸引物pra31dk-rv。seqidno:107.寡核苷酸引物d_起始_rv。seqidno:108.寡核苷酸引物k_终止_fw。seqidno:109.寡核苷酸引物。seqidno:110.寡核苷酸引物。seqidno:111.nifd-接头-nifk多肽9氨基酸序列n端延伸区seqidno:112.ha表位氨基酸序列。seqidno:113.nife-nifnha多肽接头氨基酸序列。seqidno:114.寡核苷酸引物praen-fw。seqidno:115.寡核苷酸引物praen-rv。seqidno:116.寡核苷酸引物e_起始_rv。seqidno:117.寡核苷酸引物n_终止_fw。seqidno:118.寡核苷酸引物。seqidno:119.寡核苷酸引物。seqidno:120.scsv-s4版本1启动子的核苷酸序列。seqidno:121.scsv-s4版本2启动子的核苷酸序列。seqidno:122.scsv-s7启动子的核苷酸序列。seqidno:123.camv-35s长版本启动子的核苷酸序列。seqidno:124.camv-2×25s启动子的核苷酸序列。seqidno:125.p19病毒抑制蛋白的氨基酸序列。seqidno:126.p19肽的氨基酸序列。seqidno:127.p19肽的氨基酸序列。seqidno:128-143.nifk肽的氨基酸序列。seqidno:144-154.nifh肽的氨基酸序列。seqidno:155-160.nifb肽的氨基酸序列。seqidno:161-165.nifj肽的氨基酸序列。seqidno:166-174.nifs肽的氨基酸序列。seqidno:175-182.nifx肽的氨基酸序列。seqidno:183-186.niff肽的氨基酸序列。具体实施方式一般技术和定义除非另有明确定义,否则本文使用的所有技术和科学术语均应被视为具有与本领域(例如在细胞培养、分子遗传学、植物分子生物学、蛋白质化学和生物化学领域)普通技术人员通常理解的相同含义。除非另有说明,本发明中使用的重组蛋白、细胞培养和免疫技术都是标准程序,为本领域技术人员熟知。这些技术在以下来源的文献中都有描述和解释,例如,约翰·威利父子出版公司出版的《分子克隆实用指南》(j.perbal,apracticalguidetomolecularcloning,johnwileyandsons(1984));冷泉港实验室出版社出版的j.sambrook等人的《分子克隆:实验室手册》(j.sambrooketal.,molecularcloning:alaboratorymanual,coldspringharbourlaboratorypress(1989));irl出版社出版的t.a.brown(编者)的《基本分子生物学:实用方法》第1卷和第2卷(t.a.brown(editor),essentialmolecularbiology:apracticalapproach,volumes1and2,irlpress(1991);irl出版社出版的d.m.glover和b.d.hames(编者)的《dna克隆:一种实用的方法》第1-4卷(d.m.gloverandb.d.hames(editors),dnacloning:apracticalapproach,volumes1-4,irlpress(1995和1996);格林出版联合公司与约翰威立父子出版公司出版的f.m.ausubel等人(编者)的《最新分子生物学实验方法汇编》(f.m.ausubeletal.(editors),currentprotocolsinmolecularbiology,greenepub.associatesandwiley-interscience)(1988,包括到目前为止的所有更新);冷泉港实验室的edharlow和davidlane(编者)的《抗体:实验室手册》(edharlowanddavidlane(editors),antibodies:alaboratorymanual,coldspringharbourlaboratory,(1988));以及约翰·威利父子出版公司出版的j.e.coligan等人(编者)的《免疫学实验指南》(currentprotocolsinimmunology,johnwiley&sons)(包括目前为止的所有更新)。术语“和/或”,例如“x和/或y”应理解为“x和y”或“x或y”,并应被视为对两种含义或其中一种含义提供明确支持。在整个说明书中,单词“包括(comprise)”或诸如“包括(comprises或comprising)”之类的变化将被理解为暗示包括所述元件、整体或步骤,或一组元件、整体或步骤,但不排除任何其他元件、整体或步骤,或一组元件、整体或步骤。固氮酶是真细菌和古细菌中的酶,其催化氮(n2)的强三键还原产生氨(nh3)。固氮酶仅天然地存在于细菌中。其是可以分别被纯化地两种酶的复合物,即固二氮酶(dinitrogenase)和固二氮酶还原酶(dinitrogenasereductase)。固二氮酶,也称为组件i或钼-铁(mofe)蛋白,是两个nifd和两个nifk多肽(α2β2)的四聚体,其也含有两个“p簇”和两个“femo-辅因子”(femo-co)。每对nifd-nifk亚基含有一个p簇和一个femo-co。femo-co是由高柠檬酸分子络合的mofe3-s3簇组成的金属原子簇,该高柠檬酸分子与钼原子配位并通过三个硫配体桥接到fe4-s3簇上。femo-co分别在细胞中组装,然后掺入apo-mofe蛋白中。p簇也是金属原子簇,含有8个fe原子和7个硫原子,结构与femo-co相似但不同。p簇位于固二氮酶的αβ亚基界面,由两个亚基的半胱氨酰残基配位。固二氮酶还原酶,也称为组件ii或“fe蛋白”,是nifh多肽的二聚体,其在亚基界面也含有单个fe4-s4簇和两个mg-atp结合位点,每个亚基一个。该酶是固二氮酶的强制性电子供体,其中电子从fe4-s4簇转移到p簇,进而转移到n2还原的位点femo-co。尽管含有mo的固氮酶是细菌中最常见的固氮酶,但存在两种遗传上不同但具有相似辅因子和亚基组成的同源固氮酶,即分别由vnf(钒固氮)和anf(交替固氮)基因编码的含有钒固氮酶和仅含有fe的固氮酶。自然界中的一些细菌具有所有三种类型的固氮酶,其他细菌仅含有含mo和含v的酶或仅含有含mo的酶,例如肺炎克雷伯菌(klebsiellapneumoniae)。femo-co的生物合成和固氮酶组件催化活性形式的成熟需要多种固氮(nif)基因。已经描述了nifb、nife、nifh、nifn、nifq、nifv和nifx多肽在femo-co合成中的作用(rubio和ludden,2005)。由原核生物酶固氮酶催化的生物固氮是使用合成n2肥料的替代方案。固氮酶对氧的敏感性是工程化生物固氮(engineeringbiologicalnitrogenfixation)通过直接nif基因转移进入植物例如谷类作物中的主要障碍。本发明人认为将nif多肽靶向到植物细胞的线粒体基质(mm)可能克服氧敏感性问题。mm具有耗氧酶,其允许含有氧敏感fe-s簇的其他酶起作用。线粒体fe-s簇组装机器类似于固氮等价物(balk和pilon,2011;lill和mühlenhoff,2008)。因此,对固氮酶生物合成的一些必要条件可能已经存在于mm中,减少了重建所需nif基因的数量。辅因子高柠檬酸作为tca循环的一部分产生。atp(geigenberger和fernie,2014;mackenzie和mcintosh,1999)的还原电位和浓度也很高,这都是固氮酶催化的先决条件。另外,线粒体中谷氨酸合酶的存在为固氮酶将任何铵固定进入植物代谢提供了进入点。鉴于这些特征,以及线粒体自身是α-蛋白细菌来源的事实,本发明人认为该细胞器非常适合作为尝试功能性重建固氮酶的位置。作为在植物细胞线粒体中重建固氮酶的第一步,需要个体nif蛋白能够正确靶向mm的证据。为此目的,本发明人选择模型植物本生烟(nicotianabenthamiana)作为表达平台(wood等人,2009),单独或更重要地以组合方式提供转基因的表达。由于大多数mm定位的蛋白是核编码的,本发明人依靠使用先前表征的n端肽靶向信号(lee等人,2012)理解亚细胞信号传导和转移过程(huang等人,2009;murcha等人,2014)的最新进展。模型细菌固氮生物肺炎克雷伯菌使用16种独特的蛋白用于固氮酶的生物合成和催化功能。本发明人对来自肺炎克雷伯菌的所有16种nif蛋白进行了重新工程化用于靶向植物mm,并在本生烟叶片中评估它们的表达和加工。将所有16种nif多肽瞬时表达并测试序列特异性mm加工。本发明人已经确定,所有16种nif多肽都可以在植物叶片细胞中单独表达为mtp:nif融合多肽。此外,本发明人提供了这些蛋白可以靶向线粒体基质(mm)的证据,线粒体基质(mm)是潜在适应固氮酶功能的亚细胞定位定位(subcellularlocation),并且可以被线粒体加工蛋白酶(mpp)切割。这是第一次实际证明这种方法的可行性,并且代表了在植物中设计内源性固氮目标的重要进展。线粒体靶向肽(mtp)-nif融合多肽本发明涉及线粒体靶向肽(mtp)-nif融合多肽及其切割的多肽产物。当本发明的mtp-nif融合多肽在植物细胞中表达时,mtp-nif融合多肽和/或切割的多肽产物靶向线粒体基质(mm)。优选地,融合多肽促进植物细胞的固氮酶还原酶和/或固氮酶活性,或与细菌中相应野生型nif多肽促进的活性相同的活性。如本文所用,术语“融合多肽(fusionpolypeptide)”是指包括两个或更多个通过肽键共价连接的功能性多肽结构域的多肽。通常,融合多肽由本发明的嵌合多核苷酸编码为单多肽链。在一个实施方案中,本发明的融合多肽包括线粒体靶向肽(mtp)和nif多肽(np)。在该实施方案中,mtp的c端与np的n端翻译融合。在另一个实施方案中,本发明的融合多肽包括mtp的c端部分和np,其中c端部分由mpp切割mtp产生。在该实施方案中,mtp的c端部分的c端与np的n端翻译融合。如本文所用,术语“翻译融合到n端(translationallyfusedtothen-terminus)”是指mtp多肽的c端通过肽键共价连接到np的n端氨基酸,从而成为融合多肽。在一个实施方案中,相对于相应的野生型np,np不包括其天然翻译起始甲硫氨酸(met)残基或其两个n端met残基。在另一个实施方案中,np包括翻译起始met或野生型np多肽的两个n端met残基中的一个或两个,例如nifd。这种多肽通常通过表达嵌合蛋白编码区产生,其中编码mtp的核苷酸的翻译阅读框与编码np的核苷酸的阅读框在框内连接。本领域技术人员将理解mtp的c端可以在没有接头的情况下或通过一个或多个氨基酸残基(例如1-5个氨基酸残基)的接头与np的n端氨基酸翻译融合。这种接头也可以被认为是mtp的一部分。蛋白编码区表达之后可以在植物细胞的mm中切割mtp,并且这种切割(如果发生的话)包括在本发明融合多肽的生产概念中。融合多肽或加工的nif多肽优选具有与相应野生型nif多肽相当的功能性nif活性。融合多肽或加工的nif多肽的功能活性可以在细菌和生化互补试验(biochemicalcomplementatonassays)中测定。在优选的实施方案中,融合多肽或加工的nif多肽具有野生型nif活性的约70-100%的活性。融合多肽可以包括多于一种mtp和/或多于一种np,例如,融合多肽可以包括mtp、nifd多肽和nifk多肽。融合多肽还可以包括寡肽接头,例如连接两种np。优选地,接头具有足够的长度以允许两种或多种功能域,例如两种np,如nifd和nifk,在植物细胞中以功能构型缔合。这种接头的长度可以是8至50个氨基酸残基,优选约25至35个氨基酸,更优选约30个氨基酸残基。融合多肽可以通过常规方法获得,例如通过编码所述融合多肽的多核苷酸序列在合适细胞中的基因表达的方法。如本文所用,“基本上纯化的多肽(substantiallypurifiedpolypeptide)”是指基本上不含通常例如在细胞中与多肽缔合的成分(例如脂质、核酸、碳水化合物)的多肽。优选地,基本上纯化的多肽至少90%不含所述组件。本发明的植物细胞、转基因植物及其部分包括编码本发明多肽的多核苷酸。本发明的多肽不是天然存在于植物细胞中,特别是不存在于植物细胞的线粒体中,因此编码该多肽的多核苷酸在本文中可以称为外源多核苷酸,因为它不是天然存在于植物细胞中,而是被引入植物细胞或祖细胞(progenitorcell)中。因此,可以说产生本发明多肽的本发明的细胞、植物和植物部分产生重组多肽。在多肽的上下文中,术语“重组的”是指当细胞产生外源多核苷酸时由外源多核苷酸编码的多肽,该多核苷酸已经通过重组dna或rna技术例如转化引入细胞或祖细胞。通常,植物细胞、植物或植物部分包括非内源基因,其可在植物细胞或植物的生命周期中的至少一些时间引起一定量的多肽产生。在一个实施方案中,本发明的多肽不是天然存在的多肽。在另一个实施方案中,本发明的多肽是天然存在的,但存在于植物细胞中,优选存在于植物细胞的线粒体中,其中该多肽是非天然存在的。nif多肽本文所用术语“nif多肽”和“nif蛋白”可互换使用,是指氨基酸序列与涉及固氮酶活性的天然多肽相关的多肽,其中本发明的nif多肽选自nifd多肽、nifh多肽、nifk多肽、nifb多肽、nife多肽、nifn多肽、niff多肽、nifj多肽、nifm多肽、nifq多肽、nifs多肽、nifu多肽、nifv多肽、nifw多肽、nifx多肽、nify多肽和nifz多肽组成的组,各自如本文所定义。本发明的nif多肽包括“nif融合多肽”,本文所用的“nif融合多肽”是指天然存在的nif多肽的多肽同系物,相对于相应的天然存在的nif多肽,其具有与n端或c端或两者连接的额外氨基酸残基。如上所述,相对于相应的野生型nif多肽,nif融合多肽可能缺少翻译起始met或两个n端met残基。对应于天然存在的nif多肽的nif融合多肽的氨基酸残基,即没有连接到n端或c端或两者的额外氨基酸残基,在本文中也称为nif多肽,在这种情况下缩写为“np”,或称为nifd多肽(“nd”)等。在优选实施方案中,“连接到n端或c端或两者的额外氨基酸残基”包括连接到np的n端的线粒体靶向肽(mtp)或与np的n端连接的加工的mtp,或是np的n端或c端或两者的表位序列(“标签”),或mtp或加工的mtp和表位序列两者。天然存在的nif多肽仅存在于一些细菌中,包括固氮菌(nitrogen-fixingbacteria),包括自生固氮菌(freelivingnitrogenfixingbacteria)、联合固氮菌(associativenitrogenfixingbacteria)和共生固氮菌(symbioticnitrogenfixingbacteria)。自生固氮菌能够固定显著水平的氮,而不与其他生物体直接相互作用。非限制性地,所述自生固氮菌包括固氮菌属(azotobacter)、拜叶林克氏菌属(beijerinckia)、克雷伯氏菌属(klebsiella)、蓝藻属(cyanobacteria)(归类为好氧生物)的成员和梭状芽孢杆菌属(clostridium)、脱硫弧菌属(desulfovibrio)以及命名为紫色硫细菌、紫色非硫细菌和绿色硫细菌的成员。联合固氮细菌是能够与禾本科(杂草)的几个成员形成紧密联系的那些原核生物。这些细菌在宿主植物的根际内固定了相当数量的氮。固氮螺菌属(azospirillum)的成员是联合固氮细菌的代表。共生固氮菌是通过与宿主植物合作而共生固氮的那些细菌。植物通过光合作用提供糖分,固氮细菌利用这些糖分来提供固氮作用所需的能量。根瘤菌的成员是联合固氮细菌的代表。本发明的nif多肽或nif融合多肽选自由nifh、nifd、nifk、nifb、nife、nifn、niff、nifj、nifm、nifq、nifs、nifu、nifv、nifw、nifx、nify和nifz多肽组成的组。多肽或多肽类可以通过其氨基酸序列与参考氨基酸序列的同一性程度(%同一性),或通过与一种参考氨基酸序列比与另一种参考氨基酸序列具有更大的%同一性来定义。多肽或多肽类也可以通过具有与天然存在的nif多肽相同的生物活性来定义,除了序列的同一性程度。多肽的%同一性由gap(needleman和wunsch,1970)分析(gcg程序)确定,空位形成罚分=5,空位延伸罚分=0.3,或使用blastp2.5版或其更新版本(altschul等人,1997),其中在每种情况下,该分析将两个序列进行比对,包括参考序列整个长度上的参考序列。如本文所用,参考序列包括来自肺炎克雷伯菌的天然存在的nif多肽所示的参考序列,seqidno:1-20。在以下定义中,氨基酸序列与seqidno所示的参考序列的同一性程度由blastp2.5版或其更新版本(altschul等人,1997)确定,除目标序列最大数目设置为10,000,使用默认参数,且沿着参考氨基酸序列的全长确定。细菌中天然存在的nifh多肽是固氮酶复合物的结构组件,通常称为铁(fe)蛋白。它形成同二聚体,其中fe4s4簇结合在亚基和两个atp结合结构域之间。nifh是mofe蛋白(nifd/nifk异源四聚体)的专性电子供体,因此起固氮酶还原酶(ec1.18.6.1)的作用。nifh还参与femo-co生物合成和apo-mofe蛋白成熟。如本文所用,“nifh多肽”是指包括氨基酸的多肽,氨基酸的序列与seqidno:5所示的氨基酸序列至少具有41%同一性,并且其包括一个或多个结构域tigr01287、prk13236、prk13233和cd02040。tigr01287结构域存在于钼-铁固氮酶还原酶(nifh)、钒-铁固氮酶还原酶(vnfh)和铁-铁固氮酶还原酶(anfh)中的每一个中,但不包括来自非光依赖原叶绿素酸脂的同源蛋白。因此,本文使用的nifh多肽包括包含其序列与seqidno:5至少具有41%同一性的氨基酸的铁结合多肽的亚类,vnfh铁结合多肽和anfh铁结合多肽。天然存在的nifh多肽通常具有260-300个氨基酸的长度,天然单体具有约30kda的分子量。已经鉴定了大量的nifh多肽,并且在公众可获得的数据库中可获得许多序列。例如,已经报道了来自klebsiellamichiganensis(密歇根克雷伯菌,登录号wp_049123239.1,99%与seqidno:5同一性)、brenneriagoodwinii(wp_048638817.1,93%同一性)、sideroxydanslithotrophicus(wp_013029017.1,84%同一性)、denitrovibrioacetiphilus(wp_013010353.1,80%同一性)、desulfovibrioafricanus(非洲脱硫弧菌,wp_014258951.1,72%同一性)、chlorobiumphaeobacteroides(褐杆状绿菌,wp_011744626.1,69%同一性)、methanosaetaconcilii(甲烷鬓毛菌属,wp_013718497.1,64%同一性)、rhodobacter(红杆菌,wp_009565928.1,61%同一性)、methanocaldococcusinfernus(甲烷暖球菌,wp_013099472.1,42%同一性)和desulfosporosinusyoungiae(wp_007781874.1,41%同一性)的nifh多肽。nifh多肽已经在thiel等人(1997)、pratte等人(2006)、boison等人(2006)和staples等人(2007)中进行了描述和综述。如本文所用,功能性nifh多肽是能够与其他所需亚基(例如nifd和nifk)以及femo或其他辅因子一起形成功能性固氮酶蛋白复合物的nifh多肽。如本文所用,“nifd多肽”是指包括氨基酸的多肽,氨基酸的序列与如seqidno:6所示的氨基酸序列至少具有33%同一性,并且多肽包括(i)结构域tigr01282和cog2710中的一个或两个,两个结构域都存在于铁-钼结合多肽中,包括具有seqidno:6所示氨基酸序列的多肽,或(ii)铁-钒结合结构域tigr01860,其中nifd多肽存在于vnfd多肽的亚类中,或(iii)铁-铁结合结构域tigr1861,其中nifd多肽存在于anfd多肽的亚类中。如本文所用,nifd多肽包括铁-钼(femo-co)结合多肽的亚类,其包括序列与seqidno:6至少具有33%同一性的氨基酸、vnfd铁-钒多肽和anfd多肽。天然存在的nifd多肽通常具有470至540个氨基酸的长度。已经鉴定了大量的nifd多肽,并且在公众可获得的数据库中可获得许多序列。例如,已经报道了来自raoultellaornithinolytica(解鸟氨酸拉乌尔菌,登录号wp_044347161.1,与seqidno:6具有96%同一性)、kluyveraintermedia(中间克鲁瓦氏菌,wp_047370273.1,93%同一性)、dickeyadadantii(达旦提狄克氏菌,wp_038902190.1,89%同一性)、tolumonassp.brl6-1(甲苯单胞菌属brl6-1,wp_024872642.1,81%同一性)、magnetospirillumgryphiswaldense(格瑞菲斯瓦尔德磁螺菌,wp_024078601.1,68%同一性)、thermoanaerobacteriumthermosaccharolyticum(热解糖热厌氧杆菌,wp_013298320.1,42%同一性)、methanothermobacterthermautotrophicus(热自养甲烷热杆菌,wp_010877172.1,38%同一性)、desulfovibrioafricanus(非洲脱硫弧菌,wp_014258953.1,37%同一性)、desulfotomaculumsp.lma1(脱遛肠状菌属lma1,wp_066665786.1,37%同一性)、desulfomicrobiumbaculatum(杆状脱硫微菌,wp_015773055.1,36%同一性)、fischerellamuscicola(底栖蓝藻)的vnfd多肽(wp_016867598.1,34%同一性)和来自opitutaceaebacterium(丰祐菌)tav5的anfd多肽(wp_009512873.1,33%同一性)的nifd多肽。nifd多肽已经在lawson和smith(2002)、kim和rees(1994)、eady(1996)、robson等人(1989)、dilworth等人(1988)、dilworth等人(1993)、miller和eady(1988)、chiu等人(2001)、mayer等人(1999)和tezcan等人(2005)中进行了描述和综述。铁钼亚类的nifd多肽是固氮酶复合物的关键亚基,是固氮酶核心处α2β2mofe蛋白复合物的α亚基,是用femo辅因子还原底物的位点。如本文所用,功能性nifd多肽是能够与其他所需亚基(例如nifh和nifk)以及femo或其他辅因子一起形成功能性固氮酶蛋白复合物的nifd多肽。如本文所用,“nifk多肽”是指包括氨基酸的多肽,氨基酸的序列与如seqidno:7所示的氨基酸序列至少具有31%同一性,并且其包括保守结构域cd01974、tigr01286或cd01973中的一个或多个,在这种情况下,nifk多肽属于vnfk多肽的亚类。如本文所用,nifk多肽包括来自铁-钒固氮酶的vnfk多肽。天然存在的nifk多肽通常具有430至530个氨基酸的长度。已经鉴定了大量的nifk多肽,并且在公众可获得的数据库中可获得许多序列。例如,已经报道了来自klebsiellamichiganensis(密歇根克雷伯菌,登录号wp_049080161.1,与seqidno:7具有99%同一性)、raoultellaornithinolytica(解鸟氨酸拉乌尔菌,wp_044347163.1,96%同一性)、klebsiellavariicola(变栖克雷伯菌,sbm87811.1,94%同一性)、kluyveraintermedia(中间克鲁瓦氏菌,wp_047370272.1,89%同一性)、rahnellaaquatilis(水生拉恩菌,wp_014333919.1,82%同一性)、tolumonasauensis(wp_012728880.1,75%同一性)、pseudomonasstutzeri(斯氏假单胞菌,wp_011912506.1,68%同一性)、vibrionatriegens(需钠弧菌,wp_065303473.1,65%同一性)、azoarcustoluclasticus(wp_018989051.1,54%同一性)、frankiasp.(弗兰克氏菌属,prf||2106319a,50%同一性)和methanosarcinaacetivorans(乙酸甲烷八叠球菌,wp_011021239.1,31%同一性)的nifk多肽。在标注为“nifk”的数据库中存在一些多肽实例,其与seqidno:7具有小于31%的同一性,但不含有上文列出的任何结构域,因此不包括在本文中作为nifk多肽。nifk多肽已经在kim和rees(1994)、eady(1996)、robson等人(1989)、dilworth等人(1988)、dilworth等人(1993)、miller和eady(1988)、igarashi和seefeldt(2003)、fani等人(2000)以及rubio和ludden(2005)中进行了描述和综述。铁钼亚类的nifk多肽是固氮酶复合物的关键亚基,是固氮酶核心处α2β2mofe蛋白复合物的β亚基。如本文所用,功能性nifk多肽是能够与其他所需亚基(例如nifd和nifh)以及femo或其他辅因子一起形成功能性固氮酶蛋白复合物的nifk多肽。在一个优选的实施方案中,当与氨基酸序列seqidno:7比对时,本发明的nifk多肽的氨基酸序列在其c端具有氨基酸dlvr(seqidno:7的残基517-520),精氨酸为c端氨基酸。也就是说,本发明的nifk多肽和nifk融合多肽优选具有与天然nifk多肽相同的c端,即,它没有c端的人工添加物。这些优选的nifk多肽能更好地与nifd和nifh多肽形成功能性固氮酶复合物,如实施例所示。天然存在的细菌中的nifb多肽是参与femo-co合成,将[4fe-4s]簇转化为nifb-co的蛋白,nifb-co是具有中心c原子的更高核性的fe-s簇,其用作femo-co的前体(guo等人,2016)。因此nifb催化femo-co合成途径中的第一个关键步骤。nifb的nifb-co产物能够结合nife-nifn复合物,并且可以通过金属原子簇载体(carrier)蛋白nifx从nifb穿梭到nife-nifn。如本文所用,“nifb多肽”意指其氨基酸序列包括序列与seqidno:9所示的氨基酸序列至少具有27%同一性的氨基酸的多肽。大多数nifb多肽包括保守结构域tigr01290、nifb保守结构域cd00852中的一个或多个,nifx-nifb超家族保守结构域cl00252和s-腺苷甲硫氨酸自由基(radical_sam)保守结构域cd01335。如本文所用,nifb多肽包括已被注释为具有nifb功能但不具有这些结构域之一的天然存在的多肽。天然存在的nifb多肽通常具有440至500个氨基酸的长度,天然单体具有约50kda的分子量。已经鉴定了大量的nifb多肽,并且在公众可获得的数据库中可获得许多序列。例如,已经报道了来自raoultellaornithinolytica(解鸟氨酸拉乌尔菌,登录号wp_041145602.1,与seqidno:9具有91%同一性)、kosakoniaradicincitans(玉米联合固氮菌,wp_043953592.1,80%同一性)、dickeyachrysanthemi(菊欧文氏菌,wp_040003311.1,76%同一性)、pectobacteriumatrosepticum(黑胫病菌,wp_011094468.1,70%同一性)、brenneriagoodwinii(wp_048638849.1,63%同一性)、halorhodospirahalophila(嗜盐嗜盐红螺菌,wp_011813098.1,59%同一性)、methanosarcinabarkeri(巴氏甲烷八叠球菌,wp_048108879.1,50%同一性)、clostridiumpurinilyticum(wp_050355163.1,40%同一性)、geofilumrubicundum(gao28552.1,35%同一性)和desulfovibriosalexigens(需盐脱硫弧菌,wp_015850328.1,27%同一性)的nifb多肽。如本文所用,“功能性nifb多肽”是能够由[4fe-4s]簇形成nifb-co的nifb多肽。nifb多肽已经在curatti等人,(2006)和allen等人,(1995)中进行了描述和综述。nifen复合物是正确组装固二氮酶所需的支架复合物(scaffoldcomplex),并且在结构上也类似于固二氮酶(fay等人,2016)。nifen复合物分别由nife和nife各自的2个亚基组成,形成异源四聚体,本文称为enα2β2。天然存在于细菌中的nife多肽是具有nifn多肽的enα2β2四聚体α亚基的多肽,该enα2β2四聚体是femo-co合成所需的,并被建议用作合成femo-co的支架。如本文所用,“nife多肽”是指包括氨基酸的多肽,氨基酸的序列与seqidno:10所示的氨基酸序列至少具有32%同一性,并且其包括结构域tigr01283和prk14478中的一个或两个。tigr01283结构域蛋白家族的成员也是超家族cl02775的成员。天然存在的nife多肽通常具有440至490个氨基酸的长度,天然单体具有约50kda的分子量。已经鉴定了大量的nife多肽,并且在公众可获得的数据库中可获得许多序列。例如,已经报道了来自klebsiellamichiganensis(密歇根克雷伯菌,登录号wp_049114606.1,99%与seqidno:10同一性)、klebsiellavariicola(变栖克雷伯菌,sbm87755.1,92%同一性)、dickeyaparadisiaca(菊欧文氏杆菌香蕉致病变种,wp_012764127.1,89%同一性)、tolumonasauensis(wp_012728883.1,75%同一性)、pseudomonasstutzeri(斯氏假单胞菌,wp_003297989.1,69%同一性)、azotobactervinelandii(维涅兰德固氮菌,wp_012698965.1,62%同一性)、trichormusazollae(满江红鱼腥藻,wp_013190624.1,55%同一性)、paenibacillusdurus(坚韧类芽孢杆菌,wp_025698318.1,50%同一性)、sulfuricurvumkujiense(wp_013460149.1,44%同一性)、methanobacteriumformicicum(甲酸甲烷杆菌,ais31022.1,39%同一性)、anaeromusaacidaminophila(wp_018701501.1,35%同一性)和megasphaeracerevisiae(酿酒巨球形菌,wp_048514099.1,32%相同)的nife多肽。如本文所用,“功能性nife多肽”是能够与nifn一起形成功能性四聚体使得复合物能够合成femo-co的nife多肽。nife多肽已经在fay等人(2016)、hu等人(2005)、hu等人(2006)和hu等人(2008)中进行了描述和综述。天然存在的细菌中的niff多肽是作为nifh的电子供体的黄素氧还蛋白。如本文所用,“niff多肽”是指包括氨基酸的多肽,氨基酸的序列与seqidno:16所示的氨基酸序列至少具有34%同一性,并且其包括在来自定氮菌和其他细菌属prk09267的nif蛋白上发现的黄素氧还蛋白长结构域tigr01752和黄素氧还蛋白flda结构域中的一个或两个。niff多肽包括在非固氮细菌中与丙酮酸甲酸裂解酶活化和钴胺素依赖性甲硫氨酸合酶活性相关的黄素氧还蛋白,但排除了涉及更广泛功能的其他黄素氧还蛋白。天然存在的niff多肽通常具有160至200个氨基酸的长度,天然单体具有约19kda的分子量。已经鉴定了大量的niff多肽,并且在公众可获得的数据库中可获得许多序列。例如,已经报道了来自klebsiellamichiganensis(密歇根克雷伯菌,登录号wp_004122417.1,与seqidno:16具有99%同一性)、klebsiellavariicola(变栖克雷伯菌,wp_040968713.1,85%同一性)、kosakoniaradicincitans(玉米联合固氮菌,wp_035885760.1,76%同一性)、dickeyachrysanthemi(菊欧文氏菌,wp_039999438.1,72%同一性)、brenneriagoodwinii(wp_048638838.1,62%同一性)、methylomonasmethanica(甲烷甲基单胞菌,wp_064006977.1,56%同一性)、azotobactervinelandii(维涅兰德固氮菌,wp_012698862.1,50%同一性)、chlorobaculumtepidum(厌氧嗜热绿硫细菌,wp_010933399.1,39%同一性)、campylobactershowae(昭和弯曲菌,wp_002949173.1,37%同一性)和azotobacterchromococcum(圆褐固氮菌,wp_039801725.1,34%同一性)的niff多肽。如本文所用,“功能性niff多肽”是能够作为nifh多肽电子供体的niff多肽。niff多肽已经在drummond(1985)中进行了描述和综述。天然存在的细菌中的nifj多肽是丙酮酸盐:黄素氧还蛋白(铁氧还蛋白)氧化还原酶,其是nifh的电子供体。如本文所用,“nifj多肽”是指包括氨基酸的多肽,氨基酸的序列与seqidno:18所示的氨基酸序列至少具有40%同一性并且包括保守结构域tigr02176。天然存在的nifj多肽通常具有1100-1200个氨基酸的长度,天然单体具有约128kda的分子量。已经鉴定了大量的nifj多肽,并且在公众可获得的数据库中可获得许多序列。例如,已经报道了来自klebsiellamichiganensis(密歇根克雷伯菌,登录号wp_024360006.1,与seqdino:18具有99%同一性)、raoultellaornithinolytica(解鸟氨酸拉乌尔菌,wp_044347157.1,95%同一性)、klebsiellaquasipneumoniae(类肺炎克雷伯菌,wp_050533844.1,92%同一性)、kosakoniaoryzae(wp_064566543.1,82%同一性)、dickeyasolani(马铃薯黑胫病菌,wp_057084649.1,78%同一性)、rahnellaaquatilis水生拉恩菌(wp_014683040.1,72%同一性)、thermoanaerobactermathranii(油藏嗜热厌氧杆菌,wp_013149847.1,64%同一性)、clostridiumbotulinum(肉毒梭菌,wp_053341220.1,60%同一性)、spirochaetaafricana(wp_014454638.1,52%同一性)和vibriocholerae(霍乱弧菌,csa83023.1,40%同一性)的nifj多肽。如本文所用,“功能性nifj多肽”是能够作为nifh多肽电子供体的nifj多肽。nifj多肽已经在schmitz等人,(2001)中进行了描述和综述。天然细菌中的nifm多肽是nifh成熟所需的多肽。在没有nifm的情况下,当异源表达时,nifh在大肠杆菌和酵母中只以低水平存在,并且不能向nifd-nifk提供电子。如本文所用,“nifm多肽”是指包括氨基酸的多肽,氨基酸的序列与seqidno:19所示的氨基酸序列至少具有26%同一性并且包括结构域tigr02933。nifm多肽与肽基脯氨酰顺反异构酶同源,似乎是nifh的辅助蛋白。天然存在的nifm多肽通常具有240-300个氨基酸的长度,天然单体具有约30kda的分子量。已经鉴定了大量的nifm多肽,并且在公众可获得的数据库中可获得许多序列。例如,已经报道了来自klebsiellaoxytoca(产酸克雷伯菌,登录号wp_064342940.1,与seqidno:19具有99%同一性)、klebsiellamichiganensis(密歇根克雷伯菌,wp_004122413.1,97%同一性)、raoultellaornithinolytica(解鸟氨酸拉乌尔菌,wp_044347181.1,85%同一性)、klebsiellavariicola(变栖克雷伯菌,wp_063105800.1,75%同一性)、kosakoniaradicincitans(玉米联合固氮菌,wp_035885759.1,59%同一性)、pectobacteriumatrosepticum(黑胫病菌,wp_011094472.1,42%同一性)、brenneriagoodwinii(wp_048638837.1,33%同一性)、pseudomonasaeruginosa(绿脓杆菌)pao1(caa75544.1,28%同一性)、marinobacteriumsp.(海杆菌属)ak27(wp_051692859.1,27%同一性)和teredinibacterturnerae(wp_018415157.1,26%同一性)的nifm多肽。如本文所用,“功能性nifm多肽”是能够与nifh多肽复合以使nifh多肽成熟的nifm多肽。nifm多肽已在petrova等人(2000)中进行了描述和综述。天然存在的细菌中的nifn多肽是enα2β2四聚体与nife多肽的β亚基,enα2β2四聚体是femo-co合成所需的,并被建议用作合成femo-co的支架。如本文所用,“nifn多肽”意指(i)包括其序列与seqidno:11所示的序列具有至少76%同一性的氨基酸的多肽和/或(ii)包括其序列与seqidno:11所示的序列至少具有34%同一性的氨基酸的多肽,并且其包括保守结构域tigr01285、cd01966和prk14476中的一个或多个。nifn在结构上与钼-铁蛋白β链nifk有关。包括保守tigr01285的多肽涵盖了nifn多肽的大多数实例,但排除了一些nifn多肽,如推定的绿硫细菌nifn,因此nifn的定义不限于包括保守tigr01285结构域的多肽。prk14476结构域蛋白家族的成员也是超家族cl02775的成员。天然存在的nifn多肽通常具有410至470个氨基酸的长度,尽管当与nifb天然融合时,其可以具有约900个氨基酸残基,并且天然单体具有约50kda的分子量。已经鉴定了大量的nifn多肽,并且在公众可获得的数据库中可获得许多序列。例如,已经报道了来自klebsiellaoxytoca(产酸克雷伯菌,登录号wp_064391778.1,与seqidno:11具有97%同一性)、kluyveraintermedia(中间克鲁瓦氏菌,wp_047370268.1,80%同一性)、rahnellaaquatilis(水生拉恩菌,wp_014683026.1,70%同一性)、brenneriagoodwinii(wp_048638830.1,65%同一性)、methylobactertundripaludum(wp_027147663.1,46%同一性)、calothrixparietina(墙壁眉藻,wp_015195966.1,41%同一性)、zymomonasmobilis(运动发酵单胞菌,wp_023593609.1,37%同一性)、paenibacillusmassiliensis(马赛类芽孢杆菌,wp_025677480.1,35%同一性)和desulfitobacteriumhafniense(四氯乙烯降解菌,wp_018306265.1,34%同一性)的nifn多肽。如本文所用,“功能性nifn多肽”是能够与nife一起形成功能性四聚体使得复合物能够合成femo-co的nifn多肽。nifn多肽已经在fay等人(2016)、brigle等人(1987)、fani等人(2000)和hu等人(2005)中进行了描述和综述。天然存在的细菌中的nifq多肽是参与femo-co合成的多肽,可能存在于moo42-加工的早期过程中。保守的c端半胱氨酸残基可能参与金属结合。如本文所用,“nifq多肽”是指包括氨基酸的多肽,氨基酸的序列与seqidno:12所示的氨基酸序列至少具有34%同一性,并且是cl04826结构域蛋白家族的成员和pfam04891结构域蛋白家族的成员。天然存在的nifq多肽通常具有160至250个氨基酸的长度,尽管它们可以长达350个氨基酸残基,并且天然单体具有约20kda的分子量。已经鉴定了大量的nifq多肽,并且在公众可获得的数据库中可获得许多序列。例如,已经报道了来自klebsiellaoxytoca(产酸克雷伯菌,登录号wp_064391765.1,与seqidno:12具有95%同一性)、klebsiellavariicola(变栖克雷伯菌,ctq06350.1,75%同一性)、kluyveraintermedia(中间克鲁瓦氏菌,wp_047370257.1,63%同一性)、pectobacteriumatrosepticum(黑胫病菌,wp_043878077.1,59%同一性)、mesorhizobiummetallidurans(耐重金属中慢生根瘤菌,wp_008878174.1,46%同一性)、rhodopseudomonaspalustris(沼泽红假单胞菌,wp_011501504.1,42%同一性)、paraburkholderiasprentiae(wp_027196569.1,41%同一性)、burkholderiastabilis(稳定伯克霍尔德菌,gau06296.1,39%同一性)和cupriavidusoxalaticus(草酸盐嗜铜菌,wp_063239464.1,34%同一性)的nifq多肽。如本文所用,“功能性nifq多肽”是能够加工moo42-的nifq多肽。nifq多肽已经在allen等人(1995)和sidddivertam等人(1993)中进行了描述和综述。天然存在的细菌中的nifs多肽是参与铁-硫(fes)簇生物合成的半胱氨酸脱硫酶,例如其参与硫的活动化以用于fe-s簇合成和修复。如本文所用,“nifs多肽”意指(i)包括其序列与seqidno:13所示的氨基酸序列至少具有90%同一性的氨基酸的多肽和/或(ii)包括其序列与seqidno:13所示的序列至少具有36%同一性的氨基酸的多肽,并且其包括保守结构域tigr03402和cog1104中的一个或两个。tigr03402结构域蛋白家族包括几乎总是在延伸的氮固定系统中发现的分支加上与第一个比与iscs更紧密相关的第二个分支以及nifs样/nifu样系统的一部分。tigr03402结构域蛋白家族不延伸至在如幽门螺杆菌的ε变形菌门(文献中也称为nifs)中发现的更远的分支,而是在tigr03403中构建。cog1104结构域蛋白家族包括半胱氨酸亚磺酸盐脱硫酶/半胱氨酸脱硫酶或相关酶。一些nifs多肽包括天冬氨酸转氨酶结构域cl18945。天然存在的nifs多肽通常具有370-440个氨基酸的长度,天然单体具有约43kda的分子量。已经鉴定了大量的nifs多肽,并且在公众可获得的数据库中可获得许多序列。例如,已经报道了来自klebsiellamichiganensis(密歇根克雷伯菌,登录号wp_004138780.1,与seqidno:13具有99%同一性)、raoultellaterrigena(土生拉乌尔菌,wp_045858151.1,89%同一性)、kluyveraintermedia(中间克鲁瓦氏菌,wp_047370265.1,80%同一性)、rahnellaaquatilis(水生拉恩菌,wp_014333911.1,73%同一性)、agarivoransgilvus(wp_055731597.1,64%同一性)、azospirillumbrasilense(巴西固氮螺菌,wp_014239770.1,60%同一性)、desulfosarcinacetonica(wp_054691765.1,55%同一性)、clostridiumintestinale(肠梭菌,wp_021802294.1,47%同一性)、clostridiisalibacterpaucivorans(wp_026894054.1,36%同一性)和bacilluscoagulans(凝结芽孢杆菌,wp_061575621.1,42%同一性并位于cog1104中)的多肽。如本文所用,“功能性nifs多肽”是能够在铁-硫(fes)簇生物合成和/或修复中起作用的nifs多肽。nifs多肽已经在clausen等人(2000)、johnson等人(2005)、olson等人(2000)和yuvaniyama等人(2000)中进行了描述和综述。天然存在的细菌中的nifu多肽是参与固氮酶组件的铁-硫(fes)簇生物合成的分子支架多肽。如本文所用,“nifu多肽”是指包括氨基酸的多肽,氨基酸的序列与seqidno:14所示的序列至少具有31%同一性并且包括结构域tigr02000。tigr02000结构域蛋白家族的成员特异性地参与固氮酶成熟。nifu包括n端结构域(pfam01592)和c端结构域(pfam01106)。已经描述了三种不同但部分同源的fe-s簇组装系统:isc、suf和nif。nif系统(其中nifu是其一部分)与向多种固氮物种的固氮酶供给fe-s簇相关。具有来自螺杆菌(helicobacter)和弯曲杆菌(campylobacter)的等价域结构的isc和suf同系物在本文被排除在nifu的定义之外。因此,nifu特异于参与固氮酶成熟的nifu多肽。相关的tigr01999结构域蛋白家族的成员也被排除在本文的nifu的定义之外,其是包括nifu的n端区域的同系物的iscu蛋白(例如来自大肠杆菌和酿酒酵母和人智人)。天然存在的nifu多肽通常具有260-310个氨基酸的长度,天然单体具有约29kda的分子量。已经鉴定了大量的nifu多肽,并且在公众可获得的数据库中可获得许多序列。例如,已经报道了来klebsiellamichiganensis(密歇根克雷伯菌,登录号wp_049136164.1,与seqidno:14具有97%同一性)、klebsiellavariicola(变栖克雷伯菌,wp_050887862.1,90%同一性)、dickeyasolani(马铃薯黑胫病菌,wp_057084657.1,80%同一性)、brenneriagoodwinii(wp_048638833.1,73%同一性)、tolumonasauensis(wp_012728889.1,66%同一性)、agarivoransgilvus(wp_055731596.1,58%同一性)、desulfocurvusvexinensis(wp_028587630.1,54%同一性)、rhodopseudomonaspalustris(沼泽红假单胞菌,wp_044417303.1,49%同一性)、helicobacterpylori(幽门螺杆菌,wp_001051984.1,31%同一性)和sulfurovumsp.pc08-66(kim05011.1,31%同一性)的nifu多肽。如本文所用,“功能性nifu多肽”是能够用作参与铁-硫(fes)簇生物合成的分子支架多肽的nifu多肽。nifu多肽已经在hwang等人(1996)、mühlenhoff等人(2003)和ouzunis等人(1994)中进行了描述和综述。天然存在的细菌中的nifv多肽是高柠檬酸合酶(ec2.3.3.14),通过将乙酰基从乙酰辅酶a(乙酰-coa)转移到2-氧代戊二酸盐而产生高柠檬酸。然后将高柠檬酸用于femo-co的合成。如本文所用,“nifv多肽”是指包括氨基酸的多肽,氨基酸的序列与seqidno:20所示的氨基酸序列至少具有39%同一性并且包括结构域tigr02660和dre_tim中的一个或两个。tigr02660结构域蛋白家族的成员与包括2-异丙基苹果酸盐合酶、(r)-柠苹酸合酶和与除氮固定以外的过程相关的高柠檬酸合酶的酶同源。cd07939结构域蛋白家族还包括绿螺旋杆菌(heliobacteriumchlorum)和重氮营养葡糖酸醋杆菌(gluconacetobacterdiazotrophicus)的nifv蛋白,它们似乎与frbc直系同源。该家族属于dre-tim金属裂解酶超家族。dre-tim金属裂解酶包括2-异丙基苹果酸合酶(ipms)、α-异丙基苹果酸合酶(leua)、3-羟基-3-甲基戊二酰-coa裂解酶、高柠檬酸合酶、柠苹酸合酶、4-羟基-2-氧代戊酸醛缩酶、re-柠檬酸合成酶、转羧酶5s、丙酮酸羧化酶、aksa和frbc。这些成员都共有保守的磷酸丙糖异构酶(tim)桶形(barrel)结构域,其由核心β(8)-α(8)基序组成,八个平行的β链形成被八个α螺旋包围的封闭桶形结构。该结构域具有含有二价阳离子结合位点的催化中心,二价阳离子结合位点由覆盖桶形芯的不变残基簇形成。此外,催化位点包括三个不变的残基——天冬氨酸(d)、精氨酸(r)和谷氨酸(e)——这是域名“dre-tim”的基础。天然存在的nifv多肽通常具有360-390个氨基酸的长度,尽管一些成员长度为约490个氨基酸残基,并且天然单体具有约41kda的分子量。已经鉴定了大量的nifv多肽,并且在公众可获得的数据库中可获得许多序列。例如,已经报道了来自klebsiellamichiganensis(密歇根克雷伯菌,登录号wp_049083341.1,与seqidno:20具有同一性95%)、raoultellaornithinolytica(解鸟氨酸拉乌尔菌,wp_045858154.1,86%同一性)、kluyveraintermedia(中间克鲁瓦氏菌,wp_047370264.1,81%同一性)、dickeyadadantii(达旦提狄克氏菌,wp_038912041.1,70%同一性)、brenneriagoodwinii(wp_048638835.1,59%同一性)、magnetococcusmarinus(wp_011712856.1,46%同一性)、sphingomonaswittichii(wp_037528703.1,43%同一性)、frankiasp.(弗兰克氏菌属)ei5c(oaa29062.1,41%同一性)和clostridiumsp.maddingleymbc34-26(ekq56006.1,39%同一性)的nifv多肽。如本文所用,“功能性nifv多肽”是能够作为高柠檬酸合酶起作用的nifv多肽。nifv多肽已经在hu等人(2008)、lee等人(2000)、masukawa等人(2007)和zheng等人(1997)中进行了描述和综述。天然存在的细菌中的nifx多肽是参与femo-co合成的多肽,至少有助于将femo-co前体从nifb转移到nife-nifn。如本文所用,“nifx多肽”是指包括氨基酸的多肽,氨基酸的序列与seqidno:15所示的氨基酸序列至少具有29%同一性并且包括保守结构域tigr02663和cd00853中的一个或两个。nifx包括在包括nifb和nify的铁-钼簇结合蛋白的较大家族中,因为nifx、nafy和nifb的c端区域都包括pfam02579结构域,并且各自参与femo-co的合成。因此,一些nifx多肽在数据库中被注释为nify,反之亦然。天然存在的nifx多肽通常具有110-160个氨基酸的长度,天然单体具有约15kda的分子量。已经鉴定了大量的nifx多肽,并且在公众可获得的数据库中可获得许多序列。例如,已经报道了来自klebsiellamichiganensis(密歇根克雷伯菌,登录号wp_049070199.1,与seqidno:15具有97%同一性)、klebsiellaoxytoca(产酸克雷伯菌,wp_064342937.1,97%同一性)、raoultellaornithinolytica(解鸟氨酸拉乌尔菌,wp_044347173.1,91%同一性)、klebsiellavariicola(变栖克雷伯菌,wp_044612922.1,83%同一性)、kosakoniaradicincitans(玉米联合固氮菌,wp_043953583.1,75%同一性)、dickeyachrysanthemi(菊欧文氏菌,wp_039999416.1,68%同一性)、rahnellaaquatilis(水生拉恩菌,wp_047608097.1,58%同一性)、azotobacterchroococcum(圆褐固氮菌,wp_039800848.1,34%同一性)、beggiatoaleptomitiformis(水节霉状贝日阿托氏菌,wp_062149047.1,33%同一性)和methyloversatilisdiscipulorum(wp_020165972.1,29%同一性)的nifx多肽。如本文所用,“功能性nifx多肽”是能够将femo-co前体从nifb转移到nife-nifn的nifx多肽。nifx多肽已经在allen等人(1994)和shah等人(1999)中进行了描述和综述。天然存在的细菌中的nify多肽是参与femo-co合成的多肽,至少有助于将femo-co前体从nifb转移到nife-nifn。如本文所用,“nify多肽”是指包括氨基酸的多肽,氨基酸的序列与seqidno:8所示的氨基酸序列至少具有34%同一性并且包括保守结构域tigr02663和cd00853中的一个或两个。nify包括在包括nifb和nifx的铁-钼簇结合蛋白的较大家族中,因为nifx、nafy和nifb的c端区域都包括pfam02579结构域,并且各自参与femo-co的合成。已经鉴定了大量的nify多肽,并且在公众可获得的数据库中可获得许多序列。例如,已经报道了来自klebsiellamichiganensis(密歇根克雷伯菌,登录号wp_049089500.1,与seqidno:8具有99%同一性)、klebsiellaoxytoca(产酸克雷伯菌,wp_064342935.1,98%同一性)、klebsiellaquasipneumoniae(类肺炎克雷伯菌,wp_044524054.1,90%同一性)、klebsiellavariicola(变栖克雷伯菌,wp_049010739.1,81%同一性)、kluyveraintermedia(中间克鲁瓦氏菌,wp_047370270.1,69%同一性)、dickeyachrysanthemi(菊欧文氏菌,wp_039999411.1,62%同一性)、serratiasp.atcc39006(wp_037382461.1,57%同一性)、rahnellaaquatilis(水生拉恩菌,wp_014683024.1,47%同一性)、pseudomonasputida(恶臭假单胞菌,aex25784.1,37%同一性)和azotobactervinelandii(维涅兰德固氮菌,wp_012698835.1,34%同一性)的nify多肽。如本文所用,“功能性nify多肽”是能够将femo-co前体从nifb转移到nife-nifn的nify多肽。天然存在的细菌中的nifz多肽是参与fe-s簇合成的多肽,特别是第二fe4s4对偶联所需的多肽。如本文所用,“nifz多肽”是指包括氨基酸的多肽,氨基酸的序列与seqidno:17所示的序列至少具有28%同一性并且包括保守结构域pfam04319。发现该约75个氨基酸残基的结构域独立地存在于在一些成员中和较长nifz蛋白的氨基端一半中。天然存在的nifz多肽通常具有70至150个氨基酸的长度,并且天然单体具有约9至约16kda的分子量。已经鉴定了大量的nifz多肽,并且在公众可获得的数据库中可获得许多序列。例如,已经报道了来自klebsiellamichiganensis(密歇根克雷伯菌,登录号wp_057173223.1,与seqidno:17具有93%同一性)、klebsiellaoxytoca(产酸克雷伯菌,wp_064342939.1,95%同一性)、klebsiellavariicola(变栖克雷伯菌,wp_043875005.1,77%同一性)、kosakoniaradicincitans(玉米联合固氮菌,wp_043953588.1,67%同一性)、kosakoniasacchari(wp_065368553.1,58%同一性)、ferriphaselusamnicola(wp_062627625.1,47%同一性)、paraburkholderiaxenovorans(wp_011491838.1,41%同一性)、acidithiobacillusferrivoranswu(wp_014029050.1,35%同一性)和bradyrhizobiumoligotrophicum(wp_015665422.1,28%同一性)的nifz多肽。如本文所用,“功能性nifz多肽”是能够在fe-s簇合成中偶联fe4s4簇的nifz多肽。nifz多肽已经在cotton(2009)和hu等人(2004)中进行了描述和综述。天然存在的细菌中的nifw多肽是与nifz多肽缔合形成更高级复合物的多肽(lee等人,1998),并参与mofe蛋白(nifd-nifk)合成或活性。nifw和nifz似乎参与mofe蛋白的形成或累积(paul和merrick,1989)。如本文所用,“nifw多肽”是指这样的多肽,其氨基酸序列包括其序列与seqidno:74所示的氨基酸序列至少具有28%同一性的氨基酸,并且其包括保守的nifw超家族蛋白结构域,结构id号10505077,并且在p家族pf03206中。已经鉴定了多种nifw多肽,并且在公众可获得的数据库中可获得许多序列。例如,已经报道来自klebsiellaoxytoca(产酸克雷伯菌,登录号wp_064342938.1,与seqidno:74具有98%同一性)、klebsiellamichiganensis(密歇根克雷伯菌,wp_049080155.1,94%同一性)、enterobactersp.(肠杆菌属)10-1(wp_095103586.1,90%同一性)、klebsiellaquasipneumoniae(类肺炎克雷伯菌,wp_065877373.1,81%同一性)、pectobacteriumpolaris(wp_095699971.1,69%同一性)、dickeyaparadisiaca(菊欧文氏杆菌香蕉致病变种,wp_012764136.1,58%同一性)、brenneriagoodwinii(wp_053085547.1,36%同一性)、aquaspirillumsp.(水螺菌属)lm1(wp_077299824.1,44%同一性)、candidatusmuproteobacteriabacteriumrbg_16_64_10(ogi40729,34%同一性)、azotobactervinelandii(维涅兰德固氮菌,aco76430.1,32%同一性)和methylocaldummarinum(bba37427.1,28%同一性)的nifw多肽。如本文所用,“功能性nifw多肽”是促进或增强mofe蛋白的一种或多种形成、累积或活性的nifw多肽。功能性nifw可以与nifz相互作用和/或在mofe蛋白的氧保护中起作用(gavini等人,(1998))。关于定义的多肽,应当理解,高于以上提供的那些的%同一性数字将包括优选的实施方案。因此,在适用时,根据最小%同一性数字,优选多肽包括至少30%、更优选至少35%、更优选至少40%、更优选至少45%、更优选至少50%、更优选至少55%、更优选至少60%、更优选至少65%、更优选至少70%、更优选至少75%、更优选至少80%、更优选至少85%、更优选至少90%、更优选至少91%、更优选至少92%、更优选至少93%、更优选至少94%、更优选至少95%、更优选至少96%、更优选至少97%、更优选至少98%、更优选至少99%、更优选至少99.1%、更优选至少99.2%、更优选至少99.3%、更优选至少99.4%、更优选至少99.5%、更优选至少99.6%、更优选至少99.7%、更优选至少99.8%、甚至更优选与相关命名的seqidno至少具有99.9%同一性的氨基酸序列。本文定义的多肽的氨基酸序列突变体可以通过向本文定义的核酸中引入适当的核苷酸变化,或通过所需多肽的体外合成来制备。这样的突变体包括例如一个或多个氨基酸缺失、插入或替换。可以进行缺失、插入和替换突变的组合以获得最终的构建体,条件是最终的多肽产物具有所需的特征。优选的氨基酸序列突变体相对于参照野生型多肽仅有一个、两个、三个、四个或少于10个氨基酸的变化。突变体(改变的)多肽可使用本领域已知的任何技术制备,例如使用定向进化或合理设计策略(参见下文)。可以使用本文描述的技术容易地筛选来源于突变/改变的dna的产物,以确定它们在植物中的表达是否相对于相应的野生型植物改变了其表型,例如,如果它们的表达导致相对于相应的野生型植物在产量、生物质、生长速率、活力、来源于生物固氮的氮增益、氮利用效率、非生物胁迫抗性和/或对营养缺乏的耐受性等方面有所增加。在设计氨基酸序列突变体时,突变位点的位置和突变的性质将取决于待修饰的特征。突变位点可以单独或串联修饰,例如,通过(1)首先用保守的氨基酸选择替换,然后根据获得的结果用更多的自由基选择替换,(2)删除目标残基或(3)插入邻近定位位点的其他残基。氨基酸序列缺失通常为约1至15个残基,更优选约1至10个残基,通常约1至5个连续残基。替换突变体使多肽分子中的至少一个氨基酸残基被去除,并在其位置插入不同的残基。当需要维持某种活性时,优选在相关蛋白家族中高度保守的氨基酸位置不进行或仅进行保守性替换。保守性替换的实例示于表1的“示例性替换”标题下。在优选的实施方案中,突变体/变异体多肽与天然存在的多肽相比具有一个或两个或三个或四个保守氨基酸改变。表1提供了保守氨基酸改变的细节。在优选的实施方案中,在本发明不同多肽之间高度保守的一个或多个基序或结构域中没有改变。本领域技术人员将意识到,当在重组细胞中表达时,可以合理地预测这种微小变化不会改变多肽的活性。基于与密切相关多肽的比较,本发明多肽的一级氨基酸序列可用于设计其变异体/突变体。如本领域技术人员将理解的,紧密相关的蛋白中高度保守的残基不太可能被改变,尤其是非保守性替换,并且活性保持低于较不保守的残基(参见上文)。鉴定保守氨基酸残基更严格的测试是比对具有相同功能的关系更远的多肽。应该保持高度保守的残基以保持功能,而非保守的残基更易于在保持功能的同时进行替换或缺失。也包括在本发明范围内的是本发明的多肽,其在细胞中合成期间或之后被差异修饰,例如通过糖基化、乙酰化、磷酸化或蛋白水解切割。表1.示例性替换合理设计可以在已知蛋白结构和折叠信息的基础上合理设计蛋白。这可以通过从头设计(从新设计)或通过基于天然支架的重新设计(参见例如hellinga,1997;和lu和berry的《蛋白质手册2:蛋白质结构设计与工程》(proteinstructuredesignandengineering,handbookofproteins2),1153-1157(2007))。参见例如本文的实施例10。蛋白设计通常涉及识别折叠成给定或目标结构的序列,并且可以使用计算机模型完成。计算蛋白质设计算法在序列构象空间中搜索折叠到目标结构时能量较低的序列。计算蛋白设计算法使用蛋白能量学模型来评估突变如何影响蛋白的结构和功能。这些能量函数通常包括分子力学、统计学(即基于知识)和其他经验项的组合。合适的可用软件包括ipro(迭代蛋白重新设计和优化)、egad(用于蛋白设计的遗传算法)、rosettadesign、sharpen和abalone。植物中线粒体蛋白的导入几乎所有线粒体蛋白都是核编码的并且在胞质溶胶中翻译的,因此需要它们易位到线粒体中。多肽内的信号序列将它们导入四个不同的线粒体内位置:外膜(om)、膜间间隙(is)、内膜(im)或基质(mm)。这些信号序列的区别在于它们的生物化学特性,并且通过至少四种不同的导入途径指导转运,导入途径将多肽导向四个位置中的一个或多个(chacinska等人,2009)。这四种途径是:(1)一般导入途径,也称为“经典”前导序列途径,其将多肽导向mm、is或im;(2)用于转移至im的载体(carrier)导入途径,(3)线粒体膜间间隙(mia)组装途径,和(4)用于将多肽转移至om的分选和组装机器(sam)途径。一般导入途径导入具有可切割的前导序列(也称为信号序列)的多肽。这些多肽也可以具有疏水分选信号(hss)。载体(carrier)导入途径导入具有内部前导序列样信号和疏水区的多肽。mia途径导入具有双半胱氨酸残基的多肽。sam途径导入含有β信号和推定的tom20信号的多肽。所有这些途径都利用外膜的移位酶(tom),第一途径和第二途径也利用膜间复合物的tim23移位酶。只有第一途径使用基质加工肽酶(基质作用蛋白酶,mpp)。所有线粒体靶向多肽的共同特征是在多肽内存在至少一个指导转运到正确位置的结构域。其中研究得最好的是“经典的”n端前导序列结构域,其在基质中被mpp切割(murcha等人,2004)。据估计约70%的植物和动物线粒体蛋白具有可切割的前导序列,但也发现了内部和c端信号序列(综述见pfanner和geissler(2001)、schleiff和soll(2000))。在拟南芥(arabidopsis)中,这些前导序列的长度为11至109个氨基酸残基,平均长度为50个氨基酸残基。尽管没有完全限定第一途径的前导序列的共有序列,但它们倾向于含有高比例的疏水和带正电荷的氨基酸。另一个特征是它们形成两亲性α螺旋的能力,通常在前10个氨基酸残基内开始(roise等人,1986)。这些结构域富含疏水的(ala、leu、phe、val)、羟基化的(ser、thr)和带正电荷的(arg、lys)氨基酸残基,并且缺乏酸性氨基酸。在大量线粒体蛋白中,丝氨酸(16-17%)和丙氨酸(12-13%)在线粒体信号肽中大大过量表达,精氨酸丰富(12%)。对于大多数前导序列,mpp切割点是通过保守精氨酸残基的存在来定义的,通常在p2位(距易裂键约-2个氨基酸),或在大多数其他情况下在p3位(huang等人,2009)。线粒体前导序列通过疏水残基与tom20受体相互作用。研究表明,α-螺旋的疏水表面促进tom导入复合物的tom20组件识别肽,而正电荷被tom22亚基识别(abe等人,2000)。最后,大多数前导序列引导与hsp70相关的多肽的运输,因此几乎所有的植物前导序列都至少含有一个hsp70分子伴侣的结合基序(zhang和glaser,2002)。伴侣hsp70参与蛋白折叠,防止蛋白聚集,并起到分子马达的作用,拉动前体穿过线粒体膜。跨内膜的电膜电位(δψ)(-100mv,内部负)也通过电泳效应驱动带正电荷的前导序列易位。大多数具有可切割前导序列的蛋白通过一般导入途径到达线粒体基质,该途径利用外膜(tom)复合物转运蛋白和内膜23复合物(tim23)转运蛋白。然而,一些具有可切割前导序列的蛋白可以在内膜(murcha等人,2005)或膜间间隙中组装,如果它们还含有疏水性分选信号(hss)(glick等人,1992)。很少有前导序列未被切割的基质定位蛋白的实例。在拟南芥中,在具有未加工的全长前导序列的基质中仅发现谷氨酸脱氢酶(huang等人,2009)。对于不是基质靶向的蛋白,使用各种内部不可切割的定位信号。这些通常与特定的运输途径相关,并且额外针对特定种类的蛋白进行定制。在植物中,迄今为止没有研究确定是什么精确地构成膜间间隙蛋白的内部信号序列。然而,看来具有双半胱氨酸残基的基序与经线粒体膜间间隙组装途径(mia)的转移相关(carrie等人,2010;darshi等人,2012)。最后,不可切割的内部序列也被通过载体(carrier)途径到达内膜的蛋白利用,其利用tom和tim22器插入具有多个跨膜区的蛋白(kerscher等人,1997;sirrenberg等人,1996)。这些序列通常含有疏水区,其后是前导序列样内部序列,因此与n端前导序列相似,但区别在于它们在它们的同源蛋白内的内部位置。在光合生物体中,核编码的线粒体蛋白需要在叶绿体和线粒体转运(mitochondrialtrafficking)之间进行区分,尽管这两种细胞器和它们的蛋白质组之间存在许多相似性。大部分出现在线粒体前导序列中的α-螺旋通常不存在于叶绿体前导序列中(zhang和glaser,2002),其倾向于更加非结构化并显示高β片结构域结构(bruce,2001)。在植物中,mpp锚定于内膜结合的cytbc1复合物,尽管活性mpp位点位于面向基质的位置,并且两种蛋白的功能是独立的(glaser和dessi,1999)。线粒体靶向肽如本文所用,术语“线粒体靶向肽”或“mtp”是指这样的氨基酸序列,其包括至少10个氨基酸,优选长度为10至约80个氨基酸残基,其将靶蛋白导向线粒体,并且其可以异源地用于mtp-靶蛋白翻译融合中以将选定的靶蛋白,例如nif多肽、gus、gfp等导向线粒体。mtp通常在其n端包括衍生它的多肽的翻译起始子甲硫氨酸。mtp通过与对应于靶蛋白起始子met的met残基的肽键与nif多肽或“靶蛋白”翻译融合,或者met残基可以省略并且肽键直接与野生型中作为靶蛋白第二氨基酸的氨基酸残基融合。mtp通常富含碱性和羟基化的氨基酸,并且通常缺乏酸性氨基酸或延长的疏水性伸展。mtp可以形成两亲性螺旋。不希望受理论的限制,mtp通常包括与线粒体外膜上的受体结合的摄取靶向序列。当与外膜结合时,融合多肽优选经历膜易位以转移通道蛋白,并通过线粒体的双层膜到达线粒体基质(mm)。然后通常切割摄取靶向序列并折叠成熟融合蛋白。mtp可包括随后将蛋白靶向线粒体的不同区域如线粒体基质(mm)的额外信号。在一个实施方案中,摄取靶向序列是基质靶向序列。当翻译融合到nif多肽时,mtp可以是可切割的或不可切割的。在一个实施方案中,细胞中产生的mtp-nif融合多肽的至少50%在细胞中被切割。在另一个实施方案中,少于50%的mtp-nif融合多肽在细胞中被切割,例如mtp未被切割。在一个实施方案中,mtp不包括mpp的切割位点。mtp可以包括切割位点(在mtp序列内)。切割后,所得加工产物(即成熟np)的n端部分可包括mtp的一个或多个c端氨基酸。或者,切割位点可以位于融合多肽内,使得整个mtp序列被切割,例如,接头可以包括切割序列。天然线粒体靶向肽位于前体蛋白的n端,并且n端部分通常在导入线粒体期间或之后被切除。切割通常由一般的基质作用蛋白酶(mpp)催化,其在植物中整合到呼吸链的bc1复合物中。该蛋白酶识别近1000种前体蛋白的切割位点,这些蛋白具有广泛的氨基酸序列,几乎没有保守性。在一个实施方案中,mtp包括mpp的蛋白酶切割位点。在另一个实施方案中,通过mpp切割mtp内的融合蛋白产生加工产物。在一个实施方案中,mtp不被切割。本发明人已经证明,mtp的掺入并不总是导致nif蛋白的完全加工。在一些情况下(nifx-flag、nifd-haopt1和nifdk-ha),加工和未加工的nif蛋白都能观察到。考虑到mtp无通用共有序列,并且内部蛋白序列可以影响线粒体靶向(becker等人,2012),本发明人发现nif蛋白之间加工效率的差异也许并不令人惊讶。可用于本发明上下文的合适mtp包括但不限于具有vonheijne(1986)或roise和schatz(1988)定义的一般结构的肽。mtp的非限制性实例是vonheijne(1986)的表i中定义的线粒体靶向肽。在一个实施方案中,mtp是f1-atpγ酶亚基(pfaγ)。合适的pfaγmtp的实例是来自拟南芥(lee等人,2012)的。在一个实施方案中,pfaγmtp长度为77个氨基酸,mmp对其的切割在融合多肽的n端留下38个mtp残基。在优选的实施方案中,pfaγmtp的长度小于77个氨基酸。例如,pfaγmtp的长度可以是约51个氨基酸,mmp对其的切割在融合多肽的n端留下9个mtp残基。本领域技术人员将理解,存在用于预测线粒体蛋白及其靶向序列的软件,例如mitoprotii、psort、targetp、nnpsl。mitoprotii是基于若干物理化学参数(例如,n端部分中的氨基酸组成,或17个残基窗口的最高总疏水性)预测序列的线粒体定位的程序。psort是基于各种序列衍生特征(例如序列基序和氨基酸组成的存在)预测亚细胞定位的程序。targetp基于任何n端序列的预测存在预测真核蛋白的亚细胞定位:叶绿体转移肽、线粒体靶向肽或分泌途径信号肽。利用早期的二进制预测器、signalp和chlorop,targetp需要n端序列作为两层人工神经网络(ann)的导入。对于预测含有n端前导序列的序列,也可以预测潜在的切割位点。nnpsl是另一种基于ann的方法,其使用氨基酸组成将四种亚细胞定位(胞质、胞外、核和线粒体)之一分配给查询序列。基于常规方法和本文公开的方法,本领域技术人员能够容易地确定所选择的mtp是否将融合多肽靶向至线粒体基质。本发明人选择以前证明能够在拟南芥原生质体中转移gfp的靶向肽(lee等人,2012),并且该靶向肽相对较长,以助于检测加工的蛋白。如本文实施例中所示,选择的mtp将所有选择的固氮酶蛋白靶向至mm。这一结论基于以下证据。首先,观察到的本生烟表达的nif多肽的大小与mm肽酶加工产生的预期大小一致。这也反映在细菌(全长未加工)和植物线粒体表达的小尺寸nifs(niff和nifz)之间观察到的尺寸差异上。此外,mtp的突变,使其不能被线粒体导入机器加工,产生用于nifd和gfp融合的更大条带,这与加工和未加工蛋白之间的大小差异一致。最后,示例性融合多肽的质谱分析确定mtp-nifh在mtp的残基42-43之间被切割,如预测的那样用于基质中的特定加工。在本发明的一些实施方案中,使用选定mtp的多个串联拷贝可能是有用的。复制的或倍增的靶向肽的编码序列可以通过基因工程从现有的mtp获得。mtp的量可以通过细胞分级来测量,然后,例如,定量免疫印迹分析。因此,在本发明中,术语“线粒体靶向肽”或“mtp”包括将靶nif蛋白导向线粒体的一个氨基酸肽的一个或多个拷贝。在优选实施方案中,mtp包括选定mtp的两个拷贝。在另一实施方案中,mtp包括选定mtp的三个拷贝。在另一实施方案中,mtp包括选定mtp的四个拷贝或更多。本领域技术人员将理解mtp序列不限于天然mtp序列,而是可以包括相对于天然存在的mtp的氨基酸替换、缺失和/或插入,条件是序列变异体仍然起线粒体靶向的作用。本领域技术人员将理解,作为克隆策略的结果,mtp可以在其n或c端侧接氨基酸,并且可以用作接头。这些额外的氨基酸可被认为形成mtp的一部分。本领域技术人员还将理解mtp可以在n或c端与寡肽接头和/或标签如表位标签融合。在一个优选的实施方案中,在植物细胞中产生的本发明的一种或多种或所有nif融合多肽相对于相应的野生型nif多肽缺少附加的表位标签。接头本文在多肽的上下文中使用的术语“接头(linker)”或“寡肽接头”是指共价连接两个或多个功能域,例如mtp和np、两种np、np和标签的一个或多个氨基酸。氨基酸通过肽键共价连接,既在接头内也在接头和功能域之间。接头可以提供一个功能域相对于另一个功能域的自由移动,而不会对两个或更多个功能域的功能造成显著的有害影响。接头可以帮助促进一个或两个功能域的适当折叠和功能。本领域技术人员将理解接头的大小可以凭经验确定或可以基于蛋白折叠信息建模。接头可包括蛋白酶如mpp的切割位点。这种接头也可以被认为是mtp的一部分。本领域技术人员将理解mtp的c端可以在没有接头的情况下或通过一个或多个氨基酸残基(例如1-5个氨基酸残基)的接头与np的n端氨基酸翻译融合。这种接头也可以被认为是mtp的一部分。在实施方案中,接头包括至少1个氨基酸、至少2个氨基酸、至少3个氨基酸、至少4个氨基酸、至少5个氨基酸、至少6个氨基酸、至少7个氨基酸、至少8个氨基酸、至少9个氨基酸、至少10个氨基酸、至少12个氨基酸、至少14个氨基酸、至少16个氨基酸、至少18个氨基酸、至少20个氨基酸、至少25个氨基酸、至少30个氨基酸、至少35个氨基酸、至少40个氨基酸、至少45个氨基酸、至少50个氨基酸、至少60个氨基酸、至少70个氨基酸、至少80个氨基酸、至少90个氨基酸、或约100个氨基酸。在实施方案中,接头的最大长度是100个氨基酸,优选60个氨基酸,更优选40个氨基酸。在一些实施方案中,接头允许一个功能域相对于另一个功能域移动以增加融合多肽的稳定性。如果需要,接头可以包括聚甘氨酸的重复或甘氨酸、脯氨酸和丙氨酸残基的组合。用于连接两个nif多肽(例如nifd-接头-nifk和nife-接头-nifn)的接头优选地基于几个标准来选择接头中氨基酸的数量和序列。这些是:缺少半胱氨酸残基以避免形成不需要的二硫键,很少或优选没有带电荷的残基(glu、asp、arg、lys)以降低不需要的表面盐桥相互作用的可能性,很少或没有疏水残基(phe、trp、tyr、met、val、ile、leu),因为这些残基可以促进穿透多肽表面的倾向。并且缺乏可翻译后修饰的氨基酸。在本文中,“少数带电残基”是指少于接头中氨基酸残基的10%,“少数疏水残基”是指少于接头中氨基酸残基的15%。在一个实施方案中,接头不包括半胱氨酸残基。在一个实施方案中,接头包括四个、三个或两个、或一个、或不包括带电残基。优选地,接头总共包括四个、三个或两个、或一个、或不包括谷氨酸、天冬氨酸、精氨酸和赖氨酸残基。在一个实施方案中,接头包括四个、三个或两个、或一个或不包括疏水残基。优选地,接头总共包括四个、三个或两个、或一个或不包括苯丙氨酸、色氨酸、酪氨酸、甲硫氨酸、缬氨酸、异亮氨酸和亮氨酸残基。在一个实施方案中,至少70%、或至少80%、或至少90%的接头包括选自苏氨酸、丝氨酸、甘氨酸和丙氨酸的残基。寡肽接头在修饰多肽中的应用综述于chen等人,(2013)和zhang等人,(2009)。标签在一个特定的实施方案中,融合多肽包括至少一个适于检测或纯化融合多肽或其加工产物的标签(tag)。标签通常与融合多肽的c端或n端结构域结合。在一个优选的实施方案中,标签与nif多肽的c端结合。标签通常是能够以高亲和力与一个或多个配体,例如亲和基质(例如色谱支持物(support)或珠子(bead))的一个或多个配体,或抗体结合的肽或氨基酸序列。本领域技术人员将理解,一旦mtp在导入线粒体后被切除,标签优选位于融合蛋白中不导致标签从np去除的位置。此外,标签不应干扰线粒体导入机制。在优选的实施方案中,本发明的多核苷酸编码融合多肽,该融合多肽以n端到c端顺序包括n端mtp、nif多肽和检测/纯化标签。在另一个实施方案中,融合多肽以n端到c端的顺序包括n端mtp、检测/纯化标签和nif多肽。用于检测、分离或纯化融合多肽或其加工产物的标签的其他说明性、非限制性实例包括人流感血凝素(ha)标签、包括例如6或8个组氨酸残基的组氨酸标签、如荧光素的荧光标签、试卤灵(resourfin)和其衍生物、arg-标签、flag-标签、strep-标签、能够被抗体识别的表位,例如c-myc-标签(被抗c-myc抗体识别)、sbp-标签、s-标签、钙调蛋白结合肽、纤维素结合结构域、几丁质结合结构域、谷胱甘肽s-转移酶-标签、麦芽糖结合蛋白、nusa、trxa、dsba、avi标签等。涉及nif多肽的翻译融合如科学文献中报道的,已经对几种nif多肽进行了翻译融合。这些总结于表2和buren和rubio(2018)的综述中。它们中的大多数涉及为了检测和纯化的目的向蛋白中人工添加表位或结合结构域,例如组氨酸标签或strep标签,并且仅有一些已在植物细胞中表达。有一些关于细菌中nif多肽之间天然存在的融合的报道。对于细菌宿主中的测定,将不同长度的his标签(7-10个组氨酸)加入到nifd(christiansen等人,1998)、nife(goodwin等人,1998)、nifm(gavini等人,2006)以及nifb的全长和截短版本(fay等人,2015)中。在每种情况下,经修饰的nif多肽都保留nif功能,如在细菌或体外固氮酶重建试验中所证实的。thiel等人,(1995)在蓝藻多变鱼腥藻(anabaenavariabilis)中发现了天然存在的29个核苷酸的缺失,因此删除了9个氨基酸和nife终止密码子,这些密码子位于蓝藻多变鱼腥藻中的nife和nifn基因之间的基因间隔区。缺失导致nife-nifn多肽融合,其保留了nife和nifn多肽的至少一些固氮酶功能。nife-nifn融合多肽在融合连接区也具有19个其他氨基酸替换,其可能影响nif功能但方式未知。该融合基因仅在严格厌氧条件下表达。没有报道相对于未融合基因活性是否降低。suh等人,(1996)通过包括nifd的终止密码子和nifk的翻译起始密码子(atg)的缺失,在维涅兰德固氮菌(a.vinelandii)的染色体的nifd和nifk基因之间建立了人工连接,形成了名为pbg1404的载体(vector)。缺失导致nifk多肽的氨基酸2-10中3个氨基酸和7个氨基酸替换的净损失。相对于相应的野生型细菌,含有pbg1404的维涅兰德固氮菌宿主细胞在低氮培养基中的生长受到损害。wiig等人(2011)使用在巴氏梭菌(clostridiumpastuerianum)中发现的nifn和nifb基因之间天然存在的翻译融合,并确定其在细菌和生化互补试验中具有nifn和nifb活性的功能。该融合是直接的,没有任何肽接头,即nifn的c端直接共价连接到nifb的n端。在酵母和植物细胞中,已经使用翻译融合将细胞核中编码的蛋白导向线粒体基质。在酵母表达测定中,线粒体靶向肽(mtp)和一些nif多肽(nifh、nifm、nifs和nifu)的翻译融合在有氧条件下生长时显示出功能(lopez-torrrjon等人,2016)。表位融合多肽(flag和his)在与nifh、nifm、nifs和nifu融合时也显示出功能,尽管这些融合多肽旨在定位于酵母细胞质内,并且仅在酵母在厌氧条件下生长时才有功能。buren等人,(2017)表明,当从酵母的线粒体中重新分离时,nifb可溶性变异体的线粒体基质靶向版本在体外互补试验中具有功能。nifb的这种版本包括n端mtp、nifb的截短变异体(没有nifx样结构域)和c端10×his表位标签。在酵母表达测定中也产生大量mtp-nif融合。然而,这种共表达蛋白的大量集合在酵母中没有显示活性(buren等人,2017)。将来自cpn-60基因的mtp融合到nifh、nifm、nifs和nifu的n端,并通过体外互补试验证明,当从在氧张力降低的10%氧下生长的植物中重新分离铁蛋白时,该基因具有功能(us2016/0304842)。表2.文献报道的nif多肽的基因融合概述多核苷酸术语“多核苷酸”和“核酸”在本文种可互换使用。它们是指任何长度的核苷酸的聚合形式,脱氧核糖核苷酸或核糖核苷酸,或其类似物。本文定义的多核苷酸可以是基因组、cdna、半合成或合成来源、单链或优选双链的并且由于其来源或操作:(1)与天然与其缔合的多核苷酸(例如,不包括天然启动子编码序列的nif多核苷酸)的全部或部分不缔合,(2)连接到天然与其连接的多核苷酸以外的多核苷酸(例如,连接到mtp编码核苷酸序列和/或非天然启动子编码序列的nif多核苷酸),或(3)天然不存在(例如,本发明的编码mtp-nif融合多肽的多核苷酸)。以下是多核苷酸的非限制性实例:基因或基因片段的编码或非编码区,由接头分析定义的多个基因座(单个基因座)、外显子、内含子、信使rna(mrna)、转移rna(trna)、核糖体rna(rrna)、核酶、cdna、重组多核苷酸、分支多核苷酸、质粒、载体(vector)、任何序列的分离dna、任何序列的分离rna、任何序列的嵌合dna、核酸探针和引物。多核苷酸可以包括修饰的核苷酸如甲基化核苷酸和核苷酸类似物。如果存在,可以在组装聚合物之前或之后对核苷酸结构进行修饰。核苷酸序列可以被非核苷酸组件中断。多核苷酸可以在聚合后进一步修饰,例如通过与标签组件结合。“分离的多核苷酸”基本上不含通常与多核苷酸连接的组件(例如调节序列)或缔合的组件。因此,当通过重组技术生产时,分离的多核苷酸基本上不含其他细胞物质或培养基,或者当化学合成时,基本上不含化学前体或其他化学物质。优选地,分离的多核苷酸至少60%,更优选至少75%,更优选至少90%不含所述组件。如本文所用,术语“基因”以其最广泛的上下文来理解,并且包括脱氧核糖核苷酸序列,该脱氧核糖核苷酸序列包括结构基因的转录区和(如果翻译的话)蛋白编码区,并且包括位于5'和3'端上的编码区相邻的序列,在两端至少约2kb的距离,并且参与该基因的表达。在这点上,基因包括控制信号,例如与给定基因天然相关的启动子、增强子、翻译和转录终止和/或聚腺苷酸化信号、或异源控制信号,在这种情况下,基因被称为“嵌合基因”。位于蛋白编码区5'并存在于mrna上的序列称为5'非翻译序列。位于蛋白编码区3'或下游并存在于mrna上的序列称为3'非翻译序列。术语“基因”包括基因的cdna和基因组形式。基因的基因组形式或克隆含有可被称为“内含子”、“间隔区”或“间隔序列”的非编码序列中断的编码区。内含子是转录成核rna(nrna)的基因片段。内含子可以含有调节元件如增强子。从细胞核或初级转录物中除去或“剪接出”内含子;因此,在mrna转录物中不存在内含子。mrna在翻译过程中起作用以确定新生多肽中氨基酸的序列或顺序。术语“基因”包括编码本文所述的本发明的全部或部分蛋白的合成或融合分子和与上述任一种互补的核苷酸序列。如本文所用,“嵌合dna”,在本文中也称为“dna构建体”,是指不是天然存在的,而是人工地将两个dna部分连接到单个分子中的任何dna分子,单个分子的每个部分可以天然存在,但整体天然不存在。例如,编码本发明mtp-nif融合多肽的dna构建体。通常,嵌合dna包括本质上非天然存在的调节和转录或蛋白编码序列(例如,与非天然启动子编码序列连接的nif多核苷酸)。因此,嵌合dna可包括来源于不同来源的调节序列和编码序列,或来源于相同来源但以不同于天然存在的方式排列的调节序列和编码序列。开放阅读框可以或可以不与其天然的上游和下游调控元件连接。开放阅读框可以掺入到,例如,植物基因组中、非天然位置、或非天然发现的复制子或载体(vector)中,如细菌质粒或病毒载体(vector)。术语“嵌合dna”不限于可在宿主中复制的dna分子,而是包括能够通过例如特异性连接体序列连接到复制子中的dna。“转基因”是通过转化方法引入基因组的基因。术语包括引入其祖细胞基因组的子代细胞、植物、种子、非人生物体或其部分中的基因。这样的子代细胞等可以是来自初级转化细胞的祖细胞的至少第3或第4代子代。子代可以通过有性繁殖或营养性繁殖产生,例如来自马铃薯中的块茎或甘蔗中的截根苗。术语“基因修饰的”及其变异是更广泛的术语,包括通过转化或转导将基因引入细胞,突变细胞中的基因并遗传改变或调节细胞或如上所述修饰的任何细胞的子代中基因的调节。本文所用的“基因组区”是指基因组内转基因或转基因组(本文也称为簇)已插入细胞或其前体的位置。这些区域仅包括已通过人的介入(例如通过本文所述的方法)掺入的核苷酸。本发明的“重组多核苷酸”是指通过人工重组方法构建或修饰的核酸分子。与其天然状态相比,重组多核苷酸可以以改变的量存在于细胞中或以改变的速率(例如,在mrna的情况下)表达。在一个实施方案中,将多核苷酸引入不天然包括多核苷酸的细胞中。通常,外源dna用作mrna转录的模板,然后在转化细胞内将mrna翻译成编码本发明多肽的氨基酸残基的连续序列。在另一个实施方案中,多核苷酸对于细菌细胞是内源的,并且其表达通过重组方式改变,例如,将外源控制序列引入目的内源基因的上游,以使转化细胞能够表达由该基因编码的多肽。本发明的重组多核苷酸包括未从其所在的基于细胞或无细胞表达系统的其他组件中分离出来的多核苷酸,以及在所述基于细胞或无细胞系统中生成的多核苷酸,其随后从至少某些其他组件中纯化。多核苷酸可以是天然存在的连续核苷酸片段(例如nif多核苷酸),或包括来自不同来源(天然存在的和/或合成的)的两个或更多个连续核苷酸片段,其连接形成单个多核苷酸(例如与mtp编码核苷酸序列和/或非天然启动子编码序列连接的nif多核苷酸)。通常,这样的嵌合多核苷酸包括至少一个编码本发明多肽的开放阅读框,该开放阅读框与适合于驱动该开放阅读框在目的细胞中转录的启动子可操作地连接。关于定义的多核苷酸,应当理解,高于以上提供的那些的%同一性数字将包括优选的实施方案。因此,在适用时,根据最小%同一性数字,优选多核苷酸包括至少60%、更优选至少65%、更优选至少70%、更优选至少75%、更优选至少80%、更优选至少85%、更优选至少90%、更优选至少91%、更优选至少92%、更优选至少93%、更优选至少94%、更优选至少95%、更优选至少96%、更优选至少97%、更优选至少98%、更优选至少99%、更优选至少99.1%、更优选至少99.2%、更优选至少99.3%、更优选至少99.4%、更优选至少99.5%、更优选至少99.6%、更优选至少99.7%、更优选至少99.8%、甚至更优选与相关命名的seqidno具有至少99.9%同一性的多核苷酸序列。本发明的或对本发明有用的多核苷酸可以在严格的条件下选择性地与本文定义的多核苷酸杂交。本文所用的严格条件是:(1)在杂交过程中使用甲酰胺等变性剂,例如:50%(v/v)甲酰胺加0.1%(w/v)牛血清白蛋白,0.1%菲可(ficoll),0.1%聚乙烯吡咯烷酮,42℃下ph为6.5的50mm磷酸钠缓冲液,含750mmnacl和75mm柠檬酸钠;或(2)使用50%甲酰胺,5×ssc(0.75mnacl,0.075m柠檬酸钠),50mm磷酸钠(ph6.8),0.1%焦磷酸钠,5×denhardt's溶液,42℃下在0.2×ssc和0.1%sds中超声处理的鲑鱼精dna(50g/ml)、0.1%sds和10%葡聚糖硫酸酯,和/或(3)使用低离子强度和高温洗涤,例如50℃下0.015mnacl/0.0015m柠檬酸钠/0.1%sds。与天然存在的分子相比,本发明的多核苷酸可以具有一个或多个突变,该突变是核苷酸残基的缺失、插入或替换。相对于参考序列具有突变的多核苷酸可以是天然存在的(即,从天然来源分离)或合成的(例如,通过如上所述对核酸进行定点诱变或dna改组)。可以对本发明的多核苷酸进行密码子修饰以在植物细胞中表达。本领域技术人员将理解,蛋白编码区可以相对于例如固氮细菌中天然存在的多核苷酸的编码区进行密码子优化。核酸构建体本发明包括含有一种或多种本发明多核苷酸的核酸构建体,含有这些的载体(vector)和宿主细胞,它们的生产和使用的方法,以及它们的用途。本发明涉及可操作地连接或连接的元件。“可操作地连接”或“可操作地连接”等是指功能关系的多核苷酸元件接头。通常,可操作连接的核酸序列是连续连接的,并且当需要连结两个蛋白编码区时,是连续的并且在阅读框中。当rna聚合酶将两个编码序列转录成单个rna时,编码序列“可操作地连接到”另一编码序列,如果被翻译,则单个rna被翻译成具有衍生自两个编码序列的氨基酸的单个多肽。编码序列不必彼此邻接,只要所表达的序列最终被加工以产生所需蛋白即可。如本文所用,术语“顺式作用序列”、“顺式作用元件”或“顺式调节区”或“调节区”或类似术语应理解为意指核苷酸的任何序列,当相对于可表达的遗传序列适当地定位和连接时,其能够至少部分地调节遗传序列的表达。本领域技术人员将意识到顺式调节区能够在转录或转录后水平激活、沉默、增强、阻遏或改变基因序列的表达水平和/或细胞类型特异性和/或发育特异性。在本发明的优选实施方案中,顺式作用序列是增强或刺激可表达遗传序列表达的激活物序列。“可操作地连接”启动子或增强子元件与可转录的多核苷酸是指将可转录的多核苷酸(例如,编码蛋白的多核苷酸或其他转录物)置于启动子的调控之下,然后启动子控制多核苷酸的转录。在异源启动子/结构基因组合的构建中,通常优选将启动子或其变异体定位于距可转录多核苷酸的转录起始位点的距离,该距离与该启动子和它在其天然环境中控制的蛋白编码区之间的距离大致相同;即,衍生启动子的基因。如本领域已知的,可以在不损失功能的情况下适应该距离的一些变化。类似地,调节序列元件(例如,操纵子、增强子等)相对于置于其控制之下的可转录多核苷酸的优选定位由元件在其天然环境中的定位来定义;即,衍生它的基因。本文所用的“启动子”或“启动子序列”是指基因的区域,通常为rna编码区的上游(5'),其控制目的细胞中转录的起始和水平。“启动子”包括经典基因组基因的转录调节序列,如tata盒和ccaat盒序列,以及响应发育和/或环境刺激或以组织特异性或细胞类型特异性方式改变基因表达的额外调节元件(即上游活化序列、增强子和沉默子)。启动子通常,但不是必须的(例如,一些poliii启动子),位于结构基因的上游,其调节其表达。此外,包括启动子的调节元件通常位于基因转录起始位点的2kb内。启动子可以含有额外的特异性调节元件,其位于离起始位点更远的位置以进一步增强细胞中的表达,和/或改变与其可操作地连接的结构基因的表达的时间或诱导性。“组成型启动子”是指指导可操作地连接的转录序列在生物体如植物的许多或所有组织中表达的启动子。本文所用的术语“组成型”不一定表示基因在所有细胞类型中以相同水平表达,而是该基因在广泛的细胞类型中表达,尽管水平上的一些变化通常是可检测的。本文所用的“选择性表达”是指几乎排他地在例如植物的特定器官中表达,例如胚乳、胚、叶片、果实、块茎或根。在优选的实施方案中,启动子选择性地或优先地在植物,优选谷类植物的根、叶片和/或茎中表达。因此,选择性表达可以与组成型表达形成对比,组成型表达是指在植物经历的大多数或所有条件下在植物的许多或所有组织中的表达。选择性表达也可导致特定植物组织、器官或发育阶段中基因表达产物的区室化。在特定亚细胞定位例如质体、胞质溶胶、液泡或质外体空间中的区室化可以通过在基因产物的结构中包括适当的信号来实现,例如,信号肽,用于转移到所需的细胞区室,或在半自主细胞器(质体和线粒体)的情况下,通过将转基因与适当的调节序列直接整合到细胞器基因组中。“组织特异性启动子”或“器官特异性启动子”是相对于许多其他组织或器官优选在一个组织或器官中表达的启动子,优选大多数(如果不是所有)其他组织或器官在例如植物中表达。通常,启动子在特定组织或器官中的表达水平比其他组织或器官高10倍。在一个实施方案中,启动子是茎特异性启动子、叶片特异性启动子或指导植物地上部分(至少茎和叶片)基因表达的启动子(绿色组织特异性启动子),例如核酮糖-1,5-二磷酸羧化酶加氧酶(rubisco)启动子。茎特异性启动子的实例包括但不限于us5,625,136和bam等人(2008)描述的那些。在一个实施方案中,启动子是根特异性启动子,根特异性启动子的实例包括但不限于酸性壳多糖酶基因的启动子和camv35s启动子的特异性亚结构域。本发明预期的启动子对于待转化的宿主植物可以是天然的,或者可以衍生自替代来源,其中该区域在宿主植物中是功能性的。其他来源包括农杆菌t-dna基因,例如用于胭脂碱、章鱼碱、甘露氨酸或其他阿片启动子的生物合成的基因的启动子,组织特异性启动子(参见例如us5,459,252和wo91/13992);来自病毒(包括宿主特异性病毒)的启动子,或部分或全部合成的启动子。在单和双子叶片植物中有功能的许多启动子是本领域熟知的(参见例如greve,1983;salomon等人,1984;garfinkel等人,1983;barker等人,1983);包括从植物和病毒分离的各种启动子,例如花椰菜花叶病毒启动子(camv35s,19s)。medberry等人(1992,1993)、sambrook等人(1989,上文)和us5,164,316公开了评估启动子活性的非限制性方法。或者或另外,启动子可以是诱导型启动子或发育调节型启动子,其能够驱动引入的多核苷酸在例如植物的适当发育阶段表达。可以使用的其他顺式作用序列包括转录和/或翻译增强子。增强子区域是本领域技术人员熟知的,并且可以包括atg翻译起始密码子和相邻序列。当包括起始密码子时,起始密码子应与编码序列阅读框同相,其涉及外来或外源多核苷酸,以确保整个序列的翻译,如果要翻译的话。翻译起始区可由转录起始区的来源提供,或由外来或外源多核苷酸提供。该序列还可以来源于选择用于驱动转录的启动子的来源,并且可以被特异性修饰以增加mrna的翻译。本发明的核酸构建体可以包括约50—1,000个核苷酸碱基对的3'非翻译序列,其可以包括转录终止序列。3'非翻译序列可以含有转录终止信号,其可以包括或不包括聚腺苷酸化信号和能够影响mrna加工的任何其他调节信号。聚腺苷酸化信号用于将聚腺苷酸段添加到mrna前体的3'端。聚腺苷酸化信号通常通过存在与标准型5'aataaa-3'的同源性而被识别,尽管变异并不少见。不包括聚腺苷酸化信号的转录终止序列包括poli或poliii聚合酶的终止子,其包括一组4个或更多个胸腺嘧啶的序列。合适的3'非翻译序列的实例是含有来自根癌土壤杆菌(agrobacteriumtumefaciens)的章鱼碱合酶(ocs)基因或胭脂碱合酶(nos)基因的聚腺苷酸化信号的3'转录非翻译区(bevan等人,1983)。合适的3'非翻译序列也可以来源于植物基因,例如核酮糖-1,5-二磷酸羧化酶(ssrubisco)基因,尽管也可以使用本领域技术人员已知的其他3'元件。由于插入转录起始位点和编码序列起点之间的dna序列,即非翻译的5'前导序列(5'utr),可以影响基因表达,如果它被翻译和转录,也可以使用特定的前导序列。合适的前导序列包括那些包括选择用于指导外来或内源dna序列最佳表达的序列的前导序列。例如,这样的前导序列包括优选的共有序列,其可以增加或维持mrna稳定性并防止翻译的不适当启动,例如joshi(1987)所描述的那样。载体(vector)本发明包括载体(vector)用于操作或转移基因构建体的用途。载体(vector)是可用于人工携带外来遗传物质的核酸分子,优选dna分子;进入另一个细胞中,在那里复制或表达。含有外来dna的载体(vector)称为“重组载体(vector)”。载体(vector)的实例包括但不限于质粒、病毒载体(vector)、粘粒、染色体外元件、微染色体、人工染色体。载体(vector)可包括转座元件。载体(vector)优选是双链dna并且含有一个或多个独特的限制性位点,并且能够在包括靶细胞或组织或祖细胞或其组织的确定的宿主细胞中自主复制,或者能够整合到确定宿主的基因组,优选核基因组中,使得克隆序列可再现。因此,载体(vector)可以是自主复制载体(vector),即作为染色体外实体存在的载体(vector),其复制独立于染色体复制,例如线性或闭合环状质粒、染色体外元件、微染色体或人工染色体。载体(vector)可以含有确保自我复制的任何手段。或者,载体(vector)可以是当引入细胞时整合到受体细胞基因组,优选核基因组中并与其整合的染色体一起复制的载体(vector)。载体(vector)系统可包括单一载体(vector)或质粒、两种或多种载体(vector)或质粒,它们一起含有待引入宿主细胞的总dna、或转座子(transposon)。载体(vector)的选择通常取决于载体(vector)与载体(vector)将要引入的细胞的相容性。载体(vector)还可包括选择标志,例如抗生素抗性基因、除草剂抗性基因或其他可用于选择合适转化体的基因。这些基因的实例通常为本领域技术人员已知。本发明的核酸构建体可以引入载体(vector),如质粒。质粒载体(vector)通常包括提供原核和真核细胞中表达盒的容易选择、扩增和转化的额外核酸序列,例如puc来源的载体(vector)、psk来源的载体(vector)、pgem来源的载体(vector)、psp来源的载体(vector)、pbs来源的载体(vector)或含有一个或多个t-dna区的二元载体(vector)。额外核酸序列包括复制起源,以提供载体(vector)的自主复制;可选择的标志基因,优选编码抗生素或抗除草剂性;独特的多个克隆位点,提供多个位点以插入核酸序列或核酸构建体中编码的基因;以及增强原核细胞和真核细胞(特别是植物细胞)转化的序列。所谓“标志基因(markergene)”是指将一种独特的表型转入表达该标志基因的细胞,从而使这种转化细胞能够与没有该标志物的细胞区分开来。可选择的标志基因促进了可以基于对选择剂(例如除草剂、抗生素、辐射、热或其他破坏未转化细胞的处理)的抗性来“选择”的性状。可筛选标志基因(或报告基因)促进了可以通过观察或测试,即通过“筛选”(例如,β-葡糖醛酸酶、荧光素酶gfp或未转化细胞中不存在的其他酶活性)鉴定的性状。标志基因和目的核苷酸序列不必连接。为了便于鉴定转化体,核酸构建体理想地包括可选择的或可筛选的标志基因作为外来或外源多核苷酸,或除了外来或外源多核苷酸之外还包括可选择的或可筛选的标志基因。标志的实际选择并不重要,只要它与宿主细胞,优选植物宿主细胞组合是功能性的(即选择性的)。标志基因和外来或外源目的多核苷酸不必连接,因为例如us4,399,216中描述的未连接基因的共转化也是植物转化的有效方法。细菌选择性标志物的实例是促进抗生素抗性的标志物,例如氨苄青霉素、红霉素、氯霉素或四环素抗性,优选卡那霉素抗性。用于选择植物转化体的示例性可选择的标志包括但不限于编码潮霉素b抗性的hyg基因;促进卡那霉素、巴龙霉素、g418抗性的新霉素磷酸转移酶(nptii)基因;来自大鼠肝脏的促进对谷胱甘肽衍生的除草剂抗性的谷胱甘肽-s-转移酶基因,如例如ep256223中描述;谷氨酰胺合酶基因,其在过表达时促进对谷氨酰胺合酶抑制剂如膦丝菌素的抗性,如例如wo87/05327中描述;来自绿色产色链霉菌(streptomycesviridochromogenes)的乙酰转移酶基因,其促进对选择性试剂膦丝菌素的抗性,如例如ep275957中描述;编码5-烯醇莽草酸酯-3-磷酸合酶(epsps)的基因,其促进对n-膦酰基甲基甘氨酸的抗性,如hinchee等人(1988)描述。促进抗双丙氨酰膦抗性的bar基因,如例如wo91/02071中描述;腈水解酶基因,例如来臭鼻克雷伯菌(klebsiellaozaenae)的bxn,其促进对溴苯腈的抗性(stalker等人,1988);促进氨甲蝶呤抗性的二氢叶片酸还原酶(dhfr)基因(thillet等人,1988);突变的乙酰乳酸合酶基因(als),其促进对咪唑啉酮、磺酰脲或其他als抑制化学品的抗性(ep154,204);突变的邻氨基苯甲酸盐合酶基因,其促进对5-甲基色氨酸的抗性;或促进除草剂抗性的茅草枯脱卤素酶基因。优选的可筛选标记包括但不限于编码β-葡糖醛酸酶(gus)酶的uida基因;编码显色底物已知的酶的β-半乳糖苷酶基因;水母发光蛋白基因(prasher等人,1985),其可用于钙敏感生物发光检测;绿色荧光蛋白基因(niedz等人,1995)或其衍生物;荧光素酶(luc)基因(ow等人,1986),其允许生物发光检测,以及本领域已知的其他基因。本说明书中使用的“报告分子”是指通过其化学性质提供分析上可鉴定的信号的分子,信号通过参考蛋白产物促进启动子活性的测定。优选地,核酸构建体稳定地掺入到例如植物的基因组中。因此,核酸包括允许分子掺入基因组的合适元件,或将构建体置于可掺入植物细胞染色体的合适载体(vector)中。本发明的一个实施方案包括重组载体(recombinantvector),其包括至少一种本文定义的多核苷酸,并且能够将多核苷酸递送到宿主细胞中。这种载体(vector)含有异源核酸序列,即不是天然发现的与本发明的核酸分子相邻的核酸序列,并且优选衍生自不同于衍生核酸分子的物种的物种。载体(vector)可以是rna或dna、原核或真核,通常是病毒或质粒。本发明的重组载体包括导致核酸分子表达为融合蛋白的融合序列。重组载体还可以包括在本文定义的多核苷酸的核酸序列周围和/或内部的间隔和/或非翻译序列。优选地,将重组载体(vector)稳定地掺入宿主细胞如植物细胞的基因组中。因此,重组载体可包括合适的元件,其允许载体(vector)掺入到基因组或细胞的染色体中。重组细胞本发明的另一个实施方案包括重组细胞,例如重组植物细胞,其是用本发明的一种或多种多核苷酸、构建体或载体(vector)或其子代细胞转化的宿主细胞。术语“重组细胞”在本文中可与术语“转基因细胞”互换使用。核酸分子向细胞的转化可以通过将核酸分子插入细胞的任何方法来完成。转化技术包括但不限于转染、电穿孔、显微注射脂质转染、吸附和原生质体融合。重组细胞可以保持单细胞或可以生长成组织、器官或多细胞生物体。本发明的转化核酸分子可以保留在染色体外,或者可以整合到转化细胞染色体内的一个或多个位点中,从而保留它们的表达能力。优选的宿主细胞是植物细胞,更优选谷类植物的细胞,更优选大麦或小麦细胞,甚至更优选小麦细胞。重组细胞可以是培养中的细胞、体外细胞、或生物体例如植物中的细胞,或器官例如根、叶片或茎中的细胞。优选地,细胞在植物中,更优选在植物的根、叶片和/或茎中。在一个实施方案中,活性nifdk在植物细胞中的表达需要nifd、nifk、nifh、nifb、nife、nifn和任选的nifu、nifs、nifo、nifv、nify、nifw和/或nifz的表达。在另一个或进一步的实施方案中,活性nifh在植物细胞中的表达需要nifh和nifm以及任选地nifu和/或nifn的表达。在一个实施方案中,在植物细胞中重建固氮酶活性至少需要表达nifd、nifk、nifh、nifb、nife、nifn和nifm。本领域技术人员将理解,较小的nif蛋白子集可导致植物细胞中功能性固氮酶的重建。就发明人所知,将固氮酶基因转移到任何光合生物体的唯一报道描述了衣藻叶绿体基因组中nifh的引入(cheng等人,2005)。nifh能够补充叶绿素生物合成突变体,尽管nifh生物合成前体蛋白nifm、nifs和nifu没有共表达。这证明内源性真核等同物可功能性替代某些nif蛋白。事实上,最近的报道表明,大肠杆菌仅使用8种nif蛋白即可重建固氮酶功能(wang等人,2013),这意味着在植物中实现功能可能不如表达nif蛋白的完整补体复杂尽管本发明人还没有建立植物原位(inplanta)nif蛋白的功能,但生物合成和功能性nif蛋白的所有组成成分(repertoire)有希望可以在潜在支持固氮酶功能的环境中表达。转基因植物如本文中作为名词使用的术语“植物”是指整株植物并且是指植物界的任何成员,但是如作为形容词使用是指存在于植物中、从植物获得、衍生自植物或与植物相关的任何物质,例如植物器官(例如叶片、茎、根、花)、单细胞(例如花粉)、种子、植物细胞等。从中产生根和芽的植物和发芽的种子,也包括在“植物”的含义范围内。本文所用的术语“植物部分”是指从植物获得的并且包括植物基因组dna的一种或多种植物组织或器官。植物部分包括营养结构(例如叶片、茎)、根、花器官/结构、种子(包括胚、子叶片和种皮)、植物组织(例如维管组织,基本组织等)、细胞及其子代。本文所用的术语“植物细胞”是指从植物或植物中获得的细胞,包括原生质体或来源于植物的其他细胞,配子产生细胞和再生为完整植物的细胞。植物细胞可以是培养中的细胞。“植物组织”是指植物中的分化组织或从植物(“外植体”)获得的分化组织或衍生自未成熟或成熟胚、种子、根、芽、果实、块茎、花粉、肿瘤组织(例如冠瘿)和培养中植物细胞的聚集体的各种形式(例如愈伤组织)的未分化组织。种子中或来自种子的示例性植物组织是子叶片、胚和胚轴。因此,本发明包括植物和植物部分以及包括这些的产物。如本文所用,术语“种子”是指植物的“成熟种子”,其可以准备好收获或者已经从植物中收获,例如通常在田间商业收获,或者作为“发育中的种子”,在受精后和种子休眠建立之前和收获之前,在植物中发生。本文所用的“转基因植物”是指含有在相同物种、品种或栽培品种的野生型植物中未发现的核酸构建体的植物。也就是说,转基因植物(转化的植物)含有在转化之前不含有的遗传物质(转基因)。转基因可以包括从植物细胞、或另一种植物细胞、或非植物来源、或合成序列获得或衍生的遗传序列。通常,已经通过人的操作例如通过转化将转基因引入植物,但是本领域技术人员可以使用任何方法。遗传物质优选稳定整合到植物基因组,优选核基因组。引入的遗传物质可包括天然存在于相同物种但以重排顺序或元件的不同排列的序列,例如反义序列。含有这些序列的植物包括在“转基因植物”中。在一个优选的实施方案中,转基因植物对于已经引入的每个基因(转基因)是纯合的,使得它们的子代不会因所需的表型分离。转基因植物对于引入的转基因也可以是杂合的,例如在已经从杂交种子生长的f1子代中。这样的植物可以提供本领域熟知的优点,例如杂种优势。本发明上下文中定义的转基因植物包括已经用重组技术经基因修饰的植物的子代,其中子代包括目的转基因。这样的子代可以通过初级转基因植物的自体受精或通过将这样的植物与相同物种的另一种植物杂交而获得。这通常将调节本文定义的至少一种蛋白在所需植物或植物器官中的产生。转基因植物部分包括含有转基因的所述植物的所有部分和细胞,例如培养组织、愈伤组织和原生质体。可以使用本领域中已知的技术来生产转基因植物,例如牛津大学出版社出版的a.slater等人的《植物生物技术—植物的遗传操作》(plantbiotechnology-thegeneticmanipulationofplants,oxforduniversitypress)(2003)),以及约翰·威利父子出版公司的p.christou和h.klee的《植物生物技术手册》(handbookofplantbiotechnology,johnwileyandsons)(2004)中一般描述的技术。“非转基因植物”是没有通过重组dna技术引入遗传物质进行基因修饰的植物。如本文所用,术语“与同基因植物比较”或类似短语是指相对于转基因植物是同基因的但没有目的转基因的植物。优选地,相应的非转基因植物是与目的转基因植物的始祖相同的栽培品种或品种,或缺乏通常称为“分离体(segregant)”的构建体的同胞植物系,或用“空载体(emptyvector)”构建体转化的相同栽培品种或品种的植物,并且可以是非转基因植物。如本文所用,“野生型”是指未根据本发明修饰的细胞、组织或植物。野生型细胞、组织或植物可用作对照,以比较外源核酸的表达水平或如本文所述修饰的细胞、组织或植物的性状修饰的程度和性质。本发明上下文中定义的转基因植物包括已经用重组技术经基因修饰的植物的子代,其中子代包括目的转基因。这样的子代可以通过初级转基因植物的自体受精或通过将这样的植物与相同物种的另一种植物杂交而获得。转基因植物部分包括含有转基因的所述植物的所有部分和细胞,例如培养组织、愈伤组织和原生质体。预期用于实施本发明的植物包括单子叶片植物和双子叶片植物。目标植物包括但不限于以下:谷类(例如,小麦、大麦、黑麦、燕麦、水稻、玉米、高粱和相关作物);葡萄;甜菜(甜菜和饲料甜菜);梨果、核果和无核小水果(苹果、梨、李子、桃、杏仁、樱桃、草莓、树莓和黑莓);豆科植物(豆角、扁豆、豌豆、大豆);油料植物(油菜或其他芸苔属、芥菜、罂粟、橄榄、向日葵、红花、亚麻、椰子、蓖麻油植物、可可豆、花生);黄瓜植物(西葫芦、黄瓜、甜瓜);纤维植物(棉花、亚麻、大麻、黄麻);柑桔果实(橙子、柠檬、葡萄柚、柑橘);蔬菜(菠菜、莴苣、芦笋、白菜、胡萝卜、洋葱、番茄、马铃薯、辣椒);樟科(鳄梨、肉桂、樟脑);或植物,例如玉米、烟草、坚果、咖啡、甘蔗、茶、葡萄、啤酒花、草皮、香蕉和天然橡胶植物,以及观赏植物(花、灌木、阔叶片树和常青树、例如针叶片树)。优选地,植物是谷类植物,更优选小麦、水稻、玉米、黑小麦、燕麦或大麦,甚至更优选小麦。如本文所用,术语“小麦”是指小麦属的任何物种,包括其始祖(progenitor),以及通过与其他物种杂交产生的其子代。小麦包括具有包括42条染色体的aabbdd基因组结构的“六倍体小麦”和具有包括28条染色体的aabb基因组结构的“四倍体小麦”。六倍体小麦包括普通小麦(t.aestivum)、斯卑尔脱小麦(t.spelta)、莫迦小麦(t.macha)、密穗小麦(t.compactum)、印度圆粒小麦(t.sphaerococcum)、瓦维洛夫小麦(t.vavilovii)及其种间杂交。六倍体小麦的优选物种是普通小麦(t.aestivumssp)(也称为“面包小麦”)。四倍体小麦包括硬粒小麦(t.durum)(本文也称为硬质小麦或圆锥小麦(triticumturgidumssp.durum)),野生二粒麦(t.dicoccoides)、二粒小麦(t.dicoccum)、波兰小麦(t.polonicum)及其种间杂交。此外,术语“小麦”包括六倍体或四倍体小麦的潜在始祖,如a基因组的乌拉尔图小麦(t.urartu)、一粒小麦(t.monococcum)或野生一粒小麦(t.boeoticum),b基因组的拟斯卑尔托山羊草(aegilopsspeltoides),d基因组的节节麦(t.tauschii)(又称为方穗山羊草(aegilopssquarrosa)或粗山羊草(aegilopstauschii))。特别优选的始祖是a基因组始祖,更优选a基因组始祖是一粒小麦(t.monococcum)。用于本发明的小麦栽培品种可以属于但不限于任何上面列出的物种。还包括用传统技术以小麦属(triticumsp.)作为亲本与非小麦物种(如黑麦[secalecereale])有性杂交培育的植物,包括但不限于小黑麦。如本文所用,术语“大麦”是指大麦属的任何物种,包括其祖细胞,以及通过与其他物种杂交产生的其子代。优选植物是商业栽培的大麦属植物,例如大麦的品种或栽培品种或菌株,或适合于谷物的商业生产。已经描述了将基因直接递送到细胞中的四种一般方法:(1)化学方法(graham等人,1973);(2)物理方法如显微注射(capecchi,1980);电穿孔(参见,例如,wo87/06614、us5,472,869、5,384,253、wo92/09696和wo93/21335);和基因枪(参见例如us4,945,050和us5,141,131);(3)病毒载体(vector)(clapp,1993;lu等人,1993;eglitis等人,1988);和(4)受体介导的机制(curiel等人,1992;wagner等人,1992)。可以使用的加速方法包括例如基因枪法等。将转化核酸分子递送至植物细胞的方法的一个实例是微粒轰击。英国牛津出版社出版的yang等人的《基因转移的粒子轰击技术》(particlebombardmenttechnologyforgenetransfer,oxfordpress,england(1994))综述了该方法。可以用核酸包被并通过推动力递送到细胞中的非生物颗粒(微粒)。示例性的颗粒包括由钨、金、铂等组成的那些。基因枪法的一个特别的优点是,除了它是可重复转化单子叶片植物的有效方法之外,既不需要分离原生质体,也不需要农杆菌感染的易感性。适用于本发明的颗粒输送系统是氦加速pds-1000/he枪,可从bio-radlaboratories获得。对于轰击,未成熟胚或衍生的靶细胞如来自未成熟胚的盾片或愈伤组织可以排列在固体培养基上。在另一个可选择的实施方案中,质体可以被稳定地转化。公开的用于高等植物中质体转化的方法包括粒子枪递送含有可选择的标志dna和通过同源重组将dna靶向质体基因组(us5,451,513、us5,545,818、us5,877,402、us5,932479和wo99/05265)。农杆菌介导的转移是用于将基因引入植物细胞的广泛适用的系统,因为dna可被引入整个植物组织中,从而不需要从原生质体再生完整植物。使用农杆菌介导的植物整合载体(vector)将dna引入植物细胞是本领域熟知的(参见例如us5,177,010、us5,104,310、us5,004,863、us5,159,135)。此外,t-dna的整合是导致极少重排的相对精确的过程。待转移的dna区域由边界序列限定,间插dna通常插入植物基因组中。农杆菌转化载体(agrobacteriumtransformationvector)能够在大肠杆菌和农杆菌中复制,以便于如(klee等人植物dna传染性病原体,(kleeetal.,plantdnainfectiousagents,hohnandschell,(editors),springer-verlag,newyork,(1985):179-203)描述的进行操作。此外,农杆菌介导的基因转移载体(vector)的技术进步改善了载体(vector)中基因和限制性位点的排列,有利于构建能够表达多种多肽编码基因的载体(vector)。所述载体(vector)具有方便的多接头区域,其侧接启动子和聚腺苷酸化位点,用于直接表达插入的多肽编码基因,并且适用于本发明目的。此外,含有武装的和解除武装的ti基因的农杆菌可用于转化。在那些农杆菌介导的转化是有效的植物品种中,由于基因转移的简单易行和明确的性质,农杆菌介导的转化成为选择的方法。使用农杆菌转化方法形成的转基因植物通常在一个染色体上含有单个基因座。这种转基因植物可被称为添加基因的半合子植物。更优选的是对于所添加的结构基因是纯合的转基因植物;即,含有两个添加基因的转基因植物,一个基因位于染色体对的每个染色体上的相同基因座。纯合转基因植物可通过将含有单一添加基因的独立分离体转基因植物进行有性杂交(自交),使一些产生的种子萌发,并对产生的植株进行目的基因分析。还应当理解,也可以使两种不同的转基因植物交配以产生含有两种独立分离的外源基因的子代。合适子代的自交可以产生对两种外源基因都纯合的植物。也考虑了与亲本植物的回交和与非转基因植物的远交,如营养繁殖。关于其他常用于不同性状和作物的育种方法的描述可以在威斯康星州麦迪逊市美国农学会fehr的《用于栽培种发育的育种方法》(fehr,breedingmethodsforcultivardevelopment(),j.wilcox(editor)americansocietyofagronomy,madisonwis.(1987))中找到。植物原生质体的转化可以使用基于磷酸钙沉淀、聚乙二醇处理、电穿孔和这些处理的组合的方法来实现。这些系统在不同植物品种中的应用取决于从原生质体再生该特定植物株的能力。描述了从原生质体再生谷物的说明性方法(fujimura等人,1985;toriyama等人,1986;abdullah等人,1986)。也可以使用细胞转化的其他方法,包括但不限于通过将dna直接转移到花粉中,通过将dna直接注射到植物的生殖器官中,或通过将dna直接注入未成熟胚的细胞中,然后再水化干燥的胚而将dna引入植物中。从单个植物原生质体转化体或从各种转化的外植体再生、发育和培养植物是本领域熟知的(学术出版社出版的weissbach等人《植物分子生物学方法》(methodsforplantmolecularbiology,academicpress,sandiego,(1988))。这种再生和生长过程通常包括以下步骤:选择转化的细胞,通过胚胎发育的通常阶段通过生根的幼苗阶段培养这些个体化的细胞。类似地再生转基因胚和种子。然后将得到的转基因生根芽种植在合适的植物生长培养基如土壤中。含有外来、外源基因的植物的发育或再生是本领域熟知的。优选地,再生植物自花授粉以提供纯合转基因植物。否则,将从再生植物获得的花粉与农业上重要品系的种子生长植物杂交。相反,来自这些重要品系植物的花粉用于给再生植物授粉。使用本领域技术人员熟知的方法培养含有所需外源核酸的本发明转基因植物。主要通过使用根癌土壤杆菌转化双子叶片植物并获得转基因植物的方法已经公开用于棉花(us5,004,863、us5,159,135、us5,518,908);大豆(us5,569,834、us5,416,011);芸苔属(brassica)(us5,463,174);花生(cheng等人,1996);和豌豆(grant等人,1995)。用于通过引入外源核酸将遗传变异引入植物来转化谷类植物如小麦和大麦和用于从原生质体或未成熟植物胚再生植物的方法是本领域熟知的,参见例如ca2,092,588、au61781/94、au667939、us6,100,447、wo97/048814、us5,589,617、us6,541,257和在wo99/14314中列出的其他方法。优选地,转基因小麦或大麦植物通过根癌土壤杆菌介导的转化方法产生。可以将携带所需核酸构建体的载体(vector)引入组织培养植物的可再生小麦细胞或外植体、或合适的植物系统如原生质体中。可再生的小麦细胞优选来自未成熟胚的盾片、成熟胚、来自这些胚的愈伤组织或分生组织。为了证实转基因在转基因细胞和植物中的存在,可以使用本领域技术人员已知的方法进行聚合酶链式反应(pcr)扩增或southernblot(southern印迹杂交)分析。转基因的表达产物可以以多种方式中的任何一种检测,这取决于产物的性质,并且包括westernblot和酶测定。一种特别有用的定量蛋白表达和检测不同植物组织中复制的方法是使用报告基因,如gus。一旦获得转基因植物,它们可以生长以产生具有所需表型的植物组织或部分。可以收获植物组织或植物部分,和/或收集种子。种子可用作培育具有所需特征的组织或部分的额外植物的来源。“聚合酶链式反应”(pcr)是一种利用由“上游”和“下游”引物组成的“引物对”或“引物组”,以及聚合催化剂(如dna聚合酶)和通常是热稳定聚合酶,对目标多核苷酸进行复制拷贝的反应。pcr的方法是本领域已知的,例如,参见牛津大学bios科学出版社有限公司出版的mcpherson和s.gmoller(编者)的“pcr”(m.j.mcphersonands.gmoller(editors),biosscientificpublishersltd,oxford,(2000))。可以对从表达本发明多核苷酸的植物细胞分离的逆转录mrna获得的cdna进行pcr。然而,如果对从植物分离的基因组dna进行pcr,通常将更容易。引物是能够以序列特异性方式与靶序列杂交并在pcr过程中延伸的寡核苷酸序列。扩增子或pcr产物或pcr片段或扩增产物是包括引物和新合成的靶序列拷贝的延伸产物。多重pcr系统含有多组导致同时产生一个以上扩增子的引物。引物可以与靶序列完全匹配,或者它们可以含有内部错配碱基,其可以导致在特定靶序列中引入限制性酶或催化性核酸识别/切割位点。引物还可以含有额外的序列和/或含有修饰的或标记的核苷酸以促进扩增子的捕获或检测。dna热变性、引物与其互补序列退火和退火引物用聚合酶延伸的重复循环导致靶序列指数扩增。术语靶或靶序列或模板是指扩增的核酸序列。核苷酸序列的直接测序方法是本领域技术人员熟知的,可以在例如ausubel等人,(同上)和sambrook等人,(同上)中找到。测序可以通过任何合适的方法进行,例如双脱氧测序、化学测序或其变化形式。直接测序具有确定特定序列的任何碱基对变化的优点。植物/谷物加工可以使用本领域已知的任何技术加工本发明的谷物/种子,优选谷类谷物或本发明的其他植物部分以生产食品配料、食品或非食物产品。在一个实施方案中,产品是全谷物粉,例如超细碾磨的全谷物粉,或由约100%谷物制成的面粉。全谷物粉包括精制面粉成分(精制面粉或精制面粉)和粗粒级(超细碾磨的粗粒级)。精制面粉可以是例如通过研磨和筛选清洁的谷物如小麦或大麦谷物而制备的面粉。精制面粉的粒度被描述为这样的面粉,其中不少于98%的面粉可通过一种网(cloth),其具有不大于指定为“212微米(美国70号金属丝)(212micrometers(u.s.wire70))”的金属丝编织网的孔洞(opening)。粗粒级包括麸皮和胚芽中的至少一种。例如,胚芽是在谷物中发现的胚胎植物。胚芽包括脂类、纤维、维生素、蛋白、矿物质和植物营养素,例如黄酮。麸皮包括若干细胞层并且具有显著量的脂质、纤维、维生素、蛋白、矿物质和植物营养素,例如黄酮。此外,粗粒级可以包括糊粉层,糊粉层还包括脂质、纤维、维生素、蛋白、矿物质和植物营养素,例如黄酮。糊粉层虽然在技术上被认为是胚乳的一部分,但表现出许多与麸皮相同的特征,因此通常在碾磨过程中与麸皮和胚芽一起除去。糊粉层含有蛋白、维生素和植物营养素,例如阿魏酸。此外,粗粒级可以与精制面粉成分混合。粗粒级可以与精制面粉成分混合以形成全谷物粉,从而提供与精制面粉相比具有营养价值、纤维含量和抗氧化能力增加的全谷物粉。例如,粗粒级或全谷物粉可以以各种量使用以代替烘焙食品、零食产品和食物产品中的精制或全谷物粉。本发明的全谷物粉(即超细碾磨的全谷物粉)也可以直接销售给消费者用于其自制烘焙产品。在一个示例性实施方案中,全谷物粉的颗粒分布使得98%重量的全谷物粉颗粒小于212微米。在进一步的实施方案中,在全谷物粉和/或粗粒级的麸皮和胚芽中发现的酶被灭活,以便稳定全谷物粉和/或粗粒级。稳定化是使用蒸汽、热、辐射或其他处理来灭活麸皮和胚芽层中发现的酶的方法。已经稳定的面粉保持其烹饪特性并具有较长的保质期。在额外的实施方案中,全谷物粉、粗粒级或精制面粉可以是食物产品的组件(配料)并且可以用于生产食物产品。例如,食物产品可以是百吉饼、饼干、面包、小圆面包、羊角面包、饺子、英式松饼、松饼、皮塔饼、快速面包、冷藏/冷冻面团产品、面团、烘焙豆、墨西哥卷饼、辣椒、墨西哥煎玉米粉卷(taco)、玉米面包卷的辣味肉饼(tamale)、玉米粉圆饼(tortilla)、波特派(potpie)、即食谷物、即食膳食、馅料、可微波膳食、布朗尼(brownie)、蛋糕、奶酪蛋糕、咖啡蛋糕、曲奇饼、甜点、糕点、小甜面包、糖果条、馅饼外壳、饼馅、婴儿食品、烘培混合物、稀面糊、面包屑、肉汁混合物、肉增量剂、肉代用品、调料混合物、汤料混合物、肉汁、油面酱、沙拉调料、汤、酸奶油、意大利面食、日本拉面、炒面面条、捞面面条、冰淇淋夹杂物、雪糕、蛋卷筒、冰淇淋三明治、薄脆饼干、烤面包丁、甜甜圈、蛋卷、挤压零食、水果和谷物棒、可微波的零食产品、营养棒、煎饼、半烘焙的烘培食品、椒盐卷饼、布丁、基于格兰诺拉麦片的产品、零食片、零食食品、零食混合物、华夫饼、比萨饼皮、动物性食品或宠物食品。在可选择的实施方案中,全谷物粉、精制面粉或粗粒级可以是营养补充剂的组件。例如,营养补充剂可以是添加到含有一种或多种额外成分的饮食中的产品,额外成分通常包括:维生素、矿物质、草药、氨基酸、酶、抗氧化剂、草药、香料、益生菌、提取物、益生元和纤维。本发明的全谷物粉、精制面粉或粗粒级包括维生素、矿物质、氨基酸、酶和纤维。例如,粗粒级含有浓缩量的膳食纤维以及其他必需营养素,如维生素b、硒、铬、锰、镁和抗氧化剂,它们对于健康饮食是必需的。例如,22克本发明的粗粒级提供了33%的个体每日推荐纤维消耗量。营养补充剂可包括有助于个体总体健康的任何已知营养成分,实例包括但不限于维生素、矿物质、其他纤维组件、脂肪酸、抗氧化剂、氨基酸、肽、蛋白、叶片黄素、核糖、ω-3脂肪酸和/或其他营养成分。补充剂可以以下形式递送,但不限于以下形式:速溶饮料混合物、即饮饮料、营养棒、威化饼、饼干、薄脆饼干、凝胶丸(gelshots)、胶囊、咀嚼物、咀嚼片和丸剂。一个实施方案以调味摇动或麦芽型饮料的形式递送纤维补充剂,该实施方案作为儿童的纤维补充剂可能特别有吸引力。在另一个实施方案中,碾磨方法可用于制备杂粮面粉或杂粮粗粒级。例如,来自一种类型谷物的麸皮和胚芽可以被磨碎并与磨碎的胚乳或另一种类型谷物的全谷物谷类粉混合。或者,可以将一种类型谷物的麸皮和胚芽磨碎并与磨碎的胚乳或另一种类型谷物的全谷物粉混合。预期本发明包括混合一种或多种麸皮、胚芽、胚乳和一种或多种谷物的全谷物粉的任意组合。这种杂粮方法可用于制作定制面粉,并利用多种类型谷类谷物的品质和营养含量来制作一种面粉。预期本发明的全谷物粉、粗粒级和/或谷物产品可以通过本领域已知的任何碾磨方法生产。示例性实施方案包括在单个流中研磨谷物而不将谷物的胚乳、麸皮和胚芽分离成单独的流。清洁和回火的谷物被输送到第一通道研磨机,例如锤磨机、辊磨机、针磨机、冲击磨机、盘磨机、空气研磨机、凹口碾磨机等。研磨后,将谷物排出并输送到筛子中。此外,预期本发明的全谷物粉、粗粒级和/或谷物产品可以通过许多其他方法改性或增强,例如发酵、速溶、挤出、包封、烘、烤等。麦芽制造本发明提供的麦芽基饮料包括酒精饮料(包括蒸馏饮料)和通过使用麦芽作为其原料的一部分或全部生产的非酒精饮料。实例包括啤酒、发泡酒(happoshu,低麦芽啤酒饮料)、威士忌酒、低醇麦芽基饮料(例如含有少于1%乙醇的麦芽基饮料)和非醇饮料。麦芽制造是控制浸泡和发芽,然后干燥谷物如大麦和小麦谷物的过程。该事件序列对于许多引起谷物修饰的酶的合成是重要的,谷物修饰主要是解聚死亡的胚乳细胞壁并调动谷物营养素的过程。在随后的干燥过程中,由于化学褐变反应产生香味和颜色。尽管麦芽的主要用途是用于饮料生产,但它也可用于其他工业过程中,例如作为烘焙工业中的酶源,或作为食品工业中的调味剂和着色剂,例如作为麦芽或作为麦芽粉,或间接作为麦芽糖浆等。在一个实施方案中,本发明涉及生产麦芽组合物的方法。该方法优选包括以下步骤:(i)提供本发明的谷物,例如大麦或小麦谷物,(ii)浸泡所述谷物,(iii)在预定条件下使浸泡的谷物发芽(iv)干燥所述发芽的谷物。例如,麦芽可以用美国谷物化学师协会出版的hoseney的《谷物科学与技术原理》第二版(principlesofcerealscienceandtechnology,secondedition,1994:americanassociationofcerealchemists,st.paul,minn.)中描述的任何方法生产。然而,任何其他合适的生产麦芽的方法也可以用于本发明,例如生产特殊麦芽的方法,包括但不限于烘焙麦芽的方法。麦芽主要用于酿造啤酒,也用于生产蒸馏酒精。酿造包括麦芽汁生产、主发酵和二次发酵以及后处理。首先将麦芽研磨、搅拌入水中并加热。在该“糖化”过程中,麦芽制造中活化的酶将谷粒的淀粉降解为可发酵的糖。澄清产生的麦芽汁,加入酵母,发酵混合物并进行后处理。固氮酶复合物检测固氮酶复合物的检测可以通过允许检测nifdk蛋白复合物和nifh蛋白之间相互作用的任何方法进行。适于检测nifdk蛋白复合物和nifh蛋白之间相互作用的方法包括本领域已知的用于检测蛋白-蛋白相互作用的任何方法,包括免疫共沉淀、亲和印迹、蛋白质体外结合实验(pulldown)、荧光共振能量转移(fret)等。或者,可以通过测量所得固氮酶复合物的活性来检测固氮酶复合物。适于测量固氮酶活性的方法包括本领域已知的用于检测将固二氮酶经酶还原成氨的任何方法,其中电子从nifh蛋白转移到nifdk蛋白复合物。例如,可以通过乙炔还原检测来估算固氮活性。简言之,该技术是一种间接方法,其利用固氮酶复合物的能力减少三重结合底物。固氮酶将乙炔(c2h2)还原为乙烯(c2h4)。两种气体都可以用气相色谱法定量。氮固定也可以通过析氢测定来测量。h2是n2固定的专性副产物。由此可以通过使用流通式h2传感器或气相色谱法定量气流中的h2浓度来获得固氮酶活性的间接测量。n2固定的检测固氮可以通过确定植物-土壤系统的总n的净增加来估计(n平衡法);2)将植物n分离成从土壤中吸收的级分和衍生自n2固定的级分(n差异,15n天然丰度,15n同种型稀释和酰脲方法)和3)测量固氮酶的活性(乙炔还原和析氢检测)。实施例实施例1.材料和方法植物细胞瞬时表达系统中基因的表达基本上如wood等人,(2009)描述的,基因利用瞬时表达系统在植物细胞中表达。将含有通过强组成型35s启动子在植物细胞中表达的编码区的二元载体(vector)引入根癌土壤杆菌菌株agl1或gv3101中。如wo2010/057246中描述,将用于表达p19病毒沉默抑制因子的嵌合二元载体(vector)35s:p19分别引入agl1中。在添加50mg/l卡那霉素和50mg/l利福平的lb液体培养基中,将重组根癌土壤杆菌细胞在28℃的温度下培养至稳定期。然后在室温下以5000g离心5分钟将细菌沉淀,接着在含有10mmmesph5.7,10mmmgcl2和100μm乙酰丁香酮的压渗缓冲液中重悬至od600=1.0。然后将细胞在28℃下振荡孵育3小时,之后测量od600,并将达到od600=0.125的终浓度所需体积的每种培养物,包括病毒抑制因子构建体35s:p19,加入到新鲜管中。用压渗缓冲液补足最终体积。然后用培养混合物压渗叶片,通常在压渗后再生长3至5天,然后回收叶盘进行分析。为了以组合方式过度表达一个以上的目的基因,每个额外的基因分别引入根癌土壤杆菌菌株中,并像以前一样生长。混合细菌悬浮液,使得每种细菌菌株的终浓度为od600=0.125。含有编码病毒沉默抑制因子35s:p19的细菌菌株以相同浓度包括在所有混合物中。例如,为了在瞬时叶片测定中表达四种基因并包括病毒抑制因子构建体,压渗混合物的最终od600为5×0.125=0.625单位。以前已经证明了以瞬时测定形式在植物细胞内同时过表达各自来自单独t-dna载体(vector)的至少5种基因(wood等人,2009)。叶片组织蛋白提取为了分析t-dna引入后植物细胞中产生的多肽,通过在压渗后5天从压渗区域切除约2×2cm的叶片碎片来获取本生烟叶片样品(除非另有说明)。这些立即冷冻在液氮中并在2mleppendorf管中研磨成粉末。将300μl的缓冲液添加至每个粉末样品。缓冲液含有125mmtris-hclph6.8、4%十二烷基硫酸钠(sds)、20%甘油、60mm二硫苏糖醇(dtt)。将样品在95℃加热3分钟,然后在12000g离心2分钟。除去含有提取的多肽的上清液,根据待检测多肽的预期水平,取10μl至100μl用于westernblot。westernblot分析提取的样品中的多肽通过sds-聚丙烯酰胺凝胶电泳(sds-page)在nupagebistris4-12%凝胶(thermofisher)上在200v下分离约1小时。根据供应商的说明书(thermosfier),使用半干装置将分离的多肽从每个凝胶转移到pvdf膜上。印迹后,凝胶用考马斯亮蓝染料染色1小时,然后在水中漂洗以观察剩余的蛋白,以显示发生了多肽的转移。将具有结合多肽的膜在含有5%脱脂奶粉的tbst缓冲液中于4℃封闭过夜。tbst缓冲液是:购自sigma的抗ha和抗flag抗体。由leilablackman(澳大利亚,堪培拉,澳大利亚国立大学)赠送的抗gfp抗体。抗体以1∶5000稀释加入含5%脱脂奶粉的tbst中,并将膜在溶液中孵育2小时。然后用tbst洗涤膜3×20分钟。第二抗体,immun-star山羊抗小鼠(gam)-hrp缀合物(biorad),以1∶5000加入含5%脱脂牛奶的tbst中并将膜孵育1小时,然后用tbst洗涤膜3×15分钟。对于第二抗体检测,使用amershamecl试剂,用x射线显影剂或在amersham成像仪(amersham)上显影膜。原生质体制备为了从叶片组织分离原生质体,对breuers等人(2012)的方案进行如下修改。压渗后3天(3dpi),切除2cm见方面积的压渗叶片,切成小块并转移到5ml注射器中。加入2ml含有1.5%(w/v)纤维素酶r-10,0.4%(w/v)离析酶(macerozyme)r-10、0.4m甘露醇、20mmkcl、20mmmesph5.6、10mmcacl2和0.1%(w/v)bsa的消解液,并手动施加轻微真空以促进溶液进入叶片组织中的细胞间隙。将溶液和叶片碎片转移到2mleppendorf管中并将混合物在室温下孵育1小时。通过手动翻转试管轻轻地提取得到原生质体。用镊子除去叶片碎片,使原生质体沉淀,然后用成像溶液(0.4m甘露醇、20mmkcl、20mmmesph5.6、10mmcacl2、0.1%bsa)代替溶液。共聚焦激光扫描显微技术与线粒体染色使用具有40×水浸物镜的立式莱卡(leica)共聚焦激光扫描显微镜对原生质体成像。gfp在488nm处激发并在499-535nm范围内记录发射情况。线粒体用100nmmitotrackerredcmxros(thermofisherscientific(赛默飞世尔科技))溶液染色10-20分钟。mitotrackerrredcmxros在561nm处激发并在570-624nm范围内记录发射情况。rna提取、cdna合成与分析为了从已由农杆菌压渗的本生烟细胞中提取rna,约2×2cm左右的叶片用液氮冷冻,研磨成粉末,每个样品加入500μl的trizol缓冲液(thermofisherscientific)。随后,除了这些修改,按照trizol供应商的说明书:重复氯仿萃取并在37℃溶解rna。提取的rna用rq1dna酶(promega)处理以去除任何提取的dna。然后使用植物rnaeasy柱(qiagen)进一步纯化rna制备物。当进行操作时,根据供应商的方案使用superscriptiii逆转录酶(thermofisherscientific)用寡聚dt引物进行cdna合成。对每个rna样品的rt-pcr分析,进行三个单独的cdna合成反应。将20μl的cdna反应在无核酸酶的水中稀释20倍。在qiagenrotorgeneq实时pcr机器上进行qrt-pcr。将9.6μl的每种cdna加入到10μl的2×sensifastnoroxsybrtaq(bioline)和0.4μl的正向和反向引物中,每种引物为10μmol,最终反应体积为20μl。所有qpcr反应(对于参考基因和特异性基因两者)在以下循环条件下重复进行三次:95℃/5分钟进行1个循环,95℃/15秒、60℃/15秒和72℃/20秒进行45个循环。在72℃的步骤中测量荧光。然后进行55℃至99℃熔化循环。利用rotorgene软件包中的比较定量程序,通过对组成型表达的本生烟gadphmrna对照扩增,使基因表达正常化。将代表三次测定平均值的每组三种cdna的值取平均值,从而计算平均值的标准误差(sem)。串联质谱分析在液态n2下研磨压渗的本生烟组织,然后用retschtissue-lyser(高通量组织研磨仪)在2ml的eppendorf管中进行加工,其中含50mmtris-hclph7.5、1mmedta、150mmnacl、0.2%sds、10%甘油、5mmdtt、0.5mmpmsf和植物的1%蛋白酶抑制剂混合物(sigma,货号p9599)。通过离心澄清蛋白提取物,将上清液直接用作导入物,与缀合到琼脂糖珠(sigma,货号a2095)的单克隆抗ha抗体孵育过夜。通过用150mmtris-hclph7.5、5mmedta、150mmnacl、0.1%tritonx-100、5%甘油、5mmdtt、0.5mmpmsf和1%蛋白酶抑制剂混合物进行一系列洗涤以除去未结合的蛋白。通过将琼脂糖珠在laemmli缓冲液(50mmtris-hcl,ph6.8、2%(w/v)sds、0.1%(w/v)溴酚蓝、10%(v/v)甘油、100mmdtt)中于95℃孵育10分钟来洗脱结合的蛋白。通过sds-page分离导入和免疫沉淀的蛋白质样品,并通过从复制凝胶中同时进行western分析确定含有融合多肽(mtp::nifh::ha)的凝胶面积。切除含有融合多肽的区域,用凝胶内胰蛋白酶消化处理,使用与agilentq-tof6550质谱仪联接的agilentchipcube系统通过串联质谱分析分析胰蛋白酶产物(campbell等人,2014)。对从例如添加的胰蛋白酶和角蛋白等常见污染物的胰蛋白酶消化肽段获得的质谱进行鉴定,然后使用剩余的质谱数据搜索数据库,该数据库含有ncbi(数据库编号10/3/2015)中来自nicotiana物种的所有蛋白质序列,加上使用spectrummill软件(agilentrevb.04.01.141sp1),前体质量公差为15ppm,产品质量公差为50ppm,默认q-tof评分和严格的默认‘自动验证’设置的ha序列。丙烯酰胺对半胱氨酸残基的修饰是必需的修饰,甲硫氨酸的氧化是可变的修饰。最初,需要胰蛋白酶切割,并且允许多达两个遗漏的切割。在证实肽匹配后,重复搜索,剩余的不匹配光谱允许非胰蛋白酶切割。分子建模型软件使用如accelrysdiscoverystudio3.5中实现的modeller程序(sali和blundell,2013)构建所有同源性模型。使用针对布鲁克海文蛋白质数据库(brookhavenproteindatabank)的blast搜索确定了建立同源模型的合适模型。使用如discoverystudio3.5中实现的clustalw算法(sali和blundell,2013)进行所有序列比对。所有分子动力学模拟都采用amber12进行。维涅兰德固氮菌的转化根据dossantos(2011)的方法将质粒转化到维涅兰德固氮菌中。简言之,将维涅兰德固氮菌dj1271敲入到缺乏来自dmso储备液的钼酸盐的固态burk's培养基上,再传代一段额外的时间以除去dmso污染的细胞。将一铂环量的细胞接种在50ml缺乏钼酸盐的改良burk's培养基上并在125ml锥形瓶中加入铁。培养物在28℃下以160rpm振荡孵育20-24小时。将1ng所需质粒dna加入到50μl等分试样的感受态细胞中并在室温下孵育20分钟。然后将细胞-dna混合物加入到3.8ml改良的burk's培养基中,并在28℃下以160rpm振荡回收24小时。将回收细胞的等分试样铺在含有6μg/ml卡那霉素和20μg/ml氨苄青霉素的固体改良burk's培养基上,以选择携带pmmb66eh及其衍生物。将板在28℃孵育3-5天。单菌落在固体改良burk's培养基上再传代以获得单菌落分离物。保留氨苄青霉素抗性的再传代分离物用于接种固体改良burk培养基以制备dmso储备液并进一步测试。实施例2.使用来自酵母coxiv基因的mtp将nif多肽靶向至植物线粒体就发明人所知,没有关于在高等植物种,包括例如植物线粒体中生产细菌固氮酶(nif)多肽的公开报道。为了在线粒体中尝试这种生产,本发明人开发了基于植物的,在本生烟叶片中的瞬时表达系统,以确定是否可以在植物细胞中产生和检测nif多肽,并通过使用线粒体靶向肽(mtp)测试基于细菌nif多肽的融合多肽是否可以靶向植物细胞中的线粒体。为了测试对线粒体的定位,选择来源于酵母细胞色素-c氧化酶亚基iv(coxiv)蛋白n端区域的mtp。已经显示coxivmtp在植物细胞中为线粒体定位和融合多肽的加工提供gfp(kohler等人,1997)。coxivmtp(seqidno:1)在切割前仅有29个氨基酸长,比许多其他mtp都短(huang等人,2009)。当在酵母细胞中加工该mtp时,去除17或25个氨基酸(hurt等人,1985),可能从连接到多肽n端的mtp中留下少至4个氨基酸残基。然而,后来在植物细胞中的研究(huang等人,2009)预测,在线粒体基质中,线粒体基质蛋白酶(mmp)加工会在20-21氨基酸处切断丝氨酸-丝氨酸上游的肽,产生一个加工过的融合多肽,其具有来自融合多肽n端的mtp的10个氨基酸残基。本发明人认为较短的mtp序列可能是有利的,特别是如果切割后融合物仅添加10个氨基酸。设计coxivmtp的衍生物,本文称为dcoxiv(seqidno:2)。根据huang等人(2009),dcoxiv包括其参与将多肽引入和加工到线粒体中的基序xrxxxssx(seqidno:3)中的保守精氨酸和丝氨酸残基,他们在线粒体靶向和加工位点的全基因组研究中发现在植物线粒体基质(mm)中导入和加工的最重要的残基是在切割位点上游3或4个残基(-3r或-4r)处的精氨酸和紧接在切割位点(+1s,+2s)之后的两个丝氨酸残基。dcoxiv具有两个额外的插入到n端的氨基酸,并且在seqidno:2中的位置28处也具有谷氨酸,而非天然coxivmtp中的相应谷氨酰胺。预期这些变化不会影响dcoxiv肽的定位或切割。载体(vector)pcw440和pcw441的构建为了将基因引入植物细胞,设计并制备了多用途植物/细菌表达载体(vector)pcw440。这是基于二元载体(vector)以允许在大肠杆菌和根癌土壤杆菌细菌中复制和选择,以及含有用于将来自根癌土壤杆菌的基因转移到植物细胞的t-dna区域。为了制备pcw440,处理pore1(coutu等人,2007)以去除植物可选择的标志基因,产生pore1-null。该载体(vector)含有35s启动子和nos3'转录终止子区。合成dna片段以依次含有t7rna聚合酶的启动子、5'utr序列、由atg起始密码子起始的编码dcoxivmtp的核苷酸序列、限制性酶asci的克隆位点和t7rna聚合酶的转录终止区。该片段的序列部分基于pet14b载体(vector)(novogene)。该片段侧接限制性位点,以允许连接到35s启动子和nos3'区之间的pore-null中,从而产生pcw440。pcw440的t-dna区的核苷酸序列如seqidno:4所示。载体(vector)的t-dna中表达盒的组件在转录方向上依次是camv35s启动子(seqidno:4的核苷酸219-1564),其侧接hindiii位点和xhoi位点便于克隆,以驱动下游蛋白编码区在植物细胞中表达,从而产生融合多肽;然后是t7启动子(核苷酸1571-1587),其允许在编码区的合适大肠杆菌细胞中表达,从而产生相同的多肽;然后编码dcoxivmtp的核苷酸(核苷酸1650-1742),其由atg起始密码子起始;接着是asci的限制性酶切位点(核苷酸1743-1750),其作为插入蛋白编码区的克隆位点;然后是t7rna聚合酶转录终止序列(核苷酸1810-1856)以及最后是nos3'植物转录终止序列(核苷酸1861-2084)。pcw440的遗传图谱如图1示意性地示出。将编码mtp的核苷酸序列插入到pcw440中以允许将任何期望的蛋白编码区插入到asci位点,来提供所编码的蛋白与翻译多肽中dcoxiv氨基酸c端的框内融合。载体(vector)及其衍生物在大肠杆菌中稳定并可用于通过t7聚合酶启动子系统生产蛋白(studier和moffatt,1986)。因此,将多用途pcw440设计为使用市售t7rna聚合酶细胞系,如bl21gold,在细菌中表达融合多肽的基础载体(vector),其中t7启动子和终止子控制融合蛋白的表达。由于细菌缺乏线粒体,大肠杆菌中不会发生对包括mtp的全长融合多肽的加工。在植物细胞中可以使用相同的载体(vector)在35s启动子的控制下表达相同的融合多肽,并且在植物细胞中nos3'终止子使用内源转录机器控制基因表达。为了产生pcw441,通过pcr扩增编码gfp(brosnan等人,2007)的开放阅读框而没有其起始甲硫氨酸残基的dna片段,侧接asci位点以允许在asci位点插入pcw440并允许dcoxiv和gfp之间的翻译融合。asci位点的插入在融合多肽的连接处引入三个额外的氨基酸。将dna片段插入pcw440以产生pcw441。为了测试dcoxiv区域是否能够将gus融合多肽导向植物线粒体,使用实施例1中的方法将包括pcw441(dcoxiv::gfp)的根癌土壤杆菌细胞压渗至本生烟叶片。已知与gfp的n端融合通常不影响其荧光活性(kohler等人,1997),因此如果dcoxiv::gfp多肽表达,预期其能保持荧光活性。同时用根癌土壤杆菌进行对照压渗,根癌土壤杆菌含有表达细胞质定位的gfp、puq214的构建体(brosnan等人,2007)。所有压渗均包括包含编码沉默病毒抑制因子p19的构建体的根癌土壤杆菌细胞,其也被单独用作对照压渗。压渗4天后,用在488nm处蓝光激发的光学显微镜和gfp滤器检查压渗叶片区的荧光,以检测任何gfp多肽。在用pcw441压渗的叶片细胞中观察到荧光。在显微镜下,观察到许多小的亚细胞结构发荧光,结果与以前的报道一致(kohler等人,1997)。这些结构是高度可移动的并且倾向于聚集在细胞边缘,和它们是线粒体一致。相反,已经引入了编码细胞质gfp基因的细胞发出的荧光更均匀。由此得出结论,dcoxiv足以将dcoxiv::gfp多肽引导到植物线粒体中,植物细胞内小的线粒体样颗粒运动就证明了这一点。载体(vector)pcw446、pcw447、pcw448和pcw449的设计和构建基于观察到dcoxiv::gfp融合多肽在瞬时表达后在植物细胞中产生并表现为定位于线粒体,设计并构建了一系列载体(vector)以提供dcoxiv::nif融合多肽的表达。最初,设计并制备基因构建体以表达与肺炎克雷伯菌nifh、nifd、nifk和nify的融合。野生型肺炎克雷伯菌nifh、nifd、nifk和nify的氨基酸序列分别如seqidno:5、6、7和8所示。为了提高翻译效率,使用人类密码子偏倚对这些多肽的蛋白质编码区进行密码子修饰,并由商业供应商(geneart)从核苷酸序列中去除隐蔽剪接位点、隐蔽聚腺苷酸化信号和内部重复序列。对于每种融合多肽,省略nif起始子甲硫氨酸。此外,每个开放阅读框包括编码每个多肽的c端延伸区的核苷酸序列,该延伸包括ha表位或flag表位(例如,wood等人,2006),以便利用来自商业来源的表位的抗体对多肽进行更容易地检测。当翻译的多肽具有相似的大小时,加入不同的表位以通过使用市售的不同抗体来区分蛋白。例如,nifd和nifk融合多肽具有相似的大小,因此nifd与flag表位融合,nifk与ha表位融合。在每个c端添加的氨基酸序列用于ha表位的如seqidno:21所示和用于flag表位的如seqidno:22所示。包括dcoxivmtp和表位的融合多肽的氨基酸序列分别如seqidno:23、24、25和26所示。编码nifh::ha、nifd::flag、nifk::ha和nify::ha多肽的密码子优化的核苷酸序列分别如seqidno:27、28、29和30所示。具有这些核苷酸序列的dna片段由商业供应商合成,每个都包括侧翼asci限制位点,并且每个都插入pcw440的asci位点以产生载体(vector)pcw446(pcoxiv::nifh::ha)、pcw447(pcoxiv::nifd::flag)、pcw448(pcoxiv::nifk::ha)和pcw449(pcoxiv::nify::ha)。融合多肽在细菌中的表达这些载体(vector)的多用途性质允许在合适的细菌细胞中表达基因和产生融合多肽,以提供可在凝胶电泳和免疫检测实验中用作对照的多肽来源。因此,将这些载体(vector)引入大肠杆菌菌株bl21.1-gold(stratagene),其通过在37℃的过夜表达培养基(emd-millipore)中生长进行诱导后,允许在基因构建体中从t7启动子开始表达。离心含有pcw446、pcw447、pcw448或pcw449的细菌培养物以收集细胞。在室温下将细胞在半体积bugbuster试剂(emd-millipore)中裂解2分钟。将裂解物进一步离心以收集包涵体,将其在半体积bugbuster试剂中进一步洗涤两次。将包涵体溶解在含有sds的标准laemmli缓冲液中,并根据需要进一步稀释以在westernblot上产生透明信号。用市售的识别ha或flag表位的抗体(1∶5000稀释度)和兔抗小鼠hrp二抗,chemstar化学发光试剂(amersham)作为最终检测溶液对westernblot进行探测。融合多肽在植物细胞中的表达使用实施例1中描述的方法,将用于表达dcoxiv::nif::ha或flag标记的融合多肽的每个基因构建体分别引入到本生烟叶中。如上所述,所有压渗都包括根癌土壤杆菌细胞,其包括编码沉默病毒抑制因子p19的构建体,以减少基因沉默反应。5天后,通过采用结合ha或flag表位的抗体使用westernblot技术,如实施例1所描述的和上述对细菌产生提取物所做的那样,对压渗区进行收获并处理。这检测了多肽并测定了它们的大小和相对表达水平。在凝胶电泳步骤中,将细菌和植物提取物的等分试样施用于相邻的泳道以允许检测多肽大小可能的较小变化,该较小变化是在dcoxivmtp切割时预测的。例如,全长dcoxiv::nify::ha多肽的大小约为30kda,而预测加工的dcoxiv::nify::ha多肽的大小为28kda。当检查westernblot(图2)时,在引入pcw446的t-dna的样品中容易观察到对应于dcoxiv::nifh::ha多肽的条带存在。根据条带的强度,观察到这种nifh融合多肽在叶片细胞中的表达比nifk和nify融合多肽更强。在植物细胞中产生的nifh融合多肽的大小表现为与细菌表达的多肽相同,在凝胶电泳中约为40kda。此大小与全长dcoxiv::nifh::ha融合蛋白的预期大小一致。以类似的方式,对于已将pcw448的t-dna引入叶片细胞的样品,观察到对应于dcoxiv::nifk::ha多肽的条带,并且对于已引入pcw449的t-dna的样品,观察到对应于dcoxiv::nify::ha的条带。在每种情况下,植物表达的多肽表现为与相应的细菌表达的多肽大小相同,nifk多肽大约60kda,nify多肽大约30kda。基于细菌表达的多肽和植物表达的多肽的迁移率无显著差异,本发明人推断dcoxivmtp在每种情况下在植物细胞中没有经过任何显著地切割。对于dcoxiv::nifd::flag融合多肽,与细菌细胞相比,在植物细胞中观察到了相当不同的结果。当融合多肽在细菌中从pcw447表达,并且使用抗体通过westernblot分析样品以检测flag标记的多肽时,在55kda观察到强条带,正如预期的全长融合多肽(图2)。然而,当将来自pcw447的t-dna引入植物细胞时,dcoxiv::nifd::flag多肽无可检测到的条带,即使在通过成像技术延长westernblot曝光时间后也没有检测到。这表明包含nifd序列的融合多肽没有产生或非常快速地被转换。重复几次使用nifd构建体的实验以检查结果。同样,在植物细胞的提取物中未检测到dcoxiv::nifd::flag多肽,即使在延长westernblot曝光时间后也未检测到。通过用nifd特异性和35s特异性引物测序来检查基因构建体,以证实pcw440骨架中启动子和nifd片段的正确序列。显然,开放阅读框架是完整的,因为相同的构建体在大肠杆菌菌株中表达良好。其他nif融合多肽在植物细胞中的表达利用每种情况下的肺炎克雷伯菌序列,并使用pcw440作为基础载体(vector),构建了类似的基因构建体,编码较大组的nif融合多肽。这包括蛋白编码区的密码子优化\天然nif起始子甲硫氨酸的缺失以及向每个融合多肽添加含有ha或flag表位标签的c端延伸区。这些构建体是pcw452(dcoxiv::nifb::ha;氨基酸序列seqidno:31)、pcw454(dcoxiv::nife::ha;seqidno:32)、pcw455(dcoxiv::nifn::flag;seqidno:33)、pcw456(dcoxiv::nifq::ha;seqidno:34)、pcw450(dcoxiv::nifs::ha;seqidno:35)、pcw451(dcoxiv::nifu::flag;seqidno:36)和pcw453(dcoxiv:nifx::flag;seqidno:37)。对于其中的每一种,在从农杆菌中引入相关的t-dna后,很容易检测到在本生烟细胞中全长融合多肽的产生。在每种情况下,检测到的全长多肽的大小与相应的细菌产生的多肽的大小相同,表明在植物细胞中缺少mmp对mtp的切割。在一些情况下,在westernblot上观察到多个条带。特别地,dcoxiv翻译融合多肽对nifh::ha、nifs::ha和nifn::flag的表达在westernblot上产生了一些较小的条带,可能是由于翻译起始于开放阅读框内的隐藏的内部起始密码子,或由于植物细胞中多肽的切割或在提取过程中,使得c端的表位仍然存在于检测到的条带中。从该分析中,nifh、nifs、nifu和nifx融合多肽的产量表现为比其他多肽高,而nifk、nify、nife、nifn、nifb和nifq融合多肽则检测到在中至低水平。再次,未检测到植物细胞中产生nifd融合多肽。发明人得出结论,除了nifd多肽之外的所有nif融合多肽都可以使用该方法在植物细胞中表达,尽管在密码子使用参数相似、启动子和聚腺苷酸化调控序列相同的情况下,表达水平不同,或者在丰度不同的情况下累积。重要的是,本发明人得出结论,nifd的融合多肽或构建体有些不同寻常,因为它显然是最突出的。实施例3.进一步尝试检测植物细胞中nifd融合多肽的产生鉴于没有检测到如实施例2所述的dcoxiv::nifd::flag融合多肽,发明人假设该nif融合多肽可能对降解敏感,可能与氧浓度、叶片细胞中的光合作用或潜在的折叠错误有关,因此多肽不稳定,可能是由于缺乏推定的伴侣蛋白(ribbe和burgess,2001)。为了验证这些假设,将含有载体(vector)pcw447(其t-dna编码pcoxiv::nifd::flag多肽)的根癌土壤杆菌压渗到本生烟叶片中。切除压渗植物组织并在不同条件下保持在液体培养基上24小时。还测试了这些变化的每一种的组合。变化包括将植物组织维持在氧浓度为21%(环境氧浓度)、5%或1%的黑暗或光照条件下。在一些压渗物中还进行了共同引入基因构建体,以表达groel多肽(ribbe和burgess,2001)或豆血红蛋白(ott等人,2005)(lhb;图6和图3)。这是通过将groel和lbh编码序列插入pore1-35s表达载体(vector)(wood等人,2009)实现的,分别产生pcw-groel和pcw444构建体。将两种构建体转化到农杆菌中并用于如前所述的瞬时叶片表达。所有这些条件或基因的变化都没有导致在本生烟细胞中检测到dcoxiv::nifd::flag融合多肽,即使对照压渗产生了大量诸如dcoxiv::nifn:flag多肽等其他nif融合多肽。本发明人从这些实验得出结论,nifd融合多肽在植物细胞中的表达提出了需要解决的问题。实施例4.在植物细胞中表达nif多肽组合的多种载体(vector)的表达本发明人还测试了四种nif融合多肽是否可以在本生烟叶片系统中共表达,以及这是否可以提高dcoxiv::nifd多肽的产生水平。这是通过混合四种根癌土壤杆菌细胞悬液进行的,每个悬液含有不同的表达基因,即dcoxiv融合到nify::ha、nifd::flag、nifk::ha和nifh::ha,以及含有p19构建体的根癌土壤杆菌。当压渗叶片组织的提取物通过westernblot如前所述进行检测时,这四个nif基因共压渗实验产生了dcoxiv::nifk、dcoxiv::nify和dcoxiv::nifh的条带,尽管每一个都在比单独表达时低得多的水平上检测,但未检测到dcoxivnifd条带。因此,nifd融合多肽再次表现为不同于其他nif多肽。dcoxiv::nifk::ha、dcoxiv::nifh::ha和dcoxiv::nify::ha的组合没有将共同表达的dcoxiv::nifd::flag的产生提高到可检测的水平。讨论在上述实验中,合成并使用基因构建体,每种编码分别与10种不同nif多肽融合的酵母线粒体靶向肽(mtp)dcoxiv的衍生物。这些多肽也被融合到每个c端的ha或flag表位标签上,以检测在细菌或植物细胞中产生的多肽,并在根癌土壤杆菌引入t-dna后在叶片组织中表达。通过仔细比较从细菌和本生烟细胞中提取的多肽的大小来测试线粒体中mtp的加工。观察到,在大肠杆菌中产生dcoxiv-nif融合多肽后,在westernblot分析中很容易检测到所有10种dcoxiv-nif融合多肽,但在引入本生烟细胞后只检测到9种多肽。除了dcoxiv::nifd::flag多肽始终未检测到。改变维持植物组织的环境条件不会导致检测多肽还原氧浓度,将植物组织维持在黑暗与光照中,或者存在共表达的豆血红蛋白或groel伴侣蛋白。nifd是固氮酶复合物的关键组件。因此,进行以下实施例中的实验以试图减轻nifd融合多肽表达的缺乏。实施例5.pfaγmtp引导蛋白进入植物线粒体的验证如实施例2-4所述,证明11个测试的nif融合多肽中的10个在使用dcoxivmtp的本生烟叶片细胞中产生,唯一的例外是nifd融合多肽。在每种情况下,没有观察到靶向肽的加工。因此,本发明人测试了与nifd和其他nif多肽融合的不同mtp序列是否将提供植物细胞中可检测的表达和融合多肽的切割。为此,发明人从拟南芥f1-atp酶γ-亚基(pfaγ)中选择了mtp。长度为77个氨基酸残基的mtp在拟南芥原生质体(lee等人,2012)中通过与gfp报告多肽融合而得到功能性验证。通过基质作用蛋白酶(mpp)切割pfaγmtp序列发生在氨基酸42之后,留下35个氨基酸的c端部分与融合多肽的n端融合。尚不清楚35种氨基酸中有多少是在本生烟叶片细胞中进行线粒体定位和加工所需的,因此发明者决定使用整个77个氨基酸序列。因此,pfaγmtp将融合的nif多肽运输到完整植物叶片细胞的mm的能力用本生烟瞬时叶片测定系统进行了测试。化学合成了970bp的dna片段,其编码由与gfp融合的pfaγmtp的77个氨基酸(seqidno:38的1-77个氨基酸)组成的319氨基酸多肽,gfp(gfp65t,基因库登录号u43284;haas等人,1996)由三个氨基酸gly-ala-pro(gap)分开,并且还包括侧翼ncoi和asci限制位点。ncoi和asci(部分)消化后,将片段插入pcw441的ncoi至asci位点,产生载体(vector)pra01。用asci消化pra01切除gfp编码区,但留下pfaγmtp序列和gap氨基酸,产生载体(vector)pra00,其用作克隆nif融合多肽的基础载体(vector)。当通过在asci位点插入dna序列将nif或其他多肽融合到pfaγmtp和gap氨基酸的下游时,所得到的基因构建体编码pfaγmtp和位于融合连接处的gap氨基酸,从而向nif或其他多肽添加了80个氨基酸的n端延伸区(seqidno:38)。预期线粒体中pfaγmtp的加工从包括起始子(initiator)甲硫氨酸的n端切除42个氨基酸,将表达的多肽的大小减小约4.6kda。因此,预期mtp的切割会从融合至nif或其他多肽n端的c端延伸区留下38个氨基酸。作为编码相同长度n端延伸区但不通过mpp提供线粒体加工的对照载体(vector),制备编码pfaγmtp失效版本的第二载体(vector)。在该载体(vector)中,在mtp的线粒体识别和加工所需的区域引入24个氨基酸替换(lee等人,2012),包括pfaγ切割位点的氨基酸。每个替换用丙氨酸残基取代pra1的pfaγmtp中的野生型氨基酸(图3)。因此,pfaγmtp的这种修饰版本(本文称为mfaγ)将不能被正确加工,而是将产生具有与相应的未加工pfaγ融合多肽相同数目的氨基酸残基的全长融合多肽。因此,该修饰的载体(vector)用作表达融合多肽的基础载体(basevector),以在westernblot分析中提供未加工的分子量对照。载体(vector)pra00及其mfaγ衍生物都是二元载体(vector),用于将它们的t-dna从根癌土壤杆菌转移到植物细胞中。修饰的mfaγn端延伸区的氨基酸序列包括在其c端的gap三联体,如seqidno:39所示。为了测试pfaγmtp将融合多肽定位于植物细胞线粒体的能力,在瞬时本生烟叶片分析中使用pra01。作为检测植物细胞中pfaγ::gfp融合多肽的基质加工的对照,还制作了编码mfaγ::gfp融合多肽的相应载体(vector),命名为pra21(表3)。在以含有pra01或pra21的有根癌土壤杆菌对叶片压渗5天后,从压渗区获得叶片样品,并制备如实施例1所述的蛋白质提取物。使用gfp抗体对蛋白提取物进行sds-page凝胶电泳和westernblot。通过引入pra01(pfaγ::gfp),在具有预期大小(~30kda)的已在预测位点切割的gfp多肽的westernblot上观察到多肽条带,而对于pra21(mfaγ::gfp),观察到未加工融合多肽预期大小的较大条带(~35kda)(图4)。pra01和pra21在~28kda处均有较模糊的条带,在缺乏编码gfp多肽基因的阴性对照中未观察到。这可能代表gfp多肽的降解产物,或来自替代转录或翻译的产物。可见pfaγ::gfp融合多肽的加工是有效的,因为切割形式的条带比未切割形式的条带更强(图4)。发明人的结论是,pfaγ::gfp融合多肽正在完整的本生烟叶片细胞的mm中加工,这意味着至少融合多肽的mtp部分正被运输到mm中并可被mpp获得。为了可视化gfp多肽的定位,如实施例1所述,从含有pra01(pfaγ::gfp)的叶片组织制备原生质体,并通过共聚焦显微技术进行检查。使用具有40×水浸物镜的立式莱卡(leica)共聚焦激光扫描显微镜对原生质体成像。gfp在488nm处激发并在499-535nm范围内记录发射情况。作为鉴定线粒体的复染色,还使用mitotrackerrredcmxros(thermofisherscientific,货号m7512)的100nm溶液将原生质体染色10-20分钟。mitotrackerrredcmxros在561nm处激发并在570-624nm范围内记录发射情况。这样,两种荧光团的荧光图像可以重叠。通过这些方法,观察到gfp荧光与mitotrackerr共定位,并因此定位于线粒体。因此,发明人的结论是,至少对于pfaγ::gfp,mtp正在将融合多肽易位到本生烟叶片细胞的mm,并且还提供了mpp对其进行切割(图4)。这一结论是基于mpp对mtp的处理仅发生在mm中的知识。表3.基因构建体编码nif融合多肽实施例6.nif融合多肽向植物线粒体的易位发明人接下来想要测试pfaγmtp是否能够将nif融合多肽易位至mm,以及它是否能够提供mpp对mtp的切割。为了首先测试这一点,选择两种nif蛋白(niff和nifz)用于构建融合多肽,因为它们的分子量相对较小,这将能够通过westernblot清楚地区分切割和未切割的多肽。与ha或flag表位融合作为c端融合物的肺炎克雷伯菌niff和nifz多肽的蛋白编码区是人密码子优化的,并由商业供应商合成。将dna片段插入pra00的asci位点,使得nif开发阅读框与n端pfaγmtp翻译融合,产生载体(vector)pra05和pra04(表3)。ha和flag表位分别掺入niff和nifz多肽的c端,以允许通过相应的抗体检测。为了产生这些pfaγ::nif融合多肽的未加工版本作为对照,同样的构建体通过t7rna聚合酶在大肠杆菌中表达。考虑到mtp不在没有mpp的细菌中加工,植物和细菌表达的多肽之间的大小差异使得加工能够通过凝胶电泳和westernblot检测。加工前的pfaγ::niff::ha和pfaγ::nifz::flag融合多肽的氨基酸序列分别如seqidno:40和41所示。westernblot(图4)显示,在每种情况下,从本生烟叶片中检测到的多肽的大小都小于大肠杆菌中产生的相应多肽的大小。对于pra05(pfaγ::niff::ha)和pra04(pfaγ::nifz::flag)中的每一个,从植物细胞中检测到的多肽对应于在其mtp中的融合多肽的切割的预期大小,而从大肠杆菌提取物中检测到的多肽是未加工的pfaγ::nif融合多肽的预期大小。本发明人从这些数据得出结论,pfaγmtp能够将至少nif融合多肽的mtp部分转移到mm中,并提供植物细胞中mpp对mtp的切割。实施例7.质谱分析显示mtp切割为了证明融合多肽的pfaγ部分中预期的mtp加工位点被mpp在线粒体中切割,用质谱分析对肽产品进行分析。为此,本发明人设计并制备了基于pra00的基因构建体,其编码融合多肽pfaγ::nifh::ha,命名为pra10(表3)。该融合多肽在加工前的氨基酸序列如seqidno:42所示。由于nifh作为固氮酶复合物的核心组件的重要性和先前在植物细胞中观察到的dcoxiv::nifh::ha多肽的高水平表达,选择nifh融合多肽(实施例2)。将pra10构建体引入本生烟叶片细胞,并于4天后收获压渗组织。在液氮下研磨组织,然后使用retschtissue-lyser(高通量组织研磨仪)在2mleppendorf管中进行加工,其中存在含有50mmtris-hcl,ph7.5、1mmedta、150mmnacl、10%甘油、5mmdtt、0.5mmpmsf和1%植物蛋白酶抑制剂混合物的蛋白提取物缓冲液(peb)(sigma,货号p9599)。为了提高靶向至线粒体的nifh融合多肽的溶解度,将0.2%(w/v)sds加入到peb中。粗蛋白提取物经离心清除,然后在与琼脂糖珠(sigma,货号a2095)缀合的单克隆抗ha抗体存在下孵育,以免疫沉淀含有ha的多肽。通过用150mmtris-hclph7.5、5mmedta、150mmnacl、0.1%tritonx-100、5%甘油、5mmdtt、0.5mmpmsf和1%植物蛋白酶抑制剂混合物系列洗涤除去未结合的蛋白。通过将珠子在laemmli缓冲液中于95℃孵育10分钟洗脱结合的蛋白,然后通过变性sds-page电泳进一步纯化。通过同时进行的westernblot分析确定含有pfaγ::nifh::ha融合多肽的凝胶区域被切除,并用胰蛋白酶进行凝胶内消化,随后使用偶联到agilentq-tof6550质谱仪的agilentchipcube系统对所得多肽进行串联质谱分析,如实施例1所述(campbell等人,2014)。该分析发现5个完全消化的胰蛋白酶肽与nifh内的区域相同,第六个半胰蛋白酶消化肽段与残基42和43之间mtp的精确切割一致(图5)。没有观察到从未加工mtp获得的胰蛋白酶消化肽段sistqvvr(seqidno:43)。相反,检测到的大多数n端肽是半胰蛋白酶istqvvr(seqidno:44),其ms/ms光谱中的一系列完整的γ-离子证实了这一点。这些数据最终证明,至少pfaγ::nifh::ha多肽的mtp部分已经易位到mm,并在n端延伸区内的mtp中的预选位点被mpp切割。数据暗示pfaγmtp含有mm中易位和加工所必需的所有信号。实施例8.多种固氮酶蛋白在植物线粒体基质中的表达鉴于在本生烟叶片细胞中融合到pfaγmtp的gfp、niff、nifz和nifh多肽的线粒体种成功表达和加工,发明人试图将剩余的13个nif多肽作为融合多肽表达到pfaγmtp中。在固氮生物模型肺炎克雷伯菌中,有16个nif蛋白参与固氮酶的生物合成或功能,另外4个蛋白的功能未知或参与转录调控(oldroyd和dixon,2014)。鉴于实施例2-4中给出的数据,本发明人特别关注pfaγmtp是否将提供nifd融合多肽的生产和切割。获得编码肺炎克雷伯菌的16个nif多肽的dna片段的密码子优化版本,每个片段分别插入pra00的asci位点以构建一系列基因构建体(表3)。每个基因构建体编码融合多肽,该融合多肽具有n端pfaγmtp,然后是nif序列(有或没有其起始子甲硫氨酸),然后是包括ha或flag表位的c端延伸区,用于通过合适的抗体检测。对于植物表达,每个构建体包括35s启动子和蛋白编码区侧翼的nos3'转录终止子区。16种融合多肽的氨基酸序列如seqidno:40-42和46-58所示。根癌土壤杆菌细胞分别含有16个基因构建体,分别压渗至本生烟叶片中,4天后,并像以前一样通过凝胶电泳和westernblot进行分析和制备蛋白提取物。对于编码ha标记的pfaγ-nif多肽的每种构建体,在westernblot上检测到大约与mpp预期的大小接近的条带(表3,图6)。在ha标记的多肽中蛋白丰度不同,最容易检测的是nifb、nifh、nifk、nifs和nify融合多肽。niff、nife和nifm多肽以低水平存在,而nifq多肽的检测需要更长的印迹曝光时间以可见。有趣的是,对于nifb、nifs、nifh和nify构建体的压渗,检测到额外的更高分子量的条带(图6),这些构建体对于每个单独的nif::ha结构具有特定大小。与初级条带相比,这些额外的条带的分子量大约是原来的两倍,这向发明者表明,尽管在凝胶电泳过程中存在变性条件,但仍有多肽二聚体形成。据报道,nifb、nifs和nifh蛋白在细菌中起同型二聚体的作用(rubio和ludden,2008;yuvaniyama等人,2000)。在包括nifj、nifn、nifv、nifu、nifx和nifz序列在内的每个构建体的westernblot中观察到flag标记的pfaγ::nif::flag融合多肽(图6,右上组)。flag抗体从本生烟提取物中产生的背景条带比ha抗体多。然而,结果与ha标记的蛋白的结果相似。观察到不同nif::flag融合多肽的信号强度有相当大的变化。此外,观察到另一个对nifu构建体特异的较高分子量条带(图6)。pfaγ::nifx::flag构建体还产生了额外的较小条带,其强度比预期的加工分子量更大。值得注意的是,考虑到16个基因构建体中的15个获得了可检测到的多肽产生,并且尽管大量的压渗复制,但发明人无法在这些植物分析中检测到pfaγ::nifd::flag构建体(pra07)的任何特定条带,即使相同的基因构建体在大肠杆菌中的表达容易产生预期分子量的可见条带(图7)。事实上,即使是细菌产生的提取物以1∶100稀释也能产生一条很容易在westernblot上观察到的条带。来自细菌提取物的结果证实基因构建体pra07是功能性的,至少对于蛋白编码区是功能性的。因此,除了值得注意的pfaγ::nifd::flag多肽外,固氮酶生物合成和功能所需的全套必需nif多肽中的每一种都通过与pfaγmtp融合成功地在植物叶细胞中表达。此外,观察到的条带的分子量与mm中融合多肽的加工一致(表3)。由于固氮酶活性需要许多nif蛋白的协同作用,因此预期植物中功能性固氮酶重建将需要固氮细菌用于生物合成和功能的许多nif蛋白。因此,发明人想要确定多个nif蛋白是否可以使用pfaγmtp在本生烟mm中表达。为了检验这个概念,选择编码四种不同大小的nif融合多肽的基因构建体,其能够通过westernblot分析鉴定所得多肽,即编码pfaγ::nifb::ha(pra03)、pfaγ::nifs::ha(pra16)、pfaγ::nifh::ha(pra10)和pfaγ::nify::ha(pra12)的构建体。用这些构建物转化的四种根癌土壤杆菌培养物以等量混合,然后将混合物压渗到本生烟叶片中。将累积的多肽水平与各培养物分别压渗时进行比较。观察到当从单个构建体表达时,每种多肽比从四个基因组合表达时更丰富。然而,在来自基因组合的蛋白提取物中容易检测到所有四种nif融合多肽,并且对于个体和组合压渗,观察到的每种多肽的分子量是相同的。这表明nif融合多肽的组合可以在植物细胞中产生,并且每个nif融合多肽都可以对线粒体进行预期的靶向和加工。实施例9.本生烟中提高nifd融合多肽产量的尝试鉴于nifd在固氮酶催化活性中的关键作用,发明者试图确定缺少nifd融合多肽产生的原因,并测试了几种提高植物分析中nifd丰度的方法。首先,他们通过测量mrna表达水平来测试nifd融合多肽产生/累积的缺乏是否可归因于低转基因转录或mrna不稳定性。为此,首先通过实施例1中描述的qrt-pcr测定来自pra07的压渗本生烟细胞中的mrna水平,并将其与从编码pfaγ::nifu::flag(pra15)的构建体转录的mrna水平进行比较。该第二构建体用作对照,因为它如上所述在植物细胞中提供高水平的多肽产生。为了去除扩增效率的任何偏倚,使用寡核苷酸引物,其在两个nif融合基因共有的pfaγmtp区内退火。rt-pcr测定结果表明,pfaγ::nifd::flagmrna的表达水平是pfaγ::nifu::flagmrna水平的3倍以上。其次,在植物中由pra07转录的mrna合成cdna,克隆并测序,证明其核苷酸序列碱基完整。因此,pfaγ::nifd::flag融合多肽缺乏累积不是由于mrna低表达或不稳定所致。这些实验还表明,编码pfaγ::nifd::flag的pra07中的t-dna是完全功能的,并且该构建体中的35s启动子也是完全功能的。因此,无法在本生烟细胞中检测到nifd融合多肽并不是由于mrna表达的任何损伤。由于编码pfaγ::nifd::flag的基因的转录和mrna累积显然不限制nifd融合多肽的产生,因此对基因构建体进行了若干次修改,试图克服nifd融合多肽累积的缺乏。首先,考虑并测试了在c端延伸区中flag表位的存在导致nifd融合多肽产生缺乏或不稳定的可能性。为了测试这种可能性,设计并制备了一种构建体,其中用ha表位取代flag表位,命名为pra19(pfaγ::nifd::ha)。ha表位允许累积和检测已经用该表位测试的每个标记的nif融合多肽(表3)。该融合多肽的氨基酸序列如seqid59所示。其次,对nifd::ha开放阅读框架的密码子使用进行了修饰,使其更接近于拟南芥中的密码子使用,而不是针对人类细胞中的翻译进行优化,以试图确定不同的mrna序列是否可以提高翻译效率。该构建体命名为pra24;其编码相同的pfaγ::nifd::ha多肽(seqidno:59)作为pra19。此外,制备了编码具有突变的mfaγn端延伸区的nifd融合多肽版本的基因构建体,而不是如实施例5中描述的pfaγ(mfaγ::nifd::ha)。制备该构建体(pra22)是为了测试线粒体靶向和/或加工(如果它们发生的话)是否至少部分导致nifd融合多肽产生的缺乏。表3列出了为解决这些问题而设计的三种构建体。将含有这些构建体和从压渗组织中制备的蛋白质提取物的根癌土壤杆菌压渗到本生烟叶片并进行分析。值得注意的是,当引入pra19或pra24(pfaγ::nifd::ha)构建体时,westernblot显示基质加工的和未加工的nifd融合多肽预期的分子量都显示含有ha的条带(图8)。pra22的引入仅产生较大的(未加工的)融合多肽。在本实验中,将pra19引入植物细胞,在westernblot中产生比pra24更强的nifd::ha融合多肽条带,但在随后的重复实验中,强度差异不显著。通过将条带的位置与pra22(mfaγ::nifd::ha)和细菌产生的多肽的位置进行比较来验证基质作用(图8)。对大小对应于加工和未加工形式的pfaγ::nifd::ha的两条带的观察表明,该nifd融合多肽的加工效率不如上述其他nif融合多肽。尽管表达构建体设计和表达条件相同,但观察到的pfaγ::nifd::ha多肽的水平仍远低于用作阳性对照的pfaγ::nifk::ha融合多肽(泳道2)的水平。此外,对于三个修饰的nifd::ha构建体中的每一个,在westernblot上观察到更低分子量的额外的条带,其中一些是特定pfaγ::nifd::ha版本特有的。例如,所有修饰的nifd::ha构建体都存在一个约50kda的强条带,但当引入pra22时,出现了样品所特有的一个约40kda的不同的强条带。由于这些条带不存在于pfaγ::nifk::ha或gfp对照中,它们可能代表nifd::ha降解产物或可能是替代转录或翻译起始信号的产物。发明人得出结论,用ha表位替换flag表位至少部分导致nifd融合多肽累积提高。讨论在上述实验中,发明人证明了克雷伯氏菌属中固氮酶功能所需的所有16个nif多肽都可以从植物叶细胞中的基因构建体中表达为mtp::nif融合多肽,并且这些多肽以加工形式累积在线粒体中。此外,实验显示这些蛋白可以靶向mm,mm是潜在适应固氮酶功能的亚细胞定位。发明人相信这些实验是这种方法可行性的第一个实际证明。nif融合多肽靶向mm的结论基于几行证据。首先,每种植物表达的nif多肽的大小与mpp加工产生的预期大小一致。与细菌产生的多肽(全长,未加工的)相比,观察到植物表达的nif融合多肽的较小分子量表明mpp加工nif融合多肽。另外,当mtp序列突变时,使得mtp不能被线粒体导入机加工,观察到nifd和gfp融合多肽的较大的多肽,这与加工和未加工多肽之间的大小差异一致。最后得出的结论是,质谱分析确定pfaγ::nifh如预测的那样在mtp的残基42-43之间被切割,以便在基质中进行特定加工,表明mtp序列在融合到nif多肽时可以被切割。mtp的存在并不总是导致nif蛋白的完全加工。例如,nifx::flag构建体和两个nifd::ha构建体导致加工的和未加工的融合多肽的累积。此外,尽管使用了强的、组成性的35s启动子,但观察到各种nif融合多肽的多肽累积水平有相当程度的可变性。另外,对于一些nif融合多肽,通过在多肽c端存在的表位进行检测,观察到比预期短的多肽。在所有nif融合多肽中,nifd多肽最难以以可检测水平产生。这并不是由于缺少nifd基因表达或mrna不稳定,而是翻译不好和/或蛋白质不稳定可能限制了nifd融合多肽的丰度。鉴于nifd在固氮酶功能中的重要作用,包括其在细菌中高度表达的要求(poza-carrion等人,2014),这个问题需要克服。实施例10.使用计算机模拟(insilico)建模研究固氮酶多肽的结构和功能细菌固氮酶是由需要缔合形成活性酶的多种不同nif多肽组成的金属蛋白酶复合物。本发明人考虑了相对于每个相应的野生型多肽,在每个nif多肽的n端或c端的氨基酸延伸是否可能干扰每个单独多肽的生化功能以及植物细胞内酶复合物的组装和功能。在上述实施例中,使用线粒体靶向肽(mtp)将由引入植物细胞的转基因编码的nif融合多肽导向线粒体基质。然而,该方法用mpp切割前的较长序列或切割后的较短序列延伸每个固氮酶多肽的n端。为了测试延伸是否可能通过干扰功能所需的催化中心或蛋白-蛋白界面而影响固氮酶复合物的功能,发明人首先使用细菌固氮酶亚基的同源性模型来预测n端延伸区和c端延伸区对固氮酶多肽nifh、nifd、nifk、nife、nifn、nifs和nifu的影响。类似地,表位标签如flag、ha和myc表位通常添加到多肽的c端以允许从细胞提取物中检测或纯化。形成野生型细菌固氮酶的许多单个组件和蛋白质复合物已经结晶,并且已经研究了它们作为单多肽或多蛋白复合物的结构。因此,发明人使用这些高分辨率结构来模拟各种变化的影响,例如基于肺炎克雷伯菌氨基酸序列的每个nif多肽的延伸和翻译融合。方法:分子建模软件每个nif多肽和含有这些多肽的复合物的同源模型是使用modeleller程序(sali和blundell,2013)构建的,如在accelrysdiscoverystudio3.5中实现的。使用针对布鲁克海文蛋白质数据库(brookhavenproteindatabank)的blast搜索确定了建立同源模型的合适模板。模板多肽列于表4,其中大部分来自维涅兰德固氮菌。使用如discoverystudio3.5中实现的clustalw算法(sali和blundell,2013)进行序列比对。所有分子动力学模拟都采用amber12进行。表4.用于分子建模的模板多肽1sorlie等人,(2001);2schindelin等人,(1997);3kaiser等人;4cupp-vickery等人,(2003)结果:nifh结构来自nifd-nifk-nifh复合物的维涅兰德固氮菌nifh结构在c端比肺炎克雷伯菌nifh序列短17个氨基酸残基,但在其他方面,两个nifh多肽比其他测试的nif多肽同源性更高(表4)。因此,在从序列中省略了这17个残基之后,构建了如下使用肺炎克雷伯菌nifh多肽序列构建的同源模型。nifd-nifk和nifd-nifk-nifh复合物利用来自肺炎克雷伯菌的nifd和nifk多肽构建了nifd-nifkα2β2异源四聚体的结构模型,并通过分子动力学模拟进行了验证。使用accelrysdiscoverystudio3.5中实现的modeller算法,使用维涅兰德固氮菌的nifd-nifk复合物的晶体结构(pdbid:1fp4,仅nifd和nifk)作为模板,构建该模型。肺炎克雷伯菌与维涅兰德固氮菌的nifd同一性为73.2%,nifk的同一性为65%。然后使用pdbid构建第二个模型:1n2c用于包括肺炎克雷伯菌nifd、nifk和nifh多肽(nifd-nifk-nifhα2β2γ4异源八聚体)的复合物。这样做是为了测试第二个模型是否显示nifd-nifk与nifh亚基的相互作用,该相互作用可能已经受到c端延伸区或n端延伸区的不利影响。本发明人从第二个模型观察到nifk亚基的c端包埋在复合物的核心中,并与相邻nifk和nifd亚基的几个残基接触,包括nifk的5个c端残基和相邻亚基之间的氢键和盐桥。这与在nifd-nifk模型中观察到的一致。发明人得出结论,相对于野生型肺炎克雷伯菌序列的nifk多肽的任何c端延伸区都可能导致复合物的破坏,导致相对于固氮酶活性的不太稳定的形式或完全无功能的复合物。相反,nifd和nifh亚基的c端位于复合物的表面,因此预期可以对nifd和nifh多肽的c端进行延伸而不破坏酶复合物的形成和功能。维涅兰德固氮菌nifh结构的模型预测,由于nifd-nifk-nifh复合物中没有17个残基延伸区可及的‘孔(hole)’,肺炎克雷伯菌c端遗漏的17个氨基酸残基也将终止于表面。类似地,nifd、nifk和nifh亚基的n端也位于复合物的表面,并且推断这些n端应当适合于增加延伸。nifen复合物虽然在布鲁克海文蛋白质数据库(brookhavenproteindatabank)中可以获得nife-nifnα2β2异源四聚体结构与肺炎克雷伯菌nife-nifn序列有合理的同源性,但在nifn结构中的c端缺失了23个残基,这些残基可能折叠回到复合物的核心中,如可获得的等效nifd-nifkα2β2异源四聚体结构中所见。另外,nife结构的n端缺失22个残基,这将给模型增加不必要的不确定性。由于nife-nifnα2β2异源四聚体与来自维涅兰德固氮菌的nifd-nifkα2β2异源四聚体共享相同的折叠(pdbid:1n2c含有nifh-nifd-nifk-nifh复合物,对于nife-nifn和nifd-nifk复合物,叠加rmsd为),因此nifd-nifk结构被用作模板,以在其上构建nife-nifn同源模型。其另一个优点是nife和nifn序列端几乎完全覆盖。这也允许检查nife-nifn端和nifh之间可能的相互作用,如果它们存在的话,以及任何亚基上的端区域的改变是否潜在地干扰复合物形成或活性位点可及性。因此,在维涅兰德固氮菌nifd-nifk结构上构建了肺炎克雷伯菌nife-nifnα2β2异源四聚体模型。类似于nifd-nifk模型,nife和nifn亚基的c端和n端终止于复合物的表面,除了终止于结构核心的nifn的c端。肺炎克雷伯菌nife序列的n端比nifd的n端短18个氨基酸残基,其中19个氨基酸残基保守性较好,与相邻的nifk亚基(同源模型中的nifn)接触。从该模型得出结论,预期nife的c端或n端的延伸不会对复合物的结构或功能产生有害影响,但是nifn的c端不太可能允许延伸。在维涅兰德固氮菌nifk结构的n端存在51个氨基酸残基的相对长的序列,但在肺炎克雷伯菌的nifn序列中没有相应的序列。在该模型中,额外的氨基酸残基位于nifd和nifk单元之间界面的外侧。将催化芯与该序列屏蔽开。从这些观察结果得出结论,pfaγmtp序列在被mpp切割后,留下35个残基的n端延伸区,该35个残基在生理ph下带正电荷(+8),将很可能保持非结构化形貌并保持溶剂暴露。此外,来自nifd-nifk模板结构的先例支持了肺炎克雷伯菌nifn的n端的额外氨基酸残基可被耐受的结论。没有任何迹象表明nifn中的这种n端延伸区将干扰任何单元的催化功能或组装。另一方面,预期nifn的c端包埋在酶的核心内。值得注意的是,它比nifk的高度保守的c端长6个残基,因此这些残基可能参与相互作用的确切性质仍然未知。基于维涅兰德固氮菌nifd-nifkα2β2异源四聚体和肺炎克雷伯菌nife-nifnα2β2异源四聚体在折叠上的相似性,预测对nifn的c端的改变,特别是延伸,将对复合物的二级结构和四元结构产生不利影响。结论是应该使用与野生型nifn多肽相同的c端。nifs模型在功能单元为同源二聚体的大肠杆菌半胱氨酸脱硫酶的结构基础上构建了肺炎克雷伯菌nifs的同源模型。肺炎克雷伯菌nifs的c端的最后11个氨基酸残基不包括在模型中。将肺炎克雷伯菌nifs的同源模型构建为同源二聚体,观察到n端和c端位于结构的表面。对于大多数分析的nif多肽,预测mpp加工后剩余的pfaγmtp氨基酸残基不会对同型二聚体产生不利影响。已经显示nifu和nifs多肽可以相互作用(yuvaniyama等人,2000),然而相互作用的精确物理性质仍然未知。如果存在nifu和nifs之间的物理相互作用,那么n端或c端的延伸可能会破坏nifu和nifs之间的物理相互作用。nifu模型布鲁克海文蛋白质数据库中与肺炎克雷伯菌nifu最接近的匹配是pdbid:2z7e,来自超嗜热菌的铁硫簇蛋白(iscu)结晶为同源三聚体。尽管比对显示出2z7e单体的良好同源性(120个残基),但nifu序列相当长(271个残基),在c端留下约150个残基未模建。因此,以超嗜热菌的铁硫簇蛋白(iscu)为模板,构建了不含其c端151个残基的肺炎克雷伯菌nifu的同源模型。n端被很好地覆盖并聚集在亚基的‘顶部’。如果nifu多肽的n端与mtp序列融合,则不能确定地预测该结构是否允许与nifs多肽相互作用。总之,预期预计可以允许在大多数nif多肽的n端有氨基酸延伸,而没有必要的功能损失。预期对nifk和nifn的c端的延伸对复合物形成和功能的破坏最敏感。这些结果随后被发明人用来帮助指导功能性固氮酶多肽的设计,用于从核基因组中的转基因表达并易位到植物线粒体基质中。实施例11.nifd和其他nif融合多肽的共表达本发明人试图进一步改进nifd融合多肽的产生和累积的方法。在固氮细菌中,nifd、nifk和nifh通过氢键,静电相互作用和疏水相互作用强烈相互作用,形成固氮酶复合物的催化核心。实施例10中描述的蛋白质结构建模表明,nifk与另外两个多肽nifd和nifh之间的有效复合物形成依赖于nifk中存在野生型c端氨基酸序列,即作为c端最后四个残基的氨基酸残基dlvr(seqidno:69)(例如,seqidno:7的残基517-520)。在上述实验中,发明人使用了添加了17个氨基酸的c端序列(包括9个氨基酸的ha表位)的nifk融合多肽。实施例10中描述的建模预测由于c端延伸区,nifk序列对于与nifd相互作用不是最佳的。因此,本发明人决定测试具有野生型c端的nifk多肽与nifd构建体的共表达是否可以改善nifd融合多肽的共累积。以pra00为基础载体(vector),制备了编码无c端延伸区的pfaγ::nifk融合的基因构建体。该构建体命名为pra25(表3)。多肽的氨基酸序列如seqidno:60所示。用pra25(pfaγ::nifk)与pra24(pfaγ::nifd::ha)的组合对本生烟叶片进行压渗,并与以前使用的pra11(pfaγ::nifk::ha)与pra24进行比较。使用ha抗体通过凝胶电泳和westernblot比较nifd多肽的水平。令人惊讶的是,与pra11(pfaγ::nifk无c端延伸区)共压渗相比,与pra25(pfaγ::nifk无c端延伸区或表位)共压渗显著增加了nifd融合多肽的累积。在该实验中,在混合物中包括pra11似乎没有增强nifd融合多肽的累积,但是在重复实验中使用pra11有一些增加。图9显示了nifd构建体单独或与nifk和nifh构建体组合压渗后的westernblot照片。对于图9所示的印迹,当pra19自身被压渗时,需要更长的曝光时间来观察nifd融合多肽的条带(泳道11)。对于其他压渗物,将pfaγ::nifh::ha构建体pra10添加到nifd和nifk的组合中(图9的泳道1-3)。这进一步增强了nifd融合多肽的累积水平。这表明具有野生型c端的nifk多肽的存在稳定了来自pra19的nifd翻译产物,并且添加nifh构建体进一步增强了nifd融合多肽的累积,可能是通过保护其不受蛋白酶活性影响。为了测试nifd融合多肽的改进累积是否在线粒体基质中发生,pfaγ::nifh::ha(pra10)和pfaγ::nifk(pra25)构建体与具有非功能mtp(pra22,mfaγ::nifd::ha)的nifd版本共表达。该融合多肽与pfaγ::nifd::ha的不同之处仅在于n端结构域的氨基酸。观察到,非基质靶向的mfaγ::nifd::ha融合多肽与基质靶向的pfaγ::nifd::ha多肽的累积水平不同(比较泳道2和3,图9)。这证实nifd多肽的增加的累积可能发生在mm中,并提示它们相互作用以形成蛋白复合物。通过观察到在nifk和nifh多肽存在下,与nifd::ha融合的mtp序列的加工程度增加,这一结论得到了加强。当密码子优化的构建体pra24与nifk(无c端延伸区)和nifh构建体一起使用时,nifd融合多肽以甚至更高的水平累积(泳道1,图9)。随着另一次压渗,观察到nifk和nifh构建体的共表达增加了切割的:未切割的pfaγ::nifd::ha融合多肽。为了确定编码nifk的构建体(有或没有c端延伸区)的共表达是否也能增强pfaγ::nifd::flag的累积,将pra25和pra07的组合引入叶片细胞中。在一些压渗物中也进行与pra10的进一步组合。使用检测flag表位的抗体,在westernblot(上组,图10)中检测到nifd::flag融合多肽,表明nifd::flag多肽的累积通过与nifk::ha融合多肽的共同表达而增强。这是发明人第一次检测到nifd::flag融合多肽;参见实施例2-5中报道的先前没有检测的结果。nifd::flag多肽累积的水平在更大程度上通过编码缺少c端延伸区的nifk的构建体的共同表达而得到增强。当nifh和nifk(无c端延伸区)构建体被共压渗时,观察到nifd::flag融合多肽的最高水平(图10,泳道1)。也就是说,向组合物中加入nifh融合多肽进一步提高了nifd融合多肽水平。这一结果表明,无论表位标签(ha或flag)如何,nifd的累积都由nifh:ha和nifk增强,而无c端延伸区。进一步压渗的westernblot如图11所示。本实验还表明,共引入pra10(pfaγ::nifh::ha)和pra25(pfaγ::nifk)可提高pra07的pfaγ::nifd::flag累积水平。再次将和pra25的组合与和pra11(nifk,有c端延伸区)的组合进行比较,参见图11的泳道10和11。当nifd条带的强度被量化并归一化为背景条带之一的强度时,确定与pra11相比,与pra25共表达可使nifd::flag累积水平增加约50%。因此,nifk(无c端延伸区)和nifh的组合有助于除ha标记的nifd多肽之外的nifd融合多肽的累积,并且比具有c端延伸区的nifk多肽增加了nifd融合多肽的累积。总之,这些结果表明nifh、nifk和nifd多肽的组合改善了nifd融合多肽在植物线粒体中的累积。据发明人所知,这是第一个通过与nifh和nifk共表达增强植物细胞中nifd累积的报道,特别是当靶向线粒体时。nifk的野生型c端有利于nifd的累积,nifh进一步增加了nifd融合多肽的丰度。通过nifd编码区的密码子优化,进一步增强了nifh和nifk多肽对nifd累积的积极作用。还注意到,图9的泳道1中nifd融合多肽的丰度接近pfaγ::nifk::ha多肽的丰度。发明人认为这两种关键组件的等摩尔丰度将有益于植物细胞中的固氮酶功能。基于nifk和nifh对nifd融合多肽水平的有益作用,发明人接下来测试了nifs融合多肽的共表达是否可以增强nifd累积。nifs是参与固氮酶组装早期的半胱氨酸脱硫酶,因此它移动硫(s)形成fe-s簇。这些fe-s簇是固氮酶复合物的组成部分。为此,将各自含有一个构建体的根癌土壤杆菌的组合共压渗至本生烟叶片。本实验使用编码pfaγ::nifd::flag(pra07)融合多肽的构建体。令人惊讶的是,pfaγ::nifs::ha构建体(pra16)与nifd::flag构建体的组合也增加了后一种多肽的累积(比较图10中的泳道6和11)。结论是nifs融合多肽的共生产增加了nifd融合多肽的累积。本发明人在该实验中观察到另一个令人惊讶和意想不到的现象。如图10所示,当nifd::flag构建体也被共同引入时,与单独引入这些构建体相比,从它们相应的构建体中检测到的nifh::ha、nifk::ha和nifs::ha融合多肽中的每一个的水平都大大降低。将泳道5与7、6与9和3与8进行比较,的减少百分比nifh::ha为42%、nifs::ha为37%和nifk::ha为36%。可见pfaγ::nifd::flag结构(pra07)的共同引入对共同引入的第二个nif融合构建体的表达产生了负面影响。进一步研究了这种现象。为了显示这些多肽在植物细胞中缔合,引入包括pra07的基因组合后的蛋白提取物与结合flag表位的抗体一起孵育以免疫沉淀nifd::flag多肽和与其结合的任何其他多肽。将沉淀的多肽电泳并用抗ha表位的抗体探测印迹。检测免疫沉淀的并且含有ha表位的多肽,例如pfa::nifh::ha。非变性条件下的尺寸排除色谱也可用于显示多肽蛋白复合物的形成,特别是nifd、nifh和nifk融合多肽的缔合。实施例12.nifdk融合多肽的构建nifh和nifk融合多肽的共同表达对稳定植物细胞中的nifd水平有影响,这一发现促使发明者设计了一种方法,将固氮酶中的nifd异源四聚体配偶体nifk融合到nifk多肽上。据认为该策略还可以保护nifd多肽免受可能的蛋白酶活性的影响,发明人认为这可能是导致低水平的nifd累积的原因。此外,在细菌中,nifd和nifk多肽以几乎相等的量存在(poza-carrion等人,2014),并且在固氮酶的晶体结构中发现亚基的比例为1∶1(schmid等人,2002)。本发明人认为两种多肽的直接融合将确保多肽的比例相等。然而,发现该策略需要插入用于连接nifd和nifk单元的氨基酸接头,以提供单多肽链的适当蛋白折叠,如下所述。以维涅兰德固氮菌(pdbid:1fp4,(schmid等人,2002))的nifd-k复合物的晶体结构为模板,构建了肺炎克雷伯菌nifd-kα2β2异源四聚体的同源模型。将肺炎克雷伯菌与相应的维涅兰德固氮菌多肽进行比对,nifd多肽的氨基酸序列同源性为72.2%,nifk多肽的氨基酸序列同源性为67.3%。肺炎克雷伯菌d2k2异源四聚体的结构模型中,每个nifd亚基的c端距离其nifk配偶体的n端约因此,设计该长度的接头以连接两个单元。接头长度为30个残基,由来自红褐肉座菌外切纤维素酶ii的已知非结构化接头区的11个残基组成(登录号:aag39980.1,atpppgstttr;seqidno:61),其中最后的精氨酸被丙氨酸替换,然后是8个残基的flag表位(dykddk;seqidno:62),最后是11个残基的非结构化接头序列的另一个拷贝,其中精氨酸被丙氨酸(完整接头序列:atpppgstttadykddddkatpppgsttta;seqidno:63)替换。精氨酸的置换被设计为减少与接头的中心flag表位中的负电荷残基的潜在静电相互作用。为了确保在nifd-nifk单元和融合多肽的接头之间或者在接头本身之间不存在可能损害d和k单元在其天然取向上缔合的能力的明显相互作用或构象应变,在八面体tip3p水箱中对复合融合多肽的二聚体(不包括任何金属中心)在恒压下进行几何优化和平衡,该水箱与溶质的边界距离最小。使用amber12进行计算,在总共5ns的298k使用ff99sb力场,使用2fs时间步长和shake约束。检测了nifd-接头-nifk复合多肽(包括其30个氨基酸的接头)的预测结构(图12),表明加入接头允许完整融合多肽折叠成模拟天然nifd-nifk复合物的结构。组装第二个模型,其进一步包括与nifd-接头-nifk多肽的二聚体结合的两个nifh亚基(图13),这表明加入接头似乎不妨碍nifh亚基的结合。因此,基于具有包括pfaγmtp的n端延伸区的pra00,设计并制备了编码融合多肽的构建体(pra02)。pfaγ::nifd-接头-nifk融合多肽的氨基酸序列如seqidno:64所示。在第二构建体中,将包括ha表位的c端延伸区添加到编码的多肽的nifk单位的c端,以使得能够用ha抗体检测融合蛋白,否则编码多肽的剩余部分与pra02相同。第二个构建体命名为pra20。pfaγ::nifd-接头-nifk::ha融合多肽的氨基酸序列如seqidno:65所示。第一个编码的融合多肽具有结构组件pfaγ::nifd-接头(flag)-nifk,而第二个是pfaγ::nifd-接头(flag)-nifk::ha。当将pra20引入到本生烟细胞和蛋白质提取物中时,通过westernblot检查(图14),在预测的大小范围内检测到两条特异性条带,该多肽的大小约为120kda。两条带的紧密程度表明mtp前导序列的未加工形式和加工形式都存在。上条带的强度大于下条带的强度,表明加工仅部分发生。pfaγ::nifd-接头(flag)-nifk::ha多肽的累积水平明显高于平行压渗的pfaγ::nifd::ha,但远低于pfaγ::nifk::ha(图14)。实施例13.用于植物原位共表达固氮酶多肽的多基因构建体的创建为了在植物细胞中产生功能性固氮酶,需要在细胞中协同引入和表达编码三种核心结构蛋白nifd、nifh和nifk以及包括nifb、nife和nifn在内的多种辅助nif蛋白的基因。使用在一个t-dna内含有多个表达盒的转化构建体可以实现多个转基因的基因组整合,每个表达盒指导不同nif多肽的表达。如果需要,这种设计将提供t-dna整合到一个基因组位置。因此,本发明人设计并制备了三个独立的多基因构建体,如果在单个转化事件中组合,其允许将包括15个转基因的三个t-dna整合到植物基因组中。三个t-dna各自含有不同可选择的标志基因,因此它们可以独立地选择,如下所述。从thomasvanhercke博士(csiroagricultureandfood(澳大利亚联邦科学与工业研究组织农业与食品部),canberra,australia)获得的二元载体(vector)pdcot用作起始载体(vector)。它具有与二元载体(vector)pore_o4相同的主链序列和左边界和右边界t-dna序列(coutu等人,2007;genbankaccessionno.ay562542.1)。除了可选择的标志基因盒之外,pdcot的t-dna区含有四个表达盒,每个表达盒包括强的、组成型启动子和转录终止子/聚腺苷酸化区,其侧翼是用于克隆目的的独特限制性位点。在每个启动子之后,每个表达盒还含有烟草花叶病毒ω前导序列(tmvω),其紧接在用于从植物细胞中产生的mrna优化翻译的独特克隆位点之前。改变不同盒中的启动子和终止子序列以限制载体(vector)内的序列重复,考虑到这将减少转化后植物内表达的沉默。另外,盒1和盒2,以及盒4和可选择的标志盒各自被基质相关区域(mar)序列分开,以减少从一个表达盒到另一个表达盒的转录干扰。pdcot中的可选择的标志基因盒是促进对抗生素卡那霉素抗性的nptii基因。pdcot载体(vector)在表达盒的排列中是模块化的,因为每个表达盒依次包括启动子、tmvω、克隆位点和终止子/聚腺苷酸化序列,侧接独特的限制性位点,允许盒的直接交换。为了增加转化载体(vector)中单个基因表达盒的数目,如下将第五个表达盒插入到pdcot中存在于原始pdcot载体(vector)中的独特kpni/aatii位点之间。设计并合成第五表达盒的dna片段以含有增强的camv35s启动子,随后是tmvω前导序列,然后是转基因插入的独特克隆位点(bstz171/sbfi),最后是nos3'终止子区。将侧接5'kpni位点和3'aatii位点的整个选择盒插入pdcot,使用限制性位点定向克隆到pdcot中。这将第五表达盒插入mar序列和可选择的标志基因盒之间。pkt100的结构如图15所示。pdcot及其衍生物pkt100中的一些启动子来自拟南芥(a.thaliana)核酮糖-1,5-二磷酸羧化酶/加氧酶(rubisco)小亚基基因(arath-rubisco/ssu),该基因在光照下提供了绿色组织中的转基因表达,而载体(vector)中的其他盒使用camv35s启动子序列进行组成性表达。对于用于表达nif多肽的起始构建体,期望所有的都应该是组成型表达的,因此arath-rubisco/ssu启动子被其他基于植物病毒的组成型启动子替代,同时减少最终载体(vector)内的序列重复。选择的替代组成型启动子是洋丁香黄曲叶病毒(cestrumyellowleafcurlingvirus,cmylcv)启动子cm4(sahoo等人,2014)和地三叶草矮化病毒(subterraneancloverstuntvirus,scsv)启动子s7和s4的衍生物(schunmann等人,2003)。所用的cm4启动子序列包括相对于cmylcv转录起始位点为-271至+5的核苷酸(sahoo等人,2014),以避免5'端的mlui位点和任何不必要的3'utr序列的存在。每个启动子序列包括5'utr中的tmv前导序列,并且侧接与它们要替换的启动子所使用的限制性位点相匹配的独特限制性位点,特别是cmylcvcm4的mlui/bamhi、scsvs7的nrui/apai和scsvs4的xmai/noti。每个启动子序列由商业供应商合成。标准限制性消化和定向克隆用于以顺序方式交换每个启动子序列以产生pkt-hc。为了使用三种载体(vector)在植物细胞中协调表达多达15个转基因,希望每种载体(vector)具有不同的可选择的标志基因。pdcot选择盒中的可选择的标志基因编码促进卡那霉素抗性的nptii酶(bevan等人,1983)。选择分别促进潮霉素b和草胺磷/草丁磷(basta)抗性的编码hpt(waldron等人,1985)和bar(block等人,1987)的替代基因,因为这些基因适于在植物中选择。编码这些多肽的dna片段由供应商制备,侧翼是fsei/asci限制性位点,其与pdcot选择盒中侧接nptii编码区的限制性位点匹配。标准限制性消化和定向克隆用于用hpt编码区或bar编码区替换pkt-hc中的nptii编码区,产生一组三个多基因载体(vector),称为pkt-hc、pkt-ic和pkt-jc。每个都具有如图15所示的基因结构,组件总结于表5。表5.t-dna载体(vector)中多表达盒组件实施例14.由多基因载体(vector)在植物细胞中表达nif多肽为了证明和比较多基因载体(vector)中每个表达盒的转基因表达能力,制备了一组5个构建体,其中将编码pfaγ::nifh::ha融合多肽的蛋白编码区分别插入每个表达盒。正确插入mtp-nifh-ha的构建体通过其hindiii限制性酶消化模式鉴定,并用于转化根癌土壤杆菌菌株gv3101。然后使用本生烟瞬时叶片表达系统(实施例1)来评估蛋白表达,用三个gv3101菌株的混合物压渗叶片,即一个含有pfaγ::nifh::ha构建体的菌株、第二菌株pra01以及包括表达p19沉默抑制蛋白的载体(vector)的第三菌株,每个菌株由35s启动子驱动。在该实验中,pra01作为gfp荧光合成蛋白的阳性对照。4天后收获叶片组织,如上所述通过凝胶电泳和westernblot分析蛋白质提取物。用抗ha和抗gfp第一抗体和与辣根过氧化物酶(biorad)连接的山羊抗小鼠第二抗体探测westernblot。通过应用amershameclwesternblot检测试剂(gehealthcarelifesciences)产生化学发光信号,并用ccd成像仪记录。从每个单独的表达盒中观察到pfaγ::nifh::ha的表达(图16),从而验证了多基因载体(vector)中的每个启动子在瞬时引入t-dna后以强的、组成性的方式在叶片细胞中表达nif融合多肽的能力。然后构建多基因载体(vector),其基于pkt-hc载体(vector)在一个t-dna中编码5个单独的nif融合多肽。通过每个表达盒中独特的克隆位点以连续方式插入每个nif编码区。pfaγ::nifh::ha的编码区插入盒1,pfaγ::nifs::ha的一个编码区插入盒2,pfaγ::nifm::myc的一个编码区插入盒带3,pfaγ::nifu::ha的一个编码区插入盒4,pfaγ::nifz::myc的一个编码区插入盒5(图4)。pfaγ::nifm::myc融合多肽的氨基酸序列如seqidno:67所示。最终构建体称为hc13,用于转化根癌土壤杆菌菌株gv3101以及转化的细胞用于使用上述方法压渗本生烟叶片。westernblot分析显示从hc13表达的多肽可以在pfaγ::nifh::ha、pfaγ::nifm::myc、pfaγ::nifs::ha和pfaγ::nifu::ha的预期大小下被清楚地检测到(图17)。nifm和nifu融合多肽以比nifh和nifs融合多肽低的水平累积,但仍然可以清楚地检测到。也在对照pfaγ::gfp(泳道2)的泳道中观察到的背景信号与pfaγ::nifz::myc的预期条带处于相同的位置(图17b),尽管泳道4中的信号强度比泳道2中的强得多。本发明人得出结论,所有5种nif融合多肽均由单个、连续的基因构建体表达。植物稳定转化含有基因构建体的根癌土壤杆菌菌株用于通过浸渍法(bechtold等人,1993)转化拟南芥,特别是hc13。从处理过的植物中收集种子。将种子铺在含有适当选择剂的组织培养基上以选择稳定转化的植物,将其转移到土壤中以生长至成熟。收集来自显示为转基因的植物的种子并播种以产生子代植物。鉴定和选择转基因纯合的子代植物。通过标准qrt-pcr方法分析转基因的表达水平,如上述瞬时表达实验,用westernblot方法测定含有表位的融合多肽的累积水平。实施例15.nifd构建体对共表达转基因的影响如实施例11所述,出乎意料地观察到,当编码特异性nifd融合多肽(pra07,pra19)的构建体与其他nif表达的构建体共压渗时,所有nif表达的构建体的表达显著降低(图10)。特异性构建体pra19具有根据人密码子使用优化的核苷酸序列。因此,发明人试图理解简化表述的范围和原因,以试图避开它。首先,为了观察该作用是否特异于pra19和pra01的组合,将来自pra01(pfaγ::gfp)的t-dna单独或与pra19(pfaγ::nifd::ha,优化的人类密码子)或pra16(pfaγ::nifs::ha)组合,或将三个pra01、pra19和pra16组合在一起引入本生烟叶片细胞。如前所述,包括表达沉默抑制因子p19的构建体的根癌土壤杆菌包括在包括对照在内的所有压渗中。压渗后4天,将压渗的本生烟叶的背面暴露在紫外线(uv)光下并拍照以确定gfp表达的强度。与用pra01压渗的区域观察到的荧光相比,pra19+pra01的gfp荧光显著降低,荧光几乎不可见。相反,pra16+pra01的荧光是清晰可见的,尽管与单独的pra01相比观察到了荧光有所降低,如使用两个t-dna时从稀释物中可以预期的那样。接着,通过凝胶电泳和使用抗ha和抗gfp抗体的westernblot比较gfp和nifs融合多肽累积的水平。与观察到的gfp荧光水平一致,当pra19与pra01共压渗时,pfaγ::gfp累积的水平降低,而当pra16和pra01共同引入时,pfaγ::gfp的水平仅略微下降(图18)。当所有三种构建体共同引入时,pfaγ::nifs::ha累积的水平与不包括pra19时相比也显著降低。因此,由从pra19引入t-dna引起的减少不限于自pra01的pfaγ::gfp的减少。为了进一步检查这种下调的范围和模式,单独或与pra19或编码nifd融合多肽的其他构建体组合引入额外d的构建体,分析蛋白质和mrna转基因表达水平以及uv光下的荧光强度。如前所述,来自pra19的t-dna的引入导致来自共同引入的pra16的gfp荧光以及gfp和pfaγ::nifs::ha融合多肽的蛋白质水平降低。然而,当共引入pra24(pfaγ::nifd::ha,通过拟南芥密码子使用优化密码子)或pra20,编码nifd-nifk融合多肽(pfaγ::nifd-接头-nifk::ha)时,情况并非如此。值得注意的是,pra24和pra20编码与pra19相同的nifd氨基酸序列,但使用不同的核苷酸序列,因为它们是基于拟南芥密码子使用进行密码子优化的。基于这些观察,本发明人认为pra19的下调作用可能由从pra19的t-dna转录的特异性nifd编码rna序列触发。为了确定下调作用是否发生在蛋白质和/或rna水平,从相同的压渗区域提取rna并通过qrt-pcr分析。使用gfp特异性引物,发现pra19+pra01组合的gfpmrna水平相对于单独pra01的水平降低到约25%。当pra16和pra01组合时,与单独的pra01相比,减少小于50%,正如当组合两种构建体时所预期的。接下来,使用与编码pfaγ肽的rna部分退火的引物,其中引物将扩增来自同时表达的两种转基因的mrna区域,qrt-pcr测定确定pra19+pra01组合的转基因降低水平比pra16+pra01高约50%。综上所述,这些结果表明,将pra19引入本生烟叶片细胞具有降低共表达转基因的mrna和蛋白质水平的效果。这表现出非一般的mrna/蛋白质下调效应,因为对照基因(gadph)的表达水平在所有压渗实验中是恒定的,并且总蛋白质合成水平对于所有压渗表现为相似,如凝胶的考马斯亮蓝染色所证明的。总之,这些结果表明编码由pra19的t-dna产生的nifd多肽的mrna的内在特性导致在mrna和蛋白质水平上共同引入的转基因的下调。已知一种rna介导的基因沉默的公认模式受到长度为21-24个核苷酸的sirna的影响,这些sirna引导argonaute蛋白减少具有互补或近互补序列的多核苷酸的表达,这一过程被称为rna干扰(rnai)。然而,pra19的人密码子优化的编码区的rna序列与pra01(gfp)或pra16(nifs)编码的rna序列的比对没有发现足以提供argonaute介导的沉默所需的序列互补性的同源性片段。基于该发现,假设尽管特异性nifd的rna序列引发共引入转基因的沉默,但沉默效应物存在于pra19构建体中与gfp和其他转基因共有序列互补性的区域中的其他地方。为了验证这一假设,将pra22(mfaγ::nifd::ha)与pra01共同引入,pra22(mfaγ::nifd::ha)与pra19相同,但含有mmtp,因此在mtp区域的部分区域具有不同的rna序列。为了比较,还引入pra19+pra01和pra24+pra01组合。从荧光强度看来,pra22+pra01组合以与pra24+pra01组合大约相同的水平表达gfp。westernblot结果显示,pra24+pra01和pra22+pra01组合均显示gfp蛋白累积减少不大,而pra19+pra01则显示gfp蛋白明显减少。这些结果表明,将mtp序列修饰为与其他转基因的互补性降低的序列减轻了人密码子优化的nifd序列的下调。这表明pra19中的人密码子优化的nifd的rna与pfaγ序列组合触发转基因下调。或者,但不相互排斥,nifd核苷酸序列本身足以引发下调的起始,但nifd的rna上游的区域需要与转基因互补以引发它们的下调。为了证实人密码子优化的nifd对其他转基因的下调作用是nifd的rna而不是nifd蛋白的特性,设计了一种构建体,其具有与pra19几乎相同的rna序列,但是具有两个核苷酸改变,这两个核苷酸改变在mtp内产生提前终止密码子和移码。这称为pra26(δfaγ::nifd::ha)。如果下调的机制是rna介导的而不是蛋白介导的,那么预期pra26下调gfp和类似于pra19的其他nif构建体。作为进一步的测试,还共引入人密码子优化的nifd构建体pra07(pfaγ::nifd::flag)。pra07将在nifd区而不是表位区表达与pra19相同的rna序列,因为ha表位被flag取代。5天后,在压渗区中观察到荧光水平。与前面的实验一致,pra19+pra01组合的gfp荧光降低。pra26+pra01和pra7+pra01组合的荧光也显著降低。westernblot表明pra26与pra19相比在降低gfp表达方面具有相似的作用。结论是人密码子优化的nifd的mrna能够降低其他转基因的表达,即使当没有nifd蛋白产生或如果表位改变时。结合早期结果显示人类密码子优化的nifd的rna序列的改变阻止了转基因的下调,最终证明pra19中的nifd序列减少转基因表达是特异性nifd人类密码子优化的rna序列的结果。pra19中的pfaγ::nifd::ha融合多肽编码区的核苷酸序列如seqidno:68所示。实施例16.测试nif多肽功能的细菌表达实验如前面实施例中所述,制造和测试了用于在植物细胞中生产nif融合多肽的基因构建体,举例说明了利用根癌土壤杆菌引入合成基因的本生烟叶片细胞系统。融合多肽被设计成具有添加到nif多肽n端的线粒体靶向肽(mtp)和添加到c端延伸区的表位标签的框内融合。尽管蛋白折叠和结合的模型化(实施例10)预测大多数n端和c端延伸区不应阻止复合物形成和功能,但本发明人希望在体内生物系统中测试这些延伸区是否可能影响融合多肽相对于天然nif多肽的功能。设计用于在植物细胞中表达并易位到线粒体中的nif融合多肽及其加工的多肽产品在细菌固氮酶表达系统中进行了功能测试。本发明人建立了一种系统,其允许通过添加到固氮酶系统中,或迭代地或甚至与其他修饰的nif多肽组合来单独测定每种修饰的多肽。该系统在大肠杆菌中使用野生型固氮酶基因簇,该基因簇通过交换编码待测试的nif融合多肽的单个nif基因,而不是编码野生型nif多肽的相应基因来修饰。随后分别使用制乙烯试验和制氢试验测定固氮酶或固氮酶还原酶活性,以确定nif融合多肽是否可以代替相应的野生型nif多肽起作用。基于smanski等人(2014)的细菌固氮酶载体(vector)系统是这种情况的基础。在该系统中,固氮酶活性所需的所有野生型基因都包括在单一的、宽宿主范围表达载体(vector)pmit2.1中,其中基因的表达由来自第二质粒pn249的诱导型启动子/t7-rna聚合酶系统控制。当在大肠杆菌中表达时,产生全套野生型细菌nif多肽,并一起提供固氮酶复合物,其活性可通过标准乙炔还原检测(ara,固氮酶活性的事实测量)中由乙炔产生乙烯来测定,或通过固氮酶还原酶活性的产氢来测定。pmit2.1及其衍生物的使用方法如下。用分别促进对氯霉素和壮观霉素产生耐药性的两个质粒pmit2.1和pn249转化大肠杆菌菌株dh5α细胞。通过在含有氯霉素(34mg/l)和壮观霉素(80mg/l)的lb培养基(10g/l胰蛋白胨,5g/l酵母提取物,10g/lnacl)上生长来选择转化的细胞。转化的细胞在含有抗生素的lb培养基中37℃有氧生长过夜至光密度值为1.0。将培养物以10,000g离心1分钟,弃去上清液。将细胞重悬于1倍体积的不含n源,含有25g/lna2hpo4、3g/lkh2po4、0.25g/lmgso4.7h2o、1g/lnacl、0.1g/lcacl2.2h2o、2.9mg/lfecl3、0.25mg/lna2moo4.2h2o和20g/l补充有1.5ml/l的10%丝氨酸的蔗糖(基本培养基)的诱导培养基中。将原液过滤灭菌。为了诱导nif基因表达,除非另有说明,培养基中补充有异丙基-β-d-1-硫代半乳糖苷(iptg;goldbio#i2481c25259)的终浓度为0.1mm、0.5mm或1.0mm,通常为1.0mm。将细胞悬浮液转移到13cc培养瓶中并使用卷边锁定系统用气密橡胶密封件封盖,并用纯n2气体鼓泡顶部空间20分钟。然后将悬浮液在30℃下以200rpm振荡孵育5小时。此后,通过注入1.5cc纯c2h2(boc气体,仪器级)开始乙炔还原检测(ara),并进一步孵育3小时,然后至18小时。使用agilent6890ngc仪器,通过具有火焰离子化检测(flameionisationdetection,gc-fid)的气相色谱法测量两个时间点的乙烯产量。以10∶1分流模式将顶部空间样品(0.5cc)手动注射到分流/无分流入口中。该仪器在以下参数下操作:入口和fid温度为200℃,载体(carrier)he的平均速度为35cm/sec,等温烘箱温度为120℃。使用rt-aluminabond/mapd柱(30m×0.32mm×5μm),其中5m颗粒捕获柱偶联到检测器端。通过运行合适的空白和标准物评估仪器的分析性能。在这些条件下,乙烯在约2.3分钟时从柱中排出,乙炔在约3.1分钟时从柱中排出。该gc系统能够在低至0.00001%atm的水平下检测乙烯,其中乙炔作为该形式中唯一的其他可检测峰具有清晰的分辨率,因此非常灵敏。对于乙烯生产,在加入iptg后的不同时间点,通常在3小时和18小时,取0.5cc的顶部空间样品。使用大肠杆菌dh5α菌株中的野生型pmit2.1和pn249作为阳性对照的测定系统在生长培养基中不添加iptg时仅产生微量乙烯,而在生长培养基中加入0.1mm、0.5mm或1.0mm的iptg则大大增加了乙烯的生成量。从3小时取样到18小时,乙烯产生速率大大增加,并且随着iptg浓度增加,表明随着nif基因表达增加,固氮酶活性增加。这些结果表明iptg在该表达系统中快速诱导nif基因表达和固氮酶形成,与smanski等人(2014)的报告一致。如前面实施例中所述,pfaγmtp氨基酸序列在植物线粒体中被切割,留下38个氨基酸残基与加工的nif融合多肽的nif多肽的n端融合。为了测试这种n端延伸区是否会改变融合多肽的功能,并证明细菌测定系统起作用,使用pra10dna作为模板通过pcr合成117bp的dna片段(seqidno:70)。该dna片段编码38个氨基酸,其序列与pfaγmtp的c端部分相同,只是n端ile残基被met替换用于翻译起始,加上c端的额外thr残基。其设计使得,当直接在编码任一nif多肽的基因的起始密码子上游框内融合时,嵌合基因将编码到所选择的nif多肽的翻译融合多肽。39个氨基酸多肽的氨基酸序列,下文称为pfaγ-c,如seqidno:71所示。通过各种技术,包括平端连接、剪接重叠pcr和引导连接酶循环技术(dekok等人,2014),将pfaγ-c的dna片段直接插入pmit2.1中编码nifh、nifm、nifd、nifk、nife\nifn或nifb的基因的翻译起始密码子上游。例如,117bp的dna片段与pmit2.1上nifd基因的蛋白编码区框内融合,如下所述。使用引物扩增pmit2.1dna以产生线性dna产物,使得nifd基因的起始密码子(atg)位于5'端,并且翻译起始密码子的紧邻5'的nifd基因的核苷酸序列形成产物的3'端。为此,使用引物nifd-f5’-atgatgactaatgctactggcgaacgtaacctg-3'(seqidno:72)和nifd-r5’-ccggctcctccgctagataaaaatgtga-3'(seqidno:73)进行pcr。使用具有pmit2.1dna模板的phusion-hf校正聚合酶(neb,目录m05081l)进行扩增,pcr方案为96℃30秒,然后30个循环的92℃20秒、55℃20秒和72℃6分钟,然后72℃10分钟。反应产物用dpni处理以去除载体(vector)模板dna。pcr产物基本上是pmit2.1的线性化形式,开始于紧邻nifd翻译起始密码子的上游。使用t4dna连接酶将117bp片段连接到线性化pmit2.1,产生pcw1001。117bp片段的连接也通过使用illuminaampligase(illumina,a8101)的连接循环反应条件(lcr)和dekok等人描述的条件实现。(2014)。为了确定pmit2.1dna上扩增反应的保真度和准确性并证明野生型nif基因中没有发生核苷酸替换,连接线性化pmit2.1dna样品以再生环状分子并用于转化大肠杆菌dh5α。如上所述,对含有重新连接的pmit2.1的大约10个单独的菌落进行了从乙炔产生乙烯的检测,以证明失活突变未通过pcr扩增并入dna中。还可以对载体(vector)测序以确保构建的准确性和保真度。将载体(vector)pcw1001和pn249引入dh5α并用氯霉素和壮观霉素选择转化细胞。从这些细胞在诱导培养基中生长的培养物在ara分析中测试乙烯的产生,使用1mmiptg诱导基因表达,n2气体作为顶部空间,添加乙炔气体作为底物。乙烯的产生表明对于固氮酶活性可以允许39个氨基酸的n端延伸区到另外的野生型nifd多肽。使用类似的方法修饰pmit2.1中的其他nif基因,包括例如nifh、nifk、nifb、nife、nifm和nifn,在每种情况下使用合适的引物对在翻译起始密码子上游紧邻的位置线性化pmit2.1dna,并在该位置插入117bp片段。还进行连续几轮的nif基因修饰,以在单个载体(vector)中产生两个或更多个nif基因修饰的组合,以测试修饰的nif融合多肽的组合是否在其他野生型固氮酶复合物中保持功能。测试nif融合多肽的生物学功能的另一种检测系统是使用在特异性nif基因中突变的维涅兰德固氮菌的突变体的细菌互补试验。为了实现这一点,编码nif融合多肽的基因序列被单独插入到宽宿主范围的质粒中,例如可以在维涅兰德固氮菌菌株中繁殖的pmmb66eh(furste等人,1986)每个产生的基因构建体被引入到维涅兰德固氮菌突变株中,例如从内源性nifh基因的5’端缺少200p的菌株dj1271,用于测试nifh融合多肽功能。其他缺乏单个nif活性的突变菌株列于表6。这些突变使得突变体不能在合适的生长条件下固定大气中的氮。功能性nifh基因在dj1271中的表达例如恢复了固氮酶活性。因此,当与其他nif多肽缔合时,其是用于评估修饰的nifh融合多肽功能的有用测试系统。为了评估这些多肽的功能,可以在无氮培养基上评估转化体的生长。在生长培养基中没有添加氮源的情况下,维涅兰德固氮菌必须固定大气中的氮才能生长。如果修饰的nifh融合多肽是功能性的,那么这些多肽在dj1271中的过表达应该恢复在缺乏添加氮的固体培养基上生长的能力。或者,在上述方法中,可以通过乙炔还原检测(ara)测定固氮酶功能。ara测量固氮酶活性的灵敏度高于在无氮培养基上生长的灵敏度。这些表达系统测试nif融合多肽在细菌中的功能,其中固氮酶功能所需的所有其他nif组件在细菌中编码。表6.可用于测试nif融合多肽固氮酶功能(细菌互补)的维涅兰德固氮菌突变菌株。菌株id注释还原乙炔?dj11δnifhdk,kpni缺失否dj13δnifk框内psti缺失(aa136–>>aa293)否dj33δnifdkkpni缺失否dj97nifz:kn否dj99nifu:kn否dj100δnifd框内kpni缺失(aa103–>>aa376)否dj112δnifzm否dj349nifm::knnif-dj525δnifh,去除nifh的5’编码区否dj771δnifu::kn否dj1025nifh::kanr+δh–d茎环否dj1151δnifh::kanr.否dj1271nifh:kanr否dj1469δnifus,pscr-lacz:kn否dj262δnifus否dj536δnifusvwzm否wt野生型nif+实施例17.mtp序列的修饰及其与nif多肽的用途如实施例5至9所描述和使用的长度为77个氨基酸残基的pfaγmtp序列(seqidno:38的氨基酸1-77)来自拟南芥f1-atpase-γ-亚基(lee等人,2012),其片段在此命名为mtp-faγ77。在实施例5中用于制造编码mtp-faγ77和远端nif多肽之间的翻译融合多肽的基因序列的克隆方案在融合连接处使用插入的9核苷酸接头,该接头由asci克隆位点和额外的碱基组成以维持阅读框。使用该接头在mtp-faγ77的c端和目的多肽(如gfp或nif)的n端之间产生另外三个氨基酸,即gly-ala-pro(gap)。因此,在线粒体中切割之前,编码的多肽包括80个氨基酸的n端延伸区,其显示有效地将融合多肽转移到线粒体中并在预测位点被线粒体基质肽酶(mmp)切割。如实施例5-9描述,推断mtp-faγ77能够将16种不同的nif融合多肽导向植物线粒体基质,并且mtp-faγ77序列在线粒体内被mmp加工。切割发生在42个氨基酸之后,留下与目的gfp或nif多肽融合的38个氨基酸残基的n端延伸区,35个残基来自mtp-faγ77加上gap。该n端延伸区在此称为faγ-scar38。本发明人试图从mtp-faγ77的77个氨基酸缩短mtp序列,用于在植物细胞中与nif多肽一起使用,同时仍然保持mtp功能。发明人检查了是否可以从mtp-faγ77的c端修剪26个氨基酸以产生命名为mtp-faγ51(seqidno:75)的mtp。发明人预测mtp-faγ51将在氨基酸42之后被mpp切割,在加工的融合多肽的n端留下来自mtp-faγ51的9个氨基酸(istqvvrnr;seqidno:76)。该9氨基酸序列命名为faγ-scar9。为了测试mtp-faγ51的功能,制备编码与nifh或gfp融合的mtp的基因构建体。这些构建体包括修饰的nifh或gfp基因,其与pra10中的nifh基因(seqidno:42)或pra01中的gfp基因(seqidno:38)相同,除了mtp-faγ51融合到每个多肽的n端而不是mtp-faγ77加上gap。所得构建体命名为pra34(mtp-faγ51::nifh)和pra35(mtp-faγ51::gfp)。以pra01为例,通过连接循环反应(dekok等人2014)进行截短。使用引物5’-atggtgagcaagggcgaggag-3'(seqidno.xxx)和5’-gcggttacgcaccacttgagttg-3'(seqidno.xxx)以及pra01作为模板,扩增出没有编码mtp-faγ77的c端29个氨基酸和编码氨基酸gap的额外9个核苷酸的pra01。用桥接寡聚物5'-ctcctcgcccttgctcaccatgcggttacgcaccacttgagttg-3'和illuminaampligase(illumina,a8101)连接所得pcr产物,条件如dekok等人所述。(2014)。这些构建体在本生烟叶片系统中进行了测试,并与具有mtp-faγ77加上gap的相应构建体进行了比较。对于pra34和pra10,从压渗叶片组织产生蛋白提取物。使用ha-抗体对提取物进行sdspage和westernblot分析以评估蛋白表达水平和mpp加工效率用于比较。来自大肠杆菌的蛋白提取物表达编码mtp-faγ51::nifh::ha(pra34)和mtp-faγ77+gap::nifh::ha(pra10)的蛋白质提取物,用作未加工融合多肽的对照,产生未加工mtp::nifh的预期大小的多肽条带。与相应的细菌提取物相比,被这些构建物压渗的本生烟叶片组织的蛋白质提取物产生的多肽条带尺寸较小,与预期的加工多肽的尺寸相一致。例如,根据这两个多肽之间预期大小的差异,mtp-faγ51::nifh::ha多肽的表达比mtp-faγ77+gap::nifh::ha在更小的mw处产生一条带。因此推断mtp-faγ51氨基酸序列能够将nif多肽靶向植物细胞中的线粒体基质,并提供给mpp加工。多肽表达水平和加工效率与较长mtp一样好。此外,较小尺寸的多肽条带被认为是降解产物,在pra34的印迹中检测到ha抗体。发明人得出结论,较短的mtp序列出乎意料地降低了n端mtp::nif降解。也用编码融合gfp多肽的pra35进行实验,将其与pra01进行比较。为了通过荧光观察多肽的定位,共聚焦显微镜检查了经pra01(pfaγ77+gap::gfp)或pra35(faγ51::gfp)编码构建体压渗的叶片组织。使用具有40×水浸物镜的立式leica显微镜对叶片样品成像。gfp在488nm激发并记录荧光发射。对于两种构建体,观察到gfp荧光定位于具有相同定位模式的小亚细胞体中。基于与如实施例1所述测试线粒体定位的pra01的比较和pra34的加工数据,本发明人得出结论,缩短的mtp-faγ51能够将合成的融合多肽导向植物细胞的线粒体,并且能够通过线粒体中的mpp提供给它们的加工。测试了一系列不同的mtp序列以评估它们在将nif多肽转移到植物细胞的线粒体基质中的性能。goldengate克隆系统(weber等人,2011)用于组装不同的基因元件,包括启动子、5'-utr、3'-utr、n端和c端延伸区和终止子。每个元件具有限定的边界,允许模块化组装和元件的容易更换。因此,将具有engler等人,(2014)描述组件的克隆系统用于测试大量不同的基因构建体,用于生产mtp::nif融合多肽。由于goldengate克隆系统利用了在其识别序列之外切割的iis型限制性酶,因此可以避免使用连接序列内的限制性酶克隆位点。这允许在pra00中没有gly-ala-pro序列的情况下构建编码mtp::nif融合多肽的基因,并且避免了如实施例5-9中所描述的asci限制性位点的使用。取而代之的是mtp::多肽融合结合处的gly-gly桥,以适应goldengate系统。由于甘氨酸通常出现在mtp序列的-1位,因此选择甘氨酸作为该接头的标准氨基酸。选择一系列不同长度(30-70个氨基酸残基)的不同mtp,预测在mpp切割后留下融合在nif多肽n端的不同剩余氨基酸残基(“scar”)(表7)。scar序列的长度为0-36个氨基酸残基。使用goldengate克隆系统,使用这些mtp与几个nif(表8)的组合组装17种不同的基因构建体,用于在植物细胞中表达。用于这些融合的nifd和nifs多肽序列是用于实施例5-9的序列的变异体,而不是使用根据temme等人,(2012)的序列。变异体氨基酸序列分别在seqidno:95和seqidno:96中提供。变异体nifd氨基酸序列(seqidno:95)在39、41、87、96、355和483位有6个氨基酸替换不同于seqidno:6所示的483个氨基酸的序列。对于所有这些载体(vector),启动子、5'和3'utr和终止子是相同的。含有这些构建体的农杆菌培养物,每个与产生p19沉默抑制蛋白的构建体混合,分别引入本生烟叶片和压渗后5天产生的蛋白提取物。对蛋白提取物进行sds-page和westernblot分析。对于具有mtp::nifd构建体的压渗,pra25(pfaγ::nifk)被共压渗,因为无c端延伸区的nifk的共同表达已经被证明可以增强nifd的丰度(实施例11)。表8总结了检测植物细胞中产生的多肽并通过mpp(加工)对每个mtp::nif融合多肽进行切割的结果。表7.使用goldengate系统在植物中进行测试时使用的mtp的详细信息,连接处的gly-gly残基以粗体突出显示(cpn60/no接头除外)。kdaf/p:已加工、未加工mtp的大小。表8.将构建体(由sn编号表示)引入叶片细胞后产生的多肽的westernblot分析,以表达nifh、nifd、nifm、nifs和nifu。pro:加工的。(p)=部分-少于50%的westernblot检测到的加工形式。ud:无法根据条带大小区分加工。当翻译融合为mtp::nif融合多肽时,mtp-faγ51为所有测试的nif多肽产生了切割的mtp::nif多肽(表7)。当与faγ51融合时,nifm融合多肽只被部分加工,而其他nif融合多肽的加工效率更高,表明对于一个mtp,不同nif的加工效率可能不同。测试了faγ51的一个版本,其ha表位在mtp的c端融合,然后融合到nifh(sn29)。通过westernblot分析检测由该构建体表达的多肽(表8)。测试了融合到nifd的cpn60mtp的两个版本。在一种版本中,融合mtp,使得gly-gly接头置于cpn60mtp(seqid:77)和nifd(sn11)之间。在另一个版本(sn4)中,cpn60mtp(seqid:78)直接与nifd多肽的第一个甲硫氨酸融合。由于预测cpn60在其氨基酸序列中的c端酪氨酸之后立即被切割,该结构理论上将产生具有野生型n端的nifd多肽,即没有“scar”,而sn11构建体被预测在mtp(glygly)::nifd融合被切割后留下gly-gly延伸。令人惊奇的是,这些非常相似的构建体产生不同的结果,如westernblot分析所证明的:sn11产生未加工的cpn60(glygly)::nifd预期大小的多肽条带,而sn4产生对应于加工和未加工多肽的条带,其中存在比加工多肽更多的未加工多肽。此外,当通过westernblot将来自sn4压渗的蛋白质与从平行的pra24+pra25(faγ77+gap::nifd::ha+faγ77+gap::nifk)压渗中提取的蛋白质进行比较时(也见实施例11),显然,sn4构建体产生的正确加工的多肽比pra24构建体少得多。因此,可见尽管cpn60mtp能够靶向融合多肽并允许基质作用以产生野生型nifd多肽,但表达水平和加工效率都低(us2016/0304842)。对于sn11,cpn60和nifd之间的gly-gly接头可能阻止mtp的加工。还测试了来自超氧化物歧化酶多肽的各种mtp,无论是作为单mtp还是串联mtp,以及在gly-gly接头之前在c末端包括或不包括ile和gln的情况下。对于含有不含ile和gln残基的sodmtp的版本(sn15,seqidno:81和sn16,seqidno:82),westernblot分析未检测到多肽,而保留ile和gln残基的版本(sn12,seqidno:79和sn13,seqidno:80)确实产生了可检测的多肽,尽管看起来他们没有被mpp加工过。相反,测试的另一mtp,l29(sn17,seqidno:83)当与nifd融合时产生强多肽信号。由于使用此mtp加工的形式和未加工的形式之间的大小差异很小,将需要额外的实验来确定加工效率。预期l29mtp以有效的方式产生切割的nif多肽。最后,发明人用twinstrep标签测试了coxivmtp(burénetal.2017),twinstrep标签融合在c端但位于gly-gly接头上游(sn19,seqidno:86)。当与nifd融合时,该mtp通过westernblot分析给出强信号,其大小与线粒体基质作用一致。细菌系统中与nif多肽融合的不同scar序列的功能检测在mit2.1系统中评估了掺入各种mtp和切割后保留在nif融合多肽n端的序列对nif多肽功能的影响。例如,通过引导连接酶循环技术,编码pfaγ-scar9的dna片段以met代替ile作为第一个氨基酸被插入到pmit2.1中nif基因的翻译起始密码子的上游(dekok等人,2014)。用于制备构建体的方法如实施例16中nifb所述。将编码pfaγ-scar9的27bpdna片段融合到pmit2.1中各种nif基因的5'端。利用乙炔还原检测进行大肠杆菌转化和固氮酶功能测试,如实施例16所示。这些测定显示,与用未修饰的pmit2.1观察到的乙烯产生水平相比,向nifb的n端添加9个氨基酸延伸区不改变固氮酶功能。以类似的方式,nifh、nifd、nifk、nife和nifn也在其各自的n端允许9个氨基酸的延伸,对nifh具有完全活性,但在其他情况下活性有所降低。nifd、nifk、nife和nifn的n端的9个氨基酸延伸区产生的乙炔还原活性水平分别为未修饰的pmit2.1的50%,70%,30%和50%。其他nif多肽,即nifj、nify、nifq、niff、nifu、nifs、nifv、nifw、nifz和nifm,以相同的方式进行测试。使用这些测定法,为每个nif多肽选择最佳mtp序列。实施例18.添加到nifk的c端破坏其活性如实施例10所述,发明人通过对来自肺炎克雷伯菌的nifk多肽的结构建模得出结论,nifk亚基的c端埋藏在nifd-nifk-nifh蛋白复合物的核心中,并且相对于野生型序列对nifk多肽的任何c端延伸区都可能导致复合物的破坏。通过pmit2.1质粒的修饰,使用实施例16中描述的细菌固氮酶系统进行功能性测试。如下通过添加氨基酸序列ypydvpdya(seqidno:97),修饰质粒以表达相对于野生型序列具有9个氨基酸c端延伸区的修饰的nifk多肽。为了引入nifk修饰,将编码氨基酸ypydvpdya(seqidno:97)的核苷酸序列(密码子优化为在大肠杆菌中表达)添加到nifk的3'端的反向引物的5'端。该引物,5'-tcatcaagcgtaatcaggaacatcgtaggggtaacgaaccagatcgaaagaatagtcgg-3'(seqidno:98)和正向引物5'-aagggcgaattccagcacactgg-3'(seqidno:99)用于pcr,以ptopoh-j(实施例16)dna为模板,含有pmit2.1的前半部分,得到7909bp产物。使用引物5'-cctgattgtatccgcatctgatgctac-3'(seqidno:100)和5'-aacctgcagggctaactaactaaccacggacaaaaaacc-3'(seqidno:101),也用pcr扩增剩余的7222bp。以与实施例16中描述相似的方式,然后使用连接循环反应(lcr)将两个pcr片段与引物bo1,5'-ggtagcatcagatgcggatacaatcaggtcatcaagcgtaatcaggaacatcgagg-3'(seqidno:102)和bo2,5'-ccagtgtgctggaattcgcccttaacctgcagggctaactaactaaccacg-3'(seqidno:103)使用dekok等人(2014)的方法连接在一起。连接的dna片段用sbfi消化并连接到来自pb-ori的含有未修饰的nifbqfusvwzm基因的sbfi片段,产生编码具有添加到nifk的c端延伸区的融合多肽的修饰的mit2.1载体(vector)。将得到的基因构建体引入含有pn249的大肠杆菌菌株dh5α中,并如实施例16所述,培育用两种载体(vector)转化的细胞的培养物。在乙炔还原检测中测试培养物的乙烯产量。结果表明,添加到nifk中的9氨基酸c端延伸区完全消除了乙烯的产生。未修饰的对照pmit2.1产生阳性乙烯产量。发明人得出结论,nifk的c端相对于野生型c端的翻译融合延伸消除了如通过乙炔还原测量的固氮酶活性,与实施例10中描述的建模研究一致。考虑到上述生物化学测试,证明延伸到nifk的c端消除了固氮酶功能,并且以前的植物原位(inplanta)实验显示nifd和nifk与野生型c端的共表达提高了nifd的丰度(实施例11),发明人推断nifk多肽中的野生型c端对于nifd-nifk异源四聚体的稳定性和功能以及因此的固氮酶活性都是关键的。通过表达nifd和具有野生型c端(pra25)的nifk的不同版本的基因进一步研究了这一点,并评估了nifd丰度。实施例20描述了一系列各自包括nifd基因的基因构建体,其用于评估本生烟中nifd的丰度。本实验中的nifd包括c端ha表位。在这些构建体(表8)共压渗后,在表达沉默抑制因子p19的基因构建体存在的情况下,如用于所有本生烟压渗,以及有或没有共压渗的pra25(nifk),通过sdspage和使用抗ha抗体的westernblot分析评估nifd::ha的蛋白质丰度和mpp加工。分析(图19)显示在每种情况下加入pra25都大大提高了nifd丰度,特别是对于sn7和pra25的组合。最重要的是,通过加入pra25提高了加工的nifd与未加工的nifd的比例。这些结果进一步支持了以下结论:nifk对nifd的稳定发生在线粒体基质中,并且关键是依赖于具有野生型c端的nifk多肽来实现这一点。进一步值得注意的是,对于所有测试的构建体,观察到大约48kda的多肽条带并且认为是nifd的降解产物。由于这种多肽是用抗ha抗体检测的,并且在凝胶上观察到清晰限定的多肽条带,因此它必须代表包括nifd的c端的特异性切割产物。出乎意料地,pra25(nifk)的共表达也提高了该产物的量。这表明了nifd和nifk多肽之间的相互作用。基于这些观察,本发明人认为nifd降解是线粒体基质特异性的并且为nifd和nifk在mm中的相互作用提供支持。关于当将nifd::ha编码构建体引入植物细胞时产生的大约48kda的多肽被测试的一种可能性是,48kda多肽是从第二次下游翻译起始产生的。nifd基因在可能导致48kda翻译产物的区域中具有对应于met残基的多种atg密码子。为了测试这种可能性,制备了与pra24相同的基因构建体(pra30),除了nifd起始密码子下游的第二至第五met密码子被ala密码子替代。分别将pra30和pra24压渗到本生烟叶片中并进行sds-page和westernblot分析后,pra24和pra30的带型相同,仍存在约48kda的多肽产物。这一观察推翻了当nifd::ha基因表达时看到的48kda大小的条带是由于交替翻译起始的假设。结论是该多肽是通过mm中未知的降解过程产生的。实施例19.评估nifd-nifk和nife-nifn融合多肽实施例12描述了编码nifd-nifk融合多肽的基因的设计和构建,使用由nifd的c端和nifk的n端之间的30个氨基酸残基组成的接头。接头包括用于检测多肽的flag表位。现在设计并构建了第二基因,其具有与nifd-(flag)接头-nifk相同的排列,除了flag表位被ha表位替代。该第二个基因的接头编码的氨基酸序列(也有30个氨基酸残基)为atpppgstttaypydvpdyatpppgsttta(seqidno:104),接头中的ha表位为氨基酸12-20。在实施例16中描述的基于pmit2.1载体(vector)的细菌系统中,通过乙炔还原检测测试这两种融合多肽的功能性,如下所述。首先,用引物pra31dk-fw5'-atgatgactaacgctacaggagaa-3'(seqidno:105),和pra31dk-rv5'-tcatcatctaaccaaatcaaaactgtaatctg-3'(seqidno:106)扩增来自pra31的nifd::ha接头::nifk基因。该基因中的核苷酸序列被密码子优化以在拟南芥中表达。使用引物d_起始_rv5'-ccggctcctccgctagataaaaatgtgaatttttgcatgcagcc-3'(seqidno:107),和k_end_fw5'-cctgattgtatccgcatctgatgctaccgtggttga-3'(seqidno:108),也通过pcr产生了不含nifd和nifk基因的pmit2.1的前半部分。扩增的pcr产物通过连接循环反应使用桥接寡核苷酸5'-ttctcctgtagcgttagtcatcatccggctcctccgcta-3'(seqidno:109)和5'-cagatgcggatacaatcaggtcatcatctaaccaaatcaaaactg-3'(seqidno:110)连接,并用sbfi消化。将消化的dna片段连接到也已经用sbfi消化的pb-oridna上,产生pmit2.1的修饰形式,包括nifd::ha接头::nifk融合基因。在大肠杆菌(dh5α)细胞中的功能测试表明,含有nifd::ha接头::nifk基因的修饰pmit2.1载体(vector)可以将乙炔还原为乙烯,约为未修饰pmit2.1水平的10%。也就是说,即使在细菌系统中活性降低10倍,在其他野生型nifd和nifk多肽之间具有30个氨基酸接头的融合多肽也具有明显的功能。为了增加融合多肽的固氮酶活性,进行了两次改变并且基本上重复实验。这些改变首先是使用nifd的变异体氨基酸序列(temme等人,2012),参见实施例17,其次,使用对肺炎克雷伯菌而不是拟南芥进行密码子优化的核苷酸序列。构建体以与上述相同的方式产生。也就是说,编码单独的nifd和nifk多肽的未修饰的pmit2.1中的nifd和nifk之间的操纵子结构被编码nifd-接头-nifk的翻译融合多肽的核苷酸序列替换,从而对于每个含有flag和ha的接头,在修饰的pmit2.1中形成nifd::接头::nifk基因。在大肠杆菌(dh5α)细胞中的功能测试表明,pmit2.1中修饰的nifd::flag接头::nifk和nifd::ha接头::nifk基因分别减少乙炔产生乙烯的量约为未修饰pmit2.1的量的100%和80%。结论是第一个nifd-接头-nifk融合多肽的活性降低是由于不太理想的nifd氨基酸序列,或植物优化的密码子使用,或两者。这证明了根据宿主细胞使用合适的多肽序列和密码子优化的重要性。为了在植物细胞中表达并导入线粒体基质,将需要mtp序列,并且预期在mtp内切割后保留n端延伸区。例如,使用mtp-faγ51(实施例17),然后用mpp切割,将留下与nif多肽融合的9-氨基酸n端延伸区。因此,测试了向nifd-接头-nifk多肽添加这种9个氨基酸延伸区的效果,除了用met残基替换9(ile)的第一个残基以提供有效翻译。使用如上所述的克隆方法,将9-氨基酸序列添加到nifd::接头::nifk融合多肽的nifd部分的n端。然后在pmit2.1系统中测试这两种改性组合的乙炔还原能力。为了产生构建体,对于接头中的每个flag和ha表位,将编码9个氨基酸延伸区(mstqvvrnr,seqidno:111)的27个核苷酸的dna序列添加到nifd::接头::nifk融合基因的5'端。在含有构建体的大肠杆菌上的乙炔还原检测表明,向nifd::ha接头::nifk多肽添加9氨基酸n端延伸区产生的乙烯产量与nifd::ha接头::nifk在n端缺少9氨基酸延伸区的水平大致相同,即n端延伸区耐受性良好且不降低固氮酶活性。这与先前的发现一致,即可以将9氨基酸延伸区添加到野生型nifd中而不降低其活性。本发明人还通过从未修饰的mit2.1去除nifd基因的终止密码子、间隔序列和nifk编码区的核糖体结合位点,测试了在mit2.1系统中没有任何接头序列的nifd和nifk之间翻译融合的功能。与阳性对照相比,这种编码nifd-nifk多肽(本文称为nifd-无接头-nifk)的阅读框的直接融合将乙炔还原活性降低至5%。nifd-无接头-nifk功能的这种降低显示了包括约30个氨基酸残基的柔性接头的益处,所述柔性接头允许融合多肽的nifd和nifk部分之间的正确构象缔合以形成功能性固二氮酶结构。nife-nifn融合类似于nifd和nifk,已知nife和nifn形成异源四聚体。该复合物在固氮酶活性方面的功能是接受来自nifb的femo辅因子核心、来自nifq的钼以及来自nifv的用于femo辅助因子成熟的高柠檬酸(hu和ribbe,2011)。发明人认识到nife和nifn应该以等摩尔水平表达以获得最佳的固氮酶功能,并且融合两种多肽将减少固氮酶重组表达所需的启动子、mtp和终止子的数量。因此,根据nifd和nifk融合(上述)的原理,设计并制备了编码nife和nifn之间的融合多肽的基因,除了使用46个氨基酸残基的接头代替nifd-接头-nifk融合多肽的30个氨基酸残基。其设计、构造和测试如下。以维涅兰德固氮菌(pdbid:3pdi,(kaiser等人,2011))的nife-nifn复合物的晶体结构为模板,构建了肺炎克雷伯菌nife-nifnα2β2异源四聚体的同源模型。将肺炎克雷伯菌与相应的维涅兰德固氮菌多肽进行比对,nife多肽的氨基酸序列同源性为68.1%,nifn多肽的氨基酸序列同源性为44.2%。发明人意识到,在肺炎克雷伯菌α2β2异源四聚体(nife2nifn2)的结构模型中,每个nife亚基的c端距其nifn配偶体的n端约因此,设计合适长度的接头以连接具有46个氨基酸残基的两个单元。发明人认为接头应围绕异源四聚体的表面弯曲并具有约的长度,因此选择了46个氨基酸残基的数目。本发明人还认为接头应最佳地具有若干其他特性,包括柔性。所选择的46个残基接头的氨基酸序列基于来自绳状篮状菌(talaromycesfuniculosus,uniprotkb:q8wzj4)的外切纤维素酶的碳水化合物/糖类结合结构域和催化结构域之间的天然连接体。选择它是基于几个标准,包括缺少半胱氨酸残基以避免形成不需要的二硫键,很少或没有带电荷的残基(glu、asp、arg、lys)以降低不需要的表面盐桥相互作用的可能性,很少或没有疏水残基(phe、trp、tyr、met、val、ile、leu),因为这些残基可以增加穿透多肽表面的倾向,并且接头序列缺少翻译后修饰的氨基酸-参见uniprotentrywww.uniprot.org/uniprot/q8wzj4,其中接头是残基457-493。外切纤维素酶序列中第2位的phe残基用ser替换以降低疏水性。ha表位序列ypydvpdya(seqidno:112)插入接头的氨基酸16和17之间,仅仅是为了帮助检测和定量融合多肽,接头不需要这样的表位来发挥作用。ha表位位于比接头中心更靠近n端的位置,以便将其与保留的单个带正电荷的赖氨酸残基隔开。所得接头序列是tssggtstggsttttaypydvpdyasgttstkasttstsststgtg(seqidno:113),含有作为氨基酸17-25的ha表位和在位置32保留的赖氨酸。为了产生基因构建体并测试nife-接头-nifn融合多肽的功能,pmit2.1中nife和nifn顺反子之间的区域用编码包括46个残基接头的翻译融合多肽的核苷酸序列替换,使用与先前构建体在修饰pmit2.1中相同的克隆方法。对拟南芥的核苷酸序列进行密码子优化,即使融合基因将在大肠杆菌中进行检测。利用引物praen-fw5'-atgaagggaaatgagattcttgctctt-3'(seqidno:114)和praen-rv5'-tcatcaatcagcagcataagcacc-3'(seqidno:115)从pra01-en中以pcr扩增出nife::ha接头::nifn基因。使用引物e_起始_rv5'-ttgtaataacctccagtgatgaattgaata-3'(seqidno:116)和n_终止_fw5'-ctagagattaatatggagaaattaagcatg-3'(seqidno:117),也通过pcr产生了pmit2.1的前半部分,没有nife和nifn。用sbfi消化两个dna后,将未修饰的pmit2.1(pb-ori)的后半部分与修饰后的前半部分连接,使用桥接寡核苷酸5'-aagagcaagaatctcatttcccttcatttgtaataacctccagtgatgaattgatagtg-3'(seqidno:118)和5'-tagttttcatgcttaatttctccatattaatctctagtcatcaatcagcagcataagcacc-3'(seqidno:119)用连接循环反应连接扩增pcr产物。所得构建体命名为atnife::接头::nifn/mit2.1。通过乙炔还原检测(acetylenereductionassays)对大肠杆菌细胞中含有atnife::接头::nifn基因的修饰的mit2.1载体(vector)的功能测试表明,相对于未修饰的mit2.1,修饰的mit2.1载体的乙烯产量约为40%。为了在植物细胞中表达并导入线粒体基质,需要mtp序列,并且在mtp序列内经mpp切割后保留n端延伸区。例如,使用mpp切割mtp-faγ77将在融合多肽上留下38个氨基酸的n端延伸区。因此,通过将编码它的核苷酸序列添加到atnife::接头::nifn基因中,测试添加这样的38个氨基酸的延伸,用met残基替换第一ile残基以提供有效翻译的效果。该基因的翻译将因此将38个氨基酸的延伸添加到nife::接头::nifn融合多肽的nife的n端。使用与前述相同的克隆方法,在pmit2.1系统中测试这两种修饰的组合对固氮酶活性的影响。经修饰的pmit2.1载体(vector)包括大肠杆菌中的38aa::atnife::接头::nifn基因,其乙烯产量约为未修饰pmit2.1水平的25%。也就是说,相对于未修饰的pmit2.1,在n端使用38个氨基酸的延伸产生了阳性、但降低了的固氮酶活性。发明人认为,这种降低可能至少部分是由于拟南芥密码子的使用对于大肠杆菌中的有效翻译来说是是次佳的,这可以通过创建具有细菌密码子优化的天然克雷伯氏菌属nife和nifn的翻译融合多肽来容易地进行测试。发明人考虑了可以优化nife-接头-nifn融合多肽功能的其他方式,如下所述。nife的n端的38个氨基酸延伸可能干扰从nife2nifn2异源四聚体释放有效的femo辅因子,因为异源四聚体的nife亚基的n端非常接近femo辅因子所在的腔(kaiser等人,2011)。nife2nifn2异源四聚体活性可通过使用在mpp加工时完全除去的mtp,或通过截短nife的n端编码区以适应当使用未完全除去的mtp序列时的额外延伸而增加。还认为46个氨基酸接头可能环出以部分阻断femo辅因子腔的进入,从而干扰从nifb接收femo辅因子核心或释放成熟的femo辅因子。在这种情况下,接头可以缩短,或者nife的c端可以截短,或者两者都进行,以避免接头区域阻塞空腔。使用pmit2.1系统可以容易地测试根据这些策略的变异体并进行优化。总结nifd-接头-nifk和nif-接头-nifn融合多肽都能够支持pmit2.1细菌系统中乙炔的还原,即具有提供固氮酶活性的功能。向nifd-接头-nifk添加9个氨基酸的n端延伸区提供了完全的固氮酶活性。向nife-接头-nifn融合多肽添加更长的38个氨基酸的n端延伸区也显示出功能,尽管相对于没有n端延伸区的多肽活性降低。发明人得出结论,在植物细胞中使用n端mtp序列与其他所需nif多肽组合表达这些融合多肽的策略将允许表达固氮酶活性。本文举例说明的nifd-nifk和nife-nifn融合策略的另外优点包括产生等摩尔水平的亚基以获得最佳固氮酶功能,并且融合两种多肽或两对多肽将减少植物细胞中固氮酶重组表达所需的启动子、mtp和终止子的数目。实施例20.检测植物细胞中nif融合多肽表达的不同启动子评估了一系列不同的启动子在植物细胞中表达nif基因以产生nif多肽的有效性,例如在本生烟叶片细胞中。为此目的选择了五个不同的启动子,即来自地三叶草矮化病毒的s4启动子(scsv-s4)的两个版本(schünmann等人,2003a;schünmann等人,2003b),scsv-s7启动子(schünmann等人,2003a)和camv35s启动子的两个版本,即35s启动子的较长形式和包括用于最大化基因表达的复制增强子(2×35s)的增强形式(表9)。scsv启动子在大多数植物组织中强烈表达并被认为是组成型启动子。在这些测试中用作nif多肽实例的nifd多肽是实施例17中使用的版本(seqidno:95)。在本实验中选择nifd作为nif多肽的实例,因为在所有测试的nif多肽(实施例5-9)中,其最难以表达。5种载体(vector)分别使用不同的启动子,但其他方面相同,包括编码faγ51mtp的序列(表9),用于在植物线粒体中转移和加工。使用goldengate克隆方法(weber等人,2011,engler等人,2014)产生5种构建体,允许模块化基因组装方法。含有这些载体(vector)的农杆菌细胞,每个与含有产生p19沉默抑制蛋白的构建体的农杆菌细胞混合,分别引入本生烟叶片和压渗后5天产生的蛋白提取物。相应地,与上述构建体进行配对压渗,加或不加pra25(pfaγ77+gap::nifk),因为已证明不含c端延伸区的nifk共表达可增强nifd丰度(实施例11)。使用抗ha抗体对蛋白提取物进行sds-page和westernblot分析(图19-见实施例18)。表9总结了检测植物细胞中产生的多肽并通过mpp(加工)对每个faγ51::nifd融合多肽进行切割的结果。当引入植物细胞时,所有5种构建体都产生用抗体检测的多肽(图19)。在每种情况下,观察到的多肽条带的大小与faγ51::nifd的加工形式和未加工形式一致,尽管多肽丰度和加工多肽∶未加工多肽的比率(即加工效率)存在相当大的变化(表9)。最重要的是,对于每个构建体sn6、sn7和sn8,pra25的pfaγ77+gap::nifk的内含物既提高了nifd的绝对水平,也意外地提高了nifd蛋白的加工形式和未加工形式的比率。这一点对于构建体sn7最为明显,其中无论有没有pfaγ77+gap::nifk,未加工的nifd的水平都是相同的,但是由于nifk多肽的存在,加工的nifd的水平大大增加。这些结果表明,令人惊讶的是,尽管每个构建体都使用相同的mtp,但不同的启动子不仅会对蛋白质表达水平产生实质性影响,而且还会对线粒体基质靶向nif蛋白的加工效率产生实质性影响。这些结果也证实了在没有c端延伸区的情况下nifk表达对nifd丰度的增强作用。表9.测试不同的启动子以评估mtp::nifd::ha在本生烟中的表达,以及融合多肽的加工效率。启动子seqidnosn#加工效率scsv-s4版本11206部分,<50%切割scsv-s4版本21217>50%scsv-s71228>50%camv-35s长版本1239部分,<50%切割camv-2×25s12410部分,<50%切割实施例21.测试nifs和nifu的固氮酶功能冗余性对于固氮酶的功能,存在两种nif蛋白,它们将铁硫片段提供给不同的固氮酶组件,用于它们各自的铁硫(fe-s)簇生物合成。nifs是吡哆醛p依赖的半胱氨酸脱硫酶,它激活硫以提供硫化物(zheng等人,1993),而nifu是从nifs接收硫化物并组装fe-s簇的分子支架(agar等人,2000)。它们一起向nifb提供fe-s簇用于femo辅因子核心(l-簇)组装(hu和ribbe2011),以及在肽基脯氨酰顺/反式异构酶nifm的协助下为[4fe-4s]提供fe蛋白(nifh)(gavini等人,2006),并在nifz的帮助下连续形成p簇([fe8-s7])(lee等人,2009)。在真核生物中,存在nifs和nifu的直系同源物,其为含有fe-s簇的各种酶提供fe-s片段(couturier等人,2013)。特别地,植物线粒体分别具有nfsi(at5g65720)和isui(at4g22220)多肽作为nifs和nifu的直系同源物,而在质体中nfs2(at1g08490)和sufb(at4g04770)分别是nifs和nifu的功能等同物。线粒体和质体系统都由另外的辅助蛋白辅助,以完成fe-s簇组装并在它们的天然细胞器环境中转移至它们的目标酶。最近在酵母线粒体和烟草质体中已经证明nifs和nifu对于重组fe蛋白活性不是必需的(lopez-torrrejon等人,2016;ivleva等人,2016)。为了在大肠杆菌pmit2.1系统中测试nifs和nifu的植物直系同源物的功能,将克雷伯氏菌属nifs和nifu基因替换为植物nfsi和isui基因,或者单独替换,或者组合两个替换。通过乙炔还原检测评估修饰的pmit2.1的固氮酶功能。另外,将拟南芥nfsi突变体(salk_083681)和isui突变体(salk_006332)分别以含有克雷伯氏菌属nifs和nifu基因的基因构建体转化,靶向至线粒体基质,以观察细菌nif酶是否可以补充这些植物突变。该实验旨在证明细菌nif蛋白可以功能性替换植物等价物(frazzon等人,2007)。成功的互补证明了所选的mtp在功能状态下将固氮酶蛋白靶向线粒体的能力。本实验首次证明了植物中nif多肽的固氮酶相关功能,在这种情况下为nifs和nifu。实施例22.使用质谱分析测定nif多肽表达为了开发不使用westernblot和抗体检测方法并因此避免使用要添加到nif多肽的表位的nif多肽的检测和定量技术,本发明人寻求使用质谱分析方法,特别是使用蛋白水解消化自下向上(bottom-up)的ms/ms方案直接检测和定量各种nif蛋白。这利用了oritrapfusiontribrid质谱仪(thermofisher),其具有三个并行工作的质量分析器,以在全扫描中利用母离子的orbitrap质量分析器提供高质量分辨率和质量精度。相比于目前可获得的其他仪器,该仪器的方法学和灵敏度被认为能够从复杂的、未纯化的蛋白混合物获得更多的蛋白和肽识别。该仪器首先通过以数据依赖的方式分析样品进行测试,以允许在表达nif蛋白的细菌,特别是转化并表达编码16种不同nif多肽的pmit2.1的大肠杆菌细胞中对多肽和蛋白质进行非靶向检测。用胰蛋白酶处理粗蛋白混合物以提供用于检测的分子量范围内的肽。一旦鉴定出由胰蛋白酶消化nif多肽产生的特异性肽,就可以创建靶向列表,其在预期的保留时间特异性地分离目的肽的亲本质量。这种靶向方法有助于消除由于低丰度而丢失来自nif蛋白的肽或由于大量内源细菌或植物胰蛋白酶消化肽段而未通过数据依赖性方法选择来自nif蛋白的肽的可能性。这种基于质谱分析的方案成功地检测到在用pmit2.1质粒转化的大肠杆菌中表达的16种不同nif蛋白的整体表达。在16种nif多肽中,每一种至少检测到一种特异性肽。几乎所有的nif多肽都检测到多个特异性胰蛋白酶消化肽段,对于nif多肽最多可达19种肽,占整个nif多肽序列的4-55%(表10)。许多nif多肽的总pep评分非常高(表10),表明该方法特异性检测和测量nif多肽水平的能力完全可信。在本文中,总后验误差概率(pep)评分是肽光谱匹配的pep值的负对数之和。总pep值越大,检测的假阳性可能性越小。然后,在表达各种mtp::nif基因构建体的本生烟细胞的粗提取物上测试该方法,使用下面描述的方法来处理用含有目的载体(vector)的农杆菌压渗的植物叶片样品。所有的压渗物包括与含有共表达p19沉默抑制蛋白(seqidno:125)的基因构建体的农杆菌的混合物。检测来自p19病毒抑制蛋白的两种特异性胰蛋白酶消化肽段(seqidno:126;seqidno:127)的用作瞬时表达系统成功的阳性对照。该方法成功地特异性检测了在植物细胞中瞬时表达时来自nifk、nifb、nifh、niff、nifj、nifs和nifx多肽中每一种的3-16种特异性胰蛋白酶消化肽段。例如,在植物细胞中从这些nif融合多肽检测到的特异性肽如下所示。对于每个瞬时表达实验,还检测了来自p19的两种特异性肽。在一些使用基因表达其他nif多肽的实验中,在没有检测到特异性nif多肽的情况下,也没有检测到来自p19的两个特异性肽。这表明在这些实验中转化效率较差,可能是由于植物健康状况不佳。用更好的植物重复实验。表10.含有pmit2.1的大肠杆菌中表达的nif多肽的检测示例性地,在植物细胞中检测到来自nifk的特异性肽:inscyplfeqdeyqelfr(seqidno:128)qleeahdaqr(seqidno:129)ealtvdpak(seqidno:130)tftadyqgqpgk(seqidno:131)lpklnlvtgfetylgnfr(seqidno:132)mmeqmavpcsllsdpsevldtpadghyr(seqidno:133)mysggttqqemk(seqidno:134)eapdaidtlllqpwqllk(seqidno:135)fglygdpdfvmgltr(seqidno:136)dsevfincdlwhfr(seqidno:137)qpdfmignsygk(seqidno:138)afevplir(seqidno:139)lgfplfdr(seqidno:140)qttwgyegamnivttlvnavlek(seqidno:141)ldsdtsqlgk(seqidno:142)tdysfdlvr(seqidno:143)在植物细胞中检测到来自nifh的特异性肽lgtqmihfvpr(seqidno:144)mtvieydpack(seqidno:145)vmivgcdpk(seqidno:146)caesggpepgvgcagr(seqidno:147)stttqnlvaalaemgk(seqidno:148)stttqnlvaalaemgkk(seqidno:149)qtdredeliialaek(seqidno:150)aqeiyivcsgemmamyaannisk(seqidno:151)avqgaptmr(seqidno:152)lgglicnsr(seqidno:153)aqntimemaaevgsvedleledvlqigygdvr(seqidno:154)在植物细胞中检测到来自nifb的特异性肽lclstnglvlpdavdr(seqidno:155)faailelladvk(seqidno:156)geseaddaclvavassr(seqidno:157)avqgaptscssfsggk(seqidno:158)insvlipgindsgmagvsr(seqidno:159)qvaqaipqlsvvgiagpgdplaniar(seqidno:160)在植物细胞中检测到来自nifj的特异性肽。iagellpgvfhvsar(seqidno:161)gtaqnpdiyfqer(seqidno:162)ipfvnffdgfr(seqidno:163)ievleyeqlatlldrpaldsfr(seqidno:164)sggitvshlr(seqidno:165)在植物细胞中检测到的来自nifs的特异性肽。ekeidyvvatlppiidr(seqidno:166)eidyvvatlppiidr(seqidno:167)iavdgegaldmaqfr(seqidno:168)eiitsvvehpatlaacehmer(seqidno:169)idmlscsahk(seqidno:170)amnipytaahgtir(seqidno:171)qvyldnnattr(seqidno:172)ipiavgqtr(seqidno:173)gvgclylr(seqidno:174)在植物细胞中检测到来自nifx的特异性肽。vpadttivgllqeiqlywydk(seqidno:175)qfdmvhsdewsmk(seqidno:176)vvdfsvenghqtek(seqidno:177)ragdykddddkpg(seqidno:178)argdykddddkpg(seqidno:179)hvdqhfgatpr(seqidno:180)vafassdyr(seqidno:181)llqeqewhgdpdpr(seqidno:182)在植物细胞中检测到的来自niff的特异性肽laswleeikr(seqidno:183)vlgswtgdsvnyaasr(seqidno:184)qlgeladapvninr(seqidno:185)fvglvldqdnqfdqtear(seqidno:186)方法植物叶片样品用液氮冷冻并用研钵和研杵在液氮下研磨。将两倍体积(v/v)的尿素/sds缓冲液(6m尿素,2%sds,62.5mmph6.8的tris-hcl,65mmdtt)加入到研磨的叶盘中,并在室温下放置(30分钟)用于细胞裂解和用dtt还原蛋白。样品以16,000g离心5分钟。将10-20μl的可溶部分加到含有100μl尿素缓冲液的microcon-30mwco过滤器上,并以10,000g离心10分钟。加入100μl尿素缓冲液和3.5μl40%丙烯酰胺,在室温下孵育30分钟,然后以10,000g离心10分钟,以用丙烯酰胺灭活半胱氨酸残基。在,用100μl碳酸氢铵(水中浓度为25mm)进行2次洗涤和如上所述的离心,然后用稀释在60μlabc中的0.5μg胰蛋白酶(promega)消化,并在37℃下孵育过夜。通过10,000g离心10分钟洗脱胰蛋白酶消化物中的肽。使用280nm处的光密度值(nanodrop)来测定肽浓度,并且将样品在lcms加载缓冲液中稀释至50ng/μl。将每个样品中的250ng胰蛋白酶消化物注入直接偶联到orbitrapfusiontribrid质谱仪的dionexnanomate3000(thermofisher)nanolc系统上。将肽在acclaimpepmapc18(5mm×300μm)捕获柱上用负载溶剂以10μl/min的流速脱盐5分钟,并且在acclaimpepmapc18(150mm×0.075mm)柱上以0.3μl/min的流速在35℃下分离。在60分钟内使用5%到40%溶剂b的线性梯度,然后在5分钟内用40-99%的b洗涤并重新平衡,在99%的b下保持5分钟,在6分钟内恢复至5%的b,并保持7分钟。使用的溶剂为:(a)0.1%甲酸,99.9%水;(b)0.08%甲酸,80%乙腈,19.92%水。纳米lc直接偶联到orbitrapfusionms的nanosprayflex离子源。离子喷雾电压设定为2400v,吹扫气体设定为1arb,离子转移管温度设定为300℃。在数据依赖采集模式中采集数据,其包括orbitap-ms测量扫描,随后并行采集分辨率为120,000的高分辨率orbitrap扫描和线性离子阱中的多个ms/ms事件,采集时间为3秒。第一阶段ms分析以正离子模式在m/z400-1500的质量范围内进行,agc目标为4×105,最大注入时间为50毫秒。在离子阱中获得了前体离子的串联质谱,该前体离子在电荷态为2-7的情况下,超过了1000个计数的强度阈值。使用四极分离获得光谱,隔离窗口为1.6m/z,根据前体离子的大小和电荷将(高能碰撞解离)hcd设置为28%,以实现最佳的肽片段化。将离子阱扫描速率设定为快速,agc靶标为4×103,最大注入时间为300毫秒,将仪器设置为在orbitrap采集高分辨率ms光谱的3秒窗口内利用最大可并行时间将离子注入阱。设定动态排除以在15秒间隔和10ppm的质量公差下一次出现后排除前体离子。使用sequest算法在proteomediscovererv2.1(thermofisher)中进行蛋白鉴定。选择脲甲基作为烷化剂,胰蛋白酶作为消化酶,选择对h、m、w和去酰胺化的n和q进行氧化的动态修饰,最多三个修饰。在nif构建体的内部数据库、常见污染物和由uniprot注释的生物体特异性数据库中搜索串联质谱分析数据。使用由proteomediscoverer软件内的内置fdr工具确定的1%的全局错误发现率(fdr)手动整理数据库搜索结果以产生蛋白识别。本领域技术人员应当理解,在不脱离本公开的广义范围的情况下,可以对上述实施方案行多种变化和/或修改。因此,本实施方案在所有方面都被认为是说明性的而非限制性的。本文讨论和/或引用的所有出版物均全文引入本文。包括在本说明书中的文件、动作、材料、装置、物品等的任何讨论仅仅是为了提供本发明的上下文。不应视为承认这些内容中的任何或全部形成现有技术基础的一部分或者是与本发明相关的领域中的公知常识,因为其存在于本申请的每个权利要求的优先权日之前。参考文献abe等人,(2000).细胞(cell)100:551-560.allen等人,(1994).生物技术综述(crit.rev.biotechnol.)14:225-249.allen等人,(1995).生物化学杂志(j.biol.chem.)270:26890-26896.agar等人,(2000).生物无机化学杂志(journalofbiologicalinorganicchemistry)5:167-177.balk和pilon(2011).植物科学的趋势(trendsplantsci)16:218-226.bechtold等人,(1993).法国科学院学报(c.r.acad.sci.,comptesrendusdel'academiedessciences)paris316:1194-1199.becker等人,(2012).生物化学趋势(trendsinbiochemicalsciences)37:85-91.bevan等人,(1983).自然(nature)304:184-187.boison等人,(2006).微生物学文献集(arch.microbiol.)186:367-376.birchler,j.a.(2015).染色体研究(chromosomeres)23:77-85.block等人,(1987).欧洲分子生物学学会杂志(theembojournal)6:2513-2518.breuers等人,(2012).植物科学前沿3(frontplantsci3).brigle等人,(1987).细菌学杂志(j.bacteriol.)169:1547-1553.brosnan等人,(2007).美国科学院院报(proc.natl.acad.sci.usa),104:14741-14746.bruce(2001).生物化学与生物物理学报——分子细胞研究(biochimicaetbiophysicaacta(bba)-molecularcellresearch)1541:2-21.burén等人,(2017a).植物科学前沿(frontplantsci)8:1567.burén等人,(2017b).美国化学学会合成生物学杂志(acssyntheticbiology)6(6):1043-1055.buren和rubio(2018)欧洲微生物学会联合会微生物学快报(femsmicrobiollett)365:fnx274;doi:10.1093/femsle/fnx274.campbell等人,(2014).昆虫生物化学和分子生物学(insectbiochemmolbiol)48:40-50.carrie等人,(2010).生物化学杂志(journalofbiologicalchemistry)285:36138-36148.chacinska等人,(2009).细胞(cell)138:628-644.chen等人,(2013).先进药物输送评论(advanceddrugdeliveryreviews)65:1357-1369.cheng等人,(2005).生物化学与生物物理学研究通讯(biochemicalandbiophysicalresearchcommunications)329:966-975.chiu等人,(2001).生物化学(biochemistry)40:641-650.christiansen等人,(1998)生物化学-美国(biochemistry-us)37:12611-12623.clausen等人,(2000).美国科学院院报(proc.natl.acad.sci.u.s.a.)97:3856-3861.cotton(2009).美国化学协会期刊(j.am.chem.soc.)131:4558-4559.coutu等人,(2007).转基因研究(transgenicres)16:771-781.couturier等人,(2013).植物科学前沿(frontiersinplantscience)4,art.259.cui等人,(2013).美国科学院院报(proceedingsofthenationalacademyofsciences)110,2052-2057.cupp-vickery等人,(2003).分子生物学杂志(j.mol.biol.)330:1049-1059.curatti等人,(2006).美国科学院院报(proc.natl.acad.sci.u.s.a.)103:5297-5301.curatti和rubio(2014).植物科学(plantsci)225:130-137.darshi等人,(2012).生物化学杂志(journalofbiologicalchemistry)287:39480-39491.de'ath等人,(2012).美国科学院院报(proc.natl.acad.sci.usa)109:17995-17999.debruijn(2015).见:生物固氮(in:biologicalnitrogenfixation)pp.1087-1101.约翰·威利父子出版公司(johnwiley&sons,inc.)dekok等人,(2014).acs合成生物学(acssynth.biol.)3:97–106.dilworth等人,(1988).生物化学杂志(biochem.j.)249:745-751.dilworth等人,(1993).生物化学杂志(biochem.j.)289:395-400.dossantos(2011).分子生物学方法(methods.mol.biol.)766:81-92.dossantos等人,(2012).bm基因组学(bmcgenomics)13:162.drummond(1985).生物化学杂志(biochem.j.)232:891-896.eady(1996).化学评论(chem.rev.)96:3013-3030.engler等人,,美国化学学会合成生物学杂志(acssyntheticbiology)3(11):839-843.fani等人,(2000).分子进化杂志(j.mol.evol.)51:1-11.fay等人,(2015)美国科学院院报(procnatlacadsciusa)112:14829-14833.fay等人,(2016).美国科学院院报(proc.natl.acad.sci.u.s.a.)2016:9504-9508.frazzon等人,(2007).植物分子生物学(plantmolecularbiology)64:225-240.furste,等人,(1986).基因(gene)48:119-131.gavini等人,(1998).生物化学与生物物理学研究通讯(biochemicalandbiophysicalresearchcommunications).244(2):498-504.gavini等人,(2006).细菌学杂志(journalofbacteriology)188:6020-6025.geddes等人,(2015).生物技术新观点(curropinbiotech)32:216-222.geigenberger和fernie(2014).抗氧化剂与氧化还原信号(antioxidredoxsign)21:1389-1421.glaser和deshi(1999).生物能学与生物膜杂志(jbioenergbiomembr)31:259-274.glibert等人,(2014).环境研究快报(environreslett)9:e105001;doi.org/10.1088/1748-9326/9/10/105001.glick等人,(1992).细胞(cell)69:809-822.goodwin等人,(1998)生物化学-美国(biochemistry-us)37:10420-10428.good和beatty(2011).公共科学图书馆·生物学(plosbiol)9,e1001124.hu等人,(2004).j.生物化学杂志(biol.chem.)279:54963-54971.hu等人,(2005).美国科学院院报(proc.natl.acad.sci.u.s.a.)102:3236-3241.hu等人,(2006).美国科学院院报(proc.natl.acad.sci.u.s.a.)103:17119-17124.hu等人,(2008).生物化学(biochemistry)47:3973-3981.hu和ribbe(2011).配位化学评述(coordinationchemistryreviews)255:1218-1224.hu和ribbe(2013).生化与生物物理学报:生物能学(bba-bioenergetics)1827:1112-1122.huang等人,(2009).植物生理学(plantphysiology)150(3):1272-1285.hurt等人,(1985).欧洲分子生物学学会杂志(emboj.)4:2061-2068.hwang等人,(1996).分子进化杂志(j.mol.evol.)nov;43:536-540.igarashi和seefeldt(2003).生物化学与分子生物学评论(crit.rev.biochem.mol.biol.)38:351-384.ivleva等人,(2016).公共科学图书馆·综合(plosone)11,e0160951.johnson等人,(2005).生物化学学会学报(biochem.soc.trans.)33:90-93.kaiser等人,(2011).科学(science)331:91-94.kerscher等人,(1997).细胞生物学杂志(thejournalofcellbiology)139:1663-1675.kim和rees(1994).生物化学(biochemistry)33:389-397.kohler等人,(1997).植物杂志(plantj.)11:613-621.lahiri等人,(2005).生物化学与生物物理学研究通讯(biochemicalandbiophysicalresearchcommunications)337:677-684.lawson和smith(2002).金属离子生物系统(metionsbiolsyst);39:75-119.lee等人,(2000).细菌学杂志(j.bacteriol.)182:7088-7091.lee等人,(2009).美国科学院院报(proc.natl.acad.sci.u.s.a.)106,18474-18478.lee等人,(2012).植物细胞(plantcell)24:5037-5057.lill和mühlenhoff(2008).生物化学年报(annualreviewofbiochemistry)77:669-700.lopez-torrejon等人,(2016).自然通讯(naturecommunications)7:11426.mackenzie和mcintosh(1999).植物细胞(plantcell)11:571-585.marques等人,(2014).晶体学报(actacrystallographicasection)f70(5):669-672.masukawa等人,(2007).应用与环境微生物学(appl.environ.microbiol.)73:7562-7570.mayer等人,(1999).分子生物学杂志(j.mol.biol.)292:871-891.merrick和dixon(1984).生物技术趋势(trendsbiotechnol)2:162-166.miller和eady(1988).生物化学杂志(biochem.j.)256:429-432.mueller等人,(2012).自然(nature)490:254-257.mühlenhoff等人,(2003).欧洲分子生物学学会杂志(emboj.)22:4815-4825.murcha等人,(2004).分子生物学杂志(jmolbiol)344:443-454.murcha等人,(2014).生物化学与生物物理学报:综合学科(bba-gensubjects)1840:1233-1245.oldroyd和dixon(2014)生物技术新观点(curropinbiotechnol)26:19-24.olson等人,(2000).生物化学(biochemistry);39:16213-16219.等人,(1996).科学(science)273:1392-1395.ouzounis等人,(1994).生物化学科学前沿(trendsbiochem.sci.)19:199-200.petrie(2014).公共科学图书馆·综合(plosone)9:issue1,e85061.petrova等人,(2000).生物化学与生物物理研究通讯(biochem.biophys.res.commun.)270:863-867.pfanner和geissler(2001)自然评论分子细胞生物学(nat.rev.mol.cellbiol.)2:339-349.poza-carrion等人,(2014).细菌学杂志(journalofbacteriology)196:595-603.prasad等人,(1992).植物分子生物学(plantmolecularbiology)18(5):873-885.pratte等人,(2006).细菌学杂志(j.bacteriol.)188:5806-5811.rees等人,(2005).皇家学会哲学汇刊a辑:数学、物理学与工程学(philostransamathphysengsci.)363:971-984.robson和postgate(1980).微生物学年度评论(annualreviewofmicrobiology)34:183-207.robson等人,(1989).欧洲分子生物学学会杂志(emboj.)8:1217-1224.rockstrom等人,(2009).自然(nature)461:472-475.roise等人,(1986)欧洲分子生物学学会杂志(theembojournal)5:1327-1334.roise和schatz(1988).生物化学杂志(j.biol.chem.)263:4509-4511.rubio等人,(2004)生物化学杂志(jbiolchem279):19739-19746.rubio和ludden(2008).微生物学年度评论(annurevmicrobiol)62:93-111.sahoo等人,(2014).植物(planta)240:855-875.sali和blundell,(1993).分子生物学杂志(j.mol.biol.)234:779-815.santi等人,(2013).植物学年报(annbot)111:743-767.schleiff和soll(2000)植物(planta)211:449–456.schmid等人,(2002).科学(science)296:352-356.schmitz等人,(2001).欧洲微生物学会联合会微生物学快报(femsmicrobiollett).195:97-102.schunmann等人,(2003a).功能植物生物学植物(functionalplantbiology)30(4):443-452.schunmann等人,(2003b).功能植物生物学植物(functionalplantbiology)30(4):453-460.seefeldt等人,(2009).生物化学年报(annurevbiochem)78:701-722.shah等人,(1999).细菌学杂志(j.bacteriol.)181:2797-2801.siddavattam等人,(1993).分子遗传学和基因组学(mol.gen.genet.)239:435-440.smil(2002).ambio31:126-131.smanski等人,(2014).自然生物技术(naturebiotechnology)32:1241-1249.sorlie等人,(2001).生物化学(biochem.)40:1540-1549.schindelin等人,(1997).自然(nature)387:370-376.staples等人,(2007).细菌学杂志(j.bacteriol.)189:7392-7398.studier和moffatt(1986).分子生物学杂志(jmolbiol)189:113-130.suh等人,(2003).生物化学杂志(journalofbiologicalchemistry)278:5353-5360.sutton等人,(2008).环境污染(environpollut)156:583-604.temme等人,(2012).美国科学院院报(proc.natl.acad.sci.u.s.a.)109(18):7085-7090.tezcan等人,(2005).科学(science)309:1377-1380.thiel等人,(1995)美国科学院院报(procnatlacadsciusa)92:9358-9362.thiel等人,(1997).细菌学杂志(j.bacteriol.)179:5222-5225.voinnet等人,(2003).植物杂志(plantjournal)33:949-956.vonheijne(1986).欧洲分子生物学学会杂志(emboj.)5:1335-1342.waldron等人,(1985).植物分子生物学(plantmolbiol)5:103-108.wang等人,(2013).公共科学图书馆·遗传学(plosgenet)9,e1003865.weber等人,(2011)公共科学图书馆·综合(plosone).6(2),pp.e16765.wiig等人,(2011)美国科学院院报(procnatlacadsciusa)108:8623-8627.wood等人,(2006).美国科学院院报(proc.natl.acad.sci.usa).103:14631-14636.wood等人,(2009).植物生物技术杂志(plantbiotechnolj.)7:914-924.yuvaniyama等人,(2000).美国科学院院报(proc.natl.acad.sci.u.s.a.)97:599-604.zhang和glaser(2002).植物科学的趋势(trendsplantsci)7:14-21.zhang等人,(2009).自然科学进展(progressinnaturalscience)19:1197-1200.zheng等人,(1993).美国科学院院报(proc.natl.acad.sci.u.s.a.)90:2754-2758zheng等人,(1997).细菌学杂志(j.bacteriol.)179:5963-5966.序列表<110>澳大利亚联邦科学与工业研究组织<120>固氮酶<130>527134<150>au2017900359<151>2017-02-06<160>186<170>patentin3.5版<210>1<211>29<212>prt<213>酿酒酵母<400>1metleuserleuargglnserileargphephelysproalathrarg151015thrleucysserserargtyrleuleuglnglnlyspro2025<210>2<211>31<212>prt<213>人工序列<220><223>衍生序列<400>2metglyleuserleuleuargglnserileargphephelysproala151015thrargthrleucysserserargtyrleuleugluglnlyspro202530<210>3<211>8<212>prt<213>人工序列<220><223>基序<220><221>x<222>(1)..(1)<223>任意氨基酸<220><221>x<222>(3)..(5)<223>任意氨基酸<220><221>x<222>(8)..(8)<223>任意氨基酸<400>3xaaargxaaxaaxaaserserxaa15<210>4<211>2346<212>dna<213>人工序列<220><223>载体序列<400>4ctcctgtggttggcatgcacatacaaatggacgaacggataaaccttttcacgccctttt60aaatatccgattattctaataaacgctcttttctcttaggtttacccgccaatatatcct120gtcaaacactgatagtttaaactgaaggcgggaaacgacaatctgctagtggatctccca180gtcacgacgttgtaaaacgggcgccccgcggaaagctttcgacgaattaattccaatccc240acaaaaatctgagcttaacagcacagttgctcctctcagagcagaatcgggtattcaaca300ccctcatatcaactactacgttgtgtataacggtccacatgccggtatatacgatgactg360gggttgtacaaaggcggcaacaaacggcgttcccggagttgcacacaagaaatttgccac420tattacagaggcaagagcagcagctgacgcgtacacaacaagtcagcaaacagacaggtt480gaacttcatccccaaaggagaagctcaactcaagcccaagagctttgctaaggccctaac540aagcccaccaaagcaaaaagcccactggctcacgctaggaaccaaaaggcccagcagtga600tccagccccaaaagagatctcctttgccccggagattacaatggacgatttcctctatct660ttacgatctaggaaggaagttcgaaggtgaaggtgacgacactatgttcaccactgataa720tgagaaggttagcctcttcaatttcagaaagaatgctgacccacagatggttagagaggc780ctacgcagcaggtctcatcaagacgatctacccgagtaacaatctccaggagatcaaata840ccttcccaagaaggttaaagatgcagtcaaaagattcaggactaattgcatcaagaacac900agagaaagacatatttctcaagatcagaagtactattccagtatggacgattcaaggctt960gcttcataaaccaaggcaagtaatagagattggagtctctaaaaaggtagttcctactga1020atctaaggccatgcatggagtctaagattcaaatcgaggatctaacagaactcgccgtga1080agactggcgaacagttcatacagagtcttttacgactcaatgacaagaagaaaatcttcg1140tcaacatggtggagcacgacactctggtctactccaaaaatgtcaaagatacagtctcag1200aagaccaaagggctattgagacttttcaacaaaggataatttcgggaaacctcctcggat1260tccattgcccagctatctgtcacttcatcgaaaggacagtagaaaaggaaggtggctcct1320acaaatgccatcattgcgataaaggaaaggctatcattcaagatctctctgccgacagtg1380gtcccaaagatggacccccacccacgaggagcatcgtggaaaaagaagacgttccaacca1440cgtcttcaaagcaagtggattgatgtgacatctccactgacgtaagggatgacgcacaat1500cccactatccttcgcaagacccttcctctatataaggaagttcatttcatttggagagga1560cacgctcgagtaatacgactcactatagggagaccacaacggtttccctctagaaataat1620tttgtttaactttaagaaggagatataccatgggcttgtcactactacgtcaatctataa1680gatttttcaagccagccacaagaactttgtgtagctctagatatctgcttgagcaaaaac1740ccggcgcgccggctgctaacaaagcccgaaaggaagctgagttggctgctgccaccgctg1800agcaataactagcataaccccttggggcctctaaacgggtcttgaggggttttttgctgc1860ttaagattgaatcctgttgccggtcttgcgatgattatcatataatttctgttgaattac1920gttaagcatgtaataattaacatgtaatgcatgacgttatttatgagatgggtttttatg1980attagagtcccgcaattatacatttaatacgcgatagaaaacaaaatatagcgcgcaaac2040taggataaattatcgcgcgcggtgtcatctatgttactagatccctagggaagttcctat2100tccgaagttcctattctctgaaaagtataggaacttctttgcgtattgggcgctcttggc2160ctttttggccaccggtcgtacggttaaaaccaccccagtacattaaaaacgtccgcaatg2220tgttattaagttgtctaagcgtcaatttgtttacaccacaatatatcctgccaccagcca2280gccaacagctccccgaccggcagctcggcacaaaatcaccactcgatacaggcagcccat2340cagtcc2346<210>5<211>293<212>prt<213>肺炎克雷伯菌<400>5metthrmetargglncysalailetyrglylysglyglyileglylys151015serthrthrthrglnasnleuvalalaalaleualaglumetglylys202530lysvalmetilevalglycysaspprolysalaaspserthrargleu354045ileleuhisalalysalaglnasnthrilemetglumetalaalaglu505560valglyservalgluaspleugluleugluaspvalleuglnilegly65707580tyrglyaspvalargcysalagluserglyglyprogluproglyval859095glycysalaglyargglyvalilethralaileasnpheleugluglu100105110gluglyalatyrgluaspaspleuaspphevalphetyraspvalleu115120125glyaspvalvalcysglyglyphealametproilearggluasnlys130135140alaglngluiletyrilevalcysserglyglumetmetalamettyr145150155160alaalaasnasnileserlysglyilevallystyralalyssergly165170175lysvalargleuglyglyleuilecysasnserargglnthrasparg180185190gluaspgluleuileilealaleualaglulysleuglythrglnmet195200205ilehisphevalproargaspasnilevalglnargalagluilearg210215220argmetthrvalileglutyraspproalacyslysglnalaasnglu225230235240tyrargthrleualaglnlysilevalasnasnthrmetlysvalval245250255prothrprocysthrmetaspgluleugluserleuleumetgluphe260265270glyilemetgluglugluaspthrserileileglylysthralaala275280285glugluasnalaala290<210>6<211>483<212>prt<213>肺炎克雷伯菌<400>6metmetthrasnalathrglygluargasnleualaleuileglnglu151015valleugluvalpheprogluthralaarglysgluargarglyshis202530metmetvalseraspprolysmetlysservalglylyscysileile354045serasnarglysserglnproglyvalmetthrvalargglycysala505560tyralaglyserlysglyvalvalpheglyproilelysaspmetala65707580hisileserhisglyproalaglycysglyglntyrserargalaglu859095argargasntyrtyrthrglyvalserglyvalaspserpheglythr100105110leuasnphethrserasppheglngluargaspilevalpheglygly115120125asplyslysleuserlysleuilegluglumetgluleuleuphepro130135140leuthrlysglyilethrileglnserglucysprovalglyleuile145150155160glyaspaspileseralavalalaasnalaserserlysalaleuasp165170175lysprovalileprovalargcysgluglypheargglyvalsergln180185190serleuglyhishisilealaasnaspvalvalargasptrpileleu195200205asnasnarggluglyglnprophegluthrthrprotyraspvalala210215220ileileglyasptyrasnileglyglyaspalatrpalaserargile225230235240leuleugluglumetglyleuargvalvalalaglntrpserglyasp245250255glythrleuvalglumetgluasnthrprophevallysleuasnleu260265270valhiscystyrargsermetasntyrilealaarghismetgluglu275280285lyshisglnileprotrpmetglutyrasnphepheglyprothrlys290295300ilealagluserleuarglysilealaaspglnpheaspaspthrile305310315320argalaasnalaglualavalilealaargtyrgluglyglnmetala325330335alaileilealalystyrargproargleugluglyarglysvalleu340345350leutyrileglyglyleuargproarghisvalileglyalatyrglu355360365aspleuglymetgluileilealaalaglytyrgluphealahisasn370375380aspasptyraspargthrleuproaspleulysgluglythrleuleu385390395400pheaspaspalasersertyrgluleuglualaphevallysalaleu405410415lysproaspleuileglyserglyilelysglulystyrilephegln420425430lysmetglyvalpropheargglnmethissertrpasptyrsergly435440445protyrhisglytyraspglyphealailephealaargaspmetasp450455460metthrleuasnasnproalatrpasngluleuthralaprotrpleu465470475480lysserala<210>7<211>520<212>prt<213>肺炎克雷伯菌<400>7metserglnthrileasplysileasnsercystyrproleupheglu151015glnaspglutyrglngluleupheargasnlysargglnleugluglu202530alahisaspalaglnargvalglngluvalphealatrpthrthrthr354045alaglutyrglualaleuasnpheargargglualaleuthrvalasp505560proalalysalacysglnproleuglyalavalleucysserleugly65707580phealaasnthrleuprotyrvalhisglyserglnglycysvalala859095tyrpheargthrtyrpheasnarghisphelysgluproilealacys100105110valseraspsermetthrgluaspalaalavalpheglyglyasnasn115120125asnmetasnleuglyleuglnasnalaseralaleutyrlysproglu130135140ileilealavalserthrthrcysmetalagluvalileglyaspasp145150155160leuglnalapheilealaasnalalyslysaspglyphevalaspser165170175serilealavalprohisalahisthrproserpheileglyserhis180185190valthrglytrpaspasnmetphegluglyphealalysthrphethr195200205alaasptyrglnglyglnproglylysleuprolysleuasnleuval210215220thrglyphegluthrtyrleuglyasnpheargvalleulysargmet225230235240metgluglnmetalavalprocysserleuleuseraspproserglu245250255valleuaspthrproalaaspglyhistyrargmettyrserglygly260265270thrthrglnglnglumetlysglualaproaspalaileaspthrleu275280285leuleuglnprotrpglnleuleulysserlyslysvalvalglnglu290295300mettrpasnglnproalathrgluvalalaileproleuglyleuala305310315320alathraspgluleuleumetthrvalserglnleuserglylyspro325330335ilealaaspalaleuthrleugluargglyargleuvalaspmetmet340345350leuaspserhisthrtrpleuhisglylyslyspheglyleutyrgly355360365aspproaspphevalmetglyleuthrargpheleuleugluleugly370375380cysgluprothrvalileleuserhisasnalaasnlysargtrpgln385390395400lysalametasnlysmetleuaspalaserprotyrglyargaspser405410415gluvalpheileasncysaspleutrphispheargserleumetphe420425430thrargglnproaspphemetileglyasnsertyrglylyspheile435440445glnargaspthrleualalysglylysalaphegluvalproleuile450455460argleuglypheproleupheasparghishisleuhisargglnthr465470475480thrtrpglytyrgluglyalametasnilevalthrthrleuvalasn485490495alavalleuglulysleuaspseraspthrserglnleuglylysthr500505510asptyrserpheaspleuvalarg515520<210>8<211>220<212>prt<213>肺炎克雷伯菌<400>8metseraspasnaspthrleuphetrpargmetleualaleuphegln151015serleuproaspleuglnproalaglnilevalasptrpleualagln202530gluserglygluthrleuthrprogluargleualathrleuthrgln354045proglnleualaalaserpheproseralathralavalmetserpro505560alaargtrpserargvalmetalaserleuglnglyalaleuproala65707580hisleuargilevalargproalaglnargthrproglnleuleuala859095alaphecysserglnaspglyleuvalileasnglyhispheglygln100105110glyargleuphepheiletyralapheaspgluglnglyglytrpleu115120125tyraspleuargargtyrproseralaprohisglnglnglualaasn130135140gluvalargalaargleuilegluaspcysglnleuleuphecysgln145150155160gluileglyglyproalaalaalaargproilearghisargilehis165170175prometlysalaglnproglythrthrileglnalaglncysgluala180185190ileasnthrleuleualaglyargleuproprotrpleualalysarg195200205leuasnargaspasnproleuglugluargvalphe210215220<210>9<211>468<212>prt<213>肺炎克雷伯菌<400>9metthrsercysserserpheserglyglylysalacysargproala151015aspaspseralaleuthrproleuvalalaasplysalaalaalahis202530procystyrserarghisglyhishisargphealaargmethisleu354045provalalaproalacysasnleuglncysasntyrcysasnarglys505560pheaspcysserasngluserargproglyvalserserthrleuleu65707580thrprogluglnalavalvallysvalargglnvalalaglnalaile859095proglnleuservalvalglyilealaglyproglyaspproleuala100105110asnilealaargthrpheargthrleugluleuilearggluglnleu115120125proaspleulysleucysleuserthrasnglyleuvalleuproasp130135140alavalaspargleuleuaspvalglyvalasphisvalthrvalthr145150155160ileasnthrleuaspalagluilealaalaglniletyralatrpleu165170175trpleuaspglygluargtyrserglyargglualaglygluileleu180185190ilealaargglnleugluglyvalargargleuthralalysglyval195200205leuvallysileasnservalleuileproglyileasnaspsergly210215220metalaglyvalserargalaleuargalaserglyalapheilehis225230235240asnilemetproleuilealaargprogluhisglythrvalphegly245250255leuasnglyglnprogluproaspalagluthrleualaalathrarg260265270serargcysglygluvalmetproglnmetthrhiscyshisglncys275280285argalaaspalaileglymetleuglygluaspargserglnglnphe290295300thrglnleuproalaprogluserleuproalatrpleuproileleu305310315320hisglnargalaglnleuhisalaserilealathrargglygluser325330335glualaaspaspalacysleuvalalavalalaserserargglyasp340345350valileaspcyshispheglyhisalaaspargphetyriletyrser355360365leuseralaalaglymetvalleuvalasngluargphethrprolys370375380tyrcysglnglyargaspaspcysgluproglnaspasnalaalaarg385390395400phealaalaileleugluleuleualaaspvallysalavalphecys405410415valargileglyhisthrprotrpglnglnleugluglngluglyile420425430gluprocysvalaspglyalatrpargprovalsergluvalleupro435440445alatrptrpglnglnargargglysertrpproalaalaleuprohis450455460lysglyvalala465<210>10<211>457<212>prt<213>肺炎克雷伯菌<400>10metlysglyasngluileleualaleuleuaspgluproalacysglu151015hisasnhislysglnlysserglycysseralaprolysproglyala202530thralaalaglycysalapheaspglyalaglnilethrleuleupro354045ilealaaspvalalahisleuvalhisglyproileglycysalagly505560sersertrpaspasnargglyseralaserserglyprothrleuasn65707580argleuglyphethrthraspleuasngluglnaspvalilemetgly859095argglygluargargleuphehisalavalarghisilevalthrarg100105110tyrhisproalaalavalpheiletyrasnthrcysvalproalamet115120125gluglyaspaspleuglualavalcysglnalaalaglnthralathr130135140glyvalprovalilealaileaspalaalaglyphetyrglyserlys145150155160asnleuglyasnargproalaglyaspvalmetvallysargvalile165170175glyglnarggluproalaprotrpprogluserthrleuphealapro180185190gluglnarghisaspileglyleuileglyglupheasnilealagly195200205gluphetrphisileglnproleuleuaspgluleuglyileargval210215220leuglyserleuserglyaspglyargphealagluileglnthrmet225230235240hisargalaglnalaasnmetleuvalcysserargalaleuileasn245250255valalaargalaleugluglnargtyrglythrprotrppheglugly260265270serphetyrglyileargalathrseraspalaleuargglnleuala275280285alaleuleuglyaspaspaspleuargglnargthrglualaleuile290295300alaargglugluglnalaalagluleualaleuglnprotrpargglu305310315320glnleuargglyarglysalaleuleutyrthrglyglyvallysser325330335trpservalvalseralaleuglnaspleuglymetthrvalvalala340345350thrglythrarglysserthrglugluasplysglnargileargglu355360365leumetglygluglualavalmetleuglugluglyasnalaargthr370375380leuleuaspvalvaltyrargtyrglnalaaspleumetilealagly385390395400glyargasnmettyrthralatyrlysalaargleupropheleuasp405410415ileasnglngluarggluhisalaphealaglytyrglnglyileval420425430thrleualaargglnleucysglnthrileasnserproiletrppro435440445glnthrhisserargalaprotrparg450455<210>11<211>461<212>prt<213>肺炎克雷伯菌<400>11metalaaspilepheargthrasplysproleualavalserproile151015lysthrglyglnproleuglyalaileleualaserleuglyileglu202530hisserileproleuvalhisglyalaglnglycysseralapheala354045lysvalphepheileglnhisphehisaspprovalproleuglnser505560thralametaspprothrserthrilemetglyalaaspglyasnile65707580phethralaleuaspthrleucysglnargasnasnproglnalaile859095valleuleuserthrglyleuserglualaglnglyseraspileser100105110argvalvalargglnphearggluglutyrproarghislysglyval115120125alaileleuthrvalasnthrproaspphetyrglysermetgluasn130135140glypheseralavalleugluservalilegluglntrpvalpropro145150155160alaproargproalaglnargasnargargvalasnleuleuvalser165170175hisleucysserproglyaspileglutrpleuargargcysvalglu180185190alapheglyleuglnproileileleuproaspleualaglnsermet195200205aspglyhisleualaglnglyasppheserproleuthrglnglygly210215220thrproleuargglnilegluglnmetglyglnserleucysserphe225230235240alaileglyvalserleuhisargalaserserleuleualaproarg245250255cysargglygluvalilealaleuprohisleumetthrleugluarg260265270cysaspalapheilehisglnleualalysileserglyargalaval275280285proglutrpleugluargglnargglyglnleuglnaspalametile290295300aspcyshismettrpleuglnglyglnargmetalailealaalaglu305310315320glyaspleuleualaalatrpcysaspphealaasnserglnglymet325330335glnproglyproleuvalalaprothrglyhisproserleuarggln340345350leuprovalgluargvalvalproglyaspleugluaspleuglnthr355360365leuleucysalahisproalaaspleuleuvalalaasnserhisala370375380argaspleualagluglnphealaleuproleuvalargalaglyphe385390395400proleupheasplysleuglyglupheargargvalargglnglytyr405410415serglymetargaspthrleuphegluleualaasnleuileargglu420425430arghishishisleualahistyrargserproleuargglnasnpro435440445gluserserleuserthrglyglyalatyralaalaasp450455460<210>12<211>167<212>prt<213>肺炎克雷伯菌<400>12metproproleuasptrpleuargargleutrpleuleutyrhisala151015glylysglyserpheproleuargmetglyleuserproargasptrp202530glnalaleuargargargleuglygluvalgluthrproleuaspgly354045gluthrleuthrargargargleumetalagluleuasnalathrarg505560gluglugluargglnglnleuglyalatrpleualaglytrpmetgln65707580glnaspalaglyprometalaglnileilealagluvalserleuala859095pheasnhisleutrpglnaspleuglyleualaserargalagluleu100105110argleuleumetseraspcyspheproglnleuvalvalmetasnglu115120125hisasnmetargtrplyslysphephetyrargglnargcysleuleu130135140glnglnglygluvalilecysargserprosercysaspglucystrp145150155160gluargseralacyspheglu165<210>13<211>400<212>prt<213>肺炎克雷伯菌<400>13metlysglnvaltyrleuaspasnasnalathrthrargleuasppro151015metvalleuglualametmetpropheleuthraspphetyrglyasn202530proserserilehisasppheglyileproalaglnalaalaleuglu354045argalahisglnglnalaalaalaleuleuglyalaglutyrproser505560gluileilephethrsercysalathrglualathralathralaile65707580alaseralailealaleuleuprogluargarggluileilethrser859095valvalgluhisproalathrleualaalacysgluhismetgluarg100105110gluglytyrargilehisargilealavalaspglygluglyalaleu115120125aspmetalaglnpheargalaalaleuserproargvalalaleuval130135140servalmettrpalaasnasngluthrglyvalleupheproilegly145150155160glumetalagluleualahisgluglnglyalaleuphehiscysasp165170175alavalglnvalvalglylysileproilealavalglyglnthrarg180185190ileaspmetleusercysseralahislysphehisglyprolysgly195200205valglycysleutyrleuargargglythrargpheargproleuleu210215220argglyglyhisglnglutyrglyargargalaglythrgluasnile225230235240cysglyilevalglymetglyalaalacysgluleualaasnilehis245250255leuproglymetthrhisileglyglnleuargasnargleugluhis260265270argleuleualaservalproservalmetvalmetglyglyglygln275280285proalavalproglythrvalasnleualapheglupheileglugly290295300glualaileleuleuleuleuasnglnalaglyilealaalaserser305310315320glyseralacysthrserglyserleugluproserhisvalmetarg325330335alametasnileprotyrthralaalahisglythrileargpheser340345350leuserargtyrthrargglulysgluileasptyrvalvalalathr355360365leuproproileileaspargleuargalaleuserprotyrtrpgln370375380asnglylysproargproalaaspalavalphethrprovaltyrgly385390395400<210>14<211>274<212>prt<213>肺炎克雷伯菌<400>14mettrpasntyrserglulysvallysasphisphepheasnproarg151015asnalaargvalvalaspasnalaasnalavalglyaspvalglyser202530leusercysglyaspalaleuargleumetleuargvalaspprogln354045sergluileilegluglualaglypheglnthrpheglycysglyser505560alailealaserserseralaleuthrgluleuileileglyhisthr65707580leualaglualaglyglnilethrasnglnglnilealaasptyrleu859095aspglyleuproproglulysmethiscysservalmetglyglnglu100105110alaleuargalaalailealaasnpheargglygluserleugluglu115120125gluhisaspgluglylysleuilecyslyscyspheglyvalaspglu130135140glyhisileargargalavalglnasnasnglyleuthrthrleuala145150155160gluvalileasntyrthrlysalaglyglyglycysthrsercyshis165170175glulysilegluleualaleualagluileleualaglnglnprogln180185190thrthrproalavalalaserglylysaspprohistrpglnserval195200205valaspthrilealagluleuargprohisileglnalaaspglygly210215220aspmetalaleuleuservalthrasnhisglnvalthrvalserleu225230235240serglysercysserglycysmetmetthraspmetthrleualatrp245250255leuglnglnlysleumetgluargthrglycystyrmetgluvalval260265270alaala<210>15<211>156<212>prt<213>肺炎克雷伯菌<400>15metproproileasnargglnpheaspmetvalhisseraspglutrp151015sermetlysvalalaphealaserserasptyrarghisvalaspgln202530hispheglyalathrproargleuvalvaltyrglyvallysalaasp354045argvalthrleuileargvalvalasppheservalgluasnglyhis505560glnthrglulysilealaargargilehisalaleugluaspcysval65707580thrleuphecysvalalaileglyaspalavalpheargglnleuleu859095glnvalglyvalargalagluargvalproalaaspthrthrileval100105110glyleuleuglngluileglnleutyrtrptyrasplysglyglnarg115120125lysasnglnargglnargaspprogluargphethrargleuleugln130135140gluglnglutrphisglyaspproaspproargarg145150155<210>16<211>176<212>prt<213>肺炎克雷伯菌<400>16metalaasnileglyilephepheglythraspthrglylysthrarg151015lysilealalysmetilehislysglnleuglygluleualaaspala202530provalasnileasnargthrthrleuaspaspphemetalatyrpro354045valleuleuleuglythrprothrleuglyaspglyglnleuprogly505560leuglualaglycysgluserglusertrpserglupheilesergly65707580leuaspaspalaserleulysglylysthrvalalaleupheglyleu859095glyaspglnargglytyrproaspasnphevalserglymetargpro100105110leupheaspalaleuseralaargglyalaglnmetileglysertrp115120125proasngluglytyrglupheseralaserseralaleugluglyasp130135140argphevalglyleuvalleuaspglnaspasnglnpheaspglnthr145150155160glualaargleualasertrpleuglugluilelysargthrvalleu165170175<210>17<211>148<212>prt<213>肺炎克雷伯菌<400>17metargprolysphethrpheserglugluvalargvalvalargala151015ileargasnaspglythrvalalaglyphealaproglyalaleuleu202530valargargglyserthrglyphevalargasptrpglyvalpheleu354045glnaspglnileiletyrglnilehispheprogluthraspargile505560ileglycysarggluglngluleuileproilethrglnprotrpleu65707580alaglyasnleuglntyrargaspservalthrcysglnmetalaleu859095alavalasnglyaspvalvalvalseralaglyglnargglyargval100105110glualathraspargglygluleuglyaspsertyrthrvalaspphe115120125serglyargtrppheargvalprovalglnalailealaleuileglu130135140gluarggluglu145<210>18<211>1171<212>prt<213>肺炎克雷伯菌<400>18metserglylysmetlysthrmetaspglyasnalaalaalaalatrp151015ilesertyralaphethrgluvalalaalailetyrproilethrpro202530serthrprometalagluasnvalaspglutrpalaalaglnglylys354045lysasnleupheglyglnprovalargleumetglumetglnserglu505560alaglyalaalaglyalavalhisglyalaleuglnalaglyalaleu65707580thrthrthrtyrthralaserglnglyleuleuleumetileproasn859095mettyrlysilealaglygluleuleuproglyvalphehisvalser100105110alaargalaleualathrasnserleuasnilepheglyasphisgln115120125aspvalmetalavalargglnthrglycysalametleualagluasn130135140asnvalglnglnvalmetaspleuseralavalalahisleualaala145150155160ilelysglyargileprophevalasnphepheaspglypheargthr165170175serhisgluileglnlysilegluvalleuglutyrgluglnleuala180185190thrleuleuaspargproalaleuaspserpheargargasnalaleu195200205hisproasphisprovalileargglythralaglnasnproaspile210215220tyrpheglngluargglualaglyasnargphetyrglnalaleupro225230235240aspilevalglusertyrmetthrglnileseralaleuthrglyarg245250255glutyrhisleupheasntyrthrglyalaalaaspalagluargval260265270ileilealametglyservalcysaspthrvalglngluvalvalasp275280285thrleuasnalaalaglyglulysvalglyleuleuservalhisleu290295300pheargpropheserleualahisphephealaglnleuprolysthr305310315320valglnargilealavalleuaspargthrlysgluproglyalagln325330335alagluproleucysleuaspvallysasnalaphetyrhishisasp340345350aspalaproleuilevalglyglyargtyralaleuglyglylysasp355360365valleuproasnaspilealaalavalpheaspasnleuasnlyspro370375380leuprometaspglyphethrleuglyilevalaspaspvalthrphe385390395400thrserleuproproargglnglnthrleualavalserhisaspgly405410415ilethralacyslysphetrpglymetglyseraspglythrvalgly420425430alaasnlysseralailelysileileglyasplysthrproleutyr435440445alaglnalatyrphesertyraspserlyslysserglyglyilethr450455460valserhisleuargpheglyaspargproileasnserprotyrleu465470475480ilehisargalaasppheilesercysserglnglnsertyrvalglu485490495argtyraspleuleuaspglyleulysproglyglythrpheleuleu500505510asncyssertrpseraspalagluleugluglnhisleuprovalgly515520525phelysargtyrleualaarggluasnilehisphetyrthrleuasn530535540alavalaspilealaarggluleuglyleuglyglyargpheasnmet545550555560leumetglnalaalaphephelysleualaalaileileaspprogln565570575thralaalaasptyrleulysglnalavalglulyssertyrglyser580585590lysglyalaalavalileglumetasnglnargalailegluleugly595600605metalaserleuhisglnvalthrileproalahistrpalathrleu610615620aspgluproalaalaglnalaseralametmetproasppheilearg625630635640aspileleuglnprometasnargglncysglyaspglnleuproval645650655seralaphevalglymetgluaspglythrpheproserglythrala660665670alatrpglulysargglyilealaleugluvalprovaltrpglnpro675680685gluglycysthrglncysasnglncysalapheilecysprohisala690695700alaileargproalaleuleuasnglyglugluhisaspalaalapro705710715720valglyleuleuserlysproalaglnglyalalysglutyrhistyr725730735hisleualaileserproleuaspcysserglycysglyasncysval740745750aspilecysproalaargglylysalaleulysmetglnserleuasp755760765serglnargglnmetalaprovaltrpasptyralaleualaleuthr770775780prolysserasnprophearglysthrthrvallysglyserglnphe785790795800gluthrproleuleuglupheserglyalacysalaglycysglyglu805810815thrprotyralaargleuilethrglnleupheglyaspargmetleu820825830ilealaasnalathrglycysserseriletrpglyalaseralapro835840845serileprotyrthrthrasnhisargglyhisglyproalatrpala850855860asnserleuphegluaspasnalaglupheglyleuglymetmetleu865870875880glyglyglnalavalargglnglnilealaaspaspmetthralaala885890895leualaleuprovalseraspgluleuseraspalametargglntrp900905910leualalysglnaspgluglygluglythrarggluargalaasparg915920925leusergluargleualaalaglulysgluglyvalproleuleuglu930935940glnleutrpglnasnargasptyrphevalargargserglntrpile945950955960pheglyglyaspglytrpalatyraspileglypheglyglyleuasp965970975hisvalleualaserglygluaspvalasnileleuvalpheaspthr980985990gluvaltyrserasnthrglyglyglnserserlysserthrproval99510001005alaalailealalysphealaalaglnglylysargthrarglys101010151020lysaspleuglymetmetalametsertyrglyasnvaltyrval102510301035alaglnvalalametglyalaasplysaspglnthrleuargala104010451050ilealaglualaglualatrpproglyproserleuvalileala105510601065tyralaalacysileasnhisglyleulysalaglymetargcys107010751080serglnargglualalysargalavalglualaglytyrtrphis108510901095leutrpargtyrhisproglnargglualagluglylysthrpro110011051110phemetleuaspserglugluproglugluserpheargaspphe111511201125leuleuglygluvalargtyralaserleuhislysthrthrpro113011351140hisleualaaspalaleupheserargthrglugluaspalaarg114511501155alaargphealaglntyrargargleualaglygluglu116011651170<210>19<211>266<212>prt<213>肺炎克雷伯菌<400>19metasnprotrpglnargphealaargglnargleualaargserarg151015trpasnargaspproalaalaleuaspproalaaspthrproalaphe202530gluglnalatrpglnargglncyshismetgluglnthrilevalala354045argvalprogluglyaspileproalaalaleuleugluasnileala505560alaserleualailetrpleuaspgluglyaspphealaproproglu65707580argalaalailevalarghishisalaargleugluleualapheala859095aspilealaargglnalaproglnproaspleuserthrvalglnala100105110trptyrleuarghisglnthrglnphemetargprogluglnargleu115120125thrarghisleuleuleuthrvalaspasnaspargglualavalhis130135140glnargileleuglyleutyrargglnileasnalaserargaspala145150155160phealaproleualaglnarghisserhiscysproseralaleuglu165170175gluglyargleuglytrpileserargglyleuleutyrproglnleu180185190gluthralaleupheserleualagluasnalaleuserleuproile195200205alasergluleuglytrphisleuleutrpcysglualaileargpro210215220alaalaprometgluproglnglnalaleugluseralaargasptyr225230235240leutrpglnglnserglnglnarghisglnargglntrpleuglugln245250255metileserargglnproglyleucysgly260265<210>20<211>381<212>prt<213>肺炎克雷伯菌<400>20metgluargvalleuileasnaspthrthrleuargaspglyglugln151015serproglyvalalapheargthrserglulysvalalailealaglu202530alaleutyralaalaglyilethralametgluvalglythrproala354045metglyaspglugluilealaargileglnleuvalargargglnleu505560proaspalathrleumetthrtrpcysargmetasnalaleugluile65707580argglnseralaaspleuglyileasptrpvalaspileserilepro859095alaserasplysleuargglntyrlysleuarggluproleualaval100105110leuleugluargleualametpheilehisleualahisthrleugly115120125leulysvalcysileglycysgluaspalaserargalaserglygln130135140thrleuargalailealagluvalalaglnasnalaproalaalaarg145150155160leuargtyralaaspthrvalglyleuleuaspprophethrthrala165170175alaglnileseralaleuargaspvaltrpserglygluileglumet180185190hisalahisasnaspleuglymetalathralaasnthrleualaala195200205valseralaglyalathrservalasnthrthrvalleuglyleugly210215220gluargalaglyasnalaalaalatrplysproseralaleuglyleu225230235240gluargcysleuglyvalgluthrglyvalhispheseralaleupro245250255alaleucysglnargvalalaglualaalaglnargalaileasppro260265270glnglnproleuvalglygluleuvalphethrhisgluserglyval275280285hisvalalaalaleuleuargaspserglusertyrglnserileala290295300proserleumetglyargsertyrargleuvalleuglylyshisser305310315320glyargglnalavalasnglyvalpheaspglnmetglytyrhisleu325330335asnalaalaglnileasnglnleuleuproalaileargargpheala340345350gluasntrplysargserprolysasptyrgluleuvalalailetyr355360365aspgluleucysglygluseralaleuargalaarggly370375380<210>21<211>17<212>prt<213>人工序列<220><223>c端延伸<400>21alaglyglyglyglyglytyrprotyraspvalproasptyralapro151015gly<210>22<211>12<212>prt<213>人工序列<220><223>c端延伸<400>22alaglyasptyrlysaspaspaspasplysprogly1510<210>23<211>343<212>prt<213>人工序列<220><223>融合多肽<400>23metglyleuserleuleuargglnserileargphephelysproala151015thrargthrleucysserserargtyrleuleugluglnlysprogly202530alaprothrmetargglncysalailetyrglylysglyglyilegly354045lysserthrthrthrglnasnleuvalalaalaleualaglumetgly505560lyslysvalmetilevalglycysaspprolysalaaspserthrarg65707580leuileleuhisalalysalaglnasnthrilemetglumetalaala859095gluvalglyservalgluaspleugluleugluaspvalleuglnile100105110glytyrglyaspvalargcysalagluserglyglyprogluprogly115120125valglycysalaglyargglyvalilethralaileasnpheleuglu130135140glugluglyalatyrgluaspaspleuaspphevalphetyraspval145150155160leuglyaspvalvalcysglyglyphealametproilearggluasn165170175lysalaglngluiletyrilevalcysserglyglumetmetalamet180185190tyralaalaasnasnileserlysglyilevallystyralalysser195200205glylysvalargleuglyglyleuilecysasnserargglnthrasp210215220arggluaspgluleuileilealaleualaglulysleuglythrgln225230235240metilehisphevalproargaspasnilevalglnargalagluile245250255argargmetthrvalileglutyraspproalacyslysglnalaasn260265270glutyrargthrleualaglnlysilevalasnasnthrmetlysval275280285valprothrprocysthrmetaspgluleugluserleuleumetglu290295300pheglyilemetgluglugluaspthrserileileglylysthrala305310315320alaglugluasnalaalaalaglyglyglyglyglytyrprotyrasp325330335valproasptyralaprogly340<210>24<211>527<212>prt<213>人工序列<220><223>融合多肽<400>24metglyleuserleuleuargglnserileargphephelysproala151015thrargthrleucysserserargtyrleuleugluglnlysprogly202530alaprothrasnalathrglygluargasnleualaleuileglnglu354045valleugluvalpheprogluthralaarglysgluargarglyshis505560metmetvalseraspprolysmetlysservalglylyscysileile65707580serasnarglysserglnproglyvalmetthrvalargglycysala859095tyralaglyserlysglyvalvalpheglyproilelysaspmetala100105110hisileserhisglyproalaglycysglyglntyrserargalaglu115120125argargasntyrtyrthrglyvalserglyvalaspserpheglythr130135140leuasnphethrserasppheglngluargaspilevalpheglygly145150155160asplyslysleuserlysleuilegluglumetgluleuleuphepro165170175leuthrlysglyilethrileglnserglucysprovalglyleuile180185190glyaspaspileseralavalalaasnalaserserlysalaleuasp195200205lysprovalileprovalargcysgluglypheargglyvalsergln210215220serleuglyhishisilealaasnaspvalvalargasptrpileleu225230235240asnasnarggluglyglnprophegluthrthrprotyraspvalala245250255ileileglyasptyrasnileglyglyaspalatrpalaserargile260265270leuleugluglumetglyleuargvalvalalaglntrpserglyasp275280285glythrleuvalglumetgluasnthrprophevallysleuasnleu290295300valhiscystyrargsermetasntyrilealaarghismetgluglu305310315320lyshisglnileprotrpmetglutyrasnphepheglyprothrlys325330335ilealagluserleuarglysilealaaspglnpheaspaspthrile340345350argalaasnalaglualavalilealaargtyrgluglyglnmetala355360365alaileilealalystyrargproargleugluglyarglysvalleu370375380leutyrileglyglyleuargproarghisvalileglyalatyrglu385390395400aspleuglymetgluileilealaalaglytyrgluphealahisasn405410415aspasptyraspargthrleuproaspleulysgluglythrleuleu420425430pheaspaspalasersertyrgluleuglualaphevallysalaleu435440445lysproaspleuileglyserglyilelysglulystyrilephegln450455460lysmetglyvalpropheargglnmethissertrpasptyrsergly465470475480protyrhisglytyraspglyphealailephealaargaspmetasp485490495metthrleuasnasnproalatrpasngluleuthralaprotrpleu500505510lysseralaalaglyasptyrlysaspaspaspasplysprogly515520525<210>25<211>570<212>prt<213>人工序列<220><223>融合多肽<400>25metglyleuserleuleuargglnserileargphephelysproala151015thrargthrleucysserserargtyrleuleugluglnlysprogly202530alaproserglnthrileasplysileasnsercystyrproleuphe354045gluglnaspglutyrglngluleupheargasnlysargglnleuglu505560glualahisaspalaglnargvalglngluvalphealatrpthrthr65707580thralaglutyrglualaleuasnpheargargglualaleuthrval859095aspproalalysalacysglnproleuglyalavalleucysserleu100105110glyphealaasnthrleuprotyrvalhisglyserglnglycysval115120125alatyrpheargthrtyrpheasnarghisphelysgluproileala130135140cysvalseraspsermetthrgluaspalaalavalpheglyglyasn145150155160asnasnmetasnleuglyleuglnasnalaseralaleutyrlyspro165170175gluileilealavalserthrthrcysmetalagluvalileglyasp180185190aspleuglnalapheilealaasnalalyslysaspglyphevalasp195200205serserilealavalprohisalahisthrproserpheileglyser210215220hisvalthrglytrpaspasnmetphegluglyphealalysthrphe225230235240thralaasptyrglnglyglnproglylysleuprolysleuasnleu245250255valthrglyphegluthrtyrleuglyasnpheargvalleulysarg260265270metmetgluglnmetalavalprocysserleuleuseraspproser275280285gluvalleuaspthrproalaaspglyhistyrargmettyrsergly290295300glythrthrglnglnglumetlysglualaproaspalaileaspthr305310315320leuleuleuglnprotrpglnleuleulysserlyslysvalvalgln325330335glumettrpasnglnproalathrgluvalalaileproleuglyleu340345350alaalathraspgluleuleumetthrvalserglnleuserglylys355360365proilealaaspalaleuthrleugluargglyargleuvalaspmet370375380metleuaspserhisthrtrpleuhisglylyslyspheglyleutyr385390395400glyaspproaspphevalmetglyleuthrargpheleuleugluleu405410415glycysgluprothrvalileleuserhisasnalaasnlysargtrp420425430glnlysalametasnlysmetleuaspalaserprotyrglyargasp435440445sergluvalpheileasncysaspleutrphispheargserleumet450455460phethrargglnproaspphemetileglyasnsertyrglylysphe465470475480ileglnargaspthrleualalysglylysalaphegluvalproleu485490495ileargleuglypheproleupheasparghishisleuhisarggln500505510thrthrtrpglytyrgluglyalametasnilevalthrthrleuval515520525asnalavalleuglulysleuaspseraspthrserglnleuglylys530535540thrasptyrserpheaspleuvalargalaglyglyglyglyglytyr545550555560protyraspvalproasptyralaprogly565570<210>26<211>270<212>prt<213>人工序列<220><223>融合多肽<400>26metglyleuserleuleuargglnserileargphephelysproala151015thrargthrleucysserserargtyrleuleugluglnlysprogly202530alaproseraspasnaspthrleuphetrpargmetleualaleuphe354045glnserleuproaspleuglnproalaglnilevalasptrpleuala505560glngluserglygluthrleuthrprogluargleualathrleuthr65707580glnproglnleualaalaserpheproseralathralavalmetser859095proalaargtrpserargvalmetalaserleuglnglyalaleupro100105110alahisleuargilevalargproalaglnargthrproglnleuleu115120125alaalaphecysserglnaspglyleuvalileasnglyhisphegly130135140glnglyargleuphepheiletyralapheaspgluglnglyglytrp145150155160leutyraspleuargargtyrproseralaprohisglnglngluala165170175asngluvalargalaargleuilegluaspcysglnleuleuphecys180185190glngluileglyglyproalaalaalaargproilearghisargile195200205hisprometlysalaglnproglythrthrileglnalaglncysglu210215220alaileasnthrleuleualaglyargleuproprotrpleualalys225230235240argleuasnargaspasnproleuglugluargvalphealaglygly245250255glyglyglytyrprotyraspvalproasptyralaprogly260265270<210>27<211>950<212>dna<213>人工序列<220><223>asci片段<400>27ggcgcgccaactatgagacaatgtgctatctatggaaagggtggaatcggaaagtctact60actactcagaaccttgttgctgctcttgctgagatgggaaagaaagttatgatcgttgga120tgcgatcctaaggctgattctactagacttatcctccatgctaaggctcagaacaccatt180atggaaatggctgctgaggttggatctgttgaagatcttgagcttgaggatgttctccaa240atcggatacggtgatgttagatgtgctgaatctggtggacctgaacctggtgttggatgt300gctggaagaggtgttattaccgctatcaacttccttgaagaagagggagcttacgaggat360gatctcgatttcgttttctacgatgtgctcggagatgttgtttgtggtggatttgctatg420cctatcagagagaacaaggctcaagagatctacatcgtttgctctggtgagatgatggct480atgtatgctgctaacaacatctctaagggaatcgtgaagtacgctaagtctggaaaggtt540agacttggaggactcatctgtaactctagacagactgatagagaggatgagcttattatc600gctttggctgagaagttgggaactcagatgatccatttcgtgccaagagataacatcgtt660cagagagctgagatcagaaggatgactgttatcgaatacgatcctgcttgcaaacaagct720aacgagtatagaactctcgctcagaagatcgttaacaacactatgaaggtggtgcctact780ccttgtactatggatgaacttgagtctctcctcatggaatttggaatcatggaagaagag840gatacctctatcatcggaaagactgctgctgaagaaaacgctgctgccggcggtggaggt900ggatacccttacgacgttcctgattacgctcccgggtgatgaggcgcgcc950<210>28<211>1502<212>dna<213>人工序列<220><223>asci片段<400>28ggcgcgccaactaatgctactggtgaaagaaacctcgctctcatccaagaggttttggaa60gtttttcctgagactgctaggaaagaaaggcgtaagcacatgatggtgtctgatcctaag120atgaagtctgtgggaaagtgcatcatctctaacagaaagtctcagcctggtgtgatgact180gttagaggatgtgcttatgctggatctaagggtgttgttttcggacctatcaaggatatg240gctcatatctctcatggacctgctggatgtggacaatattctagagctgagcgtaggaac300tactacactggtgtttctggtgtggattctttcggaactctcaacttcacctctgatttc360caagagcgtgatatcgttttcggaggtgataagaagctctctaagttgatcgaagagatg420gaactcttgttcccactcactaagggaatcactatccaatctgagtgccctgttggactt480atcggagatgatatttctgctgtggctaacgcttcttctaaggctcttgataagcctgtt540atccctgttagatgtgaaggattcaggggagtttctcagtctcttggacatcatatcgct600aacgatgtggtgagagattggatccttaacaacagagagggacaacctttcgagactact660ccttacgatgttgctatcatcggagactacaacattggaggtgatgcttgggcttctaga720atccttcttgaagagatgggactcagagttgttgctcaatggtctggtgatggtactctt780gttgagatggaaaacacccctttcgttaagctcaacctcgttcattgctaccgtagcatg840aactacattgctagacacatggaagagaagcaccaaattccttggatggagtacaacttc900ttcggacctactaagatcgctgagtctcttagaaagatcgctgatcagttcgatgatacc960atcagagctaatgctgaggctgttattgctagatacgagggacaaatggctgctattatc1020gctaagtacagacctagactcgagggaagaaaggttttgctttacatcggaggactcaga1080cctagacatgttattggagcttacgaggatctcggaatggaaattattgctgctggatac1140gagttcgctcacaacgatgattacgatagaactctccctgacctcaaagagggaactctt1200cttttcgatgacgcttcttcatacgagcttgaggcttttgttaaggctcttaagcctgat1260ctcatcggatctggaatcaaagagaagtacatcttccagaagatgggagttcctttcaga1320cagatgcactcttgggattattctggaccttaccatggatacgacggatttgctatcttc1380gctagggatatggatatgactctcaacaatcctgcttggaacgaacttactgctccttgg1440cttaagtctgctgccggcgactacaaagacgatgatgacaaacccgggtgatgaggcgcg1500cc1502<210>29<211>1631<212>dna<213>人工序列<220><223>asci片段<400>29ggcgcgccatctcaaactattgataagatcaacagctgctacccactctttgagcaagat60gaataccaagagcttttccgtaacaagagacagcttgaagaggctcatgatgctcagaga120gttcaagaagttttcgcttggactactactgctgaatacgaggctctcaactttagaaga180gaggctcttactgttgaccctgctaaggcttgtcaacctcttggagctgttctttgttct240cttggattcgctaacacccttccttatgttcatggatctcagggatgtgtggcttacttc300agaacctacttcaacaggcacttcaaagaacctatcgcttgcgttagcgattctatgact360gaagatgctgctgttttcggaggtaacaacaacatgaaccttggacttcagaacgcttct420gctctttacaagcctgagattatcgctgtgtctactacttgcatggctgaagttatcgga480gatgatctccaggctttcattgctaacgctaagaaggatggattcgtcgattcttctatc540gctgttcctcatgctcacactccatctttcatcggatctcatgttaccggatgggataat600atgttcgagggattcgctaagaccttcactgctgattatcaaggacagcctggaaagttg660cctaagttgaatctcgttaccggattcgagacttacctcggaaacttcagagtgctcaag720agaatgatggaacagatggctgtgccttgctctcttctttctgatccttctgaagtgctc780gatactcctgctgatggacattataggatgtactctggtggaactacccagcaagaaatg840aaggaagctcctgacgctatcgatactcttcttcttcaaccttggcaactcctcaagtct900aagaaagttgtgcaagagatgtggaaccagcctgctactgaagttgctattcctcttgga960cttgctgctactgatgagcttctcatgactgtttctcagctttctggaaagcctatcgct1020gatgctcttactcttgagagaggtagactcgttgatatgatgctcgattctcatacttgg1080ctccacggaaagaagtttggactttacggagatcctgacttcgttatgggacttactaga1140ttcttgctcgagcttggatgtgagcctactgttatcctttctcacaacgctaacaagagg1200tggcaaaaggctatgaacaagatgcttgatgcttctccatacggaagggattctgaggtt1260ttcatcaactgcgatctctggcatttccgttctctcatgttcactagacagcctgatttc1320atgatcggaaacagctacggaaagttcatccagagagatactctcgctaagggaaaggct1380ttcgaagttcctcttatcagacttggattcccactcttcgatagacatcatctccacaga1440caaactacttggggatatgaaggtgctatgaacatcgttactaccctcgttaacgctgtt1500cttgagaagttggattctgatacttctcagctcggaaagaccgattactctttcgatctc1560gtgagagccggcggtggaggtggatacccttacgacgttcctgattacgctcccgggtga1620tgaggcgcgcc1631<210>30<211>731<212>dna<213>人工序列<220><223>asci片段<400>30ggcgcgccatctgataatgatacacttttttggaggatgctcgctcttttccagtctctt60cctgatcttcaacctgctcaaatcgttgattggcttgctcaagaatctggtgagactctt120actcctgagagacttgctactcttactcaacctcaactcgctgcttcttttccttctgct180actgctgttatgtctcctgctagatggtctagagttatggcttctcttcagggtgctctc240ccagctcatcttagaatcgttagacctgctcaaagaactcctcaacttcttgctgctttc300tgttctcaggatggacttgttatcaacggacatttcggacagggtagactctttttcatc360tacgcttttgatgagcagggaggatggctttacgatcttagaagatatccttctgctcct420catcagcaagaggctaatgaagttagagctagacttatcgaggattgccagcttttgttc480tgtcaagaaattggaggacctgctgctgctagacctattagacatagaatccaccctatg540aaggctcaacctggaactactatccaagctcaatgtgaggctatcaacactcttcttgct600ggtagacttcctccttggcttgctaaaagactcaacagagataacccgctcgaagagagg660gttttcgccggcggtggaggtggatacccttacgacgttcctgattacgctcccgggtga720tgaggcgcgcc731<210>31<211>518<212>prt<213>人工序列<220><223>融合多肽<400>31metglyleuserleuleuargglnserileargphephelysproala151015thrargthrleucysserserargtyrleuleugluglnlysprogly202530alaprothrsercysserserpheserglyglylysalacysargpro354045alaaspaspseralaleuthrproleuvalalaasplysalaalaala505560hisprocystyrserarghisglyhishisargphealaargmethis65707580leuprovalalaproalacysasnleuglncysasntyrcysasnarg859095lyspheaspcysserasngluserargproglyvalserserthrleu100105110leuthrprogluglnalavalvallysvalargglnvalalaglnala115120125ileproglnleuservalvalglyilealaglyproglyaspproleu130135140alaasnilealaargthrpheargthrleugluleuileargglugln145150155160leuproaspleulysleucysleuserthrasnglyleuvalleupro165170175aspalavalaspargleuleuaspvalglyvalasphisvalthrval180185190thrileasnthrleuaspalagluilealaalaglniletyralatrp195200205leutrpleuaspglygluargtyrserglyargglualaglygluile210215220leuilealaargglnleugluglyvalargargleuthralalysgly225230235240valleuvallysileasnservalleuileproglyileasnaspser245250255glymetalaglyvalserargalaleuargalaserglyalapheile260265270hisasnilemetproleuilealaargprogluhisglythrvalphe275280285glyleuasnglyglnprogluproaspalagluthrleualaalathr290295300argserargcysglygluvalmetproglnmetthrhiscyshisgln305310315320cysargalaaspalaileglymetleuglygluaspargserglngln325330335phethrglnleuproalaprogluserleuproalatrpleuproile340345350leuhisglnargalaglnleuhisalaserilealathrargglyglu355360365serglualaaspaspalacysleuvalalavalalaserserarggly370375380aspvalileaspcyshispheglyhisalaaspargphetyriletyr385390395400serleuseralaalaglymetvalleuvalasngluargphethrpro405410415lystyrcysglnglyargaspaspcysgluproglnaspasnalaala420425430argphealaalaileleugluleuleualaaspvallysalavalphe435440445cysvalargileglyhisthrprotrpglnglnleugluglnglugly450455460ilegluprocysvalaspglyalatrpargprovalsergluvalleu465470475480proalatrptrpglnglnargargglysertrpproalaalaleupro485490495hislysglyvalalaalaglyglyglyglyglytyrprotyraspval500505510proasptyralaprogly515<210>32<211>507<212>prt<213>人工序列<220><223>融合多肽<400>32metglyleuserleuleuargglnserileargphephelysproala151015thrargthrleucysserserargtyrleuleugluglnlysprogly202530alaprolysglyasngluileleualaleuleuaspgluproalacys354045gluhisasnhislysglnlysserglycysseralaprolysprogly505560alathralaalaglycysalapheaspglyalaglnilethrleuleu65707580proilealaaspvalalahisleuvalhisglyproileglycysala859095glysersertrpaspasnargglyseralaserserglyprothrleu100105110asnargleuglyphethrthraspleuasngluglnaspvalilemet115120125glyargglygluargargleuphehisalavalarghisilevalthr130135140argtyrhisproalaalavalpheiletyrasnthrcysvalproala145150155160metgluglyaspaspleuglualavalcysglnalaalaglnthrala165170175thrglyvalprovalilealaileaspalaalaglyphetyrglyser180185190lysasnleuglyasnargproalaglyaspvalmetvallysargval195200205ileglyglnarggluproalaprotrpprogluserthrleupheala210215220progluglnarghisaspileglyleuileglyglupheasnileala225230235240glygluphetrphisileglnproleuleuaspgluleuglyilearg245250255valleuglyserleuserglyaspglyargphealagluileglnthr260265270methisargalaglnalaasnmetleuvalcysserargalaleuile275280285asnvalalaargalaleugluglnargtyrglythrprotrppheglu290295300glyserphetyrglyileargalathrseraspalaleuargglnleu305310315320alaalaleuleuglyaspaspaspleuargglnargthrglualaleu325330335ilealaargglugluglnalaalagluleualaleuglnprotrparg340345350gluglnleuargglyarglysalaleuleutyrthrglyglyvallys355360365sertrpservalvalseralaleuglnaspleuglymetthrvalval370375380alathrglythrarglysserthrglugluasplysglnargilearg385390395400gluleumetglygluglualavalmetleuglugluglyasnalaarg405410415thrleuleuaspvalvaltyrargtyrglnalaaspleumetileala420425430glyglyargasnmettyrthralatyrlysalaargleupropheleu435440445aspileasnglngluarggluhisalaphealaglytyrglnglyile450455460valthrleualaargglnleucysglnthrileasnserproiletrp465470475480proglnthrhisserargalaprotrpargalaglyglyglyglygly485490495tyrprotyraspvalproasptyralaprogly500505<210>33<211>505<212>prt<213>人工序列<220><223>融合多肽<400>33metglyleuserleuleuargglnserileargphephelysproala151015thrargthrleucysserserargtyrleuleugluglnlysprogly202530alaproalaaspilepheargthrasplysproleualavalserpro354045ilelysthrglyglnproleuglyalaileleualaserleuglyile505560gluhisserileproleuvalhisglyalaglnglycysseralaphe65707580alalysvalphepheileglnhisphehisaspprovalproleugln859095serthralametaspprothrserthrilemetglyalaaspglyasn100105110ilephethralaleuaspthrleucysglnargasnasnproglnala115120125ilevalleuleuserthrglyleuserglualaglnglyseraspile130135140serargvalvalargglnphearggluglutyrproarghislysgly145150155160valalaileleuthrvalasnthrproaspphetyrglysermetglu165170175asnglypheseralavalleugluservalilegluglntrpvalpro180185190proalaproargproalaglnargasnargargvalasnleuleuval195200205serhisleucysserproglyaspileglutrpleuargargcysval210215220glualapheglyleuglnproileileleuproaspleualaglnser225230235240metaspglyhisleualaglnglyasppheserproleuthrglngly245250255glythrproleuargglnilegluglnmetglyglnserleucysser260265270phealaileglyvalserleuhisargalaserserleuleualapro275280285argcysargglygluvalilealaleuprohisleumetthrleuglu290295300argcysaspalapheilehisglnleualalysileserglyargala305310315320valproglutrpleugluargglnargglyglnleuglnaspalamet325330335ileaspcyshismettrpleuglnglyglnargmetalailealaala340345350gluglyaspleuleualaalatrpcysaspphealaasnserglngly355360365metglnproglyproleuvalalaprothrglyhisproserleuarg370375380glnleuprovalgluargvalvalproglyaspleugluaspleugln385390395400thrleuleucysalahisproalaaspleuleuvalalaasnserhis405410415alaargaspleualagluglnphealaleuproleuvalargalagly420425430pheproleupheasplysleuglyglupheargargvalargglngly435440445tyrserglymetargaspthrleuphegluleualaasnleuilearg450455460gluarghishishisleualahistyrargserproleuargglnasn465470475480progluserserleuserthrglyglyalatyralaalaalaglyasp485490495tyrlysaspaspaspasplysprogly500505<210>34<211>217<212>prt<213>人工序列<220><223>融合多肽<400>34metglyleuserleuleuargglnserileargphephelysproala151015thrargthrleucysserserargtyrleuleugluglnlysprogly202530alaproproproleuasptrpleuargargleutrpleuleutyrhis354045alaglylysglyserpheproleuargmetglyleuserproargasp505560trpglnalaleuargargargleuglygluvalgluthrproleuasp65707580glygluthrleuthrargargargleumetalagluleuasnalathr859095arggluglugluargglnglnleuglyalatrpleualaglytrpmet100105110glnglnaspalaglyprometalaglnileilealagluvalserleu115120125alapheasnhisleutrpglnaspleuglyleualaserargalaglu130135140leuargleuleumetseraspcyspheproglnleuvalvalmetasn145150155160gluhisasnmetargtrplyslysphephetyrargglnargcysleu165170175leuglnglnglygluvalilecysargserprosercysaspglucys180185190trpgluargseralacyspheglualaglyglyglyglyglytyrpro195200205tyraspvalproasptyralaprogly210215<210>35<211>450<212>prt<213>人工序列<220><223>融合多肽<400>35metglyleuserleuleuargglnserileargphephelysproala151015thrargthrleucysserserargtyrleuleugluglnlysprogly202530alaprolysglnvaltyrleuaspasnasnalathrthrargleuasp354045prometvalleuglualametmetpropheleuthraspphetyrgly505560asnproserserilehisasppheglyileproalaglnalaalaleu65707580gluargalahisglnglnalaalaalaleuleuglyalaglutyrpro859095sergluileilephethrsercysalathrglualathralathrala100105110ilealaseralailealaleuleuprogluargarggluileilethr115120125servalvalgluhisproalathrleualaalacysgluhismetglu130135140arggluglytyrargilehisargilealavalaspglygluglyala145150155160leuaspmetalaglnpheargalaalaleuserproargvalalaleu165170175valservalmettrpalaasnasngluthrglyvalleupheproile180185190glyglumetalagluleualahisgluglnglyalaleuphehiscys195200205aspalavalglnvalvalglylysileproilealavalglyglnthr210215220argileaspmetleusercysseralahislysphehisglyprolys225230235240glyvalglycysleutyrleuargargglythrargpheargproleu245250255leuargglyglyhisglnglutyrglyargargalaglythrgluasn260265270ilecysglyilevalglymetglyalaalacysgluleualaasnile275280285hisleuproglymetthrhisileglyglnleuargasnargleuglu290295300hisargleuleualaservalproservalmetvalmetglyglygly305310315320glnproalavalproglythrvalasnleualapheglupheileglu325330335glyglualaileleuleuleuleuasnglnalaglyilealaalaser340345350serglyseralacysthrserglyserleugluproserhisvalmet355360365argalametasnileprotyrthralaalahisglythrileargphe370375380serleuserargtyrthrargglulysgluileasptyrvalvalala385390395400thrleuproproileileaspargleuargalaleuserprotyrtrp405410415glnasnglylysproargproalaaspalavalphethrprovaltyr420425430glyalaglyglyglyglyglytyrprotyraspvalproasptyrala435440445progly450<210>36<211>319<212>prt<213>人工序列<220><223>融合多肽<400>36metglyleuserleuleuargglnserileargphephelysproala151015thrargthrleucysserserargtyrleuleugluglnlysprogly202530alaprotrpasntyrserglulysvallysasphisphepheasnpro354045argasnalaargvalvalaspasnalaasnalavalglyaspvalgly505560serleusercysglyaspalaleuargleumetleuargvalasppro65707580glnsergluileilegluglualaglypheglnthrpheglycysgly859095seralailealaserserseralaleuthrgluleuileileglyhis100105110thrleualaglualaglyglnilethrasnglnglnilealaasptyr115120125leuaspglyleuproproglulysmethiscysservalmetglygln130135140glualaleuargalaalailealaasnpheargglygluserleuglu145150155160glugluhisaspgluglylysleuilecyslyscyspheglyvalasp165170175gluglyhisileargargalavalglnasnasnglyleuthrthrleu180185190alagluvalileasntyrthrlysalaglyglyglycysthrsercys195200205hisglulysilegluleualaleualagluileleualaglnglnpro210215220glnthrthrproalavalalaserglylysaspprohistrpglnser225230235240valvalaspthrilealagluleuargprohisileglnalaaspgly245250255glyaspmetalaleuleuservalthrasnhisglnvalthrvalser260265270leuserglysercysserglycysmetmetthraspmetthrleuala275280285trpleuglnglnlysleumetgluargthrglycystyrmetgluval290295300valalaalaalaglyasptyrlysaspaspaspasplysprogly305310315<210>37<211>201<212>prt<213>人工序列<220><223>融合多肽<400>37metglyleuserleuleuargglnserileargphephelysproala151015thrargthrleucysserserargtyrleuleugluglnlysprogly202530alaproproproileasnargglnpheaspmetvalhisseraspglu354045trpsermetlysvalalaphealaserserasptyrarghisvalasp505560glnhispheglyalathrproargleuvalvaltyrglyvallysala65707580aspargvalthrleuileargvalvalasppheservalgluasngly859095hisglnthrglulysilealaargargilehisalaleugluaspcys100105110valthrleuphecysvalalaileglyaspalavalpheargglnleu115120125leuglnvalglyvalargalagluargvalproalaaspthrthrile130135140valglyleuleuglngluileglnleutyrtrptyrasplysglygln145150155160arglysasnglnargglnargaspprogluargphethrargleuleu165170175glngluglnglutrphisglyaspproaspproargargalaglyasp180185190tyrlysaspaspaspasplysprogly195200<210>38<211>80<212>prt<213>人工序列<220><223>n端延伸<400>38metalametalavalpheargarggluglyargargleuleuproser151015ilealaalaargproilealaalaileargserproleuserserasp202530glnglugluglyleuleuglyvalargserileserthrglnvalval354045argasnargmetlysservallysasnileglnlysilethrlysala505560metlysmetvalalaalaserlysleuargalavalglnglyalapro65707580<210>39<211>80<212>prt<213>人工序列<220><223>n端延伸<400>39metalametalavalpheargarggluglyargalaalaalaalaala151015alaalaalaargproilealaalaalaalaalaalaalaalaalaala202530alaglugluglyleuleualaalaalaalaalaalaalaglnvalval354045argasnargmetlysservallysasnileglnlysilethrlysala505560metlysmetvalalaalaserlysleuargalavalglnglyalapro65707580<210>40<211>267<212>prt<213>人工序列<220><223>融合多肽<400>40metalametalavalpheargarggluglyargargleuleuproser151015ilealaalaargproilealaalaileargserproleuserserasp202530glnglugluglyleuleuglyvalargserileserthrglnvalval354045argasnargmetlysservallysasnileglnlysilethrlysala505560metlysmetvalalaalaserlysleuargalavalglnglyalapro65707580metalaasnileglyilephepheglythraspthrglylysthrarg859095lysilealalysmetilehislysglnleuglygluleualaaspala100105110provalasnileasnargthrthrleuaspaspphemetalatyrpro115120125valleuleuleuglythrprothrleuglyaspglyglnleuprogly130135140leuglualaglycysgluserglusertrpserglupheilesergly145150155160leuaspaspalaserleulysglylysthrvalalaleupheglyleu165170175glyaspglnargglytyrproaspasnphevalserglymetargpro180185190leupheaspalaleuseralaargglyalaglnmetileglysertrp195200205proasngluglytyrglupheseralaserseralaleugluglyasp210215220argphevalglyleuvalleuaspglnaspasnglnpheaspglnthr225230235240glualaargleualasertrpleuglugluilelysargthrvalleu245250255tyrprotyraspvalproasptyralaprogly260265<210>41<211>238<212>prt<213>人工序列<220><223>融合多肽<400>41metalametalavalpheargarggluglyargargleuleuproser151015ilealaalaargproilealaalaileargserproleuserserasp202530glnglugluglyleuleuglyvalargserileserthrglnvalval354045argasnargmetlysservallysasnileglnlysilethrlysala505560metlysmetvalalaalaserlysleuargalavalglnglyalapro65707580metargprolysphethrpheserglugluvalargvalvalargala859095ileargasnaspglythrvalalaglyphealaproglyalaleuleu100105110valargargglyserthrglyphevalargasptrpglyvalpheleu115120125glnaspglnileiletyrglnilehispheprogluthraspargile130135140ileglycysarggluglngluleuileproilethrglnprotrpleu145150155160alaglyasnleuglntyrargaspservalthrcysglnmetalaleu165170175alavalasnglyaspvalvalvalseralaglyglnargglyargval180185190glualathraspargglygluleuglyaspsertyrthrvalaspphe195200205serglyargtrppheargvalprovalglnalailealaleuileglu210215220gluargglugluasptyrlysaspaspaspasplysprogly225230235<210>42<211>389<212>prt<213>人工序列<220><223>融合多肽<400>42metalametalavalpheargarggluglyargargleuleuproser151015ilealaalaargproilealaalaileargserproleuserserasp202530glnglugluglyleuleuglyvalargserileserthrglnvalval354045argasnargmetlysservallysasnileglnlysilethrlysala505560metlysmetvalalaalaserlysleuargalavalglnglyalapro65707580thrmetargglncysalailetyrglylysglyglyileglylysser859095thrthrthrglnasnleuvalalaalaleualaglumetglylyslys100105110valmetilevalglycysaspprolysalaaspserthrargleuile115120125leuhisalalysalaglnasnthrilemetglumetalaalagluval130135140glyservalgluaspleugluleugluaspvalleuglnileglytyr145150155160glyaspvalargcysalagluserglyglyprogluproglyvalgly165170175cysalaglyargglyvalilethralaileasnpheleuglugluglu180185190glyalatyrgluaspaspleuaspphevalphetyraspvalleugly195200205aspvalvalcysglyglyphealametproilearggluasnlysala210215220glngluiletyrilevalcysserglyglumetmetalamettyrala225230235240alaasnasnileserlysglyilevallystyralalysserglylys245250255valargleuglyglyleuilecysasnserargglnthraspargglu260265270aspgluleuileilealaleualaglulysleuglythrglnmetile275280285hisphevalproargaspasnilevalglnargalagluileargarg290295300metthrvalileglutyraspproalacyslysglnalaasnglutyr305310315320argthrleualaglnlysilevalasnasnthrmetlysvalvalpro325330335thrprocysthrmetaspgluleugluserleuleumetgluphegly340345350ilemetgluglugluaspthrserileileglylysthralaalaglu355360365gluasnalaalaalaglyglyglyglyglytyrprotyraspvalpro370375380asptyralaprogly385<210>43<211>8<212>prt<213>人工序列<220><223>胰蛋白酶消化肽段<400>43serileserthrglnvalvalarg15<210>44<211>7<212>prt<213>人工序列<220><223>胰蛋白酶消化肽段<400>44ileserthrglnvalvalarg15<210>45<211>9<212>prt<213>人工序列<220><223>胰蛋白酶消化肽段<400>45alavalglnglyalaprothrmetarg15<210>46<211>564<212>prt<213>人工序列<220><223>融合多肽<400>46metalametalavalpheargarggluglyargargleuleuproser151015ilealaalaargproilealaalaileargserproleuserserasp202530glnglugluglyleuleuglyvalargserileserthrglnvalval354045argasnargmetlysservallysasnileglnlysilethrlysala505560metlysmetvalalaalaserlysleuargalavalglnglyalapro65707580thrsercysserserpheserglyglylysalacysargproalaasp859095aspseralaleuthrproleuvalalaasplysalaalaalahispro100105110cystyrserarghisglyhishisargphealaargmethisleupro115120125valalaproalacysasnleuglncysasntyrcysasnarglysphe130135140aspcysserasngluserargproglyvalserserthrleuleuthr145150155160progluglnalavalvallysvalargglnvalalaglnalailepro165170175glnleuservalvalglyilealaglyproglyaspproleualaasn180185190ilealaargthrpheargthrleugluleuilearggluglnleupro195200205aspleulysleucysleuserthrasnglyleuvalleuproaspala210215220valaspargleuleuaspvalglyvalasphisvalthrvalthrile225230235240asnthrleuaspalagluilealaalaglniletyralatrpleutrp245250255leuaspglygluargtyrserglyargglualaglygluileleuile260265270alaargglnleugluglyvalargargleuthralalysglyvalleu275280285vallysileasnservalleuileproglyileasnaspserglymet290295300alaglyvalserargalaleuargalaserglyalapheilehisasn305310315320ilemetproleuilealaargprogluhisglythrvalpheglyleu325330335asnglyglnprogluproaspalagluthrleualaalathrargser340345350argcysglygluvalmetproglnmetthrhiscyshisglncysarg355360365alaaspalaileglymetleuglygluaspargserglnglnphethr370375380glnleuproalaprogluserleuproalatrpleuproileleuhis385390395400glnargalaglnleuhisalaserilealathrargglygluserglu405410415alaaspaspalacysleuvalalavalalaserserargglyaspval420425430ileaspcyshispheglyhisalaaspargphetyriletyrserleu435440445seralaalaglymetvalleuvalasngluargphethrprolystyr450455460cysglnglyargaspaspcysgluproglnaspasnalaalaargphe465470475480alaalaileleugluleuleualaaspvallysalavalphecysval485490495argileglyhisthrprotrpglnglnleugluglngluglyileglu500505510procysvalaspglyalatrpargprovalsergluvalleuproala515520525trptrpglnglnargargglysertrpproalaalaleuprohislys530535540glyvalalaalaglyglyglyglyglytyrprotyraspvalproasp545550555560tyralaprogly<210>47<211>573<212>prt<213>人工序列<220><223>融合多肽<400>47metalametalavalpheargarggluglyargargleuleuproser151015ilealaalaargproilealaalaileargserproleuserserasp202530glnglugluglyleuleuglyvalargserileserthrglnvalval354045argasnargmetlysservallysasnileglnlysilethrlysala505560metlysmetvalalaalaserlysleuargalavalglnglyalapro65707580thrasnalathrglygluargasnleualaleuileglngluvalleu859095gluvalpheprogluthralaarglysgluargarglyshismetmet100105110valseraspprolysmetlysservalglylyscysileileserasn115120125arglysserglnproglyvalmetthrvalargglycysalatyrala130135140glyserlysglyvalvalpheglyproilelysaspmetalahisile145150155160serhisglyproalaglycysglyglntyrserargalagluargarg165170175asntyrtyrthrglyvalserglyvalaspserpheglythrleuasn180185190phethrserasppheglngluargaspilevalpheglyglyasplys195200205lysleuserlysleuilegluglumetgluleuleupheproleuthr210215220lysglyilethrileglnserglucysprovalglyleuileglyasp225230235240aspileseralavalalaasnalaserserlysalaleuasplyspro245250255valileprovalargcysgluglypheargglyvalserglnserleu260265270glyhishisilealaasnaspvalvalargasptrpileleuasnasn275280285arggluglyglnprophegluthrthrprotyraspvalalaileile290295300glyasptyrasnileglyglyaspalatrpalaserargileleuleu305310315320gluglumetglyleuargvalvalalaglntrpserglyaspglythr325330335leuvalglumetgluasnthrprophevallysleuasnleuvalhis340345350cystyrargsermetasntyrilealaarghismetgluglulyshis355360365glnileprotrpmetglutyrasnphepheglyprothrlysileala370375380gluserleuarglysilealaaspglnpheaspaspthrileargala385390395400asnalaglualavalilealaargtyrgluglyglnmetalaalaile405410415ilealalystyrargproargleugluglyarglysvalleuleutyr420425430ileglyglyleuargproarghisvalileglyalatyrgluaspleu435440445glymetgluileilealaalaglytyrgluphealahisasnaspasp450455460tyraspargthrleuproaspleulysgluglythrleuleupheasp465470475480aspalasersertyrgluleuglualaphevallysalaleulyspro485490495aspleuileglyserglyilelysglulystyrilepheglnlysmet500505510glyvalpropheargglnmethissertrpasptyrserglyprotyr515520525hisglytyraspglyphealailephealaargaspmetaspmetthr530535540leuasnasnproalatrpasngluleuthralaprotrpleulysser545550555560alaalaglyasptyrlysaspaspaspasplysprogly565570<210>48<211>553<212>prt<213>人工序列<220><223>融合多肽<400>48metalametalavalpheargarggluglyargargleuleuproser151015ilealaalaargproilealaalaileargserproleuserserasp202530glnglugluglyleuleuglyvalargserileserthrglnvalval354045argasnargmetlysservallysasnileglnlysilethrlysala505560metlysmetvalalaalaserlysleuargalavalglnglyalapro65707580lysglyasngluileleualaleuleuaspgluproalacysgluhis859095asnhislysglnlysserglycysseralaprolysproglyalathr100105110alaalaglycysalapheaspglyalaglnilethrleuleuproile115120125alaaspvalalahisleuvalhisglyproileglycysalaglyser130135140sertrpaspasnargglyseralaserserglyprothrleuasnarg145150155160leuglyphethrthraspleuasngluglnaspvalilemetglyarg165170175glygluargargleuphehisalavalarghisilevalthrargtyr180185190hisproalaalavalpheiletyrasnthrcysvalproalametglu195200205glyaspaspleuglualavalcysglnalaalaglnthralathrgly210215220valprovalilealaileaspalaalaglyphetyrglyserlysasn225230235240leuglyasnargproalaglyaspvalmetvallysargvalilegly245250255glnarggluproalaprotrpprogluserthrleuphealaproglu260265270glnarghisaspileglyleuileglyglupheasnilealaglyglu275280285phetrphisileglnproleuleuaspgluleuglyileargvalleu290295300glyserleuserglyaspglyargphealagluileglnthrmethis305310315320argalaglnalaasnmetleuvalcysserargalaleuileasnval325330335alaargalaleugluglnargtyrglythrprotrpphegluglyser340345350phetyrglyileargalathrseraspalaleuargglnleualaala355360365leuleuglyaspaspaspleuargglnargthrglualaleuileala370375380argglugluglnalaalagluleualaleuglnprotrpargglugln385390395400leuargglyarglysalaleuleutyrthrglyglyvallyssertrp405410415servalvalseralaleuglnaspleuglymetthrvalvalalathr420425430glythrarglysserthrglugluasplysglnargilearggluleu435440445metglygluglualavalmetleuglugluglyasnalaargthrleu450455460leuaspvalvaltyrargtyrglnalaaspleumetilealaglygly465470475480argasnmettyrthralatyrlysalaargleupropheleuaspile485490495asnglngluarggluhisalaphealaglytyrglnglyilevalthr500505510leualaargglnleucysglnthrileasnserproiletrpprogln515520525thrhisserargalaprotrpargalaglyglyglyglyglytyrpro530535540tyraspvalproasptyralaprogly545550<210>49<211>1261<212>prt<213>人工序列<220><223>融合多肽<400>49metalametalavalpheargarggluglyargargleuleuproser151015ilealaalaargproilealaalaileargserproleuserserasp202530glnglugluglyleuleuglyvalargserileserthrglnvalval354045argasnargmetlysservallysasnileglnlysilethrlysala505560metlysmetvalalaalaserlysleuargalavalglnglyalapro65707580metserglylysmetlysthrmetaspglyasnalaalaalaalatrp859095ilesertyralaphethrgluvalalaalailetyrproilethrpro100105110serthrprometalagluasnvalaspglutrpalaalaglnglylys115120125lysasnleupheglyglnprovalargleumetglumetglnserglu130135140alaglyalaalaglyalavalhisglyalaleuglnalaglyalaleu145150155160thrthrthrtyrthralaserglnglyleuleuleumetileproasn165170175mettyrlysilealaglygluleuleuproglyvalphehisvalser180185190alaargalaleualathrasnserleuasnilepheglyasphisgln195200205aspvalmetalavalargglnthrglycysalametleualagluasn210215220asnvalglnglnvalmetaspleuseralavalalahisleualaala225230235240ilelysglyargileprophevalasnphepheaspglypheargthr245250255serhisgluileglnlysilegluvalleuglutyrgluglnleuala260265270thrleuleuaspargproalaleuaspserpheargargasnalaleu275280285hisproasphisprovalileargglythralaglnasnproaspile290295300tyrpheglngluargglualaglyasnargphetyrglnalaleupro305310315320aspilevalglusertyrmetthrglnileseralaleuthrglyarg325330335glutyrhisleupheasntyrthrglyalaalaaspalagluargval340345350ileilealametglyservalcysaspthrvalglngluvalvalasp355360365thrleuasnalaalaglyglulysvalglyleuleuservalhisleu370375380pheargpropheserleualahisphephealaglnleuprolysthr385390395400valglnargilealavalleuaspargthrlysgluproglyalagln405410415alagluproleucysleuaspvallysasnalaphetyrhishisasp420425430aspalaproleuilevalglyglyargtyralaleuglyglylysasp435440445valleuproasnaspilealaalavalpheaspasnleuasnlyspro450455460leuprometaspglyphethrleuglyilevalaspaspvalthrphe465470475480thrserleuproproargglnglnthrleualavalserhisaspgly485490495ilethralacyslysphetrpglymetglyseraspglythrvalgly500505510alaasnlysseralailelysileileglyasplysthrproleutyr515520525alaglnalatyrphesertyraspserlyslysserglyglyilethr530535540valserhisleuargpheglyaspargproileasnserprotyrleu545550555560ilehisargalaasppheilesercysserglnglnsertyrvalglu565570575argtyraspleuleuaspglyleulysproglyglythrpheleuleu580585590asncyssertrpseraspalagluleugluglnhisleuprovalgly595600605phelysargtyrleualaarggluasnilehisphetyrthrleuasn610615620alavalaspilealaarggluleuglyleuglyglyargpheasnmet625630635640leumetglnalaalaphephelysleualaalaileileaspprogln645650655thralaalaasptyrleulysglnalavalglulyssertyrglyser660665670lysglyalaalavalileglumetasnglnargalailegluleugly675680685metalaserleuhisglnvalthrileproalahistrpalathrleu690695700aspgluproalaalaglnalaseralametmetproasppheilearg705710715720aspileleuglnprometasnargglncysglyaspglnleuproval725730735seralaphevalglymetgluaspglythrpheproserglythrala740745750alatrpglulysargglyilealaleugluvalprovaltrpglnpro755760765gluglycysthrglncysasnglncysalapheilecysprohisala770775780alaileargproalaleuleuasnglyglugluhisaspalaalapro785790795800valglyleuleuserlysproalaglnglyalalysglutyrhistyr805810815hisleualaileserproleuaspcysserglycysglyasncysval820825830aspilecysproalaargglylysalaleulysmetglnserleuasp835840845serglnargglnmetalaprovaltrpasptyralaleualaleuthr850855860prolysserasnprophearglysthrthrvallysglyserglnphe865870875880gluthrproleuleuglupheserglyalacysalaglycysglyglu885890895thrprotyralaargleuilethrglnleupheglyaspargmetleu900905910ilealaasnalathrglycysserseriletrpglyalaseralapro915920925serileprotyrthrthrasnhisargglyhisglyproalatrpala930935940asnserleuphegluaspasnalaglupheglyleuglymetmetleu945950955960glyglyglnalavalargglnglnilealaaspaspmetthralaala965970975leualaleuprovalseraspgluleuseraspalametargglntrp980985990leualalysglnaspgluglygluglythrarggluargalaasparg99510001005leusergluargleualaalaglulysgluglyvalproleuleu101010151020gluglnleutrpglnasnargasptyrphevalargargsergln102510301035trpilepheglyglyaspglytrpalatyraspileglyphegly104010451050glyleuasphisvalleualaserglygluaspvalasnileleu105510601065valpheaspthrgluvaltyrserasnthrglyglyglnserser107010751080lysserthrprovalalaalailealalysphealaalaglngly108510901095lysargthrarglyslysaspleuglymetmetalametsertyr110011051110glyasnvaltyrvalalaglnvalalametglyalaasplysasp111511201125glnthrleuargalailealaglualaglualatrpproglypro113011351140serleuvalilealatyralaalacysileasnhisglyleulys114511501155alaglymetargcysserglnargglualalysargalavalglu116011651170alaglytyrtrphisleutrpargtyrhisproglnarggluala117511801185gluglylysthrprophemetleuaspserglugluprogluglu119011951200serpheargasppheleuleuglygluvalargtyralaserleu120512101215hislysthrthrprohisleualaaspalaleupheserargthr122012251230glugluaspalaargalaargphealaglntyrargargleuala123512401245glyglugluasptyrlysaspaspaspasplysprogly125012551260<210>50<211>616<212>prt<213>人工序列<220><223>融合多肽<400>50metalametalavalpheargarggluglyargargleuleuproser151015ilealaalaargproilealaalaileargserproleuserserasp202530glnglugluglyleuleuglyvalargserileserthrglnvalval354045argasnargmetlysservallysasnileglnlysilethrlysala505560metlysmetvalalaalaserlysleuargalavalglnglyalapro65707580serglnthrileasplysileasnsercystyrproleupheglugln859095aspglutyrglngluleupheargasnlysargglnleuglugluala100105110hisaspalaglnargvalglngluvalphealatrpthrthrthrala115120125glutyrglualaleuasnpheargargglualaleuthrvalasppro130135140alalysalacysglnproleuglyalavalleucysserleuglyphe145150155160alaasnthrleuprotyrvalhisglyserglnglycysvalalatyr165170175pheargthrtyrpheasnarghisphelysgluproilealacysval180185190seraspsermetthrgluaspalaalavalpheglyglyasnasnasn195200205metasnleuglyleuglnasnalaseralaleutyrlysprogluile210215220ilealavalserthrthrcysmetalagluvalileglyaspaspleu225230235240glnalapheilealaasnalalyslysaspglyphevalaspserser245250255ilealavalprohisalahisthrproserpheileglyserhisval260265270thrglytrpaspasnmetphegluglyphealalysthrphethrala275280285asptyrglnglyglnproglylysleuprolysleuasnleuvalthr290295300glyphegluthrtyrleuglyasnpheargvalleulysargmetmet305310315320gluglnmetalavalprocysserleuleuseraspprosergluval325330335leuaspthrproalaaspglyhistyrargmettyrserglyglythr340345350thrglnglnglumetlysglualaproaspalaileaspthrleuleu355360365leuglnprotrpglnleuleulysserlyslysvalvalglnglumet370375380trpasnglnproalathrgluvalalaileproleuglyleualaala385390395400thraspgluleuleumetthrvalserglnleuserglylysproile405410415alaaspalaleuthrleugluargglyargleuvalaspmetmetleu420425430aspserhisthrtrpleuhisglylyslyspheglyleutyrglyasp435440445proaspphevalmetglyleuthrargpheleuleugluleuglycys450455460gluprothrvalileleuserhisasnalaasnlysargtrpglnlys465470475480alametasnlysmetleuaspalaserprotyrglyargaspserglu485490495valpheileasncysaspleutrphispheargserleumetphethr500505510argglnproaspphemetileglyasnsertyrglylyspheilegln515520525argaspthrleualalysglylysalaphegluvalproleuilearg530535540leuglypheproleupheasparghishisleuhisargglnthrthr545550555560trpglytyrgluglyalametasnilevalthrthrleuvalasnala565570575valleuglulysleuaspseraspthrserglnleuglylysthrasp580585590tyrserpheaspleuvalargalaglyglyglyglyglytyrprotyr595600605aspvalproasptyralaprogly610615<210>51<211>357<212>prt<213>人工序列<220><223>融合多肽<400>51metalametalavalpheargarggluglyargargleuleuproser151015ilealaalaargproilealaalaileargserproleuserserasp202530glnglugluglyleuleuglyvalargserileserthrglnvalval354045argasnargmetlysservallysasnileglnlysilethrlysala505560metlysmetvalalaalaserlysleuargalavalglnglyalapro65707580metasnprotrpglnargphealaargglnargleualaargserarg859095trpasnargaspproalaalaleuaspproalaaspthrproalaphe100105110gluglnalatrpglnargglncyshismetgluglnthrilevalala115120125argvalprogluglyaspileproalaalaleuleugluasnileala130135140alaserleualailetrpleuaspgluglyaspphealaproproglu145150155160argalaalailevalarghishisalaargleugluleualapheala165170175aspilealaargglnalaproglnproaspleuserthrvalglnala180185190trptyrleuarghisglnthrglnphemetargprogluglnargleu195200205thrarghisleuleuleuthrvalaspasnaspargglualavalhis210215220glnargileleuglyleutyrargglnileasnalaserargaspala225230235240phealaproleualaglnarghisserhiscysproseralaleuglu245250255gluglyargleuglytrpileserargglyleuleutyrproglnleu260265270gluthralaleupheserleualagluasnalaleuserleuproile275280285alasergluleuglytrphisleuleutrpcysglualaileargpro290295300alaalaprometgluproglnglnalaleugluseralaargasptyr305310315320leutrpglnglnserglnglnarghisglnargglntrpleuglugln325330335metileserargglnproglyleucysglytyrprotyraspvalpro340345350asptyralaprogly355<210>52<211>551<212>prt<213>人工序列<220><223>融合多肽<400>52metalametalavalpheargarggluglyargargleuleuproser151015ilealaalaargproilealaalaileargserproleuserserasp202530glnglugluglyleuleuglyvalargserileserthrglnvalval354045argasnargmetlysservallysasnileglnlysilethrlysala505560metlysmetvalalaalaserlysleuargalavalglnglyalapro65707580alaaspilepheargthrasplysproleualavalserproilelys859095thrglyglnproleuglyalaileleualaserleuglyilegluhis100105110serileproleuvalhisglyalaglnglycysseralaphealalys115120125valphepheileglnhisphehisaspprovalproleuglnserthr130135140alametaspprothrserthrilemetglyalaaspglyasnilephe145150155160thralaleuaspthrleucysglnargasnasnproglnalaileval165170175leuleuserthrglyleuserglualaglnglyseraspileserarg180185190valvalargglnphearggluglutyrproarghislysglyvalala195200205ileleuthrvalasnthrproaspphetyrglysermetgluasngly210215220pheseralavalleugluservalilegluglntrpvalproproala225230235240proargproalaglnargasnargargvalasnleuleuvalserhis245250255leucysserproglyaspileglutrpleuargargcysvalgluala260265270pheglyleuglnproileileleuproaspleualaglnsermetasp275280285glyhisleualaglnglyasppheserproleuthrglnglyglythr290295300proleuargglnilegluglnmetglyglnserleucysserpheala305310315320ileglyvalserleuhisargalaserserleuleualaproargcys325330335argglygluvalilealaleuprohisleumetthrleugluargcys340345350aspalapheilehisglnleualalysileserglyargalavalpro355360365glutrpleugluargglnargglyglnleuglnaspalametileasp370375380cyshismettrpleuglnglyglnargmetalailealaalaglugly385390395400aspleuleualaalatrpcysaspphealaasnserglnglymetgln405410415proglyproleuvalalaprothrglyhisproserleuargglnleu420425430provalgluargvalvalproglyaspleugluaspleuglnthrleu435440445leucysalahisproalaaspleuleuvalalaasnserhisalaarg450455460aspleualagluglnphealaleuproleuvalargalaglyphepro465470475480leupheasplysleuglyglupheargargvalargglnglytyrser485490495glymetargaspthrleuphegluleualaasnleuilearggluarg500505510hishishisleualahistyrargserproleuargglnasnproglu515520525serserleuserthrglyglyalatyralaalaalaglyasptyrlys530535540aspaspaspasplysprogly545550<210>53<211>263<212>prt<213>人工序列<220><223>融合多肽<400>53metalametalavalpheargarggluglyargargleuleuproser151015ilealaalaargproilealaalaileargserproleuserserasp202530glnglugluglyleuleuglyvalargserileserthrglnvalval354045argasnargmetlysservallysasnileglnlysilethrlysala505560metlysmetvalalaalaserlysleuargalavalglnglyalapro65707580proproleuasptrpleuargargleutrpleuleutyrhisalagly859095lysglyserpheproleuargmetglyleuserproargasptrpgln100105110alaleuargargargleuglygluvalgluthrproleuaspglyglu115120125thrleuthrargargargleumetalagluleuasnalathrargglu130135140glugluargglnglnleuglyalatrpleualaglytrpmetglngln145150155160aspalaglyprometalaglnileilealagluvalserleualaphe165170175asnhisleutrpglnaspleuglyleualaserargalagluleuarg180185190leuleumetseraspcyspheproglnleuvalvalmetasngluhis195200205asnmetargtrplyslysphephetyrargglnargcysleuleugln210215220glnglygluvalilecysargserprosercysaspglucystrpglu225230235240argseralacyspheglualaglyglyglyglyglytyrprotyrasp245250255valproasptyralaprogly260<210>54<211>496<212>prt<213>人工序列<220><223>融合多肽<400>54metalametalavalpheargarggluglyargargleuleuproser151015ilealaalaargproilealaalaileargserproleuserserasp202530glnglugluglyleuleuglyvalargserileserthrglnvalval354045argasnargmetlysservallysasnileglnlysilethrlysala505560metlysmetvalalaalaserlysleuargalavalglnglyalapro65707580lysglnvaltyrleuaspasnasnalathrthrargleuasppromet859095valleuglualametmetpropheleuthraspphetyrglyasnpro100105110serserilehisasppheglyileproalaglnalaalaleugluarg115120125alahisglnglnalaalaalaleuleuglyalaglutyrproserglu130135140ileilephethrsercysalathrglualathralathralaileala145150155160seralailealaleuleuprogluargarggluileilethrserval165170175valgluhisproalathrleualaalacysgluhismetgluargglu180185190glytyrargilehisargilealavalaspglygluglyalaleuasp195200205metalaglnpheargalaalaleuserproargvalalaleuvalser210215220valmettrpalaasnasngluthrglyvalleupheproileglyglu225230235240metalagluleualahisgluglnglyalaleuphehiscysaspala245250255valglnvalvalglylysileproilealavalglyglnthrargile260265270aspmetleusercysseralahislysphehisglyprolysglyval275280285glycysleutyrleuargargglythrargpheargproleuleuarg290295300glyglyhisglnglutyrglyargargalaglythrgluasnilecys305310315320glyilevalglymetglyalaalacysgluleualaasnilehisleu325330335proglymetthrhisileglyglnleuargasnargleugluhisarg340345350leuleualaservalproservalmetvalmetglyglyglyglnpro355360365alavalproglythrvalasnleualapheglupheilegluglyglu370375380alaileleuleuleuleuasnglnalaglyilealaalasersergly385390395400seralacysthrserglyserleugluproserhisvalmetargala405410415metasnileprotyrthralaalahisglythrileargpheserleu420425430serargtyrthrargglulysgluileasptyrvalvalalathrleu435440445proproileileaspargleuargalaleuserprotyrtrpglnasn450455460glylysproargproalaaspalavalphethrprovaltyrglyala465470475480glyglyglyglyglytyrprotyraspvalproasptyralaprogly485490495<210>55<211>365<212>prt<213>人工序列<220><223>融合多肽<400>55metalametalavalpheargarggluglyargargleuleuproser151015ilealaalaargproilealaalaileargserproleuserserasp202530glnglugluglyleuleuglyvalargserileserthrglnvalval354045argasnargmetlysservallysasnileglnlysilethrlysala505560metlysmetvalalaalaserlysleuargalavalglnglyalapro65707580trpasntyrserglulysvallysasphisphepheasnproargasn859095alaargvalvalaspasnalaasnalavalglyaspvalglyserleu100105110sercysglyaspalaleuargleumetleuargvalaspproglnser115120125gluileilegluglualaglypheglnthrpheglycysglyserala130135140ilealaserserseralaleuthrgluleuileileglyhisthrleu145150155160alaglualaglyglnilethrasnglnglnilealaasptyrleuasp165170175glyleuproproglulysmethiscysservalmetglyglngluala180185190leuargalaalailealaasnpheargglygluserleuglugluglu195200205hisaspgluglylysleuilecyslyscyspheglyvalaspglugly210215220hisileargargalavalglnasnasnglyleuthrthrleualaglu225230235240valileasntyrthrlysalaglyglyglycysthrsercyshisglu245250255lysilegluleualaleualagluileleualaglnglnproglnthr260265270thrproalavalalaserglylysaspprohistrpglnservalval275280285aspthrilealagluleuargprohisileglnalaaspglyglyasp290295300metalaleuleuservalthrasnhisglnvalthrvalserleuser305310315320glysercysserglycysmetmetthraspmetthrleualatrpleu325330335glnglnlysleumetgluargthrglycystyrmetgluvalvalala340345350alaalaglyasptyrlysaspaspaspasplysprogly355360365<210>56<211>471<212>prt<213>人工序列<220><223>融合多肽<400>56metalametalavalpheargarggluglyargargleuleuproser151015ilealaalaargproilealaalaileargserproleuserserasp202530glnglugluglyleuleuglyvalargserileserthrglnvalval354045argasnargmetlysservallysasnileglnlysilethrlysala505560metlysmetvalalaalaserlysleuargalavalglnglyalapro65707580metgluargvalleuileasnaspthrthrleuargaspglyglugln859095serproglyvalalapheargthrserglulysvalalailealaglu100105110alaleutyralaalaglyilethralametgluvalglythrproala115120125metglyaspglugluilealaargileglnleuvalargargglnleu130135140proaspalathrleumetthrtrpcysargmetasnalaleugluile145150155160argglnseralaaspleuglyileasptrpvalaspileserilepro165170175alaserasplysleuargglntyrlysleuarggluproleualaval180185190leuleugluargleualametpheilehisleualahisthrleugly195200205leulysvalcysileglycysgluaspalaserargalaserglygln210215220thrleuargalailealagluvalalaglnasnalaproalaalaarg225230235240leuargtyralaaspthrvalglyleuleuaspprophethrthrala245250255alaglnileseralaleuargaspvaltrpserglygluileglumet260265270hisalahisasnaspleuglymetalathralaasnthrleualaala275280285valseralaglyalathrservalasnthrthrvalleuglyleugly290295300gluargalaglyasnalaalaalatrplysproseralaleuglyleu305310315320gluargcysleuglyvalgluthrglyvalhispheseralaleupro325330335alaleucysglnargvalalaglualaalaglnargalaileasppro340345350glnglnproleuvalglygluleuvalphethrhisgluserglyval355360365hisvalalaalaleuleuargaspserglusertyrglnserileala370375380proserleumetglyargsertyrargleuvalleuglylyshisser385390395400glyargglnalavalasnglyvalpheaspglnmetglytyrhisleu405410415asnalaalaglnileasnglnleuleuproalaileargargpheala420425430gluasntrplysargserprolysasptyrgluleuvalalailetyr435440445aspgluleucysglygluseralaleuargalaargglyasptyrlys450455460aspaspaspasplysprogly465470<210>57<211>247<212>prt<213>人工序列<220><223>融合多肽<400>57metalametalavalpheargarggluglyargargleuleuproser151015ilealaalaargproilealaalaileargserproleuserserasp202530glnglugluglyleuleuglyvalargserileserthrglnvalval354045argasnargmetlysservallysasnileglnlysilethrlysala505560metlysmetvalalaalaserlysleuargalavalglnglyalapro65707580proproileasnargglnpheaspmetvalhisseraspglutrpser859095metlysvalalaphealaserserasptyrarghisvalaspglnhis100105110pheglyalathrproargleuvalvaltyrglyvallysalaasparg115120125valthrleuileargvalvalasppheservalgluasnglyhisgln130135140thrglulysilealaargargilehisalaleugluaspcysvalthr145150155160leuphecysvalalaileglyaspalavalpheargglnleuleugln165170175valglyvalargalagluargvalproalaaspthrthrilevalgly180185190leuleuglngluileglnleutyrtrptyrasplysglyglnarglys195200205asnglnargglnargaspprogluargphethrargleuleuglnglu210215220glnglutrphisglyaspproaspproargargalaglyasptyrlys225230235240aspaspaspasplysprogly245<210>58<211>316<212>prt<213>人工序列<220><223>融合多肽<400>58metalametalavalpheargarggluglyargargleuleuproser151015ilealaalaargproilealaalaileargserproleuserserasp202530glnglugluglyleuleuglyvalargserileserthrglnvalval354045argasnargmetlysservallysasnileglnlysilethrlysala505560metlysmetvalalaalaserlysleuargalavalglnglyalapro65707580seraspasnaspthrleuphetrpargmetleualaleupheglnser859095leuproaspleuglnproalaglnilevalasptrpleualaglnglu100105110serglygluthrleuthrprogluargleualathrleuthrglnpro115120125glnleualaalaserpheproseralathralavalmetserproala130135140argtrpserargvalmetalaserleuglnglyalaleuproalahis145150155160leuargilevalargproalaglnargthrproglnleuleualaala165170175phecysserglnaspglyleuvalileasnglyhispheglyglngly180185190argleuphepheiletyralapheaspgluglnglyglytrpleutyr195200205aspleuargargtyrproseralaprohisglnglnglualaasnglu210215220valargalaargleuilegluaspcysglnleuleuphecysglnglu225230235240ileglyglyproalaalaalaargproilearghisargilehispro245250255metlysalaglnproglythrthrileglnalaglncysglualaile260265270asnthrleuleualaglyargleuproprotrpleualalysargleu275280285asnargaspasnproleuglugluargvalphealaglyglyglygly290295300glytyrprotyraspvalproasptyralaprogly305310315<210>59<211>574<212>prt<213>人工序列<220><223>融合多肽<400>59metalametalavalpheargarggluglyargargleuleuproser151015ilealaalaargproilealaalaileargserproleuserserasp202530glnglugluglyleuleuglyvalargserileserthrglnvalval354045argasnargmetlysservallysasnileglnlysilethrlysala505560metlysmetvalalaalaserlysleuargalavalglnglyalapro65707580thrasnalathrglygluargasnleualaleuileglngluvalleu859095gluvalpheprogluthralaarglysgluargarglyshismetmet100105110valseraspprolysmetlysservalglylyscysileileserasn115120125arglysserglnproglyvalmetthrvalargglycysalatyrala130135140glyserlysglyvalvalpheglyproilelysaspmetalahisile145150155160serhisglyproalaglycysglyglntyrserargalagluargarg165170175asntyrtyrthrglyvalserglyvalaspserpheglythrleuasn180185190phethrserasppheglngluargaspilevalpheglyglyasplys195200205lysleuserlysleuilegluglumetgluleuleupheproleuthr210215220lysglyilethrileglnserglucysprovalglyleuileglyasp225230235240aspileseralavalalaasnalaserserlysalaleuasplyspro245250255valileprovalargcysgluglypheargglyvalserglnserleu260265270glyhishisilealaasnaspvalvalargasptrpileleuasnasn275280285arggluglyglnprophegluthrthrprotyraspvalalaileile290295300glyasptyrasnileglyglyaspalatrpalaserargileleuleu305310315320gluglumetglyleuargvalvalalaglntrpserglyaspglythr325330335leuvalglumetgluasnthrprophevallysleuasnleuvalhis340345350cystyrargsermetasntyrilealaarghismetgluglulyshis355360365glnileprotrpmetglutyrasnphepheglyprothrlysileala370375380gluserleuarglysilealaaspglnpheaspaspthrileargala385390395400asnalaglualavalilealaargtyrgluglyglnmetalaalaile405410415ilealalystyrargproargleugluglyarglysvalleuleutyr420425430ileglyglyleuargproarghisvalileglyalatyrgluaspleu435440445glymetgluileilealaalaglytyrgluphealahisasnaspasp450455460tyraspargthrleuproaspleulysgluglythrleuleupheasp465470475480aspalasersertyrgluleuglualaphevallysalaleulyspro485490495aspleuileglyserglyilelysglulystyrilepheglnlysmet500505510glyvalpropheargglnmethissertrpasptyrserglyprotyr515520525hisglytyraspglyphealailephealaargaspmetaspmetthr530535540leuasnasnproalatrpasngluleuthralaprotrpleulysser545550555560alaalaglytyrprotyraspvalproasptyralaprogly565570<210>60<211>599<212>prt<213>人工序列<220><223>融合多肽<400>60metalametalavalpheargarggluglyargargleuleuproser151015ilealaalaargproilealaalaileargserproleuserserasp202530glnglugluglyleuleuglyvalargserileserthrglnvalval354045argasnargmetlysservallysasnileglnlysilethrlysala505560metlysmetvalalaalaserlysleuargalavalglnglyalapro65707580serglnthrileasplysileasnsercystyrproleupheglugln859095aspglutyrglngluleupheargasnlysargglnleuglugluala100105110hisaspalaglnargvalglngluvalphealatrpthrthrthrala115120125glutyrglualaleuasnpheargargglualaleuthrvalasppro130135140alalysalacysglnproleuglyalavalleucysserleuglyphe145150155160alaasnthrleuprotyrvalhisglyserglnglycysvalalatyr165170175pheargthrtyrpheasnarghisphelysgluproilealacysval180185190seraspsermetthrgluaspalaalavalpheglyglyasnasnasn195200205metasnleuglyleuglnasnalaseralaleutyrlysprogluile210215220ilealavalserthrthrcysmetalagluvalileglyaspaspleu225230235240glnalapheilealaasnalalyslysaspglyphevalaspserser245250255ilealavalprohisalahisthrproserpheileglyserhisval260265270thrglytrpaspasnmetphegluglyphealalysthrphethrala275280285asptyrglnglyglnproglylysleuprolysleuasnleuvalthr290295300glyphegluthrtyrleuglyasnpheargvalleulysargmetmet305310315320gluglnmetalavalprocysserleuleuseraspprosergluval325330335leuaspthrproalaaspglyhistyrargmettyrserglyglythr340345350thrglnglnglumetlysglualaproaspalaileaspthrleuleu355360365leuglnprotrpglnleuleulysserlyslysvalvalglnglumet370375380trpasnglnproalathrgluvalalaileproleuglyleualaala385390395400thraspgluleuleumetthrvalserglnleuserglylysproile405410415alaaspalaleuthrleugluargglyargleuvalaspmetmetleu420425430aspserhisthrtrpleuhisglylyslyspheglyleutyrglyasp435440445proaspphevalmetglyleuthrargpheleuleugluleuglycys450455460gluprothrvalileleuserhisasnalaasnlysargtrpglnlys465470475480alametasnlysmetleuaspalaserprotyrglyargaspserglu485490495valpheileasncysaspleutrphispheargserleumetphethr500505510argglnproaspphemetileglyasnsertyrglylyspheilegln515520525argaspthrleualalysglylysalaphegluvalproleuilearg530535540leuglypheproleupheasparghishisleuhisargglnthrthr545550555560trpglytyrgluglyalametasnilevalthrthrleuvalasnala565570575valleuglulysleuaspseraspthrserglnleuglylysthrasp580585590tyrserpheaspleuvalarg595<210>61<211>11<212>prt<213>红褐肉座菌<400>61alathrproproproglyserthrthrthrarg1510<210>62<211>8<212>prt<213>人工序列<220><223>flag表位<400>62asptyrlysaspaspaspasplys15<210>63<211>30<212>prt<213>人工序列<220><223>接头<400>63alathrproproproglyserthrthrthralaasptyrlysaspasp151015aspasplysalathrproproproglyserthrthrthrala202530<210>64<211>1110<212>prt<213>人工序列<220><223>融合多肽<400>64metalametalavalpheargarggluglyargargleuleuproser151015ilealaalaargproilealaalaileargserproleuserserasp202530glnglugluglyleuleuglyvalargserileserthrglnvalval354045argasnargmetlysservallysasnileglnlysilethrlysala505560metlysmetvalalaalaserlysleuargalavalglnglyalapro65707580thrasnalathrglygluargasnleualaleuileglngluvalleu859095gluvalpheprogluthralaarglysgluargarglyshismetmet100105110valseraspprolysmetlysservalglylyscysileileserasn115120125arglysserglnproglyvalmetthrvalargglycysalatyrala130135140glyserlysglyvalvalpheglyproilelysaspmetalahisile145150155160serhisglyproalaglycysglyglntyrserargalagluargarg165170175asntyrtyrthrglyvalserglyvalaspserpheglythrleuasn180185190phethrserasppheglngluargaspilevalpheglyglyasplys195200205lysleuserlysleuilegluglumetgluleuleupheproleuthr210215220lysglyilethrileglnserglucysprovalglyleuileglyasp225230235240aspileseralavalalaasnalaserserlysalaleuasplyspro245250255valileprovalargcysgluglypheargglyvalserglnserleu260265270glyhishisilealaasnaspvalvalargasptrpileleuasnasn275280285arggluglyglnprophegluthrthrprotyraspvalalaileile290295300glyasptyrasnileglyglyaspalatrpalaserargileleuleu305310315320gluglumetglyleuargvalvalalaglntrpserglyaspglythr325330335leuvalglumetgluasnthrprophevallysleuasnleuvalhis340345350cystyrargsermetasntyrilealaarghismetgluglulyshis355360365glnileprotrpmetglutyrasnphepheglyprothrlysileala370375380gluserleuarglysilealaaspglnpheaspaspthrileargala385390395400asnalaglualavalilealaargtyrgluglyglnmetalaalaile405410415ilealalystyrargproargleugluglyarglysvalleuleutyr420425430ileglyglyleuargproarghisvalileglyalatyrgluaspleu435440445glymetgluileilealaalaglytyrgluphealahisasnaspasp450455460tyraspargthrleuproaspleulysgluglythrleuleupheasp465470475480aspalasersertyrgluleuglualaphevallysalaleulyspro485490495aspleuileglyserglyilelysglulystyrilepheglnlysmet500505510glyvalpropheargglnmethissertrpasptyrserglyprotyr515520525hisglytyraspglyphealailephealaargaspmetaspmetthr530535540leuasnasnproalatrpasngluleuthralaprotrpleulysser545550555560alaalathrproproproglyserthrthrthralaasptyrlysasp565570575aspaspasplysalathrproproproglyserthrthrthralaser580585590glnthrileasplysileasnsercystyrproleuphegluglnasp595600605glutyrglngluleupheargasnlysargglnleugluglualahis610615620aspalaglnargvalglngluvalphealatrpthrthrthralaglu625630635640tyrglualaleuasnpheargargglualaleuthrvalaspproala645650655lysalacysglnproleuglyalavalleucysserleuglypheala660665670asnthrleuprotyrvalhisglyserglnglycysvalalatyrphe675680685argthrtyrpheasnarghisphelysgluproilealacysvalser690695700aspsermetthrgluaspalaalavalpheglyglyasnasnasnmet705710715720asnleuglyleuglnasnalaseralaleutyrlysprogluileile725730735alavalserthrthrcysmetalagluvalileglyaspaspleugln740745750alapheilealaasnalalyslysaspglyphevalaspserserile755760765alavalprohisalahisthrproserpheileglyserhisvalthr770775780glytrpaspasnmetphegluglyphealalysthrphethralaasp785790795800tyrglnglyglnproglylysleuprolysleuasnleuvalthrgly805810815phegluthrtyrleuglyasnpheargvalleulysargmetmetglu820825830glnmetalavalprocysserleuleuseraspprosergluvalleu835840845aspthrproalaaspglyhistyrargmettyrserglyglythrthr850855860glnglnglumetlysglualaproaspalaileaspthrleuleuleu865870875880glnprotrpglnleuleulysserlyslysvalvalglnglumettrp885890895asnglnproalathrgluvalalaileproleuglyleualaalathr900905910aspgluleuleumetthrvalserglnleuserglylysproileala915920925aspalaleuthrleugluargglyargleuvalaspmetmetleuasp930935940serhisthrtrpleuhisglylyslyspheglyleutyrglyasppro945950955960aspphevalmetglyleuthrargpheleuleugluleuglycysglu965970975prothrvalileleuserhisasnalaasnlysargtrpglnlysala980985990metasnlysmetleuaspalaserprotyrglyargaspsergluval99510001005pheileasncysaspleutrphispheargserleumetphethr101010151020argglnproaspphemetileglyasnsertyrglylyspheile102510301035glnargaspthrleualalysglylysalaphegluvalproleu104010451050ileargleuglypheproleupheasparghishisleuhisarg105510601065glnthrthrtrpglytyrgluglyalametasnilevalthrthr107010751080leuvalasnalavalleuglulysleuaspseraspthrsergln108510901095leuglylysthrasptyrserpheaspleuvalarg110011051110<210>65<211>1119<212>prt<213>人工序列<220><223>融合多肽<400>65metalametalavalpheargarggluglyargargleuleuproser151015ilealaalaargproilealaalaileargserproleuserserasp202530glnglugluglyleuleuglyvalargserileserthrglnvalval354045argasnargmetlysservallysasnileglnlysilethrlysala505560metlysmetvalalaalaserlysleuargalavalglnglyalapro65707580thrasnalathrglygluargasnleualaleuileglngluvalleu859095gluvalpheprogluthralaarglysgluargarglyshismetmet100105110valseraspprolysmetlysservalglylyscysileileserasn115120125arglysserglnproglyvalmetthrvalargglycysalatyrala130135140glyserlysglyvalvalpheglyproilelysaspmetalahisile145150155160serhisglyproalaglycysglyglntyrserargalagluargarg165170175asntyrtyrthrglyvalserglyvalaspserpheglythrleuasn180185190phethrserasppheglngluargaspilevalpheglyglyasplys195200205lysleuserlysleuilegluglumetgluleuleupheproleuthr210215220lysglyilethrileglnserglucysprovalglyleuileglyasp225230235240aspileseralavalalaasnalaserserlysalaleuasplyspro245250255valileprovalargcysgluglypheargglyvalserglnserleu260265270glyhishisilealaasnaspvalvalargasptrpileleuasnasn275280285arggluglyglnprophegluthrthrprotyraspvalalaileile290295300glyasptyrasnileglyglyaspalatrpalaserargileleuleu305310315320gluglumetglyleuargvalvalalaglntrpserglyaspglythr325330335leuvalglumetgluasnthrprophevallysleuasnleuvalhis340345350cystyrargsermetasntyrilealaarghismetgluglulyshis355360365glnileprotrpmetglutyrasnphepheglyprothrlysileala370375380gluserleuarglysilealaaspglnpheaspaspthrileargala385390395400asnalaglualavalilealaargtyrgluglyglnmetalaalaile405410415ilealalystyrargproargleugluglyarglysvalleuleutyr420425430ileglyglyleuargproarghisvalileglyalatyrgluaspleu435440445glymetgluileilealaalaglytyrgluphealahisasnaspasp450455460tyraspargthrleuproaspleulysgluglythrleuleupheasp465470475480aspalasersertyrgluleuglualaphevallysalaleulyspro485490495aspleuileglyserglyilelysglulystyrilepheglnlysmet500505510glyvalpropheargglnmethissertrpasptyrserglyprotyr515520525hisglytyraspglyphealailephealaargaspmetaspmetthr530535540leuasnasnproalatrpasngluleuthralaprotrpleulysser545550555560alaalathrproproproglyserthrthrthralaasptyrlysasp565570575aspaspasplysalathrproproproglyserthrthrthralaser580585590glnthrileasplysileasnsercystyrproleuphegluglnasp595600605glutyrglngluleupheargasnlysargglnleugluglualahis610615620aspalaglnargvalglngluvalphealatrpthrthrthralaglu625630635640tyrglualaleuasnpheargargglualaleuthrvalaspproala645650655lysalacysglnproleuglyalavalleucysserleuglypheala660665670asnthrleuprotyrvalhisglyserglnglycysvalalatyrphe675680685argthrtyrpheasnarghisphelysgluproilealacysvalser690695700aspsermetthrgluaspalaalavalpheglyglyasnasnasnmet705710715720asnleuglyleuglnasnalaseralaleutyrlysprogluileile725730735alavalserthrthrcysmetalagluvalileglyaspaspleugln740745750alapheilealaasnalalyslysaspglyphevalaspserserile755760765alavalprohisalahisthrproserpheileglyserhisvalthr770775780glytrpaspasnmetphegluglyphealalysthrphethralaasp785790795800tyrglnglyglnproglylysleuprolysleuasnleuvalthrgly805810815phegluthrtyrleuglyasnpheargvalleulysargmetmetglu820825830glnmetalavalprocysserleuleuseraspprosergluvalleu835840845aspthrproalaaspglyhistyrargmettyrserglyglythrthr850855860glnglnglumetlysglualaproaspalaileaspthrleuleuleu865870875880glnprotrpglnleuleulysserlyslysvalvalglnglumettrp885890895asnglnproalathrgluvalalaileproleuglyleualaalathr900905910aspgluleuleumetthrvalserglnleuserglylysproileala915920925aspalaleuthrleugluargglyargleuvalaspmetmetleuasp930935940serhisthrtrpleuhisglylyslyspheglyleutyrglyasppro945950955960aspphevalmetglyleuthrargpheleuleugluleuglycysglu965970975prothrvalileleuserhisasnalaasnlysargtrpglnlysala980985990metasnlysmetleuaspalaserprotyrglyargaspsergluval99510001005pheileasncysaspleutrphispheargserleumetphethr101010151020argglnproaspphemetileglyasnsertyrglylyspheile102510301035glnargaspthrleualalysglylysalaphegluvalproleu104010451050ileargleuglypheproleupheasparghishisleuhisarg105510601065glnthrthrtrpglytyrgluglyalametasnilevalthrthr107010751080leuvalasnalavalleuglulysleuaspseraspthrsergln108510901095leuglylysthrasptyrserpheaspleuvalargtyrprotyr110011051110aspvalproasptyrala1115<210>66<211>10<212>prt<213>人工序列<220><223>c端延伸<400>66gluglnlysleuileserglugluaspleu1510<210>67<211>356<212>prt<213>人工序列<220><223>融合多肽<400>67metalametalavalpheargarggluglyargargleuleuproser151015ilealaalaargproilealaalaileargserproleuserserasp202530glnglugluglyleuleuglyvalargserileserthrglnvalval354045argasnargmetlysservallysasnileglnlysilethrlysala505560metlysmetvalalaalaserlysleuargalavalglnglyalapro65707580metasnprotrpglnargphealaargglnargleualaargserarg859095trpasnargaspproalaalaleuaspproalaaspthrproalaphe100105110gluglnalatrpglnargglncyshismetgluglnthrilevalala115120125argvalprogluglyaspileproalaalaleuleugluasnileala130135140alaserleualailetrpleuaspgluglyaspphealaproproglu145150155160argalaalailevalarghishisalaargleugluleualapheala165170175aspilealaargglnalaproglnproaspleuserthrvalglnala180185190trptyrleuarghisglnthrglnphemetargprogluglnargleu195200205thrarghisleuleuleuthrvalaspasnaspargglualavalhis210215220glnargileleuglyleutyrargglnileasnalaserargaspala225230235240phealaproleualaglnarghisserhiscysproseralaleuglu245250255gluglyargleuglytrpileserargglyleuleutyrproglnleu260265270gluthralaleupheserleualagluasnalaleuserleuproile275280285alasergluleuglytrphisleuleutrpcysglualaileargpro290295300alaalaprometgluproglnglnalaleugluseralaargasptyr305310315320leutrpglnglnserglnglnarghisglnargglntrpleuglugln325330335metileserargglnproglyleucysglygluglnlysleuileser340345350glugluaspleu355<210>68<211>1723<212>dna<213>人工序列<220><223>融合多肽<400>68atggcaatggctgttttccgtcgcgaagggaggcgtctcctcccttgaatacgccgctcg60cccaatcgctgctatccgatctcctctctcttctgaccaggaggaaggacttcttggagt120tcgatctatctcaactcaagtggtgcgtaaccgcatgaagagtgttaagaacatccaaaa180gatcacaaaggcaatgaagatggttgctgcttccaagcttagagcagttcaaggcgcgcc240aactaatgctactggtgaaagaaacctcgctctcatccaagaggttttggaagtttttcc300tgagactgctaggaaagaaaggcgtaagcacatgatggtgtctgatcctaagatgaagtc360tgtgggaaagtgcatcatctctaacagaaagtctcagcctggtgtgatgactgttagagg420atgtgcttatgctggatctaagggtgttgttttcggacctatcaaggatatggctcatat480ctctcatggacctgctggatgtggacaatattctagagctgagcgtaggaactactacac540tggtgtttctggtgtggattctttcggaactctcaacttcacctctgatttccaagagcg600tgatatcgttttcggaggtgataagaagctctctaagttgatcgaagagatggaactctt660gttcccactcactaagggaatcactatccaatctgagtgccctgttggacttatcggaga720tgatatttctgctgtggctaacgcttcttctaaggctcttgataagcctgttatccctgt780tagatgtgaaggattcaggggagtttctcagtctcttggacatcatatcgctaacgatgt840ggtgagagattggatccttaacaacagagagggacaacctttcgagactactccttacga900tgttgctatcatcggagactacaacattggaggtgatgcttgggcttctagaatccttct960tgaagagatgggactcagagttgttgctcaatggtctggtgatggtactcttgttgagat1020ggaaaacacccctttcgttaagctcaacctcgttcattgctaccgtagcatgaactacat1080tgctagacacatggaagagaagcaccaaattccttggatggagtacaacttcttcggacc1140tactaagatcgctgagtctcttagaaagatcgctgatcagttcgatgataccatcagagc1200taatgctgaggctgttattgctagatacgagggacaaatggctgctattatcgctaagta1260cagacctagactcgagggaagaaaggttttgctttacatcggaggactcagacctagaca1320tgttattggagcttacgaggatctcggaatggaaattattgctgctggatacgagttcgc1380tcacaacgatgattacgatagaactctccctgacctcaaagagggaactcttcttttcga1440tgacgcttcttcatacgagcttgaggcttttgttaaggctcttaagcctgatctcatcgg1500atctggaatcaaagagaagtacatcttccagaagatgggagttcctttcagacagatgca1560ctcttgggattattctggaccttaccatggatacgacggatttgctatcttcgctaggga1620tatggatatgactctcaacaatcctgcttggaacgaacttactgctccttggcttaagtc1680tgctgccggctacccttacgacgttcctgattacgctcccggg1723<210>69<211>4<212>prt<213>肺炎克雷伯菌<400>69aspleuargval1<210>70<211>117<212>dna<213>人工序列<220><223>融合多肽<400>70atgtcaactcaagtggtgcgtaaccgcatgaagagtgttaagaacatccaaaagatcaca60aaggcaatgaagatggttgctgcttccaagcttagagcagttcaaggcgcgccaact117<210>71<211>39<212>prt<213>人工序列<220><223>融合多肽<400>71metserthrglnvalvalargasnarglysservallysasnilegln151015lysilethrlysalametlysmetvalalaalaserlysleuargala202530valglnglyalaprothrmet35<210>72<211>33<212>dna<213>人工序列<220><223>引物<400>72atgatgactaatgctactggcgaacgtaacctg33<210>73<211>28<212>dna<213>人工序列<220><223>引物<400>73ccggctcctccgctagataaaaatgtga28<210>74<211>86<212>prt<213>肺炎克雷伯菌<400>74metmetglutrpphetyrglnileproglyvalaspgluleuargser151015alagluserphepheglnphephealavalprotyrglnprogluleu202530leuglyargcysserleuprovalleualathrphehisarglysleu354045argalagluvalproleuglnasnargleugluaspasnaspargala505560protrpleuleualaargargleuleualaglusertyrglnglngln65707580pheglngluserglythr85<210>75<211>51<212>prt<213>人工序列<220><223>n端延伸<400>75metalametalavalpheargarggluglyargargleuleuproser151015ilealaalaargproilealaalaileargserproleuserserasp202530glnglugluglyleuleuglyvalargserileserthrglnvalval354045argasnarg50<210>76<211>9<212>prt<213>人工序列<220><223>mtpscar<400>76ileserthrglnvalvalargasnarg15<210>77<211>33<212>prt<213>人工序列<220><223>n端延伸<400>77mettyrargphealaserasnleualaserlysalaargilealagln151015asnalaargglnvalserserargmetsertrpserargasntyrgly202530gly<210>78<211>31<212>prt<213>人工序列<220><223>n端延伸<400>78mettyrargphealaserasnleualaserlysalaargilealagln151015asnalaargglnvalserserargmetsertrpserargasntyr202530<210>79<211>31<212>prt<213>人工序列<220><223>n端延伸<400>79metalaileargcysvalalaserarglysthrleualaglyleulys151015gluthrserserargleuleuargileargglyileglnglygly202530<210>80<211>60<212>prt<213>人工序列<220><223>n端延伸<400>80metalaileargcysvalalaserarglysthrleualaglyleulys151015gluthrserserargleuleuargileargglyileglnmetalaile202530argcysvalalaserarglysthrleualaglyleulysgluthrser354045serargleuleuargileargglyileglnglygly505560<210>81<211>29<212>prt<213>人工序列<220><223>n端延伸<400>81metalaileargcysvalalaserarglysthrleualaglyleulys151015gluthrserserargleuleuargileargglyglygly2025<210>82<211>56<212>prt<213>人工序列<220><223>n端延伸<400>82metalaileargcysvalalaserarglysthrleualaglyleulys151015gluthrserserargleuleuargileargglymetalaileargcys202530valalaserarglysthrleualaglyleulysgluthrserserarg354045leuleuargileargglyglygly5055<210>83<211>34<212>prt<213>人工序列<220><223>n端延伸<400>83metpheleuthrargphevalglyargargpheleualaalaalaser151015alaargsergluserthrthralaalaalaalaalaserthrilearg202530glygly<210>84<211>70<212>prt<213>人工序列<220><223>n端延伸<400>84metalaserthrargvalleualaserargleualaserglnmetala151015alaseralalysvalalaargproalavalargvalalaglnvalser202530lysargthrileglnthrglyserproleuglnthrleulysargthr354045glnmetthrserilevalasnalathrthrargglnalapheglnlys505560argalatyrserglygly6570<210>85<211>53<212>prt<213>人工序列<220><223>n端延伸<400>85metalametalavalpheargarggluglyargargleuleuproser151015ilealaalaargproilealaalaileargserproleuserserasp202530glnglugluglyleuleuglyvalargserileserthrglnvalval354045argasnargglygly50<210>86<211>61<212>prt<213>人工序列<220><223>n端延伸<400>86metleuserleuargglnserileargphephelysproalathrarg151015thrleucysserserargtyrleuleuglnglnlysproseralatrp202530serhisproglnpheglulysglyglyglyserglyglyglysergly354045glyseralatrpserhisproglnpheglulysglygly505560<210>87<211>43<212>prt<213>人工序列<220><223>n端延伸<400>87metleuserleuargglnserileargphephelysproalathrarg151015thrleucysserserargtyrleuleuglnglnlysproglyglyhis202530hishishishishishishishishisglygly3540<210>88<211>4<212>prt<213>人工序列<220><223>预测的scar<400>88ileglnglygly1<210>89<211>4<212>prt<213>人工序列<220><223>预测的scar<400>89ileglnglygly1<210>90<211>15<212>prt<213>人工序列<220><223>预测的scar<400>90gluserthrthralaalaalaalaalaserthrileargglygly151015<210>91<211>4<212>prt<213>人工序列<220><223>预测的scar<400>91tyrserglygly1<210>92<211>11<212>prt<213>人工序列<220><223>预测的scar<400>92ileserthrglnvalvalargasnargglygly1510<210>93<211>36<212>prt<213>人工序列<220><223>预测的scar<400>93glnglnlysproseralatrpserhisproglnpheglulysglygly151015glyserglyglyglyserglyglyseralatrpserhisproglnphe202530glulysglygly35<210>94<211>16<212>prt<213>人工序列<220><223>预测的scar<400>94lysproglyglyhishishishishishishishishishisglygly151015<210>95<211>488<212>prt<213>人工序列<220><223>变异体<400>95metmetthrasnalathrglygluargasnleualaleuileglnglu151015valleugluvalpheprogluthralaarglysgluargarglyshis202530metmetvalseraspproglumetgluservalglylyscysileile354045serasnarglysserglnproglyvalmetthrvalargglycysala505560tyralaglyserlysglyvalvalpheglyproilelysaspmetala65707580hisileserhisglyprovalglycysglyglntyrserargalagly859095argargasntyrtyrthrglyvalserglyvalaspserpheglythr100105110leuasnphethrserasppheglngluargaspilevalpheglygly115120125asplyslysleuserlysleuilegluglumetgluleuleuphepro130135140leuthrlysglyilethrileglnserglucysprovalglyleuile145150155160glyaspaspileseralavalalaasnalaserserlysalaleuasp165170175lysprovalileprovalargcysgluglypheargglyvalsergln180185190serleuglyhishisilealaasnaspvalvalargasptrpileleu195200205asnasnarggluglyglnprophegluthrthrprotyraspvalala210215220ileileglyasptyrasnileglyglyaspalatrpalaserargile225230235240leuleugluglumetglyleuargvalvalalaglntrpserglyasp245250255glythrleuvalglumetgluasnthrprophevallysleuasnleu260265270valhiscystyrargsermetasntyrilealaarghismetgluglu275280285lyshisglnileprotrpmetglutyrasnphepheglyprothrlys290295300ilealagluserleuarglysilealaaspglnpheaspaspthrile305310315320argalaasnalaglualavalilealaargtyrgluglyglnmetala325330335alaileilealalystyrargproargleugluglyarglysvalleu340345350leutyrmetglyglyleuargproarghisvalileglyalatyrglu355360365aspleuglymetgluileilealaalaglytyrgluphealahisasn370375380aspasptyraspargthrleuproaspleulysgluglythrleuleu385390395400pheaspaspalasersertyrgluleuglualaphevallysalaleu405410415lysproaspleuileglyserglyilelysglulystyrilephegln420425430lysmetglyvalpropheargglnmethissertrpasptyrsergly435440445protyrhisglytyraspglyphealailephealaargaspmetasp450455460metthrleuasnasnproalatrpasngluleuthralaprotrpleu465470475480lysserglyvalargproargser485<210>96<211>400<212>prt<213>人工序列<220><223>变异体<400>96metlysglnvaltyrleuaspasnasnalathrthrargleuasppro151015metvalleuglualametmetpropheleuthraspphetyrglyasn202530proserserilehisasppheglyileproalaglnalaalaleuglu354045argalahisglnglnalaalaalaleuleuglyalaglutyrproser505560gluileilephethrsercysalathrglualathralathralaile65707580alaseralailealaleuleuprogluargarggluileilethrser859095valvalgluhisproalathrleualaalacysgluhisleugluarg100105110glnglytyrargilehisargilealavalaspsergluglyalaleu115120125aspmetalaglnpheargalaalaleuserproargvalalaleuval130135140servalmettrpalaasnasngluthrglyvalleupheproilegly145150155160glumetalagluleualahisgluglnglyalaleuphehiscysasp165170175alavalglnvalvalglylysileproilealavalglyglnthrarg180185190ileaspmetleusercysseralahislysphehisglyprolysgly195200205valglycysleutyrleuargargglythrargpheargproleuleu210215220argglyglyhisglnglutyrglyargargalaglythrgluasnile225230235240cysglyilevalglymetglyalaalacysgluleualaasnilehis245250255leuproglymetthrhisileglyglnleuargasnargleugluhis260265270argleuleualaservalproservalmetvalmetglyglyglygln275280285proargvalproglythrvalasnleualapheglupheileglugly290295300glualaileleuleuleuleuasnglnalaglyilealaalaserser305310315320glyseralacysthrserglyserleugluproserhisvalmetarg325330335alametasnileprotyrthralaalahisglythrileargpheser340345350leuserargtyrthrargglulysgluileasptyrvalvalalathr355360365leuproproileileaspargleuargalaleuserprotyrtrpgln370375380asnglylysproargproalaaspalavalphethrprovaltyrgly385390395400<210>97<211>9<212>prt<213>人工序列<220><223>c端延伸<400>97tyrprotyraspvalproasptyrala15<210>98<211>59<212>dna<213>人工序列<220><223>寡核苷酸引物<400>98tcatcaagcgtaatcaggaacatcgtaggggtaacgaaccagatcgaaagaatagtcgg59<210>99<211>23<212>dna<213>人工序列<220><223>寡核苷酸引物<400>99aagggcgaattccagcacactgg23<210>100<211>27<212>dna<213>人工序列<220><223>寡核苷酸引物<400>100cctgattgtatccgcatctgatgctac27<210>101<211>39<212>dna<213>人工序列<220><223>寡核苷酸引物<400>101aacctgcagggctaactaactaaccacggacaaaaaacc39<210>102<211>56<212>dna<213>人工序列<220><223>寡核苷酸引物<400>102ggtagcatcagatgcggatacaatcaggtcatcaagcgtaatcaggaacatcgagg56<210>103<211>51<212>dna<213>人工序列<220><223>寡核苷酸引物<400>103ccagtgtgctggaattcgcccttaacctgcagggctaactaactaaccacg51<210>104<211>30<212>prt<213>人工序列<220><223>接头<400>104alathrproproproglyserthrthrthralatyrprotyraspval151015proasptyralathrproproproglyserthrthrthrala202530<210>105<211>24<212>dna<213>人工序列<220><223>寡核苷酸引物<400>105atgatgactaacgctacaggagaa24<210>106<211>32<212>dna<213>人工序列<220><223>寡核苷酸引物<400>106tcatcatctaaccaaatcaaaactgtaatctg32<210>107<211>44<212>dna<213>人工序列<220><223>寡核苷酸引物<400>107ccggctcctccgctagataaaaatgtgaatttttgcatgcagcc44<210>108<211>36<212>dna<213>人工序列<220><223>寡核苷酸引物<400>108cctgattgtatccgcatctgatgctaccgtggttga36<210>109<211>39<212>dna<213>人工序列<220><223>寡核苷酸引物<400>109ttctcctgtagcgttagtcatcatccggctcctccgcta39<210>110<211>45<212>dna<213>人工序列<220><223>寡核苷酸引物<400>110cagatgcggatacaatcaggtcatcatctaaccaaatcaaaactg45<210>111<211>9<212>prt<213>人工序列<220><223>接头<400>111metserthrglnvalvalargasnarg15<210>112<211>9<212>prt<213>人工序列<220><223>ha表位序列<400>112tyrprotyraspvalproasptyrala15<210>113<211>46<212>prt<213>人工序列<220><223>接头<400>113thrserserglyglythrserthrglyglyserthrthrthrthrala151015tyrprotyraspvalproasptyralaserglythrthrserthrlys202530alaserthrthrserthrserserthrserthrglythrgly354045<210>114<211>27<212>dna<213>人工序列<220><223>寡核苷酸引物<400>114atgaagggaaatgagattcttgctctt27<210>115<211>24<212>dna<213>人工序列<220><223>寡核苷酸引物<400>115tcatcaatcagcagcataagcacc24<210>116<211>30<212>dna<213>人工序列<220><223>寡核苷酸引物<400>116ttgtaataacctccagtgatgaattgaata30<210>117<211>30<212>dna<213>人工序列<220><223>寡核苷酸引物<400>117ctagagattaatatggagaaattaagcatg30<210>118<211>59<212>dna<213>人工序列<220><223>寡核苷酸引物<400>118aagagcaagaatctcatttcccttcatttgtaataacctccagtgatgaattgatagtg59<210>119<211>61<212>dna<213>人工序列<220><223>寡核苷酸引物<400>119tagttttcatgcttaatttctccatattaatctctagtcatcaatcagcagcataagcac60c61<210>120<211>545<212>prt<213>人工序列<220><223>启动子<400>120thralaalathrthrglythrthralathrthralathrcysalaala151015thralaalaalaalaglyalaalathrthrthrthrthralathrthr202530glythrthralathrthrglythrglythrthralathrthrthrgly354045glythralaalathrthrthralathrglycysthrthralathrala505560alaglythralaalathrthrcysthralathrglyalathrthrala65707580alathrthrglythrglyalaalathrthralaalathralaalagly859095alacysthralaalathrglyalaglyglyalathralaalathrala100105110alathrthrglyalaalathrthrthrglyalathrthralaalaala115120125thrthralaalacysthrcysthrglycysglyalaalaglycyscys130135140alathralathrglythrcysthrthrthrcysalacysglythrgly145150155160alaglyalaglythrcysalacysglythrglyalathrglythrcys165170175thrcyscysglycysglyalacysalaglyglycysthrglyglycys180185190alacysglyglyglyglycysthrthralaglythralathrthrala195200205cyscyscyscyscysglythrglycyscysglyglyglyalathrcys210215220alaglyalaglyalacysalathrthrthrglyalacysthralaala225230235240alathrglythrthrglyalacysthrthrglyglyalaalathrala245250255alathralaglycyscyscysthrthrglyglyalathrthralagly260265270alathrglyalacysalacysglythrglyglyalacysglycysthr275280285cysalaglyglyalathrcysthrglythrglyalathrglycysthr290295300alaglythrglyalaalaglycysglycysthrthralaalaglycys305310315320thrglyalaalacysglyalaalathrcysthrglyalacysglygly325330335alaalaglyalaglycysglyglyalacysalaalaalacysglycys340345350alacysalathrglyglyalacysthralathrglyglycyscyscys355360365alacysthrglycysthrthrthralathrthralaalaalaglyala370375380alaglythrglyalaalathrglyalacysalaglycysthrglythr385390395400cysthrthrthrglycysthrthrcysalaalaglyalacysglyala405410415alaglythralaalaalaglyalaalathralaglythrglyglyala420425430alaalaalacysglycysglythralaalaalaglyalaalathrala435440445alaglycysglythralacysthrcysalaglythralacysglycys450455460thrthrcysglythrglyglycysthrthrthralathralaalaala465470475480thralaglythrglycysthrthrcysglythrcysthrthralathr485490495thrcysthrthrcysglythrthrglythralathrcysalathrcys500505510alaalacysglyalaalaglyalaalaglythrthralaalaglycys515520525thrthrthrglythrthrcysthrglycysglythrthrthrthrala530535540ala545<210>121<211>528<212>prt<213>人工序列<220><223>启动子<400>121thralaalathrthrglythrthralathrthralathrcysalaala151015thralaalaalaalaglyalaalathrthrthrthrthralathrthr202530glythrthralathrthrglythrglythrthralathrthrthrgly354045glythralaalathrthrthralathrglycysthrthralathrala505560alaglythralaalathrthrcysthralathrglyalathrthrala65707580alathrthrglythrglyalaalathrthralaalathralaalagly859095alacysthralaalathrglyalaglyglyalathralaalathrala100105110alathrthrglyalaalathrthrthrglyalathrthralaalaala115120125thrthralaalacysthrcysthrglycysglyalaalaglycyscys130135140alathralathrglythrcysthrthrthrcysalacysglythrgly145150155160alaglyalaglythrcysalacysglythrglyalathrglythrcys165170175thrcyscysglycysglyalacysalaglyglycysthrglyglycys180185190alacysglyglyglyglycysthrthralaglythralathrthrala195200205cyscyscyscyscysglythrglycyscysglyglyglyalathrcys210215220alaglyalaglyalacysalathrthrthrglyalacysthralaala225230235240alathrglythrthrglyalacysthrthrglyglyalaalathrala245250255alathralaglycyscyscysthrthrglyglyalathrthralagly260265270alathrglyalacysalacysglythrglyglyalacysglycysthr275280285cysalaglyglyalathrcysthrglythrglyalathrglycysthr290295300alaglythrglyalaalaglycysglycysthrthralaalaglycys305310315320thrglyalaalacysglyalaalathrcysthrglyalacysglygly325330335alaalaglyalaglycysglyglyalacysalaalaalacysglycys340345350alacysalathrglyglyalacysthralathrglyglycyscyscys355360365alacysthrglycysthrthrthralathrthralaalaalaglyala370375380alaglythrglyalaalathrglyalacysalaglycysthrglythr385390395400cysthrthrthrglycysthrthrcysalaalaglyalacysglyala405410415alaglythralaalaalaglyalaalathralaglythrglyglyala420425430alaalaalacysglycysglythralaalaalaglyalaalathrala435440445alaglycysglythralacysthrcysalaglythralacysglycys450455460thrthrcysglythrglyglycysthrthrthralathralaalaala465470475480thralaglythrglycysthrthrcysglythrcysthrthralathr485490495thrcysthrthrcysglythrthrglythralathrcysalathrcys500505510alaalacysglyalaalaglyalaalaglythrthralaalaglycys515520525<210>122<211>569<212>prt<213>人工序列<220><223>启动子<400>122thralaalathrthralaalathralaglythralaalathrthrala151015thrglyalathrthralaalathrthralathrglyalaglyalathr202530alaalaglyalaglythrthrglythrthralathrthralaalathr354045glycysthrthralathrglyalaglyglyalaalathralaalaala505560glyalaalathrglyalathrthralaalathralathrthrglythr65707580thrthralaalathrthrthrthralathrthrcyscysglycysgly859095alaalaglycysglyglythrglythrglythrthralathrglythr100105110thrthrthrthrglythrthrglyglyalaglyalacysalathrcys115120125alacysglythrglyalacysthrcysthrcysalacysglythrgly130135140alathrglythrcysthrcyscysglycysglyalacysalaglygly145150155160cysthrglyglycysalacysglyglyglyglycysthrthralagly165170175thralathrthralacyscyscyscyscysglythrglycyscysgly180185190glyglyalathrcysalaglyalaglyalacysalathrthrthrgly195200205alacysthralaalaalathralathrthrglyalacysthrthrgly210215220glyalaalathralaalathralaglycyscyscysthrthrglygly225230235240alathrthralaglyalathrglyalacysalacysglythrglygly245250255alacysglycysthrcysalaglyglyalathrcysthrglythrgly260265270alathrglycysthralaglythrglyalaalaglycysglycysthr275280285thralaalaglycysthrglyalaalacysglyalaalathrcysthr290295300glyalacysglyglyalaalaglyalaglycysglyglyalacysala305310315320thralacysglycysalacysalathrglyglyalathrthralathr325330335glyglycyscyscysalacysalathrglythrcysthralaalaala340345350glythrglythralathrcysthrcysthrthrthralacysalagly355360365cysthralathralathrcysglyalathrglythrglyalacysgly370375380thralaalaglyalathrglycysthrthrthralacysthrthrcys385390395400glycysthrthrcysglyalaalaglythralaalaalaglythrala405410415glyglyalaalaalathrthrglycysthrcysglycysthralaala420425430glythrthralathrthrcysthrthrthrthrcysthrglyalaala435440445alaglyalaalaalathrthralaalathrthrthralaalathrthr450455460cysthralaalaalathrthralaalaalathrthralaalaalathr465470475480glyalaglythrglyglycysthralathralaalaalathralagly485490495thrglythrcysglyalathrglycysthralacyscysthrcysala500505510cysalathrcysglythralathrthrcysthrthrcysthrthrcys515520525glycysalathrcysglythrcysthrglythrthrcysthrglygly530535540thrthrthrthralaalaglyglyalaalathrthrcysthralagly545550555560alaglythrcysalaglyalacyscys565<210>123<211>1428<212>dna<213>人工序列<220><223>启动子<400>123ggaggaattccaatcccacaaaaatctgagcttaacagcacagttgctcctctcagagca60gaatcgggtattcaacaccctcatatcaactactacgttgtgtataacggtccacatgcc120ggtatatacgatgactggggttgtacaaaggcggcaacaaacggcgttcccggagttgca180cacaagaaatttgccactattacagaggcaagagcagcagctgacgcgtacacaacaagt240cagcaaacagacaggttgaacttcatccccaaaggagaagctcaactcaagcccaagagc300tttgctaaggccctaacaagcccaccaaagcaaaaagcccactggctcacgctaggaacc360aaaaggcccagcagtgatccagccccaaaagagatctcctttgccccggagattacaatg420gacgatttcctctatctttacgatctaggaaggaagttcgaaggtgaaggtgacgacact480atgttcaccactgataatgagaaggttagcctcttcaatttcagaaagaatgctgaccca540cagatggttagagaggcctacgcagcaagtctcatcaagacgatctacccgagtaacaat600ctccaggagatcaaataccttcccaagaaggttaaagatgcagtcaaaagattcaggact660aattgcatcaagaacacagagaaagacatatttctcaagatcagaagtactattccagta720tggacgattcaaggcttgcttcataaaccaaggcaagtaatagagattggagtctctaaa780aaggtagttcctactgaatctaaggccatgcatggagtctaagattcaaatcgaggatct840aacagaactcgccgtcaagactggcgaacagttcatacagagtcttttacgactcaatga900caagaagaaaatcttcgtcaacatggtggagcacgacactctggtctactccaaaaatgt960caaagatacagtctcagaagatcaaagggctattgagacttttcaacaaaggataatttc1020gggaaacctcctcggattccattgcccagctatctgtcacttcatcgaaaggacagtaga1080aaaggaaggtggctcctacaaatgccatcattgcgataaaggaaaggctatcattcaaga1140tctctctgccgacagtggtcccaaagatggacccccacccacgaggagcatcgtggaaaa1200agaagaggttccaaccacgtctacaaagcaagtggattgatgtgacatctccactgacgt1260aagggatgacgcacaatcccactatccttcgcaagacccttcctctatataaggaagttc1320atttcatttggagaggacacgctcgagtataagagctcatttttacaacaattaccaaca1380acaacaaacaacaaacaacattacaattacatttacaattatcgatac1428<210>124<211>839<212>dna<213>人工序列<220><223>启动子<400>124gtcaacatggtggagcacgacactctggtctactccaaaaatgtcaaagatacagtctca60gaagatcaaagggctattgagacttttcaacaaaggataatttcgggaaacctcctcgga120ttccattgcccagctatctgtcacttcatcgaaaggacagtagaaaaggaaggtggctcc180tacaaatgccatcattgcgataaaggaaaggctatcattcaagatctctctgccgacagt240ggtcccaaagatggacccccacccacgaggagcatcgtggaaaaagaagaggttccaacc300acgtctacaaagcaagtggattgatgtgataacatggtggagcacgacactctggtctac360tccaaaaatgtcaaagatacagtctcagaagatcaaagggctattgagacttttcaacaa420aggataatttcgggaaacctcctcggattccattgcccagctatctgtcacttcatcgaa480aggacagtagaaaaggaaggtggctcctacaaatgccatcattgcgataaaggaaaggct540atcattcaagatctctctgccgacagtggtcccaaagatggacccccacccacgaggagc600atcgtggaaaaagaagaggttccaaccacgtctacaaagcaagtggattgatgtgacatc660tccactgacgtaagggatgacgcacaatcccactatccttcgcaagacccttcctctata720taaggaagttcatttcatttggagaggacacgctcgagtataagagctcatttttacaac780aattaccaacaacaacaaacaacaaacaacattacaattacatttacaattatcgatac839<210>125<211>172<212>prt<213>番茄丛矮病毒属<400>125metgluargalaileglnglyasnaspalaarggluglnalaasnser151015gluargtrpaspglyglyserglyserserthrserpropheglnleu202530proaspgluserprosertrpthrglutrpargleuhisasnaspglu354045thrasnserasnglnaspasnproleuglyphelysglusertrpgly505560pheglylysvalvalphelysargtyrleuargtyrgluargthrglu65707580thrserleuhisargvalleuglysertrpthrglyaspservalasn859095tyralaalaserargphepheglyvalasnglnileglycysthrtyr100105110serileargpheargglyvalservalthrileserglyglyserarg115120125thrleuglnhisleucysglumetalaileargserlysglngluleu130135140leuglnleuthrprovalgluvalgluserasnvalserargglycys145150155160progluglyvalgluthrphegluglugluserglu165170<210>126<211>17<212>prt<213>人工序列<220><223>肽<400>126leuhisasnaspgluthrasnserasnglnaspasnproleuglyphe151015lys<210>127<211>16<212>prt<213>人工序列<220><223>肽<400>127valleuglysertrpthrglyaspservalasntyralaalaserarg151015<210>128<211>18<212>prt<213>人工序列<220><223>肽<400>128ileasnsercystyrproleuphegluglnaspglutyrglngluleu151015phearg<210>129<211>10<212>prt<213>人工序列<220><223>肽<400>129glnleugluglualahisaspalaglnarg1510<210>130<211>9<212>prt<213>人工序列<220><223>肽<400>130glualaleuthrvalaspproalalys15<210>131<211>12<212>prt<213>人工序列<220><223>肽<400>131thrphethralaasptyrglnglyglnproglylys1510<210>132<211>18<212>prt<213>人工序列<220><223>肽<400>132leuprolysleuasnleuvalthrglyphegluthrtyrleuglyasn151015phearg<210>133<211>28<212>prt<213>人工序列<220><223>肽<400>133metmetgluglnmetalavalprocysserleuleuseraspproser151015gluvalleuaspthrproalaaspglyhistyrarg2025<210>134<211>12<212>prt<213>人工序列<220><223>肽<400>134mettyrserglyglythrthrglnglnglumetlys1510<210>135<211>18<212>prt<213>人工序列<220><223>肽<400>135glualaproaspalaileaspthrleuleuleuglnprotrpglnleu151015leulys<210>136<211>15<212>prt<213>人工序列<220><223>肽<400>136pheglyleutyrglyaspproaspphevalmetglyleuthrarg151015<210>137<211>14<212>prt<213>人工序列<220><223>肽<400>137aspsergluvalpheileasncysaspleutrphisphearg1510<210>138<211>12<212>prt<213>人工序列<220><223>肽<400>138glnproaspphemetileglyasnsertyrglylys1510<210>139<211>8<212>prt<213>人工序列<220><223>肽<400>139alaphegluvalproleuilearg15<210>140<211>8<212>prt<213>人工序列<220><223>肽<400>140leuglypheproleupheasparg15<210>141<211>23<212>prt<213>人工序列<220><223>肽<400>141glnthrthrtrpglytyrgluglyalametasnilevalthrthrleu151015valasnalavalleuglulys20<210>142<211>10<212>prt<213>人工序列<220><223>肽<400>142leuaspseraspthrserglnleuglylys1510<210>143<211>9<212>prt<213>人工序列<220><223>肽<400>143thrasptyrserpheaspleuvalarg15<210>144<211>11<212>prt<213>人工序列<220><223>肽<400>144leuglythrglnmetilehisphevalproarg1510<210>145<211>11<212>prt<213>人工序列<220><223>肽<400>145metthrvalileglutyraspproalacyslys1510<210>146<211>9<212>prt<213>人工序列<220><223>肽<400>146valmetilevalglycysaspprolys15<210>147<211>16<212>prt<213>人工序列<220><223>肽<400>147cysalagluserglyglyprogluproglyvalglycysalaglyarg151015<210>148<211>16<212>prt<213>人工序列<220><223>肽<400>148serthrthrthrglnasnleuvalalaalaleualaglumetglylys151015<210>149<211>17<212>prt<213>人工序列<220><223>肽<400>149serthrthrthrglnasnleuvalalaalaleualaglumetglylys151015lys<210>150<211>15<212>prt<213>人工序列<220><223>肽<400>150glnthrasparggluaspgluleuileilealaleualaglulys151015<210>151<211>23<212>prt<213>人工序列<220><223>肽<400>151alaglngluiletyrilevalcysserglyglumetmetalamettyr151015alaalaasnasnileserlys20<210>152<211>9<212>prt<213>人工序列<220><223>肽<400>152alavalglnglyalaprothrmetarg15<210>153<211>9<212>prt<213>人工序列<220><223>肽<400>153leuglyglyleuilecysasnserarg15<210>154<211>32<212>prt<213>人工序列<220><223>肽<400>154alaglnasnthrilemetglumetalaalagluvalglyservalglu151015aspleugluleugluaspvalleuglnileglytyrglyaspvalarg202530<210>155<211>16<212>prt<213>人工序列<220><223>肽<400>155leucysleuserthrasnglyleuvalleuproaspalavalasparg151015<210>156<211>12<212>prt<213>人工序列<220><223>肽<400>156phealaalaileleugluleuleualaaspvallys1510<210>157<211>17<212>prt<213>人工序列<220><223>肽<400>157glygluserglualaaspaspalacysleuvalalavalalaserser151015arg<210>158<211>16<212>prt<213>人工序列<220><223>肽<400>158alavalglnglyalaprothrsercysserserpheserglyglylys151015<210>159<211>19<212>prt<213>人工序列<220><223>肽<400>159ileasnservalleuileproglyileasnaspserglymetalagly151015valserarg<210>160<211>26<212>prt<213>人工序列<220><223>肽<400>160glnvalalaglnalaileproglnleuservalvalglyilealagly151015proglyaspproleualaasnilealaarg2025<210>161<211>15<212>prt<213>人工序列<220><223>肽<400>161ilealaglygluleuleuproglyvalphehisvalseralaarg151015<210>162<211>13<212>prt<213>人工序列<220><223>肽<400>162glythralaglnasnproaspiletyrpheglngluarg1510<210>163<211>11<212>prt<213>人工序列<220><223>肽<400>163ileprophevalasnphepheaspglyphearg1510<210>164<211>22<212>prt<213>人工序列<220><223>肽<400>164ilegluvalleuglutyrgluglnleualathrleuleuaspargpro151015alaleuaspserphearg20<210>165<211>10<212>prt<213>人工序列<220><223>肽<400>165serglyglyilethrvalserhisleuarg1510<210>166<211>17<212>prt<213>人工序列<220><223>肽<400>166glulysgluileasptyrvalvalalathrleuproproileileasp151015arg<210>167<211>15<212>prt<213>人工序列<220><223>肽<400>167gluileasptyrvalvalalathrleuproproileileasparg151015<210>168<211>15<212>prt<213>人工序列<220><223>肽<400>168ilealavalaspglygluglyalaleuaspmetalaglnphearg151015<210>169<211>21<212>prt<213>人工序列<220><223>肽<400>169gluileilethrservalvalgluhisproalathrleualaalacys151015gluhismetgluarg20<210>170<211>10<212>prt<213>人工序列<220><223>肽<400>170ileaspmetleusercysseralahislys1510<210>171<211>14<212>prt<213>人工序列<220><223>肽<400>171alametasnileprotyrthralaalahisglythrilearg1510<210>172<211>11<212>prt<213>人工序列<220><223>肽<400>172glnvaltyrleuaspasnasnalathrthrarg1510<210>173<211>9<212>prt<213>人工序列<220><223>肽<400>173ileproilealavalglyglnthrarg15<210>174<211>8<212>prt<213>人工序列<220><223>肽<400>174glyvalglycysleutyrleuarg15<210>175<211>21<212>prt<213>人工序列<220><223>肽<400>175valproalaaspthrthrilevalglyleuleuglngluileglnleu151015tyrtrptyrasplys20<210>176<211>13<212>prt<213>人工序列<220><223>肽<400>176glnpheaspmetvalhisseraspglutrpsermetlys1510<210>177<211>14<212>prt<213>人工序列<220><223>肽<400>177valvalasppheservalgluasnglyhisglnthrglulys1510<210>178<211>13<212>prt<213>人工序列<220><223>肽<400>178argalaglyasptyrlysaspaspaspasplysprogly1510<210>179<211>13<212>prt<213>人工序列<220><223>肽<400>179alaargglyasptyrlysaspaspaspasplysprogly1510<210>180<211>11<212>prt<213>人工序列<220><223>肽<400>180hisvalaspglnhispheglyalathrproarg1510<210>181<211>9<212>prt<213>人工序列<220><223>肽<400>181valalaphealaserserasptyrarg15<210>182<211>14<212>prt<213>人工序列<220><223>肽<400>182leuleuglngluglnglutrphisglyaspproaspproarg1510<210>183<211>10<212>prt<213>人工序列<220><223>肽<400>183leualasertrpleuglugluilelysarg1510<210>184<211>16<212>prt<213>人工序列<220><223>肽<400>184valleuglysertrpthrglyaspservalasntyralaalaserarg151015<210>185<211>14<212>prt<213>人工序列<220><223>肽<400>185glnleuglygluleualaaspalaprovalasnileasnarg1510<210>186<211>18<212>prt<213>人工序列<220><223>肽<400>186phevalglyleuvalleuaspglnaspasnglnpheaspglnthrglu151015alaarg当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1