α-酮戊二酸光学探针及其制备方法和应用与流程
本发明涉及α-酮戊二酸的检测探针,更具体地涉及用于检测α-酮戊二酸的光学探针。本发明还涉及上述检测探针的制备方法及其在检测、定量α-酮戊二酸、筛选化合物或药物中的应用。本发明还涉及包含上述检测探针的试剂盒。
背景技术:
α-酮戊二酸(α-kg)是三羧酸循环中的重要中间物质,它是连接能量代谢,谷氨酰胺分解以及脂质合成的重要节点,与细胞的增殖、分化、衰老和死亡等生命活动有着紧密的关系。α-kg对于表观遗传学的调控起着重要作用(careyb.w.,nature,2015,518:413;hwangi.y.,cellmetab,2016,24:494;teslaat.,cellmetab,2016,24:485),它还可以促进蛋白质合成以及骨骼生长(wun.,biomolecules&therapeutics,2016,24:1),同时还具有延缓衰老,延长寿命的作用(inokik.,trendsincellbiology.,2006,16:206;chinr.m.,nature,2014,510:397)。除此之外,α-kg还起到增强免疫系统功能(klyszd.,sciencesignaling,2015,8:ra97)以及机体寒冷耐受性的作用(bayliakm.m.,journalofthermalbiology,2016,60:1)。鉴于α-kg的上述功能,对于α-kg的检测也显得尤为重要。
哺乳动物细胞内α-kg的浓度大约在0.2mm(fanj.,molecularsystemsbiology,2013,9:712),传统的测定α-kg浓度的方法主要有色谱-质谱联用分析(rocchicciolif.,biomedmassspectrom,1984,11:24),同位素比率分析方法(bennettb.d.,natureprotocol,2008,3:1299),核磁共振分析方法(tengr.,nmrinbiomedicine,2009,22:292),虽然上述方法能实现较精确的测量,但是操作步骤繁琐,处理样品的时间较长,不能对样品进行高通量检测,此外,也不能用于活细胞、亚细胞以及活体动物水平α-kg的检测。上述缺点限制了它们在临床疾病诊断及药物前体研究等邻域的应用,并且在一定程度上制约了α-kg相关研究领域的发展。
因此,本领域亟需发展一种高特异性的α-kg检测技术,特别是一种适合细胞水平和亚细胞水平的原位、实时、动态、高通量和高时空分辨率的检测方法。
近年来出现的基因编码光学探针提供了一种全新且方便的测定体内代谢物质的方法。相对于传统的检测技术来说,基因编码的荧光蛋白探针在活细胞以及活体动物的研究中存在很大的优势。首先,其可以对活细胞或者活体动物内代谢物的水平进行实时监测,此外,还可以根据需要定位到细胞的不同亚细胞器结构,不同组织,不同器官之中。基因编码光学探针主要的构建方式包括两种,基于荧光共振能量转移(fret)的构建方式和基于单荧光蛋白(例如cpfps)的构建方式。本技术中采用的构建方式为基于单荧光蛋白的构建方式。
最早发现的荧光蛋白是从维多利亚发光水母(aequoreavictoria)中提取出来的绿色荧光蛋白gfp(seqidno:23),其分子量为26kda,包括238个氨基酸。gfp的荧光来自三肽发色团(ser65-tyr66-gly67),在氧气存在的情况下,其会自发产生荧光,不需要辅因子,而且荧光非常稳定,是一种良好的成像工具。gfp有两个激发峰,395nm的主峰可产生508nm的发射光,而肩峰475nm的激发光照射则会产生的503nm的发射光(heim,r.等procnatlacadsci1994)。随着对gfp蛋白突变的研究不断深入,目前产生了很多不同颜色的突变体,例如黄色荧光蛋白(yfp)、水鸭蓝色荧光蛋白(cfp)、蓝色荧光蛋白(bfp)等。除此之外,科学家还在海洋珊瑚中发现了第一种红色荧光蛋白,经过不断的改造产生了多种商业化的红色荧光蛋白突变体,其中最常用的红色荧光蛋白,一种是mcherry,它的激发峰在587nm,发射峰在621nm(tsienr.y.,naturemethods,2008,5:545);另一种是mkate,它的光谱和mcherry类似(shcherbod.,naturemethods,2007,4:741)。
随着对gfp蛋白突变的研究不断深入,研究者对其进行环状重排,将gfp的原始n端和c端通过一段柔性的短肽链连接,而在野生型gfp近生色团位置(如y144和n145位氨基酸)制造一个新的n端和c端,创造出了对空间构象变化非常敏感的环状排列荧光蛋白(circularlypermutedfluorescentprotein)。目前已经创造了多种环状重排的荧光蛋白(cpfp)用于光学探针的构建,如环状重排蓝色荧光蛋白(cpbfpseqidno:16),环状重排水鸭蓝色荧光蛋白(cptfpseqidno:7),环状重排绿色荧光蛋白(cpgfpseqidno:18),环状重排黄色荧光蛋白(cpyfpseqidno:14),环状重排橙色荧光蛋白(cpmorangeseqidno:20),环状重排苹果红荧光蛋白(cpmappleseqidno:17),环状重排红色荧光蛋白(cpmkateseqidno:21)等(zhao,y.,2011),其中环状排列黄色荧光蛋白cpyfp(cpyfpseqidno:14)在光学探针的构建和应用中非常普遍(nagai,t.,procnatlacadsci,2001)。本文的示例性荧光蛋白包括环状排列的黄色荧光蛋白(cpyfpseqidno:14)及其突变体cpmvenus(seqidno:15)。
本文中,示例性的α-kg的识别元件包括glnk1(seqidno:1)蛋白,其由112个氨基酸组成,分子量大概在12kda左右,通常以三聚体的形式存在,它是来自詹氏甲烷球菌(methanococcusjannaschii)的一种调节细菌内氮代谢的pii蛋白,其在谷氨酰胺合成酶(gs)腺苷化的调节,氮调节基因(ntr)的转录调节中起着至关重要的作用(truan,d.,2010)。
2007年,ozkanyildiz等人揭秘了glnk1结合各种效应分子之后(atp,adp,α-kg)的晶体结构。glnk1蛋白含有三个loop区域,分别为b-loop区,c-loop区和t-loop区。没有结合效应分子的glnk1三聚体的t-loop的结构都是延展的,且该蛋白带正电;在有mg-atp存在的情况下,glnk1三聚体可以结合三个mg-atp,glnk1蛋白的三聚体中的t-loop变成了紧凑的结构,并且蛋白显著的带了负电;在体系中同时加入α-kg和mg-atp之后,glnk1三聚体的三个口袋都结合atp,但是只有一个口袋结合mg-atp,结合mg-atp的t-loop是紧凑的,其他的两个t-loop是松散的,而α-kg只结合在结合了mg-atp的紧凑的t-loop上。α-kg与glnk1的结合依赖于ile52、val53和asp54的主链n和α-kg的结合。在延展的t-loop结构中,α-kg结合位点是不存在的,只有结合了mg-atp之后,α-kg的结合位点才能暴露出来。α-kg与glnk1蛋白的第一个位点结合后会带负电,第二个和第三个位点结合α-kg会受到排斥,因此第二个和第三个位点对α-kg的亲和力会降低20-30倍,这也就是glnk1只有一个位点结合α-kg的原因(yildizo.,embojournal,2007,26:589)。因此,glnk1蛋白是一个很好的细胞内atp,α-kg检测探针的候选者。
发明人发现,荧光蛋白与α-kg的识别元件融合后的光学探针,可以将α-kg的识别元件与α-kg的结合实时、定量且直观的显示出来。本发明的一系列重组荧光蛋白能够满足在哺乳动物细胞水平及亚细胞水平上检测α-kg的迫切需要。
不应认为对本文所述参考文献的引用或讨论意味着承认这些参考文献是本发明的现有技术。
技术实现要素:
本发明的目的在于提供一种在细胞内外实时、原位、高通量、定量检测α-kg的α-kg光学探针。
本发明提供一种光学探针,包含:响应α-酮戊二酸的多肽和光学活性多肽,其中所述光学活性多肽位于所述响应α-酮戊二酸的多肽的序列内。
在一个实施方式中,光学活性多肽是荧光蛋白或其功能片段或变体。在一个实施方式中,荧光蛋白选自蓝色荧光蛋白、水鸭蓝色荧光蛋白、绿色荧光蛋白、黄色荧光蛋白、橘黄色荧光蛋白、苹果红荧光蛋白、红色荧光蛋白。优选地,光学活性多肽是黄色荧光蛋白。在一个实施方式中,荧光蛋白具有seqidno:14-25中任一所示的序列。
在一个实施方式中,响应α-酮戊二酸的多肽包含glnk的t-loop区。在一个实施方式中,响应α-酮戊二酸的多肽源自氮调节蛋白或其功能片段。在一个实施方式中,响应α-酮戊二酸的多肽源自pii蛋白或其功能片段。在一个实施方式中,响应α-酮戊二酸的多肽源自对α-酮戊二酸敏感的pii蛋白或其功能片段。在一个实施方式中,响应α-酮戊二酸的多肽来源于glnk蛋白或其功能片段。在一个实施方式中,响应α-酮戊二酸的多肽选自glnk蛋白或其功能片段。在一个实施方式中,响应α-酮戊二酸的多肽是来自詹氏甲烷球菌的glnk1蛋白或其功能片段。
在一个实施方式中,响应α-酮戊二酸的多肽具有seqidno:1或2所示的序列或其功能片段,或与其有35%、40%、50%、60%、70%、80%、85%、90%、95%、99%序列相同性的序列。
在一个实施方式中,光学探针还包含侧接所述光学活性多肽的一个或多个接头。本发明所述接头可以是任何长度的任何氨基酸序列。在一个实施方式中,光学活性多肽侧翼包含不超过5个氨基酸的接头,例如0、1、2、3、4个氨基酸的接头。在一个实施方式中,光学活性多肽侧翼的接头包含sag或gt。在一个实施方式中,接头sag、gt分别位于光学活性多肽的n端和c端。在一个实施方式中,接头sag、gt分别位于光学活性多肽的c端和n端。在一个实施方式中,光学探针如下所示:glnk1(1-51)-sag-cpyfp-gt-glnk1(52-112)。在一个实施方式中,光学探针如下所示:glnk1(1-51)-sag-cpyfp-gt-glnk1(52-108)。在一个实施方式中,本发明光学探针不包含接头。
在一个实施方式中,本发明光学探针还包含定位序列,用于将探针定位到例如细胞的特定细胞器。本发明所述光学活性多肽可以位于响应α-kg的多肽的任何位置。
在一个实施方式中,光学活性多肽位于所述响应α-酮戊二酸的多肽的残基48-54之间,编号对应于所述响应α-酮戊二酸的多肽的全长。在一个实施方式中,光学活性多肽置换所述响应α-酮戊二酸的多肽的残基48-54之间的一个或多个氨基酸,编号对应于所述响应α-酮戊二酸的多肽的全长。
在一个实施方式中,光学活性多肽插入响应α-酮戊二酸的多肽的下述位点:48/49,48/50,48/51,48/52,48/53,48/54,49/50,49/51,49/52,49/53,49/54,50/51,50/52,50/53,50/54,51/52,51/53,51/54,52/53,52/54或53/54。本文中,如果以“x/y”形式表示的位点中的两个数字是连续的数字,则表示光学活性多肽插入该数字所述的氨基酸之间。例如插入位点52/53表示光学活性多肽插入响应α-酮戊二酸的多肽的氨基酸52与53之间。如果以“x/y”形式表示的位点中的两个数字不是连续的数字,则表示光学活性多肽置换该数字所示氨基酸之间的氨基酸。例如插入位点48/54表示光学活性多肽置换α-酮戊二酸的多肽的氨基酸49-53。优选地,光学活性多肽插入响应α-酮戊二酸的多肽的下述位点:51/52,49/53,49/54,50/51。
在一个实施方式中,本发明光学探针具有下述序列或由其组成:glnk1蛋白的氨基酸序列的氨基酸1-51(seqidno:3)或其变体、黄色荧光蛋白(seqidno:14)或其变体和glnk1蛋白的氨基酸序列的氨基酸52-112(seqidno:4)或氨基酸52-108(seqidno:5)或其变体。
优选地,上述序列以3’-5’的顺序具有下述序列或由其组成:glnk1蛋白的氨基酸序列的氨基酸1-51(seqidno:3)或其变体、黄色荧光蛋白(seqidno:14)或其变体和glnk1蛋白的氨基酸序列的氨基酸52-112(seqidno:4)或氨基酸52-108(seqidno:5)或其变体。
本发明提供的光学探针包含氨基酸序列seqidno:26-29中任一或其变体。在一个实施方式中,本发明提供的光学探针包含与氨基酸序列seqidno:26-29中任一有35%、40%、50%、60%、70%、80%、85%、90%、95%、99%序列相同性的序列。在优选实施方式中,本发明提供的光学探针包含与氨基酸序列seqidno:26-29中任一实质上相似或相同的序列。在更优选的实施方式中,本发明提供的光学探针包含seqidno:26。
在一些实施方式中,光学探针中的响应α-酮戊二酸的多肽包含一个或多个氨基酸突变。所述氨基酸突变包括氨基酸的修饰、取代、缺失或截短。在一个实施方式中,所述突变是v43f和/或e50v。
在一些实施方式中,氨基酸残基发生突变而不改变多肽或蛋白质的总体构型和功能,即功能保守突变。氨基酸突变包括但不限于相似性质的氨基酸置换,所述性质包括极性或非极性特征、尺寸、形状和电荷。对于许多常见已知非遗传性编码氨基酸的保守氨基酸取代本领域已知。其他非编码氨基酸的保守取代可基于其物理性质与遗传上编码的氨基酸的性质的比较来确定。
本发明还提供一种串联光学探针,包含:第一响应α-酮戊二酸的多肽,光学活性多肽,和其中α-酮戊二酸结合位点被删除的其他响应α-酮戊二酸的多肽,其中光学活性多肽位于第一响应α-酮戊二酸的多肽的序列内。在一个实施方式中,串联光学探针可包含一个或多个其中α-酮戊二酸结合位点被删除的其他响应α-酮戊二酸的多肽。在一个实施方式中,本发明提供一种由下述组成的串联光学探针:第一响应α-酮戊二酸的多肽,光学活性多肽,和一个或多个其中α-酮戊二酸结合位点被删除的其他响应α-酮戊二酸的多肽,其中光学活性多肽位于第一响应α-酮戊二酸的多肽的序列内。上述任何串联光学探针任选具有降低蛋白对α-酮戊二酸的结合能力的突变。在一个实施方式中,所述突变是其中α-酮戊二酸结合位点被删除的其他响应α-酮戊二酸的多肽中的突变。在一个实施方式中,插入有光学活性多肽的第一响应α-酮戊二酸的多肽任选具有氨基酸突变。
在一个实施方式中,第一响应α-酮戊二酸的多肽包含glnk的t-loop区。在一个实施方式中,第一响应α-酮戊二酸的多肽源自氮调节蛋白或其功能片段。在一个实施方式中,第一响应α-酮戊二酸的多肽源自pii蛋白或其功能片段。在一个实施方式中,第一响应α-酮戊二酸的多肽源自对α-酮戊二酸敏感的pii蛋白或其功能片段。在一个实施方式中,第一响应α-酮戊二酸的多肽来源于glnk蛋白或其功能片段。在一个实施方式中,第一响应α-酮戊二酸的多肽是glnk蛋白或其功能片段。在一个实施方式中,第一响应α-酮戊二酸的多肽是来自詹氏甲烷球菌的glnk1蛋白或其功能片段。在一个实施方式中,第一响应α-酮戊二酸的多肽可为seqidno:1或2或其功能片段。
在一个实施方式中,其他响应α-酮戊二酸的多肽源自氮调节蛋白或其功能片段。在一个实施方式中,其他响应α-酮戊二酸的多肽源自pii蛋白或其功能片段。在一个实施方式中,其他响应α-酮戊二酸的多肽源自对α-酮戊二酸敏感的pii蛋白或其功能片段。在一个实施方式中,其他响应α-酮戊二酸的多肽来源于glnk蛋白或其功能片段。在一个实施方式中,其他响应α-酮戊二酸的多肽是glnk蛋白或其功能片段。在一个实施方式中,其他响应α-酮戊二酸的多肽是来自詹氏甲烷球菌的glnk1蛋白或其功能片段。在一个实施方式中,其中α-酮戊二酸结合位点被删除的其他响应α-酮戊二酸的多肽可以是glnk1蛋白截短突变体。在一个实施方式中,其中α-酮戊二酸结合位点被删除的其他响应α-酮戊二酸的多肽或其变体的序列包括seqidno:6-13的序列中任一。
在一个实施方式中,第一响应α-酮戊二酸的多肽和其他响应α-酮戊二酸的多肽可源自相同或不同的蛋白。例如,第一响应α-酮戊二酸的多肽和其他响应α-酮戊二酸的多肽均可源自对α-酮戊二酸敏感的pii蛋白或其功能片段。
在串联光学探针包括两个或多个其中α-酮戊二酸结合位点被删除的其他响应α-酮戊二酸的多肽的情况中,所述两个或多个其中α-酮戊二酸结合位点被删除的其他响应α-酮戊二酸的多肽可源自相同或不同的蛋白。在一个实施方式中,两个或多个其中α-酮戊二酸结合位点被删除的其他响应α-酮戊二酸的多肽可位于第一响应α-酮戊二酸的多肽的n端或c端或n端和c端。
本发明还提供一种串联光学探针,包含:第一响应α-酮戊二酸的多肽,光学活性多肽,和一个或多个其中α-酮戊二酸结合位点被删除的第二响应α-酮戊二酸的多肽,其中光学活性多肽位于第一响应α-酮戊二酸的多肽的序列内。在一个实施方式中,本发明提供一种由下述组成的串联光学探针:第一响应α-酮戊二酸的多肽,光学活性多肽,和一个或多个其中α-酮戊二酸结合位点被删除的第二响应α-酮戊二酸的多肽,其中光学活性多肽位于第一响应α-酮戊二酸的多肽的序列内。上述任何串联光学探针任选具有降低蛋白对α-酮戊二酸的结合能力的突变。在一个实施方式中,所述突变是其中α-酮戊二酸结合位点被删除的第二响应α-酮戊二酸的多肽中的突变。在一个实施方式中,插入有光学活性多肽的第一响应α-酮戊二酸的多肽任选具有氨基酸突变。
在一个实施方式中,第一响应α-酮戊二酸的多肽包含glnk的t-loop区。在一个实施方式中,第一响应α-酮戊二酸的多肽源自氮调节蛋白或其功能片段。在一个实施方式中,第一响应α-酮戊二酸的多肽源自pii蛋白或其功能片段。在一个实施方式中,第一响应α-酮戊二酸的多肽源自对α-酮戊二酸敏感的pii蛋白或其功能片段。在一个实施方式中,第一响应α-酮戊二酸的多肽来源于glnk蛋白或其功能片段。在一个实施方式中,第一响应α-酮戊二酸的多肽是glnk蛋白或其功能片段。在一个实施方式中,第一响应α-酮戊二酸的多肽是来自詹氏甲烷球菌的glnk1蛋白或其功能片段。在一个实施方式中,第一响应α-酮戊二酸的多肽可为seqidno:1或2或其功能片段。
第一响应α-酮戊二酸的多肽和第二响应α-酮戊二酸的多肽可源自相同或不同的蛋白。在一个实施方式中,第二响应α-酮戊二酸的多肽源自氮调节蛋白或其功能片段。在一个实施方式中,第二响应α-酮戊二酸的多肽源自pii蛋白或其功能片段。在一个实施方式中,第二响应α-酮戊二酸的多肽源自对α-酮戊二酸敏感的pii蛋白或其功能片段。在一个实施方式中,第二响应α-酮戊二酸的多肽来源于glnk蛋白或其功能片段。在一个实施方式中,第二响应α-酮戊二酸的多肽是glnk蛋白或其功能片段。在一个实施方式中,第二响应α-酮戊二酸的多肽是来自詹氏甲烷球菌的glnk1蛋白或其功能片段。在一个实施方式中,其中α-酮戊二酸结合位点被删除的第二响应α-酮戊二酸的多肽可以是glnk1蛋白截短突变体。在一个实施方式中,其中α-酮戊二酸结合位点被删除的第二响应α-酮戊二酸的多肽的序列包括seqidno:6-13中任一。在一个实施方式中,两个或多个其中α-酮戊二酸结合位点被删除的第二响应α-酮戊二酸的多肽可位于第一响应α-酮戊二酸的多肽的n端或c端或n端和c端。
本发明还提供一种串联光学探针,包含:响应α-酮戊二酸的多肽,其序列中插入有光学活性多肽,和响应α-酮戊二酸的多肽变体,其序列中α-酮戊二酸结合位点被删除。上述串联光学探针可包含一个或多个其中α-酮戊二酸结合位点被删除的响应α-酮戊二酸的多肽变体。在一个实施方式中,本发明提供一种由下述组成的串联光学探针:响应α-酮戊二酸的多肽,其序列中插入有光学活性多肽,和一个或多个响应α-酮戊二酸的多肽变体,其序列中α-酮戊二酸结合位点被删除。上述任何串联光学探针任选具有降低蛋白对α-酮戊二酸的结合能力的突变。在一个实施方式中,所述突变是其中α-酮戊二酸结合位点被删除的响应α-酮戊二酸的多肽变体中的突变。在一个实施方式中,插入有光学活性多肽的响应α-酮戊二酸的多肽任选具有氨基酸突变。
在一个实施方式中,待插入有光学活性多肽的响应α-酮戊二酸的多肽,即在将光学活性多肽纳入之前的响应α-酮戊二酸的多肽,包含glnk的t-loop区。在一个实施方式中,所述响应α-酮戊二酸的多肽源自氮调节蛋白或其功能片段。在一个实施方式中,所述响应α-酮戊二酸的多肽源自pii蛋白或其功能片段。在一个实施方式中,所述响应α-酮戊二酸的多肽源自对α-酮戊二酸敏感的pii蛋白或其功能片段。在一个实施方式中,所述响应α-酮戊二酸的多肽来源于glnk蛋白或其功能片段。在一个实施方式中,所述响应α-酮戊二酸的多肽是glnk蛋白或其功能片段。在一个实施方式中,所述响应α-酮戊二酸的多肽是来自詹氏甲烷球菌的glnk1蛋白或其功能片段。在一个实施方式中,所述响应α-酮戊二酸的多肽可为seqidno:1或2或其功能片段。
在一个实施方式中,其中α-酮戊二酸结合位点被删除的响应α-酮戊二酸的多肽变体源自氮调节蛋白或其功能片段。在一个实施方式中,所述多肽变体源自pii蛋白或其功能片段。在一个实施方式中,所述多肽变体源自对α-酮戊二酸敏感的pii蛋白或其功能片段。在一个实施方式中,所述多肽变体来源于glnk蛋白或其功能片段。在一个实施方式中,所述多肽变体是glnk蛋白或其功能片段。在一个实施方式中,所述多肽变体是来自詹氏甲烷球菌的glnk1蛋白或其功能片段。在一个实施方式中,其中α-酮戊二酸结合位点被删除的响应α-酮戊二酸的多肽变体可以是glnk1蛋白截短突变体。在一个实施方式中,其中α-酮戊二酸结合位点被删除的响应α-酮戊二酸的多肽变体的序列包括seqidno:6-13中任一或其变体。在一个实施方式中,两个或更多个其中α-酮戊二酸结合位点被删除的响应α-酮戊二酸的多肽变体可位于插入有光学活性多肽的响应α-酮戊二酸的多肽的n端或c端或n端和c端。
本发明还提供一种串联光学探针,包含:
第一响应α-酮戊二酸的多肽,
光学活性多肽,和
其中α-酮戊二酸结合位点被删除的第二响应α-酮戊二酸的多肽,
其中光学活性多肽位于第一响应α-酮戊二酸的多肽的序列内。
本发明还提供一种串联光学探针,包含:
第一响应α-酮戊二酸的多肽,
光学活性多肽,
其中α-酮戊二酸结合位点被删除的第二响应α-酮戊二酸的多肽,和
其中α-酮戊二酸结合位点被删除的第三响应α-酮戊二酸的多肽,
其中光学活性多肽位于第一响应α-酮戊二酸的多肽的序列内。上述任何串联光学探针任选具有降低蛋白对α-酮戊二酸的结合能力的突变。在一个实施方式中,所述突变是其中α-酮戊二酸结合位点被删除的第二和/或第三响应α-酮戊二酸的多肽中的突变。在一个实施方式中,插入有光学活性多肽的第一响应α-酮戊二酸的多肽任选具有氨基酸突变。
在一个实施方式中,如上所述的串联光学探针还包含其中α-酮戊二酸结合位点被删除的第四响应α-酮戊二酸的多肽、其中α-酮戊二酸结合位点被删除的第五响应α-酮戊二酸的多肽和/或其中α-酮戊二酸结合位点被删除的第六响应α-酮戊二酸的多肽。在一个实施方式中,第三、第四、第五和/或第六α-酮戊二酸结合位点被删除的第二和/或第三响应α-酮戊二酸的多肽包含突变,所述突变降低蛋白对α-酮戊二酸的结合能力。
在一个实施方式中,光学活性多肽是荧光蛋白或其功能片段或变体。在一个实施方式中,荧光蛋白选自蓝色荧光蛋白、水鸭蓝色荧光蛋白、绿色荧光蛋白、黄色荧光蛋白、橘黄色荧光蛋白、苹果红荧光蛋白、红色荧光蛋白。优选地,光学活性多肽是黄色荧光蛋白。在一个实施方式中,荧光蛋白具有seqidno:2-13中任一所示的序列。
本发明的第一、第二、第三、第四、第五或第六响应α-酮戊二酸的多肽可源自相同或不同的蛋白。在一个实施方式中,第一、第二、第三、第四、第五或第六响应α-酮戊二酸的多肽包含glnk的t-loop区。在一个实施方式中,这些响应α-酮戊二酸的多肽源自氮调节蛋白或其功能片段。在一个实施方式中,这些响应α-酮戊二酸的多肽源自pii蛋白或其功能片段。在一个实施方式中,这些响应α-酮戊二酸的多肽源自对α-酮戊二酸敏感的pii蛋白或其功能片段。在一个实施方式中,这些响应α-酮戊二酸的多肽来源于glnk蛋白或其功能片段。
在一个实施方式中,第一响应α-酮戊二酸的多肽是glnk蛋白或其功能片段。在一个实施方式中,第一响应α-酮戊二酸的多肽是来自詹氏甲烷球菌的glnk1蛋白或其功能片段。在一个实施方式中,第一响应α-酮戊二酸的多肽可为seqidno:1或2或其功能片段。
在一个实施方式中,第二、第三、第四、第五或第六响应α-酮戊二酸的多肽可以是把天然的响应α-kg的多肽上的α-kg结合口袋删除。在一个实施方式中,这些多肽还具有突变,例如0-4个氨基酸的突变。本发明示例性glnk1蛋白截短突变体是把天然的glnk1蛋白上的t-loop区删掉(42-53位),并在38位、39位进行突变(v38a,q39g)。这些突变可以改变glnk1蛋白结合α-kg的性质,例如降低glnk1蛋白对α-kg的结合能力。在一个实施方式中,第二、第三、第四、第五或第六多肽可以是glnk1蛋白截短突变体。在一个实施方式中,第二、第三、第四、第五或第六多肽或其变体的序列包括seqidno:6-13中任一。在一个实施方式中,第二、第三、第四、第五和/或第六响应α-酮戊二酸的多肽可位于第一响应α-酮戊二酸的多肽的n端或c端或n端和c端。
本发明还提供一种串联光学探针,包含:其中插入有光学活性多肽的响应α-酮戊二酸的多肽,和其中α-酮戊二酸结合位点被删除的响应α-酮戊二酸的多肽,所述串联光学探针任选具有降低蛋白对α-酮戊二酸的结合能力的突变。上述串联光学探针可包含一个或多个其中α-酮戊二酸结合位点被删除的其他响应α-酮戊二酸的多肽。在一个实施方式中,本发明提供由下述组成的串联光学探针:其中插入有光学活性多肽的响应α-酮戊二酸的多肽,和一个或多个其中α-酮戊二酸结合位点被删除的响应α-酮戊二酸的多肽,所述串联光学探针任选具有降低蛋白对α-酮戊二酸的结合能力的突变。
在一个实施方式中,响应α-酮戊二酸的多肽包含glnk的t-loop区。在一个实施方式中,响应α-酮戊二酸的多肽源自氮调节蛋白或其功能片段。在一个实施方式中,响应α-酮戊二酸的多肽源自pii蛋白或其功能片段。在一个实施方式中,响应α-酮戊二酸的多肽源自对α-酮戊二酸敏感的pii蛋白或其功能片段。在一个实施方式中,响应α-酮戊二酸的多肽来源于glnk蛋白或其功能片段。在一个实施方式中,响应α-酮戊二酸的多肽是glnk蛋白或其功能片段。在一个实施方式中,响应α-酮戊二酸的多肽是来自詹氏甲烷球菌的glnk1蛋白或其功能片段。
在一个实施方式中,响应α-酮戊二酸的多肽具有seqidno:1或2所示的序列或其功能片段,或与其有35%、40%、50%、60%、70%、80%、85%、90%、95%、99%序列相同性的序列。
在一个实施方式中,串联光学探针包含两个或更多个其中α-酮戊二酸结合位点被删除的响应α-酮戊二酸的多肽。在一个实施方式中,两个或更多个其中α-酮戊二酸结合位点被删除的响应α-酮戊二酸的多肽源自相同的响应α-酮戊二酸的多肽。在一个实施方式中,两个或更多个其中α-酮戊二酸结合位点被删除的响应α-酮戊二酸的多肽源自glnk1蛋白。在一个实施方式中,两个或更多个其中α-酮戊二酸结合位点被删除的响应α-酮戊二酸的多肽源自不同的响应α-酮戊二酸的多肽。在一个实施方式中,两个或更多个其中α-酮戊二酸结合位点被删除的响应α-酮戊二酸的多肽是序列不同的多肽。在一个实施方式中,两个或更多个其中α-酮戊二酸结合位点被删除的响应α-酮戊二酸的多肽是序列相同的多肽。在一个实施方式中,串联光学探针包含两个其中α-酮戊二酸结合位点被删除的第二响应α-酮戊二酸的多肽。
在一个实施方式中,一个或多个其中α-酮戊二酸结合位点被删除的响应α-酮戊二酸的多肽位于其中插入有光学活性多肽的响应α-酮戊二酸的多肽的n端或c端。在一个实施方式中,两个或更多个其中α-酮戊二酸结合位点被删除的响应α-酮戊二酸的多肽位于其中插入有光学活性多肽的响应α-酮戊二酸的多肽的n端或c端或n端和c端。
在一个实施方式中,其中α-酮戊二酸结合位点被删除的响应α-酮戊二酸的多肽可以是把天然的响应α-kg的多肽上的α-kg结合口袋删除。在一个实施方式中,其中α-酮戊二酸结合位点被删除的响应α-酮戊二酸的多肽还具有突变,例如0-4个氨基酸的突变。本发明示例性glnk1蛋白截短突变体是把天然的glnk1蛋白上的t-loop区删掉(42-53位),并在38位、39位进行突变(v38a,q39g)。这些突变可以改变glnk1蛋白结合α-kg的性质,例如降低glnk1蛋白对α-kg的结合能力。
在一个实施方式中,其中α-酮戊二酸结合位点被删除的响应α-酮戊二酸的多肽可以是glnk1蛋白截短突变体。在一个实施方式中,其中α-酮戊二酸结合位点被删除的响应α-酮戊二酸的多肽或其变体的序列包括seqidno:6-13中任一。
在一个实施方式中,串联光学探针还包含一个或多个接头。在一个实施方式中,接头位于光学活性多肽侧翼。在一个实施方式中,接头位于两个响应α-酮戊二酸的多肽之间。在一个实施方式中,接头位于其中插入有光学活性多肽的响应α-酮戊二酸的多肽与一个或多个其中α-酮戊二酸结合位点被删除的响应α-酮戊二酸的多肽之间。在一个实施方式中,接头位于两个或更多个其中α-酮戊二酸结合位点被删除的响应α-酮戊二酸的多肽之间。本发明所述接头可以是任何长度的任何氨基酸序列。在一个实施方式中,本发明所述接头长度不超过4个氨基酸。在一个实施方式中,本发明所述接头长度不超过15个,例如0、1、2、3、4、5、6、7、8、9、10、11、12、13、14或15个氨基酸的接头。在一个实施方式中,接头包括sag、gt、asgggsggggasg或asgggggsggasg。在一个实施方式中,光学活性多肽侧翼包含不超过4个氨基酸的接头。在一个实施方式中,光学活性多肽侧翼的接头包含sag或gt。在一个实施方式中,接头sag、gt分别位于光学活性多肽的n端和c端。在一个实施方式中,接头sag、gt分别位于光学活性多肽的c端和n端。在一个实施方式中,其中插入有光学活性多肽的响应α-酮戊二酸的多肽与其中α-酮戊二酸结合位点被删除的响应α-酮戊二酸的多肽之间包含13个氨基酸的接头,优选为asgggsggggasg或asgggggsggasg。在一个实施方式中,两个或更多个其中α-酮戊二酸结合位点被删除的响应α-酮戊二酸的多肽之间包含13个氨基酸的接头,优选为asgggsggggasg或asgggggsggasg。在一个实施方式中,串联光学探针如下所示:glnk1(1-51)-sag-cpyfp-gt-glnk1(52-112)-asgggsggggasg-glnk1(1-41)-glnk1(54-112)-asgggggsggasg-glnk1(1-41)-glnk1(54-112)。在一个实施方式中,串联光学探针如下所示:glnk1(1-51)-sag-cpyfp-gt-glnk1(52-108)-asgggsggggasg-glnk1(1-41)-glnk1(54-108)-asgggggsggasg-glnk1(1-41)-glnk1(54-112)。在一个实施方式中,串联光学探针如下所示:glnk1(1-51)-sag-cpyfp-gt-glnk1(52-108)-asgggsggggasg-glnk1(1-41)-glnk1(54-108)-asgggggsggasg-glnk1(1-41)-glnk1(54-108)。在一个实施方式中,串联光学探针如下所示:glnk1(1-51)-sag-cpyfp-gt-glnk1(52-108)-asgggsggggasg-glnk1(1-41)-glnk1(54-112)-asgggggsggasg-glnk1(1-41)-glnk1(54-108)。在一个实施方式中,串联光学探针不包含接头。
在一个实施方式中,串联光学探针还包含定位序列,用于将探针定位到例如细胞的特定细胞器。光学活性多肽可以位于响应α-kg的多肽的任何位置。
在一个实施方式中,光学活性多肽位于响应α-酮戊二酸的多肽的残基48-54之间,编号对应于所述响应α-酮戊二酸的多肽的全长。在一个实施方式中,光学活性多肽置换响应α-酮戊二酸的多肽的残基48-54之间的一个或多个氨基酸,编号对应于所述响应α-酮戊二酸的多肽的全长。
在一个实施方式中,光学活性多肽插入响应α-酮戊二酸的多肽的下述位点:48/49,48/50,48/51,48/52,48/53,48/54,49/50,49/51,49/52,49/53,49/54,50/51,50/52,50/53,50/54,51/52,51/53,51/54,52/53,52/54和/或53/54。本文中,如果以“x/y”形式表示的位点中的两个数字是连续的数字,则表示光学活性多肽插入该数字所述的氨基酸之间。例如插入位点52/53表示光学活性多肽插入响应α-酮戊二酸的多肽的氨基酸52与53之间。如果以“x/y”形式表示的位点中的两个数字不是连续的数字,则表示光学活性多肽置换该数字所示氨基酸之间的氨基酸。例如插入位点48/54表示光学活性多肽置换α-酮戊二酸的多肽的氨基酸49-53。
优选地,光学活性多肽插入响应α-酮戊二酸的多肽的下述位点:51/52,49/53,49/54和/或50/51。
在一个实施方式中,本发明光学探针具有下述序列或由其组成:glnk1蛋白的氨基酸序列的氨基酸1-51(seqidno:3)或其变体、黄色荧光蛋白(seqidno:14)或其变体、glnk1蛋白的氨基酸序列的氨基酸52-112(seqidno:4)或氨基酸52-108(seqidno:5)其变体、和两个删除t-loop区域的glnk1蛋白截短突变体(seqidno:6-13)或其变体。优选地,上述序列以3’-5’的顺序具有下述序列或由其组成:glnk1蛋白的氨基酸序列的氨基酸1-51(seqidno:3)或其变体、黄色荧光蛋白(seqidno:14)或其变体、glnk1蛋白的氨基酸序列的氨基酸52-112(seqidno:4)或氨基酸52-108(seqidno:5)或其变体、和两个删除t-loop区域的glnk1蛋白截短突变体(seqidno:6-13)或其变体。
本发明提供的串联光学探针包含氨基酸序列seqidno:30-41中任一或其变体。在一个实施方式中,本发明提供的串联光学探针包含与氨基酸序列seqidno:30-41中任一有35%、40%、50%、60%、70%、80%、85%、90%、95%、99%序列相同性的序列。在一个实施方式中,在优选实施方式中,本发明提供的串联光学探针包含与氨基酸序列seqidno:30-41中任一实质上相似或相同的序列。在另一优选实施方式中,本发明提供的串联光学探针包含与氨基酸序列seqidno:34-41中任一实质上相似或相同的序列。在具体实施方式中,本发明提供的串联光学探针包含seqidno:35、37、39或41中任一或其变体。在更优选的实施方式中,本发明提供的串联光学探针包含seqidno:35。
在一些实施方式中,相对于野生型的响应α-酮戊二酸的多肽或其部分,本发明的各种光学探针中响应α-酮戊二酸的多肽包含一个或多个氨基酸突变。在一些实施方式中,本发明的光学探针中的插入有光学活性多肽的响应α-酮戊二酸的多肽包含一个或多个氨基酸突变。在一些实施方式中,本发明的光学探针中的一个或多个其中α-酮戊二酸结合位点被删除的响应α-酮戊二酸的多肽中的任意包含一个或多个氨基酸突变。在一些实施方式中。本发明的光学探针中的其中插入有光学活性多肽的响应α-酮戊二酸的多肽包含一个或多个氨基酸突变,并且,本发明的光学探针中的一个或多个其中α-酮戊二酸结合位点被删除的响应α-酮戊二酸的多肽中的任意包含一个或多个氨基酸突变。所述氨基酸突变包括氨基酸的修饰、取代、缺失或截短。使氨基酸发生突变的方法本领域已知。
在一些实施方式中,氨基酸残基发生突变而不改变多肽或蛋白质的总体构型和功能,即功能保守突变。氨基酸突变包括但不限于相似性质的氨基酸置换,所述性质包括极性或非极性特征、尺寸、形状和电荷。对于许多常见已知非遗传性编码氨基酸的保守氨基酸取代本领域已知。其他非编码氨基酸的保守取代可基于其物理性质与遗传上编码的氨基酸的性质的比较来确定。
在一些实施方式中,氨基酸突变的位置选自v38、q39、v43和e50。在一个实施方式中,氨基酸突变的位点是v43和/或e50,编号对应于glnk1蛋白的全长。在一个实施方式中,氨基酸突变的位点是v38和/或q39,编号对应于glnk1蛋白的全长。在一个实施方式中,插入有光学活性多肽的响应α-酮戊二酸的多肽中氨基酸突变是v43f和/或e50v。在一个实施方式中,其中α-酮戊二酸结合位点被删除的响应α-酮戊二酸的多肽中氨基酸突变是v38a和/或q39g。
在一些实施方式中,突变的响应α-酮戊二酸的多肽(例如glnk1蛋白)包括与本文的氨基酸序列具有70%、75%、80%、85%、90%、95%或100%相同性的氨基酸序列。
在一些实施方式中,本发明光学探针对α-kg的最高响应达到荧光蛋白的6.7倍以上。
本发明还提供融合多肽,包含本文所述光学探针和其它多肽。在一些实施方式中,本文所述光学探针还包含与之融合的其它多肽。本文所述其他多肽不影响光学探针的性质。在一些实施方式中,其他多肽位于所述光学探针的n端和/或c端。在一些实施方式中,其他多肽包括将光学探针定位到不同细胞器或亚细胞器的多肽、用于纯化的标签或者用于免疫印迹的标签。
本文所述亚细胞器包括细胞浆、线粒体、细胞核、内质网、细胞膜、高尔基体、溶酶体和过氧化物酶体等。在一些实施方式中,用于纯化的标签或者用于免疫印迹的标签包括6组氨酸(6*his)、谷胱甘肽硫转移酶(gst)、flag。
本文所述融合多肽中的光学探针和其它多肽之间可具有接头。在一些实施方式中,该接头为柔性氨基酸组成的短肽链的重复单元,例如ggggs,其数目不超过30个,优选为10-20个。
本发明还提供编码本文所述光学探针或串联光学探针或融合多肽的核酸序列或其互补序列。例如seqidno30:glnk1-cpmvenustandem51/52;seqidno31:glnk1-cpmvenustandem51/5243f;seqidno32:glnk1-cpmvenustandem51/5243f50v;seqidno:33:glnk1-cpyfptandem51/52、seqidno:34:glnk1-cpyfptandem51/5243f、seqidno:35:glnk1-cpyfptandem51/5243f50v。
在一个实施方式中,本发明提供一种核酸序列,包含核苷酸序列seqidno:30-35中任一或其变体。在优选实施方式中,本发明提供一种核酸序列,包含与核苷酸序列seqidno:42-47中任一具有99%、95%、90%、80%、70%或50%相同性的序列。在另一优选实施方式中,本发明提供一种核酸序列,包含与核苷酸序列seqidno:42-47中任一实质上相似或相同的核苷酸序列。
本发明还涉及上述核酸序列的互补序列或其变体,其可包含编码本发明光学探针或融合蛋白的片段、类似物、衍生物、可溶性片段和变体的核酸序列或其互补序列。
本发明中的氨基酸序列和核酸序列优选以分离形式提供,更优选地被纯化至均质。
本发明还提供包含与表达控制序列操作性连接的本发明所述的核酸序列或其互补序列的表达载体,该核酸序列编码本发明所述光学探针或串联光学探针或融合多肽。在一些实施方式中,表达载体选自原核表达载体、真核表达载体和病毒载体。在一些实施方式中,原核表达载体优选由质粒prsetb与本文所述的核酸序列操作性连接得到。在一些实施方式中,表达控制序列包括复制起点、启动子、增强子、操纵子、终止子、核糖体结合位点。
本发明还提供包含本发明所述表达载体的细胞,该表达载体包含与表达控制序列操作性连接的本发明所述的核酸序列或其互补序列。所述细胞表达本文所述光学探针或串联光学探针或融合多肽。
本发明提供制备本文所述光学探针的方法,包括:提供包含表达本文所述光学探针或串联光学探针或融合多肽的载体的细胞,在所述细胞表达的条件下培养所述细胞,和分离光学探针或融合多肽。
在一个实施方式中,制备本文所述光学探针或串联光学探针或融合多肽的方法包括:
a.将本发明表达载体转移到宿主细胞大肠杆菌jm109(de3)菌株中,
b.在适合所述宿主细胞表达的条件下培养所述宿主细胞,
c.由所述宿主细胞利用例如his-tag的表达载体分离光学探针或融合多肽。
本发明还提供检测样品中α-kg的方法,包括:使本文所述光学探针或串联光学探针或融合多肽或如本文所述方法制备的光学探针或串联光学探针或融合多肽与样品接触,和检测光学活性多肽的变化。所述检测可以在体内、体外、亚细胞或原位进行。
本文还提供所述光学探针或串联光学探针或融合多肽或如本文所述方法制备的光学探针或串联光学探针或融合多肽在检测样品中的α-kg中的用途。所述检测可以在体内、体外、亚细胞或原位进行。
本发明还提供筛选化合物的方法,包括:使本文所述光学探针或串联光学探针或融合多肽或如本文所述方法制备的光学探针或串联光学探针或融合多肽与候选化合物接触,检测光学活性多肽的变化,和根据光学活性多肽的变化筛选化合物。
本发明还提供本文所述光学探针或串联光学探针或融合多肽或如本文所述方法制备的光学探针或串联光学探针或融合多肽在高通量化合物筛选中的应用。
本发明还提供定量样品中α-kg的方法,包括:使本文所述光学探针或串联光学探针或融合多肽或如本文所述方法制备的光学探针或串联光学探针或融合多肽与样品接触,检测光学活性多肽的变化,和根据光学活性多肽的变化定量样品中的α-kg。
本发明还提供本文所述光学探针或串联光学探针或融合多肽或如本文所述方法制备的光学探针或串联光学探针或融合多肽在定量样品中α-kg中的应用。
本发明还提供筛选药物的方法,包括:使本文所述光学探针或串联光学探针或融合多肽或如本文所述方法制备的光学探针或串联光学探针或融合多肽与候选药物接触,检测光学活性多肽的变化,和根据光学活性多肽的变化筛选药物。
本发明还提供了检测和/或定量α-kg的试剂盒,其中包含本文所述光学探针或串联光学探针或融合多肽或如本文所述方法制备的光学探针或串联光学探针或融合多肽。所述检测和/或定量可以在体内、体外、亚细胞或原位进行。在一个实施方式中,本发明试剂盒可用于定位检测。
本发明还提供通过检测和/或定量α-kg来筛选化合物的试剂盒,其中包含本文所述光学探针或串联光学探针或融合多肽或如本文所述方法制备的光学探针或串联光学探针或融合多肽。
本发明的有益效果:本发明所述的光学探针易于成熟,荧光动态变化大,特异性好,并且能够通过基因操作的方法在细胞中表达,可在细胞内外实时定位、高通量、定量检测α-kg,省去了耗时的处理样品步骤。实验效果表明本申请所提供的α-kg光学探针对α-kg的最高响应达到6.7倍以上,可以在细胞浆、线粒体、细胞核、内质网、细胞膜、高尔基体、溶酶体和过氧化物酶体等亚细胞结构中对细胞进行定位检测;并且可以进行高通量的化合物筛选。
附图说明
下面结合附图和实施例对本发明作进一步说明。
图1为实施例2所述的α-kg光学探针的sds-page分析图;
图2为实施例2所述的四种荧光蛋白在glnk1蛋白不同插入位点形成的α-kg光学探针对α-kg的响应变化图;
图3为实施例3所述的α-kg串联光学探针的sds-page分析图;
图4为实施例3所述的四种荧光蛋白在glnk1蛋白不同插入位点形成的α-kg串联光学探针对α-kg的响应变化图;
图5为实施例4所述的定点突变的α-kg串联光学探针对α-kg的响应变化图;
图6为实施例5所述的α-kg串联光学探针的荧光光谱性质图;
图7为实施例5所述的α-kg串联光学探针对不同浓度α-kg的滴定曲线;
图8为实施例6所述的α-kg串联光学探针在哺乳动物细胞中亚细胞器定位分布图;
图9为实施例7所述的α-kg光学探针在哺乳动物细胞中的实时检测;
图10为实施例8所述的在活细胞水平基于α-kg光学探针进行高通量化合物筛选分析图;
图11为实施例9所述的α-kg光学探针检测活细胞中α-kg的定量分析图。
具体实施方式
i.定义:
在给出数值或范围时,本文所用术语“约”指该数值或范围在给定数值或范围的20%以内、10%以内和5%以内。
本文所用术语“包含”、“包括”和其等同形式包括“含有”以及“由……组成”的含义,例如“包含”x的组合物可仅由x组成或可含有其它物质,例如x+y。
本文所用术语“响应α-kg的多肽”或“响应多肽”指对α-kg产生响应的多肽,所述响应包括与响应多肽的相互作用相关的多肽的化学,生物学,电学或生理学参数的任何响应。响应包括小的变化,例如,响应多肽的氨基酸或肽片段的方向的变化以及例如多肽的一级,二级或三级结构的变化,包括例如质子化,电化学势和/或构象的变化。“构象”是分子中包含侧基的分子的一级,二级和三级结构的三维排列;当分子的三维结构发生变化时,构象发生变化。构象变化的实例包括从α-螺旋转变为β-折叠或从β-折叠转变为α-螺旋。可以理解的是,只要荧光蛋白部分的荧光被改变,可检测到的改变不需要是构象改变。
本发明所述响应α-kg的多肽包括但不限于“glnk蛋白”或与glnk1蛋白有90%以上同源性的其他pii蛋白突变体。本发明所述示例性glnk1蛋白来源于詹氏甲烷球菌(methanococcusjannaschii),含有glnk蛋白所具有的典型的t-loop结构,在mg-atp存在的情况下可以结合α-kg。glnk1蛋白可以响应细胞内α-kg浓度的变化,在α-kg浓度动态变化的过程中glnk1蛋白的空间构象也会发生很大改变。示例性glnk1蛋白如seqidno:1所示。示例性glnk1蛋白的变体为seqidno:2。
本文所用术语删除α-kg结合位点的响应α-kg的多肽是指把天然的响应α-kg的多肽上的α-kg结合口袋删除,并任选对其进行突变,氨基酸的突变个数在0-4之间。优选这些突变可以改变glnk1蛋白结合α-kg的性质,降低glnk1蛋白对α-kg的结合能力。本发明所示示例性glnk1蛋白截短突变体是把天然的glnk1蛋白上的t-loop区删掉,并在38位、39位发生了突变。示例性的glnk1蛋白截短突变体或其变体如seqidno:6-13中任一所示。
本文所用术语“截短”指的是采用分子克隆的方法将编码光学探针蛋白的部分氨基酸序列的核苷酸序列进行缩短或者删除的操作。截短可以直接将蛋白的一个结构域(例如glnk1蛋白的t-loop区)缩短或者删除。截短主要通过反向pcr的分子操作方法来实现,即通过一对特异性的引物与截短区域外的蛋白序列两端匹配,这样通过反向pcr扩增就可以产生删除某段核苷酸的线性化质粒,之后形成的重组质粒编码产生的蛋白就不再含有特定的氨基酸。这种截短方法在荧光蛋白探针的构建中很常用,适用于探针的性质优化和改造。
本文所用术语“光学探针”是指与荧光蛋白融合的响应α-kg的多肽,所述响应α-kg的多肽具体可以是glnk蛋白,其利用与α-kg结合后引起的荧光蛋白的构象变化,进而导致荧光蛋白的荧光发生改变,并借助不同α-kg浓度下测定的荧光蛋白的荧光绘制标准曲线,进而检测并分析α-kg的存在和/或水平。
在本发明的光学探针中,光学活性多肽(例如荧光蛋白)可操作地插入响应α-kg的多肽中。基于蛋白质的“光学活性多肽”是具有发射荧光能力的多肽。荧光是光学活性多肽的一种光学性质,其可用作检测本发明的光学探针或响应多肽的响应性的手段。如本文所用,术语“荧光性质”是指适当激发波长下的摩尔消光系数,荧光量子效率,激发光谱或发射光谱的形状,激发波长最大值和发射波长最大值,两个不同波长激发的振幅,两个不同波长的发射振幅比,激发态寿命或荧光各向异性。
活性和无活性状态之间的这些性质中的任何一个的可测量的差异足以用于本发明的荧光蛋白底物在活性测定中的效用。可测量的差异可通过确定任何定量荧光性质的量来确定,例如,特定波长处的荧光量或荧光在发射光谱上的积分。优选地,选择蛋白质底物以具有在未激活和活化的构象状态下容易区分的荧光特性。
“接头”或“连接区”指在本发明多肽、蛋白质或核酸中连接两个部分的氨基酸或核苷酸序列。本发明中glnk1与荧光蛋白的连接区氨基端的氨基酸数目选择的是3个,羧基端的氨基酸数目选择的是2个;当glnk1-荧光蛋白结构作为基本单元,串联两个glnk1蛋白截短突变体时,两个连接区的氨基酸数目选择的都是13个;而当重组荧光蛋白探针作为基本单元和功能蛋白连接时,可以融合在重组荧光蛋白探针的氨基酸或羧基端;接头序列为柔性氨基酸组成的短肽链的重复单元,如ggggs,其数目不超过30个,优选为10-20个。
本文所用术语“生色团”,“荧光团”与“荧光蛋白”同义,指在激发光照射下发出荧光的蛋白质。荧光蛋白作为生物科学领域的基础检测手段,例如生物技术领域常用的绿色荧光蛋白gfp及由该蛋白突变衍生出的环状重排的蓝色荧光蛋白(cpbfp)、环状重排的绿色荧光蛋白(cpgfp)、环状重排的黄色荧光蛋白(cpyfp)等;还有本技术领域常用的红色荧光蛋白rfp,及由该蛋白衍生出来的环状重排的蛋白,如cpmapple,cpmorange,cpmkate等。示例性荧光蛋白的序列如seqidno:14-25中任一所示。
本文所用术语“gfp”指绿色荧光蛋白,最初是从维多利亚发光水母(aequoreavictoria)中提取出来的,由238个氨基酸构成,分子量约为26kda。gfp是由12条β-折叠链形成了独特的桶状结构,其内包裹着生色三肽(ser65-tyr66-gly67)。当在氧气存在下,它会自发形成对-羟基苯亚甲基咪唑啉酮的生色团结构而产生荧光。gfp产生荧光不需要辅因子,而且荧光非常稳定,是一种良好的成像工具。gfp有两个激发峰,395nm的主峰可产生508nm的发射光,而肩峰475nm的激发光照射则会产生的503nm的发射光。
本文所用术语“yfp”指黄色荧光蛋白,该蛋白衍生自绿色荧光蛋白gfp,其氨基酸序列与gfp同源性高达90%以上,yfp相比于gfp关键改变在于第203位氨基酸由苏氨酸突变为酪氨酸(t203y)。相比于原始的avgfp,yfp的主激发峰的波长红移至514nm而发射波长则改变为527nm。在此基础上对yfp第65位氨基酸进行定点突变(s65t)可获得荧光增强型黄色荧光蛋白eyfp。
本文所用术语“rfp”指红色荧光蛋白,最初是从海洋中的珊瑚中提取的,野生的rfp是寡聚体蛋白不利于生物体的融合表达,随后在rfp的基础上进一步衍生出了不同颜色波段的红色荧光蛋白,其中最常用的是mcherry和mkate等。
本文所用术语“cpfp”指环状重排的荧光蛋白,该蛋白最早衍生自绿色荧光蛋白gfp,是将gfp的原始n端和c端通过一段柔性的短肽链连接,而在野生型gfp近生色团位置(如y144和n145位氨基酸)制造一个新的n端和c端,将原第145-238位氨基酸部分作为新蛋白的n端,原第1-144位氨基酸作为新蛋白的c端,两片段间通过5-9个具有柔性的短肽链,如vdggsggtg或ggsgg等连接,形成一个对空间变化敏感的环状排列绿色荧光蛋白cpgfp(seqidno:6),其氨基酸序列与gfp同源性高达90%以上。目前已经创造了多种环状重排的荧光蛋白(cpfp)用于光学探针的构建,其中应用最广泛一种是cpyfp,其氨基酸序列为seqidno:14。
在本发明其他实施方式中,所述荧光蛋白还可以为氨基酸序列如seqidno:6或seqidno:23所示绿色荧光蛋白、氨基酸序列如seqidno:19所示水鸭蓝色荧光蛋白cptfp、氨基酸序列如seqidno:16或seqidno:22所示蓝色荧光蛋白、氨基酸序列如seqidno:20所示橘黄色荧光蛋白、氨基酸序列如seqidno:17所示苹果红荧光蛋白、氨基酸序列如seqidno:21或seqidno:25所示红色荧光蛋白cpmkate和氨基酸序列如seqidno:24所示红色荧光蛋白mcherry中的一种或多种。
在本发明的光学探针中,荧光蛋白或其功能片段或变体可以位于响应多肽的任何位置。在一个实施方式中,荧光蛋白或其功能片段或变体以n-c方向位于n-c方向的响应多肽的任何位置。具体地,荧光蛋白或其功能片段或变体位于响应多肽的柔性区域,所述的柔性区域是指蛋白质高级结构中存在的一些特定的如环状结构域等结构,这些结构域相比于蛋白质的其他高级结构具有更高的移动性和柔性,并且该区域可以在该蛋白质和配体结合后,空间结构构象发生动态变化。本发明中所述的柔性区域可为响应α-kg的多肽中的插入位点所在区域。
光学探针可在荧光蛋白两端包含接头。在一个实施方式中,光学探针可在荧光蛋白两端包含0-15个氨基酸的接头,例如0、1、2、3、4、5、6、7、8、9、10、11、12、13、14或15个氨基酸的接头。在一个实施方式中,光学探针可在荧光蛋白两端包含0-4个氨基酸的接头。在一个实施方式中,荧光蛋白两端的接头包含sag或gt。
例如,seqidno:14所示的cpyfp可以插入seqidno:1或2所示的glnk蛋白的氨基酸48/49、48/50、48/51、48/52、48/53、48/54、49/50、49/51、49/52、49/53、49/54、50/51、50/52、50/53、50/54、51/52、51/53、51/54、52/53、52/54或53/54位点。示例性地,seqidno:26示出了seqidno:14所示的cpyfp插入seqidno:2所示的glnk蛋白的氨基酸51/52位点并包含接头的光学探针的序列。本领域技术人员可以相同的方式获得cpyfp插入上述其他位点的光学探针的氨基酸序列。
例如,seqidno:15所示的cpmvenus可以插入seqidno:1或2所示的glnk蛋白的氨基酸48/49、48/50、48/51、48/52、48/53、48/54、49/50、49/51、49/52、49/53、49/54、50/51、50/52、50/53、50/54、51/52、51/53、51/54、52/53、52/54或53/54位点。示例性地,seqidno:27示出了seqidno:15所示的cpmvenus插入seqidno:2所示的glnk蛋白的氨基酸51/52位点并包含接头的光学探针的序列。本领域技术人员可以相同的方式获得cpmvenus插入上述其他位点的光学探针的氨基酸序列。
例如,seqidno:16所示的cpbfp可以插入seqidno:1或2所示的glnk蛋白的48/49、48/50、48/51、48/52、48/53、48/54、49/50、49/51、49/52、49/53、49/54、50/51、50/52、50/53、50/54、51/52、51/53、51/54、52/53、52/54或53/54位点。示例性地,seqidno:28示出了seqidno:16所示的cpbfp插入seqidno:2所示的glnk蛋白的氨基酸51/52位点并包含接头的光学探针的序列。本领域技术人员可以相同的方式获得cpbfp插入上述其他位点的光学探针的氨基酸序列。
例如,seqidno:17所示的cpmapple可以插入seqidno:1或2所示的glnk蛋白的48/49、48/50、48/51、48/52、48/53、48/54、49/50、49/51、49/52、49/53、49/54、50/51、50/52、50/53、50/54、51/52、51/53、51/54、52/53、52/54或53/54位点。示例性地,seqidno:29示出了seqidno:17所示的cpmapple插入seqidno:2所示的glnk蛋白的氨基酸51/52位点并包含接头的光学探针的序列。本领域技术人员可以相同的方式获得cpmapple插入上述其他位点的光学探针的氨基酸序列。
本文所用术语“串联光学探针”是指包含第一部分和第二部分的光学探针,第一部分是其中插入有光学活性多肽(例如荧光蛋白或其功能片段或变体)的响应α-酮戊二酸的多肽,第二部分是一个或多个其中α-酮戊二酸结合位点被删除的响应α-酮戊二酸的多肽。示例性的第二部分可以是一个或多个glnk1蛋白截短突变体。
如上所述,荧光蛋白或其功能片段或变体可以位于响应多肽的任何位置。在一个实施方式中,荧光蛋白或其功能片段或变体以n-c方向位于n-c方向的响应多肽的任何位置。具体地,荧光蛋白或其功能片段或变体位于响应多肽的柔性区域。本发明中所述的柔性区域可为响应α-kg的多肽中的插入位点所在区域。串联光学探针可在荧光蛋白两端包含接头。在一个实施方式中,光学探针可在荧光蛋白两端包含0-15个氨基酸的接头,例如0、1、2、3、4、5、6、7、8、9、10、11、12、13、14或15个氨基酸的接头。在一个实施方式中,光学探针可在荧光蛋白两端包含0-4个氨基酸的接头。在一个实施方式中,荧光蛋白两端的接头包含sag或gt。
串联光学探针中的第二部分可以位于第一部分的任何位置。在一个实施方式中,串联光学探针中的第二部分可以位于第一部分的n和/或c端。在一个实施方式中,串联光学探针中的第二部分可以位于第一部分的c端。串联光学探针可在第一部分和第一部分之间包含接头。在一个实施方式中,串联光学探针可在第一部分和第一部分之间包含0-15个氨基酸的接头,例如0、1、2、3、4、5、6、7、8、9、10、11、12、13、14或15个氨基酸的接头。在一个实施方式中,串联光学探针可在第一部分和第一部分之间包含13个氨基酸的接头。在一个实施方式中,串联光学探针可在第一部分和第一部分之间包含接头:asgggsggggasg或asgggggsggasg。
例如,seqidno:14所示的cpyfp可以插入seqidno:1或2所示的glnk蛋白的氨基酸48/49、48/50、48/51、48/52、48/53、48/54、49/50、49/51、49/52、49/53、49/54、50/51、50/52、50/53、50/54、51/52、51/53、51/54、52/53、52/54或53/54位点。所得序列的c端融合有例如seqidno:6-13所示的glnk1蛋白截短突变体。示例性地,seqidno:30示出了将seqidno:14所示的cpyfp插入seqidno:2所示的glnk蛋白的氨基酸51/52位点,在所得序列的c端融合有seqidno:11和seqidno:7所示的glnk1蛋白截短突变体,并包含接头的串联光学探针的序列。本领域技术人员可以相同的方式获得cpyfp插入上述其他位点的串联光学探针的氨基酸序列。
例如,seqidno:15所示的cpmvenus可以插入seqidno:1或2所示的glnk蛋白的氨基酸48/49、48/50、48/51、48/52、48/53、48/54、49/50、49/51、49/52、49/53、49/54、50/51、50/52、50/53、50/54、51/52、51/53、51/54、52/53、52/54或53/54位点。所得序列的c端融合有例如seqidno:6-13所示的glnk1蛋白截短突变体。示例性地,seqidno:31示出了将seqidno:15所示的cpmvenus插入seqidno:2所示的glnk蛋白的氨基酸51/52位点,在所得序列的c端融合有seqidno:11和seqidno:7所示的glnk1蛋白截短突变体,并包含接头的串联光学探针的序列。本领域技术人员可以相同的方式获得cpmvenus插入上述其他位点的串联光学探针的氨基酸序列。
例如,seqidno:16所示的cpbfp可以插入seqidno:1或2所示的glnk蛋白的氨基酸48/49、48/50、48/51、48/52、48/53、48/54、49/50、49/51、49/52、49/53、49/54、50/51、50/52、50/53、50/54、51/52、51/53、51/54、52/53、52/54或53/54位点。所得序列的c端融合有例如seqidno:6-13所示的glnk1蛋白截短突变体。示例性地,seqidno:32示出了将seqidno:16所示的cpbfp插入seqidno:2所示的glnk蛋白的氨基酸51/52位点,在所得序列的c端融合有seqidno:11和seqidno:7所示的glnk1蛋白截短突变体,并包含接头的串联光学探针的序列。本领域技术人员可以相同的方式获得cpbfp插入上述其他位点的串联光学探针的氨基酸序列。
例如,seqidno:17所示的cpmapple可以插入seqidno:1所示的glnk蛋白的氨基酸48/49、48/50、48/51、48/52、48/53、48/54、49/50、49/51、49/52、49/53、49/54、50/51、50/52、50/53、50/54、51/52、51/53、51/54、52/53、52/54或53/54位点。所得序列的c端融合有例如seqidno:6-13所示的glnk1蛋白截短突变体。示例性地,seqidno:33示出了将seqidno:17所示的cpmapple插入seqidno:2所示的glnk蛋白的氨基酸51/52位点,在所得序列的c端融合有seqidno:11和seqidno:7所示的glnk1蛋白截短突变体,并包含接头的串联光学探针的序列。本领域技术人员可以相同的方式获得cpmapple插入上述其他位点的串联光学探针的氨基酸序列。
本发明的光学探针可为具有突变的变体。提到某多肽或蛋白时,本发明所用术语“变体”,“突变体”或“衍生突变体”包括具有所述多肽或蛋白相同功能、但序列不同的变体。这些变体包括(但并不限于):在所述多肽或蛋白的序列中缺失、插入和/或取代一个或多个(通常为1-30个,较佳地1-20个,更佳地1-10个,最佳地1-5个)氨基酸,以及在其羧基末端和/或氨基末端添加一个或数个(通常为20个以内,较佳地为10个以内,更佳地为5个以内)氨基酸获得的序列。例如,在本领域中,用性能相近或相似的氨基酸进行取代时,通常不会改变多肽或蛋白的功能。在本领域中,性能相似的氨基酸往往指具有相似侧链的氨基酸家族,在本领域已有明确定义。这些家族包括具有碱性侧链的氨基酸(例如赖氨酸、精氨酸、组氨酸)、具有酸性侧链的氨基酸(例如天冬氨酸、谷氨酸)、具有不带电荷的极性侧链的氨基酸(例如甘氨酸、天冬酰胺、谷氨酰胺、丝氨酸、苏氨酸、酪氨酸、半胱氨酸)、具有非极性侧链的氨基酸(例如丙氨酸、缬氨酸、亮氨酸、异亮氨酸、脯氨酸、苯丙氨酸、甲硫氨酸、色氨酸)、具有β-分支侧链的氨基酸(例如苏氨酸、缬氨酸、异亮氨酸)和具有芳香侧链的氨基酸(例如酪氨酸、苯丙氨酸、色氨酸、组氨酸)。又比如,在氨基末端和/或羧基末端添加一个或数个氨基酸通常也不会改变多肽或蛋白的功能。本领域技术人员公知,在基因克隆操作中,常常需要设计合适的酶切位点,这势必在所表达的多肽或蛋白末端引入了一个或多个不相干的残基,而这并不影响目的多肽或蛋白的活性。又如为了构建融合蛋白、促进重组蛋白的表达、获得自动分泌到宿主细胞外的重组蛋白、或利于重组蛋白的纯化,常常需要将一些氨基酸添加至重组蛋白的n-末端、c-末端或该蛋白内的其它合适区域内,例如,包括但不限于,适合的接头肽、信号肽、前导肽、末端延伸、谷胱甘肽s-转移酶(gst)、麦芽糖e结合蛋白、蛋白a、如6his或flag的标签,或xa因子或凝血酶或肠激酶的蛋白水解酶位点。多肽或蛋白的变体可包括:同源序列、保守性变体、等位变体、天然突变体、诱导突变体。这些变体还可包含与所述多肽或蛋白的序列相同性为至少约70%、至少约75%、至少约80%、至少约85%、至少约90%、至少约95%、至少约98%、至少约99%或100%的多肽或蛋白。
具有突变的串联光学探针可具有seqidno:34-41中任一所示的序列。例如,如seqidno:34所示的序列为:seqidno:14所示的cpyfp插入seqidno:2所示的glnk蛋白的51/52之间,其中glnk蛋白在氨基酸43处突变为f,并在glnk蛋白的c端融合有seqidno:11和seqidno:7所示的glnk1蛋白截短突变体。
例如,如seqidno:35所示的序列为:seqidno:14所示的cpyfp插入seqidno:2所示的glnk蛋白的51/52之间,其中glnk蛋白在氨基酸43处突变为f和在氨基酸50处突变为v,并在glnk蛋白的c端融合有seqidno:11和seqidno:7所示的glnk1蛋白截短突变体。
例如,如seqidno:36所示的序列为:seqidno:15所示的cpmvenus插入seqidno:2所示的glnk蛋白的51/52之间,其中glnk蛋白在氨基酸43处突变为f,并在glnk蛋白的c端融合有seqidno:11和seqidno:7所示的glnk1蛋白截短突变体。
例如,如seqidno:37所示的序列为:seqidno:15所示的cpmvenus插入seqidno:2所示的glnk蛋白的51/52之间,其中glnk蛋白在氨基酸43处突变为f和在氨基酸50处突变为v,并在glnk蛋白的c端融合有seqidno:11和seqidno:7所示的glnk1蛋白截短突变体。
例如,如seqidno:38所示的序列为:seqidno:16所示的cpbfp插入seqidno:2所示的glnk蛋白的51/52之间,其中glnk蛋白在氨基酸43处突变为f,并在glnk蛋白的c端融合有seqidno:11和seqidno:7所示的glnk1蛋白截短突变体。
例如,如seqidno:39所示的序列为:seqidno:16所示的cpbfp插入seqidno:2所示的glnk蛋白的51/52之间,其中glnk蛋白在氨基酸43处突变为f和在氨基酸50处突变为v,并在glnk蛋白的c端融合有seqidno:11和seqidno:7所示的glnk1蛋白截短突变体。
例如,如seqidno:40所示的序列为:seqidno:17所示的cpmapple插入seqidno:2所示的glnk蛋白的51/52之间,其中glnk蛋白在氨基酸43处突变为f,并在glnk蛋白的c端融合有seqidno:11和seqidno:7所示的glnk1蛋白截短突变体。
例如,如seqidno:41所示的序列为:seqidno:17所示的cpmapple插入seqidno:2所示的glnk蛋白的51/52之间,其中glnk蛋白在氨基酸43处突变为f和在氨基酸50处突变为v,并在glnk蛋白的c端融合有seqidno:11和seqidno:7所示的glnk1蛋白截短突变体。
在两种或多种多肽或核酸分子序列中,术语“相同性”或“相同性百分数”指在比较窗口或指定区域上,采用本领域已知方法如序列比较算法,通过手工比对和目测检查来比较和比对最大对应性时,两个或多个序列或子序列相同或其中在指定区域有一定百分数的氨基酸残基或核苷酸相同(例如,60%、65%、70%、75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%相同)。例如,适合测定序列相同性百分数和序列相似性百分数的优选算法是blast和blast2.0算法,分别可参见altschul等(1977)nucleicacidsres.25:3389和altschul等(1990)j.mol.biol.215:403。
本文所用术语“功能片段”、“衍生物”、“突变体”和“类似物”是指基本上保持与原始多肽或蛋白(例如glnk1蛋白或荧光蛋白)相同的生物学功能或活性的蛋白。本发明的多肽或蛋白(例如glnk1蛋白或荧光蛋白)的功能片段、衍生物、突变体或类似物可以是(i)有一个或多个保守或非保守性氨基酸残基(优选保守性氨基酸残基)被取代的蛋白,而这样的取代的氨基酸残基可以是也可以不是由遗传密码编码的,或(ii)在一个或多个氨基酸残基中具有取代基团的蛋白,或(iii)成熟蛋白与另一个化合物(比如延长蛋白半衰期的化合物,例如聚乙二醇)融合所形成的蛋白,或(iv)附加的氨基酸序列融合到此蛋白序列而形成的蛋白(如前导序列或分泌序列或用来纯化此蛋白的序列或蛋白原序列,或与抗原igg片段的形成的融合蛋白)。根据本文的教导,这些功能片段、衍生物和类似物属于本领域熟练技术人员公知的范围。
所述类似物与原始多肽或蛋白的差别可以是氨基酸序列上的差异,也可以是不影响序列的修饰形式上的差异,或者兼而有之。这些蛋白包括天然或诱导的遗传变体。诱导变体可以通过各种技术得到,如通过辐射或暴露于诱变剂而产生随机诱变,还可通过定点诱变法或其他已知分子生物学的技术得到。
所述类似物还包括具有不同于天然l-氨基酸的残基(如d-氨基酸)的类似物,以及具有非天然存在的或合成的氨基酸(如β、γ-氨基酸)的类似物。应理解,本发明的glnk蛋白并不限于上述列举的代表性蛋白、片段、衍生物和类似物。修饰(通常不改变一级结构)形式包括:体内或体外的蛋白的化学衍生形式如乙酰化或羧基化。修饰还包括糖基化,如那些在蛋白的合成和加工中或进一步加工步骤中进行糖基化修饰而产生的蛋白。这种修饰可以通过将蛋白暴露于进行糖基化的酶(如哺乳动物的糖基化酶或去糖基化酶)而完成。修饰形式还包括具有磷酸化氨基酸残基(如磷酸酪氨酸,磷酸丝氨酸,磷酸苏氨酸)的序列。还包括被修饰从而提高了其抗蛋白水解性能或优化了溶解性能的蛋白。
本发明所用术语“核酸”可以是dna形式或rna形式。dna形式包括cdna、基因组dna或人工合成的dna。dna可以是单链的或是双链的。dna可以是编码链或非编码链。
提到核酸时,本文所用术语“变体”可以是天然发生的等位变体或非天然发生的变体。这些核苷酸变体包括简并变体、取代变体、缺失变体和插入变体。如本领域所知的,等位变体是一个核酸的替换形式,它可能是一个或多个核苷酸的取代、缺失或插入,但不会从实质上改变其编码的蛋白的功能。本发明核酸可包含与所述核酸序列的序列相同性为至少约70%、至少约75%、至少约80%、至少约85%、至少约90%、至少约95%、至少约98%、至少约99%或100%的核苷酸序列。
如此处所用,术语“在严谨条件下杂交”是用来描述典型的相互间至少60%同源的核苷酸序列仍可相互杂交的杂交和清洗条件。优选的,严谨条件为这样的条件,在此条件下相互间有至少65%、更优的至少70%、且甚至更优选的至少80%或更高同源性的序列一般仍可相互杂交。此严谨条件为本领域普通技术人员所公知。严谨条件的一个优选,非限制性实例为:(1)在较低离子强度和较高温度下的杂交和洗脱,如0.2×ssc,0.1%sds,0℃;或(2)杂交时加有变性剂,50%(v/v)甲酰胺,0.1%小牛血清/0.1%ficoll,42℃等;或(3)仅在两条序列之间的相同性至少在90%以上,更好是95%以上时才发生杂交。
本发明还涉及与上述的序列杂交的核酸片段。如本文所用,“核酸片段”的长度至少含15个核苷酸,较好是至少30个核苷酸,更好是至少50个核苷酸,最好是至少100个核苷酸以上。核酸片段可用于核酸的扩增技术(如pcr)。
本发明光学探针或融合蛋白的全长序列或其片段通常可以用pcr扩增法、重组法或人工合成的方法获得。对于pcr扩增法,可根据本发明所公开的有关核苷酸序列,尤其是开放阅读框序列来设计引物,并用市售的cdna库或按本领域技术人员已知的常规方法所制备的cdna库作为模板,扩增而得有关序列。当序列较长时,常常需要进行两次或多次pcr扩增,然后再将各次扩增出的片段按正确次序拼接在一起。
可以用重组法来大批量地获得有关序列。这通常是将其克隆入载体,再转入细胞,然后通过常规方法从增殖后的宿主细胞中分离和纯化得到有关多肽或蛋白。
此外,还可用人工合成的方法来合成有关序列,尤其是片段长度较短时。通常,通过先合成多个小片段,然后再进行连接可获得序列很长的片段。
目前,已经可以完全通过化学合成来得到编码本发明蛋白(或其片段、衍生物、类似物或变体)的dna序列。然后可将该dna序列引入本领域中已知的各种现有的dna分子(如载体)和细胞中。可通过突变pcr或化学合成等方法将突变引入本发明蛋白序列中。
本文所用的术语“表达载体”和“重组载体”可互换使用,指本领域熟知的原核或真核载体,例如细菌质粒、噬菌体、酵母质粒、植物细胞病毒、哺乳动物细胞病毒如腺病毒、逆转录病毒或其他载体,这些载体能够在宿主体内复制和稳定表达,这些重组载体的一个重要特征是通常含有表达控制序列。本文所用术语“表达控制序列”指调控目的基因的转录、翻译和表达的可以与目的基因操作性连接的元件,可以是复制起点、启动子、标记基因或翻译控制元件,包括增强子、操纵子、终止子、核糖体结合位点等,表达控制序列的选择取决于所用的宿主细胞。在本发明中适用的重组载体包括但不限于细菌质粒。在重组表达载体中,“操作性连接”是指目的的核苷酸序列与调节序列以允许核苷酸序列表达的方式连接。本领域的技术人员熟知能用于构建含本发明融合蛋白编码序列和合适的转录/翻译控制信号的表达载体的方法。这些方法包括体外重组dna技术、dna合成技术、体内重组技术等。所述的dna序列可有效连接到表达载体中的适当启动子上,以指导mrna合成。这些启动子的代表性例子有:大肠杆菌的lac或trp启动子;λ噬菌体pl启动子;真核启动子包括cmv立即早期启动子、hsv胸苷激酶启动子、早期和晚期sv40启动子、反转录病毒的ltr和其他一些已知的可控制基因在原核或真核细胞或其病毒中表达的启动子。表达载体还包括翻译起始用的核糖体结合位点和转录终止子。
本领域普通技术人员将理解重组表达载体的设计可取决于如欲转化的宿主细胞的选择、所需的蛋白质表达水平等因素。此外,重组表达载体优选地包含一个或多个选择性标记基因,以提供用于选择转化的宿主细胞的表型性状,如用于真核细胞的二氢叶酸还原酶、新霉素抗性,或用于大肠杆菌的四环素或氨苄青霉素抗性。
在一种实施方式中,将本发明光学探针或融合蛋白的编码序列经bamhi和hindiii双酶切后与bamhi和hindiii双酶切的prsetb载体连接,得到大肠杆菌重组表达载体。可以将本发明的表达载体转移到宿主细胞中,以产生包括融合蛋白的蛋白或肽。此种转移过程可用转化或转染等本领域技术人员熟知的常规技术进行。
本文所用术语“宿主细胞”或“细胞”又称为受体细胞,是指能够接收和容纳重组dna分子的细胞,是重组基因扩增的场所。理想的受体细胞应该满足易于获取和增殖两个条件。本发明的“宿主细胞”可包括原核细胞和真核细胞,具体包括细菌细胞、酵母细胞、昆虫细胞和哺乳动物细胞。
本发明的表达载体可用于在原核或真核细胞中表达本发明光学探针或融合蛋白。从而,本发明涉及已导入本发明表达载体的宿主细胞、优选大肠杆菌。宿主细胞可为任何原核或真核细胞,代表性例子有:大肠杆菌,链霉菌属,鼠伤寒沙门氏菌的细菌细胞,真菌细胞如酵母,植物细胞,果蝇s2或sf9的昆虫细胞,cho、cos、hek293细胞、或bowes黑素瘤细胞的动物细胞等,其中包括但不限于上述的那些宿主细胞。所述宿主细胞优选各种利于基因产物表达或发酵生产的细胞,此类细胞已为本领域熟知并常用,例如各种大肠杆菌细胞和酵母细胞。在本发明的一个实施方式中,选用大肠杆菌mach1构建表达本发明融合蛋白的宿主细胞。本领域一般技术人员都清楚如何选择适当的载体、启动子、增强子和宿主细胞。
本文所用术语“转化”和“转染”、“接合”和“转导”意指本领域内公知的各种将外源核酸,例如,线性dna或rna(例如,线性化载体或无载体的单独的基因构建体)或载体形式的核酸(例如,质粒、粘粒、噬菌体、噬粒、噬菌粒、转座子或其它dna)导入宿主细胞的技术,包括磷酸钙或氯化钙共沉淀、deae-甘露聚糖-介导的转染、脂质体转染、天然感受态、化学介导的转移或电穿孔。当宿主为原核生物如大肠杆菌时,能吸收dna的感受态细胞可在指数生长期后收获,用cacl2法处理,所用的步骤在本领域众所周知。另一种方法是使用mgcl2。如果需要,转化也可用电穿孔的方法进行。当宿主细胞是真核细胞时,可选用如下的dna转染方法:磷酸钙共沉淀法,常规机械方法如显微注射、电穿孔、脂质体包装等。
可以用适合所述宿主细胞表达的常规方法培养获得的转化细胞,表达本发明融合蛋白。根据所用的宿主细胞,培养中所用的培养基可以是各种常规培养基。在适于宿主细胞生长的条件下进行培养。当宿主细胞生长到适当的细胞密度后,用合适的方法(如温度转换或化学诱导)诱导选择的启动子,将细胞再培养一段时间。
在上面的方法中的重组蛋白可在细胞内或在细胞膜上表达,或分泌到细胞外。如果需要,可利用其物理的、化学的和其它特性通过各种分离方法分离或纯化重组的蛋白。这些方法是本领域技术人员所熟知的。这些方法的例子包括但并不限于:常规的复性处理、用蛋白沉淀剂处理(盐析方法)、离心、渗透破菌、超声处理、超离心、分子筛层析(凝胶过滤)、吸附层析、离子交换层析、高效液相层析(hplc)和其它各种液相层析技术及这些方法的结合。
在一个实施方式中,通过包含本发明融合蛋白编码序列的大肠杆菌发酵生产本发明光学探针或融合蛋白,并通过超声破碎,亲和层析和凝胶层析纯化得到了纯形式的本发明光学探针或融合蛋白。
本发明光学探针或融合蛋白的用途包括但不限于:检测α-kg、在生理状态下检测α-kg、在亚细胞水平检测α-kg、筛选化合物或药物,和定量α-kg。
本发明还提供了所述光学探针在α-kg实时定位、定量检测以及高通量化合物筛选中的应用。在一个实施方式中,所述的光学探针优选与细胞不同部位的信号肽连接,转入到细胞中,通过检测细胞中荧光信号的强弱,进行α-kg的实时定位;通过α-kg标准滴加曲线进行α-kg的定量检测。在一个实施方式中,α-kg标准滴加曲线是根据α-kg光学探针在不同浓度α-kg的情况下的荧光信号绘制而成。本发明所述光学探针直接转入细胞中,在α-kg实时定位和定量检测过程中不需要耗时的样品处理过程,更加准确。本发明光学探针在进行高通量化合物筛选时,将不同的化合物添加到细胞培养液中,测定α-kg含量的变化,从而筛选出对α-kg含量变化有影响的化合物。通常,本发明中所述应用不涉及疾病的诊断和治疗。
在本文中,浓度、含量、百分数和其它数值均可用范围的形式表示。也应理解,使用这种范围形式只是为了方便和简洁,应该被弹性地解读为包括范围上下限所明确提及的数值,还应包括该范围内包括的所有单个数值或子范围。
实施例
下面结合具体实施例,进一步阐述本发明。应理解,这些实施例仅用于说明本发明而不用于限制本发明的范围。
下列实施例中未注明具体条件的实验方法是本领域普通技术人员所熟知的,通常按照常规条件如sambrook等的《分子克隆:实验室指南》(美国纽约州:冷泉港实验室出版社(coldspringharborlaboratorypress),1989);简·罗斯凯姆斯等的《分子生物学实验参考手册》,j.萨姆布鲁克,d.w.拉塞尔著,黄培堂等译:《分子克隆实验指南》(第三版,2002年8月,科学出版社出版,北京);费雷谢尼等的《动物细胞培养:基本技术指南》(第五版),章静波,徐存拴等译;j.s.博尼费斯农,m.达索等的《精编细胞生物学实验指南》,章静波等译;或按照制造厂商所建议的条件进行。在本文中,除非另外说明,百分比和份数均按重量计算。本领域普通技术人员按照以下实施例,不难根据具体情况略作修改和变换而成功实施本发明,这些修改和变换均落在本申请权利要求的范围内。
i.实验材料和试剂
实施例中所用的基于prsetb-cpyfp,prsetb-cpmvenus,prsetb-cpbfp,prsetb-cpmapple,prsetb-glnk1质粒由华东理工大学蛋白质实验室构建,prsetb质粒载体购自invitrogen公司。所有用于pcr的引物均由上海捷瑞生物工程技术有限公司合成、纯化和经质谱法鉴定正确。实施例中构建的表达质粒都经过序列测定,序列测定由华大基因公司和杰李测序公司完成。各实施例所用的taqdna聚合酶购自东盛生物,pfudna聚合酶购自天根生化科技(北京)有限公司,primestardna聚合酶购自takara公司,三种聚合酶购买时都附带赠送对应聚合酶缓冲液和dntp。bamhi、bglii、hindiii、ndei、xhoi、ecori、spei等限制性内切酶、t4连接酶、t4磷酸化酶(t4pnk)购自fermentas公司,购买时附带有相对应的缓冲液等。hieffclonetmmultionestepcloningkit购自上海翊圣生物科技有限公司。α-kg,atp,adp等均购自merck公司。除非特别声明,无机盐类等化学试剂均购自sigma-aldrich公司。hepes盐,氨苄青霉素(amp)和嘌呤霉素购自ameresco公司;96孔荧光检测黑板购自whb公司;384孔荧光检测黑板购自greiner公司。
实施例中所用的dna纯化试剂盒购自bbi公司(加拿大),普通质粒小抽试剂盒购自天根生化科技(北京)有限公司,转染级别质粒小抽试剂盒购自omega公司。克隆菌株mach1购自invitrogen公司。镍柱亲和层析柱和脱盐柱填料均来自gehealthcare公司。
实施例中所用的hela等细胞购自atcc细胞保藏库,胰酶,澳洲特级胎牛血清购自gibco公司,lipofectamine2000购自美国invitrogen公司,磷酸盐缓冲液(pbs)和dmem培养基购自hyclone公司,小干扰rna(sirna)由上海吉玛公司合成。
实施例中用到的主要仪器:bioteksynergy2多功能酶标仪(美国bio-tek公司),x-15r高速冷冻离心机(美国beckman公司),microfuge22r台式高速冷冻离心机(美国beckman公司),pcr扩增仪(德国biometra公司),超声破碎仪(宁波新芝公司),核酸电泳仪(申能博彩公司),荧光分光光度计(美国varian公司),co2恒温细胞培养箱(sanyo),倒置荧光显微镜(日本尼康公司),活体成像系统(美国kodak公司),ariaii流式细胞仪(美国bd公司)。
ii.分子生物学方法和细胞实验方法
(一)聚合酶链式反应(pcr):
1.目的片段扩增pcr:
该方法主要用于基因片段扩增和菌落pcr鉴定阳性克隆。所述pcr扩增的反应体系如表1所示,扩增程序如表2所示。
表1.pcr扩增反应体系
表2.pcr扩增程序
2.长片段(>2500bp)扩增pcr:
本发明中使用的长片段扩增,主要是反向pcr扩增载体,在下述实施例中用于获得定点突变的一种技术。在变异部位设计反向pcr引物,其中一条引物的5’端包含变异的核苷酸序列。扩增后的产物就含有相应的突变位点。长片段扩增pcr反应体系如表3所示,扩增程序如表4或表5所示。
表3.长片段(>2500bp)扩增pcr反应体系
表4.长片段(>2500bp)扩增pcr扩增程序
表5.长片段(>2500bp)扩增pcr扩增程序
(二)核酸内切酶酶切反应:
对质粒载体进行双酶切的体系如表6所示,其中n代表使体系达到总体积所需要加入的灭菌超纯水μl量。
表6.质粒载体双酶切体系
(三)dna片段5’端磷酸化反应
从微生物中抽提出的质粒或者基因组末端都含有磷酸基团,而pcr产物没有,故需对pcr产物的5’端碱基进行磷酸基团加成反应,只有末端含有磷酸基团dna分子才能发生连接反应。磷酸化反应体系如表7所示,其中t4pnk为t4多聚核苷酸激酶的简写,用于对dna分子的5’端磷酸基团的加成反应。
表7.磷酸化反应体系
(四)目的片段和载体的连接反应
不同的片段和载体之间的连接方法有所差异,本发明中使用了三种连接方法
1.平末端短片段和线性化载体的平末端连接
该方法的原理是pcr获得的平末端产物在t4pnk作用下对dna片段的5’末端进行磷酸化反应后,与线性化的载体在peg4000和t4dna连接酶的作用下连接获得重组质粒。同源重组连接体系如表8所示。
表8.平末端片段连接反应体系
2.含有粘性末端的dna片段和含有粘性末端载体片段的连接
通过限制性内切酶切割的dna片段通常会产生突出的粘性末端,因此可以和含有序列互补的粘性末端载体片段连接,形成重组质粒。连接反应体系如表9所示。
表9.粘性末端连接反应体系
注:pcr产物片段与载体双酶切产物的质量比大致在2:1-6:1之间。
3.反向pcr引入定点突变后5’端磷酸化的dna片段产物自身环化的连接反应
将5’端磷酸化的dna片段通过自身环化连接反应将线性化载体的3’端和5’端连接反应得到重组质粒。自身环化连接反应体系如表10所示。
表10.自身环化连接反应体系
4.含有同源序列的多个dna片段与含有同源序列的载体片段的连接反应
将含有同源序列的多个dna片段与含有同源序列的载体片段混合,利用多片段一步法快速克隆试剂盒通过重组反应得到重组质粒。
表11.同源重组连接反应体系
线性化载体片段与pcr产物片段的物质的量之比为1:2。
(五)感受态细胞的制备与转化
感受态细胞的制备:
1.挑取单菌落(如mach1)接种于5mllb培养基中,37℃摇床过夜。
2.取0.5-1ml过夜培养的菌液转种到50mllb培养基中,37℃,220rpm培养3至5小时,直到od600达到0.5。
3.冰浴预冷细胞2小时。
4.4℃,4000rpm离心10分钟。
5.弃上清,用5ml预冷的缓冲液重悬细胞,待均匀后再加入重悬缓冲液至终体积为50ml。
6.冰浴45分钟。
7.4℃4000rpm离心10分钟,用5ml冰预冷的储存缓冲液重悬细菌。
8.每个ep管中放100μl菌液,-80℃或液氮冻存。
重悬缓冲液:cacl2(100mm)、mgcl2(70mm)、naac(40mm)
储存缓冲液:0.5mldmso、1.9ml80%甘油、1ml10×cacl2(1m)、1ml10×mgcl2(700mm)、1ml10×naac(400mm)、4.6mlddh2o
转化:
1.取100μl感受态细胞于冰浴上融化。
2.加入适当体积的连接产物,轻轻吹打混匀,冰浴30分钟。通常加入的连接产物的体积少于感受态细胞体积的1/10。
3.将菌液放入42℃水浴中热激90秒,迅速转移至冰浴中放置5分钟。
4.加入500μllb,于37℃恒温摇床上200rpm培养1小时。
5.将菌液4000rpm离心3分钟,留200μl上清将菌体吹匀,均匀涂布于含适当抗生素的琼脂平板表面,平板于37℃恒温培养箱内倒置过夜。
(六)蛋白质的表达,纯化和荧光检测
1.将prsetb为基础的α-kg探针质粒转化到jm109(de3)中,倒置培养过夜,从平板上挑取克隆到250ml锥形瓶中,置于37℃摇床,220rpm培养至od=0.4-0.8,加入1/1000(v/v)的iptg(1m),18℃诱导表达24-36小时。
2.诱导表达完成后,4000rpm,30分钟离心收菌,加入50mm的tris-hcl缓冲液重悬菌体沉淀,超声破碎至菌体澄清。9600rpm,4℃离心20分钟。
3.离心上清通过自装的镍柱亲和层析柱纯化获得蛋白,镍柱亲和层析后的蛋白再通过自装的脱盐柱获得溶解在100mmmops缓冲液(100mmmops,50mmkcl,ph7.4)的蛋白。
4.纯化的α-kg光学探针蛋白经过sds-page鉴定后,使用检测缓冲液(100mmmops,50mmkcl,2mmmg-atpph7.4)把探针成终浓度为0.5μm的探针溶液。
5.取100μl0.5μm的光学探针溶液,37℃温育5分钟,测定探针蛋白的荧光光谱;加入终浓度为10mmα-kg溶液后,测定探针蛋白的荧光光谱。对样品的荧光光谱的测定通过荧光分光光度计完成。
6.取100μl0.5μm的光学探针溶液,37℃温育5分钟,加入不同浓度梯度的α-kg溶液滴定,测定蛋白485nm光激发后528nm发射的荧光强度以及蛋白420nm光激发后528nm发射的荧光强度。对样品的荧光测定利用多功能荧光酶标仪完成。
(七)哺乳动物细胞的转染和荧光检测
1.取对数生长期的细胞,吸出细胞培养板中的旧培养液,用磷酸盐缓冲液pbs洗涤细胞一次。
2.加入0.5ml胰酶,37℃或者常温作用数分钟,于光学显微镜下进行观察,当细胞呈现圆粒状将要离壁时,就可以终止消化。
3.加入无抗生素的含有胎牛血清的培养基,轻拍培养板使细胞脱落,用吸管轻轻吹匀打散细胞团,将单细胞悬液铺板到96孔全黑细胞培养板或者35mm玻璃底培养板。
4.约12小时后转染,使用lipofectamine2000将合适量的质粒或者sirna转染到细胞中,4-6小时后更换培养基。
5.显微镜荧光成像:将转染后的细胞培养基移除,加入含有25mm葡萄糖的磷酸盐缓冲液pbs,将样品置于倒置荧光显微镜载物台上,选择好合适的条件进行拍照。
6.酶标仪荧光检测:去掉96孔黑底的荧光检测板中的培养基,加含有25mm葡萄糖的磷酸盐缓冲液pbs洗两遍后,加入检测药物或试剂,放入酶标仪进行荧光检测。
实施例1
prsetb-glnk1质粒构建
通过生物合成获得glnk1基因,通过pcr扩增该基因,glnk1的pcr产物凝胶电泳后回收后,用bamhi和hindiii酶切;同时对prsetb载体进行相应的双酶切。用t4dnaligase连接后,连接产物转化mach1,转化的mach1涂布于lb平板(氨苄青霉素100μg/ml),置于37℃培养过夜。将生长mach1转化子进行质粒抽提后,进行pcr鉴定。阳性质粒经过测序正确后进行后续的质粒构建。
实施例2
prsetb-glnk1-cpyfp,prsetb-glnk1-cpmvenus,prsetb-glnk1-cpbfp,prsetb-glnk1-cpmapple光学探针不同插入位点的质粒构建和检测
本实施例中,我们以prsetb-glnk1为基础质粒根据glnk1晶体结构选择了48/49,48/50,48/51,48/52,48/53,48/54,49/50,49/51,49/52,49/53,49/54,50/51,50/52,50/53,50/54,51/52,51/53,51/54,52/53,52/54,53/54位点插入cpyfp,cpmvenus,cpbfp,cpmapple。
利用pcr产生cpyfp,cpmvenus,cpbpf,cpmapple的dna片段,对该dna片段使用5’末端的加磷操作后灭活,同时通过反向pcr扩增产生含有不同断裂位点的prsetb-glnk1线性化载体,将线性化的prsetb-glnk1与5’末端磷酸化的cpyfp,cpmvenus,cpbpf,cpmapple片段在peg4000和t4dnaligase的作用下连接产生重组质粒,连接产物转化mach1,转化的mach1涂布于lb平板(氨苄青霉素100μg/ml),置于37℃培养过夜。将生长mach1转化子进行质粒抽提后,进行pcr鉴定。阳性质粒经过测序正确后进行后续的质粒构建。
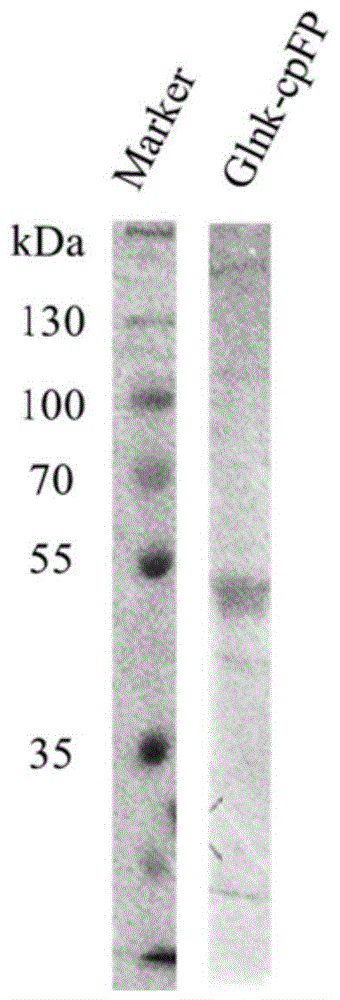
经过测序正确后,将重组质粒转化到jm109(de3)中诱导表达,并纯化蛋白质,通过sds-page电泳鉴定蛋白大小在47.8kda。该大小符合prsetb-glnk1-cpyfp,prsetb-glnk1-cpmvenus,prsetb-glnk1-cpbfp,prsetb-glnk1-cpmapple表达出的含his-tag纯化标签的glnk1-cpyfp,glnk1-cpmvenus,glnk1-cpbfp和glnk1-cpmapple融合蛋白质的大小。结果如图1所示。
将纯化的glnk1-cpyfp,glnk1-cpmvenus,glnk1-cpbfp和glnk1-cpmapple融合蛋白质进行α-kg响应筛选,将含有10mmα-kg的融合荧光蛋白质的检测信号除以α-kg的融合荧光蛋白质的检测信号得到标准化后的荧光信号。
结果如图2所示,检测结果显示对α-kg响应较大的插入位点是51/52位,其中,插入cpyfp时变化倍数约为1.9;插入cpmvenus时,变化倍数约为1.7;插入cpbfp时,变化倍数约为1.3;插入cpmapple时,变化倍数约为1.25(如图2所示)。
实施例3
prsetb-glnk1-cpyfptandem,prsetb-glnk1-cpmvenustandem,prsetb-glnk1-cpbfptandem,prsetb-glnk1-cpmappletandem光学探针不同插入位点的质粒构建和检测
本实施例中,我们以实施例2中prsetb-glnk1-cpyfp,prsetb-glnk1-cpmvenus,prsetb-glnk1-cpbfp,prsetb-glnk1-cpmapple荧光蛋白不同插入位点的质粒为基础质粒在其后连接两个删除t-loop区域的glnk1蛋白截短突变体。
以prset-glnk1质粒为基础质粒利用反向pcr和自身环化连接反应对glnk1进行截短突变(seqidno:11和seqidno:7),突变完成后,利用pcr产生两个截短突变后的glnk1dna片段。同时通过反向pcr扩增产生含有不同插入位点的prsetb-glnk1-cpyfp,prsetb-glnk1-cpmvenus,prsetb-glnk1-cpbfp,prsetb-glnk1-cpmapple线性化载体。将线性化的prsetb-glnk1-cpyfp,prsetb-glnk1-cpmvenus,prsetb-glnk1-cpbfp,prsetb-glnk1-cpmapple与两个glnk1截短突变体的dna片段在同源重组酶的作用下连接产生重组质粒,连接产物转化mach1,转化的mach1涂布于lb平板(氨苄青霉素100μg/ml),置于37℃培养过夜。将生长mach1转化子进行质粒抽提后,进行pcr鉴定。阳性质粒经过测序正确后进行后续的质粒构建。
经过测序正确后,将重组质粒转化到jm109(de3)中诱导表达,并纯化蛋白质,通过sds-page电泳大小在66.4kda。该大小符合prsetb-glnk1-cpyfptandem,prsetb-glnk1-cpmvenustandem,prsetb-glnk1-cpbfptandem,prsetb-glnk1-cpmappletandem表达出的含his-tag纯化标签的glnk1-cpyfptandem,glnk1-cpmvenustandem,glnk1-cpbfptandem和glnk1-cpmappletandem融合蛋白质的大小,结果如图3所示。
将纯化的glnk1-cpyfptandem,glnk1-cpmvenustandem,glnk1-cpbfptandem和glnk1-cpmappletandem融合蛋白质进行α-kg响应筛选,将含有10mmα-kg的融合荧光蛋白质的检测信号除以α-kg的融合荧光蛋白质的检测信号。
结果如图4所示,检测结果显示对α-kg响应较大的插入位点是51/52位,其中,插入cpyfp时变化倍数约为2.1;插入cpmvenus时,变化倍数约为1.8;插入cpbfp时,变化倍数约为1.4;插入cpmapple时,变化倍数约为1.3(如图4所示)。
实施例4
prsetb-glnk1-cpyfptandem51/52,prsetb-glnk1-cpmvenustandem51/52,prsetb-glnk1-cpbfptandem51/52,prsetb-glnk1-cpmappletandem51/52光学探针突变体的质粒构建和检测
本实施例中,我们以实施例2中prsetb-glnk1-cpyfp51/52,prsetb-glnk1-cpmvenus51/52,prsetb-glnk1-cpbfp51/52,prsetb-glnk1-cpmapple51/52为基础质粒,在其基础上利用反向pcr和自身环化连接反应进行定点突变,获得含有特定突变位点的prsetb-glnk1-cpyfp51/52,prsetb-glnk1-cpmvenus,prsetb-glnk1-cpbfp51/52,prsetb-glnk1-cpmapple51/52质粒。并以上述质粒为模板质粒,在其后连接两个删除t-loop区域的glnk1蛋白截短突变体,最终获得含有不同突变位点的prsetb-glnk1-cpyfp51/52tandem,prsetb-glnk1-cpmvenus51/52tandem,prsetb-glnk1-cpbfp51/52tandem,prsetb-glnk1-cpmapple51/52tandem质粒。突变位点包括6s、36t、43f和/或50v。
结果如图5所示,其中perceval为glnk1-cpmvenustandem51/526s36t,percevalhr为glnk1-cpmvenustandem51/526s36t43f50v,。根据图5,对于glnk1-cpyfp51/52tandem来说,43位的缬氨酸突变为苯丙氨酸,探针对α-kg响应的变化倍数增加到3.0倍,同时把43位的缬氨酸突变为苯丙氨酸,50位的谷氨酸突变为缬氨酸,探针对α-kg响应的变化倍数增加到6.7倍;对于glnk1-cpmvenus51/52tandem来说,43位的缬氨酸突变为苯丙氨酸,探针对α-kg响应的变化倍数增加到2.9倍,同时把43位的缬氨酸突变为苯丙氨酸,50位的谷氨酸突变为缬氨酸,探针对α-kg响应的变化倍数增加到3.5倍;对于glnk1-cpbfp51/52tandem来说,43位的缬氨酸突变为苯丙氨酸,探针对α-kg响应的变化倍数增加到1.5倍,同时把43位的缬氨酸突变为苯丙氨酸,50位的谷氨酸突变为缬氨酸,探针对α-kg响应的变化倍数增加到1.7倍;对于glnk1-cpmapple51/52tandem来说,43位的缬氨酸突变为苯丙氨酸,探针对α-kg响应的变化倍数增加到1.4倍,同时把43位的缬氨酸突变为苯丙氨酸,50位的谷氨酸突变为缬氨酸,探针对α-kg响应的变化倍数增加到1.8倍。
实施例5
glnk1-cpyfptandem51/5243f,glnk1-cpyfptandem51/5243f50v,glnk1-cpmvenustandem51/5243f,glnk1-cpmvenustandem51/5243f50v光学探针性质检测
本实施例中,选取对α-kg响应变化倍数较大的glnk1-cpyfptandem51/5243f,glnk1-cpyfptandem51/5243f50v,glnk1-cpmvenustandem51/5243f,glnk1-cpmvenustandem51/5243f50v探针进行性质检测。
首先,进行光谱性质的检测,将纯化的探针蛋白用检测缓冲液稀释到0.5μm,再分别使用0mm和10mmα-kg处理10分钟,使用荧光分光光度计进行荧光谱的检测。对激发谱的测定,固定发射在530nm处,进行350-515nm区间的激发谱检测;对发射谱的测定,固定激发在420nm或者485nm处,对505~600nm区间的发射谱进行检测。光学探针的光谱曲线如图6所示,glnk1-cpyfptandem51/5243f,glnk1-cpyfptandem51/5243f50v,glnk1-cpmvenustandem51/5243f,glnk1-cpmvenustandem51/5243f50v光学探针结合α-kg后,其在420nm处的荧光值上升,在485nm处的荧光值下降。cpyfp和cpvenus对a-kg没有明显响应,如图6中的e、f所示。
其次,进行kd值的测定,将纯化的探针蛋白用检测缓冲液稀释到0.5μm,再分别使用梯度浓度的α-kg处理10分钟后,检测420nm激发528nm发射处荧光强度和485nm激发528nm发射处荧光强度比值的变化,检测结果如图7所示。以上结果说明上述光学探针具有不同的kd值,其中glnk1-cpyfptandem51/5243f的kd值约为0.55mm,glnk1-cpyfptandem51/5243f50v的kd值约为2.72mm,glnk1-cpmvenustandem51/5243f的kd值约为1.13mm,glnk1-cpmvenustandem51/5243f50v的kd值约为5.6mm。cpyfp和cpvenus对高达20-50mm的a-kg都没有明显响应,如图7中的e、f所示,这表明上述4个探针展现了很好的特异性和响应性。
表12滴定曲线测定结果:
实施例6
glnk1-cpyfptandem51/5243f50v光学探针在不同亚细胞器内定位表达
本实施例中,选择的glnk1-cpyfptandem51/5243f50v光学探针进行不同亚细胞器的定位,我们使用不同的定位信号肽与glnk1-cpyfptandem51/5243f50v探针进行融合,将其定位到不同的细胞器中。不添加任何信号序列作为细胞浆定位。
典型的细胞器定位序列如下:
细胞核定位信号肽:dpkkkrkvdpkkkrkvdpkkkrkv
线粒体定位信号肽:msvltplllrgltgsarrlpvprakihslgdlsvltplllrg
ltgsarrlpvprakihslgd
细胞膜定位信号肽:mrgghkggrcacprvirkvlakcgccfardppvat
高尔基体定位信号肽:mrlrepllsgsaampgaslqracrllvavcalhlgvtlv
yylagrdlsrlpqlvgvstpl
融合不同定位信号肽的重组质粒转染hela细胞36小时后,使用pbs冲洗之后,置于hbss溶液中使用倒置荧光显微镜进行fitc通道下进行荧光检测。如图8所示。
实验结果表明glnk1-cpyfptandem51/5243f50v探针可以准确的定位于细胞浆、线粒体、细胞核、细胞膜、溶酶体、高尔基体、内质网、过氧化物酶体等亚细胞器中,不同的亚细胞结构中都显示有荧光,并且荧光的分布和强度各不相同。
实施例7
glnk1-cpyfptandem51/5243f50v光学探针在哺乳动物细胞中的实时检测
本实例中,选择的glnk1-cpyfptandem51/5243f50v光学探针进行哺乳动物细胞中的实时检测,按照实施例6的方法在hela细胞胞浆内表达glnk1-cpyfptandem51/5243f50v光学探针,通过在培养基中外加二甲基酮戊二酸(dmkg)来调控细胞内α-kg的浓度,使用多功能酶标仪对样品的荧光值进行检测,检测结果如图9所示,实验结果表明该探针能够实时地检测细胞内α-kg水平。
实施例8
活细胞水平基于α-kg光学探针进行高通量化合物筛选
本实施例中,我们选用胞浆表达glnk1-cpyfptandem51/5243f50v光学探针的hela细胞进行了高通量化合物筛选。将经过转染glnk1-cpyfptandem51/5243f50v基因的hela细胞,使用pbs冲洗之后,置于hbss溶液中处理1小时后,使用10μm的化合物进行处理1小时。使用酶标仪记录420nm激发528nm发射处荧光强度和485nm激发528nm发射处荧光强度的比值变化。以未用任何化合物处理的样品为标准,结果如图10所示,我们发现使用600种化合物处理的细胞中,绝大部分的化合物对细胞内α-kg的浓度没有影响。有6种化合物能够明显提高细胞内α-kg的浓度,另外有5种化合物能够明显降低细胞内α-kg的浓度。鉴于α-kg与表观遗传学的调控、蛋白质的合成,骨骼的生长和衰老等生理过程密切相关,基于α-kg探针的化合物筛选对于细胞代谢的研究以及新型药物的发现具有重要的意义。
实施例9
α-kg光学探针对胞浆内α-kg的定量
本实施例中,我们选用胞浆表达glnk1-cpyfptandem51/5243f50v光学探针的hela细胞进行半定量。将经过转染glnk1-cpyfptandem51/5243f50v基因的hela细胞,使用pbs冲洗之后,分成两组,一组未经处理,另一组置于hbss溶液中处理1小时后,使用10mm的dmkg进行处理1小时。使用酶标仪记录420nm激发528nm发射处荧光强度和485nm激发528nm发射处荧光强度比值变化。如图11,我们可以对细胞内的α-kg进行定量。探针和α-kg的相互作用服从下面的等式:
vα-kg=[α-kg]/(kd+[α-kg])
vα-kg=(r-rmin)/(rmax-rmin)
[α-kg]=kd(r-rmin)/(rmax-r)
其中,vα-kg表示α-kg探针的饱和率,[α-kg]是游离的α-kg的浓度。kd代表α-kg荧光探针的解离常数,即探针响应达到最大变化的一半时底物的浓度。rmin和rmax分别表示不加和存在10mmα-kg时,f420nm/f485nm比率。r代表样品的f485nm/f420nm比率。
由以上实施例可知,本发明提供的光学探针的蛋白分子量相对较小且易于成熟,荧光动态变化大,特异性好,并且能够通过基因操作的方法在细胞中表达,可在细胞内外实时、定位、定量检测α-kg;并且能够进行高通量的化合物筛选。
其它实施方式
本说明书描述了许多实施方式。然而应理解,本领域技术人员通过阅读本说明书获知的不背离本发明的构思和范围的各种改进,也应包括在所附权利要求书的范围内。
序列表
<110>华东理工大学
<120>α-酮戊二酸光学探针及其制备方法和应用
<130>184275cncn
<160>47
<170>patentinversion3.5
<210>1
<211>112
<212>prt
<213>詹氏甲烷球菌(methanococcusjannaschii)
<400>1
metlyslysvalglualaileileargproglulysleugluileval
151015
lyslysalaleuseraspalaglytyrvalglymetthrvalserglu
202530
vallysglyargglyvalglnglyglyilevalgluargtyrarggly
354045
argglutyrilevalaspleuileprolysvallysilegluleuval
505560
vallysglugluaspvalaspasnvalileaspileilecysgluasn
65707580
alaargthrglyasnproglyaspglylysilephevalileproval
859095
gluargvalvalargvalargthrlysglugluglylysglualaleu
100105110
<210>2
<211>108
<212>prt
<213>人工序列
<400>2
metlyslysvalglualaileileargproglulysleugluileval
151015
lyslysalaleuseraspalaglytyrvalglymetthrvalserglu
202530
vallysglyargglyvalglnglyglyilevalgluargtyrarggly
354045
argglutyrilevalaspleuileprolysvallysilegluleuval
505560
vallysglugluaspvalaspasnvalileaspileilecysgluasn
65707580
alaargthrglyasnproglyaspglylysilephevalileproval
859095
gluargvalvalargvalargthrlysgluglugly
100105
<210>3
<211>51
<212>prt
<213>人工序列
<400>3
metlyslysvalglualaileileargproglulysleugluileval
151015
lyslysalaleuseraspalaglytyrvalglymetthrvalserglu
202530
vallysglyargglyvalglnglyglyilevalgluargtyrarggly
354045
argglutyr
50
<210>4
<211>61
<212>prt
<213>人工序列
<400>4
ilevalaspleuileprolysvallysilegluleuvalvallysglu
151015
gluaspvalaspasnvalileaspileilecysgluasnalaargthr
202530
glyasnproglyaspglylysilephevalileprovalgluargval
354045
valargvalargthrlysglugluglylysglualaleu
505560
<210>5
<211>57
<212>prt
<213>人工序列
<400>5
ilevalaspleuileprolysvallysilegluleuvalvallysglu
151015
gluaspvalaspasnvalileaspileilecysgluasnalaargthr
202530
glyasnproglyaspglylysilephevalileprovalgluargval
354045
valargvalargthrlysgluglugly
5055
<210>6
<211>100
<212>prt
<213>人工序列
<400>6
metlyslysvalglualaileileargproglulysleugluileval
151015
lyslysalaleuseraspalaglytyrvalglymetthrvalserglu
202530
vallysglyargglyvalglnglyglyaspleuileprolysvallys
354045
ilegluleuvalvallysglugluaspvalaspasnvalileaspile
505560
ilecysgluasnalaargthrglyasnproglyaspglylysilephe
65707580
valileprovalgluargvalvalargvalargthrlysgluglugly
859095
lysglualaleu
100
<210>7
<211>100
<212>prt
<213>人工序列
<400>7
metlyslysvalglualaileileargproglulysleugluileval
151015
lyslysalaleuseraspalaglytyrvalglymetthrvalserglu
202530
vallysglyargglyalaglyglyglyaspleuileprolysvallys
354045
ilegluleuvalvallysglugluaspvalaspasnvalileaspile
505560
ilecysgluasnalaargthrglyasnproglyaspglylysilephe
65707580
valileprovalgluargvalvalargvalargthrlysgluglugly
859095
lysglualaleu
100
<210>8
<211>100
<212>prt
<213>人工序列
<400>8
metlyslysvalglualaileileargproglulysleugluileval
151015
lyslysalaleuseraspalaglytyrvalglymetthrvalserglu
202530
vallysglyargglyalaglnglyglyaspleuileprolysvallys
354045
ilegluleuvalvallysglugluaspvalaspasnvalileaspile
505560
ilecysgluasnalaargthrglyasnproglyaspglylysilephe
65707580
valileprovalgluargvalvalargvalargthrlysgluglugly
859095
lysglualaleu
100
<210>9
<211>100
<212>prt
<213>人工序列
<400>9
metlyslysvalglualaileileargproglulysleugluileval
151015
lyslysalaleuseraspalaglytyrvalglymetthrvalserglu
202530
vallysglyargglyvalglyglyglyaspleuileprolysvallys
354045
ilegluleuvalvallysglugluaspvalaspasnvalileaspile
505560
ilecysgluasnalaargthrglyasnproglyaspglylysilephe
65707580
valileprovalgluargvalvalargvalargthrlysgluglugly
859095
lysglualaleu
100
<210>10
<211>96
<212>prt
<213>人工序列
<400>10
metlyslysvalglualaileileargproglulysleugluileval
151015
lyslysalaleuseraspalaglytyrvalglymetthrvalserglu
202530
vallysglyargglyvalglnglyglyaspleuileprolysvallys
354045
ilegluleuvalvallysglugluaspvalaspasnvalileaspile
505560
ilecysgluasnalaargthrglyasnproglyaspglylysilephe
65707580
valileprovalgluargvalvalargvalargthrlysgluglugly
859095
<210>11
<211>96
<212>prt
<213>人工序列
<400>11
metlyslysvalglualaileileargproglulysleugluileval
151015
lyslysalaleuseraspalaglytyrvalglymetthrvalserglu
202530
vallysglyargglyalaglyglyglyaspleuileprolysvallys
354045
ilegluleuvalvallysglugluaspvalaspasnvalileaspile
505560
ilecysgluasnalaargthrglyasnproglyaspglylysilephe
65707580
valileprovalgluargvalvalargvalargthrlysgluglugly
859095
<210>12
<211>96
<212>prt
<213>人工序列
<400>12
metlyslysvalglualaileileargproglulysleugluileval
151015
lyslysalaleuseraspalaglytyrvalglymetthrvalserglu
202530
vallysglyargglyalaglnglyglyaspleuileprolysvallys
354045
ilegluleuvalvallysglugluaspvalaspasnvalileaspile
505560
ilecysgluasnalaargthrglyasnproglyaspglylysilephe
65707580
valileprovalgluargvalvalargvalargthrlysgluglugly
859095
<210>13
<211>96
<212>prt
<213>人工序列
<400>13
metlyslysvalglualaileileargproglulysleugluileval
151015
lyslysalaleuseraspalaglytyrvalglymetthrvalserglu
202530
vallysglyargglyvalglyglyglyaspleuileprolysvallys
354045
ilegluleuvalvallysglugluaspvalaspasnvalileaspile
505560
ilecysgluasnalaargthrglyasnproglyaspglylysilephe
65707580
valileprovalgluargvalvalargvalargthrlysgluglugly
859095
<210>14
<211>246
<212>prt
<213>人工序列
<400>14
tyrasnseraspasnvaltyrilemetalaasplysglnlysasngly
151015
ilelysalaasnphelysilearghisasnvalgluaspglyserval
202530
glnleualaasphistyrglnglnasnthrproileglyaspglypro
354045
valleuleuproaspasnhistyrleuserpheglnservalleuser
505560
lysaspproasnglulysargasphismetvalleuleuglupheval
65707580
thralaalaglyilethrleuglymetaspgluleutyrasnvalasp
859095
glyglyserglyglythrglyserlysglyglugluleuphethrgly
100105110
valvalproileleuvalgluleuaspglyaspvalasnglyhislys
115120125
pheservalserglygluglygluglyaspalathrtyrglylysleu
130135140
thrleulysleuilecysthrthrglylysleuprovalprotrppro
145150155160
thrleuvalthrthrleuglytyrglyleulyscysphealaargtyr
165170175
proasphismetlysglnhisaspphephelysseralametproglu
180185190
glytyrvalglngluargthrilephephelysaspaspglyasntyr
195200205
lysthrargalagluvallysphegluglyaspthrleuvalasnarg
210215220
ilegluleulysglyileglyphelysgluaspglyasnileleugly
225230235240
hislysleuglutyrasn
245
<210>15
<211>244
<212>prt
<213>人工序列
<400>15
tyrasnseraspasnvaltyrilethralaasplysglnlysasngly
151015
ilelysalaasnphelysilearghisasnilegluaspglyglyval
202530
glnleualaasphistyrglnglnasnthrproileglyaspglypro
354045
valleuleuproaspasnhistyrleuserpheglnserlysleuser
505560
lysaspproasnglulysargasphismetvalleuleuglupheval
65707580
thralaalaglyilethrleuglymetaspgluleutyrlysglygly
859095
serglyglymetvalserlysglyglugluleuphethrglyvalval
100105110
proileleuvalgluleuaspglyaspvalasnglyhislyspheser
115120125
valserglygluglygluglyaspalathrtyrglylysleuthrleu
130135140
lysleuilecysthrthrglylysleuprovalprotrpprothrleu
145150155160
valthrthrleuglytyrglyleuglncysphealaargtyrproasp
165170175
hismetlysglnhisaspphephelysseralametprogluglytyr
180185190
valglngluargthrilephephelysaspaspglyasntyrlysthr
195200205
argalagluvallysphegluglyaspthrleuvalasnargileglu
210215220
leulysglyileaspphelysgluaspglyasnileleuglyhislys
225230235240
leuglutyrasn
<210>16
<211>243
<212>prt
<213>人工序列
<400>16
asnvaltyrilelysalaasplysglnlysasnglyilelysalaasn
151015
phelysilearghisasnilegluglyglyglyvalglnleualatyr
202530
histyrglnglnasnthrproileglyaspglyprovalleuleupro
354045
aspasnhistyrleuservalglnserileleuserlysaspproasn
505560
glulysargasphismetvalleuleugluphevalthralaalagly
65707580
ilethrleuglymetaspgluleutyrlysglyglythrglyglyser
859095
glusermetvalserlysglyglugluleuphethrglyvalvalpro
100105110
ileglnvalgluleuaspglyaspvalasnglyhislyspheserval
115120125
serglygluglygluglyaspalathrtyrglylysleuthrleulys
130135140
pheilecysthrthrglylysleuprovalprotrpprothrleuval
145150155160
thrthrleuserhisglyvalglncyspheserargtyrproasphis
165170175
metlysglnhisaspphephelysseralametproglyglytyrile
180185190
glngluargthrilephephelysaspaspglyasntyrlysthrarg
195200205
alagluvallysphegluglyaspthrleuvalasnargilegluleu
210215220
lysglyileaspphelysgluaspglyasnileleuglyhislysleu
225230235240
glutyrasn
<210>17
<211>242
<212>prt
<213>人工序列
<400>17
valsergluargmettyrprogluaspglyalaleulyssergluile
151015
lyslysglyleuargleulysaspglyglyhistyralaalagluval
202530
lysthrthrtyrlysalalyslysprovalglnleuproglyalatyr
354045
ilevalaspilelysleuaspilevalserhisasngluasptyrthr
505560
ilevalgluglncysgluargalagluglyarghisserthrglygly
65707580
metaspgluleutyrlysglyglythrglyglyserleuvalserlys
859095
glyglugluaspasnmetalaileilelysgluphemetargphelys
100105110
valhismetgluglyservalasnglyhisgluphegluileglugly
115120125
gluglygluglyargprotyrglualapheglnthralalysleulys
130135140
valthrlysglyglyproleuprophealatrpaspileleuserpro
145150155160
glnphemettyrglyserlysalatyrilelyshisproalaaspile
165170175
proasptyrphelysleuserpheprogluglypheargtrpgluarg
180185190
valmetasnphegluaspglyglyileilehisvalasnglnaspser
195200205
serleuglnaspglyvalpheiletyrlysvallysleuargglythr
210215220
asnpheproproaspglyprovalmetglnlyslysthrmetglytrp
225230235240
gluala
<210>18
<211>241
<212>prt
<213>人工序列
<400>18
asnvaltyrilelysalaasplysglnlysasnglyilelysalaasn
151015
phelysilearghisasnilegluaspglyglyvalglnleualatyr
202530
histyrglnglnasnthrproileglyaspglyprovalleuleupro
354045
aspasnhistyrleuservalglnserileleuserlysaspproasn
505560
glulysargasphismetvalleuleugluphevalthralaalagly
65707580
ilethrleuglymetaspgluleutyrlysglyglythrglyglyser
859095
metvalserlysglyglugluleuphethrglyvalvalproilegln
100105110
valgluleuaspglyaspvalasnglyhislyspheservalsergly
115120125
gluglygluglyaspalathrtyrglylysleuthrleulyspheile
130135140
cysthrthrglylysleuprovalprotrpprothrleuvalthrthr
145150155160
leuthrtyrglyvalglncyspheserargtyrproasphismetlys
165170175
glnhisaspphephelysseralametprogluglytyrileglnglu
180185190
argthrilephephelysaspaspglyasntyrlysthrargalaglu
195200205
vallysphegluglyaspthrleuvalasnargilegluleulysgly
210215220
ileaspphelysgluaspglyasnileleuglyhislysleuglutyr
225230235240
asn
<210>19
<211>241
<212>prt
<213>人工序列
<400>19
asnvaltyrilemetalaasplysglnlysasnglyilelysalaasn
151015
phelysilearghisasnilegluaspglyglyvalglnleualaasp
202530
histyrglnglnasnthrproileglyaspglyprovalleuleupro
354045
aspasnhistyrleuserileglnserlysleuserlysaspproasn
505560
glulysargasphismetvalleuleugluphevalthralaalagly
65707580
ilethrhisglymetaspgluleutyrlysglyglythrglyglyser
859095
metvalserlysglyglugluleuphethrglyvalvalproileleu
100105110
valgluleuaspglyaspvalasnglyhislyspheservalsergly
115120125
gluglygluglyaspalathrtyrglylysleuthrleulyspheile
130135140
cysthrthrglylysleuprovalprotrpprothrleuvalthrthr
145150155160
phesertyrglyvalmetvalphealaargtyrproasphismetlys
165170175
glnhisaspphephelysseralametprogluglytyrvalglnglu
180185190
argthrilephephelysaspaspglyasntyrlysthrargalaglu
195200205
vallysphegluglyaspthrleuvalasnargilegluleulysgly
210215220
ileaspphelysgluaspglyasnileleuglyhislysleuglutyr
225230235240
asn
<210>20
<211>242
<212>prt
<213>人工序列
<400>20
valsergluargmettyrprogluaspglyvalleulyssergluile
151015
lyslysglyleuargleulysaspglyglyhistyralaalagluval
202530
lysthrthrtyrlysalalyslysprovalglnleuproglyalatyr
354045
ilevalaspilelysleuaspilevalserhisasngluasptyrthr
505560
ilevalgluglncysgluargalagluglyarghisprothrglygly
65707580
argaspgluleutyrlysglyglythrglyglyserleuvalserlys
859095
glyglugluaspasnmetalaileilelysgluphemetargphelys
100105110
valhismetgluglyservalasnglyhisgluphegluileglugly
115120125
gluglygluglyargprotyrglualapheglnthralalysleulys
130135140
valthrlysglyglyproleuprophealatrpaspileleuserpro
145150155160
glnphethrtyrglyserlysalatyrilelyshisproalaaspile
165170175
proasptyrphelysleuserpheprogluglypheargtrpgluarg
180185190
valmetasnphegluaspglyglyileilehisvalasnglnaspser
195200205
serleuglnaspglyvalpheiletyrlysvallysleuargglythr
210215220
asnpheproproaspglyprovalmetglnlyslysthrmetglytrp
225230235240
gluala
<210>21
<211>250
<212>prt
<213>人工序列
<400>21
metglyglyargserlyslysproalalysasnleulysmetprogly
151015
valtyrtyrvalaspargargleugluargilelysglualaasplys
202530
gluthrtyrvalgluglnhisgluvalalavalalaargtyrcysasp
354045
leuproserlysleuglyhislysleuasnglyglythrglyglyser
505560
metvalserlysglyglugluleuilelysgluasnmethismetlys
65707580
leutyrmetgluglythrvalasnasnhishisphelyscysthrser
859095
gluglygluglylysprotyrgluglythrglnthrmetargilelys
100105110
valvalgluglyglyproleuprophealapheaspileleualathr
115120125
serphemettyrglyserlysthrpheileasnhisthrglnglyile
130135140
proaspphephelysglnserpheprogluglyphethrtrpgluarg
145150155160
valthrthrtyrgluaspglyglyvalleuthralathrglnaspthr
165170175
serleuglnaspglycysleuiletyrasnvallysileargglyval
180185190
asnpheproserasnglyprovalmetglnlyslysthrleuglytrp
195200205
glualaserthrglumetleutyrproalaaspglyglyleuglugly
210215220
argseraspmetalaleulysleuvalglyglyglyhisleuilecys
225230235240
asnleulysthrthrtyrargserlyslys
245250
<210>22
<211>239
<212>prt
<213>人工序列
<400>22
metvalserlysglyglugluleuphethrglyvalvalproileleu
151015
valgluleuaspglyaspvalasnglyhislyspheservalsergly
202530
gluglygluglyaspalathrtyrglylysleuthrleulyspheile
354045
cysthrthrglylysleuprovalprotrpprothrleuvalthrthr
505560
leuserhisglyvalglncyspheserargtyrproasphismetlys
65707580
glnhisaspphephelysseralametprogluglytyrvalglnglu
859095
argthrilephephelysaspaspglyasntyrlysthrargalaglu
100105110
vallysphegluglyaspthrleuvalasnargilegluleulysgly
115120125
ileaspphelysgluaspglyasnileleuglyhislysleuglutyr
130135140
asnpheasnserhisasnvaltyrilemetalaasplysglnlysasn
145150155160
glyilelysalaasnphelysilearghisasnilegluaspglyser
165170175
valglnleualaasphistyrglnglnasnthrproileglyaspgly
180185190
provalleuleuproaspserhistyrleuserthrglnseralaleu
195200205
serlysaspproasnglulysargasphismetvalleuleugluphe
210215220
valthralaalaglyilethrleuglymetaspgluleutyrlys
225230235
<210>23
<211>238
<212>prt
<213>维多利亚发光水母(aequoreavictoria)
<400>23
metserlysglyglugluleuphethrglyvalvalprovalleuval
151015
gluleuaspglyaspvalasnglyglnlyspheservalserglyglu
202530
glygluglyaspalathrtyrglylysleuthrleuasnpheilecys
354045
thrthrglylysleuprovalprotrpprothrleuvalthrthrphe
505560
sertyrglyvalglncyspheserargtyrproasphismetlysgln
65707580
hisaspphephelysseralametprogluglytyrvalglngluarg
859095
thrilephetyrlysaspaspglyasntyrlysthrargalagluval
100105110
lysphegluglyaspthrleuvalasnargilegluleulysglyile
115120125
aspphelysgluaspglyasnileleuglyhislysmetglutyrasn
130135140
tyrasnserhisasnvaltyrilemetglyasplysprolysasngly
145150155160
ilelysvalasnphelysilearghisasnilelysaspglyserval
165170175
glnleualaasphistyrglnglnasnthrproileglyaspglypro
180185190
valleuleuproaspasnhistyrleuserthrglnseralaleuser
195200205
lysaspproasnglulysargasphismetileleuleuglupheval
210215220
thralaalaargilethrhisglymetaspgluleutyrlys
225230235
<210>24
<211>236
<212>prt
<213>人工序列
<400>24
metvalserlysglyglugluaspasnmetalaileilelysgluphe
151015
metargphelysvalhismetgluglyservalasnglyhisgluphe
202530
gluilegluglygluglygluglyargprotyrgluglythrglnthr
354045
alalysleulysvalthrlysglyglyproleuprophealatrpasp
505560
ileleuserproglnphemettyrglyserlysalatyrvallyshis
65707580
proalaaspileproasptyrleulysleuserpheprogluglyphe
859095
lystrpgluargvalmetasnphegluaspglyglyvalvalthrval
100105110
thrglnaspserserleuglnaspglyglupheiletyrlysvallys
115120125
leuargglythrasnpheproseraspglyprovalmetglnlyslys
130135140
thrmetglytrpglualasersergluargmettyrprogluaspgly
145150155160
alaleulysglygluilelysglnargleulysleulysaspglygly
165170175
histyraspalagluvallysthrthrtyrlysalalyslysproval
180185190
glnleuproglyalatyrasnvalasnilelysleuaspilethrser
195200205
hisasngluasptyrthrilevalgluglntyrgluargalaglugly
210215220
arghisserthrglyglymetaspgluleutyrlys
225230235
<210>25
<211>233
<212>prt
<213>人工序列
<400>25
metsergluleuilethrgluasnmethismetlysleutyrmetglu
151015
glythrvalasnasnhishisphelyscysthrsergluglyglugly
202530
lysprotyrgluglythrglnthrmetargilelysvalvalglugly
354045
glyproleuprophealapheaspileleualathrserphemettyr
505560
glyserlysthrpheileasnhisthrglnglyileproaspphephe
65707580
lysglnserpheprogluglyphethrtrpgluargvalthrthrtyr
859095
gluaspglyglyvalleuthralathrglnaspthrserleuglnasp
100105110
glycysleuiletyrasnvallysileargglyvalasnpheproser
115120125
asnglyprovalmetglnlyslysthrleuglytrpglualaserthr
130135140
glumetleutyrproalaaspglyglyleugluglyargalaaspmet
145150155160
alaleulysleuvalglyglyglyhisleuilecysasnleulysthr
165170175
thrtyrargserlyslysproalalysasnleulysmetproglyval
180185190
tyrtyrvalaspargargleugluargilelysglualaasplysglu
195200205
thrtyrvalgluglnhisgluvalalavalalaargtyrcysaspleu
210215220
proserlysleuglyhislysleuasn
225230
<210>26
<211>359
<212>prt
<213>人工序列
<400>26
metlyslysvalglualaileileargproglulysleugluileval
151015
lyslysalaleuseraspalaglytyrvalglymetthrvalserglu
202530
vallysglyargglyvalglnglyglyilevalgluargtyrarggly
354045
argglutyrseralaglytyrasnseraspasnvaltyrilemetala
505560
asplysglnlysasnglyilelysalaasnphelysilearghisasn
65707580
valgluaspglyservalglnleualaasphistyrglnglnasnthr
859095
proileglyaspglyprovalleuleuproaspasnhistyrleuser
100105110
pheglnservalleuserlysaspproasnglulysargasphismet
115120125
valleuleugluphevalthralaalaglyilethrleuglymetasp
130135140
gluleutyrasnvalaspglyglyserglyglythrglyserlysgly
145150155160
glugluleuphethrglyvalvalproileleuvalgluleuaspgly
165170175
aspvalasnglyhislyspheservalserglygluglygluglyasp
180185190
alathrtyrglylysleuthrleulysleuilecysthrthrglylys
195200205
leuprovalprotrpprothrleuvalthrthrleuglytyrglyleu
210215220
lyscysphealaargtyrproasphismetlysglnhisaspphephe
225230235240
lysseralametprogluglytyrvalglngluargthrilephephe
245250255
lysaspaspglyasntyrlysthrargalagluvallyspheglugly
260265270
aspthrleuvalasnargilegluleulysglyileglyphelysglu
275280285
aspglyasnileleuglyhislysleuglutyrasnglythrileval
290295300
aspleuileprolysvallysilegluleuvalvallysglugluasp
305310315320
valaspasnvalileaspileilecysgluasnalaargthrglyasn
325330335
proglyaspglylysilephevalileprovalgluargvalvalarg
340345350
valargthrlysgluglugly
355
<210>27
<211>357
<212>prt
<213>人工序列
<400>27
metlyslysvalglualaileileargproglulysleugluileval
151015
lyslysalaleuseraspalaglytyrvalglymetthrvalserglu
202530
vallysglyargglyvalglnglyglyilevalgluargtyrarggly
354045
argglutyrseralaglytyrasnseraspasnvaltyrilethrala
505560
asplysglnlysasnglyilelysalaasnphelysilearghisasn
65707580
ilegluaspglyglyvalglnleualaasphistyrglnglnasnthr
859095
proileglyaspglyprovalleuleuproaspasnhistyrleuser
100105110
pheglnserlysleuserlysaspproasnglulysargasphismet
115120125
valleuleugluphevalthralaalaglyilethrleuglymetasp
130135140
gluleutyrlysglyglyserglyglymetvalserlysglygluglu
145150155160
leuphethrglyvalvalproileleuvalgluleuaspglyaspval
165170175
asnglyhislyspheservalserglygluglygluglyaspalathr
180185190
tyrglylysleuthrleulysleuilecysthrthrglylysleupro
195200205
valprotrpprothrleuvalthrthrleuglytyrglyleuglncys
210215220
phealaargtyrproasphismetlysglnhisaspphephelysser
225230235240
alametprogluglytyrvalglngluargthrilephephelysasp
245250255
aspglyasntyrlysthrargalagluvallysphegluglyaspthr
260265270
leuvalasnargilegluleulysglyileaspphelysgluaspgly
275280285
asnileleuglyhislysleuglutyrasnglythrilevalaspleu
290295300
ileprolysvallysilegluleuvalvallysglugluaspvalasp
305310315320
asnvalileaspileilecysgluasnalaargthrglyasnprogly
325330335
aspglylysilephevalileprovalgluargvalvalargvalarg
340345350
thrlysgluglugly
355
<210>28
<211>356
<212>prt
<213>人工序列
<400>28
metlyslysvalglualaileileargproglulysleugluileval
151015
lyslysalaleuseraspalaglytyrvalglymetthrvalserglu
202530
vallysglyargglyvalglnglyglyilevalgluargtyrarggly
354045
argglutyrseralaglyasnvaltyrilelysalaasplysglnlys
505560
asnglyilelysalaasnphelysilearghisasnilegluglygly
65707580
glyvalglnleualatyrhistyrglnglnasnthrproileglyasp
859095
glyprovalleuleuproaspasnhistyrleuservalglnserile
100105110
leuserlysaspproasnglulysargasphismetvalleuleuglu
115120125
phevalthralaalaglyilethrleuglymetaspgluleutyrlys
130135140
glyglythrglyglyserglusermetvalserlysglyglugluleu
145150155160
phethrglyvalvalproileglnvalgluleuaspglyaspvalasn
165170175
glyhislyspheservalserglygluglygluglyaspalathrtyr
180185190
glylysleuthrleulyspheilecysthrthrglylysleuproval
195200205
protrpprothrleuvalthrthrleuserhisglyvalglncysphe
210215220
serargtyrproasphismetlysglnhisaspphephelysserala
225230235240
metproglyglytyrileglngluargthrilephephelysaspasp
245250255
glyasntyrlysthrargalagluvallysphegluglyaspthrleu
260265270
valasnargilegluleulysglyileaspphelysgluaspglyasn
275280285
ileleuglyhislysleuglutyrasnglythrilevalaspleuile
290295300
prolysvallysilegluleuvalvallysglugluaspvalaspasn
305310315320
valileaspileilecysgluasnalaargthrglyasnproglyasp
325330335
glylysilephevalileprovalgluargvalvalargvalargthr
340345350
lysgluglugly
355
<210>29
<211>355
<212>prt
<213>人工序列
<400>29
metlyslysvalglualaileileargproglulysleugluileval
151015
lyslysalaleuseraspalaglytyrvalglymetthrvalserglu
202530
vallysglyargglyvalglnglyglyilevalgluargtyrarggly
354045
argglutyrseralaglyvalsergluargmettyrprogluaspgly
505560
alaleulyssergluilelyslysglyleuargleulysaspglygly
65707580
histyralaalagluvallysthrthrtyrlysalalyslysproval
859095
glnleuproglyalatyrilevalaspilelysleuaspilevalser
100105110
hisasngluasptyrthrilevalgluglncysgluargalaglugly
115120125
arghisserthrglyglymetaspgluleutyrlysglyglythrgly
130135140
glyserleuvalserlysglyglugluaspasnmetalaileilelys
145150155160
gluphemetargphelysvalhismetgluglyservalasnglyhis
165170175
gluphegluilegluglygluglygluglyargprotyrglualaphe
180185190
glnthralalysleulysvalthrlysglyglyproleupropheala
195200205
trpaspileleuserproglnphemettyrglyserlysalatyrile
210215220
lyshisproalaaspileproasptyrphelysleuserpheproglu
225230235240
glypheargtrpgluargvalmetasnphegluaspglyglyileile
245250255
hisvalasnglnaspserserleuglnaspglyvalpheiletyrlys
260265270
vallysleuargglythrasnpheproproaspglyprovalmetgln
275280285
lyslysthrmetglytrpglualaglythrilevalaspleuilepro
290295300
lysvallysilegluleuvalvallysglugluaspvalaspasnval
305310315320
ileaspileilecysgluasnalaargthrglyasnproglyaspgly
325330335
lysilephevalileprovalgluargvalvalargvalargthrlys
340345350
gluglugly
355
<210>30
<211>581
<212>prt
<213>人工序列
<400>30
metlyslysvalglualaileileargproglulysleugluileval
151015
lyslysalaleuseraspalaglytyrvalglymetthrvalserglu
202530
vallysglyargglyvalglnglyglyilevalgluargtyrarggly
354045
argglutyrseralaglytyrasnseraspasnvaltyrilemetala
505560
asplysglnlysasnglyilelysalaasnphelysilearghisasn
65707580
valgluaspglyservalglnleualaasphistyrglnglnasnthr
859095
proileglyaspglyprovalleuleuproaspasnhistyrleuser
100105110
pheglnservalleuserlysaspproasnglulysargasphismet
115120125
valleuleugluphevalthralaalaglyilethrleuglymetasp
130135140
gluleutyrasnvalaspglyglyserglyglythrglyserlysgly
145150155160
glugluleuphethrglyvalvalproileleuvalgluleuaspgly
165170175
aspvalasnglyhislyspheservalserglygluglygluglyasp
180185190
alathrtyrglylysleuthrleulysleuilecysthrthrglylys
195200205
leuprovalprotrpprothrleuvalthrthrleuglytyrglyleu
210215220
lyscysphealaargtyrproasphismetlysglnhisaspphephe
225230235240
lysseralametprogluglytyrvalglngluargthrilephephe
245250255
lysaspaspglyasntyrlysthrargalagluvallyspheglugly
260265270
aspthrleuvalasnargilegluleulysglyileglyphelysglu
275280285
aspglyasnileleuglyhislysleuglutyrasnglythrileval
290295300
aspleuileprolysvallysilegluleuvalvallysglugluasp
305310315320
valaspasnvalileaspileilecysgluasnalaargthrglyasn
325330335
proglyaspglylysilephevalileprovalgluargvalvalarg
340345350
valargthrlysglugluglyalaserglyglyglyserglyglygly
355360365
glyalaserglymetlyslysvalglualaileileargproglulys
370375380
leugluilevallyslysalaleuseraspalaglytyrvalglymet
385390395400
thrvalsergluvallysglyargglyalaglyglyglyaspleuile
405410415
prolysvallysilegluleuvalvallysglugluaspvalaspasn
420425430
valileaspileilecysgluasnalaargthrglyasnproglyasp
435440445
glylysilephevalileprovalgluargvalvalargvalargthr
450455460
lysglugluglyalaserglyglyglyglyglyserglyglyalaser
465470475480
glymetlyslysvalglualaileileargproglulysleugluile
485490495
vallyslysalaleuseraspalaglytyrvalglymetthrvalser
500505510
gluvallysglyargglyalaglyglyglyaspleuileprolysval
515520525
lysilegluleuvalvallysglugluaspvalaspasnvalileasp
530535540
ileilecysgluasnalaargthrglyasnproglyaspglylysile
545550555560
phevalileprovalgluargvalvalargvalargthrlysgluglu
565570575
glylysglualaleu
580
<210>31
<211>579
<212>prt
<213>人工序列
<400>31
metlyslysvalglualaileileargproglulysleugluileval
151015
lyslysalaleuseraspalaglytyrvalglymetthrvalserglu
202530
vallysglyargglyvalglnglyglyilevalgluargtyrarggly
354045
argglutyrseralaglytyrasnseraspasnvaltyrilethrala
505560
asplysglnlysasnglyilelysalaasnphelysilearghisasn
65707580
ilegluaspglyglyvalglnleualaasphistyrglnglnasnthr
859095
proileglyaspglyprovalleuleuproaspasnhistyrleuser
100105110
pheglnserlysleuserlysaspproasnglulysargasphismet
115120125
valleuleugluphevalthralaalaglyilethrleuglymetasp
130135140
gluleutyrlysglyglyserglyglymetvalserlysglygluglu
145150155160
leuphethrglyvalvalproileleuvalgluleuaspglyaspval
165170175
asnglyhislyspheservalserglygluglygluglyaspalathr
180185190
tyrglylysleuthrleulysleuilecysthrthrglylysleupro
195200205
valprotrpprothrleuvalthrthrleuglytyrglyleuglncys
210215220
phealaargtyrproasphismetlysglnhisaspphephelysser
225230235240
alametprogluglytyrvalglngluargthrilephephelysasp
245250255
aspglyasntyrlysthrargalagluvallysphegluglyaspthr
260265270
leuvalasnargilegluleulysglyileaspphelysgluaspgly
275280285
asnileleuglyhislysleuglutyrasnglythrilevalaspleu
290295300
ileprolysvallysilegluleuvalvallysglugluaspvalasp
305310315320
asnvalileaspileilecysgluasnalaargthrglyasnprogly
325330335
aspglylysilephevalileprovalgluargvalvalargvalarg
340345350
thrlysglugluglyalaserglyglyglyserglyglyglyglyala
355360365
serglymetlyslysvalglualaileileargproglulysleuglu
370375380
ilevallyslysalaleuseraspalaglytyrvalglymetthrval
385390395400
sergluvallysglyargglyalaglyglyglyaspleuileprolys
405410415
vallysilegluleuvalvallysglugluaspvalaspasnvalile
420425430
aspileilecysgluasnalaargthrglyasnproglyaspglylys
435440445
ilephevalileprovalgluargvalvalargvalargthrlysglu
450455460
gluglyalaserglyglyglyglyglyserglyglyalaserglymet
465470475480
lyslysvalglualaileileargproglulysleugluilevallys
485490495
lysalaleuseraspalaglytyrvalglymetthrvalsergluval
500505510
lysglyargglyalaglyglyglyaspleuileprolysvallysile
515520525
gluleuvalvallysglugluaspvalaspasnvalileaspileile
530535540
cysgluasnalaargthrglyasnproglyaspglylysilepheval
545550555560
ileprovalgluargvalvalargvalargthrlysglugluglylys
565570575
glualaleu
<210>32
<211>578
<212>prt
<213>人工序列
<400>32
metlyslysvalglualaileileargproglulysleugluileval
151015
lyslysalaleuseraspalaglytyrvalglymetthrvalserglu
202530
vallysglyargglyvalglnglyglyilevalgluargtyrarggly
354045
argglutyrseralaglyasnvaltyrilelysalaasplysglnlys
505560
asnglyilelysalaasnphelysilearghisasnilegluglygly
65707580
glyvalglnleualatyrhistyrglnglnasnthrproileglyasp
859095
glyprovalleuleuproaspasnhistyrleuservalglnserile
100105110
leuserlysaspproasnglulysargasphismetvalleuleuglu
115120125
phevalthralaalaglyilethrleuglymetaspgluleutyrlys
130135140
glyglythrglyglyserglusermetvalserlysglyglugluleu
145150155160
phethrglyvalvalproileglnvalgluleuaspglyaspvalasn
165170175
glyhislyspheservalserglygluglygluglyaspalathrtyr
180185190
glylysleuthrleulyspheilecysthrthrglylysleuproval
195200205
protrpprothrleuvalthrthrleuserhisglyvalglncysphe
210215220
serargtyrproasphismetlysglnhisaspphephelysserala
225230235240
metproglyglytyrileglngluargthrilephephelysaspasp
245250255
glyasntyrlysthrargalagluvallysphegluglyaspthrleu
260265270
valasnargilegluleulysglyileaspphelysgluaspglyasn
275280285
ileleuglyhislysleuglutyrasnglythrilevalaspleuile
290295300
prolysvallysilegluleuvalvallysglugluaspvalaspasn
305310315320
valileaspileilecysgluasnalaargthrglyasnproglyasp
325330335
glylysilephevalileprovalgluargvalvalargvalargthr
340345350
lysglugluglyalaserglyglyglyserglyglyglyglyalaser
355360365
glymetlyslysvalglualaileileargproglulysleugluile
370375380
vallyslysalaleuseraspalaglytyrvalglymetthrvalser
385390395400
gluvallysglyargglyalaglyglyglyaspleuileprolysval
405410415
lysilegluleuvalvallysglugluaspvalaspasnvalileasp
420425430
ileilecysgluasnalaargthrglyasnproglyaspglylysile
435440445
phevalileprovalgluargvalvalargvalargthrlysgluglu
450455460
glyalaserglyglyglyglyglyserglyglyalaserglymetlys
465470475480
lysvalglualaileileargproglulysleugluilevallyslys
485490495
alaleuseraspalaglytyrvalglymetthrvalsergluvallys
500505510
glyargglyalaglyglyglyaspleuileprolysvallysileglu
515520525
leuvalvallysglugluaspvalaspasnvalileaspileilecys
530535540
gluasnalaargthrglyasnproglyaspglylysilephevalile
545550555560
provalgluargvalvalargvalargthrlysglugluglylysglu
565570575
alaleu
<210>33
<211>577
<212>prt
<213>人工序列
<400>33
metlyslysvalglualaileileargproglulysleugluileval
151015
lyslysalaleuseraspalaglytyrvalglymetthrvalserglu
202530
vallysglyargglyvalglnglyglyilevalgluargtyrarggly
354045
argglutyrseralaglyvalsergluargmettyrprogluaspgly
505560
alaleulyssergluilelyslysglyleuargleulysaspglygly
65707580
histyralaalagluvallysthrthrtyrlysalalyslysproval
859095
glnleuproglyalatyrilevalaspilelysleuaspilevalser
100105110
hisasngluasptyrthrilevalgluglncysgluargalaglugly
115120125
arghisserthrglyglymetaspgluleutyrlysglyglythrgly
130135140
glyserleuvalserlysglyglugluaspasnmetalaileilelys
145150155160
gluphemetargphelysvalhismetgluglyservalasnglyhis
165170175
gluphegluilegluglygluglygluglyargprotyrglualaphe
180185190
glnthralalysleulysvalthrlysglyglyproleupropheala
195200205
trpaspileleuserproglnphemettyrglyserlysalatyrile
210215220
lyshisproalaaspileproasptyrphelysleuserpheproglu
225230235240
glypheargtrpgluargvalmetasnphegluaspglyglyileile
245250255
hisvalasnglnaspserserleuglnaspglyvalpheiletyrlys
260265270
vallysleuargglythrasnpheproproaspglyprovalmetgln
275280285
lyslysthrmetglytrpglualaglythrilevalaspleuilepro
290295300
lysvallysilegluleuvalvallysglugluaspvalaspasnval
305310315320
ileaspileilecysgluasnalaargthrglyasnproglyaspgly
325330335
lysilephevalileprovalgluargvalvalargvalargthrlys
340345350
glugluglyalaserglyglyglyserglyglyglyglyalasergly
355360365
metlyslysvalglualaileileargproglulysleugluileval
370375380
lyslysalaleuseraspalaglytyrvalglymetthrvalserglu
385390395400
vallysglyargglyalaglyglyglyaspleuileprolysvallys
405410415
ilegluleuvalvallysglugluaspvalaspasnvalileaspile
420425430
ilecysgluasnalaargthrglyasnproglyaspglylysilephe
435440445
valileprovalgluargvalvalargvalargthrlysgluglugly
450455460
alaserglyglyglyglyglyserglyglyalaserglymetlyslys
465470475480
valglualaileileargproglulysleugluilevallyslysala
485490495
leuseraspalaglytyrvalglymetthrvalsergluvallysgly
500505510
argglyalaglyglyglyaspleuileprolysvallysilegluleu
515520525
valvallysglugluaspvalaspasnvalileaspileilecysglu
530535540
asnalaargthrglyasnproglyaspglylysilephevalilepro
545550555560
valgluargvalvalargvalargthrlysglugluglylysgluala
565570575
leu
<210>34
<211>581
<212>prt
<213>人工序列
<400>34
metlyslysvalglualaileileargproglulysleugluileval
151015
lyslysalaleuseraspalaglytyrvalglymetthrvalserglu
202530
vallysglyargglyvalglnglyglyilephegluargtyrarggly
354045
argglutyrseralaglytyrasnseraspasnvaltyrilemetala
505560
asplysglnlysasnglyilelysalaasnphelysilearghisasn
65707580
valgluaspglyservalglnleualaasphistyrglnglnasnthr
859095
proileglyaspglyprovalleuleuproaspasnhistyrleuser
100105110
pheglnservalleuserlysaspproasnglulysargasphismet
115120125
valleuleugluphevalthralaalaglyilethrleuglymetasp
130135140
gluleutyrasnvalaspglyglyserglyglythrglyserlysgly
145150155160
glugluleuphethrglyvalvalproileleuvalgluleuaspgly
165170175
aspvalasnglyhislyspheservalserglygluglygluglyasp
180185190
alathrtyrglylysleuthrleulysleuilecysthrthrglylys
195200205
leuprovalprotrpprothrleuvalthrthrleuglytyrglyleu
210215220
lyscysphealaargtyrproasphismetlysglnhisaspphephe
225230235240
lysseralametprogluglytyrvalglngluargthrilephephe
245250255
lysaspaspglyasntyrlysthrargalagluvallyspheglugly
260265270
aspthrleuvalasnargilegluleulysglyileglyphelysglu
275280285
aspglyasnileleuglyhislysleuglutyrasnglythrileval
290295300
aspleuileprolysvallysilegluleuvalvallysglugluasp
305310315320
valaspasnvalileaspileilecysgluasnalaargthrglyasn
325330335
proglyaspglylysilephevalileprovalgluargvalvalarg
340345350
valargthrlysglugluglyalaserglyglyglyserglyglygly
355360365
glyalaserglymetlyslysvalglualaileileargproglulys
370375380
leugluilevallyslysalaleuseraspalaglytyrvalglymet
385390395400
thrvalsergluvallysglyargglyalaglyglyglyaspleuile
405410415
prolysvallysilegluleuvalvallysglugluaspvalaspasn
420425430
valileaspileilecysgluasnalaargthrglyasnproglyasp
435440445
glylysilephevalileprovalgluargvalvalargvalargthr
450455460
lysglugluglyalaserglyglyglyglyglyserglyglyalaser
465470475480
glymetlyslysvalglualaileileargproglulysleugluile
485490495
vallyslysalaleuseraspalaglytyrvalglymetthrvalser
500505510
gluvallysglyargglyalaglyglyglyaspleuileprolysval
515520525
lysilegluleuvalvallysglugluaspvalaspasnvalileasp
530535540
ileilecysgluasnalaargthrglyasnproglyaspglylysile
545550555560
phevalileprovalgluargvalvalargvalargthrlysgluglu
565570575
glylysglualaleu
580
<210>35
<211>581
<212>prt
<213>人工序列
<400>35
metlyslysvalglualaileileargproglulysleugluileval
151015
lyslysalaleuseraspalaglytyrvalglymetthrvalserglu
202530
vallysglyargglyvalglnglyglyilephegluargtyrarggly
354045
argvaltyrseralaglytyrasnseraspasnvaltyrilemetala
505560
asplysglnlysasnglyilelysalaasnphelysilearghisasn
65707580
valgluaspglyservalglnleualaasphistyrglnglnasnthr
859095
proileglyaspglyprovalleuleuproaspasnhistyrleuser
100105110
pheglnservalleuserlysaspproasnglulysargasphismet
115120125
valleuleugluphevalthralaalaglyilethrleuglymetasp
130135140
gluleutyrasnvalaspglyglyserglyglythrglyserlysgly
145150155160
glugluleuphethrglyvalvalproileleuvalgluleuaspgly
165170175
aspvalasnglyhislyspheservalserglygluglygluglyasp
180185190
alathrtyrglylysleuthrleulysleuilecysthrthrglylys
195200205
leuprovalprotrpprothrleuvalthrthrleuglytyrglyleu
210215220
lyscysphealaargtyrproasphismetlysglnhisaspphephe
225230235240
lysseralametprogluglytyrvalglngluargthrilephephe
245250255
lysaspaspglyasntyrlysthrargalagluvallyspheglugly
260265270
aspthrleuvalasnargilegluleulysglyileglyphelysglu
275280285
aspglyasnileleuglyhislysleuglutyrasnglythrileval
290295300
aspleuileprolysvallysilegluleuvalvallysglugluasp
305310315320
valaspasnvalileaspileilecysgluasnalaargthrglyasn
325330335
proglyaspglylysilephevalileprovalgluargvalvalarg
340345350
valargthrlysglugluglyalaserglyglyglyserglyglygly
355360365
glyalaserglymetlyslysvalglualaileileargproglulys
370375380
leugluilevallyslysalaleuseraspalaglytyrvalglymet
385390395400
thrvalsergluvallysglyargglyalaglyglyglyaspleuile
405410415
prolysvallysilegluleuvalvallysglugluaspvalaspasn
420425430
valileaspileilecysgluasnalaargthrglyasnproglyasp
435440445
glylysilephevalileprovalgluargvalvalargvalargthr
450455460
lysglugluglyalaserglyglyglyglyglyserglyglyalaser
465470475480
glymetlyslysvalglualaileileargproglulysleugluile
485490495
vallyslysalaleuseraspalaglytyrvalglymetthrvalser
500505510
gluvallysglyargglyalaglyglyglyaspleuileprolysval
515520525
lysilegluleuvalvallysglugluaspvalaspasnvalileasp
530535540
ileilecysgluasnalaargthrglyasnproglyaspglylysile
545550555560
phevalileprovalgluargvalvalargvalargthrlysgluglu
565570575
glylysglualaleu
580
<210>36
<211>579
<212>prt
<213>人工序列
<400>36
metlyslysvalglualaileileargproglulysleugluileval
151015
lyslysalaleuseraspalaglytyrvalglymetthrvalserglu
202530
vallysglyargglyvalglnglyglyilephegluargtyrarggly
354045
argglutyrseralaglytyrasnseraspasnvaltyrilethrala
505560
asplysglnlysasnglyilelysalaasnphelysilearghisasn
65707580
ilegluaspglyglyvalglnleualaasphistyrglnglnasnthr
859095
proileglyaspglyprovalleuleuproaspasnhistyrleuser
100105110
pheglnserlysleuserlysaspproasnglulysargasphismet
115120125
valleuleugluphevalthralaalaglyilethrleuglymetasp
130135140
gluleutyrlysglyglyserglyglymetvalserlysglygluglu
145150155160
leuphethrglyvalvalproileleuvalgluleuaspglyaspval
165170175
asnglyhislyspheservalserglygluglygluglyaspalathr
180185190
tyrglylysleuthrleulysleuilecysthrthrglylysleupro
195200205
valprotrpprothrleuvalthrthrleuglytyrglyleuglncys
210215220
phealaargtyrproasphismetlysglnhisaspphephelysser
225230235240
alametprogluglytyrvalglngluargthrilephephelysasp
245250255
aspglyasntyrlysthrargalagluvallysphegluglyaspthr
260265270
leuvalasnargilegluleulysglyileaspphelysgluaspgly
275280285
asnileleuglyhislysleuglutyrasnglythrilevalaspleu
290295300
ileprolysvallysilegluleuvalvallysglugluaspvalasp
305310315320
asnvalileaspileilecysgluasnalaargthrglyasnprogly
325330335
aspglylysilephevalileprovalgluargvalvalargvalarg
340345350
thrlysglugluglyalaserglyglyglyserglyglyglyglyala
355360365
serglymetlyslysvalglualaileileargproglulysleuglu
370375380
ilevallyslysalaleuseraspalaglytyrvalglymetthrval
385390395400
sergluvallysglyargglyalaglyglyglyaspleuileprolys
405410415
vallysilegluleuvalvallysglugluaspvalaspasnvalile
420425430
aspileilecysgluasnalaargthrglyasnproglyaspglylys
435440445
ilephevalileprovalgluargvalvalargvalargthrlysglu
450455460
gluglyalaserglyglyglyglyglyserglyglyalaserglymet
465470475480
lyslysvalglualaileileargproglulysleugluilevallys
485490495
lysalaleuseraspalaglytyrvalglymetthrvalsergluval
500505510
lysglyargglyalaglyglyglyaspleuileprolysvallysile
515520525
gluleuvalvallysglugluaspvalaspasnvalileaspileile
530535540
cysgluasnalaargthrglyasnproglyaspglylysilepheval
545550555560
ileprovalgluargvalvalargvalargthrlysglugluglylys
565570575
glualaleu
<210>37
<211>579
<212>prt
<213>人工序列
<400>37
metlyslysvalglualaileileargproglulysleugluileval
151015
lyslysalaleuseraspalaglytyrvalglymetthrvalserglu
202530
vallysglyargglyvalglnglyglyilephegluargtyrarggly
354045
argvaltyrseralaglytyrasnseraspasnvaltyrilethrala
505560
asplysglnlysasnglyilelysalaasnphelysilearghisasn
65707580
ilegluaspglyglyvalglnleualaasphistyrglnglnasnthr
859095
proileglyaspglyprovalleuleuproaspasnhistyrleuser
100105110
pheglnserlysleuserlysaspproasnglulysargasphismet
115120125
valleuleugluphevalthralaalaglyilethrleuglymetasp
130135140
gluleutyrlysglyglyserglyglymetvalserlysglygluglu
145150155160
leuphethrglyvalvalproileleuvalgluleuaspglyaspval
165170175
asnglyhislyspheservalserglygluglygluglyaspalathr
180185190
tyrglylysleuthrleulysleuilecysthrthrglylysleupro
195200205
valprotrpprothrleuvalthrthrleuglytyrglyleuglncys
210215220
phealaargtyrproasphismetlysglnhisaspphephelysser
225230235240
alametprogluglytyrvalglngluargthrilephephelysasp
245250255
aspglyasntyrlysthrargalagluvallysphegluglyaspthr
260265270
leuvalasnargilegluleulysglyileaspphelysgluaspgly
275280285
asnileleuglyhislysleuglutyrasnglythrilevalaspleu
290295300
ileprolysvallysilegluleuvalvallysglugluaspvalasp
305310315320
asnvalileaspileilecysgluasnalaargthrglyasnprogly
325330335
aspglylysilephevalileprovalgluargvalvalargvalarg
340345350
thrlysglugluglyalaserglyglyglyserglyglyglyglyala
355360365
serglymetlyslysvalglualaileileargproglulysleuglu
370375380
ilevallyslysalaleuseraspalaglytyrvalglymetthrval
385390395400
sergluvallysglyargglyalaglyglyglyaspleuileprolys
405410415
vallysilegluleuvalvallysglugluaspvalaspasnvalile
420425430
aspileilecysgluasnalaargthrglyasnproglyaspglylys
435440445
ilephevalileprovalgluargvalvalargvalargthrlysglu
450455460
gluglyalaserglyglyglyglyglyserglyglyalaserglymet
465470475480
lyslysvalglualaileileargproglulysleugluilevallys
485490495
lysalaleuseraspalaglytyrvalglymetthrvalsergluval
500505510
lysglyargglyalaglyglyglyaspleuileprolysvallysile
515520525
gluleuvalvallysglugluaspvalaspasnvalileaspileile
530535540
cysgluasnalaargthrglyasnproglyaspglylysilepheval
545550555560
ileprovalgluargvalvalargvalargthrlysglugluglylys
565570575
glualaleu
<210>38
<211>578
<212>prt
<213>人工序列
<400>38
metlyslysvalglualaileileargproglulysleugluileval
151015
lyslysalaleuseraspalaglytyrvalglymetthrvalserglu
202530
vallysglyargglyvalglnglyglyilephegluargtyrarggly
354045
argglutyrseralaglyasnvaltyrilelysalaasplysglnlys
505560
asnglyilelysalaasnphelysilearghisasnilegluglygly
65707580
glyvalglnleualatyrhistyrglnglnasnthrproileglyasp
859095
glyprovalleuleuproaspasnhistyrleuservalglnserile
100105110
leuserlysaspproasnglulysargasphismetvalleuleuglu
115120125
phevalthralaalaglyilethrleuglymetaspgluleutyrlys
130135140
glyglythrglyglyserglusermetvalserlysglyglugluleu
145150155160
phethrglyvalvalproileglnvalgluleuaspglyaspvalasn
165170175
glyhislyspheservalserglygluglygluglyaspalathrtyr
180185190
glylysleuthrleulyspheilecysthrthrglylysleuproval
195200205
protrpprothrleuvalthrthrleuserhisglyvalglncysphe
210215220
serargtyrproasphismetlysglnhisaspphephelysserala
225230235240
metproglyglytyrileglngluargthrilephephelysaspasp
245250255
glyasntyrlysthrargalagluvallysphegluglyaspthrleu
260265270
valasnargilegluleulysglyileaspphelysgluaspglyasn
275280285
ileleuglyhislysleuglutyrasnglythrilevalaspleuile
290295300
prolysvallysilegluleuvalvallysglugluaspvalaspasn
305310315320
valileaspileilecysgluasnalaargthrglyasnproglyasp
325330335
glylysilephevalileprovalgluargvalvalargvalargthr
340345350
lysglugluglyalaserglyglyglyserglyglyglyglyalaser
355360365
glymetlyslysvalglualaileileargproglulysleugluile
370375380
vallyslysalaleuseraspalaglytyrvalglymetthrvalser
385390395400
gluvallysglyargglyalaglyglyglyaspleuileprolysval
405410415
lysilegluleuvalvallysglugluaspvalaspasnvalileasp
420425430
ileilecysgluasnalaargthrglyasnproglyaspglylysile
435440445
phevalileprovalgluargvalvalargvalargthrlysgluglu
450455460
glyalaserglyglyglyglyglyserglyglyalaserglymetlys
465470475480
lysvalglualaileileargproglulysleugluilevallyslys
485490495
alaleuseraspalaglytyrvalglymetthrvalsergluvallys
500505510
glyargglyalaglyglyglyaspleuileprolysvallysileglu
515520525
leuvalvallysglugluaspvalaspasnvalileaspileilecys
530535540
gluasnalaargthrglyasnproglyaspglylysilephevalile
545550555560
provalgluargvalvalargvalargthrlysglugluglylysglu
565570575
alaleu
<210>39
<211>578
<212>prt
<213>人工序列
<400>39
metlyslysvalglualaileileargproglulysleugluileval
151015
lyslysalaleuseraspalaglytyrvalglymetthrvalserglu
202530
vallysglyargglyvalglnglyglyilephegluargtyrarggly
354045
argvaltyrseralaglyasnvaltyrilelysalaasplysglnlys
505560
asnglyilelysalaasnphelysilearghisasnilegluglygly
65707580
glyvalglnleualatyrhistyrglnglnasnthrproileglyasp
859095
glyprovalleuleuproaspasnhistyrleuservalglnserile
100105110
leuserlysaspproasnglulysargasphismetvalleuleuglu
115120125
phevalthralaalaglyilethrleuglymetaspgluleutyrlys
130135140
glyglythrglyglyserglusermetvalserlysglyglugluleu
145150155160
phethrglyvalvalproileglnvalgluleuaspglyaspvalasn
165170175
glyhislyspheservalserglygluglygluglyaspalathrtyr
180185190
glylysleuthrleulyspheilecysthrthrglylysleuproval
195200205
protrpprothrleuvalthrthrleuserhisglyvalglncysphe
210215220
serargtyrproasphismetlysglnhisaspphephelysserala
225230235240
metproglyglytyrileglngluargthrilephephelysaspasp
245250255
glyasntyrlysthrargalagluvallysphegluglyaspthrleu
260265270
valasnargilegluleulysglyileaspphelysgluaspglyasn
275280285
ileleuglyhislysleuglutyrasnglythrilevalaspleuile
290295300
prolysvallysilegluleuvalvallysglugluaspvalaspasn
305310315320
valileaspileilecysgluasnalaargthrglyasnproglyasp
325330335
glylysilephevalileprovalgluargvalvalargvalargthr
340345350
lysglugluglyalaserglyglyglyserglyglyglyglyalaser
355360365
glymetlyslysvalglualaileileargproglulysleugluile
370375380
vallyslysalaleuseraspalaglytyrvalglymetthrvalser
385390395400
gluvallysglyargglyalaglyglyglyaspleuileprolysval
405410415
lysilegluleuvalvallysglugluaspvalaspasnvalileasp
420425430
ileilecysgluasnalaargthrglyasnproglyaspglylysile
435440445
phevalileprovalgluargvalvalargvalargthrlysgluglu
450455460
glyalaserglyglyglyglyglyserglyglyalaserglymetlys
465470475480
lysvalglualaileileargproglulysleugluilevallyslys
485490495
alaleuseraspalaglytyrvalglymetthrvalsergluvallys
500505510
glyargglyalaglyglyglyaspleuileprolysvallysileglu
515520525
leuvalvallysglugluaspvalaspasnvalileaspileilecys
530535540
gluasnalaargthrglyasnproglyaspglylysilephevalile
545550555560
provalgluargvalvalargvalargthrlysglugluglylysglu
565570575
alaleu
<210>40
<211>577
<212>prt
<213>人工序列
<400>40
metlyslysvalglualaileileargproglulysleugluileval
151015
lyslysalaleuseraspalaglytyrvalglymetthrvalserglu
202530
vallysglyargglyvalglnglyglyilephegluargtyrarggly
354045
argglutyrseralaglyvalsergluargmettyrprogluaspgly
505560
alaleulyssergluilelyslysglyleuargleulysaspglygly
65707580
histyralaalagluvallysthrthrtyrlysalalyslysproval
859095
glnleuproglyalatyrilevalaspilelysleuaspilevalser
100105110
hisasngluasptyrthrilevalgluglncysgluargalaglugly
115120125
arghisserthrglyglymetaspgluleutyrlysglyglythrgly
130135140
glyserleuvalserlysglyglugluaspasnmetalaileilelys
145150155160
gluphemetargphelysvalhismetgluglyservalasnglyhis
165170175
gluphegluilegluglygluglygluglyargprotyrglualaphe
180185190
glnthralalysleulysvalthrlysglyglyproleupropheala
195200205
trpaspileleuserproglnphemettyrglyserlysalatyrile
210215220
lyshisproalaaspileproasptyrphelysleuserpheproglu
225230235240
glypheargtrpgluargvalmetasnphegluaspglyglyileile
245250255
hisvalasnglnaspserserleuglnaspglyvalpheiletyrlys
260265270
vallysleuargglythrasnpheproproaspglyprovalmetgln
275280285
lyslysthrmetglytrpglualaglythrilevalaspleuilepro
290295300
lysvallysilegluleuvalvallysglugluaspvalaspasnval
305310315320
ileaspileilecysgluasnalaargthrglyasnproglyaspgly
325330335
lysilephevalileprovalgluargvalvalargvalargthrlys
340345350
glugluglyalaserglyglyglyserglyglyglyglyalasergly
355360365
metlyslysvalglualaileileargproglulysleugluileval
370375380
lyslysalaleuseraspalaglytyrvalglymetthrvalserglu
385390395400
vallysglyargglyalaglyglyglyaspleuileprolysvallys
405410415
ilegluleuvalvallysglugluaspvalaspasnvalileaspile
420425430
ilecysgluasnalaargthrglyasnproglyaspglylysilephe
435440445
valileprovalgluargvalvalargvalargthrlysgluglugly
450455460
alaserglyglyglyglyglyserglyglyalaserglymetlyslys
465470475480
valglualaileileargproglulysleugluilevallyslysala
485490495
leuseraspalaglytyrvalglymetthrvalsergluvallysgly
500505510
argglyalaglyglyglyaspleuileprolysvallysilegluleu
515520525
valvallysglugluaspvalaspasnvalileaspileilecysglu
530535540
asnalaargthrglyasnproglyaspglylysilephevalilepro
545550555560
valgluargvalvalargvalargthrlysglugluglylysgluala
565570575
leu
<210>41
<211>577
<212>prt
<213>人工序列
<400>41
metlyslysvalglualaileileargproglulysleugluileval
151015
lyslysalaleuseraspalaglytyrvalglymetthrvalserglu
202530
vallysglyargglyvalglnglyglyilephegluargtyrarggly
354045
argvaltyrseralaglyvalsergluargmettyrprogluaspgly
505560
alaleulyssergluilelyslysglyleuargleulysaspglygly
65707580
histyralaalagluvallysthrthrtyrlysalalyslysproval
859095
glnleuproglyalatyrilevalaspilelysleuaspilevalser
100105110
hisasngluasptyrthrilevalgluglncysgluargalaglugly
115120125
arghisserthrglyglymetaspgluleutyrlysglyglythrgly
130135140
glyserleuvalserlysglyglugluaspasnmetalaileilelys
145150155160
gluphemetargphelysvalhismetgluglyservalasnglyhis
165170175
gluphegluilegluglygluglygluglyargprotyrglualaphe
180185190
glnthralalysleulysvalthrlysglyglyproleupropheala
195200205
trpaspileleuserproglnphemettyrglyserlysalatyrile
210215220
lyshisproalaaspileproasptyrphelysleuserpheproglu
225230235240
glypheargtrpgluargvalmetasnphegluaspglyglyileile
245250255
hisvalasnglnaspserserleuglnaspglyvalpheiletyrlys
260265270
vallysleuargglythrasnpheproproaspglyprovalmetgln
275280285
lyslysthrmetglytrpglualaglythrilevalaspleuilepro
290295300
lysvallysilegluleuvalvallysglugluaspvalaspasnval
305310315320
ileaspileilecysgluasnalaargthrglyasnproglyaspgly
325330335
lysilephevalileprovalgluargvalvalargvalargthrlys
340345350
glugluglyalaserglyglyglyserglyglyglyglyalasergly
355360365
metlyslysvalglualaileileargproglulysleugluileval
370375380
lyslysalaleuseraspalaglytyrvalglymetthrvalserglu
385390395400
vallysglyargglyalaglyglyglyaspleuileprolysvallys
405410415
ilegluleuvalvallysglugluaspvalaspasnvalileaspile
420425430
ilecysgluasnalaargthrglyasnproglyaspglylysilephe
435440445
valileprovalgluargvalvalargvalargthrlysgluglugly
450455460
alaserglyglyglyglyglyserglyglyalaserglymetlyslys
465470475480
valglualaileileargproglulysleugluilevallyslysala
485490495
leuseraspalaglytyrvalglymetthrvalsergluvallysgly
500505510
argglyalaglyglyglyaspleuileprolysvallysilegluleu
515520525
valvallysglugluaspvalaspasnvalileaspileilecysglu
530535540
asnalaargthrglyasnproglyaspglylysilephevalilepro
545550555560
valgluargvalvalargvalargthrlysglugluglylysgluala
565570575
leu
<210>42
<211>1740
<212>dna
<213>人工序列
<400>42
atgaaaaaggtggaagccatcatcaggcccgaaaagctggagatcgttaagaaggctctc60
tcggacgctggatatgtgggtatgaccgtctctgaagtcaaaggccggggcgtccagggc120
ggcatcgtcgagaggtaccgaggaagggagtactctgcaggctacaacagcgacaacgtc180
tacatcaccgccgacaagcagaagaacggcatcaaggccaacttcaagatccgccacaac240
atcgaggacggcggcgtgcagctcgccgaccactaccagcagaacacccccatcggcgac300
ggccccgtgctgctgcccgacaaccactacctgagcttccagtccaagctgagcaaagac360
cccaacgagaagcgcgatcacatggtcctgctggagttcgtgaccgccgccgggatcact420
ctcggcatggacgagctgtacaagggcggttccggaggcatggtgagcaagggcgaggag480
ctgttcaccggggtggtgcccatcctggtcgagctggacggcgacgtaaacggccacaag540
ttcagcgtgtccggcgagggcgagggcgatgccacctacggcaagctgaccctgaagctg600
atctgcaccaccggcaagctgcccgtgccctggcccaccctcgtgaccaccctgggctac660
ggtctccaatgcttcgcccgctaccccgaccacatgaagcagcacgacttcttcaagtcc720
gccatgcccgaaggctacgtccaggagcgcaccatcttcttcaaggacgacggcaactac780
aagacccgcgccgaggtgaagttcgagggcgacaccctggtgaaccgcatcgagctgaag840
ggcatcgacttcaaggaggacggcaacatcctggggcacaagcttgagtacaacggcacc900
atagtagatctgatccctaaggtaaaaattgaactagtggtgaaggaggaggacgtcgat960
aacgtgatagacattatttgcgaaaatgcccgcacaggtaaccccggcgacggtaagata1020
tttgtgatcccagtggagcgagtggtcagggtgcgaaccaaagaggagggagcatctggt1080
ggtggatccggtggtggcggtgcatctggtatgaaaaaggtggaagccatcatcaggccc1140
gaaaagctggagatcgttaagaaggctctctcggacgctggatatgtgggtatgaccgtc1200
tctgaagtcaaaggccggggcgcaggtggcggcgatctgatccctaaggtaaaaattgaa1260
ctagtggtgaaggaggaggacgtcgataacgtgatagacattatttgcgaaaatgcccgc1320
acaggtaaccccggcgacggtaagatatttgtgatcccagtggagcgagtggtcagggtg1380
cgaaccaaagaggagggagcctccggcggtggcggtggctccggtggcgccagcggcatg1440
aaaaaggtggaagccatcatcaggcccgaaaagctggagatcgttaagaaggctctctcg1500
gacgctggatatgtgggtatgaccgtctctgaagtcaaaggccggggcgcaggtggcggc1560
gatctgatccctaaggtaaaaattgaactagtggtgaaggaggaggacgtcgataacgtg1620
atagacattatttgcgaaaatgcccgcacaggtaaccccggcgacggtaagatatttgtg1680
atcccagtggagcgagtggtcagggtgcgaaccaaagaggagggaaaggaagcactgtga1740
<210>43
<211>1740
<212>dna
<213>人工序列
<400>43
atgaaaaaggtggaagccatcatcaggcccgaaaagctggagatcgttaagaaggctctc60
tcggacgctggatatgtgggtatgaccgtctctgaagtcaaaggccggggcgtccagggc120
ggcatcttcgagaggtaccgaggaagggagtactctgcaggctacaacagcgacaacgtc180
tacatcaccgccgacaagcagaagaacggcatcaaggccaacttcaagatccgccacaac240
atcgaggacggcggcgtgcagctcgccgaccactaccagcagaacacccccatcggcgac300
ggccccgtgctgctgcccgacaaccactacctgagcttccagtccaagctgagcaaagac360
cccaacgagaagcgcgatcacatggtcctgctggagttcgtgaccgccgccgggatcact420
ctcggcatggacgagctgtacaagggcggttccggaggcatggtgagcaagggcgaggag480
ctgttcaccggggtggtgcccatcctggtcgagctggacggcgacgtaaacggccacaag540
ttcagcgtgtccggcgagggcgagggcgatgccacctacggcaagctgaccctgaagctg600
atctgcaccaccggcaagctgcccgtgccctggcccaccctcgtgaccaccctgggctac660
ggtctccaatgcttcgcccgctaccccgaccacatgaagcagcacgacttcttcaagtcc720
gccatgcccgaaggctacgtccaggagcgcaccatcttcttcaaggacgacggcaactac780
aagacccgcgccgaggtgaagttcgagggcgacaccctggtgaaccgcatcgagctgaag840
ggcatcgacttcaaggaggacggcaacatcctggggcacaagcttgagtacaacggcacc900
atagtagatctgatccctaaggtaaaaattgaactagtggtgaaggaggaggacgtcgat960
aacgtgatagacattatttgcgaaaatgcccgcacaggtaaccccggcgacggtaagata1020
tttgtgatcccagtggagcgagtggtcagggtgcgaaccaaagaggagggagcatctggt1080
ggtggatccggtggtggcggtgcatctggtatgaaaaaggtggaagccatcatcaggccc1140
gaaaagctggagatcgttaagaaggctctctcggacgctggatatgtgggtatgaccgtc1200
tctgaagtcaaaggccggggcgcaggtggcggcgatctgatccctaaggtaaaaattgaa1260
ctagtggtgaaggaggaggacgtcgataacgtgatagacattatttgcgaaaatgcccgc1320
acaggtaaccccggcgacggtaagatatttgtgatcccagtggagcgagtggtcagggtg1380
cgaaccaaagaggagggagcctccggcggtggcggtggctccggtggcgccagcggcatg1440
aaaaaggtggaagccatcatcaggcccgaaaagctggagatcgttaagaaggctctctcg1500
gacgctggatatgtgggtatgaccgtctctgaagtcaaaggccggggcgcaggtggcggc1560
gatctgatccctaaggtaaaaattgaactagtggtgaaggaggaggacgtcgataacgtg1620
atagacattatttgcgaaaatgcccgcacaggtaaccccggcgacggtaagatatttgtg1680
atcccagtggagcgagtggtcagggtgcgaaccaaagaggagggaaaggaagcactgtga1740
<210>44
<211>1740
<212>dna
<213>人工序列
<400>44
atgaaaaaggtggaagccatcatcaggcccgaaaagctggagatcgttaagaaggctctc60
tcggacgctggatatgtgggtatgaccgtctctgaagtcaaaggccggggcgtccagggc120
ggcatcttcgagaggtaccgaggaagggtgtactctgcaggctacaacagcgacaacgtc180
tacatcaccgccgacaagcagaagaacggcatcaaggccaacttcaagatccgccacaac240
atcgaggacggcggcgtgcagctcgccgaccactaccagcagaacacccccatcggcgac300
ggccccgtgctgctgcccgacaaccactacctgagcttccagtccaagctgagcaaagac360
cccaacgagaagcgcgatcacatggtcctgctggagttcgtgaccgccgccgggatcact420
ctcggcatggacgagctgtacaagggcggttccggaggcatggtgagcaagggcgaggag480
ctgttcaccggggtggtgcccatcctggtcgagctggacggcgacgtaaacggccacaag540
ttcagcgtgtccggcgagggcgagggcgatgccacctacggcaagctgaccctgaagctg600
atctgcaccaccggcaagctgcccgtgccctggcccaccctcgtgaccaccctgggctac660
ggtctccaatgcttcgcccgctaccccgaccacatgaagcagcacgacttcttcaagtcc720
gccatgcccgaaggctacgtccaggagcgcaccatcttcttcaaggacgacggcaactac780
aagacccgcgccgaggtgaagttcgagggcgacaccctggtgaaccgcatcgagctgaag840
ggcatcgacttcaaggaggacggcaacatcctggggcacaagcttgagtacaacggcacc900
atagtagatctgatccctaaggtaaaaattgaactagtggtgaaggaggaggacgtcgat960
aacgtgatagacattatttgcgaaaatgcccgcacaggtaaccccggcgacggtaagata1020
tttgtgatcccagtggagcgagtggtcagggtgcgaaccaaagaggagggagcatctggt1080
ggtggatccggtggtggcggtgcatctggtatgaaaaaggtggaagccatcatcaggccc1140
gaaaagctggagatcgttaagaaggctctctcggacgctggatatgtgggtatgaccgtc1200
tctgaagtcaaaggccggggcgcaggtggcggcgatctgatccctaaggtaaaaattgaa1260
ctagtggtgaaggaggaggacgtcgataacgtgatagacattatttgcgaaaatgcccgc1320
acaggtaaccccggcgacggtaagatatttgtgatcccagtggagcgagtggtcagggtg1380
cgaaccaaagaggagggagcctccggcggtggcggtggctccggtggcgccagcggcatg1440
aaaaaggtggaagccatcatcaggcccgaaaagctggagatcgttaagaaggctctctcg1500
gacgctggatatgtgggtatgaccgtctctgaagtcaaaggccggggcgcaggtggcggc1560
gatctgatccctaaggtaaaaattgaactagtggtgaaggaggaggacgtcgataacgtg1620
atagacattatttgcgaaaatgcccgcacaggtaaccccggcgacggtaagatatttgtg1680
atcccagtggagcgagtggtcagggtgcgaaccaaagaggagggaaaggaagcactgtga1740
<210>45
<211>1746
<212>dna
<213>人工序列
<400>45
atgaaaaaggtggaagccatcatcaggcccgaaaagctggagatcgttaagaaggctctc60
tcggacgctggatatgtgggtatgaccgtctctgaagtcaaaggccggggcgtccagggc120
ggcatcgtcgagaggtaccgaggaagggagtactctgcaggctacaacagcgacaacgtc180
tatatcatggccgacaagcagaagaacggcatcaaggccaacttcaagatccgccacaac240
gtcgaggacggcagcgtgcagctcgccgaccactaccagcagaacacccccatcggcgac300
ggccccgtgctgctgcccgacaaccactacctgagcttccagtccgtcctgagcaaagac360
cccaacgagaagcgcgatcacatggtcctgctggagttcgtgaccgccgccgggatcact420
ctcggcatggacgagctgtacaacgtggatggcggtagcggtggcaccggcagcaagggc480
gaggagctgttcaccggggtggtgcccatcctggtcgagctggacggcgacgtaaacggc540
cacaagttcagcgtgtccggcgagggcgagggcgatgccacctacggcaagctgaccctg600
aagctgatctgcaccaccggcaagctgcccgtgccctggcccaccctcgtgaccaccctc660
ggctacggcctgaagtgcttcgcccgctaccccgaccacatgaagcagcacgacttcttc720
aagtccgccatgcccgaaggctacgtccaggagcgcaccatcttcttcaaggacgacggc780
aactacaagacccgcgccgaggtgaagttcgagggcgacaccctggtgaaccgcatcgag840
ctgaagggcatcggcttcaaggaggacggcaacatcctggggcacaagctggagtacaac900
ggcaccatagtagatctgatccctaaggtaaaaattgaactagtggtgaaggaggaggac960
gtcgataacgtgatagacattatttgcgaaaatgcccgcacaggtaaccccggcgacggt1020
aagatatttgtgatcccagtggagcgagtggtcagggtgcgaaccaaagaggagggagca1080
tctggtggtggatccggtggtggcggtgcatctggtatgaaaaaggtggaagccatcatc1140
aggcccgaaaagctggagatcgttaagaaggctctctcggacgctggatatgtgggtatg1200
accgtctctgaagtcaaaggccggggcgcaggtggcggcgatctgatccctaaggtaaaa1260
attgaactagtggtgaaggaggaggacgtcgataacgtgatagacattatttgcgaaaat1320
gcccgcacaggtaaccccggcgacggtaagatatttgtgatcccagtggagcgagtggtc1380
agggtgcgaaccaaagaggagggagcctccggcggtggcggtggctccggtggcgccagc1440
ggcatgaaaaaggtggaagccatcatcaggcccgaaaagctggagatcgttaagaaggct1500
ctctcggacgctggatatgtgggtatgaccgtctctgaagtcaaaggccggggcgcaggt1560
ggcggcgatctgatccctaaggtaaaaattgaactagtggtgaaggaggaggacgtcgat1620
aacgtgatagacattatttgcgaaaatgcccgcacaggtaaccccggcgacggtaagata1680
tttgtgatcccagtggagcgagtggtcagggtgcgaaccaaagaggagggaaaggaagca1740
ctgtga1746
<210>46
<211>1746
<212>dna
<213>人工序列
<400>46
atgaaaaaggtggaagccatcatcaggcccgaaaagctggagatcgttaagaaggctctc60
tcggacgctggatatgtgggtatgaccgtctctgaagtcaaaggccggggcgtccagggc120
ggcatcttcgagaggtaccgaggaagggagtactctgcaggctacaacagcgacaacgtc180
tatatcatggccgacaagcagaagaacggcatcaaggccaacttcaagatccgccacaac240
gtcgaggacggcagcgtgcagctcgccgaccactaccagcagaacacccccatcggcgac300
ggccccgtgctgctgcccgacaaccactacctgagcttccagtccgtcctgagcaaagac360
cccaacgagaagcgcgatcacatggtcctgctggagttcgtgaccgccgccgggatcact420
ctcggcatggacgagctgtacaacgtggatggcggtagcggtggcaccggcagcaagggc480
gaggagctgttcaccggggtggtgcccatcctggtcgagctggacggcgacgtaaacggc540
cacaagttcagcgtgtccggcgagggcgagggcgatgccacctacggcaagctgaccctg600
aagctgatctgcaccaccggcaagctgcccgtgccctggcccaccctcgtgaccaccctc660
ggctacggcctgaagtgcttcgcccgctaccccgaccacatgaagcagcacgacttcttc720
aagtccgccatgcccgaaggctacgtccaggagcgcaccatcttcttcaaggacgacggc780
aactacaagacccgcgccgaggtgaagttcgagggcgacaccctggtgaaccgcatcgag840
ctgaagggcatcggcttcaaggaggacggcaacatcctggggcacaagctggagtacaac900
ggcaccatagtagatctgatccctaaggtaaaaattgaactagtggtgaaggaggaggac960
gtcgataacgtgatagacattatttgcgaaaatgcccgcacaggtaaccccggcgacggt1020
aagatatttgtgatcccagtggagcgagtggtcagggtgcgaaccaaagaggagggagca1080
tctggtggtggatccggtggtggcggtgcatctggtatgaaaaaggtggaagccatcatc1140
aggcccgaaaagctggagatcgttaagaaggctctctcggacgctggatatgtgggtatg1200
accgtctctgaagtcaaaggccggggcgcaggtggcggcgatctgatccctaaggtaaaa1260
attgaactagtggtgaaggaggaggacgtcgataacgtgatagacattatttgcgaaaat1320
gcccgcacaggtaaccccggcgacggtaagatatttgtgatcccagtggagcgagtggtc1380
agggtgcgaaccaaagaggagggagcctccggcggtggcggtggctccggtggcgccagc1440
ggcatgaaaaaggtggaagccatcatcaggcccgaaaagctggagatcgttaagaaggct1500
ctctcggacgctggatatgtgggtatgaccgtctctgaagtcaaaggccggggcgcaggt1560
ggcggcgatctgatccctaaggtaaaaattgaactagtggtgaaggaggaggacgtcgat1620
aacgtgatagacattatttgcgaaaatgcccgcacaggtaaccccggcgacggtaagata1680
tttgtgatcccagtggagcgagtggtcagggtgcgaaccaaagaggagggaaaggaagca1740
ctgtga1746
<210>47
<211>1746
<212>dna
<213>人工序列
<400>47
atgaaaaaggtggaagccatcatcaggcccgaaaagctggagatcgttaagaaggctctc60
tcggacgctggatatgtgggtatgaccgtctctgaagtcaaaggccggggcgtccagggc120
ggcatcttcgagaggtaccgaggaagggtgtactctgcaggctacaacagcgacaacgtc180
tatatcatggccgacaagcagaagaacggcatcaaggccaacttcaagatccgccacaac240
gtcgaggacggcagcgtgcagctcgccgaccactaccagcagaacacccccatcggcgac300
ggccccgtgctgctgcccgacaaccactacctgagcttccagtccgtcctgagcaaagac360
cccaacgagaagcgcgatcacatggtcctgctggagttcgtgaccgccgccgggatcact420
ctcggcatggacgagctgtacaacgtggatggcggtagcggtggcaccggcagcaagggc480
gaggagctgttcaccggggtggtgcccatcctggtcgagctggacggcgacgtaaacggc540
cacaagttcagcgtgtccggcgagggcgagggcgatgccacctacggcaagctgaccctg600
aagctgatctgcaccaccggcaagctgcccgtgccctggcccaccctcgtgaccaccctc660
ggctacggcctgaagtgcttcgcccgctaccccgaccacatgaagcagcacgacttcttc720
aagtccgccatgcccgaaggctacgtccaggagcgcaccatcttcttcaaggacgacggc780
aactacaagacccgcgccgaggtgaagttcgagggcgacaccctggtgaaccgcatcgag840
ctgaagggcatcggcttcaaggaggacggcaacatcctggggcacaagctggagtacaac900
ggcaccatagtagatctgatccctaaggtaaaaattgaactagtggtgaaggaggaggac960
gtcgataacgtgatagacattatttgcgaaaatgcccgcacaggtaaccccggcgacggt1020
aagatatttgtgatcccagtggagcgagtggtcagggtgcgaaccaaagaggagggagca1080
tctggtggtggatccggtggtggcggtgcatctggtatgaaaaaggtggaagccatcatc1140
aggcccgaaaagctggagatcgttaagaaggctctctcggacgctggatatgtgggtatg1200
accgtctctgaagtcaaaggccggggcgcaggtggcggcgatctgatccctaaggtaaaa1260
attgaactagtggtgaaggaggaggacgtcgataacgtgatagacattatttgcgaaaat1320
gcccgcacaggtaaccccggcgacggtaagatatttgtgatcccagtggagcgagtggtc1380
agggtgcgaaccaaagaggagggagcctccggcggtggcggtggctccggtggcgccagc1440
ggcatgaaaaaggtggaagccatcatcaggcccgaaaagctggagatcgttaagaaggct1500
ctctcggacgctggatatgtgggtatgaccgtctctgaagtcaaaggccggggcgcaggt1560
ggcggcgatctgatccctaaggtaaaaattgaactagtggtgaaggaggaggacgtcgat1620
aacgtgatagacattatttgcgaaaatgcccgcacaggtaaccccggcgacggtaagata1680
tttgtgatcccagtggagcgagtggtcagggtgcgaaccaaagaggagggaaaggaagca1740
ctgtga1746
- 还没有人留言评论。精彩留言会获得点赞!