一种基于EST-SSR标记的罗氏沼虾分类方法与流程
本发明涉及一种虾类的分类方法,尤其是一种基于est-ssr标记的罗氏沼虾分类方法。
背景技术:
罗氏沼虾属于我国重要的淡水虾类养殖品种之一。本发明属于虾类分子生物学dna标记技术与应用领域,具体涉及罗氏沼虾est数据中微卫星标记筛选,及利用这些标记开展罗氏沼虾不同群体之间遗传距离分析。
技术实现要素:
本发明旨在筛选到12个罗氏沼虾est—ssr分子标记位点,并建立一种准确、有效地利用分子标记检测不同来源罗氏沼虾遗传结构和遗传背景的方法,以鉴定出不同来源罗氏沼虾的遗传结构,为筛选和建立罗氏沼虾选育基础群体提供参考。即提供了一种基于est-ssr标记的罗氏沼虾分类方法。
本发明所提供的技术方案是这样的:
一种基于est-ssr标记的罗氏沼虾分类方法,包括提取罗氏沼虾的dna,使用est-ssr标记作为引物进行扩增;根据扩增结果对罗氏沼虾进行分类;所述est-ssr标记对为快速鉴定不同来源罗氏沼虾的遗传结构提供分子技术手段。
pcr的反应体系总体积为:
pcr的反应条件为:
1)94℃预变性5分钟;
2)94℃变性30秒;
3)55℃退火30秒;
4)72℃延伸30秒;
5)72℃延伸7分钟;
6)循环上述步骤1)-5)35次。
本发明与现有技术相比,具有以下优点:利用筛选到的12个具有丰富多态的est--ssr标记,对不同群体的罗氏沼虾dna样品进行pcr检测,利用str基因分型和生物技术和数据分析软件进行相关分析,能将生产中来源不同的罗氏沼虾群体进行其遗传结构和遗传聚类分析,为罗氏沼虾选育基础群体的选定和罗氏沼虾良种选育奠定基础。该方法与传统的方法相比,具有目的性强,作用效果直接的优点。且操作简单、检测快速、检测成本低,便于广泛推广使用。
附图说明
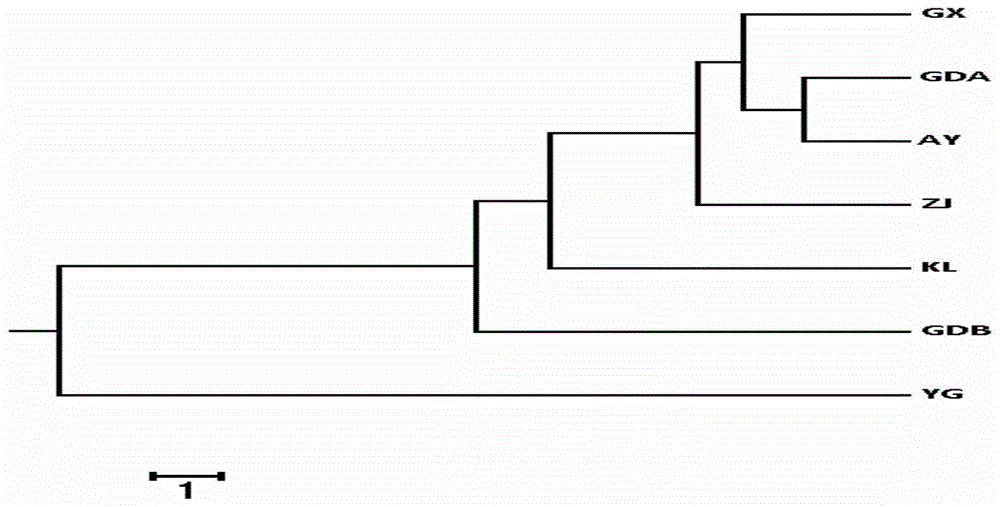
图1是七个罗氏沼虾群体的聚类分析图。
具体实施方式
下面结合具体实施例对权利要求做进一步的详细说明,但不限于以下实施例。
实施例1
本申请人2018年构建了罗氏沼虾脑、性腺等组织cdna文库,去冗余处理后得到87672条est序列,平均序列长度为1477bp,用trf(tandemrepeatsfinder)软件从该序列中查找到12138个微卫星。选取重复次数在5次以上的双碱基重复序列和重复次数在4次以上的三碱基或四碱基重复序列,使用引物设计软件primerprimer5.0进行引物设计,获得est-ssr引物60对。
本申请人利用获得60对est-ssr引物对10尾来自不同群体的罗氏沼虾样本进行pcr扩增,其中51对引物扩增出pcr条带、9对引物无扩增产物。经过筛选,最终获得12对具有多态性片段的引物作为鉴定罗氏沼虾群体的标记位点,见表1。
表112对微卫星引物序列及退火温度
本申请人在2018年4月-7月对7个不同来源罗氏沼虾群体进行了样品采集,分别为马来西亚养殖群体、泰国养殖群体、缅甸野生群体、浙江群体、广西群体、广东中山群体及广东佛山群体,每个群体30尾,共210尾。采样地点和数量见表2。每个样本剪取鳍条,保存于95%的乙醇中备用。
表27个罗氏沼虾群体的采样情况表
将12对微卫星引物对7个罗氏沼虾群体共210尾罗氏沼虾样本进行多态性检测,共获得89个等位基因,每对引物检测到的等位基因数为3~16个,平均为8.94个;其多态信息为0.591,具体信息见表3。
表312个微卫星位点的遗传多样性参数
本申请人利用数据统计及分析软件对210尾罗氏沼虾样本进行遗传多样性分析,群体遗传多样性的统计分析结果见表4,7个罗氏沼虾群体的平均等位基因数为(na)4.94~6.88,平均有效等位基因数为(ne)2.801~3.816,平均观测杂合度为(ho)0.313~0.3770,平均期望杂合度为(he)0.569~0.630,平均多态信息含量为(pic)
0.503~0.581;缅甸野生群体平均多态信息含量和平均期望杂合度最高为0.581和0.630;广西养殖群体的平均观察杂合度和平均期望杂合度最低为0.313和0.569。
表4七个罗氏沼虾群体的遗传多样性参数
本申请人对不同罗氏沼虾群体间进行遗传相似系数与遗传距离分析,依据nei(1978)计算群体间的遗传距离和遗传相似系数,结果表明:缅甸群体与广东中山群体遗传距离最远(0.274),说明两者亲缘关系较远;而广西群体与广东中山群体遗传距离最近(0.031),说明两者亲缘关系最近,详见表5。根据群体间的遗传距离矩阵构建7个群体的upgma聚类图(图1)。可以看出,7个罗氏沼虾群体聚成了两支,缅甸野生群体单独聚成一支;其他六个养殖群体聚成一支。
表5七个罗氏沼虾群体遗传距离与遗传相似系数
注:对角线以下为fst值,对角线以上为遗传距离.
具体的检测方法如下:
模板dna的提取
(1)取待检测虾的肌肉组织3mg剪碎后,加入0.5ml的裂解液(10mmol/ltris-hcl;0.1mol/ledta;0.5%sds;30mg/lrnase;100mg/l蛋白酶k,ph8.0),55℃消化1小时,其间不时轻轻摇动。
(2)加入等体积的酚/氯仿/异戊醇(25:24:1),颠倒混匀,室温下静置5分钟后,12000转/分钟离心10分钟,取上清液,再用氯仿抽提一次,室温下静置5分钟后,12000转/分钟离心10分钟,取上清液。
(3)加入2倍体积的无水乙醇,室温静置10分钟沉淀dna,12000转/分钟离心10分钟。
(4)用70%乙醇洗涤1次,12000转/分钟离心2分钟,吸去上清,室温静置干燥10分钟,加入50μlte(10mmol/ltris-hcl;1mmol/ledta,ph8.0)溶解dna,4℃贮存备用。
pcr反应体系为:
pcr反应条件为:
1)94℃预变性5分钟;
2)94℃变性30秒;
3)55℃退火30秒;
4)72℃延伸30秒;
5)72℃延伸7分钟;
6)循环上述步骤1)-5)35次
基因分型:使用dyy-8型稳压稳流电泳仪、凝胶成像系统和3730xl测序分析仪进行str序列分析,根据每个扩增条带的分子量的差异性,判断每个个体各基因座的基因型。
数据统计与分析:
1)用popgen32软件计算有效等位基因数(effectivenumbersofallele,ne),观测杂合度(observedheterozygosity,ho),期望杂合度(expectedheterozygosity,he),相对遗传距离(geneticdistance,d),遗传相似系数(geneticsimilarityindex,s),遗传分化指数(geneticdifferentiationindex,fst)和hardy-weinberg遗传偏离平衡指数(d)。
有效等位基因数(ne):
式中pi为第i个等位基因的频率,n为等位基因数目。
平均观测杂合度(ho):ho=观察到的杂合子数/观察个体总数;
平均期望杂合度(he):he=1-∑pi2
式中pi为第i个等位基因的频率。
遗传相似系数(s):sij=nij/[ni+nj+nij]
式中sij为任意两个个体间的遗传相似系数,nij为i个体和j个体共享位点数,ni、nj分别为i个体和j个体各自扩增的位点总数。
相对遗传距离(d):
式中sij为两种群间的遗传相似系数,si,sj分别表示种群i和种群j的遗传相似系数。
群本间遗传分化指数(fst):1-fst=(1-fis)×(1-fit)
式中fis、fst、fit分别代表群体内近交系数、群体间分化系数和总群体近交系数。
hardy-weinberg遗传偏离平衡指数(d):d=ho-he/he
2)多态信息含量(polymorphicinformationcontent,pic),
式中pi、pj分别为第i和第j个等位基因的频率,n为等位基因数目。
3)用mega4软件根据群体间的遗传相似性指数和遗传距离,采用非加权配对算术平均法(unweithtedpairgroupmethodusingarithmetic,upgma)构建5个罗氏沼虾群体的系统树,以分析群体间的亲缘关系。
本检测方法大约可以在三天内操作完成,能为要进行的不同来源罗氏沼虾群体遗传背景检测提供快速准确的检测结果。我们通过对不同等位基因的鉴定,是在dna水平上对罗氏沼虾遗传背景进行评估,目的性更强。用该方法检测一个样品所需的成本大约为0.6元,成本低,适合推广应用。
12对est-ssr标记序列如下:
unigene038721
unigene082899
unigene032300
unigene001327
unigene041668
unigene018861
unigene034642
unigene041463
unigene036095
unigene001885
unigene015914
unigene044258
备注:加粗加下划线核苷酸序列为est-ssr的重复序列区域。
序列表
<110>中国水产科学研究院珠江水产研究所
<120>一种基于est-ssr标记的罗氏沼虾分类方法
<130>1
<160>24
<170>siposequencelisting1.0
<210>1
<211>20
<212>dna
<213>人工引物()
<400>1
tgggcttggtccatatgagt20
<210>2
<211>20
<212>dna
<213>人工引物()
<400>2
tctccatcgaactgactccc20
<210>3
<211>20
<212>dna
<213>人工引物()
<400>3
atttctgctgtgagggcagt20
<210>4
<211>20
<212>dna
<213>人工引物()
<400>4
acaggtgtcgctttggactt20
<210>5
<211>20
<212>dna
<213>人工引物()
<400>5
tctgagggcttcttccatgt20
<210>6
<211>20
<212>dna
<213>人工引物()
<400>6
ggggccactgactcaaaaat20
<210>7
<211>20
<212>dna
<213>人工引物()
<400>7
cgggagggtattgtagcgta20
<210>8
<211>21
<212>dna
<213>人工引物()
<400>8
ccttgcagaaaggaccaatag21
<210>9
<211>20
<212>dna
<213>人工引物()
<400>9
tcgtttaagtccaaaggcag20
<210>10
<211>20
<212>dna
<213>人工引物()
<400>10
cttgtctcttgaagcccctg20
<210>11
<211>20
<212>dna
<213>人工引物()
<400>11
gcaagtgaggagggtagcag20
<210>12
<211>20
<212>dna
<213>人工引物()
<400>12
gcaagtgaggagggtagcag20
<210>13
<211>20
<212>dna
<213>人工引物()
<400>13
cgaatggggtcacattaggt20
<210>14
<211>20
<212>dna
<213>人工引物()
<400>14
tcacggtttgtgctcagttt20
<210>15
<211>19
<212>dna
<213>人工引物()
<400>15
gtgggcgatctccaaatag19
<210>16
<211>20
<212>dna
<213>人工引物()
<400>16
gctcaggggcacgtttaata20
<210>17
<211>20
<212>dna
<213>人工引物()
<400>17
ttcctcagcaccgtaacctc20
<210>18
<211>20
<212>dna
<213>人工引物()
<400>18
cgctcattcgatcacctgta20
<210>19
<211>19
<212>dna
<213>人工引物()
<400>19
cacattgagctcgctgaga19
<210>20
<211>18
<212>dna
<213>人工引物()
<400>20
agccatgcctgctcacat18
<210>21
<211>21
<212>dna
<213>人工引物()
<400>21
gcagctccgtagcagctataa21
<210>22
<211>20
<212>dna
<213>人工引物()
<400>22
agaaaagacgaagacgcgag20
<210>23
<211>20
<212>dna
<213>人工引物()
<400>23
gtggttaacgtcgctccttc20
<210>24
<211>20
<212>dna
<213>人工引物()
<400>24
catgttcgtgtgggagagaa20
- 还没有人留言评论。精彩留言会获得点赞!