一类稳定性提高的木聚糖酶突变体及其编码基因和应用的制作方法
本发明属于基因工程领域,具体涉及一类稳定性提高的新型木聚糖酶突变体及其编码基因和应用。
背景技术:
在养殖行业中,饲料中的木聚糖在动物消化系统中不能被有效的降解,同时还会影响其他营养成分的吸收利用,大大降低了饲料的转化效率。木聚糖酶(xylanase),通过随机的内切水解木聚糖主链的1,4-β-D-糖苷键,产生木糖或低聚木糖,是木聚糖降解过程中的关键酶。通过在饲料中添加木聚糖酶转化抗营养因子木聚糖对提高饲料转化率非常经济有效。
饲料生产时,酶制剂与饲料混匀后要经过高温造粒,在此过程中酶易变性失活;同时添加在饲料中的酶制剂通常需要在动物消化道内发挥作用,消化道内的蛋白酶对添加的酶制剂有降解作用,而胃液的酸性条件也极易使得酶制剂变性,高温、胃酸和蛋白酶对酶制剂的破坏作用极大的影响了酶制剂在饲料中的添加效果。
目前市场上大多数木聚糖酶最适温度在40~70℃之间,最适pH在5.0-7.0之间,在经过高温或在消化道中极易失活,解决这类问题的一种方法是利用包被剂和载体来提高酶的稳定性,这不但会增加酶制剂的生产成本,而且会严重影响酶制剂的生物利用率;另一种经济有效的方法就是通过改良酶的基因提高其稳定性。从酶的基因和结构入手,筛选得到适宜耐热、耐酸、耐内源蛋白酶的木聚糖酶对降低目前饲用木聚糖酶的生产成本,提高其利用效率具有重要的意义。
易错PCR技术是通过PCR反应扩增目的片段时,通过调整反应条件增加扩增时的突变频率,从而向目的基因中引入随机突变,构建突变体库,从而筛选需要的正向突变体。易错PCR技术可以很好地应用于蛋白质的分子改造中。
技术实现要素:
本发明提供了一类优化的木聚糖酶突变体及其编码基因和应用,通过人工基因突变和大量筛选的方法,对木聚糖酶(GenbankID:CAD48747.1)进行改良,筛选得到耐热性提高的突变体,并将该突变体基因转化到毕赤酵母中,构建得到重组木聚糖酶表达菌株。经验证所述重组木聚糖酶的耐热性以及在胃酸胃液中的稳定性均有所提高,从而有利于提高其在饲料工业中的应用效果。
为实现上述发明目的,本发明采用以下技术方案予以实现:
本发明提供了一种木聚糖酶突变体XYNTH1,所述XYNTH1的氨基酸序列如SEQ ID NO:3所示,XYNTH1是由氨基酸序列为SEQ ID NO:1的木聚糖酶XYNTH0第66位氨基酸由丝氨酸突变为苏氨酸得到。
本发明提供了所述的木聚糖酶突变体XYNTH1的编码基因,其核苷酸序列如SEQ ID NO:4所示。
本发明提供了一种木聚糖酶突变体XYNTH2,所述XYNTH2的氨基酸序列如SEQ ID NO:5所示,XYNTH2是由氨基酸序列为SEQ ID NO:1的木聚糖酶XYNTH0第183位氨基酸由丝氨酸突变为天冬酰胺得到。
本发明提供了所述的木聚糖酶突变体XYNTH2的编码基因,其核苷酸序列如SEQ ID NO:6所示。
本发明提供了一种木聚糖酶突变体XYNTH3,所述XYNTH3的氨基酸序列如SEQ ID NO:7所示,XYNTH3是由氨基酸序列为SEQ ID NO:1的木聚糖酶XYNTH0第66位氨基酸由丝氨酸突变为苏氨酸,同时第183位氨基酸由丝氨酸突变为天冬酰胺得到。
本发明提供了所述的木聚糖酶突变体XYNTH3的编码基因,其核苷酸序列如SEQ ID NO:8所示。
本发明提供了包含木聚糖酶编码基因的重组表达载体,所述载体分别包含核苷酸序列为SEQ ID NO:4、SEQ ID NO:6或SEQ ID NO:8的编码基因。
本发明提供了包含所述的重组表达载体的基因工程菌,所述基因工程菌为毕赤酵母GS115或毕赤酵母X33。
本发明提供了氨基酸序列如SEQ NO:1所示的天然木聚糖酶XYNTH0、所述的XYNTH1、所述的XYNTH2和所述的XYNTH3在制备用于酶解饲料的添加剂中的应用。
本发明提供了氨基酸序列如SEQ NO:1所示的天然木聚糖酶XYNTH0、所述的XYNTH1、所述的XYNTH2和所述的XYNTH3在制备用于提高动物生长性能的动物饲料添加剂中的应用。
与现有技术相比,本发明的优点和技术效果是:本发明通过对热多孢菌属来源的木聚糖酶XYNTH0进行突变,经过大量筛选,发现S66T单位点突变以及S183N单位点突变能引起其稳定性改变;继续整合两个突变位点得到S66T/S183N双位点突变体发现其稳定性进一步提高。
实验证明,本发明中的木聚糖酶突变体XYNTH1、XYNTH2和XYNTH3在80℃处理5min后的酶活残留率由突变前XYNTH0的57.3%分别提高到了75.3%、77.1%和88.5%,分别为原始木聚糖酶XYNTH0的1.31倍、1.35倍和1.54倍;90℃处理5min后的酶活残留率由XYNTH0的33.9%分别提高到了57.1%、54.3%和62.1%,分别为原始木聚糖酶XYNTH0的1.68倍、1.60倍和1.83倍;在人工胃液中处理2小时后的酶活残留率由XYNTH0的38.2%提高到58.3%、49.3%和68.4%,分别为原始木聚糖酶XYNTH0的1.53倍、1.29倍和1.76倍。
体外酶解麸皮实验证明本发明中的木聚糖酶能提高麸皮原料在消化道中的消化效率;保育猪养殖试验证明本发明中的木聚糖酶添加到饲料中能提高保育猪的生长性能。
通过本发明技术方案得到的木聚糖酶突变体的热稳定性和胃液耐受性较野生型有较大提高,使其在饲料添加领域有很好的应用潜力。
附图说明
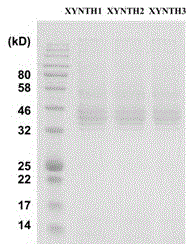
图1是本发明中的木聚糖酶突变体蛋白电泳图;
图2是本发明中的木聚糖酶耐热性比较结果;
图3是本发明中的木聚糖酶内源蛋白酶耐受性比较结果;
图4是本发明中的木聚糖酶体外酶解麸皮原料的应用实验结果。
具体实施方式
为了便于理解本发明,以下将结合较佳的实施例对本文发明做更全面、细致地描述,但本发明的保护范围并不限于以下具体实施例。
以下实施例中未作具体说明的分子生物学实验方法,可以参照《分子克隆实验指南》(第三版)J.萨姆布鲁克一书中所列的具体方法进行,或者按照试剂盒和产品说明书进行。具体实施例中使用的试剂和生物材料如无特殊说明均可从商业途径获得。
1.菌株和载体
毕赤酵母GS115,质粒pPIC9K,大肠杆菌DH5α,大肠杆菌BL21,质粒pET21a(+)购自Invitrogen公司。
2.试剂与培养基
质粒提取试剂盒,片段纯化回收试剂盒,限制性内切酶等购自宝生物工程(大连)有限公司;GeneMorph II随机突变PCR试剂盒购自Stratagene公司;氨苄青霉素,遗传霉素,IPTG等购自生工生物工程(上海)股份有限公司。
LB培养基:1%胰蛋白胨,0.5%酵母提取物,1%NaCl;
MD培养基:1.34%YNB,0.4mg/L生物素,2%葡萄糖;
YPD培养基:1%酵母提取物,2%蛋白胨,2%葡萄糖;
BMGY培养基:1%酵母提取物,2%蛋白胨,100mmol/L磷酸钾缓冲液(pH6.0),1.34%YNB,0.4mg/L生物素,1%甘油;
BMMY培养基:1%酵母提取物,2%蛋白胨,100mmol/L磷酸钾缓冲液(pH6.0),1.34%YNB,0.4mg/L生物素,1%甲醇;
以上培养基为固体时加入2%的琼脂粉。
3.实验方法
菌株培养条件:大肠杆菌37℃培养,酵母30℃培养。液体培养时摇床转速为200rpm。
毕赤酵母GS115转化方法:将活化的毕赤酵母GS115接种到含有20mL YPD液体培养基中,30℃摇瓶培养至OD600为1.2~1.5后低温离心收集菌体,依次用20mL冰冷无菌水和5mL浓度为1mol/L的冰冷山梨醇溶液清洗菌体,最后用1mL山梨醇溶液重悬菌体制得酵母感受态悬液。将100μL酵母感受态与10μL线性化载体混匀后转移到预冷的电转杯中电击转化,电转化的条件为1.5kV,6ms。电击后加入1mL山梨醇溶液,转移至1.5mL离心管中30℃温育1h。5000rpm离心5min,收集菌体涂布于MD筛选平板上倒置培养,直至长出阳性单克隆。
木聚糖酶活性检测根据中华人民共和国国家标准《GB/T 23874-2009》进行;木聚糖酶活性定义是指样品在温度37℃、pH值5.5的条件下,每分钟从5.0mg/mL木聚糖溶液中释放1μmol还原糖所需要的酶量,即为一个木聚糖酶活性单位,以U表示。
实施例1:突变筛选木聚糖酶XYNTH0的突变体
参考XYNTH0的氨基酸序列(SEQ ID NO:1),根据毕赤酵母密码子偏好性获得优化后的基因序列,其核苷酸序列如SEQ ID NO:2所示,合成该基因,并在5’端设计EcoR I限制性酶切位点,3’端设计Not I限制性酶切位点。
SEQ ID NO:1:
SEQ ID NO:2:
用GeneMorph II随机突变PCR试剂盒,以木聚糖酶XYNTH0的基因为模版,进行随机突变,使用的引物序列如下:
XYNTHF:5’-AGAATTCGATACCACTATTAC-3’;
XYNTHR:5’-CGCGGCCGCTTAGTTGGCGGT-3’;
将扩增好的随机突变PCR产物用EcoR I和Not I双酶切,纯化回收后连接到pET-21a(+)载体上,转化大肠杆菌BL21-DE3,氨苄青霉素抗性LB平板筛选阳性克隆,得到pET-XYNTHx。同样方法将合成的原始基因连接到pET-21a(+)载体上并转化大肠杆菌BL21-DE3,得到pET-XYNTH0。
将筛选的单菌落接种到96孔深孔板中。每个平板接入2个表达XYNTH0的单菌落为对照。每孔装入300uL LB液体培养基(含有100μg/mL氨苄青霉素),37℃200rpm震荡培养4小时后,转移50uL菌液到新的96孔平板保种,在平板剩余菌液中添加200uL含有IPTG的LB-Amp培养基,使IPTG终浓度为1mM,氨苄青霉素终浓度为100μg/mL,37℃200rpm摇床培养10h诱导表达木聚糖酶。
将诱导完成的菌液反复冻融进行破碎,将破碎后的细胞液离心取上清,进行热处理(80℃水浴5min),然后检测木聚糖酶的剩余活性。将剩余酶活高于对照的突变体基因进行测序。
筛选到以XYNTH0为出发模板的耐热性提高的单位点突变体S66T(命名为XYNTH1)和S183N(命名为XYNTH2)。
所述XYNTH1的氨基酸序列如SEQ ID NO:3所示,编码基因的核苷酸序列如SEQ ID NO:4所示。
SEQ ID NO:3:
SEQ ID NO:4:
所述XYNTH2的氨基酸序列如SEQ ID NO:5所示,编码基因的核苷酸序列如SEQ ID NO:6所示。
SEQ ID NO:5:
SEQ ID NO:6:
实施例2:融合PCR整合木聚糖酶正向突变位点
以XYNTH1的基因序列为模版,PCR扩增其基因中1~513bp位置的DNA片段,所用引物如下:
XYNTHF:5’-AGAATTCGATACCACTATTAC-3’;
XYNTHR(513):5’-TTGGTAATCATGAGATCCTAAG-3’;
以XYNTH2的基因序列为模版,PCR扩增其基因中491~902bp位置的DNA片段,所用引物如下:
XYNTHF(491):5’-CTTAGGATCTCATGATTACCAA-3’;
XYNTHR:5’-CGCGGCCGCTTAGTTGGCGGT-3’。
将以上扩增的两段PCR产物切胶回收,经PCR融合,以融合产物为模版,用上述XYNTHF/XYNTHR引物进行扩增,得到的PCR产物测序验证。
测序结果证明已得到序列如SEQ ID NO:8所示的DNA,说明经过融合PCR将S66T和S183N两个突变位点成功地整合到两一条基因序列中,将该序列对应的木聚糖酶突变体S66T/S183N命名为XYNTH3。
所述XYNTH3的氨基酸序列如SEQ ID NO:7所示,编码基因的核苷酸序列如SEQ ID NO:8所示。
SEQ ID NO:7:
SEQ ID NO:8:
实施例4:木聚糖酶突变体毕赤酵母高表达菌株构建
木聚糖酶突变体基因XYNTH1、XYNTH2和XYNTH3分别用EcoR I和Not I双酶切位点连接到pPIC9K质粒上并转化至大肠杆菌DH5α中,得到各突变体在毕赤酵母中的表达载体pPIC-XYNm,测序验证;将各突变体的表达载体用Sal I酶切线性化后电转入毕赤酵母GS115中,MD平板筛选得到突变体表达菌株的转化子。同样方法得到表达木聚糖酶XYNTH0的转化子。将各突变体的转化子用无菌水洗下,适当稀释,依次涂布于含不同浓度的遗传霉素(1mg/mL、2mg/mL、4mg/mL、8mg/mL)的YPD平板上,筛选高拷贝转化子;将高拷贝的转化子在YPD平板上划线活化后接于BMGY摇瓶中,30℃振荡培养18h后,加1%甲醇诱导,继续振荡培养,此后每24h添加培养体积1%的甲醇;诱导表达96h后,将培养液离心获得上清,蛋白电泳检测木聚糖酶表达情况,结果如图1。
实施例5:木聚糖酶突变体稳定性测定
按照常规方法测定温度耐受性和胃/胰蛋白酶耐受性。
1.热稳定性测定:
将XYNTH0、XYNTH1、XYNTH2和XYNTH3酶液分别在80℃、90℃水浴处理5min,以未处理酶液的酶活定义为100%,计算相对酶活力。木聚糖酶热稳定性检测结果如图2所示。
2.内源蛋白酶耐受性测定:
将XYNTH0、XYNTH1、XYNTH2和XYNTH3于pH2.0的人工胃液中40℃水浴消化2h,在pH5.5、37℃条件下测定其酶活,以未处理酶液的酶活定义为100%,计算相对酶活力。
将XYNTH0、XYNTH1、XYNTH2和XYNTH3于pH6.8的人工肠液中40℃水浴消化4h,在pH5.5、37℃条件下测定其酶活,以未处理酶液的酶活定义为100%,计算相对酶活力。
木聚糖酶内源蛋白酶耐受性测定结果如图3。结果显示XYNTH1、XYNTH2和XYNTH3在80℃处理5min后的酶活残留率由突变前XYNTH0的57.3%分别提高到了75.3%、77.1%和88.5%;90℃处理5min后的酶活残留率由XYNTH0的33.9%分别提高到了57.1%、54.3%和62.1%;在人工胃液中处理2小时后的酶活残留率由XYNTH0的38.2%提高到58.3%、49.3%和68.4%。
以上结果表明将XYNTH0第66位的丝氨酸突变为苏氨酸,或将183位的丝氨酸突变为天冬酰胺能提高其热稳定性和胃蛋白酶耐受性;整合两突变位点,将第66位的丝氨酸突变为苏氨酸的同时将183位的丝氨酸突变为天冬酰胺能进一步提高其热稳定性和胃蛋白酶耐受性。
实施例6:木聚糖酶体外酶解麸皮实验
以麸皮为底物,不添加酶制剂组为空白对照组;处理组分为XYNTH0处理组、XYNTH1处理组、XYNTH2处理组和XYNTH3处理组,分别添加300U/g木聚糖酶XYNTH0、XYNTH1、XYNTH2和XYNTH3;每组3个重复。
酶体外酶解麸皮过程包括以下步骤:
(1)用缓冲液调节pH至5.26,41℃,120rpm恒温振荡消化1h;
(2)用缓冲液调节pH至2.7,41℃,120rpm恒温振荡消化1h;
(3)用缓冲液调节pH至6.8,41℃,120rpm恒温振荡消化4h。
消化完成之后,测定消化后放入食糜干重和木聚糖含量(地衣酚-盐酸法),并计算干物质消失率、还原糖增长率和木聚糖降解率,结果如图4。
实验数据表明经突变体XYNTH1、XYNTH2或XYNTH3消化后的麸皮干物质消失率分别由突变前XYNTH0的21.1%提高到27.3%、30.2%和38.47%;而木聚糖降解率分别由XYNTH0的15.6%提高到了25.6、26.1%和35.9%。这说明本发明中突变后的木聚糖酶XYNTH1、XYNTH2和XYNTH3在模拟消化道环境中降解麸皮原料效率更高。
本实施例为了便于体现本发明所述木聚糖酶突变体的在消化饲料原料中应用,并不局限于对麸皮的酶解应用,所述的木聚糖酶突变体可以应用于其它饲料原料的酶解,并提高饲料利用率。
实施例7、本发明中木聚糖酶突变体对保育猪生产性能的影响
试验目的:通过检测保育猪生产性能指标,验证本发明中的木聚糖酶在保育猪养殖中的应用效果。
试验产品:木聚糖酶XYNTH0/XYNTH1/XYNTH2/XYNTH3(10万U/g)
试验设计:选用健康体重相近,产期相近的13.5kg左右的良种保育猪20栏,每栏11头猪,按照头数和均重相近的原则分成5个组,每个组4个重复。对照组饲喂普通基础日粮,其余试验组饲喂添加100ppm相应的木聚糖酶的基础日粮,验料表明各组饲料其它成分基本无差异。
试验测定:在试验的开始及结束以重复为单位进行试猪体重的测定。以重复为单位记录饲料消耗、腹泻头数,以检测平均采食量、平均日增重和腹泻频率。
表1保育猪料中使用木聚糖酶对保育猪生产性能的影响
从以上动物数据可以看出,使用本发明的木聚糖酶后,采食量和日增重有所提高,料重比较不使用木聚糖酶的对照组分别降低了2.4%、3.4%、4.4%和2.7%,腹泻都很少,没有发现差异。综合以上分析得出结论,使用本发明的木聚糖酶保育猪具有更好的生长性能。
本实施例7为了便于体现本发明所述木聚糖酶的应用,并不局限于在保育猪养殖中的应用,所述的木聚糖酶可以添加到基础日粮中,并应用于其它畜禽养殖或水产养殖中,可以在鸡、鸭、牛等养殖过程中配合饲料添加饲喂。
以上实施例仅用以说明本发明的技术方案,而非对其进行限制;尽管参照前述实施例对本发明进行了详细的说明,对于本领域的普通技术人员来说,依然可以对前述实施例所记载的技术方案进行修改,或者对其中部分技术特征进行等同替换;而这些修改或替换,并不使相应技术方案的本质脱离本发明所要求保护的技术方案的精神和范围。
序列表
<110> 青岛红樱桃生物技术有限公司
<120> 一类稳定性提高的木聚糖酶突变体及其编码基因和应用
<160> 8
<170> SIPOSequenceListing 1.0
<210> 1
<211> 301
<212> PRT
<213> 热多孢菌(Thermopolyspora flexuosa)
<400> 1
Asp Thr Thr Ile Thr Gln Asn Gln Thr Gly Tyr Asp Asn Gly Tyr Phe
1 5 10 15
Tyr Ser Phe Trp Thr Asp Ala Pro Gly Thr Val Ser Met Thr Leu His
20 25 30
Ser Gly Gly Ser Tyr Ser Thr Ser Trp Arg Asn Thr Gly Asn Phe Val
35 40 45
Ala Gly Lys Gly Trp Ser Thr Gly Gly Arg Arg Thr Val Thr Tyr Asn
50 55 60
Ala Ser Phe Asn Pro Ser Gly Asn Ala Tyr Leu Thr Leu Tyr Gly Trp
65 70 75 80
Thr Arg Asn Pro Leu Val Glu Tyr Tyr Ile Val Glu Ser Trp Gly Thr
85 90 95
Tyr Arg Pro Thr Gly Thr Tyr Lys Gly Thr Val Thr Thr Asp Gly Gly
100 105 110
Thr Tyr Asp Ile Tyr Glu Thr Trp Arg Tyr Asn Ala Pro Ser Ile Glu
115 120 125
Gly Thr Arg Thr Phe Gln Gln Phe Trp Ser Val Arg Gln Gln Lys Arg
130 135 140
Thr Ser Gly Thr Ile Thr Ile Gly Asn His Phe Asp Ala Trp Ala Arg
145 150 155 160
Ala Gly Met Asn Leu Gly Ser His Asp Tyr Gln Ile Met Ala Thr Glu
165 170 175
Gly Tyr Gln Ser Ser Gly Ser Ser Thr Val Ser Ile Ser Glu Gly Gly
180 185 190
Asn Pro Gly Asn Pro Gly Asn Pro Gly Asn Pro Gly Asn Pro Gly Asn
195 200 205
Pro Gly Asn Pro Gly Gly Gly Cys Val Ala Thr Leu Ser Ala Gly Gln
210 215 220
Gln Trp Ser Asp Arg Tyr Asn Leu Asn Val Ser Val Ser Gly Ser Asn
225 230 235 240
Asn Trp Thr Val Arg Met Asp Val Pro Tyr Pro Ala Arg Ile Ile Ala
245 250 255
Thr Trp Asn Ile His Ala Gln Trp Pro Glu Ser Gln Val Leu Ile Ala
260 265 270
Arg Pro Asn Gly Asn Gly Asn Asn Trp Gly Val Thr Ile Gln His Asn
275 280 285
Gly Asn Trp Thr Trp Pro Thr Val Thr Cys Thr Ala Asn
290 295 300
<210> 2
<211> 906
<212> DNA
<213> 热多孢菌(Thermopolyspora flexuosa)
<400> 2
gataccacta ttactcaaaa ccaaactggt tacgataacg gatactttta ctccttttgg 60
actgatgccc caggtactgt ttctatgacc ttgcattctg gtggatcata ctccacctca 120
tggcgtaaca ccggcaactt tgttgccggt aagggatggt ccaccggagg acgtcgtact 180
gttacctaca acgcctcttt taacccatcc ggaaacgctt acttgacctt atacggttgg 240
actcgtaacc ctcttgttga atactacatt gttgaatcat ggggaaccta cagacctacg 300
ggaacctaca agggaactgt taccactgat ggaggaactt acgatattta cgaaacttgg 360
cgttacaacg ctccttccat tgaaggtact agaacctttc aacaattttg gtcagttcgt 420
caacaaaaga gaacttccgg tactattact attggaaacc attttgatgc ttgggctaga 480
gctggtatga acttaggatc tcatgattac caaattatgg ccaccgaagg ataccaatct 540
tccggatcat ccaccgtttc tatttccgaa ggtggaaacc caggtaaccc aggaaaccca 600
ggaaaccctg gtaaccctgg taacccaggt aacccaggag gtggttgtgt tgccactttg 660
tctgccggtc aacaatggtc agatagatac aacttaaacg tttcagtttc aggttctaac 720
aactggaccg ttcgtatgga tgttccatac cctgctagaa ttattgccac ttggaacatt 780
catgctcaat ggcctgaatc acaagtttta attgctcgtc ctaacggaaa cggaaacaac 840
tggggagtta ctattcaaca taacggaaac tggacttggc ctaccgttac ctgtaccgcc 900
aactaa 906
<210> 3
<211> 301
<212> PRT
<213> 热多孢菌(Thermopolyspora flexuosa)
<400> 3
Asp Thr Thr Ile Thr Gln Asn Gln Thr Gly Tyr Asp Asn Gly Tyr Phe
1 5 10 15
Tyr Ser Phe Trp Thr Asp Ala Pro Gly Thr Val Ser Met Thr Leu His
20 25 30
Ser Gly Gly Ser Tyr Ser Thr Ser Trp Arg Asn Thr Gly Asn Phe Val
35 40 45
Ala Gly Lys Gly Trp Ser Thr Gly Gly Arg Arg Thr Val Thr Tyr Asn
50 55 60
Ala Thr Phe Asn Pro Ser Gly Asn Ala Tyr Leu Thr Leu Tyr Gly Trp
65 70 75 80
Thr Arg Asn Pro Leu Val Glu Tyr Tyr Ile Val Glu Ser Trp Gly Thr
85 90 95
Tyr Arg Pro Thr Gly Thr Tyr Lys Gly Thr Val Thr Thr Asp Gly Gly
100 105 110
Thr Tyr Asp Ile Tyr Glu Thr Trp Arg Tyr Asn Ala Pro Ser Ile Glu
115 120 125
Gly Thr Arg Thr Phe Gln Gln Phe Trp Ser Val Arg Gln Gln Lys Arg
130 135 140
Thr Ser Gly Thr Ile Thr Ile Gly Asn His Phe Asp Ala Trp Ala Arg
145 150 155 160
Ala Gly Met Asn Leu Gly Ser His Asp Tyr Gln Ile Met Ala Thr Glu
165 170 175
Gly Tyr Gln Ser Ser Gly Ser Ser Thr Val Ser Ile Ser Glu Gly Gly
180 185 190
Asn Pro Gly Asn Pro Gly Asn Pro Gly Asn Pro Gly Asn Pro Gly Asn
195 200 205
Pro Gly Asn Pro Gly Gly Gly Cys Val Ala Thr Leu Ser Ala Gly Gln
210 215 220
Gln Trp Ser Asp Arg Tyr Asn Leu Asn Val Ser Val Ser Gly Ser Asn
225 230 235 240
Asn Trp Thr Val Arg Met Asp Val Pro Tyr Pro Ala Arg Ile Ile Ala
245 250 255
Thr Trp Asn Ile His Ala Gln Trp Pro Glu Ser Gln Val Leu Ile Ala
260 265 270
Arg Pro Asn Gly Asn Gly Asn Asn Trp Gly Val Thr Ile Gln His Asn
275 280 285
Gly Asn Trp Thr Trp Pro Thr Val Thr Cys Thr Ala Asn
290 295 300
<210> 4
<211> 906
<212> DNA
<213> 热多孢菌(Thermopolyspora flexuosa)
<400> 4
gataccacta ttactcaaaa ccaaactggt tacgataacg gatactttta ctccttttgg 60
actgatgccc caggtactgt ttctatgacc ttgcattctg gtggatcata ctccacctca 120
tggcgtaaca ccggcaactt tgttgccggt aagggatggt ccaccggagg acgtcgtact 180
gttacctaca acgccacctt taacccatcc ggaaacgctt acttgacctt atacggttgg 240
actcgtaacc ctcttgttga atactacatt gttgaatcat ggggaaccta cagacctacg 300
ggaacctaca agggaactgt taccactgat ggaggaactt acgatattta cgaaacttgg 360
cgttacaacg ctccttccat tgaaggtact agaacctttc aacaattttg gtcagttcgt 420
caacaaaaga gaacttccgg tactattact attggaaacc attttgatgc ttgggctaga 480
gctggtatga acttaggatc tcatgattac caaattatgg ccaccgaagg ataccaatct 540
tccggatcat ccaccgtttc tatttccgaa ggtggaaacc caggtaaccc aggaaaccca 600
ggaaaccctg gtaaccctgg taacccaggt aacccaggag gtggttgtgt tgccactttg 660
tctgccggtc aacaatggtc agatagatac aacttaaacg tttcagtttc aggttctaac 720
aactggaccg ttcgtatgga tgttccatac cctgctagaa ttattgccac ttggaacatt 780
catgctcaat ggcctgaatc acaagtttta attgctcgtc ctaacggaaa cggaaacaac 840
tggggagtta ctattcaaca taacggaaac tggacttggc ctaccgttac ctgtaccgcc 900
aactaa 906
<210> 5
<211> 301
<212> PRT
<213> 热多孢菌(Thermopolyspora flexuosa)
<400> 5
Asp Thr Thr Ile Thr Gln Asn Gln Thr Gly Tyr Asp Asn Gly Tyr Phe
1 5 10 15
Tyr Ser Phe Trp Thr Asp Ala Pro Gly Thr Val Ser Met Thr Leu His
20 25 30
Ser Gly Gly Ser Tyr Ser Thr Ser Trp Arg Asn Thr Gly Asn Phe Val
35 40 45
Ala Gly Lys Gly Trp Ser Thr Gly Gly Arg Arg Thr Val Thr Tyr Asn
50 55 60
Ala Ser Phe Asn Pro Ser Gly Asn Ala Tyr Leu Thr Leu Tyr Gly Trp
65 70 75 80
Thr Arg Asn Pro Leu Val Glu Tyr Tyr Ile Val Glu Ser Trp Gly Thr
85 90 95
Tyr Arg Pro Thr Gly Thr Tyr Lys Gly Thr Val Thr Thr Asp Gly Gly
100 105 110
Thr Tyr Asp Ile Tyr Glu Thr Trp Arg Tyr Asn Ala Pro Ser Ile Glu
115 120 125
Gly Thr Arg Thr Phe Gln Gln Phe Trp Ser Val Arg Gln Gln Lys Arg
130 135 140
Thr Ser Gly Thr Ile Thr Ile Gly Asn His Phe Asp Ala Trp Ala Arg
145 150 155 160
Ala Gly Met Asn Leu Gly Ser His Asp Tyr Gln Ile Met Ala Thr Glu
165 170 175
Gly Tyr Gln Ser Ser Gly Asn Ser Thr Val Ser Ile Ser Glu Gly Gly
180 185 190
Asn Pro Gly Asn Pro Gly Asn Pro Gly Asn Pro Gly Asn Pro Gly Asn
195 200 205
Pro Gly Asn Pro Gly Gly Gly Cys Val Ala Thr Leu Ser Ala Gly Gln
210 215 220
Gln Trp Ser Asp Arg Tyr Asn Leu Asn Val Ser Val Ser Gly Ser Asn
225 230 235 240
Asn Trp Thr Val Arg Met Asp Val Pro Tyr Pro Ala Arg Ile Ile Ala
245 250 255
Thr Trp Asn Ile His Ala Gln Trp Pro Glu Ser Gln Val Leu Ile Ala
260 265 270
Arg Pro Asn Gly Asn Gly Asn Asn Trp Gly Val Thr Ile Gln His Asn
275 280 285
Gly Asn Trp Thr Trp Pro Thr Val Thr Cys Thr Ala Asn
290 295 300
<210> 6
<211> 905
<212> DNA
<213> 热多孢菌(Thermopolyspora flexuosa)
<400> 6
ataccactat tactcaaaac caaactggtt acgataacgg atacttttac tccttttgga 60
ctgatgcccc aggtactgtt tctatgacct tgcattctgg tggatcatac tccacctcat 120
ggcgtaacac cggcaacttt gttgccggta agggatggtc caccggagga cgtcgtactg 180
ttacctacaa cgcctctttt aacccatccg gaaacgctta cttgacctta tacggttgga 240
ctcgtaaccc tcttgttgaa tactacattg ttgaatcatg gggaacctac agacctacgg 300
gaacctacaa gggaactgtt accactgatg gaggaactta cgatatttac gaaacttggc 360
gttacaacgc tccttccatt gaaggtacta gaacctttca acaattttgg tcagttcgtc 420
aacaaaagag aacttccggt actattacta ttggaaacca ttttgatgct tgggctagag 480
ctggtatgaa cttaggatct catgattacc aaattatggc caccgaagga taccaatctt 540
ccggaaactc caccgtttct atttccgaag gtggaaaccc aggtaaccca ggaaacccag 600
gaaaccctgg taaccctggt aacccaggta acccaggagg tggttgtgtt gccactttgt 660
ctgccggtca acaatggtca gatagataca acttaaacgt ttcagtttca ggttctaaca 720
actggaccgt tcgtatggat gttccatacc ctgctagaat tattgccact tggaacattc 780
atgctcaatg gcctgaatca caagttttaa ttgctcgtcc taacggaaac ggaaacaact 840
ggggagttac tattcaacat aacggaaact ggacttggcc taccgttacc tgtaccgcca 900
actaa 905
<210> 7
<211> 301
<212> PRT
<213> 热多孢菌(Thermopolyspora flexuosa)
<400> 7
Asp Thr Thr Ile Thr Gln Asn Gln Thr Gly Tyr Asp Asn Gly Tyr Phe
1 5 10 15
Tyr Ser Phe Trp Thr Asp Ala Pro Gly Thr Val Ser Met Thr Leu His
20 25 30
Ser Gly Gly Ser Tyr Ser Thr Ser Trp Arg Asn Thr Gly Asn Phe Val
35 40 45
Ala Gly Lys Gly Trp Ser Thr Gly Gly Arg Arg Thr Val Thr Tyr Asn
50 55 60
Ala Thr Phe Asn Pro Ser Gly Asn Ala Tyr Leu Thr Leu Tyr Gly Trp
65 70 75 80
Thr Arg Asn Pro Leu Val Glu Tyr Tyr Ile Val Glu Ser Trp Gly Thr
85 90 95
Tyr Arg Pro Thr Gly Thr Tyr Lys Gly Thr Val Thr Thr Asp Gly Gly
100 105 110
Thr Tyr Asp Ile Tyr Glu Thr Trp Arg Tyr Asn Ala Pro Ser Ile Glu
115 120 125
Gly Thr Arg Thr Phe Gln Gln Phe Trp Ser Val Arg Gln Gln Lys Arg
130 135 140
Thr Ser Gly Thr Ile Thr Ile Gly Asn His Phe Asp Ala Trp Ala Arg
145 150 155 160
Ala Gly Met Asn Leu Gly Ser His Asp Tyr Gln Ile Met Ala Thr Glu
165 170 175
Gly Tyr Gln Ser Ser Gly Asn Ser Thr Val Ser Ile Ser Glu Gly Gly
180 185 190
Asn Pro Gly Asn Pro Gly Asn Pro Gly Asn Pro Gly Asn Pro Gly Asn
195 200 205
Pro Gly Asn Pro Gly Gly Gly Cys Val Ala Thr Leu Ser Ala Gly Gln
210 215 220
Gln Trp Ser Asp Arg Tyr Asn Leu Asn Val Ser Val Ser Gly Ser Asn
225 230 235 240
Asn Trp Thr Val Arg Met Asp Val Pro Tyr Pro Ala Arg Ile Ile Ala
245 250 255
Thr Trp Asn Ile His Ala Gln Trp Pro Glu Ser Gln Val Leu Ile Ala
260 265 270
Arg Pro Asn Gly Asn Gly Asn Asn Trp Gly Val Thr Ile Gln His Asn
275 280 285
Gly Asn Trp Thr Trp Pro Thr Val Thr Cys Thr Ala Asn
290 295 300
<210> 8
<211> 906
<212> DNA
<213> 热多孢菌(Thermopolyspora flexuosa)
<400> 8
gataccacta ttactcaaaa ccaaactggt tacgataacg gatactttta ctccttttgg 60
actgatgccc caggtactgt ttctatgacc ttgcattctg gtggatcata ctccacctca 120
tggcgtaaca ccggcaactt tgttgccggt aagggatggt ccaccggagg acgtcgtact 180
gttacctaca acgccacctt taacccatcc ggaaacgctt acttgacctt atacggttgg 240
actcgtaacc ctcttgttga atactacatt gttgaatcat ggggaaccta cagacctacg 300
ggaacctaca agggaactgt taccactgat ggaggaactt acgatattta cgaaacttgg 360
cgttacaacg ctccttccat tgaaggtact agaacctttc aacaattttg gtcagttcgt 420
caacaaaaga gaacttccgg tactattact attggaaacc attttgatgc ttgggctaga 480
gctggtatga acttaggatc tcatgattac caaattatgg ccaccgaagg ataccaatct 540
tccggaaact ccaccgtttc tatttccgaa ggtggaaacc caggtaaccc aggaaaccca 600
ggaaaccctg gtaaccctgg taacccaggt aacccaggag gtggttgtgt tgccactttg 660
tctgccggtc aacaatggtc agatagatac aacttaaacg tttcagtttc aggttctaac 720
aactggaccg ttcgtatgga tgttccatac cctgctagaa ttattgccac ttggaacatt 780
catgctcaat ggcctgaatc acaagtttta attgctcgtc ctaacggaaa cggaaacaac 840
tggggagtta ctattcaaca taacggaaac tggacttggc ctaccgttac ctgtaccgcc 900
aactaa 906
- 还没有人留言评论。精彩留言会获得点赞!