一种重组红霉素工程菌及其构建方法和筛选方法和应用与流程
本申请涉及红霉素生物合成
技术领域:
,尤其涉及一种重组红霉素工程菌及其构建方法和筛选方法,以及该重组红霉素工程菌在生产红霉素中的应用。
背景技术:
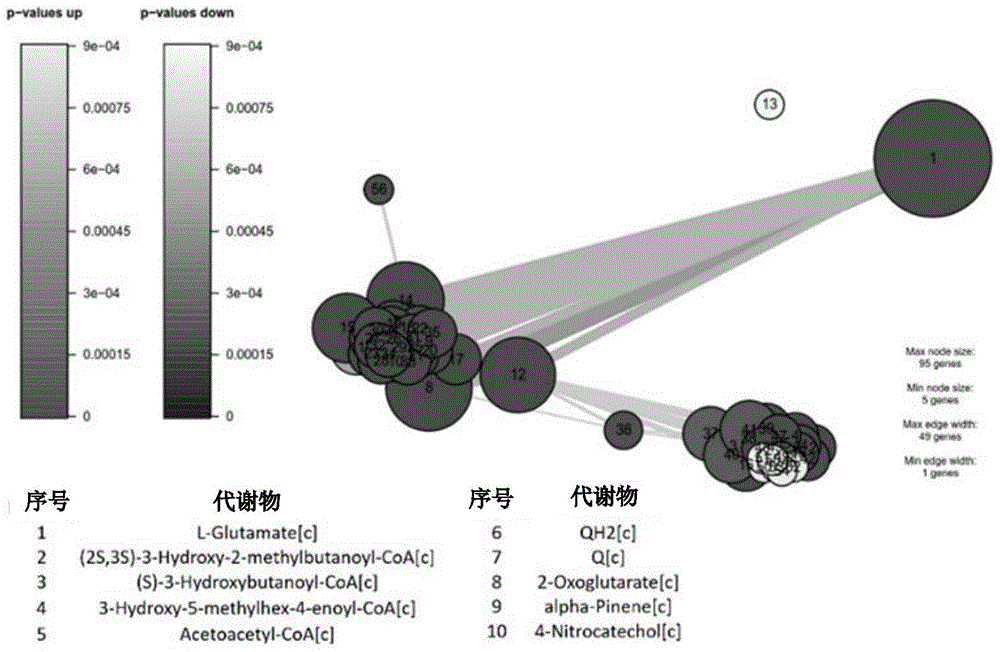
:红霉素(erythromycin,er)是由红霉素高产菌株红色糖多孢菌(saccharopolysporaerythraea)合成的次级代谢产物。在工业发酵生产中,红霉素的生产单位远低于青霉素等其他抗生素浓度,在老药新用及确保国家完整产业链的背景下,提高红霉素产量仍具有重要的战略意义。在现有技术中,工业上提高合成红霉素产量的常规手段为:采用诱变育种技术对工业红霉素产生菌进行改造,然后经过多轮筛选获得高产红霉素的红霉素产生菌突变株进行工业发酵生产。例如,红霉素高产菌s.erythraeahl3168-e3(或称红霉素高产菌hl3168-e3)为诱变获得的高产红霉素工业菌株。但是,一方面,采用诱变育种技术改造红霉素产生菌往往需要耗费大量劳动力,并且诱变所得菌稳定性差,其生理性状经多代培养之后可能发生改变;另一方面,由于诱变育种多是随机诱变,所以筛选获得的高产突变株可能存在过度诱变的问题,即:包含若干个本质上不利于红霉素产量提高的突变。此外,现有技术中存在利用代谢工程技术提高红霉素产生菌生物合成红霉素产量的策略,其多聚焦于两点:1、采用基因工程技术,提高红霉素前体的积累,进而提高红霉素的浓度;2、通过转录调控的控制技术,以提高红霉素生物合成基因簇的转录,进而提高红霉素的浓度,但是只有极少数策略在工业菌株中得到有效验证。随着核酸测序技术的快速发展,比较组学技术逐渐被应用于发掘提高工业微生物合成次级代谢产物的研究中,具有全面、有效、靶向精准度高的优点。但目前,尚无应用比较组学的定向代谢策略来提高红霉素的生物合成产量。技术实现要素:本申请实施例提供了一种重组红霉素工程菌及其筛选方法与在生产红霉素中的应用,通过比较组学的定向代谢策略来预测与红霉素产量相关的基因,将所述与红霉素产量相关的基因作为改造靶点,构建多个工程菌,然后通过发酵试验筛选重组红霉素工程菌,从而有效提高红霉素的生物合成产量。根据本申请的一方面,提供一种重组红霉素工程菌,是在红霉素产生菌中同时过量表达suca基因和sucb基因所得,所述suca基因的核苷酸序列如seqidno.1所示,所述sucb基因的核苷酸序列如seqidno.2所示。根据本申请的另一方面,提供上述重组红霉素工程菌在生产红霉素中的应用。在一些实施例中,所述应用为:使用上述重组红霉素工程菌发酵生产红霉素,以提高红霉素的生物合成产量。根据本申请的另一方面,提供上述重组红霉素工程菌的构建方法,包括步骤:构建重组质粒pib-sucb:以红霉素产生菌的基因组作为模板,以引物1和引物2进行pcr扩增,获得sucb基因的扩增片段;将所述sucb基因的扩增片段连接至含启动子erme*p的质粒载体中,获得重组质粒pib-sucb;构建重组质粒pib-sucba:以红霉素高产菌的基因组作为模板,采用引物对引物3和引物4进行pcr扩增,获得suca基因的扩增片段;将所述suca基因的扩增片段连接至所述重组质粒pib-sucb中,获得重组质粒pib-sucba;以及,将所述重组质粒pib-sucba转入至红霉素高产菌中,获得所述重组红霉素工程菌;其中,所述引物1的核苷酸序列如seqidno.3所示,所述引物2的核苷酸序列如seqidno.4所示,所述引物3的核苷酸序列如seqidno.5所示,所述引物4的核苷酸序列如seqidno.6所示。在上述构建方法中,尤其选择红霉素高产菌hl3168-e3作为原始菌株(或出发菌株)。即,在上述构建方法中,所述红霉素产生菌为红霉素高产菌hl3168-e3。在一些实施例中,在所述构建重组质粒pib-sucb的步骤中,以gibson组装克隆法将所述sucb基因的扩增片段连接至含启动子erme*p的质粒载体中。在一些实施例中,在所述构建重组质粒pib-sucba的步骤中,以gibson组装克隆法将所述suca基因的扩增片段连接至重组质粒pib-sucb中。根据本申请的另一方面,提供上述重组红霉素工程菌的筛选方法。包括以下步骤:获得多个预测基因:利用比较组学技术,预测红霉素产生菌中与红霉素产量相关的基因,以获得多个预测基因,所述多个预测基因包括suca基因和所述sucb基因,其中,所述suca基因的核苷酸序列为seqidno.1,所述sucb基因的核苷酸序列为seqidno.2;构建多个重组质粒:各个所述重组质粒携带有所述多个预测基因中的至少一个基因,其中,一重组质粒上同时携带有所述suca基因和所述sucb基因;构建多个工程菌:将各个所述重组质粒分别转入至所述红霉素产生菌中,获得多个工程菌,其中,一工程菌体内重组质粒上同时携带有所述suca基因和所述sucb基因;以及筛选获得重组红霉素工程菌:以所述红霉素产生菌作为对照,在相同培养条件下,对所述多个工程菌分别进行红霉素合成的发酵培养,检测发酵液中红霉素的浓度,并将各个工程菌发酵液中红霉素的浓度与所述红霉素产生菌发酵液中红霉素的浓度进行对比,以筛选重组红霉素工程菌,其中,体内重组质粒上同时携带有所述suca基因和所述sucb基因的工程菌对应发酵液中红霉素的浓度最高,所述体内重组质粒上同时携带有所述suca基因和所述sucb基因的工程菌即为重组红霉素工程菌。在上述筛选方法中,尤其选择红霉素高产菌hl3168-e3作为研究对象。即,在上述筛选方法中,所述红霉素产生菌为红霉素高产菌hl3168-e3。需要说明的是,所述与红霉素产量相关的基因是促进红霉素合成的基因或抑制红霉素合成的基因。因此,构建所述重组红霉素工程菌可以是在所述红霉素产生菌中过表达促进红霉素合成的基因,如:同时过表达suca基因和所述sucb基因。所述红霉素高产菌hl3168-e3是已有报道的红霉素工业生产菌,相关报道可见中国专利申请cn200910194419.4、cn201010154463.5和cn200710173703.4。在一些实施例中,所述获得多个预测基因的步骤进一步包括以下步骤:确定单碱基突变:以红色糖多孢菌标准株nrrl23338全基因组作为对照,识别所述红霉素高产菌hl3168-e3全基因组发生的所有单碱基突变;建立基因组规模代谢模型:建立基因组规模代谢模型:将所述红霉素高产菌hl3168-e3全基因组上的基因间区单碱基突变,以及编码区上的非同义单碱基突变和同义单碱基突变对映至代谢途径,以建立基因组规模代谢模型;预测关键代谢节点和代谢途径中的至少一者:通过所述基因组规模代谢模型,以预测与红霉素产量相关的关键代谢节点和代谢途径中的至少一者;预测报告代谢产物:分别对所述红色糖多孢菌标准株nrrl23338和所述红霉素高产菌hl3168-e3在生长对数期的转录组,以及在稳定期的转录组进行测序比对,获得两者之间在生长对数期的差异表达基因和在稳定期的差异表达基因,并分别对映至所述基因组规模代谢模型中,预测报告代谢物;获得多个预测基因:所述报告代谢物周围发生非同义单碱基突变的基因和基因间区单碱基突变的基因,即预测为与红霉素产量相关的基因。在一些实施例中,在所述预测关键代谢节点和代谢途径中的至少一者中,是通过评估各代谢途径,以及各代谢途径中各个代谢节点中非同义单碱基突变和基因间区单碱基突变发生的频率来进行预测。在一些实施例中,在所述预测报告代谢物中,是将周边代谢途径中发生多个转录变化的代谢物,以及预测的所述关键代谢节点和代谢途径中的至少一者中涉及多个非同义单碱基突变和/或基因间区单碱基突变的代谢物预测为报告代谢物。在一些实施例中,所述预测报告代谢物为l-谷氨酸、l-谷氨酰胺和α-酮戊二酸中的至少一者。在本申请中,采用比较组学的定向代谢策略来预测与红霉素产量相关的基因,具有全面、有效、靶向精准度高的优点。因此,在本申请中,基于比较组学数据,yi现有常规的红霉素高产菌hl3168-e3作为研究对象,首次确定了suca基因和sucb基因作为基因改造靶点,获得了一一种相较于红霉素高产菌hl3168-e3更为高产的重组红霉素工程菌。该重组红霉素工程菌是在红霉素高产菌hl3168-e3中同时过量表达suca基因和sucb基因所得。本申请所述重组红霉素工程菌应用于红霉素生产中可提高红霉素的浓度。经实验表明,将本申请所述重组红霉素工程菌和红霉素高产菌hl3168-e3在相同培养条件下分别进行摇瓶发酵,以合成红霉素,发酵结束后,相较于红霉素高产菌hl3168-e3发酵液中红霉素的浓度,所述重组红霉素工程菌发酵液中红霉素的浓度提高了40%,红霉素的浓度达到700mg/l。附图说明下面结合附图,通过对本申请的具体实施方式详细描述,将使本申请的技术方案及其它有益效果显而易见。图1为本申请实施例中利用红霉素高产菌hl3168-e3和红色糖多孢菌标准株nrrl23338在菌体指数生长期的转录组,以及红霉素高产菌hl3168-e3基因组规模代谢模型预测的报告代谢产物图谱。图2为本申请实施例中利用红霉素高产菌hl3168-e3和红色糖多孢菌标准株nrrl23338在菌体生长稳定期的转录组,以及红霉素高产菌hl3168-e3基因组规模代谢模型预测的报告代谢产物图谱。图3为基础培养基中红霉素生物合成途径(简化版)及基因靶点的筛选策略。图4为本申请实施例中工程菌e3-icd、e3-suca、e3-sucb和e3-sucba菌落pcr验证。图5为本申请实施例中工程菌e3-icd、e3-suca、e3-sucb和e3-sucba,以及红霉素高产菌hl3168-e3摇瓶发酵合成红霉素的结果分析图。其中,图1和图2中,图中点代表报告代谢物,点中间的数字对应图下方代谢物的名称(仅列出排序前十的代谢物名称,依照报告代谢物预测结果中的得分由高至低进行排序),点的颜色表示p值大小,点的大小表示代谢物周围基因数,代谢物后缀[c]表示胞内(intracellular),qh2表示泛醇,q表示泛醌,xmp表示5-磷酸黄嘌呤核苷,imp表示肌苷磷酸。图3中,每个菌种发酵合成红霉素的浓度结果为盒状示意,每个菌种发酵合成红霉素的浓度结果均来源于6个平行发酵液样品;盒状的上下边界分别表示6个平行发酵液样品结果中的第一个四分位数和第三个四分位数,盒中横线表示6个平行发酵液样品结果的中间值,小正方形表示6个平行发酵液样品结果的平均值,盒外横线表示6个平行发酵液样品结果中的最大值或最小值,黑色菱形表示6个平行发酵液样品结果中较为异常的数值。图4中泳道1:模板e3,引物pibc1/icdc;泳道2:模板e3-icd,引物pibc1/icdc;泳道3:模板e3-suca,引物pibc1/sucac;泳道4:模板e3,引物pibc1/sucac;泳道5:模板e3-sucb,引物pibc1/sucbc;泳道6:模板e3,引物pibc1/sucbc;泳道7:模板e3-sucba,引物b1/a2;泳道8:模板e3,引物b1/a2。由于e3基因组上无pibc1序列,所以对照泳道1、4、6不能扩增出片段;e3中suca与sucb在基因组上距离过远,因此使用来自sucb内部引物b1和suca内部引物a2不能扩增出片段。具体实施方式下面将结合本申请实施例中的附图,对本申请实施例中的技术方案进行清楚、完整地描述。显然,所描述的实施例仅仅是本申请一部分实施例,而不是全部的实施例。基于本申请中的实施例,本领域技术人员在没有作出创造性劳动前提下所获得的所有其他实施例,都属于本申请保护的范围。下列实施例中未注明具体条件的实验方法,通常按照常规条件如sambrook等人,分子克隆:实验室手册(newyork:coldspringharborlaboratorypress,1989)中所述的条件,或按照制造厂商所建议的条件。除非另行定义,文中所使用的所有专业与科学用语与本领域技术人员所熟悉的意义相同。此外,任何与所记载内容相似或均等的方法及材料皆可应用于本发明中。文中所述的较佳实施方法与材料仅作示范之用,但不能限制本申请的内容。一、本申请实施例中涉及培养基的配方与配制如下所述:(1)斜面培养基每100ml斜面培养基中,包括:1.2g的玉米浆,1.0g的淀粉,0.3g的nacl,0.3g的(nh4)2so4,0.5g的caco3以及2.0g的琼脂。100ml斜面培养基的配制:首先,按照配方称取各个成分;然后,将各个成分分别溶解于去离子水中,定容至100ml,用naoh调节ph至7.0;接着,将配制好的培养基置于高压灭菌锅中,在121℃下高压灭菌15min;最后,将灭菌后的培养基分装至多个试管,并进行斜面摆台,制得斜面培养基。(2)种子培养基每100ml种子培养基中,包括:2.0g的蛋白胨,4.0g的淀粉,2.0g的糊精,0.4g的nacl,0.02g的kh2po4,0.025g的mgso4,0.6g的豆油以及0.6g的caco3。100ml种子培养基的配制:首先,按照配方称取各个成分;然后,将各个成分分别溶解于去离子水中,定容至100ml,用naoh调节ph至7.0;接着,将配制好的培养基置于高压灭菌锅中,在121℃下高压灭菌15min;最后,向灭菌后的培养基中加入葡萄糖,培养基中葡萄糖的终浓度为1g/ml,制得种子培养基。(3)基础培养基每800ml基础培养基中,包括:20.0g的(nh4)2so4,50.0g的酪蛋白水解物,0.6g的mgso4·7h2o,1.0g的znso4·7h2o,1.0g的feso4·7h2o,1.0g的mncl2·4h2o以及1.0g的cacl2。800ml基础培养基的配制:首先,按照配方称取各个成分;然后,将各个成分分别溶解于去离子水中,定容至800ml;接着,将配制好的培养基置于高压灭菌锅中,在121℃下高压灭菌15min;接着,向灭菌后的培养基中加入150ml的nah2po4/k2hpo4缓冲液(浓度为0.1mol/l,ph=6.8);最后,向培养基中加入重量体积比为25%的葡萄糖,培养基中葡萄糖的终浓度为20g/l,制得基础培养基。(4)lb培养基每100ml的lb液体培养基中,包括:1.0g的蛋白胨,0.5g的酵母粉,以及1.0g的nacl。100mllb液体培养基的配制:按照配方称取各个成分;然后,将各个成分分别溶解于去离子水中,定容至100ml;接着,将配制好的培养基置于高压灭菌锅中,在121℃下高压灭菌20min,制得lb液体培养基。对于lb固体培养基,是在lb液体培养基配方的基础上添加20g/l的琼脂。需要说明的是,上述用于配制各个培养基的成分均可购自于试剂公司,其中,酪蛋白水解物购自于bd公司,蛋白胨和酵母粉购自于oxoid公司。(5)tsb培养基,胰酪大豆胨液体培养基,市售通用商品,例如购自于sigma-aldrich公司。(6)isp4培养基,用于接合转移,成分(g/100ml):可溶性淀粉1,k2hpo4·3h2o0.131,mgso4·7h2o0.1,nacl0.1,(nh4)2so40.2,caco30.2,琼脂2。灭菌前加入100μl微量元素,其母液含0.01g/mlfeso4·7h2o,mncl2·4h2o,znso4·7h2o。ph7.0。121℃灭菌15min。灭菌后每升加入mgcl2使终浓度30mm。。二、本申请实施例中涉及菌株和质粒的说明详见下表1:表1为本申请实施例中涉及的菌株和质粒三、本申请实施例中涉及的试剂、溶液以及引物说明本申请实施例中涉及的试剂,包括:分子生物学试剂(如:各种酶、pcr试剂、试剂盒、电泳试剂、rnalatertm存储液等)、化学试剂(如:酚、氯仿、异戊醇、醋酸钠、乙醇、异丙醇、磷酸等)和抗生素(如:红霉素、氨苄青霉素、安普霉素等),均为商业化产品,均有市售。本申请实施例中涉及的溶液,如:tes缓冲液、te缓冲液、pbs缓冲液、0.1g/ml十二烷基硫酸钠(sds)、5mol/l的nacl、0.1g/ml的ctab、3mol/l的醋酸钠、10mol/l的磷酸、70%乙醇,均为现有技术中的已知溶液,依据本领域已知技术手段自行配制。本申请实施例中涉及的引物均由华大基因公司合成。本申请实施例中涉及的全基因组测序、转录组测序、转化子测序验证和接合子测序均由华大基因公司完成。下面对本申请的实施例进行详细说明,本实施例在以本申请技术方案为前提下进行实施,给出了详细的实施方式和具体的操作过程。实施例1:红霉素高产菌hl3168-e3的全基因组提取红霉素高产菌hl3168-e3的全基因组提取步骤如下所述:s1.1、制备红霉素高产菌hl3168-e3的菌丝重悬液。具体的,首先,挑取红霉素高产菌hl3168-e3的单菌落接种于30ml的tsb液体培养基中,置于34℃振荡培养48~72h,以使红霉素高产菌hl3168-e3处于对数生长期;然后,取0.4~0.6ml的培养液在12000r/min转速下离心3min,去上清液以收集沉淀,所述沉淀即为红霉素高产菌hl3168-e3菌丝;接着,将收集的红霉素高产菌hl3168-e3菌丝用tes缓冲液洗涤数次;最后,将洗涤后的红霉素高产菌hl3168-e3菌丝重置于装有0.7mltes缓冲液的ep管中,获得红霉素高产菌hl3168-e3的菌丝重悬液。其中,所述tes缓冲液的浓度为0.05mol/l,ph为8.0。需要说明的是,可制备多管红霉素高产菌hl3168-e3菌丝重悬液,以具有多个平行样。s1.2、向所述红霉素高产菌hl3168-e3的菌丝重悬液中加入溶菌酶和核糖核酸酶(rnase),然后置于37℃孵育,直至管内液体变得粘稠;具体的,向每管菌丝重悬液中加入溶菌酶和rnase,直至溶菌酶的终浓度为5mg/ml,rnase的终浓度为40μg/ml。所述孵育的时间为1h,并每隔15min轻轻颠倒ep管数次以使菌丝细胞充分裂解。所述溶菌酶的浓度为50mg/ml,所述rnase的浓度为4mg/ml。s1.3、向管内液体中依次加入0.1g/mlsds和蛋白酶k,然后置于37℃孵育,直至管内液体变得澄清透明。具体的,sds的作用是:进一步破坏细胞膜、核膜,使蛋白质变性,以促进蛋白质与dna分离;加入sds,直至sds在管内液体中的终浓度为0.01~0.02g/ml。所述蛋白酶k用于将蛋白质分解为小分子物质,每管液体中蛋白酶k的添加量为5μl。所述孵育时间为30min,每隔10min上下剧烈颠倒ep管数次。s1.4、向管内液体中加入预热至65℃的5mol/lnacl和0.1g/mlctab,然后置于65℃孵育,以进一步沉淀胞内蛋白。具体的,每管液体中,所述nacl的添加量为100μl,所述ctab的添加量为50μl。所述孵育时间为1h,每隔15min上下颠倒ep管数次。s1.5、对管内液体进行抽提dna操作。具体的,首先,向管内液体中加入等体积的dna抽提液;然后,漩涡振荡15s以充分混匀;接着,将ep管在12000r/min转速下离心10min,收集上清液并转移至新ep管中;最后,对收集的上清液加入等体积的dna抽提液,并依次进行漩涡振荡、离心和收集上清液操作,重复数次,最终获得的上清液中含有高纯度的dna。所述dna抽提液包括酚、氯仿和异戊醇,且所述酚、氯仿和异戊醇的体积比为25:24:1。优选的,所述dna抽提液还包括tris和edta,所述tris在所述dna抽提液中的浓度为10mmol/l的tris,所述edta在所述dna抽提液中的浓度为1mmol/l,且所述edta的ph为8.0。s1.6、将步骤s5中最终获得的上清液进行dna沉淀操作。具体的,首先,向管内上清液中加入3mol/l的醋酸钠,所述醋酸钠在所述上清液中的终浓度为0.3mol/l;然后,向管内加入等体积的4℃异丙醇,以冲洗管壁上黏附的dna;接着,将ep管置于4℃下12000r/min离心1min,离心后去除上清液;然后,加入2ml的70%乙醇,所述70%乙醇的温度优选为4℃,再置于4℃下12000r/min离心10min,离心后去除上清液;最后,加入1ml的4℃70%乙醇,再置于4℃下12000r/min离心5min,离心后去除上清液,将ep管倒置于吸液纸上,以排出ep管内的液体,dna沉淀于ep管内。s1.7、制备全基因组溶液。具体的,向步骤s6装有dna沉淀的ep管中加入预热至55℃的te缓冲液,te缓冲液的加入量为30~50μl/管,获得红霉素高产菌hl3168-e3的全基因组溶液,其中,基因组浓度可采用微量紫外分光光度计测量。实施例2:红霉素高产菌hl3168-e3全基因组的高通量测序将所述步骤s1.7中红霉素高产菌hl3168-e3的全基因组溶液用于高通量测序。高通量测序工作是在华大基因公司的illuminahiseq2000平台进行。序列获得原始读段后,会去除大量重复片段和含有n碱基的片段。所述原始读段的处理方法包括如下步骤:s2.1、采用软件soapdenovo2.04和platanus1.2.4对红霉素高产菌hl3168-e3的全基因组原始读段进行组装,将所有原始读段组装成重叠群(contig)。s2.2、根据双末端测序和重叠的关系,对步骤s1.1组装的重叠群进一步优化,通过软件fgapl来修补全基因组中位于同一染色体的两个contig之间中断空缺部分(gap)。s2.3、利用glimmer3.02软件预测红霉素高产菌hl3168-e3全基因组中的编码区。s2.4、通过kegg、cog、swissprot、trembl、phi和iprscan数据库,对红霉素高产菌hl3168-e3全基因组中编码区的序列功能进行预测。需要说明的是,对于红霉素高产菌hl3168-e3全基因组中出现的重复序列,利用软件repeatmasker和trf转入转座子数据库中,以进行比对确定。红霉素高产菌hl3168-e3全基因组经高通量测序,共获得1.2×107个长度为150bp的原始核酸读段。将上述读段组装成contig后继续拼装获得两个非连续的基因组骨架(scaffold)。实施例3:比对分析红霉素高产菌hl3168-e3全基因组和红色糖多孢菌标准株nrrl23338全基因组红色糖多孢菌标准株nrrl23338全基因组已于2007年公开,其全基因组序列可在ncbi上查询获得。利用软件mummer对所述红霉素高产菌hl3168-e3全基因组和所述红色糖多孢菌标准株nrrl23338全基因组进行比对分析,以红色糖多孢菌标准株nrrl23338全基因组作为对照,识别所述红霉素高产菌hl3168-e3全基因组发生的所有单碱基突变,其中,利用软件lastz1.01.50识别插入或缺失型突变。所述红霉素高产菌hl3168-e3全基因组和所述红色糖多孢菌标准株nrrl23338全基因组的区别点详见下表2:表2红色糖多孢菌标准株nrrl23338和为红霉素高产菌hl3168-e3的全基因组特点名称nrrl23338hl3168-e3长度8,212,805bp8,181,083bpg+c含量71.10%71.18%编码密度82.40%87.19%编码序列数量72647669rrna1612trna5050蛋白编码序列数量71987607蛋白编码序列平均长度968bp931bp由表2可知,红霉素高产菌hl3168-e3全基因组与红色糖多孢菌标准株nrrl23338全基因组高度相似。红霉素高产菌hl3168-e3的非编码rna(即:trna)数量与红色糖多孢菌标准株nrrl23338相同,说明经红色糖多孢菌标准株nrrl23338诱变获得的红霉素高产菌hl3168-e3的翻译过程较为保守。经软件mummer比对分析,相较于所述红色糖多孢菌标准株nrrl23338全基因组,所述红霉素高产菌hl3168-e3全基因组上共发生255个单碱基突变,其中,160个单碱基突变位于红霉素高产菌hl3168-e3全基因组的核心部位。在255个单碱基突变中,128个单碱基突变分布在118个基因编码序列之中,基因编码序列中的非同义突变可能会引起其编码蛋白的结构性变化,从而引起编码蛋白活性的改变;51个单碱基突变发生在44个编码序列的基因间区之中,这51个单碱基突变可能影响多个编码区转录本的稳定性,从而影响对应转录本中部分mrna的功能稳定性。为了揭示所述红霉素高产菌hl3168-e3相较于所述红色糖多孢菌标准株nrrl23338高产红霉素的本质原因,对255个单碱基突变进行了聚类分析,发现含有单碱基突变较多的基因的细胞功能为:转录,氨基酸代谢和转运,能量合成与转化,糖类代谢和转运,以及次级代谢产物合成、转运和分解。实施例4:建立红霉素高产菌hl3168-e3基因组规模代谢模型以及预测关键代谢节点和代谢途径分析红霉素高产菌hl3168-e3全基因组编码区上的所有单碱基突变,将其中的非同义单碱基突变和同义单碱基突变通过软件ipath对映至代谢途径,并将红霉素高产菌hl3168-e3全基因组上的基因间区单碱基突变亦通过软件ipath对映至代谢途径,形成基因组规模代谢模型。通过评估各代谢途径,以及各代谢途径中各个代谢节点中非同义单碱基突变和基因间区单碱基突变发生的频率,来预测可能对于红霉素生物合成较为关键的代谢途径和代谢节点。经过评估,预测的关键代谢途径和关键代谢节点基本均与红霉素前体合成和辅因子供应,或信号传导相关。所述关键代谢途径包括:经由异柠檬酸至α-酮戊二酸,以及α-酮戊二酸至乙醛酸循环的代谢途径;氧化磷酸化代谢途径;经由α-酮戊二酸至谷氨酸的代谢途径;脂质代谢途径;s-甲硫氨酸代谢途径;支链氨基酸代谢途径;丙酸盐代谢途径;丙酮酸代谢途径;硫胺素代谢途径;色氨酸/酪氨酸代谢途径;磷酸戊糖代谢途径等。所述关键代谢节点包括:α-酮戊二酸、异柠檬酸、l-谷氨酸/谷氨酰胺等。在发生非同义单碱基突变或基因间区单碱基突变的基因中,编码异柠檬酸裂解酶的基因sace_1449、编码异柠檬酸脱氢酶的基因sace_6636以及编码α-酮戊二酸脱氢酶的基因sace_1638/6358均围绕在α-酮戊二酸节点周围。实施例5:红霉素高产菌hl3168-e3和红色糖多孢菌标准株nrrl23338的转录组提取本实施例仅详细阐述红霉素高产菌hl3168-e3的转录组提取方法,而红色糖多孢菌标准株nrrl23338的转录组提取方法参照红霉素高产菌hl3168-e3的转录组提取方法进行即可。s5.1、制备红霉素高产菌hl3168-e3的转录组提取样品。具体的,首先,挑取红霉素高产菌hl3168-e3的单菌落接种于30ml的基础培养基中,置于34℃振荡培养,分别取培养10h(对应指数生长期)的菌液和培养50h(对应稳定期)的菌液作为转录组提取样品,并且各培养时间点的菌液取两个平行样,每个转录组提取样品为10ml。s5.2、制备各个转录组提取样品的菌体重悬液。具体的,首先,将步骤s3.1制得的各个转录组提取样品分别在4℃下4000×g离心10min,去除上清液所得的沉淀即为菌体,收集获得各个转录组提取样品的菌体;然后,将各个转录组提取样品的菌体分别重悬于1.5~2ml的rnalatertm存储液中,获得各个转录组提取样品的菌体重悬液;最后,将各个菌体重悬液置于室温4h,再转移至4℃保存。s5.3、抽提各个菌体重悬液的转录组(总rna)。仅描述一个重悬液的总rna抽提方法,其他菌体重悬液总rna的抽提均参照这个重悬液的总rna抽提方法进行。具体的,首先,取0.5ml的菌体重悬液转移至一个规格为2ml的ep管中,向ep管内的菌体重悬液中加入适量玻璃砂;然后,将装有菌体重悬液和玻璃砂的ep管置于均质破碎仪中剧烈振荡30s,重复数次以充分破碎细胞,获得细胞破碎液;最后,用试剂盒rnaeasyplusminikit对所述细胞破碎液进行rna抽提。其中,将试剂盒rneasyplusminikit(50)(市售商品,例如狗子qiagen)中去除dna的步骤,改为采用无核糖核酸酶的dna脱氧核糖核酸酶试剂盒(rnase-freednaseset(50),市售商品,购自qiagen)处理。利用1%琼脂糖凝胶电泳初步预测抽提的总rna完整度,当电泳显示三条清晰且较少拖尾的条带时,说明抽提的总rna较为完整,然后用细胞分析仪(bioanalyzer)精确分析抽提的总rna质量。将抽提的总rna保存以用作转录组测序样品。实施例6:红霉素高产菌hl3168-e3和红色糖多孢菌标准株nrrl23338的转录组高通量测序及比对用于转录组高通量测序的总rna样品需同时满足条件:rin≥7.0,且23s/16s≥1.0,且od260/280≥1.8,且od260/230≥1.8,且浓度>40ng/μl,且总量>1μg。红霉素高产菌hl3168-e3和红色糖多孢菌标准株nrrl23338的转录组高通量测序,是在华大基因公司的bgiseq500平台进行,每种菌株的总rna样品取自在基础培养基中培养10h和培养50h的菌体,并且各培养时间点具有两个平行样,即:每种菌株共有4个总rna样品。高通量测序仪生成的原始测序图谱通过碱基识别转换为序列数据,并以fastq的文件形式呈现,每个总rna样品获得至少30兆长度为100bp的原始读段。测序完成后,所有原始读段会去除适配序列、污染片段和低质量片段。经转录组高通量测序,红霉素高产菌hl3168-e3的总rna样品共测得1.07亿个长度大小为100bp的原始转录片段,对应为红霉素高产菌hl3168-e3的转录组测序数据;红色糖多孢菌标准株nrrl23338的总rna样品共测得2.05亿个长度大小为150bp的原始转录片段,对应为红色糖多孢菌标准株nrrl23338的转录组测序数据。采用软件geneiousprime处理红霉素高产菌hl3168-e3和红色糖多孢菌标准株nrrl23338的转录组测序数据,即:将双端测序片段进行修剪和过滤操作,对映到注释程度最高的红色糖多孢菌标准株nrrl23338全基因组上,各个编码区序列的表达值利用软件geneiousprime计算。通过deseq2算法分析红霉素高产菌hl3168-e3和红色糖多孢菌标准株nrrl23338之间的差异表达基因,获得两者在指数生长期的差异表达基因,以及两者在稳定期的差异表达基因。差异表达基因的聚类分析借助于ncbi序列批量提取功能和eggnog数据库,并且通过软件morpheus完成。实施例7:预测报告代谢产物和与红霉素产量相关的基因所述预测报告代谢产物,是将周边代谢途径中发生多个转录变化的代谢物,以及预测的所述关键代谢节点和代谢途径中的至少一者中涉及多个非同义单碱基突变和/或基因间区单碱基突变的代谢物预测为报告代谢物。将红霉素高产菌hl3168-e3和红色糖多孢菌标准株nrrl23338在指数生长期的差异表达基因,和在稳定期的差异表达基因分别通过软件ipath,对映至红霉素高产菌hl3168-e3基因组规模代谢模型中,然后利用软件r包piano预测报告代谢产物,得到图1和图2。如图1和图2所示,在指数生长期和稳定期中,l-谷氨酸/谷氨酰胺、α-酮戊二酸(2-oxoglutarate或α-ketoglutarate)、红霉素合成前体和电子传递相关物质均在排序前十中,而l-谷氨酸/谷氨酰胺和α-酮戊二酸均为氮代谢中的重要节点,证明红霉素高产菌中红霉素合成的启动与氮缺乏紧密相关。综上所述,α-酮戊二酸是报告代谢产物之一,而在α-酮戊二酸周围的基因icd、基因suca和基因sucb上发现了若干个非同义单碱基突变和基因间区单碱基突变,其可能促进α-酮戊二酸脱氢形成琥珀酰辅酶a,从而提高红霉素的生物合成量,因此,预测icd基因、suca基因和sucb基因与红霉素产量相关。其中,所述suca基因的核苷酸序列为seqidno.1,所述sucb基因的核苷酸序列为seqidno.2,所述icd基因的核苷酸序列为seqidno.7。实施例8:构建工程菌e3-icd、e3-suca、e3-sucb和e3-sucba本实施例中,工程菌e3-icd、e3-suca、e3-sucb和e3-sucba的构建包括如下步骤:s8.1、分别构建重组质粒pib-icd、pib-suca、pib-sucb以及pib-sucba。涉及的引物详见下表3:表3为本申请实施涉及的引物信息基于同源序列识别原理,采用gibson一步克隆,正向引物sucaf3未保留酶切位点。采用实施例1中红霉素高产菌hl3168-e3的全基因组作为模板,以进行相关基因片段的pcr扩增。其中,icdf/icdr引物对用于扩增icd基因以构建重组质粒pib-icd,sucaf/sucar引物对用于扩增suca基因以构建重组质粒pib-suca,sucbf/sucbr引物对用于扩增sucb基因以构建重组质粒pib-sucb,sucaf3/sucar3引物对用于扩增suca基因以构建重组质粒pib-sucba。具体的,各个基因片段的pcr反应体系详见下表4:表4为本申请实施涉及的pcr反应体系(25μl体系)试剂用量/μl5×gc高保真聚合酶缓冲液5dna聚合酶(phusion聚合酶,购自neb)0.25模板0.5上游引物(10μm)1下游引物(10μm)1dntp4双蒸水12.5总体系25具体的,各个基因片段的pcr扩增程序为:①98℃预变性3min;②98℃变性10s;③66℃退火30s;④72℃延伸1.5min;⑤重复②~④35个循环;⑥72℃,10min。采用gibson无缝克隆试剂盒(即:gibson一步克隆法),将icd基因的扩增片段插入至质粒pib139的ndei和xbai酶切位点之间,获得重组质粒pib-icd。采用gibson无缝克隆试剂盒(即:gibson一步克隆法),将sucb基因的扩增片段插入至质粒pib139的ndei和xbai酶切位点之间,获得重组质粒pib-sucb。采用gibson无缝克隆试剂盒(即:gibson一步克隆法),将以sucaf/sucar为引物pcr扩增的suca基因片段插入至质粒pib139的ndei和xbai酶切位点之间,获得重组质粒pib-suca。采用gibson无缝克隆试剂盒(即:gibson一步克隆法),将以sucaf3/sucar3为引物pcr扩增的suca基因片段插入至重组质粒pib-sucb的ecori酶切位点处,获得重组质粒pib-sucba。s8.2、采用接合转移方式,将(1)中构建的重组质粒pib-icd、pib-suca、pib-sucb以及pib-sucba分别转入至红霉素高产菌hl3168-e3中。以重组质粒pib-sucba为例,pib-sucba接合转移至红霉素高产菌hl3168-e3中,具体包括如下步骤:s8.21、将pib-sucba转化至大肠杆菌et12567/puz8002感受态细胞中,筛选获得转化子。具体的,转化的操作方法为:首先,向冰预冷的含有100μl大肠杆菌et12567/puz8002感受态细胞的离心管中,加入10μl的pib-sucba,手指轻叩离心管底部,小心混匀,冰浴30min。然后,将装有pib-sucba和大肠杆菌et12567/puz8002感受态细胞的离心管置于42℃恒温水浴中,以热激90s,其间不要摇动离心管。热激后,快速将离心管置于冰浴中保持5min,获得转化细胞。转化结束后,向装有转化细胞的离心管中加入800μl/管的lb液体培养基,颠倒混匀,置于37℃、150r/min的条件下振荡温育1h,以促进转化细胞繁殖。温育结束后,将培养液涂布于含有硫酸卡那霉素(25μg/ml)和安普霉素(50μg/ml)的lb抗性平板表面,然后于37℃倒置培养(不超过16h)。挑取lb抗性平板表面上生长的单菌落进行菌落pcr验证,验证正确的转化子进行测序验证,将正确的转化子命名pib-sucba/et12567。s8.22、将步骤s.21获得的pib-sucba/et12567,基于φc31位点特异性整合酶识别位点attb/attp,接合转移至红霉素高产菌hl3168-e3中,过表达基因整合至hl3168-e3基因组上。具体的,制备供体菌的方法为:首先,挑取步骤s.21获得的pib-sucba/et12567/于lb液体培养基中,37℃振荡培养过夜,至菌体浓度od600为0.4~0.6;然后,取适量培养液离心,以收集菌体;接着,用无菌的lb液体培养基漂洗菌体数次;最后,将漂洗后的菌体重悬于lb液体培养基以备用,获得供体菌。制备受体菌的方法为:首先,挑取108个红霉素高产菌hl3168-e3的孢子,悬浮于500μl的tes缓冲液中;然后,在50℃下热激10min;接着,加入等体积的tsb液体培养基,37℃220rpm培养萌发2h,以获得菌丝;接着,用无菌的lb液体培养基漂洗菌丝数次;最后,将漂洗后的菌丝重悬于lb液体培养基以备用,获得受体菌。将供体菌和受体菌按照1:1的细胞数量比混匀,然后涂布于无抗性的isp4平板上,34℃培养24h后,在平板上均匀覆盖1ml含有30μl安普霉素(100mg/ml)的tes缓冲液,超净工作台中晾干,34℃继续培养7~8天。挑取在isp4抗性平板上生长的单菌落,进行菌落pcr验证,验证正确的接合子可通过测序进行进一步的验证,验证正确的接合子即为工程菌e3-sucba。同理,按照上述方法,构建获得工程菌e3-icd、e3-suca和e3-sucb。图3所示为基础培养基中红霉素生物合成途径(简化版)及基因靶点的筛选策略。图中基因代号acea/icd/suca/sucb分别显示在基因所控制代谢途径旁边,#表示该基因编码区上游发生基因间区突变;*表示该基因编码区内部发生非同义突变。右上角插图显示上述四个基因在两株菌两个培养时期内的转录水平变化。工程菌e3-icd、e3-suca、e3-sucb和e3-sucba的pcr验证结果请见图4。图4中泳道1:模板e3,引物pibc1/icdc;泳道2:模板e3-icd,引物pibc1/icdc;泳道3:模板e3-suca,引物pibc1/sucac;泳道4:模板e3,引物pibc1/sucac;泳道5:模板e3-sucb,引物pibc1/sucbc;泳道6:模板e3,引物pibc1/sucbc;泳道7:模板e3-sucba,引物b1/a2;泳道8:模板e3,引物b1/a2。由于e3基因组上无pibc1序列,所以对照泳道1、4、6不能扩增出片段;e3中suca与sucb在基因组上距离过远,因此使用来自sucb内部引物b1和suca内部引物a2不能扩增出片段。实施例9:筛选重组红霉素工程菌以所述红霉素高产菌hl3168-e3作为对照,在相同培养条件下,对工程菌e3-icd、e3-suca、e3-sucb和e3-sucba分别进行基础培养基中红霉素合成的发酵培养,检测发酵液中红霉素的浓度,并将各个工程菌发酵液中红霉素的浓度与所述红霉素高产菌发酵液中红霉素的浓度进行对比,以筛选重组红霉素工程菌。具体包括如下步骤:s9.1、将各个工程菌分别接种于含有安普霉素(50μg/ml)的斜面培养基上,红霉素高产菌hl3168-e3接种于斜面培养基上,34℃培养6天,以生长孢子。s9.2、在各个工程菌的斜面培养基上,各取1cm2长满孢子的培养基,并分别接种至一300ml摇瓶中,所述300ml摇瓶中装有30ml含有安普霉素(50μg/ml)的种子培养基,在34℃、220r/min下振荡培养48h,获得各个工程菌的种子培养液;在红霉素高产菌hl3168-e3的斜面培养基上,取1cm2长满孢子的培养基,接种至一300ml摇瓶中,所述300ml摇瓶中装有30ml种子培养基,在34℃、220r/min下振荡培养48h,获得红霉素高产菌hl3168-e3的种子培养液。s9.3、每个工程菌的种子培养液各取3ml,并取3ml的红霉素高产菌hl3168-e3种子培养液,然后将取得的各个种子培养液分别重悬于1mlpbs缓冲液(ph=7.4)中,再分别接种至一300ml摇瓶中,所述300ml摇瓶中装有27ml基础液体培养基,在34℃、220r/min下振荡培养5天,以发酵生产红霉素。s9.4、发酵结束后,检测各个工程菌和红霉素高产菌hl3168-e3的发酵液中红霉素浓度,以筛选重组红霉素工程菌。其中,在摇瓶发酵生产红霉素中,各个工程菌和红霉素高产菌hl3168-e3各具有6个平行样,对每个平行样的发酵液都要进行红霉素浓度检测。具体的,是采用磷酸法检测各个发酵液样品中的红霉素浓度。其中,制作用于红霉素浓度测定的标准曲线包括如下步骤:s9.41、制备各浓度红霉素标准液:称取定量红霉素,溶于少量乙醇后,加入去离子水,配置浓度为100μg/l、250μg/l、500μg/l、750μg/l和1000μg/l的各浓度红霉素标准液。s9.42、制备用于绘制标准曲线的实验样品:分别取80μl各浓度红霉素标准液分别置于一2mlep管中,每个ep管中加入400μl浓度为10mol/l的磷酸,漩涡振荡后置于80℃水浴3min,然后将各个ep管从水浴中取出并置于自来水中降温1min,再向各个ep管中加入520μl浓度为10mol/l的磷酸,获得各浓度红霉素实验样品。s9.43、分别取200μl的各浓度红霉素实验样品,采用96孔板酶标仪分别测量各浓度红霉素实验样品的od485值。s9.44、制备用于绘制标准曲线的对照样品:分别取80μl各浓度红霉素标准液置于一2mlep管中,每个ep管中直接加入920μl浓度为10mol/l的磷酸,获得各浓度红霉素对照样品。s9.45、分别取200μl的各浓度红霉素对照样品,采用96孔板酶标仪分别测量各浓度红霉素对照样品od485值。s9.46、计算各浓度的红霉素实验样品与红霉素对照样品之间的od485差值。s9.47、绘制标准曲线:以各浓度的红霉素实验样品与红霉素对照样品之间的od485差值作为横坐标,以不同的红霉素浓度作为纵坐标,绘制标准曲线。对于检测各个发酵液样品中的红霉素浓度,具体流程如下:取80μl某一发酵液样品置于一2mlep管中,然后向ep管中加入400μl浓度为10mol/l的磷酸,漩涡振荡后置于80℃水浴3min,接着将ep管从水浴中取出并置于自来水中降温1min,再向ep管中加入520μl浓度为10mol/l的磷酸,获得某一发酵液的实验样品,采用96孔板酶标仪测量其od485值。取80μl所述某一发酵液样品置于一2mlep管中,然后向ep管中直接加入920μl浓度为10mol/l的磷酸,获得所述某一发酵液的对照样品,采用96孔板酶标仪测量其od485值。计算所述某一发酵液的实验样品与其对照样品之间的od485差值,然后将该od485差值对映至所述标准曲线中,获得所述某一发酵液中的红霉素浓度。通过上述方法,获得工程菌e3-icd、e3-suca、e3-sucb和e3-sucba,以及红霉素高产菌hl3168-e3发酵液中红霉素的浓度,其中,发酵液中红霉素的浓度与红霉素的产量呈正相关。如图5所示,相较于红霉素高产菌hl3168-e3发酵液中红霉素的浓度,工程菌e3-icd的发酵液中红霉素浓度降低了45%,工程菌e3-sucba的发酵液中红霉素浓度提高了40%(红霉素浓度为700mg/l)。工程菌e3-suca和工程菌e3-sucb的发酵液中红霉素浓度差异不显著,并且均与红霉素高产菌hl3168-e3发酵液中红霉素浓度相近。因此,筛选获得工程菌e3-sucba为重组红霉素工程菌,即:在红霉素产生菌,尤其是红霉素高产菌hl3168-e3中同时过量表达suca基因和sucb基因,可有效提高红霉素的生物合成产量。以上对本申请实施例所提供的一种,进行了详细介绍。本文中应用了具体个例对本申请的原理及实施方式进行了阐述,以上实施例的说明只是用于帮助理解本申请的技术方案及其核心思想;本领域的普通技术人员应当理解:其依然可以对前述各实施例所记载的技术方案进行修改,或者对其中部分技术特征进行等同替换;而这些修改或者替换,并不使相应技术方案的本质脱离本申请实施例的技术方案的范围。序列表<110>华东理工大学<120>一种重组红霉素工程菌及其构建方法和筛选方法和应用<160>17<170>siposequencelisting1.0<210>1<211>3621<212>dna<213>人工序列(artificialsequence)<400>1atgtacgagcagttcctgcacgatcccacctccgtagactcggcgtggcacgaattcttc60gccgactacaagccgggccaggccactgaaaccaccgcagccgcgaccggttctgcagcg120gccccgaaggcgacggccaccgcggcacgcgtggtcgagaccaacgggcagaccccgccc180ccgtccgccaccgcaccgagcaagccgaagcccgccgagcccaagccctccccgcaggag240gccgccaaggccgcccccgtcaaggaggcccccgcgggcgagcaggccaagccgctgcgc300ggcgccgcggccgcgatcgccaagaacatggagcagtcgctcaccgtgccgaccgcgacc360agcgtgcgcgcggtgccggccaagctgctcttcgacaaccgcatcgtcatcaacaaccac420ctcaagcggaacaagggcgggaaggtctccttcacgcacctgatcggctacgcgctgatc480cgggcgctgcggaaccaccccgacatgaaccgccactacggcgaggacgccaagggcaag540cccgccgtggtcacccccgagcacgtcaacctcggcctggcgatcgacatgccggccaag600gacggctcgcgcagcctggtggtcgcctccatcaagggctgcgaggagatgaccttccag660cagttctggcaggcctacgaggacatcatccgcaaggcccgcaacagcgcgctgaccgcc720gacgacttctccggcaccaccatctcgctgaccaaccccggccccagcggcaccaaccac780tcggtgccgcggctgaccaagggccagagcgcgatcatcggcgtcggcgcgatggactac840cccgccgagttccagggcgccagcgagcaggcgctggtcgacatggggatcagcaagatc900gtgacgctgacctccacctacgaccaccgcgtcatccagggcgcggagtccggtgacttc960ctgcgcacggtgcaccagctgctgctcggcgagaacgggttctacgacgacatcttcacg1020tcgctgcggatcccctacgagccggtccgctggacccgcgacatcccagagggcgcggtc1080gacaagaccgcccgggtgctggagctgatcgacgcctaccgcacccgcggccacctgatg1140gccgacatcgacccgctgaactaccgccagcgccgccacgaggacctcgacgtcctctcc1200cacagcctcaccctgtgggacctggaccgcacgttcgccgtcggcggcttcgcgggcaag1260gagcggatgaagctgcgcgacgtgctcggcgtgctgcgcgactcctactgccgcaccgtc1320ggcgtcgagtacatgcacatcctcgagcccgacgagcgcgagtggctgcagggccgggtc1380gagaagccgcacaccaagcccgagccgaccgagcagaagtacatcctgtccaagctcaac1440gccgccgaggcgttcgagaccttcctgcagaccaagtacgtcggccagaagcgcttctcg1500ctggagggcgccgagaccgtcgtgccgctgctggacgcggtgctcgacaccgccgcggcc1560tccgagctggacgaggtcgtcatcggcatgccgcaccgcggccgcctcaacgtgctggcc1620aacatcgtcggcaagccgatctcgcagatcttccgcgagttcgagggcaacctcgacccg1680ggccaggcacacggctccggcgacgtgaagtaccacctcggcgccgagggcaagtacttc1740cggatgttcggcgacggcgagaccaaggtgtcgctgacctccaacccctcgcacctggag1800gccgtggacccggtgctggagggcatcgtccgcgccaagcaggacatcctggacaagggc1860caggagggcttcacggtgctgccggtgctgctgcacggcgacgccgcgttcgcgggccag1920ggcgtggtcgccgagacgctgaacctgtcgctgctgcgcggctaccgcaccggcggcacg1980gtccacgtgatcgtcaacaaccaggtcggctacaccaccgccccggagcactcgcggtcg2040agcaagtactccaccgacgtggcgaagatgatcggcgcgccggtcttccacgtcaacggt2100gacgaccccgaggcctgcgtctgggtcgccaagctggccgtggagtaccgccaggcgttc2160ggcaaggacgtcgtgatcgacatggtctgctaccgccgccggggtcacaacgagggcgac2220gacccgtccatgacgcagccggcgatgtacgacgcgatcgacaagatgcgcagcgtccgc2280aagacctacaccgaggccctcatcggccgtggcgacatcacggtcgaagaggccgagaag2340gcgctgaaggactacgccagccagctggagcacgtgttcaacgaggtccgcgagctggag2400aagcacccgccggagcccagcccgtcggtggagtccgagcaggtggtcccgcaggggctg2460gcgaccgcgatcccggtcgacacactcaagcggatcgccgacgcgcaggtcaacatgccc2520gagggcttcacgccgcactcgcgggtcaagcccgtgctggagcgccgcgcgaagatggcc2580accgagggcggcatcgactgggcgttcggcgagctgctcgcgttcggctcgctgaccatg2640gagggccgcccggtccgcctgaccggccaggacagccgtcgcggcacgttcggccagcgg2700cactcggtgctcatcgaccgcaagaccggtgccgagtacacgccgctgcagaacctgagc2760gaggaccaggcgaagttcctggtctacgactcggcgctgtcggagttcgcggccatgggc2820ttcgagtacggctactcggtggccaacccggacgccctggtgctgtgggaggcgcagttc2880ggcgacttcttcaacggcgcgcagtcgatcatcgacgagttcatctcctccggtgaggcc2940aagtggggccagcgctccgacgtcgtgctgctgctgccgcacggccacgagggccagggt3000ccggaccacagctcggcgcgcatcgagcggtggctgcagctctgcgccgaggggtcgatg3060acggtggcgatgccgtcgacgccggcgaactacttccacctgctgcgccgccacgcgctg3120gacggcatccaccggccgctggtcgtcttcacgccgaagtcgatgctgcggctcaaggcg3180gccaccagcccggtcgaggacttcaccgagggcaagttcacctcggtgatcgacgacccg3240acgcagccggacccggcctcggtgcgccgcgtggtgctgtgcaccggcaagctctactac3300gagctggccgccgagaaggccaagcagggccacgacgacaccgcggtggtgcggctggag3360cagctctacccgctgccgcaccgcaagctgggcaggctgctcgagcggtactccaacgcc3420accgacgtccggtgggtgcaggaggagccggcgaaccagggtgcctggccgttcctcggc3480ctggcgctgccggagctgttcccggagcgcctggcgggcctgcgccgcgtgtcgcggcgg3540ccgatggccgccccggcgaccggtatggccaaggtgcacgaggtcgagcaggccgaggtc3600gtgcagggcgccttcgcctga3621<210>2<211>1830<212>dna<213>人工序列(artificialsequence)<400>2atggccttctccgtccagatgccggcactcggcgagagcgtcaccgagggaacgatcacc60aggtggctcaagcaggagggcgacaccgtcgaggtcgacgaacccctgctggaggtctcc120accgacaaggtcgacaccgagatcccctccccggccgccggtgtgctccagcgcatcgtc180gcccaggaggacgacaccatcgagatcggcggcgagctcgccgtgatcggtgagtcgggc240gagtccggcggcggcgagtccgcccccgcggagcagccccagcaggccgagccgcagcag300cccgagccccagcaggccgaggccgcgcagccggagcccgagcagcagcccgccccggcc360cagcaggaggccccggcgcagagctccggtgccgcccagtccggttcggcgcagggcacc420gaggtgccgatgcccgcgctgggcgagagcgtcaccgagggaacgatcaccaggtggctc480aagcaggtcggcgacaccgtcgaggtcgacgaacccctgctggaggtctccaccgacaag540gtcgacaccgagatcccctccccggtggccgggaccctgctggagatctccgccggtgag600gacgacaccgtcgaggtcggcgccaagctggccgtggtcggtgagcagggcgccgcgccg660tcggccccggccgcgccgccggaggagcagaccgccccgcagcaggccgcgccacagcag720gccgcgccacagcagtcggccccggccgagcagcaggcaccggctcagcagcaggctccg780gcccagcccgctccgcagcccgtgcaggagcagccccagcaggccgccccggcgcagccc840gcccagccgtcggctccggccgaggccccctcgggcgccgcgccgtacgtgacgccgctg900gtgcgcaagctggccaacgagcacggcatcgacctgtccaagatcaagggcagcggtgtc960ggcggccggatccgcaagcaggacgtgcaggccgccgtcgacgccgccaaggccgagcag1020gaggcgcccgccgcggctccgtcggctccgtcccggcccggcccggcggccgcccaggcc1080gtggagccctcggaagaggccaaggcgctgcgcggcaccacccagaagatgacgcggctg1140cgccagcttctggcccgccgcatggtcgagtcgctgcagaccgccgcccagctcaccacg1200gtggtcgaggtcgacgtcaccagaatcgcccgcctgcgcgaccgcgccaagcagaacttc1260gaggcggccgaaggcgtcaagctctccttcctgcccttcttcgccaaggccgcggccgag1320gcgctgaagctgcacccgaagctcaacgcttcggtggacgaggagaacaaggaggtcacc1380taccacgccgccgagcacctggcgatcgcggtcgacaccgagcgcggcctggtctcgccg1440gtgatccacgacgcgggtgacctcaacctcggcgggctggcccgcaagatcgccgacctg1500gcggcgcgcacccgcaacaacaagatcaagccggacgagctcagcggcggcacgttcacg1560ctgaccaacaccggcagccggggcgcgctgttcgacacgccgatcctgaacccgccgcag1620gtgggcatgctgggcaccggcaccgtggtgaagcggccggtcgtggtcaccgacgagaac1680ggcggcgacacgatcgccattcgctcgatggtgtacctcgtgctgtcctacgaccaccgc1740ctggtcgacggcgcggacgcggcccggttcctggccaccctcaagcagcggctcgaggaa1800ggcgccttcgaggccgacctgggcctctga1830<210>3<211>39<212>dna<213>人工序列(artificialsequence)<400>3ttggtaggatccacatatgatggccttctccgtccagat39<210>4<211>42<212>dna<213>人工序列(artificialsequence)<400>4gcgcggccgcggatcctctagatcagaggcccaggtcggcct42<210>5<211>40<212>dna<213>人工序列(artificialsequence)<400>5ccgcggccgcgcgcgatatcgaccggatcgtcgtggtcga40<210>6<211>45<212>dna<213>人工序列(artificialsequence)<400>6acagctatgacatgattacactagttcaggcgaaggcgccctgca45<210>7<211>1221<212>dna<213>人工序列(artificialsequence)<400>7atgagcaagatcaaggtccagggcaccatcgccgagctcgacggcgacgagatgacgcgg60atcatctggcagttcatcaaggacaagctgatccacccctacctggacgtcaacctcgac120tactacgacctcggcatcgagcaccgggacgccaccgacgaccaggtcaccgtcgacgcc180gcgaacgccatcaagaagtacggcgtgggcgtgaagtgcgccaccatcaccccggacgag240gcccgcgtggaggagttcggcctgaagaagatgtggcggagcccgaacgggacgatccgc300aacatcctcggtggcgtcatcttccgcgagccgatcgtcatctccaacatcccgcgctac360gtaccgacgtggaccaagccgatcgtcatcggccgccacgcgcacggcgaccagtacaag420gcgaccgacttcaaggtgccgggcccgggcacggtgaccgtcacctacacgccggaggac480ggcggcgaaccgatcgagttcgaggtcgccaagttcggtgaggacggcggcgtggcgatg540gccatgtacaactacaggcgctccatcgaggagttcgcccgcgcgtccttccgctacggc600ctcgagcgcggctacccggtgtacatgtccaccaagaacacgatcctcaaggcctacgac660ggcctgttcaaggacgtgttccaggaggtcttcgacaacgagtacaaggccgacttcgac720gccaagggcctgacctacgagcaccggctgatcgacgacatggtcgccaccgccatgaag780tgggagggcggctacgtctgggcctgcaagaactacgacggtgacgtgcagtccgacacc840gtcgcgcagggcttcggctcgctgggcctgatgacctcggtgctgatgaccgaggacggc900aaggtcgaggccgaggccgcgcacggcacggtcacccggcacttccgccagcaccagcag960ggcaagccgacctcgaccaacccgatcgcgtcgatcttcgcgtggacccgcggcctgcag1020caccgcggcaagctggactcgacccccgaggtcgtcggcttcgccgagacgctggagaag1080gtcgtcatcgagaccgtcgagagcggccggatgaccaaggacctggcgctgctggtcggc1140ggcgaccagggctaccagaccaccgaggagttcctcgcgacgctggacgacaacctgcag1200aagaggatggccaaccgctga1221<210>8<211>41<212>dna<213>人工序列(artificialsequence)<400>8ttggtaggatccacatatgatgagcaagatcaaggtccagg41<210>9<211>42<212>dna<213>人工序列(artificialsequence)<400>9gcgcggccgcggatcctctagatcagcggttggccatcctct42<210>10<211>41<212>dna<213>人工序列(artificialsequence)<400>10ttggtaggatccacatatgatgtacgagcagttcctgcacg41<210>11<211>45<212>dna<213>人工序列(artificialsequence)<400>11gcgcggccgcggatcctctagatcagaggcccaggtcggcctcct45<210>12<211>20<212>dna<213>人工序列(artificialsequence)<400>12ctgttgtgggcacaatcgtg20<210>13<211>21<212>dna<213>人工序列(artificialsequence)<400>13agcgccaggtccttggtcatc21<210>14<211>20<212>dna<213>人工序列(artificialsequence)<400>14ccgtagtggcggttcatgtc20<210>15<211>20<212>dna<213>人工序列(artificialsequence)<400>15agcctgctgctgagccggtg20<210>16<211>21<212>dna<213>人工序列(artificialsequence)<400>16gtcgtggtcaccgacgagaac21<210>17<211>21<212>dna<213>人工序列(artificialsequence)<400>17tcggcgaagaattcgtgccac21当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1