一株高效刺激免疫反应的沙门菌突变株及构建方法和应用与流程
本发明属于生物工程技术领域,具体涉及一株高效刺激宿主产生针对志贺氏菌o抗原多糖免疫反应的沙门菌突变株及构建方法和应用。
背景技术:
志贺氏菌属(shigellaspp.)细菌是一类具有高度传染性以及致病性,并且会引起人类严重腹泻甚至死亡的革兰氏阴性菌,俗称痢疾杆菌,是人类细菌性痢疾最为常见的病原菌。痢疾是一种全世界范围内高发的肠道传染病,据保守估计,全世界每年的感染人数超过1.6亿,死亡人数超过110万人,痢疾尤其在发展中国家存在多发现象。
志贺氏菌属细菌分为四个群体。a群:痢疾志贺氏菌(sh.dysenteriae),通称志贺氏痢疾杆菌;b群:福氏志贺氏菌(sh.flexneri),通称福氏痢疾杆菌;c群:鲍氏志贺氏菌(sh.boydii),通称鲍氏痢疾杆菌;d群:宋内氏志贺氏菌(sh.sonnei),通称宋内氏痢疾杆菌。上述志贺氏菌属中各菌株都有强烈的内毒素,作用于肠壁,使通透性增高,从而促使毒素的吸收,继而作用于中枢神经系统及心血管系统,引起临床上一系列毒血症症状,如发热、神志障碍,甚至中毒休克。其毒素破坏粘膜,形成炎症、溃疡,呈现典型的痢疾脓血便。毒素作用于肠壁植物神经,使肠道功能紊乱,肠蠕动共济失调和痉挛,尤其直肠括约肌最明显,因而发生腹痛、里急后重等症状。
志贺氏菌属的发现虽已有一百多年,且针对细菌性痢疾的疫苗研究从20世纪40、50年代就已经开展,但是目前仍有问题需要解决。现有研究已经发现了一些具有潜力的候选疫苗靶标,其中志贺氏菌的脂多糖(lipopolysaccharide)能够表现较好的保护力,有研究证明志贺氏菌脂多糖与蛋白的复合体能够激发小鼠很高的血清抗体滴度,并且能够表现一定的保护力,临床实验表明,福氏2a(shigellaflexneri2a)的o抗原多糖能够引起特异的免疫保护,但脂多糖最关键的问题在于进入宿主体内,不能有效的到达宿主免疫系统被免疫系统识别,从而有一定概率导致免疫失败。这一缺陷极大的限制了志贺氏菌疫苗在实际临床中的应用效果,因此开发新的疫苗迫在眉捷,如何开发新的疫苗能够保证高效刺激宿主产生免疫反应,这一问题目前亟待解决。
技术实现要素:
本发明的目的就是为了解决上述技术问题,而提供一株能够高效刺激宿主产生针对志贺氏菌福氏2a血清型o抗原多糖的免疫反应的沙门菌突变菌株及其构建方法和应用。本发明的突变菌株是通过对沙门菌外膜结构的改造,使其更高效的作为多糖递呈载体来应用,同时将志贺氏菌的o抗原多糖表达至沙门菌外膜中,并纯化外膜囊泡,利用外膜囊泡能够高效刺激宿主产生免疫反应的特性,从而递呈异源多糖抗原作为疫苗,用于防治志贺氏菌的感染。
本发明的目的之一是提供一株高效刺激宿主产生针对志贺氏菌o抗原多糖的免疫反应的沙门菌突变株,该突变株发生以下基因缺失突变δflic、δfljb、δompa、δompc、δompd,并敲除利于志贺氏菌o抗原多糖表达的基因rfbp,同时转入能够表达志贺氏菌o抗原的表达质粒。
本发明提供的上述突变菌株,采用基因工程改造的方法,通过对沙门菌外膜结构的改造,使其更高效的作为多糖递呈载体来应用,通过缺失rfbp基因去掉沙门菌自身o抗原;去掉干扰沙门菌外膜囊泡免疫反应的flic和fljb鞭毛蛋白;去掉沙门菌自身非必须的外膜蛋白ompa、ompc和ompd,以排除自身外膜蛋白对所递呈多糖的免疫干扰;利用表达质粒表达完整志贺氏菌福氏2a菌株的o抗原多糖,通过沙门菌自身waal氨基转移酶连接至沙门菌外膜上。
本发明首次将志贺氏菌的o抗原多糖表达至沙门菌外膜中,并纯化突变株的外膜囊泡,利用外膜囊泡能够高效刺激宿主产生免疫反应的特性,从而递呈异源多糖抗原作为疫苗。此种疫苗设计策略在全球范围内均属首次,同时该技术策略的成功构建也能够运用于其他重要革兰氏阴性肠道致病菌的多糖重组疫苗的研发。
具体的,本发明上述突变菌株的命名为:salmonellaentericaserovartyphimuriumstr.uk-1δflicδfljbδompaδompcδompdδrfbp,保藏号为cctccno:m2020142,保藏于中国典型培养物保藏中心,保藏中心地址为:中国.武汉.武汉大学,保藏时间为2020年5月25日。
具体的,编码所述flic蛋白的基因核苷酸序列如seqidno.1所示,编码所述fljb蛋白的基因核苷酸序列如seqidno.2所示。
具体的,编码所述ompa蛋白的基因核苷酸序列如seqidno.3所示,编码所述ompc蛋白的基因核苷酸序列如seqidno.4所示,编码所述ompd蛋白的基因核苷酸序列如seqidno.5所示。
具体的,所述引入rfbp的突变基因的核苷酸序列如seqidno.6所示。
具体的,所述编码志贺菌o抗原的基因簇序列如seqidno.7所示。
本发明的目的之二是提供上述高效刺激宿主产生针对志贺氏菌o抗原多糖的免疫反应的沙门菌突变株的构建方法,其包括以下步骤:
1)通过同源重组的方法,在鼠伤寒沙门氏菌野生型菌株s100中敲除沙门菌干扰外膜囊泡免疫反应的鞭毛蛋白flic和fljb;
2)通过同源重组的方法,去掉沙门菌自身非必须的外膜蛋白ompa、ompc和ompd;
3)将截断志贺氏菌o抗原多糖表达的突变基因δrfbp引入步骤2)的突变株中,构建得到沙门菌突变菌株。
所述步骤1)、2)和3)的同源重组的方法包括如下步骤:
1)构建同源重组质粒pre112-flic、pre112-fljb、pre112-ompa、pre112-ompc、pre112-ompd、pre112-rfbp以及表达志贺氏菌o抗原的表达质粒;
2)制备鼠伤寒沙门氏菌s100电转化感受态细胞χ7232;
3)将步骤1)所得同源重组质粒通过电转的方式转化入鼠伤寒沙门氏菌s100电转化感受态细胞χ7232中,将电转杯移入电转仪,2500伏电击4毫秒;
4)将电击杯中的液体加入100μl预热的lb培养基,轻轻吹熄混匀后涂布与含有氯霉素抗性(cmr)的平板上,37℃培养过夜,产生的单克隆即为电转成功的菌;
5)通过pcr筛选得到不含flic、fljb、ompa、ompc、ompd和rfbp的突变菌株。
所述鼠伤寒沙门氏菌s100电转化感受态细胞χ7232的制备方法包括如下步骤:
1)挑取单菌落χ7232接种于lb液体培养基中,37℃振荡培养过夜,取400μl母液加入到100ml装有lb培养基的三角瓶中,37℃、180r/min振荡培养至od600=0.8-1;
2)冰浴30min,4℃,4500r/min离心10min收集菌体,沉淀用冰浴预冷的超纯水洗两遍,弃上清;
3)用预冷的10%甘油洗一遍,弃上清,菌体用60μl预冷的10%甘油重悬,即制备好感受态备用
所述同源重组质粒pre112-flic、pre112-fljb、pre112-ompa、pre112-ompc、pre112-ompd、pre112-rfbp的构建步骤为:
1)含有flic基因上下游同源臂的同源重组质粒pre112-flic的构建:
根据genbank上已发表沙门氏菌uk-1,全基因组序列,序列号为:gcf_000006945,设计flic基因的左右同源臂,运用vecternti软件设计下述引物:
flic-1f:5’cgttctttgtcaggtctgtc3’
flic-2r:5’gattagcggccgcgatcttttccttatcaatta3’
flic-2f:5’aagatcgcggccgctaatccggcgattgattcac3’
flic-2r:5’tgtacccggcacagacggtc3’
提取处于对数生长期的鼠伤寒沙门氏菌野生型菌株s100的基因组dna作为模板,分别扩增对应的上下游同源臂片段,得到其左右同源臂的扩增产物,扩增产物利用1f和2r引物进行重叠pcr扩增,用重叠pcr方法连接前后臂片段;再用ahdi酶切自杀质粒载体pya4278,酶切产物与预先pcr扩增的融合片段进行连接并转入鼠伤寒沙门氏菌s100电转化感受态细胞χ7232中,得到重组质粒pre112-flic。
2)含有fljb基因上下游同源臂的同源重组质粒pre112-fljb的构建:
根据genbank上已发表沙门氏菌uk-1,全基因组序列,序列号为:gcf_000006945,设计flic基因的左右同源臂,运用vecternti软件设计下述引物:
fljb-1f:5’agtgagctccacgttcatgt3’
fljb-1r:5’aattagcggccgcaaaattttccttttggaagg3’
fljb-2f:5’attttgcggccgctaatttatttcgttttattc3’
fljb-2r:5’gtcattacctgataattcttc3’
提取处于对数生长期的鼠伤寒沙门氏菌野生型菌株s100的基因组dna作为模板,分别扩增对应的上下游同源臂片段,得到其左右同源臂的扩增产物,扩增产物利用1f和2r引物进行重叠pcr扩增,用重叠pcr方法连接前后臂片段;再用ahdi酶切自杀质粒载体pya4278,酶切产物与预先pcr扩增的融合片段进行连接并转入鼠伤寒沙门氏菌s100电转化感受态细胞χ7232中,得到重组质粒pre112-fljb。
3)含有ompa基因上下游同源臂的同源重组质粒pre112-ompa的构建:
根据genbank上已发表沙门氏菌uk-1,全基因组序列,序列号为:gcf_000006945,设计ompa基因的左右同源臂,运用vecternti软件设计下述引物:
ompa-1f:5’catcctctcacacaacgagac3’
ompa-1r:
5’ctgcaggaatgcggccgccgggggatctgctcaatatt3’
ompa-2f:
5’cggccgcattcctgcaggtaagttatcgtctggtagaaaaac3’
ompa-2r:5’catatgaatccggaactggtc3’
提取处于对数生长期的鼠伤寒沙门氏菌野生型菌株s100的基因组dna作为模板,分别扩增对应的上下游同源臂片段,得到其左右同源臂的扩增产物,扩增产物利用1f和2r引物进行重叠pcr扩增,用重叠pcr方法连接前后臂片段;再用ahdi酶切自杀质粒载体pya4278,酶切产物与预先pcr扩增的融合片段进行连接并转入鼠伤寒沙门氏菌s100电转化感受态细胞χ7232中,得到重组质粒pre112-ompa。
4)含有ompc基因上下游同源臂的同源重组质粒pre112-ompc的构建:
根据genbank上已发表沙门氏菌uk-1,全基因组序列,序列号为:gcf_000006945,设计ompc基因的左右同源臂,运用vecternti软件设计下述引物:
ompc-1f:5’gccaatacgcagcgccgaggtcacg3’
ompc-1r:
5’acctgcaggatgcggccgcggtcagcaaaagatg3’
ompc-2f:
5’ccgcggccgcatcctgcaggtgttattaaccctctg3’
ompc-2r:5’ataggggtaaacagacattcagaag3’
提取处于对数生长期的鼠伤寒沙门氏菌野生型菌株s100的基因组dna作为模板,分别扩增对应的上下游同源臂片段,得到其左右同源臂的扩增产物,扩增产物利用1f和2r引物进行重叠pcr扩增,用重叠pcr方法连接前后臂片段;再用ahdi酶切自杀质粒载体pya4278,酶切产物与预先pcr扩增的融合片段进行连接并转入鼠伤寒沙门氏菌s100电转化感受态细胞χ7232中,得到重组质粒pre112-ompc。
5)含有ompd基因上下游同源臂的同源重组质粒pre112-ompd的构建:
根据genbank上已发表沙门氏菌uk-1,全基因组序列,序列号为:gcf_000006945,设计ompd基因的左右同源臂,运用vecternti软件设计下述引物:
ompd-1f:5’aaagttaatgatgatagcgg3’
ompd-1r:5’ctgcaggaatgcggccgcgttattaaccctctgttata3’
ompd-2f:5’cggccgcattcctgcaggtaatctcgatggatatcgaac3’
ompd-2r:5’cgttaaagcgcatcagcgcg3’
提取处于对数生长期的鼠伤寒沙门氏菌野生型菌株s100的基因组dna作为模板,分别扩增对应的上下游同源臂片段,得到其左右同源臂的扩增产物,扩增产物利用1f和2r引物进行重叠pcr扩增,用重叠pcr方法连接前后臂片段;再用ahdi酶切自杀质粒载体pya4278,酶切产物与预先pcr扩增的融合片段进行连接并转入鼠伤寒沙门氏菌s100电转化感受态细胞χ7232中,得到重组质粒pre112-ompd。
6)含有rfbp基因上下游同源臂的同源重组质粒pre112-rfbp的构建:
根据genbank上已发表沙门氏菌uk-1,全基因组序列,序列号为:gcf_000006945,设计rfbp基因的左右同源臂,运用vecternti软件设计下述引物:
rfbp-1f:5’caactgataaaagtcaatcc3’
rfbp-1r:
5’gtaagcttacctgcaggttaatcctcaccctctga3’
rfbp-2f:
5’gattaacctgcaggtaagcttaccgagaagtactga3’
rfbp-2r:5’atacgacgaggcgtttcgag3’
提取处于对数生长期的鼠伤寒沙门氏菌野生型菌株s100的基因组dna作为模板,分别扩增对应的上下游同源臂片段,得到其左右同源臂的扩增产物,扩增产物利用1f和2r引物进行重叠pcr扩增,用重叠pcr方法连接前后臂片段;再用ahdi酶切自杀质粒载体pya4278,酶切产物与预先pcr扩增的融合片段进行连接并转入鼠伤寒沙门氏菌s100电转化感受态细胞χ7232中,得到重组质粒pre112-rfbp。
同源重组质粒由于不能在沙门氏菌中不能复制存活,将含有同源片段的同源重组质粒通过电转化的方式转入大肠杆菌δasd缺失株χ7213中,通过同源片段的同源重组作用,能够使左右同源臂中缺失目的基因的全片段与基因组中的目的基因上、下游的同源臂发生替换从而将基因敲除。最后利用pcr的方法鉴定得到沙门氏菌突变体δflicfljbδompaδompcδompdδrfbp。
7)志贺氏菌o抗原表达载体的构建:
根据genbank上已发表志贺氏菌福氏2a,全基因组序列,序列号为:gcf_002950215.1,设计o抗原基因的左右同源臂,运用vecternti软件设计下述引物:
2a-f-vector:5’cgcctgcatatagcccatttcagaatggccggccgcagtc3’
2a-r-vector:5’gaaagtcgtggttataccgctcatgagacaataaccctg3’
2a-1f:5’ccgccattctgaaatgggctatatgcaggcgtttgtgaa3’
2a-1r:5’aatgcatttcttactcgataataac3’
2a-2f:5’ctgtgggattaacataccaattcac3’
2a-2r:5’attgtctcatgagcggtataaccacgactttcgatgttg3’
提取处于对数生长期的福氏2a志贺氏菌的基因组dna作为模板,分别扩增对应的上下游同源臂片段,得到其左右同源臂的扩增产物。以其基因组为模板,用lataq聚合酶进行扩增,通过两对引物2a-1f/2a-1r和2a-2f/2a-2r来扩增完整的o抗原基因簇,大小分别约为8000bp,并以载体psc101为模板扩增载体片段,大小约为6000bp,用浓度为0.8%的结晶紫琼脂糖凝胶回收片段,用gibson组装试剂盒(gibsonassemblymastermix,neb),通过在设计引物时在片段中加入overlap片段,从而片段能够通过重叠片段,并通过gibson组装酶连接成一个完整的质粒。加好体系后在50摄氏度作用1小时,取2微升用电转化的方法将连接物转化入top10感受态细胞内,涂布于kan平板上,放置与37℃待菌落长出后挑菌用相应引物进行pcr鉴定,pcr鉴定为阳性的菌落再次进行对其表达o抗原的后续鉴定。
本发明的目的之三是提供上述高效刺激宿主产生志贺氏菌o抗原多糖免疫反应的沙门菌突变株的应用,是将该突变菌株纯化其自发产生的外膜囊泡用于制备成疫苗用于防治志贺氏菌的感染。
与现有技术相比,本发明的有益效果如下:
1)本发明首次将志贺氏菌的o抗原多糖表达至沙门菌外膜上,通过纯化其外膜囊泡,利用外膜囊泡能够高效刺激宿主产生免疫反应的特性,从而递呈异源多糖抗原作为疫苗。
2)本发明通过对沙门菌外膜结构的改造,使其更高效的作为多糖递呈载体来应用,包括:缺失rfbp基因去掉沙门菌自身o抗原;去掉干扰沙门菌外膜囊泡免疫反应的flic和fljb鞭毛蛋白;去掉沙门菌自身非必须的外膜蛋白ompa、ompc和ompd,以排除自身外膜蛋白对所递呈多糖的免疫干扰;利用表达质粒表达完整志贺氏菌福氏2a菌株的o抗原多糖,通过沙门菌自身waal氨基转移酶连接至沙门菌外膜上。
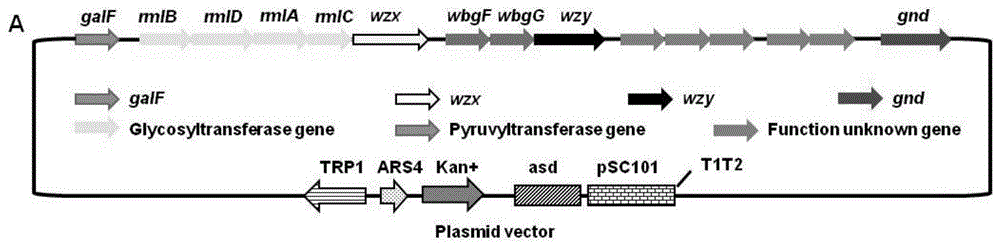
附图说明
图1为构建得到的表达lps、s.flexneri2ao-抗原基因簇的质粒。
图2为利用cryo-em鉴定所纯化得到的能够表达志贺氏菌o抗原多糖的沙门菌突变株的外膜囊泡外观形态。
图3为采用银染法对表达志贺氏菌o抗原多糖的鼠伤寒沙门氏菌抗原递呈疫苗的外膜囊泡脂多糖图谱的鉴定结果。
图4为采用免疫印迹法对表达志贺氏菌o抗原多糖的鼠伤寒沙门氏菌抗原递呈疫苗的外膜囊泡的鉴定结果。
图5为验证突变菌株外膜囊泡介导志贺菌o抗原多糖诱导免疫应答的测试结果。
具体实施方式
为了使本发明的目的、技术方案及优点更加清楚明白,以下结合实施例对本发明进行具体描述,有必要指出的是,以下实施例仅仅用于对本发明进行解释和说明,并不用于限定本发明。本领域技术人员根据上述发明内容所做出的一些非本质的改进和调整,仍属于本发明的保护范围。
实施例1
突变菌株的构建方法:
以鼠伤寒沙门菌uk-1菌株为模板,对基因进行敲除,构建突变株qs001(δflicδfljbδompaδompcδompd)。纯化其外膜囊泡,通过sds-page验证外膜囊泡中不含鞭毛蛋白以及ompacd。验证构建成功后,再将截断脂多糖o抗原链的突变(δrfbp)引入突变株qs001中,构建o抗原突变株,并通过pcr以及银染(sliverstaining)观察其脂多糖图谱的方法鉴定所构建的突变株。
实验例1
构建表达lps、s.flexneri2ao-抗原基因簇的质粒:
1、δfilc引物设计和pcr扩增
根据已报到的沙门氏菌s100菌株序列,(参照genbank序列号为gcf_000006945)设计两对融合pcr引物分别扩增filc基因的上游同源臂以及下游同源臂,扩增片段大小分别为300bp,上述引物均由北京华大基因公司合成,引物序列如下:
flic-1f:cgttctttgtcaggtctgtc
flic-1r:gattagcggccgcgatcttttccttatcaatta
flic-2f:aagatcgcggccgctaatccggcgattgattcac
flic-2r:tgtacccggcacagacggtc
2、沙门氏菌filc基因的上、下游同源臂的扩增
煮沸裂解法制备鼠伤寒沙门氏菌s100的基因组dna模板:挑取鼠伤寒沙门氏菌s100单菌落于lb培养基37℃过夜培养,第二天取0.5ml菌液12000r/min离心3min,弃上清,收集菌体用超纯水洗一次,并重悬。放于沸水中煮10min,冷却过后12000r/min离心3min,以上清作为模板备用。
pcr的扩增反应在50μl的体系中进行,反应体系如下:10×pcrbuffer(mg2+)2.5μl,dntp(10mm)1.5μl,primer1(50mm)0.25μl,primer2(50mm)0.25μl,dna模板2μl,超纯水17μl,taqdna高保真酶0.5μl。
扩增条件为:98℃变性3min后进入循环;循环参数为98℃解链10sec,55℃退火30sec,72℃延伸30sec,30个循环后,72℃保温10min,最终4℃保存。扩增的pcr产物经1%的琼脂糖凝胶电泳分析,扩增两个片段大小分别为300bp,与预期大小相当。回收产物过后,利用1f和2r引物进行重叠pcr扩增,用重叠pcr方法连接前后臂片段,回收纯化连接产物为δflic片段。
3、pre112-filc同源重组质粒的构建
用ahdi酶切自杀质粒载体pre112,由于酶切之后载体会出现一个t-terminal,切胶回收该片段,将得到的载体酶切产物在t4连接酶的作用下与预先pcr扩增已+a的δflic融合片段进行连接,连接体系为:pre112载体片段5μl,δflic融合片段5μl,t4连接酶buffer,2.5μl,t4连接酶1μl,16℃水浴过夜,将连接产物电转化入感受态细胞χ7232,得到重组质粒pre112-filc。
首先制备电转感受态细胞χ7232,挑取单菌落χ7232接种于lb液体培养基中,37℃振荡培养过夜。取400μl母液加入到100ml装有lb培养基的三角瓶中,37℃、180r/min振荡培养至od600=0.8-1,冰浴30min,4℃,4500r/min离心10min收集菌体,沉淀用冰浴预冷的超纯水洗两遍,最后一步用预冷的10%甘油洗一遍,最终用60μl预冷的10%甘油重悬备用。将连接产物加入到感受态细胞中充分混匀,加入到直径为2mm的电击杯中,冰浴使电击杯冷却,在电压2.5kv的参数下进行电击,随即在电击杯中加入100μl预热的lb培养基,轻轻吹熄混匀后涂布与含有氯霉素抗性(cmr)的平板上,37℃培养过夜,待菌落长出后用pcr进行筛选鉴定,鉴定结果证明pre112-flic质粒构建成功。
4、filc基因缺失突变株的构建与鉴定
利用重组自杀质粒pre112-flic在大肠杆菌δasd缺失株χ7213中可以复制,但在沙门氏菌中不能复制存活的特点,将在χ7232菌株中构建得到的自杀质粒进行提取质粒,将其电转入感受态χ7213中,该菌缺失了asd基因,所以其需要dap才能够生长,将自杀质粒转入χ7213之后,培养含有质粒pre112-flic的χ7213菌以及鼠伤寒沙门氏菌s100,待其培养至od值约为0.8左右时进行结合转移,待结合培养24小时过后,挑取菌苔划线于氯霉素平板上,同源片段与载体片段导入沙门氏菌基因组的细菌能够在抗性平板上生长,随机挑取能够在抗性平板上生长的细菌进行第二次同源重组,将单菌落挑取至lb液体培养基培养,置于37℃振荡培养2-3个小时,待菌od600值达到0.6时,将菌进行1:100稀释后取100μl涂布含有5%的蔗糖平板进行第二次筛选,插入有sacb敏感基因的菌株在蔗糖平板上不生长,无抗性的突变菌株与回复突变菌株能够在蔗糖平板上生长,最后将得到对抗生素敏感而对蔗糖抗性的克隆,进行pcr鉴定,pcr鉴定大小结果显示,突变株成功缺失了大小约为900bp的flic基因,即得到pre112δfilc突变菌株。
pcr鉴定:提取flic突变菌株pre112δfilc基因组dna,用引物filc-1f和filc-2r扩增上下游同源臂片段,看能否扩增出大小为800bp左右的缺失了flic基因的上下游同源臂片段,如果能扩增出相应片段,则表明结果与预期相符,可初步鉴定突变株构建成功(命名为s.typhimuriums100δflic)。
实施例2
1、δfljb引物设计和pcr扩增
根据已报到的沙门氏菌s100菌株序列,(参照genbank序列号为gcf_000006945)设计两对融合pcr引物分别扩增fljb基因的上游同源臂以及下游同源臂,扩增片段大小分别为300bp,上述引物均由北京华大基因公司合成,引物序列如下:
fljb-1f:agtgagctccacgttcatgt
fljb-1r:aattagcggccgcaaaattttccttttggaagg
fljb-2f:attttgcggccgctaatttatttcgttttattc
fljb-2r:gtcattacctgataattcttc
2、沙门氏菌fljb基因的上、下游同源臂的扩增
煮沸裂解法制备鼠伤寒沙门氏菌s100的基因组dna模板:挑取鼠伤寒沙门氏菌s100单菌落于lb培养基37℃过夜培养,第二天取0.5ml菌液12000r/min离心3min,弃上清,收集菌体用超纯水洗一次,并重悬。放于沸水中煮10min,冷却过后12000r/min离心3min,以上清作为模板备用。
pcr的扩增反应在25μl的体系中进行,反应体系如下:10×pcrbuffer(mg2+)2.5μl,dntp(10mm)1.5μl,primer1(50mm)0.25μl,primer2(50mm)0.25μl,dna模板2μl,超纯水17μl,taqdna高保真酶0.5μl。
扩增条件为:98℃变性3min后进入循环;循环参数为98℃解链10sec,55℃退火30sec,72℃延伸30sec,30个循环后,72℃保温10min,最终4℃保存。扩增的pcr产物经1%的琼脂糖凝胶电泳分析,扩增两个片段大小分别为300bp,与预期大小相当。回收产物过后,利用1f和2r引物进行重叠pcr扩增,用重叠pcr方法连接前后臂片段,回收纯化连接产物为δfljb片段。
3、pre112-fljb同源重组质粒的构建
用ahdi酶切自杀质粒载体pre112,由于酶切之后载体会出现一个t-terminal,切胶回收该片段,将得到的载体酶切产物在t4连接酶的作用下与预先pcr扩增已+a的δfljb融合片段进行连接,连接体系为:pre112载体片段5μl,δflic融合片段5μl,t4连接酶buffer,2.5μl,t4连接酶1μl,16℃水浴过夜,将连接产物电转化入感受态细胞χ7232,得到重组质粒pre112-fljb。
首先制备电转感受态细胞χ7232,挑取单菌落χ7232接种于lb液体培养基中,37℃振荡培养过夜。取400μl母液加入到100ml装有lb培养基的三角瓶中,37℃、180r/min振荡培养至od600=0.8-1,冰浴30min,4℃,4500r/min离心10min收集菌体,沉淀用冰浴预冷的超纯水洗两遍,最后一步用预冷的10%甘油洗一遍,最终用60μl预冷的10%甘油重悬备用。将连接产物加入到感受态细胞中充分混匀,加入到直径为2mm的电击杯中,冰浴使电击杯冷却,在电压2.5kv的参数下进行电击,随即在电击杯中加入100μl预热的lb培养基,轻轻吹熄混匀后涂布与含有氯霉素抗性(cmr)的平板上,37℃培养过夜,待菌落长出后用pcr进行筛选鉴定,鉴定结果证明pre112-fljb质粒构建成功。
4、沙门氏菌δflicδfljb缺失株的突变与鉴定
利用重组自杀质粒pre112-fljb在大肠杆菌δasd缺失株χ7213中可以复制,但在沙门氏菌中不能复制存活的特点,将在χ7232菌株中构建得到的自杀质粒进行提取质粒,将其电转入感受态χ7213中,该菌缺失了asd基因,所以其需要dap才能够生长,将自杀质粒转入χ7213之后,培养含有质粒pre112-fljb的χ7213菌以及鼠伤寒沙门氏菌δflic突变缺失株(命名为s.typhimuriums100δfljb),待其培养至od值约为0.8左右时进行结合转移,待结合培养24小时过后,挑取菌苔划线于氯霉素平板上,同源片段与载体片段导入沙门氏菌基因组的细菌能够在抗性平板上生长,随机挑取能够在抗性平板上生长的细菌进行第二次同源重组,将单菌落挑取至lb液体培养基培养,置于37℃振荡培养2-3个小时,待菌od600值达到0.6时,将菌进行1:100稀释后取100μl涂布含有5%的蔗糖平板进行第二次筛选,插入有sacb敏感基因的菌株在蔗糖平板上不生长,无抗性的突变菌株与回复突变菌株能够在蔗糖平板上生长,最后将得到对抗生素敏感而对蔗糖抗性的克隆,进行pcr鉴定,pcr鉴定大小结果显示,突变株成功缺失了大小约为800bp的fljb基因,即得到pre112δfilcδfljb突变菌株。
pcr鉴定:提取δfljb突变菌株pre112δfilcδfljb基因组dna,用引物fljb-1f和fljb-2r扩增上下游同源臂片段,看能否扩增出大小为800bp左右的缺失了fljb基因的上下游同源臂片段,如果能扩增出相应片段,则表明结果与预期相符,可初步鉴定突变株构建成功(命名为s.typhimuriums100δflicδfljb)。
实施例3
1、δompa引物设计和pcr扩增
根据已报到的沙门氏菌s100菌株序列,(参照genbank序列号为gcf_000006945)设计两对融合pcr引物分别扩增ompa基因的上游同源臂以及下游同源臂,扩增片段大小分别为400bp,上述引物均由北京华大基因公司合成,引物序列如下:
ompa-1f:5’catcctctcacacaacgagac3’
ompa-1r:
5’ctgcaggaatgcggccgccgggggatctgctcaatatt3’
ompa-2f:
5’cggccgcattcctgcaggtaagttatcgtctggtagaaaaac3’
ompa-2r:5’catatgaatccggaactggtc3’
2、沙门氏菌ompa基因的上、下游同源臂的扩增
煮沸裂解法制备鼠伤寒沙门氏菌s100的基因组dna模板:挑取鼠伤寒沙门氏菌s100单菌落于lb培养基37℃过夜培养,第二天取0.5ml菌液12000r/min离心3min,弃上清,收集菌体用超纯水洗一次,并重悬。放于沸水中煮10min,冷却过后12000r/min离心3min,以上清作为模板备用。
pcr的扩增反应在25μl的体系中进行,反应体系如下:10×pcrbuffer(mg2+)2.5μl,dntp(10mm)1.5μl,primer1(50mm)0.25μl,primer2(50mm)0.25μl,dna模板2μl,超纯水17μl,taqdna高保真酶0.5μl。
扩增条件为:98℃变性3min后进入循环;循环参数为98℃解链10sec,55℃退火30sec,72℃延伸30sec,30个循环后,72℃保温10min,最终4℃保存。扩增的pcr产物经1%的琼脂糖凝胶电泳分析,扩增两个片段大小分别为400bp,与预期大小相当。回收产物过后,利用1f和2r引物进行重叠pcr扩增,用重叠pcr方法连接前后臂片段,回收纯化连接产物为δompa片段。
3、pre112-ompa同源重组质粒的构建
用ahdi酶切自杀质粒载体pya4278,由于酶切之后载体会出现一个t-terminal,切胶回收该片段,将得到的载体酶切产物在t4连接酶的作用下与预先pcr扩增已+a的δompa融合片段进行连接,连接体系为:pre112载体片段5μl,δompa融合片段5μl,t4连接酶buffer,2.5μl,t4连接酶1μl,16℃水浴过夜,将连接产物电转化入感受态细胞χ7232,得到重组质粒pre112-ompa。
首先制备电转感受态细胞χ7232,挑取单菌落χ7232接种于lb液体培养基中,37℃振荡培养过夜。取400μl母液加入到100ml装有lb培养基的三角瓶中,37℃、180r/min振荡培养至od600=0.8-1,冰浴30min,4℃,4500r/min离心10min收集菌体,沉淀用冰浴预冷的超纯水洗两遍,最后一步用预冷的10%甘油洗一遍,最终用60μl预冷的10%甘油重悬备用。将连接产物加入到感受态细胞中充分混匀,加入到直径为2mm的电击杯中,冰浴使电击杯冷却,在电压2.5kv的参数下进行电击,随即在电击杯中加入100μl预热的lb培养基,轻轻吹熄混匀后涂布与含有氯霉素抗性(cmr)的平板上,37℃培养过夜,待菌落长出后用pcr进行筛选鉴定,鉴定结果证明pre112-ompa质粒构建成功。
4、沙门氏菌δflicδfljbδompa缺失株的突变与鉴定
利用重组自杀质粒pre112-ompa在大肠杆菌δasd缺失株χ7213中可以复制,但在沙门氏菌中不能复制存活的特点,将在χ7232菌株中构建得到的自杀质粒进行提取质粒,将其电转入感受态χ7213中,该菌缺失了asd基因,所以其需要dap才能够生长,将自杀质粒转入χ7213之后,培养含有质粒pre112-ompa的χ7213菌以及鼠伤寒沙门氏菌δflicδfljb突变缺失株(命名为s.typhimuriums100δflicδfljb),待其培养至od值约为0.8左右时进行结合转移,待结合培养24小时过后,挑取菌苔划线于氯霉素平板上,同源片段与载体片段导入沙门氏菌基因组的细菌能够在抗性平板上生长,随机挑取能够在抗性平板上生长的细菌进行第二次同源重组,将单菌落挑取至lb液体培养基培养,置于37℃振荡培养2-3个小时,待菌od600值达到0.6时,将菌进行1:100稀释后取100μl涂布含有5%的蔗糖平板进行第二次筛选,插入有sacb敏感基因的菌株在蔗糖平板上不生长,无抗性的突变菌株与回复突变菌株能够在蔗糖平板上生长,最后将得到对抗生素敏感而对蔗糖抗性的克隆,进行pcr鉴定,pcr鉴定大小结果显示,突变株成功缺失了大小约为900bp的ompa基因,即得到pre112δfilcδfljbδompa突变菌株。
pcr鉴定:提取δompa突变菌株pre112δfilcδfljbδompa基因组dna,用引物ompa-1f和ompa-2r扩增上下游同源臂片段,看能否扩增出大小为900bp左右的缺失了ompa基因的上下游同源臂片段,如果能扩增出相应片段,则表明结果与预期相符,可初步鉴定突变株构建成功(命名为s.typhimuriums100δflicδfljbδompa)。
实施例4
1、δompc引物设计和pcr扩增
根据已报到的沙门氏菌s100菌株序列,(参照genbank序列号为gcf_000006945)设计两对融合pcr引物分别扩增ompc基因的上游同源臂以及下游同源臂,扩增片段大小分别为400bp,上述引物均由北京华大基因公司合成,引物序列如下:
ompc-1f:5’gccaatacgcagcgccgaggtcacg3’
ompc-1r:5’acctgcaggatgcggccgcggtcagcaaaagatg3’
ompc-2f:5’ccgcggccgcatcctgcaggtgttattaaccctctg3’
ompc-2r:5’ataggggtaaacagacattcagaag3’
2、沙门氏菌ompc基因的上、下游同源臂的扩增
煮沸裂解法制备鼠伤寒沙门氏菌s100的基因组dna模板:挑取鼠伤寒沙门氏菌s100单菌落于lb培养基37℃过夜培养,第二天取0.5ml菌液12000r/min离心3min,弃上清,收集菌体用超纯水洗一次,并重悬。放于沸水中煮10min,冷却过后12000r/min离心3min,以上清作为模板备用。
pcr的扩增反应在25μl的体系中进行,反应体系如下:10×pcrbuffer(mg2+)2.5μl,dntp(10mm)1.5μl,primer1(50mm)0.25μl,primer2(50mm)0.25μl,dna模板2μl,超纯水17μl,taqdna高保真酶0.5μl。
扩增条件为:98℃变性3min后进入循环;循环参数为98℃解链10sec,55℃退火30sec,72℃延伸30sec,30个循环后,72℃保温10min,最终4℃保存。扩增的pcr产物经1%的琼脂糖凝胶电泳分析,扩增两个片段大小分别为400bp,与预期大小相当。回收产物过后,利用1f和2r引物进行重叠pcr扩增,用重叠pcr方法连接前后臂片段,回收纯化连接产物为δompc片段。
3、pre112-ompc同源重组质粒的构建
用ahdi酶切自杀质粒载体pya4278,由于酶切之后载体会出现一个t-terminal,切胶回收该片段,将得到的载体酶切产物在t4连接酶的作用下与预先pcr扩增已+a的δompc融合片段进行连接,连接体系为:pre112载体片段5μl,δompc融合片段5μl,t4连接酶buffer,2.5μl,t4连接酶1μl,16℃水浴过夜,将连接产物电转化入感受态细胞χ7232,得到重组质粒pre112-ompc。
首先制备电转感受态细胞χ7232,挑取单菌落χ7232接种于lb液体培养基中,37℃振荡培养过夜。取400μl母液加入到100ml装有lb培养基的三角瓶中,37℃、180r/min振荡培养至od600=0.8-1,冰浴30min,4℃,4500r/min离心10min收集菌体,沉淀用冰浴预冷的超纯水洗两遍,最后一步用预冷的10%甘油洗一遍,最终用60μl预冷的10%甘油重悬备用。将连接产物加入到感受态细胞中充分混匀,加入到直径为2mm的电击杯中,冰浴使电击杯冷却,在电压2.5kv的参数下进行电击,随即在电击杯中加入100μl预热的lb培养基,轻轻吹熄混匀后涂布与含有氯霉素抗性(cmr)的平板上,37℃培养过夜,待菌落长出后用pcr进行筛选鉴定,鉴定结果证明pre112-ompc质粒构建成功。
4、沙门氏菌δflicδfljbδompaδompc缺失株的突变与鉴定
利用重组自杀质粒pre112-ompc在大肠杆菌δasd缺失株χ7213中可以复制,但在沙门氏菌中不能复制存活的特点,将在χ7232菌株中构建得到的自杀质粒进行提取质粒,将其电转入感受态χ7213中,该菌缺失了asd基因,所以其需要dap才能够生长,将自杀质粒转入χ7213之后,培养含有质粒pre112-ompc的χ7213菌以及鼠伤寒沙门氏菌δflicδfljbδompa突变缺失株(命名为s.typhimuriums100δflicδfljbδompa),待其培养至od值约为0.8左右时进行结合转移,待结合培养24小时过后,挑取菌苔划线于氯霉素平板上,同源片段与载体片段导入沙门氏菌基因组的细菌能够在抗性平板上生长,随机挑取能够在抗性平板上生长的细菌进行第二次同源重组,将单菌落挑取至lb液体培养基培养,置于37℃振荡培养2-3个小时,待菌od600值达到0.6时,将菌进行1:100稀释后取100μl涂布含有5%的蔗糖平板进行第二次筛选,插入有sacb敏感基因的菌株在蔗糖平板上不生长,无抗性的突变菌株与回复突变菌株能够在蔗糖平板上生长,最后将得到对抗生素敏感而对蔗糖抗性的克隆,进行pcr鉴定,pcr鉴定大小结果显示,突变株成功缺失了大小约为900bp的ompc基因,即得到pre112δfilcδfljbδompaδompc突变菌株。
pcr鉴定:提取δompc突变菌株pre112δfilcδfljbδompaδompc基因组dna,用引物ompc-1f和ompc-2r扩增上下游同源臂片段,看能否扩增出大小为900bp左右的缺失了ompc基因的上下游同源臂片段,如果能扩增出相应片段,则表明结果与预期相符,可初步鉴定突变株构建成功(命名为s.typhimuriums100δflicδfljbδompaδompc)。
实施例5
1、δompd引物设计和pcr扩增
根据已报到的沙门氏菌s100菌株序列,(参照genbank序列号为gcf_000006945)设计两对融合pcr引物分别扩增ompd基因的上游同源臂以及下游同源臂,扩增片段大小分别为400bp,上述引物均由北京华大基因公司合成,引物序列如下:
ompd-1f:aaagttaatgatgatagcgg
ompd-1r:ctgcaggaatgcggccgcgttattaaccctctgttata
ompd-2f:cggccgcattcctgcaggtaatctcgatggatatcgaac
ompd-2r:cgttaaagcgcatcagcgcg
2、沙门氏菌ompd基因的上、下游同源臂的扩增
煮沸裂解法制备鼠伤寒沙门氏菌s100的基因组dna模板:挑取鼠伤寒沙门氏菌s100单菌落于lb培养基37℃过夜培养,第二天取0.5ml菌液12000r/min离心3min,弃上清,收集菌体用超纯水洗一次,并重悬。放于沸水中煮10min,冷却过后12000r/min离心3min,以上清作为模板备用。
pcr的扩增反应在25μl的体系中进行,反应体系如下:10×pcrbuffer(mg2+)2.5μl,dntp(10mm)1.5μl,primer1(50mm)0.25μl,primer2(50mm)0.25μl,dna模板2μl,超纯水17μl,taqdna高保真酶0.5μl。
扩增条件为:98℃变性3min后进入循环;循环参数为98℃解链10sec,55℃退火30sec,72℃延伸30sec,30个循环后,72℃保温10min,最终4℃保存。扩增的pcr产物经1%的琼脂糖凝胶电泳分析,扩增两个片段大小分别为400bp,与预期大小相当。回收产物过后,利用1f和2r引物进行重叠pcr扩增,用重叠pcr方法连接前后臂片段,回收纯化连接产物为δompd片段。
3、pre112-ompd同源重组质粒的构建
用ahdi酶切自杀质粒载体pya4278,由于酶切之后载体会出现一个t-terminal,切胶回收该片段,将得到的载体酶切产物在t4连接酶的作用下与预先pcr扩增已+a的δompa融合片段进行连接,连接体系为:pre112载体片段5μl,δompd融合片段5μl,t4连接酶buffer,2.5μl,t4连接酶1μl,16℃水浴过夜,将连接产物电转化入感受态细胞χ7232,得到重组质粒pre112-ompd。
首先制备电转感受态细胞χ7232,挑取单菌落χ7232接种于lb液体培养基中,37℃振荡培养过夜。取400μl母液加入到100ml装有lb培养基的三角瓶中,37℃、180r/min振荡培养至od600=0.8-1,冰浴30min,4℃,4500r/min离心10min收集菌体,沉淀用冰浴预冷的超纯水洗两遍,最后一步用预冷的10%甘油洗一遍,最终用60μl预冷的10%甘油重悬备用。将连接产物加入到感受态细胞中充分混匀,加入到直径为2mm的电击杯中,冰浴使电击杯冷却,在电压2.5kv的参数下进行电击,随即在电击杯中加入100μl预热的lb培养基,轻轻吹熄混匀后涂布与含有氯霉素抗性(cmr)的平板上,37℃培养过夜,待菌落长出后用pcr进行筛选鉴定,鉴定结果证明pre112-ompd质粒构建成功。
4、沙门氏菌δflicδfljbδompaδompcδompd缺失株的突变与鉴定
利用重组自杀质粒pre112-ompd在大肠杆菌δasd缺失株χ7213中可以复制,但在沙门氏菌中不能复制存活的特点,将在χ7232菌株中构建得到的自杀质粒进行提取质粒,将其电转入感受态χ7213中,该菌缺失了asd基因,所以其需要dap才能够生长,将自杀质粒转入χ7213之后,培养含有质粒pre112-ompd的χ7213菌以及鼠伤寒沙门氏菌δflicδfljbδompaδompc突变缺失株,待其培养至od值约为0.8左右时进行结合转移,待结合培养24小时过后,挑取菌苔划线于氯霉素平板上,同源片段与载体片段导入沙门氏菌基因组的细菌能够在抗性平板上生长,随机挑取能够在抗性平板上生长的细菌进行第二次同源重组,将单菌落挑取至lb液体培养基培养,置于37℃振荡培养2-3个小时,待菌od600值达到0.6时,将菌进行1:100稀释后取100μl涂布含有5%的蔗糖平板进行第二次筛选,插入有sacb敏感基因的菌株在蔗糖平板上不生长,无抗性的突变菌株与回复突变菌株能够在蔗糖平板上生长,最后将得到对抗生素敏感而对蔗糖抗性的克隆,进行pcr鉴定,pcr鉴定大小结果显示,突变株成功缺失了大小约为900bp的ompd基因,即得到pre112δfilcδfljbδompaδompcδompd突变菌株。
pcr鉴定:提取δompd突变菌株pre112δfilcδfljbδompaδompcδompd基因组dna,用引物ompd-1f和ompd-2r扩增上下游同源臂片段,看能否扩增出大小为900bp左右的缺失了ompd基因的上下游同源臂片段,如果能扩增出相应片段,则表明结果与预期相符,可初步鉴定突变株构建成功(命名为s.typhimuriums100δflicδfljbδompaδompcδompd)。
实施例6:
1、δrfbp引物设计和pcr扩增
根据已报到的沙门氏菌s100菌株序列,(参照genbank序列号为gcf_000006945)设计两对融合pcr引物分别扩增rfbp基因的上游同源臂以及下游同源臂,扩增片段大小分别为400bp,上述引物均由北京华大基因公司合成,引物序列如下:
rfbp-1f:5’caactgataaaagtcaatcc3’
rfbp-1r:5’gtaagcttacctgcaggttaatcctcaccctctga3’
rfbp-2f:5’gattaacctgcaggtaagcttaccgagaagtactga3’
rfbp-2r:5’atacgacgaggcgtttcgag3’
2、沙门氏菌rfbp基因的上、下游同源臂的扩增
煮沸裂解法制备鼠伤寒沙门氏菌s100的基因组dna模板:挑取鼠伤寒沙门氏菌s100单菌落于lb培养基37℃过夜培养,第二天取0.5ml菌液12000r/min离心3min,弃上清,收集菌体用超纯水洗一次,并重悬。放于沸水中煮10min,冷却过后12000r/min离心3min,以上清作为模板备用。
pcr的扩增反应在25μl的体系中进行,反应体系如下:10×pcrbuffer(mg2+)2.5μl,dntp(10mm)1.5μl,primer1(50mm)0.25μl,primer2(50mm)0.25μl,dna模板2μl,超纯水17μl,taqdna高保真酶0.5μl。
扩增条件为:98℃变性3min后进入循环;循环参数为98℃解链10sec,55℃退火30sec,72℃延伸30sec,30个循环后,72℃保温10min,最终4℃保存。扩增的pcr产物经1%的琼脂糖凝胶电泳分析,扩增两个片段大小分别为400bp,与预期大小相当。回收产物过后,利用1f和2r引物进行重叠pcr扩增,用重叠pcr方法连接前后臂片段,回收纯化连接产物为δrfbp片段。
3、pre112-rfbp同源重组质粒的构建
用ahdi酶切自杀质粒载体pya4278,由于酶切之后载体会出现一个t-terminal,切胶回收该片段,将得到的载体酶切产物在t4连接酶的作用下与预先pcr扩增已+a的δrfbp融合片段进行连接,连接体系为:pre112载体片段5μl,δrfbp融合片段5μl,t4连接酶buffer,2.5μl,t4连接酶1μl,16℃水浴过夜,将连接产物电转化入感受态细胞χ7232,得到重组质粒pre112-rfbp。
首先制备电转感受态细胞χ7232,挑取单菌落χ7232接种于lb液体培养基中,37℃振荡培养过夜。取400μl母液加入到100ml装有lb培养基的三角瓶中,37℃、180r/min振荡培养至od600=0.8-1,冰浴30min,4℃,4500r/min离心10min收集菌体,沉淀用冰浴预冷的超纯水洗两遍,最后一步用预冷的10%甘油洗一遍,最终用60μl预冷的10%甘油重悬备用。将连接产物加入到感受态细胞中充分混匀,加入到直径为2mm的电击杯中,冰浴使电击杯冷却,在电压2.5kv的参数下进行电击,随即在电击杯中加入100μl预热的lb培养基,轻轻吹熄混匀后涂布与含有氯霉素抗性(cmr)的平板上,37℃培养过夜,待菌落长出后用pcr进行筛选鉴定,鉴定结果证明pre112-rfbp质粒构建成功。
4、沙门氏菌δflicδfljbδompaδompcδompdδrfbp缺失株的突变与鉴定
利用重组自杀质粒pre112-rfbp在大肠杆菌δasd缺失株χ7213中可以复制,但在沙门氏菌中不能复制存活的特点,将在χ7232菌株中构建得到的自杀质粒进行提取质粒,将其电转入感受态χ7213中,该菌缺失了asd基因,所以其需要dap才能够生长,将自杀质粒转入χ7213之后,培养含有质粒pre112-rfbp的χ7213菌以及鼠伤寒沙门氏菌δflicδfljbδompaδompcδompd突变缺失株,待其培养至od值约为0.8左右时进行结合转移,待结合培养24小时过后,挑取菌苔划线于氯霉素平板上,同源片段与载体片段导入沙门氏菌基因组的细菌能够在抗性平板上生长,随机挑取能够在抗性平板上生长的细菌进行第二次同源重组,将单菌落挑取至lb液体培养基培养,置于37℃振荡培养2-3个小时,待菌od600值达到0.6时,将菌进行1:100稀释后取100μl涂布含有5%的蔗糖平板进行第二次筛选,插入有sacb敏感基因的菌株在蔗糖平板上不生长,无抗性的突变菌株与回复突变菌株能够在蔗糖平板上生长,最后将得到对抗生素敏感而对蔗糖抗性的克隆,进行pcr鉴定,pcr鉴定大小结果显示,突变株成功缺失了大小约为900bp的rfbp基因,即得到pre112δfilcδfljbδompaδompcδompdδrfbp突变菌株。
pcr鉴定:提取δrfbp突变菌株pre112δfilcδfljbδompaδompcδompdδrfbp基因组dna,用引物rfbp-1f和rfbp-2r扩增上下游同源臂片段,看能否扩增出大小为900bp左右的缺失了rfbp基因的上下游同源臂片段,如果能扩增出相应片段,则表明结果与预期相符,可初步鉴定突变株构建成功(命名为s.typhimuriums100δflicδfljbδompaδompcδompdδrfbp)。
实施例6
1、o抗原基因簇引物设计和pcr扩增
根据已报到的志贺氏菌福氏2a菌株序列,(参照genbank序列号为gcf_002950215.1)设计两对pcr引物来扩增完整的o抗原基因簇,大小分别约为8000bp,引物序列如下:
2a-f-vector:
cgcctgcatatagcccatttcagaatggccggccgcagtc
2a-r-vector:
gaaagtcgtggttataccgctcatgagacaataaccctg
2a-1f:
5’ccgccattctgaaatgggctatatgcaggcgtttgtgaa3’
2a-1r:5’aatgcatttcttactcgataataac3’
2a-2f:5’ctgtgggattaacataccaattcac3’
2a-2r:
5’attgtctcatgagcggtataaccacgactttcgatgttg3’
2、志贺氏菌福氏2ao抗原基因簇的扩增:
煮沸裂解法制备志贺氏菌福氏2a的基因组dna模板,挑取单菌落37℃过夜培养。取0.5ml菌液12000r/min离心3min,弃上清,收集菌体用超纯水洗一次,并重悬。放于沸水中煮10min,冷却过后12000r/min离心3min,以上清作为模板备用。
pcr的扩增反应在25μl的体系中进行,反应体系如下:10×pcrbuffer(mg2+)2.5μl,dntp(10mm)3μl,primer1(50mm)0.25μl,primer2(50mm)0.25μl,dna模板2μl,超纯水17μl,lataqdna聚合酶0.5μl。
扩增条件为:95℃预变性3min后进入循环,循环参数为98℃10sec,55℃30sec,68℃8min。30个循环后,72℃保温10min,最终4℃保存。扩增的pcr产物经0.8%的琼脂糖凝胶电泳分析,扩增的两个片段大小分别为8000bp,与预期大小相当。
3、质粒构建
质粒构建需要用到gibson组装试剂盒(gibsonassemblymastermix,neb),通过在设计引物时在片段中加入overlap片段,从而片段能够通过重叠片段,并通过gibson组装酶连接成一个完整的质粒。将片段按照相同摩尔数比控制总体积在20微升加好反应体系:组装片段(2-3个)xμl,总量为0.02-0.5pmols,gibson组装酶10μl,超纯水10-xμl,加好体系后在50摄氏度作用1小时,取2微升用电转化的方法将连接物转化入top10感受态细胞内,涂布于kan平板上,放置与37℃待菌落长出后挑菌用相应引物进行pcr鉴定。鉴定结果证实构建质粒正确。
4、top10电转化感受态细胞制作流程如下:
挑取对数生长期的单菌落大肠杆菌top10接种于lb液体培养基中,37℃振荡培养过夜。取400μl母液加入到100ml装有lb培养基的三角瓶中,37℃、180r/min振荡培养至od600=0.8-1,冰浴30min,4℃,低转速4500r/min离心10min收集菌体,重复洗涤三次,最终用60μl预冷的10%甘油重悬,即制备好感受态备用。
如图1所示,为构建得到的表达lps、s.flexneri2ao-抗原基因簇的质粒所包含的基因实验例2
验证表达o抗原突变株构建成功:
1、志贺氏菌o抗原表达质粒的鉴定
lps图谱鉴定:过夜培养5mlpcr鉴定为阳性的包含有质粒的大肠杆菌top10菌株,离心收集菌体,取200μl缓冲液a(组份为:0.5mtris-clph6.8,10%glycerol,10%sds以及5%beta-mercaptoethanol)重悬菌体,样品充分混匀后在沸水中煮10分钟,待样品冷却后离心15分钟以除去未溶解的杂质,离心完成后,将上清按照1:10(10μl加至90μl)的比例加入缓冲液b(组份为:0.5mtris-clph6.8,10%glycerol,0.05%bromophenolblue)中,并加入1μl浓度为20mg/ml的蛋白酶k。充分混匀后将样品放置于37℃。一小时过后,将样品取15μl在15%浓度的sds-page胶进行电泳,待胶跑完后,将胶进行银氨染色,若表达志贺氏菌o抗原,则能够看到有条带,若不表达,则top10的脂多糖结构表现为rough型。
2、染色步骤简要如下:
1)固定:将胶放入固定液中固定过夜,之后用超纯水漂洗三次,每次10min;
2)敏化:向染色盘中加入敏化液,轻摇10min,之后用超纯水洗三次,每次10min;
3)上银:向染色盘中加入银染液,轻摇10min,之后用超纯水洗三次,每次10min;
4)显色:向染色盘中加入显色液,轻轻振荡,待显出条带后,立即换超纯水,将胶漂洗至不变色为止,进行照相保存分析。
3、westernblot鉴定:
将处理好的脂多糖样品通过sds-page胶分离,待电泳完成后,将lps条带通过转印仪转印至硝酸纤维素膜(nitrocellulosemembrane)上。待转膜完成,用含有5%脱脂牛奶的tris缓冲液封闭2小时,随即加入特异性针对志贺氏菌福氏2ao抗原的兔抗血清(1:100稀释)放置于室温孵育2小时。孵育完成后,用碱性磷酸酶标记的羊抗兔igg二抗以1:10000倍稀释进行孵育,两小时后用tbs缓冲液洗三遍。免疫印记条带通过加入bcip显色液进行显色。反应终止则通过将膜放入超纯水中清洗,则终止反应。其中亲本志贺氏菌福氏2a菌株为阳性对照,未转入质粒的top10菌株为阴性对照。
如图2所示利用cryo-em鉴定所纯化得到的能够表达志贺氏菌o抗原多糖的沙门菌突变株的外膜囊泡外观形态。
如图3所示采用银染法对表达志贺氏菌o抗原多糖的鼠伤寒沙门氏菌抗原递呈疫苗的外膜囊泡脂多糖图谱的鉴定结果。
如图4所示采用免疫印迹法对表达志贺氏菌o抗原多糖的鼠伤寒沙门氏菌抗原递呈疫苗的外膜囊泡的鉴定结果。
实验例3
验证突变菌株omv介导志贺菌o抗原诱导免疫应答的能力:
免疫:本研究采用4-6周龄(16-22g)的雌性balb/c小鼠,购买于成都达硕生物技术有限公司,并采用两种免疫途径,包括滴鼻(intranasal)与腹腔注射(intraperitoneal)来评估沙门氏菌外膜囊泡引起机体产生的免疫效力与保护野生毒株攻击的能力。将新购进的小鼠饲养一周以适应新环境,将小鼠按照每组8只分为若干组,从第一次免疫算起,0天和第30天分别作为首免和加强免疫(二免)。免疫计量分别为:滴鼻免疫组为20μg/10μldpbs浓度的外膜囊泡;腹腔免疫组为5μg/100μldpbs浓度的外膜囊泡。对照组则免疫相应体积的dpbs缓冲液作为阴性对照。血清样品采集:采用眼眶窦静脉从穿刺采血,于一免后4周以及二免后4周采集,将采集到的血液静置于37℃一小时后放于4℃过夜,待血清析出后,采集血清放置于-80℃冰箱备用。阴道分泌物样品的采集:取150μlpbs缓冲液在阴道口来回冲洗,并收集阴道分泌物放于-80℃冰箱备用。
攻毒保护效力评价:我们利用志贺氏菌小鼠肺部病理模型来评估沙门氏菌多糖抗原递呈疫苗对野生型志贺氏菌的攻击保护效力,具体实施如下:
(1)于二免后第五周进行攻毒,用致死计量109cfu/10μldpbs的志贺氏菌通过滴鼻的方式进行攻毒,攻毒完24小时后,分别于免疫组以及对照组取3只小鼠解剖,取其肺脏进行病理切片制作,将组织依次在乙醇和二甲苯中浸泡,然后用石蜡进行包埋,并使组织脱水,脱水完成后将石蜡包埋样品切成5mm的切片,进行苏木精-伊红染色。染色完成的结果在电子显微镜下进行照相观察分析,通过病理切片以及病理损伤分数的统计来反应疫苗的保护效力。
(2)将攻毒完后24小时的部分小鼠取其肺脏灌洗液进行细胞因子的测定,肺脏灌洗液采集步骤简要如下:将小鼠处死过后,对颈部进行解刨,暴露气管,利用穿刺针或者4.5号针头进行插管,用预冷的pbs缓冲液0.8ml进行灌洗,灌洗3-5次即可,收集的灌洗液放置于-80℃备用。最终检测其肺泡灌洗液中iga、tnf-α、ifn-γ和il-6的含量,检测方法利用elisa检测试剂盒(r&dsystem)进行,所有的操作步骤严格按照试剂盒说明书严格进行。
(3)剩余小鼠则继续观察其体重变化以及是否死亡,记录其每天死亡小鼠的数量,观察持续到攻毒后14天,最终统计结果进行保护效力分析。
抗体含量测定:间接酶联免疫吸附实验(elisa)被用于抗体含量的测定,针对志贺氏菌脂多糖(lps)的抗体含量被测定,具体步骤如下:
(1)我们采用绘制不同抗体特异性的标准曲线的方法,来定量不同个体中所含有的抗体含量,所以需要将纯化的鼠ig亚型标准品从0.5μg/μl,按照2倍的倍数进行倍比稀释,按照同样的方法进行包被,同时待测样品则以1μg每孔的脂多糖稀释于100μl包被缓冲液中加至96孔elisa板中,放置于4℃过夜孵育。
(2)孵育过夜后,将elisa板用pbst(pbs包含0.1%tween20)洗三遍,随即用2%bsa缓冲液进行封闭,于室温静置2小时。
(3)将不同的血清样品与阴道分泌物样品以一定的稀释比例加100μl入孔中,每个样品进行三个重复,加好样品后放置于室温1小时。
(4)待pbst洗三遍结束之后,分别在不同板子里加入不同的生物素标记的羊抗鼠igg、igg1、igg2a和iga,测定不同的ig亚型含量,同上仍在室温放置2小时。
(5)pbst洗三遍过后,加入碱性磷酸酶标记的链霉亲和素作用1小时。
(6)最终用pnpp(p-nitrophenylphosphatesubstrate)进行显色。颜色反应(吸光度)测定其在波长为405nm时的吸光度。数值大于pbs对照的被认为是阳性。
(7)标准曲线的绘制使用curveexpert软件进行,利用log-logregressioncurve曲线函数得到标准曲线,将测得的样品数值带入,即可得到样品中抗体含量,最终的ig抗体含量需要换算样品的稀释倍数,得到最终的抗体含量。
统计分析:所有数据均已mean±sd表示,采用软件graphprim6对数据进行绘图分析,用软件spssstatistics17.0对数据进行单因素方差分析。当p<0.05被认为是微显著差异,当p<0.01被认为是显著差异,当p<0.001被认为是极显著差异。
注:动物实验是按照动物福利法案和有关动物实验的规定进行的。本研究严格遵循实验动物的使用和关爱原则。所有对实验动物的不利影响在不影响实验结果的情况下都控制在最小范围。
如图5所示:验证突变菌株外膜囊泡介导志贺菌o抗原多糖诱导免疫应答的测试结果。
序列表
<110>南昌大学
<120>一株高效刺激免疫反应的沙门菌突变株及构建方法和应用
<141>2020-06-22
<160>7
<170>siposequencelisting1.0
<210>1
<211>1488
<212>dna
<213>salmonellaenterica
<400>1
atggcacaagtcattaatacaaacagcctgtcgctgttgacccagaataacctgaacaaa60
tcccagtccgctctgggcaccgctatcgagcgtctgtcttccggtctgcgtatcaacagc120
gcgaaagacgatgcggcaggtcaggcgattgctaaccgttttaccgcgaacatcaaaggt180
ctgactcaggcttcccgtaacgctaacgacggtatctccattgcgcagaccactgaaggc240
gcgctgaacgaaatcaacaacaacctgcagcgtgtgcgtgaactggcggttcagtctgct300
aacagcaccaactcccagtctgacctcgactccatccaggctgaaatcacccagcgcctg360
aacgaaatcgaccgtgtatccggccagactcagttcaacggcgtgaaagtcctggcgcag420
gacaacaccctgaccatccaggttggtgccaacgacggtgaaactatcgatatcgatctg480
aagcagatcaactctcagaccctgggtctggatacgctgaatgtgcaacaaaaatataag540
gtcagcgatacggctgcaactgttacaggatatgccgatactacgattgctttagacaat600
agtacttttaaagcctcggctactggtcttggtggtactgaccagaaaattgatggcgat660
ttaaaatttgatgatacgactggaaaatattacgccaaagttaccgttacggggggaact720
ggtaaagatggctattatgaagtttccgttgataagacgaacggtgaggtgactcttgct780
ggcggtgcgacttccccgcttacaggtggactacctgcgacagcaactgaggatgtgaaa840
aatgtacaagttgcaaatgctgatttgacagaggctaaagccgcattgacagcagcaggt900
gttaccggcacagcatctgttgttaagatgtcttatactgataataacggtaaaactatt960
gatggtggtttagcagttaaggtaggcgatgattactattctgcaactcaaaataaagat1020
ggttccataagtattaatactacgaaatacactgcagatgacggtacatccaaaactgca1080
ctaaacaaactgggtggcgcagacggcaaaaccgaagttgtttctattggtggtaaaact1140
tacgctgcaagtaaagccgaaggtcacaactttaaagcacagcctgatctggcggaagcg1200
gctgctacaaccaccgaaaacccgctgcagaaaattgatgctgctttggcacaggttgac1260
acgttacgttctgacctgggtgcggtacagaaccgtttcaactccgctattaccaacctg1320
ggcaacaccgtaaacaacctgacttctgcccgtagccgtatcgaagattccgactacgcg1380
accgaagtttccaacatgtctcgcgcgcagattctgcagcaggccggtacctccgttctg1440
gcgcaggcgaaccaggttccgcaaaacgtcctctctttactgcgttaa1488
<210>2
<211>1520
<212>dna
<213>salmonellaenterica
<400>2
atggcacaagtaatcaacactaacagtctgtcgctgctgacccagaataacctgaacaaa60
tcccagtccgcactgggcaccgctatcgagcgtctgtcttctggtctgcgtatcaacagc120
gcgaaagacgatgcggcaggtcaggcgattgctaaccgtttcaccgcgaacatcaaaggt180
ctgactcaggcttcccgtaacgctaacgacggtatctccattgcgcagaccactgaaggc240
gcgctgaacgaaatcaacaacaacctgcagcgtgtgcgtgaactggcggttcagtctgct300
aacagcaccaactcccagtctgacctcgactccatccaggctgaaatcacccagcgcctg360
aacgaaatcgaccgtgtatccggccagactcagttcaacggcgtgaaagtcctggcgcag420
gacaacaccctgaccatccaggttggcgccaacgacggtgaaactatcgatatcgatctg480
aagcagatcaactctcagaccctgggtctggactcactgaacgtgcagaaagcgtatgat540
gtgaaagatacagcagtaacaacgaaagcttatgccaataatggtactacactggatgta600
tcgggtcttgatgatgcagctattaaagcggctacgggtggtacgaatggtacggcttct660
gtaaccggtggtgcggttaaatttgacgcagataataacaagtactttgttatattggtg720
gctttactggtgctgatgccgccaaaaatggcgattatgaagttaacgttgctactgacg780
gtacagtaacccttgcggctggcgcaactaaaaccacaatgcctgctggtgcgacaacta840
aaacagaagtacaggagttaaaagatacaccggcagttgtttcagcagatgctaaaaatg900
ccttaattgctggcggcgttgacgctaccgatgctaatggcgctgagttggtcaaaatgt960
cttataccgataaaaatggtaagacaattgaaggcggttatgcgcttaaagctggcgata1020
agtattacgccgcagattacgatgaagcgacaggagcaattaaagctaaaactacaagtt1080
atactgctgctgacggcactaccaaaacagcggctaaccaactgggtggcgtagacggta1140
aaaccgaagtcgttactatcgacggtaaaacctacaatgccagcaaagccgctggtcatg1200
atttcaaagcacaaccagagctggcggaagcagccgctaaaaccaccgaaaacccgctgc1260
agaaaattgatgccgcgctggcgcaggtggatgcgctgcgctctgatctgggtgcggtac1320
aaaaccgtttcaactctgctatcaccaacctgggcaataccgtaaacaatctgtctgaag1380
cgcgtagccgtatcgaagattccgactacgcgaccgaagtttccaacatgtctcgcgcgc1440
agattctgcagcaggccggtacttccgttctggcgcaggctaaccaggtcccgcagaacg1500
tgctgtctctgttacgttaa1520
<210>3
<211>1053
<212>dna
<213>salmonellaenterica
<400>3
atgaaaaagacagctatcgcgattgcagtggcactggctggtttcgctaccgtagcgcag60
gccgctccgaaagataacacctggtacgctggtgctaaactgggctggtctcagtaccat120
gacaccggcttcattcacaatgatggcccgactcatgaaaaccaactgggcgcaggtgct180
tttggtggttaccaggttaacccgtatgttggctttgaaatgggctacgactggttaggc240
cgtatgccgtacaaaggcgacaacatcaatggcgcttataaagctcagggcgttcagttg300
accgctaaactgggttatccaatcactgacgatctggacgtttatacccgtctgggtggt360
atggtatggcgtgcagacaccaagtctaacgtccctggcggcccgtctactaaagaccac420
gacaccggcgtttccccggtattcgcgggcggtatcgagtatgctatcacccctgaaatc480
gcaacccgtctggaataccagtggactaacaacatcggtgatgccaacaccatcggcacc540
cgtccggacaacggcctgctgagcgtaggtgtttcctaccgtttcggccagcaagaagct600
gctccggtagtagctccggcaccagctccggctccggaagtacagaccaagcacttcact660
ctgaagtctgacgtactgttcaacttcaacaaatctaccctgaagccggaaggccagcag720
gctctggatcagctgtacagccagctgagcaacctggatccgaaagacggttccgttgtc780
gttctgggcttcactgaccgtatcggttctgacgcttacaaccagggtctgtccgagaaa840
cgtgctcagtctgttgttgattacctgatctccaaaggtattccgtctgacaaaatctcc900
gcacgtggtatgggcgaatctaacccggttaccggcaacacctgtgacaacgtgaaacct960
cgcgctgccctgatcgattgcctggctccggatcgtcgcgtagagatcgaagttaaaggc1020
gttaaagacgtggtaactcagccgcaggcttaa1053
<210>4
<211>908
<212>dna
<213>salmonellaenterica
<400>4
atgaaagttaaagtactgtccctcctggtaccagctctgctggtggcgggcgcagcgaat60
gcggctgaaatttataataaagacggcaacaaattagacctgtttggtaaagttgatggc120
ctgcactacttctctgacgacaaaggcagcgatggcgaccagacctacatgcgtatcggc180
ttcaaaggcgaaacgcaggttaacgatcagctgaccggttatggccagtgggaatatcag240
attcagggcaaccagactgaaggcagcaacgactcctggacgcgtgtggcgtttgcgggt300
ctgaaattcgctgatgcaggttccttcgattatggtcgtaactacggcgtaacctatgac360
gtgacctcctggaccgacgttctgccggagttcggcggcgacacctacggcgctgacaac420
tttatgcagcagcgtggtaacggctatgctacctaccgtaacaccgacttcttcggcctg480
gtggatggtctggacttcgcgttacagtatcagggcaaaaacggcagcgtgagcggtgaa540
aacaccaacggtcgcagcctgctgaaccagaacggcgacggttacggcggatcgctgact600
tatgcaatcggcgaaggcttctctgtcggtggcgctatcaccacgtctaaacgtactgcc660
gatcagaacaacaccgctaacgctcgcctgtatggtaacggcgatcgcgccacggtttac720
accggcggcctgaaatacgatgcgaacaacatctatctggcagcgcagtattctcagacc780
tataacgcaacccgttttggtacctctaacggtagcaacccgtccacctcttacggtttt840
gccaacaaagcgcagaactttgaagtggttgctcagtaccagttcgactttggtctgcgt900
ccgtctgt908
<210>5
<211>1089
<212>dna
<213>salmonellaenterica
<400>5
atgaaacttaagttagtggcagtggcagtgacttccctgttggcagcaggcgtggtaaac60
gcagccgaggtatataacaaagacggtaataaactggatctgtacggcaaagttcatgct120
cagcattatttctctgatgataacggaagtgatggcgacaaaacttacgcgcgactgggt180
tttaaaggcgaaacgcagatcaacgaccaactgaccggttttggtcagtgggaatatgaa240
ttcaaaggcaaccgtactgaaagccagggttctgacaaagataaaactcgtctggcattt300
gctggcctgaaattcgctgactacggttctttcgactatggccgtaactacggcgttgct360
tacgacatcggcgcatggaccgacgttctgccggaattcggcggcgacacctggactcag420
gctgatgtcttcatgactggccgtactactggcgttgcaacctaccgtaacactgacttc480
ttcggtctggttgatgggctgaattttgccgcacagtatcagggcaaaaacgaccgtgcc540
aatgcttatgagtctaacggcgacggtttcggtttgtccgcaacgtatgaatacgaaggc600
tttggcgtaggtgcggcctatgcgaaatctgaccgtactaacaaccaggttaaagctgca660
agcaatctgaacgccgcaggtaaaaatgctgaagtctgggctgctggcctgaaatatgat720
gcgaacaacatctacctggcgacgacctactccgaaacgctgaacatgaccaacttcggt780
gaagatgccgtaggtaacgcgtttatcgcaaacaaaacccagaactttgaagcggttgct840
cagtaccagtttgacttcggtctgcgtccatctatcgcttacctgaaatccaaaggtaaa900
aacctcggtactttcggtgaccaggatctggttgagtacatcgatctcggcgcaacctac960
tacttcaacaaaaacatgtctaccttcgttgattacaaaatcaacctgctggacgacagc1020
aacttcaccaaagcagctaaagtgtctaccgacaacatcgttgctgttggtctgaactat1080
cagttctaa1089
<210>6
<211>1431
<212>dna
<213>salmonellaenterica
<400>6
atggataatattgataataagtataatccacagctatgtaaaatttttttggctatatcg60
gatttgattttttttaatttagccttatggttttcattaggatgtgtctattttattttt120
gatcaagtacagcgatttattcctcaagaccaattagatacaagagttattacgcatttt180
attttgtcagtagtatgtgtcggttggttttggattcgtttgcgacattatacataccgc240
aagccattttggtatgagttaaaagaaatttttcgtacgatcgttatttttgctatattt300
gatttggctctgatagcgtttacaaaatggcagttttcacgctatgtctgggtgttttgt360
tggacttttgccctaatcctggtgcctttttttcgcgcacttacaaagcatttattgaac420
aagctaggtatctggaagaaaaaaactatcatcctggggagcggacagaatgctcgtggt480
gcatattctgcgctgcaaagtgaggagatgatggggtttgatgttatcgctttttttgat540
acggatgcgtcagatgctgaaataaatatgttgccggtgataaaggatactgagattatt600
tgggatttaaatcgtacaggtgatgtccattatatccttgcttatgaatacaccgagttg660
gagaaaacacatttttggctacgtgaactttcaaaacatcattgtcgttctgttactgta720
gtcccctcgtttagaggattgccattatataatactgatatgtcttttatctttagccat780
gaagttatgttattaaggatacaaaataacttggctaaaaggtcgtcccgttttctcaaa840
cggacatttgatattgtttgttcaataatgattcttataattgcatcaccacttatgatt900
tatctgtggtataaagttactcgagatggtggtccggctatttatggtcaccagcgagta960
ggtcggcatggaaaactttttccatgctacaaatttcgttctatggttatgaattctcaa1020
gaggtactaaaagaacttttggctaacgatcctattgccagggctgaatgggagaaagat1080
tttaaactgaaaaatgatcctcgaatcacagctgtaggtcgatttatacgtaaaactagc1140
cttgatgagttgccacaactttttaatgtactaaaaggtgatatgagcctggttggacca1200
cgacctatcgtttcggatgaactggagcgttattgtgatgatgttgattattatttgatg1260
gcaaagccgggcatgacaggtctatggcaagtgagtgggcgtaatgatgttgattatgac1320
actcgtgtttattttgattcctggtatgttaaaaactggacgctttggaatgatattgcc1380
attctgtttaaaacagcgaaagttgttttgcggcgagatggtgcgtattaa1431
<210>7
<211>7583
<212>dna
<213>salmonellaenterica
<400>7
tgaagatacttgttactggtggcgcaggatttattggttctgctgtagttcgtcacatta60
taaataatacgcaggatagtgttgttaatgtcgataaattaacgtacgccggaaacctgg120
agtcacttgctgatgtttctgactctaaacgctatgtttttgaacatgcggatatttgcg180
atgctgctgcaatggcgcggatttttgctcagcatcagccggatgcagtgatgcacctgg240
ctgctgaaagccatgtggatcgttcaattacaggccctgcggcatttattgaaaccaata300
ttgttggtacttatgtccttttggaagcggctcgcaattactggtctgctcttgatggcg360
acaagaaaaatagcttccgttttcatcatatttctactgacgaagtctatggtgatttgc420
ctcatcctgacgaagtaaataataaagaacaattacccctctttactgagacgacagctt480
acgcgcctagtagtccttattccgcatcaaaagcatccagcgatcatttagtccgtgcgt540
ggaaacgtacctatggtttaccgaccattgtgactaactgttcgaataactacggtcctt600
atcactttccggaaaaattgattccactagtaattcttaatgctctggaaggtaaggcat660
tacctatttatggcaaaggggatcaaattcgtgactggctgtatgttgaagatcatgcgc720
gtgcgttatatatcgtcgtaaccgaaggtaaagcgggtgaaacttataacattggtggac780
acaacgaaaagaaaaacatcgatgtagtgctcactatttgtgatttgttggatgagattg840
taccgaaagagaaatcttaccgcgagcaaattacttatgttgccgatcgcccgggacacg900
atcgccgttatgcgattgatgcagagaagattagccgcgaattgggctggaaaccgcagg960
aaacgtttgagagcgggattcgtaaaacggtgggatggtacctctccaatacaaaatggg1020
ttgataatgtaaaaagtggtgcctatcaatcgtggattgaacagaactatgagggccgcc1080
agtaatgaatatcctccttttcggcaaaacagggcaggtaggttgggaactacagcgtgc1140
tctggcacctctgggtaatttgattgctcttgatgttcactccactgattactgtggtga1200
ttttagtaatcctgaaggtgtagctgaaaccgtaagaagcattcggcctgatattattgt1260
caacgcagccgctcacaccgcagtagacaaagcagaatcagaaccggagtttgcacaatt1320
acttaacgcgacgagtgtcgaagcgatcgcgaaagcagccaatgaagtcggcgcctgggt1380
tattcactactctactgactacgtatttccggggaccggtgaaataccatggcaggaggc1440
ggatgcaaccgcaccgctaaatgtttacggtgaaaccaagttagctggagaaaaagcatt1500
acaagagcattgtgcgaagcacctaattttccgtacaagctgggtctatgcaggtaaagg1560
aaataacttcgccaaaacgatgttgcgtctgggaaaagagcgtgaagaattagccgttat1620
taatgatcagtttggtgcgccaacaggtgctgaactgctggctgattgtacggcacatgc1680
aattcgtgtggcactgaataaaccagaagtcgcaggcttgtaccatctggtagccactgg1740
taccacaacctggcacgattatgctgcgctggtttttgaagaggcacgaaaagcaggtat1800
tccccttgcactcaacaagctcaacgcagtaccaacaacagcttatcctacaccagctcg1860
tcgtccacataactctcgccttaatacagaaaaatttcagcaaaattttgcgcttgtttt1920
gcctgactggcaggttggcgtgaaacgaatgctcaacgaattatttacgactacagcaat1980
ttaatagtttttgcatcttgttcgtgatgatggagcaagatgaattaaaaggaatgatgt2040
aatgaaaacgcgtaaaggtattattttagcgggtggctctggtactcgtctttatcctgt2100
gactatggctgtcagtaaacagctattacctatttatgataagccgatgatctattaccc2160
gctctctacactgatgttggcgggtattcgcgatattctgattattagtacgccacagga2220
tactcctcgttttcaacaactcctgggtgatggtagccagtgggggttaaatcttcagta2280
caaagtgcaaccgagtccagatggtcttgcgcaggcatttatcatcggtgaagagtttat2340
cggtggtgatgattgtgctctggttctcggtgataatatcttctacggtcatgatctgcc2400
gaagttaatggatgtcgctgtcaacaaagaaagtggtgcaacggtatttgcctatcacgt2460
taatgatcctgaacgctacggtgttgttgagtttgataaaaacggtacggcaatcagcct2520
ggaagaaaaaccgctacaaccaaaaagtaattatgcggtaaccgggctttatttctatga2580
taacgacgttgtcgaaatggcgaaaaaccttaagccttctgcccgtggtgaactggaaat2640
taccgatattaaccgtatttatatggagcaggggcgtttatccgttgccatgatgggacg2700
tggttatgcatggctggacacggggacacatcaaagtcttattgaagcaagcaacttcat2760
tgcaacaattgaagagcgccaagggttaaaggtatcttgcctggaagagattgcttatcg2820
taaaggctttattgacgcagagcaggttaatgtattagccgaaccgctaaagaaaaatgc2880
ttatggtcagtatctgttgaaaatgattaaaggttattaaaaatgaatgtaattaaaact2940
gaaattccagatgtattaattttcgagccgaaagtttttggtgatgaacgtggttttttt3000
atggaaagctttaaccagaaagttttcgaagaggctgtagggcggaaggttgaatttgtt3060
caggataaccattctaaatcaactaagggtgtgttacgcggactgcactatcagttggaa3120
ccttatgctcaaggtaaattagttcgttgtgttgtcggtgaagtttttgatgtagcagtt3180
gatattcgtaaatcgtcacctacatttgggaaatggattggggtgaatttgtctgctgag3240
aataagcgtcagttgtggatacctgaaggatttgcgcatggatttttggtgctgagtgaa3300
acggctgagtttgtttataaaacaacaaactattacaatccaagttttgaaaaaagtatt3360
tcatactcagatcctaccattaaaattcagtggcccaatttacaggatatgcattttaaa3420
ttatcaaataaggatttgaatgctaagaacttttttaataacaatagtttaatgcaatga3480
agaaaaatatattgctcttgttcttagtacatggggcaaattatttgttcccgtttatag3540
ttcttccatatcaaactcgaatattaagcatcgagacattcgcagatgtagcaaaaattc3600
aagccgctgtgatgcttttatctttaatcgtaaattatggatataacttatcaagtacaa3660
gagctatagctagggccgtatctcaagcagaaataaataagatctatagtgagactctta3720
ttgtaaaattattattggcaaccatttgtcttgcacttggttgcgtacatttgatgtatg3780
tcaaagagtactcattgatatatccttttataatcagttcgatatatctttatggtagtg3840
cattatttgctacttggttattccaaggacttgagaaaatgaaagcggtcgttatagcaa3900
caacaatcgctaaactgactggtgtgatacttacttttattttagttaagtctccaaatg3960
atatagttgcagctctttttacacaaaacattgggatgtttataagtggtataatatcta4020
tttatttggtaaggaaaaacaaatatgcaaccgtaatatgttttcgacttaaaaatatta4080
ttgtaagcttaaaagaagcgtggccgttttttttatcattagctgcaacaagtgtatata4140
catattttaatgtgattttattatctttttatgctggcgactatgttgtggcaaatttta4200
atgctgctgataaattaagaatggctgctcaagggttacttattccaataggacaggctg4260
ttttcccacgattatctaaactagagggctatgaatatagttctaaacttaaaatttatg4320
caataaggtatgctatttttggtgtttgcattagtgcgggacttgtatttttaggtccca4380
tgttaactactatttatttaggcaaagaatattcgttgtcaggagaatatcttcaaagta4440
tgtttttactacctgccactatttcaatatcgactatactgagtcaatggatgttgatac4500
ctcaaggcaaagaaaaaatattaagcagaatctatattctaggcgccattgtccatttat4560
tatatgcatttcctttagtttactattatggggcttggggcatggtaatatcaattttat4620
ttactgaagtcttaattgtattatttatgcttaaggctgtgaaatgacttactttactgg4680
ttttattttaatattgtttgctattataattaaaagattaactccaagtcaaagcaagaa4740
aaatattgtcttaatagctaatgcgttttggggaatattgttggtaggttatgctttcaa4800
tgaacaatatttcgtaccattaagtgcaacaaccttgttttttatacttgcattcttatt4860
tttctttagtatgacttatattttaattgctaggagtggaagggttgttttttctttcgg4920
tactggttttatagaaagcaaatatatttactggtttgctgggatgattaatattattag4980
tatctgctttggcattatccttttatataataatcatttttctttaaaagtaatgagaga5040
aggaattttagatggttctattagtgggtttggattggggataagtttgccactttcctt5100
ctgctgtatgtatttagcaagacatgagaataaaaaaaattatttctattgttttacact5160
actttcattcttgcttgcggtgttatcaacttcaaagatcttcttaatattattccttgt5220
atatattgttggaataaatagttatgtaagcaaaaagaaattgcttatttatggagtgtt5280
tgtatttggactgttcgctttatcaagtattatcttgggtaagttctcttcagaccctga5340
aggcaagattatttcagcaatatttgatacgttaagggtttatcttttctcgggattggc5400
agcctttaatctttatgttgaaaagaatgccacgctccccgaaaatttacttttgtatcc5460
atttaaggaggtttgggggacgacaaaagatattcccaaaactgatattttgccttggat5520
caacattggtgtatgggacacgaatgtatatacagcttttgcaccatggtatcagtcatt5580
gggattatatgcagctataattattggtattctcttagggttttattacgggatatggtt5640
tagctttcgtcaaaatttagctgtgggtttttatcaaacatttttgtgttttcctctttt5700
aatgttgtttttccaggagcattatttgttgtcatggaaaatgcattttatttatttttt5760
atgtgcaattttattagcgatgagaaaagcattagagtatgaataaatattgtatcttag5820
tactatttaatccagatataagtgtttttattgataatgtcaaaaagattttatctttgg5880
atgtaagtttatttgtatatgacaattcagcaaataaacatgcattccttgctctatcct5940
cacaagagcaaacaaagataaattacttttcgatatgtgaaaatatcggattgtcgaaag6000
cttataatgagacactaaggcatattcttgaatttaataagaatgtgaaaaataaaagca6060
ttaatgatagtgtgctttttctcgaccaagactctgaagttgatttaaattccatcaata6120
ttttgtttgaaactatatcagcagcagagtctaatgtgatgatagtcgcggggaatccca6180
taaggagagatggactaccgtatatagattacccccacactgtaaacaatgtaaaatttg6240
taattagtagttatgctgtgtatcgcttagacgcatttagaaacatcggcttgtttcaag6300
aagatttttttatagatcatatcgatagtgatttttgttcaaggctgataaaaagcaatt6360
accaaattctccttagaaaagatgcctttttttatcaaccaataggaataaaaccattca6420
atctctgtggtagatatttattccctatcccatcacaacaccgaacatattttcaaatta6480
gaaatgcttttttaagttacaggcgcaatggtgttacatttaattttttatttagggaaa6540
ttgtaaatagattgattatgagtatattctcaggccttaacgagaaagacttattgaaac6600
gattgcatttatatttaaaaggaataaaagatggtcttaaaatgtaattcttggctagaa6660
gtgggggcgttgtgattaaaaaaaaagtggcggcgataattataacatataatccagatc6720
taacaattctgcgagaaagttatacgagtctatataagcaagtcgataaaataattctta6780
ttgataacaactctacaaactatcaagaacttaagaagttattcgaaaaaaaagaaaaaa6840
taaaaatagtgcccttgagtgataatataggactagcagcagctcaaaatttaggtttga6900
acttagctattaaaaataactatacttatgctattttattcgatcaggatagcgtcttac6960
aagacaatggaattaacagtttcttttttgaatttgagaaattagttagtgaagaaaaat7020
taaatatagttgccattgggccaagtttttttgacgaaaagacaggaagacgctttcggc7080
ctacaaaatttatcggtccctttttatatccctttcgtaaaataaccacaaaaaatcctc7140
taacagaagttgacttcttgattgcttctggttgtttcataaaattggagtgtattaaat7200
cagccggaatgatgactgaatcgttattcatcgattatattgatgttgaatggtcatatc7260
gtatgcgttcgtatggctataagctatatattcataatgatattcacatgagtcatttag7320
tgggagaatctcgagttaatttaggattgaaaactatttctttacatgggccgctaagac7380
gatattacttatttaggaattatatttcaattttaaaagtgagatatataccgttaggat7440
ataaaatacgtgagggtttttttaatatcggaagatttttggtaagtatgattataacta7500
aaaatagaaaaactttaattttatacactataaaagcaattaaggacggaataaataatg7560
aaatggggaaatataaaggctaa7583
- 还没有人留言评论。精彩留言会获得点赞!