高效分泌表达几丁二糖脱乙酰酶的枯草芽孢杆菌及其应用
本发明属于代谢工程技术领域,尤其涉及一种高效分泌表达几丁二糖脱乙酰酶的枯草芽孢杆菌及其应用。
背景技术:
氨基葡萄糖glcn作为一种天然的水溶性单糖,在医药保健、皮肤美容、农业生产等领域有着重要应用。目前,在许多国家,氨基葡萄糖glcn已被列为医药品行列。此外,利用氨基葡萄糖glcn与阿魏酸结合,可获得具有良好水溶性的新型化妆品材料,具有美白补水的功效。另一方面,氨基葡萄糖glcn可作为农作物的排毒剂的主要成分,当在植物生长期内使用时,可排出植物体内的重金属及农药污染物等有害物质。
近年来,氨基葡萄糖glcn的市场需求量逐年递增,人们开始对氨基葡萄糖glcn的工业化生产进行关注。目前工业上氨基葡萄糖glcn的生产方法是化学水解法及微生物发酵法。其中化学水解法常用强酸热解几丁质脱除乙酰基,容易导致严重的环境污染问题,且获得产品中乙酰基的脱除程度不一致,产品均一性差。利用微生物发酵法生产氨基葡萄糖glcn,安全无毒,大大降低了强酸强碱的使用量,但发酵液中无法实现高浓度的积累,众多研究将研究重点放在生产氨基葡萄糖glcn的前体物质n-乙酰氨基葡萄糖glcnac上,实现在发酵液中大量积累n-乙酰氨基葡萄糖glcnac后,再利用酸解法将n-乙酰氨基葡萄糖glcnac水解为氨基葡萄糖glcn,但该过程需利用盐酸进行加热反应,使用大量强酸试剂,依然会造成环境污染及设备腐蚀等问题。综上所述,这两种方法普遍存在制备步骤复杂、反应条件苛刻、环境污染严重、生产设备易腐蚀和制备周期长等问题。因此,为了解决目前存在的问题,需要找到一种安全高效且以环境友好型的方式生产氨基葡萄糖glcn。
相较于上述两种方法,酶法催化具有底物来源充足,不受原材料限制,制备条件温和,环境污染小,反应专一性高,体系中杂质少易于下游分离纯化,生产成本低等优点。目前市场上存在的脱乙酰酶,多是针对以寡聚体或多聚体形式存在的乙酰氨基葡萄进行作用的,而针对单体形式作用的脱乙酰酶鲜有报道。而来源于热球菌属的几丁二糖脱乙酰酶dac属于糖类酯酶,可以利用n-乙酰氨基葡萄糖glcnac为底物进行单酶催化反应,实现氨基葡萄糖glcn的一步生产。因此,建立几丁二糖脱乙酰酶dac高效表达体系能够为氨基葡萄糖glcn的生产提供便利。
枯草芽孢杆菌已经被美国食品和药物管理局(fda)列为原则上认为安全的微生物(gras)。作为非致病的土壤微生物,其细胞壁组成成分简单,只含有肽聚糖和磷壁质,当分泌蛋白跨过细胞膜后,就被加工和直接释放到培养基中。但野生型的枯草芽孢杆菌bacillussubtilis168会在其对数生长末期大量表达和分泌各种蛋白酶,造成外源蛋白降解,有时甚至得不到目标蛋白,因此当生产胞外表达的外源蛋白时,一般选用枯草芽孢杆菌bacillussubtiliswb600作为表达宿主。课题组在前期的工作中,构建重组质粒p43nmkmut-yncm-dac,当以b.subtiliswb600为表达宿主时,摇瓶中胞外酶活达到1548.7u/ml,但是仍难以满足工业化利用酶法制备氨基葡萄糖glcn的最低酶活需求,氨基葡萄糖glcn在转化过程中的产量和转化率还较低,所以需要进一步提高氨基葡萄糖glcn的产量。
技术实现要素:
为解决几丁二糖脱乙酰酶dac催化效率有限,酶活不够高的问题,本发明提供了一种提高几丁二糖脱乙酰酶dac催化效率,进而提升胞外酶活的方法。
本发明的第一个目的是提供一种几丁二糖脱乙酰酶,其氨基酸序列如seqidno.2所示。
本发明的第二个目的是提供一种编码所述的几丁二糖脱乙酰酶的基因。
本发明的第三个目的是提供一种包括所述的基因的表达载体。
本发明的第四个目的是提供一种高效分泌表达所述的几丁二糖脱乙酰酶的枯草芽孢杆菌,所述的枯草芽孢杆菌以p43nmk为载体,融合表达了氨基酸序列如seqidno.2所示的几丁二糖脱乙酰酶与核苷酸序列如seqidno.3所示的yncm信号肽,并在几丁二糖脱乙酰酶基因的5’非翻译区引入seqidno.4所示的dna片段,引入位点位于启动子p43的+1转录起始位点后的8个碱基之后。
进一步地,所述的枯草芽孢杆菌还包括采用核苷酸序列如seqidno.5或seqidno.6所示的rbs序列调控几丁二糖脱乙酰酶的表达。
进一步地,所述的枯草芽孢杆菌的宿主为b.subtiliswb600。
本发明的第五个目的是提供所述的枯草芽孢杆菌在提高氨基葡萄糖产量中的应用,以所述的枯草芽孢杆菌发酵上清液作为催化剂,以n-乙酰氨基葡糖为底物,在35~45℃催化底物,得到氨基葡萄糖。
进一步地,所述的枯草芽孢杆菌发酵上清液是通过将枯草芽孢杆菌种子液按照3~5%接种量接种至发酵培养基中,在35~38℃培养50~70h,经10000~15000rpm离心1~3min得到。
进一步地,所述的发酵培养基成分包括:蛋白胨10~15g/l、酵母提取物20~30g/l、甘油3~5ml,kh2po42~2.5g/l和k2hpo412~13g/l。
进一步地,所述的种子液为od600为4.0~6.0的枯草芽孢杆菌。
进一步地,n-乙酰氨基葡萄糖的添加量为80~120g/l。
借由上述方案,本发明至少具有以下优点:
本发明通过对几丁二糖脱乙酰酶dac的分子结构及表达载体pp43mut-n1-yncm-dac的rbs序列进行改造,提高几丁二糖脱乙酰酶dac的催化速率及翻译表达速率,提高酶活,并构建了可以高产氨基葡萄糖glcn的枯草芽孢杆菌。与现有技术相比,几丁二糖脱乙酰酶胞外酶活可达3769.5u/ml。本转化方法条件温和,对环境污染小,而且底物选择性高,目标产物专一,反应速率快,产物得率高。此法有利于解决化学合成法中污染严重,步骤繁琐等问题,而且工艺简单,易于控制,方便推广应用。
上述说明仅是本发明技术方案的概述,为了能够更清楚了解本发明的技术手段,并可依照说明书的内容予以实施,以下以本发明的较佳实施例并配合详细说明如后。
附图说明
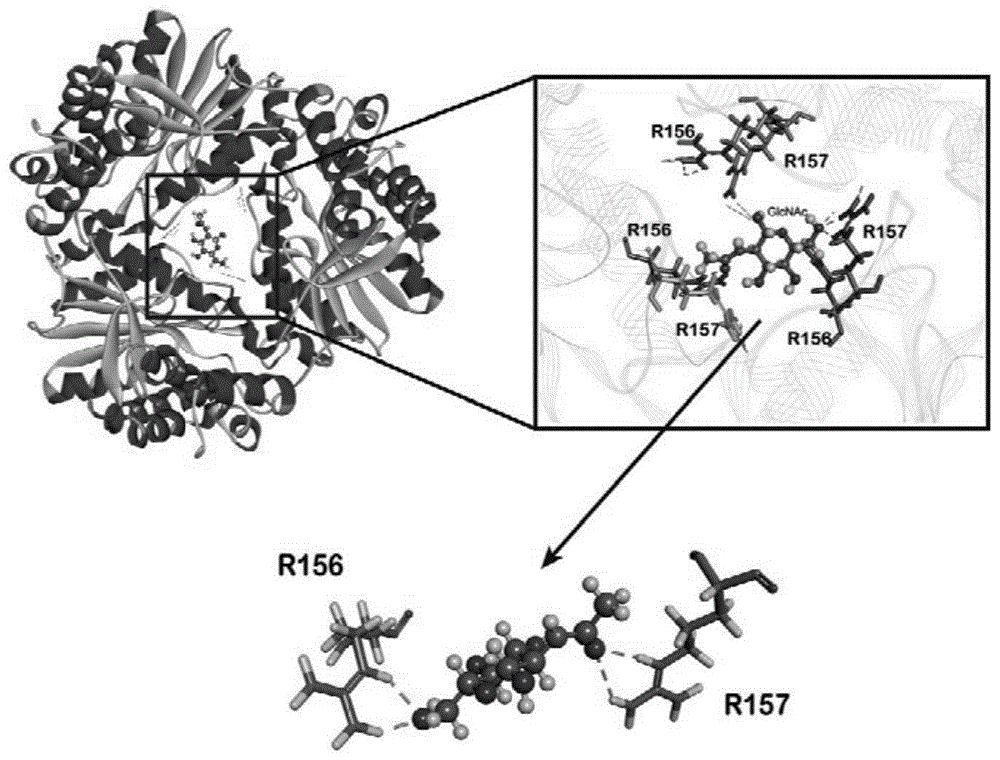
图1为底物分子glcnac与蛋白分子dac三维结构分子对接示意图;
图2为蛋白分子dac中与底物分子glcnac相互作用的关键位点;
图3为蛋白分子dac定点饱和突变体库的大肠杆菌escherichiacolidh5α转化菌落图;
图4为蛋白分子dac突变菌株摇瓶放大实验的胞外酶活;
图5为蛋白分子dac原始菌株及突变菌株的动力学检测参数;
图6为构建rbs优化库时的核酸电泳验证;
图7为rbs优化库的大肠杆菌escherichiacolidh5α转化菌落图;
图8为rbs优化筛选出的突变菌株摇瓶放大实验的胞外酶活。
具体实施方式
涉及的检测方法:
(1)几丁二糖脱乙酰酶酶活力定义
在40℃反应条件下,1h能转化1μmol底物n-乙酰氨基葡萄糖glcnac为1μmol产物氨基葡萄糖glcn所需的酶量,称为一个酶活力单位,即1u=1μmol/h。
(2)几丁二糖脱乙酰酶酶酶活力检测方法
邻苯二甲醛显色法
(3)检测试剂
底物n-乙酰氨基葡萄糖溶液(100g/l):5gn-乙酰氨基葡萄糖glcnac溶于pb1中,用pb1定容至50ml。
pb1磷酸钠缓冲液(200mmol/l,ph8.0):配制浓度为200mmol/l的nah2po4溶液和na2hpo4溶液。18mlnah2po4溶液与390mlna2hpo4溶液混匀。用ph测定仪测定ph值,确定ph8.0,室温储存。
pb2碳酸钠缓冲液(100mmol/l,ph10.5):配制浓度为100mmol/l的naco3溶液和nahco3溶液。18mlnah2po4溶液与390mlna2hpo4溶液混匀。用ph测定仪测定ph值,确定ph10.5,室温储存。
dtt二硫苏糖醇(2mol/l):称取308.5mg二硫苏糖醇于1mlddh2o中,混合均匀,密封低温保存。
10×opa检测试剂:500mg邻苯二甲醛opa加入100mlpb2中,混合均匀,密封低温避光保存。
1×opa检测试剂:3ml10×opa,300μl无水乙醇,15μldtt,加pb2定容到30ml,混匀后,密封低温避光保存。现用现配。
hcl终止剂(0.5mol/l):取1800μl盐酸于40mlddh2o中。
实施例1:分子对接及饱和突变位点的选择
利用软件discoverystudio的cdocker模块进行分子对接,根据discoverystudio软件的操作手册,将几丁二糖脱乙酰酶dac模型的三维蛋白结构文件和配体n-乙酰氨基葡萄糖glcnac的三维结构文件,进行分子对接,如图1所示。
当底物分子与蛋白分子成功对接后,在底物分子glcnac与蛋白分子dac结合位置周围,与glcnac存在相互作用力的氨基酸是蛋白分子dac中与底物分子glcnac相互作用的关键位点,如图2所示。
根据文献报道,影响几丁二糖脱乙酰酶催化效应的关键位点为152位组氨酸h152及120位酪氨酸y120。结合软件discoverystudio模拟的氨基酸饱和突变结果,160位苯丙氨酸f160是与底物分子相互作用的重要位点,选择r156、r157、h152、y120和f160五个位点进行定点饱和突变。
实施例2:定点饱和突变库的建立
根据几丁二糖脱乙酰酶dac的碱基序列,设计简并引物,如表1。以pp43mut-yncm-dac(构建方法参见申请号为cn201910193636.5)为模板,进行pcr扩增,及大肠杆菌escherichiacolidh5α转化(如图3),将全部大肠转化子制备混合菌液并提质粒后,转入表达宿主枯草芽孢杆菌bacillussubtiliswb600。
表1
实施例3:定点饱和突变库的高通量筛选与放大验证
(1)高通量筛选:
使用灭菌牙签在每个饱和突变点库中挑选100个左右单克隆到48孔板进行种子培养。每孔含有600μl添加卡那抗生素(10mg/ml)的液体lb培养基,(lb培养基成分如下:胰蛋白胨10g/l、酵母粉5g/l、nacl10g/l),于37℃孔板摇床培养12h得到种子液,之后在新的48孔板中,将200μl种子液转接至每孔含有600μl添加卡那抗生素(10mg/ml)的液体tb培养基进行发酵培养,(tb培养基成分如下:甘油4g/l、胰蛋白胨12g/l、酵母粉24g/l、磷酸氢二钾12.54g/l、磷酸二氢钾2.31g/l),于37℃,700rpm培养6h后结束发酵,以4200rpm,4℃的条件进行孔板离心30min,取离心后得到的上清液100μl加入每孔装有100μl50g/lglcnac溶液(溶剂为pb1缓冲液)的96孔板中,于40℃,900rpm震荡摇床上反应10min,然后每孔加入100μl终止剂(0.5mol/lhcl)终止反应。每孔加入300μlddh2o对反应液进行稀释,混合均匀后,4200rpm,离心5min,吸取上清液10μl加入到100μl1×opa检测试剂中,在330nm下检测吸光值。
将产量高于对照组的菌株按上述方法复筛两遍后进行锥形瓶发酵放大验证。
(2)放大验证:
使用灭菌牙签将高通量筛选得到的157-2、157-5、157-163株菌克隆子在50ml离心管中进行种子培养。每管含有5ml添加卡那抗生素(10mg/ml)的液体lb培养基,于37℃弹簧摇床培养12h得到种子液,在500ml锥形瓶中,将种子液以4%的接种量转接至96ml添加卡那抗生素(10mg/ml)的液体tb培养基进行发酵培养,通过邻苯二甲醛显色法进行粗酶液酶活检测,具体方式如下:
取发酵60h的发酵液1ml,以1200rpm,4℃,2min条件离心,上清液即为粗酶液,底物为100g/l的n-乙酰氨基葡萄糖glcnac溶液。将待测粗酶液与底物各100μl分装于1.5mlep管中,40℃预热5min。将预热后的100μl粗酶液加入100μl底物中,40℃震荡反应2min,精确计时,加入0.5mol/lhcl终止剂终止反应。以1200rpm,2min条件离心,取上清50μl于950μlpb1中,稀释20倍,混合均匀后取5μl加入100μl1×opa检测试剂中,震荡2min。利用酶标仪检测其在330nm处的吸光值。以失活后的酶液为反应空白对照。
glcn的标准曲线绘制,精确称取标准品(精确到0.0001g)配制0.5g/l、1g/l、2g/l、3g/l、4g/l、5g/lglcn溶液,取5μl加入100μlopa检测试剂中,震荡1min,30℃保温2min。利用酶标仪检测其在330nm处的吸光值,绘制标准曲线。
酶活力计算公式如下所示:
式中:
x:粗酶液酶活力(u/ml);
a:粗酶液体积0.1(ml);
b:底物溶液体积0.1(ml);
m:加入终止剂后样品稀释倍数1.5;
c:反应液glcn浓度(g/l);
d:空白液glcn浓度(g/l)。
t:反应时间2(min);
60:1h为60min;
215.6:标准样品氨基葡萄糖盐酸盐摩尔质量(g/mol);
0.1:底物溶液体积数(ml)
x:酶液稀释倍数20。
通过锥形瓶放大验证,157-2、157-5、157-163株突变菌株胞外酶活均有提升,如图4,发酵60h菌株157-5胞外酶活达到2042.8u/ml,提升31.9%;菌株157-2达1609.9u/ml,提升3.95%;菌株157-16达1573.4u/ml,提升1.59%。提质粒送测后发现菌株157-2为157位的精氨酸突变为色氨酸(r157w),菌株157-5为157位的精氨酸突变为苏氨酸(r157t),菌株157-16为157位的精氨酸突变为组氨酸(r157h),得到突变质粒p43nmk-r157t-dac、p43nmk-r157w-dac和p43nmk-r157h-dac。
实施例4:几丁二糖脱乙酰酶的动力学参数的测定
(1)将实施例3中筛到的r157t、r157w、r157h3株菌进行蛋白纯化
取60h发酵液50ml以8000rpm离心15min,取上清液于85℃条件下进行水浴加热,再以8000rpm离心15min,将上清液置于pb1缓冲液中进行过夜透析。将所得透析液利用0.45μm的滤膜进行过滤,过滤所得粗酶液通过阴离子交换柱(hitrapqhp柱),利用akta蛋白纯化仪进行纯化,具体纯化步骤如下:
a)利用5-10个柱体积的ddh2o清洗柱子,流速2ml/min。
b)利用5-10个柱体积的蛋白纯化a液平衡柱子,流速2ml/min。
c)将准备好的蛋白样品加入到蛋白纯化柱中,流速1ml/min。
d)待上完适量的蛋白样品后,利用蛋白纯化b液梯度洗脱,流速1ml/min。
e)将所获蛋白液置于pb1缓冲液进行过夜透析。
f)获得目标蛋白,4℃保存。
(2)在40℃反应温度下,以不同浓度的底物glcnac溶液(浓度范围:0.9-22.6mmol/l),进行动力学参数的km和vmax值的测定,根据下述公式,利用双倒数作图法进行作图,如图5,计算动力学参数,如表2。
式中:
v:反应速率(μm/s)
km:米氏常数(mm)
[s]:底物浓度(mm)
vmax:最大反应速率(μm/s)
表2
突变体r157t表现出最高的催化效率。突变体r157t的km值从原始dac的值6.75mm降低为5.15mm,vmax值由原来的4.94μm/s提高到了7.56μm/s。
实施例5:rbs优化库的建立
借助工具在线网站rbscalculator进行反向工程,预测实施例3中所述突变质粒p43nmk-r157t-dac上已有的mrna翻译强度,以提供的翻译速率作为参考,对数据区间上下游浮动进行正向工程设计,优化rbs翻译强度,以p43nmk-r157t-dac为模板,设计简并引物,如表3,进行pcr扩增,如图6,进行大肠杆菌escherichiacolidh5α转化,如图7,将全部大肠转化子制备混合菌液并提质粒后,转入表达宿主枯草芽孢杆菌bacillussubtiliswb600。
表3
实施例6:rbs优化库的高通量筛选与放大验证
参照实施例3中所述的方法,获得枯草芽孢杆菌重组菌株(p43nmk-r2-r157t-dac),胞外酶活达3769.5u/ml,较原始菌株提升了1.43倍,较实施例3中获得的菌株,胞外酶活提升了84.53%,如图8,其中r1~r5的核苷酸序列分别如seqidno.5~9所示。
以上仅是本发明的优选实施方式,并不用于限制本发明,应当指出,对于本技术领域的普通技术人员来说,在不脱离本发明技术原理的前提下,还可以做出若干改进和变型,这些改进和变型也应视为本发明的保护范围。
序列表
<110>山东润德生物科技有限公司,江南大学
<120>高效分泌表达几丁二糖脱乙酰酶的枯草芽孢杆菌及其应用
<160>21
<170>siposequencelisting1.0
<210>1
<211>272
<212>prt
<213>(人工序列)
<400>1
metvalvalasnmetphegluaspileaspthrphegluglualaphe
151015
asnlysleuleuarggluvalleuglupheaspleuglnasnprophe
202530
lysaspalalyslysvalleucysilegluprohisproaspaspcys
354045
valileglymetglyglythrilelyslysleuseraspmetglyval
505560
gluvaliletyrvalcysmetthraspglytyrmetglythrthrasp
65707580
gluserleuserglyhisgluleualaalaileargarglysgluglu
859095
glugluseralaargleuleuglyvallyslysiletyrtrpleuasn
100105110
tyrargaspthrgluleuprotyrserarggluvalarglysaspleu
115120125
thrlysileleuarglysgluglnproaspglyvalphealaproasp
130135140
protrpleuprotyrgluserhisproasphisargargthrglyphe
145150155160
leualailegluservalalapheserglnleuproasnpheserasn
165170175
thraspleuaspileglyleuasnprotyrasnserglyserpheile
180185190
alaleutyrtyrthrhislysproasntyrilevalaspilethrasp
195200205
leumetgluleulysleulysalaileargvalhisargserglnphe
210215220
proaspaspiletrpglulystrpglupropheleuargthrileala
225230235240
metphetyrglyglulysileglyvalargtyrglygluglyphearg
245250255
ilemetproglyleuphetyrhisilethrprophethraspleuile
260265270
<210>2
<211>272
<212>prt
<213>(人工序列)
<400>2
metvalvalasnmetphegluaspileaspthrphegluglualaphe
151015
asnlysleuleuarggluvalleuglupheaspleuglnasnprophe
202530
lysaspalalyslysvalleucysilegluprohisproaspaspcys
354045
valileglymetglyglythrilelyslysleuseraspmetglyval
505560
gluvaliletyrvalcysmetthraspglytyrmetglythrthrasp
65707580
gluserleuserglyhisgluleualaalaileargarglysgluglu
859095
glugluseralaargleuleuglyvallyslysiletyrtrpleuasn
100105110
tyrargaspthrgluleuprotyrserarggluvalarglysaspleu
115120125
thrlysileleuarglysgluglnproaspglyvalphealaproasp
130135140
protrpleuprotyrgluserhisproasphisargthrthrglyphe
145150155160
leualailegluservalalapheserglnleuproasnpheserasn
165170175
thraspleuaspileglyleuasnprotyrasnserglyserpheile
180185190
alaleutyrtyrthrhislysproasntyrilevalaspilethrasp
195200205
leumetgluleulysleulysalaileargvalhisargserglnphe
210215220
proaspaspiletrpglulystrpglupropheleuargthrileala
225230235240
metphetyrglyglulysileglyvalargtyrglygluglyphearg
245250255
ilemetproglyleuphetyrhisilethrprophethraspleuile
260265270
<210>3
<211>126
<212>dna
<213>(人工序列)
<400>3
atggcgaaaccactatcaaaagggggaattttggtgaaaaaagtattgattgcaggtgca60
gtaggaacagcagttcttttcggaaccctttcatcaggtataccaggtttacccgcggca120
gacgct126
<210>4
<211>73
<212>dna
<213>(人工序列)
<400>4
ggtaccattataggtaagagaggaatgtacacatggtcgtcaacatgttcgaggacatcg60
acacgttcgagga73
<210>5
<211>19
<212>dna
<213>(人工序列)
<400>5
ggaaaggaggaatagagac19
<210>6
<211>19
<212>dna
<213>(人工序列)
<400>6
ggaagggaggaatagagac19
<210>7
<211>19
<212>dna
<213>(人工序列)
<400>7
ggaaaggaggaattgagac19
<210>8
<211>19
<212>dna
<213>(人工序列)
<400>8
ggaagggaggcatagagac19
<210>9
<211>19
<212>dna
<213>(人工序列)
<400>9
ggaaaggaggaacagagac19
<210>10
<211>33
<212>dna
<213>(人工序列)
<400>10
tcacatccggatcatnnkcgcacaggctttctg33
<210>11
<211>33
<212>dna
<213>(人工序列)
<400>11
cagaaagcctgtgcgmnnatgatccggatgtga33
<210>12
<211>32
<212>dna
<213>(人工序列)
<400>12
atccggatcatcgtnnkaccggttttttagcc32
<210>13
<211>32
<212>dna
<213>(人工序列)
<400>13
ggctaaaaaaccggtmnnacgatgatccggat32
<210>14
<211>32
<212>dna
<213>(人工序列)
<400>14
catagacgcacaggcnnkctggcgattgaatc32
<210>15
<211>32
<212>dna
<213>(人工序列)
<400>15
gattcaatcgccagmnngcctgtgcgtctatg32
<210>16
<211>34
<212>dna
<213>(人工序列)
<400>16
gaattagcagcaatcnnkcgcaaagaagaagaag34
<210>17
<211>34
<212>dna
<213>(人工序列)
<400>17
cttcttcttctttgcgmnngattgctgctaattc34
<210>18
<211>33
<212>dna
<213>(人工序列)
<400>18
cttccgtacgaatcannkccggatcatagacgc33
<210>19
<211>33
<212>dna
<213>(人工序列)
<400>19
gcgtctatgatccggmnntgattcgtacggaag33
<210>20
<211>49
<212>dna
<213>(人工序列)
<400>20
aabsgaggaatwrabacatgaaaaaaatcacaacaaacgaacaatttaa49
<210>21
<211>45
<212>dna
<213>(人工序列)
<400>21
catgtvtywattcctcsvttbcctataatggtacctcctcgaacg45
- 还没有人留言评论。精彩留言会获得点赞!