创制水稻大长粒型新种质的方法与流程
1.本发明涉及植物育种技术领域,具体涉及创制水稻大长粒型新种质的方法。
背景技术:
2.水稻是中国主要的粮食作物,全国60%以上的人口以稻米为主食。水稻籽粒形状是影响产量的重要性状之一,粒型增大与水稻增产直接相关。同时籽粒形状也影响稻米的外观品质,随着人民生活的日益改善,对米质要求越来越高。在目前中国的餐饮市场上,长粒米广受青睐。部分水稻品种的其它性状很好,却因为粒型不够长而限制了其市场份额。
3.基因编辑技术是近年来分子育种领域的热点技术,它能够实现亲本目标性状的快速精准改良,且不涉及转基因安全问题。该技术在育种中的应用大多数是通过对目标基因进行碱基的添加或删除,从而使基因功能失活,获得相应的目标性状。利用基因编辑技术修饰水稻基因、创制增大增长粒型的水稻种质对于水稻的增产具有重要意义。
技术实现要素:
4.本发明的目的是提供一种创制水稻大长粒型新种质的方法。
5.本发明首先发现了osd-h基因和loc_os01g10460基因的新功能,将osd-h基因和loc_os01g10460基因同时失活,能够使得水稻的籽粒增大、增长,提高水稻的千粒重。水稻osd-h基因编码与ga信号传导途径有关的蛋白,已有研究表明的osd-h影响水稻的株高和籽粒大小,osd-h基因63bp缺失在粳稻中导致了矮杆和小粒的表型。但未有osd-h基因与水稻籽粒增大增长相关的研究。本发明发现的osd-h基因和loc_os01g10460基因联合失活的新功能可用于水稻大长粒型新种质的创制。
6.具体地,本发明提供以下技术方案:
7.第一方面,本发明提供水稻osd-h和loc_os01g10460基因、其编码蛋白或水稻osd-h和loc_os01g10460基因的抑制因子在调控植物籽粒大小或千粒重中的应用。
8.优选地,所述调控植物籽粒大小为使得水稻籽粒增大、增长,所述调控千粒重为提高千粒重。
9.进一步优选地,通过使得植物osd-h和loc_os01g10460基因丧失生物学功能,使得植物籽粒增大、增长,千粒重提高。
10.第二方面,本发明提供水稻osd-h和loc_os01g10460基因、其编码蛋白或水稻osd-h和loc_os01g10460基因的抑制因子在植物育种中的应用。
11.优选地,所述水稻育种为植物籽粒性状或千粒重性状的育种。
12.进一步优选地,所述植物育种为大长粒植物或增加千粒重的育种。
13.第三方面,本发明提供水稻osd-h和loc_os01g10460基因、其编码蛋白或水稻osd-h和loc_os01g10460基因的抑制因子在植物种质资源改良中的应用。
14.优选地,所述植物种质资源改良为植物籽粒性状或千粒重性状的种质资源改良。
15.进一步优选地,所述植物种质资源改良为大长粒型植物或增加千粒重的种质资源
改良。
16.本发明中,所述osd-h基因的编码蛋白的氨基酸序列如seq id no:1所示,或者为与其具有至少70%的序列同一性的保留其功能的活性变体的氨基酸序列;loc_os01g10460基因的编码蛋白的氨基酸序列如seq id no:2所示,或者为与其具有至少70%的序列同一性的保留其功能的活性变体的氨基酸序列。
17.优选地,所述序列同一性为至少80%、至少85%、至少90%、至少95%、至少98%、至少99%。
18.本发明中,osd-h基因的核苷酸序列如seq id no:3所示,或者为与其具有至少90%的序列同一性的保留其功能的活性变体的核苷酸序列;loc_os01g10460基因的核苷酸序列如seq id no:4所示,或者为与其具有至少90%的序列同一性的保留其功能的活性变体的核苷酸序列。
19.优选地,所述序列同一性为至少80%、至少85%、至少90%、至少95%、至少98%、至少99%。
20.本发明中,osd-h基因、loc_os01g10460基因的抑制因子为能够破坏所述基因的生物学功能的核酸分子或蛋白分子。
21.本发明中,所述植物优选为水稻,更优选为粳稻或籼稻。
22.第四方面,本发明提供创制水稻大长粒型新种质的方法,该方法包括:使水稻osd-h和loc_os01g10460基因的生物学功能丧失。
23.优选地,osd-h基因的编码蛋白的氨基酸序列如seq id no:1所示,或者为与其具有至少90%的序列同一性的保留其功能的活性变体的氨基酸序列;loc_os01g10460基因的编码蛋白的氨基酸序列如seq id no:2所示,或者为与其具有至少90%的序列同一性的保留其功能的活性变体的氨基酸序列。
24.上述方法中,使水稻osd-h和loc_os01g10460基因的生物学功能丧失可通过本领域常用的技术手段实现,包括但不限于基因编辑等。
25.本发明优选通过基因编辑方法使得水稻osd-h和loc_os01g10460基因的生物学功能丧失;所述基因编辑方法为通过选自以下的序列特异性核酸酶中的一种或多种进行:crispr/cas9、crispr/cpf1、crispr/cas12a、talen、大范围核酸酶和zfn。
26.优选地,所述基因编辑利用crispr/cas9基因编辑载体进行。
27.由于osd-h基因和loc_os01g10460基因有部分区域为反向重叠区域,本发明以两者的重叠区域为靶标区域,通过一次crispr/cas9基因编辑同时实现osd-h基因和loc_os01g10460基因的失活。
28.优选地,所述crispr/cas9基因编辑载体包含第一sgrna和第二sgrna,所述第一sgrna和第二sgrna分别靶向seq id no:5-6所示的核苷酸序列。
29.上述crispr/cas9基因编辑可导致osd-h基因发生缺失、插入、取代或以上的组合,导致loc_os01g10460位点基因区段发生缺失、插入、取代或以上的组合。
30.在一些具体的实施方案中,所述基因编辑导致osd-h基因、loc_os01g10460基因缺失、添加或取代3的倍数数量的碱基,例如3bp、6bp、9bp。
31.第五方面,本发明提供一种大长粒型水稻,该水稻的osd-h和loc_os01g10460基因的生物学功能丧失。
32.优选地,所述大长粒型水稻的osd-h和loc_os01g10460基因的重叠区域发生缺失、插入、取代或以上的组合。
33.进一步优选地,所述大长粒型水稻的osd-h和loc_os01g10460基因的重叠区域缺失、添加或取代3的倍数数量的碱基,例如3bp、6bp、9bp碱基发生缺失、插入、取代。
34.本发明提供一种大长粒水稻,该水稻在osd-h和loc_os01g10460基因的重叠区域缺失65bp。
35.第六方面,本发明提供利用所述创制水稻大长粒型新种质的方法创制得到的水稻在水稻育种或种质资源改良中的应用。
36.第七方面,本发明提供由所述创制水稻大长粒型新种质的方法创制得到的水稻种子制备得到的产品,所述产品为选自食品、饲料或工业原材料中的一种或多种。
37.第八方面,本发明提供靶向水稻osd-h和loc_os01g10460基因的sgrna,其包括第一sgrna和第二sgrna,所述第一sgrna包含seq id no:7所示的核苷酸序列,所述第二sgrna包含seq id no:8所示的核苷酸序列。
38.第九方面,本发明提供靶向水稻osd-h和loc_os01g10460基因的crispr/cas9基因编辑载体,所述载体包含第一表达盒和第二表达盒,所述第一表达盒表达cas9,所述第二表达盒表达sgrna,所述sgrna包含seq id no:7和/或seq id no:8所示的核苷酸序列,或与其具有至少70%的序列同一性的保留其功能的活性变体序列。
39.优选地,所述cas9包含seq id no:9所示的核苷酸序列或与其具有至少70%序列同一性并编码相同功能的蛋白质的活性变体序列。
40.作为本发明的优选方案,所述第一表达盒以甘蔗ubi4启动子驱动cas9的表达,所述第二表达盒以水稻u3启动子驱动sgrna的表达。
41.第十方面,本发明提供所述sgrna或所述crispr/cas9基因编辑载体在创制水稻大长粒型新种质中的应用。
42.本发明的有益效果在于:本发明发现同时失活水稻中osd-h和loc_os01g10460基因,能够使得水稻的籽粒显著增大、增长,千粒重显著提高。水稻中osd-h和loc_os01g10460基因的失活可用于大长粒型水稻新种质的创制。
43.本发明提供一种创制大长粒型水稻新种质的方法,该方法利用基因编辑技术创制osd-h基因及loc_os01g10460位点基因突变体以改良水稻粒型,利用基因编辑技术的定向打靶功能来实现基因突变,并筛选出特定突变类型。该方法简单易行、成本低、并且效率高。利用该方法可获得粒型明显增大增长的优质水稻品种。本发明利用该方法对osd-h基因及loc_os01g10460位点基因进行定点编辑,成功获得了粒型显著增长增大的籼稻品种。该方法为利用基因编辑技术快速改良水稻品种的粒型提供一种有效的策略,特别是对于一些其它性状良好,粒型尚需改良的品种,提供了一种便捷高效的改良策略。
附图说明
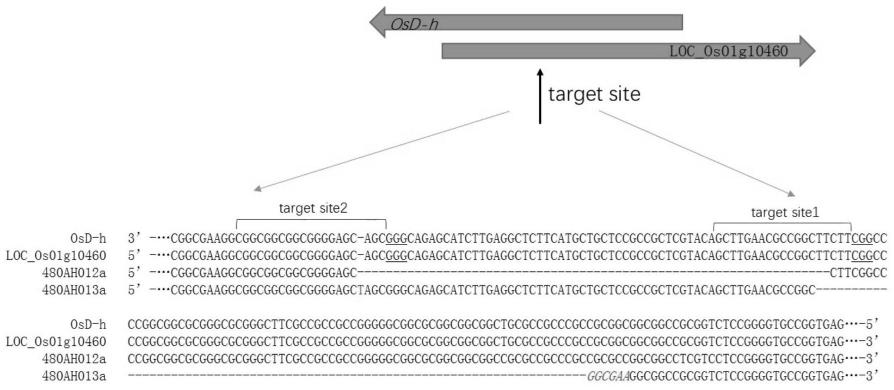
44.图1为本发明实施例1中osd-h基因、loc_os01g10460基因的序列和实施例4中编辑株系osd-h基因、loc_os01g10460基因的序列及2个基因的序列重叠关系,其中osd-h、loc_os01g10460:野生型;480ah012a和480ah013a:编辑株系;下划线表示pam序列;虚线表示缺失序列;方框表示外显子;斜体字表示插入序列。
45.图2为本发明实施例1中基因编辑载体的结构示意图。
46.图3为本发明实施例4中野生型株系和编辑株系osd-h基因编码的氨基酸序列,osd-h:野生型;480ah012a和480ah013a:编辑株系;虚线表示缺失的氨基酸,斜体字红字表示无法与野生型匹配的氨基酸。
47.图4为本发明实施例4中野生型株系和编辑株系loc_os01g10460基因编码的氨基酸序列,loc_os01g10460:野生型;480ah012a和480ah013a:编辑株系;虚线表示缺失的氨基酸,斜体字表示无法与野生型匹配的氨基酸。
48.图5为本发明实施例5中osd-h基因、loc_os01g10460基因编辑株系与野生型株系的籽粒比较,其中wt:野生型;480ah012a和480ah013a:编辑株系,a为谷子的照片,b为米粒的照片。
具体实施方式
49.关于本文使用的术语解释如下:
50.本文使用的术语“基因编辑(gene editing)”或“基因组编辑(genome edting)”,是一种新兴的比较精确的能对生物体基因组特定目标基因进行修饰的一种基因工程技术。基因编辑指能够对目标基因进行定点“编辑”,实现对特定dna片段的修饰。基因编辑依赖于经过基因工程改造的核酸酶,也称“分子剪刀”,在基因组中特定位置产生位点特异性双链断裂(dsb),诱导生物体通过非同源末端连接(nhej)或同源重组(hr)来修复dsb,由于这个修复过程容易出错,从而导致靶向突变。
51.本文使用的术语“crispr/cas9”是指使用rna引导链靶向内切核酸酶切割位点的内切核酸酶。参见,jinek等人,science 337:816-821(2013);cong等人,science(2013年1月3日);和mali等人,science(2013年1月3日)。目前发现的crispr/cas9系统有三种不同类型即i型、ii型和iii型,它们存在于大约40%已测序的真细菌和90%已测序的古细菌中。其中ii型的组成较为简单,以cas9蛋白以及向导rna(grna)为核心组成,也是目前研究得最深入的类型。当细菌抵御噬菌体等外源dna入侵时,在前导区的调控下,crispr被转录为长的rna前体(pre-crrna),然后加工成一系列短的含有保守重复序列和间隔区的成熟crrna,最终识别并结合到与其互补的外源dna序列上发挥剪切作用。crispr/cas9的剪切位点位于crrna互补序列下游临近的pam区(protospacer adjacent motif)的5
’‑
gg-n18-ngg-3’特征区域中的ngg位点,而这种特征的序列在每128bp的随机dna序列中就重复出现一次。
52.本文使用的术语“crispr/cpf1”是一类新型的crispr-cas系统。目前得到广泛应用的crispr/cas9系统经常会发生脱靶效应。作为crispr/cas9系统的一种潜在的对手,crispr-cpf1系统作为基因编辑的新工具,进一步扩大了基因编辑靶位点的选择范围,同时几乎没有脱靶效应。cpf1是一种新型crispr效应蛋白,具有许多与cas9不同的特性,有利于克服crispr/cas9应用中的一些限制。
53.本文使用的术语“crispr/cas12a”也是一类新型的crispr-cas系统。相较于cas9蛋白来说,cas12a蛋白更具准确度,同时也更安全。crispr/cas9工作时,cas9蛋白识别pam序列(rna编写的遗传密码),通过grna解开部分双螺旋,在这个过程中,cas9蛋白一旦找到匹配较好的序列时,便会紧密贴合在该段dna上,而在这个过程中,也许会出现一些错配的现象,但这种结合是不可逆的。而在这一方面,cas12a就显得机智许多,它在寻找“靶标”时,
会对沿途的dna序列进行单碱基的识别,如若发现有匹配不好的碱基,便会离开重新寻找,在寻找到pam序列时,cas12a蛋白会与pam序列形成一种半封闭的r-环(r-loop),当识别到正确的序列时,才会完全结合成封闭的r-环,所以这种结合是可逆的,这也体现出了它更安全的一面。
54.本文使用的术语“转录激活子样效应器核苷酶”或“tal效应器核苷酶”或“talen”是指一类通过使tal效应器dna结合结构域与dna切割结构域融合而产生的人工限制内切核酸酶。
55.本文使用的术语“锌指核酸酶(zfn)”由一个dna识别域以及一个dna剪切域组成。dna识别域为3~4个zf串联结构,每个zf约含30个氨基酸,被1个锌离子所固定,可识别并结合1个特异的三联体碱基,dna剪切域由非特异性核酸内切酶fok i羧基端的96个氨基酸残基组成。每个fok i单体与1个zfp相连构成1个zfn,并识别特定的位点,当2个识别位点相距恰当的距离时(6~8bp),2个单体zfn相互作用产生酶切功能,形成双链断裂,从而介导dna定点剪切。
56.本文使用的术语“大范围核酸酶(meganuclease)”是指能够识别14-40碱基长度的核酸序列的归巢核酸内切酶。一些大范围核酸酶可以容忍小的归巢位点序列差异,大的识别区域仍然能够保证这些酶的高度特异性,而这反过来又可保持低水平的基因组内非特异性裂解和较低的毒性。大范围核酸酶由自我剪接rna内含子或自我剪接蛋白内含子序列的移动序列内的开放阅读框架编码。
57.本文使用的术语“同一性”,在两个或更多个核酸或多肽的情况下,指相同或具有相同核苷酸或氨基酸的指定百分比(即在指定区域内,当在比较窗或指定区域内比较和比对最大一致性时,约60%的同一性,优选65%、70%、75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或更高的同一性)的两条或更多条序列或亚序列,如利用blast或blast 2.0序列比较算法以默认参数或通过手工比对和可视检查(参阅如ncbi网站等)所测量的。
58.关于本发明的具体实施方式说明如下:
59.本发明提出了一种以基因编辑技术创制osd-h基因失活或loc_os01g10460位点失活或这2个基因位点同时失活来改良水稻粒型的方法,其适应于所有含有功能型osd-h基因或loc_os01g10460位点基因的水稻品种中,包括以下步骤:
60.(1)拟改良水稻品种osd-h基因或loc_os01g10460位点基因的序列分析,在选择osd-h基因或loc_os01g10460位点或2个基因重叠位点合适位点设计靶标;
61.(2)构建编辑载体,将拟改良水稻品种的愈伤组织作为遗传转化的受体材料,用农杆菌介导法将编辑载体导入愈伤组织细胞,并再生成水稻株系;
62.(3)对osd-h基因和loc_os01g10460位点基因编辑株系的基因型检测和分析,选取目标基因编辑株系进行性状分析,考察粒型情况;
63.(4)经过传代分离、鉴定筛选,得到编辑纯合且非转基因的大粒长粒表型水稻种质。
64.本发明的创制水稻大长粒型新种质的方法,具体包括以下步骤:
65.(1)水稻中osd-h基因和loc_os01g10460位点基因的检测;
66.(2)水稻中osd-h基因和loc_os01g10460位点基因编辑载体的构建;
67.(3)将籼稻的愈伤组织作为遗传转化的受体材料,用农杆菌介导法将编辑载体导入,鉴定编辑株系中osd-h基因和loc_os01g10460位点基因的编辑情况,获得不同编辑类型的osd-h基因和loc_os01g10460位点等位基因。经过传代分离、鉴定筛选,得到编辑纯合且非转基因的株系,最终获得粒型增大增长的改良品系。
68.本发明具体涉及如下技术方案。
69.在第一方面,本发明提供水稻osd-h和loc_os01g10460基因、其编码蛋白或水稻osd-h和loc_os01g10460基因的抑制因子在调控植物籽粒大小或千粒重中的应用。
70.在第二方面,本发明提供水稻osd-h和loc_os01g10460基因、其编码蛋白或水稻osd-h和loc_os01g10460基因的抑制因子在植物育种中的应用。
71.在第三方面,本发明提供水稻osd-h和loc os01g10460基因、其编码蛋白或水稻osd-h和loc_os01g10460基因的抑制因子在植物种质资源改良中的应用。
72.在第四方面,本发明提供了创制水稻大长粒型新种质的方法,其包括修饰水稻中的osd-h基因和loc_os01g10460位点,使其生物学功能丧失。
73.在一些实施方案中,所述修饰通过基因编辑进行。
74.在一些实施方案中,所述osd-h基因包含以下序列或由以下序列组成:seq id no:3所示的核苷酸序列或与其具有至少约70%,75%,80%,85%,86%,87%,88%,89%,90%,91%,92%,93%,94%,95%,96%,97%,98%,99%或更高的同一性并编码相同功能的蛋白质的活性变体序列,或者编码seq id no:1所示的氨基酸序列或与其具有至少约70%,75%,80%,85%,86%,87%,88%,89%,90%,91%,92%,93%,94%,95%,96%,97%,98%,99%或更高的序列同一性的保留其功能的活性变体序列的核苷酸序列。
75.在一些实施方案中,所述loc_os01g10460位点基因包含以下序列或由以下序列组成:seq id no:4所示的核苷酸序列或与其具有至少约70%,75%,80%,85%,86%,87%,88%,89%,90%,91%,92%,93%,94%,95%,96%,97%,98%,99%或更高的同一性并编码相同功能的蛋白质的活性变体序列,或者编码seq id no:2所示的氨基酸序列或与其具有至少约70%,75%,80%,85%,86%,87%,88%,89%,90%,91%,92%,93%,94%,95%,96%,97%,98%,99%或更高的序列同一性的保留其功能的活性变体序列的核苷酸序列。
76.本文所用的术语“活性变体序列”指基本上相似的序列。对于核苷酸序列,活性变体序列包括由于遗传密码子简并性而编码相同功能的蛋白质的那些序列。诸如天然存在的等位基因变体可以使用公知的分子生物学技术例如聚合酶链式反应(pcr)和杂交技术来鉴定。例如,在本发明中,与籼稻x12的osd-h基因具有至少约70%,75%,80%,85%,86%,87%,88%,89%,90%,91%,92%,93%,94%,95%,96%,97%,98%,99%或更高的同一性并且也编码功能性osd-h蛋白的来自其他水稻品种的基因,包括在本发明定义的“活性变体序列”中。同一性的确定是通过本文描述的序列比对程序,所述程序使用默认参数。核苷酸的活性变体序列与该核苷酸序列的差异可以少至1-15个核苷酸、少至1-10个(例如6-10个),少至5个,少至4,3,2或甚至1个核苷酸。
77.本文所用的术语“活性变体序列”指基本上相似的序列。对于核苷酸序列,活性变体序列包括由于遗传密码子简并性而编码相同功能的蛋白质的那些序列。诸如天然存在的等位基因变体可以使用公知的分子生物学技术例如聚合酶链式反应(pcr)和杂交技术来鉴
定。例如,在本发明中,与籼稻x12的loc_os01g10460基因具有至少约70%,75%,80%,85%,86%,87%,88%,89%,90%,91%,92%,93%,94%,95%,96%,97%,98%,99%或更高的同一性并且也编码功能性loc_os01g10460蛋白的来自其他水稻品种的基因,包括在本发明定义的“活性变体序列”中。同一性的确定是通过本文描述的序列比对程序,所述程序使用默认参数。核苷酸的活性变体序列与该核苷酸序列的差异可以少至1-15个核苷酸、少至1-10个(例如6-10个),少至5个,少至4,3,2或甚至1个核苷酸。
78.对于蛋白质序列,术语“活性变体序列”包括衍生自天然蛋白的多肽,所述衍生是通过缺失(所谓的截短)天然蛋白n末端和/或c末端的一个或多个氨基酸或将一个或多个氨基酸添加至所述天然蛋白的n末端和/或c末端;在天然蛋白的一个或多个位点缺失或添加一个或多个氨基酸;或者在天然蛋白的一个或多个位点取代一个或多个氨基酸。因而,就蛋白质而言,术语“活性变体序列”包括天然蛋白的生物活性片段,其包含保留天然蛋白生物学活性,例如具有osd-h蛋白功能的足够数目的连续氨基酸残基。这样的功能相对天然蛋白可以是不同的或者是改良的,或者可以是不变的,只要保留了osd-h蛋白功能。同一性的确定是通过本文描述的序列比对程序,所述程序使用默认参数。蛋白的活性变体序列与该蛋白的差异可以少至1-15个氨基酸残基、少至1-10个(例如6-10个),少至5个,少至4,3,2或甚至1个氨基酸残基。
79.对于蛋白质序列,术语“活性变体序列”包括衍生自天然蛋白的多肽,所述衍生是通过缺失(所谓的截短)天然蛋白n末端和/或c末端的一个或多个氨基酸或将一个或多个氨基酸添加至所述天然蛋白的n末端和/或c末端;在天然蛋白的一个或多个位点缺失或添加一个或多个氨基酸;或者在天然蛋白的一个或多个位点取代一个或多个氨基酸。因而,就蛋白质而言,术语“活性变体序列”包括天然蛋白的生物活性片段,其包含保留天然蛋白生物学活性,例如具有loc_os01g10460蛋白功能的足够数目的连续氨基酸残基。这样的功能相对天然蛋白可以是不同的或者是改良的,或者可以是不变的,只要保留了loc_os01g10460蛋白功能。同一性的确定是通过本文描述的序列比对程序,所述程序使用默认参数。蛋白的活性变体序列与该蛋白的差异可以少至1-15个氨基酸残基、少至1-10个(例如6-10个),少至5个,少至4,3,2或甚至1个氨基酸残基。
80.在优选的实施方案中,所述水稻为籼稻或粳稻。
81.在一些实施方案中,所述基因编辑通过选自以下的序列特异性核酸酶中的一种或多种进行:crispr/cas9、crispr/cpf1、crispr/cas12a、talen、大范围核酸酶和zfn。优选地,所述基因编辑通过crispr/cas9进行。
82.在一些实施方案中,所述crispr/cas9包含cas9,其中所述cas9包含seq id no:9所示的核苷酸序列或与其具有至少约70%,75%,80%,85%,86%,87%,88%,89%,90%,91%,92%,93%,94%,95%,96%,97%,98%,99%或更高的序列同一性并编码相同功能的蛋白质的活性变体序列。
83.在一些实施方案中,所述crispr/cas9包含导向rna(sgrna),其中所述sgrna靶向osd-h基因。
84.在一些实施方案中,所述crispr/cas9包含导向rna(sgrna),其中所述sgrna靶向loc_os01g10460基因。
85.在一些具体的实施方案中,所述sgrna靶向seq id no:5或seq id no:6所示的核
苷酸序列。
86.在一些具体的实施方案中,所述sgrna包含seq id no:7或seq id no:8所示的核苷酸序列或与其具有至少约70%,75%,80%,85%,86%,87%,88%,89%,90%,91%,92%,93%,94%,95%,96%,97%,98%,99%或更高的序列同一性的保留其功能的活性变体序列。
87.在一些实施方案中,所述修饰导致osd-h基因发生缺失、插入、取代或以上的组合。
88.在一些实施方案中,所述修饰导致loc_os01g10460基因发生缺失、插入、取代或以上的组合。
89.在一些具体的实施方案中,所述修饰导致osd-h基因功能丧失。
90.在一些具体的实施方案中,所述修饰导致loc_os01g10460基因功能丧失。
91.在第五方面,本发明提供了通过第四方面所述的方法创制的水稻用于育种的用途。
92.在第六方面,本发明提供了通过第四方面所述的方法创制的水稻用于改良种质资源的用途。
93.在第七方面,本发明提供了通过第四方面所述的方法获得的水稻的种子制成的产品,其选自食品、饲料或工业原材料中的一种或多种。
94.在第八方面,本发明提供了能够靶向水稻osd-h基因及loc_os01g10460基因的sgrna,其包含seq id no:7或seq id no:8所示的核苷酸序列或由seq id no:7或seq id no:8所示的核苷酸序列组成。
95.在第九方面,本发明提供了能够靶向水稻osd-h基因及loc_os01g10460的crispr/cas9编辑载体,其包含表达cas9的第一表达盒和表达sgrna的第二表达盒,其中所述cas9包含seq id no:9所示的核苷酸序列或与其具有至少约70%,75%,80%,85%,86%,87%,88%,89%,90%,91%,92%,93%,94%,95%,96%,97%,98%,99%或更高的序列同一性并编码相同功能的蛋白质的活性变体序列,并且所述sgrna包含seq id no:7或seq id no:8所示的核苷酸序列或与其具有至少约70%,75%,80%,85%,86%,87%,88%,89%,90%,91%,92%,93%,94%,95%,96%,97%,98%,99%或更高的序列同一性的保留其功能的活性变体序列。
96.在第十方面,本发明提供了创制水稻大长粒型新种质的方法,其包括通过第九方面所述的crispr/cas9编辑载体导入含有osd-h基因及loc_os01g10460的水稻中。
97.在一些实施方案中,所述osd-h基因包含以下序列或由以下序列组成:seq id no:3所示的核苷酸序列或与其具有至少90%、95%、99%或更高的序列同一性并编码相同功能的蛋白质的活性变体序列,或者编码seq id no:1所示的氨基酸序列或与其具有至少90%、95%、99%或更高的序列同一性的保留其功能的活性变体序列的核苷酸序列。
98.在一些实施方案中,所述loc_os01g10460基因包含以下序列或由以下序列组成:seq id no:4所示的核苷酸序列或与其具有至少90%、95%、99%或更高的序列同一性并编码相同功能的蛋白质的活性变体序列,或者编码seq id no:2所示的氨基酸序列或与其具有至少90%、95%、99%或更高的序列同一性的保留其功能的活性变体序列的核苷酸序列。
99.在优选的实施方案中,所述水稻为一些其它性状良好,粒型尚需改良的品种,例如籼稻或粳稻。在优选的实施方案中,所述水稻为籼稻x12。
100.本文中,词语“包括”、“包含”和“含有”意指“包括但不限于”,且并非意图排除其他部分、添加物、组分、或步骤。
101.应该理解,在本发明的特定方面、实施方案或实施例中描述的特征、特性、组分或步骤,可适用于本文所描述的任何其他的方面、实施方案或实施例,除非与之矛盾。
102.以下实施例用于说明本发明,但不用来限制本发明的范围。在不背离本发明精神和实质的情况下,对本发明方法、步骤或条件所作的修改或替换,均属于本发明的范围。
103.若未特别指明,实施例中所用的化学试剂均为常规市售试剂,实施例中所用的技术手段为本领域技术人员所熟知的常规手段。
104.以下实施例中使用的水稻品种为籼稻x12,由中国种子集团有限公司提供。
105.实施例1crispr/cas9编缉载体的构建
106.1、水稻osd-h基因及loc_os01g10460基因的序列分析及基因编辑靶点设计
107.水稻osd-h基因的序列如seq id no:3所示,水稻loc_os01g10460基因的序列如seq id no:4所示(2个基因重叠区域见图1)。序列分析显示,osd-h基因含有1个外显子,loc_os01g10460基因含有1个外显子。本实施例所设的靶序列在籼稻x12材料中loc_os01g10460基因第253-272位碱基(agcttgaacgccggcttctt(seq id no:5))和第188-207位碱基(cggcggcggcggggagcagc(seq id no:6)),如图1所示。
108.2、crispr/cas9编缉载体的构建
109.本实施例采用的基因编辑技术为第三代基因编辑技术crispr/cas9,所使用的载体根据liu等人(hao liu,yuduan ding,yanqing zhou,wenqi jin,kabin xie*,ling-ling chen*.crispr-p 2.0:an improved crispr/cas9 tool for genome editing in plants.mol plant,2017,10(3):530-532)的方法设计,利用crispr-p2.0(http://crispr.hzau.edu.cn/crispr2/)在线工具设计了导向rna(sgrna)(sgrna的序列seq id no:7或seq id no:8,sgrna是由引导rna(grna)和骨架rna(grna scaffold)核苷酸序列连接起来的一个整体分子。该植物表达载体包含甘蔗ubi4启动子(即甘蔗泛素4启动子)驱动的cas9(cas9的序列seq id no:9)表达盒,以及水稻u3启动子驱动的sgrna表达盒,编辑载体同时包含两个sgrna(seq id no:7及seq id no:8)。构建的crispr/cas9编缉载体的图谱见图2。
110.实施例2籼稻x12的遗传转化
111.将实施例1构建的编辑载体转入农杆菌菌株eha105(华中农业大学馈赠)中并进行测序鉴定。由籼稻x12种子诱导产生胚性愈伤组织,与含有上述编缉载体的农杆菌eha105共同孵育,经过筛选再生等一系列步骤完成遗传转化。农杆菌侵染水稻愈伤组织及筛选、分化流程参照nishimura等的文献报道(a protocol for agrobacterium-mediated transformation in rice.nature protocols,2006,6:2 796-2 802)。具体流程包括,水稻愈伤组织与农杆菌共培养3天后,将愈伤组织于250mg/lcarbenicillin的ddh2o中浸泡30min,去除多余的农杆菌后置于含250mg/l carbenicillin的筛选培养基上筛选3轮,然后将抗性愈伤转移至再生培养基上培养,待分化出2~3cm的小苗后,将其转移至生根培养基中继续培养2周。
112.实施例3t0代osd-h基因及loc_os01g10460基因编辑水稻的筛选和鉴定
113.将实施例2中的再生植株送至温室种植,取再生的t0代小苗叶片,通过ctab法提取
植物基因组dna作为模板。dna样品用荧光定量pcr方法进行阳性检测。荧光定量pcr方法选择筛选标记基因cp4作为目标基因和水稻actin1基因作为内参基因,于荧光定量pcr仪上进行扩增和荧光值检测,根据rq值筛选出基因编辑阳性的植株(cp4基因rq值》0.1)。用于扩增筛选标记基因cp4的引物序列为csp356:cagcacaggttaagtctg(seq id no:10)和csp357:gtctgtctcaacggtaag(seq id no:11);用于扩增actin1基因的引物序列为csp106:tgctatgtacgtcgccatccag(seq id no:12)和csp107:aatgagtaaccacgctccgtca(seq id no:13)。
114.用osd-h基因及loc_os01g10460基因靶位点特异性检测引物(csp6305:5
’‑
tcactgccgaccaggagact-3’(seq id no:14);反向引物csp6306:5
’‑
caggtggtccagatcctcac-3’(seq id no:15))和q5超保真dna聚合酶进行pcr扩增。待反应结束后,利用琼脂糖电泳检测pcr扩增成功与否。之后利用abi3730xl测序仪进行毛细管电泳,之后用软件分析扩增片段的碱基长度。结果显示在164株t0代水稻中39株的osd-h基因和loc_os01g10460的重叠区域发生大片段缺失,突变率为23.7%。该结果为目标编辑结果,即同时编辑osd-h基因及loc_os01g10460基因。另外,所有发生编辑的株系中,osd-h基因的两个拷贝都被成功编辑。这些数据表明,实施例1的编辑载体能够高效地编辑osd-h基因。
115.实施例4t1代osd-h基因及loc_os01g10460基因编辑纯合非转基因水稻的筛选和鉴定
116.将实施例3中鉴定的阳性水稻株系移入温室进行移栽种植,收取其自交t1代种子。选取17株编辑结果大片段缺失的t1代种子(300粒),进行发芽、育苗,提取幼苗的dna进行转基因成分检测及编辑位点pcr检测,去掉含有转基因成分、没有编辑或者编辑位点杂合的株系,保留不携带转基因成分,并且编辑位点纯合的株系,其编辑情况主要为2种大片段缺失类型,选取2种编辑情况各一个株系将其分别命名为480ah012a和480ah013a。转基因成分检测方法请参见《基因编辑后代材料gmo检测流程》,其是根据中国国家标准农业部953号公告6-2007号公告gb/t 19495—2004转基因产品检测标准要求进行。为确认480ah012a和480ah013a编辑株系中osd-h基因及loc_os01g10460基因编辑情况,对其靶位点区域进行pcr扩增和测序。结果显示,480ah012a缺失了65bp核苷酸,和480ah013a缺失了74个核苷酸另外有2处共有7bp核苷酸插入(图1),突变后osd-h基因及loc_os01g10460基因编码蛋白的氨基酸序列如图3和图4所示,480ah012a株系的osd-h基因及loc_os01g10460基因的编码蛋白序列分别如seq id no.16和seq id no.17所示,480ah013a株系的osd-h基因及loc_os01g10460基因的编码蛋白序列分别如seq id no.18和seq id no.19所示。
117.实施例5编辑株系的籽粒表型鉴定
118.将480ah012a和480ah013a t2代纯合编辑株系及对照种子(野生型,wt)进行表型拍照(图5)。从图中可以看出,无论是稻谷还是米粒,粒长方面,编辑材料都明显大于对照;粒宽方面,编辑材料也略大于对照。从整体上看,编辑材料的籽粒明显大于对照。用近红外仪对籽粒糙米的长、宽进行测量,并算出长宽比(表1),结果显示,编辑材料的籽粒长度和长宽比较野生型对照显著增加(经统计学分析具有显著性差异)。
119.表1osd-h基因及loc_os01g10460基因编辑株系与野生型株系的籽粒糙米长、宽及长宽比(n》20)
120.材料糙米长均值(mm)糙米宽均值(mm)糙米长宽比
wt6.8481.963.507480ah012a7.5852.0723.669480ah013a7.6852.0423.777
121.由于粒型明显增大,编辑材料稻谷的千粒重也明显增大(表2)。
122.表2osd-h基因及loc_os01g10460基因编辑株系与野生型株系的籽粒稻谷千粒重
123.材料千粒重(g)(n=3)wt23.28
±
0.56480ah012a28.2
±
0.72**480ah013a27.8
±
0.68**
124.注:3次重复,**p<0.01。
125.从上述结果中可以看出,本发明利用crispr/cas9基因编辑技术获得了osd-h基因及loc_os01g10460基因突变体。与talen和zfn技术相比,crispr/cas9基因编辑技术操作过程简单方便,节约成本,一般实验室都可操作,是一种快速获得籽粒粒型增大增长水稻品种或种质资源的生物技术育种方法。
126.虽然,上文中已经用一般性说明及具体实施方案对本发明作了详尽的描述,但在本发明基础上,可以对之作一些修改或改进,这对本领域技术人员而言是显而易见的。因此,在不偏离本发明精神的基础上所做的这些修改或改进,均属于本发明要求保护的范围。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1