生物标志物组合及其在预测胃癌治疗效果中的应用的制作方法
1.本发明属于生物信息学领域,具体涉及一种生物标志物组合及其在预测赫赛汀联合xelox化疗对胃癌治疗效果中的应用。
背景技术:
2.胃癌(gastric cancer,gc)是世界上第四大最常见的恶性肿瘤,也是全球第二大癌症相关死亡原因。胃癌早期检出率低、晚期治愈率低。晚期胃癌病人的首选治疗方法是外科手术。对于一些没有手术治疗机会的患者,只能靠药物提高生活质量甚至维持生存。尽管近年来一线治疗药物的迭代更新、二线治疗药物以及靶向药物曲妥珠单抗(herceptin,赫赛汀)和雷莫芦单抗(ramucirumab;cyramza)的深入研发取得了重大进展,但是药物耐受问题仍未得到解决,胃癌治疗的总体预后仍处于较差水平。在临床上,肿瘤的治疗效果存在较大的个体差异,但是药物个性化的选择依据过少,临床急需指导个体化精准医疗的标志物,从而缓解药物耐受问题。
3.因此,为了实现胃癌的个体化治疗,有必要根据分子遗传学和病理学特征来识别亚类,发现并应用相应的靶基因。此外,在胃癌研究中,已经报道了可以根据胃癌的亚型对胃癌的预后进行分类的结果。目前已涌现的多项研究性专利均是基于基因组和转录组的基因表达水平实现对胃癌的分类,如预测进展期胃癌患者的术后预后或抗癌药物适合性的系统(专利号cn110168106a),基于胃癌生物学特征的集群分类及预后预测系统(专利号cn110177886a)。然而在临床中,针对胃癌患者的一线治疗方案赫赛汀靶向治疗或联合其他化疗方案(例如联合卡培他滨/5-fu/氟尿嘧啶类,或奥沙利铂/铂类)仍未实现有效的治疗方案抉择。
4.蛋白质组学在揭示肿瘤发生的复杂分子事件,如肿瘤发生、侵袭、转移和对治疗耐受上起了重大作用。蛋白质组学肿瘤诊断具有灵敏度高、特异性强、背景机理明确的优点,近年来被越来越多地运用于肿瘤检测。而且,这些肿瘤标志物的研究往往是基于一定量的实验数据,所涉及的癌症种类和样本量都相对有限。因此,通过收集蛋白质组数据,利用大数据分析方法,建立预测治疗有效的模型,有助于实现个性化化疗,对患者推荐合适的治疗方案具有重要的临床意义。
技术实现要素:
5.针对现有技术中缺少预测赫赛汀联合xelox化疗治疗胃癌效果的技术方案的缺陷,本发明提供了一种生物标志物组合及其在预测赫赛汀联合xelox化疗治疗胃癌效果中的应用。所述生物标志物组合至少包括acox1、actl6a、aip、ambp、b4galnt3、brd8和bsdc1,此时可达到验证集中预测准确率、诊断灵敏度和特异性均为100%,所述生物标志物组合至多可扩展至包括如本发明所述的336种蛋白质分子生物标志物,其预测准确率、诊断灵敏度和特异性均较高。
6.为解决上述技术问题,本发明提供的一个技术方案为:一种生物标志物组合,所述
生物标志物组合包括以下生物标志物:acox1、actl6a、aip、ambp、b4galnt3、brd8和bsdc1。
7.优选地,所述生物标志物组合还包括atp5d。
8.更优选地,所述生物标志物组合还包括以下一个或多个生物标志物:abcd1、abcd3、acads、acbd5、acpp、acsf2、adap1、adh4、adpgk、agl、ago1、akap9、alyref、anapc13、ank2、ap1g1、apaf1、apbb1ip、apip、arhgap4、arhgef17、arl1、arl6ip4、arrb2、atp11b、atp5a1、atp5d、atp6v0a2、b3gnt6、b4galt4、baiap2、bckdk、bms1、bop1、c10orf112、c10orf76、c11orf31、c14orf169、c17orf85、c1orf198、c2orf47、c4orf33、c6orf211、cab39、cadm1、cant1、capn5、capn8、casp6、ccdc47、cdc42ep1、cdk5rap3、ceacam1、cfl2、chi3l1、chordc1、cldn7、clic2、cltc、cnot7、col15a1、col4a1、commd3、cops4、cops5、cops6、cox15、creg1、cryl1、cstb、cstf2t、cyp2s1、cytb、dazap1、dctn5、ddah2、ddhd2、ddr1、ddx27、ddx60、ddx60l、decr2、des、dhcr24、dhfr、dhx38、dido1、dnaja1、dnajc11、dnase2、dock11、dopey2、dpysl4、dsg2、ecm1、edf1、eif2ak2、eif3f、eif5a、elf1、entpd5、ephx1、eps15、eps8l3、erap1、erbb2ip、erlin2、esyt2、etaa1、fam103a1、fam105b、fam162a、fam96a、fis1、fkbp10、flg2、fmnl1、fn3krp、fut2、gale、gba2、gemin7、gid8、gmfg、golim4、golt1b、gosr1、grap2、gtdc2、gtf2e1、gtf2f1、gucy1a3、guk1、heph、herc2、hgd、hla-a、hnmt、hnrnpa1、hnrnph3、hnrnpll、hnrnpr、hspa4l、hspa6、hspb6、hspb7、hspe1、htra3、ict1、ifit5、il1rn、impad1、inppl1、ints9、isca2、itgb5、itsn2、kdelr1、kdm2a、khnyn、kiaa0195、kiaa0319l、kiaa1522、kiaa1704、krt73、krt80kxd1、lamc2、lax1、lgals4、llgl2、lmf2、lpcat3、lrrc1、lsm2、lss、man2b1、me1、med14、med4、memo1、mfsd5、mgst1、mgst3、mlxipl、mmp7、mnf1、mon2、mpc2、mpv17、mrpl3、mrpl48、mrps14、mrps18a、mrvi1、msln、mtg1、mtif2、muc5ac、nbas、nd2、ndufb7、nhej1、nif3l1、nipsnap3a、nras、nsfl1c、nt5c、oca2、ociad2、osbpl1a、pafah1b3、parp12、pawr、pbk、pbx1、pdcd11、pdcd6、pea15、pfkp、pglyrp2、phc2、pik3ca、pip4k2b、pkp2、plin4、podn、polr1a、ppfibp1、ppig、ppp1cc、ppp3cc、prph、psma6、ptdss1、ptges3、ptgr1、ptprcap、qki、qser1、qsox1、rab10、rab3gap1、rangap1、rbm3、rbm39、rbmxl1、rcsd1、rheb、rhoc、rhot1、rnf181、rnf25、rpl18a、rpl23、rpp38、rps4x、rps6ka3、rragc、runx1、ruvbl1、s100a16、scaf11、scin、scrn3、sel1l3、sepsecs、sf3a3、sh3bgrl2、slc25a35、slc30a5、slc35d1、slc44a1、slc44a2、smpdl3b、smtn、snap23、snrpg、soat1、spon1、srsf2、srsf9、st14、st6galnac1、steap4、stk17b、stk39、stxbp3、sugp2、syk、syncrip、synpo、szrd1、tacstd2、taf6、tbcc、telo2、tep1、timm13、tm9sf2、tmem45b、tmem63a、tnfaip8l2、tnik、tnrc6b、tomm6、tp53bp1、tpp2、tram1、trappc2l、trim14、tspan8、ttc39a、ttn、tufm、ube2q1、ube2s、ubxn6、uqcrq、usp34、usp9y、vps51、vti1a、wbscr22、wdr26、wdr81、wibg、ybx3和znf706。
9.为解决上述技术问题,本发明提供的一个技术方案为:一种用于检测如本文所述的生物标志物组合的表达水平的试剂,所述试剂包括分别或同时与所述生物标志物特异性杂交的生物分子,所述生物分子例如引物、探针和/或抗体;或,所述试剂包括用于检测如本文所述的生物标志物组合的表达水平的基因组、转录组和/或蛋白质组测序的试剂。
10.为解决上述技术问题,本发明提供的一个技术方案为:一种试剂盒,包括如本文所述的生物标志物组合和/或如本文所述的试剂。
11.为解决上述技术问题,本发明提供的一个技术方案为:如本文所述的生物标志物
组合、如本文所述的试剂或如本文所述的试剂盒在制备预测赫赛汀联合xelox化疗治疗胃癌效果的制剂中的应用。优选地,所述制剂为诊断产品。
12.为解决上述技术问题,本发明提供的一个技术方案为:一种用于预测赫赛汀联合xelox化疗治疗胃癌效果的系统,所述系统包括:
13.数据处理模块,用于将接收或输入胃癌患者的如本文所述的生物标志物组合的表达数据;
14.判断并输出模块,用于在所述接收或输入完成后,通过firmiana软件处理所述表达数据,预设为基于广义线性回归模型的机器学习算法,构建预测模型,分别预测胃癌患者的敏感预测概率和不敏感预测概率,得到胃癌患者的敏感预测概率和不敏感预测概率,判断所述表达数据是否符合预设的判断条件,以预测赫赛汀联合xelox化疗治疗胃癌效果,并输出预测结果;
15.其中,在所述判断并输出模块中,当所述表达数据满足所述判断条件时,所述判断条件为敏感预测概率大于或等于非敏感预测概率,输出预测结果为“赫赛汀联合xelox化疗对胃癌具有治疗效果”;当所述表达数据不满足所述判断条件时,即敏感预测概率小于非敏感预测概率,输出预测结果为“赫赛汀联合xelox化疗对胃癌不具有治疗效果”。
16.优选地,所述预设中,训练集参数设置为80%,验证集参数设置为20%。
17.优选地,所述系统还包括数据收集模块,用于收集患者胃癌组织样本中所述生物标志物组合的表达数据,并将其传输给数据处理模块。
18.为解决上述技术问题,本发明提供的一个技术方案为:一种计算机辅助的预测胃癌赫赛汀联合xelox化疗治疗效果的方法,其包括以下步骤:
19.(1)接收或输入胃癌患者的如本文所述的生物标志物组合的表达数据,通过firmiana软件处理所述表达数据,预设为基于广义线性回归模型的机器学习算法,构建预测模型,分别预测胃癌患者的敏感预测概率和不敏感预测概率;
20.(2)判断所述表达数据是否符合预设的判断条件,所述判断条件为敏感预测概率大于或等于非敏感预测概率,以预测赫赛汀联合xelox化疗治疗胃癌效果,并输出预测结果;
21.其中,在步骤(2)中,当所述表达数据满足所述判断条件时,输出预测结果为“赫赛汀联合xelox化疗对胃癌具有治疗效果”;当所述表达数据不满足所述判断条件时,输出预测结果为“赫赛汀联合xelox化疗对胃癌不具有治疗效果”。
22.为解决上述技术问题,本发明提供的一个技术方案为:一种计算机可读存储介质,其存储有计算机程序,所述计算机程序被处理器执行时,可实现如本文所述的系统的功能,或实现如本文所述的方法的步骤。
23.为解决上述技术问题,本发明提供的一个技术方案为:一种电子设备,其包括存储器和处理器,所述存储器存储有计算机程序,所述处理器用于执行所述计算机程序以实现如本文所述的系统的功能,或如本文所述的方法的步骤。
24.在符合本领域常识的基础上,上述各优选条件,可任意组合,即得本发明各较佳实例。
25.本发明所用试剂和原料均市售可得。
26.本发明的积极进步效果在于:
27.本发明提供的生物标志物组合在胃癌患者不同治疗反应的临床样本中的表达水平存在显著变化,因此本发明提供生物标志物组合可以预测胃癌患者接受赫赛汀联合xelox化疗治疗的胃癌效果,具有高灵敏度和高特异性的优点,为预测胃癌患者接受赫赛汀联合xelox化疗治疗提供有利的技术支持。
28.基于本发明的生物标志物组合研制相应的系统、计算机可读存储介质、电子设备和开发的预测方法,具有广泛的科研价值并为胃癌患者提供个性化预测,为患者推荐是否适合接受该赫赛汀联合xelox化疗治疗的方案。
附图说明
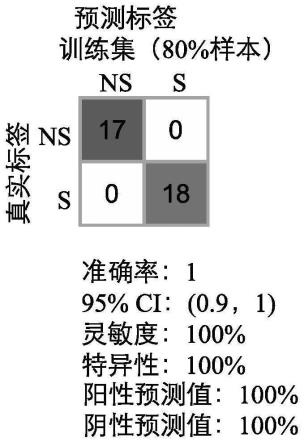
29.图1为本发明所述标志物组合在训练集中的灵敏度和特异性结果。
30.图2为本发明所述标志物组合在验证集中的灵敏度和特异性结果。
31.图3为预测胃癌铂类加氟尿嘧啶类联合紫杉醇类药物治疗效果的系统的结构示意图;
32.图4为电子设备的结构示意图。
具体实施方式
33.下面通过实施例的方式进一步说明本发明,但并不因此将本发明限制在所述的实施例范围之中。下列实施例中未注明具体条件的实验方法,按照常规方法和条件,或按照商品说明书选择。
34.实施例中所需的接受赫赛汀靶向治疗或联合其他化疗方案治疗,其中包括赫赛汀单独治疗2例(2.9%)、赫赛汀联合紫杉醇类治疗3例(4.3%)、赫赛汀联合氟尿嘧啶类治疗6例(8.7%)、赫赛汀联合喜树碱治疗3例(4.3%)、赫赛汀联合铂类治疗1例(1.4%)、赫赛汀联合帕妥珠单抗治疗1例(1.4%)、赫赛汀联合xelox化疗44例(64%)和赫赛汀联合氟尿嘧啶加紫杉醇类治疗9例(13.0%)的胃癌患者的治疗前的临床样本,共44例,包括治疗敏感组、非敏感组分别为35例、9例。本研究的设计和实施由医学伦理委员会通过伦理投票批准和监督。已获得所有患者的书面知情同意。
35.实施例1胃癌治疗前的临床样本的预处理
36.临床样本为福尔马林固定石蜡包埋组织。样品预处理:从石蜡块上取3-10μm厚的切片进行宏观解剖,二甲苯脱蜡,乙醇洗涤,风干处理,获得白片,同时苏木精-伊红对3μm厚的切片进行染色作为肿瘤镜下对切片中肿瘤细胞含量进行评估判断并划选肿瘤区域。10μm厚的切片收集对应的肿瘤样本至离心管中,-80冻存备用。
37.实施例2临床样本的蛋白质及肽段提取
38.将等量的ffpe(实施例1中制备的肿瘤样本)组织收集于ep管,加入裂解缓冲液(0.1m tris-hcl ph 8.0中,0.1m dtt,1mm pmsf),然后用磨棒研磨3分钟;加入十二烷基硫酸钠(sds)使终浓度为4%,99℃,1800rpm震荡2-2.5小时;12,000g离心5分钟收集上清于ep管,加入4倍体积的丙酮,放置-20℃4小时或过夜;12,000g于4℃离心1分钟弃上清保留沉淀,冷丙酮洗涤沉淀三次,超净台风干处理蛋白沉淀;用8m urea和50mm nh4hco3将蛋白沉淀复溶后加在fasp管中,反复用50mm nh4hco3离心去除urea;将溶有5.5μg胰蛋白酶的50μl 50mm nh4hco3加入到fasp管中,于37℃温育18-20小时进行酶解;12,800g离心15分钟收集
肽段,为提高肽段得率,用200μl ms水洗涤两次,一并收集;60℃真空抽干,获得用于质谱检测所需的肽段。
39.实施例3临床样本的质谱检测
40.用q-exactive hf-x混合四极轨道阱质谱仪(thermo fisher scientific,rockford,il,usa)和高效液相色谱系统(easy nlc 1200,thermo fisher)进行检测,并得到该肽样品对应的质谱数据。具体操作为:
41.抽干的肽样品重新溶解在溶剂a(0.1%甲酸的水溶液)中,上样至trap柱(100μm
×
2cm;粒子大小,3μm;孔径大小,),后在分析柱上进行分离(150μm
×
12cm,粒子大小,1.9μm;孔径大小,),梯度为5-35%流动相b(80%乙腈和0.1%甲酸)洗脱,流速为600nl/min,合计洗脱75分钟。对qe-hfx进行ms分析,一次全扫描(300-1400m/z,分辨率=12000),离子阱中允许进入的最大离子数(automatic gain control target,agc target)为3e+06离子,随后进行高能量碰撞诱导离解(隔离窗口为1.6m/z,碰撞能量为27%,agc target为5e+04离子,最大注入时间为30ms,动态排除设置为18秒)。液相色谱串联质谱系统使用xcalibur软件(thermo scientific)控制进行数据采集。
42.实施例4预测赫赛汀联合xelox化疗治疗胃癌的效果
43.所有数据均使用firmiana(v1.0)进行处理。firmiana是一个基于galaxy系统的工作流,由用户登录界面、原数据、识别与量化、数据分析和知识挖掘等多个功能模块组成,本实施例选择的预设为基于广义线性回归模型的机器学习算法。原始文件是根据人类国家生物技术信息中心(ncbi)的refseq蛋白质数据库(updated on 04-07-2013,32,015entries)检索的。选择胰蛋白酶作为蛋白水解酶,最大允许两个漏切位点,固定修饰为carbamidomethyl(c),动态修饰为protein acetyl(protein n-term),oxidation(m)。第一个搜索质量耐受性为20ppm,主要搜索肽耐受性为0.5da。肽谱匹配(psms)和蛋白质的错误发现率(fdr)均小于1%。将所鉴定的肽段定量结果记为所有参考谱库中色谱碎片离子峰面积的平均值。使用无标签的基于强度的绝对定量(ibaq)方法进行蛋白质定量。我们计算了峰面积值作为相应蛋白质的一部分。总分数(fot)用于表示样品中特定蛋白质的标准化丰度。fot定义为蛋白质的ibaq除以样品中所有已鉴定蛋白质的总ibaq。选择具有至少一条专属肽段(unique peptide)且1%fdr的蛋白质。将上述44例临床样本分为包括35例的训练集和9例的内部验证集,以下各组均使用相同的训练集和验证集,见如下组:
44.第一组
45.对胃癌患者临床样本中的7种蛋白质分子生物标志物(acox1、actl6a、aip、ambp、b4galnt3、brd8和bsdc1)的相对表达水平计算其预测准确度、灵敏度及特异性,其中包括35例训练集,预测准确率为100%,灵敏度和特异性分别为100%和100%;剩余9例为测试集,预测准确率为100%(图1),灵敏度和特异性分别为100%和100%(图2)。对于待治疗胃癌患者,根据蛋白质分子生物标志物的表达水平,得到该患者使用赫赛汀联合xelox化疗的不同反应的输出结果,从而向该患者推荐或不推荐该治疗方案(见表1)。
46.表1第1组标志物的预测及输出结果
[0047][0048]
第2组
[0049]
在第1组生物标志物的基础上,增加蛋白质分子生物标志物atp5d,对该8种蛋白质分子生物标志物(acox1、actl6a、aip、ambp、atp5d、b4galnt3、brd8、bsdc1)组成蛋白质分子生物标志物组合的相对表达水平计算其预测准确度,灵敏度及特异性。在训练集中,预测准确率为100%,诊断灵敏度100.00%,特异性100.00%;内部验证集中,预测准确率为100%,诊断灵敏度100.00%,特异性100%。对于待治疗胃癌患者,根据蛋白质分子生物标志物的表达水平,得到该患者使用赫赛汀联合xelox化疗的不同反应的输出结果,从而向该患者推荐或不推荐该治疗方案(见表2)。
[0050]
表2第2组标志物的预测及输出结果
[0051][0052]
第3组
[0053]
在第二组的基础上,扩展为336种蛋白质分子生物标志物(abcd1、abcd3、acads、acbd5、acox1、acpp、acsf2、actl6a、adap1、adh4、adpgk、agl、ago1、aip、akap9、alyref、ambp、anapc13、ank2、ap1g1、apaf1、apbb1ip、apip、arhgap4、arhgef17、arl1、arl6ip4、arrb2、atp11b、atp5a1、atp5d、atp6v0a2、b3gnt6、b4galnt3、b4galt4、baiap2、bckdk、bms1、bop1、brd8、bsdc1、c10orf112、c10orf76、c11orf31、c14orf169、c17orf85、c1orf198、c2orf47、c4orf33、c6orf211、cab39、cadm1、cant1、capn5、capn8、casp6、ccdc47、cdc42ep1、cdk5rap3、ceacam1、cfl2、chi3l1、chordc1、cldn7、clic2、cltc、cnot7、col15a1、col4a1、commd3、cops4、cops5、cops6、cox15、creg1、cryl1、cstb、cstf2t、cyp2s1、cytb、dazap1、dctn5、ddah2、ddhd2、ddr1、ddx27、ddx60、ddx60l、decr2、des、dhcr24、dhfr、dhx38、dido1、dnaja1、dnajc11、dnase2、dock11、dopey2、dpysl4、dsg2、ecm1、edf1、eif2ak2、eif3f、eif5a、elf1、entpd5、ephx1、eps15、eps8l3、erap1、erbb2ip、erlin2、esyt2、etaa1、fam103a1、fam105b、fam162a、fam96a、fis1、fkbp10、flg2、fmnl1、fn3krp、fut2、gale、gba2、gemin7、gid8、gmfg、golim4、golt1b、gosr1、grap2、gtdc2、gtf2e1、gtf2f1、gucy1a3、guk1、heph、herc2、hgd、hla-a、hnmt、hnrnpa1、hnrnph3、hnrnpll、hnrnpr、hspa4l、hspa6、hspb6、hspb7、hspe1、htra3、ict1、ifit5、il1rn、impad1、inppl1、ints9、isca2、itgb5、itsn2、kdelr1、kdm2a、khnyn、kiaa0195、kiaa0319l、kiaa1522、kiaa1704、krt73、krt80 kxd1、lamc2、lax1、lgals4、llgl2、lmf2、lpcat3、lrrc1、lsm2、lss、man2b1、me1、med14、med4、memo1、mfsd5、mgst1、mgst3、mlxipl、mmp7、mnf1、mon2、mpc2、mpv17、mrpl3、mrpl48、mrps14、
mrps18a、mrvi1、msln、mtg1、mtif2、muc5ac、nbas、nd2、ndufb7、nhej1、nif3l1、nipsnap3a、nras、nsfl1c、nt5c、oca2、ociad2、osbpl1a、pafah1b3、parp12、pawr、pbk、pbx1、pdcd11、pdcd6、pea15、pfkp、pglyrp2、phc2、pik3ca、pip4k2b、pkp2、plin4、podn、polr1a、ppfibp1、ppig、ppp1cc、ppp3cc、prph、psma6、ptdss1、ptges3、ptgr1、ptprcap、qki、qser1、qsox1、rab10、rab3gap1、rangap1、rbm3、rbm39、rbmxl1、rcsd1、rheb、rhoc、rhot1、rnf181、rnf25、rpl18a、rpl23、rpp38、rps4x、rps6ka3、rragc、runx1、ruvbl1、s100a16、scaf11、scin、scrn3、sel1l3、sepsecs、sf3a3、sh3bgrl2、slc25a35、slc30a5、slc35d1、slc44a1、slc44a2、smpdl3b、smtn、snap23、snrpg、soat1、spon1、srsf2、srsf9、st14、st6galnac1、steap4、stk17b、stk39、stxbp3、sugp2、syk、syncrip、synpo、szrd1、tacstd2、taf6、tbcc、telo2、tep1、timm13、tm9sf2、tmem45b、tmem63a、tnfaip8l2、tnik、tnrc6b、tomm6、tp53bp1、tpp2、tram1、trappc2l、trim14、tspan8、ttc39a、ttn、tufm、ube2q1、ube2s、ubxn6、uqcrq、usp34、usp9y、vps51、vti1a、wbscr22、wdr26、wdr81、wibg、ybx3和znf706),对这336种蛋白质分子生物标志物的相对表达水平计算其预测准确度,其中包括35例训练集,预测准确率为100%,诊断灵敏度100.00%,特异性100.00%,剩余9例为内部验证集,预测准确率为100%,诊断灵敏度100%,特异性100%。对于待治疗胃癌患者,根据蛋白质分子标志物的表达水平,得到该患者使用赫赛汀联合xelox化疗的不同反应的输出结果,从而向该患者推荐或不推荐该治疗方案(见表3)
[0054]
表3第3组标志物的预测及输出结果
[0055][0056]
由上述结果可知,将胃癌患者的临床样本中的7种蛋白质分子生物标志物(见第1组)联用,可用于预测胃癌患者接受赫赛汀联合xelox化疗后是否可以产生肿瘤消退的有效治疗效果。
[0057]
在此基础上增加蛋白分子生物标志物(如第2组和第3组),均可很好地预测胃癌患者接受赫赛汀联合xelox化疗后是否可以产生肿瘤消退的有效治疗效果。
[0058]
实施例5预测胃癌铂类联合氟尿嘧啶类药物治疗效果的系统
[0059]
预测胃癌铂类联合氟尿嘧啶类药物治疗效果的系统61:数据处理模块52和判断并输出模块53,还包括数据收集模块51(图3)。
[0060]
数据收集模块51用于收集患者胃癌组织样本中所述生物标志物组合的表达数据,并将其传输给数据处理模块。
[0061]
数据处理模块52用于将接收或输入生物标志物组合的表达数据按如实施例4所述的数据分析方法进行分析,得到计算结果。其中,所述生物标志物组合的表达数据可通过数据收集模块51进行收集,亦可从其他来源获取所述生物标志物组合的表达数据。
[0062]
判断并输出模块53用于判断所述的计算结果是否符合预设的判断条件,即敏感预测概率大于非敏感预测概率,以预测铂类联合氟尿嘧啶类药物治疗胃癌效果,并输出预测结果;其中,在所述判断并输出模块中,当所述表达数据满足所述判断条件敏感预测概率大
于或等于非敏感预测概率时,输出预测结果为“铂类联合氟尿嘧啶类药物对胃癌具有治疗效果”;当所述表达数据不满足所述判断条件敏感预测概率小于非敏感预测概率时,输出预测结果为“铂类联合氟尿嘧啶类药物对胃癌不具有治疗效果”。
[0063]
实施例6电子设备
[0064]
本实施例提供了一种电子设备,电子设备可以通过计算设备的形式表现(例如可以为服务器设备),包括存储器、处理器及存储在存储器上并可在处理器上运行的计算机程序,其中处理器执行计算机程序时可以实现本发明实施例4中预测胃癌铂类联合氟尿嘧啶类药物治疗效果的方法。
[0065]
图4示出了本实施例的硬件结构示意图,电子设备9具体包括:
[0066]
至少一个处理器91、至少一个存储器92以及用于连接不同系统组件(包括处理器91和存储器92)的总线93,其中:
[0067]
总线93包括数据总线、地址总线和控制总线。
[0068]
存储器92包括易失性存储器,例如随机存取存储器(ram)921和/或高速缓存存储器922,还可以进一步包括只读存储器(rom)923。
[0069]
存储器92还包括具有一组(至少一个)程序模块924的程序/实用工具925,这样的程序模块924包括但不限于:操作系统、一个或者多个应用程序、其它程序模块以及程序数据,这些示例中的每一个或某种组合中可能包括网络环境的实现。
[0070]
处理器91通过运行存储在存储器92中的计算机程序,从而执行各种功能应用以及数据处理,例如本发明实施例4的数据分析方法。
[0071]
电子设备9进一步可以与一个或多个外部设备94(例如键盘、指向设备等)通信。这种通信可以通过输入/输出(i/o)接口95进行。并且,电子设备9还可以通过网络适配器96与一个或者多个网络(例如局域网(lan),广域网(wan)和/或公共网络,例如因特网)通信。网络适配器96通过总线93与电子设备9的其它模块通信。应当明白,尽管图中未示出,可以结合电子设备9使用其它硬件和/或软件模块,包括但不限于:微代码、设备驱动器、冗余处理器、外部磁盘驱动阵列、raid(磁盘阵列)系统、磁带驱动器以及数据备份存储系统等。
[0072]
应当注意,尽管在上文详细描述中提及了电子设备的若干单元/模块或子单元/模块,但是这种划分仅仅是示例性的并非强制性的。实际上,根据本技术的实施方式,上文描述的两个或更多单元/模块的特征和功能可以在一个单元/模块中具体化。反之,上文描述的一个单元/模块的特征和功能可以进一步划分为由多个单元/模块来具体化。
[0073]
实施例7计算机可读存储介质
[0074]
本发明实施例提供了一种计算机可读存储介质,其上存储有计算机程序,程序被处理器执行时实现本发明实施例4中预测胃癌铂类联合氟尿嘧啶类药物治疗效果的方法的步骤。
[0075]
其中,可读存储介质可以采用的更具体可以包括但不限于:便携式盘、硬盘、随机存取存储器、只读存储器、可擦拭可编程只读存储器、光存储器件、磁存储器件或上述的任意合适的组合。
[0076]
在可能的实施方式中,本发明还可以实现为一种程序产品的形式,其包括程序代码,当所述程序产品在终端设备上运行时,所述程序代码用于使所述终端设备执行实现本发明实施例4中预测胃癌铂类联合氟尿嘧啶类药物治疗效果的方法的步骤。
[0077]
其中,可以以一种或多种程序设计语言的任意组合来编写用于执行本发明的程序代码,所述程序代码可以完全地在用户设备上执行、部分地在用户设备上执行、作为一个独立的软件包执行、部分在用户设备上部分在远程设备上执行或完全在远程设备上执行。
[0078]
对比例1种类更少的蛋白质分子生物标志物组合的预测效果
[0079]
在第1组的7种蛋白分子生物标志物的基础上减少蛋白质分子生物标志物种类形成种类更少的蛋白质分子生物标志物组合,以与实施例1-4相同的数据库、分析方法、训练集和验证集,计算各组的预测准确度。
[0080]
第4组
[0081]
在缺失bsdc1蛋白的情况下,标志物组合为acox1、actl6a、aip、ambp、b4galnt3和brd。在训练集上,准确率为97.14%,灵敏度和特异性分别为94.12%和100%;测试集上准确率为100%,灵敏度和特异性分别为100%和100%。
[0082]
第5组
[0083]
在缺失bsdc1、brd8蛋白的情况下,标志物组合为acox1、actl6a、aip、ambp和b4galnt3。在训练集上,准确率为77.14%,灵敏度和特异性分别为64.71%和88.89%;测试集上准确率为88.89%,灵敏度和特异性分别为100%和83.33%。
[0084]
第6组
[0085]
在缺失bsdc1、brd8、b4galnt3蛋白的情况下,标志物组合为acox1、actl6a、aip和ambp。在训练集上,准确率为68.57%,灵敏度和特异性分别为70.59%和66.67%;测试集上准确率为88.89%,灵敏度和特异性分别为100%和83.33%。
[0086]
第7组
[0087]
在缺失bsdc1、brd8、b4galnt3、ambp蛋白的情况下,标志物组合为acox1、actl6a和aip。在训练集上,准确率为62.86%,灵敏度和特异性分别为58.82%和66.67%;测试集上准确率为77.78%,灵敏度和特异性分别为66.67%和83.33%。
[0088]
第8组
[0089]
在缺失bsdc1、brd8、b4galnt3、ambp、aip蛋白的情况下,标志物组合为acox1和actl6a。在训练集上,准确率为71.43%,灵敏度和特异性分别为52.94%和88.89%;测试集上准确率为66.67%,灵敏度和特异性分别为33.33%和83.33%。
[0090]
此结果表明,随着最少标志物组合蛋白质数目的逐次缺失,标志物组合预测的准确率下降,并且灵敏度和特异性也降低;因此,标志物组合acox1、actl6a、aip、ambp、b4galnt3、brd8和bsdc1中的蛋白质是保证预测准确率,灵敏度和特异性的必需蛋白质标志物。
[0091]
最后,上述具体实施方法仅用以说明本发明的技术方案,而非对其限制。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1