一种丛枝菌根真菌共生作用下的根际简化细菌菌群的筛选方法
本发明涉及菌群筛选,更具体的说是涉及一种丛枝菌根真菌共生作用下的根际简化细菌菌群的筛选方法。
背景技术:
1、根际对植物健康至关重要,它包含一个复杂的微生物群落,被称为植物的第二基因组。植物根系向根际释放养分、分泌物、边缘细胞和粘液,细菌、真菌(包括丛枝菌根真菌)、卵菌、病毒和古细菌被根际沉积物吸引并以它们为食。然而,根和微生物之间的直接相互作用是最重要的,并且极大地影响植物的生长、健康和适应。植物对根际微生物群落的相对丰度和组成具有物种特异性影响,不同植物生长在同一土壤中会发展出不同的微生物群落。除了植物的调节外,微生物相互作用还可以调节根际微生物群落。
2、丛枝菌根(am)真菌与超过三分之二的陆生植物形成共生关系。在共生关系中,am真菌以多种方式促进植物生长,尤其是通过增加对磷、氮和钾的吸收。但是,对几种am真菌全基因组进行研究发现,与腐生真菌或外生菌根真菌的基因组相比,am真菌具有特别少的外酶谱,其编码碳水化合物降解酶(cazys)的基因较少,尤其是缺乏糖苷水解酶(ghs),如gh6、gh7。此外,am真菌缺乏编码植酸酶的基因,该酶能水解土壤中有机磷的主要形式—植酸。这表明,与其他真菌相比,am真菌对土壤有机养分的直接活化能力相对较弱。然而,定殖在am真菌菌丝际的土壤微生物可能极大地促进了土壤有机养分的周转,弥补了am真菌缺失的功能。迄今为止,大多数研究都集中在微生物-根系的相互作用上,但am真菌和根际微生物的相互作用及其对植物生长的影响在很大程度上仍未得到探索。菌根生物技术最近在改善退化土壤和保护环境的生物修复中变得不可或缺。am真菌通过联合根际微生物群来开发土壤中的有机养分(如氮和磷)资源,通过菌根途径提高养分吸收效率,具有重要的生态和农学意义。
3、因此,发明一种丛枝菌根真菌共生作用下的根际简化细菌菌群的筛选方法,对于am真菌共生效应的基础研究及广泛应用,具有重要意义。
技术实现思路
1、有鉴于此,本发明提供了一种丛枝菌根真菌共生作用下的根际简化细菌菌群的筛选方法。
2、为了实现上述目的,本发明采用如下技术方案:
3、一种丛枝菌根真菌共生作用下的根际简化细菌菌群的筛选方法,包括如下步骤:
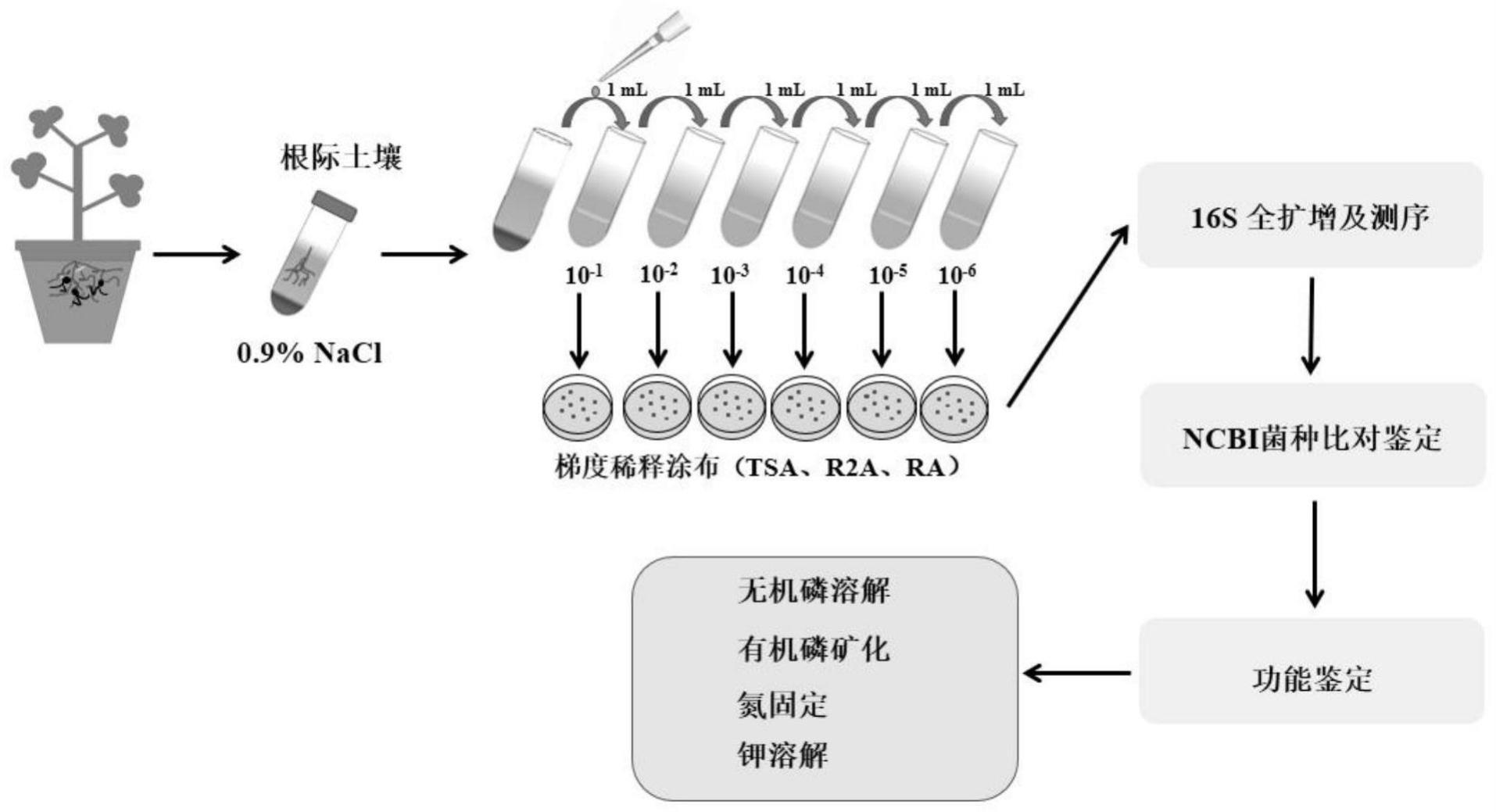
4、步骤一:丛枝菌根真菌共生作用下的根际细菌分离
5、收集丛枝菌根真菌共生的根际土壤,利用质量浓度0.9%的nacl无菌溶液对根际土壤进行重悬和梯度稀释,将稀释液涂布到细菌培养基中,对根际细菌进行分离;
6、步骤二:根际微生物群的筛选
7、1)对根际土壤样品dna进行提取,制备插入片段长度为500bp的文库,进行宏基因组测序;同时,以根际土壤样品dna为模板进行16s rrna pcr扩增并纯化,构建pe文库,进行16s rrna基因高通量测序;
8、2)将16s rrna基因高通量测序数据进行拼接和质量控制,得到优化数据,进行asv聚类,对asv注释,之后进行物种相对丰度分析、α多样性分析、β多样性分析以及物种差异与相关性网络分析;
9、3)对宏基因组测序的原始数据进行处理,得到有效数据,之后计算基因在根际样本的丰度信息,将基因集与nr、kegg、eggnog、ardb、cazy、seed进行比对,获得基因的物种注释信息和功能注释信息;
10、4)对16s rrna基因高通量测序数据的分析结果(指的是步骤2)的分析结果)和宏基因组数据(步骤3)的有效数据)进行整合分析,利用随机森林分析、lefse分析和相关性网络分析,结合宏基因组的功能分析结果(步骤3)的分析结果),筛选出丛枝菌根真菌介导的关键根际细菌菌群;
11、步骤三:简化细菌菌群的构建
12、1)对步骤一分离到的根际细菌进行种属鉴定;
13、2)将第1)步鉴定的种属与步骤二分析得到的丛枝菌根真菌介导的关键根际细菌菌群,进行整合分析,找到步骤一和步骤二中相同的菌属,构建简化细菌菌群,并对简化菌群进行纯化培养和保藏;
14、步骤四:简化细菌菌群功能鉴定
15、对简化细菌菌群的固氮、解磷和解钾功能进行鉴定,最终确定丛枝菌根真菌共生作用下的根际简化细菌菌群。
16、所取得的有益效果:丛枝菌根真菌共生作用下的根际细菌的筛选和鉴定是本发明的第一个难点。主要依靠多种组学分析技术,扩增子高通量测序可以明确根际富集的菌属,宏基因组测序可以明确根际富集的菌属的功能,两者结合,就可以分析出丛枝菌根真菌共生作用下介导养分吸收的根际细菌。
17、根际细菌的分离和功能鉴定是本发明的第二个难点,因为土壤中可分离的菌属占比非常小,分离特定种属一直是个挑战,本发明采取多种培养基结合的方式进行分离,并采用鉴定培养基对特定功能菌属进行筛选。
18、进一步的,所述细菌培养基为牛肉膏蛋白胨培养基、r2a培养基和大豆酪蛋白琼脂培养基。
19、所取得的有益效果:上述三种培养基可以培养的细菌种类很多,同时也可以达到互补分离的效果。
20、进一步的:所述步骤二中的16s rrna基因高通量测序区域为v3-v4;
21、测序平台为illumnia mi-seq 2×300bp;
22、测序引物为:
23、341f:cctacgggnggcwgcag,seq id no:1;
24、805r:gactachvgggtatctaatcc,seq id no:2。
25、所取得的有益效果:上述测序区域具有较高的简并度有利于较高的覆盖率。
26、进一步的,步骤二的具体操作为:
27、1)对步骤一分离收集到的根际土壤,对根际土壤样品dna进行提取,制备插入片段长度为500bp左右的文库,在illuminatm二代测序平台进行宏基因组测序。同时,以土壤样品dna为模板进行16s rrna pcr扩增并纯化,构建pe文库,利用illumina测序平台进行16srrna基因高通量测序。
28、2)将16s rrna基因高通量测序下机的原始bcl格式数据转化为fastq文件,然后对数据进行拼接和质量控制,优化得到数据;利用usearch进行asv聚类,利用已有的物种注释数据库对asv注释。利用已注释的asv进行物种相对丰度分析、α多样性分析、β多样性分析以及物种差异与相关性网络分析。
29、3)对宏基因组测序的原始数据通过fastqc进行质量评估,并通过trimmomatic进行过滤处理,得到相对准确的有效数据。对各样本clean reads进行拼接组装,对拼接结果进行orf预测,对于各样本的基因预测结果进行去冗余,以获得非冗余的基因集,然后将各样本clean reads比对到非冗余基因集序列上,计算基因在各样本的丰度信息。将基因集与nr、kegg、eggnog、ardb、cazy、seed进行比对,获得基因的物种注释信息和功能注释信息。
30、4)对16s rrna基因高通量测序数据和宏基因组数据进行整合分析,利用随机森林分析、lefse分析和相关性网络分析,结合宏基因组的功能分析,筛选出丛枝菌根真菌介导的关键根际细菌菌群。
31、进一步的,所述步骤四中的氮、磷和钾筛选培养基为nfm培养基、opd b培养基、无机磷筛选培养基和解钾细菌筛选培养基,各培养基组分如下:
32、每1000g nfm培养基包括如下组分:k2hpo40.20g,kh2po40.50g,mgso4.7h2o 0.20g,feso4·7h2o 0.10g,na2moo4·2h2o 0.005g,nacl 0.20g,glucose 10.00g和琼脂15.00g;
33、每1000g opdb培养基包括如下组分:葡萄糖10.0g,(nh4)2so40.5g,nacl 0.3g,kcl0.3g,yeast extract 0.5g,mgso4·7h2o 0.3g,feso4·7h2o 0.03g,mnso4·4h2o 0.03g,caco35.0g,soybean lecithin15.0g和bacto-agar 15.0g;
34、每1000g无机磷筛选培养基包括如下组分:葡萄糖10.0g,(nh4)2so40.5g,nacl0.3g,kcl 0.3g,yeast extract 0.5g,mgso4·7h2o 0.3g,feso4·7h2o 0.03g,mnso4·4h2o0.03g,caco35.0g,ca3(po4)215.0g和bacto-agar 15.0g;
35、每1000g解钾细菌筛选培养基包括如下组分:glucose 3.50g,mgso4.7h2o 0.50g,caco30.10g,fecl30.0005g,ca3po42.00g和不溶性钾长石粉1.0g。
36、经由上述的技术方案可知,与现有技术相比,本发明取得的有益效果为:本发明利用16s rrna基因高通量测序技术与宏基因测序技术结合,使筛选的根际细菌更具有可靠性。本发明利用根际细菌分离纯化,并使用不同功能筛选培养基去鉴定其菌群菌株的功能,以筛选和鉴定与am真菌相互作用的促生根际细菌,对于am真菌共生效应的基础研究及广泛应用,具有重要意义。
- 还没有人留言评论。精彩留言会获得点赞!