一种基于多色探针杂交的基因芯片编解码方法与流程
本发明涉及生物芯片,具体涉及一种基于多色探针杂交的基因芯片编解码方法。
背景技术:
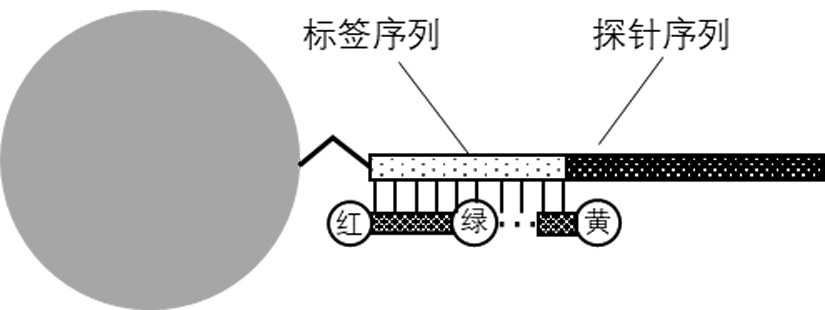
1、基因芯片技术最成熟的结构形式为微珠芯片,其包括微珠和与微珠连接的探针分子,探针分子上靠近微珠的一段为标签序列,标签序列具有特异性,标签序列的设计即为编码;连接有探针分子的微珠装载到芯片上后,由于装载过程具有随机性,需采取某种方法来获取芯片上每个微珠所对应的探针种类信息,即获取每个微珠的标签序列信息,这一过程即为解码。
2、传统的基因芯片编解码方法采用单色荧光探针互补杂交的原理进行。具体地,利用碱基互补配对原理,使微珠上探针的标签序列与其互补序列(解码序列)进行杂交反应,由于解码序列上带有荧光基团,微珠上就带上了相应的荧光信号,通过荧光扫描获取荧光信号,再转换成对应的标签序列,即可获知微珠上所载探针种类。由于荧光基团颜色种类有限,对于高密度基因芯片而言,采用单色荧光探针需要多轮次显色为每种微珠形成颜色编码,成本较高,同时多轮次杂交显色涉及多次dna变性和复性过程,对微珠上寡核苷酸链有损伤,不利于后续分析检测。
3、在现有技术上,也有研究提出多段标签序列依次杂交的杂交方式,即一种探针有多段标签序列,第一次解码实验与第一段标签序列杂交,第二次解码实验与第二段标签序列杂交,以此类推,但是此种方式增加了体系复杂性,而且多段标签序列占据了更多的序列可能性,当编码的微珠种类非常多时,需要生成大量的标签序列,易出现重复序列导致dna杂交特异性变差,准确度下降。因此基因芯片编解码方式亟待创新。
4、比如,公开号为cn113416766a的发明申请公开了一种生物芯片的解码方法,所述生物芯片的解码方法通过x段标签序列同时与x个带有荧光基团的解码序列发生y步特异性杂交反应,x≥2,y≥1,使得生物芯片上的每一个孔位位置得到一个实际荧光颜色标签序列,所述荧光基团选自至少四种不同颜色的荧光基团中的任一种,将每一个孔位位置对应的实际荧光颜色标签序列与预设荧光颜色标签序列进行比对,即可得到所有孔位的探针种类-孔位位置的对应信息,即完成生物芯片的解码。该现有技术中采用单色荧光解码,单个标签,每轮次一个位置显示一种颜色,每次解码信息有限,需要更多轮次解码。
5、再比如,公开号为cn113106090a的发明申请公开了一种生物芯片的带有复合标签序列的探针及生物芯片,所述复合标签序列包括至少2段标签序列,每段标签序列的长度为8-45mer,每段标签序列均符合预设原则。使用本技术的技术方案生成的标签序列的特异性更高,与目标基因组的相似度更低;并且,本技术的技术方案中的探针带有复合标签序列,每一段标签序列分别同时与其互补解码序列发生杂交反应,因此在一次解码实验中获得的信息量增大,解码相同数量的探针所需的解码实验次数减少,从而极大地提高了在生产环节中确定生物芯片的解码文件时的解码效率。该现有技术中采用单色荧光解码、多个标签,每轮次一个位置显示多种颜色,每次解码信息增加,增加了体系复杂性,而且多段标签序列占据了更多的序列可能性,当编码的微珠种类非常多时,需要生成大量的标签序列,易出现重复序列导致dna杂交特异性变差,准确度下降。
技术实现思路
1、针对现有技术中存在的上述不足,本发明提供了一种基于多色探针杂交的基因芯片编解码方法及基因芯片,以解决现有基因芯片编解码方法中效率低、成本高、对寡核苷酸链损伤大的缺点。
2、一种基于多色探针杂交的基因芯片编解码方法,包括以下步骤:
3、a)提供一种基因芯片,所述基因芯片包括:
4、k种寡核苷酸链修饰的微珠,其中,每个微珠上修饰一种寡核苷酸链,k≥1,
5、寡核苷酸链包括标签序列和探针序列, k种寡核苷酸链中的探针序列各不相同,所述探针序列用于捕获目标物,所述标签序列与探针序列一一对应;
6、b)使所述基因芯片中的k种不同序列寡核苷酸链修饰的微珠与k种m色荧光探针一起孵育,其中m≥2,
7、k种m色荧光探针中各m色荧光探针的序列与k种不同序列寡核苷酸链的标签序列一一对应且为互补序列,每种m色荧光探针上修饰有m种不同荧光颜色的荧光基团;
8、c)基于步骤b)中m色荧光探针,选择对应的荧光通道进行荧光拍照,然后判断是否能确定所述k种寡核苷酸链中的所述标签序列;
9、d)如果不能,使用碱溶液与荧光拍照后的寡核苷酸链修饰的微珠一起孵育,然后重复步骤b)和c),直到能确定所述k种寡核苷酸链中的所述标签序列。
10、标签序列是探针序列的编码id,探针序列是捕获目标物的序列,通过获知标签序列即可获知探针序列,标签序列和探针序列具有一一对应的关系。
11、使用一种具体序列的探针序列用于捕获一种目标物序列,而一种具体序列的探针序列对应于一种标签序列。实际使用时,可以根据所需捕获目标物序列的多少来设置探针序列和标签序列的种类,也就是寡核苷酸链的种类。
12、m色荧光探针上修饰有m种不同荧光颜色的荧光基团,荧光基团连接在荧光探针上的具体位置并没有特别的要求,可以连接在两端,也可以连在中间碱基上或骨架上。
13、本发明中使用的荧光基团可以是本领域常规使用的荧光基团,比如,amca、fam、rox、cy2、cy3、cy5、cy5.5、cy7。可以根据具体的情况来合理选择需要的荧光基团。
14、优选的,寡核苷酸链修饰的微珠中寡核苷酸链的5’端与微珠偶联,所述标签序列位于寡核苷酸链的5’端,所述探针序列位于寡核苷酸链的3’端。
15、寡核苷酸链与磁珠的偶联可以使用现有技术中常规的方法。更优选的,寡核苷酸链的末端修饰有氨基,微珠具有羧基修饰,寡核苷酸链通过末端修饰的氨基与微珠上的羧基反应偶联。利用氨基和羧基之间缩合反应生成酰胺键将寡核苷酸链共价耦合于微珠上。
16、微珠可以使用现有技术中常用的材质,优选的,微珠为羧基修饰的聚苯乙烯微珠或二氧化硅微珠。
17、优选的,步骤(1)中,各寡核苷酸链中含有的连续的相同的碱基的数量不超过3个,gc含量为30%-60%,发夹结构的长度不超过6个碱基,所述标签序列之间的自互补片段不超过6个碱基,并且所述标签序列与目标基因组之间的相似性低于0.05。序列相似性使用blast比对。
18、优选的,所述标签序列的长度为16-30 mer。
19、优选的,碱溶液为naoh或koh水溶液,浓度为0.01~0.2 m。碱性环境可以使dna双链变性解体,碱溶液浓度过低,双链解体不完全,碱溶液浓度过高,对dna双链造成不可逆伤害。所以,使用碱溶液与杂交后的寡核苷酸链修饰的微珠进行孵育,可以使寡核苷酸链与对应的荧光探针之间解体,以为下一次的孵育做准备。
20、本发明又提供了一种基于多色探针杂交的基因芯片,包括k种寡核苷酸链修饰的微珠,其中,每个微珠上修饰一种寡核苷酸链,k≥1,
21、寡核苷酸链包括标签序列和探针序列, k种寡核苷酸链中的探针序列各不相同。
22、优选的,寡核苷酸链修饰的微珠中寡核苷酸链的5’端与微珠偶联,所述标签序列位于寡核苷酸链的5’端,所述探针序列位于寡核苷酸链的3’端。
23、优选的,寡核苷酸链的末端修饰有氨基,微珠具有羧基修饰,寡核苷酸链通过末端修饰的氨基与微珠上的羧基反应偶联。
24、优选的,各寡核苷酸链中含有的连续的相同的碱基的数量不超过3个,gc含量为30%-60%,发夹结构的长度不超过6个碱基,所述标签序列之间的自互补片段不超过6个碱基,并且所述标签序列与目标基因组之间的相似性低于0.05。序列相似性使用blast比对。
25、本发明基于多色探针杂交的基因芯片的编解码方法解码利用多色探针读取,多个荧光基团可以在一次解码实验中全部呈现,通过多个荧光基团自由组合,可以使得一次解码实验中获得信息量增大,解码相同数量的探针所需的解码实验次数减少。使用多色荧光解码、单个标签,每轮次一个位置显示多种颜色,每次解码信息增加,体系简单,成本低;每个位置只需要一个标签序列,当编码大量微珠时,不易出现重复序列,特异性较好。
26、比如,使用2种荧光基团构建单色、双色荧光探针进行解码,在一次解码实验中,芯片上每个位置可能带上的信号有3种,分别为2种单色和1种双色,比单独使用单色的2种每次解码要多1种。如果使用3种荧光基团构建单色、双色、三色荧光探针进行解码,在一次解码实验中,芯片上每个位置可能带上的信号有7中,分别为3种单色、3种双色和1种三色,比单独使用单色的3种每次解码要多4种。如果使用4种荧光基团构建单色、双色、三色、四色荧光探针进行解码,在一次解码实验中,芯片上每个位置可能带上的信号有15种,分别为4种单色、6种双色、4种三色和1种四色,比单独使用单色的4种每次解码要多11种。可见,随着一次解码实验中使用的荧光基团数量的增加,芯片上每个位置可能带上的信号迅速增加。
27、相比较传统的单色荧光探针编解码,本发明方法可以大大减少解码次数,尤其是对于高密度生物芯片,通过合理搭配荧光基团,可以极大地提高在生产环节中确定生物芯片的解码文件时的解码效率,节约成本,同时也可以一定程度的减轻多次解码实验对生物芯片上生物信息的损伤,更有利于后续分析检测实验。
- 还没有人留言评论。精彩留言会获得点赞!