一种检测mRNA的polyA尾长的方法与流程
本发明涉及生物,具体涉及一种检测mrna的polya尾长度的方法。
背景技术:
1、在真核生物中,大多数mrna分子在3'末端被多聚腺苷酸化,形成的polya尾巴和与其结合的蛋白质有助于保护mrna免于被核酸外切酶降解。3'端加尾对于转录终止、从细胞核输出mrna和翻译也很重要。polya长度被发现和mrna的翻译与稳定性密切相关(nicholson, angela l.; pasquinelli, amy e.trends in cell biology (2019), 29(3), 191-200)。早期人们认为,polya越长越能保护mrna不被降解,但随着研究的深入,特别是针对polya的测序技术发展,人们发现真相可能不像想象中那样简单。一些研究表明,相对较短的polya通常出现在高表达的、翻译效率较高mrna中,而较长的polya则更多出现地在低丰度的、翻译效率较低的mrna中。同时,polya长度似乎与其在胞内的半衰期呈负相关。
2、mrna药物,在体外制备mrna时,通常会采用两种方法将polya添加到rna 3'端:(1)在体外转录模板上设计固定长度的polya序列,通过体外转录直接添加;(2)在转录完成后用polya聚合酶添加。由于两种方法所用酶的特性,rna产物中polya的长度不可避免地会出现参差不齐的情况。鉴于polya长度对于mrna药物的重要性,各国药监机构出台的mrna药物指导原则中均规定需要对mrna的polya长度进行严格地检测。
3、中国专利cn105734053b公开了“一种高通量测序分析polya尾长度文库的构建方法”,该专利提供了一种测定polya尾长度的建库方法,即利用试剂盒对待测的样品添加3'接头,然后转录成cdna,再利用建库试剂盒富集待测样品序列的3'端信息,进而利用高通量测序仪illuminanextseq500 sr300测定mrna的polya尾长度。但该方法并非直接测定mrna的polya尾长度,而是要经历由mrna逆转录先生成互补的cdna,再由dna聚合酶合成第二条链形成dna双链后再进行建库测序,测序过程中又需要使用dna聚合酶进行边合成边测序。由于对长的多聚a尾进行反转录或pcr聚合酶合成的过程中,逆转录酶和dna聚合酶常常都会发生误读导致产生多余的a或缺失的a,从而引入较多的测序的系统误差。而且整个流程步骤繁多,受多个试剂盒效率、人员操作熟练水平影响多,mrna样品多步骤处理后的质量下降等因素制约,都会影响最终检测结果的准确性。
4、中国专利cn110452951b公开了一种“监测mrna polya尾长度的方法及应用”,该专利提供了一种监测mrna polya尾长度的方法,该方法通过测定标准加尾反应中不同反应时间点的atp消耗量和/或ppi生成量,绘制出标准mrna加尾长度和反应时间的标准曲线,然后通过待测mrna反应液中atp消耗量,来确定mrna polya加尾长度。然而,该方法的原理是基于生化反应动力学的统计学计算间接推断polya的个数,其结果是化学反应的平均结果,难以直接判断样品中polya的长度范围,且仅适用于需要单独进行加尾反应的mrna合成或生产流程,并不适合目前工业生产中普遍采用的把polya直接放到转录模板质粒中的生成策略,并且该方法使用过程中,一旦体系中各个物料发生变化时,均会对检测结果产生影响。
5、目前其他测定mrna polya尾长度的方法还包括:northern印迹法、lc-ms法等,这些方法也有各自优点和适用的局限性。比如northern印迹法相对简单,但属于半定量方法,不够准确;lc-ms方法测分子量的精确度高,但成本昂贵,其结果也基于统计学的计算,难以直接判断样品中polya的长度范围。因此,开发一套实验流程既简单、经济、又能准确直接定量的检测mrna样品中polya尾长度范围的方法,是mrna药物研究和药物生产质检方法学面临的挑战之一。
技术实现思路
1、在一方面,本发明提供了一种检测rna polya长度的方法,包括如下步骤:
2、1)采用具rna剪切活性的dna核酶特异性剪切mrna,生成带有polya结构的rna片段以及不带有 polya结构的rna片段;
3、mrna包括天然的含有polya尾的mrna或者人工合成的含有polya尾的mrna。utr(untranslated regions)即非翻译区,是信使rna(mrna)分子两端的非编码片段。5'-utr从mrna起点的甲基化鸟嘌呤核苷酸帽延伸至aug起始密码子,3'-utr从编码区末端的终止密码子延伸至多聚a尾巴(poly-a)之前。原核生物和真核生物的mrna中都存在非编码区域(utr),但它们的长度和组成都有所不同。原核生物中,5'非翻译区通常为3至10个核苷酸残基的长度。但在真核生物中,5'非翻译区可以长达成百上千个核苷酸残基的长度。与原核生物相比,真核生物的基因组的复杂性更高,3'非翻译区的长度也不同。
4、由于mrna的3'端utr区并不固定,因此为了使检测结果更加精确,针对不同的3'端utr区,需要对特定位点的具rna剪切活性的dna核酶进行设计和/或对该剪切位点进行筛选,从中获得最优的dna核酶,本发明提供的具体3'端utr区及对应设计的dna核酶,并不限制本发明的保护范围,任何3'端utr区及任何能够特异性有效切割3'端utr区的dna核酶均可应用于本发明提供的检测方法。
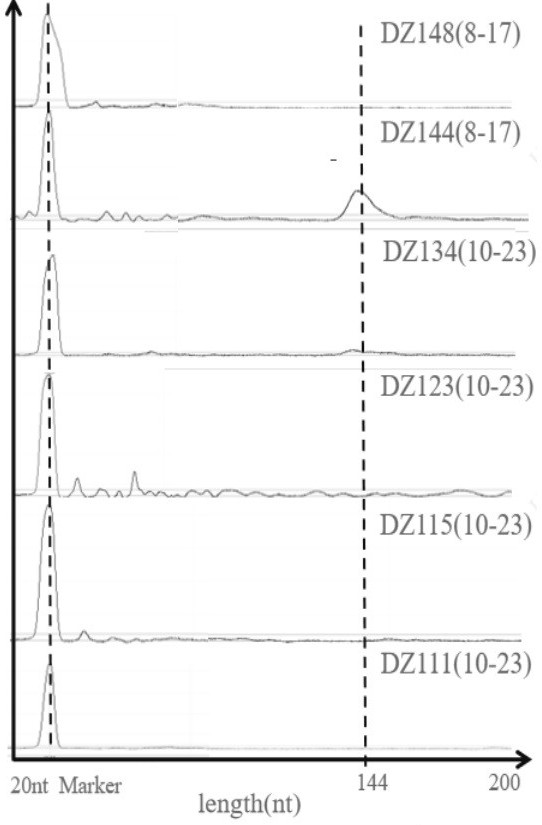
5、本技术提供的检测方法,筛选出能够特异性定点切割mrna的dna核酶,其切割位点位于3'-utr区的包括polya尾在内不超过200个碱基的位点。当切割后的带有polya结构的rna片段碱基数超过200时,毛细管电泳仪的检测结果精度会随着碱基个数的增加而逐渐下降。
6、在一些实施方式中,剪切酶为能够特异性切割rna的脱氧核酶,具体地,可以是脱氧核酶dnazyme(10-23)、dnazyme(8-17)。在一些实施方式中,剪切酶为dnazyme(10-23),在一些实施方式中,剪切酶为dnazyme(8-17)。在一些实施方式中,dnazyme(10-23)或dnazyme(8-17)的两臂臂长可以在7~15 nt范围,本实施方案中优选为14 nt。本技术提供的核酶并不对本技术进行限制。任何与本技术提供的utr序列不相同或相似的序列也可通过本技术提供的方法筛选出合适的核酶并进行下一步的检测。
7、在一些实施方式中,mrna 的3'端的utr区包括seq id no: 1所示序列,剪切酶为dnazyme(8-17)或dnazyme(10-23)。
8、5'-ctcgagctggtactgcatgcacgcaatgctagctgcccctttcccgtcctgggtaccccgagtctcccccgacctcgggtcccaggtatgctcccacctccacctgccccactcaccacctctgctagttccagacacctcccaagcacgcagcaatgcagctcaaaacgcttagcctagccacacccccacgggaaacagcagtgattaacctttagcaataaacgaaagtttaactaagctatactaaccccagggttggtcaatttcgtgccagccacaccctggagctagc-3'(seq id no: 1)
9、seq id no: 1所示序列长295 nt,是一种较为常见的utr序列,实际应用时mrna样品的3'utr也可能有所不同,可进行类似的筛选流程获得适合的dna核酶。在一些实施方式中,可以位于3'端utr区的末端并连接polya。
10、在一些实施方式中,3'端utr区的末端连接的polya长度为110 nt,如seq id no:2所示序列:
11、5'-aaaaaaaaaaaaaaaaaaaaaaaaaaaaaagcatatgactaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaa-3'(seq id no: 2)
12、在一些实施方式中,3'端utr区的末端连接的polya长度为92 nt,如seq id no: 3所示序列:
13、5'-aaaaaaaaaaaaaaaaaaaaaaaaaaaaaagcatatgactaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaa-3'(seq id no:3)
14、在一些实施方式中,3'端utr区的末端连接的polya长度为73 nt,如seq id no: 4所示序列:
15、5'-aaaaaaaaaaaaaaaaaaaaaaaaaaaaaagcatatgactaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaa-3'(seq id no: 4)
16、在一些实施方式中,3'端utr区的末端连接的polya长度为32 nt,如seq id no: 5所示序列:
17、5'-aaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaa-3'(seq id no:5)
18、在一些实施方式中,对连接polya的utr区进行切割,优选地,切割后的带有polya片段的rna片段不大于200 nt。
19、本发明采用的脱氧核酶可以特异性切割mrna的某一固定位点,因此切割后得到的带有polya结构的rna片段中的utr序列部分是固定和已知的,通过毛细管电泳精确测得了片段总长度范围后,减去片段中utr固定序列的长度,即可以得到mrna中polya的长度。另外,由于本发明采用的剪切rna样品的酶是dna核酶,在切割反应之后很容易用dnase i消化除去,因而可以最小限度减少蛋白酶类(常可能含有rna酶残留从而降解rna)或rna核酶(也是短小的rna)对后续分析切割后的短片段rna的干扰,从而保障了片段长度分析的高精度要求。
20、在一些实施方式中,mrna 的3'端的utr区的序列如seq id no: 1所示;进一步地,剪切酶为dnazyme(8-17);在一些实施方式中,dnazyme(8-17)的序列如seq id no: 11所示。本技术采用的如seq id no: 1所示的utr区是一种较为常见的utr序列,任何采用如seqid no: 1所示的utr序列或类似的utr序列均可使用本技术提供的seq id no: 11所示的核酶进行切割。本技术提供的utr序列及对应的核酶并不对本技术进行限制。任何与本技术提供的utr序列不相同或相似的序列也可通过本技术提供的方法筛选核酶并进行下一步的检测。
21、在一些实施方式中,dnazyme(8-17)的序列如seq id no: 11所示,任何utr区包含如seq id no: 11所示dnazyme(8-17)核酶靶向序列的加尾mrma均可使用dnazyme(8-17)所示的核酶进行切割,靶向序列如seq id no: 13所示。
22、在一些实施方式中,根据mrna的3'端utr区设计并筛选剪切酶,剪切酶为能够特异性切割mrna的酶;
23、2)提取带有polya结构的rna片段;
24、本发明提供的检测mrna polya长度的方法,可应用于各种长度的mrna,尤其是长度较长,达到几百至数千的mrna,剪切后的带有polya结构的rna片段与不带有polya结构的rna片段一般情况下具有显著的长度差异,可通过各种常见方式(如磁珠法,柱层析法,电泳法等等)对带有polya结构的rna片段进行提纯。在一些实施方式中,采用磁珠法提取带有polya结构的rna片段。在一些实施方式中,采用偶联了oligo(dt)的1μm顺磁性微球,从纯化的总rna混合物中分离polya+ rna片段。磁性分离技术可从小体积样品中分离出完整的mrna,避免了mrna沉淀的步骤。整个操作过程可在1 h内完成
25、3)采用毛细管电泳检测读取所述带有 polya的所述rna片段的长度。
26、毛细管电泳(capillary electrophoresis,ce)技术是一种高灵敏和高精度的基于电荷和分子量大小差异分离分析生物分子(核酸、蛋白质、多肽、糖等)的方法。由于毛细管电泳可以达到很高的精度(对短片段核酸可以达到1nt的精度)和灵敏度,常被用于精确分析相对较短的dna或rna片段,如引物扩增产物或rna小分子。利用电场将带电生物分子在带电毛细管中移动,可分离不同大小、电荷和形状的分子。mrna是由核苷酸线性连接而成的、带负电荷性质的单链生物大分子,因此在电场作用下,基于mrna的大小、形态和电荷密度的不同可在毛细管中移动并差异分离。本发明采用毛细管电泳检测直接读取剪切后的含polya尾的短片段,再进行简单的计算即可得到polya尾的长度数值。
27、在另一方面,本发明还提供了一种mrna polya长度检测盒,其包括上述的脱氧核酶dnazyme(8-17),dnazyme(8-17)的序列如seq id no: 11所示。
28、本发明提供的rna polya长度的检测方法,首先根据mrna的3'端utr区设计并筛选能够特异性切割rna的剪切酶;然后采用剪切酶处理mrna,生成带有polya结构的rna片段以及不带有 polya结构的rna片段;接着提取带有polya结构的rna片段;最后采用毛细管电泳检测读取带有 polya的rna片段的长度。该方法不仅简单、经济、高效,而且可以精确读数,直接测定出 mrna样品中polya的长度范围。
- 还没有人留言评论。精彩留言会获得点赞!