基于全卷积网络的脑部MRI肿瘤分割方法与流程
本发明属于医学图像处理技术领域。具体来说,涉及一种以分割出脑部核磁共振成像(mri)中的肿瘤为目的的基于全卷积网络的脑部mri肿瘤分割方法。
背景技术:
脑肿瘤是患病率和死亡率最高的肿瘤之一。mri是一种在临床上评估脑肿瘤特别有效的手段。对脑肿瘤和肿瘤内部结构的精确分割不仅对辅助医生的治疗计划十分重要,而且也对接下来的随访评估十分重要。然而,人工分割方法是十分耗时的,而且容易受到主观因素的影响导致误分割。因此,寻找一种精确的脑部肿瘤分割方法是必需的。然而,由于肿瘤区域的形状、结构和位置都是高度可变的,此外,由于成像器件和环境等原因,导致mri图像本身质量也存在一些问题,如同一患者在不同仪器设备上成像的灰度分布不一致等。所以找到一种高精度的肿瘤分割方法十分困难。
当前,有些学者提出了一系列概率模型用于脑肿瘤分割领域。这些模型通常包含一个与观测数据和先验模型一致的似然函数。受到形状和连续性的约束,有学者将肿瘤区域看成是一种异常区域,从而将它从正常区域中分割出来。用马尔科夫随机场结合肿瘤区域附近的区域也可以实现光滑的图像分割。如zhang等人提出了一种基于马尔科夫随机场的分割方法,先利用基于直方图的似然函数估计法初步分割,然后利用基于马尔科夫随机场的分割方法再次分割。根据menze等人的调查,生成模型对潜在数据也有较好的生成效果,但是它不能很好地将先验知识应用到适当的概率模型中。
另一类分割方法直接从已有数据中学习数据分布,这类方法不依赖与某种特定的模型。这类方法认为图像中的像素点都是独立同分布的,而且像素间的信息也会被作为特征进行训练。所以,一些孤立的像素点和一些很小的类簇会被错误分类。为了解决这个问题,一些学者在条件随机场中加入了一个概率性预测。近年来,随机森林被成功地应用到脑肿瘤分割当中。由于随机森林比较适合处理多分类问题和大特征向量,所以它被广泛应用于分割问题。tustison等人提出了一种基于随机森林的二阶分割框架,用第一阶段的分割器的输出结果来提高第二阶段的分割结果。geremia等人提出了一种空间自适应的随机森林模型用来分割脑部肿瘤。深度学习自2006年由hinton等人提出以来,在很多研究领域得到了成功的应用。深度学习技术源于神经网络学习技术,神经网络技术通过模拟人脑的运行模式来建立学习结构和模型。虽然神经网络有很多缺点,但是hinton等一批人坚持了他们的研究,提出了许多新的方法解决深度网络难以训练,容易过拟合的问题。如采用新的线性修正单元(relu)激活函数、数据池化处理、丢弃(dropout)训练法等,这些新的方法有效的解决了深度神经网络容易过拟合的问题,在语音识别、图像识别等领域取得了成功的应用。pereira等人在2015年提出了一种基于卷积神经网络的方法对脑部mri图像进行肿瘤分割,将图像分割问题转换为对像素点的分类,并且取得了比一般方法更好的分割效果。jonathanlong等人在2015年提出了全卷积网络用于图像语义分割,这是一种端对端的分割模型,是深度学习用于图像分割领域的一个重大突破。本发明将全卷积网络用于脑部mri肿瘤分割。
技术实现要素:
为了对脑部肿瘤区域和肿瘤内部结构进行精确分割,本发明设计了一种脑部肿瘤分割方法,将脑部肿瘤分割这个任务分成两步,训练两个全卷积网络,第一个网络用于对脑部肿瘤图像进行初步分割,检测脑部肿瘤所在区域,第二个网络用来对脑部肿瘤区域的内部结构进行精细分割。实验表明,这种分割方法有较好的分割效果。该方法包括:
训练粗分割全卷积网络模型,用于检测原始mri图像中的肿瘤区域;
训练精细分割全卷积网络模型,用于对肿瘤区域的内部结构进行精细分割;
利用训练好的两个全卷积网络模型对输入脑部mri图像进行分割。
其中,根据所述用处理过的训练样本训练粗分割全卷积网络模型,包括:
对脑部mri图像数据集进行“去黑色背景”预处理;
设计粗分割全卷积网络结构:为了充分学习脑部mri图像的肿瘤特征,本网络采用五层池化结构,池化层1、2的前面各有两层卷积层,池化层3、4、5的前面各有三层卷积层,池化层5后面有三层卷积层;原始图像经过五层池化处理之后,尺寸变为原来的1/32,此时得到的是包含高维特征的热图,经过32倍上采样(反卷积)和裁剪处理得到与原始图像尺寸大小相同图像,再与标签图像进行比较计算出损失值,最后通过反向传播调整各层之间的权值与偏置参数,这个网络称作fcn-32s;直接进行32倍上采样得到的结果往往非常粗糙,为了得到更加精细的分割效果,结合池化层3和池化层4的特征图谱,将五层池化处理后的热图进行2倍上采样,与四层池化处理后的热图进行求和,再经过16倍上采样就得到与原始图像尺寸大小相同的图像,最后经过反向传播调整网络参数,这个网络称作fcn-16s;同理,将上一步求和后的热图进行2倍上采样与三层池化处理后的热图进行求和,再经过8倍上采样就得到与原始图像尺寸大小相同的图像,最后经过反向传播调整网络参数,这个网络称作fcn-8s;
用预处理后的脑部mri图像作为训练集训练粗分割全卷积网络模型,先训练fcn-32s网络,再用训练好的参数去初始化并训练fcn-16s网络,最后用训练好的参数去初始化并训练fcn-8s网络。
其中,根据所述用检测出的肿瘤区域作为训练样本训练精细分割全卷积网络模型,包括:
根据专家分割模板提取原始数据集中的肿瘤区域;
设计精细分割全卷积网络结构,将粗分割全卷积网络结构进行修改,由于肿瘤区域尺寸较小,这里采用两层池化结构就能取得较好的分割效果,在未分类的特征图谱之前的每层卷积层之后都加了一个规范化层和归一化层,用来消除不同肿瘤图像之间的灰度差异性;
用提取出的肿瘤区域作为训练集训练精细分割全卷积网络模型。
其中,根据所述用训练好的全卷积网络模型对输入脑部mri图像进行分割,包括:
对输入脑部mri图像进行“去黑色背景”预处理;
用训练好的粗分割全卷积网络模型对输入脑部mri图像进行粗分割,检测出肿瘤区域;
用训练好的精细分割全卷积网络模型对肿瘤区域内部结构进行精细分割。
附图说明
附图用来提供对本发明技术方案的进一步理解,并且构成说明书的一部分,与本申请的实施例一起用于解释本发明的技术方案,并不构成对本发明技术方案的限制。
图1基于全卷积网络的脑部mri肿瘤分割方法流程图;
图2图像预处理示意图;
图3fcn-32s网络结构;
图4fcn-16s网络结构;
图5fcn-8s网络结构;
图6粗分割结果;
图7肿瘤区域提取过程示意图;
图8精细分割网络结构;
图9精细分割结果;
图10将分割出的肿瘤区域还原到原始图像中的结果;
图11其他实施例的分割结果。
具体实施方式
以下将结合附图及实施例来详细说明本发明的实施方式,借此对本发明如何应用技术手段来解决技术问题,并达成技术效果的实现过程能充分理解并据以实施。
本申请实施例的基于全卷积网络的脑部mri肿瘤分割方法,用于脑部mri肿瘤分割。
如图1所示,本申请实施例的基于全卷积网络的脑部mri肿瘤分割方法,主要包括以下步骤:
步骤1训练粗分割全卷积网络模型,用于检测原始图像中的肿瘤区域;
步骤2训练精细分割全卷积网络模型,用于对肿瘤区域的内部结构进行精细分割;
步骤3利用训练好的两个全卷积网络模型对输入脑部mri图像进行分割。
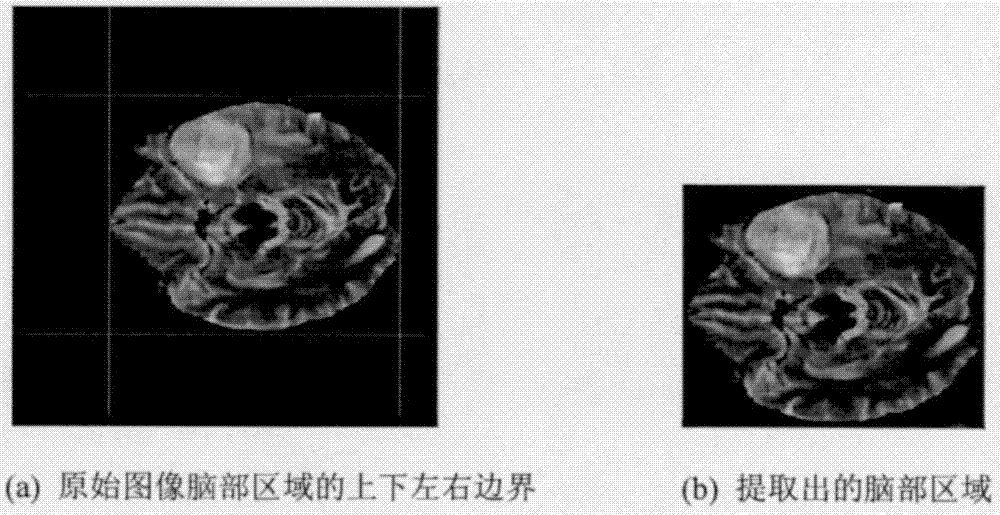
本申请实施例中,根据所述对脑部mri图像数据集进行预处理。本实施例用到的数据集来自brats2015数据库(https://www.smir.ch/brats/start2015),数据库中的脑部mri图像分为四个模态,分别是t1、t1c、t2和flair,本实施例用t2模态的图像进行训练和测试。原始数据集中的图像四周有较大的黑色背景区域,而这些区域中并不包含有用信息,我们将其去掉可以减少图像尺寸,从而提高全卷积网络的运算速度。
如图2所示,我们可以确定脑组织的上下左右边界,从而可以将脑组织部分提取出来,去除原始图像周围的黑色边框。
本申请实施例中,根据所述训练粗分割全卷积网络模型,用于对脑部肿瘤区域的初步分割,确定肿瘤区域在原始mri图像中的大致位置和初始轮廓。粗分割全卷积网络用预处理后的mri图像作为训练样本。粗分割全卷积网络分为三个训练阶段,即先训练fcn-32s,再训练fcn-16s,最后训练fcn-8s。网络结构如图3,图4和图5所示。
具体训练步骤是:先用vgg16的参数微调训练fcn-32s网络;再用训练好的fcn-32s参数初始化fcn-16s网络,进行微调训练;最后用训练好的fcn-16s参数初始化fcn-8s网络,进行微调训练。此时fcn-8s训练出来的网络模型就是最终的粗分割全卷积网络模型,用来对输入脑部mri图像进行粗分割。粗分割的结果如图6所示。
本申请实施例中,根据所述训练精细分割全卷积网络模型,用于对上一步检测出的肿瘤区域内部结构进行精细分割。此网络是用原始脑部mri图像中的肿瘤区域作为训练样本。提取肿瘤区域的方法和图像预处理的方法类似,先找出手工分割模板中肿瘤区域的上下左右边界,将肿瘤区域提取出来,然后将原始mri图像中对应区域提取出来。提取过程如图7所示。由于肿瘤区域尺寸较小,所以在粗分割网络的基础上进行了修改,将池化层由五层改为两层,另外在卷积层之后加上规范化层和归一化层,网络结构如图8所示。
本申请实施例中,根据所述利用训练好的全卷积网络模型对输入图像进行分割。先对输入脑部mri图像进行预处理,去除黑色背景区域;然后利用粗分割全卷积网络模型对脑部肿瘤区域进行初步分割,检测原始图像中肿瘤区域,将肿瘤区域提取出来;再利用精细分割全卷积网络模型对提取出来的肿瘤区域内部结构进行精细分割,最后将分割出的肿瘤区域还原到原始mri图像中。精细分割的结果如图9所示,将分割出的肿瘤区域还原到原始mri图像中的结果如图10所示。本发明对其他实施例的分割效果如图11所示。
为了检验本发明对脑部mri肿瘤区域的分割精度,下面分别对肿瘤区域的三个区域用三个评价指标进行分析,并与其他分割方法进行比较。本发明采的数据集中的脑部mri肿瘤图像中的肿瘤分为四种类型,分别是水肿、坏死、未增强肿瘤和增强肿瘤。在说明书附图中我们分别对这四种类型分别用绿、红、蓝和黄色进行区分。在计算评价指标时将这四种类型分为三个区域:全部区域(包括所有四种类型)、核心区域(包括坏死,未增强和增强肿瘤)、增强肿瘤区域。评价指标采用骰子相似系数(dsc,dicesimilaritycoefficient),积极预测值(ppv,positivepredictivevalue)和敏感度(sensitivity)。其中dsc定义为:
ppv定义为:
sensitivity定义为:
其中,tp代表本发明分割出的区域与专家手工分割模板的重叠区域,fp代表本发明未分割出的专家手工分割模板中的区域,fn代表本发明分割出的多余部分。与其他分割方法的比较如表1所示。
表1本发明与其他分割方法的评价指标对比
通过对比,证明本发明有较好的分割效果,并且本发明对增强区域的分割效果最好,另外,本发明对所有区域的dsc参数指标都有最好效果。
- 还没有人留言评论。精彩留言会获得点赞!