一种植物细胞三维重构方法
1.本发明涉及三维图像重构领域,尤其涉及一种植物细胞三维重构方法。
背景技术:
2.植物细胞的三维重构对研究植物细胞组织结构和模拟植物细胞生长过程都具有重要作用。因为植物细胞的形态及其结构均难以用肉眼观察得到,需要借助电子计算机断层扫描技术[1]获得植物细胞的连续切片图像,或是使用透射电子显微镜[2]获得植物细胞的显微图像来进行三维重构。目前业界常用的植物细胞三维重构软件有visualize、amira、review和image j等。通过上述三维重构软件重构出来的通常为难以直接动态扩展的三维植物细胞模型且所包括的可计算的形态参数较少,而动态的或能动态扩展的三维植物细胞模型和相对较丰富的形态参数是植物细胞生长形态变化过程数值模拟的不可或缺的部分。
技术实现要素:
[0003]
本发明设计了一种用于生成能动态扩展的三维植物细胞模型的植物细胞三维重构方法。该方法不仅可支持能动态扩展的多类植物细胞的三维模型的重构,而且提供了可用于植物细胞生长模拟中形态变化可视化的数据结构。本发明所设计的植物细胞三维重构方法的技术方案如下所述。
[0004]
一种植物细胞三维重构方法,所述方法包括:
[0005]
根据得到的目标植物组织细胞连续切片图像的分辨率和目标植物细胞的大小建立三维笛卡尔坐标系;
[0006]
根据目标植物细胞所需采集的数据信息和所需计算的参数,设计用于存储对应数据的数据结构;
[0007]
基于三维笛卡尔坐标系对目标植物细胞的连续切片图像进行采样,在每张切片图像中使用平行于x轴或y轴的等间距线对目标植物细胞细胞壁的内外两侧分别进行点采样,并按照点采样的顺序将采样点的xyz轴坐标值以及采样点的序号对应存入设计好的数据结构内部;
[0008]
利用采集和存入完成的数据结构分别计算目标植物细胞的表面积、重心、体积和细胞壁厚度参数。
[0009]
其中,所述根据得到的目标植物组织细胞连续切片图像的分辨率和目标植物细胞的大小建立三维笛卡尔坐标系具体为:
[0010]
确定目标细胞从首次在切片图像中完整出现到完全消失的前一张所覆盖的n张切片图像,首张目标植物细胞切片图像的z轴坐标值为1
×
l,目标植物细胞首次出现的切片图像的图像序号为1,再乘上比例值l,最后一张目标植物细胞切片图像的z轴坐标值为n
×
l,即目标植物细胞在切片图像中完全消失的前一张切片图像的图像序号为n,再乘上比例值l;
[0011]
将起始切片图像放置于三维笛卡尔坐标系的第一象限中,得到的切片图像分辨率
为w
×
h,其中切片图像边长为w像素所对应的边与x轴平行且水平距离为1
×
l,边长为h像素所对应的边与y轴平行且水平距离为1
×
l。
[0012]
进一步地,所述用于存储对应数据的数据结构为:
[0013]
一级字段信息:params_ovrl整体参数、array_pcells[]各植物细胞数据;其中一级字段的params_ovrl含有二级字段:name_ptype植物类别名称、num_total细胞总数量、name_ctypes[]细胞类别名称、num_ctypes[]每类细胞的数量、area_tar_ctypes[]每类细胞的目标表面积和volm_tar_ctypes[]每类细胞的目标体积,同时一级字段的array_pcells[]含有二级字段:nctpid细胞类别名称序号、area_cur当前细胞面积、volm_cur当前细胞体积、avethk_cellwall_cur当前细胞细胞壁平均厚度、cengra当前细胞重心点、outer细胞外侧数据和inner细胞内侧数据;其中二级字段的outer和inner均包含三级字段:points[]点、edges[]边和triaglfcs[]三角面,同时:三级字段points[]包含四级字段:ptid点的序号、cirid切片图像序号、xval、yval和zval;三级字段edges[]包含四级字段:edgid边序号、ptid1起点端点序号和ptid2末端端点序号;三级字段triaglfcs[]包含四级字段:trifclid三角面序号、edgid1-edgid3三角边的序号。
[0014]
其中,所述在每张切片图像中使用平行于x轴或y轴的等间距线对目标植物细胞细胞壁的内外两侧分别进行点采样具体为:
[0015]
根据目标植物细胞出现在其所覆盖的切片图像上的大小来确定等间距线方向应选取平行于x轴或y轴;
[0016]
采集目标植物细胞的内外侧点的xy轴坐标值,等间距线的间距大小根据目标植物细胞在切片图像上表现的大小来调整;
[0017]
目标植物细胞与等间距线在细胞左上角处通过计算得到的第一个内或外侧点为1号采样点,按顺时针排序对目标细胞的内外侧进行点采样,下一张切片图像中目标植物细胞的第一个内或外侧点序号为上一张最后一个内或外侧点的序号值加1,并按顺时针排序对目标细胞的内外侧进行采点;
[0018]
内侧点为等间距线与目标植物细胞的细胞壁内侧的交点,外侧点由两相邻细胞的内侧点来确定;
[0019]
分别采集并记录每个内外侧点的点序号、xyz轴数值,存入对应细胞内外侧数据的三级字段points[]中的四级字段:ptid、xval、yval和zval。
[0020]
进一步地,所述按照采点顺序将所采点的xyz轴坐标值以及点的序号对应存入设计好的数据结构内部具体为:
[0021]
将得到的三角面和对应的三条边的数据信息分别存入数据结构的outer和inner内三级字段triaglfcs[]中的四级字段trifclid、edgid1、edgid2和edgid3;
[0022]
计算单个外侧三角面的面积,通过遍历操作计算目标植物细胞的外侧所有单个三角面的面积,将所有单个三角面的面积进行累加得到目标植物细胞外侧的表面积,并将得到的数据存入数据结构的二级字段area_cur中;
[0023]
使用数据结构中细胞外侧数据中四级字段xval、yval和zval的值分别进行求和操作,再对得到的xyz三轴坐标值各自进行取平均值操作,得到的三个结果分别对应着目标植物细胞外侧细胞重心点的xyz三轴坐标值;再使用细胞内侧数据中四级字段xval、yval和zval的值进行同样操作,得到目标植物细胞内细胞重心点的xyz三轴坐标值,对细胞外侧和
内侧的两个细胞重心点的xyz三轴坐标值进行取平均值操作,将目标植物细胞划分为若干个四面体,并通过先计算单个四面体的体积再进行累加的方式得到目标植物细胞的体积。
[0024]
其中,所述计算目标植物细胞的表面积、重心、体积和细胞壁厚度参数为:
[0025]
使用细胞外侧点所有四级字段xval、yval和zval的值和与重心点p
cg
的xyz三轴坐标值分别进行求点到点之间距离的计算操作,再对所有计算得到的值取平均值,得到的d
ot
值为目标植物细胞所有外侧点到该细胞重心点的平均距离;
[0026]
使用步骤细胞内侧点所有四级字段xval、yval和zval的值和与重心点的xyz三轴坐标值进行计算操作,再对所有计算得到的值取平均值,得到的d
in
值为目标植物细胞所有内侧点到该细胞重心点的平均距离;
[0027]
用目标植物细胞所有外侧点到该细胞重心点的平均距离减去目标植物细胞所有内侧点到该细胞重心点的平均距离,得到的d
cw
值为目标植物细胞细胞壁的平均厚度,并将得到的数据存入数据结构的avethk_cellwall_cur中。
[0028]
本发明提供的技术方案的有益效果是:
[0029]
1)本发明所设计的植物细胞三维重构方法可用于植物细胞生长过程中形态变化的多分辨率的连续观测。
[0030]
2)本发明所设计的植物细胞三维重构方法的重构成本较低,重构时间较短,计算资源要求较少。
[0031]
3)本发明所设计的植物细胞三维重构方法可用于辅助分析植物细胞壁的木质化程度及进一步的细胞板形成建模。
附图说明
[0032]
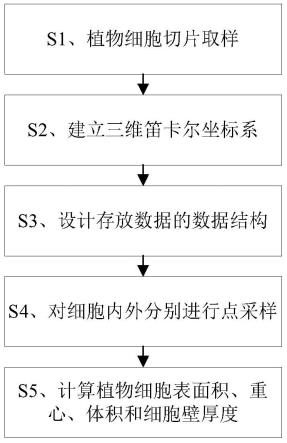
图1为一种植物细胞三维重构方法的流程图;
[0033]
图2为三维笛卡尔坐标系建立的示意图;
[0034]
图3为多植物细胞的数据结构的展开图;
[0035]
图4为目标植物细胞等间距线设置的示意图;
[0036]
图5为目标植物细胞三维点采样计算的示意图。
具体实施方式
[0037]
为使本发明的目的、技术方案和优点更加清楚,下面对本发明实施方式作进一步地详细描述。
[0038]
一种植物细胞三维重构方法,如图1所示,该方法包括如下步骤(1)至步骤(5):
[0039]
(1)首先对植物组织的茎部(根部和叶片)进行取样,样本大小规格需在40cm
×
40cm
×
40cm以内,然后利用高分辨率ct扫描仪器对目标植物组织样本进行扫描取样,以此得到m张高分辨率的目标植物组织细胞连续切片图像;
[0040]
(2)如图2所示,根据步骤(1)得到的目标植物组织细胞连续切片图像分辨率和目标植物细胞的大小建立三维笛卡尔坐标系;
[0041]
(3)如图3所示,根据目标植物细胞所需采集的数据信息和所需计算的参数,设计
用于存储对应数据的数据结构;
[0042]
(4)如图4和图5所示,基于步骤(2)建立好的三维笛卡尔坐标系对目标植物细胞的连续切片图像进行点采样,在每张切片图像中使用平行于x轴或y轴的等间距线对目标植物细胞细胞壁的内外两侧分别进行点采样,并按照采样顺序将采样点的xyz轴坐标值以及点的序号对应存入步骤(3)中设计好的数据结构里面;
[0043]
(5)利用步骤(4)采集和存入完成的数据结构分别计算目标植物细胞的表面积、重心、体积和细胞壁厚度参数。
[0044]
进一步地,如图2所示,该步骤(2)根据步骤(1)得到的植物细胞连续切片图像分辨率和目标植物细胞的大小建立三维笛卡尔坐标系包含如下步骤(2.1)至步骤(2.2):
[0045]
(2.1)在高分辨率ct扫描仪器扫描完成后得到的m张植物细胞连续切片图像中,实际的目标植物细胞的大小和扫描的目标植物细胞的大小一般都以μm为单位,它们两者之间存在一定的比例值l。其中体素是数字数据于三维空间中分割的最小单位,像素是二维数字图像中分割的最小单位,令目标植物细胞切片所在三维空间中的1体素为一个1μm
×
1μm
×
1μm的立方体,那么一张切片图像的厚度为1μm,切片图像中的1像素对应的是一个1μm
×
1μm的正方形。每张切片图像中都包含着相同数量的像素点。然后按如下流程选择需要进行三维重构的单类型目标细胞:
[0046]
首先使用windows操作系统自带的照片查看器对步骤(1)得到的m张连续切片图像进行判读选取有效的植物细胞,假设单个目标植物细胞首次出现在m张切片图像中的第g张,并且是一个完整的细胞形态,则此张首次出现完整的单个目标植物细胞的切片图像的图像序号设置为1。该目标植物细胞的形态面积在照片查看器从第g张切片图像向后开始连续滚动查看的情况下应表现为先逐渐变大再逐渐缩小直至消失的一个过程,需要满足从第g张切片图像开始,该目标植物细胞的形态面积需要大于上一张切片图像目标植物细胞的形态面积,且当该目标植物细胞的形态面积到达一个极大值时,该目标植物细胞的形态面积会开始逐渐缩小,直至消失在切片图像中。由此确定该目标细胞从首次在切片图像中完整出现到完全消失的前一张所覆盖的n(通常目标细胞为完整细胞的情况下0《n≤m)张切片图像,其中首张目标植物细胞切片图像的z轴坐标值为1
×
l,因目标植物细胞首次出现的切片图像的图像序号为1,再乘上比例值l,最后一张目标植物细胞切片图像的z轴坐标值为n
×
l,即目标植物细胞在切片图像中完全消失的前一张切片图像的图像序号为n,再乘上比例值l。
[0047]
(2.2)将步骤(2.1)中的起始切片图像放置于图2中三维笛卡尔坐标系的第一象限中,假设得到的切片图像分辨率为w
×
h,其中切片图像边长为w像素所对应的边与x轴平行且水平距离为1
×
l(μm),边长为h像素所对应的边与y轴平行且水平距离为1
×
l(μm)。
[0048]
更进一步地,参见图3,该步骤(3)中d3d_pcells(三维植物细胞数据)含有两个一级字段:params_ovrl(整体参数)和array_pcells[](各植物细胞数据),其中params_ovrl含有以下二级字段信息:name_ptype(植物类别名称)、num_total(细胞总数量)、name_ctypes[](细胞类别名称)、num_ctypes[](每类细胞的数量)、area_tar_ctypes[](每类细胞的目标表面积)和volm_tar_ctypes[](每类细胞的目标体积),同时array_pcells[]含有以下二级字段信息:nctpid(细胞类别名称序号)、area_cur(当前细胞面积)、volm_cur(当前细胞体积)、avethk_cellwall_cur(当前细胞细胞壁平均厚度)、cengra(当前细胞重
心点)、outer(细胞外侧数据)和inner(细胞内侧数据),其中二级字段的outer和inner均需分别包含如下相同结构的三级字段:points[](点)、edges[](边)和triaglfcs[](三角面),同时:
①
三级字段points[]均需包含如下四级字段:ptid(点的序号)、cirid(切片图像序号)、xval(x轴坐标值)、yval(y轴坐标值)和zval(z轴坐标值);
②
三级字段edges[]均需包含如下四级字段:edgid(边序号)、ptid1(起点端点序号)和ptid2(末端端点序号);
③
三级字段triaglfcs[]均需包含如下四级字段:trifclid(三角面序号)、edgid1(其三角边边1的序号)、edgid2(其三角边边2的序号)和edgid3(其三角边边3的序号)。
[0049]
更进一步地,该步骤(4)对每层切片图像中目标植物细胞的内外两侧分别进行点采样包含如下步骤(4.1)至步骤(4.3):
[0050]
(4.1)首先需要将目标植物细胞的类型名称以及该植物细胞的序号存入二级字段nctpid中(细胞类型名称:目标植物细胞属于什么类型细胞,序号:目标植物细胞采集顺序的序号),以便用于后续在该植物细胞生长模拟中形态变化的可视化。
[0051]
(4.2)如图4(a)为将单张切片图像放置于图2三维坐标系的xoy二维平面坐标系中进行点采样,图4(b)为对图4(a)的单张切片图像中单个目标植物细胞进行局部放大的示例图,根据目标植物细胞出现在其所覆盖的切片图像上的大小来确定等间距线方向应选取平行于x轴或y轴(注:等间距线的方向与x轴平行还是与y轴平行,取决于哪个方向的等间距线能够与切片图像中单个目标植物细胞的细胞壁内外侧产生更多如图5(a)中的内外侧点,如平行于x轴的等间距线与单个目标植物细胞的细胞壁内外侧产生的内外侧点数多于平行于y轴的等间距线,则选取平行于x轴的等间距线,反之则选取平行于y轴的等间距线,若两者相等则任选其一即可),其中等间距线的作用为采集目标植物细胞的内外侧点的xy轴坐标值,等间距线的间距大小根据目标植物细胞在切片图像上表现的大小来调整(单个目标植物细胞在切片图像中形态面积的范围内一般需包含六条以上等间距线)。
[0052]
(4.3)如图5(a)所示,单个目标植物细胞的点采样顺序如下:目标植物细胞与等间距线在该细胞左上角处通过计算得到的第一个内(外)侧点为1号采样点,按顺时针排序对目标细胞的内外侧进行点采样(图5(a)中白色点为内侧点,黑色为外侧点),依此类推,下一张切片图像中目标植物细胞的第一个内(外)侧点的序号为上一张最后一个内(外)侧点的序号值加1,并按顺时针排序对目标细胞的内外侧进行点采样。如图5(b)所示为图5(a)中黑色方框处放大示意图,其中内侧点为等间距线与目标植物细胞的细胞壁内侧的交点,外侧点由两相邻细胞1和细胞2的内侧点来确定,其中细胞1和细胞2外侧点的y轴坐标值分别为和均由细胞1内侧点的y轴坐标值与细胞2内侧点的y坐标值相加再求均值而得到,即x轴坐标值因处于同一条等间距线上而相等,z轴坐标值为当前切片图像的图像序号乘上比例值l。需要分别采集并记录每个内外侧点的点序号、xyz轴数值,存入步骤(3)中对应细胞内外侧数据的三级字段points[]中的四级字段:ptid、xval、yval和zval。
[0053]
更进一步地,该步骤(5)利用步骤(4.2)采集和存入完成的数据结构分别计算目标植物细胞的表面积、重心、体积和细胞壁厚度参数包含如下步骤(5.1)至步骤(5.5):
[0054]
(5.1)需要将目标植物细胞的外(内)侧表面分割成若干个三角面来表示以便于相关参数的计算,这里使用步骤(4.2)存入完成后的数据结构中outer和inner中三级字段
points[]的四级字段成员数据信息进行三角剖分计算,将得到的三角面和对应的三条边的数据信息分别存入步骤(3)中数据结构的outer和inner内三级字段triaglfcs[]中的四级字段trifclid、edgid1、edgid2和edgid3;
[0055]
(5.2)目植物细胞的表面积采用外侧表面积的计算流程:
①
首先计算步骤(5.1)中单个外侧三角面的面积,即使用三角面三个点的坐标值计算对应三条边的长度和其中其中sqrt为平方根函数,1≤r≤n_tri,n_tri为三角面的数量,i∈{1,2,3}为第r个三角面中的一条边,pid(r,i,1)和pid(r,i,2)分别为第r个三角面中第i条边的两个端点的ptid1值和ptid2值,并使用海伦公式计算第r个三角面的表面积:的ptid1值和ptid2值,并使用海伦公式计算第r个三角面的表面积:其中
②
其次通过遍历操作计算目标植物细胞的外侧所有单个三角面的面积,
③
然后将所有单个三角面的面积进行累加即可得到目标植物细胞外侧的表面积,并将得到的数据存入步骤(3)中数据结构的二级字段area_cur中;
[0056]
(5.3)目标植物细胞的重心点p
cg
=(x
cg
,y
cg
,z
cg
)的坐标值的计算流程为:分别计算出外侧细胞的重心,然后取平均值。其中先使用步骤(4.2)存入完成后的数据结构中细胞外侧数据中四级字段xval、yval和zval的值分别进行求和操作,再对其得到的xyz三轴坐标值各自进行取平均值操作,取平均值得到的三个结果分别对应着目标植物细胞外侧细胞重心点的xyz三轴坐标值;再使用步骤(4.2)中细胞内侧数据中四级字段xval、yval和zval的值进行同样的操作步骤,得到目标植物细胞内细胞重心点的xyz三轴坐标值,然后对细胞外侧和内侧的两个细胞重心点的xyz三轴坐标值进行取平均值操作,即可得到目标植物细胞的重心参数x
cg
,y
cg
和z
cg
,并将得到的数据p
cg
存入步骤(3)中数据结构的二级字段cengra;
[0057]
(5.4)通常情况下目标植物细胞为非规则球体,所以需将目标植物细胞划分为若干个四面体,并通过先计算单个四面体的体积再进行累加的方式得到目标植物细胞的体积,其中:
①
所有四面体的共同顶点(注:此处的顶点均是以步骤(5.1)中得到的外侧三角面作为四面体的底面,该底面所对的点为该四面体的顶点)为目标植物细胞的重心点,即数据使用的是步骤(5.3)中计算得到的重心点p
cg
,由于四面体的底面均为步骤(5.1)中得到的三角面,因此四面体的底面积使用的是步骤(5.2)中的外侧表面积。
②
第t个四面体体积计算使用底面积与顶点到底面距离的积再取三分之一,即其中1≤t≤n_thd,n_thd为四面体的数量。然后对目标植物细胞所分割成的所有四面体进行体积值累加,即此处得到累加的结果v
cl
就是目标植物细胞的体积参数,将得到的v
cl
存入步骤(3)中数据结构的二级字段volm_cur;
[0058]
(5.5)目标植物细胞的细胞壁平均厚度计算为:
①
首先使用步骤(4.2)中细胞外侧点所有四级字段xval、yval和zval的值和与步骤(5.3)中重心点p
cg
的xyz三轴坐标值分别进行求点到点之间距离的计算操作,即
再对所有计算得到的值取平均值,即值取平均值,即得到的d
ot
值为目标植物细胞所有外侧点到该细胞重心点的平均距离,
②
其次使用步骤(4.2)中细胞内侧点所有四级字段xval、yval和zval的值和与步骤(5.3)中重心点的xyz三轴坐标值进行同样的计算操作,即步骤(5.3)中重心点的xyz三轴坐标值进行同样的计算操作,即再对所有计算得到的值取平均值,即得到的d
in
值为目标植物细胞所有内侧点到该细胞重心点的平均距离,
③
然后用目标植物细胞所有外侧点到该细胞重心点的平均距离减去目标植物细胞所有内侧点到该细胞重心点的平均距离,即d
cw
=d
ot-d
in
,得到的d
cw
值为目标植物细胞细胞壁的平均厚度,并将得到的数据存入步骤(3)中数据结构的二级字段avethk_cellwall_cur中。
[0059]
实施例1
[0060]
如图1所示,本发明实施例以毛竹细胞为例提供了一种植物细胞三维重构方法,该方法包括如下步骤s1-s5:
[0061]
s1:以毛竹细胞为例,对毛竹组织取大小规格为0.0503cm
×
0.0518cm
×
0.1905cm的样本,用高分辨率ct扫描仪器对毛竹组织进行扫描取样,得到四千张分辨率为1939
×
1995的毛竹组织细胞连续切片图像;
[0062]
s2:根据毛竹细胞切片图像分辨率和毛竹细胞的大小建立三维笛卡尔坐标系,其中毛竹细胞切片图像的分辨率为1939
×
1995像素,切片图像中的毛竹细胞与实际毛竹细胞大小的比例值为l=0.2956,所选毛竹细胞首次在切片图像中完整出现的图像为第77张切片图像即图像序号设置为1,所选毛竹细胞完全消失的前一张为第514张切片图像即图像序号设置为438,即该毛竹细胞三维重构采样对应的x轴坐标值(注:1939
×
0.2956=573.1684)的起止范围为:0.2956μm~573.1684μm,y轴坐标值(注:1995
×
0.2956=589.722)的起止范围为:0.2956μm~589.722μm,z轴坐标值(注:438
×
0.2956=129.4728)的起止范围为:0.2956μm~129.4728μm;
[0063]
s3:如图3所示,毛竹细胞采样的点信息存储在d3d_pcells数据结构下,其中包含两个两个一级字段:params_ovrl和array_pcells[],其中:
①
一级字段params_ovrl包含了以下二级字段:name_ptype、num_total、name_ctypes[]、num_ctypes[]、area_tar_ctypes[]和volm_tar_ctypes[],
②
array_pcells[]包含了以下二级字段:nctpid、area_cur、volm_cur、avethk_cellwall_cur、cengra、outer和inner,
③
outer和inner两个二级字段均包含了以下三个三级字段:points[]、edges[]和triaglfcs[],
④
三级字段points[]均包含了五个四级字段ptid、cirid、xval、yval和zval,
④
三级字段edges[]均包含了三个四级字段edgid、ptid1和ptid2,
⑤
三级字段triaglfcs[]均包含了四个四级字段trifclid、edgid1、edgid2和edgid3;
[0064]
s4:本实施例中所进行采样的毛竹细胞为维管束细胞,首先需要将此信息存入二级字段nctpid中(细胞类型名称:维管束细胞,序号:1),以便用于后续毛竹细胞生长模拟中形态变化的可视化,再对毛竹细胞单张切片图像进行点采样,其中所选的单个毛竹细胞的细胞壁在切片图像中与平行于y轴的等间距线产生的内(外)侧点的点数多于平行于x轴的
等间距线,因此采用平行于y轴的等间距线进行点采样,并且在s2所选毛竹细胞的438张切片图像中,其中单个毛竹细胞最小的形态面积在以x轴方向为准跨越了140个像素左右,而目标植物细胞切片图像中形态面积的范围内需包含至少六条等间距线,因此本实例选择间距为20像素且平行于y轴的等间距线对该毛竹细胞切片图像进行点采样;
[0065]
如图5(a)所示,对毛竹细胞的细胞壁内侧点进行采样,以左上角第一个内侧交点为序号1的点对所有的内侧点进行顺时针排序。将对内侧1号点采集并记录对应的点序号存入二级字段inner中三级字段points[]的四级字段的ptid中、当前采样点切片图像对应的图像序号则存入四级字段中的cirid中、x轴坐标值存入四级字段的xval中,y轴坐标值存入四级字段的yval中和z轴坐标值存入四级字段中的zval中,以此类推对毛竹细胞切片图像的所有内侧点进行上述采样记录直至图像序号为438的切片图像。
[0066]
如图5(a)所示,对毛竹细胞的细胞壁外侧点进行采样,以左上角第一个外侧点为序号1的点对所有的外侧点进行顺时针排序。将对外侧1号点采集并记录对应的点序号存入二级字段outer中三级字段points[]的四级字段的ptid中、当前采样点切片图像对应的图像序号则存入四级字段中的cirid中、x轴坐标值存入四级字段的xval中,y轴坐标值存入四级字段的yval中和z轴坐标值存入四级字段中的zval中,以此类推对毛竹细胞切片图像的所有外侧点进行上述采样记录直至图像序号为438的切片图像停止;
[0067]
s5:将s4中采集完成的毛竹细胞外侧点,即二级字段outer中三级字段的points[]成员信息进行三角剖分算法之后得到三角面信息存入对应的三级字段的triaglfcs[]成员中,其中三角面序号录入四级字段的trifcid,三条三角边的序号对应存入四级字段的edgid1、edgid2和edgid3中,其中三角边的序号在三角剖分算法中得到,各三角边两个端点对应的点序号与s4中的ptid相同,将它们再次分别存入三级字段edges[]中四级字段的ptid1和ptid2中,对于毛竹细胞内侧点使用同样的步骤并将各参数存入对应的内侧点数据结构中;
[0068]
计算毛竹细胞表面积即毛竹细胞的外侧表面积,对于毛竹细胞外侧表面积先计算外侧单个三角面的面积,首先根据二级字段outer中三级字段triaglfcs[]的四级字段edgid1、edgid2、edgid3所对应的ptid1和ptid2中的点序号值,可以与s4中采集完成的细胞外侧点中三级字段points[]的四级字段ptid里面的序号值对应上,然后使用对应序号点的四级字段xval、yval和zval值计算出三条边的长度和再使用三角形表面积海伦公式得出单个三角面的面积sr,再将目标毛竹细胞的二级字段outer中三级字段triaglfcs[]的四级字段trifcid的所有三角面循环进行面积计算的结构进行累加即可得到毛竹细胞外侧的表面积,并将得到的数据存入二级字段area_cur中。
[0069]
毛竹细胞的重心计算分为三步,先使用二级字段outer中三级字段points[]的四级字段xval、yval和zval进行求和再求平均值,然后使用二级字段inner中points[]的四级字段xval、yval和zval进行求和再求平均值,最后将前两者的平均值相加再求平均得到毛竹细胞的重心点数据p
cg
并存入二级字段cengra中。
[0070]
毛竹细胞的体积参数计算如下:目标毛竹细胞形态为非规则球体,因此这里将毛竹细胞整体进行分割为若干个四面体分别进行体积计算再累加,其中此处所有四面体的共
同顶点(注:此处的顶点均为由三角剖分算法得到的外侧三角面作为四面体的底面,该底面所对的点为该四面体的顶点,同时为目标植物细胞的重心点)数据均使用二级字段cengra中的数据信息,单个四面体的底面均使用三角形表面积海伦公式计算得到对应底面积面积参数记为计算毛竹细胞重心点到各个四面体底面的距离参数其中单个四面体体积的计算使用公式然后对该毛竹细胞所分割得到的所有四面体进行累加,得到该毛竹细胞的体积参数v
cl
并将其存入二级字段volm_cur中。
[0071]
毛竹细胞的细胞壁的平均厚度计算为:
①
计算毛竹细胞所有外侧点到该细胞重心点的平均距离d
ot
。首先,遍历读取该毛竹细胞数据结构一级字段array_pcells[]中的二级字段outer下的三级字段points[]中所有外侧点的xval、yval和zval值,并将第i个外侧点的坐标值存入其次,读取data_pcell中的cengra值并存入p
cg
,计算i个外侧点到细胞重心点的距离:然后,执行运算:
②
计算毛竹细胞所有内侧点到该细胞重心点的平均距离d
in
。首先,遍历读取一级字段array_pcells[]中的二级字段inner下的三级字段points[]中所有内侧点的值,并将第j个内侧点的坐标值存入其次,计算j个内侧点到细胞重心点的距离:然后,执行运算:
③
通过d
cw
=d
ot-d
in
计算毛竹细胞细胞壁的平均厚度,并将所计算的d
cw
存入二级字段avethk_cellwall_cur中。
[0072]
参考文献
[0073]
[1]k.toyooka,b.h.kang,“reconstructing plant cells in 3d by serial section electron tomography,”plant cell morphogenesis.humana press,totowa,nj,2014:159-170.
[0074]
[2]薛秀花,张晓嫣,郑东,等,“激光共聚焦显微成像技术在植物细胞微丝骨架三维动态观察中的应用,”现代仪器,2010(5):50-54.
[0075]
本领域技术人员可以理解附图只是一个优选实施例的示意图,上述本发明实施例序号仅仅为了描述,不代表实施例的优劣。
[0076]
以上所述仅为本发明的较佳实施例,并不用以限制本发明,凡在本发明的精神和原则之内,所作的任何修改、等同替换、改进等,均应包含在本发明的保护范围之内。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1